Method Article
Cristallographie aux rayons X pour étudier la transition oligomérique de l’État de la Thermotoga maritima M42 Aminopeptidase TmPep1050
Dans cet article
Résumé
Ce protocole a été développé pour étudier la transition dimer-dodecamer de TmPep1050, un Aminopeptidase M42, au niveau structurel. Il s’agit d’un pipeline simple qui part de la purification des protéines au traitement des données aux rayons X. Crystallogenesis, indexation d’ensemble de données, et remplacement moléculaire ont été soulignés par un cas d’étude, TmPep1050H60A H307A variante.
Résumé
Les aminopeptidases M42 forment des complexes fonctionnellement actifs faits de 12 sous-units. Leur processus d’assemblage semble être régulé par leurs cofacteurs d’ions métalliques déclenchant une transition dimer-dodecamer. Lors de la liaison d’ion métallique, plusieurs modifications structurelles se produisent dans le site actif et à l’interface d’interaction, façonnant les dimers pour favoriser l’auto-assemblage. Pour observer de telles modifications, les oligomers stables doivent être isolés avant l’étude structurale. Rapporté ici est une méthode qui permet la purification de dodecamers stables et dimers de TmPep1050, un Aminopeptidase M42 de T. maritima, et leur détermination de la structure par cristallographie aux rayons X. Dimers ont été préparés à partir de dodecamers en enlevant les ions métalliques avec un agent chélateur. Sans leur cofacteur, les dodecamers sont devenus moins stables et ont été entièrement dissociés au chauffage. Les structures oligomériques ont été résolues par l’approche moléculaire simple de remplacement. Pour illustrer la méthodologie, la structure d’une variante tmpep1050, totalement altérée dans la reliure d’ion métallique, est présentée ne montrant aucune autre dégradation des dimers aux monomères.
Introduction
L’oligomérisation est un processus prédominant qui dicte les fonctions biologiques de nombreuses protéines. Dans Escherichia coli, on estime que seulement 35% des protéines sont monomères1. Certaines protéines, appelées morphéines, peuvent même adopter plusieurs états oligomériques avec des sous-unités ayant une structure distincte dans chaque état oligomérique2. La transition entre leurs états oligomériques est souvent un moyen de réguler l’activité protéique car chaque état oligomérique peut avoir une activité ou une fonction spécifique différente. Plusieurs exemples de morphétines ont été bien documentés dans la littérature, notamment la synthase porphobilinogen3, HPr kinase/phosphatase4, Lon protease5, lactate dehydrogenase6, glyceraldehyde-3-phosphate dehydrogenase7, pyruvate kinase8, synthase citrate9, et ribonuclease A10. Récemment, nous avons décrit l’aminopeptidase M42 TmPep1050, un autre exemple d’enzyme avec un comportement morphéine, dont l’activité dépend de ses états oligomériques11. La transition entre ses états oligomériques est médiée par ses cofacteurs métalliques qui induisent plusieurs modifications structurelles des sous-unités.
La famille des aminopeptidase M42 appartient au clan MH12,13, et est largement répartie entre bactéries et archée14. Les aminopeptidases M42 sont de véritables enzymes dinucléaires dégradant les peptides jusqu’à 35 résidus d’acides aminés delongueur 15. Ils adoptent une structure particulière en forme de tétraèdre faite de 12 sous-units avec leurs sites actifs orientés vers une cavité intérieure. Un tel arrangement est souvent décrit comme une nano-compartimentation de l’activité pour éviter la protéolyse incontrôlée. La fonction physiologique des aminopeptidases M42 peut être associée aux peptides hydrolysants protéasomes résultant de la dégradation desprotéines 16,17. Pyrococcus horikoshii possède quatre aminopeptidases M42, chacune présentant des spécificités distinctes mais complémentaires18,19,20,21. Singulièrement, des hétérocomplexes faits de deux types différents de sous-unités ont été décrits dans P. horikoshii,suggérant l’existence des complexes peptidasome22,23.
Plusieurs structures de M42 aminopeptidases ont été décrites dans lalittérature 11,16,18,19,20,24,25,26. Le sous-unité est composé de deux domaines distincts, d’un domaine catalytique et d’un domaine de dimérisation. Le domaine catalytique adopte un pli α/β commun conservé dans tout le clan MH, le domaine catalytique archétypal étant l’aminopeptidase Ap1 de Vibrio proteolyticus27. Le domaine de la dimérisation adopte un pli16 en forme de PDZ et peut avoir, en plus de son rôle dans l’oligomérisation, un rôle dans le contrôle de l’accès au substrat et la liaison dans la cavitéinterne 11. Comme le bloc de base est un dimère, le dodécaamer est souvent décrit comme l’association de six dimers, chaque dimer étant placé à chaque bord du tétraèdre16. L’oligomérisation des aminopeptidases M42 repose sur la disponibilité de ses cofacteurs métalliques. Les ions métalliques Divalent, souvent Zn2+ et Co2+,sont catalytiques impliqués dans la liaison peptidique et l’hydrolyse. On les trouve dans deux sites de liaison distincts, à savoir les sites M1 et M2. Les deux ions métalliques conduisent et accordent finement l’oligomérisation comme démontré pour PhTET2, PhTET3, PfTET3, et TmPep105011,24,28,29. Lorsque les cofacteurs métalliques sont épuisés, le dodecamer se démonte en dimers, comme dans PhTET2, PhTET3, et TmPep105011,16,28, ou même des monomères, comme dans PhTET2 et PfTET324,29.
Présenté ici est un protocole utilisé pour étudier les structures des oligomers TmPep1050. Ce protocole est un ensemble de méthodes communes comprenant la purification de protéine, le criblage protéolytique d’activité, la cristallisation, la diffraction de rayon X, et le remplacement moléculaire. Les subtilités inhérentes au traitement des métalloenzymes, de l’oligomérisation des protéines, de la cristallisation des protéines et du remplacement moléculaire sont soulignées. Un cas d’étude est également présenté pour montrer si tmPep1050 dodecamers peut se dissocier davantage en monomères ou non. Pour répondre à cette question, une variante tmpep1050, TmPep1050H60A H307A, a été étudiée dont les sites de liaison métallique sont altérés par la mutation de His-60 (site M2) et His-307 (site M1) en résidus d’Ala. Ce protocole peut être hébergé pour étudier d’autres aminopeptidases M42 ou tout métalloenzymes avec un comportement morpheein-like.
Protocole
1. Production et purification de tmpep1050 recombinant
NOTE: Ci-après sont décrits la procédure de clonage et la purification de type sauvage TmPep1050 adapté d’une étude précédente11. Alternativement, le clonage peut être fait à l’aide d’un gène synthétique. Pour générer des variantes TmPep1050, la mutagenèse dirigée par le site peut être effectuée à la suite, par exemple, des réactions d’amorce unique dans la méthode du protocole parallèle (SPRINP)30. Le protocole de purification peut être utilisé pour les variantes tmpep1050. L’utilisation de son étiquette doit être évitée car elle interfère avec la liaison irion métallique.
- Conception de vecteur d’expression
- Acquérir l’ADN génomique de Thermotoga maritima MSB8 (ATCC 43589) ou TmCD00089984 (Joint Center for Structural Genomics).
- Amplifier TM_1050 de lecture ouverte (ORF) à l’aide d’ADN génomique ou de plasmide modèle, une polymése d’ADN haute fidélité, et les amorces suivantes: ocej419 (5'- TTTAACTTTAAGAAGGAGATATACATCCATGAAGGAACTGATCAGAAAGCTG) et ocej420 (5'- ATCCGCCAAAACAGCCAAGCTGGAGACCGTTTACGCCCCCAGATACCTGATGAG). Exécutez le criblage de réaction en chaîne de polymése (PCR) selon le schéma suivant : 5 min à 95 °C, 30 cycles de 3 étapes (30 s à 95 °C, 30 s à 55 °C, 90 s à 72 °C) et 10 min à 72 °C comme dernière étape.
- Cloner le fragment pcr en un vecteur d’expression approprié (Tableau des matériaux) par recombinaison homologue ( Figure1) dans E. coli selon le protocole SLiCE31. À 50 ng de vecteur linéarisé, ajouter le fragment pcr dans un rapport molaire de 10:1 de fragment par vecteur, 1 μL d’extrait de souche PPY, 50 mM Tris-HCl pH 7,5, 100 mM MgCl2, 10 mM ATP et 10 mM dithiothreitol (DTT) pour un volume de réaction de 10 μL. Incuber pendant 1 h à 37 °C.
- Transformez la souche bleue E. coli XL1 chimiquement compétente (ou toute souche recA- appropriée) avec 1 μL de réaction de recombinaison. Plaquez les cellules sur le milieu LB contenant 100 μg/mL d’ampicilline. Incuber les assiettes toute la nuit à 37 °C.
- Cueillez les colonies sur des assiettes LB fraîches avec de l’ampicilline de 100 μg/mL. Incuber les assiettes à 37 °C pendant au moins 8 h.
- Écran pour les candidats positifs par pcr colonie en utilisant la paire d’amorce appropriée (5'- ATGCCATAGCATTTTTATCC et 5'- ATTTAATCTGTATCAGGC si vous utilisez le vecteur recommandé énumérés dans le tableau des matériaux). À l’aide d’une extrémité microtipée, gratter une colonie choisie et transférer les cellules à 20 μL de mélange de réaction contenant 0,5 μM de chaque amorce et 10 μL d’un mélange commercial de polymélase d’ADN Taq.
- Exécutez le criblage PCR selon le schéma suivant : 5 min à 95 °C comme étape de dénaturation, 30 cycles de 3 étapes (30 s à 95 °C, 30 s à 55 °C, 90 s à 72 °C) et 10 min à 72 °C comme dernière étape.
REMARQUE : Les réactions PCR peuvent être stockées pendant la nuit dans la machine PCR à 12 °C. - Chargez 10 μL de chaque réaction PCR sur un gel d’agarose de 0,8 % préparé dans le tampon Tris-acétate-EDTA (TAE). Faire fonctionner l’électrophorèse pendant 25 min à 100 V.
REMARQUE : Un amplicon de 1,1 kbp est attendu. - Extraire les plasmides des candidats à l’aide d’un kit commercial(Tableau des matériaux)et les séquencer à l’aide de la même paire d’amorce utilisée à l’étape 1.1.6.
- Culture cellulaire
REMARQUE : Lorsqu’un candidat approprié a été identifié par séquençage, le clone peut être directement utilisé comme expression si l’on utilise le vecteur recommandé (Tableau des matériaux). Dans ce cas, l’expression est contrôlée par le promoteur arabinose-inducible PBAD 32.- Inoculer 10 mL de milieu LB contenant 100 μg/mL d’ampicilline avec le candidat et incuber la préculture pendant la nuit à 37 °C sous secousse orbitale. Ajouter 5 mL de préculture à 1 L de LB moyen avec 100 μg/mL d’ampicilline. Attention à respecter un rapport air/liquide de 3.
- Laissez les cellules se développer à 37 °C sous la secousse orbitale. Surveiller la densité optique à 660 nm (OD660).
- Lorsque l’OD660 a atteint 0,5−0,6, refroidissez rapidement la culture pendant 5 min sur la glace et transférez-la dans un incubateur fixé à 18 °C.
- Ajouter 0,2 g/L d’arabinose pour induire l’expression des gènes et incuber pendant 12−18 h à 18 °C.
- Récoltez les cellules en centrifugant la culture à 6 000 x g pendant 30 min à 4 °C. Jetez le supernatant et lavez les cellules avec 100 mL de 0,9% (w/v) NaCl.
- Centrifugeuse à nouveau à 6.000 x g pendant 15 min à 4 °C et jeter le supernatant.
REMARQUE : Les granulés cellulaires peuvent être utilisés directement pour l’extraction des protéines ou stockés à -80 °C.
- Purification des protéines
- Resuspendez les granulés cellulaires dans 40 mL de MOPS de 50 mM, 1 mM CoCl2, pH 7,2. Ajouter 1 μL de 25 U/μL d’ADN/ARN endonuclease et un comprimé de cocktail inhibiteur de la protéase qui ne contient pas d’EDTA. Sonicate la suspension en mode impulsion sous refroidissement pendant 30 min.
- Centrifugeuse l’extrait brut à 20.000 x g pendant 30 min à 4 °C. Recueillir le surnatant et le chauffer dans un bain d’eau à 70 °C pendant 10 min.
- Centrifugeuse l’extrait de cellules dénaturées à 20.000 x g pendant 30 min à 4 °C et recueillir le supernatant pour la purification.
- Utilisez de la résine d’échanged’anion appropriée( Table of Materials ) emballée dans une colonne d’environ 15 mL de volume. Consultez les recommandations du fabricant concernant le taux de débit de travail et la limite de pression des colonnes. Equilibrer la résine avec 50 mM MOPS, 1 mM CoCl2, pH 7,2.
- Chargez le supernatant recueilli à partir de l’étape 1.3.3 dans la colonne. Surveiller l’absorption de l’élitate à 280 nm. Quand il a atteint la ligne de base, procéder à l’élitution.
- Appliquer un gradient de 0 à 0,5 M NaCl en 50 mM MOPS, 1 mM CoCl2, pH 7,2 pour 5 volumes de colonnes (CV). Attendez que la conductivité soit stabilisée et que l’absorption ait atteint la ligne de base.
- Appliquer un gradient final de 0,5 à 1 M NaCl en 50 mM MOPS, 1 mM CoCl2, pH 7,2 pour 1 CV.
- Analyser certaines fractions (voir la figure 2A pour obtenir des indications) par électrophoresis de gel polyacrylamide de sulfate de dodecyl de sodium (SDS-PAGE).
NOTE: TmPep1050 apparaît comme un groupe de 36 kDa après coomassie coloration. Par ailleurs, la présence de TmPep1050 peut être confirmée par un test d’activité (voir la section 2.1). À cette étape, les fractions peuvent être stockées à 4 °C pendant la nuit. - Mettre en commun les fractions contenant TmPep1050 et ajouter de la poudre finement moulue de (NH4)2SO4 pour obtenir une concentration de 1,5 M (NH4)2SO4. Mélanger délicatement en inversant le tube à l’envers jusqu’à dissolution complète.
- Utilisez de la résine d’interaction hydrophobe(Tableau desmatériaux) emballée dans une colonne d’environ 30 mL de volume. Consultez les recommandations du fabricant concernant le taux de débit de travail et la limite de pression des colonnes. Equilibrer la résine avec 50 mM MOPS, 1,5 M (NH4)2SO4, 1 mM CoCl2, pH 7,2.
- Chargez l’échantillon sur la colonne et surveillez l’absorption de l’élitisme à 280 nm. Lorsque l’absorption a atteint la ligne de base, elute protéines liées en appliquant un gradient de 1,5 M à 0 M (NH4)2SO4 en 50 mM MOPS, 1 mM CoCl2, pH 7,2 pour 5 CV.
- Analyser certaines fractions (voir la figure 2B pour obtenir des conseils) par SDS-PAGE.
NOTE: TmPep1050 apparaît comme un groupe de 36 kDa après coomassie coloration. Par ailleurs, la présence de TmPep1050 peut être confirmée par un test d’activité (voir la section 2.1). À cette étape, les fractions peuvent être stockées à 4 °C pendant la nuit. - Mettre en commun les fractions contenant TmPep1050 et se concentrer à 2 mL à l’aide d’unités d’ultrafiltration avec 30 kDa de coupure (Tableau des matériaux). Passez à l’article 1.4 pour déterminer le poids moléculaire.
- Chromatographie d’exclusion de taille
- Utilisez de la résine d’exclusionde taille( Tableau des matériaux ) emballé dans une colonne d’environ 120 mL de volume. Consultez les recommandations du fabricant concernant le taux de débit de travail et la limite de pression des colonnes. Equilibrer la résine avec 50 mM MOPS, 0,5 M (NH4)2SO4, 1 mM CoCl2, pH 7,2.
- Chargez l’échantillon sur la colonne et surveillez l’absorption de l’élitisme à 280 nm. Fractionner du volume mort de la colonne (~0,33 CV) jusqu’à la fin de l’élitution (1 CV).
- Mesurer le volume d’élitution pour chaque pic observé.
REMARQUE: Pour les conseils, dodecameric TmPep1050 elutes à ~ 82 mL (Figure 3A) dans les conditions expérimentales actuelles, tandis que dimeric TmPep1050, tels que le TmPep1050H60A H307A variante, elutes à ~ 95 mL (Figure 3B). Certains TmPep1050 peuvent adopter les deux formes oligomériques, comme TmPep1050H60A (Figure 3C). - Analyser les fractions correspondant aux maxima et aux queues des pics observés à l’aide de SDS-PAGE.
NOTE: TmPep1050 apparaît comme un groupe de 36 kDa après coomassie coloration. - Mettre en commun les fractions de chaque pic et se concentrer à l’aide d’unités d’ultrafiltration avec 30 kDa de coupure(tableau des matériaux)pour obtenir une concentration d’environ 300 μM.
- Mesurez l’absorption à 280 nm sur un spectrophotomètre nano-volume et calculez la concentration à l’aide du coefficient d’extinction moléculaire de 18 910 M-1 cm-1.
- Conserver la protéine purifiée à -18 °C.
- Pour déterminer le poids moléculaire, calibrer la colonne de chromatographie d’exclusion de taille (SEC) à l’aide de normes de poids moléculaire (Tableau des matériaux). Analyser les normes à l’aide de 50 mM MOPS, 0,5 M (NH4)2SO4, 1 mM CoCl2 pH 7.2 comme tampon en cours d’exécution.
2. Analyse d’activité et préparation d’apo-enzyme
REMARQUE : À l’origine, l’apo-enzyme a été préparée en diluant 1 volume de TmPep1050 en 10 volumes de pH 7.0 d’acide malique de 2,1 M et en se concentrant de nouveau à 1 volume avant la dialyse11. Ci-dessous est présenté une procédure alternative en utilisant 1,10-phénanthroline, un chélateur d’ion métallique. Cette procédure réduit la perte de protéines et donne les mêmes résultats que la méthode précédemment publiée.
- Analyse d’activité
- Préparer une solution de stock de 100 mM L-Leucine-p-nitroanilide (Tableau des matériaux) en méthanol.
- Ajouter 25 μL de 100 mM L-Leucine-p-nitroanilide en 965 μL de 50 mM MOPS, 250 μM CoCl2, pH 7,2, 10% méthanol. Préincuber le mélange de réaction à 75 °C dans un bain sec.
- Diluer l’enzyme en 50 mM mops pH 7,2 à une concentration de 1 μM. Ajouter 10 μL au mélange de réaction, vortex, et incuber à 75 °C soit jusqu’à ce qu’il soit devenu jaunâtre ou pendant 1 h.
- Arrêtez la réaction en ajoutant 1 mL d’acide acétique de 20%. Vortex bien et laissez-le refroidir à température ambiante.
- Transférer le mélange de réaction dans une cellule spectrophotomètre. Lire l’absorption à 410 nm contre un contrôle négatif (mélange de réaction incubée sans enzyme).
- Préparation en apo-enzymes
- Préparer une solution de stock de 1 M 1,10-phénanthroline dans l’éthanol. Ajouter 10 μL de solution de stock de 1,10 phénanthroline à 890 μL de 50 mM MOPS, 0,5 M (NH4)2SO4, pH 7,2. Ajouter 100 μL de TmPep1050 purifié (concentration de 300 μM−1 mM).
- Vérifiez la perte d’activité à l’aide de l’analyse d’activité décrite à la section 2.1 sans ajouter cocl2 dans le mélange de réaction.
- Transférer l’échantillon dans un tube de dialyse. Dialyze contre 200 mL de 50 mM MOPS, 0,5 M (NH4)2SO4, pH 7,2 à 4 °C. Échangez trois fois le dialyse avec un tampon frais pendant la dialyse de 48 h.
- Prélouillez l’échantillon du tube de dialyse et concentrez-vous jusqu’à 100 μL à l’aide d’unités d’ultrafiltration avec coupure de 30 kDa(tableau des matériaux). Vérifiez la concentration en lisant l’absorption à 280 nm à l’aide d’un spectrophotomètre nano-volume.
- Préparation dimer
- Diluer l’apo-enzyme à une concentration de 1 μM dans 50 mM MOPS, 0,5 M (NH4)2SO4, pH 7,2. Incuber pendant 2 h à 75 °C dans un bain sec, puis laisser refroidir l’échantillon à température ambiante.
- Concentrez l’échantillon sur une concentration enzymatique d’au moins 50 μM. Vérifiez le poids moléculaire par sec (voir la section 1.4). Le pic d’élitution doit passer de ~82 mL à ~95 mL (dans les conditions expérimentales actuelles).
3. Cristallisation TmPep1050
REMARQUE : La cristallisation des protéines demeure une science empirique car il s’agit d’un phénomène multifactoriel33. Bien que certains paramètres puissent être identifiés et contrôlés (comme la température, le pH, la concentration d’agents de précipitation), d’autres peuvent influencer de façon insaisissable la cristallisation (comme la pureté des protéines et des produits chimiques, la protéolyse, l’historique des échantillons). Aujourd’hui, la cristallisation des protéines est abordée de manière rationnelle et systématique grâce à un tas de conditions de cristallisation commerciale et d’automatisation. L’optimisation d’une condition de cristallisation, cependant, repose principalement sur une approche d’essai et d’erreur. Par la suite sont décrits un plan pour cristalliser les protéines et plusieurs conseils pour optimiser les conditions de cristallisation.
- Dépistage de cristallisation
REMARQUE : À l’aide de kits de cristallisation commerciale, des cristaux de tmpep1050 dodécatérique ont été obtenus dans 2,2 M d’acide dla-malique pH 7,0, 0,1 M Bis-Tris propane pH 7,0 et 0,18 M de citrate de tri-ammonium, 20 % polyéthylène glycol (PEG) 3350. Des cristaux de tmpep1050 dimérique ont été obtenus dans 0,1 M de citrate de sodium pH 5,6, 0,2 M d’acétate d’ammonium, 30% PEG4000. Les cristaux de dodécaamers apparaissent dans la semaine et atteignent leur pleine taille en un mois. Cristaux de dimers apparaissent généralement dans les 24 h et se développent à pleine taille en une semaine.- Acquérir plusieurs kits de cristallisation commerciale (voir Table of Materials par exemple).
- Mettre en place des plaques de cristallisation (Table of Materials) pour la méthode de chute suspendue. Remplissez les puits de 500 μL de chaque solution d’une trousse de criblage de cristallisation.
- Pour chaque puits, mettre en place un support de cristallisation. Sur le support, déposer une goutte de 1 μL de protéines purifiées (généralement ~10 mg/mL).
- Pipet immédiatement 1 μL de solution de cristallisation à partir du puits. Ajoutez-le soigneusement à la goutte de protéines et mélangez doucement en pipetting à l’envers trois fois. La chute doit rester semi-sphérique sans bulles.
- Vissez le support sur le puits correspondant. Répétez l’opération pour l’ensemble du kit.
- Après avoir mis en place les plaques, observez chaque goutte avec une jumelle. Consultez le guide d’interprétation du kit de cristallisation (chute claire, séparation des phases, précipité, aiguilles, etc.).
- Incuber les assiettes à 20 °C. Vérifiez les plaques une fois par jour pendant la première semaine et une fois par semaine par la suite.
- Marquez chaque puits à l’aide de la feuille de pointage et du guide utilisateur fourni avec les kits de cristallisation.
- Optimisation de la cristallisation
REMARQUE : Les conditions initiales de cristallisation du tmpep1050 dodécamérique ont été optimisées à 2,1 M d’acide dla-malique pH 6,75 et 0,18 M de citrate de tri-ammonium pH 7,5, 40% (w/v) PEG3350 tandis que l’état de cristallisation du dimeric TmPep1050 a été déplacé à 0,1 M de citrate de sodium pH 6,0, 10% (w/v) PEG3350. Un cycle d’ensemencement a été nécessaire pour améliorer la cristallinité. Ci-après est décrit comment la cristallisation de TmPep1050H60A H307A variante a été optimisée.- Préparez des solutions de stock de tampon de citrate de sodium de 0,5 M à différents pH (4,5, 5,2 et 6,0) et de 50 % (w/v) solution PEG3350.
- Mettre en place une plaque de cristallisation comme matrice de pH par rapport à l’agent des précipitations (voir la figure 4A).
- Incuber la plaque à 20 °C. Observez chaque puits avec une jumelle une fois par jour pendant une semaine.
- Marquez chacun bien en fonction de la taille et de la forme du cristal. Sélectionnez une condition donnant des cristaux d’au moins 50 μm. Procéder au microsédage.
- Microsédage
REMARQUE : Le microsédage est une méthode puissante pour améliorer la forme, la taille et la cristallinité des cristaux protéiques34. Une approche d’ensemencement plus rapide est l’ensemencement des stries à l’aide d’une moustache pour chat. Voir figure 4 à titre d’exemple comment l’optimisation de la cristallisation et le microsédage ont amélioré la forme et la taille des cristaux pour TmPep1050H60A H307A.- Préparer les graines à l’aide du puits sélectionné à l’étape 3.2.4.
- Augmentez le volume d’une goutte contenant des cristaux à 10 μL en ajoutant la solution de cristallisation du puits. Pipet la goutte et ajouter 90 μL de solution de cristallisation à partir du puits. Vortex complètement et garder les graines sur la glace.
- Préparer plusieurs dilutions des graines : 1x, 10x, 25x et 100x. Vortex bien les graines avant pipetting. Gardez les dilutions sur la glace.
- Pour chaque dilution des graines, configurer une plaque de cristallisation comme matrice de pH par rapport à l’agent de précipitations (voir la figure 4B). Utilisez les solutions de stock préparées à l’étape 3.2.1.
- Lors de la chute, ajouter 0,2 μL de graines pour une goutte de 2 μL. Incuber la plaque à 20 °C. Observez chaque puits avec une jumelle une fois par jour pendant une semaine.
REMARQUE : La distribution et la forme de la taille du cristal doivent être améliorées, voir la figure 4C comme exemple pour TmPep1050H60A H307A. Pour d’autres utilisations, les graines peuvent être stockées à -20 °C.
4. Diffraction aux rayons X
- Cueillette de cristaux
REMARQUE : La préparation de l’échantillon dépend de l’installation source de rayons X (installation à domicile vs synchrotron). Utilisez les dispositifs de stockage (flacons et panier porte-fiole) en conséquence. L’ajout de cryoprotecteur (comme le glycérol) peut être nécessaire en fonction de la concentration d’agents de sel et de précipitations. Pour les cristaux TmPep1050, un cryoprotecteur n’est pas nécessaire car peg ou concentration tampon est suffisamment élevée pour éviter les cristaux d’eau.- Préparer un bain rempli d’azote liquide, plonger les flacons ou le panier utilisés pour la manipulation de l’échantillon.
- Configurer des boucles de cueillette d’échantillons de différentes tailles : 100, 150 et 200 μm(Tableau des matériaux). Choisissez les tailles en fonction de la taille du cristal.
- À l’aide d’une jumelle, vérifiez la goutte contenant des cristaux et repèrez les cristaux isolés (les plus faciles à cueillir). À l’aide d’une boucle, choisissez doucement un cristal par le bas. Plongez immédiatement la boucle dans de l’azote liquide et placez la boucle dans un flacon approprié.
- Collecte de données
REMARQUE : La collecte de données peut varier considérablement selon la source des rayons X (installation à domicile par rapport au synchrotron) et la sensibilité du détecteur. La stratégie de collecte peut également différer considérablement d’un échantillon à l’autre, selon la résolution, l’intensité ponctuelle, le groupe spatial, etc. Le sujet a été largement examiné par Dauter35.- Montez la boucle portant le cristal sur la tête du goniomètre du diffractomètre.
- Réglez la tête du goniomètre le long des axes XYZ pour aligner le cristal avec la trajectoire du faisceau de rayons X.
- Réglez la longueur d’onde à 0,98 Å et déplacez le détecteur pour obtenir 2 Å de résolution.
- Commencez une courte collecte de données en acquérant des images dans au moins deux orientations cristallines différentes. Prenez 10 images (1 image par 0,1°) à 0° et 90°.
- Vérifiez les images recueillies à l’aide d’un logiciel approprié (p. ex., ADXV, XDS-Viewer ou Albula Viewer). Déterminez l’intensité de la tache et la résolution la plus élevée où les taches sont vues. Vérifiez également la monocrystallinité et la séparation ponctuelle.
- Éventuellement, répétez les étapes 4.2.3−4.2.5 en changeant la position du détecteur pour une résolution plus élevée ou inférieure et le temps d’exposition conformément à l’observation.
- Démarrez la collecte de données autour de 360° avec 1 image prise pour 0,1°. N’oubliez pas de définir la position du détecteur et le temps d’exposition de façon optimale.
5. Indexation, remplacement moléculaire et construction de modèles
- Indexation
REMARQUE : L’indexation est une méthode pour mesurer l’intensité des taches de diffraction, donnant aux amplitudes des facteurs de structure36. Quatre logiciels sont couramment utilisés pour le traitement des images collectées: Mosflm37, HKL200038, DIALS39, et XDS40. Ce dernier a été utilisé pour indexer les ensembles de données obtenus à partir de la diffraction cristalline TmPep1050.- Installez le paquet XDS et XDSME41. Si vous traitez des fichiers HDF5, installez le plugin XDS Neggia (disponible sur le site Dectris). Pour plus d’informations, visitez la page Web du wiki XDS https://strucbio.biologie.uni-konstanz.de/xdswiki/index.php/Main_Page et la page Web de XDSME https://github.com/legrandp/xdsme.
- Avant de traiter les données, créez un dossier à partir de l’endroit où XDS sera exécuté. Localiser le chemin vers les images.
- Pour exécuter XDSME, tapez xdsme /path_to_images/image.extension dans une fenêtre terminale.
- Une fois que XDS a terminé le travail, vérifiez le CORRECT. Fichier LP. Notez la probabilité de la détermination du groupe spatial, de l’exhaustivité des données, de la résolution la plus élevée, de la mosaïque cristalline et de la qualité des données. Vérifiez également XDS_pointless.log pour obtenir la probabilité de groupes spatiaux.
REMARQUE : Voir la figure 5 comme exemple de sortie. - Réexécuter XDSME avec différentes solutions de groupe spatial proposées par XDS dans un dossier séparé pour éviter de dépasser le processus précédent. Type xdsme -s space_group_name -c « unit_cell_parameters » /path_to_images/image.extension (par exemple, xdsme -s P21 -c « 43.295 137.812 61.118 90.000 110.716 90.000).
- Vérifiez le CORRECT. LP fichiers et choisir la meilleure solution basée sur les statistiques de données.
- Exécutez XSCALE en tapant xscale.py XDS_ASCII. HKL. Exécutez XDSCONV en tapant xdsconv.py XSCALE. HKL ccp4.
REMARQUE : Dans certains cas, XDSME ne parvient pas à identifier le groupe spatial ou ne parvient pas à réduire correctement la plage de résolution ou génère des statistiques de données bizarres. Si un tel problème est rencontré, il vaut la peine d’exécuter XDS nativement. Plusieurs paramètres doivent être introduits dans le XDS. Fichier d’initiation INP (voir page wiki XDS). Lors de l’utilisation de XDS, la probabilité de groupes spatiaux possibles peut être vérifiée en utilisant Inutile, une partie du paquet CCP442. Pour réduire la résolution de l’ensemble de données, Rmeas < 60% et I/σ ~2 sont généralement acceptés pour déterminer la résolution la plusélevée 43. Le remplacement moléculaire et le raffinement du modèle peuvent toutefois être améliorés en étendant la résolution à I/σ ~0.5−1.5 et CC1/2 jusqu’à 0.2−0.444.
- Remplacement moléculaire
REMARQUE : Les données expérimentales donnent accès à l’amplitude des facteurs structurels mais, sans connaître la phase, elles sont inutiles. La phase peut être déterminée expérimentalement par différentes méthodes s’appuyant sur un signal anormal (à partir d’un atome lourd, par exemple)45. Le remplacement moléculaire est une autre méthode pour déterminer la phase sans un atome de diffusion anormal46,47. Cette méthode utilise les coordonnées d’une molécule apparenté pour trouver et améliorer la phase itérativement. Nous utilisons phaser48 dans Phenix GUI49 pour le remplacement moléculaire.- Préparez le modèle de départ pour le remplacement moléculaire à l’aide des coordonnées 4P6Y. À partir du fichier pdb, extraire le monomère A et tronquer ses aminoacées en alanine à l’aide de l’éditeur de fichiers PDB dans Phenix (sous l’onglet Outils modèle).
- Exécutez Xtriage dans Phenix (sous l’onglet Analyse de données) avec le fichier de réflexion généré par XDSCONV (5.1.9) et la séquence sous forme d’entrées.
- Vérifiez le fichier journal de Xtriage. Notez l’exhaustivité, le nombre de sous-unités dans l’unité asymétrique, l’anisotropie, la présence d’anneaux de glace et l’apparition de jumelage.
- Exécutez Phaser-MR dans Phenix (sous l’onglet remplacement moléculaire) pour le remplacement moléculaire à l’aide du fichier de réflexion, de la séquence et du modèle 4P6Y de départ tronqué en poly alanine (étape 5.2.1).
- Une fois terminé, vérifiez si un modèle a été trouvé et le score du remplacement moléculaire. Un facteur de traduction Z-score (TFZ) d’au moins 8 indique que la solution est définitivement correcte.
- Construction de modèles
REMARQUE : Après avoir déterminé la phase par remplacement moléculaire, le modèle doit être construit et affiné. Ce protocole utilise Phenix GUI49 pour la construction automatique et les raffinements itératifs, et Coot50 pour la construction manuelle de la structure et le raffinement.- Après remplacement moléculaire à l’aide de phase-MR dans Phenix, sélectionnez Run Autobuild. Tous les fichiers requis seront automatiquement ajoutés. Il suffit d’appuyer sur Exécuter pour démarrer la construction automobile.
- Une fois terminé, vérifiez le modèle à Coot. Construire et affiner manuellement le modèle selon la carte de densité électronique de Coot.
- Affiner le modèle manuellement dans Phenix (dans l’onglet Raffinement) en utilisant le modèle, la séquence et les données de diffraction comme entrées. Référez-vous à Phenix aider à choisir la bonne stratégie.
- Après raffinement, vérifiez les résultats : le travail Rlibre et R doit diminuer, les indicateurs Molprobity51 doivent être respectés, et les valeurs aberrantes avec une faible corrélation entre l’espace réel doivent être limitées.
- Répétez les étapes 5.3.2−5.3.4 jusqu’à ce que le meilleur modèle raffiné soit généré.
- Exécutez Molprobity sur le serveur : http://molprobity.biochem.duke.edu/. Vérifiez toutes les valeurs aberrantes identifiées par Molprobity.
- Répétez éventuellement les étapes 5.3.2−5.3.6 jusqu’à ce que le meilleur modèle raffiné soit obtenu.
Résultats
Pour étudier une dissociation possible du dodécaamer en monomères dans TmPep1050, les codons His-60 et His-307 ont été remplacés par de l’alanine codon à l’aide d’un gène synthétique. Ce gène a ensuite été cloné dans le vecteur pBAD pour l’expression et la purification de la variante tmpep1050 correspondante par la suite nommée TmPep1050H60A H307A. La chromatographie d’exclusion de taille (figure 3B) a prouvé que la protéine purifiée a eu un poids moléculaire apparent de 56 kDa (poids moléculaire du monomère étant 36.0 kDa). Un poids moléculaire apparent semblable, 52 kDa, a été rapporté pour TmPep1050dimère 11. Par conséquent, l’état oligomérique de TmPep1050H60A H307A pourrait être déduit comme dimeric. En ce qui concerne son activité spécifique, TmPep1050H60A H307A était complètement inactif sur L-Leu-p NA comme substrat, même en présence d’ions cobalt. Ce résultat suggère fortement que les variantes ne peuvent lier aucun ions métalliques.
L’état de cristallisation de TmPep1050H60A H307A a été optimisé par une concentration variable de pH par rapport à peg (figure 4) autour de l’état du dimère (c.-à-d. 0,1 M de citrate de sodium pH 6,0 10 % PEG3350). Les meilleurs cristaux de TmPep1050H60A H307A ont été obtenus en citrate de sodium pH 0,1 M 5,2 20% PEG3350 avec un cycle de microsédage pour améliorer la monocrystallinité. Un ensemble complet de données a été recueilli à proxima 2 beamline (SOLEIL synchrotron) à une résolution 2.36 Å (Tableau 1). L’indexation des données a montré que le groupe spatial du cristal TmPep1050H60A H307A est C2221, mais XDS a proposé une autre solution, le groupe spatial mP (voir figure 5). Selon Inutile, la probabilité de groupes spatiaux C2221 et P21 était de 0,711 et 0,149, respectivement. Selon l’analyse de la qualité des données, deux monomères se trouvent dans l’unité asymétrique. L’analyse de Xtriage a révélé que l’ensemble de données est probablement jumelé, mais le jumelage dans le groupe spatial C2221 est peuprobable 52. Le jumelage résulte d’une anomalie de croissance cristalline où plusieurs domaines définis ont certaines de leurs directions de treillis parallèles les uns auxautres 53. Le jumelage peut également résulter d’une symétrie cristalline plus élevée, indiquant une indexation erronée des données. Par conséquent, un jumeau pseudo-merohedral peut exister de sorte qu’un treillis en cristal P21 ressemble à un C2221. L’ensemble de données a ensuite été indexé dans le groupe spatial P21 et testé en remplacement moléculaire. L’analyse de Xtriage de l’ensemble de données indexée dans P21 a indiqué un jumeau pseudo-merohedral suivant une loi jumelle h, -k, -h-l.
En utilisant les coordonnées d’un monomère de dodecameric TmPep1050 (code PDB 4P6Y), une solution de remplacement moléculaire a été trouvée pour l’ensemble de données indexées en P21 seulement, avec un score TFZ de 28,9. Par conséquent, les données de diffraction ont été traitées comme un ensemble de données jumelées pour la construction de modèles. Pour minimiser le biais du remplacement moléculaire, un premier modèle a été construit en utilisant phenix.autobuild54,55. La structure de TmPep1050H60A H307A a été achevée après plusieurs cycles de raffinement automatisé et manuel à Phenix et Coot (tableau 1 et figure 6A). La structure a confirmé l’état oligomérique avec une surface d’interface de 1710 Å2 entre les monomères et un ΔiG de -16,2 kcal mol-1 tel que calculé par PDBe Pisa56. En comparaison, la surface de l’interface et ΔiG de dimeric TmPep10502-mer est de 1673 Å2 et -16,7 kcal mol-1, respectivement.
La structure de TmPep1050H60A H307A est très similaire à la structure de dimère de type sauvage avec RMS de 0,774 Å lors de l’alignement. Fait important, les mêmes modifications structurelles sont observées dans les deux structures : une grande flexibilité des hélices α8 et α10, le site actif désordonné Gln-196-Val-202, et le déplacement de Lys-229-Ala-235 et Lys-247-Ser 254. Ces modifications ont été corrélées précédemment avec l’obstacle de la formation de dodecamer en l’absence de son cofacteuren métal 11. Les deux mutations de His-60 et His-307, cependant, ont eu un léger effet sur les chaînes latérales d’Asp-168 et d’Asp-62. Ils semblaient enfermés dans une conformation différente de la dimère de type sauvage (Figure 6B). L’Asp-168 carboxylate est tourné de 40° en raison de l’absence de His-60 et His-307. Par conséquent, les deux résidus d’histidine sont importants pour positionner correctement le carboxylate Asp-168 pour combler les deux ions métalliques. La chaîne latérale Asp-62 est orientée vers le carboxylate Glu-18, à l’extérieur du site catalytique. Asp-62 peut avoir un rôle important dans la catalyse car il est supposé moduler le pKa de His-60 et, par conséquent, influencer la liaison irion métallique dans le site M2. En outre, il pourrait être impliqué structurellement dans la stabilisation du site catalytique sur la liaison d’ion métallique, favorisant la formation du dodecamer.

Figure 1 : Représentation schématique du clonage TM_1050 ORF en vecteur pBAD par recombinaison homologue.
L’ORF est flanqué de deux séquences de 30 bp homologues au promoteur BAD end et à la séquence en amont du site de restriction PmeI. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
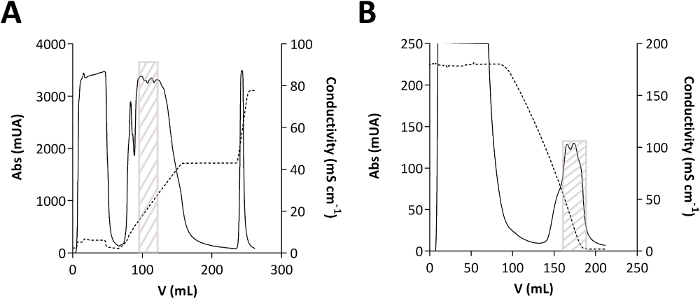
Figure 2 : Chromatogrammes de purification TmPep1050.
(A) Chromatographie d’échange d’anion. (B) Chromatographie d’interaction hydrophobe. L’absorption (Abs), exprimée en milliunits d’absorption (MUA), est montrée en ligne claire. La conductivité, exprimée en mS cm-1,est affichée en ligne pointillée. La boîte grise indique où TmPep1050 s’allonge sur les chromatogrammes. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 3 : Chromatographie d’exclusion de taille de (A) TmPep1050 dodecamer, (B) TmPep1050H60A H307A, et (C) TmPep1050H60A.
Des échantillons ont été analysés à l’aide de résine SEC emballée dans une colonne de 120 mL. Absorbance (Abs) est exprimée en milliunits d’absorption (MUA). (D) Étalonnage de la colonne SEC à l’aide de thyroglobuline (T), ferritine (F), aldolase (Ald), conalbumin (C), et albumine (Alb) comme normes. La corrélation entre le logarithme de la masse relative et le volume d’élitution est linéaire, avec un R2 de 0,91. Les intervalles de confiance de 95 % sont représentés sous forme de points. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 4 : Optimisation de la cristallisation de TmPep1050H60A H307A.
(A) La première stratégie d’optimisation consiste en un pH variable (entre 4,5 et 6,0) par rapport à la concentration peg3350 (entre 5% et 25%). La plaque de cristallisation est schématisée, et les puits sont codés en couleur : rouge pour précipité, jaune pour les polycrystals et vert pour les monocrystals. (B) La deuxième stratégie d’optimisation comprend l’utilisation de graines diluées 25x avec une variation plus étroite de pH par rapport à PEG3350. (C) Forme et taille en cristal avant (image supérieure) et après l’optimisation et le microsédage de cristallisation (image inférieure). S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 5 : Extraits de la sortie du journal CORRECT. LP de TmPep1050H60A H307A indexation des données par XDS.
Panneau supérieur, les treillis bravais possibles, le plus probable étant mC, mP, et oC. Panneau intermédiaire, statistiques globales des données indexées dans le groupe spatial C2221. Panneau inférieur, statistiques globales des données indexées dans le groupe spatial P21. S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.

Figure 6 : Structure de TmPep1050H60A H307A.
(A) Alignement structurel d’une sous-unité TmPep1050H60A H307A (rouge, code PDB 5NE9) contre une sous-unité dodecamer (blanc, code PDB 6NW5) et une sous-unité dimer (bleu, code PDB 5NE6). Les flèches indiquent les dissemblables structurels entre les dodecamers et les dimers. (B) Gros plan du site actif TmPep1050H60A H307A (rouge) par rapport au site actif de TmPep1050 dimère (bleu) et dodécaamer (blanc). S’il vous plaît cliquez ici pour voir une version plus grande de ce chiffre.
| TmPep1050 H60A H307A TmPep1050H60A H307A | |
| Collecte de données | |
| Température (K) | 100 |
| Source de rayonnement | SOLEIL Proxima 2 |
| Longueur d’onde (Å) | 0.9801 |
| Détecteur | Dectris Eiger X 9M |
| Plage d’oscillation (°) | 0.1 |
| Temps d’exposition (s) | 0.025 |
| Groupe spatial | P 1 21 1 |
| Paramètres des cellules unitaires | |
| α, β, γ (°) | 90.00, 110.69, 90.00 |
| a, b, c (Å) | 43.24, 137.79, 61.11 |
| Résolution | 43.99 – 2.37 (2.52-2.37) |
| Réflexions uniques | 26.902 |
| Rmerge (%) | 0.14 |
| Redondance | 6,815 |
| 8.64 (2.12) | |
| Exhaustivité (%) | 99.6 (97.9) |
| CC1/2 (%) | 99.2 (84.1) |
| Raffinement | |
| Résolution | 43.99 – 2.37 |
| Réflexions | 26.9 |
| Rnombre de tests libres | 1345 |
| Rtravail/Rgratuit | 0.206/0.234 |
| Molécules protéiques par ASU | 2 |
| VM (Å3/Da) | 2.37 |
| Teneur en solvants (%) | 49.0 |
| Atomes protéiques/solvants | 4,559/96 |
| longueurs .m.s.d. obligations (Å) | 0.31 |
| r.m.s.d. angles de liaison (°) | 0.51 |
| Facteurs B moyens (Å2) | 57.0 |
| Ramachandran φ/ψ (%) | 95.02 / 0.17 |
| Loi jumelle | h, -k, -h-l |
| Code PDB | 5NE9 (en) |
Tableau 1 : Statistiques sur la collecte et le raffinement des données. Les valeurs entre parenthèses sont pour la coquille la plus haute résolution.
Discussion
Le protocole décrit ci-après permet de comprendre la transition dimer-dodecamer de TmPep1050 au niveau structurel. La méthodologie a été expérimentée précédemment pour déterminer la structure des deux oligomers TmPep105011. L’étape la plus difficile a été de trouver des conditions favorisant la dissociation des dodecamers en dimers stables. De telles conditions ont dû être assez douces pour permettre la réassociation des dimers dans des dodecamers quand le cofacteur d’ion en métal a été ajouté. La séparation des oligomères a également été une étape cruciale car elle conditionnait les études structurelles et la caractérisation biochimique (p. ex., étudier la réassociation du dodécaamer à diverses doses de Co2+). Le remplacement moléculaire, une méthode éprouvée pour la détermination de phase, a été employé pour résoudre les structures des oligomers de TmPep1050 et de ses variantes. Le protocole proposé peut être adapté pour étudier d’autres métallo-enzymes dont les états d’oligomérisation dépendent de la disponibilité de leurs cofacteurs métalliques.
Pour illustrer le protocole, un cas d’étude a été présenté, TmPep1050H60A H307A dont les emplacements de liaison en métal ont été altérés en mutant His-60 et His-307 à l’alanine. Ces résidus lient co2+ sur les sites M2 et M1, respectivement. Interférer dans la liaison métallique aurait pu perturber l’état d’oligomérisation et conduire à une dissociation complète en monomères. Des preuves d’un tel phénomène ont été rapportées pour PhTET2 et PfTET3, deux aminopeptidases M42 de P. horikoshii et P. furiosus,respectivement 24,29. TmPep1050H60A H307A ne s’est pas comporté comme prévu que cette variante formé dimers seulement. Sa structure a montré les mêmes modifications que le dimère de type sauvage, mais à deux petites exceptions près. En effet, les chaînes latérales d’Asp-168 et d’Asp-62 semblaient enfermées dans une orientation non conventionnelle empêchant la stabilisation du site actif. Leur orientation semblait être imposée par His-60 et His-307 car de telles modifications n’ont pas été observées dans les variantes de mutation à point unique.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Nous remercions Martine Roovers d’avoir relu ce document et d’avoir formulé des commentaires constructifs. L’accès à la ligne de faisceau Proxima 2 (synchrotron SOLEIL) faisait partie des groupes d’allocation de blocs 20151139.
matériels
| Name | Company | Catalog Number | Comments |
| 1,10-phenanthroline | Sigma-Aldrich | 13, 137-7 | |
| Amicon Ultra 0.5 ml Centrifugal Filters Ultracel 30K | Merck Millipore | UFC503096 | |
| Amicon Ultra 15 Centrifugal Filters Ultracel 30K | Merck Millipore | UFC903024 | |
| Benzonase Nuclease | Merck Millipore | 70664-3 | |
| CCP4 | N/A | visit http://www.ccp4.ac.uk/ | |
| cOmplete EDTA-free | Roche | 5056489001 | |
| Coot | N/A | visit https://www2.mrc-lmb.cam.ac.uk/personal/pemsley/coot/ | |
| Crystal Screen I | Hampton Research | HR2-110 | |
| Crystal Screen II | Hampton Research | HR2-112 | |
| DreamTaq Green PCR Master Mix | ThermoFisher Scientific | K1082 | |
| EasyXtal 15-well tool | NeXtal | 132007 | |
| Escherichia coli PPY strain | N/A | see reference 31 | |
| Escherichia coli XL1 blue strain | Agilent | 200249 | |
| Gel Filtration Calibration Kit HMW | GE Healthcare Life Sciences | 28-4038-42 | |
| Gel Filtration Calibration Kit LMW | GE Healthcare Life Sciences | 28-4038-41 | |
| Gel Filtration Standard | Biorad | 1511901 | |
| GeneJET Plasmid Miniprep Kit | ThermoFisher Scientific | K0503 | |
| Index | Hampton Research | HR2-144 | |
| Litholoops | Molecular Dimensions | ||
| L-leucine-p-nitroanilide | Bachem AG | 40010720025 | |
| Natrix 1 | Hampton Research | HR2-116 | |
| Natrix 2 | Hampton Research | HR2-117 | |
| Neggia plugin | Dectris | N/A | visit https://www.dectris.com/ |
| NeXtal Tubes JCSG Core Suite I | NeXtal | 130724 | |
| NeXtal Tubes JCSG Core Suite II | NeXtal | 130725 | |
| NeXtal Tubes JCSG Core Suite III | NeXtal | 130726 | |
| NeXtal Tubes JCSG Core Suite IV | NeXtal | 130727 | |
| pBAD-TOPO | ThermoFisher Scientific | K430001 | |
| Phenix | N/A | visit https://www.phenix-online.org/ | |
| Phusion High-Fidelity DNA polymerase | ThermoFisher Scientific | F-530L | |
| Salt RX 1 | Hampton Research | HR2-107 | |
| Salt RX 2 | Hampton Research | HR2-109 | |
| SnakeSkin Dialysis Tubing, 3.5K MWCO | ThermoFisher Scientific | 88242 | |
| Source 15Phe | GE Healthcare Life Sciences | 17014702 | |
| Source 15Q | GE Healthcare Life Sciences | 17094705 | |
| Superdex 200 prep grade | GE Healthcare Life Sciences | 17104301 | |
| Thermotoga maritima MSB8 strain | American Type Culture Collection | ATCC 43589 | |
| TmCD00089984 | DNASU Plasmid Repository | N/A | |
| XDS | N/A | visit http://xds.mpimf-heidelberg.mpg.de/ | |
| xdsme | N/A | visit https://github.com/legrandp/xdsme |
Références
- Levy, E. D., Teichmann, S. A. Structural, Evolutionary, and Assembly Principles of Protein Oligomerization. Progress in Molecular Biology and Translational Science. 117, 25-51 (2013).
- Selwood, T., Jaffe, E. K. Dynamic dissociating homo-oligomers and the control of protein function. Archives of Biochemistry and Biophysics. 519 (2), 131-143 (2012).
- Jaffe, E. K. The Remarkable Character of Porphobilinogen Synthase. Accounts of Chemical Research. 49 (11), 2509-2517 (2016).
- Ramström, H., et al. Properties and Regulation of the Bifunctional Enzyme HPr Kinase/Phosphatase in Bacillus subtilis. Journal of Biological Chemistry. 278 (2), 1174-1185 (2003).
- Rudyak, S. G., Brenowitz, M., Shrader, T. E. Mg2+-Linked Oligomerization Modulates the Catalytic Activiy of the Lon (La) Protease from Mycobacterium smegmatis. Biochemistry. 40 (31), 9317-9323 (2001).
- Yamamoto, S., Storey, K. B. Dissociation-Association of lactate dehydrogenase Isozymes: Influences on the formation of tetramers vs. dimers of M4-LDH and H4-LDH. International Journal of Biochemistry. 20 (11), 1261-1265 (1988).
- Sirover, M. A. Structural analysis of glyceraldehyde-3-phosphate dehydrogenase functional diversity. The International Journal of Biochemistry & Cell Biology. 57, 20-26 (2014).
- Gupta, V., Bamezai, R. N. K. Human pyruvate kinase M2: A multifunctional protein: Multifunctional Human PKM2. Protein Science. 19 (11), 2031-2044 (2010).
- Wiegand, G., Remington, S. J. Citrate synthase: Structure, Control, and Mechanism. Annual Review of Biophysics and Biophysical Chemistry. 15, 97-117 (1986).
- Libonati, M., Gotte, G. Oligomerization of bovine ribonuclease A: structural and functional features of its multimers. Biochemical Journal. 380 (2), 311-327 (2004).
- Dutoit, R., et al. How metal cofactors drive dimer-dodecamer transition of the M42 aminopeptidase TmPep1050 of Thermotoga maritima. Journal of Biological Chemistry. 294 (47), 17777-17789 (2019).
- Rawlings, N. D., et al. The MEROPS database of proteolytic enzymes, their substrates and inhibitors in 2017 and a comparison with peptidases in the PANTHER database. Nucleic Acids Research. 46 (1), 624-632 (2018).
- Neuwald, A. F., Liu, J. S., Lipman, D. J., Lawrence, C. E. Extracting protein alignment models from the sequence database. Nucleic Acids Research. 25 (9), 1665-1677 (1997).
- Dutoit, R., Brandt, N., Legrain, C., Bauvois, C. Functional Characterization of Two M42 Aminopeptidases Erroneously Annotated as Cellulases. PLoS ONE. 7 (11), 50639(2012).
- Franzetti, B., et al. Tetrahedral aminopeptidase: a novel large protease complex from archaea. The EMBO Journal. 21 (9), 2132-2138 (2002).
- Borissenko, L., Groll, M. Crystal Structure of TET Protease Reveals Complementary Protein Degradation Pathways in Prokaryotes. Journal of Molecular Biology. 346 (5), 1207-1219 (2005).
- Appolaire, A., et al. TET peptidases: A family of tetrahedral complexes conserved in prokaryotes. Biochimie. 122, 188-196 (2016).
- Russo, S., Baumann, U. Crystal Structure of a Dodecameric Tetrahedral-shaped Aminopeptidase. Journal of Biological Chemistry. 279 (49), 51275-51281 (2004).
- Schoehn, G., et al. An Archaeal Peptidase Assembles into Two Different Quaternary Structures: A tetrahedron and a giant octahedron. Journal of Biological Chemistry. 281 (47), 36327-36337 (2006).
- Durá, M. A., et al. The structural and biochemical characterizations of a novel TET peptidase complex from Pyrococcus horikoshii reveal an integrated peptide degradation system in hyperthermophilic Archaea: Characterization of P. horikoshii TET3 peptidase. Molecular Microbiology. 72 (1), 26-40 (2009).
- Basbous, H., Appolaire, A., Girard, E., Franzetti, B. Characterization of a Glycyl-Specific TET Aminopeptidase Complex from Pyrococcus horikoshii. Journal of Bacteriology. 200 (17), 00059(2018).
- Appolaire, A., et al. Small-angle neutron scattering reveals the assembly mode and oligomeric architecture of TET, a large, dodecameric aminopeptidase. Acta Crystallographica Section D Biological Crystallography. 70 (11), 2983-2993 (2014).
- Appolaire, A., et al. The TET2 and TET3 aminopeptidases from P yrococcus horikoshii form a hetero-subunit peptidasome with enhanced peptide destruction properties: TET aminopeptidase multi-subunit complex. Molecular Microbiology. 94 (4), 803-814 (2014).
- Colombo, M., Girard, E., Franzetti, B. Tuned by metals: the TET peptidase activity is controlled by 3 metal binding sites. Scientific Reports. 6 (1), 20876(2016).
- Petrova, T. E., et al. Structure of the dodecamer of the aminopeptidase APDkam598 from the archaeon Desulfurococcus kamchatkensis. Acta Crystallographica Section F Structural Biology Communications. 71 (3), 277-285 (2015).
- Kim, D., et al. Structural basis for the substrate specificity of PepA from Streptococcus pneumoniae, a dodecameric tetrahedral protease. Biochemical and Biophysical Research Communications. 391 (1), 431-436 (2010).
- Chevrier, B., et al. Crystal structure of Aeromonas proteolytica aminopeptidase: a prototypical member of the co-catalytic zinc enzyme family. Structure. 2, 283-291 (1994).
- Rosenbaum, E., Ferruit, M., Durá, M. A., Franzetti, B. Studies on the parameters controlling the stability of the TET peptidase superstructure from Pyrococcus horikoshii revealed a crucial role of pH and catalytic metals in the oligomerization process. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1814 (10), 1289-1294 (2011).
- Macek, P., et al. Unraveling self-assembly pathways of the 468-kDa proteolytic machine TET2. Science Advances. 3 (4), 1601601(2017).
- Edelheit, O., Hanukoglu, A., Hanukoglu, I. Simple and efficient site-directed mutagenesis using two single-primer reactions in parallel to generate mutants for protein structure-function studies. BMC Biotechnology. 9 (1), 61(2009).
- Zhang, Y., Werling, U., Edelmann, W. SLiCE: a novel bacterial cell extract-based DNA cloning method. Nucleic Acids Research. 40 (8), 55(2012).
- Schleif, R. AraC protein, regulation of the l-arabinose operon in Escherichia coli, and the light switch mechanism of AraC action. FEMS Microbiology Reviews. 34 (5), 779-796 (2010).
- McPherson, A., Gavira, J. A. Introduction to protein crystallization. Acta Crystallographica Section F Structural Biology Communications. 70 (1), 2-20 (2014).
- Bergfors, T. Seeds to crystals. Journal of Structural Biology. 142 (1), 66-76 (2003).
- Dauter, Z. Collection of X-Ray Diffraction Data from Macromolecular Crystals. Protein Crystallography. 1607, 165-184 (2017).
- Powell, H. R. X-ray data processing. Bioscience Reports. 37 (5), 0227(2017).
- Battye, T. G. G., Kontogiannis, L., Johnson, O., Powell, H. R., Leslie, A. G. W. iMOSFLM a new graphical interface for diffraction-image processing with MOSFLM. Acta Crystallographica Section D Biological Crystallography. 67 (4), 271-281 (2011).
- Otwinowski, Z., Minor, W. Processing of X-ray diffraction data collected in oscillation mode. Methods in Enzymology. 276, 307-326 (1997).
- Clabbers, M. T. B., Gruene, T., Parkhurst, J. M., Abrahams, J. P., Waterman, D. G. Electron diffraction data processing with DIALS. Acta Crystallographica Section D Structural Biology. 74 (6), 506-518 (2018).
- Kabsch, W. XDS. Acta Crystallographica Section D Biological Crystallography. 66 (2), 125-132 (2010).
- Legrand, P. legrandp/xdsme: March 2019 version working with the latest XDS version. , Jan 26, 2018. Zenodo (2019).
- Evans, P. Scaling and assessment of data quality. Acta Crystallographica Section D Biological Crystallography. 62 (1), 72-82 (2006).
- Wlodawer, A., Minor, W., Dauter, Z., Jaskolski, M. Protein crystallography for non-crystallographers, or how to get the best (but not more) from published macromolecular structures: Protein crystallography for non-crystallographers. FEBS Journal. 275 (1), 1-21 (2008).
- Karplus, P. A., Diederichs, K. Assessing and maximizing data quality in macromolecular crystallography. Current Opinion in Structural Biology. 34, 60-68 (2015).
- Taylor, G. L. Introduction to phasing. Acta Crystallographica Section D Biological Crystallography. 66 (4), 325-338 (2010).
- Rossmann, M. G., Blow, D. M. The detection of sub-units within the crystallographic asymmetric unit. Acta Crystallographica. 15, 24-31 (1962).
- Rossmann, M. G. The molecular replacement method. Acta Crystallographica Section A Foundations of Crystallography. 46 (2), 73-82 (1990).
- McCoy, A. J., et al. Phaser crystallographic software. Journal of Applied Crystallography. 40 (4), 658-674 (2007).
- Adams, P. D., et al. PHENIX: a comprehensive Python-based system for macromolecular structure solution. Acta Crystallographica Section D Biological Crystallography. 66 (2), 213-221 (2010).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Crystallographica Section D Biological Crystallography. 66 (4), 486-501 (2010).
- Chen, V. B., et al. MolProbity: all-atom structure validation for macromolecular crystallography. Acta Crystallographica Section D Biological Crystallography. 66 (1), 12-21 (2010).
- Zwart, P. H., Grosse-Kunstleve, R. W., Lebedev, A. A., Murshudov, G. N., Adams, P. D. Surprises and pitfalls arising from (pseudo)symmetry. Acta Crystallographica Section D Biological Crystallography. 64 (1), 99-107 (2008).
- Yeates, T. O. Detecting and overcoming crystal twinning. Methods in Enzymology. 276, 344-358 (1997).
- Terwilliger, T. C. Using prime-and-switch phasing to reduce model bias in molecular replacement. Acta Crystallographica Section D Biological Crystallography. 60 (12), 2144-2149 (2004).
- Terwilliger, T. C., et al. Iterative model building, structure refinement and density modification with the PHENIX AutoBuild wizard. Acta Crystallographica Section D Biological Crystallography. 64 (1), 61-69 (2008).
- Krissinel, E., Henrick, K. Inference of Macromolecular Assemblies from Crystalline State. Journal of Molecular Biology. 372 (3), 774-797 (2007).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon