Method Article
Cristalografía de rayos X para estudiar la transición del estado oligomérico de la Thermotoga maritima M42 Aminopeptidasse TmPep1050
En este artículo
Resumen
Este protocolo ha sido desarrollado para estudiar la transición dimer-dodecamer de TmPep1050, un M42 aminopeptidasa, a nivel estructural. Es una tubería sencilla que va desde la purificación de proteínas hasta el procesamiento de datos de rayos X. La cristalgénesis, la indexación de conjuntos de datos y el reemplazo molecular se han enfatizado a través de un caso de estudio, la variante TmPep1050H60A H307A.
Resumen
Los aminopeptidases M42 forman complejos funcionalmente activos hechos de 12 subunidades. Su proceso de montaje parece estar regulado por sus cofactores de iones metálicos que desencadenan una transición dimer-dodecamer. Tras la unión de iones metálicos, se producen varias modificaciones estructurales en el sitio activo y en la interfaz de interacción, dando forma a los dimers para promover el autoensamble. Para observar tales modificaciones, los oligómeros estables deben aislarse antes del estudio estructural. Reportado aquí es un método que permite la purificación de dodecamers estables y dimers de TmPep1050, un M42 aminopeptidasa de T. maritima,y su determinación de la estructura por cristalografía de rayos X. Los dimers se preparaban a partir de dodecamers mediante la eliminación de iones metálicos con un agente quelante. Sin su cofactor, los dodecamers se volvieron menos estables y se disociaron por completo al calentarse. Las estructuras oligoméricas fueron resueltas por el enfoque de reemplazo molecular sencillo. Para ilustrar la metodología, la estructura de una variante TmPep1050, totalmente deteriorada en la unión de iones metálicos, se presenta mostrando ninguna descomposición adicional de dimers a monómeros.
Introducción
La oligomerización es un proceso predominante que dicta las funciones biológicas de muchas proteínas. En Escherichia coli, se estima que sólo el 35% de las proteínas son monoméricas1. Algunas proteínas, llamadas morfeínas, pueden incluso adoptar varios estados oligoméricos con subunidades que tienen una estructura distinta en cada estado oligomérico2. La transición entre sus estados oligoméricos es a menudo un medio para regular la actividad proteica ya que cada estado oligomérico puede tener una actividad o función específica diferente. Varios ejemplos de morfeeeinas han sido bien documentados en la literatura, en particular el porphobilinogen synthase3, HPr kinasa/phosphatase4, Lon proteasa5, lactato deshidrogenasa6, gliceraldehído-3-fosfato deshidrogenasa7, pirateuvasa8, cito de lactato9, y ribonusa10 . Recientemente, describimos el M42 aminopeptidasa TmPep1050, otro ejemplo de enzima con comportamiento similar a la morfeína, cuya actividad depende de sus estados oligoméricos11. La transición entre sus estados oligoméricos está mediada por sus cofactores metálicos que inducen varias modificaciones estructurales de las subunidades.
La familia M42 aminopeptidasa pertenece al clan MH12,13,y se distribuye ampliamente entre Bacterias y Archaea14. Los aminopeptidases M42 son enzimas dinucleares genuinas degradando péptidos hasta 35 residuos de aminoácidos en longitud15. Adoptan una peculiar estructura en forma de tetraedro hecha de 12 subunidades con sus sitios activos orientados hacia una cavidad interna. Tal disposición se describe a menudo como una nano-compartimentación de la actividad para evitar la proteólisis incontrolada. La función fisiológica de las aminopeptidasas M42 puede estar asociada con el proteosoma, péptidos hidrolizantes resultantes de la degradación de proteínas16,17. Pyrococcus horikoshii posee cuatro Aminopeptidases M42, cada uno presentando especificidades distintas pero complementarias18,19,20,21. Singularmente, heterocomplejos hechos de dos tipos diferentes de subunidades se han descrito en P. horikoshii, sugiriendo la existencia de complejos peptidasome22,23.
Varias estructuras de M42 aminopeptidases se han descrito en la literatura11,16,18,19,20,24,25,26. La subunidad se compone de dos dominios distintos, un dominio catalítico y un dominio de dimerización. El dominio catalítico adopta un pliegue común α/β conservado en todo el clan MH, siendo el dominio catalítico arquetípico la aminopeptidasa Ap1 de Vibrio proteolyticus27. El dominio de dimerización adopta un pliegue similar a laPDS 16 y puede tener, además de su papel en la oligomerización, un papel en el control del acceso al sustrato y la unión en la cavidad interna11. Como el bloque de construcción básico es un dimer, el dodecamer se describe a menudo como la asociación de seis dimers, cada dimer se coloca en cada borde del tetraedro16. La oligomerización de los aminopeptidases M42 depende de la disponibilidad de sus cofactores metálicos. Los iones metálicos divalentes, a menudo Zn2+ y Co2+,participan catalíticamente en la unión a péptidos y la hidrólisis. Se encuentran en dos sitios de enlace distintos, a saber, los sitios M1 y M2. Los dos iones metálicos también conducen y afinan finamente la oligomerización como se demuestra para PhTET2, PhTET3, PfTET3 y TmPep105011,24,28,29. Cuando se agotan los cofactores metálicos, el dodecamer se desmonta en dimers, como en PhTET2, PhTET3, y TmPep105011,16,28,o incluso monómeros, como en PhTET2 y PfTET324,29.
Aquí se presenta un protocolo utilizado para estudiar las estructuras de los oligómeros TmPep1050. Este protocolo es un conjunto de métodos comunes que incluyen purificación de proteínas, cribado de actividad proteolítica, cristalización, difracción de rayos X y reemplazo molecular. Se enfatizan las sutilezas inherentes al tratamiento de las metaloenzimas, la oligomerización de proteínas, la cristalización de proteínas y el reemplazo molecular. También se presenta un caso de estudio para mostrar si los dodecamers TMPep1050 pueden disociarse aún más en monómeros o no. Para abordar esta pregunta, se ha estudiado una variante TmPep1050, TmPep1050H60A H307A,cuyos sitios de unión de metales se ven deteriorados al mutar His-60 (sitio M2) e His-307 (sitio M1) a los residuos de Ala. Este protocolo puede ser acomodado para estudiar otros Aminopeptidases M42 o cualquier metaloenzimas con comportamiento morfeina-como.
Protocolo
1. Producción y purificación del recombinante TmPep1050
NOTA: A continuación se describe el procedimiento de clonación y purificación de tmpep1050 de tipo salvaje adaptado de un estudio anterior11. Alternativamente, la clonación se puede hacer usando un gen sintético. Para generar variantes TmPep1050, se puede realizar mutagénesis dirigida por el sitio siguiendo, por ejemplo, las reacciones de imprimación única en el método30del protocolo paralelo (SPRINP). El protocolo de purificación se puede utilizar para las variantes TmPep1050. El uso de Su etiqueta debe evitarse ya que interfiere con la unión de iones metálicos.
- Diseño vectorial de expresión
- Adquirir ADN genómico de Thermotoga maritima MSB8 (ATCC 43589) o TmCD00089984 (Centro Conjunto de Genómica Estructural).
- Amplificar TM_1050 marco de lectura abierto (ORF) utilizando ADN genómico o plásmido de plantilla, una polimerasa de ADN de alta fidelidad, y las siguientes imprimaciones: ocej419 (5'- TTTAACTTTAAGAAGGAGATATACATACCCATGAAGGAACTGATGAAAGCTG) y ocej420 (5'- ATCCGCCAAAACAGCCAGCCAGCTAGACCGTTTAGCCCCCACCTGATGATGAG). Ejecutar el cribado de reacción en cadena de la polimerasa (PCR) de acuerdo con el siguiente esquema: 5 min a 95oC, 30 ciclos de 3 pasos (30 s a 95oC, 30 s a 55oC, 90 s a 72oC) y 10 min a 72oC como paso final.
- Clonar el fragmento de PCR en un vector de expresión adecuado (Tabla de materiales) por recombinación homóloga ( Figura1) en E. coli según el protocolo SLiCE31. A 50 ng de vector linealizado, agregue el fragmento de PCR en una relación molar 10:1 de fragmento a vector, 1 ml de extracto de cepa de PPY, pH Tris-HCl de 50 mM 7,5, 100 mM MgCl2, 10 mM de ATP y 10 mM de ditiothreitol (TDT) para un volumen de reacción de 10 l. Incubar durante 1 h a 37oC.
- Transforme la cepa azul E. coli XL1 químicamente competente (o cualquier cepaadecuada de recA) con 1 ml de reacción de recombinación. Encaje las células en el medio LB que contenga ampicilina de 100 g/ml. Incubar las placas durante la noche a 37oC.
- Escoge colonias en placas LB frescas con ampicilina de 100 g/ml. Incubar las placas a 37oC durante un mínimo de 8 h.
- Pantalla para candidatos positivos por colonia PCR utilizando el par de imprimación adecuado (5'- ATGCCATAGCATTTTTATCC y 5'- ATTTAATCTGTATCAGGC si se utiliza el vector recomendado listado en la Tabla de Materiales). Con un extremo de microtipo, rasque una colonia escogida y transfiera las células a 20 l de mezcla de reacción que contenga 0,5 m de cada imprimación y 10 ml de una mezcla comercial de polimerasa de ADN Taq.
- Ejecute el cribado de PCR de acuerdo con el siguiente esquema: 5 min a 95 oC como paso de desnaturalización, 30 ciclos de 3 pasos (30 s a 95 oC, 30 s a 55 oC, 90 s a 72 oC) y 10 min a 72 oC como paso final.
NOTA: Las reacciones de PCR se pueden almacenar durante la noche en la máquina PCR a 12 oC. - Cargue 10 l de cada reacción de PCR en un gel de agarosa del 0,8% preparado en tampón Tris-acetate-EDTA (TAE). Ejecute la electroforesis durante 25 min a 100 V.
NOTA: Se espera un amplicon de 1,1 kbp. - Extraiga plásmidos de los candidatos utilizando un kit comercial(Tabla de materiales)y secuenciarlos utilizando el mismo par de imprimación utilizado en el paso 1.1.6.
- Cultivo celular
NOTA: Cuando se ha identificado un candidato adecuado mediante la secuenciación, el clon se puede utilizar directamente como expresión si se utiliza el vector recomendado (Tabla de materiales). En ese caso, la expresión está controlada por el promotor PBAD inducible en la arabinosa32.- Inocular 10 ml de medio LB que contenga 100 g/ml de ampicilina con el candidato e incubar la precultivo durante la noche a 37oC bajo temblor orbital. Añadir 5 mL de la precultivo a 1 L de medio LB con ampicilina de 100 g/ml. Mente respetar una relación aire-líquido de 3.
- Deje que las células crezcan a 37 oC bajo temblor orbital. Monitoree la densidad óptica a 660 nm (OD660).
- Cuando la OD660 haya alcanzado los 0,5 x 0,6, enfríe rápidamente el cultivo durante 5 minutos sobre hielo y transfiéralo a una incubadora establecida en 18 oC.
- Añadir 0,2 g/L de arabinosa para inducir la expresión génica e incubar durante 12-18 h a 18 oC.
- Cosecha las células centrifugando el cultivo a 6.000 x g durante 30 min a 4oC. Deseche el sobrenadante y lave las células con 100 ml de 0,9% (p/v) naCl.
- Centrifugar de nuevo a 6.000 g durante 15 min a 4 oC y deseche el sobrenadante.
NOTA: Los pellets celulares se pueden utilizar directamente para la extracción de proteínas o almacenarse a -80 oC.
- Purificación de proteínas
- Resuspender los gránulos celulares en 40 mL de 50 mM MOPS, 1 mM CoCl2, pH 7.2. Añadir 1 l de 25 U/L DNA/ARN endonucleasa y un comprimido de cóctel inhibidor de la proteasa que no contenga EDTA. Sonicar la suspensión en modo de pulso bajo enfriamiento durante 30 min.
- Centrifugar el extracto crudo a 20.000 x g durante 30 min a 4oC. Recoger el sobrenadante y calentarlo en un baño de agua a 70 oC durante 10 min.
- Centrifugar el extracto de célula desnaturalada a 20.000 x g durante 30 min a 4 oC y recoger el sobrenadante para su purificación.
- Utilice resina de intercambio de aniones adecuada (Tabla de materiales) embalada en una columna de 15 ml de volumen. Consulte las recomendaciones del fabricante para conocer el caudal de trabajo y el límite de presión de columna. Equilibrar la resina con 50 mM MOPS, 1 mM CoCl2, pH 7.2.
- Cargue el sobrenadante recopilado del paso 1.3.3 en la columna. Monitorear la absorbancia del eluido a 280 nm. Cuando haya alcanzado la línea de base, proceda a la elución.
- Aplicar un gradiente de 0 a 0,5 M NaCl en 50 mM MOPS, 1 mM CoCl2, pH 7.2 para 5 volúmenes de columna (CV). Espere hasta que la conductividad se estabite y la absorbancia haya alcanzado la línea de base.
- Aplicar un gradiente final de 0,5 a 1 M naCl en 50 mM MOPS, 1 mM CoCl2,pH 7.2 para 1 CV.
- Analice algunas fracciones (consulte la Figura 2A para obtener orientación) mediante electroforesis de gel de poliacrilamida de dodecil sulfato de sodio (SDS-PAGE).
NOTA: TmPep1050 aparece como una banda de 36 kDa después de la tinción de Coomassie. Alternativamente, la presencia de TmPep1050 puede confirmarse mediante un ensayo de actividad (ver sección 2.1). En este paso, las fracciones se pueden almacenar a 4 oC durante la noche. - Agrupar las fracciones que contienen TmPep1050 y añadir polvo finamente molido de (NH4)2SO4 para obtener una concentración de 1,5 M (NH4)2SO4. Mezcle suavemente invirtiendo el tubo boca abajo hasta la disolución completa.
- Utilice resina de interacción hidrófoba (Tabla de materiales) embalada en una columna de 30 ml de volumen. Consulte las recomendaciones del fabricante para conocer el caudal de trabajo y el límite de presión de columna. Equilibrar la resina con 50 mM MOPS, 1,5 M (NH4)2SO4, 1 mM CoCl2, pH 7,2.
- Cargue la muestra en la columna y supervise la absorbancia del eluido a 280 nm. Cuando la absorbancia haya alcanzado la línea de base, eluir las proteínas unidas aplicando un gradiente de 1,5 M a 0 M (NH4)2SO4 en 50 mM MOPS, 1 mM CoCl2, pH 7.2 para 5 CV.
- Analice algunas fracciones (consulte la figura 2B para obtener orientación) por SDS-PAGE.
NOTA: TmPep1050 aparece como una banda de 36 kDa después de la tinción de Coomassie. Alternativamente, la presencia de TmPep1050 puede confirmarse mediante un ensayo de actividad (ver sección 2.1). En este paso, las fracciones se pueden almacenar a 4 oC durante la noche. - Agrupa las fracciones que contienen TmPep1050 y concentra a 2 ml utilizando unidades de ultrafiltración con límite de 30 kDa (Tabla de Materiales). Proceda a la sección 1.4 para determinar el peso molecular.
- Cromatografía de exclusión de tamaño
- Utilice resina de exclusión de tamaño (Tabla de materiales) embalada en una columna de 120 ml de volumen. Consulte las recomendaciones del fabricante para conocer el caudal de trabajo y el límite de presión de columna. Equilibrar la resina con 50 mM MOPS, 0,5 M (NH4)2SO4, 1 mM CoCl2, pH 7,2.
- Cargue la muestra en la columna y supervise la absorbancia del eluido a 280 nm. Fraccionar desde el volumen muerto de la columna (0,33 CV) hasta el final de la elución (1 CV).
- Mida el volumen de elución para cada pico observado.
NOTA: Para obtener orientación, eldemecérico TmPep1050 eluta a 82 ml(Figura 3A) en condiciones experimentales actuales, mientras que el dimerico TmPep1050, como la variante TmPep1050H60A H307A, eluda a 95 mL(Figura 3B). Algunos TMPep1050 pueden adoptar ambas formas oligoméricas, como TmPep1050H60A (Figura 3C). - Analizar fracciones correspondientes a las máximas y colas de los picos observados utilizando SDS-PAGE.
NOTA: TmPep1050 aparece como una banda de 36 kDa después de la tinción de Coomassie. - Acople las fracciones de cada pico y concéntrese utilizando unidades de ultrafiltración con un límite de 30 kDa(Tabla de materiales)para obtener una concentración de 300 oM.
- Mida la absorbancia a 280 nm en un espectrofotómetro de nano vol volumen y calcule la concentración utilizando el coeficiente de extinción molecular de 18.910 M-1 cm-1.
- Conservar la proteína purificada a -18oC.
- Para determinar el peso molecular, calibrar la columna de cromatografía de exclusión de tamaño (SEC) utilizando estándares de peso molecular (Tabla de materiales). Analice los estándares utilizando 50 mM MOPS, 0.5 M (NH4)2SO4, 1 mM CoCl2 pH 7.2 como búfer de ejecución.
2. Ensayo de actividad y preparación de apoenzimas
NOTA: Originalmente, la apoenzima se preparó diluyendo 1 volumen de TmPep1050 en 10 volúmenes de 2,1 M de ácido málico pH 7.0 y concentrándose de nuevo a 1 volumen antes de la diálisis11. A continuación se presenta un procedimiento alternativo utilizando 1,10-fenantrolina, un quelante de iones metálicos. Este procedimiento reduce la pérdida de proteínas y da los mismos resultados que el método publicado anteriormente.
- Ensayo de actividad
- Preparar una solución de 100 mM L-leucina-p-nitroanilida(Tabla de materiales)en metanol.
- Añadir 25 l de 100 mM L-leucina-p-nitroanilida en 965 l de 50 mM MOPS, 250 mC-2,pH 7,2, 10% metanol. Preincubinar la mezcla de reacción a 75oC en un baño seco.
- Diluir la enzima en 50 mM de pH MOPS 7,2 a una concentración de 1 m. Añadir 10 ml a la mezcla de reacción, vórtice, e incubar a 75 oC hasta que se haya vuelto amarillento o durante 1 h.
- Detener la reacción añadiendo 1 ml de 20% de ácido acético. Vórtice bien y deje que se enfríe a temperatura ambiente.
- Transfiera la mezcla de reacción en una célula espectrofotómetro. Lea la absorbancia a 410 nm contra un control negativo (mezcla de reacción incubada sin enzima).
- Preparación de la apoenzila
- Preparar una solución de 1 M 1,10-fenantrolina en etanol. Añadir 10 l de solución en stock de 1,10-fenantrolina a 890 ml de 50 mM MOPS, 0,5 M (NH4)2SO4, pH 7,2. Añadir 100 l de tmpep1050 purificado (concentración de 300 m-1 mM).
- Compruebe la pérdida de actividad utilizando el ensayo de actividad descrito en la sección 2.1 sin añadir CoCl2 en la mezcla de reacciones.
- Transfiera la muestra en un tubo de diálisis. Dialyze contra 200 mL de 50 mM MOPS, 0,5 M (NH4)2SO4, pH 7,2 a 4 oC. Intercambie tres veces el dialato con un tampón fresco durante la diálisis de 48 h.
- Recoger la muestra del tubo de diálisis y concentrar de nuevo a 100 l utilizando unidades de ultrafiltración con límite de 30 kDa (Tabla de materiales). Compruebe la concentración leyendo la absorbancia a 280 nm utilizando un espectrofotómetro de nano-volumen.
- Preparación de dimer
- Diluir la apoenzida a una concentración de 1 mM en 50 mM DE MOPS, 0,5 M (NH4)2SO4, pH 7,2. Incubar durante 2 h a 75oC en un baño seco, luego dejar que la muestra se enfríe a temperatura ambiente.
- Concentrar la muestra en una concentración enzimática de al menos 50 oM. Compruebe el peso molecular según la SEC (ver sección 1.4). El pico de elución debe oscilar entre 82 ml y 95 ml (en condiciones experimentales actuales).
3. Cristalización TmPep1050
NOTA: La cristalización de proteínas sigue siendo una ciencia empírica, ya que es un fenómeno multifactorial33. Mientras que algunos parámetros pueden ser identificados y controlados (como la temperatura, el pH, la concentración del agente de precipitación), otros pueden influir esquivamente la cristalización (como la pureza de proteínas y químicas, proteólisis, historial de muestras). Hoy en día la cristalización de proteínas se aborda de manera racional y sistemática gracias a un montón de condiciones comerciales de cribado de cristalización y automatización. La optimización de una condición de cristalización, sin embargo, se basa principalmente en un enfoque de ensayo y error. A continuación se describe un plano para cristalizar proteínas y varios consejos para optimizar las condiciones de cristalización.
- Examen de cristalización
NOTA: Utilizando kits de cristalización comercial, se han obtenido cristales de dodecameric TmPep1050 en 2,2 M de ácido dH-meico DL 7,0, 0,1 M Bis-Tris propano pH 7,0 y 0,18 M citrato de triamoniio, 20% de polietilenglicol (PEG) 3350. Cristales de dimerico TmPep1050 se han obtenido en 0,1 M citrato de sodio pH 5,6, 0,2 M acetato de amonio, 30% PEG4000. Cristales de dodecamers aparecen dentro de una semana y alcanzan su tamaño completo en un mes. Los cristales de los dimers suelen aparecer dentro de las 24 h y crecen hasta el tamaño completo en una semana.- Adquiera varios kits de cristalización comercial (consulte la Tabla de materiales para ver ejemplos).
- Configure placas de cristalización (Tabla de materiales) para el método de caída colgante. Llene los potes con 500 ml de cada solución de un kit de cribado de cristalización.
- Para cada pozo, configure un soporte de cristalización. En el soporte, deposite una gota de 1 l de proteína purificada (generalmente 10 mg/ml).
- Inmediatamente pipetear 1 l de solución de cristalización del pozo. Añádalo cuidadosamente a la gota de proteína y mezcle suavemente pipeteando al revés tres veces. La caída debe permanecer semiesférica sin burbujas.
- Atornille el soporte en la parte superior del pozo correspondiente. Repita la operación para todo el kit.
- Después de configurar las placas, observe cada gota con un binocular. Consulte la guía del usuario del kit de cristalización para la interpretación (gota clara, separación de fases, precipitado, agujas, etc.).
- Incubar las placas a 20oC. Revise las placas una vez al día durante la primera semana y una vez por semana después.
- Punte cada pozo utilizando la hoja de puntuación y la guía del usuario proporcionada con los kits de cristalización.
- Optimización de la cristalización
NOTA: Las condiciones iniciales de cristalización de dodecameric TmPep1050 se han optimizado a 2,1 M DL-ácido málico pH 6,75 y 0,18 M de citrato de triamonio pH 7,5, 40% (p/v) PEG3350 mientras que la condición de cristalización de dimerico TmPep1050 se ha desplazado a 0,1 M citrato de sodio pH 6,0, 10% (p/v) PEG3350. Se ha necesario un ciclo de siembra para mejorar la cristalinidad. A partir de entonces se describe cómo se ha optimizado la cristalización de la variante TmPep1050H60A H307A.- Preparar soluciones de stock de tampón de citrato sódico de 0,5 M a diferentes pH (4,5, 5,2 y 6,0) y 50% (p/v) solución PEG3350.
- Configure una placa de cristalización como una matriz de pH frente a agente de precipitación (véase la figura 4A).
- Incubar la placa a 20oC. Observar cada pozo con un binocular una vez al día durante una semana.
- Puntuar cada pozo de acuerdo con el tamaño y la forma del cristal. Seleccione una condición que dé cristales de al menos 50 m. Proceda a la microsedición.
- Microsempresa
NOTA: La microsedredación es un método potente para mejorar la forma, el tamaño y la cristalinidad de los cristales proteicos34. Un enfoque de siembra más rápido es la siembra de rayas usando un bigote de gato. Vea la Figura 4 como un ejemplo de cómo la optimización de la cristalización y la microsedadura han mejorado la forma y el tamaño del cristal para TmPep1050H60A H307A.- Prepare las semillas usando el pozo seleccionado en el paso 3.2.4.
- Aumente el volumen de una gota que contenga cristales a 10 ml añadiendo solución de cristalización desde el pozo. Entuere la gota y añada 90 l de solución de cristalización del pozo. Vórtice a fondo y mantener las semillas en hielo.
- Preparar varias diluciones de las semillas: 1x, 10x, 25x y 100x. Vortex bien las semillas antes de pipetear. Mantenga las diluciones en hielo.
- Para cada dilución de semillas, configure una placa de cristalización como una matriz de pH frente a agente de precipitación (ver Figura 4B). Utilice las soluciones de stock preparadas en el paso 3.2.1.
- Al hacer la caída, añadir 0,2 l de semillas para una gota de 2 l. Incubar la placa a 20oC. Observar cada pozo con un binocular una vez al día durante una semana.
NOTA: Se debe mejorar la distribución y la forma del tamaño del cristal, consulte la Figura 4C como ejemplo para TmPep1050H60A H307A. Para usos posteriores, las semillas se pueden almacenar a -20 oC.
4. Difracción de rayos X
- Recogida de cristales
NOTA: La preparación de la muestra depende de la instalación de la fuente de rayos X (instalaciones domésticas frente a sincrotrón). Utilice dispositivos de almacenamiento (viales y cesta del porta viales) en consecuencia. La adición de crioprotector (como el glicerol) puede ser necesaria dependiendo de la concentración del agente de sal/precipitación. Para los cristales TmPep1050, un crioprotector no es necesario ya que la concentración de PEG o tampón es lo suficientemente alta como para evitar cristales de agua.- Prepare un baño lleno de nitrógeno líquido, sumerja los viales o la cesta utilizados para la manipulación de muestras.
- Configure bucles de selección de muestras de diferentes tamaños: 100, 150 y 200 m(Tabla de materiales). Elija los tamaños de acuerdo con el tamaño del cristal.
- Usando un binocular, compruebe la gota que contiene cristales y detecte cristales aislados (los más fáciles de recoger). Con un lazo, elija suavemente un cristal de la parte inferior. Sumerja inmediatamente el lazo en nitrógeno líquido y coloque el lazo en un vial adecuado.
- Recopilación de datos
NOTA: La recopilación de datos puede variar mucho dependiendo de la fuente de rayos X (instalaciones domésticas frente a sincrotrón) y la sensibilidad del detector. La estrategia de recolección también puede diferir mucho de una muestra a otra, dependiendo de la resolución, intensidad de punto, grupo espacial, etc. El tema ha sido ampliamente revisado por Dauter35.- Monte el lazo que lleva el cristal en la cabeza del goniómetro del difractómetro.
- Ajuste el cabezal del goniómetro a lo largo de los ejes XYZ para alinear el cristal con la trayectoria del haz de rayos X.
- Establezca la longitud de onda en 0,98o y mueva el detector para obtener 2o de resolución.
- Inicie una breve recopilación de datos adquiriendo imágenes en al menos dos orientaciones de cristal diferentes. Tome 10 imágenes (1 imagen por cada 0,1o) a 0o y 90o.
- Compruebe las imágenes recopiladas con el software adecuado (por ejemplo, ADXV, XDS-Viewer o Albula Viewer). Determine la intensidad del punto y la resolución más alta donde se ven los puntos. Compruebe también la monocristalinidad y la separación de puntos.
- Eventualmente, repita los pasos 4.2.3-4.2.5 cambiando la posición del detector para una resolución mayor o menor y el tiempo de exposición de acuerdo con la observación.
- Inicie la recopilación de datos en torno a 360o con 1 imagen tomada por cada 0,1o. Recuerde ajustar la posición del detector y el tiempo de exposición de forma óptima.
5. Indexación, reemplazo molecular y construcción de modelos
- Indexación
NOTA: La indexación es un método para medir la intensidad de los puntos de difracción, dando las amplitudes de los factores de estructura36. Cuatro paquetes de software se utilizan comúnmente para el procesamiento de imágenes recogidas: Mosflm37, HKL200038, DIALS39y XDS40. Este último se ha utilizado para indexar los conjuntos de datos obtenidos de la difracción de cristal TmPep1050.- Instale el paquete XDS y XDSME41. Si procesa archivos HDF5, instale el plugin XDS Neggia (disponible en el sitio web de Dectris). Para obtener más información, visite la página web wiki de XDS https://strucbio.biologie.uni-konstanz.de/xdswiki/index.php/Main_Page y la página web de XDSME https://github.com/legrandp/xdsme.
- Antes de procesar los datos, cree una carpeta desde la que se ejecutará XDS. Localice la ruta de acceso a las imágenes.
- Para ejecutar XDSME, escriba xdsme /path_to_images/image.extension en una ventana de terminal.
- Después de que XDS haya terminado el trabajo, compruebe correcto. Archivo LP. Tenga en cuenta la probabilidad de la determinación del grupo espacial, la integridad de los datos, la resolución más alta, la mosaico de cristales y la calidad de los datos. Compruebe también XDS_pointless.log para obtener la probabilidad de grupos espaciales.
NOTA: Vea la figura 5 como un ejemplo de salida. - Vuelva a ejecutar XDSME con diferentes soluciones de grupos de espacio propuestas por XDS en una carpeta independiente para evitar sobrescribir el proceso anterior. Escriba xdsme -s space_group_name -c "unit_cell_parameters" /path_to_images/image.extension (por ejemplo, xdsme -s P21 -c "43.295 137.812 61.118 90.000 110.716 90.000).
- Compruebe el CORRECTO. LP y elegir la mejor solución basada en estadísticas de datos.
- Ejecute XSCALE escribiendo xscale.py XDS_ASCII. HKL. Ejecute XDSCONV escribiendo xdsconv.py XSCALE. HKL ccp4.
NOTA: En algunos casos, XDSME no identifica el grupo de espacio o no puede reducir el rango de resolución correctamente o genera estadísticas de datos extrañas. Si se encuentra un problema de este tipo, vale la pena ejecutar XDS de forma nativa. Se deben introducir varios parámetros en el XDS. Archivo de iniciación INP (consulte la página wiki de XDS). Cuando se utiliza XDS, la probabilidad de posibles grupos espaciales se puede comprobar mediante Pointless, parte del paquete CCP442. Para reducir la resolución del conjunto de datos, Rmeas < 60% y I/σ 2 se aceptan comúnmente para determinar la resolución más alta43. Sin embargo, el reemplazo molecular y el refinamiento del modelo se pueden mejorar ampliando la resolución a I/σ 0,5 x 1,5 y CC1/2 hasta 0,2 a 0,444.
- Reemplazo molecular
NOTA: Los datos experimentales dan acceso a la amplitud de los factores estructurales pero, sin conocer la fase, son inútiles. La fase se puede determinar experimentalmente mediante diferentes métodos basados en una señal anómala (de un átomo pesado, por ejemplo)45. El reemplazo molecular es otro método para determinar la fase sin un átomo de dispersión anómalo46,47. Este método utiliza las coordenadas de una molécula relacionada para encontrar y mejorar la fase de forma iterativa. Utilizamos Phaser48 en Phenix GUI49 para reemplazo molecular.- Prepare el modelo inicial para el reemplazo molecular utilizando coordenadas 4P6Y. Del archivo pdb, extraiga el monómero A y truncar sus aminoácidos en alanina utilizando el editor de archivos PDB en Phoenix (bajo la pestaña Herramientas del modelo).
- Ejecute Xtriage en Phoenix (en la pestaña Análisis de datos) con el archivo de reflexión generado por XDSCONV (5.1.9) y la secuencia como entradas.
- Compruebe el archivo de registro de Xtriage. Observe la integridad, el número de subunidades en la unidad asimétrica, la anisotropía, la presencia de anillos de hielo y la ocurrencia de hermanamiento.
- Ejecute Phaser-MR en Phoenix (en la pestaña Reemplazo molecular) para el reemplazo molecular utilizando el archivo de reflexión, la secuencia y el modelo 4P6Y inicial truncado en poli alanina (paso 5.2.1).
- Al finalizar, compruebe si se ha encontrado un modelo y la puntuación del reemplazo molecular. Un factor de traducción Z-puntuación (TFZ) de al menos 8 indica que la solución es definitivamente correcta.
- Construcción de modelos
NOTA: Después de determinar la fase por reemplazo molecular, el modelo debe ser construido y refinado. Este protocolo utiliza Phenix GUI49 para la construcción automática y refinamientos iterativos, y Coot50 para la construcción de estructura manual y el refinamiento.- Después del reemplazo molecular usando Phase-MR en Phoenix, seleccione Ejecutar Autobuild. Todos los archivos necesarios se añadirán automáticamente. Simplemente presione Ejecutar para iniciar la autoconstrucción.
- Al finalizar, compruebe el modelo en Coot. Construir y refinar el modelo manualmente según el mapa de densidad de electrones en Coot.
- Refinar el modelo curado manualmente en Phoenix (en la pestaña Refinamiento) utilizando el modelo, la secuencia y los datos de difracción como entradas. Consulte la ayuda de Phoenix para elegir la estrategia correcta.
- Después del refinamiento, compruebe los resultados: Eltrabajo sin R y R debe disminuir, los indicadores de Molprobity51 deben ser respetados y los valores atípicos con baja correlación en el espacio real deben ser limitados.
- Repita los pasos 5.3.2 a 5.3.4 hasta que se genere el mejor modelo refinado.
- Ejecute Molprobity en el servidor: http://molprobity.biochem.duke.edu/. Compruebe los valores atípicos identificados por Molprobity.
- Eventualmente repita los pasos 5.3.2 a 5.3.6 hasta obtener el mejor modelo refinado.
Resultados
Para estudiar una posible disociación dodecamer en monómeros en TmPep1050, los codones His-60 e His-307 fueron reemplazados por codón de alanina usando un gen sintético. Este gen fue clonado en vector pBAD para la expresión y purificación de la variante tmPep1050 correspondiente posteriormente denominada TmPep1050H60A H307A. La cromatografía de exclusión de tamaño (Figura 3B) mostró que la proteína purificada tenía un peso molecular aparente de 56 kDa (el peso molecular del monómero era de 36,0 kDa). Se ha notificado un peso molecular aparente similar, 52 kDa, para el dimer TMPep105011. Por lo tanto, el estado oligomérico de TmPep1050H60A H307A podría deducirse como dimerico. En cuanto a su actividad específica, TmPep1050H60A H307A fue completamente inactivo en L-Leu-pNA como sustrato, incluso en presencia de iones de cobalto. Este resultado sugiere fuertemente que las variantes no pueden unir ningún iones metálicos.
La condición de cristalización de TmPep1050H60A H307A se optimizó variando el pH frente a la concentración de PEG (Figura 4) alrededor de la condición del dimer (es decir, 0,1 M citrato de sodio pH 6,0 10% PEG3350). Los mejores cristales de TmPep1050H60A H307A se obtuvieron en 0,1 M citrato de sodio pH 5,2 20% PEG3350 con un ciclo de microsedrado para mejorar la monocristalinidad. Se recopiló un conjunto de datos completo en la línea de haz de Próxima 2 (sincrotrón SOLEIL) con una resolución de 2,36o (Tabla 1). La indexación de datos mostró que el grupo espacial del cristal TmPep1050H60A H307A es C2221, pero XDS propuso otra solución, el grupo espacial mP (ver Figura 5). Según Pointless, la probabilidad de grupos espaciales C2221 y P21 eran 0,711 y 0,149, respectivamente. Según el análisis de calidad de datos, dos monómeros se encuentran en la unidad asimétrica. El análisis de Xtriage reveló que el conjunto de datos probablemente está hermanado pero el hermanamiento en el grupo espacial C2221 es poco probable52. El hermanamiento resulta de la anomalía de crecimiento del cristal donde varios dominios definidos tienen algunas de sus direcciones de celosía paralelas entre sí53. El hermanamiento también puede ser el resultado de una simetría de cristal más alta, lo que indica una indexación de datos errónea. Por lo tanto, un gemelo pseudo-merohedral puede existir para que una celosía de cristal P21 se vea como un C2221. El conjunto de datos se indexó posteriormente en el grupo espacial P21 y se probó en reemplazo molecular. El análisis de Xtriage del conjunto de datos indexado en P21 reveló un gemelo pseudo-merohedral siguiendo una ley gemela h, -k, -h-l.
Utilizando las coordenadas de un monómero de dodecameric TmPep1050 (código PDB 4P6Y), se encontró una solución de reemplazo molecular para el conjunto de datos indexado en P21 solamente, con una puntuación TFZ de 28.9. Por lo tanto, los datos de difracción se trataron como un conjunto de datos hermanado para la creación de modelos. Para minimizar el sesgo del reemplazo molecular, se construyó un primer modelo mediante el uso de phenix.autobuild54,55. La estructura de TmPep1050H60A H307A se completó después de varios ciclos de refinamiento automatizado y manual en Phoenix y Coot(Tabla 1 y Figura 6A). La estructura confirmó el estado oligomérico con una superficie de interfaz de 1.710s2 entre ambos monómeros y uniG de -16,2 kcal mol-1 calculado por PDBe Pisa56. En comparación, la superficie de la interfaz yeli G de dimerico TmPep10502-mer es 1,673 á2 y -16,7 kcal mol-1, respectivamente.
La estructura de TmPep1050H60A H307A es muy similar a la estructura de dimer de tipo salvaje con RMS de 0.774 s al alineado. Es importante destacar que las mismas modificaciones estructurales se observan en ambas estructuras: alta flexibilidad de los hélices de 8 y 10, sitio activo desordenado Gln-196–Val-202, y el desplazamiento de Lys-229–Ala-235 y Lys-247–Ser 254. Estas modificaciones se correlacionaron previamente con el obstáculo de la formación dodecamer en ausencia de su cofactor metálico11. Las dos mutaciones de His-60 e His-307, sin embargo, tuvieron un ligero efecto en las cadenas laterales de Asp-168 y Asp-62. Parecían estar encerrados en una conformación diferente del dimer de tipo salvaje(Figura 6B). El carboxilato Asp-168 se gira 40o debido a la ausencia de His-60 e His-307. Por lo tanto, ambos residuos de histidina son importantes para colocar el carboxilato Asp-168 correctamente para salvar los dos iones metálicos. La cadena lateral Asp-62 está orientada hacia el carboxilato Glu-18, fuera del sitio catalítico. Asp-62 puede tener un papel importante en la catálisis, ya que se supone que modula elpK a de His-60 y, por lo tanto, influir en la unión de iones metálicos en el sitio M2. Además, podría estar implicado estructuralmente en la estabilización del sitio catalítico sobre la unión de iones metálicos, favoreciendo la formación del dodecamer.

Figura 1: Representación esquemática de la clonación de ORF TM_1050 en vector pBAD por recombinación homóloga.
El ORF está flanqueado por dos secuencias de 30 bp homólomas al extremo BAD promotor y el sitio de restricción PmeI aguas arriba de la secuencia. Haga clic aquí para ver una versión más grande de esta figura.
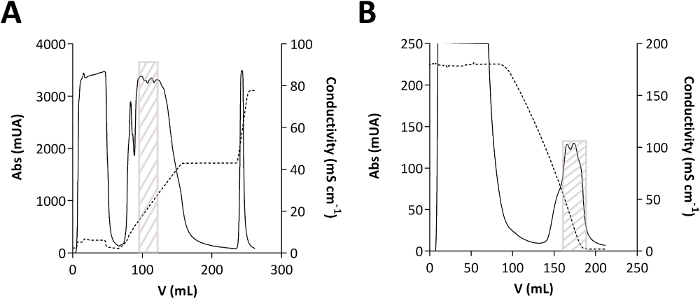
Figura 2: Cromatogramas de la purificación tmPep1050.
(A) Cromatografía de intercambio de aniones. (B) Cromatografía de interacción hidrófoba. La absorbancia (Abs), expresada en miliuniones de absorbancia (mUA), se muestra en línea plana. La conductividad, expresada en mS cm-1, se muestra en línea discontinua. La caja gris indica dónde elue en los cromatogramas tmPep1050. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Cromatografía de exclusión de tamaño de (A) tóp1050 dodecamer, (B) TmPep1050H60A H307Ay (C) TmPep1050H60A.
Las muestras se analizaron utilizando resina SEC empaquetada en una columna de 120 ml. La absorbancia (Abs) se expresa en miliuniones de absorbancia (mUA). (D) Calibración de la columna SEC utilizando tiroglobulina (T), ferritina (F), aldolasa (Ald), conalbumina (C) y albúmina (Alb) como estándares. La correlación entre el logaritmo de la masa relativa y el volumen de elución es lineal, con un R2 de 0.91. Los intervalos de confianza del 95% se representan como puntos. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Optimización de la cristalización TmPep1050H60A H307A.
(A) La primera estrategia de optimización consiste en variar el pH (entre 4,5 y 6,0) frente a la concentración de PEG3350 (entre el 5% y el 25%). La placa de cristalización está esquemática, y los pozos están codificados por colores: rojo para precipitado, amarillo para policristales y verde para monocristales. (B) La segunda estrategia de optimización incluye el uso de semillas diluidas 25x con una variación más estrecha de pH frente a PEG3350. (C) Forma y tamaño de cristal antes (imagen superior) y después (imagen inferior) optimización de cristalización y microsempresa. Haga clic aquí para ver una versión más grande de esta figura.

Figura 5: Extractos de la salida del registro CORRECTO. LP de la indexación de datos TmPep1050H60A H307A por XDS.
Panel superior, las posibles celosías Bravais, siendo las más probablemente mC, mP y OC. Panel medio, estadísticas generales de datos indexados en el grupo espacial C2221. Panel inferior, estadísticas generales de datos indexados en el grupo espacial P21. Haga clic aquí para ver una versión más grande de esta figura.

Figura 6: Estructura de TmPep1050H60A H307A.
(A) Alineación estructural de una subunidad TmPep1050H60A H307A (rojo, código PDB 5NE9) frente a una subunidad dodecamer (blanco, código PDB 6NW5) y una subunidad dimer (azul, código PDB 5NE6). Las flechas indican las diferencias estructurales entre dodecamers y dimers. (B) Primer plano del sitio activo TmPep1050H60A H307A (rojo) en comparación con el sitio activo de TmPep1050 dimer (azul) y dodecamer (blanco). Haga clic aquí para ver una versión más grande de esta figura.
| TmPep1050H60A H307A | |
| Recopilación de datos | |
| Temperatura (K) | 100 |
| Fuente de radiación | Axis proxima SOLEIL 2 |
| Longitud de onda (o) | 0.9801 |
| Detector | Dectris Eiger X 9M |
| Rango de oscilación (o) | 0.1 |
| Tiempo de exposición (s) | 0.025 |
| Grupo espacial | P 1 21 1 |
| Parámetros de celda de unidad | |
| α, β, γ (°) | 90.00, 110.69, 90.00 |
| a, b, c (o) | 43.24, 137.79, 61.11 |
| Resolución | 43.99 – 2.37 (2.52-2.37) |
| Reflejos únicos | 26.902 |
| Rmerge (%) | 0.14 |
| Redundancia | 6,815 |
| 8.64 (2.12) | |
| Integridad (%) | 99.6 (97.9) |
| CC1/2 (%) | 99.2 (84.1) |
| Refinamiento | |
| Resolución | 43.99 – 2.37 |
| Reflexiones | 26.9 |
| R conteo de pruebas de setlibre | 1345 |
| Rtrabajo/Rlibre | 0.206/0.234 |
| Moléculas de proteínas por ASU | 2 |
| VM (3/Da) | 2.37 |
| Contenido de disolvente (%) | 49.0 |
| Atomes de proteína/disolvente | 4,559/96 |
| longitudes de unión r.m.s.d. (o) | 0.31 |
| r.m.s.d. ángulos de unión (o) | 0.51 |
| Factores B promedio (n.o2) | 57.0 |
| Ramachandran φ/ψ favorecido/no permitido (%) | 95.02 / 0.17 |
| Ley gemela | h, -k, -h-l |
| Código PDB | 5NE9 |
Tabla 1: Estadísticas de recopilación y refinamiento de datos. Los valores entre paréntesis son para el shell de mayor resolución.
Discusión
El protocolo descrito aquí permite comprender la transición dimer-dodecamer de TmPep1050 a nivel estructural. La metodología fue experimentada anteriormente para determinar la estructura de ambos oligómeros TmPep105011. El paso más difícil fue encontrar condiciones que promuevan la disociación de los dodecamers en dimers estables. Tales condiciones tenían que ser lo suficientemente suaves como para permitir la reasociación de los dimers en los dodecamers cuando se añadió el cofactor de iones metálicos. La separación de los oligómeros también fue un paso crítico, ya que condiciona los estudios estructurales y una mayor caracterización bioquímica (por ejemplo, el estudio de la reasociación dodecamer en varias dosis de Co2+). El reemplazo molecular, un método probado para la determinación de fases, se utilizó para resolver las estructuras de los oligómeros TmPep1050 y sus variantes. El protocolo propuesto puede adaptarse para estudiar otras enzimas metalónmicas cuyos estados de oligomerización dependen de la disponibilidad de sus cofactores metálicos.
Para ilustrar el protocolo, se presentó un caso de estudio, TmPep1050H60A H307A cuyos sitios de unión de metales se vieron afectados por la mutación de His-60 y His-307 a alanina. Estos residuos unen Co2+ en los sitios M2 y M1, respectivamente. Interferir en la unión de metales podría haber perturbado el estado de oligomerización y llevado a una disociación completa en monómeros. Se han notificado evidencias de tal fenómeno para PhTET2 y PfTET3, dos Aminopeptidases M42 de P. horikoshii y P. furiosus,respectivamente24,29. TmPep1050H60A H307A no se comportó como se esperaba ya que esta variante formaba dimers solamente. Su estructura mostró las mismas modificaciones que el dimer de tipo salvaje, pero con dos pequeñas excepciones. De hecho, las cadenas laterales de Asp-168 y Asp-62 parecían estar bloqueadas en una orientación no convencional que impedía la estabilización del sitio activo. Su orientación parecía ser impuesta por His-60 y His-307 como tales modificaciones no fueron observadas en las variantes de mutación de un solo punto.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Agradecemos a Martine Roovers por revisar este documento y dar comentarios constructivos. El acceso a la línea de haz Próxima 2 (sincrotrón SOLEIL) estaba dentro de los grupos de asignación de bloques 20151139.
Materiales
| Name | Company | Catalog Number | Comments |
| 1,10-phenanthroline | Sigma-Aldrich | 13, 137-7 | |
| Amicon Ultra 0.5 ml Centrifugal Filters Ultracel 30K | Merck Millipore | UFC503096 | |
| Amicon Ultra 15 Centrifugal Filters Ultracel 30K | Merck Millipore | UFC903024 | |
| Benzonase Nuclease | Merck Millipore | 70664-3 | |
| CCP4 | N/A | visit http://www.ccp4.ac.uk/ | |
| cOmplete EDTA-free | Roche | 5056489001 | |
| Coot | N/A | visit https://www2.mrc-lmb.cam.ac.uk/personal/pemsley/coot/ | |
| Crystal Screen I | Hampton Research | HR2-110 | |
| Crystal Screen II | Hampton Research | HR2-112 | |
| DreamTaq Green PCR Master Mix | ThermoFisher Scientific | K1082 | |
| EasyXtal 15-well tool | NeXtal | 132007 | |
| Escherichia coli PPY strain | N/A | see reference 31 | |
| Escherichia coli XL1 blue strain | Agilent | 200249 | |
| Gel Filtration Calibration Kit HMW | GE Healthcare Life Sciences | 28-4038-42 | |
| Gel Filtration Calibration Kit LMW | GE Healthcare Life Sciences | 28-4038-41 | |
| Gel Filtration Standard | Biorad | 1511901 | |
| GeneJET Plasmid Miniprep Kit | ThermoFisher Scientific | K0503 | |
| Index | Hampton Research | HR2-144 | |
| Litholoops | Molecular Dimensions | ||
| L-leucine-p-nitroanilide | Bachem AG | 40010720025 | |
| Natrix 1 | Hampton Research | HR2-116 | |
| Natrix 2 | Hampton Research | HR2-117 | |
| Neggia plugin | Dectris | N/A | visit https://www.dectris.com/ |
| NeXtal Tubes JCSG Core Suite I | NeXtal | 130724 | |
| NeXtal Tubes JCSG Core Suite II | NeXtal | 130725 | |
| NeXtal Tubes JCSG Core Suite III | NeXtal | 130726 | |
| NeXtal Tubes JCSG Core Suite IV | NeXtal | 130727 | |
| pBAD-TOPO | ThermoFisher Scientific | K430001 | |
| Phenix | N/A | visit https://www.phenix-online.org/ | |
| Phusion High-Fidelity DNA polymerase | ThermoFisher Scientific | F-530L | |
| Salt RX 1 | Hampton Research | HR2-107 | |
| Salt RX 2 | Hampton Research | HR2-109 | |
| SnakeSkin Dialysis Tubing, 3.5K MWCO | ThermoFisher Scientific | 88242 | |
| Source 15Phe | GE Healthcare Life Sciences | 17014702 | |
| Source 15Q | GE Healthcare Life Sciences | 17094705 | |
| Superdex 200 prep grade | GE Healthcare Life Sciences | 17104301 | |
| Thermotoga maritima MSB8 strain | American Type Culture Collection | ATCC 43589 | |
| TmCD00089984 | DNASU Plasmid Repository | N/A | |
| XDS | N/A | visit http://xds.mpimf-heidelberg.mpg.de/ | |
| xdsme | N/A | visit https://github.com/legrandp/xdsme |
Referencias
- Levy, E. D., Teichmann, S. A. Structural, Evolutionary, and Assembly Principles of Protein Oligomerization. Progress in Molecular Biology and Translational Science. 117, 25-51 (2013).
- Selwood, T., Jaffe, E. K. Dynamic dissociating homo-oligomers and the control of protein function. Archives of Biochemistry and Biophysics. 519 (2), 131-143 (2012).
- Jaffe, E. K. The Remarkable Character of Porphobilinogen Synthase. Accounts of Chemical Research. 49 (11), 2509-2517 (2016).
- Ramström, H., et al. Properties and Regulation of the Bifunctional Enzyme HPr Kinase/Phosphatase in Bacillus subtilis. Journal of Biological Chemistry. 278 (2), 1174-1185 (2003).
- Rudyak, S. G., Brenowitz, M., Shrader, T. E. Mg2+-Linked Oligomerization Modulates the Catalytic Activiy of the Lon (La) Protease from Mycobacterium smegmatis. Biochemistry. 40 (31), 9317-9323 (2001).
- Yamamoto, S., Storey, K. B. Dissociation-Association of lactate dehydrogenase Isozymes: Influences on the formation of tetramers vs. dimers of M4-LDH and H4-LDH. International Journal of Biochemistry. 20 (11), 1261-1265 (1988).
- Sirover, M. A. Structural analysis of glyceraldehyde-3-phosphate dehydrogenase functional diversity. The International Journal of Biochemistry & Cell Biology. 57, 20-26 (2014).
- Gupta, V., Bamezai, R. N. K. Human pyruvate kinase M2: A multifunctional protein: Multifunctional Human PKM2. Protein Science. 19 (11), 2031-2044 (2010).
- Wiegand, G., Remington, S. J. Citrate synthase: Structure, Control, and Mechanism. Annual Review of Biophysics and Biophysical Chemistry. 15, 97-117 (1986).
- Libonati, M., Gotte, G. Oligomerization of bovine ribonuclease A: structural and functional features of its multimers. Biochemical Journal. 380 (2), 311-327 (2004).
- Dutoit, R., et al. How metal cofactors drive dimer-dodecamer transition of the M42 aminopeptidase TmPep1050 of Thermotoga maritima. Journal of Biological Chemistry. 294 (47), 17777-17789 (2019).
- Rawlings, N. D., et al. The MEROPS database of proteolytic enzymes, their substrates and inhibitors in 2017 and a comparison with peptidases in the PANTHER database. Nucleic Acids Research. 46 (1), 624-632 (2018).
- Neuwald, A. F., Liu, J. S., Lipman, D. J., Lawrence, C. E. Extracting protein alignment models from the sequence database. Nucleic Acids Research. 25 (9), 1665-1677 (1997).
- Dutoit, R., Brandt, N., Legrain, C., Bauvois, C. Functional Characterization of Two M42 Aminopeptidases Erroneously Annotated as Cellulases. PLoS ONE. 7 (11), 50639(2012).
- Franzetti, B., et al. Tetrahedral aminopeptidase: a novel large protease complex from archaea. The EMBO Journal. 21 (9), 2132-2138 (2002).
- Borissenko, L., Groll, M. Crystal Structure of TET Protease Reveals Complementary Protein Degradation Pathways in Prokaryotes. Journal of Molecular Biology. 346 (5), 1207-1219 (2005).
- Appolaire, A., et al. TET peptidases: A family of tetrahedral complexes conserved in prokaryotes. Biochimie. 122, 188-196 (2016).
- Russo, S., Baumann, U. Crystal Structure of a Dodecameric Tetrahedral-shaped Aminopeptidase. Journal of Biological Chemistry. 279 (49), 51275-51281 (2004).
- Schoehn, G., et al. An Archaeal Peptidase Assembles into Two Different Quaternary Structures: A tetrahedron and a giant octahedron. Journal of Biological Chemistry. 281 (47), 36327-36337 (2006).
- Durá, M. A., et al. The structural and biochemical characterizations of a novel TET peptidase complex from Pyrococcus horikoshii reveal an integrated peptide degradation system in hyperthermophilic Archaea: Characterization of P. horikoshii TET3 peptidase. Molecular Microbiology. 72 (1), 26-40 (2009).
- Basbous, H., Appolaire, A., Girard, E., Franzetti, B. Characterization of a Glycyl-Specific TET Aminopeptidase Complex from Pyrococcus horikoshii. Journal of Bacteriology. 200 (17), 00059(2018).
- Appolaire, A., et al. Small-angle neutron scattering reveals the assembly mode and oligomeric architecture of TET, a large, dodecameric aminopeptidase. Acta Crystallographica Section D Biological Crystallography. 70 (11), 2983-2993 (2014).
- Appolaire, A., et al. The TET2 and TET3 aminopeptidases from P yrococcus horikoshii form a hetero-subunit peptidasome with enhanced peptide destruction properties: TET aminopeptidase multi-subunit complex. Molecular Microbiology. 94 (4), 803-814 (2014).
- Colombo, M., Girard, E., Franzetti, B. Tuned by metals: the TET peptidase activity is controlled by 3 metal binding sites. Scientific Reports. 6 (1), 20876(2016).
- Petrova, T. E., et al. Structure of the dodecamer of the aminopeptidase APDkam598 from the archaeon Desulfurococcus kamchatkensis. Acta Crystallographica Section F Structural Biology Communications. 71 (3), 277-285 (2015).
- Kim, D., et al. Structural basis for the substrate specificity of PepA from Streptococcus pneumoniae, a dodecameric tetrahedral protease. Biochemical and Biophysical Research Communications. 391 (1), 431-436 (2010).
- Chevrier, B., et al. Crystal structure of Aeromonas proteolytica aminopeptidase: a prototypical member of the co-catalytic zinc enzyme family. Structure. 2, 283-291 (1994).
- Rosenbaum, E., Ferruit, M., Durá, M. A., Franzetti, B. Studies on the parameters controlling the stability of the TET peptidase superstructure from Pyrococcus horikoshii revealed a crucial role of pH and catalytic metals in the oligomerization process. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1814 (10), 1289-1294 (2011).
- Macek, P., et al. Unraveling self-assembly pathways of the 468-kDa proteolytic machine TET2. Science Advances. 3 (4), 1601601(2017).
- Edelheit, O., Hanukoglu, A., Hanukoglu, I. Simple and efficient site-directed mutagenesis using two single-primer reactions in parallel to generate mutants for protein structure-function studies. BMC Biotechnology. 9 (1), 61(2009).
- Zhang, Y., Werling, U., Edelmann, W. SLiCE: a novel bacterial cell extract-based DNA cloning method. Nucleic Acids Research. 40 (8), 55(2012).
- Schleif, R. AraC protein, regulation of the l-arabinose operon in Escherichia coli, and the light switch mechanism of AraC action. FEMS Microbiology Reviews. 34 (5), 779-796 (2010).
- McPherson, A., Gavira, J. A. Introduction to protein crystallization. Acta Crystallographica Section F Structural Biology Communications. 70 (1), 2-20 (2014).
- Bergfors, T. Seeds to crystals. Journal of Structural Biology. 142 (1), 66-76 (2003).
- Dauter, Z. Collection of X-Ray Diffraction Data from Macromolecular Crystals. Protein Crystallography. 1607, 165-184 (2017).
- Powell, H. R. X-ray data processing. Bioscience Reports. 37 (5), 0227(2017).
- Battye, T. G. G., Kontogiannis, L., Johnson, O., Powell, H. R., Leslie, A. G. W. iMOSFLM a new graphical interface for diffraction-image processing with MOSFLM. Acta Crystallographica Section D Biological Crystallography. 67 (4), 271-281 (2011).
- Otwinowski, Z., Minor, W. Processing of X-ray diffraction data collected in oscillation mode. Methods in Enzymology. 276, 307-326 (1997).
- Clabbers, M. T. B., Gruene, T., Parkhurst, J. M., Abrahams, J. P., Waterman, D. G. Electron diffraction data processing with DIALS. Acta Crystallographica Section D Structural Biology. 74 (6), 506-518 (2018).
- Kabsch, W. XDS. Acta Crystallographica Section D Biological Crystallography. 66 (2), 125-132 (2010).
- Legrand, P. legrandp/xdsme: March 2019 version working with the latest XDS version. , Jan 26, 2018. Zenodo (2019).
- Evans, P. Scaling and assessment of data quality. Acta Crystallographica Section D Biological Crystallography. 62 (1), 72-82 (2006).
- Wlodawer, A., Minor, W., Dauter, Z., Jaskolski, M. Protein crystallography for non-crystallographers, or how to get the best (but not more) from published macromolecular structures: Protein crystallography for non-crystallographers. FEBS Journal. 275 (1), 1-21 (2008).
- Karplus, P. A., Diederichs, K. Assessing and maximizing data quality in macromolecular crystallography. Current Opinion in Structural Biology. 34, 60-68 (2015).
- Taylor, G. L. Introduction to phasing. Acta Crystallographica Section D Biological Crystallography. 66 (4), 325-338 (2010).
- Rossmann, M. G., Blow, D. M. The detection of sub-units within the crystallographic asymmetric unit. Acta Crystallographica. 15, 24-31 (1962).
- Rossmann, M. G. The molecular replacement method. Acta Crystallographica Section A Foundations of Crystallography. 46 (2), 73-82 (1990).
- McCoy, A. J., et al. Phaser crystallographic software. Journal of Applied Crystallography. 40 (4), 658-674 (2007).
- Adams, P. D., et al. PHENIX: a comprehensive Python-based system for macromolecular structure solution. Acta Crystallographica Section D Biological Crystallography. 66 (2), 213-221 (2010).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Crystallographica Section D Biological Crystallography. 66 (4), 486-501 (2010).
- Chen, V. B., et al. MolProbity: all-atom structure validation for macromolecular crystallography. Acta Crystallographica Section D Biological Crystallography. 66 (1), 12-21 (2010).
- Zwart, P. H., Grosse-Kunstleve, R. W., Lebedev, A. A., Murshudov, G. N., Adams, P. D. Surprises and pitfalls arising from (pseudo)symmetry. Acta Crystallographica Section D Biological Crystallography. 64 (1), 99-107 (2008).
- Yeates, T. O. Detecting and overcoming crystal twinning. Methods in Enzymology. 276, 344-358 (1997).
- Terwilliger, T. C. Using prime-and-switch phasing to reduce model bias in molecular replacement. Acta Crystallographica Section D Biological Crystallography. 60 (12), 2144-2149 (2004).
- Terwilliger, T. C., et al. Iterative model building, structure refinement and density modification with the PHENIX AutoBuild wizard. Acta Crystallographica Section D Biological Crystallography. 64 (1), 61-69 (2008).
- Krissinel, E., Henrick, K. Inference of Macromolecular Assemblies from Crystalline State. Journal of Molecular Biology. 372 (3), 774-797 (2007).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados