Method Article
X線結晶学は 、熱利子マリティマ M42アミノペプチダーゼTmPepT050のオリゴマー状態推移を研究する
要約
このプロトコルは、構造レベルでTmPep1050、M42アミノペプチダーゼのダイマー-ドデカマー転移を研究するために開発されました。タンパク質精製からX線データ処理まで、簡単なパイプラインです。結晶生成、データセットインデックス化、および分子置換は、研究の場合、TmPep1050H60A H307A 変異体を通じて強調されてきた。
要約
M42アミノペプチダーゼは、12個のサブユニットからなる機能的に活性な複合体を形成する。彼らの組み立てプロセスは、二量体ドーデカメア転移を引き起こす金属イオン補因子によって調節されているようだ。金属イオン結合の際に、アクティブ部位および相互作用界面で、自己集合を促進するためにダイマーを形成するいくつかの構造修飾が起こる。このような修飾を観察するには、構造研究の前に安定したオリゴマーを単離する必要があります。ここで報告されているのは、TmPep1050の安定したドーデカマーおよびダイマーの精製 、T.マリティマのM42アミノペプチダーゼ、およびX線結晶学によるそれらの構造決定を可能にする方法である。ダイマーは、キレート剤で金属イオンを除去することによってドーデカマーから調製した。彼らの補因子がなければ、ドデカメルは安定性が低くなり、加熱時に完全に解解化された。オリゴマー構造は、単純な分子置換アプローチによって解明された。方法論を例示するために、金属イオン結合において完全に障害を受けたTmPep1050変異体の構造は、単量体に対するダイマーのさらなる分解を示さない提示される。
概要
オリゴマー化は、多くのタンパク質の生物学的機能を決定する主要なプロセスです。大腸菌では、タンパク質の35%だけが単量体1であると推定されている。モルフェインと呼ばれるいくつかのタンパク質は、各オリゴマー状態2に明確な構造を有するサブユニットを有するいくつかのオリゴマー状態を採用することさえできる。それらのオリゴマー状態間の移行は、各オリゴマー状態が異なる特異的活性または機能を有し得るタンパク質活性を調節する意味であることが多い。モルフェインのいくつかの例は、文献、特にポルフォビリノーゲン合成酵素3、HPrキナーゼ/ホスファターゼ4、ロンプロテアーゼ5、乳酸脱水素酵素6、グリセアルデヒド-3-リン酸脱水素酵素7、ピルビン酸キナーゼ8、クエン酸シターゼ9、およびリブコンA10において十分に文書化されている。最近、我々は、M42アミノペプチダーゼTmPep1050、モルフェイン様な挙動を有する酵素の別の例を説明し、その活性はそのオリゴマー状態11に依存する。そのオリゴマー状態間の遷移は、サブユニットのいくつかの構造修飾を誘発する金属補因子によって媒介される。
M42アミノペプチダーゼファミリーはMH族12、13に属し、細菌と古細菌14の間で広く分布している。M42アミノペプチダーゼは、長さ15のアミノ酸残基を最大35個までペプチドを分解する本物の二核酵素である。彼らは、内側の空洞に向けられたアクティブな部位を持つ12のサブユニットで作られた独特の四面体型の構造を採用しています。このような配置は、制御されていないタンパク質分解を避けるために活性のナノ区画化としてしばしば記述される。M42アミノペプチダーゼの生理機能は、プロテアソームに関連付けてもよいし、タンパク質分解から生じるペプチドを加水分解する16、17。ピロコッカス堀越は4つのM42アミノペプチダーゼを持ち、それぞれが18、19、20、21の別個の、しかし相補的な特異性を提示する。単独では、2種類のサブユニットからなるヘテロ複合体がP.堀越しに記載されており、ペプチダソーム複合体22,23の存在を示唆している。
M42アミノペプチダーゼのいくつかの構造は、文献11、16、18、19、20、24、25、26に記載されている。サブユニットは、触媒ドメインと二量体化ドメインの2つの異なるドメインで構成されています。触媒ドメインは、MH族全体に保存された共通のα/βフォールドを採用し、ビブリオプロテオリチクス27のアミノペプチダーゼAp1である原型触媒ドメインである。二量体化ドメインはPDZ様の折り目16を採用し、オリゴマー化におけるその役割に加えて、内腔11における基板アクセスおよび結合を制御する役割を有し得る。基本的なビルディングブロックがダイマーであるため、ドーデカマーは、6つのダイマーの関連としてしばしば記述され、各ダイマーは四面体16の各縁に位置付けられている。M42アミノペプチダーゼのオリゴマー化は、その金属補因子の可用性に依存しています.二価金属イオンは、しばしばZn2+およびCo2+、ペプチド結合および加水分解に触媒的に関与している。これらは、M1 サイトと M2 サイトという 2 つの異なるバインド サイトに存在します。また、この2つの金属イオンは、PhTET2、PhTET3、PfTET3、およびTmPep105011、24、28、29に対して実証されているように、オリゴマー化を駆動し、細かく調整する。金属補因子が枯渇すると、ドーデカマーは、PhTET2、PhTET3、およびTmPep105011、16、28、あるいはPhTET2およびPfTET324、29のような単量体のようにダイマーに分解する。
ここで提示されるプロトコルは、TmPep1050オリゴマーの構造を研究するために使用されます。このプロトコルは、タンパク質精製、タンパク質分解活性スクリーニング、結晶化、X線回折、分子置換などの一般的な方法のセットです。メタロ酵素、タンパク質オリゴマー化、タンパク質の結晶化、分子置換に固有の機微が強調されています。研究のケースはまた、TmPep1050ドーデカマーがモノマーにさらに解離することができるかどうかを示すために提示されています。この質問に対処するために、TmPep1050変異体であるTmPep1050H60A H307Aは、His-60(M2部位)およびHis-307(M1部位)をアラ残基に変異させることによって金属結合部位が損なわれる研究を行った。このプロトコルは、他のM42アミノペプチダーゼまたはモルフェイン様の挙動を有する任意のメタロ酵素を研究するために収容されてもよい。
プロトコル
1. 組換えTmPep1050の製造と精製
注:以下、以前の研究11から適応した野生型TmPep1050のクローニング手順と精製について説明する。あるいは、クローニングは、合成遺伝子を用いて行うことができる。TmPep1050変異体を生成するために、部位指向変異誘発は、例えば、単一プライマー反応を並列プロトコル(SPRINP)法30で以下に行うことができる。精製プロトコルは、TmPep1050の変異体に使用することができます。Hisタグの使用は、金属イオン結合を妨げるため避けるべきである。
- 式ベクトル設計
- サーモトガ・マリティマMSB8(ATCC 43589)またはTmCD00089984(構造ゲノム研究共同センター)のゲノムDNAを取得する。
- ゲノムDNAまたはテンプレートプラスミドのいずれかを使用して、TM_1050開き読み取りフレーム(ORF)を増幅し、 高忠実度のDNAポリメラーゼと次のプライマー:ocej419(5'-TTTAACTTTATTAAGAAGATATCATGAAGAGCTG)およびocej420(5'-ATCCGCCAAACAGCCACAAAGCTGGAGACCGTクトッタクトCGGCGCGCGCACCACCACCポリメラーゼ連鎖反応(PCR)は、95°Cで5分、30サイクル(95°Cで30s、55°Cで30s、72°Cで90s)、最終ステップとして72°Cで10分の3回の方法で行います。
- SLiCEプロトコル31に従って大腸菌中の相同組換え(図1)により、PCRフラグメントを適切な発現ベクター(材料表)にクローン化する。50 ngのリニア化ベクターに、PCRフラグメントをベクターに対するフラグメントの10:1モル比、PPY株抽出物の1μL、50 mM Tris-HCl pH 7.5、100 mM MgCl2、10mM ATP、および10 mMジチオトライト(DTT)を10μL°Cで10μLの反応量に加える。
- 化学に有能 な大腸菌 XL1ブルー株(または任意の recA- 適切な株)を1 μLの再結合反応で変換します。100 μg/mL アンピシリンを含む LB 培地に細胞を入火します。プレートを一晩37°Cでインキュベートする。
- 100 μg/mL アンピシリンを使用した新鮮な LB プレートにコロニーを選びます。プレートを37°Cで少なくとも8時間インキュベートします。
- 適切なプライマーペアを用いたコロニーPCRによる陽性候補のスクリーニング(5'-ATGCCATAGTTTTATCCおよび5'-ATTTAATCTGTATCAggC)が 材料表に記載されている推奨ベクターを使用する場合)。マイクロチップエンドで、摘んだコロニーをスクラッチし、各プライマーの0.5 μMと市販のTaq DNAポリメラーゼミックスの10 μLを含む反応ミックスの20 μLに細胞を移します。
- 変性工程として95°Cで5分、3ステップ(30回95°Cで30、55°Cで30s、72°Cで90s)、最終ステップとして72°Cで10分のPCRスクリーニングを実行します。
注:PCR反応は12°CのPCRマシンに一晩保存することができます。 - トリス-酢酸-EDTA(TAE)バッファーで調製した0.8%アガロースゲルに各PCR反応の10 μLをロードします。電気泳動を100Vで25分間実行します。
注: 1.1 kbp アンプリコンが必要です。 - 市販キット(材料表)を使用して候補からプラスミドを抽出し、ステップ1.1.6で使用したのと同じプライマーペアを使用してそれらをシーケンスします。
- 細胞培養
注: 適切な候補がシーケンスによって識別された場合、推奨ベクター (表/材料) を使用する場合、クローンを式として直接使用できます。その場合、その発現はアラビノース誘導性PBAD プロモーター32によって制御される。- 候補と100 μg/mLアンピシリンを含むLB培地を10mL接種し、軌道揺れの下で37°Cで一晩前培養をインキュベートする。100 μg/mL アンピシリンを用いてLB培地1 Lに5mLの前培養を加えます。空気と液体の比を3に尊重するように気をつけてください。
- 細胞は軌道揺れの下で37°Cで成長させる。光密度を660 nm(OD660)で監視します。
- OD660 が0.5-0.6に達したら、氷上で5分間培養液を急速に冷やし、18°Cに設定したインキュベーターに移します。
- 0.2 g/L アラビノースを加えて遺伝子発現を誘導し、18°C で 12-18 時間インキュベートします。
- 培養物を遠心分離して4°Cで30分間30分間培養して細胞を収穫する。 上清を捨て、100 mLの0.9%(w/v)NaClで細胞を洗浄します。
- 遠心分離機は6,000xgで再び4°Cで15分間、上清を捨てます。
注:細胞ペレットは、タンパク質抽出に直接使用するか、-80°Cで保存することができます。
- タンパク質精製
- 50 mM MOPS、1 mM CoCl2、pH7.2 の 40 mL で細胞ペレットを再懸濁します。25 U/μL DNA/RNAエンドヌクレアーゼの1 μLと、EDTAを含まないプロテアーゼ阻害剤カクテルの1錠を加えます。30分間冷却下のパルスモードでサスペンションを超音波処理します。
- 粗エキスを20,000xgで4°Cで30分間遠心分離する。 上清を集め、70°Cの水浴で10分間加熱します。
- 変性細胞抽出物を4°Cで30分間20,000xgで遠心分離し、清浄のために上清を回収する。
- 容積の15mLのコラムに詰め、適当なアニオン交換樹脂(材料表)を使用してください。作業流量とカラム圧力制限については、メーカーの推奨事項を参照してください。樹脂を50 mM MOPS、1 mM CoCl2、pH7.2と平衡化します。
- ステップ 1.3.3 から収集した上清を列にロードします。溶出水の吸光度を280nmで監視します。ベースラインに達したら、溶出に進みます。
- 50 mM MOPS、1 mM CoCl 2、pH7.2の 5 つのカラム ボリューム (CV) に 0 ~ 0.5 M NaCl を適用します。導電率が安定し、吸光度がベースラインに達するまで待ちます。
- 50 mM MOPS、1 mM CoCl 2、pH7.2で 0.5 から 1 M NaCl までの最終勾配を 1 CV に適用します。
- ドデシル硫酸ポリアクリルアミドゲル電気泳動(SDS-PAGE)によるいくつかの画分(ガイダンスについては 図2A を参照)を分析します。
注:TmPep1050は、クマシー染色後に36 kDaバンドとして表示されます。あるいは、TmPep1050の存在は、活性アッセイ(セクション2.1を参照)によって確認することができる。この工程では、分画を一晩4°Cで保存することができます。 - TmPep1050を含む分数をプールし、(NH4)2SO4の細かく粉砕粉末を加え、1.5M(NH4)2SO4の濃度を得る。完全に溶解するまでチューブを逆さまに反転して、穏やかに混ぜます。
- 体積の30mLのカラムに詰め込んだ疎水性相互作用樹脂(材料表)を使用してください。作業流量とカラム圧力制限については、メーカーの推奨事項を参照してください。樹脂を50 mM MOPS、1.5 M(NH4)2SO4、1mM CoCl2、pH7.2と平衡化します。
- サンプルをカラムに積み込み、280 nmで溶出の吸光度を監視します。吸光度がベースラインに達すると、50mM MOPSに1.5Mから0M(NH4)2SO4の勾配を適用することにより、結合タンパク質を溶出し、1mMCoCl2、pH7.2を5CVに適用する。
- SDS-PAGE を使用して、いくつかの分数を分析します (ガイダンスについては 図 2B を参照してください)。
注:TmPep1050は、クマシー染色後に36 kDaバンドとして表示されます。あるいは、TmPep1050の存在は、活性アッセイ(セクション2.1を参照)によって確認することができる。この工程では、分画を一晩4°Cで保存することができます。 - TmPep1050を含む画分をプールし、30 kDaカットオフ(材料表)を備えた限外濾過ユニットを使用して2mLに濃縮する。1.4項に進み、分子量を決定する。
- サイズ除外クロマトグラフィー
- ●体積の120mL~120mLの列に詰め込まれたサイズ排除樹脂(材料表)を使用してください。作業流量とカラム圧力制限については、メーカーの推奨事項を参照してください。樹脂を50 mM MOPS、0.5 M(NH4)2SO4、1mM CoCl2、pH7.2と平衡化します。
- サンプルをカラムに積み込み、280 nmで溶出の吸光度を監視します。カラムデッドボリューム(〜0.33CV)から溶出の終わり(1CV)までの分画。
- 観測された各ピークの溶出量を測定します。
注:ガイダンスについては、ドデカメリックTmPep1050は現在の実験条件下で〜82 mL(図3A)でエルチし、TmPep1050のようなダイメリックTmPep1050は、TmPep1050H60A H307Aバリアント、〜95 mLで溶出する(図3B)。TmPep1050の中には、TmPep1050H60Aのようなオリゴマー形態の両方を採用するものもあります (図3C)。 - SDS-PAGEを使用して、観測されたピークの最大値と尾に対応する分数を分析します。
注:TmPep1050は、クマシー染色後に36 kDaバンドとして表示されます。 - 各ピークの分率をプールし、30 kDaカットオフ(材料表)を使用して除水ユニットを使用して濃縮し、〜300μMの濃度を得ます。
- ナノ体積分光光度計で280nmの吸光度を測定し、18,910 M-1 cm-1の分子吸光係数を用いて濃度を計算します。
- 精製したタンパク質を-18°Cで保存します。
- 分子量を決定するには、分子量基準を用いてサイズ排除クロマトグラフィー(SEC)カラムを較正する(材料表)。50 mM MOPS、0.5 M (NH4)2SO4、1mM CoCl 2 pH7.2を実行バッファとして使用して、標準を分析します。
2. 活性アッセイとアポ酵素製剤
注:もともと、アポ酵素は、TmPep1050の1体積を10体積の2.1 Mリンゴ酸pH 7.0で希釈し、透析11の前に1体積に濃縮して調製した。以下に、金属イオンキレート体である1,10フェナントロリンを用いた代替手順を提示する。この手順は、タンパク質の損失を減少させ、以前に公開された方法と同じ結果を与えます。
- 活動アッセイ
- メタノール中の100 mM L-ロイシン-p-ニトロアニリド(材料表)のストック溶液を調製する。
- 100 mM L-ロイシン-p-ニトロアニリドを50 mM MOPSの965 μL、250 μM CoCl2、pH7.2、10%メタノールの25 μLを加えます。乾燥浴中に75°Cで反応ミックスをプレインキュベートします。
- 酵素を50 mM MOPS pH 7.2で1μMの濃度に希釈し、反応ミックスに10μLを加え、渦を加え、黄色が出るまで75°Cでインキュベートします。
- 20%酢酸の1 mLを加えて反応を停止する。渦をよくして、室温まで冷やします。
- 分光光度計細胞内で反応ミックスを移動します。負のコントロール(酵素を含まないインキュベート反応ミックス)に対して410 nmの吸光度を読み取る。
- アポ酵素製剤
- エタノールに1M 1,10フェナントロリンのストック溶液を調製する。1,10-フェナントロリンストック溶液の10 μLを50 mM MOPS、0.5 M(NH4)2SO4、pH7.2の890 μLに加えます。精製されたTmPep1050(300 μM-1 mM濃度)を100 μL加えます。
- 反応ミックスにCoCl2 を添加することなく、セクション2.1に記載の活性アッセイを用いて活性損失を確認する。
- 透析管内でサンプルを移します。200 mL の 50 mM MOPS に対する透析、 0.5 M (NH4)2SO4、 pH 7.2 (4 °C) 48時間の透析中に新鮮なバッファーと透析液を交換します。
- 透析管からサンプルを収集し、30 kDaカットオフ(材料表)を備えた除水ユニットを使用して100 μLに戻す。ナノ体積分光光度計を用いて280nmの吸光度を読み取って濃度を確認します。
- ダイマー調製物
- アポ酵素を50 mM MOPS、0.5 M(NH4)2SO4、pH7.2で1μMの濃度に希釈します。乾燥浴中に75°Cで2時間インキュベートし、その後、試料を室温まで冷まします。
- サンプルを少なくとも50μMの酵素濃度に濃縮し、SEC(1.4項参照)で分子量を確認します。溶出ピークは、約82mLから~95mL(現在の実験条件下)にシフトする必要があります。
3. TmPep1050結晶化
注意:タンパク質の結晶化は、多因子現象である33の経験科学のままです。いくつかのパラメータは特定および制御することができますが(温度、pH、沈殿剤濃度など)、他のパラメータは結晶化(タンパク質および化学的純度、タンパク質分解、サンプル履歴など)に影響を与える可能性があります。現在では、タンパク質の結晶化は、商業的な結晶化スクリーニング条件と自動化の束のおかげで合理的かつ体系的な方法で取り組まれています。しかし、結晶化条件の最適化は、主に試行錯誤のアプローチに依存します。以下、結晶化条件を最適化するためのタンパク質の結晶化のための青写真といくつかのヒントについて説明する。
- 結晶化スクリーニング
注:市販の結晶化キットを使用して、ドデカメリックTmPep1050の結晶は、2.2 M DL-リンゴ酸pH 7.0、0.1 MビストリプロパンpH 7.0および0.18 Mトリアンモニウムクエン酸、20%ポリエチレングリコール(PEG)3350で得られています。二量体TmPep1050の結晶は、0.1 Mクエン酸ナトリウムpH 5.6、0.2 M酢酸アンモニウム、30%PEG4000で得られている。ドデカメの結晶は1週間以内に現れ、1ヶ月でフルサイズに達する。ダイマーの結晶は、通常24時間以内に現れ、1週間でフルサイズに成長します。- いくつかの商業結晶化キットを入手します(例については 、資料表 を参照)。
- 吊り下げ方法の結晶板(材料表)を設定します。結晶化スクリーニングキットの各溶液の500 μLでウェルを充填します。
- 各井戸に対して、結晶化サポートを設定します。支持体に、精製タンパク質の1μL滴(通常〜10mg/mL)を沈着させます。
- すぐにウェルから結晶化溶液の1 μLをパイプ。タンパク質のドロップに慎重に追加し、逆さまに3回ピペットすることによって穏やかに混ぜます。ドロップは、泡なしで半球形のままでなければなりません。
- 対応するウェルの上にサポートをねじ込みます。キット全体に対して操作を繰り返します。
- プレートを設定した後、各ドロップを双眼鏡で観察します。解釈のための結晶化キットのユーザーガイド(明確な低下、相分離、沈殿、針など)を参照してください。
- プレートを20°Cでインキュベートする。 最初の週に1日1回、その後週に1回プレートをチェックしてください。
- スコアシートと結晶化キットを提供するユーザーガイドを使用して、各ウェルをスコア。
- 結晶化の最適化
注:ドデカメリックTmPep1050の初期結晶化条件は、2.1 M DL-リンゴ酸pH 6.75および0.18 Mクエン酸三アンモニウムpH 7.5に最適化されています。 40%(w/v) PEG3350 ジメリック TmPep1050 の結晶化条件が 0.1 M クエン酸ナトリウム pH 6.0, 10% (w/v) PEG3350.結晶性を改善するために、1サイクルの播種が必要であった。以下、TmPep1050H60A H307A 変異体の結晶化がどのように最適化されたかについて説明する。- 異なるpH(4.5、5.2、および6.0)で0.5 Mクエン酸ナトリウムバッファーのストック溶液を準備し、50%(w/v)PEG3350溶液を調製します。
- 結晶化プレートをpH対沈殿剤のマトリックスとして設定します( 図4Aを参照)。
- プレートを20°Cでインキュベートする。 1週間に1日1回双眼鏡で各井戸を観察します。
- 結晶の大きさと形状に応じて各ウェルをスコア。少なくとも50 μmの結晶を与える条件を選択してください。マイクロシードに進みます。
- マイクロシード
注:マイクロシードは、タンパク質結晶34の形状、サイズ、結晶性を改善するための強力な方法です。より速い播種アプローチは、猫のウィスカーを使用したストリークシードです。結晶化の最適化とマイクロシードがTmPep1050H60A H307Aの結晶形状とサイズをどのように改善したかを例示して図4を参照してください。- ステップ3.2.4で選択した井戸を使用して種子を準備します。
- ウェルから結晶化液を添加して、結晶を含むドロップの体積を10μLに上げます。滴をピペットし、井戸から90 μLの結晶化溶液を加えます。徹底的に渦を、氷の上に種子を保ちます。
- 種子のいくつかの希釈を準備します:1x、10x、25x、および100x。ピペット化する前によく種子をボルテックス。希釈液を氷の上に置いておきなさい。
- 各種子希釈について、pH対沈殿剤のマトリックスとして結晶化板を設定する( 図4Bを参照)。ステップ 3.2.1 で準備したストックソリューションを使用します。
- ドロップを行う場合は、0.2 μLのシードを加えて2 μLのドロップを行います。プレートを20°Cでインキュベートする。 1週間に1日1回双眼鏡で各井戸を観察します。
注:結晶サイズの分布と形状を改善する必要があります、TmPep1050H60A H307Aの例として図4Cを参照してください。さらなる使用のために、種子は-20 °Cで貯えることができる。
4. X線回折
- クリスタルピッキング
注:サンプルの準備は、X線源施設(家庭用施設対シンクロトロン)によって異なります。それに応じて、ストレージデバイス(バイアルとバイアルホルダーバスケット)を使用してください。塩・沈殿剤濃度によっては、凍結保護剤(グリセロールなど)の添加が必要な場合があります。TmPep1050結晶の場合、PEGまたはバッファー濃度が水晶を避けるのに十分な高さであるため、凍結保護剤は必要ありません。- 液体窒素で満たされた浴場を準備し、サンプル処理に使用されるバイアルまたはバスケットを突っ込みます。
- さまざまなサイズのサンプルピッキングループを設定します:100、150、および200 μm(材料表)。結晶サイズに合わせてサイズを選択します。
- 双眼鏡を使用して、結晶とスポット分離結晶を含むドロップを確認してください(最も簡単に選ぶ)。ループで、下からクリスタルをそっと選びます。すぐに液体窒素でループを突っ込み、適切なバイアルにループを置きます。
- データ収集
注:データ収集は、X線源(家庭用施設対シンクロトロン)および検出器感度によって大きく異なる場合があります。また、分解能、スポット強度、空間群などによって、収集方法はサンプルによって大きく異なる場合があります。このトピックはダウター35によって広くレビューされています。- 回折計のゴニオメーターヘッドに、結晶を載せたループを取り付ける。
- X線ビームパスに合わせて、XYZ軸に沿ってゴニオメーターヘッドを調整します。
- 波長を0.98Åに設定し、検出器を動かして2Åの分解能を得ます。
- 少なくとも2つの異なる結晶方向の画像を取得して、短いデータ収集を開始します。0°と90°で10枚の画像(0.1°あたり1枚)を撮影します。
- 適切なソフトウェア(例えば、ADXV、XDSビューアまたはアルブラビューア)で収集した画像を確認してください。スポットの強度とスポットが見える最高解像度を決定します。単結晶性とスポット分離も確認してください。
- 最終的には、観測に応じて、検出器の位置を高い解像度または低解像度に変更し、露光時間を変更して、ステップ 4.2.3-4.2.5 を繰り返します。
- 0.1°あたり1画像を撮影して、約360°のデータ収集を開始します。検出器の位置と露出時間を最適に設定することを忘れないでください。
5. インデックス化、分子置換、モデル構築
- インデクセーション
注:インデックス化は、回折スポット強度を測定するための方法であり、構造因子36の振幅を与える。収集したイメージの処理には、一般的に 4 つのソフトウェア パッケージが使用されます: Mosflm37、HKL200038、DIALS39、および XDS40。後者は、TmPep1050結晶回折から得られたデータセットのインデックス作成に使用されている。- XDS パッケージと XDSME41をインストールします。HDF5ファイルを処理する場合は、XDS Neggiaプラグインをインストールします(Dectrisのウェブサイトで入手可能)。詳細については、XDS wiki のウェブページ https://strucbio.biologie.uni-konstanz.de/xdswiki/index.php/Main_Page および XDSME の web ページ https://github.com/legrandp/xdsmeをご覧ください。
- データを処理する前に、XDS を実行するフォルダーを作成します。イメージへのパスを見つけます。
- XDSME を実行するには、ターミナル ウィンドウに xdsme /path_to_images/image.extension と入力します。
- XDS がジョブを終了した後、正しい値を確認します。LP ファイル。スペースグループの決定の確率、データの完全性、最高解像度、結晶モザイク性、およびデータ品質に注意してください。また、スペースグループの尤度を取得するためにXDS_pointless.logもチェックします。
注: 出力の例として 図 5 を参照してください。 - 前のプロセスを上書きしないように、XDS が提案した別のスペース グループ ソリューションを別のフォルダに配置して XDSME を再実行します。 xdsme -s space_group_name -c "unit_cell_parameters" /path_to_images/image.extension (例えば 、xdsme -s P21 -c "43.295 137.812 61.118 90.000 110.716 90.000)と入力します。
- 正しいかどうかを確認してください。LPファイルとデータ統計に基づいて最適なソリューションを選択します。
- 「xscale.py XDS_ASCII」と入力して、XSCALE を実行 します。HKL.「XSCALE」と入力して 、XDSCONV xdsconv.py実行します。HKL ccp4.
注: 場合によっては、XDSME がスペース グループを識別できないか、解像度の範囲を適切にカットできないか、または奇妙なデータ統計情報を生成します。このような問題が発生した場合は、XDS をネイティブに実行する価値があります。XDS に複数のパラメーターを導入する必要があります。INP の開始ファイル (XDS wiki ページを参照)。XDS を使用する場合、CCP4 パッケージ42の一部である Pointless を使用して、可能なスペース グループの可能性を確認できます。データ セットの解像度を削減するために、Rmeas < 60% および I/σ ~2 が一般的に受け入れられ、最高解像度の 43を決定します。ただし、分子の置換とモデルの改良は、解像度を I/σ ~0.5-1.5 および CC1/2 に 0.2-0.444まで拡張することで改善できます。
- 分子置換
メモ:実験データは、構造因子の振幅へのアクセスを提供しますが、フェーズを知らなくても、それらは役に立ちません。この位相は、(例えば重原子から)異常な信号に依存する異なる方法によって実験的に決定することができる45。分子置換は、異常散乱原子46,47を伴わない位相を決定する別の方法である。この方法は、関連する分子の座標を使用して、相を反復的に見つけて改善します。分子置換にはフェニックスGUI49のフェイザー48を使用しています。- 4P6Y座標を用いて分子置換の開始モデルを準備します。pdb ファイルからモノマー A を抽出し、Phenix の PDB ファイル エディタ ([モデル ツール ] タブの下) を使用してアラニンのアミノ酸を切り捨てます。
- XDSCONV (5.1.9) によって生成されたリフレクション ファイルと入力としてシーケンスを使用して、Phenix ( データ分析 タブの下) で Xtriage を実行します。
- Xtriage からログ ファイルを確認します。完全性、非対称単位のサブユニット数、異方性、氷のリングの存在、およびツイニング発生に注意してください。
- ポリアラニンで切り捨てられた配列および開始4P6Yモデルを使用して分子置換のためのフェニックス( 分子置換 タブの下)でPhaser-MRを実行します(ステップ5.2.1)。
- 完成したら、モデルが見つかったかどうか、および分子置換のスコアを確認します。少なくとも8の変換係数Zスコア(TFZ)は、解が決定的に正しいことを示します。
- モデル構築
注: 分子置換によって相を決定した後、モデルを構築し、改良する必要があります。このプロトコルは、自動構築と反復的な改良のためにPhenix GUI49 を使用し、手動構造の構築と改良のためにクート50 を使用します。- フェニックスでPhase-MRを使用して分子置換を行った後で、[ 自動構築を実行]を選択します。必要なファイルはすべて自動的に追加されます。[ 実行] を押して自動ビルドを開始します。
- 完了したら、クートでモデルを確認します。クートの電子密度マップに従ってモデルを手動で作成し、改良します。
- モデル、シーケンス、および回折データを入力として使用して、Phenix([ 絞り込み ]タブ)で手動でキュレーションされたモデルを調整します。正しい戦略を選択するには、Phenix ヘルプを参照してください。
- 絞り込んだ後、結果を確認してください:Rフリー とRの作業 は減少する必要があり、Molprobity51 指標は尊重され、現実空間相関が低い外れ値は制限されなければなりません。
- 最適な改良モデルが生成されるまで、ステップ 5.3.2~ 5.3.4 を繰り返します。
- サーバー上で Molprobity を実行 http://molprobity.biochem.duke.edu/します。Molprobityによって識別された外れ値を確認してください。
- 最終的に、最適な改良モデルが得られるまで、ステップ 5.3.2-5.3.6 を繰り返します。
結果
TmPep1050で単量体へのドーデカマー解離の可能性を研究するために、His-60およびHis-307コドンは合成遺伝子を使用してアラニンコドンに置き換えられました。この遺伝子は、次に、TmPep1050変異体の発現および精製のためにpBADベクターでクローン化された後、TmPep1050H6050 H307Aと名付けた。サイズ排除クロマトグラフィー(図3B)は、精製タンパク質が56kDa(モノマーの分子量が36.0kDaである)の見かけの分子量を示した。同様の見かけの分子量である52 kDaが、TmPep1050ダイマー11について報告されている。したがって、TmPep1050H60A H307Aのオリゴマー状態は、二量体として推論され得る。その特異的な活性に関しては、TmPep1050H60A H307Aは、コバルトイオンの存在下でも、基質としてL-Leu-p NA上で完全に不活性であった。この結果は、バリアントが金属イオンを結合できないことを強く示唆している。
TmPep1050H60A H307Aの結晶化条件は、ダイマーの状態(すなわち、0.1 Mクエン酸ナトリウムpH 6.0 10%PEG3350)の周囲のpH対PEG濃度(図4)を変化させることによって最適化された。TmPep1050H60A H307Aの最高の結晶は、0.1 Mクエン酸ナトリウムpH 5.2 20%PEG3350で得られ、単結晶性を改善するためのマイクロシードの1サイクルが得られました。完全なデータセットは、Proxima 2ビームライン(SOLEILシンクロトロン)で分解能2.36Å(表1)で収集された。データインデックス化は、TmPep1050H60A H307A結晶の空間群がC2221であることを示したが、XDSは別の解決策、m P空間群を提案した(図5を参照)。Pointlessによると、C2221とP21のスペースグループの尤度はそれぞれ0.711と0.149でした。データ品質分析によると、非対称単位には2つのモノマーが見られます。Xtriageによる分析は、データセットはおそらく双子であるが、C2221宇宙群の双子は52である可能性は低いことを明らかにした。いくつかの明確なドメインが互いに平行にそれらの格子方向のいくつかを持っている結晶成長異常から結果をツイニングする53.また、高い結晶対称性から生じる可能性があり、誤ったデータインデックス化を示します。したがって、P21結晶格子がC2221のように見えるように、疑似メロエドラル双生児が存在する可能性があります。データセットは、その後、宇宙群P21でインデックス化され、分子置換で試験された。P21でインデックス付けされたデータセットのXtriage分析は、双子の法則h、-k、-h-lに続く擬似メロエドラル双子を明らかにした。
ドデカメリックTmPep1050(PDBコード4P6Y)からのモノマーの座標を用いて、分子置換溶液がP21のみでインデックス化されたデータセットに対して、TFZスコア28.9で発見された。したがって、回折データはモデル構築のための組み合わせたデータセットとして扱われました。分子置換のバイアスを最小限に抑えるために、phenix.autobuild54,55を使用して最初のモデルを構築しました。TmPep1050H60A H307Aの構造は、フェニックスとクートでの自動および手動の改良のいくつかのサイクルの後に完了しました(表1および図6A)。この構造は、PDBeピサ56で計算した、−16.2kcalmol-1の両方のモノマーとΔiGの間の1,710Å2の界面を有するオリゴマー状態を確認した。これに対して、ダイメリックTmPep10502-merの界面面とΔiGはそれぞれ1,673Å2および-16.7kcalmol-1である。
TmPep1050H60A H307A の構造は、アライメント時に0.774 ÅのRMSを有する野生型ダイマー構造と非常に類似している。重要なことに、α8とα10ヘリの高い柔軟性、無秩序な活性部位Gln-196-Val-202、およびLys-229-Ala-235およびLys-247-Ser 254の変位の両方の構造で同じ構造変化が観察される。これらの改変は、その金属補因子11の存在しない場合のドーデカマー形成の障害と以前に相関していた。しかし、His-60とHis-307の2つの突然変異は、Asp-168およびAsp-62の側鎖にわずかな影響を及ぼした。それらは野生型ダイマーとは異なる立体構造でロックされているように見えた(図6B)。Asp-168カルボキシレートは、His-60とHis-307の不在のため40°回転します。したがって、両方のヒスチジン残基は、2つの金属イオンをブリッジングするために正しくAsp-168カルボキシレートを配置するために重要です。Asp-62側鎖は触媒の場所の外のGlu-18カルボキシレートに向いている。Asp-62は、His-60のpKa を調節すると仮定され、したがって、M2部位における金属イオン結合に影響を与えるとして、触媒に重要な役割を有し得る。さらに、金属イオン結合時の触媒部位の安定化に構造的に関与し、ドーデカマーの形成を支持する可能性がある。

図1:相同組換えによるpBADベクターへのクローニングTM_1050 ORFの模式図。
ORFは、プロモーターBAD末端および配列上流 PmeI制限部位に相同の2つの30bp配列によって横たわる。 この図の大きなバージョンを表示するには、ここをクリックしてください。
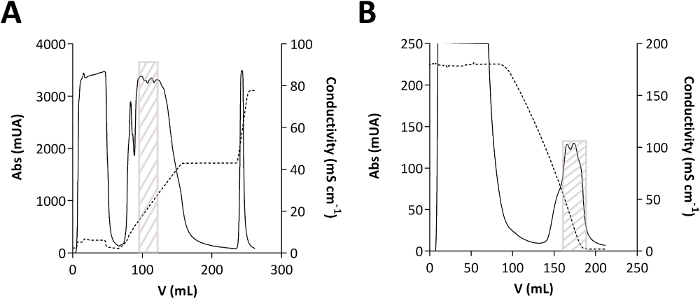
図2:TmPep1050精製のクロマトグラム。
(A) アニオン交換クロマトグラフィー(B) 疎水性相互作用クロマトグラフィー.吸光度(Abs)は、吸光度(mUA)のミリ単位で表され、平易な線で示される。mS cm-1で表される導電率は、破線で示されます。灰色のボックスは、TmPep1050がクロマトグラム上で溶出する場所を示しています。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図3:(A)のサイズ排除クロマトグラフィー(A)ドーデカマー、(B)TmPep1050H60A H307A、および(C)TmPep1050H60A H60A。
サンプルは、120 mLカラムに詰め込まれたSEC樹脂を用いて分析した。吸光度(Abs)は、吸光度(mUA)のミリ単位で表されます。(D)チログロブリン(T)、フェリチン(F)、アルドラーゼ(Ald)、コンアルブミン(C)、アルブミン(Alb)を標準として使用したSECカラムの較正。相対質量と溶出体積の対数の相関は線形であり、R2 は0.91である。95% の信頼区間はドットで表されます。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図4: TmPep1050H60A H307A 結晶化の最適化
(A) 最初の最適化戦略は、pH (4.5~ 6.0) と PEG3350 濃度 (5% ~ 25%)結晶板は、スキーマ化され、ウェルは色分けされています:沈殿のための赤、多結晶の黄色、単結晶の緑。(B) 第2の最適化戦略には、pH対PEG3350のばらつきが狭い25倍の希釈種子の使用が含まれる。(C)結晶の形状とサイズ(上の画像)と後(下の画像)結晶化最適化とマイクロシード。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図 5: ログ出力からの抜粋が正しい。XDSによるTmPep1050H60A H307A データインデックスのLP。
上部パネルは、可能なブラヴァイス格子、最も可能性が高い m C、mP、および oC.ミドルパネル、C2221 空間群でインデックス化されたデータの全体的な統計である。P21 スペース・グループで索引付けされたデータの、下部パネル、全体の統計。 この図の大きなバージョンを表示するには、ここをクリックしてください。

図 6: TmPep1050H60A H307A の構造 .
()TmPep1050H6050 H307A サブユニット(赤、PDB コード 5NE9)対ドーデカマーサブユニット(白、PDB コード 6NW5)とダイマーサブユニット(青、PDB コード5NE6)の構造的アライメント。矢印は、ドーデカマーとダイマーの構造上の相違を示します。(B) TmPep1050H60A H307A 活性部位(赤色)のクローズアップ(赤)は、TmPep1050ダイマー(青)とドデカマー(白)の活性部位と比較した。 この図の大きなバージョンを表示するには、ここをクリックしてください。
| TmPep1050H60A H307A | |
| データ収集 | |
| 温度 (K) | 100 |
| 放射線源 | ソレイル・プロキシマ 2 |
| 波長(Å) | 0.9801 |
| 検出 器 | デクトリス アイガー X 9M |
| 振動範囲 (°) | 0.1 |
| 露光時間(複数可) | 0.025 |
| スペースグループ | P 1 21 1 |
| 単位セル パラメータ | |
| α, β, γ (°) | 90.00, 110.69, 90.00 |
| a, b, c (Å) | 43.24, 137.79, 61.11 |
| 解像 度 | 43.99 – 2.37 (2.52-2.37) |
| ユニークな反射 | 26.902 |
| マージ (%) | 0.14 |
| 冗長性 | 6,815 |
| 8.64 (2.12) | |
| 完全性 (%) | 99.6 (97.9) |
| CC1/2 (%) | 99.2 (84.1) |
| 絞り込み | |
| 解像 度 | 43.99 – 2.37 |
| 反射 | 26.9 |
| Rフリー セット テストカウント | 1345 |
| R作業/R無料 | 0.206/0.234 |
| ASUあたりのタンパク質分子 | 2 |
| VM (Å3/Da) | 2.37 |
| 溶剤含有量(%) | 49.0 |
| タンパク質/溶媒原子 | 4,559/96 |
| r.m.s.d. ボンド長さ (Å) | 0.31 |
| r.m.s.d. ボンド角度 (°) | 0.51 |
| 平均B因子 (Å2) | 57.0 |
| 好まれる/許可されていないラマチャンドランφ/ψ (%) | 95.02 / 0.17 |
| ツイン法 | h, -k, -h-l |
| PDB コード | 5NE9 |
表 1: データ収集および絞り込み統計 括弧内の値は、最高解像度のシェル用です。
ディスカッション
本明細書に記載されたプロトコルは、構造レベルでのTmPep1050のダイマー・ドデカマー転移を理解することを可能にする。方法論は、両方のTmPep1050オリゴマー11の構造を決定するために以前に経験した。最も困難なステップは、安定したダイマーへのドーデカマーの解離を促進する条件を見つけることでした。このような条件は、金属イオン補因子が添加されたときにドーマーをドーデカマーに再関連付けできるように十分に軽度でなければならなかった。オリゴマーの分離は、構造研究およびさらなる生化学的特徴付けを条件とする重要なステップでもあった(例えば、種々の用量のCo2+におけるドーデカマー再結合の研究)。分子置換は、相決定のための実証済みの方法であり、TmPep1050オリゴマーおよびその変異体の構造を解くために使用された。提案されたプロトコルは、オリゴマー化状態が金属補因子の入手可能性に依存する他の金属酵素を研究するために適応され得る。
プロトコルを説明するために、研究の事例が提示された、TmPep1050H60A H307Aその金属結合部位は、その金属結合部位は、その金属結合部位は、アラニンにHis-60およびHis-307を変異させることによって損なわれた。これらの残基は、それぞれM2およびM1部位でCo2+に結合する。金属結合の干渉は、オリゴマー化状態を摂動させ、単量体への完全な解離につながった可能性がある。このような現象の証拠は、PhTET2およびPfTET3、P.堀越しおよびP.フリオススからの2つのM42アミノペプチダーゼ、それぞれ24、29について報告されている。TmPep1050H60A H307Aは、この変異体形成されたダイマーのみとして期待どおりに動作しなかった。その構造は野生型ダイマーと同じ変更を示したが、2つの小さな例外があった。実際、Asp-168とAsp-62の側鎖は、活動部位の安定化を妨げる型破りな向きでロックされているように見えた。彼らの向きは、単一点突然変異変異変異体ではこのような改変が認められなかったため、His-60およびHis-307によって課されたように見えた。
開示事項
著者らは開示するものは何もない。
謝辞
この論文を校正し、建設的なコメントをしてくれたマーティン・ルーバーに感謝します。Proxima 2ビームライン(SOLEILシンクロトロン)へのアクセスは、ブロック割り当てグループ20151139内に含まれました。
資料
| Name | Company | Catalog Number | Comments |
| 1,10-phenanthroline | Sigma-Aldrich | 13, 137-7 | |
| Amicon Ultra 0.5 ml Centrifugal Filters Ultracel 30K | Merck Millipore | UFC503096 | |
| Amicon Ultra 15 Centrifugal Filters Ultracel 30K | Merck Millipore | UFC903024 | |
| Benzonase Nuclease | Merck Millipore | 70664-3 | |
| CCP4 | N/A | visit http://www.ccp4.ac.uk/ | |
| cOmplete EDTA-free | Roche | 5056489001 | |
| Coot | N/A | visit https://www2.mrc-lmb.cam.ac.uk/personal/pemsley/coot/ | |
| Crystal Screen I | Hampton Research | HR2-110 | |
| Crystal Screen II | Hampton Research | HR2-112 | |
| DreamTaq Green PCR Master Mix | ThermoFisher Scientific | K1082 | |
| EasyXtal 15-well tool | NeXtal | 132007 | |
| Escherichia coli PPY strain | N/A | see reference 31 | |
| Escherichia coli XL1 blue strain | Agilent | 200249 | |
| Gel Filtration Calibration Kit HMW | GE Healthcare Life Sciences | 28-4038-42 | |
| Gel Filtration Calibration Kit LMW | GE Healthcare Life Sciences | 28-4038-41 | |
| Gel Filtration Standard | Biorad | 1511901 | |
| GeneJET Plasmid Miniprep Kit | ThermoFisher Scientific | K0503 | |
| Index | Hampton Research | HR2-144 | |
| Litholoops | Molecular Dimensions | ||
| L-leucine-p-nitroanilide | Bachem AG | 40010720025 | |
| Natrix 1 | Hampton Research | HR2-116 | |
| Natrix 2 | Hampton Research | HR2-117 | |
| Neggia plugin | Dectris | N/A | visit https://www.dectris.com/ |
| NeXtal Tubes JCSG Core Suite I | NeXtal | 130724 | |
| NeXtal Tubes JCSG Core Suite II | NeXtal | 130725 | |
| NeXtal Tubes JCSG Core Suite III | NeXtal | 130726 | |
| NeXtal Tubes JCSG Core Suite IV | NeXtal | 130727 | |
| pBAD-TOPO | ThermoFisher Scientific | K430001 | |
| Phenix | N/A | visit https://www.phenix-online.org/ | |
| Phusion High-Fidelity DNA polymerase | ThermoFisher Scientific | F-530L | |
| Salt RX 1 | Hampton Research | HR2-107 | |
| Salt RX 2 | Hampton Research | HR2-109 | |
| SnakeSkin Dialysis Tubing, 3.5K MWCO | ThermoFisher Scientific | 88242 | |
| Source 15Phe | GE Healthcare Life Sciences | 17014702 | |
| Source 15Q | GE Healthcare Life Sciences | 17094705 | |
| Superdex 200 prep grade | GE Healthcare Life Sciences | 17104301 | |
| Thermotoga maritima MSB8 strain | American Type Culture Collection | ATCC 43589 | |
| TmCD00089984 | DNASU Plasmid Repository | N/A | |
| XDS | N/A | visit http://xds.mpimf-heidelberg.mpg.de/ | |
| xdsme | N/A | visit https://github.com/legrandp/xdsme |
参考文献
- Levy, E. D., Teichmann, S. A. Structural, Evolutionary, and Assembly Principles of Protein Oligomerization. Progress in Molecular Biology and Translational Science. 117, 25-51 (2013).
- Selwood, T., Jaffe, E. K. Dynamic dissociating homo-oligomers and the control of protein function. Archives of Biochemistry and Biophysics. 519 (2), 131-143 (2012).
- Jaffe, E. K. The Remarkable Character of Porphobilinogen Synthase. Accounts of Chemical Research. 49 (11), 2509-2517 (2016).
- Ramström, H., et al. Properties and Regulation of the Bifunctional Enzyme HPr Kinase/Phosphatase in Bacillus subtilis. Journal of Biological Chemistry. 278 (2), 1174-1185 (2003).
- Rudyak, S. G., Brenowitz, M., Shrader, T. E. Mg2+-Linked Oligomerization Modulates the Catalytic Activiy of the Lon (La) Protease from Mycobacterium smegmatis. Biochemistry. 40 (31), 9317-9323 (2001).
- Yamamoto, S., Storey, K. B. Dissociation-Association of lactate dehydrogenase Isozymes: Influences on the formation of tetramers vs. dimers of M4-LDH and H4-LDH. International Journal of Biochemistry. 20 (11), 1261-1265 (1988).
- Sirover, M. A. Structural analysis of glyceraldehyde-3-phosphate dehydrogenase functional diversity. The International Journal of Biochemistry & Cell Biology. 57, 20-26 (2014).
- Gupta, V., Bamezai, R. N. K. Human pyruvate kinase M2: A multifunctional protein: Multifunctional Human PKM2. Protein Science. 19 (11), 2031-2044 (2010).
- Wiegand, G., Remington, S. J. Citrate synthase: Structure, Control, and Mechanism. Annual Review of Biophysics and Biophysical Chemistry. 15, 97-117 (1986).
- Libonati, M., Gotte, G. Oligomerization of bovine ribonuclease A: structural and functional features of its multimers. Biochemical Journal. 380 (2), 311-327 (2004).
- Dutoit, R., et al. How metal cofactors drive dimer-dodecamer transition of the M42 aminopeptidase TmPep1050 of Thermotoga maritima. Journal of Biological Chemistry. 294 (47), 17777-17789 (2019).
- Rawlings, N. D., et al. The MEROPS database of proteolytic enzymes, their substrates and inhibitors in 2017 and a comparison with peptidases in the PANTHER database. Nucleic Acids Research. 46 (1), 624-632 (2018).
- Neuwald, A. F., Liu, J. S., Lipman, D. J., Lawrence, C. E. Extracting protein alignment models from the sequence database. Nucleic Acids Research. 25 (9), 1665-1677 (1997).
- Dutoit, R., Brandt, N., Legrain, C., Bauvois, C. Functional Characterization of Two M42 Aminopeptidases Erroneously Annotated as Cellulases. PLoS ONE. 7 (11), 50639(2012).
- Franzetti, B., et al. Tetrahedral aminopeptidase: a novel large protease complex from archaea. The EMBO Journal. 21 (9), 2132-2138 (2002).
- Borissenko, L., Groll, M. Crystal Structure of TET Protease Reveals Complementary Protein Degradation Pathways in Prokaryotes. Journal of Molecular Biology. 346 (5), 1207-1219 (2005).
- Appolaire, A., et al. TET peptidases: A family of tetrahedral complexes conserved in prokaryotes. Biochimie. 122, 188-196 (2016).
- Russo, S., Baumann, U. Crystal Structure of a Dodecameric Tetrahedral-shaped Aminopeptidase. Journal of Biological Chemistry. 279 (49), 51275-51281 (2004).
- Schoehn, G., et al. An Archaeal Peptidase Assembles into Two Different Quaternary Structures: A tetrahedron and a giant octahedron. Journal of Biological Chemistry. 281 (47), 36327-36337 (2006).
- Durá, M. A., et al. The structural and biochemical characterizations of a novel TET peptidase complex from Pyrococcus horikoshii reveal an integrated peptide degradation system in hyperthermophilic Archaea: Characterization of P. horikoshii TET3 peptidase. Molecular Microbiology. 72 (1), 26-40 (2009).
- Basbous, H., Appolaire, A., Girard, E., Franzetti, B. Characterization of a Glycyl-Specific TET Aminopeptidase Complex from Pyrococcus horikoshii. Journal of Bacteriology. 200 (17), 00059(2018).
- Appolaire, A., et al. Small-angle neutron scattering reveals the assembly mode and oligomeric architecture of TET, a large, dodecameric aminopeptidase. Acta Crystallographica Section D Biological Crystallography. 70 (11), 2983-2993 (2014).
- Appolaire, A., et al. The TET2 and TET3 aminopeptidases from P yrococcus horikoshii form a hetero-subunit peptidasome with enhanced peptide destruction properties: TET aminopeptidase multi-subunit complex. Molecular Microbiology. 94 (4), 803-814 (2014).
- Colombo, M., Girard, E., Franzetti, B. Tuned by metals: the TET peptidase activity is controlled by 3 metal binding sites. Scientific Reports. 6 (1), 20876(2016).
- Petrova, T. E., et al. Structure of the dodecamer of the aminopeptidase APDkam598 from the archaeon Desulfurococcus kamchatkensis. Acta Crystallographica Section F Structural Biology Communications. 71 (3), 277-285 (2015).
- Kim, D., et al. Structural basis for the substrate specificity of PepA from Streptococcus pneumoniae, a dodecameric tetrahedral protease. Biochemical and Biophysical Research Communications. 391 (1), 431-436 (2010).
- Chevrier, B., et al. Crystal structure of Aeromonas proteolytica aminopeptidase: a prototypical member of the co-catalytic zinc enzyme family. Structure. 2, 283-291 (1994).
- Rosenbaum, E., Ferruit, M., Durá, M. A., Franzetti, B. Studies on the parameters controlling the stability of the TET peptidase superstructure from Pyrococcus horikoshii revealed a crucial role of pH and catalytic metals in the oligomerization process. Biochimica et Biophysica Acta (BBA) - Proteins and Proteomics. 1814 (10), 1289-1294 (2011).
- Macek, P., et al. Unraveling self-assembly pathways of the 468-kDa proteolytic machine TET2. Science Advances. 3 (4), 1601601(2017).
- Edelheit, O., Hanukoglu, A., Hanukoglu, I. Simple and efficient site-directed mutagenesis using two single-primer reactions in parallel to generate mutants for protein structure-function studies. BMC Biotechnology. 9 (1), 61(2009).
- Zhang, Y., Werling, U., Edelmann, W. SLiCE: a novel bacterial cell extract-based DNA cloning method. Nucleic Acids Research. 40 (8), 55(2012).
- Schleif, R. AraC protein, regulation of the l-arabinose operon in Escherichia coli, and the light switch mechanism of AraC action. FEMS Microbiology Reviews. 34 (5), 779-796 (2010).
- McPherson, A., Gavira, J. A. Introduction to protein crystallization. Acta Crystallographica Section F Structural Biology Communications. 70 (1), 2-20 (2014).
- Bergfors, T. Seeds to crystals. Journal of Structural Biology. 142 (1), 66-76 (2003).
- Dauter, Z. Collection of X-Ray Diffraction Data from Macromolecular Crystals. Protein Crystallography. 1607, 165-184 (2017).
- Powell, H. R. X-ray data processing. Bioscience Reports. 37 (5), 0227(2017).
- Battye, T. G. G., Kontogiannis, L., Johnson, O., Powell, H. R., Leslie, A. G. W. iMOSFLM a new graphical interface for diffraction-image processing with MOSFLM. Acta Crystallographica Section D Biological Crystallography. 67 (4), 271-281 (2011).
- Otwinowski, Z., Minor, W. Processing of X-ray diffraction data collected in oscillation mode. Methods in Enzymology. 276, 307-326 (1997).
- Clabbers, M. T. B., Gruene, T., Parkhurst, J. M., Abrahams, J. P., Waterman, D. G. Electron diffraction data processing with DIALS. Acta Crystallographica Section D Structural Biology. 74 (6), 506-518 (2018).
- Kabsch, W. XDS. Acta Crystallographica Section D Biological Crystallography. 66 (2), 125-132 (2010).
- Legrand, P. legrandp/xdsme: March 2019 version working with the latest XDS version. , Jan 26, 2018. Zenodo (2019).
- Evans, P. Scaling and assessment of data quality. Acta Crystallographica Section D Biological Crystallography. 62 (1), 72-82 (2006).
- Wlodawer, A., Minor, W., Dauter, Z., Jaskolski, M. Protein crystallography for non-crystallographers, or how to get the best (but not more) from published macromolecular structures: Protein crystallography for non-crystallographers. FEBS Journal. 275 (1), 1-21 (2008).
- Karplus, P. A., Diederichs, K. Assessing and maximizing data quality in macromolecular crystallography. Current Opinion in Structural Biology. 34, 60-68 (2015).
- Taylor, G. L. Introduction to phasing. Acta Crystallographica Section D Biological Crystallography. 66 (4), 325-338 (2010).
- Rossmann, M. G., Blow, D. M. The detection of sub-units within the crystallographic asymmetric unit. Acta Crystallographica. 15, 24-31 (1962).
- Rossmann, M. G. The molecular replacement method. Acta Crystallographica Section A Foundations of Crystallography. 46 (2), 73-82 (1990).
- McCoy, A. J., et al. Phaser crystallographic software. Journal of Applied Crystallography. 40 (4), 658-674 (2007).
- Adams, P. D., et al. PHENIX: a comprehensive Python-based system for macromolecular structure solution. Acta Crystallographica Section D Biological Crystallography. 66 (2), 213-221 (2010).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Crystallographica Section D Biological Crystallography. 66 (4), 486-501 (2010).
- Chen, V. B., et al. MolProbity: all-atom structure validation for macromolecular crystallography. Acta Crystallographica Section D Biological Crystallography. 66 (1), 12-21 (2010).
- Zwart, P. H., Grosse-Kunstleve, R. W., Lebedev, A. A., Murshudov, G. N., Adams, P. D. Surprises and pitfalls arising from (pseudo)symmetry. Acta Crystallographica Section D Biological Crystallography. 64 (1), 99-107 (2008).
- Yeates, T. O. Detecting and overcoming crystal twinning. Methods in Enzymology. 276, 344-358 (1997).
- Terwilliger, T. C. Using prime-and-switch phasing to reduce model bias in molecular replacement. Acta Crystallographica Section D Biological Crystallography. 60 (12), 2144-2149 (2004).
- Terwilliger, T. C., et al. Iterative model building, structure refinement and density modification with the PHENIX AutoBuild wizard. Acta Crystallographica Section D Biological Crystallography. 64 (1), 61-69 (2008).
- Krissinel, E., Henrick, K. Inference of Macromolecular Assemblies from Crystalline State. Journal of Molecular Biology. 372 (3), 774-797 (2007).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved