Method Article
共焦点顕微鏡による単細胞自然蛍光シグネチャの再構築
要約
ここでは、3次元空間に分布する全ての個々の生細胞から自然な細胞蛍光シグネチャ(すなわち細胞自家蛍光)を光学的に抽出およびカタログするためのプロトコルが提示される。この方法は、細菌、真菌、酵母、植物、および動物からの細胞を含む単一細胞分解能で、多様な生物学的系の自然蛍光シグネチャを研究するのに適しています。
要約
ここで説明するのは、共焦点反射顕微鏡支援単一細胞自然蛍光分析(CRIF)であり、3次元(3D)空間に分布する集団内の各個体の生細胞からの自然な細胞蛍光シグネチャを再構築するための低侵襲的な方法である。細胞の自然蛍光シグネチャは、細胞内の様々な生体分子によって放出される蛍光シグナルの集合体です。これまでの研究では、自然蛍光シグネチャは、様々な細胞特性と生理学的状態の違いを反映し、細胞の特性評価と同定のための豊富な情報源であることを確立しました。自然蛍光シグネチャは、伝統的に母集団レベルで分析されてきたが、単細胞レベルではクローン培養を必要としない。CRIFは、個々の細胞からの蛍光シグナルの3D分解能および/または選択的抽出を必要とする研究に特に適しています。蛍光シグネチャは細胞の自然な性質であるため、CRIFは、無傷および単一細胞のタイプおよび/または生理学的状態のタグフリー予測にも適しています。この方法は、異種集団における各単一細胞の表現型を、細胞タグ付けなしで顕微鏡下で自己蛍光シグネチャによって直接評価することができる、合理化された細胞分析のための強力なツールであり得る。
概要
セル1内の多様な生体分子は自己蛍光シグナルを放出し、細胞の自然蛍光シグネチャはこれらのシグナルの集合体から成る。このシグネチャー蛍光は、様々な細胞特性および生理学的状態の違いを反映しています。自然な蛍光の分析は、低侵襲であり、完全な細胞破壊に軽度の代謝修飾から痕跡の範囲を残す伝統的な、より侵襲的な微生物学的プローブを補完することができます。DNAや細胞内容抽出2,3、蛍光インストハイブリゼーション4、および蛍光レポーター遺伝子のゲノムへの導入は、細胞の種類や生理学的状態の決定に有効であるが、一般的には細胞の操作または侵襲的なタグ付けのいずれかが必要である。
バルク微生物培養懸濁液5,6、活性汚泥7、哺乳動物組織8,9、および哺乳動物細胞1,10を含む様々な生きた微生物コロニーの自然な蛍光の研究は、自然な蛍光分析が細胞型および生理学的状態のタグフリー分析を促進することを示した。自然蛍光シグネチャは、従来、単細胞レベルではなく集団レベルで分析されてきたため、クローン培養が必要です。これに対し、このコンフォーカル・レンレクシス法は、コンフォーカル・レンレクシス法を支援する単細胞中華離性の分析(CRIF)技術111を、個々の生きた微生物細胞の自然な細胞蛍光シグネチャを再構成およびカタログ化する。さらに、CRIFは、3次元(3D)空間に分布する集団内の単一の微生物細胞の自然蛍光シグネチャを体系的に照合することができます。
プロトコル
1. サンプルの調製
- グラススライドに、厚さ1mmのシリコーンガスケットとウェルを置きます。
- シリコーンガスケットのウェルに厚さ0.8%(w/v)のアガローススラブを1mm入れる。
- 任意の微生物細胞培養物の細胞密度を600 nm(OD660)=1.0の光学密度に希釈します。
- アガローススラブに5μLの細胞懸濁液を置きます。
- ガラスカバースリップでやさしく覆います。
2. 顕微鏡のセットアップ
注意:CRIF技術は、共焦点反射顕微鏡(CRM)と多チャンネル共焦点顕微鏡を組み合わせたものです。CRMは、細胞の自然な蛍光から独立している細胞形態と空間局在化のための情報源として機能します。多チャンネル共焦点顕微鏡は、細胞の自然蛍光のスペクトル情報を提供します。以下のプロトコルにおいて、CRMまたは共焦点蛍光顕微鏡で取得した任意の画像は、それぞれCRM画像または多チャネル共焦点顕微鏡画像と呼ばれる。
- 反射スペクトルチャンネルを持つ共焦点顕微鏡を、光増倍管(PMT)またはGaAsP検出器に接続します。
注: これらの設定は、いくつかの製造元から入手できます。 - 顕微鏡に十分な倍率を持つ高い数値開口(NA)の目的を装備します。
注: 細菌細胞の解析には、NA > 1.4 の 63x の目的が推奨されます。 - 細胞の形態を可視化するために入射光の細胞散乱に依存するCRMに対応するために、半反射ミラー(例えば、NT 80/20)を顕微鏡に装備します。
- 多チャンネル共焦点顕微鏡では、顕微鏡に二色性ミラーを装備します。たとえば、405、458、488、488、488、514、543、または633 nmのexの場合は、MBS InVis405、MBS458、MBS458/514、MBS488/543/513、またはMBS 488/543/633ビームスプリッターを使用します。
- レーザーパワーメーターを使用して、励起波長ごとに照明強度を調整します。励起波長を通して一定の顕微鏡の下で出力を保つ(例えば、63xの目的で50 μWを使用)。
3. 画像取得
- 顕微鏡ソフトを使用してピンホールサイズを1 AUに設定します。
- 各励起波長のピクセルのドウェル時間(すなわち、走査速度)を設定します。
注: ピクセルの長い時間が長すぎると、セルに損傷を与える可能性があります。細胞への光の損傷を最小限に抑えるために、過度に長いピクセルのドウェル時間を避けてください。細菌サンプルの場合、ピクセルドウェルタイム<55.6 μs/μm2 (顕微鏡下での放射出力が〜17μW/cm2の場合)は、通常、増殖阻害を避けるために適しています。このパラメータは、生物や実験の設定によって異なる場合があります。 - スキャンの解像度を設定します。細菌などの小さな細胞の場合は、1,024 x 1,024のスキャンエリアを使用してください。
- 対象領域がカバーされるように Z スキャン範囲を設定します。
注:アガローススラブに分布する細菌および酵母細胞サンプルの場合、Zスキャン範囲は通常〜15μmで十分です。 - スキャン解除された検出器を設定して、可視波長範囲(例:416~691 nm)をキャプチャします。8-10 nm のスペクトル ウィンドウを使用します。
- 蛍光画像のZスタックを作成するために、長いから最短の励起波長までのシーケンスで多チャンネル共焦点顕微鏡画像を取得します。
- CRMイメージを取得します。
- 取得したイメージを 16 ビットの TIFF ファイルとしてディレクトリに保存します。名前付け規則 XXXcYYzZZ を使用してファイルに名前を付けます.tif XXX は励起波長、YY は検出チャネル番号、ZZ は Z スライス番号、"c"と "z" はそれぞれ検出番号と Z スライス番号のプレフィックスです。
- 例えば、多チャンネル共焦点顕微鏡画像が励起波長405nmで撮影された場合、検出器アレイの第1検出器チャネル、およびZスタックの第5のスライスである場合、その名前は「405c01z05.tif。CRM イメージの場合は、XXX の代わりに文字列 "CRM" を使用します ("CRMc01z05.tifなど)。
注: 2D または 3D セグメンテーションが画像データに最適かどうかを決定します。小さな細胞が2D平面に拘束される状況(例えば、ガラス表面に付着した細菌集団)で、2Dセグメンテーション法を使用します。細胞集団が3次元空間(例えば、バイオフィルムおよび組織サンプル)に分布している場合、または細胞サイズが光学スライスの厚さ(例えば、酵母細胞、哺乳類細胞)よりも大きい場合には、3Dセグメンテーション法を使用する。2D セグメンテーションについては、セクション 4 を参照してください。3D セグメンテーションについては、セクション 5 を参照してください。
- 例えば、多チャンネル共焦点顕微鏡画像が励起波長405nmで撮影された場合、検出器アレイの第1検出器チャネル、およびZスタックの第5のスライスである場合、その名前は「405c01z05.tif。CRM イメージの場合は、XXX の代わりに文字列 "CRM" を使用します ("CRMc01z05.tifなど)。
4. 2D画像解析
- ワークステーションに画像解析ソフトウェア(MATLABなど)を装備します。
- 単細胞の自然蛍光シグネチャの細胞のセグメンテーションと再構築を行います。
- 画像解析ソフトウェアを開きます。
- 表示されているスクリプトの 1 つをダブルクリックして開きます "Script2D.m" 。
- [エディタ] タブに移動し、[実行] をクリックします。フォルダ選択ウィンドウが表示されます。
- ステップ 3.8 で作成したディレクトリを選択し、[ 開く ] をクリックして続行します。セグメンテーション パラメータの入力を求めるダイアログ ボックスが自動的に表示されます。
注: テスト目的で、提供されたデータセット (「Sample_2D」) を選択します。サンプル データセットは圧縮ファイルとして提供され、事前に抽出する必要があります。 - セグメンテーション パラメータを入力します: イメージ 二値化のしきい値 ( 0.45)、 セル領域の上限しきい値 (ピクセル単位) = 200、 セル領域の下限 (ピクセル単位) = 10、 検出器の数 = 32。[ OK] を クリックして続行します。
注: これらのパラメータは、画質に応じて調整が必要な場合があります。 - CRM イメージを表示する新しいイメージ ウィンドウが表示されます。背景の減算に使用する任意の背景領域 (セルが存在しない領域) を選択します。CRM イメージ内の四角形をマウスでドラッグして描画します。選択した領域内をダブルクリックして、選択を確定します。
- 手順 4.2.4 で選択した同じディレクトリで Signature という名前の新しいディレクトリを見つけます。
注: 指定されたコードは、このディレクトリを自動的に作成します。「署名」ディレクトリは、一般的な接頭辞「署名」の後に連続して番号が付.pngファイルとして、集団内の各微生物細胞の自然蛍光シグネチャを格納します。
5.3D画像解析
- ワークステーションに画像解析ソフトウェア(材料表)を装備します。
- 単細胞の自然蛍光シグネチャの細胞のセグメンテーションと再構築を行います。
- 画像解析ソフトウェアを開きます。
- ダブルクリックして、提供されているスクリプト "Script3D.m" を開きます。
- [エディタ] タブに移動し、[実行] をクリックします。フォルダ選択ウィンドウが表示されます。
- ステップ 3.8 で作成したディレクトリを選択し、[ 開く ] をクリックして続行します。セグメンテーション パラメータの入力を求めるダイアログ ボックスが自動的に表示されます。
注: テスト目的で、提供されたデータセット (「Sample_3D」) を選択します。サンプル データセットは圧縮ファイルとして提供され、事前に抽出する必要があります。 - セグメンテーション パラメータを入力: イメージ 二値化のしきい値 (0 –1) のイメージ の二値化 = 0.01、 セル体積の上限しきい値(ピクセル数)=1,000、セル領域の下限しきい値(ピクセル単位)=20、Xピクセルサイズ[μm/ピクセル]=0.26、Yピクセルサイズ[μm/pixel]=0.26、Zピクセルサイズ[μm/ピクセル]=0.42、検出器数= 32。[OK] をクリックして続行します。励起波長の数の入力を促すダイアログボックスが表示されます。
注: これらのパラメータは、画質に応じて調整が必要な場合があります。 - 画像取得に使用する波長数を入力します(例えば、405、488、561、630を使用する場合は、ダイアログボックスに 4 と入力します)。[ OK] をクリックします。
- 最短(すなわち、ボックス名: 励起番号1)から最長波長までのシーケンスで励起波長を入力して、ダイアログボックスに入れます。[ OK] を クリックして続行します。CRM イメージを表示する新しいイメージ ウィンドウがポップアップ表示されます。
- 背景の減算に使用する任意の背景領域(つまり、セルが存在しない領域)を選択します。CRM イメージ内の四角形をマウスでドラッグして描画します。選択した領域内をダブルクリックして、選択を確定します。
- 手順 5.2.4 で選択したディレクトリで 、Signature という名前のディレクトリを見つけます。
注: 指定されたコードは、このディレクトリを自動的に作成します。「署名」ディレクトリは、一般的な接頭辞「署名」の後に連続番号が付いた.pngファイルとして、集団内の各単一の微生物細胞の自然蛍光シグネチャを格納します。
6. 統計分析
注:細胞集団のハイパースペクトルの分布を可視化するために、寸法縮小技術(例えば、主成分分析[PCA])を行います。提供されたスクリプト(PCA.py)は、2つのセル集団(すなわち、2つのクラス)に対してPCAを実行する。
- ワークステーションにプログラミング言語と付属のライブラリとモジュールを装備します (資料表)。
- C ドライブ (またはそれと同等) に空のディレクトリを作成し、ディレクトリに 「Parent_directory」という名前を付けます (C:/Parent_ディレクトリ)。
- 2つの細胞集団のそれぞれについて、2つの細胞集団の蛍光シグネチャ(例えば、ステップ4.2.7で生成された.pngファイル)を2つの別々のディレクトリに格納します。
注: 2 つのディレクトリは両方とも "Parent_directory" に配置する必要があります。
C:/Parent_directory/ プティダKT2440/
プティダKT2440/
 Signature01.png
Signature01.png
 シグネチャー02.png
シグネチャー02.png
 :
: プティダKT2442/
プティダKT2442/
 Signature01.png
Signature01.png
 シグネチャー02.png
シグネチャー02.png
 :
: - PCA.py を「Parent_directory」にダウンロードします。
- ワークステーションのコマンド・ライン・インターフェースを開きます。
- コマンドラインインターフェイスに「python C:/Parent_directory/PCA.py」と入力します。
- 「対象ディレクトリの選択」というメッセージが表示されたら、「Parent_directory」を選択します。
- 「C:/Parent_directory」で、結果として得られるPCAプロットを含む「PCA.png」を見つけます。
結果
図1Aは 、従来のスペクトルプロット(上)およびヒートマップ(中央)として提示される細菌細胞の典型的な単細胞蛍光シグネチャを示す。 図1B は、土壌細菌の集団の元のCRM画像に重ね合わせた正確な2D細胞セグメンテーションの結果を示す(シュードモナスプチダ KT2440)12。結果として生じる母集団の自然蛍光シグネチャは 、図1Dのヒートマップとして提示される。なお、細胞のセグメント化が成功した後、母集団内変動は比較的軽微であった。 図1Bに示すように、P .プチダ の同じ集団に重ね合わされる、不正確な細胞セグメンテーションの例を 図1Cに示す。不正確な細胞セグメンテーションが母集団の自然蛍光シグネチャに及ぼす影響は、かなりの数の外れ値から容易に明らかである(図1E、赤色の三角形)。不正確なセルセグメンテーションは、PCA後のクラスターが、正確なセルセグメンテーションに従って得られたタイトなクラスタと比較して緩いクラスターとなった(図1F)。
典型的には、種内変動にもかかわらず、異なる細胞タイプの自然な蛍光シグネチャは、異なるクラスターを形成する。図2Cは、分類学的に近い株(P.プチダ株KT2440および株KT244213)に対するPCA分析の結果を示す。各個体数(図2A、B)内で小さな変動が認められたにもかかわらず、各集団はPCA分析プロット上に異なるクラスタを形成した(図2C)。
図3Aは、出芽酵母サッカロミセス・セレビシエYM427114の集団の元のCRM画像に重ね合わせた正確な3D細胞セグメンテーションの結果を示す。図3Bは、その集団に対する結果として生じる蛍光シグネチャを示す。

図1:セグメンテーションの精度と明らかな種内変動(A)細菌細胞の典型的な単細胞の自然蛍光シグネチャ(P.プチダKT2440)は、スペクトルプロット(上)およびヒートマップ(中央)として提示した。発光波長は虹色マップ(下)として示されます。スペクトルプロットのY軸とヒートマップの色は、いずれも相対的な蛍光強度を示しています。下の数字は励起波長を示しています。画像解析アルゴリズムによる正確な(B)および不正確な(C)細胞領域認識の視覚的表現。薄い青色のシェーディングは、土壌細菌P.プチダKT 2440の集団に対して検出された細胞領域を示す。パネル C に赤い番号が付いた BLOB は、アルゴリズムが不適切な二値化しきい値設定のために非細胞領域 (つまり、バックグラウンド ノイズ) をセル領域として分類した偽検出の例です。スケールバー= 全体で1 μm。パネルDとEは、それぞれ正確で不正確な細胞セグメンテーションで生成された同じ集団の自然蛍光シグネチャを描写しています。 各列は、1 x 192マトリックスとして提示された再構成された単一細胞ハイパースペクトルを示し、黄色から青色のカラーマップは相対的な蛍光強度を示す。(F) PCA を使用して可視化された、正確な (水色) と不正確な (赤) セグメンテーションにおける自然蛍光シグネチャの分散。各軸ラベルは、コンポーネント番号と PCA 分析に対するその累積寄与率を示します。この図の大きなバージョンを表示するには、ここをクリックしてください。
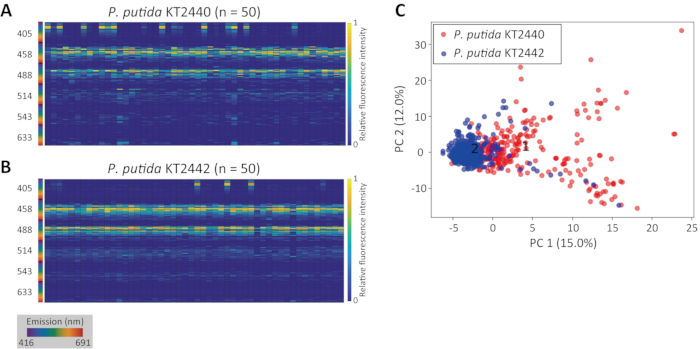
図2:自然蛍光シグネチャの内および種間変動性。P.プチダKT2440(A)およびP.プチダKT2442(B)の集団のためのヒートマップとして提示された自然蛍光シグネチャ。(C)PCAを用いて可視化された2つの自然蛍光シグネチャペアの投影は、P.プチダKT2440(赤、n=288)およびP.プチダKT2442(青、n=373)に相当する。X軸とY軸はそれぞれPC1とPC2を表します。インセット数は、各クラスタの中心を示します(1:P.プチダKT2440、2:P.プティダKT2442)。この図の大きなバージョンを表示するには、ここをクリックしてください。
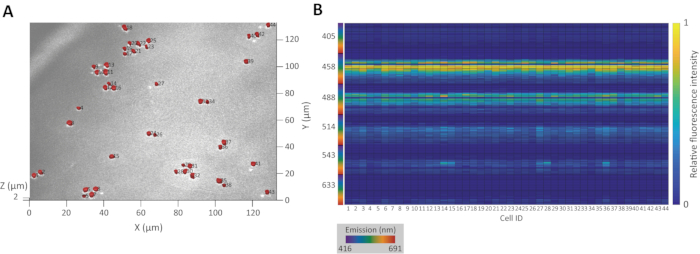
図3:3Dセグメンテーションと自然蛍光シグネチャ抽出の例。 (A)出芽酵母S.セレビシエYM4271の細胞集団として認識される領域の3D投影。差し込み番号は識別番号です。(B)3D領域から抽出された自然蛍光シグネチャのヒートマップ。X 軸番号は、識別番号に対応します。この図の大きなバージョンを表示するには、ここをクリックしてください。
ディスカッション
再現性のある結果を得るために密接に従う必要があるこの方法の2つの重要なポイントがあります:1)励起波長と実験を通じて顕微鏡目的の下でレーザー出力を一貫して保ち、2)正確な細胞セグメンテーションを行います。
最初の点は、異なる実験間で自然蛍光シグネチャを比較する際に特に重要です。最大出力出力はレーザーライン間で大きさが大きく異なる場合があるため、励起波長に同じ「パーセント出力」設定を適用しないようにしてください(すなわち、405、488、514、および530 nmのレーザーラインのすべてに対して5%の出力を使用します)。さらに、目的と内部光学は、通常、不均一な光学吸収特性を有し、また考慮する必要がある。これらの理由から、実際にレーザーパワーメータ(材料表)を使用して目的の出力を測定することをお勧めします。この測定は、レーザーラインからの出力が数年にわたって著しく減衰する可能性があるため、数回の実験、または定期的に行うことをお勧めします。
第2の点は、正確な細胞セグメンテーション、内または種間変動が懸念される状況において特に重要となる。不正確な細胞セグメンテーション(図1D)は外れ値のデータポイント(図1E、赤い三角形で示される)と、より大きな明らかな種内変動をもたらす。セグメンテーション パラメータは慎重に決定し、セグメンテーション結果を元の CRM イメージにオーバーレイしてセルセグメンテーションの精度をチェックしてから、さらに分析または機械学習モデルをトレーニングする必要があります。画質の悪い画像を扱う場合、正確なセグメンテーションを実現するために追加の画像処理が必要になる可能性があります。「ガウスフィルター」または「中央値フィルタ」のいずれかを使用すると、粒状のアーティファクトを除去することができ、二国間フィルタ処理はストライプされた背景をカウンタします。
品質の悪い光学系(非共焦点グレードの目的)や検出器の感度が不十分な場合、微弱な自然蛍光シグネチャの検出に対処することは困難である可能性があります。ここで説明した設定を使用して、以前11,000の細胞サンプルでは、自然蛍光がかすかすぎて自然な蛍光シグネチャを再構築できないタイプの細胞サンプルには遭遇しないことに注意してください。励起のためのより高い放出出力およびより長いピクセルのドウェル時間はより強い放出信号を得るために使用することができるが、これは細胞サンプルに損傷を与える可能性がある。微弱な自然の蛍光が問題となる場合の潜在的な解決策は、例えば、培養液の代わりにバッファーまたは合成培地中の細胞を懸濁させることによって、バックグラウンドシグナル強度を低下させることである。ガラス底皿やマイクロ流体デバイスなどの細胞容器の種類は、アガローススラブがここおよび以前の研究で細胞を位置に保持するために使用されたが、この技術に適しており、適切である。
共焦点顕微鏡法は、接着細胞集団、バイオフィルム、組織サンプルの空間分解能および場解析を提供します。この方法論の潜在的なトレードオフは、例えば、フローサイトメーターと比較した最大スループットです。単一のスキャンセットで数百から千の自然な蛍光シグネチャを収集するコンフォーカルスキャンの完全なセットは、数分かかることがあります。フローサイトメーターは光学設計による感度が低くなりますが、スループットを数桁大きくする可能性があります。
自然の蛍光シグネチャの特定のピークを担う化合物を厳密に同定することは、その複雑な性質のために、典型的には困難です。しかし、ビタミン(例えば、フラビンアデニンジヌクレオチド[FAD])、補酵素(例えば、ニコチンアミドアデニンジヌクレオチド[NADH])、およびリポフスチン色素が細胞内の主要なフルオロフォアとなり得るなどの生物学的に関連する分子の数が示唆されている。例えば、FAD は励起の最大値が 350 ~ 450 nm、放出ピークが約 525 nm16 です。遊離NADHは、それぞれ340nmおよび460nmの励起最大値と発光ピークを有するが、タンパク質結合NADHの発光スペクトルは16,17,18と異なる。ストレスや老化した細胞と関連することが知られているリポ色素は、広い励起範囲(350〜500 nm)および放出範囲(450〜600 nm)16,18を有する。
CRMの使用は、細胞の輪郭を識別するための独立した情報源を提供し、したがって、CRIFのもう一つの重要な利点を提供する:3D空間に分布する個々の細胞からの蛍光信号の選択的抽出。これは、微生物コロニー全体またはバルク培養懸濁液の蛍光特性の従来の分析からの一歩前進を表し、これは培地成分、分泌代謝物、および細胞外マトリックスからの非細胞シグナルで汚染された膨大な数の細胞からのシグナルの平均混合物である。 単一細胞の自然蛍光シグネチャの解析は、無傷細胞の生理的状態の予測的特徴を可能にし、細胞の分布および生理状態の一時的な発達を解決するための潜在的な解決策を提供する。
開示事項
著者らは開示するものは何もない。
謝辞
本研究は、文部科学省(18K04843)からY.八幡、JST ERATO(JPMJER1502)から野村への科学研究助成によって支えられた。
資料
| Name | Company | Catalog Number | Comments |
| Agarose | Wako Chemicals | 312-01193 | |
| Beam splitters | Carl Zeiss, Nikon | MBSInVis405, MBS458, MBS488, MBS458/514, MBS488/543, or MBS 488/543/633 beam splitters (Carl Zeiss) | |
| Confocal microscope | Carl Zeiss, Nikon | Model LSM 880 (Carl Zeiss), Model A1R (Nikon) | |
| Cover slips | Matsunami Glass | C024601 | |
| Glass slides | Matsunami Glass | S011120 | |
| Half-reflection mirror | Carl Zeiss, Nikon | NT80/20 | |
| Laser power meter | Thorlabs | PM400 (power meter console) and S175C (sensor) | |
| LB Broth | Nacalai tesque | 20066-95 | For bacteria culture |
| Image analysis software | The MathWorks | MATLAB version 2019a or later, Image Processing Toolbox is needed | |
| Microscope objective | Carl Zeiss, Nikon | 440762-9904 | e.g. 63x plan Apochomat NA = 1.4 (Carl Zeiss) |
| Microscope software | Carl Zeiss, Nikon | ZEN (Carl Zeiss),NIS-elements (Nikon) | |
| PBS(-) | Wako Chemicals | 166-23555 | |
| Programming language | Python and libraries, modules (numpy, scikit-learn, scikit-image, os, glob, matplotlib, tkinter) are rquired to run the supplied PCA script. | ||
| Silicone gasket | ThermoFisher Scientific | P24744 | |
| Workstation | A high-performance workstation with discrete GPUs is recommended. | ||
| Yeast extract-peptone-dextrose (YPD) agar medium | Sigma-Aldrich | Y1500-250G | For yeast culture |
| YPD medium | Sigma-Aldrich | Y1375-250G |
参考文献
- Monici, M. Cell and tissue autofluorescence research and diagnostic applications. Biotechnology Annual Review. 11, 227-256 (2005).
- Tang, J. Microbial metabolomics. Current Genomics. 12, 391-403 (2011).
- Woo, P. C., Lau, S. K., Teng, J. L., Tse, H., Yuen, K. Y. Then and now: use of 16S rDNA gene sequencing for bacterial identification and discovery of novel bacteria in clinical microbiology laboratories. Clinical Microbiology and Infection. 14, 908-934 (2008).
- Amman, R., Fuchs, B. M. Single-cell identification in microbial communities by improved fluorescence in situ hybridization techniques. Nature Reviews Microbiology. 6, 339-348 (2008).
- Giana, H. E., Silveira, L., Zângaro, R. A., Pacheco, M. T. T. Rapid identification of bacterial species by fluorescence spectroscopy and classification through principal components analysis. Journal of Fluorescence. 13 (6), 489-493 (2003).
- Leblanc, L., Dufour, E. Monitoring the identity of bacteria using their intrinsic fluorescence. FEMS Microbiology Letters. 211 (2), 147-153 (2002).
- Hou, X., Liu, S., Feng, Y. The autofluorescence characteristics of bacterial intracellular and extracellular substances during the operation of anammox reactor. Scientific Reports. 7, 39289 (2017).
- Ramanujam, N., et al. In vivo diagnosis of cervical intraepithelial neoplasia using 337-nm-excited laser-induced fluorescence. Proceeding National Academy of Sciences of the United States of America. 91 (21), 10193-10197 (1994).
- Zhang, J. C., et al. Innate cellular fluorescence reflects alterations in cellular proliferation. Lasers in Surgery and Medicine. 20 (3), 319-331 (1997).
- Gosnell, M. E., et al. Quantitative non-invasive cell characterisation and discrimination based on multispectral autofluorescence features. Scientific Reports. 6, 23453 (2016).
- Yawata, Y., et al. Intra- and interspecies variability of single-cell innate fluorescence signature of microbial cell. Applied and Environmental Microbiology. 85, 00608-00619 (2019).
- Nelson, K. E., et al. Complete genome sequence and comparative analysis of the metabolically versatile Pseudomonas putida KT2440. Environmental Microbiology. 4 (12), 799-808 (2002).
- Bagdasarian, M., et al. Specific-purpose plasmid cloning vectors. II. Broad host range, high copy number, RSF1010-derived vectors, and a host-vector system for gene cloning in Pseudomonas. Gene. 16 (1-3), 237-247 (1981).
- Luo, Y., Vijaychander, S., Stile, J., Zhu, L. Cloning and analysis of DNA-binding proteins by yeast one-hybrid and one-two-hybrid systems. Biotechniques. 20 (4), 564-568 (1996).
- Yawata, Y., et al. Monitoring biofilm development in a microfluidic device using modified confocal reflection microscopy. Journal of Bioscience and Bioengineering. 110 (3), 377-380 (2010).
- Lakowicz, J. R. . Principles of Fluorescence Spectroscopy. Third edition. , (2006).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using NADH and NADPH autofluorescence. Free Radical Biology & Medicine. 100, 53-65 (2016).
- Croce, A. C., Bottiroli, G. Autofluorescence spectroscopy and imaging: A tool for biomedical research and diagnosis. European Journal of Histochemistry. 58 (4), 2461 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved