Method Article
שחזור חתימות פלואורסצנטיות מולדות חד-תאיות על ידי מיקרוסקופיה קונפוקלית
In This Article
Summary
כאן, מוצג פרוטוקול לחילוץ וקיטלוג אופטי של חתימות פלואורסצנטיות תאיות מולדות (כלומר, אוטופלואורסצנטיות תאית) מכל תא חי בודד המופץ במרחב תלת מימדי. שיטה זו מתאימה לחקר חתימת הפלואורסצנטיות המולדת של מערכות ביולוגיות מגוונות ברזולוציה של תא אחד, כולל תאים מחיידקים, פטריות, שמרים, צמחים ובעלי חיים.
Abstract
מתואר כאן הוא השתקפות קונפוקלית בסיוע מיקרוסקופיה ניתוח פלואורסצנטיות מולדת (CRIF), שיטה זעיר פולשנית לשחזור חתימת הפלואורסצנטיות התאית המולדת מכל תא חי בודד באוכלוסייה המופצת במרחב תלת מימדי (תלת-ממדי). חתימת הפלואורסצנטיות המולדת של תא היא אוסף של אותות פלואורסצנטיות הנפלטים על ידי ביומולקולים שונים בתוך התא. מחקרים קודמים קבעו כי חתימות פלואורסצנטיות מולדות משקפות תכונות תאיות שונות והבדלים במצב הפיזיולוגי ומהוות מקור מידע עשיר לאפיון וזיהוי תאים. חתימות פלואורסצנטיות מולדות נותחו באופן מסורתי ברמת האוכלוסייה, מה שמחייב תרבות כלונסאות, אך לא ברמת התא הבודד. CRIF מתאים במיוחד למחקרים הדורשים רזולוציה תלת-ממדית ו/או מיצוי סלקטיבי של אותות פלואורסצנטיות מתאים בודדים. מכיוון שחתימת הפלואורסצנטיות היא מאפיין מולד של תא, CRIF מתאים גם לחיזוי ללא תגים של סוג ו /או מצב פיזיולוגי של תאים שלמים ובודדים. שיטה זו עשויה להיות כלי רב עוצמה לניתוח תאים יעיל, שבו הפנוטיפ של כל תא בודד באוכלוסייה הטרוגנית יכול להיות מוערך ישירות על ידי חתימת autofluorescence שלה תחת מיקרוסקופ ללא תיוג תאים.
Introduction
ביומולקולים מגוונים בתוך תא 1 פולטים אותות פלואורסצנטיות אוטומטית, וחתימת הפלואורסצנטיות המולדת של תא מורכבת מהרכבה של אותות אלה. פלואורסצנטיות חתימה זו משקפת תכונות תאיות שונות וגם הבדלים במצב הפיזיולוגי. ניתוח של פלואורסצנטיות מולדת הוא זעיר פולשני ויכול להשלים בדיקות מיקרוביולוגיות מסורתיות ו פולשניות יותר שמשאירות מגוון של עקבות משינוי מטבולי קל להרס תאים מלא. בעוד טכניקות מסורתיות כגון DNA או חילוץ תוכן תא2,3, פלואורסצנטית בהכלאה במקום4, והכנסת גנים עיתונאי פלואורסצנטי לגנום יעילים בקביעת סוג התא או מצב פיזיולוגי, הם בדרך כלל דורשים מניפולציה של התאים או תיוג פולשני.
מחקרים על הפלואורסצנטיות המולדת של מושבות מיקרוביאליות חיות ושלמות שונות, כולל השעיות של תרבית מיקרואורגניאלית בתפזורת5,6, בוצה פעילה7, רקמות יונקים8,9 ותאי יונקים1,10, הראו כי ניתוח פלואורסצנטיות מולד מאפשר ניתוח ללא תגים של סוגי תאים ומצב פיזיולוגי. חתימות פלואורסצנטיות מולדות נותחו באופן מסורתי ברמת האוכלוסייה ולא ברמת התא הבודד, ולכן מחייבות תרבות כלונסאות. לעומת זאת, טכניקת ה-innate fluorescence של innate (CRIF) בסיוע מיקרוסקופיה confocal,11 המתוארת כאן משחזרת ומקטלגת את חתימת הפלואורסצנטיות התאית המולדת של כל תא מיקרוביאלי חי. יתר על כן, CRIF יכול לאסוף באופן שיטתי את חתימת הפלואורסצנטיות המולדת של תא מיקרוביאלי יחיד בתוך אוכלוסייה המופצת במרחב תלת מימדי (תלת-ממדי).
Protocol
1. הכנת המדגם
- מניחים אטם סיליקון בעובי 1 מ"מ עם בארות על מגלשת זכוכית.
- מניחים לוח אגרוז בעובי 1 מ"מ (w/v) בבאר אטם הסיליקון.
- לדלל את צפיפות התא של תרבית תאים מיקרוביאליים שרירותית לצפיפות אופטית ב 600 ננומטר (OD660) = 1.0.
- מניחים 5 μL aliquot של השעיית תא על לוח אגרוז.
- מכסים בעדינות עם כיסוי זכוכית.
2. התקנה של מיקרוסקופ
הערה: טכניקת CRIF משלבת מיקרוסקופיה של השתקפות קונפוקלית (CRM) ומיקרוספקטרוסקופיה קונפוקלית רב-ערוצית. CRM משמש כמקור המידע למורפולוגיה תאית ולוקליזציה מרחבית, שאינה תלויה בפלואורסצנטיות מולדת תאית. מיקרוספקטרוסקופיה קונפוקלית רב-ערוצית מספקת את המידע הספקטרלי של פלואורסצנטיות תאית מולדת. בפרוטוקול הבא, כל תמונה שנרכשה עם CRM או מיקרוספקטרוסקופיה פלואורסצנטית קונפוקלית מכונה תמונת CRM או תמונה מיקרוספקטרוסקופית קונפוקלית רב ערוצית, בהתאמה.
- חבר מיקרוסקופ קונפוקלי עם ערוצים ספקטרליים מסולקים לצינור פוטומולטיפלייר (PMT) או גלאי GaAsP.
הערה: הגדרות אלה זמינות ממספר יצרנים. - צייד את המיקרוסקופ במטרה צמצם מספרי גבוה (NA) בהגדלה נאותה.
הערה: יעד 63x עם NA > 1.4 מומלץ לניתוח תאים חיידקיים. - צייד את המיקרוסקופ במראה חצי השתקפות (למשל, NT 80/20) כדי להתאים ל- CRM, הנשען על הפיזור התאי של אור האירוע כדי לדמיין את המורפולוגיה של התאים.
- למיקרוספקטרוסקופיה קונפוקלית רב-ערוצית, ציידו את המיקרוסקופ במראות דיכרואיות. לדוגמה, השתמש ב- MBS InVis405, MBS458, MBS488, MBS458/514, MBS488/543 או מפצלי קרן MBS 488/543/633 עבור 405, 458, 488, 514, 543 או 633 ננומטר עירור, בהתאמה.
- התאם את עוצמת התאורה עבור כל אורך גל עירור באמצעות מד כוח לייזר. שמור על הפלט מתחת לקוביות המיקרוסקופ דרך אורכי גל עירור (למשל, השתמש ב- 50 μW עם המטרה 63x).
3. רכישת תמונות
- הגדר את גודל חור הסיכה ל- 1 AU באמצעות תוכנת המיקרוסקופ.
- הגדר את זמן השתהות הפיקסלים (כלומר, מהירות הסריקה) עבור כל אורך גל של עירור.
הערה: זמן השתהות של פיקסלים ארוך מדי עלול לגרום נזק לתאים. הימנעו מזמן השתהות פיקסלים ארוך מדי כדי למזער פוטודאם לתאים. עבור דגימות חיידקים, זמן התעכבות פיקסל <55.6 μm2 (כאשר פלט ההקרנה מתחת למיקרוסקופ הוא ~ 17 μW / cm2) מתאים בדרך כלל כדי למנוע עיכוב צמיחה. פרמטר זה עשוי להשתנות בהתאם לאורגניזמים ולהגדרות ניסיוניות. - הגדר את רזולוציית הסריקה. עבור תאים קטנים כגון חיידקים, השתמש באזור סריקה של 1,024 x 1,024.
- הגדר את טווח סריקת Z כך שאזור העניין יכוסה.
הערה: עבור דגימות תאי חיידקים ושמרים המופצות על לוח אגרוז, טווח סריקת Z של ~ 15 מיקרומטר הוא בדרך כלל מספיק. - הגדר את הגלאי descanned כדי ללכוד את טווח אורך הגל הנראה (למשל, 416−691 ננומטר). השתמש בחלון ספקטרלי של 8−10 ננומטר.
- לרכוש תמונות מיקרוספקטרוסקופיה קונפוקלית רב ערוצית ברצף מאורכי גל עירור הארוכים ביותר עד הקצרים ביותר כדי ליצור ערימות Z של תמונות פלואורסצנטיות.
- השג תמונות CRM.
- שמור את התמונות שנרכשו כקבצי tiff של 16 סיביות בספריה. תן שם לקבצים באמצעות מוסכמות מתן השמות XXXcYYZZZ.tif, כאשר XXX הוא אורך הגל של העירור, YY הוא מספר ערוץ הגלאי, ZZ הוא מספר פרוסת Z, ו- "c" ו- "z" הן קידומת עבור מספר הגלאי ומספר פרוסת Z, בהתאמה.
- לדוגמה, אם תמונה מיקרוספקטרוסקופית קונפוצלית רב-ערוצית צולמה עם אורך גל של 405 ננומטר, ערוץ גלאי ראשון של מערך הגלאים, והיא הפרוסה החמישית של מחסנית Z, קרא לה "405c01z05.tif". עבור תמונות CRM, השתמש במחרוזת "CRM" במקום XXX (לדוגמה, "CRMc01z05.tif").
הערה: החלט אם פילוח 2D או תלת-ממדי מתאים ביותר לנתוני התמונה. השתמש בשיטת פילוח דו-ממדית במצבים שבהם תאים קטנים מוגבלים למישור דו-ממדי (למשל, אוכלוסייה חיידקית הנצמדת למשטח זכוכית). השתמש בשיטת הפילוח התלת-ממדית במצבים שבהם אוכלוסיית התאים מופצת במרחב תלת מימדי (למשל, ביופילמים ודגימות רקמות) או שגודל התא גדול מעובי הפרוסה האופטית (למשל, תאי שמרים, תאי יונקים). עבור פילוח 2D עיין בסעיף 4; עבור פילוח תלת-ממדי, עיין בסעיף 5.
- לדוגמה, אם תמונה מיקרוספקטרוסקופית קונפוצלית רב-ערוצית צולמה עם אורך גל של 405 ננומטר, ערוץ גלאי ראשון של מערך הגלאים, והיא הפרוסה החמישית של מחסנית Z, קרא לה "405c01z05.tif". עבור תמונות CRM, השתמש במחרוזת "CRM" במקום XXX (לדוגמה, "CRMc01z05.tif").
4. ניתוח תמונה 2D
- צייד תחנת עבודה בתוכנת ניתוח תמונה (למשל, MATLAB).
- בצע פילוח תאים ושחזור של חתימות פלואורסצנטיות מולדות חד-תאיות.
- פתח את תוכנת ניתוח התמונות.
- לחץ פעמיים ופתח את אחד מהקבצים שסופקו "Script2D.m".
- עבור לכרטיסיה עורך ולאחר מכן לחץ על הפעל. אמור להופיע חלון בחירת תיקיה.
- בחר את הספריה שנוצרה בשלב 3.8 ולאחר מכן לחץ על פתח כדי להמשיך. תיבת דו-שיח המבקשת את הקלט של פרמטר הפילוח תופיע באופן אוטומטי.
הערה: למטרות בדיקה בחר את ערכת הנתונים שסופקה ("Sample_2D"). ערכת הנתונים לדוגמה מסופקת כקובץ דחוס ויש לחלץ אותה מראש. - הזן את פרמטרי הפילוח: סף הבינאריזציה של התמונה (0−1) עבור בינאריזציה של תמונה = 0.45, סף עליון עבור אזור תא (בפיקסלים) = 200, סף תחתון עבור אזור תא (בפיקסלים) = 10 ומספר הגלאים = 32. לחץ על אישור כדי להמשיך.
הערה: פרמטרים אלה עשויים לדרוש התאמה בהתאם לאיכות התמונה. - חלון תמונה חדש המציג תמונת CRM אמור להופיע. בחר אזור רקע שרירותי (כלומר, אזור שבו תאים נעדרים) לשימוש עבור חיסור רקע. צייר מלבן בתוך תמונת CRM על-ידי גרירת עכבר. לחץ פעמיים בתוך האזור הנבחר כדי לאשר את הבחירה.
- חפש ספריה חדשה בשם חתימה באותה ספריה שנבחרה בשלב 4.2.4.
הערה: הקוד שסופק יוצר ספריה זו באופן אוטומטי. הספריה "חתימה" מאחסנת את חתימת הפלואורסצנטיות המולדת של כל תא מיקרוביאלי באוכלוסייה .png קבצים ממוספרים באופן סדרתי לאחר קידומת נפוצה "חתימה".
5.3D ניתוח תמונה
- צייד תחנת עבודה בתוכנת ניתוח התמונה (טבלת חומרים).
- בצע פילוח תאים ושחזור של חתימות פלואורסצנטיות מולדות חד-תאיות.
- פתח את תוכנת ניתוח התמונות.
- לחץ פעמיים ופתח את קובץ ה- Script שסופק "Script3D.m".
- עבור לכרטיסיה עורך ולאחר מכן לחץ על הפעל. אמור להופיע חלון בחירת תיקיה.
- בחר את הספריה שנוצרה בשלב 3.8 ולאחר מכן לחץ על פתח כדי להמשיך. תיבת דו-שיח המבקשת את הקלט של פרמטרי הפילוח תופיע באופן אוטומטי.
הערה: למטרות בדיקה בחר את ערכת הנתונים שסופקה ("Sample_3D"). ערכת הנתונים לדוגמה מסופקת כקובץ דחוס ויש לחלץ אותה מראש. - הזן את פרמטרי הפילוח: סף הבינאריזציה של התמונה (0-1) עבור בינריזציה של תמונה = 0.01, סף עליון עבור אמצעי אחסון של תאים (בפיקסלים) = 1,000, סף תחתון עבור אזור תא (בפיקסלים) = 20, גודל פיקסל X [μm/pixel] = 0.26, גודל פיקסל Y [μm/פיקסל] = 0.26, פיקסל Z Size [μm/pixel] = 0.42 ומספר הגלאים = 32. לחץ על אישור כדי להמשיך; תופיע תיבת דו-שיח המבקשת את הקלט עבור מספר אורכי הגל של העירור.
הערה: פרמטרים אלה עשויים לדרוש התאמה בהתאם לאיכות התמונה. - הזן את מספר אורכי הגל המשמשים לרכישת תמונה (לדוגמה, אם נעשה שימוש ב- 405, 488, 561, 630, הזן 4 בתיבת הדו-שיח). לחץ על אישור.
- הזן את אורכי הגל של העירור ברצף מהקצר ביותר (כלומר, שם תיבה: עירור מס ' 1) אל אורך הגל הארוך ביותר לתוך תיבות הדיאלוג. לחץ על אישור כדי להמשיך; חלון תמונה חדש המציג תמונת CRM אמור לצוץ.
- בחר את אזור הרקע השרירותי (כלומר, האזור שבו תאים נעדרים) שישמש לתחינת רקע. צייר מלבן בתוך תמונת CRM על-ידי גרירת עכבר. לחץ פעמיים בתוך האזור הנבחר כדי לאשר את הבחירה.
- חפש את הספריה בשם חתימה בספריה שנבחרה בשלב 5.2.4.
הערה: הקוד שסופק יוצר ספריה זו באופן אוטומטי. הספריה "חתימה" מאחסנת את חתימת הפלואורסצנטיות המולדת של כל תא מיקרוביאלי בודד באוכלוסייה .png קבצים ממוספרים באופן סדרתי לאחר קידומת נפוצה "חתימה".
6. ניתוח סטטיסטי
הערה: בצע טכניקות הפחתה ממדיות (למשל, ניתוח רכיבים עיקרי [PCA]) כדי לדמיין את ההתפלגות של היפרספקטרומים של אוכלוסיות התאים. קובץ ה- Script שסופק (PCA.py) מבצע PCA עבור שתי אוכלוסיות תאים (כלומר, שתי מחלקות).
- צייד תחנת עבודה בשפת התכנות ובספריות ובמודולים הנלווים (טבלת חומרים).
- צור ספריה ריקה בכונן C (או שווה ערך) ותן שם לספריה "Parent_directory" (כלומר, C:/ Parent_ הספריה).
- אחסן את חתימות הפלואורסצנטיות (למשל, קבצי .png שנוצרו בשלב 4.2.7) של כל אחת משתי אוכלוסיות התאים לשתי ספריות נפרדות.
הערה: שתי הספריות צריכות להיות ממוקמות ב"Parent_directory".
ג:/Parent_directory/ putidaKT2440/
putidaKT2440/
 חתימה01.png
חתימה01.png
 חתימה02.png
חתימה02.png
 :
: putidaKT2442/
putidaKT2442/
 חתימה01.png
חתימה01.png
 חתימה02.png
חתימה02.png
 :
: - הורד PCA.py לתוך "Parent_directory".
- פתח את ממשק שורת הפקודה של תחנת העבודה.
- הקלד "python C:/Parent_directory/PCA.py" בממשק שורת הפקודה.
- בחר את "Parent_directory" לאחר הצגת ההודעה 'בחר ספריית יעד'.
- ב"C:/Parent_directory", מצא "PCA.png", המכיל עלילת PCA וכתוצאה מכך.
תוצאות
איור 1A מציג את חתימת הפלואורסצנטיות האופיינית לתא יחיד של תא חיידקי המוצג כעלילה מסורתית בספקטרום (למעלה) וכמפת חום (באמצע). איור 1B מציג את התוצאה של פילוח תאים 2D מדויק על גבי תמונת CRM המקורית של אוכלוסייה של חיידקי קרקע (פסאודומונס putida KT2440)12. חתימות הפלואורסצנטיות המולדות המתקבלות עבור האוכלוסייה מוצגות כמפת חום באיור 1D. שים לב כי השתנות ריבוי תוך-כבידתי הייתה מינורית יחסית לאחר פילוח תאים מוצלח. דוגמה לפילוח תאים לא מדויק מוצג באיור 1C, שמונח על אותה אוכלוסייה של P. putida כפי שמוצג באיור 1B. ההשפעה של פילוח תאים לא מדויק על חתימות הפלואורסצנטיות המולדות של האוכלוסייה ניכרת בקלות ממספרם הניכר של חריגים (איור 1E, משולשים אדומים). פילוח תאים לא מדויק הביא לאשכול רופף יותר לאחר PCA בהשוואה לאשכול ההדוק שהושג בעקבות פילוח תאים מדויק (איור 1F).
בדרך כלל, למרות השונות בין המינים, חתימות פלואורסצנטיות מולדות של סוגי תאים שונים יוצרות אשכולות נפרדים. איור 2C מציג את התוצאה של ניתוחי PCA עבור זוג זנים קרובים מבחינה טקסונומית (זן P. putida KT2440 ומתח KT244213). למרות השונות הקטנה שנצפתה בכל אוכלוסייה נפרדת (איור 2A,B), כל אוכלוסייה יצרה אשכול נפרד על חלקת הניתוח PCA (איור 2C).
איור 3A מציג את התוצאה של פילוח תאים תלת-ממדי מדויק על גבי תמונת ה- CRM המקורית של אוכלוסייה של שמרים ניצנים Saccharomyces cerevisiae YM427114. איור 3B מציג את חתימות הפלואורסצנטיות המולדות הנובעות מכך עבור האוכלוסייה.

איור 1: דיוק הפילוח והשונות התוך-המינית הנראית לעין. (A) חתימת פלואורסצנטיות מולדת טיפוסית של תא חיידקי (P. putida KT2440), המוצגת כעלילה ספקטרום (למעלה) ומפת חום (אמצע). אורך גל הפליטה מצוין כמפת צבעי הקשת (למטה). ציר ה- Y של התוויית הספקטרום וצבע מפת החום מצביעים שניהם על עוצמות הפלואורסצנטיות היחסיות. המספרים בתחתית מצביעים על אורך גל העירור. ייצוג חזותי של זיהוי מדויק (B) ולא מדויק (C) של אזור התא על-ידי אלגוריתם ניתוח התמונה. הצללה כחולה בהירה מציינת אזורי תאים שזוהו עבור אוכלוסייה של חיידק קרקע P. putida KT 2440. כתמים עם מספור אדום בחלונית C הם דוגמאות לזיהוי כוזב, כאשר האלגוריתם סיווג אזורים שאינם תאים (כלומר, רעשי רקע) כאזורים בתא עקב הגדרות סף בינאריזציה לא הולמות. סרגלי קנה מידה = 1 מיקרומטר לאורך כל הדרך. לוחות D ו - E מתארים את חתימות הפלואורסצנטיות המולדות עבור אותה אוכלוסייה, שנוצרו עם פילוח תאים מדויק ולא מדויק, בהתאמה. כל עמודה מציגה היפרספקטרום של תא יחיד משוחזר המוצג כמטריצת 1 x 192, כאשר מפת הצבעים הצהובה-כחולה מציינת את עוצמת הפלואורסצנטיות היחסית. (ו) השונות של חתימות פלואורסצנטיות מולדות בסגמנטים מדויקים (כחול בהיר) ולא מדויקים (אדומים) שדמיינו באמצעות PCA. כל תווית ציר מציינת את מספר הרכיב ואת תרומת האחוזים המצטברת שלו לניתוח PCA. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
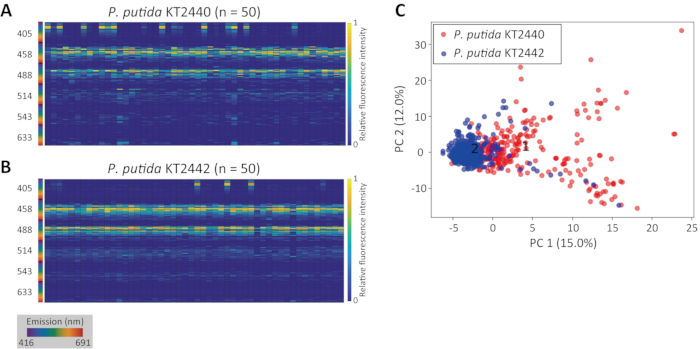
איור 2: שונות בין-גזעית של חתימות פלואורסצנטיות מולדות. חתימות פלואורסצנטיות מולדות המוצגות כמפות חום לאוכלוסייה של P. putida KT2440 (A) ו - P. putida KT2442 (B). (ג) הקרנה של שני זוגות חתימות פלואורסצנטיות מולדות שדמיינו באמצעות PCA, המתאימים ל - P. putida KT2440 (אדום, n = 288) ו - P. putida KT2442 (כחול, n = 373). צירי X ו- Y מייצגים PC1 ו- PC2, בהתאמה. מספר הכניסה מציין את מרכז כל אשכול (1: P. putida KT2440, 2: P. putida KT2442). אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
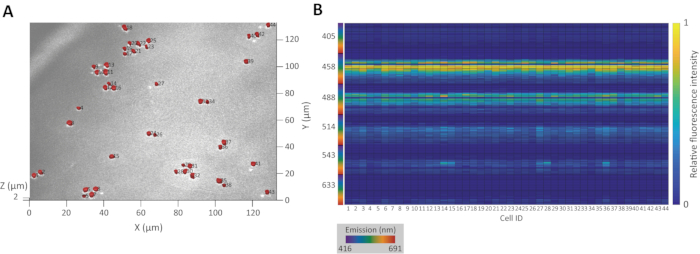
איור 3: דוגמה לסגמנטציה תלת-ממדית ומיצוי חתימת פלואורסצנטיות מולדת. (A) הקרנה תלת-ממדית של אזורים המוכרים כאוכלוסיות תאים של שמרים ניצנים S. cerevisiae YM4271. מספרי ההאצה הם מספרי הזיהוי. (B) מפת חום של חתימות פלואורסצנטיות מולדות שחולצו מאזורי תלת-ממד. מספר ציר ה- X מתאים למספר הזיהוי. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
Discussion
ישנן שתי נקודות קריטיות בשיטה זו שיש לעקוב אחריהן מקרוב כדי להשיג תוצאות ניתנות לשחזור: 1) לשמור על תפוקת כוח הלייזר תחת מטרת המיקרוסקופ עקבית באמצעות אורכי גל עירור וניסויים, ו -2) לבצע פילוח תאים מדויק.
הנקודה הראשונה חשובה במיוחד כאשר משווים את חתימת הפלואורסצנטיות המולדת בין ניסויים שונים. הימנע פשוט מהחלת אותן הגדרות "אחוז פלט" על אורכי הגל של העירור (כלומר, שימוש בתפוקת הספק של 5% עבור כל קווי הלייזר 405, 488, 514 ו- 530 ננומטר), מכיוון שפלט הכוח המרבי יכול להיות שונה עד לסדר גודל בין קווי לייזר. בנוסף, המטרה והאופטיקה הפנימית בדרך כלל יש מאפייני ספיגה אופטית אחידה, אשר גם צריך להיות בחשבון. מסיבות אלה, למעשה מומלץ למדוד את הפלט תחת המטרה באמצעות מד כוח לייזר (טבלת חומרים). לוקח מדידה זו כל כמה ניסויים, או מעת לעת, מומלץ גם, כי הפלט מקווי לייזר יכול להירקב באופן משמעותי על פני כמה שנים.
הנקודה השנייה, פילוח תאים מדויק, הופכת לחשובה במיוחד במצבים שבהם שונות בין המינים היא דאגה. פילוח תאים לא מדויק (איור 1D) מביא לנקודות נתונים חריגות יותר (איור 1E, המצוין על-ידי משולשים אדומים) ולשונות פנים-מינים גבוהה יותר. הפרמטרים פילוח צריך להיקבע בקפידה, בדיקת דיוק פילוח התא על ידי overx overlaying את תוצאות פילוח על תמונת CRM המקורית, לפני שתמשיך כל ניתוח נוסף או אימון מודלים למידת מכונה. אם אתה עובד עם תמונה באיכות ירודה, ייתכן שיידרש עיבוד תמונה נוסף כדי להשיג פילוח מדויק. שימוש ב'מסנן גאוס' או 'מסנן חציוני' יכול להסיר חפצים מגורענים, ועיבוד סינון דו-צדדי מונים בכל רקע מפוספס.
עם אופטיקה באיכות ירודה (למשל, מטרה לא קונפוצלית) או רגישות לא מספקת לגלאי, התמודדות עם זיהוי חתימות פלואורסצנטיות מולדות חלשות עשויה להיות מאתגרת. שים לב כי באמצעות ההתקנה המתוארת כאן ובעבר11, לא נתקלנו בכל סוג של דגימת תא אשר פלואורסצנטיות המולדת שלה היה קלוש מדי כדי לשחזר חתימת פלואורסצנטיות מולדת. למרות שניתן להשתמש בתפוקת פליטה גבוהה יותר עבור עירור וזמן התעכבות פיקסלים ארוך יותר כדי להשיג אותות פליטה חזקים יותר, הדבר עלול לגרום נזק לדגימות התא. פתרון פוטנציאלי למקרים שבהם פלואורסצנטיות מולדת חלשה היא בעיה היא להפחית את עוצמת אות הרקע, למשל על ידי השעיית תאים במדיה חוצץ או סינתטית במקום מרק. סוגי מיכלי תאים, כגון מנות תחתית זכוכית והתקנים מיקרופלואידיים, תואמים ומתאימים לטכניקה זו, אם כי לוח אגרוז שימש לשמירה על תאים בעמדה כאן ובמחקר קודם15.
שיטת המיקרוסקופ הקונפוקלית מציעה רזולוציה מרחבית וניתוח במקום לאוכלוסיות תאים חסידים, ביופילמים ודגימות רקמות. החלפה פוטנציאלית של מתודולוגיה זו היא התפוקה המרבית בהשוואה לציטומטר זרימה, למשל. קבוצה שלמה של סריקות קונפוקליות שאוספות כמה מאות עד אלפי חתימות פלואורסצנטיות מולדות עם סט אחד של סריקות יכולות להימשך עד כמה דקות. ציטומטר זרימה הוא פחות רגיש בשל העיצוב האופטי שלה אבל יכול פוטנציאל להשיג תפוקה מספר סדרי גודל גדול יותר.
לזהות בקפדנות תרכובת האחראית על שיא מסוים בחתימה פלואורסצנטית מולדת היא בדרך כלל מאתגרת, בשל אופיה המורכב. עם זאת, הוצע כי מספר מולקולות רלוונטיות ביולוגית, כגון ויטמינים (למשל, flavin אדנין dinucleotide [FAD]), קואנזים (למשל, nicotinamide אדנין dinucleotide [NADH]), ופיגמנטים lipofuscin יכול להיות פלואורופורים גדולים בתאים1,16. לדוגמה, ל-FAD יש מקסימום עירור בין 350-450 ננומטר ושיא פליטה של כ-525 nm16. ל-NADH חינם יש מקסימום עירור ושיא פליטה של 340 ננומטר ו-460 ננומטר, בהתאמה, אם כי ספקטרום הפליטה של NADH הקשור לחלבון שונה ב-16,17,18. ליפופיגמנטים הידועים כמקשרים לתאים לחוצים או מיושנים הם בעלי טווח עירור רחב (350-500 ננומטר) וטווח פליטה (450-600 ננומטר)16,18.
השימוש ב- CRM מציע מקור מידע עצמאי לזיהוי קווי המתאר של התאים ולכן מספק יתרון משמעותי נוסף של CRIF: החילוץ הסלקטיבי של אותות פלואורסצנטיות מתאים בודדים המופצים בחלל תלת-ממדי. זה מייצג צעד קדימה מהניתוח המסורתי של מאפייני הפלואורסצנטיות של מושבה מיקרוביאלית שלמה או השעיית תרבית בתפזורת, שהיא תערובת ממוצעת של אותות ממספר עצום של תאים מזוהמים באותות שאינם תאים מרכיבים בינוניים, מטבוליטים מופרשים ומטריצה חוץ-תאית. ניתוח של חתימות פלואורסצנטיות מולדות חד-תאיות מאפשר אפיון חיזוי של המצב הפיזיולוגי של תאים שלמים, ומספק פתרון פוטנציאלי לפתרון התפתחות זמנית של התפלגות התא ומצב פיזיולוגי.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
מחקר זה נתמך בחלקו על ידי מענק סיוע למחקר מדעי ממשרד החינוך, התרבות, הספורט והטכנולוגיה של יפן (18K04843) ל- Y. Yawata, ה- JST ERATO (JPMJER1502) ל- N. Nomura.
Materials
| Name | Company | Catalog Number | Comments |
| Agarose | Wako Chemicals | 312-01193 | |
| Beam splitters | Carl Zeiss, Nikon | MBSInVis405, MBS458, MBS488, MBS458/514, MBS488/543, or MBS 488/543/633 beam splitters (Carl Zeiss) | |
| Confocal microscope | Carl Zeiss, Nikon | Model LSM 880 (Carl Zeiss), Model A1R (Nikon) | |
| Cover slips | Matsunami Glass | C024601 | |
| Glass slides | Matsunami Glass | S011120 | |
| Half-reflection mirror | Carl Zeiss, Nikon | NT80/20 | |
| Laser power meter | Thorlabs | PM400 (power meter console) and S175C (sensor) | |
| LB Broth | Nacalai tesque | 20066-95 | For bacteria culture |
| Image analysis software | The MathWorks | MATLAB version 2019a or later, Image Processing Toolbox is needed | |
| Microscope objective | Carl Zeiss, Nikon | 440762-9904 | e.g. 63x plan Apochomat NA = 1.4 (Carl Zeiss) |
| Microscope software | Carl Zeiss, Nikon | ZEN (Carl Zeiss),NIS-elements (Nikon) | |
| PBS(-) | Wako Chemicals | 166-23555 | |
| Programming language | Python and libraries, modules (numpy, scikit-learn, scikit-image, os, glob, matplotlib, tkinter) are rquired to run the supplied PCA script. | ||
| Silicone gasket | ThermoFisher Scientific | P24744 | |
| Workstation | A high-performance workstation with discrete GPUs is recommended. | ||
| Yeast extract-peptone-dextrose (YPD) agar medium | Sigma-Aldrich | Y1500-250G | For yeast culture |
| YPD medium | Sigma-Aldrich | Y1375-250G |
References
- Monici, M. Cell and tissue autofluorescence research and diagnostic applications. Biotechnology Annual Review. 11, 227-256 (2005).
- Tang, J. Microbial metabolomics. Current Genomics. 12, 391-403 (2011).
- Woo, P. C., Lau, S. K., Teng, J. L., Tse, H., Yuen, K. Y. Then and now: use of 16S rDNA gene sequencing for bacterial identification and discovery of novel bacteria in clinical microbiology laboratories. Clinical Microbiology and Infection. 14, 908-934 (2008).
- Amman, R., Fuchs, B. M. Single-cell identification in microbial communities by improved fluorescence in situ hybridization techniques. Nature Reviews Microbiology. 6, 339-348 (2008).
- Giana, H. E., Silveira, L., Zângaro, R. A., Pacheco, M. T. T. Rapid identification of bacterial species by fluorescence spectroscopy and classification through principal components analysis. Journal of Fluorescence. 13 (6), 489-493 (2003).
- Leblanc, L., Dufour, E. Monitoring the identity of bacteria using their intrinsic fluorescence. FEMS Microbiology Letters. 211 (2), 147-153 (2002).
- Hou, X., Liu, S., Feng, Y. The autofluorescence characteristics of bacterial intracellular and extracellular substances during the operation of anammox reactor. Scientific Reports. 7, 39289 (2017).
- Ramanujam, N., et al. In vivo diagnosis of cervical intraepithelial neoplasia using 337-nm-excited laser-induced fluorescence. Proceeding National Academy of Sciences of the United States of America. 91 (21), 10193-10197 (1994).
- Zhang, J. C., et al. Innate cellular fluorescence reflects alterations in cellular proliferation. Lasers in Surgery and Medicine. 20 (3), 319-331 (1997).
- Gosnell, M. E., et al. Quantitative non-invasive cell characterisation and discrimination based on multispectral autofluorescence features. Scientific Reports. 6, 23453 (2016).
- Yawata, Y., et al. Intra- and interspecies variability of single-cell innate fluorescence signature of microbial cell. Applied and Environmental Microbiology. 85, 00608-00619 (2019).
- Nelson, K. E., et al. Complete genome sequence and comparative analysis of the metabolically versatile Pseudomonas putida KT2440. Environmental Microbiology. 4 (12), 799-808 (2002).
- Bagdasarian, M., et al. Specific-purpose plasmid cloning vectors. II. Broad host range, high copy number, RSF1010-derived vectors, and a host-vector system for gene cloning in Pseudomonas. Gene. 16 (1-3), 237-247 (1981).
- Luo, Y., Vijaychander, S., Stile, J., Zhu, L. Cloning and analysis of DNA-binding proteins by yeast one-hybrid and one-two-hybrid systems. Biotechniques. 20 (4), 564-568 (1996).
- Yawata, Y., et al. Monitoring biofilm development in a microfluidic device using modified confocal reflection microscopy. Journal of Bioscience and Bioengineering. 110 (3), 377-380 (2010).
- Lakowicz, J. R. . Principles of Fluorescence Spectroscopy. Third edition. , (2006).
- Blacker, T. S., Duchen, M. R. Investigating mitochondrial redox state using NADH and NADPH autofluorescence. Free Radical Biology & Medicine. 100, 53-65 (2016).
- Croce, A. C., Bottiroli, G. Autofluorescence spectroscopy and imaging: A tool for biomedical research and diagnosis. European Journal of Histochemistry. 58 (4), 2461 (2014).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved