Method Article
タンパク質相互作用を発見したHaloTag技術を用いたタンパク質機能の特性評価
要約
のHaloTag技術は、哺乳動物細胞から両方の小規模および大規模なタンパク質複合体の単離を大きな成功を示している多機能技術です。ここでは、既存の代替案と比較してこの技術の利点を強調し、真核生物の細胞内のタンパク質機能の多くの側面を研究するために、その有用性を実証する。
要約
プロテオミクスの研究は、ウイルス、細菌、および酵母由来のものを含む多数のプロテオームの特徴付けにつながっている質量分析能力の進歩とともに、近年、爆発的に増加している。比較すると、ヒトプロテオームの分析は部分的に起因する研究されなければならないタンパク質の膨大な数ではなく、ネットワークとの相互作用、これらの存在が複雑であるため、遅れている。ヒトプロテオームを理解する課題に対処するために、我々は、多タンパク質複合体の単離のために特に強い、タンパク質単離のためのHaloTag技術を開発し、弱い又は一時的な相互作用および/または低存在量のタンパク質のより効率的な捕捉を可能にしている。のHaloTagは、遺伝的にコードされたタンパク質の融合、共有結合のために設計されたタグ、具体的な、かつ迅速な固定化や種々のリガンドとタンパク質の標識である。これらの特性を活かして、哺乳動物細胞のための多数のアプリケーションは、文字に開発されたタンパク質の機能をIZE、ここで我々を含む方法論現在:小説相互作用または機能アッセイの発見のために使用されるタンパク質プルダウン、および細胞内局在を。我々は、速度、特異性、および融合タンパク質の共有結合捕捉の有意な利点は、他の伝統的な非共有結合性アプローチと比較して、プロテオーム解析のための表面を見つける。我々は、これらと例、人間のブロモドメインタンパクBRD4、およびヒストン脱アセチル化酵素HDAC1として二つの重要なエピジェネティックなタンパク質を用いた技術の広範な有用性を実証する。これらの例は、人間の機能的プロテオミクスのどれでしょう一緒にさらなる理解、小説の相互作用の発見を可能にし、真核生物における細胞内局在を特徴付ける、この技術の力を発揮。
概要
細胞機能、刺激に応答して、開発および/ または疾患状態の変化を理解することは、複雑に、これらの異なる動態1,2全体プロテオームのデコンボリューションに接続されています。多くは、タンパク質の数とすべての可能な相互作用を考えると、これは、ヒトプロテオーム1,3-7に対処する上で非常に困難であったが、下等生物のプロテオームを理解するために行われている。質量分析の進歩は著しく、これらの研究に着手する能力を有効にしているし、ここでは、タンパク質複合体および哺乳動物細胞8月10日からのヒトタンパク質の機能解析の両方の効率的な捕獲のためのHaloTag技術の開発で追加し、重要な進歩を提示する。この技術は、最近、新規なタンパク質機能とunderstandiへの洞察を可能にする、重要な相互作用を発見するための重要な因子であることがいくつかの研究で示されている病気11月13日のNG。
のHaloTagタンパク質融合は、当初タンパク質捕捉、精製、およびラベリング8月10日に関連するものとして、伝統的な親和性タグおよび抗体のいくつかの課題に対処するために開発されました。タンパク質の回収と精製の ためのほとんどすべてのメソッドの中では、特定のタンパク質14の濃縮に使用されている非共有結合相互作用工程がある。このステップの間に、タンパク質および/または複雑なプロセスが完了するまでに数時間ではなく数分を必要とする場合は特に、拡散による関連付けを解除することができます。この懸念に対処するために、融合タンパク質を特異的に迅速かつ不可逆的粒子、表面、又はフルオロフォア9のいずれかであり得るそのリガンドと相互作用するように操作した。それは、そのリガンドに結合されると、それゆえ、他のアフィニティータグと比較して最も差別化要因の一つである、結合したままである。また、ここに証明し、能力がある配位子9( 図1)を交互にすることによって可能である単一の構築、異なる生物学的な質問への回答が。例えば、タンパク質相互作用または機能を調べるために、表面配位子は、タンパク質または複合体( 図1)を捕捉するために使用される。別の実験において、同一の融合タンパク質は、蛍光細胞局在、輸送、またはタンパク質の代謝回転( 図1)を研究するために標識することができる。これは、リガンドが、それが表面または蛍光団であるかどうかを、バインドされると、それは不可逆的であるため、他のリガンドは、続いて同じ実験中に結合することができないことしかし覚えておくことが重要です。
タンパク質相互作用をマップするための既存の技術の分析は、効率的に、より一過性であるか、または低い存在量1,6,7,14,15であるものを含む特異的相互作用を、単離する困難性を明らかにした。数々のグループによるまた、最近の研究sは従来の単離方法の中に、特にヒトプロテオミクスの分野において、質量分析16における真の相互作用物質からの信号をマスクすることができるタンパク質混入物のかなりの数があるという懸念を示している。のHaloTagタンパク質とその共同開発した樹脂アドレスだけでなく、これらの懸念のために開発プロパティ。複合プルダウンするためのプロトコル( 図1)において、複合体の結合複合体の捕捉を促進し、8非特異的結合のレベルを低下両方、15分間で達成することができる。また、不可逆的結合は、低存在量タンパク質の効率的な捕捉を可能にし、はるかに少ない細胞の使用を可能にし、再びバックグラウンドを低減8。複合体の捕捉が確立されると、プロトコルは複雑な完全性を維持するために穏やかな洗浄条件を含む。キャプチャされた複合体の溶出は、下流の肛門の目標によって決定された方法2の方法と選択することで行うことができますysis。欲求が分析またはウェスタンブロットまたは質量分析法によって相互作用するタンパク質を発見することである場合、それは例えばSDSまたは尿素( 図1)のような変性剤との複合体を溶出することをお勧めします。これは元々のHaloTagに融合されたタンパク質は、ベイトタンパク質と呼ばれるように、質量分析のために特に有利である樹脂に結合した後に残るでしょう、したがって、質量分析で検出されたペプチドの集団を支配しません。これはまた、ベイトタンパク質は、おそらくウェスタンブロット、他の非共有結合親和性精製または共免疫沈降に比べて有意な差による分析では表示されませんしかしことを意味します。目標は、機能的研究に使用される複合体を無傷で精製することである場合、溶出は、融合タンパク質およびベイトタンパク質( 図1)との間にコードされるその同族切断配列を認識し、TEV(タバコエッチウイルス)プロテアーゼを用いて行うことができる。 Tヘセ試料は、質量分析法によって分析することができるが、後述のように示され、それらは低い存在量の検出、特定のインタラクターを防止することができるベイトタンパク質の有意な量を含有するであろう。
ここでは、二つの重要な治療上のタンパク質に適用されるブロモドメインタンパクBRD4とヒストン脱アセチル化酵素、HDAC1 17をプルダウンし、画像化プロトコルを提示する。質量分析によって決定されるように我々は、HDAC1複合体の単離のため、および両方のための適切な細胞局在した後、これらの実施例で酵素活性を、予想されるパートナーとの相互作用を示している。まとめると、これらの結果は、真核生物のタンパク質相互作用及び機能の特徴付けのための技術の多機能性の性質および強度を示す。
プロトコル
注:以下のプロトコルは、任意のHaloTag融合でかつ安定的に又は一過性にトランスフェクトされ、選択した任意の細胞株において使用することができる。これらの例のために、我々は、HEK293Tまたはヒーラ細胞内のHaloTag融合体の一過性トランスフェクションのためのプロトコルを提供します。すべての実験のために、我々は、タンパク質のみのコントロールの使用をお勧めします。
1。融合タンパク質発現試験
- 融合タンパク質構築物を取得します。
- 予め作られたのHaloTag融合ベクターを取得するか、既存のベクターを用いて構築する。利用可能な構成要素の詳細については、特定の試薬の表を参照してください。
- 任意のクローンを作成する場合、それは、DNAを検証する配列を決定することをお勧めします。
- 融合タンパク質およびコントロールの発現を試験する。
- 各サンプルについて、1×SDSローディングダイバッファー250μlの準備(60 mMのトリスHCl pH6.8のを、0.75 MMは青、12.5%グリセロール、100 mMジチオスレイトール、0.5%SDSをブロモフェノール)。
- 各融合タンパク質についてそして、制御、標準24ウェルプレート中の2-4×10 5細胞/ mlの密度で、それらの適切な培地中でHEK293TまたはHeLa細胞を0.5mlのプレート。
- 推奨されるようにトランスフェクトし、次いで、37℃で18〜24時間、5%CO 2でインキュベート。
- 24時間のトランスフェクション後、各ウェルのメディアに5mMのテトラメチルローダミン(TMR)リガンドの0.5μLを加え、穏やかにプレートをミックス。
- 37℃で15分間、5%CO 2のリガンドを含有するトランスフェクトされた細胞をインキュベートする。
- 吸引除去オフメディアはリガンドを含有し、1ミリリットルのRT PBSで優しく細胞の各ウェルを洗浄します。
- PBSをオフに吸引し、1ミリリットルのRT PBSで第二の洗浄を行う。
- PBSを除去して細胞に直接染料·バッファをロード1X SDSを200μlを加える。
- 1.5ミリリットルエッペンドルフチューブにプレートからピペット溶解物。
- 95℃で5分間溶解液を沸かす
- SDS-PAGEゲルに反応し、負荷の5〜10μLを削除します。
- 蛍光検出スキャナーを使用して、(TMR励起:555 nm、発光:585 nm)のバンドを検出する。標準として蛍光タンパク質マーカーを使用してください。
- 蛍光検知スキャナが使用できない場合は、融合タンパク質の発現を検出するために、ベイトタンパク質またはタンパク質融合タグのいずれかに対する抗体を用いてウェスタンブロットを行う。
2。タンパク質のプルダウン
- 一過性にトランスフェクトされた細胞の調製:
- 各融合や制御のために3〜4×10 5細胞/ mLまたは構築物あたりの全1〜1.2×10 7細胞での細胞の30ミリリットルと1 15cmのお皿を準備します。
- 37℃で18〜24時間インキュベートし、5%CO 2、その後、推奨されているように構築トランスフェクト。
- 24〜48時間後にトランスフェクション後、メディアを取り出し、ゆっくりと氷冷PBS 20〜25 mlの細胞層を洗浄する。
- PBS洗浄を削除し、25〜30を追加し、4℃の溶液に、PBSを冷やし、軽くプレートから細胞をこすり取る。
- コニカルチューブおよびセントリに細胞を回収2000×gで5〜10分間、4℃のためのフーガ
- 上清を捨て、30分または6ヶ月の最大値の最低-80℃で細胞ペレットを配置します。
- HaloLink樹脂の平衡化:
- 各融合または対照試料については、樹脂の平衡化/洗浄緩衝液(100mMのトリス-HCl、pH7.5、150mMのNaCl、および0.005%IGEPAL CA-630)を12mlを調製注:これは、レジンを作り、使用することが非常に重要である平衡化バッファーを希釈IGEPAL CA-630のような12時間以内に、この時間枠を超えて安定したか有効ではない。
- 優しく均一な懸濁液を得た樹脂を振ったり、混ぜる。
- 各プルダウン実験のために、1.5mlのマイクロチューブに樹脂を200μlを分注する。
- 800 XG( 例えば、微量で3,000 rpm)で1分間遠心し、注意深くチューブの底に樹脂を乱すことなく、上清(エタノール)を取り外して廃棄します。
- レジン平衡/ Waの800μl加えSHバッファチューブを数回転倒よく混ぜる。
- 800×gで2分間遠心し、慎重に取り外し、上澄みを捨てる。
- 繰り返して3回の洗浄を合計2.2.5および2.2.6 2回繰り返します。
- 乾燥から樹脂を防ぐため、以下に記載する細胞溶解物(ステップ2.3.8)を追加する準備まで、最終洗浄または上清を外さないでください。
- バインドと融合複合体の洗浄:
- 各試料について、哺乳動物溶解緩衝液(50mMのトリス-HCl、pH7.5、150mMのNaCl、1%トリトンX-100、0.1%ナトリウムデオキシコール酸)および1×TBS緩衝液1ml(100mMのトリス-HCl、500μlのを準備7.5および150mMのNaCl)。
- ピペッティングあるいは簡単にボルテックスすることにより哺乳類の溶解バッファー300μlの細胞ペレットを再懸濁を解凍する。
- 50Xプロテアーゼインヒビターカクテル(800μg/ mlのベンズアミジン塩酸、500μg/ mlのフェナントロリン、500μg/ mlのアプロチニンの6μl加え、500μg/ mlのロイペプチン、500&#。181;グラム/ mlのペプスタチンA、50mMのPMSF) 注意:これらはタンパク質融合タグが結合を妨げるとしてAEBSFを含むプロテアーゼカクテルを使用することはできません。
- 3μLRQ1 DNaseを添加し、室温で10分間反転。
- ガラスホモジナイザー2.0ミリリットルサイズのダウンス; 25-30氷上のストローク、または25または27のG針を通して細胞を渡す5〜10倍の溶解を完了するためには注意:超音波処理は、複合体がバラバラになることがあり、過加熱はタンパク質融合タグの活動に影響を与える可能性があり、推奨されません。
- 溶解液をクリアし、4℃で5分間、14,000×gで遠心します。
- 氷上の新しいチューブや場所に、明確な溶解物、約300μlの総量を転送します。
- 1X TBS緩衝液の追加の700μlの溶解物および上下にピペッティングによりよく混ぜクリアするために追加します。
- ステップ2.2.8で作成平衡化樹脂チューブを取り、チューブの底に樹脂を乱すことなく、樹脂からの最終洗浄/上清を除去。
- 各TUに追加樹脂を、希釈した溶解物を1mlのもの。
- 22℃(注)で15分間チューブローテーター(または緩やかなミキサー)で混合しながらインキュベート:この時間の間に樹脂のセトリングは、結合効率が低下する。 4°Cでの結合が所望される場合、1時間攪拌混合することによってこれを行う。
- 800×gで2分間遠心樹脂チューブ、上清を捨てる。
- ステップ2.2.1で行われた樹脂平衡化/洗浄緩衝液1ミリリットルを加え、手で数回樹脂チューブを反転させて完全に混合。
- 800×gで2分間遠心樹脂チューブおよび洗浄を捨てる。
- 2.3.12は2.3.14さらに3回に続いて手順を繰り返します。
- 樹脂平衡化/洗浄緩衝液1ミリリットルを加え、一定の回転で5分間22℃でインキュベートする。
- 800×gで2分間遠心樹脂チューブおよび洗浄を捨てる。
- 最終用途に応じて(これ以上の説明のための概要を参照してください)、セクション2.4または2.5のいずれかに進みます。
- S変性ゲル、ウェスタンブロット、または質量分析のためのDS溶出:
- 50μlのSDS溶出緩衝液中で各サンプルから樹脂を再懸濁し(1%SDSおよび50mMトリス-HCl pH7.5中)
- 室温で30分間チューブを横に振る。
- 分析のための新しいチューブに800×gで、転送溶出液で2分間遠心します。
- ウェスタンブロットまたは銀染色ゲルのため、SDS変性ゲル上の負荷5〜10μL。
- 質量分析のために、-20℃で各サンプルの40μlのを保存
- 機能アッセイ、ウェスタンブロット、または質量分析のためのTEVプロテアーゼ溶出:
- 最後の洗浄を除去した後、50μLProTEV切断緩衝液およびTEV酵素の30単位に樹脂を懸濁します。
- 1時間振盪しながら25℃でインキュベートする。
- 800×gで2分間遠心。
- 新鮮なチューブに溶出液を移します。
- 機能アッセイにそのまま使用することが4℃で保存したサンプル。
3。細胞質および核透過性リガンドを用いて蛍光標識融合タンパク質の細胞イメージング
- トランスフェクション、ラベル、および画像セル。
- ×10 5細胞/ mlの密度で1-2各融合タンパク質または対照、各ウェル中のそれらの適切な培地中でHeLa細胞のプレートを400μlのための8ウェルチャンバーのカバーガラスである。
- 推奨されるようにトランスフェクトし、次いで、37℃で18〜24時間、5%CO 2でインキュベート。
- 18〜24時間のトランスフェクション後、各ウェルに、この溶液100μlを加え、穏やかに混合する、TMRリガンド適切な細胞のメディアで1:200に希釈する。
- 37℃で15分間、5%CO 2のリガンドを含有するトランスフェクトされた細胞をインキュベートする。
- 吸引除去オフメディアはリガンドを含有し、37℃に予め温めてきた適切なメディア欠けタンパク質融合タグリガンド、500μlのと交換してください
- 合計3回の洗浄のために二度繰り返します。
- (バックインキュベーターに細胞を入れて376、Cおよび5%CO 2)で30分間。
- 吸引除去オフメディア、37℃に予め温めてきた適切なメディア、500μlのと交換してください
- 適切な取得パラメータ(:555 nm、発光:585 nmのTMR励起)を用いて顕微鏡の画像。
結果
新規の融合タンパク質を使用する場合、それはトランスフェクション後、そのタンパク質の発現のための最初のテストにとって重要であり、また、適切な分子量のタンパク質が生産されていることを検証する。のHaloTag融合タンパク質は蛍光と共有結合透過性または局在化に依存して、不浸透性のリガンドで標識することができるように、それがすぐにfluorimager上の走査に続いて、変性ゲル電気泳動に細胞溶解物を適用することにより発現を決定することが可能である。 1.2項に記載されているプロトコルを使用して、ハロ- BRD4(189 kD)のとのHaloTag単独のコントロール(CTRL)の発現は、(34 kDの、 図2A)が観察される。プロトコルで述べたように、融合タンパク質の発現はまた、抗抗体のHaloTag伝統的なウェスタンブロットを用いて検出することができる、またはそれらが利用可能である場合、ベイトタンパク質に対する抗体。可能ならば、それはより速く、より特異的であるように、代わり蛍光リガンドを使用することを推奨され、より簡単にT漢抗体検出、および10にも定量的。
適切な完全長融合タンパク質の発現が確認された後、タンパク質のプルダウンを行うことができる。 図2(b)に示すように高い再現性を実証する、SDS(議定書第2.4節)で溶出したハロ- BRD4とCtrlプルダウンの生物学的複製物の銀染色ゲルである。銀染色ゲルは、タンパク質のかなりの数が、BRD4タンパク質およびコントロール( 図2B)において非常に低いバックグラウンドと相互作用することが見出される示す。冒頭で述べたように、それが共有結合したままで、溶出のこの過程において、ハロ-BRD4は樹脂から溶出されることはありません。したがって、有意なバンドが銀染色( 図2B)またはウェスタンブロット(データは示さず)で検出され、この分子量では存在しない。これらのタンパク質はBRD4に特異的であるかどうかを決定するために、液体クロマトグラフィー質量分析(LC-MS/MS)がCで行ったomplex混合物を、SDS溶出後に得られた。 図2(c)に示すハロBRD4質量分析で見つかったBRD4 18〜20の既知の相互作用物質に対するスペクトルカウントおよび正規化されたスペクトル豊富因子(NSAF)値です。 pTEFb 18,20からの成分の高い存在ともBRD9 19タンパク質はBRD4複合体の特異的捕捉を確認する。銀染色ゲル( 図2B)によって予測されるように、多数の他のタンパク質も(データは示していない)コントロールでは観察されなかったBRD4の潜在的な相互作用物質として同定された。これらは以前は未知であるので、それらは、それが直接的または間接的にBRD4に関連付けられている場合、タンパク質は、真の相互作用物質であり、そうである場合かどうかを確認するために独立して、他の方法によって検証する必要がある。
孤立した複合体はまた活動のために研究することができる。それは彼らがfunctionaliを維持するようにTEVプロテアーゼ(議定書第2.5節)を用いた複合体を溶出することをお勧めされているTY。 図3Aに、TEVプロテアーゼを用いて樹脂から放出さハロプルダウンHDAC1複合体の銀染色ゲルを示す。 TEVプロテアーゼは、タンパク質融合タグおよびその融合パートナー、ベイトタンパク質の有意な量、この場合にはHDAC1は、観察される( 図3A)との間のリンカー領域で切断するようにこの画がHDAC1活性を含んかどうかを判断するために、溶出したTEVサンプルは発光HDACアッセイは、HDAC-グロ21で試験した。 図3Bに示すように、HDAC1は、プルダウン試料は、HDAC1活性の高レベルの特異公知のHDAC阻害剤によって阻害された(カラム1)、SAHA 22(カラム2)を示した。コントロールは、さらに特異性を実証するために、何のHDAC阻害は、関連するサーチュインファミリー阻害、EX-527 22(カラム3)と、信号が(コラム4)追加されたHDAC1プルダウンサンプルなしで緩衝液のみを使用して検出されなかったと認められなかった。
関数の重要な構成要素らプロテオミクスと理解複合体は、また、タンパク質の局在および/または輸送を理解することです。これらの同一の融合構築物は、細胞内蛍光標識することができるように、我々は、共焦点イメージングを使用してそれらの局在をモニターした。第3節でのプロトコールに従って、一過性にハロ- BRD4( 図4A)およびハロ- HDAC1( 図4B)でトランスフェクションしたHeLa細胞を蛍光TMRリガンドで標識し、画像化した。 図4Aおよび図4Bに示すように、両方の17予想通り核に局在。これらのデータは、タグの存在は、その融合パートナーの生理学的な細胞局在を変化させなかったことを示している。

図1。タンパク質プルダウンおよび共焦点イメージングアプリケーションの回路図のように使用イングル哺乳動物細胞におけるタンパク質機能を理解するための複数のアプリケーションを構築可能である。すべてについて、ハロー融合構築物のいずれかを安定的にまたは一過付着又は懸濁液哺乳動物細胞において発現される。タンパク質複合体のプルダウンのために、細胞を、次いで、複合体は共有結合樹脂上に捕捉され、溶解し、SDS溶出(左経路)又はTEV切断(右経路)のいずれかを介して溶出した。 TEV切断が機能解析を行うための最適な状態でのSDS溶出は、質量分析に進むために推奨されている。発現、細胞局在、トラフィッキングイベント、またはタンパク質の代謝回転を特徴付けるために、融合タンパク質を発現する生細胞を蛍光標識し、さらにSDS PAGEゲル上でまたは共焦点イメージングを用いて分析する。両方の細胞透過性または不透過性の蛍光リガンドは、細胞内の融合タンパク質の局在やプレゼンテーションに応じてご利用いただけます。
igure 2 "FO:コンテンツの幅=" 6インチ "SRC =" / files/ftp_upload/51553/51553fig2highres.jpg "幅=" 600 "/>
図2ハロ- BRD4タンパク質発現、プルダウン、および質量分析。 TMRリガンド(プロトコル2.1節)で標識したHEK293T細胞溶解物中のハロ- BRD4融合タンパク質、189 kDの、単独でハロータンパク質融合タグ、34 kDの、コントロール(CTRL)の発現を示す(A)の A、SDS PAGEゲル。ゲルは検出のためにfluorimagerでスキャンし、蛍光分子量マーカーを使用した。(B)SDSで溶出したハロ- BRD4とCtrlサンプルのプルダウンのための生物学的複製の銀染色ゲル。分子量の大きさは、ゲルのために示されている。(C)スペクトルカウント(左パネル)およびタンパク質ハロ-の生物学的複製物の質量分析で同定されたタンパク質の分子量を占める正規化スペクトル豊富因子(NSAF)の値(右パネル)はBRD4とCtrl。重量が相互作用することが知られている図示タンパク質pTEFb(CDK9およびサイクリンT)18,20だけでなく、BRD9 19のコンポーネントを含むi番目BRD4、。これらのタンパク質からのペプチドは、Ctrlキーで識別されなかった。

図3。ハロ- HDAC1複雑な分離と活動分析。 (A)TEV切断後、Ctrl(議定書第2.5節)からのハロ- HDAC1複合体と背景の分離を示す銀染色ゲル。著名なHDAC1バンド(55 kDa)のとTEVプロテアーゼバンドはラベルが付いています。遊離HDAC1は、樹脂( 図1)。(B)のグラフは、発光ヒストン脱アセチル化酵素アッセイ、HDAC-でHDAC1複合体の単離サンプルの活性を示す上の捕獲後のHaloTag及びHDAC1融合配列の間の最適化されたリンカー内のTEV切断によって生成されるGLO 21。グラフの列1は、高いLを示していますハロ - HDAC1プルダウンサンプル(HDAC1)で含まれているHDAC活性のevels。列2は、この活性は特異的HDAC1プルダウン試料に、HDAC阻害剤、SAHA 22の添加により減少させることができる明らかにする。対照として、第3列は、サーチュインファミリーに特異的な脱アセチル化酵素阻害剤を実証ではなく、HDACは、EX-527 22は 、HDAC1活性を阻害せず、列4は活性は緩衝液のみを用いて観察されないことを示している。
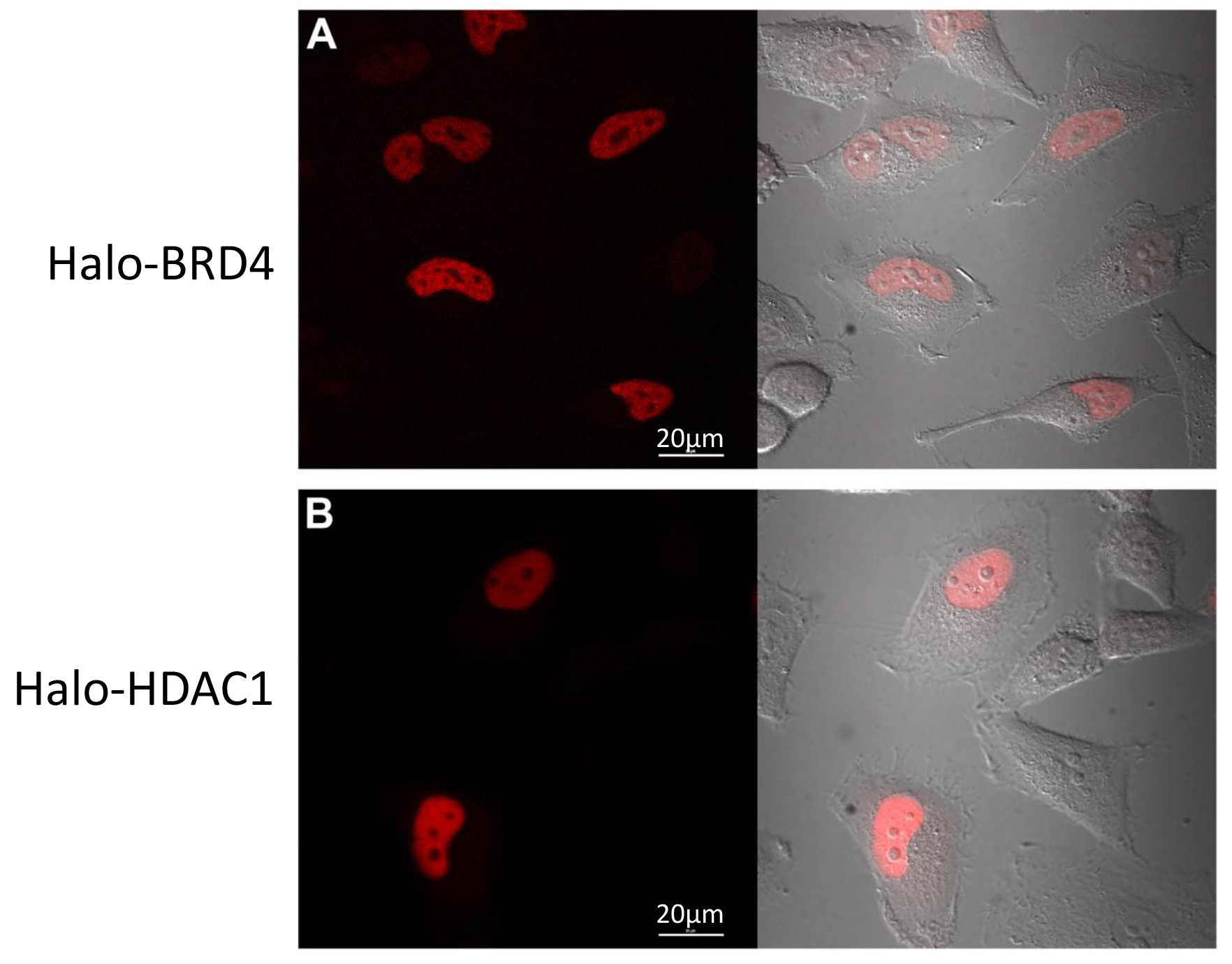
図4ハロBRD4およびハロ-HDAC1共焦点イメージング 、蛍光TMRリガンドで標識されたハロ- BRD4(A)又はハロ-HDAC1(B)でトランスフェクトされたHeLa細胞の細胞の共焦点イメージングをライブ。(A)ハロBRD4発現が制限されている核および(B)ハロHDAC1式に主にNUCですリア。パネルの左側には、蛍光チャネルであり、右側にはそれぞれにDICチャネルと蛍光チャンネルのオーバーレイである。画像は、適切なフィルターセットを用いて、37℃+ CO 2環境チャンバを備えた共焦点顕微鏡で得た。スケールバー=20μmである。
ディスカッション
ここに提示2つの融合タンパク質、ハロ - BRD4およびハロ-HDAC1、発現のための真核生物、哺乳動物細胞を特徴とする、タンパク質複合体の単離および活性、および細胞局在である。これらの様々なプロトコルを介して動作して、各実験の成功のためにいくつかの重要なステップがあります。任意の融合タンパク質の場合のように、タグ自体の発現レベルおよび配置は、生理機能を維持するために重要であることができる。これは、他のタグとの事前の知識又は作業は、選択した特定のタンパク質について実証されていない場合はN-および/またはC末端融合を設計する必要があろうことを考慮することが重要である。レベルが高すぎる発現に関しては、DNAの希釈は、より弱いプロモーターを有するベクターのトランスフェクションまたは使用中に行うことができる適切なレベルを達成することが可能である。以前の研究は、内因性ルでの高分子複合体の効率的な分離を示す行われている式の非常に低いレベルでの作業を可能にする表現8のVELS、。可能であれば、細胞局在の研究はまた、最適なタグの配置位置、並びに適切な生理機能に必要な相対的な発現レベルの洞察を提供することができるであろう。
融合タンパク質は、最大の利点の一つとして、複合体の単離の速度は、それが結合、溶解における推奨される時間枠に追従することは非常に重要であるプロトコル、および洗浄部で最大の成功を得るために、プルダウン実験の準備ができたらプロセス。これらのステップのいずれかが、このような抗体ベースの捕捉方法に必要とされるように、時間的に長くされている場合、複合体の解離または増加し、非特異的結合8の危険性がある。時間は細胞溶解または結合時に短縮された場合も、細胞が完全に溶解されていないか、効率的にそれぞれ捕獲した。洗浄の回数はDECRの場合緩和または樹脂の良好な混合が結合又は洗浄の間に発生せず、非特異的なタンパク質のバックグラウンドレベルが増加する。また、溶解の手段は、樹脂への結合効率に関連するものとして非常に重要です。サンプルを超音波処理推奨される界面活性剤を除去、SDSなどの強い界面活性剤を追加し、および/またはプロテアーゼ阻害剤AEBSFを含むしようとする試みが減少または樹脂に、融合タンパク質およびそれらの複合体の結合の喪失をもたらすであろう。
哺乳動物細胞および質量分析法16による分析のバックグラウンドを減少させるという課題に、人間のプロテオーム解析闘争から複雑な分離のための既存の方法。これは汚染タンパク質のリポジトリは数多くの質量分析グループ16によって作成されたことをとても重要なされています。質量分析は、プルダウン試料中のバックグラウンドは、の識別を防止する何かとして定義することができるベイトタンパク質の真の相互作用因子。このように、背景は、非特異的タンパク質又はもベイトタンパク質又はベイトタンパク質を沈殿させるために使用される抗体の高濃度を汚染から生じることができる。重要な仕事は、プルダウン過程における非特異的汚染タンパク質のレベルを最小限にするためにここに提示され、プルダウンプロトコルだけでなく、樹脂の両方を最適化することで行われていた。これは、銀染色ゲルおよびコントロールの質量分析( 図2)において明らかである。ベイトタンパク質または他の方法が苦労したとの汚染物質なども同様に有害である可能性が高レベルの抗体に対応するために、SDS溶出とプルダウンは(議定書2.4節)をお勧めします。による樹脂への共有結合のために、このプロセスは、樹脂に共有結合し、出発融合タンパク質の大部分を残す。餌の小パーセンテージが考えられている質量分析において観察され加水分解によって発生するが、それは、他のより弱いか、一時的な相互作用8の検出に問題はない。
冒頭で述べたように、プロテオミクスにおける重要な進歩は、質量分析法1,7の大幅な進歩によって可能にされてきた。したがって、プルダウンで得られたタンパク質の混合物をデコンボリュートするために質量分析の選択の重要なパラメータを強調することが重要である。計装は、堅牢かつ日常的かつ効率的に、<1μgのより多くの場合より少ない少量のタンパク質を含む試料を分析することができなければならない。分/ 100〜300 nlの範囲の流量で50〜75ミクロンの内径HPLCカラムを含むナノスケールのクロマトグラフィー、典型的には、小さなサンプルサイズとの互換性のために使用され、質量分析計の感度を最大にする。単一の分析状態Oで取得した情報を最大化する上記のナノスケールの分離と互換性のある時間スケールでの高分解能質量スペクトルを取得することが可能な技術の質量分析計、≥10 Hzのfは、典型的に使用される。これらの器具は、アトモルの感度レベルを有し、日常的に、サブppmの前駆体および生成物イオンの質量許容誤差を有するデータを取得することができる。これらの性能特性は、同定されたタンパク質の数の収率およびそれらの識別子に関連付けられた信頼性を高めるのに役立つ。
小説の相互作用の発見の可能性とともに、タンパク質相互作用、およびすべての単一の構築物を使用したアクティブ複合体の単離:これらのデータと、我々は生理的な細胞内局在、適切なタンパク質を示しています。実際、代替技術は、これらの異なる態様の各々について、しかしそうではない全て23,24のために使用することができる。のHaloTag技術により、多機能的なアプローチはproteomを進め、使用することができますICS研究および哺乳動物細胞におけるタンパク質機能のより完全な理解を得ることができる。
開示事項
この記事のパブリケーション料はプロメガコーポレーションが主催している。 Danette L·ダニエルズ、ジャッキー·メンデス、エレーヌBenink、アンドリュー·ナイルズ、ナンシー·マーフィーとMarjeta URHはプロメガコーポレーションのHaloTag技術とその応用の特許の譲渡による商業者の従業員である。マイケル·フォード、リチャード·ジョーンズ、ラヴィAmunugama、デビッド·アレンは、この原稿に記載された質量分析サービスを提供していMSBioworksの従業員である。
謝辞
私たちは、原稿の重要な読書のためにマーティン·ローゼンバーグ博士ゲイリータープリー、博士とキース·ウッドこの仕事のサポートのために、と博士はジェームズカリに感謝します。 DLD、JM、HB、メキシコ、AN、JC、およびMUはプロメガ社の従業員である。 MF、RJ、RA、およびDAは、MSをBioworks、LLCの従業員である。
資料
| Name | Company | Catalog Number | Comments |
| HeLa Cells | ATCC | CCL-2 | Adherent |
| HEK293T Cells | ATCC | CRL-11268 | Adherent |
| Cellular growth media | Invitrogen/Gibco | ||
| PBS - tissue culture certified | Invitrogen/Gibco | ||
| FuGENE HD Transfection Reagent | Promega | E2311 | |
| HaloTag Control Vector | Promega | G6591 | |
| HaloTag-BRD4 | Kazusa DNA Research Institute | FHC11882 | N-terminal HaloTag fusion |
| HaloTag-HDAC1 | Kazusa DNA Research Institute | FHC02563 | N-terminal HaloTag fusion |
| HaloTag Clones containing content | Promega | Various | http://www.promega.com/FindMyGene/ |
| HaloTag Flexi Vectors for cloning (N- or C-terminal) | Promega | Various | Choose based upon expression level and HaloTag orientation |
| HaloTag TMR Ligand | Promega | G8251 | |
| IGEPAL CA-630 | Sigma | 18896 | |
| HaloTag Mammalian Pull-Down and Labelling System | Promega | G6500 | Contains HaloTag TMR ligand |
| HaloTag Mammalian Pull-Down System | Promega | G6504 | |
| Protease Inhibitor Cocktail, 50X | Promega | G6521 | |
| Mammalian Lysis Buffer | Promega | G9381 | |
| HaloTag Monoclonal Antibody | Promega | G9211 | |
| ProTEV Plus | Promega | V6101 | |
| RQ1 Dnase | Promega | M6101 | |
| HaloLink Resin | Promega | G1912 | |
| HDAC-Glo | Promega | G6420 | |
| PBS + 0.1% Triton X-100 | For optional imaging | ||
| 4% paraformaldehyde/0.2 M sucrose/PBS | For optional imaging | ||
| Ethanol | |||
| Tissue culture vessels and dishes | |||
| Cell culture incubator | |||
| Disposable cell lifter | Thermo Fisher Scientific | 08-773-1 | |
| Glass homogenizer such as Kontes Dounce Tissue Grinder | Thermo Fisher Scientific | K885300-0002 | 25-27 G needle could also be used |
| Microcentrifuge | |||
| Tube rotator such as Labquake | Thermo Fisher Scientific | 4002110Q | Ideal for binding and washing steps |
| Shaking platform such as Eppendorf Thermomixer | Thermo Fisher Scientific | 05-400-200 | Ideal for SDS elution and TEV cleavage steps |
| Chambered cover glass | Thermo Fisher Scientific | 155409 | For optional imaging |
| Fluorescent Microscrope (555 nm Ex/585 nm Em) | For optional imaging |
参考文献
- Altelaar, A. F., Munoz, J., Heck, A. J. Next-generation proteomics: towards an integrative view of proteome dynamics. Nat Rev Genet. 14, 35-48 (2013).
- Tyers, M., Mann, M. From genomics to proteomics. Nature. 422, 193-197 (2003).
- Coulombe, B., et al. Interaction networks of the molecular machines that decode, replicate, and maintain the integrity of the human genome. Mol Cell Proteomics. 3, 851-856 (2004).
- Gavin, A. C., et al. Proteome survey reveals modularity of the yeast cell machinery. Nature. 440, 631-636 (2006).
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440, 637-643 (2006).
- Sardiu, M. E., et al. Probabilistic assembly of human protein interaction networks from label-free quantitative proteomics. Proc Natl Acad Sci U S A. 105, 1454-1459 (2008).
- Domon, B., Aebersold, R. Mass spectrometry and protein analysis. Science. 312, 212-217 (2006).
- Daniels, D. L., et al. Examining the complexity of human RNA polymerase complexes using HaloTag technology coupled to label free quantitative proteomics. J Proteome Res. 11, 564-575 (2012).
- Encell, L. P., et al. Development of a dehalogenase-based protein fusion tag capable of rapid, selective and covalent attachment to customizable ligands. Curr Chem Genomics. 6, 55-71 (2012).
- Ohana, R. F., et al. HaloTag-based purification of functional human kinases from mammalian cells. Protein Expr Purif. 76, 154-164 (2011).
- Black, J. C., et al. KDM4A Lysine Demethylase Induces Site-Specific Copy Gain and Rereplication of Regions Amplified in Tumors. Cell. 154, 541-555 (2013).
- Deplus, R., et al. TET2 and TET3 regulate GlcNAcylation and H3K4 methylation through OGT and SET1/COMPASS. EMBO J. 32, 645-655 (2013).
- Galbraith, M. D., et al. HIF1A Employs CDK8-Mediator to Stimulate RNAPII Elongation in Response to Hypoxia. Cell. 153, 1327-1339 (2013).
- Collins, M. O., Choudhary, J. S. Mapping multiprotein complexes by affinity purification and mass spectrometry. Curr Opin Biotechno. 19, 324-330 (2008).
- Collins, S. R., et al. Toward a comprehensive atlas of the physical interactome of Saccharomyces cerevisiae. Mol Cell Proteomics. 6, 439-450 (2007).
- Mellacheruvu, D., et al. The CRAPome: a contaminant repository for affinity purification-mass spectrometry data. Nat Methods. 10, 730-736 (2013).
- Dawson, M. A., Kouzarides, T. Cancer epigenetics: from mechanism to therapy. Cell. 150, 12-27 (2012).
- Jang, M. K., et al. The bromodomain protein Brd4 is a positive regulatory component of P-TEFb and stimulates RNA polymerase II-dependent transcription. Mol Cell. 19, 523-534 (2005).
- Rahman, S., et al. The Brd4 extraterminal domain confers transcription activation independent of pTEFb by recruiting multiple proteins, including NSD3. Mol Cell Biol. 31, 2641-2652 (2011).
- Yang, Z., et al. Recruitment of P-TEFb for stimulation of transcriptional elongation by the bromodomain protein Brd4. Mol Cell. 19, 535-545 (2005).
- Halley, F., et al. A bioluminogenic HDAC activity assay: validation and screening. J Biomol Screen. 16, 1227-1235 (2011).
- Mack, G. S. To selectivity and beyond. Nat Biotechnol. 28, 1259-1266 (2010).
- Daniels, D. L., Urh, M. Isolation of intracellular protein--DNA complexes using HaloCHIP, an antibody-free alternative to chromatin immunoprecipitation. Methods Mol Biol. 977, 111-124 (2013).
- Urh, M., Rosenberg, M. HaloTag a Platform Technology for Protein Analysis. Curr Chem Genomics. 6, 72-78 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved