Method Article
Die Entdeckung und Charakterisierung von Protein-Wechselwirkungen Protein-Funktion Mit HaloTag Technologie
In diesem Artikel
Zusammenfassung
HaloTag Technologie ist eine Technologie, die multifunktionale bedeutenden Erfolg in Isolation von kleinen und großen Proteinkomplexen aus Säugerzellen gezeigt hat. Hier beleuchten wir die Vorteile dieser Technologie im Vergleich zu bestehenden Alternativen und zeigen ihren Nutzen zu zahlreichen Aspekten der Proteinfunktion innerhalb eukaryotischen Zellen zu studieren.
Zusammenfassung
Forschung in der Proteomik hat in den letzten Jahren Fortschritte in der Massenspektrometrie-Funktionen, die auf die Charakterisierung der zahlreichen Proteomen, auch aus Viren, Bakterien und Hefe führte explodiert. Im Vergleich, die Analyse des menschlichen Proteoms hinkt, teilweise aufgrund der schieren Anzahl von Proteinen, die untersucht werden müssen, sondern auch die Komplexität von Netzwerken und Interaktionen diese vorhanden. Um den Herausforderungen der das Verständnis des menschlichen Proteoms spezifisch anzugehen, haben wir HaloTag Technologie für Proteinisolierung entwickelt, besonders stark für die Isolierung von Multiproteinkomplexen und eine effizientere Erfassung von schwachen oder transiente Wechselwirkungen und / oder Proteine in geringer Menge. HaloTag ist eine genetisch kodierte Protein-Fusion-Tag für die kovalente entworfen, spezifisch und schnelle Immobilisierung oder Markierung von Proteinen mit verschiedenen Liganden. Mit Hilfe dieser Eigenschaften wurden zahlreiche Anwendungen für Säugerzellen zum Charakter entwickeltize Proteinfunktion und hier präsentieren wir Methoden einschließlich: Protein-Pulldown-Menüs für die Entdeckung von neuen Wechselwirkungen oder funktionelle Assays verwendet, und zelluläre Lokalisation. Es ergeben sich signifikante Vorteile in der Geschwindigkeit, Genauigkeit und kovalente Erfassung von Fusionsproteinen zur Proteomanalyse Flächen im Vergleich zu anderen herkömmlichen, nicht-kovalente Ansätze. Wir zeigen diese und die breite Anwendbarkeit der Technologie mit zwei wichtige epigenetische Proteine als Beispiele, das menschliche Protein Bromodomäne BRD4 und Histon-Deacetylase HDAC1. Diese Beispiele zeigen, die Macht dieser Technologie bei, dass die Entdeckung von neuen Interaktionen und Charakterisierung von zellulären Lokalisation in Eukaryoten, die wird, gemeinsam weitere Verständnis der menschlichen funktionelle Proteomik.
Einleitung
Verständnis der zellulären Funktion, die Reaktion auf Reize, und Veränderungen in der Entwicklung und / oder Krankheitszustände ist eng gebunden an de-Faltung des Proteoms in diesen unterschiedlichen dynamischen Zustände 1,2. Es wurde schon viel getan, um das Proteom von niederen Organismen zu verstehen, aber angesichts der Anzahl von Proteinen und alle möglichen Wechselwirkungen hat sich diese sehr herausfordernd bei der Bewältigung der menschlichen Proteoms 1,3-7. Fortschritte in der Massenspektrometrie wurden stark aktiviert die Möglichkeit, diese Untersuchungen durchzuführen, und hier präsentieren wir eine zusätzliche und bedeutende Fortschritte bei der Entwicklung von HaloTag Technologie sowohl für effiziente Erfassung von Proteinkomplexen und funktionelle Charakterisierung von humanen Proteinen aus Säugerzellen 8-10. Diese Technologie wurde kürzlich in mehreren Studien gezeigt worden, daß ein kritischer Faktor für die Entdeckung der wichtigen Wechselwirkungen, so dass für einen Einblick in neue Proteinfunktion und understanding der Erkrankung 11-13.
Die HaloTag Protein-Fusion wurde zunächst auf mehrere Herausforderungen der traditionellen Affinität Tags und Antikörper als im Zusammenhang mit Protein-Capture, Reinigung und Kennzeichnung 10.8 Adresse entwickelt. In nahezu allen Verfahren zur Abtrennung und Reinigung Protein gibt es eine nicht-kovalente Wechselwirkung, die Schritt für die Anreicherung eines bestimmten Proteins 14 verwendet wird. Während dieses Schrittes kann das Protein und / oder komplexe infolge Diffusion trennen, insbesondere wenn das Verfahren erfordert Stunden anstelle von Minuten. Um dieses Problem anzugehen, wurde das Fusionsprotein speziell entwickelt, um schnell und irreversibel die Interaktion mit seinen Liganden, die entweder Teilchen, Flächen oder Fluorophore 9 sein kann. Daher, sobald es an seinen Liganden gebunden bleibt gebunden, was eine der Differenzierungsfaktor im Vergleich zu anderen Affinitätstags ist. Auch hier gezeigt, ist die Fähigkeit,zu unterschiedlichen biologischen Fragen mit einem einzigen Konstrukt, das durch abwechselnde Liganden 9 (Fig. 1) möglich ist, zu adressieren. Zum Beispiel, um Protein-Wechselwirkungen oder Funktion zu befragen, Oberflächenliganden für die Erfassung von Protein oder Komplexe (Fig. 1) verwendet. In einem separaten Experiment, kann das gleiche Fusionsprotein fluoreszenzmarkierten zelluläre Lokalisation, handeln, oder Protein-Turnover (Figur 1) zu untersuchen. Es ist jedoch wichtig zu erinnern, dass, wenn ein Ligand gebunden ist, ob es eine Oberfläche oder ein Fluorophor ist, irreversibel ist und daher andere Liganden kann dann anschließend im gleichen Experiment gebunden.
Analyse bestehender Technologien zur Protein-Interaktionen Karte zeigen die Schwierigkeit, spezifische Wechselwirkungen, einschließlich derjenigen, die mehr transiente oder sind in Hülle und Fülle unteren 1,6,7,14,15 effizient isolieren. Auch neuere Arbeiten von zahlreichen Gruppens zeigt die Sorge, dass innerhalb der traditionellen Isolationsverfahren, insbesondere im Bereich der Menschen Proteomik, gibt es eine beträchtliche Anzahl von Proteinverunreinigungen, die Signale von wahren Interaktoren in der Massenspektrometrie-Analyse 16 maskieren kann. Die für die HaloTag Protein und seine Co-Harz entwickelt Adresse auch diese Bedenken Eigenschaften entwickelt. In 15 Minuten kann das Protokoll für komplexe pulldowns (Abbildung 1), die Bindung von Komplexen erreicht werden, sowohl die Förderung der komplexen Erfassung und Verringerung der Zahl von nicht-spezifischen Bindung 8. Zusätzlich irreversibel bindenden ermöglicht eine effiziente Erfassung von Proteinen mit geringer Häufigkeit und ermöglicht die Verwendung von viel weniger Zellen wieder reduziert Hintergrund 8. Nach Erfassung der Komplexe hergestellt ist, das Protokoll enthält milde Waschbedingungen zu komplexen Integrität zu erhalten. Elution der aufgenommenen Komplexe können durch zwei Verfahren, und die Entscheidung, welche Methode wird durch das Ziel des stromabwärtigen anal diktiert geführt werdenlyse. Wenn der Wunsch zu analysieren oder zu entdecken interagierenden Proteinen durch Western-Blot oder Massenspektrometrie, dann empfiehlt es sich, Komplexe mit Denaturierungsmitteln wie Harnstoff oder SDS (Fig. 1) eluiert. Dies ist für die Massenspektrometrie besonders vorteilhaft, da das Protein ursprünglich HaloTag fusioniert, bezeichnet das Köderprotein, hinter an das Harz gebunden bleiben und daher nicht die Population von Peptiden, die in der Massenspektrometrie nachgewiesen dominieren. Das bedeutet aber auch, dass der Köder-Protein wahrscheinlich nicht sichtbar in der Analyse durch Western-Blot, ein signifikanter Unterschied im Vergleich zu anderen nicht-kovalente Affinitätsreinigungen oder Co-Immunpräzipitationen sein. Wenn das Ziel ist, einen Komplex zu reinigen intakt für Funktionsstudien verwendet werden, können die Elution unter Verwendung von TEV (Tobacco Etch Virus)-Protease, die ihre zugehörigen Spaltsequenz zwischen dem Fusionsprotein und dem Köder-Protein (Fig. 1) codiert werden, erkennt. Tiese Proben könnten auch durch Massenspektrometrie analysiert werden, aber, wie später gezeigt wird, werden sie eine erhebliche Menge an Köderprotein, das den Nachweis von unteren Hülle und Fülle, spezifische Interaktoren verhindern kann, enthalten.
Hier präsentieren wir Pulldown-und Imaging-Protokolle, um zwei wichtige therapeutische Proteine, die Protein Bromodomäne BRD4 und Histon-Deacetylase, HDAC1 17 angelegt. Wir haben mit diesen Beispielen, die Interaktion mit Partnern wie erwartet durch Massenspektrometrie, enzymatische Aktivität nach komplexen Isolation für HDAC1 und korrekte zelluläre Lokalisation für beide bestimmt gezeigt. Zusammen zeigen diese Ergebnisse die multifunktionale Art und Stärke der Technik zur Charakterisierung von eukaryontischen Protein-Wechselwirkungen und Funktion.
Protokoll
Hinweis: Das folgende Protokoll kann mit jedem HaloTag Fusion und in jeder Zelllinie der Wahl stabil oder transient transfizierten verwendet werden. Für diese Beispiele, geben wir Protokolle für die transiente Transfektion von HaloTag Fusionen in HEK293T-oder HeLa-Zellen. Für alle Versuche, empfehlen wir die Verwendung des Proteins nur Kontrolle.
1. Fusion Protein Expression-Test
- Erhalten Fusionsprotein-Konstrukt:
- Erhalten vorgefertigten HaloTag Fusionsvektor oder den Bau der vorhandenen Vektoren. Für weitere Informationen über die verfügbaren Konstrukte finden Sie Tabelle der spezifischen Reagenzien.
- Wenn sie eine Klon, ist es empfehlenswert, die DNA-Sequenz zu überprüfen.
- Testen Expression von Fusionsprotein und Kontrolle:
- Für jede Probe vorbereiten 250 ul 1X SDS Loading Dye-Puffer (60 mM Tris-HCL pH 6,8, 0,75 mM Bromphenol blau, 12,5% Glycerin, 100 mM Dithiothreitol, 0,5% SDS).
- Für jedes Fusionsproteinund Steuerung der Platte 0,5 ml HEK293T oder HeLa-Zellen in den entsprechenden Medien in einer Dichte von 2-4 x 10 5 Zellen / ml in einem Standard-24-Well-Platte.
- Inkubation für 18-24 Stunden bei 37 ° C und 5% CO 2, dann zu transfizieren, wie empfohlen.
- 24 Stunden nach der Transfektion mit 0,5 ul 5 mM Tetramethyl Rhodamin (TMR)-Liganden an die Medien aller Vertiefungen und vorsichtig mischen Platte.
- Inkubieren transfizierte Zellen enthaltenden Liganden für 15 Minuten bei 37 ° C und 5% CO 2.
- Absaugen Medien haltigen Liganden und waschen Sie jedes Well der Zellen vorsichtig mit 1 ml PBS RT.
- Absaugen PBS und führen Sie einen zweiten Waschen mit 1 ml PBS RT.
- PBS entfernen und mit 200 ul 1X SDS-Ladepuffer Farbstoff direkt zu den Zellen.
- Pipette Lysat aus Platte in 1,5-ml-Eppendorf-Röhrchen.
- Sieden Lysat 5 min bei 95 ° C.
- Entfernen von 5-10 ul der Reaktion und Last auf SDS-PAGE-Gel.
- Verwenden Sie ein Fluoreszenzdetektion Scanner(TMR Anregung: 555 nm, Emission: 585 nm), um Bands zu erkennen. Verwenden fluoreszierenden Protein-Marker als Standards.
- Wenn ein Fluoreszenzdetektions Scanner verfügbar ist, führen Western-Blots mit Antikörpern gegen entweder den Köder-Protein-oder Protein-Tag-Fusion, um die Expression des Fusionsproteins.
2. Protein Pulldowns
- Vorbereitung von transient transfizierten Zellen:
- Für jede Fusion oder Steuer bereiten ein 15-cm-Schale mit 30 ml Zellen bei 3-4 x 10 5 Zellen / ml oder 1 bis 1,2 x 10 7 Zellen pro Gesamtkonstrukt.
- Inkubation für 18-24 Stunden bei 37 ° C und 5% CO 2, dann transfizieren konstruieren, wie empfohlen.
- Nach 24 bis 48 Stunden nach der Transfektion, entfernen Sie die Medien und vorsichtig waschen die Zellschicht mit 20-25 ml eiskaltem PBS.
- PBS-Wasch entfernen, dann fügen Sie 25-30 ml 4 ° C gekühlt und vorsichtig abkratzen PBS-Zellen von der Platte.
- Sammeln Sie Zellen in konische Röhrchen und ZentrifugenFuge für 5-10 min bei 2.000 × g und 4 ° C.
- Überstand verwerfen und legen Zellpellet bei -80 ° C für mindestens 30 Minuten oder bis zu 6 Monate.
- Gleichgewichtseinstellung des Halolink Harz:
- Für jede Fusion oder Kontrollprobe, bereiten 12 ml Harz Equilibration / Wash-Puffer (100 mM Tris-HCl pH 7,5, 150 mM NaCl und 0,005% IGEPAL CA-630). Hinweis: Es ist sehr wichtig, und verwenden Sie den Harz Äquilibrationspuffer innerhalb von 12 Stunden als verdünnt IGEPAL CA-630 nicht stabil ist oder wirksam über diesen Zeitrahmen.
- Schütteln oder mischen Harz, um eine gleichmäßige Suspension zu erhalten.
- Für jeden Pull-down-Experiment verzichten 200 ul Harz in einem 1,5 ml Mikrozentrifugenröhrchen.
- Zentrifugieren für 1 min bei 800 xg (z. B. 3000 Upm in einer Mikrozentrifuge), dann sorgfältig entfernen und den Überstand verwerfen (Ethanol) ohne Harz am Boden der Röhre zu stören.
- In 800 ul von Harz Equilibration / Wash-Puffer und gründlich mischen durch Umdrehen des Röhrchens mehrmals.
- Zentrifugieren für 2 min bei 800 g, dann vorsichtig entfernen und den Überstand verwerfen.
- Wiederholen Sie die Schritte 2.2.5 und 2.2.6 zwei weitere Male für insgesamt 3 Waschungen.
- Den letzten Waschen oder Überstand, bis Sie sie unten beschrieben Zelllysaten (Schritt 2.3.8) hinzuzufügen, um das Harz vor dem Austrocknen nicht entfernen.
- Binding und Waschen der Fusionskomplexe:
- Für jede Probe vorzubereiten 500 ul Mammalian Lysepuffer (50 mM Tris-HCl pH 7,5, 150 mM NaCl, 1% Triton X-100, 0,1% Na-Desoxycholat) und 1 ml 1X TBS-Puffer (100 mM Tris-HCl pH 7,5 und 150 mM NaCl).
- Tauen die Zellpellets und resuspendieren in 300 ul Säugetier Lysepuffer durch Auf-und Abpipettieren oder kurz vortexen.
- In 6 ul 50X Protease Inhibitor Cocktail (800 ug / ml Benzamidin HCl, 500 ug / ml Phenanthrolin, 500 ug / ml Aprotinin, 500 ug / ml Leupeptin, 500 & #. 181, g / ml Pepstatin A, 50 mM PMSF) Hinweis: Protease-Cocktails, die AEBSF umfassen kann nicht verwendet werden, da diese stören Protein-Fusion-Tag verbindlich.
- In 3 ul RQ1 DNase und Invertzucker für 10 min bei RT.
- DOUNCE mit Glas-Homogenisator 2,0 ml Größe; . 25-30 Schläge auf Eis, oder übergeben Zellen durch eine 25 oder 27 G-Nadel 5-10 mal, um die Lyse zu vervollständigen Hinweis: Die Ultraschallbehandlung wird nicht als Komplexe zerfallen und Überhitzung kann die Protein-Fusion-Tag-Aktivität beeinflussen empfohlen.
- Zentrifuge bei 14.000 × g für 5 min bei 4 ° C, um das Lysat zu löschen.
- Übertragen klare Lysat, ca. 300 ul Gesamtvolumen, neue Röhre und auf Eis.
- In den Klarlysat zusätzliche 700 ul 1X TBS-Puffer und gut mischen durch Pipettieren von oben und unten.
- Nehmen äquilibriertem Harzrohre in Schritt 2.2.8 hergestellt und entfernen letzten Wasch / Stand aus Harz ohne Harz am Boden der Röhre zu stören.
- In den einzelnen tusein von Harz, die 1 ml verdünntem Lysat.
- Inkubiere unter Mischen auf einem Schwenker (oder sanft Mischer) für 15 min bei 22 º C Anmerkung: Das Absetzen des Harzes während dieser Zeit reduziert Bindungseffizienz. Wenn Bindung bei 4 º C gewünscht ist, dies zu tun, indem man 1 Stunde lang.
- Centrifuge Harzrohre für 2 min bei 800 xg und Überstand verwerfen.
- 1 ml Resin Equilibration / Wash-Puffer bei Schritt 2.2.1 gemacht und mischen durch Invertieren des Harzschlauch von Hand mehrmals.
- Centrifuge Harzrohre für 2 min bei 800 x g und entsorgen Sie die Wäsche.
- Wiederholen Sie die Schritte, gefolgt von 2.3.12 2.3.14 drei weitere Male.
- 1 ml Harz Äquilibrierung / Waschpuffer und Inkubation bei 22 ° C für 5 min mit konstanter Drehzahl.
- Centrifuge Harzrohre für 2 min bei 800 x g und entsorgen Sie die Wäsche.
- Je nach Anwendungszweck (siehe Einleitung für weitere Erläuterungen), um entweder Abschnitt 2.4 oder 2.5 gehen.
- SDS Elution zur Denaturierung Gelen, Western Blots, oder Massenspektrometrie:
- Resuspendieren des Harzes aus jeder Probe in 50 ul SDS-Elutionspuffer (1% SDS und 50 mM Tris-HCl pH 7,5)
- Schütteln Röhrchen bei RT für 30 min.
- Zentrifuge für 2 min bei 800 xg und Transfer Eluate in frische Röhrchen für die Analyse.
- Für Western-Blot-oder Silberfärbung Gel, Last 5-10 ul auf einem SDS Denaturierungsgel.
- Für Massenspektrometrie, sparen Sie 40 ul jeder Probe bei -20 ° C
- TEV-Protease Elution für funktionelle Assays, Western Blots, oder Massenspektrometrie:
- Nach dem Entfernen der letzten Waschung Resuspendieren des Harzes in 50 ul ProTEV Spaltpuffer und 30 Einheiten Enzym TEV.
- Inkubieren bei 25 ° C unter Schütteln für 1 Stunde.
- Zentrifuge für 2 Minuten bei 800 x g.
- Übertragen Eluat in frische Röhrchen.
- Bewahren Sie die Probe bei 4 ° C für den sofortigen Einsatz in funktionellen Tests.
3.Cellular Imaging von fluoreszenz markierter Fusionsproteine Mit einem zytoplasmatische und nukleäre Permeable Ligand
- Transfektion, Bezeichnung und Bildzellen:
- In einem 8-Well-Kammerdeckgläser für jedes Fusionsprotein oder Steuerung, Platte 400 ul von HeLa-Zellen in den entsprechenden Medien in jede Vertiefung in einer Dichte von 1-2 x 10 5 Zellen / ml.
- Inkubation für 18-24 Stunden bei 37 ° C und 5% CO 2, dann zu transfizieren, wie empfohlen.
- 18-24 Stunden nach der Transfektion, verdünnte TMR Ligand 1:200 in geeigneten Zell Medien, dann werden 100 ul dieser Lösung in jede Vertiefung und vorsichtig mischen.
- Inkubieren transfizierte Zellen enthaltenden Liganden für 15 Minuten bei 37 ° C und 5% CO 2.
- Absaugen Medien-Liganden und ersetzen mit 500 ul geeigneten Medien fehlt Protein-Fusion-Tag-Liganden, die seit bis 37 ° C erwärmt worden enthält
- Wiederholen zweimal für insgesamt drei Waschungen.
- Setzen Zellen zurück in Inkubator (376, C und 5% CO 2) für 30 min.
- Absaugen Medien und ersetzen mit 500 ul geeigneten Medien, das wurde auf 37 ° C erwärmt worden
- Bild auf einem Mikroskop mit geeigneten Aufnahmeparameter (TMR Anregung: 555 nm, Emission: 585 nm).
Ergebnisse
Beim Arbeiten mit jeder neuen Fusionsprotein, ist es wichtig, zuerst Test für die Expression des Proteins nach Transfektion und auch bestätigen, dass ein Protein der korrekten Molekulargewicht erzeugt wird. Als HaloTag Fusionsproteine können fluoreszierend und kovalent mit durchlässigen oder je nach Lokalisation undurchlässig Liganden markiert werden, ist es möglich, schnell zu bestimmen, indem die Expression Zellysaten denaturierende Gelelektrophorese, gefolgt von einer Abtastung auf fluorimager. Unter Verwendung der in Abschnitt 1.2, Ausdruck der Halo-BRD4 (189 kD) und dem Steuer HaloTag allein (Strg) beobachtet (34 kD, 2A) beschriebenen Protokoll. Wie im Protokoll erwähnt, kann die Expression von Fusionsproteinen auch mit herkömmlichen Western-Blots mit Anti-HaloTag Antikörper nachgewiesen werden, oder wenn sie vorhanden sind, Antikörper zu dem Köderprotein. Wenn möglich, ist es empfehlenswert, den fluoreszierenden Liganden statt dessen, wie es präziser, schneller und einfacher than-Antikörper-Nachweis und auch quantitative 10.
Nach dem Ausdruck der richtigen voller Länge Fusionsprotein verifiziert ist, können Protein pulldowns durchgeführt werden. In Abbildung 2B sind die Silber-gefärbten Gelen der biologischen Replikate von Halo-BRD4 und Strg pulldowns durch SDS (Protokoll Abschnitt 2.4) eluiert, die eine hohe Reproduzierbarkeit zu demonstrieren. Die Silberfärbung Gele zeigen eine signifikante Anzahl von Proteinen zu finden sind, mit der BRD4 Protein und sehr niedrigen Hintergrund in der Kontrolle (2B) interagieren. Wie in der Einleitung erwähnt, in diesem Prozess der Elution, die Halo-BRD4 nicht aus dem Harz eluiert werden, sie bleibt kovalent gebunden sind. Daher gibt es keine signifikante Bande bei diesem Molekulargewicht in der Silberfärbung (Fig. 2B) oder Western Blot (Daten nicht gezeigt) nachgewiesen. Zu bestimmen, ob diese Proteine spezifisch BRD4 wurde Flüssigchromatographie-Massenspektrometrie (LC-MS/MS) auf der C durchgeführtomplexe Mischung nach SDS-Elution erhalten. In Abbildung 2C sind spektrale zählt und normierte spektrale Fülle Faktorwerte (NSAF) für bekannte Interaktoren von BRD4 18-20 in der Halo-BRD4 Massenspektrometrie-Analyse gefunden. Die hohe Fülle von Komponenten aus pTEFb 18,20 und auch die BRD9 19 Protein bestätigen spezifische Erfassung von BRD4 Komplexen. Wie durch die Silberfärbung Gele (2B) vorhergesagt wurden zahlreiche andere Proteine als potentielle Interaktionspartner von BRD4, die nicht in der Kontrolle (Daten nicht gezeigt) beobachtet wurden identifiziert. Da diese bisher unbekannte, müssen sie unabhängig von anderen Methoden überprüft werden, um zu bestätigen, ob das Protein eine wahre Interaktor, und wenn ja, ob sie direkt oder indirekt mit BRD4 verbunden.
Isolierte Komplexe auch für die Aktivität untersucht werden; Es wird empfohlen, um Komplexe zu eluieren mit TEV-Protease (Protokoll Abschnitt 2.5), so dass sie Funktionalitäten zu erhaltenty. In Fig. 3A eine Silberfärbung des Gels Halo-HDAC1-Pulldown-Komplexe aus dem Harz unter Verwendung von TEV-Protease freigesetzt wird, gezeigt. Als TEV-Protease wird in einem Linkerbereich zwischen dem Fusionsprotein Tag und seinem Fusionspartner, signifikante Mengen des Köder-Proteins, in diesem Fall HDAC1, beobachtet (Fig. 3A) zu spalten. Um festzustellen, ob diese Fraktion HDAC1 Aktivität wurden eluiert TEV Proben in einem lumineszierenden HDAC-Assay HDAC-Glo 21 getestet. Wie in 3B gezeigt ist, zeigte HDAC1 Pulldown Proben hohe Aktivität HDAC1 (Spalte 1), die spezifisch mit einem bekannten HDAC-Inhibitor, SAHA 22 (Spalte 2) gehemmt wurde. Als Kontrollen weiter Spezifität zu demonstrieren, wurde keine HDAC-Inhibition mit einer verwandten Familie Sirtuin-Inhibitor, EX-527 22 (Spalte 3) und kein Signal wurde mit Puffer allein ohne die HDAC1 Pulldown-Probe gegeben (Spalte 4) erkannt beobachtet.
Ein wesentlicher Bestandteil der Funktional Proteomik und Verständnis Komplexe, ist auch das Verständnis Proteinlokalisierung und / oder Menschenhandel. Da diese gleiche Fusionskonstrukte können fluoreszenz innerhalb der Zellen markiert werden, überwacht wir deren Lokalisierung mittels konfokaler Bildgebung. Nach dem Protokoll in Abschnitt 3 wurden HeLa-Zellen transient mit Halo-BRD4 (4A) und Halo-HDAC1 (4B) transfiziert fluoreszenz mit der TMR-Liganden markiert und abgebildet. Wie in den 4A und 4B gezeigt, sowohl in den Kern, wie erwartet 17 lokalisiert. Diese Daten zeigen, dass die Anwesenheit von Tag nicht physiologischen zellulären Lokalisierung der Fusionspartner zu ändern.

Abbildung 1. Schematische Darstellung der Protein-Pulldown und konfokalen Imaging-Anwendungen. Verwendung als ingle konstruieren mehrere Anwendungen für das Verständnis der Proteinfunktion in Säugetierzellen sind möglich. Für alle, ein Halo-Fusionskonstrukt entweder stabil oder transient in adhärenten oder Suspension Säugerzellen exprimiert. Für Protein-Komplex pulldowns werden die Zellen lysiert, Komplexe kovalent an Harz eingefangen und entweder durch SDS-Elution (linke Bahn) oder TEV-Spaltung (rechte Weg) eluiert. SDS Elution wird für Sie fortfahren Massenspektrometrie-Analyse empfohlen, während TEV-Spaltung ist optimal für die Durchführung der Funktionsanalyse. Um die Expression, zelluläre Lokalisation, Menschenhandel Ereignisse oder Proteinumsatz zu charakterisieren, sind Live-Zellen, die Fusionsproteine fluoreszenzmarkierten und weiter SDS-PAGE-Gele oder mittels konfokaler Bildgebung analysiert. Beide Zelllässig oder undurchlässig fluoreszierenden Liganden verfügbar sind, abhängig von der Lokalisierung oder Darstellung des Fusionsproteins in der Zelle.
ild 2 "fo: content-width =" 6in "src =" / files/ftp_upload/51553/51553fig2highres.jpg "width =" 600 "/>
Abbildung 2. Halo-BRD4 Proteinexpression, Pull-Down-und Massenspektrometrie-Analyse. (A) Eine SDS-PAGE-Gelen, welche die Expression von Halo-BRD4 Fusionsprotein, 189 kD und Halo-Tag-Fusionsprotein allein, 34 kD, Steuerung (Strg) innerhalb eines HEK293T Zelllysat mit TMR Ligand (Protokoll Abschnitt 2.1) gekennzeichnet. Die Gele wurden mit fluorimager zur Erkennung gescannt und eine Leuchtstoffmolekulargewichtsmarker verwendet wurde. (B) Silberfärbung Gele von biologischen Wiederholungen für pulldowns von Halo-BRD4 und Strg Proben mit SDS eluiert. Molekulargewichtsgrößen für das Gel Spectral Zahlen (links) und der normierten spektralen Fülle Faktoren Werte (NSAF) (rechts), die für Protein-Molekulargewicht der Proteine in der Massenspektrometrie-Analyse von biologischen Replikaten Halo identifizierten Konto angegeben. (C) BRD4 und Strg. Dargestellt sind Proteine bekannt, w interagierenith BRD4, einschließlich Komponenten pTEFb (CDK9 und Cyclin T) 18,20 sowie BRD9 19. Keine Peptide aus dieser Proteine wurden in der Strg identifiziert.

Abbildung 3. Halo-HDAC1-Komplex Isolierung und Aktivitätsanalyse. (A) Silberfärbung Gele, die die Isolierung von Halo-HDAC1 Komplexe und Hintergrund aus der Ctrl nach TEV-Spaltung (Protokoll Abschnitt 2.5). Der prominente HDAC1 Band (55 kDa) und der TEV-Protease-Band markiert sind. Die kostenlose HDAC1 wird durch TEV-Spaltung innerhalb einer optimierten Linker zwischen dem HaloTag und HDAC1 Fusionssequenz nach der Aufnahme auf dem Harz (Abbildung 1). (B) Die Grafik zeigt die Aktivität von HDAC1-Komplex Isolierungen Proben in einem lumineszierenden Histon-Deacetylase-Assay HDAC-generierte Glo 21. Spalte 1 der Graph zeigt hohe lEvels von HDAC-Aktivität mit den Halo-HDAC1 Pulldown-Proben (HDAC1) enthalten ist. Spalte 2 zeigt, kann diese Aktivität spezifisch durch Zugabe der HDAC-Inhibitor, SAHA 22 verringert werden, um den Pull-Down-HDAC1 Proben. Als Kontrollen Spalte 3 zeigt eine Deacetylase-Inhibitor spezifisch für die Sirtuin-Familie, aber nicht HDACs, EX-527 22, nicht HDAC1-Aktivität und Spalte 4 keine Aktivität mit Puffer allein beobachtet hemmen.
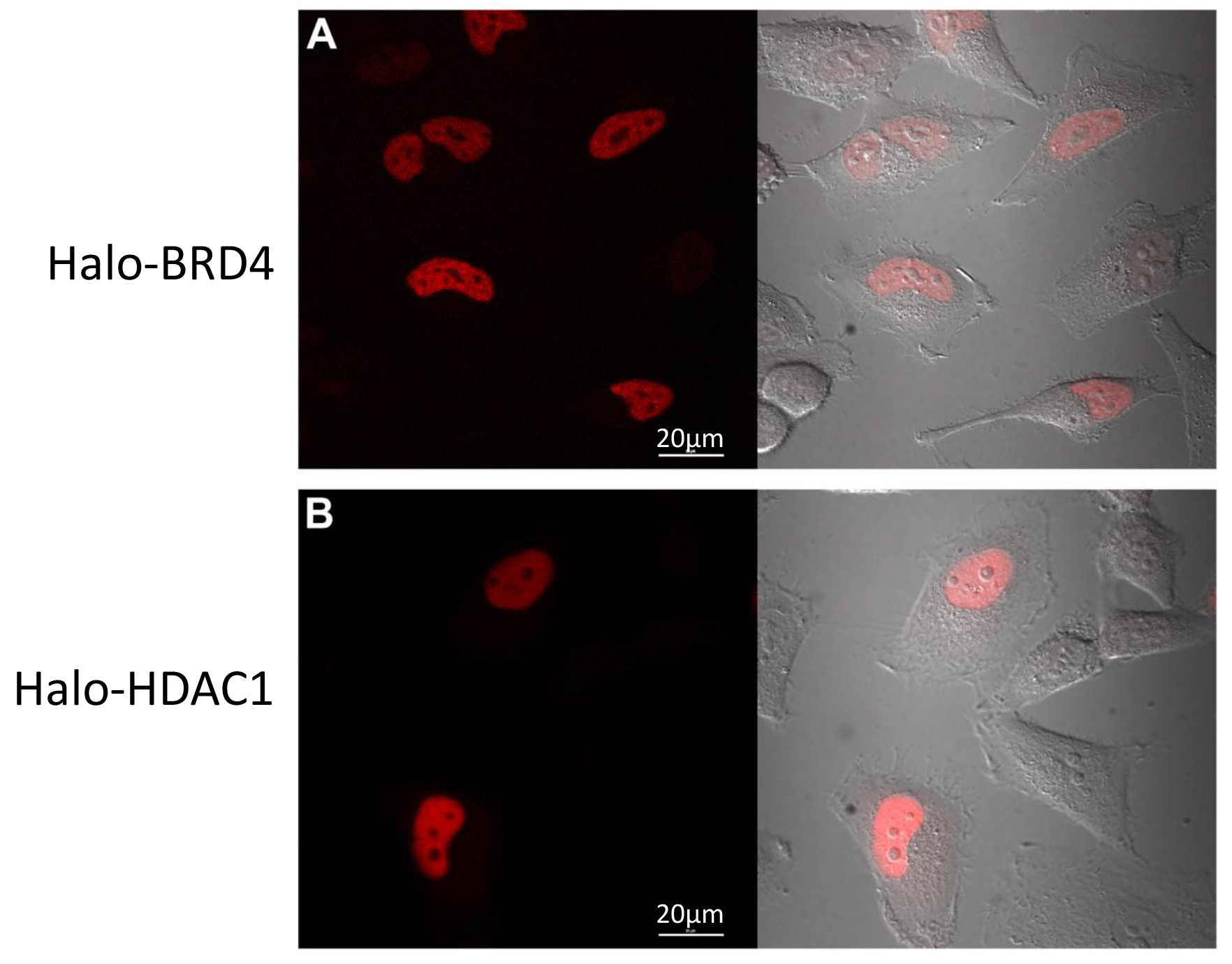
4. Halo-BRD4 und Halo-HDAC1 konfokale Bildgebung. Lebendzell konfokale Bildgebung von HeLa-Zellen mit Halo-BRD4 (A) oder Halo-HDAC1 (B) mit TMR fluoreszierend markierten Liganden transfiziert. (A) Halo-BRD4 Expression eingeschränkt zum Kern und (B) Halo-HDAC1 Expressions überwiegend nucLear. Linke Seite des Panels fluoreszierend Kanal und rechts ist eine Überlagerung der Fluoreszenzkanal mit dem Kanal für jeden DIC. Bilder wurden mit einem konfokalen Mikroskop mit einem 37 ° C + CO 2 Klimakammer mit entsprechenden Filtersätzen ausgestattet erworben. Maßstabsbalken = 20 um.
Diskussion
Präsentiert werden hier zwei Fusionsproteine, Halo-Halo-BRD4 und HDAC1, in eukaryotischen Säugetierzellen für die Expression gekennzeichnet, Protein-Komplex Isolation und Aktivität und zelluläre Lokalisation. In Arbeits durch diese verschiedenen Protokolle gibt es mehrere wichtige Schritte für den Erfolg eines jeden Versuchs. Wie es der Fall bei jedem Fusionsprotein, Expressionsniveau und die Platzierung der Markierung selbst kann für die Aufrechterhaltung Physiologie. Daher ist es wichtig zu berücksichtigen, dass N-und / oder C-terminale Fusionen müssen entwickelt werden, wenn Vorkenntnisse oder arbeiten mit einem anderen Tag wurde nicht für das jeweilige Protein der Wahl gezeigt. In Bezug auf Ausdruck, wenn der Pegel zu hoch ist, Verdünnung der DNA während der Transfektion oder Verwendung von Vektoren mit schwächeren Promotoren durchgeführt werden, werden möglich, die Höhe, die angemessen ist zu erreichen. Frühere Arbeiten durchgeführt worden ist, die effiziente Isolierung von makromolekularen Komplexen bei endogenen levels Ausdrucks 8, wodurch Arbeit bei sehr niedrigen Expressionsniveaus. Wenn möglich, würde zelluläre Lokalisation Studien auch in der Lage, Einblick in die optimale Platzierung Tag-Position als auch relative Expressionsniveau für die richtige Physiologie benötigt bereitzustellen.
Sobald die Fusionsproteine sind bereit für Pulldown-Experimente, um den maximalen Erfolg mit dem Protokoll ist es sehr wichtig, die empfohlenen Zeitrahmen bei der Lyse folgen, Bindung und Wasch Abschnitte zu erhalten, als einer der größten Vorteile ist die Geschwindigkeit des Komplexes Isolation Prozess. Wenn einer dieser Schritte werden in der Zeit verlängert wird, wie für einen Antikörper-basierten Erfassungsverfahren erforderlich ist, besteht die Gefahr von komplexen Dissoziation oder erhöhte nicht-spezifische Bindung 8. Ebenso, wenn die Zeiten während der zellulären Lyse oder verbindlich sind verkürzt, können die Zellen nicht vollständig lysiert werden oder effizient bzw. gefangen genommen. Wenn die Anzahl der Waschungen ist decrerleichtert bzw. gute Durchmischung des Harzes nicht in der Bindung oder Wasch auftreten, dann Hintergrundwerte von nicht-spezifischen Proteinen erhöht. Außerdem ist das Mittel der Lyse sehr wichtig, da im Zusammenhang mit der Bindungsaffinität zu dem Harz. Versuche, um die Proben zu beschallen, entfernen Sie das empfohlene Reinigungsmittel, fügen SDS oder andere starke Reinigungsmittel und / oder die den Protease-Inhibitor AEBSF wird reduziert oder Verlust der Bindung von Fusionsproteinen und deren Komplexen zu dem Harz führen.
Bestehende Verfahren für komplexe Isolierung aus Säugerzellen und humane Proteom-Analyse Kampf mit der Herausforderung der Reduzierung von Hintergrund in der Analyse durch Massenspektrometrie 16. Dies wurde so bedeutend, dass ein Repository von verunreinigenden Proteinen wurde von zahlreichen Gruppen-Massenspektrometrie 16 erstellt worden war. Hintergrund in der Massenspektrometrie Pulldown-Proben können als etwas, die die Identifizierung der verhindert, dass definiert werdenwahre Interaktoren der Köder-Protein. Als solches kann Hintergrund kontaminieren unspezifische Proteine oder auch hohe Konzentrationen von Köderprotein oder Antikörper verwendet werden, um Proteine auszufällen Köder entstehen. Bedeutende Arbeit wurde bei der Optimierung sowohl die Pulldown-Protokoll hier vorgestellten sowie das Harz, um das Niveau der nicht-spezifischen verunreinigende Proteine im Pulldown-Verfahren minimieren getan. Dies ist in der Silberfärbung Gele und Massenspektrometrie-Analyse der Kontrolle (Fig. 2) deutlich. Um das hohe Niveau der Köderprotein oder Antikörper, die ebenso schaden wie Verunreinigungen und mit welchen anderen Methoden kämpfen kann anzugehen, wird das Pulldown-Elution mit SDS empfohlen (Protokoll Abschnitt 2.4). Aufgrund der kovalenten Bindung an das Harz wird dieser Prozess die Mehrheit der Ausgangsfusionsprotein kovalent an das Harz gebunden verlassen. Ein kleiner Prozentsatz der Köder in der Massenspektrometrie-Analyse, von der angenommen wird beobachtetdurch Hydrolyse auftreten, aber es ist kein Problem für den Nachweis von anderen schwächer oder transiente Interaktionen 8 vorhanden.
Wie in der Einleitung erwähnt, wurden bedeutende Fortschritte in der Proteomik durch wesentliche Fortschritte in der Massenspektrometrie 1,7 aktiviert. Daher ist es wichtig, wichtige Parameter der Wahl der Massenspektrometrieanalyse zu markieren, um die Mischung von Proteinen in den pulldowns erhalten Dekonvolution. Instrumentierung muss robust und in der Lage regelmäßig und effizient Analyse von Proben, die eine kleine Menge an Protein, die oft weniger als <1 ug. Nanoscale-Chromatographie mit 50-75 um Innendurchmesser HPLC-Säulen mit Durchflussraten in der 100-300 nl / min Bereich ist in der Regel für die Kompatibilität mit der kleinen Stichprobengrößen beschäftigt und um die Empfindlichkeit des Massenspektrometers zu maximieren. Um die in einer einzigen Analyse Zustand o gewonnenen Informationen zu maximierenf die Kunst Massenspektrometer zur Akquisition hochauflösende Massenspektren auf einer Zeitskala mit oben genannten nanoskaligen Trennungen kompatibel, ≥ 10 Hz, werden typischerweise verwendet. Diese Instrumente haben attomolaren Empfindlichkeitsstufen und kann routinemäßig Daten mit Sub-ppm-Vorläufer und Produkt-Ionen-Massen-Fehlertoleranzen zu erwerben. Diese Leistungseigenschaften dienen, um die Ausbeute von der Anzahl der identifizierten Proteine und das Vertrauen mit diesen verbundenen Identifikationen zu erhöhen.
Mit diesen Daten belegen wir physiologische zelluläre Lokalisation, richtige Protein-Protein-Wechselwirkungen mit Potenzial für die Entdeckung von neuen Interaktionen und Isolierung von aktiven Komplexen alle mit einem einzigen Konstrukt. Tatsächlich alternative Technologien für jede dieser verschiedenen Aspekte verwendet werden, aber wahrscheinlich nicht für alle 23,24. Mit der HaloTag Technologie kann eine multifunktionale Ansatz verwendet werden, die Förderung ProteomICs Studien und Erhalten eines besseren Verständnis der Funktion von Proteinen in Säugerzellen.
Offenlegungen
Veröffentlichung Kosten für diesen Artikel werden von Promega Corporation gesponsert. Danette L. Daniels, Jacqui Méndez, Hélène Benink, Andrew Niles, Nancy Murphy und Marjeta Urh sind Mitarbeiter von Promega Corporation, Handels Inhaber durch Abtretung von Patenten des HaloTag Technologie und ihrer Anwendungen. Michael Ford, Richard Jones, Ravi Amunugama und David Allen sind Mitarbeiter von MSBioworks, die Masse in diesem Manuskript beschrieben Spektrometrie Dienstleistungen bietet.
Danksagungen
Wir danken Dr. Martin Rosenberg, Dr. Gary Tarpley, und Dr. Keith Wood für die Unterstützung dieser Arbeit und Dr. James Cali für das kritische Lesen des Manuskripts. DLD, JM, HB, NM, AN, JC, und MU sind Mitarbeiter von Promega Corporation. MF, RJ, RA, und DA sind Mitarbeiter von MS Bioworks, LLC.
Materialien
| Name | Company | Catalog Number | Comments |
| HeLa Cells | ATCC | CCL-2 | Adherent |
| HEK293T Cells | ATCC | CRL-11268 | Adherent |
| Cellular growth media | Invitrogen/Gibco | ||
| PBS - tissue culture certified | Invitrogen/Gibco | ||
| FuGENE HD Transfection Reagent | Promega | E2311 | |
| HaloTag Control Vector | Promega | G6591 | |
| HaloTag-BRD4 | Kazusa DNA Research Institute | FHC11882 | N-terminal HaloTag fusion |
| HaloTag-HDAC1 | Kazusa DNA Research Institute | FHC02563 | N-terminal HaloTag fusion |
| HaloTag Clones containing content | Promega | Various | http://www.promega.com/FindMyGene/ |
| HaloTag Flexi Vectors for cloning (N- or C-terminal) | Promega | Various | Choose based upon expression level and HaloTag orientation |
| HaloTag TMR Ligand | Promega | G8251 | |
| IGEPAL CA-630 | Sigma | 18896 | |
| HaloTag Mammalian Pull-Down and Labelling System | Promega | G6500 | Contains HaloTag TMR ligand |
| HaloTag Mammalian Pull-Down System | Promega | G6504 | |
| Protease Inhibitor Cocktail, 50X | Promega | G6521 | |
| Mammalian Lysis Buffer | Promega | G9381 | |
| HaloTag Monoclonal Antibody | Promega | G9211 | |
| ProTEV Plus | Promega | V6101 | |
| RQ1 Dnase | Promega | M6101 | |
| HaloLink Resin | Promega | G1912 | |
| HDAC-Glo | Promega | G6420 | |
| PBS + 0.1% Triton X-100 | For optional imaging | ||
| 4% paraformaldehyde/0.2 M sucrose/PBS | For optional imaging | ||
| Ethanol | |||
| Tissue culture vessels and dishes | |||
| Cell culture incubator | |||
| Disposable cell lifter | Thermo Fisher Scientific | 08-773-1 | |
| Glass homogenizer such as Kontes Dounce Tissue Grinder | Thermo Fisher Scientific | K885300-0002 | 25-27 G needle could also be used |
| Microcentrifuge | |||
| Tube rotator such as Labquake | Thermo Fisher Scientific | 4002110Q | Ideal for binding and washing steps |
| Shaking platform such as Eppendorf Thermomixer | Thermo Fisher Scientific | 05-400-200 | Ideal for SDS elution and TEV cleavage steps |
| Chambered cover glass | Thermo Fisher Scientific | 155409 | For optional imaging |
| Fluorescent Microscrope (555 nm Ex/585 nm Em) | For optional imaging |
Referenzen
- Altelaar, A. F., Munoz, J., Heck, A. J. Next-generation proteomics: towards an integrative view of proteome dynamics. Nat Rev Genet. 14, 35-48 (2013).
- Tyers, M., Mann, M. From genomics to proteomics. Nature. 422, 193-197 (2003).
- Coulombe, B., et al. Interaction networks of the molecular machines that decode, replicate, and maintain the integrity of the human genome. Mol Cell Proteomics. 3, 851-856 (2004).
- Gavin, A. C., et al. Proteome survey reveals modularity of the yeast cell machinery. Nature. 440, 631-636 (2006).
- Krogan, N. J., et al. Global landscape of protein complexes in the yeast Saccharomyces cerevisiae. Nature. 440, 637-643 (2006).
- Sardiu, M. E., et al. Probabilistic assembly of human protein interaction networks from label-free quantitative proteomics. Proc Natl Acad Sci U S A. 105, 1454-1459 (2008).
- Domon, B., Aebersold, R. Mass spectrometry and protein analysis. Science. 312, 212-217 (2006).
- Daniels, D. L., et al. Examining the complexity of human RNA polymerase complexes using HaloTag technology coupled to label free quantitative proteomics. J Proteome Res. 11, 564-575 (2012).
- Encell, L. P., et al. Development of a dehalogenase-based protein fusion tag capable of rapid, selective and covalent attachment to customizable ligands. Curr Chem Genomics. 6, 55-71 (2012).
- Ohana, R. F., et al. HaloTag-based purification of functional human kinases from mammalian cells. Protein Expr Purif. 76, 154-164 (2011).
- Black, J. C., et al. KDM4A Lysine Demethylase Induces Site-Specific Copy Gain and Rereplication of Regions Amplified in Tumors. Cell. 154, 541-555 (2013).
- Deplus, R., et al. TET2 and TET3 regulate GlcNAcylation and H3K4 methylation through OGT and SET1/COMPASS. EMBO J. 32, 645-655 (2013).
- Galbraith, M. D., et al. HIF1A Employs CDK8-Mediator to Stimulate RNAPII Elongation in Response to Hypoxia. Cell. 153, 1327-1339 (2013).
- Collins, M. O., Choudhary, J. S. Mapping multiprotein complexes by affinity purification and mass spectrometry. Curr Opin Biotechno. 19, 324-330 (2008).
- Collins, S. R., et al. Toward a comprehensive atlas of the physical interactome of Saccharomyces cerevisiae. Mol Cell Proteomics. 6, 439-450 (2007).
- Mellacheruvu, D., et al. The CRAPome: a contaminant repository for affinity purification-mass spectrometry data. Nat Methods. 10, 730-736 (2013).
- Dawson, M. A., Kouzarides, T. Cancer epigenetics: from mechanism to therapy. Cell. 150, 12-27 (2012).
- Jang, M. K., et al. The bromodomain protein Brd4 is a positive regulatory component of P-TEFb and stimulates RNA polymerase II-dependent transcription. Mol Cell. 19, 523-534 (2005).
- Rahman, S., et al. The Brd4 extraterminal domain confers transcription activation independent of pTEFb by recruiting multiple proteins, including NSD3. Mol Cell Biol. 31, 2641-2652 (2011).
- Yang, Z., et al. Recruitment of P-TEFb for stimulation of transcriptional elongation by the bromodomain protein Brd4. Mol Cell. 19, 535-545 (2005).
- Halley, F., et al. A bioluminogenic HDAC activity assay: validation and screening. J Biomol Screen. 16, 1227-1235 (2011).
- Mack, G. S. To selectivity and beyond. Nat Biotechnol. 28, 1259-1266 (2010).
- Daniels, D. L., Urh, M. Isolation of intracellular protein--DNA complexes using HaloCHIP, an antibody-free alternative to chromatin immunoprecipitation. Methods Mol Biol. 977, 111-124 (2013).
- Urh, M., Rosenberg, M. HaloTag a Platform Technology for Protein Analysis. Curr Chem Genomics. 6, 72-78 (2012).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten