Extracción de ADN de la comunidad de colonias bacterianas
Visión general
Fuente: Laboratorios del Dr. Ian Pepper y el Dr. Charles Gerba - Universidad de Arizona
Demostrando autor: Luisa Ikner
Métodos tradicionales de análisis de comunidades microbianas en suelos generalmente han implicado o ensayos culturales utilizando la dilución y metodología en medios selectivos y diferenciales o ensayos de recuento directo de la galjanoplastia. Conteo directo ofrece información sobre el número total de bacterias presentes, pero ninguna información sobre el número o la diversidad de las poblaciones presentes en la comunidad. Cuenta de placa permite enumeración total cultural o las poblaciones culturales y por lo tanto, proporciona información sobre las diferentes poblaciones presentes. Sin embargo, dado que menos del 1% de bacterias del suelo son fácilmente cultivable, información cultural ofrece sólo un pedazo de la imagen. La fracción real de la comunidad que puede ser cultivada depende el medio elegido para cuentas culturales. Selecciona cualquier medio solo para las poblaciones que mejor se adapten a ese medio particular.
En los últimos años han puesto de manifiesto las ventajas de estudiar la comunidad ADN extraído de muestras de suelo. Este enfoque basado en el nonculture se piensa para ser más representativo de la comunidad real presente que los enfoques basados en la cultura. Además de proporcionar información sobre los tipos de poblaciones presentes, este enfoque también puede proporcionar información acerca de su potencial genético. Como con cualquier técnica, existen limitaciones a los datos que pueden obtenerse con la extracción de ADN. Por lo tanto, muchos investigadores ahora utilizan la extracción de ADN junto con cuenta directa y cultural para aprovechar al máximo los datos obtenidos de una muestra ambiental.
Principios
Extracción de ADN de suelo puede realizarse de dos maneras diferentes (tabla 1). En el método en situ , se utiliza una combinación de técnicas químicas y mecánicas. Para esta extracción, una masa de suelo se combina con un volumen equivalente de un tampón de extracción. Granos de cristal se añaden a la suspensión junto con un volumen de detergente (dodecil sulfato de sodio, o SDS, se utiliza normalmente), y la muestra se mezcla para facilitar la separación de las partículas del suelo seguida de incubación a una temperatura elevada para promover la lisis celular. Después de la centrifugación, el sobrenadante se somete a otros pasos de incubación y extracción para purificar el producto de la DNA.
Alternativamente, las células puede ser fraccionadas (o separadas) de la matriz del suelo antes de la extracción del material genético. Una masa de muestra de suelo somete a ciclos sucesivos de la mezcla y lento centrifugación. El paso de la paliza del grano se elimina, sin embargo, para mantener las células intactas, que se centrifugaron para obtener un pellet. Una extracción de lisozima se realiza en conjunto con incubación para desarticular las paredes celulares y liberar el DNA para purificación.
Este manuscrito y video demostrará el situ método de extracción de ADN de suelo, como este procedimiento se ha demostrado para producir mayores concentraciones de ADN de muestras de suelo en relación con el método de fraccionamiento celular.
| Tema | Fraccionamiento bacteriana | In Situ Lisis de |
| Rendimiento de ADN | 1-5 μg/g | 1-20 μg/g |
| Representante de la comunidad | Menos representante debido a absorción celular | Absorción celular más representativo, |
| Fuente de ADN recuperado | Solamente las bacterias | Sobre todo las bacterias sino también hongos y protozoos |
| Grado de corte del ADN | Menos de corte | Más de corte |
| Tamaño medio de los fragmentos de ADN | 50 kb | 25 kb |
| Grado de contaminación húmica | Menos contaminados | Más contaminados |
| Facilidad de metodología | Baja, laborioso | Más rápido, requiere menos mano de obra |
Tabla 1. Comparación de metodologías de lisis bacterianas de la fraccionamiento y en situ para la recuperación de ADN de suelo.
Procedimiento
1. bacteriana comunitaria la extracción de ADN
- Para comenzar el procedimiento, pesar 100 g de suelo tamizado. Agregue esto a un recipiente de polipropileno y añadir 100 mL de tampón de extracción compuesta por Tris buffer modificado con EDTA para promover la liberación de bacterias de la matriz del suelo, luego agitar a mano.
- A continuación, pesar 100 g de perlas de vidrio y agregar al recipiente de mezcla. Agitar la muestra durante 5 min mediante una tira a dispositivo o agitador de muñeca-acción mecánica para 15 minutos agregar 10 mL 20% sodio dodecil sulfato, o SDS, a la mezcla, luego agitar durante un minuto adicional. Incubar a una temperatura elevada de 60-65 ° C durante 60 minutos.
- Igualmente distribuir la muestra entre los diferentes tubos de 50 mL y centrifugar 10 min a 6.000 x g. transferir el sobrenadante de los tubos a un único contenedor estéril. A continuación, repetir la extracción en el sedimento del suelo como se describió anteriormente, utilizando un nuevo volumen de tampón de extracción.
- A continuación, añadir el volumen de sobrenadante transformado, aproximadamente 200 mL a un tubo de 50 mL limpio llenado a mitad de volumen con una solución 30% glicol de polietileno y 1,6 M de cloruro de sodio. Invertir las botellas varias veces con la mano para mezclar e incubar a temperatura ambiente durante 2 h. muestras de centrifugar a 10.000 x g por 20 min a la pelotilla de la DNA.
- Retire el sobrenadante con cuidado el tubo de centrífuga, dejando atrás el pellet de ácidos nucleicos parcialmente purificada. Añadir 20 mL de Buffer TE y 1,5 mL de una solución de acetato de potasio de 7.5 M para Resuspender el precipitado, entonces el vórtice. Coloque la suspensión en hielo por 5 min centrifugar a 16.000 x g durante 30 min a 4 ° C para precipitar las proteínas y los polisacáridos.
- A continuación, agregue una RNasa y proteinasa K a la muestra, mezclar suavemente con la mano y dejar reposar para el momento. Añadir un volumen equivalente de alcohol fenol: cloroformo: isoamílico (mezcla de proporción de 25:24:1) a la suspensión para extraer y mezclar suavemente con la mano. Centrifugar la preparación durante 10 minutos a 13.000 x g. Retire con cuidado el recipiente de la centrífuga y nota las dos capas.
- La parte inferior, la capa más pesada se compone de alcohol fenol: cloroformo: isoamílico y extrae desechos, y la capa superior es la acuosa y contiene el ADN. Colocar la fase acuosa en un recipiente estéril, agregar un volumen equivalente de isopropanol e invierta suavemente para iniciar la precipitación de ADN. Incubar la suspensión a temperatura ambiente durante 2 h. pelotilla de la DNA purificada por centrifugación a 16.000 x g por 30 minutos Retire con cuidado el sobrenadante como ADN concentrado puede o puede no ser visible en la parte inferior de la nave y luego resuspender en 1 mL de tampón TE.
- Utilizando un espectrofotómetro o un Fluorímetro de cuantificación de ADN/ARN, medir el nivel de la DNA extraída de la muestra. Se estima la cantidad de ADN de la lectura de nm 260. Una lectura de absorbancia de 1.0 equivale a 50 μg de DNA por mL de solución. Si la concentración es demasiado alta para lecturas precisas, diluir la suspensión de 1 a 10, o 1 a 100 con agua de grado molecular.
- La pureza del ADN se calcula la relación de la lectura a 260 nm que a 280 nm. Un valor > 1,7 indica que relativamente puro ADN. El valor teórico máximo es 2.0.
Aplicación y resumen
Comunidad ADN de cultivo colonias o extraídos de suelo puede ser sometido a enfoques de la Bioinformática y la "omic" que permiten la caracterización de la bacteria original dentro de la muestra. Los omic los acercamientos incluyen metagenómica – determinación de "que" está dentro de la comunidad a través de rRNA 16S que ordena. Esto da una estimación de la diversidad dentro de la comunidad.
También se puede calcular el número de células bacterianas en la muestra de suelo original. Comunidad ADN es extraído de un suelo y cuantificado por análisis espectroscópico. Está relacionado con la cantidad estimada de DNA medida μg de DNA por mL de solución en el volumen total del ADN extraído en solución para dar una cantidad total de ADN por g de suelo. Al conocer el valor teórico de ADN por célula, se puede calcular el número total de células por g de suelo.
Ejemplo
Un suelo tiene 0.12 μg ADN por g de suelo
Si cada celda tiene 4 fg de ADN
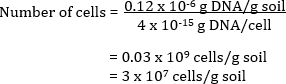
El ADN extraído de comunidad puede ser sometido a análisis de la polimerización en cadena usando las cartillas específicas para determinar si una especie en particular está presente dentro de la comunidad. Los ejemplos incluyen específicas bacterias patógenas como Clostridium perfringens o Bacillus anthracis.
Saltar a...
Vídeos de esta colección:

Now Playing
Extracción de ADN de la comunidad de colonias bacterianas
Environmental Microbiology
28.8K Vistas

Determinación del contenido de humedad en el suelo
Environmental Microbiology
359.4K Vistas

Técnica aséptica en ciencias ambientales
Environmental Microbiology
126.4K Vistas

Tinción de Gram de la Bacteria de fuentes ambientales
Environmental Microbiology
100.2K Vistas

Visualizarse los microorganismos del suelo mediante el contacto Deslice análisis y microscopia
Environmental Microbiology
42.2K Vistas

Hongos filamentosos
Environmental Microbiology
57.3K Vistas

Detección de microorganismos ambientales con la reacción en cadena de polimerasa y la electroforesis en Gel de
Environmental Microbiology
44.6K Vistas

Análisis de muestras ambientales mediante RT-PCR del ARN
Environmental Microbiology
40.4K Vistas

Cuantificación de microorganismos ambientales y virus utilizando qPCR
Environmental Microbiology
47.8K Vistas

Análisis de la calidad del agua a través de organismos indicadores
Environmental Microbiology
29.5K Vistas

Aislamiento de bacterias fecales de muestras de agua por filtración
Environmental Microbiology
39.3K Vistas

Detección de bacteriófagos en muestras ambientales
Environmental Microbiology
40.7K Vistas

Cultivo y enumeración de bacterias a partir de muestras de suelo
Environmental Microbiology
184.3K Vistas

Análisis de la curva de crecimiento bacteriano y sus aplicaciones ambientales
Environmental Microbiology
296.0K Vistas

Enumeración de las algas mediante metodología cultivable
Environmental Microbiology
13.8K Vistas
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados