Method Article
Polarizzazione e caratterizzazione di macrofagi derivati da monociti umani M1 e M2 su superfici implantari
In questo articolo
Riepilogo
Qui, presentiamo un protocollo dettagliato per valutare il potenziale immunomodulatorio delle superfici implantari in vitro, con l'obiettivo di migliorare l'affidabilità e la riproducibilità dei protocolli attuali e promuovere ulteriori ricerche. I profili delle citochine secretorie, l'espressione dell'mRNA e i marcatori della superficie cellulare sono stati monitorati utilizzando macrofagi derivati da monociti nel sangue per studiare la polarizzazione dei macrofagi coltivata sul titanio.
Abstract
La reazione da corpo estraneo (FBR), un complesso processo di guarigione immuno-mediato, svolge un ruolo cruciale nell'integrazione degli impianti nel corpo. I macrofagi, come prima linea di interazione del sistema immunitario con le superfici implantari, svolgono un ruolo bidirezionale nella modulazione dell'equilibrio infiammazione-rigenerazione. Per una comprensione approfondita e la valutazione delle reazioni tra i materiali implantari e le risposte immunitarie, sono fondamentali metodi e protocolli in vitro affidabili. Tra i diversi modelli in vitro , i macrofagi primari derivati dai monociti (MDM) rappresentano un modello eccellente per lo studio delle interazioni macrofagi-impianto. Abbiamo implementato un protocollo sperimentale per valutare la polarizzazione degli MDM in macrofagi M1 (attivati classicamente) e M2 (attivati alternativamente) sulle superfici implantari. Abbiamo isolato i monociti del sangue da donatori sani e li abbiamo differenziati in macrofagi utilizzando il fattore stimolante le colonie di macrofagi (M-CSF). I macrofagi differenziati sono stati coltivati sulle superfici dell'impianto e polarizzati nei sottotipi M1 e M2. La polarizzazione M1 è stata ottenuta in presenza di interferone (IFN)-γ e lipopolisaccaride (LPS), mentre la polarizzazione M2 è stata eseguita in un mezzo contenente interleuchina (IL)-4 e IL-13. Abbiamo valutato i fenotipi dei macrofagi mediante saggio di immunoassorbimento enzimatico (ELISA), microscopia a scansione laser confocale (CLSM) e PCR quantitativa in tempo reale (qRT-PCR) sulla base di pannelli di citochine secrete, marcatori di superficie cellulare e geni espressi. L'RNA estratto è stato trasformato in DNA complementare (cDNA) e la qRT-PCR è stata utilizzata per quantificare l'mRNA correlato ai macrofagi M1 e M2. Di conseguenza, i macrofagi M1 sono stati caratterizzati da una maggiore espressione della citochina proinfiammatoria del fattore di necrosi tumorale (TNF-α) e del marcatore di superficie CCR7 rispetto ai macrofagi M2, che mostravano livelli più elevati di CD209 e CCL13. Di conseguenza, CCR7 e CD209 sono stati identificati come marcatori specifici e affidabili dei sottotipi di macrofagi M1 e M2 mediante immunocolorazione e visualizzazione mediante CLSM. Un'ulteriore conferma è stata ottenuta dall'ELISA che ha rilevato un elevato livello di TNF-ɑ in M1 e un aumento di CCL13 nelle cellule M2. I marcatori proposti e la configurazione sperimentale possono essere utilizzati efficacemente per valutare il potenziale immunomodulatorio degli impianti.
Introduzione
I biomateriali impiantabili sono diventati una soluzione convenzionale per varie malattie umane e svolgono un ruolo importante nella ricerca biomedica, tra cui l'ingegneria tissutale, i sistemi di somministrazione di farmaci e gli impianti 1,2. Esiste un'ampia gamma di impianti realizzati con vari materiali con diverse strutture e funzionalità, come protesi d'anca, stent, reti, valvole cardiache o impianti dentali. Al momento dell'impianto, il contatto tessuto-impianto provoca una risposta immunitaria, seguita da risoluzione, rimodellamento tissutale e omeostasi. Questi processi sono influenzati dalle caratteristiche fisiche, chimiche e bioattive dei biomateriali utilizzati. Queste caratteristiche possono influenzare l'intensità e lo spettro delle risposte pro e anti-infiammatorie, la formazione di capsule fibrotiche, la degradazione dei tessuti e la fase di guarigione 3,4. Al fine di supportare e ottimizzare il processo di guarigione e l'integrazione implantare a lungo termine, un aspetto emergente della ricerca attuale è quello di studiare e mediare l'interazione tra le superfici implantari e le cellule immunitarie.
Tra le altre cellule immunitarie, i macrofagi, che si trovano in tutto il corpo, sono attori chiave nell'infiammazione e nella difesa antipatogena, nonché nei processi di guarigione e nel mantenimento dell'omeostasi tissutale 5,6. Sulla base della loro plasticità e degli stimoli microambientali tissutali locali, i macrofagi sono in grado di polarizzarsi in fenotipi funzionali distinti, che mostrano grandi differenze nel metabolismo cellulare, nelle funzioni cellulari e nei profili di secrezione di citochine. Il fenotipo M1 classicamente attivato può essere distinto dalla secrezione di citochine proinfiammatorie, come IL-1β, IL-6 e TNF-α, ed è coinvolto nella risposta infiammatoria iniziale e cronica a traumi e biomateriali estranei. Al contrario, in alternativa, i macrofagi M2 attivati, che sono attivati da citochine come IL-4 e IL-13, hanno caratteristiche come la risoluzione dell'infiammazione e la promozione della guarigione dei tessuti. I macrofagi polarizzati M2 possono essere identificati dall'espressione di marcatori di superficie cellulare come CD206 e dalla produzione di citochine come IL-10 e IL-47. Allo stesso modo, i macrofagi che sono già stati polarizzati possono riprogrammarsi in un nuovo microambiente.
Molti studi sulle interazioni cellula-biomateriale hanno dimostrato l'importanza dei macrofagi nella cascata di risposte immunologiche verso biomateriali impiantabili e nell'orchestrazione dei processi coinvolti nella guarigione delle complicanze correlate all'impianto 8,9,10. Anche se l'ingegneria biomedica ha fatto progressi significativi negli ultimi anni, sono necessarie ulteriori ricerche per capire come gli impianti modulano il comportamento e la polarizzazione dei macrofagi 11,12,13.
Nella coltura cellulare, le cellule mononucleate del sangue periferico (PBMC) derivate da monociti possono essere differenziate in macrofagi M0 aderenti seguite da polarizzazione indotta verso fenotipi M1 o M2 utilizzando rispettivamente LPS e IFN-γ o IL-4. Dopo l'incubazione in vitro con nuovi campioni di biomateriali, è possibile utilizzare i diversi recettori di superficie cellulare e i profili citochinici dei macrofagi M1 e M2 per rilevare il potenziale immunomodulatorio dei biomateriali in vitro14,15. Questo studio mirava a sviluppare un protocollo in vitro che potesse essere impiegato per studiare la polarizzazione degli MDM in risposta a diverse superfici implantari. Le analisi di espressione genica, le tecniche di microscopia e l'ELISA possono essere utilizzate per determinare i marcatori fenotipici e i profili di citochine specifici dei macrofagi M1 e M2 modulati dal biomateriale. Pertanto, le complesse interazioni tra macrofagi e superfici di biomateriali possono essere chiarite e si possono ottenere informazioni preziose per comprendere meglio le interazioni macrofagi-biomateriali. Infine, un protocollo standardizzato in vitro garantisce la riproducibilità, l'affidabilità e la comparabilità dei risultati sperimentali riducendo al minimo la variabilità nella configurazione sperimentale.
Protocollo
Il sangue periferico umano è stato ottenuto da donatori di sangue sani in conformità con il protocollo approvato dal Comitato etico della facoltà di medicina dell'Università di Tubinga (approvazione etica: 286/2021 BO). Le PBMC umane sono state isolate utilizzando la centrifugazione in gradiente di densità, come descritto in precedenza16. Il seguente protocollo è delineato per le PBMC isolate da 24 mL di sangue. Una rappresentazione schematica del protocollo è mostrata nella Figura 1.
NOTA: Il volume del sangue deve essere regolato in base al numero di macrofagi M0 utilizzati.
Un totale di 35,46 ± 9,1 milioni di PBMC sono stati ottenuti da 24 mL di sangue, risultando in 1,97 ± 0,46 milioni di macrofagi M0 (n = 5). Tutti i reagenti, i materiali di consumo e i dispositivi sono elencati nella Tabella dei materiali. I buffer sono elencati nella Tabella 1.
1. Differenziazione dei monociti del sangue umano in macrofagi
- Risospendere le PBMC isolate in 15 mL di terreno di attacco per monociti preriscaldato e trasferirle in un pallone per colture cellulari T75.
- Incubare le cellule a 37 °C e 5% di CO2 per 90 minuti per consentire l'adesione.
- Scartare il surnatante e lavare le cellule una volta con un terreno completo preriscaldato con una leggera inclinazione per rimuovere le cellule non aderenti o poco aderenti.
NOTA: Le cellule attaccate sono monociti, che costituiscono circa il 10% del totale delle PBMC originariamente aggiunte al pallone. - Aggiungere 15 mL di terreno completo contenente 10 ng/mL di fattore stimolante le colonie di macrofagi (M-CSF) alle cellule aderenti e incubare per 6 giorni per promuovere la differenziazione.
- Sostituire il terreno con un terreno fresco e completo contenente 10 ng/mL M-CSF ogni 2 giorni.
2. Coltivazione e polarizzazione di MDM sulla superficie dell'impianto in titanio
NOTA: Il giorno 6 di differenziazione, i macrofagi M0 sono stati seminati sulle superfici del biomateriale con stimoli diversi per 48 ore per ottenere macrofagi M1 o M2 completamente polarizzati. Per ogni superficie esaminata, sono stati utilizzati tre dischi per seminare i macrofagi M0, M1 e M2. Come superfici di controllo sono stati utilizzati vetrini coprioggetti in plastica trattati con colture cellulari.
- Rimuovere il terreno di coltura dai matracci T75 e lavare le cellule con 10 mL di PBS per 5 minuti.
- Staccare le cellule aderenti incubandole con 10 mL di soluzione di distacco cellulare preriscaldata per 30 minuti.
- Picchiettare delicatamente le cellule e trasferirle in una provetta da 50 ml. Staccare le cellule rimanenti raschiandole delicatamente in 10 ml di PBS.
- Trasferire le cellule staccate nella provetta da 50 mL e centrifugarle a 300 x g per 10 minuti.
- Scartare il surnatante e risospendere le cellule in 5 mL di terreno completo preriscaldato.
- Contare le cellule utilizzando la colorazione con blu di tripano e un emocitometro per determinare il numero e la vitalità delle cellule.
- Preparare la sospensione cellulare regolando il numero di cellule a 160.000 cellule per 1 mL di terreno completo.
- Pulire i dischi di biomateriale ad ultrasuoni con etanolo al 70% per 5 minuti, quindi sterilizzarli con etanolo al 70% per 30 minuti.
- Asciugare i dischi in titanio per 1-2 ore, quindi posizionarli in piastre a 24 pozzetti non trattate e aggiungere 1 mL di sospensione cellulare preparata a ciascun pozzetto.
- Per ottenere macrofagi polarizzati M1, aggiungere IFN-γ e LPS rispettivamente alla concentrazione di 50 ng/mL e 10 ng/mL. Per la polarizzazione M2, aggiungere IL-4 e IL-13 in una concentrazione di 20 ng/mL ciascuno. Assicurarsi che le cellule M0 vengano coltivate senza agenti polarizzanti. Incubare le cellule per altre 48 ore a 37 °C e 5% di CO2 per indurre la polarizzazione.
3. Caratterizzazione di macrofagi polarizzati mediante ELISA
NOTA: Il giorno 2 di polarizzazione, i campioni sono stati preparati per le analisi di caratterizzazione. La citochina TNF-ɑ e la chemochina CCL13 sono state misurate per caratterizzare rispettivamente i macrofagi polarizzati M1 e M2. La concentrazione di proteine secrete è stata normalizzata alla concentrazione totale di proteine secrete nel surnatante corrispondente.
- Raccogliere il surnatante in una provetta da 1,5 ml e centrifugare a 300 x g per 5 minuti. Trasferire il surnatante in una nuova provetta.
- Trasferire i dischi su una nuova piastra a 24 pozzetti per ulteriori esperimenti. Lo scopo è quello di eliminare le cellule morte o attaccate in modo lasco in piastre a 24 pozzetti non trattate.
NOTA: La vitalità cellulare deve essere verificata sulle superfici prima di condurre qualsiasi analisi. La vitalità delle cellule può essere determinata mediante saggi di vitalità delle cellule vive/morte o indirettamente attraverso test di proliferazione cellulare e citotossicità. - Misurare le citochine e le chemochine secondo le istruzioni specifiche fornite dal produttore. Misurare le citochine immediatamente o conservare i campioni a -80 °C per misurazioni future.
NOTA: Per ogni tipo di kit (considerando la sensibilità e i limiti di rilevamento del kit ELISA), è necessario determinare la diluizione del campione appropriata. Per il test dell'acido bicinconninico (BCA), i campioni sono stati diluiti 1:5. I campioni M1 per TNF-ɑ sono stati diluiti 1:10 e i campioni M2 per CCL13 sono stati diluiti 1:12. - Calcolare la concentrazione di citochine/chemochine secrete utilizzando la curva standard seguendo le istruzioni del produttore.
- Misurare la quantità totale di proteine utilizzando il kit per il dosaggio delle proteine BCA.
- Normalizzare la concentrazione di proteine secrete in mg di proteine totali.
4. Caratterizzazione di macrofagi polarizzati mediante CLSM
NOTA: I macrofagi polarizzati sono stati ulteriormente caratterizzati colorandoli con anticorpi contro i marcatori di superficie cellulare CD209 e CCR7. I nuclei sono stati controcolorati con DRAQ5. CD68 o altri marcatori possono essere utilizzati come marcatori pan-macrofagi.
- Lavare le cellule 2 volte in 800 μL di PBS. Incubare i dischi per 20 minuti a temperatura ambiente (RT) in 400 μL di tampone di fissaggio.
- Dopo aver rimosso il tampone di fissaggio, lavare tre volte in 400 μl di PBS. Colorare immediatamente i campioni o conservarli a 4 °C in 1 mL di tampone di conservazione.
NOTA: Con questo protocollo di fissazione e conservazione, i campioni sono stati colorati e sottoposti a imaging con successo entro 1-6 settimane dalla fissazione. - Prima del passaggio successivo, lavare i dischi due volte con 800 μL di PBS dopo la conservazione.
- Incubare i dischi con 400 μL di tampone bloccante per 30 minuti a RT per bloccare i siti di legame non specifici.
- Eliminare il tampone bloccante e incubare i dischi a RT per 1 ora con anticorpi primari diluiti in 400 μl di tampone di colorazione.
- Eseguire una procedura di immunofluorescenza utilizzando la doppia colorazione per esaminare l'espressione di CCR7 e CD209 in un campione. A tale scopo, combinare anticorpi primari allevati da specie diverse (topo e coniglio) nella stessa fase di colorazione.
NOTA: Per ottenere un segnale forte con un fondo minimo, ottimizzare la concentrazione di anticorpi. L'anticorpo CCR7 è stato utilizzato a una concentrazione finale di 10 μg/mL e l'anticorpo CD209 è stato diluito 1/400.
- Eseguire una procedura di immunofluorescenza utilizzando la doppia colorazione per esaminare l'espressione di CCR7 e CD209 in un campione. A tale scopo, combinare anticorpi primari allevati da specie diverse (topo e coniglio) nella stessa fase di colorazione.
- Rimuovere gli anticorpi primari e lavare 3 volte con 400 μL di tampone di lavaggio.
- Aggiungere anticorpi secondari marcati con fluorofori diluiti in tampone di colorazione e incubare per 1 ora a RT al buio.
NOTA: La concentrazione di anticorpi secondari deve essere ottimizzata per ottenere il massimo dei segnali specifici con un fondo minimo. In questo studio, sono stati utilizzati anticorpi secondari alla concentrazione di 5 μg/mL per la colorazione. - Dopo aver rimosso il surnatante, lavare i campioni tre volte nel tampone di lavaggio per 3 minuti ciascuno.
- Aggiungere 10 μM di DRAQ5 in PBS e incubare per 15 minuti a RT, al riparo dalla luce.
- Rimuovere il surnatante e lavare i dischi una volta in PBS.
- Rimuovere il PBS rimanente e aggiungere 1 goccia di supporto di montaggio.
- Dopo 5 minuti applicare i bicchieri di copertura e lasciare asciugare i campioni per 1 ora.
- Dopo aver asciugato i campioni, sigillare i bordi con smalto trasparente e conservarli a 4 °C al buio fino all'imaging.
- Per ottenere una panoramica delle cellule, visualizzare i campioni con un ingrandimento di 25x. Per determinare ulteriormente la struttura e la localizzazione dei marcatori di superficie, acquisire immagini con ingrandimento 63x.
- Quantificare l'intensità della fluorescenza di CCR7 e CD209 utilizzando il software ImageJ.
NOTA: L'acquisizione delle immagini è stata eseguita con un fotomoltiplicatore (PMT) utilizzando un sistema CLSM, dotato di un laser ad argon (488 nm), un laser DPSS (561 nm) e un laser HeNe (633 nm).
5. Caratterizzazione di macrofagi polarizzati mediante qRT-PCR
NOTA: Per l'isolamento dell'RNA, sono stati utilizzati due dischi per campione al fine di ottenere abbastanza RNA per la sintesi del cDNA.
- Lavare i dischi 2 volte con 800 μL di PBS.
- Aggiungere 350 μl di tampone di lisi al primo disco e lisare le cellule pipettando su e giù.
- Trasferire il lisato sul secondo disco e ripetere il processo di lisi.
- Aggiungere 350 μl di etanolo al 70% al lisato e pipettare su e giù fino a ottenere un composto omogeneo.
- Trasferire il lisato nella colonna di centrifuga e seguire le istruzioni del produttore per l'isolamento dell'RNA.
- Quantifica la quantità di RNA utilizzando una nanogoccia o un altro dispositivo.
- Normalizzare le concentrazioni di RNA per diversi campioni e sintetizzare il cDNA secondo le istruzioni del produttore utilizzando il sistema RT-PCR per la sintesi del cDNA del primo filamento.
- Sintetizzare il cDNA utilizzando 350 ng di RNA seguendo il protocollo del produttore e conservarlo a -20 °C fino all'esecuzione dell'analisi qRT-PCR.
NOTA: Qui, 350 ng di RNA purificato sono stati utilizzati per sintetizzare il cDNA utilizzando 4 μL di RT Mix (5x) in 20 μL. - Esecuzione di qRT-PCR su un sistema di PCR in tempo reale in piastre a 96 pozzetti e reazioni individuali da 15 μL (1x Syber green master mix, 0,2 μM di primer diretti e inversi e 4,5 μL di cDNA diluito 1:10).
NOTA: Il programma PCR inizia con un coperchio riscaldato (105 °C) seguito da tre fasi: denaturazione iniziale a 95 °C per 3 minuti, seguita da 15 s a 95 °C e 30 s a 55 °C per 40 cicli. - Normalizzare i livelli di espressione di vari geni del gene GAPDH (o di altri geni come la β-actina).
- Calcolare l'espressione genica relativa utilizzando il metodo 2−ΔΔCt prendendo come riferimento le cellule M0 coltivate su vetrini coprioggetti di coltura tissutale. La Tabella 2 elenca tutti i primer utilizzati in questo studio.
6. Analisi statistica
- Presentare tutti i dati come media ± SEM. Ripetere tutti i test (in questo studio, i test sono stati ripetuti cinque volte) per garantire la riproducibilità. Valutare le differenze statisticamente significative tra i dati normalmente distribuiti utilizzando un'analisi della varianza a una via (ANOVA) seguita dal test multiplo di Tukey.
- Utilizza il test di Friedman e il test di confronto multiplo di Dunn per analizzare set di dati non parametrici. Utilizzare un software di analisi dei dati appropriato per analizzare i dati e definire la significatività statistica come un valore p inferiore a 0,05.
Risultati
I risultati di questo studio dimostrano il successo della differenziazione e della polarizzazione degli MDM su superfici in titanio, seguita dalla caratterizzazione dei macrofagi polarizzati M1 o M2. Nella prima fase, abbiamo caratterizzato gli MDM polarizzati utilizzando CLSM. Sulla base dei nostri studi preliminari, CD209 e CCR7 sono stati utilizzati come marcatori specifici per differenziare M1 dagli MDM polarizzati M2. Come mostrato nella Figura 2A, B, gli MDM si sono polarizzati con successo nei macrofagi M1 e M2. Sulla superficie del titanio, CCR7 è stato espresso più fortemente nei macrofagi polarizzati M1 rispetto a CD209, espresso specificamente nei macrofagi polarizzati M2. Inoltre, la quantificazione dell'intensità relativa della fluorescenza ha facilitato l'assegnazione di marcatori ai sottotipi M1 o M2 (Figura 2C).
La Figura 3 mostra un'analisi qRT-PCR rappresentativa degli MDM su superfici in titanio e vetrino coprioggetti. I risultati hanno mostrato che gli MDM su entrambe le superfici sono stati polarizzati con successo, come dimostrato dall'elevata espressione dei marcatori di polarizzazione M1 (CCR7 e TNF-ɑ) e M2 (CD209 e CCL13). Ciò è stato ulteriormente confermato a livello proteico osservando alti livelli di citochine infiammatorie di TNF-α (Figura 4A) e chemochine IL-13 (Figura 4B) rispettivamente nelle cellule polarizzate M1 e M2.
Figura 1: Rappresentazione schematica del protocollo. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Caratterizzazione di MDM polarizzati utilizzando CLSM. L'arricchimento dei macrofagi polarizzati M1 e M2 è stato confermato dalla colorazione anticorpale specifica e dall'analisi CLSM utilizzando anticorpi CCR7 e CD209 al giorno 2 della post-semina. Secondo la colorazione a fluorescenza e l'analisi CLSM, (A) ingrandimento 25x e (B) ingrandimento 63x, le cellule M1 esprimevano un CCR7 più elevato (in verde) rispetto a M0 o M2. Le cellule M2 mostrano un significativo pattern di espressione di CD209 (in verde). (C) L'analisi quantitativa dell'intensità relativa di fluorescenza di CCR7 e CD209. I nuclei sono stati colorati con DRAQ5 (in viola). I risultati sono rappresentativi di 5 esperimenti simili eseguiti in modo indipendente. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 3: Caratterizzazione di MDM polarizzati utilizzando profili di espressione genica. La polimerasi quantitativa a trascrizione inversa è stata utilizzata per studiare i livelli di mRNA di diversi geni associati alla polarizzazione M1 (CCR7 e TNF-ɑ) e M2 (CD209 e CCL13). GAPDH è stato usato come gene per le pulizie. I dati sono presentati come media ± SEM (n = 5). Clicca qui per visualizzare una versione più grande di questa figura.
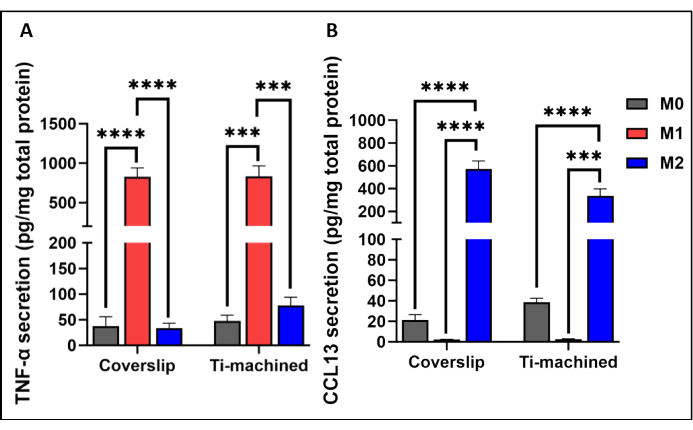
Figura 4: Produzione di citochine da parte dei macrofagi M0, M1 e M2 coltivati su vetrini in titanio e plastica. (A) Il livello di citochine TNF-ɑ e (B) i livelli di CCL13 nei surnatanti di coltura cellulare sono stati misurati mediante ELISA. La secrezione di citochine è stata normalizzata alla secrezione proteica totale misurata mediante saggio BCA. I grafici a barre rappresentano la media ± SEM (n = 5). Clicca qui per visualizzare una versione più grande di questa figura.
| Buffer/Soluzione | Contenuto |
| Tampone di colorazione/blocco | PBS + 1% BSA + 0,1% Interpolazione 20 |
| Tampone di lavaggio | PBS + 0,1% Interpolazione 20 |
| Tampone di fissazione | 3% di paraformaldeide (PFA) nel PBS |
| Buffer di archiviazione | 1% di penicillina e streptomicina nella PBS |
Tabella 1: Elenco dei buffer.
| Nome del primer | Sequenze di primer in avanti | Sequenze di primer inverse |
| GAPDH | 5'-GAGTCAACGGATTTGGTCGT-3' | 5'-TTGATTTTGGAGGGATCTCG-3' |
| CCR7 | 5'- TGGTGATCGGCTTTCTGGTC-3' | 5'- CACCTTGATGGCCTTGTTGC-3' |
| CD209 | 5'- GGAGCAGAACTTCCTACAGC-3' | 5'- CAACGTTGTTGGGCTCCT-3' |
| CCL13 | 5'-ATCTCCTTGCAGAGGCTGAA-3' | 5'-ACTTCTCTTTGGGTCAGCA-3' |
| TNF-ɑ | 5'- GCTGCACTTTGGAGTGATCG-3' | 5'- TCACTCGGGGTTCGAGAAGA-3' |
Tabella 2. Sequenze di primer utilizzati nella qRT-PCR.
Discussione
Una comprensione approfondita del comportamento dei macrofagi è essenziale per comprendere le proprietà immunomodulatorie dei materiali impiantabili. Diversi studi hanno riportato marcatori eterogenei, una varietà di modelli cellulari e protocolli per caratterizzare la polarizzazione dei macrofagi in vitro 17,18,19,20. Per migliorare la riproducibilità, l'affidabilità e la comparabilità dei risultati sperimentali, sono essenziali protocolli standardizzati e verificati, insieme a un modello cellulare adatto e a marcatori di caratterizzazione di consenso. Di conseguenza, una simulazione accurata delle proprietà immunomodulatorie dei materiali implantari richiede prima un modello cellulare adeguato. Diversi studi hanno utilizzato un'ampia gamma di modelli cellulari, come macrofagi tissutali isolati, macrofagi differenziati derivati da monociti del sangue e linee cellulari monocitiche immortalizzate. Sebbene i macrofagi isolati nei tessuti possano essere considerati più rappresentativi delle condizioni in vivo, sono tecnicamente ed eticamente impegnativi21,22. I macrofagi sono anche frequentemente ottenuti da linee cellulari monocitiche immortalizzate, come le cellule THP1 23,24,25,26. Sebbene queste cellule possano offrire una maggiore omogeneità nella risposta cellulare come fonte illimitata di cellule non senescenti, di solito sono ottenute da pazienti con neoplasie ematologiche e le loro risposte possono differire significativamente dalle cellule normali. È stato riportato che le cellule THP1 monocitiche, ad esempio, sono più reattive ai simulatori M1 e hanno maggiori probabilità di mostrare le caratteristiche M122. I risultati di questo studio sono in linea con quelli del nostro studio preliminare (dati non presentati qui).
Inoltre, poiché i monociti derivati dal sangue sono considerati i precursori dei macrofagi residenti nei tessuti e possono essere facilmente ottenuti in rese più elevate, possono essere una valida alternativa per i macrofagi 27,28,29. Sulla base del nostro studio che ha utilizzato macrofagi derivati da monociti del sangue, abbiamo scoperto che queste cellule erano ugualmente sensibili agli stimolatori M1 e M2 sia su vetrini in titanio che trattati con colture cellulari. Inoltre, hanno mostrato più marcatori di consenso M1 e M2, alcuni dei quali sono mostrati nei risultati rappresentativi. I risultati hanno dimostrato che gli MDM possono essere utilizzati come modello in vitro fattibile per simulare le interazioni impianto-macrofago.
Per ulteriori progressi negli studi di immunomodulazione, sono essenziali marcatori di caratterizzazione costanti e specifici. Gli studi hanno introdotto una varietà di marcatori per la caratterizzazione dei macrofagi che differiscono non solo tra macrofagi provenienti da fonti diverse, ma anche tra macrofagi provenienti dalla stessa fonte 17,18,19,24. Un pannello di marcatori M1 e M2 caratteristici degli MDM è stato determinato e verificato attraverso la valutazione di diversi marcatori riportati. Alcuni dei più importanti indicatori chiave sono presentati in questo articolo.
Anche la determinazione dei metodi di rilevamento più appropriati è una parte cruciale del processo di valutazione. Le tecniche di analisi comunemente utilizzate per valutare i marcatori della superficie cellulare richiedono in genere la rimozione delle cellule dai biomateriali. Tuttavia, è stato osservato che questo processo influisce negativamente sulle cellule danneggiando i loro marcatori di superficie e provocando un basso numero di cellule staccate30. Di conseguenza, la citometria a flusso, che richiede il distacco delle cellule, non è appropriata per valutare i macrofagi che sono strettamente attaccati agli impianti. In questo studio, il rilevamento dei marcatori della superficie cellulare è stato condotto utilizzando il CLSM. Utilizzando i marcatori appropriati e ottimizzando il processo di colorazione, siamo stati in grado di caratterizzare con successo i sottotipi M1 e M2 rispetto l'uno all'altro e alle cellule M0. È importante notare che la colorazione a fluorescenza è semiquantitativa, che è uno dei suoi limiti. Ciò può complicare la valutazione delle cellule utilizzando marcatori espressi in tutti i sottotipi senza differenze significative. CCR7 e CD209 sono stati selezionati dopo aver testato diversi marcatori per caratterizzare gli MDM utilizzando CLSM. CCR7 e CD209 erano costantemente più alti espressi rispettivamente nei sottotipi M1 e M2.
Entro i limiti di questo studio, i risultati evidenziano l'utilità e l'efficacia dei protocolli implementati nella polarizzazione dei macrofagi sulle superfici implantari e nella loro caratterizzazione accurata in termini di espressione genica, proteine secrete e marcatori della superficie cellulare. Inoltre, l'analisi dei marcatori descritti ha rivelato modelli di espressione coerenti e specifici che possono essere utilizzati per distinguere diversi sottotipi di MDM. Questo modello in vitro, tuttavia, non riflette pienamente la diversità fenotipica e la plasticità dei macrofagi umani. Diversi sottotipi di macrofagi (M2a, M2b, M2c, M2d) sono ora in fase di identificazione, il che indica la necessità di modelli in vitro più diversificati per studiare come vari biomateriali e le loro caratteristiche (ad esempio, proprietà fisico-chimiche) influenzano la plasticità e la polarizzazione dei macrofagi31,32. Sebbene non sia possibile rispecchiare la complessa situazione in vivo utilizzando modelli in vitro, molti risultati possono essere ottenuti utilizzando il protocollo in vitro presentato per descrivere efficacemente il potenziale immunomodulatorio di nuovi biomateriali impiantabili9. Infine, ma non meno importante, è necessario un ulteriore approfondimento per caratterizzare i macrofagi in modelli più complessi in vitro o in vivo che coinvolgono il ruolo di altri attori in contesti fisiologici complessi. Nel complesso, questo studio contribuirà allo sviluppo e alla progettazione di biomateriali immunomodulatori per migliorare e promuovere processi di rigenerazione tissutale favorevoli e un'integrazione di successo dell'impianto, nonché per prevenire l'infiammazione cronica associata all'impianto.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
I dischi sono stati gentilmente forniti da Medentis Medical, Bad-Neuenahr-Ahrweiler, Germania. Gli autori riconoscono il supporto del Dipartimento di Chirurgia Orale e Maxillo-Facciale (Ospedale Universitario di Tubinga).
Materiali
| Name | Company | Catalog Number | Comments |
| 24-well plate, not-treated | Corning Incorporated (Kennebunk, USA) | 144530 | |
| Absorbance reader Infinite F50 | TECAN Austria GmbH (Grödig, Austria) | TCAT91000001 | |
| Accutase in DPBS, 0.5 mM EDTA | EMD Millipore Corp. (Burlington, USA) | SCR005 | |
| Anti-Fade Fluorescence Mounting Medium -Aqueous, Fluoroshield | abcam (Cambridge, UK) | ab104135 | |
| Bio-Rad MJ Research PTC-200 Peltier Thermal Cycler | Bio-Rad / MJ Research | 7212 | |
| Bovine serum albumin (BSA) | VWR International bvba (Leuven, Belgium) | 422361V | |
| Centrifuge 5804 R | Eppendorf SE (Hamburg, Germany) | 5804 R | |
| DC-SIGN (D7F5C) XP Rabbit mAb | Cell Signaling Technology | 13193 | |
| Dimethyl sulfoxide | Sigma Aldrich Co. (St.Louis, MO, USA) | D2438-5X10ML | |
| DRAQ5 Staining Solution | Milteny Biotec (Bergisch Gladbach, Germany) | 130-117-344 | |
| Ethanol ≥99.8% for molecularbiology | Carl Roth GmbH + CO. KG (Karlsruhe, Germany) | 1HPH.1 | |
| Goat Anti-Mouse IgG (H&L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor Plus 488 | Invitrogen | A32723TR | |
| Goat anti-Rabbit IgG (H+L) Cross-Adsorbed Secondary Antibody, Cyanine3 | Invitrogen | A10520 | |
| GraphPad Prism | GraphPad | Version 9.4.1 | |
| Human CCR7 Antibody | R&D Systems | MAB197 | |
| Human IFN-gamma Recombinant | Invitrogen (Rockford, USA) | RIFNG100 | |
| Human IL-13 | Milteny Biotec (Bergisch Gladbach, Germany) | 5230901032 | |
| Human IL-4 | Milteny Biotec (Bergisch Gladbach, Germany) | 130-095-373 | |
| Human M-CSF | Peprotech (Cranbury, USA) | 300-25 | |
| Leica TCS SP5 | Leica Microsystems CMS GmbH (Mannheim, Germany) | https://www.leica-microsystems.com/products/confocal-microscopes/p/leica-tcs-sp5/ | |
| Lipopolysaccharides from Escherichia | Sigma Aldrich Co. (Missouri, USA) | L4391-1MG | |
| Luna Universal qPCR Master Mix | New England Biolabs | NEB #M3003 | |
| LunaScript RT SuperMix Kit | New England Biolabs | E3010L | |
| Lymphocyte Separation Medium 1077 | PromoCell (Heidelberg, Germany) | C-44010 | |
| MCP-4/CCL13 Human ELISA Kit | Invitrogen | EHCCL13 | |
| MicroAmp Fast 96-Well Reaction Plate (0.1 mL) | Applied Biosystems (Waltham, USA) | 4346907 | |
| MicroAmp Optical Adhesive Film | Life Technologies Corporation (Carlsbad, USA) | 4311971 | |
| MicroAmp Splash Free 96-Well Base | Applied Biosystems (Waltham, USA) | 4312063 | |
| Microlitercentrifuge CD-3124R | Phoenix Instrument (Germany) | 9013111121 | |
| Microscope Cover Glasses, 10 mm | Carl Roth GmbH, Karlsruhe, Germany | 4HX4.1 | |
| Monocyte Attachment Medium | PromoCell (Heidelberg, Germany) | C-28051 | |
| Multiply-Pro Gefäß 0.5 mL, PP | Sarstedt AG & CO (Nümbrecht, Germany) | 7,27,35,100 | |
| Nanodrop One | Thermo Scientific (USA) | ND-ONE-W | |
| QuantStudio 3 System | Life Technologies GmbH (St. Leon-Rot, Germany) | A28567 | |
| RNeasy Micro Kit | Qiagen | 74007 | |
| RPMI 1640, 1x, with L-glutamine | Mediatech, Inc. (Manassas, USA) | 10-040-CV | |
| Sterile bench, LaminarAir HB 2472 | Heraeus instruments (Hanau, Germany) | 51012197 | |
| Tissue Culture Coverslips 13 mm (Plastic) | Sarstedt Inc. (Newton, USA) | 83,18,40,002 | |
| Titanium machinied discs 12 cm | Medentis Medical (Bad-Neuenahr-Ahrweiler, Germany) | N/A | |
| TNF alpha Human ELISA Kit | Invitrogen | KHC3011 | |
| Trypan blue solution 0.4% | Carl Roth GmbH + Co. KG (Karlsruhe, Germany) | 1680.1 |
Riferimenti
- Othman, Z., Pastor, B. C., van Rijt, S., Habibovic, P. Understanding interactions between biomaterials and biological systems using proteomics. Biomaterials. 167, 191-204 (2018).
- Ikada, Y. Challenges in tissue engineering. J R Soc Interface. 3 (10), 589-601 (2006).
- Aamodt, J. M., Grainger, D. W. Extracellular matrix-based biomaterial scaffolds and the host response. Biomaterials. 86, 68-82 (2016).
- Batool, F., et al. Modulation of immune-inflammatory responses through surface modifications of biomaterials to promote bone healing and regeneration. J Tissue Eng. 12, 20417314211041428 (2021).
- Mantovani, A., Biswas, S. K., Galdiero, M. R., Sica, A., Locati, M. Macrophage plasticity and polarization in tissue repair and remodelling. J Pathol. 229 (2), 176-185 (2013).
- Shrivastava, R., Shukla, N. Attributes of alternatively activated (M2) macrophages. Life Sci. 224, 222-231 (2019).
- Gordon, S., Taylor, P. R. Monocyte and macrophage heterogeneity. Nat Rev Immunol. 5 (12), 953-964 (2005).
- Mosser, D. M., Edwards, J. P. Exploring the full spectrum of macrophage activation. Nat Rev Immunol. 8 (12), 958-969 (2008).
- Murray, P. J., et al. Macrophage activation and polarization: nomenclature and experimental guidelines. Immunity. 41 (1), 14-20 (2014).
- Kzhyshkowska, J., et al. Macrophage responses to implants: prospects for personalized medicine. J Leukoc Biol. 98 (6), 953-962 (2015).
- Browne, S., Pandit, A. Biomaterial-mediated modification of the local inflammatory environment. Front Bioeng Biotechnol. 3, 67 (2015).
- Piatnitskaia, S., et al. Modelling of macrophage responses to biomaterials in vitro: state-of-the-art and the need for the improvement. Front Immunol. 15, 1349461 (2024).
- Rayahin, J. E., Gemeinhart, R. A. Activation of macrophages in response to biomaterials. Results Probl Cell Differ. 62, 317-351 (2017).
- Murray, P. J. Macrophage polarization. Annu Rev Physiol. 79, 541-566 (2017).
- Gordon, S., Pluddemann, A. Tissue macrophages: heterogeneity and functions. BMC Biol. 15 (1), 53 (2017).
- Salma Iqbal, A. K. Characterization of in vitro generated human polarized macrophages. J Clin Cell Immunol. 6, 1-8 (2015).
- Hotchkiss, K. M., et al. Titanium surface characteristics, including topography and wettability, alter macrophage activation. Acta Biomater. 31, 425-434 (2016).
- Wang, Y., Zhang, Y., Sculean, A., Bosshardt, D. D., Miron, R. J. Macrophage behavior and interplay with gingival fibroblasts cultured on six commercially available titanium, zirconium, and titanium-zirconium dental implants. Clin Oral Investig. 23 (8), 3219-3227 (2019).
- Abaricia, J. O., Shah, A. H., Ruzga, M. N., Olivares-Navarrete, R. Surface characteristics on commercial dental implants differentially activate macrophages in vitro and in vivo. Clin Oral Implants Res. 32 (4), 487-497 (2021).
- Lu, W., et al. Improved osseointegration of strontium-modified titanium implants by regulating angiogenesis and macrophage polarization. Biomater Sci. 10 (9), 2198-2214 (2022).
- Murray, P. J., Wynn, T. A. Obstacles and opportunities for understanding macrophage polarization. J Leukoc Biol. 89 (4), 557-563 (2011).
- Nascimento, C. R., Fernandes, N. A. R., Maldonado, L. A. G., Junior, C. R. Comparison of monocytic cell lines U937 and THP-1 as macrophage models for in vitro studies. Biochem Biophys Rep. 32, 101383 (2022).
- Freytes, D. O., Kang, J. W., Marcos-Campos, I., Vunjak-Novakovic, G. Macrophages modulate the viability and growth of human mesenchymal stem cells. J Cell Biochem. 114 (1), 220-229 (2013).
- Zhang, Y., et al. Macrophage type modulates osteogenic differentiation of adipose tissue MSCs. Cell Tissue Res. 369 (2), 273-286 (2017).
- Cerqueira, A., et al. Evaluation of the inflammatory responses to sol-gel coatings with distinct biocompatibility levels. J Biomed Mater Res A. 109 (9), 1539-1548 (2021).
- Zhang, Y., Cheng, X., Jansen, J. A., Yang, F., van den Beucken, J. J. Titanium surfaces characteristics modulate macrophage polarization. Mater Sci Eng C Mater Biol Appl. 95, 143-151 (2019).
- Nobs, S. P., Kopf, M. Tissue-resident macrophages: guardians of organ homeostasis. Trends Immunol. 42 (6), 495-507 (2021).
- Sreejit, G., Fleetwood, A., Murphy, A., Nagareddy, P. Origins and diversity of macrophages in health and disease. Clin Transl Immunology. 9 (12), e1222 (2020).
- Parisi, L., et al. Preparation of human primary macrophages to study the polarization from monocyte-derived macrophages to pro-or anti-inflammatory macrophages at biomaterial interface in vitro. J Dent Sci. 18 (4), 1630-1637 (2023).
- Feuerer, N., et al. Macrophage retrieval from 3D biomaterials: A detailed comparison of common dissociation methods. J Immunol Regen Med. 11, 100035 (2021).
- Shapouri-Moghaddam, A., et al. Macrophage plasticity, polarization, and function in health and disease. J Cell Physiol. 233 (9), 6425-6440 (2018).
- Sridharan, R., Cameron, A. R., Kelly, D. J., Kearney, C. J., O'Brien, F. J. Biomaterial based modulation of macrophage polarization: a review and suggested design principles. Mater Today. 18 (6), 313-325 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon