Method Article
Polarisation und Charakterisierung von M1- und M2-humanen Monozyten-abgeleiteten Makrophagen auf Implantatoberflächen
In diesem Artikel
Zusammenfassung
Hier stellen wir ein detailliertes Protokoll zur Bewertung des immunmodulatorischen Potenzials von Implantatoberflächen in vitro vor, um die Zuverlässigkeit und Reproduzierbarkeit aktueller Protokolle zu verbessern und die weitere Forschung zu fördern. Sekretorische Zytokinprofile, mRNA-Expression und Zelloberflächenmarker wurden mit Hilfe von Blutmonozyten-abgeleiteten Makrophagen überwacht, um die Makrophagenpolarisation auf Titan zu untersuchen.
Zusammenfassung
Die Fremdkörperreaktion (FBR), ein immunvermittelter komplexer Heilungsprozess, spielt eine entscheidende Rolle bei der Integration von Implantaten in den Körper. Makrophagen als erste Linie der Interaktion des Immunsystems mit den Implantatoberflächen spielen eine bidirektionale Rolle bei der Modulation des Gleichgewichts zwischen Entzündung und Regeneration. Für ein tiefes Verständnis und die Bewertung der Reaktionen zwischen Implantatmaterialien und Immunantworten sind zuverlässige in vitro Methoden und Protokolle von entscheidender Bedeutung. Unter den verschiedenen in vitro Modellen stellen primäre Monozyten-abgeleitete Makrophagen (MDMs) ein hervorragendes Modell für die Untersuchung von Makrophagen-Implantat-Interaktionen dar. Wir haben ein experimentelles Protokoll implementiert, um die Polarisation von MDMs in M1 (klassisch aktivierte) und M2 (alternativ aktivierte) Makrophagen auf Implantatoberflächen zu bewerten. Wir isolierten Blutmonozyten von gesunden Spendern und differenzierten sie mit Hilfe des Makrophagen-Kolonie-stimulierenden Faktors (M-CSF) in Makrophagen. Differenzierte Makrophagen wurden auf Implantatoberflächen kultiviert und in M1- und M2-Subtypen polarisiert. Die M1-Polarisation wurde in Gegenwart von Interferon (IFN)-γ und Lipopolysaccharid (LPS) erreicht, während die M2-Polarisation in einem Medium durchgeführt wurde, das Interleukin (IL)-4 und IL-13 enthielt. Wir untersuchten Makrophagen-Phänotypen mittels Enzyme-linked Immunosorbent Assay (ELISA), konfokaler Laser-Scanning-Mikroskopie (CLSM) und quantitativer Echtzeit-PCR (qRT-PCR) auf der Grundlage von Panels von sezernierten Zytokinen, Zelloberflächenmarkern und exprimierten Genen. Die extrahierte RNA wurde in komplementäre DNA (cDNA) umgewandelt, und die qRT-PCR wurde verwendet, um mRNA zu quantifizieren, die mit M1- und M2-Makrophagen verwandt ist. Dementsprechend wurden M1-Makrophagen durch eine höhere Expression des proinflammatorischen Tumornekrosefaktors (TNF-α)-Zytokins und des CCR7-Oberflächenmarkers im Vergleich zu M2-Makrophagen, die höhere Spiegel von CD209 und CCL13 aufwiesen, charakterisiert. Folglich wurden CCR7 und CD209 als spezifische und zuverlässige Marker für M1- und M2-Makrophagen-Subtypen durch Immunfärbung und Visualisierung mittels CLSM identifiziert. Eine weitere Bestätigung wurde durch den ELISA erreicht, der einen erhöhten TNF-ɑ-Spiegel in M1 und einen erhöhten CCL13-Spiegel in M2-Zellen nachweiste. Die vorgeschlagenen Marker und der Versuchsaufbau können effektiv genutzt werden, um das immunmodulatorische Potenzial von Implantaten zu bewerten.
Einleitung
Implantierbare Biomaterialien sind zu einer konventionellen Lösung für verschiedene menschliche Krankheiten geworden und spielen eine große Rolle in der biomedizinischen Forschung, einschließlich Tissue Engineering, Drug Delivery Systems und Implantaten 1,2. Es gibt eine große Auswahl an Implantaten aus verschiedenen Materialien mit unterschiedlichen Strukturen und Funktionalitäten, wie z. B. Hüftprothesen, Stents, Netze, Herzklappen oder Zahnimplantate. Bei der Implantation provoziert der Gewebe-Implantat-Kontakt eine Immunantwort, gefolgt von Auflösung, Gewebeumbau und Homöostase. Diese Prozesse werden durch die physikalischen, chemischen und bioaktiven Eigenschaften der verwendeten Biomaterialien beeinflusst. Diese Eigenschaften können die Intensität und das Spektrum der pro- und antiinflammatorischen Reaktionen, die Bildung fibrotischer Kapseln, den Gewebeabbau und die Heilungsphase beeinflussen 3,4. Um den Heilungsprozess und die langfristige Implantatintegration zu unterstützen und zu optimieren, ist ein neuer Aspekt der aktuellen Forschung die Untersuchung und Vermittlung der Interaktion zwischen Implantatoberflächen und Immunzellen.
Makrophagen, die im ganzen Körper vorkommen, spielen unter anderem eine Schlüsselrolle bei Entzündungen und antipathogener Abwehr sowie bei Heilungsprozessen und der Aufrechterhaltung der Gewebehomöostase 5,6. Aufgrund ihrer Plastizität und der lokalen Mikroumgebungsreize des Gewebes sind Makrophagen in der Lage, in unterschiedliche funktionelle Phänotypen zu polarisieren, die große Unterschiede im Zellstoffwechsel, in den zellulären Funktionen und in den Zytokinsekretionsprofilen aufweisen. Der klassisch aktivierte M1-Phänotyp kann durch die Sekretion von proinflammatorischen Zytokinen wie IL-1β, IL-6 und TNF-α unterschieden werden und ist an der anfänglichen und chronischen Entzündungsreaktion auf Traumata und fremde Biomaterialien beteiligt. Im Gegensatz dazu weisen aktivierte M2-Makrophagen, die durch Zytokine wie IL-4 und IL-13 ausgelöst werden, charakteristische Merkmale wie die Auflösung von Entzündungen und die Förderung der Gewebeheilung auf. M2-polarisierte Makrophagen können durch die Expression von Zelloberflächenmarkern wie CD206 und die Produktion von Zytokinen wie IL-10 und IL-47 identifiziert werden. In ähnlicher Weise können sich Makrophagen, die bereits polarisiert wurden, in einer neuen Mikroumgebung neu programmieren.
Viele Studien zu Zell-Biomaterial-Wechselwirkungen haben die Bedeutung von Makrophagen in der Kaskade immunologischer Reaktionen auf implantierbare Biomaterialien und bei der Orchestrierung von Prozessen bei der Heilung implantatbedingter Komplikationen gezeigt 8,9,10. Auch wenn die Biomedizintechnik in den letzten Jahren erhebliche Fortschritte gemacht hat, sind weitere Forschungen erforderlich, um zu verstehen, wie Implantate das Verhalten und die Polarisation von Makrophagen modulieren 11,12,13.
In Zellkulturen können aus Monozyten stammende mononukleäre Zellen des peripheren Blutes (PBMCs) in adhärente M0-Makrophagen differenziert werden, gefolgt von einer induzierten Polarisation in Richtung M1- oder M2-Phänotypen unter Verwendung von LPS bzw. IFN-γ bzw. IL-4. Nach der Inkubation in vitro mit neuen Biomaterialproben ist es möglich, die verschiedenen Zelloberflächenrezeptoren und Zytokinprofile von M1- und M2-Makrophagen zu nutzen, um das immunmodulatorische Potenzial von Biomaterialien in vitro nachzuweisen 14,15. Ziel dieser Studie war es, ein in vitro Protokoll zu entwickeln, mit dem die Polarisation von MDMs als Reaktion auf verschiedene Implantatoberflächen untersucht werden kann. Genexpressionsanalysen, Mikroskopietechniken und ELISA können verwendet werden, um die phänotypischen Marker und spezifischen Zytokinprofile von M1- und M2-Makrophagen zu bestimmen, die durch das Biomaterial moduliert werden. So können die komplexen Wechselwirkungen zwischen Makrophagen und Biomaterialoberflächen aufgeklärt und wertvolle Informationen gewonnen werden, um Makrophagen-Biomaterial-Wechselwirkungen besser zu verstehen. Schließlich gewährleistet ein standardisiertes In-vitro-Protokoll die Reproduzierbarkeit, Zuverlässigkeit und Vergleichbarkeit der Versuchsergebnisse, indem die Variabilität im Versuchsaufbau minimiert wird.
Protokoll
Das humane periphere Blut wurde von gesunden Blutspendern gemäß dem von der Ethikkommission der Medizinischen Fakultät der Universität Tübingen genehmigten Protokoll gewonnen (Ethische Zulassung: 286/2021 BO). Humane PBMCs wurden mittels Dichtegradientenzentrifugation isoliert, wie zuvor beschrieben16. Das folgende Protokoll ist für PBMCs beschrieben, die aus 24 ml Blut isoliert wurden. Eine schematische Darstellung des Protokolls ist in Abbildung 1 dargestellt.
HINWEIS: Das Blutvolumen sollte in Abhängigkeit von der Anzahl der verwendeten M0-Makrophagen angepasst werden.
Aus 24 mL Blut wurden insgesamt 35,46 ± 9,1 Mio. PBMC gewonnen, was zu 1,97 ± 0,46 Mio. M0-Makrophagen (n = 5) führte. Alle Reagenzien, Verbrauchsmaterialien und Geräte sind in der Materialtabelle aufgeführt. Die Puffer sind in Tabelle 1 aufgeführt.
1. Differenzierung von Monozyten im menschlichen Blut zu Makrophagen
- Resuspendieren Sie die isolierten PBMCs in 15 mL vorgewärmtem Monozyten-Bindungsmedium und überführen Sie sie in einen T75-Zellkulturkolben.
- Die Zellen werden 90 Minuten lang bei 37 °C und 5 % CO2 inkubiert, um eine Adhäsion zu ermöglichen.
- Entsorgen Sie den Überstand und waschen Sie die Zellen einmal mit vorgewärmtem Vollmedium unter leichtem Kippen, um nicht anhaftende oder lose anhaftende Zellen zu entfernen.
ANMERKUNG: Bei den angehängten Zellen handelt es sich um Monozyten, die etwa 10 % der gesamten PBMC ausmachen, die ursprünglich in den Kolben gegeben wurden. - Geben Sie 15 ml vollständiges Medium mit 10 ng/ml Makrophagen-Kolonie-stimulierendem Faktor (M-CSF) zu den adhärenten Zellen und inkubieren Sie es 6 Tage lang, um die Differenzierung zu fördern.
- Tauschen Sie das Medium alle 2 Tage gegen ein frisches, vollständiges Medium mit 10 ng/mL M-CSF aus.
2. Kultivierung und Polarisation von MDMs auf der Titanimplantatoberfläche
HINWEIS: Am Tag 6 der Differenzierung wurden M0-Makrophagen 48 h lang mit unterschiedlichen Stimuli auf die Biomaterialoberflächen ausgesät, um vollständig polarisierte M1- oder M2-Makrophagen zu erhalten. Für jede untersuchte Oberfläche wurden drei Scheiben verwendet, um M0-, M1- und M2-Makrophagen zu säen. Als Kontrollflächen wurden mit Zellkulturen behandelte Kunststoffdeckgläser verwendet.
- Nehmen Sie das Kulturmedium aus den T75-Kolben und waschen Sie die Zellen 5 Minuten lang mit 10 mL PBS.
- Lösen Sie adhärente Zellen, indem Sie sie 30 Minuten lang mit 10 ml vorgewärmter Zellablösungslösung inkubieren.
- Klopfen Sie die Zellen vorsichtig ab und geben Sie sie in ein 50 mL Röhrchen. Entfernen Sie die verbleibenden Zellen, indem Sie sie vorsichtig in 10 ml PBS abkratzen.
- Übertragen Sie die abgelösten Zellen in das 50 mL Röhrchen und zentrifugieren Sie sie bei 300 x g für 10 min.
- Den Überstand verwerfen und die Zellen in 5 ml vorgewärmtem Komplettmedium resuspendieren.
- Zählen Sie die Zellen mit Trypanblau-Färbung und einem Hämozytometer, um die Zellzahl und Lebensfähigkeit zu bestimmen.
- Bereiten Sie die Zellsuspension vor, indem Sie die Zellzahl auf 160.000 Zellen pro 1 ml vollständigem Medium einstellen.
- Reinigen Sie die Biomaterial-Discs 5 Minuten lang mit Ultraschall in 70 % Ethanol, gefolgt von einer Sterilisation in 70 % Ethanol für 30 Minuten.
- Trocknen Sie die Titanscheiben 1-2 Stunden lang, legen Sie sie dann in unbehandelte 24-Well-Platten und geben Sie 1 ml vorbereitete Zellsuspension in jedes Well.
- Um M1-polarisierte Makrophagen zu erhalten, fügen Sie IFN-γ und LPS in einer Konzentration von 50 ng/ml bzw. 10 ng/ml hinzu. Für die M2-Polarisation fügen Sie IL-4 und IL-13 in einer Konzentration von jeweils 20 ng/ml hinzu. Stellen Sie sicher, dass die M0-Zellen ohne Polarisationsmittel gezüchtet werden. Inkubieren Sie die Zellen für weitere 48 Stunden bei 37 °C und 5 % CO2 , um eine Polarisation zu induzieren.
3. Charakterisierung polarisierter Makrophagen mittels ELISA
HINWEIS: Am Tag 2 der Polarisation wurden Proben für die Charakterisierungsanalysen vorbereitet. Das TNF-ɑ-Zytokin und das CCL13-Chemokin wurden gemessen, um M1- bzw. M2-polarisierte Makrophagen zu charakterisieren. Die Konzentration der sezernierten Proteine wurde auf die Gesamtkonzentration der sezernierten Proteine im entsprechenden Überstand normiert.
- Sammeln Sie den Überstand in einem 1,5-ml-Röhrchen und zentrifugieren Sie ihn 5 Minuten lang bei 300 x g . Den Überstand in ein neues Röhrchen umfüllen.
- Übertragen Sie die Discs für weitere Experimente auf eine neue 24-Well-Platte. Ziel ist es, tote oder lose anhaftende Zellen in unbehandelten 24-Well-Platten zu eliminieren.
HINWEIS: Die Viabilität der Zellen sollte an Oberflächen überprüft werden, bevor eine Analyse durchgeführt wird. Die Lebensfähigkeit von Zellen kann durch Viabilitätstests für lebende/tote Zellen oder indirekt durch Zellproliferations- und Zytotoxizitätstests bestimmt werden. - Messen Sie die Zytokine und Chemokine gemäß den spezifischen Anweisungen des Herstellers. Messen Sie die Zytokine entweder sofort oder lagern Sie die Proben bei -80 °C für zukünftige Messungen.
HINWEIS: Für jeden Kit-Typ sollte (unter Berücksichtigung der Empfindlichkeit und der Nachweisgrenzen des ELISA-Kits) eine geeignete Probenverdünnung bestimmt werden. Für den Bicinchoninsäure (BCA)-Assay wurden die Proben im Verhältnis 1:5 verdünnt. Die M1-Proben für TNF-ɑ wurden 1:10 und die M2-Proben für CCL13 1:12 verdünnt. - Berechnen Sie die Konzentration der sezernierten Zytokine/Chemokine unter Verwendung der Standardkurve gemäß den Anweisungen des Herstellers.
- Messen Sie die Gesamtproteinmenge mit dem BCA-Protein-Assay-Kit.
- Normalisieren Sie die Konzentration der sekretierten Proteine auf mg des Gesamtproteins.
4. Charakterisierung polarisierter Makrophagen mittels CLSM
HINWEIS: Polarisierte Makrophagen wurden weiter charakterisiert, indem sie mit Antikörpern gegen CD209- und CCR7-Zelloberflächenmarker gefärbt wurden. Die Kerne wurden mit DRAQ5 gegengefärbt. CD68 oder andere Marker können als Pan-Makrophagen-Marker verwendet werden.
- Waschen Sie die Zellen 2x in 800 μL PBS. Die Scheiben werden 20 Minuten lang bei Raumtemperatur (RT) in 400 μl Fixationspuffer inkubiert.
- Nach dem Entfernen des Fixierpuffers dreimal in 400 μl PBS waschen. Die Proben sofort färben oder bei 4 °C in 1 mL Lagerpuffer lagern.
HINWEIS: Mit diesem Fixierungs- und Lagerungsprotokoll wurden die Proben innerhalb von 1-6 Wochen nach der Fixierung erfolgreich gefärbt und abgebildet. - Waschen Sie die Scheiben vor dem nächsten Schritt nach der Lagerung zweimal mit 800 μl PBS.
- Inkubieren Sie Discs mit 400 μl Blockierungspuffer für 30 min bei RT, um die unspezifischen Bindungsstellen zu blockieren.
- Verwerfen Sie den Blockierungspuffer und inkubieren Sie die Discs 1 h lang bei RT mit Primärantikörpern, die in 400 μl Färbepuffer verdünnt sind.
- Führen Sie ein Immunfluoreszenzverfahren mit Doppelfärbung durch, um die Expression von CCR7 und CD209 in einer Probe zu untersuchen. Zu diesem Zweck werden Primärantikörper kombiniert, die von verschiedenen Spezies (Maus und Kaninchen) im selben Färbeschritt gezüchtet wurden.
HINWEIS: Um ein starkes Signal mit minimalem Hintergrund zu erzielen, optimieren Sie die Antikörperkonzentration. Der CCR7-Antikörper wurde in einer Endkonzentration von 10 μg/ml verwendet, und der CD209-Antikörper wurde um 1/400 verdünnt.
- Führen Sie ein Immunfluoreszenzverfahren mit Doppelfärbung durch, um die Expression von CCR7 und CD209 in einer Probe zu untersuchen. Zu diesem Zweck werden Primärantikörper kombiniert, die von verschiedenen Spezies (Maus und Kaninchen) im selben Färbeschritt gezüchtet wurden.
- Entfernen Sie die primären Antikörper und waschen Sie 3x mit 400 μl Waschpuffer.
- Fluorophor-markierte Sekundärantikörper, verdünnt in Färbepuffer, zugeben und 1 h bei RT im Dunkeln inkubieren.
HINWEIS: Die Konzentration der Sekundärantikörper sollte optimiert werden, um maximale spezifische Signale mit minimalem Hintergrund zu erhalten. In dieser Studie wurden Sekundärantikörper in einer Konzentration von 5 μg/mL zur Färbung verwendet. - Nach Entfernen des Überstands werden die Proben dreimal für jeweils 3 Minuten im Waschpuffer gewaschen.
- 10 μM DRAQ5 in PBS geben und 15 min bei RT lichtgeschützt inkubieren.
- Entfernen Sie den Überstand und waschen Sie die Scheiben einmal in PBS.
- Entfernen Sie das restliche PBS und fügen Sie 1 Tropfen Eindeckmedium hinzu.
- Nach 5 min setzen Sie die Deckgläser auf und lassen Sie die Proben 1 h trocknen.
- Nach dem Trocknen der Proben die Ränder mit klarem Nagellack versiegeln und bis zur Bildgebung bei 4 °C im Dunkeln lagern.
- Um einen Überblick über die Zellen zu erhalten, bilden Sie die Proben mit 25-facher Vergrößerung ab. Um die Struktur und Lokalisierung von Oberflächenmarkierungen weiter zu bestimmen, nehmen Sie Bilder mit 63-facher Vergrößerung auf.
- Quantifizieren Sie die Fluoreszenzintensität von CCR7 und CD209 mit der ImageJ-Software.
HINWEIS: Die Bildaufnahme wurde mit einem Photomultiplier (PMT) unter Verwendung eines CLSM-Systems durchgeführt, das mit einem Argon-Laser (488 nm), einem DPSS-Laser (561 nm) und einem HeNe-Laser (633 nm) ausgestattet war.
5. Charakterisierung polarisierter Makrophagen mittels qRT-PCR
HINWEIS: Für die RNA-Isolierung wurden zwei Scheiben pro Probe verwendet, um genügend RNA für die cDNA-Synthese zu erhalten.
- Waschen Sie die Scheiben 2x mit 800 μL PBS.
- Geben Sie 350 μl Lysepuffer auf die erste Scheibe und lysieren Sie die Zellen durch Auf- und Abpipettieren.
- Übertragen Sie das Lysat auf die zweite Scheibe und wiederholen Sie den Lysevorgang.
- Geben Sie 350 μl 70%iges Ethanol in das Lysat und pipettieren Sie es auf und ab, bis es homogen ist.
- Übertragen Sie das Lysat in die Spin-Säule und befolgen Sie die Anweisungen des Herstellers zur RNA-Isolierung.
- Quantifizieren Sie die RNA-Menge mit einem Nanotropfen oder einem anderen Gerät.
- Normalisieren Sie die RNA-Konzentrationen für verschiedene Proben und synthetisieren Sie cDNA gemäß den Anweisungen des Herstellers mit dem RT-PCR-System für die Erststrang-cDNA-Synthese.
- Synthetisieren Sie die cDNA mit 350 ng RNA gemäß dem Protokoll des Herstellers und lagern Sie sie bei -20 °C, bis die qRT-PCR-Analyse durchgeführt wird.
HINWEIS: Hier wurden 350 ng gereinigte RNA zur Synthese von cDNA unter Verwendung von 4 μl RT Mix (5x) in 20 μl verwendet. - Durchführung der qRT-PCR auf einem Real-Time-PCR-System in 96-Well-Platten und einzelnen 15-μl-Reaktionen (1x Syber Green Master Mix, 0,2 μM Forward- und Reverse-Primer und 4,5 μL 1:10 verdünnte cDNA).
HINWEIS: Das PCR-Programm beginnt mit einem beheizten Deckel (105 °C), gefolgt von drei Schritten: anfängliche Denaturierung bei 95 °C für 3 Minuten, gefolgt von 15 s bei 95 °C und 30 s bei 55 °C für 40 Zyklen. - Normalisieren Sie die Expressionsniveaus verschiedener Gene auf das Haushaltsgen GAPDH (oder andere Haushaltsgene wie β-Aktin).
- Berechnen Sie die relative Genexpression mit der 2-ΔΔCt-Methode , indem Sie die M0-Zellen, die auf Gewebekultur-Deckgläsern kultiviert wurden, als Referenz nehmen. Tabelle 2 listet alle in dieser Studie verwendeten Primer auf.
6. Statistische Analyse
- Präsentieren Sie alle Daten als Mittelwert ± REM. Wiederholen Sie alle Assays (in dieser Studie wurden die Assays fünfmal wiederholt), um die Reproduzierbarkeit zu gewährleisten. Bewerten Sie die statistisch signifikanten Unterschiede zwischen normalverteilten Daten mit einer unidirektionalen Varianzanalyse (ANOVA), gefolgt von dem Tukey-Mehrfachtest.
- Verwenden Sie den Friedman-Test und den Dunn-Mehrfachvergleichstest, um nicht-parametrische Datensätze zu analysieren. Verwenden Sie eine geeignete Datenanalysesoftware, um die Daten zu analysieren und definieren Sie die statistische Signifikanz als einen p-Wert von weniger als 0,05.
Ergebnisse
Die Ergebnisse dieser Studie zeigen die erfolgreiche Differenzierung und Polarisation von MDMs auf Titanoberflächen, gefolgt von der Charakterisierung von M1- oder M2-polarisierten Makrophagen. Im ersten Schritt haben wir polarisierte MDMs mittels CLSM charakterisiert. Basierend auf unseren Vorstudien wurden CD209 und CCR7 als spezifische Marker zur Unterscheidung von M1 von M2 polarisierten MDMs verwendet. Wie in Abbildung 2A,B gezeigt, polarisierten MDMs erfolgreich in M1- und M2-Makrophagen. Auf der Titanoberfläche wurde CCR7 in M1-polarisierten Makrophagen stärker exprimiert als CD209, spezifisch in M2-polarisierten Makrophagen. Darüber hinaus erleichterte die Quantifizierung der relativen Fluoreszenzintensität die Zuordnung von Markern zu M1- oder M2-Subtypen (Abbildung 2C).
Abbildung 3 zeigt eine repräsentative qRT-PCR-Analyse von MDMs auf Titan- und Deckglasoberflächen. Die Ergebnisse zeigten, dass MDMs auf beiden Oberflächen erfolgreich polarisiert wurden, was durch die hohe Expression der Polarisationsmarker M1 (CCR7 und TNF-ɑ) und M2 (CD209 und CCL13) gezeigt wurde. Dies wurde auf Proteinebene weiter bestätigt, indem hohe Konzentrationen von inflammatorischen TNF-α-Zytokinen (Abbildung 4A) und IL-13-Chemokinen (Abbildung 4B) in M1- bzw. M2-polarisierten Zellen beobachtet wurden.
Abbildung 1: Schematische Darstellung des Protokolls. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 2: Charakterisierung polarisierter MDMs mittels CLSM. Die Anreicherung von M1- und M2-polarisierten Makrophagen wurde durch spezifische Antikörperfärbung und CLSM-Analyse mit CCR7- und CD209-Antikörpern am Tag 2 der Nachsaat bestätigt. Laut Fluoreszenzfärbung und CLSM-Analyse (A) 25-fache Vergrößerung und (B) 63-fache Vergrößerung exprimierten M1-Zellen einen höheren CCR7-Wert (in grün) als M0 oder M2. M2-Zellen weisen ein signifikantes CD209-Expressionsmuster auf (in grün). (C) Die quantitative Analyse der relativen Fluoreszenzintensität von CCR7 und CD209. Die Zellkerne wurden mit DRAQ5 (in violett) angefärbt. Die Ergebnisse sind repräsentativ für 5 ähnliche Experimente, die unabhängig voneinander durchgeführt wurden. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Charakterisierung polarisierter MDMs anhand von Genexpressionsprofilen. Die quantitative reverse Transkriptionspolymerase wurde verwendet, um die mRNA-Spiegel verschiedener Gene zu untersuchen, die mit der Polarisation von M1 (CCR7 und TNF-ɑ) und M2 (CD209 und CCL13) assoziiert sind. GAPDH wurde als Haushalts-Gen verwendet. Die Daten werden als Mittelwert ± SEM (n = 5) dargestellt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
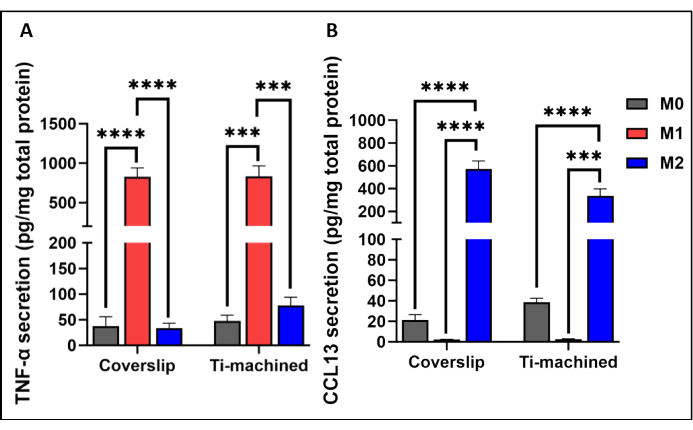
Abbildung 4: Produktion von Zytokinen durch M0-, M1- und M2-Makrophagen, die auf Titan- und Kunststoff-Deckgläsern kultiviert wurden. (A) TNF-ɑ-Zytokinspiegel und (B) CCL13-Spiegel in den Zellkulturüberständen wurden mittels ELISA gemessen. Die Zytokinsekretion wurde auf die Gesamtproteinsekretion normalisiert, gemessen mit einem BCA-Assay. Balkendiagramme stellen den Mittelwert ± SEM dar (n = 5). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
| Puffer/Lösung | Inhalt |
| Färbender/blockierender Puffer | PBS + 1 % BSA + 0,1 % Tween 20 |
| Puffer waschen | PBS + 0,1 % Tween 20 |
| Fixierungs-Puffer | 3% Paraformaldehyd (PFA) in PBS |
| Speicherpuffer | 1% Penicillin und Streptomycin in PBS |
Tabelle 1: Liste der Puffer.
| Name der Grundierung | Vorwärts-Primer-Sequenzen | Primer-Sequenzen umkehren |
| GAPDH | 5'-GAGTCAACGGATTTGGTCGT-3' | 5'-TTGATTTTGGAGGGATCTCG-3' |
| CCR7 | 5'- TGGTGATCGGCTTTCTGGTC-3' | 5'- CACCTTGATGGCCTTGTTGC-3' |
| Nr. CD209 | 5'- GGAGCAGAACTTCCTACAGC-3' | 5'- CAACGTTGTTGGGCTCTCCT-3' |
| CCL13 | 5'-ATCTCCTTGCAGAGGCTGAA-3' | 5'-ACTTCTCCTTTGGGTCAGCA-3' |
| TNF-ɑ | 5'- GCTGCACTTTGGAGTGATCG-3' | 5'- TCACTCGGGGTTCGAGAAGA-3' |
Tabelle 2. Sequenzen von Primern, die in der qRT-PCR verwendet werden.
Diskussion
Ein gründliches Verständnis des Verhaltens von Makrophagen ist unerlässlich, um die immunmodulatorischen Eigenschaften implantierbarer Materialien zu verstehen. Mehrere Studien haben über heterogene Marker, eine Vielzahl von Zellmodellen und Protokolle zur Charakterisierung der Makrophagenpolarisation in vitro berichtet 17,18,19,20. Um die Reproduzierbarkeit, Zuverlässigkeit und Vergleichbarkeit von Versuchsergebnissen zu verbessern, sind standardisierte und verifizierte Protokolle sowie ein geeignetes Zellmodell und Konsensus-Charakterisierungsmarker unerlässlich. Dementsprechend erfordert eine genaue Simulation der immunmodulatorischen Eigenschaften von Implantatmaterialien zunächst ein geeignetes Zellmodell. In verschiedenen Studien wurde eine breite Palette von Zellmodellen verwendet, wie z. B. isolierte Gewebemakrophagen, differenzierte Makrophagen aus Blutmonozyten und immortalisierte monozytäre Zelllinien. Obwohl gewebeisolierte Makrophagen als repräsentativer für In-vivo-Bedingungen angesehen werden können, sind sie technisch und ethisch anspruchsvoll21,22. Makrophagen werden häufig auch aus etablierten immortalisierten monozytären Zelllinien gewonnen, wie z. B. THP1-Zellen 23,24,25,26. Während diese Zellen als unbegrenzte Quelle für nicht-seneszente Zellen eine homogenere Zellantwort bieten können, werden sie in der Regel von Patienten mit hämatologischen Neoplasien gewonnen, und ihre Reaktionen können sich erheblich von normalen Zellen unterscheiden. Es wurde beispielsweise berichtet, dass monozytäre THP1-Zellen besser auf M1-Simulatoren ansprechen und mit größerer Wahrscheinlichkeit M1-Eigenschaften aufweisen22. Die Ergebnisse dieser Studie stimmen mit denen unserer Vorstudie überein (Daten werden hier nicht vorgestellt).
Da aus dem Blut stammende Monozyten als Vorläufer von geweberesidenten Makrophagen gelten und in höheren Ausbeuten leicht gewonnen werden können, können sie außerdem eine praktikable Alternative für Makrophagen sein 27,28,29. Basierend auf unserer Studie mit von Blutmonozyten abgeleiteten Makrophagen fanden wir heraus, dass diese Zellen gleichermaßen auf M1- und M2-Stimulatoren auf Titan- und Zellkultur-behandelten Deckgläsern reagierten. Darüber hinaus wiesen sie mehrere M1- und M2-Konsensusmarker auf, von denen einige in den repräsentativen Ergebnissen dargestellt sind. Die Ergebnisse zeigten, dass MDMs als praktikables in vitro Modell zur Simulation von Implantat-Makrophagen-Interaktionen verwendet werden können.
Für weitere Fortschritte in Immunmodulationsstudien sind konstante und spezifische Charakterisierungsmarker unerlässlich. Studien haben eine Vielzahl von Markern für die Charakterisierung von Makrophagen eingeführt, die sich nicht nur zwischen Makrophagen aus verschiedenen Quellen, sondern auch zwischen Makrophagen aus derselben Quelle unterscheiden 17,18,19,24. Ein Panel von M1- und M2-Markern, die für MDMs charakteristisch sind, wurde bestimmt und durch die Auswertung verschiedener berichteter Marker verifiziert. Einige der wichtigsten Schlüsselmarker werden in diesem Artikel vorgestellt.
Die Bestimmung der am besten geeigneten Nachweismethoden ist ebenfalls ein entscheidender Bestandteil des Bewertungsprozesses. Analysetechniken, die üblicherweise zur Beurteilung von Zelloberflächenmarkern verwendet werden, erfordern in der Regel die Entfernung von Zellen aus Biomaterialien. Es wurde jedoch beobachtet, dass sich dieser Prozess negativ auf die Zellen auswirkt, indem er ihre Oberflächenmarker beschädigt und zu einer geringen Anzahl abgelöster Zellen führt30. Folglich ist die Durchflusszytometrie, die eine Zellablösung erfordert, nicht geeignet, um Makrophagen zu bewerten, die fest an Implantaten befestigt sind. In dieser Studie wurde die Detektion von Zelloberflächenmarkern mittels CLSM durchgeführt. Durch die Verwendung der entsprechenden Marker und die Optimierung des Färbeprozesses konnten wir die M1- und M2-Subtypen erfolgreich untereinander und mit M0-Zellen vergleichen. Es ist wichtig zu beachten, dass die Fluoreszenzfärbung semi-quantitativ ist, was eine ihrer Einschränkungen ist. Dies kann die Bewertung von Zellen mit Markern erschweren, die über alle Subtypen hinweg ohne signifikante Unterschiede exprimiert werden. CCR7 und CD209 wurden nach dem Testen verschiedener Marker ausgewählt, um die MDMs mittels CLSM zu charakterisieren. CCR7 und CD209 waren in den Subtypen M1 bzw. M2 durchweg höher exprimiert.
Im Rahmen dieser Studie unterstreichen die Ergebnisse den Nutzen und die Wirksamkeit der implementierten Protokolle bei der Polarisierung von Makrophagen auf Implantatoberflächen und deren genauer Charakterisierung in Bezug auf Genexpression, sekretierte Proteine und Zelloberflächenmarker. Darüber hinaus ergab die Analyse der beschriebenen Marker konsistente und spezifische Expressionsmuster, die zur Unterscheidung verschiedener Subtypen von MDMs herangezogen werden können. Dieses In-vitro-Modell spiegelt jedoch die phänotypische Vielfalt und Plastizität menschlicher Makrophagen nicht vollständig wider. Mehrere Makrophagen-Subtypen (M2a, M2b, M2c, M2d) werden derzeit identifiziert, was auf einen Bedarf an vielfältigeren in vitro-Modellen hinweist, um zu untersuchen, wie verschiedene Biomaterialien und ihre Eigenschaften (z. B. physikalisch-chemische Eigenschaften) die Plastizität und Polarisation von Makrophagen beeinflussen31,32. Obwohl es nicht möglich ist, die komplexe In-vivo-Situation mit Hilfe von In-vitro-Modellen abzubilden, können mit dem vorgestellten In-vitro-Protokoll viele Ergebnisse erzielt werden, um das immunmodulatorische Potenzial neuer implantierbarer Biomaterialien effektiv zu beschreiben9. Nicht zuletzt ist eine weitere Etablierung notwendig, um Makrophagen in komplexeren in vitro- oder in vivo-Modellen zu charakterisieren, die die Rolle anderer Akteure in komplexen physiologischen Zusammenhängen einbeziehen. Insgesamt wird diese Studie zur Entwicklung und zum Design von immunmodulatorischen Biomaterialien beitragen, um günstige Geweberegenerationsprozesse und eine erfolgreiche Implantatintegration zu verbessern und zu fördern sowie implantatassoziierte chronische Entzündungen zu verhindern.
Offenlegungen
Die Autoren haben nichts offenzulegen.
Danksagungen
Die Scheiben wurden freundlicherweise zur Verfügung gestellt von Medentis Medical, Bad-Neuenahr-Ahrweiler, Deutschland. Die Autoren danken der Klinik für Mund-, Kiefer- und Gesichtschirurgie (Universitätsklinikum Tübingen) für die Unterstützung.
Materialien
| Name | Company | Catalog Number | Comments |
| 24-well plate, not-treated | Corning Incorporated (Kennebunk, USA) | 144530 | |
| Absorbance reader Infinite F50 | TECAN Austria GmbH (Grödig, Austria) | TCAT91000001 | |
| Accutase in DPBS, 0.5 mM EDTA | EMD Millipore Corp. (Burlington, USA) | SCR005 | |
| Anti-Fade Fluorescence Mounting Medium -Aqueous, Fluoroshield | abcam (Cambridge, UK) | ab104135 | |
| Bio-Rad MJ Research PTC-200 Peltier Thermal Cycler | Bio-Rad / MJ Research | 7212 | |
| Bovine serum albumin (BSA) | VWR International bvba (Leuven, Belgium) | 422361V | |
| Centrifuge 5804 R | Eppendorf SE (Hamburg, Germany) | 5804 R | |
| DC-SIGN (D7F5C) XP Rabbit mAb | Cell Signaling Technology | 13193 | |
| Dimethyl sulfoxide | Sigma Aldrich Co. (St.Louis, MO, USA) | D2438-5X10ML | |
| DRAQ5 Staining Solution | Milteny Biotec (Bergisch Gladbach, Germany) | 130-117-344 | |
| Ethanol ≥99.8% for molecularbiology | Carl Roth GmbH + CO. KG (Karlsruhe, Germany) | 1HPH.1 | |
| Goat Anti-Mouse IgG (H&L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor Plus 488 | Invitrogen | A32723TR | |
| Goat anti-Rabbit IgG (H+L) Cross-Adsorbed Secondary Antibody, Cyanine3 | Invitrogen | A10520 | |
| GraphPad Prism | GraphPad | Version 9.4.1 | |
| Human CCR7 Antibody | R&D Systems | MAB197 | |
| Human IFN-gamma Recombinant | Invitrogen (Rockford, USA) | RIFNG100 | |
| Human IL-13 | Milteny Biotec (Bergisch Gladbach, Germany) | 5230901032 | |
| Human IL-4 | Milteny Biotec (Bergisch Gladbach, Germany) | 130-095-373 | |
| Human M-CSF | Peprotech (Cranbury, USA) | 300-25 | |
| Leica TCS SP5 | Leica Microsystems CMS GmbH (Mannheim, Germany) | https://www.leica-microsystems.com/products/confocal-microscopes/p/leica-tcs-sp5/ | |
| Lipopolysaccharides from Escherichia | Sigma Aldrich Co. (Missouri, USA) | L4391-1MG | |
| Luna Universal qPCR Master Mix | New England Biolabs | NEB #M3003 | |
| LunaScript RT SuperMix Kit | New England Biolabs | E3010L | |
| Lymphocyte Separation Medium 1077 | PromoCell (Heidelberg, Germany) | C-44010 | |
| MCP-4/CCL13 Human ELISA Kit | Invitrogen | EHCCL13 | |
| MicroAmp Fast 96-Well Reaction Plate (0.1 mL) | Applied Biosystems (Waltham, USA) | 4346907 | |
| MicroAmp Optical Adhesive Film | Life Technologies Corporation (Carlsbad, USA) | 4311971 | |
| MicroAmp Splash Free 96-Well Base | Applied Biosystems (Waltham, USA) | 4312063 | |
| Microlitercentrifuge CD-3124R | Phoenix Instrument (Germany) | 9013111121 | |
| Microscope Cover Glasses, 10 mm | Carl Roth GmbH, Karlsruhe, Germany | 4HX4.1 | |
| Monocyte Attachment Medium | PromoCell (Heidelberg, Germany) | C-28051 | |
| Multiply-Pro Gefäß 0.5 mL, PP | Sarstedt AG & CO (Nümbrecht, Germany) | 7,27,35,100 | |
| Nanodrop One | Thermo Scientific (USA) | ND-ONE-W | |
| QuantStudio 3 System | Life Technologies GmbH (St. Leon-Rot, Germany) | A28567 | |
| RNeasy Micro Kit | Qiagen | 74007 | |
| RPMI 1640, 1x, with L-glutamine | Mediatech, Inc. (Manassas, USA) | 10-040-CV | |
| Sterile bench, LaminarAir HB 2472 | Heraeus instruments (Hanau, Germany) | 51012197 | |
| Tissue Culture Coverslips 13 mm (Plastic) | Sarstedt Inc. (Newton, USA) | 83,18,40,002 | |
| Titanium machinied discs 12 cm | Medentis Medical (Bad-Neuenahr-Ahrweiler, Germany) | N/A | |
| TNF alpha Human ELISA Kit | Invitrogen | KHC3011 | |
| Trypan blue solution 0.4% | Carl Roth GmbH + Co. KG (Karlsruhe, Germany) | 1680.1 |
Referenzen
- Othman, Z., Pastor, B. C., van Rijt, S., Habibovic, P. Understanding interactions between biomaterials and biological systems using proteomics. Biomaterials. 167, 191-204 (2018).
- Ikada, Y. Challenges in tissue engineering. J R Soc Interface. 3 (10), 589-601 (2006).
- Aamodt, J. M., Grainger, D. W. Extracellular matrix-based biomaterial scaffolds and the host response. Biomaterials. 86, 68-82 (2016).
- Batool, F., et al. Modulation of immune-inflammatory responses through surface modifications of biomaterials to promote bone healing and regeneration. J Tissue Eng. 12, 20417314211041428 (2021).
- Mantovani, A., Biswas, S. K., Galdiero, M. R., Sica, A., Locati, M. Macrophage plasticity and polarization in tissue repair and remodelling. J Pathol. 229 (2), 176-185 (2013).
- Shrivastava, R., Shukla, N. Attributes of alternatively activated (M2) macrophages. Life Sci. 224, 222-231 (2019).
- Gordon, S., Taylor, P. R. Monocyte and macrophage heterogeneity. Nat Rev Immunol. 5 (12), 953-964 (2005).
- Mosser, D. M., Edwards, J. P. Exploring the full spectrum of macrophage activation. Nat Rev Immunol. 8 (12), 958-969 (2008).
- Murray, P. J., et al. Macrophage activation and polarization: nomenclature and experimental guidelines. Immunity. 41 (1), 14-20 (2014).
- Kzhyshkowska, J., et al. Macrophage responses to implants: prospects for personalized medicine. J Leukoc Biol. 98 (6), 953-962 (2015).
- Browne, S., Pandit, A. Biomaterial-mediated modification of the local inflammatory environment. Front Bioeng Biotechnol. 3, 67 (2015).
- Piatnitskaia, S., et al. Modelling of macrophage responses to biomaterials in vitro: state-of-the-art and the need for the improvement. Front Immunol. 15, 1349461 (2024).
- Rayahin, J. E., Gemeinhart, R. A. Activation of macrophages in response to biomaterials. Results Probl Cell Differ. 62, 317-351 (2017).
- Murray, P. J. Macrophage polarization. Annu Rev Physiol. 79, 541-566 (2017).
- Gordon, S., Pluddemann, A. Tissue macrophages: heterogeneity and functions. BMC Biol. 15 (1), 53 (2017).
- Salma Iqbal, A. K. Characterization of in vitro generated human polarized macrophages. J Clin Cell Immunol. 6, 1-8 (2015).
- Hotchkiss, K. M., et al. Titanium surface characteristics, including topography and wettability, alter macrophage activation. Acta Biomater. 31, 425-434 (2016).
- Wang, Y., Zhang, Y., Sculean, A., Bosshardt, D. D., Miron, R. J. Macrophage behavior and interplay with gingival fibroblasts cultured on six commercially available titanium, zirconium, and titanium-zirconium dental implants. Clin Oral Investig. 23 (8), 3219-3227 (2019).
- Abaricia, J. O., Shah, A. H., Ruzga, M. N., Olivares-Navarrete, R. Surface characteristics on commercial dental implants differentially activate macrophages in vitro and in vivo. Clin Oral Implants Res. 32 (4), 487-497 (2021).
- Lu, W., et al. Improved osseointegration of strontium-modified titanium implants by regulating angiogenesis and macrophage polarization. Biomater Sci. 10 (9), 2198-2214 (2022).
- Murray, P. J., Wynn, T. A. Obstacles and opportunities for understanding macrophage polarization. J Leukoc Biol. 89 (4), 557-563 (2011).
- Nascimento, C. R., Fernandes, N. A. R., Maldonado, L. A. G., Junior, C. R. Comparison of monocytic cell lines U937 and THP-1 as macrophage models for in vitro studies. Biochem Biophys Rep. 32, 101383 (2022).
- Freytes, D. O., Kang, J. W., Marcos-Campos, I., Vunjak-Novakovic, G. Macrophages modulate the viability and growth of human mesenchymal stem cells. J Cell Biochem. 114 (1), 220-229 (2013).
- Zhang, Y., et al. Macrophage type modulates osteogenic differentiation of adipose tissue MSCs. Cell Tissue Res. 369 (2), 273-286 (2017).
- Cerqueira, A., et al. Evaluation of the inflammatory responses to sol-gel coatings with distinct biocompatibility levels. J Biomed Mater Res A. 109 (9), 1539-1548 (2021).
- Zhang, Y., Cheng, X., Jansen, J. A., Yang, F., van den Beucken, J. J. Titanium surfaces characteristics modulate macrophage polarization. Mater Sci Eng C Mater Biol Appl. 95, 143-151 (2019).
- Nobs, S. P., Kopf, M. Tissue-resident macrophages: guardians of organ homeostasis. Trends Immunol. 42 (6), 495-507 (2021).
- Sreejit, G., Fleetwood, A., Murphy, A., Nagareddy, P. Origins and diversity of macrophages in health and disease. Clin Transl Immunology. 9 (12), e1222 (2020).
- Parisi, L., et al. Preparation of human primary macrophages to study the polarization from monocyte-derived macrophages to pro-or anti-inflammatory macrophages at biomaterial interface in vitro. J Dent Sci. 18 (4), 1630-1637 (2023).
- Feuerer, N., et al. Macrophage retrieval from 3D biomaterials: A detailed comparison of common dissociation methods. J Immunol Regen Med. 11, 100035 (2021).
- Shapouri-Moghaddam, A., et al. Macrophage plasticity, polarization, and function in health and disease. J Cell Physiol. 233 (9), 6425-6440 (2018).
- Sridharan, R., Cameron, A. R., Kelly, D. J., Kearney, C. J., O'Brien, F. J. Biomaterial based modulation of macrophage polarization: a review and suggested design principles. Mater Today. 18 (6), 313-325 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten