Method Article
Polarización y caracterización de macrófagos derivados de monocitos humanos M1 y M2 en superficies de implantes
En este artículo
Resumen
En este artículo, presentamos un protocolo detallado para evaluar el potencial inmunomodulador de las superficies de los implantes in vitro, con el objetivo de mejorar la fiabilidad y la reproducibilidad de los protocolos actuales y promover nuevas investigaciones. Los perfiles de citocinas secretoras, la expresión de ARNm y los marcadores de superficie celular se monitorizaron utilizando macrófagos derivados de monocitos sanguíneos para investigar la polarización de los macrófagos cultivados en titanio.
Resumen
La reacción a cuerpos extraños (FBR), un complejo proceso de curación mediado por el sistema inmunitario, desempeña un papel crucial en la integración de los implantes en el cuerpo. Los macrófagos, como primera línea de interacción del sistema inmunitario con las superficies de los implantes, desempeñan un papel bidireccional en la modulación del equilibrio inflamación-regeneración. Para una comprensión profunda y la evaluación de las reacciones entre los materiales de los implantes y las respuestas inmunitarias, son fundamentales los métodos y protocolos in vitro fiables. Entre los diferentes modelos in vitro , los macrófagos primarios derivados de monocitos (MDM) presentan un excelente modelo para investigar las interacciones entre macrófagos e implantes. Hemos implementado un protocolo experimental para evaluar la polarización de MDMs en macrófagos M1 (activados clásicamente) y M2 (activados alternativamente) en superficies de implantes. Aislados monocitos sanguíneos de donantes sanos y los diferenciamos en macrófagos utilizando el factor estimulante de colonias de macrófagos (M-CSF). Los macrófagos diferenciados se cultivaron en las superficies de los implantes y se polarizaron en subtipos M1 y M2. La polarización M1 se logró en presencia de interferón (IFN)-γ y lipopolisacárido (LPS), mientras que la polarización M2 se realizó en un medio que contenía interleucina (IL)-4 e IL-13. Evaluamos los fenotipos de macrófagos mediante ensayo de inmunoabsorción ligado a enzimas (ELISA), microscopía de barrido láser confocal (CLSM) y PCR cuantitativa en tiempo real (qRT-PCR) basada en paneles de citocinas secretadas, marcadores de superficie celular y genes expresados. El ARN extraído se transformó en ADN complementario (ADNc) y se utilizó qRT-PCR para cuantificar el ARNm relacionado con los macrófagos M1 y M2. En consecuencia, los macrófagos M1 se han caracterizado por una mayor expresión de citocinas proinflamatorias del factor de necrosis tumoral (TNF-α) y el marcador de superficie CCR7 en comparación con los macrófagos M2, que exhibieron niveles más altos de CD209 y CCL13. En consecuencia, CCR7 y CD209 se identificaron como marcadores específicos y fiables de los subtipos de macrófagos M1 y M2 mediante inmunotinción y visualización por CLSM. Se logró una confirmación adicional mediante ELISA que detectó un nivel elevado de TNF-ɑ en M1 y un aumento de CCL13 en células M2. Los marcadores propuestos y la configuración experimental se pueden utilizar eficazmente para evaluar el potencial inmunomodulador de los implantes.
Introducción
Los biomateriales implantables se han convertido en una solución convencional para diversas enfermedades humanas y desempeñan un gran papel en la investigación biomédica, incluida la ingeniería de tejidos, los sistemas de administración de fármacos y los implantes 1,2. Existe una amplia gama de implantes fabricados en diversos materiales con diferentes estructuras y funcionalidades, como prótesis de cadera, stents, mallas, válvulas cardíacas o implantes dentales. Tras la implantación, el contacto tejido-implante provoca una respuesta inmunitaria, seguida de resolución, remodelación del tejido y homeostasis. Estos procesos están influenciados por las características físicas, químicas y bioactivas de los biomateriales utilizados. Estas características pueden afectar la intensidad y el espectro de las respuestas proinflamatorias y antiinflamatorias, la formación de cápsulas fibróticas, la degradación de los tejidos y la fase de curación 3,4. Con el fin de apoyar y optimizar el proceso de cicatrización y la integración a largo plazo del implante, un aspecto emergente de la investigación actual es investigar y mediar la interacción entre las superficies del implante y las células inmunitarias.
Entre otras células inmunitarias, los macrófagos, que se encuentran en todo el cuerpo, son actores clave en la inflamación y la defensa antipatógena, así como en los procesos de curación y el mantenimiento de la homeostasis de los tejidos 5,6. En función de su plasticidad y de los estímulos microambientales de los tejidos locales, los macrófagos son capaces de polarizarse en distintos fenotipos funcionales, que presentan grandes diferencias en el metabolismo celular, las funciones celulares y los perfiles de secreción de citocinas. El fenotipo M1 activado clásicamente se puede distinguir por la secreción de citocinas proinflamatorias, como IL-1β, IL-6 y TNF-α, y está involucrado en la respuesta inflamatoria inicial y crónica al trauma y a biomateriales extraños. Por el contrario, los macrófagos M2 activados, que son activados por citocinas como la IL-4 y la IL-13, tienen rasgos característicos como la resolución de la inflamación y la promoción de la curación de los tejidos. Los macrófagos polarizados M2 pueden identificarse por la expresión de marcadores de superficie celular como CD206 y la producción de citocinas como IL-10 e IL-47. Del mismo modo, los macrófagos que ya han sido polarizados pueden reprogramarse en un nuevo microambiente.
Muchos estudios sobre las interacciones célula-biomaterial han demostrado la importancia de los macrófagos en la cascada de respuestas inmunológicas hacia biomateriales implantables y en la orquestación de procesos involucrados en la curación de complicaciones relacionadas con implantes 8,9,10. A pesar de que la ingeniería biomédica ha logrado avances significativos en los últimos años, se necesita más investigación para comprender cómo los implantes modulan el comportamiento y la polarización de los macrófagos 11,12,13.
En cultivo celular, las células mononucleares de sangre periférica (PBMC) derivadas de monocitos se pueden diferenciar en macrófagos M0 adherentes seguidos de una polarización inducida hacia los fenotipos M1 o M2 utilizando LPS e IFN-γ o IL-4, respectivamente. Después de la incubación in vitro con nuevos especímenes de biomateriales, es posible utilizar los diferentes receptores de superficie celular y perfiles de citocinas de macrófagos M1 y M2 para detectar el potencial inmunomodulador de los biomateriales in vitro14,15. Este estudio tuvo como objetivo desarrollar un protocolo in vitro que pueda emplearse para investigar la polarización de los MDM en respuesta a diferentes superficies de implantes. Los análisis de expresión génica, las técnicas de microscopía y ELISA se pueden utilizar para determinar los marcadores fenotípicos y los perfiles de citocinas específicas de los macrófagos M1 y M2 modulados por el biomaterial. Por lo tanto, se pueden dilucidar las complejas interacciones entre los macrófagos y las superficies de los biomateriales, y se puede obtener información valiosa para comprender mejor las interacciones entre los macrófagos y los biomateriales. Por último, un protocolo in vitro estandarizado garantiza la reproducibilidad, fiabilidad y comparabilidad de los resultados experimentales al minimizar la variabilidad en la configuración experimental.
Protocolo
La sangre periférica humana se obtuvo de donantes de sangre sanos de acuerdo con el protocolo aprobado por el Comité de Ética de la Facultad de Medicina de la Universidad de Tübingen (aprobación ética: 286/2021 BO). Las PBMC humanas se aislaron mediante centrifugación en gradiente de densidad, como se describió anteriormente16. Se describe el siguiente protocolo para las PBMC aisladas de 24 mL de sangre. En la Figura 1 se muestra una representación esquemática del protocolo.
NOTA: El volumen sanguíneo debe ajustarse en función del número de macrófagos M0 utilizados.
Se obtuvieron un total de 35,46 ± 9,1 millones de PBMC a partir de 24 mL de sangre, lo que resultó en 1,97 ± 0,46 millones de macrófagos M0 (n = 5). Todos los reactivos, consumibles y dispositivos se enumeran en la Tabla de materiales. Los búferes se enumeran en la Tabla 1.
1. Diferenciación de monocitos sanguíneos humanos a macrófagos
- Vuelva a suspender las PBMC aisladas en 15 mL de medio de fijación de monocitos precalentado y transfiéralas a un matraz de cultivo celular T75.
- Incubar las células a 37 °C y 5% de CO2 durante 90 min para permitir la adhesión.
- Deseche el sobrenadante y lave las células una vez con un medio completo precalentado con una inclinación suave para eliminar las células no adherentes o poco adheridas.
NOTA: Las células adheridas son monocitos, que constituyen alrededor del 10% del total de PBMC añadidos originalmente al matraz. - Añadir 15 mL de medio completo que contenga 10 ng/mL de factor estimulante de colonias de macrófagos (M-CSF) a las células adherentes e incubar durante 6 días para promover la diferenciación.
- Cambie el medio por un medio fresco y completo que contenga 10 ng/mL M-CSF cada 2 días.
2. Cultivo y polarización de MDMs en la superficie del implante de titanio
NOTA: En el día 6 de diferenciación, se sembraron macrófagos M0 en las superficies del biomaterial con diferentes estímulos durante 48 h para obtener macrófagos M1 o M2 completamente polarizados. Para cada superficie examinada, se utilizaron tres discos para sembrar macrófagos M0, M1 y M2. Se utilizaron cubreobjetos de plástico tratados con cultivos celulares como superficies de control.
- Retire el medio de cultivo de los matraces T75 y lave las células con 10 mL de PBS durante 5 min.
- Separe las células adherentes incubándolas con 10 mL de solución de desprendimiento de células precalentada durante 30 min.
- Golpee suavemente las células y transfiéralas a un tubo de 50 ml. Separe las células restantes raspándolas suavemente en 10 ml de PBS.
- Transfiera las células separadas al tubo de 50 ml y centrifuérrelas a 300 x g durante 10 min.
- Deseche el sobrenadante y vuelva a suspender las células en 5 mL de medio completo precalentado.
- Cuente las células usando tinción con azul de tripán y un hemocitómetro para determinar el número de células y la viabilidad.
- Prepare la suspensión celular ajustando el número de células a 160,000 células por 1 mL de medio completo.
- Limpie los discos de biomaterial por ultrasonidos en etanol al 70% durante 5 min, seguido de la esterilización en etanol al 70% durante 30 min.
- Seque los discos de titanio durante 1-2 h, luego colóquelos en placas de 24 pocillos sin tratar y agregue 1 mL de suspensión celular preparada a cada pocillo.
- Para obtener macrófagos polarizados M1, agregue IFN-γ y LPS en la concentración de 50 ng/mL y 10 ng/mL, respectivamente. Para la polarización M2, agregue IL-4 e IL-13 en una concentración de 20 ng/mL cada una. Asegúrese de que las células M0 crezcan sin ningún agente de polarización. Incubar las células durante otras 48 h a 37 °C y 5% de CO2 para inducir la polarización.
3. Caracterización de macrófagos polarizados mediante ELISA
NOTA: En el día 2 de polarización, se prepararon muestras para los análisis de caracterización. Se midieron la citocina TNF-ɑ y la quimiocina CCL13 para caracterizar macrófagos M1 y M2 polarizados, respectivamente. La concentración de proteínas secretadas se normalizó a la concentración total de proteínas secretadas en el sobrenadante correspondiente.
- Recoja el sobrenadante en un tubo de 1,5 ml y centrifugue a 300 x g durante 5 min. Transfiera el sobrenadante a un tubo nuevo.
- Transfiera los discos a una nueva placa de 24 pocillos para realizar más experimentos. El propósito es eliminar las células muertas o sueltas en placas de 24 pocillos no tratadas.
NOTA: La viabilidad de la célula debe verificarse sobre las superficies antes de realizar cualquier análisis. La viabilidad de las células puede determinarse mediante ensayos de viabilidad de células vivas/muertas o indirectamente mediante pruebas de proliferación celular y citotoxicidad. - Mida las citocinas y quimiocinas de acuerdo con las instrucciones específicas proporcionadas por el fabricante. Mida las citocinas inmediatamente o almacene las muestras a -80 °C para futuras mediciones.
NOTA: Para cada tipo de kit (teniendo en cuenta la sensibilidad y los límites de detección del kit ELISA), se debe determinar la dilución adecuada de la muestra. Para el ensayo de ácido bicinconínico (BCA), las muestras se diluyeron 1:5. Las muestras M1 para TNF-ɑ se diluyeron 1:10 y las muestras M2 para CCL13 se diluyeron 1:12. - Calcule la concentración de citocinas/quimiocinas secretadas utilizando la curva estándar siguiendo las instrucciones del fabricante.
- Mida la cantidad total de proteína con el kit de ensayo de proteínas BCA.
- Normalizar la concentración de proteínas secretadas a mg de proteína total.
4. Caracterización de macrófagos polarizados mediante CLSM
NOTA: Los macrófagos polarizados se caracterizaron aún más tiñéndolos con anticuerpos contra los marcadores de superficie celular CD209 y CCR7. Los núcleos se contratiñeron con DRAQ5. CD68 u otros marcadores se pueden utilizar como marcadores panmacrófagos.
- Lave las células 2 veces en 800 μL de PBS. Incubar los discos durante 20 min a temperatura ambiente (RT) en 400 μL de tampón de fijación.
- Después de retirar el tampón de fijación, lavar tres veces con 400 μL de PBS. Tiñir las muestras inmediatamente o almacenar a 4 °C en 1 ml de tampón de almacenamiento.
NOTA: Con este protocolo de fijación y almacenamiento, las muestras se tiñeron y se tomaron imágenes con éxito dentro de 1 a 6 semanas después de la fijación. - Antes del siguiente paso, lave los discos dos veces con 800 μL de PBS después del almacenamiento.
- Incubar discos con 400 μL de tampón de bloqueo durante 30 min en RT para bloquear los sitios de unión inespecíficos.
- Deseche el tampón de bloqueo e incube los discos en RT durante 1 h con anticuerpos primarios diluidos en 400 μL de tampón de tinción.
- Realizar un procedimiento de inmunofluorescencia mediante tinción doble para examinar la expresión de CCR7 y CD209 en una muestra. Para ello, combine los anticuerpos primarios obtenidos de diferentes especies (ratón y conejo) en el mismo paso de tinción.
NOTA: Para lograr una señal fuerte con un fondo mínimo, optimice la concentración de anticuerpos. El anticuerpo CCR7 se utilizó a una concentración final de 10 μg/mL, y el anticuerpo CD209 se diluyó 1/400.
- Realizar un procedimiento de inmunofluorescencia mediante tinción doble para examinar la expresión de CCR7 y CD209 en una muestra. Para ello, combine los anticuerpos primarios obtenidos de diferentes especies (ratón y conejo) en el mismo paso de tinción.
- Retire los anticuerpos primarios y lave 3 veces con 400 μL de tampón de lavado.
- Añada anticuerpos secundarios marcados con fluoróforos diluidos en tampón de tinción e incube durante 1 h a RT en la oscuridad.
NOTA: La concentración de anticuerpos secundarios debe optimizarse para obtener el máximo de señales específicas con un mínimo de fondo. En este estudio, se utilizaron anticuerpos secundarios en una concentración de 5 μg/mL para la tinción. - Después de retirar el sobrenadante, lave las muestras tres veces en el tampón de lavado durante 3 minutos cada una.
- Añadir 10 μM de DRAQ5 en PBS e incubar durante 15 min en RT, protegido de la luz.
- Retire el sobrenadante y lave los discos una vez en PBS.
- Retire el PBS restante y agregue 1 gota de medio de montaje.
- Pasados 5 min aplicar los cubreobjetos y dejar secar las muestras durante 1 h.
- Después de secar las muestras, selle los bordes con esmalte de uñas transparente y guárdelas a 4 °C en la oscuridad hasta la obtención de imágenes.
- Para obtener una visión general de las celdas, obtenga imágenes de las muestras con un aumento de 25x. Para determinar aún más la estructura y la localización de los marcadores de superficie, adquiera imágenes con un aumento de 63x.
- Cuantifique la intensidad de fluorescencia de CCR7 y CD209 utilizando el software ImageJ.
NOTA: La adquisición de imágenes se realizó con un fotomultiplicador (PMT) utilizando un sistema CLSM, que estaba equipado con un láser de argón (488 nm), un láser DPSS (561 nm) y un láser HeNe (633 nm).
5. Caracterización de macrófagos polarizados mediante qRT-PCR
NOTA: Para el aislamiento de ARN, se utilizaron dos discos por muestra con el fin de obtener suficiente ARN para la síntesis de ADNc.
- Lave los discos 2 veces con 800 μL de PBS.
- Añadir 350 μL de tampón de lisis al primer disco y lisar las células pipeteando hacia arriba y hacia abajo.
- Transfiera el lisado al segundo disco y repita el proceso de lisado.
- Añadir 350 μL de etanol al 70% al lisado y pipetear hacia arriba y hacia abajo hasta que quede homogéneo.
- Transfiera el lisado a la columna de centrifugación y siga las instrucciones del fabricante para el aislamiento de ARN.
- Cuantifique la cantidad de ARN utilizando una nanogota u otro dispositivo.
- Normalice las concentraciones de ARN para diferentes muestras y sintetice el ADNc según las instrucciones del fabricante utilizando el sistema RT-PCR para la síntesis de ADNc de primera cadena.
- Sintetizar el ADNc utilizando 350 ng de ARN siguiendo el protocolo del fabricante y almacenarlo a -20 °C hasta que se realice el análisis qRT-PCR.
NOTA: Aquí, se utilizaron 350 ng de ARN purificado para sintetizar ADNc utilizando 4 μL de RT Mix (5x) en 20 μL. - Realice qRT-PCR en un sistema de PCR en tiempo real en placas de 96 pocillos y reacciones individuales de 15 μL (1x Syber green master mix, 0,2 μM de cebadores directos e inversos y 4,5 μL de ADNc diluido 1:10).
NOTA: El programa de PCR comienza con una tapa calentada (105 °C) seguida de tres pasos: desnaturalización inicial a 95 °C durante 3 min, seguida de 15 s a 95 °C y 30 s a 55 °C durante 40 ciclos. - Normalizar los niveles de expresión de varios genes al gen de mantenimiento GAPDH (u otros genes de mantenimiento como la β-actina).
- Calcule la expresión génica relativa utilizando el método 2−ΔΔCt tomando como referencia las células M0 cultivadas en cubreobjetos de cultivo de tejidos. En la Tabla 2 se enumeran todos los cebadores utilizados en este estudio.
6. Análisis estadístico
- Presentar todos los datos como la media ± SEM. Repetir todos los ensayos (en este estudio, los ensayos se repitieron cinco veces) para garantizar la reproducibilidad. Evalúe las diferencias estadísticamente significativas entre los datos distribuidos normalmente utilizando un análisis de varianza unidireccional (ANOVA) seguido de la prueba múltiple de Tukey.
- Utilice la prueba de Friedman y la prueba de comparación múltiple de Dunn para analizar conjuntos de datos no paramétricos. Utilice un software de análisis de datos adecuado para analizar los datos y definir la significación estadística como un valor p inferior a 0,05.
Resultados
Los resultados de este estudio demuestran el éxito de la diferenciación y polarización de los MDM en superficies de titanio, seguido de la caracterización de los macrófagos polarizados M1 o M2. En el primer paso, caracterizamos los MDM polarizados utilizando CLSM. Según nuestros estudios preliminares, CD209 y CCR7 se utilizaron como marcadores específicos para diferenciar los MDM polarizados M1 de los M2. Como se muestra en la Figura 2A, B, los MDM se polarizaron con éxito en macrófagos M1 y M2. En la superficie del titanio, CCR7 se expresó con más fuerza en los macrófagos polarizados M1 que CD209, expresado específicamente en los macrófagos polarizados M2. Además, la cuantificación de la intensidad relativa de fluorescencia facilitó la asignación de marcadores a los subtipos M1 o M2 (Figura 2C).
La Figura 3 muestra un análisis representativo de qRT-PCR de MDM en superficies de titanio y cubreobjetos. Los resultados mostraron que los MDM en ambas superficies se polarizaron con éxito, como lo demuestra la alta expresión de los marcadores de polarización M1 (CCR7 y TNF-ɑ) y M2 (CD209 y CCL13). Esto se confirmó aún más a nivel de proteínas al observar altos niveles de citocinas inflamatorias de TNF-α (Figura 4A) y quimiocinas IL-13 (Figura 4B) en células polarizadas M1 y M2, respectivamente.
Figura 1: Representación esquemática del protocolo. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Caracterización de MDMs polarizados utilizando CLSM. El enriquecimiento de macrófagos polarizados M1 y M2 se confirmó mediante tinción de anticuerpos específicos y análisis CLSM utilizando anticuerpos CCR7 y CD209 en el día 2 de la possiembra. De acuerdo con la tinción de fluorescencia y el análisis CLSM, (A) con un aumento de 25x y (B) con un aumento de 63x, las células M1 expresaron un CCR7 más alto (en verde) que M0 o M2. Las células M2 exhiben un patrón de expresión significativo de CD209 (en verde). (C) El análisis cuantitativo de la intensidad relativa de fluorescencia de CCR7 y CD209. Los núcleos se tiñeron con DRAQ5 (en púrpura). Los resultados son representativos de 5 experimentos similares realizados de forma independiente. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Caracterización de MDMs polarizados mediante perfiles de expresión génica. Se utilizó la polimerasa cuantitativa con transcriptasa inversa para estudiar los niveles de ARNm de diferentes genes asociados con la polarización de M1 (CCR7 y TNF-ɑ) y M2 (CD209 y CCL13). Se utilizó GAPDH como gen de limpieza. Los datos se presentan como media ± SEM (n = 5). Haga clic aquí para ver una versión más grande de esta figura.
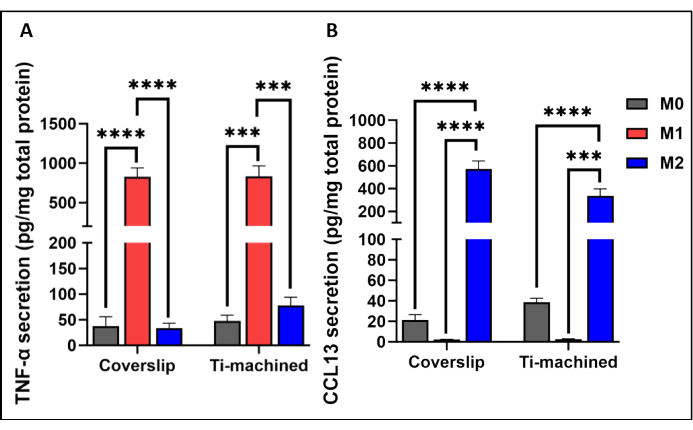
Figura 4: Producción de citocinas por macrófagos M0, M1 y M2 cultivados en cubreobjetos de titanio y plástico. (A) El nivel de citocinas TNF-ɑ y (B) los niveles de CCL13 en los sobrenadantes de cultivo celular se midieron utilizando ELISA. La secreción de citocinas se normalizó a la secreción total de proteínas medida por el ensayo BCA. Los gráficos de barras representan la media ± SEM (n = 5). Haga clic aquí para ver una versión más grande de esta figura.
| Tampón/Solución | Contenido |
| Tampón de tinción/bloqueo | PBS + 1% BSA + 0.1% Tween 20 |
| Tampón de lavado | PBS + 0.1% Preadolescente 20 |
| Tampón de fijación | 3% de paraformaldehído (PFA) en PBS |
| Búfer de almacenamiento | 1% de penicilina y estreptomicina en PBS |
Tabla 1: Lista de búferes.
| Nombre del cebador | Secuencias de cebadores hacia adelante | Secuencias de cebadores inversas |
| GAPDH | 5'-GAGTCAACGGATTTGGTCGT-3' | 5'-TTGATTTTGGAGGGATCTCG-3' |
| CCR7 | 5'- TGGTGATCGGCTCTCTCTTC-3' | 5'- CACCTTGATGGCCTTGTTGC-3' |
| CD209 | 5'- GGAGCAGAACTTCCTACAGC-3' | 5'- CAACGTTGTTGGGCTCTCCT-3' |
| CCL13 | 5'-ATCTCCTTGCAGAGGCTGAA-3' | 5'-ACTTCTCCTTTGGGTCAGCA-3' |
| TNF-ɑ | 5'- GCTGCACTTTGGAGTGATCG-3' | 5'- TCACTCGGGGTTCGAGAAGA-3' |
Tabla 2. Secuencias de cebadores utilizados en qRT-PCR.
Discusión
Una comprensión profunda del comportamiento de los macrófagos es esencial para comprender las propiedades inmunomoduladoras de los materiales implantables. Varios estudios han reportado marcadores heterogéneos, una variedad de modelos celulares y protocolos para caracterizar la polarización de macrófagos in vitro 17,18,19,20. Para mejorar la reproducibilidad, fiabilidad y comparabilidad de los resultados experimentales, son esenciales protocolos estandarizados y verificados, junto con un modelo celular adecuado y marcadores de caracterización de consenso. En consecuencia, una simulación precisa de las propiedades inmunomoduladoras de los materiales de los implantes requiere primero un modelo celular adecuado. Diferentes estudios han utilizado una amplia gama de modelos celulares, como macrófagos tisulares aislados, macrófagos diferenciados derivados de monocitos sanguíneos y líneas celulares monocíticas inmortalizadas. Aunque los macrófagos aislados en tejido pueden considerarse más representativos de las condiciones in vivo, son técnica y éticamente desafiantes21,22. Los macrófagos también se obtienen con frecuencia de líneas celulares monocíticas inmortalizadas establecidas, como las células THP1 23,24,25,26. Si bien estas células pueden ofrecer una mayor homogeneidad en la respuesta celular como una fuente ilimitada de células no senescentes, generalmente se obtienen de pacientes con neoplasias hematológicas y sus respuestas pueden diferir significativamente de las células normales. Por ejemplo, se ha informado que las células monocíticas THP1 responden mejor a los simuladores M1 y es más probable que presenten características M122. Los resultados de este estudio están en línea con los de nuestro estudio preliminar (datos no presentados aquí).
Además, dado que los monocitos derivados de la sangre se consideran los precursores de los macrófagos residentes en los tejidos y pueden obtenerse fácilmente en mayores rendimientos, pueden ser una alternativa factible para los macrófagos 27,28,29. Basándonos en nuestro estudio con macrófagos derivados de monocitos sanguíneos, descubrimos que estas células respondían por igual a los estimuladores M1 y M2 tanto en cubreobjetos tratados con titanio como en cultivos celulares. Además, exhibieron múltiples marcadores de consenso M1 y M2, algunos de los cuales se muestran en los resultados representativos. Los resultados demostraron que los MDM pueden utilizarse como un modelo in vitro factible para simular las interacciones implante-macrófago.
Para futuros avances en los estudios de inmunomodulación, los marcadores de caracterización constantes y específicos son esenciales. Los estudios han introducido una variedad de marcadores para la caracterización de macrófagos que difieren no solo entre macrófagos de diferentes fuentes, sino también entre macrófagos de la misma fuente 17,18,19,24. Se determinó y verificó un panel de marcadores M1 y M2 característicos de los MDM mediante la evaluación de diferentes marcadores reportados. En este artículo se presentan algunos de los marcadores clave más importantes.
La determinación de los métodos de detección más apropiados también es una parte crucial del proceso de evaluación. Las técnicas de análisis comúnmente utilizadas para evaluar los marcadores de la superficie celular generalmente requieren la eliminación de células de los biomateriales. Sin embargo, se ha observado que este proceso afecta negativamente a las células al dañar sus marcadores de superficie y dar lugar a un bajo número de células desprendidas30. En consecuencia, la citometría de flujo, que requiere el desprendimiento de células, no es apropiada para evaluar macrófagos que están fuertemente adheridos a los implantes. En este estudio, la detección de marcadores de superficie celular se llevó a cabo mediante CLSM. Mediante el uso de los marcadores adecuados y la optimización del proceso de tinción, pudimos caracterizar con éxito los subtipos M1 y M2 en comparación entre sí y con las células M0. Es importante tener en cuenta que la tinción de fluorescencia es semicuantitativa, lo cual es una de sus limitaciones. Esto puede complicar la evaluación de las células utilizando marcadores expresados en todos los subtipos sin diferencias significativas. CCR7 y CD209 se seleccionaron después de probar diferentes marcadores para caracterizar los MDMs utilizando CLSM. CCR7 y CD209 se expresaron consistentemente más alto en los subtipos M1 y M2, respectivamente.
Dentro de las limitaciones de este estudio, los resultados destacan la utilidad y efectividad de los protocolos implementados para polarizar macrófagos en superficies de implantes y caracterizarlos con precisión en términos de expresión génica, proteínas secretadas y marcadores de superficie celular. Además, el análisis de los marcadores descritos reveló patrones de expresión consistentes y específicos que se pueden utilizar para distinguir diferentes subtipos de MDM. Este modelo in vitro, sin embargo, no refleja completamente la diversidad fenotípica y la plasticidad de los macrófagos humanos. En la actualidad se están identificando varios subtipos de macrófagos (M2a, M2b, M2c, M2d), lo que indica la necesidad de modelos in vitro más diversos para estudiar cómo diversos biomateriales y sus características (por ejemplo, propiedades fisicoquímicas) afectan la plasticidad y polarización de los macrófagos31,32. Aunque no es posible replicar la compleja situación in vivo utilizando modelos in vitro, se pueden obtener muchos resultados utilizando el protocolo in vitro presentado para describir eficazmente el potencial inmunomodulador de nuevos biomateriales implantables9. Por último, pero no menos importante, es necesario un mayor establecimiento para caracterizar los macrófagos en modelos in vitro o in vivo más complejos que involucran el papel de otros actores en contextos fisiológicos complejos. En general, este estudio contribuirá al desarrollo y diseño de biomateriales inmunomoduladores para mejorar y promover procesos favorables de regeneración tisular y una integración exitosa de implantes, así como para prevenir la inflamación crónica asociada a los implantes.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los discos fueron amablemente proporcionados por Medentis Medical, Bad-Neuenahr-Ahrweiler, Alemania. Los autores agradecen el apoyo del Servicio de Cirugía Oral y Maxilofacial del Hospital Universitario de Tubinga.
Materiales
| Name | Company | Catalog Number | Comments |
| 24-well plate, not-treated | Corning Incorporated (Kennebunk, USA) | 144530 | |
| Absorbance reader Infinite F50 | TECAN Austria GmbH (Grödig, Austria) | TCAT91000001 | |
| Accutase in DPBS, 0.5 mM EDTA | EMD Millipore Corp. (Burlington, USA) | SCR005 | |
| Anti-Fade Fluorescence Mounting Medium -Aqueous, Fluoroshield | abcam (Cambridge, UK) | ab104135 | |
| Bio-Rad MJ Research PTC-200 Peltier Thermal Cycler | Bio-Rad / MJ Research | 7212 | |
| Bovine serum albumin (BSA) | VWR International bvba (Leuven, Belgium) | 422361V | |
| Centrifuge 5804 R | Eppendorf SE (Hamburg, Germany) | 5804 R | |
| DC-SIGN (D7F5C) XP Rabbit mAb | Cell Signaling Technology | 13193 | |
| Dimethyl sulfoxide | Sigma Aldrich Co. (St.Louis, MO, USA) | D2438-5X10ML | |
| DRAQ5 Staining Solution | Milteny Biotec (Bergisch Gladbach, Germany) | 130-117-344 | |
| Ethanol ≥99.8% for molecularbiology | Carl Roth GmbH + CO. KG (Karlsruhe, Germany) | 1HPH.1 | |
| Goat Anti-Mouse IgG (H&L) Highly Cross-Adsorbed Secondary Antibody, Alexa Fluor Plus 488 | Invitrogen | A32723TR | |
| Goat anti-Rabbit IgG (H+L) Cross-Adsorbed Secondary Antibody, Cyanine3 | Invitrogen | A10520 | |
| GraphPad Prism | GraphPad | Version 9.4.1 | |
| Human CCR7 Antibody | R&D Systems | MAB197 | |
| Human IFN-gamma Recombinant | Invitrogen (Rockford, USA) | RIFNG100 | |
| Human IL-13 | Milteny Biotec (Bergisch Gladbach, Germany) | 5230901032 | |
| Human IL-4 | Milteny Biotec (Bergisch Gladbach, Germany) | 130-095-373 | |
| Human M-CSF | Peprotech (Cranbury, USA) | 300-25 | |
| Leica TCS SP5 | Leica Microsystems CMS GmbH (Mannheim, Germany) | https://www.leica-microsystems.com/products/confocal-microscopes/p/leica-tcs-sp5/ | |
| Lipopolysaccharides from Escherichia | Sigma Aldrich Co. (Missouri, USA) | L4391-1MG | |
| Luna Universal qPCR Master Mix | New England Biolabs | NEB #M3003 | |
| LunaScript RT SuperMix Kit | New England Biolabs | E3010L | |
| Lymphocyte Separation Medium 1077 | PromoCell (Heidelberg, Germany) | C-44010 | |
| MCP-4/CCL13 Human ELISA Kit | Invitrogen | EHCCL13 | |
| MicroAmp Fast 96-Well Reaction Plate (0.1 mL) | Applied Biosystems (Waltham, USA) | 4346907 | |
| MicroAmp Optical Adhesive Film | Life Technologies Corporation (Carlsbad, USA) | 4311971 | |
| MicroAmp Splash Free 96-Well Base | Applied Biosystems (Waltham, USA) | 4312063 | |
| Microlitercentrifuge CD-3124R | Phoenix Instrument (Germany) | 9013111121 | |
| Microscope Cover Glasses, 10 mm | Carl Roth GmbH, Karlsruhe, Germany | 4HX4.1 | |
| Monocyte Attachment Medium | PromoCell (Heidelberg, Germany) | C-28051 | |
| Multiply-Pro Gefäß 0.5 mL, PP | Sarstedt AG & CO (Nümbrecht, Germany) | 7,27,35,100 | |
| Nanodrop One | Thermo Scientific (USA) | ND-ONE-W | |
| QuantStudio 3 System | Life Technologies GmbH (St. Leon-Rot, Germany) | A28567 | |
| RNeasy Micro Kit | Qiagen | 74007 | |
| RPMI 1640, 1x, with L-glutamine | Mediatech, Inc. (Manassas, USA) | 10-040-CV | |
| Sterile bench, LaminarAir HB 2472 | Heraeus instruments (Hanau, Germany) | 51012197 | |
| Tissue Culture Coverslips 13 mm (Plastic) | Sarstedt Inc. (Newton, USA) | 83,18,40,002 | |
| Titanium machinied discs 12 cm | Medentis Medical (Bad-Neuenahr-Ahrweiler, Germany) | N/A | |
| TNF alpha Human ELISA Kit | Invitrogen | KHC3011 | |
| Trypan blue solution 0.4% | Carl Roth GmbH + Co. KG (Karlsruhe, Germany) | 1680.1 |
Referencias
- Othman, Z., Pastor, B. C., van Rijt, S., Habibovic, P. Understanding interactions between biomaterials and biological systems using proteomics. Biomaterials. 167, 191-204 (2018).
- Ikada, Y. Challenges in tissue engineering. J R Soc Interface. 3 (10), 589-601 (2006).
- Aamodt, J. M., Grainger, D. W. Extracellular matrix-based biomaterial scaffolds and the host response. Biomaterials. 86, 68-82 (2016).
- Batool, F., et al. Modulation of immune-inflammatory responses through surface modifications of biomaterials to promote bone healing and regeneration. J Tissue Eng. 12, 20417314211041428 (2021).
- Mantovani, A., Biswas, S. K., Galdiero, M. R., Sica, A., Locati, M. Macrophage plasticity and polarization in tissue repair and remodelling. J Pathol. 229 (2), 176-185 (2013).
- Shrivastava, R., Shukla, N. Attributes of alternatively activated (M2) macrophages. Life Sci. 224, 222-231 (2019).
- Gordon, S., Taylor, P. R. Monocyte and macrophage heterogeneity. Nat Rev Immunol. 5 (12), 953-964 (2005).
- Mosser, D. M., Edwards, J. P. Exploring the full spectrum of macrophage activation. Nat Rev Immunol. 8 (12), 958-969 (2008).
- Murray, P. J., et al. Macrophage activation and polarization: nomenclature and experimental guidelines. Immunity. 41 (1), 14-20 (2014).
- Kzhyshkowska, J., et al. Macrophage responses to implants: prospects for personalized medicine. J Leukoc Biol. 98 (6), 953-962 (2015).
- Browne, S., Pandit, A. Biomaterial-mediated modification of the local inflammatory environment. Front Bioeng Biotechnol. 3, 67 (2015).
- Piatnitskaia, S., et al. Modelling of macrophage responses to biomaterials in vitro: state-of-the-art and the need for the improvement. Front Immunol. 15, 1349461 (2024).
- Rayahin, J. E., Gemeinhart, R. A. Activation of macrophages in response to biomaterials. Results Probl Cell Differ. 62, 317-351 (2017).
- Murray, P. J. Macrophage polarization. Annu Rev Physiol. 79, 541-566 (2017).
- Gordon, S., Pluddemann, A. Tissue macrophages: heterogeneity and functions. BMC Biol. 15 (1), 53 (2017).
- Salma Iqbal, A. K. Characterization of in vitro generated human polarized macrophages. J Clin Cell Immunol. 6, 1-8 (2015).
- Hotchkiss, K. M., et al. Titanium surface characteristics, including topography and wettability, alter macrophage activation. Acta Biomater. 31, 425-434 (2016).
- Wang, Y., Zhang, Y., Sculean, A., Bosshardt, D. D., Miron, R. J. Macrophage behavior and interplay with gingival fibroblasts cultured on six commercially available titanium, zirconium, and titanium-zirconium dental implants. Clin Oral Investig. 23 (8), 3219-3227 (2019).
- Abaricia, J. O., Shah, A. H., Ruzga, M. N., Olivares-Navarrete, R. Surface characteristics on commercial dental implants differentially activate macrophages in vitro and in vivo. Clin Oral Implants Res. 32 (4), 487-497 (2021).
- Lu, W., et al. Improved osseointegration of strontium-modified titanium implants by regulating angiogenesis and macrophage polarization. Biomater Sci. 10 (9), 2198-2214 (2022).
- Murray, P. J., Wynn, T. A. Obstacles and opportunities for understanding macrophage polarization. J Leukoc Biol. 89 (4), 557-563 (2011).
- Nascimento, C. R., Fernandes, N. A. R., Maldonado, L. A. G., Junior, C. R. Comparison of monocytic cell lines U937 and THP-1 as macrophage models for in vitro studies. Biochem Biophys Rep. 32, 101383 (2022).
- Freytes, D. O., Kang, J. W., Marcos-Campos, I., Vunjak-Novakovic, G. Macrophages modulate the viability and growth of human mesenchymal stem cells. J Cell Biochem. 114 (1), 220-229 (2013).
- Zhang, Y., et al. Macrophage type modulates osteogenic differentiation of adipose tissue MSCs. Cell Tissue Res. 369 (2), 273-286 (2017).
- Cerqueira, A., et al. Evaluation of the inflammatory responses to sol-gel coatings with distinct biocompatibility levels. J Biomed Mater Res A. 109 (9), 1539-1548 (2021).
- Zhang, Y., Cheng, X., Jansen, J. A., Yang, F., van den Beucken, J. J. Titanium surfaces characteristics modulate macrophage polarization. Mater Sci Eng C Mater Biol Appl. 95, 143-151 (2019).
- Nobs, S. P., Kopf, M. Tissue-resident macrophages: guardians of organ homeostasis. Trends Immunol. 42 (6), 495-507 (2021).
- Sreejit, G., Fleetwood, A., Murphy, A., Nagareddy, P. Origins and diversity of macrophages in health and disease. Clin Transl Immunology. 9 (12), e1222 (2020).
- Parisi, L., et al. Preparation of human primary macrophages to study the polarization from monocyte-derived macrophages to pro-or anti-inflammatory macrophages at biomaterial interface in vitro. J Dent Sci. 18 (4), 1630-1637 (2023).
- Feuerer, N., et al. Macrophage retrieval from 3D biomaterials: A detailed comparison of common dissociation methods. J Immunol Regen Med. 11, 100035 (2021).
- Shapouri-Moghaddam, A., et al. Macrophage plasticity, polarization, and function in health and disease. J Cell Physiol. 233 (9), 6425-6440 (2018).
- Sridharan, R., Cameron, A. R., Kelly, D. J., Kearney, C. J., O'Brien, F. J. Biomaterial based modulation of macrophage polarization: a review and suggested design principles. Mater Today. 18 (6), 313-325 (2015).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados