Method Article
Generazione di organoidi del cervello umano per la modellazione della malattia mitocondriale
In questo articolo
Riepilogo
Descriviamo un protocollo dettagliato per la generazione di organoidi cerebrali derivati da cellule staminali pluripotenti indotte dall'uomo e il loro uso nella modellazione delle malattie mitocondriali.
Abstract
Le malattie mitocondriali rappresentano la più grande classe di errori congeniti del metabolismo e sono attualmente incurabili. Queste malattie causano difetti dello sviluppo neurologico i cui meccanismi sottostanti rimangono da chiarire. Un importante ostacolo è la mancanza di modelli efficaci che ricapitolino la compromissione neuronale ad esordio precoce osservata nei pazienti. I progressi nella tecnologia delle cellule staminali pluripotenti indotte (iPSC) consentono la generazione di organoidi cerebrali tridimensionali (3D) che possono essere utilizzati per studiare l'impatto delle malattie sullo sviluppo e l'organizzazione del sistema nervoso. I ricercatori, compresi questi autori, hanno recentemente introdotto organoidi del cervello umano per modellare i disturbi mitocondriali. Questo documento riporta un protocollo dettagliato per la robusta generazione di organoidi cerebrali umani derivati da iPSC e il loro uso nella profilazione bioenergetica mitocondriale e nelle analisi di imaging. Questi esperimenti consentiranno l'uso di organoidi cerebrali per studiare le disfunzioni metaboliche e dello sviluppo e possono fornire informazioni cruciali per sezionare la patologia neuronale delle malattie mitocondriali.
Introduzione
Le malattie mitocondriali rappresentano la più grande classe di errori congeniti del metabolismo1. Sono causati da mutazioni genetiche che interrompono diversi processi mitocondriali, tra cui la fosforilazione ossidativa (OXPHOS)2, l'assemblaggio della catena respiratoria, la dinamica mitocondriale e la trascrizione o la replicazione del DNA mitocondriale3. I tessuti con fabbisogno energetico sono particolarmente colpiti dalla disfunzione mitocondriale4. Di conseguenza, i pazienti con malattie mitocondriali sviluppano tipicamente manifestazioni neurologiche ad esordio precoce.
Attualmente non sono disponibili trattamenti per i bambini affetti da malattie mitocondriali5. Uno dei principali ostacoli allo sviluppo di farmaci per le malattie mitocondriali è la mancanza di modelli efficaci che ricapitolino il decorso della malattia umana6. Molti dei modelli animali attualmente studiati non presentano i difetti neurologici presenti nei pazienti7. Quindi, i meccanismi alla base della patologia neuronale delle malattie mitocondriali non sono ancora completamente compresi.
Studi recenti hanno generato iPSC da pazienti affetti da malattie mitocondriali e hanno utilizzato queste cellule per ottenere cellule neuronali specifiche del paziente. Ad esempio, è stato scoperto che i difetti genetici associati alla malattia mitocondriale, la sindrome di Leigh, causano aberrazioni nella bioenergetica cellulare8,9, nella sintesi proteica10 e nell'omeostasi del calcio9,11. Questi rapporti hanno fornito importanti indizi meccanicistici sulla compromissione neuronale che si verifica nelle malattie mitocondriali, aprendo la strada alla scoperta di farmaci per queste malattie incurabili12.
Le culture bidimensionali (2D), tuttavia, non consentono lo studio della complessità architettonica e dell'organizzazione regionale degli organi 3D13. A tal fine, l'uso di organoidi cerebrali 3D derivati da iPSC specifiche del paziente14 può consentire ai ricercatori di ottenere ulteriori informazioni importanti e quindi aiutare a sezionare come le malattie mitocondriali influenzano lo sviluppo e la funzione del sistema nervoso15. Gli studi che impiegano organoidi cerebrali derivati da iPSC per studiare le malattie mitocondriali stanno iniziando a scoprire i componenti dello sviluppo neurologico delle malattie mitocondriali.
Gli organoidi del midollo spinale portatori di mutazioni associate alla malattia mitocondriale, all'encefalopatia mitocondriale, all'acidosi lattica e alla sindrome da episodi simili a ictus (MELAS), hanno mostrato neurogenesi difettosa e differenziazione ritardata dei motoneuroni16. Gli organoidi corticali derivati da pazienti con malattia mitocondriale, sindrome di Leigh, hanno mostrato dimensioni ridotte, difetti nella generazione di gemme epiteliali neurali e perdita dell'architettura corticale17. Gli organoidi cerebrali dei pazienti con sindrome di Leigh hanno dimostrato che i difetti della malattia iniziano a livello delle cellule progenitrici neurali, che non possono impegnarsi nel metabolismo mitocondriale, causando aberranti ramificazioni neuronali e morfogenesi18. Pertanto, i progenitori neurali possono rappresentare un bersaglio terapeutico cellulare per le malattie mitocondriali e le strategie che promuovono la loro funzione mitocondriale possono supportare lo sviluppo funzionale del sistema nervoso.
L'uso di organoidi cerebrali potrebbe aiutare a scoprire i componenti dello sviluppo neurologico delle malattie mitocondriali. Le malattie mitocondriali sono considerate principalmente come neurodegenerazione ad esordio precoce5. Tuttavia, i difetti dello sviluppo neurologico sono presenti anche nei pazienti affetti da malattie mitocondriali, tra cui ritardo dello sviluppo e deterioramento cognitivo19. Gli organoidi cerebrali specifici del paziente possono aiutare ad affrontare questi aspetti e chiarire come le malattie mitocondriali possono avere un impatto sullo sviluppo del cervello umano. La disfunzione mitocondriale potrebbe anche svolgere un ruolo patogenetico in altre malattie neurologiche più comuni, come il morbo di Alzheimer, il morbo di Parkinson e la malattia di Huntington4. Quindi, chiarire l'impatto dei difetti mitocondriali nel neurosviluppo usando organoidi cerebrali potrebbe anche essere strumentale per lo studio di tali malattie. Questo documento descrive un protocollo dettagliato per la generazione di organoidi cerebrali riproducibili che possono essere utilizzati per condurre la modellazione delle malattie mitocondriali.
Protocollo
NOTA: L'uso di iPSC umani può richiedere un'approvazione etica. Le iPSC utilizzate in questo studio sono state derivate da individui sani di controllo a seguito dell'approvazione etica locale (# 2019-681). Tutte le procedure di coltura cellulare devono essere eseguite sotto un cappuccio sterile di coltura cellulare, disinfettando accuratamente tutti i reagenti e i materiali di consumo prima di trasferirli sotto il cofano. Le iPSC umane utilizzate per la differenziazione dovrebbero avere un numero di passaggio inferiore a 50 per evitare potenziali aberrazioni genomiche che possono verificarsi su colture estensive. Lo stato pluripotente delle cellule dovrebbe essere convalidato prima della generazione di organoidi, ad esempio monitorando l'espressione di marcatori associati alla pluripotenza come NANOG o OCT4. I test del micoplasma dovrebbero essere condotti settimanalmente per garantire colture prive di micoplasma.
1. Generazione di organoidi cerebrali
- Cultura delle iPSC umane
- Coltivare iPSC umani in condizioni prive di alimentatore in mezzo iPSC (vedere la Tabella dei materiali) su piastre rivestite a 6 pozzetti e tenerle in un incubatore di colture tissutali umidificate a 37 °C e 5% di CO2.
NOTA: Il riporto delle cellule di alimentazione può ostacolare la differenziazione degli organoidi. Passare le cellule almeno una volta in condizioni prive di alimentatore. - Passare le iPSC all'80% di confluenza utilizzando un mezzo di distacco privo di enzimi in rapporti che vanno da 1:4 a 1:12. Per aumentare la sopravvivenza cellulare, aggiungere 10 μM di inibitore della proteina chinasi associata a Rho (ROCK) (Y27632) dopo ogni scissione.
- Coltivare iPSC umani in condizioni prive di alimentatore in mezzo iPSC (vedere la Tabella dei materiali) su piastre rivestite a 6 pozzetti e tenerle in un incubatore di colture tissutali umidificate a 37 °C e 5% di CO2.
- Dissociare le iPSC (confluenza dell'80%)-Giorno 0.
- Preparare il mezzo di differenziazione corticale I (CDMI) (Tabella 1). Media CDMI preriscaldata a temperatura ambiente (22-25 °C) prima di aggiungerlo alle celle.
- Lavare i pozzetti contenenti le iPSC con soluzione salina tamponata con fosfato (PBS) per rimuovere cellule morte e detriti.
- Aggiungere 500 μL di reagente A (Tabella dei materiali) preriscaldato a ciascun pozzetto e incubare per 5 minuti a 37 °C. Controllare al microscopio per garantire il distacco cellulare.
- Aggiungere 1 mL di mezzo iPSC per diluire il reagente A per neutralizzarne l'attività.
- Utilizzare una pipetta da 1000 μL per dissociare le cellule tubando su e giù e trasferire la sospensione cellulare in un tubo centrifugo da 15 mL.
- Centrifugare delicatamente le iPSC a 125 x g per 5 minuti a temperatura ambiente (22-25 °C).
- Aspirare accuratamente il surnatante per evitare di disturbare il pellet cellulare.
- Risospesciare il pellet con 1 mL di CDMI per ottenere una sospensione a cella singola e contare il numero di cella.
- Preparare il terreno di semina con 9.000 iPSC per 100 μL in CDMI integrato con 20 μM di inibitore ROCK, 3 μM di inibitore WNT-catenina (IWR1) e 5 μM SB431542.
- Aggiungere 100 μL di terreno di semina per pozzetto a una piastra con fondo a V da 96 pozzetti.
- Conservare la piastra in un incubatore di colture tissutali umidificate a 37 °C e al 5% di CO2.
- Generazione di neurosfere
- Il giorno 1, osserva che si stanno formando aggregati di cellule rotonde (neurosfere) con bordi lisci definiti. Si notino le cellule morte intorno agli aggregati. Continuare a coltivare nell'incubatore a 37 °C e 5% di CO2.
- Il giorno 3, agitare la piastra toccando i lati tre volte per staccare le cellule morte.
- Aggiungere 100 μL di CDMI integrati con 20 μM di inibitore ROCK, 3 μM IWR1 e 5 μM SB431542 a ciascun pozzetto.
- Riportare la piastra all'incubatore a 37 °C e al 5% di CO2.
- Il giorno 6, rimuovere con attenzione 80 μL del mezzo surnatante da ciascun pozzetto. Evitare di toccare il fondo del pozzo.
- Aggiungere 100 μL di CDMI integrati con 3 μM IWR1 e 5 μM SB431542 a ciascun pozzetto. Riportare la piastra all'incubatore a 37 °C e al 5% di CO2.
- Ripetere i passaggi 5 e 6 ogni 3 giorni fino al giorno 18.
- Trasferimento di neurosfere
- Il giorno 18, preparare Cortical Differentiation Medium II (CDMII) (Tabella 1) e aggiungere 10 ml a una piastra di coltura cellulare ad attacco ultra-basso da 100 mm.
- Utilizzare una pipetta da 200 μL con la punta tagliata per trasferire le neurosfere rotonde dalla piastra a 96 pozzetti alla piastra di coltura cellulare ad attacco ultra-basso da 100 mm.
NOTA: Sii delicato per evitare di danneggiare le neurosfere assicurandoti che l'apertura della punta sia abbastanza ampia e che gli aggregati non vengano aspirati troppo rapidamente. - Rimuovere 5 mL di mezzo dalla piastra contenente le neurosfere e aggiungere 5 mL di CDMII fresco.
NOTA: Questa procedura aiuta a ridurre la quantità di supporto CDMI che potrebbe essere stata trasferita dal trasferimento delle neurosfere. - Posizionare la piastra su uno shaker orbitale a 70 giri/min all'interno di un incubatore di colture tissutali umidificate a 37 °C e 5% di CO2.
NOTA: ispezionare visivamente le neurosfere il giorno successivo. Aumentare la velocità dello shaker orbitale se le neurosfere sono raggruppate insieme o attaccate al fondo della piastra. - Ogni 3 giorni, aspirare accuratamente il mezzo surnatante e sostituirlo con CDMII fresco. Lasciare una piccola quantità del mezzo per evitare che le neurosfere si secchino.
- Il giorno 35, preparare Cortical Differentiation Medium III (CDMIII) (Tabella 1).
NOTA: il componente della matrice deve essere sciolto in CDMIII freddo. - Aspirare il mezzo dalla piastra e aggiungere 10 ml di CDMIII freddo.
NOTA: È più efficace utilizzare il mezzo freddo in modo che il componente della matrice possa rivestire gli organoidi senza formare grumi. - Dopo aver cambiato il mezzo, riposizionare la piastra su uno shaker orbitale a 70 giri / min all'interno di un incubatore di colture tissutali umidificate a 37 ° C e 5% di CO2.
- Cambia il mezzo ogni 3-5 giorni a seconda del tasso di crescita, come indicato dal colore del mezzo.
- Il giorno 70, preparare Cortical Differentiation Medium IV (CDMIV) (Tabella 1). Utilizzare il supporto CDMIV fino al raggiungimento dell'età desiderata degli organoidi. Durante questo periodo, tenere la piastra su uno shaker orbitale impostato a 70 giri / min all'interno di un incubatore di colture tissutali umidificate (37 ° C e 5% di CO2).
- Cambia il mezzo ogni 3-5 giorni, a seconda del tasso di crescita.
2. Immunocolorazione degli organoidi cerebrali
- Preparazione dei tessuti
- Preparare la soluzione di paraformaldeide al 4% (PFA) e metterla sotto un cappuccio di sicurezza.
NOTA: Indossare dispositivi di sicurezza personali quando si maneggia PFA. - Raccogliere gli organoidi cerebrali e trasferirli delicatamente con una pipetta Pasteur di plastica da 3 mL con punta smussata in una piastra a 6 pozzetti piena di PFA.
NOTA: Utilizzare organoidi più vecchi di 40 giorni per consentire la visualizzazione di strutture con maggiore complessità cellulare. - Conservare gli organoidi nella soluzione di PFA per 1 ora a temperatura ambiente.
- Rimuovere con cura il PFA con una pipetta Pasteur in plastica da 3 ml e lavare gli organoidi fissi tre volte utilizzando PBS.
- Conservare gli organoidi fissi a 4 °C in PBS fino a nuovo utilizzo.
- Preparare la soluzione di paraformaldeide al 4% (PFA) e metterla sotto un cappuccio di sicurezza.
- Preparazione di fette di organoidi cerebrali
- Preparare una soluzione di agar al 3% e riscaldare lentamente fino a quando non si è liquefatto.
- Posizionare lo stampo (l'estremità tagliata di una siringa da 10 ml) su un pezzo di carta da filtro assorbente (lato liscio verso l'alto). Metti una goccia di agar su di esso.
- Estrarre rapidamente un singolo organoide dalla piastra a 6 pozzetti con una spatola e rimuovere l'eccessivo PBS con carta da filtro.
NOTA: Fare attenzione a non toccare l'organoide direttamente con carta da filtro. - Posizionare l'organoide sulla goccia di agar.
- Ripetere questa procedura con un massimo di tre organoidi.
NOTA: lavorare velocemente per evitare la solidificazione dell'agar durante questo passaggio. - Riempire lo stampo con l'agar fino a quando tutti gli organoidi sono completamente coperti.
- Attendere che l'agar inizi a solidificarsi, quindi trasferire delicatamente l'intero stampo contenente gli organoidi, compresa la carta da filtro assorbente, su un elemento di raffreddamento.
NOTA: Se un elemento di raffreddamento non è disponibile, conservare gli organoidi per alcuni minuti in frigorifero a 4 °C. - Nel frattempo, prepararsi per la procedura di affettamento: posizionare una lama di rasoio (pulita con acetone e lavata con acqua a doppio distillato) nel supporto del vibratoma, montare sul bagno e riempirla con PBS.
- Rimuovere lo stampo dall'agar (solidificato) e utilizzare un bisturi per tagliarlo per formare un cubo.
- Attaccare il cubo di agar contenente gli organoidi sulla piastra portante del vibratoma con gel adesivo (vedi la Tabella dei materiali) e posizionarlo nella vasca da bagno contenente PBS.
- Regolare il vibratoma (vedere la Tabella dei materiali) per tagliare le fette ad uno spessore di 150 μm.
NOTA: le impostazioni del vibratoma (angolo, ampiezza, frequenza e velocità corretti della lama) possono essere simili a quelle utilizzate per affettare il tessuto cerebrale fisso derivato dai primi animali postnatali. Tuttavia, le impostazioni ideali dipendono fortemente dal tipo di vibratoma e devono essere determinate in una prima fase per evitare distorsioni o addirittura strappi del tessuto durante il taglio. - Avviare la procedura di taglio. Utilizzare una pipetta di vetro o una spatola per trasferire delicatamente ogni fetta appena tagliata in una piastra a 24 pozzetti riempita con PBS.
- Conservare la piastra contenente le fette a 4 °C (fino a un certo giorno) fino a un'ulteriore lavorazione.
- Trasferire le fette fuori dalla piastra con una pipetta di vetro o una spatola su vetrini per microscopio. Utilizzare un minimo di 2 fette per diapositiva.
- Rimuovere con cautela l'agar e il PBS in eccesso con una siringa.
- Lasciare asciugare le fette fino a quando non aderiscono alle diapositive.
NOTA: Mentre i vetrini per microscopio possono essere conservati in camere di plastica riempite con PBS a 4 °C, devono essere colorati il prima possibile dopo la procedura di affettamento.
- Colorazione immunoistochimica
- Preparare la soluzione di blocco (Tabella 1).
- Utilizzare una penna PAP per disegnare un bordo idrofobo attorno alle fette sulla diapositiva per mantenere tutte le soluzioni sulle diapositive.
- Aggiungere con attenzione la soluzione bloccante sul vetrino e incubare per 1 ora a temperatura ambiente (22-25 °C). Per evitare di distruggere il tessuto, non aggiungere la soluzione direttamente sopra le fette.
- Aspirare la soluzione bloccante e applicare l'anticorpo primario desiderato diluito nella soluzione bloccante.
- Incubare il vetrino durante la notte in una camera umidificata a 4 °C.
- Risciacquare la diapositiva tre volte con 1x PBS per 10 minuti ciascuno.
- Incubare le fette con l'anticorpo secondario specifico diluito nella soluzione bloccante ed eseguire la colorazione Hoechst (1:2.500) per 1 ora a temperatura ambiente al buio.
NOTA: Ricordarsi di eseguire controlli negativi per confermare che non ci siano legami non specifici o autofluorescenza. - Risciacquare tre volte con 1x PBS per 10 minuti ciascuno al buio.
- Aggiungere una goccia di mezzo di montaggio alla fetta, posizionare una copertura sul bordo della goccia e posare lentamente la slitta verso la fetta per evitare bolle d'aria.
- Lasciare riposare lo scivolo durante la notte a temperatura ambiente. Applicare lo smalto sul bordo della copertina per sigillare ulteriormente la diapositiva. Per la conservazione a lungo termine, conservare a 4 °C.
- Documentazione della colorazione
- Per scansionare immagini di grandi dimensioni per cucire, utilizzare un microscopio a campo largo verticale motorizzato dotato di (vedi la Tabella dei materiali per i dettagli) un obiettivo di alta qualità; Set di filtri DAPI (ad esempio, eccitazione (EX): 340-380 nm, specchio dicroico (DM): 400 nm, filtro barriera (BA): 435-485 nm); set di filtri isotiocianati di fluoresceina (ad esempio, EX: 465-495 nm, DM: 505 nm, BA: 515-555 nm); Set di filtri Alexa594 (ad esempio, ET 575/40; T 600 LPXR; HC 623/24); fotocamera digitale; software di acquisizione ad alte prestazioni che consente operazioni automatizzate di stitches e stack.
- Per la gestione delle immagini, utilizzare un programma di elaborazione delle immagini in grado di generare file tif a 8 bit, ritagliare punti, regolare il contrasto e la luminosità, unire i canali (ad esempio, blu, verde e rosso) e aggiungere barre di scala.
- Per scansionare i dettagli, utilizzare un microscopio a scansione laser confocale motorizzato dotato di un obiettivo di alta qualità, un laser UV (EX: 408 nm), un laser Argon (EX: 488 nm), un laser elio-neon (EX: 543), software di imaging per un microscopio confocale.
- Per la gestione delle immagini dei dettagli, utilizzare un programma di elaborazione delle immagini in grado di generare proiezioni di massima intensità di z-stack confocali (ad esempio, sezioni ottiche di 0,6 μm ciascuna), generare file tif a 8 bit, regolare contrasto e luminosità, unire i canali (ad esempio, blu, verde e rosso), aggiungere barre di scala.
- Usa un editor grafico per disporre le figure.
3. Profilazione bioenergetica degli organoidi cerebrali
- Preparazione di organoidi per la profilazione bioenergetica
- Preparare la soluzione di papaina e DNasi seguendo il protocollo del produttore.
- Trasferire 3-5 organoidi in una piastra a 6 pozzetti. Lavali due volte con PBS prebellico.
- Aggiungere 2 mL di soluzione di papaina attivata preriscaldata contenente DNasi. Usando una lama, tagliare gli organoidi in piccoli pezzi.
- Posizionare la piastra su uno shaker orbitale impostato a 27 rpm all'interno di un incubatore di colture cellulari (a 37 °C, 5% DI CO2) e incubare per 15-20 min.
NOTA: Il tempo di incubazione dipende dallo stadio organoide. Gli organoidi in fase iniziale possono essere usati così come sono. Per gli organoidi di età superiore a 3 mesi, si consiglia di tagliare gli organoidi in 2-3 pezzi prima della dissociazione e incubare i pezzi a una velocità di oscillazione impostata a 27 giri / min per 15-20 minuti a 37 ° C. Questa procedura può aiutare a rimuovere il tessuto necrotico che può essere presente negli organoidi in fase successiva. - Raccogliere i tessuti digeriti in un tubo da 15 ml e aggiungere 5 mL di terreno di coltura organoide CDMIV (Tabella 1).
- Triturare il tessuto con una pipetta di plastica da 10 ml tubando su e giù 10-15 volte. Lasciare che il tessuto non dissociato si depositi sul fondo del tubo.
- Trasferire con cura la sospensione cellulare in un tubo da 15 ml, evitando qualsiasi pezzo di tessuto non dissociato. Filtrare la soluzione attraverso un filtro cellulare da 40 μm (ad esempio, tubi a fondo tondo in polistirene con tappi del filtro cellulare).
- Pellet le celle mediante centrifugazione a 300 x g per 5 minuti a temperatura ambiente.
- Valuta il numero e la qualità delle celle usando il tripano blu.
- Placcare il numero desiderato (~ 20.000 / pozzetto) di cellule su micropiastre rivestite a 96 pozzetti. Cambiare il mezzo 6-8 ore dopo la placcatura in mezzo neuronale (Tabella 1).
- Incubare la micropiastra rivestita a 96 pozzetti in un incubatore a CO2 (37 °C, 5% CO2) per 4 giorni.
- Profilazione bioenergetica
- Il giorno 3 dopo aver ripavimentato le cellule dissociate, aggiungere 200 μL di soluzione di calibrazione in ciascun pozzetto della parte inferiore della micropiastra a 96 pozzetti e posizionare la cartuccia del sensore verde superiore sulla micropiastra idratata.
NOTA: posizionare la cartuccia del sensore sulla parte superiore della micropiastra nell'orientamento corretto e assicurarsi che la soluzione calibrante copra tutti i sensori. - Incubare la micropiastra idrata a 96 pozzetti in un incubatore non a CO2 a 37 °C durante la notte.
- Accendere l'analizzatore per consentire allo strumento di stabilizzarsi a 37 °C durante la notte.
- Il giorno 4 dopo la ripaccatura, ispezionare la coltura organoide dissociata sulla micropiastra a 96 pozzetti al microscopio per assicurarsi che le cellule appaiano come un monostrato confluente.
- Preparare il mezzo di analisi (Tabella 1).
- Rimuovere Neuronal Medium da tutti i pozzetti con una pipetta senza toccare il fondo del pozzo per prevenire danni cellulari. In alternativa, capovolgere con cura l'intero piatto e quindi asciugarlo su carta pulita. Lavora rapidamente per evitare la morte cellulare.
- Lavare le cellule due volte con 200 μL preriscaldati di Assay Medium. Aggiungere Assay Medium a un volume finale di 180 μL per pozzetto. Incubare la micropiastra a 96 pozzetti in un incubatore non a CO2 a 37 °C per 1 ora.
- Preparare soluzioni da 10 μM di inibitori mitocondriali in mezzo di analisi. Si noti che la concentrazione finale dopo l'iniezione è di 1 μM.
- Caricare la cartuccia del sensore posta nella micropiastra idrata con 10x soluzioni degli inibitori mitocondriali.
- Aggiungere 18 μL di inibitore mitocondriale 1 nella porta A.
- Aggiungere 19,8 μL di inibitore mitocondriale 2 nella porta B.
- Aggiungere 21,6 μL di inibitore mitocondriale 2 nella porta C.
- Aggiungere 23,4 μL di inibitore mitocondriale 3 nella porta D.
- Posizionare la cartuccia caricata nella micropiastra idrata in un incubatore non a CO2 a 37 °C fino all'inizio del test.
- Impostare un protocollo in esecuzione nel software dello strumento (Tabella 2).
- Premere START. Prelevare la cartuccia caricata dall'incubatore non a CO2 e inserirla nell'analizzatore per la calibrazione.
NOTA: Assicurarsi che la piastra sia inserita nell'orientamento corretto e senza coperchio. - Una volta terminata la fase di calibrazione, rimuovere la piastra di calibrazione. Prendere la micropiastra a 96 pozzetti dall'incubatore non a CO2 e inserirla nell'analizzatore. Fare clic su CONTINUA per avviare le misurazioni.
- Al termine della corsa, rimuovere la micropiastra di coltura cellulare a 96 pozzetti dall'analizzatore e raccogliere il mezzo da tutti i pozzetti senza disturbare le cellule.
NOTA: Il mezzo può essere conservato a -20 °C e utilizzato successivamente per misurare la quantità di lattato rilasciata dalle cellule nel mezzo utilizzando un kit di analisi del lattato appropriato. - Lavare le cellule con 200 μL di 1x PBS in ogni pozzetto.
- Dopo aver rimosso il PBS, congelare la piastra a -20 °C.
NOTA: La piastra congelata può essere utilizzata per quantificare cellule, proteine o DNA in ciascun pozzetto della micropiastra. Questa quantificazione sarà necessaria per normalizzare i tassi bioenergetici ottenuti. Seguire le istruzioni del produttore per i test di quantificazione di cellule, proteine o DNA.
- Il giorno 3 dopo aver ripavimentato le cellule dissociate, aggiungere 200 μL di soluzione di calibrazione in ciascun pozzetto della parte inferiore della micropiastra a 96 pozzetti e posizionare la cartuccia del sensore verde superiore sulla micropiastra idratata.
Risultati
Il protocollo qui descritto facilita la robusta generazione di organoidi rotondi (Figura 1A). Gli organoidi generati contengono neuroni maturi che possono essere visualizzati utilizzando marcatori proteici specifici per assoni (SMI312) e dendriti (proteina 2 associata a microtubuli (MAP2)) (Figura 1B). Gli organoidi maturi contengono non solo cellule neuronali (MAP2-positive) ma anche cellule gliali (ad esempio, positive per il marcatore astrocitario S100 proteina B legante il calcio (S100ß)) (Figura 1B).
Analizzando gli organoidi cerebrali affettati utilizzando la microscopia confocale, è possibile identificare e monitorare la distribuzione e l'organizzazione dettagliata di diversi tipi di cellule e strutture cellulari. Ciò potrebbe fornire nuove informazioni su come le malattie mitocondriali potrebbero influenzare lo sviluppo del sistema nervoso. Ad esempio, è possibile monitorare gli assoni neuronali (SMI312-positivo) e i dendriti (MAP2-positivo) (Figura 2A) o la reciproca presenza di cellule neuronali (MAP2-positive) e cellule gliali (S100ß-positivo) (Figura 2A). Le immagini confocali possono anche aiutare a indagare in modo più dettagliato la distribuzione e l'organizzazione dei progenitori neurali ((regione di determinazione del sesso Y) box-2 (SOX2)-positivi) rispetto ai neuroni (beta-III tubulina (TUJ1)-positivi) (Figura 2B). Infine, gli organoidi cerebrali possono essere colorati per marcatori specifici dei mitocondri (come la proteina della membrana mitocondriale esterna, translocase della subunità 20 kDa della membrana esterna (TOM20)) (Figura 2C).
Il protocollo descritto consente ai ricercatori di eseguire la profilazione bioenergetica degli organoidi cerebrali. Utilizzando questa procedura, è possibile misurare sia il metabolismo mitocondriale utilizzando il tasso di consumo di ossigeno (OCR) (Figura 2D) che il metabolismo glicolitico utilizzando il tasso di acidificazione extracellulare (ECAR) (Figura 2E) . La profilazione bioenergetica consente di monitorare come le cellule possono modificare i loro profili OCR ed ECAR in risposta a una somministrazione sequenziale di inibitori mitocondriali.
In primo luogo, l'inibitore dell'ATP sintasi, oligomicina, può essere applicato. L'oligomicina provoca un calo del profilo OCR (Figura 2D) e, pertanto, identifica l'OCR necessario per la produzione di ATP. Dopo il trattamento con oligomicina, potrebbe esserci anche un aumento compensatorio dell'ECAR (Figura 2E), suggerendo che le cellule possono sovraregolare la glicolisi per prevenire lo stress metabolico causato dalla riduzione del metabolismo mitocondriale. La successiva doppia applicazione del protone ionoforo, carbonil cianuro-p-trifluorometiossifenilidrazon (FCCP), provoca la perdita del potenziale di membrana mitocondriale. Poiché le molecole di ossigeno sono ora libere di muoversi, ciò provoca un rapido aumento dell'OCR (Figura 2D).
Questi cambiamenti nel profilo OCR identificano la massima capacità respiratoria delle cellule. La somministrazione finale di rotenone più antimicina A provoca un blocco del trasporto di elettroni e, quindi, una forte diminuzione dell'OCR (Figura 2D). ECAR può mostrare fluttuazioni dopo il trattamento con FCCP e rotenone più antimicina A (Figura 2E), a seconda della capacità glicolitica residua delle cellule. I profili OCR ed ECAR possono essere drammaticamente alterati negli organoidi cerebrali derivati da pazienti mitocondriali.
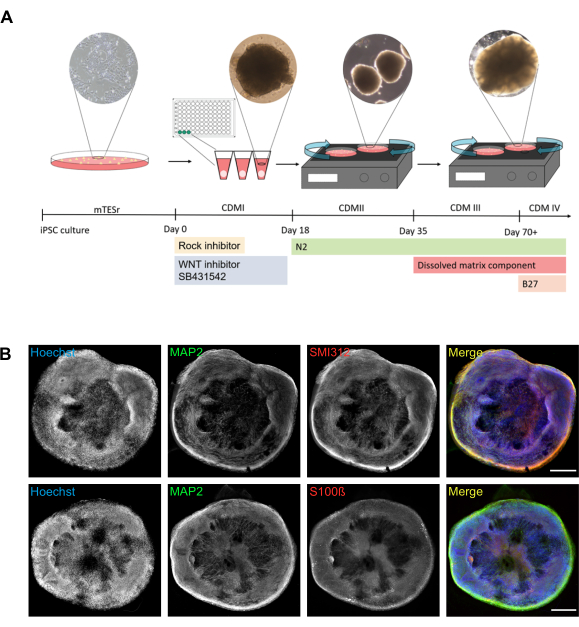
Figura 1: Generazione di organoidi cerebrali da iPSC umane. (A) Rappresentazione schematica del protocollo utilizzato per produrre organoidi cerebrali con corrispondenti immagini di trasmissione. Il giorno 0 corrisponde alla dissociazione delle iPSC e alla semina in una piastra a 96 pozzetti con fondo a V utilizzando CDMI integrato con un inibitore ROCK, un inibitore WNT e SB431542. Al giorno 18, le neurosfere vengono trasferite dalle piastre a 96 pozzetti a piastre di coltura cellulare da 100 mm con CDMII integrato con N2. Da questo punto in poi, le colture sono posizionate su uno shaker orbitale. Al giorno 35, il mezzo viene commutato da CDMII a CDMIII, che contiene anche una componente a matrice disciolta (Tabella 1). Dal giorno 70 in poi, CDMIII è passato a CDMIV integrato con B27. Un'immagine rappresentativa della neurosfera è stata scattata al giorno 12 utilizzando una fotocamera al microscopio con ingrandimento 10x. Una prima immagine organoide è stata scattata al giorno 22 utilizzando una fotocamera al microscopio con ingrandimento 4x. Un'immagine organoide matura è stata scattata al giorno 40 utilizzando una fotocamera al microscopio con ingrandimento 4x. (B) La struttura generale e l'organizzazione cellulare degli organoidi cerebrali possono essere visualizzate utilizzando la microscopia ad ampio campo. Vengono mostrate immagini rappresentative cucite ad ampio campo per visualizzare le relazioni tra dendriti (MAP2-positivo) e assoni (SMI312-positivo) e tra cellule neuronali (MAP2-positivo) e presunti astrociti (S100ß-positivo). Le cellule sono state controcolorate con Hoechst per rivelare i nuclei. Tutte le immagini sono state scattate utilizzando organoidi cerebrali di 78 giorni. La colonna di destra mostra la sovrapposizione di tre canali (unione). Barre di scala = 500 μm. Abbreviazioni: iPSC = cellula staminale pluripotente indotta; CDM = Mezzo di differenziazione corticale; ROCCIA = Rho chinasi; MAP2 = proteina 2 associata ai microtubuli; S100ß= S100 calcium-binding protein B. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Visualizzazione e profilazione bioenergetica di organoidi cerebrali per la modellazione della malattia mitocondriale. L'organizzazione dettagliata e l'architettura degli organoidi possono essere analizzate utilizzando la microscopia confocale. Tutte le immagini sono state scattate utilizzando organoidi cerebrali di 78 giorni e controinfangate con Hoechst per rivelare i nuclei. La colonna di destra mostra la sovrapposizione di tre canali (unione). Barre di scala = 50 μm. (A) Proiezioni rappresentative a fuoco esteso (44-48 piani ottici, 0,6 μm ciascuno) che affrontano l'interazione tra dendriti (MAP2-positivi, punte di freccia) e assoni (SMI312-positivo, frecce), e tra cellule neuronali (MAP2-positivo, punte di freccia) e presunti astrociti (S100ß-positivo, frecce). (B) Proiezioni rappresentative a fuoco esteso (14-31 piani ottici, 0,6 μm ciascuno) che mostrano la distribuzione dei neuroni (TUJ1-positivo, punte di freccia) rispetto ai progenitori neurali (SOX2-positivi, frecce). (C) Proiezioni rappresentative a fuoco esteso (20 piani ottici, 0,6 μm ciascuno) che mostrano la distribuzione all'interno dei neuroni (TUJ1-positivo, punte di freccia) dei mitocondri (visualizzati usando anticorpi contro la proteina di membrana mitocondriale esterna TOM20, frecce). (D) La respirazione mitocondriale degli organoidi cerebrali può essere monitorata in base al profilo dell'OCR dopo somministrazione sequenziale di diversi inibitori mitocondriali (vedere il testo per i dettagli). (E) L'attività glicolitica degli organoidi cerebrali può essere monitorata sulla base dell'ECAR dopo somministrazione sequenziale di inibitori mitocondriali (vedere il testo per i dettagli). Per la profilazione bioenergetica, circa 10-15 organoidi cerebrali sono stati dissociati per ottenere abbastanza cellule per la ripapiattaforma sulla micropiastra a 96 pozzetti. Le barre indicano i SEM in base ai risultati ottenuti in due esperimenti indipendenti. Abbreviazioni: MAP2 = proteina 2 associata ai microtubuli; S100ß = S100 proteina B legante il calcio; TUJ1 = beta-III tubulina; SOX2 = (regione di determinazione del sesso Y) box-2; TOM20 = translocase della subunità 20 kDa della membrana esterna; OCR = tasso di consumo di ossigeno; ECAR = tasso di acidificazione extracellulare; Oligom. = oligomicina; FCCP = cianuro di carbonile-p-trifluorometiossifenilidrazon; R = rotenone; Anta = antimicina A; SEM = errore standard dei mezzi. Fare clic qui per visualizzare una versione più grande di questa figura.
| Composizione multimediale | |||
| CDMI (Giorno 0-18) | Finale conc. | ||
| Glasgow-MEM | Gibco · | 11710-035 | [1:1] |
| Sostituzione del siero knockout (KSR) | Gibco · | 10828010 | 20% |
| MEM-NEAA (MEM soluzione di aminoacidi non essenziali) | Gibco · | 11140-050 | 0,1 mM |
| Piruvato di sodio | Gibco · | 11360070 | 1 mM |
| 2-mercaptonetanolo | Gibco · | 31350-010 | 0,1 mM |
| Penicillina e streptomicina | Gibco · | 15140-122 | 100 U/mL e 100 μg/mL |
| CDMII (Giorno 18-35) | Finale conc. | ||
| DMEM/F12 | Gibco · | 31330038 | [1:1] |
| Glutamax | Gibco · | 35050-061 | 2 mM |
| Supplemento N-2 (100x) | Gibco · | 17502-048 | 1% |
| Concentrato lipidico chimicamente definito | Gibco · | 11905031 | 1% |
| Penicillina e streptomicina | Gibco · | 15140-122 | 100 U/mL e 100 μg/mL |
| CDMIII (Giorno 35-70) | Finale conc. | ||
| DMEM/F12 | Gibco · | 31330038 | [1:1] |
| Glutamax | Gibco · | 35050-061 | 2 mM |
| Supplemento N-2 (100x) | Gibco · | 17502-048 | 1% |
| Concentrato lipidico chimicamente definito | Gibco · | 11905031 | 1% |
| Penicillina e streptomicina | Gibco · | 15140-122 | 100 U/mL e 100 μg/mL |
| Siero bovino fetale (FBS) | Gibco · | 10270-106 | 10% |
| Eparina | Merck | H3149-25KU | 5 μg/mL |
| Matrigel · | Corning | 356231 | 1% |
| CDMIV(Giorno 70+) | Finale conc. | ||
| DMEM/F12 | Gibco · | 31330038 | [1:1] |
| Glutamax | Gibco · | 35050-061 | 2 mM |
| Supplemento N-2 (100x) | Gibco · | 17502-048 | 1% |
| Concentrato lipidico chimicamente definito | Gibco · | 11905031 | 1% |
| Penicillina e streptomicina | Gibco · | 15140-122 | 100 U/mL e 100 μg/mL |
| Siero bovino fetale (FBS) | Gibco · | 10270-106 | 10% |
| Eparina | Merck | H3149-25KU | 5 μg/mL |
| Matrigel · | Corning | 356231 | 2% |
| Integratore B-27 con Vitamina A 50x | Gibco · | 17504044 | 2% |
| Mezzo neuronale | Finale conc. | ||
| DMEM/F12 | Gibco · | 31330038 | [1:1] |
| Supplemento N-2 | Gibco · | 17502048 | [1x] |
| Integratore B-27 con vitamina A 50x | Gibco · | 17504044 | [1x] |
| Acido L-ascorbico | Sigma Aldrich | A92902 · | [200 μM] |
| db-cAMP (adenosina monofosfato ciclico dibutirrile) | Sigma Aldrich | D0627 · | 500 μM |
| BDNF (fattore neutrotrofico derivato dal cervello) | MACS Miltenyi | 130-093-811 | [10 ng/mL] |
| GDNF (fattore neurotrofico derivato dalla linea cellulare gliale) | MACS Miltenyi | 130-096-290 | [10 ng/mL] |
| TGF-β3 umano (fattore di crescita trasformante-beta3) | MACS Miltenyi | 130-094-007 | [1 ng/mL] |
| Saggio medio | Finale conc. | ||
| Cavalluccio marino XF DMEM medio | Cavalluccio marino Bioscienza | 103680-100 | 500 ml |
| Piruvato di sodio | Gibco · | 11360070 | 1 mM |
| L-Glutammina | Lonza · | BEBP17-605E | 2 mM |
| Glucosio | Sigma Aldrich | 50-99-7 | 10 mM |
| Soluzione di blocco | Finale conc. | ||
| PBS-Interpolazione | [1:1] 0,1% Interpolazione | ||
| Siero d'asino | Sigma Aldrich | D9663 · | 10% |
| Tritone-X | Merck | X100-5ML | 1% |
Tabella 1: Dettagli dei mezzi e delle soluzioni utilizzate per la generazione di organoidi.
| Inizializzazione | Linea di base (X3) | Iniezione di oligomicina (X3) | Iniezione FCCP (X3) | Iniezione FCCP (X3) | Iniezione Anti A + Rot (X3) |
| Calibrare | Mescolare | Mescolare | Mescolare | Mescolare | Mescolare |
| (04:00) | (04:00) | (04:00) | (04:00) | (04:00) | |
| Equilibrate (12:00) | Aspettare | Aspettare | Aspettare | Aspettare | Aspettare |
| (02:00) | (02:00) | (02:00) | (02:00) | (02:00) | |
| Misura (03:00) | Misura (03:00) | Misura (03:00) | Misura | Misura (03:00) | |
| (03:00) |
Tabella 2: Configurazione del protocollo per la profilazione bioenergetica. Descrizione dei passi e della loro durata in minuti utilizzando il software Seahorse Wave Desktop. Abbreviazioni: FCCP = carbonile cianuro-p-trifluorometiossifenilidrazon; Rot = rotenone; Anti A = Antimicina A.
Discussione
Questo articolo descrive la generazione riproducibile di organoidi cerebrali umani derivati da iPSC e il loro uso per la modellazione della malattia mitocondriale. Il protocollo qui descritto viene modificato sulla base di un lavoro pubblicato in precedenza20. Uno dei principali vantaggi del presente protocollo è che non richiede l'incorporamento manuale di ciascun organoide in una matrice di impalcatura. Infatti, la soluzione della matrice viene semplicemente disciolta nel terreno di coltura cellulare. Inoltre, non è necessario impiegare costosi bioreattori, poiché gli organoidi possono essere coltivati in piastre standard di coltura tissutale a 6 pozzetti posizionate su uno shaker orbitale all'interno dell'incubatore. Questa procedura consente anche la coltivazione parallela di più piastre contenenti diversi organoidi derivati da varie singole linee, aumentando così la produttività degli esperimenti e consentendo il monitoraggio delle potenziali differenze emergenti nei profili di crescita di diversi organoidi. Abbiamo testato questo protocollo utilizzando diverse iPSC derivate da controlli sani e individui affetti da malattie mitocondriali, con risultati coerenti.
Per la modellazione della malattia mitocondriale, è essenziale utilizzare diversi marcatori per visualizzare la morfologia e l'organizzazione della rete mitocondriale. Questa procedura consente di indagare se il numero, la morfologia o la distribuzione mitocondriale possano essere alterati negli organoidi cerebrali derivati da pazienti con malattie mitocondriali. La presenza e l'organizzazione di progenitori neurali all'interno degli organoidi cerebrali potrebbe essere di cruciale importanza per la modellazione dei disturbi mitocondriali. Recentemente abbiamo scoperto che le mutazioni che causano la malattia mitocondriale, la sindrome di Leigh, interrompono l'architettura cellulare e la distribuzione delle cellule progenitrici neurali all'interno di organoidi cerebrali derivati dal paziente18.
Per eseguire la profilazione bioenergetica, abbiamo adattato un metodo precedentemente descritto per valutare la bioenergetica delle cellule staminali pluripotenti21. Un recente protocollo ha descritto come effettuare la profilazione bioenergetica di organoidi derivati da intestino tenue di topo, colon umano e tumori del colon-retto22. Tuttavia, questi organoidi sono piuttosto piccoli rispetto agli organoidi cerebrali e, pertanto, è necessario un protocollo diverso, come quello riportato qui, per gli organoidi cerebrali. Recentemente abbiamo utilizzato questo protocollo per valutare il profilo bioenergetico degli organoidi del cervello umano portatori di mutazioni nel gene surfeit locus protein 1 (SURF1) che causa la grave malattia mitocondriale, la sindrome di Leigh18. Abbiamo scoperto che il profilo OCR è particolarmente influenzato negli organoidi della sindrome di Leigh, come dimostrato da una significativa diminuzione del livello di OCR basale, del tasso di produzione di ATP e del tasso massimo di respirazione18.
In conclusione, presentiamo qui un protocollo dettagliato per la robusta generazione di organoidi del cervello umano e descriviamo come eseguire esperimenti che sarebbero importanti per lo studio dei meccanismi della malattia alla base delle malattie mitocondriali. Gli organoidi del cervello umano possono anche essere di fondamentale importanza per chiarire la diversità mitocondriale nel cervello umano e il suo ruolo nella salute e nelle malattie umane23. È importante chiarire che gli organoidi cerebrali generati con i protocolli attualmente disponibili, incluso quello qui descritto, hanno ancora dei limiti. Questi includono, ad esempio, la mancanza di vascolarizzazione e l'assenza di popolazione di microglia24. Questi aspetti devono essere presi in considerazione per interpretare correttamente i risultati.
Ad esempio, la mancanza di vascolarizzazione e microglia potrebbe limitare i meccanismi di compensazione che possono essere in atto in vivo. Gli organoidi cerebrali derivati dal paziente potrebbero quindi presentare difetti più forti di quelli osservati nei pazienti17,18. Inoltre, nonostante una riproducibilità generale di questo protocollo20, si può osservare eterogeneità da linea a linea. A tal fine, quando si eseguono studi di modellazione della malattia, è sempre importante quantificare sistematicamente l'uniformità degli organoidi di controllo e del paziente valutando i modelli di morfologia (dimensioni, strato) e la distribuzione dei marcatori molecolari tra diversi organoidi.
Infine, non è possibile generare organoidi cerebrali da una singola iPSC, limitando la fattibilità di uno screening genetico su larga scala con CRISPR/Cas9. Dato il ritmo della ricerca, è probabile che alcune delle attuali limitazioni del protocollo qui descritto saranno presto superate. Saranno disponibili protocolli ottimizzati. Si spera che questi modelli 3D delle malattie mitocondriali consentano l'eventuale scoperta di terapie implementabili per le malattie mitocondriali, che sono dannose, e per le malattie incurabili con esigenze mediche altamente insoddisfatte.
Divulgazioni
Gli autori non dichiarano interessi finanziari o non finanziari concorrenti.
Riconoscimenti
Ringraziamo Miriam Bünning per il supporto tecnico. Riconosciamo il sostegno della Deutsche Forschungsgemeinschaft (DFG) (PR1527/5-1 ad A.P.), Spark and Berlin Institute of Health (BIH) (BIH Validation Funds to A.P.), della United Mitochondrial Disease Foundation (UMDF) (Leigh Syndrome International Consortium Grant to A.P.), dell'University Hospital Duesseldorf (Forschungskommission UKD to A.P.) e del Ministero Federale Tedesco dell'Istruzione e della Ricerca (BMBF) (e: Bio giovane investigatore concede AZ 031L0211 ad A.P.). Il lavoro nel laboratorio del C.R.R. è stato sostenuto dal DFG (FOR 2795 "Synapses under stress", Ro 2327/13-1).
Materiali
| Name | Company | Catalog Number | Comments |
| 2-mercaptoethanol | Gibco | 31350-010 | |
| Affinity Designer | Serif (Europe) Ltd | Layout software; Vector graphics editor | |
| Alexa Fluor 488 donkey anti-guinea pig | Sigma Aldrich | SAB4600033-250UL | 1:300 |
| Alexa Fluor 488 donkey anti-mouse | Thermo Fisher Scientific | A-31571 | 1:300 |
| Antimycin A | Sigma Aldrich | 1397-94-0 | |
| Anti-β-Tubulin III (TUJ-1) | Sigma Aldrich | T8578 | 1:2000 |
| Argon Laser | Melles Griot | Any other Laser, e.g., diode lasers emitting 488 is fine, too | |
| Ascorbic acid | Sigma | A92902 | |
| B-27 with Vitamin A | Gibco | 17504044 | |
| Bacto Agar | Becton Dickinson | 3% in PBS, store solution at -20 °C | |
| BDNF | Miltenyi Biotec | 130-093-0811 | |
| cAMP | Sigma | D0627 | |
| Cell Star cell culture 6 well plate | Greiner-Bio-One | 657160 | |
| Chemically Defined Lipid Concentrate | Gibco | 11905031 | |
| Confocal laser scanning microscope C1 | Nikon Microscope Solutions | Modular confocal microscope system | |
| Corning Matrigel Growth Factor Reduced (GFR) Basement membrane matrix, Phenol Red-free, LDEV-free | Corning | 356231 | Matrix component |
| CyQUANT Cell Proliferation Assay Kit | Thermo Fisher | C7026 | |
| DMEM/F12 | ThermoFisher | 31330038 | |
| DMSO | Sigma | D2660-100ML | |
| Donkey anti-goat Cy3 | Merck Millipore | AP180C | 1:300 |
| Donkey anti-mouse Cy3 | Merck Millipore | AP192C | 1:300 |
| Donkey anti-rabbit Cy3 | Merck Millipore | AP182C | 1:300 |
| DPBS | Gibco | 14190250 | |
| DS-Q1Mc camera | Nikon Microscope Solutions | ||
| Eclipse 90i upright widefield microscope | Nikon Microscope Solutions | ||
| Eclipse E 600FN upright microscope | Nikon Microscope Solutions | ||
| Eclipse Ts2 Inverted Microscope | Nikon Microsope Solutions | ||
| EZ-C1 Silver Version 3.91 | Nikon Microscope Solutions | Imaging software for confocal microscope | |
| FCCP | Sigma Aldrich | 370-86-5 | |
| Fetal Bovine Serum | Gibco | 10270-106 | |
| GDNF | Miltenyi Biotec | 130-096-291 | |
| Glasgow MEM | Gibco | 11710-035 | |
| Glass Pasteur pipette | Brand | 747715 | Inverted |
| Glutamax | Gibco | 35050-061 | |
| Helium-Neon Laser | Melles Griot | Every other Laser, e.g., diode lasers emitting 594 is fine, too | |
| Heparin | Merck | H3149-25KU | |
| HERACell 240i CO2 Incubator | Thermo Scientific | 51026331 | |
| Hoechst 33342 | Invitrogen | H3570 | 1:2500 |
| Image J 1.53c | Wayne Rasband National Institute of Health | Image processing Software | |
| Injekt Solo 10 mL/ Luer | Braun | 4606108V | |
| Knockout Serum Replacement | Gibco | 10828010 | |
| Laser (407 nm) | Coherent | Any other Laser, e.g., diode lasers emitting 407 is fine, too | |
| Map2 | Synaptic Systems | No. 188004 | 1:1000 |
| Maxisafe 2030i | |||
| MEM NEAA | Gibco | 11140-050 | |
| mTeSR Plus | Stemcell Technology | 85850 | iPSC medium |
| Multifuge X3R Centrifuge | Thermo Scientific | 10325804 | |
| MycoAlert Mycoplasma Detection Kit | Lonza | # LT07-218 | |
| N2 Supplement | Gibco | 17502-048 | |
| Needle for single usage (23G x 1” TW) | Neoject | 10016 | |
| NIS-Elements Aadvanced Research 3.2 | Nikon | Imaging software | |
| Oligomycin A | Sigma Aldrich | 75351 | |
| Orbital Shaker Heidolph Unimax 1010 | Heidolph | 543-12310-00 | |
| PAP Pen | Sigma | Z377821-1EA | To draw hydrophobic barrier on slides. |
| Papain Dissociation System kit | Worthington | LK003150 | |
| Paraformaldehyde | Merck | 818715 | 4% in PBS, store solution at -20 °C |
| Pasteur pipette 7mL | VWR | 612-1681 | Graduated up to 3 mL |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Plan Apo VC 20x / 0.75 air DIC N2 ∞/0.17 WD 1.0 | Nikon Microscope Solutions | Dry Microscope Objective | |
| Plan Apo VC 60x / 1.40 oil DIC N2 ∞/0.17 WD 0.13 | Nikon Microscope Solutions | Oil Immersion Microscope Objective | |
| Polystyrene Petri dish (100 mm) | Greiner Bio-One | 664161 | |
| Polystyrene round-bottom tube with cell-strainer cap (5 mL) | Falcon | 352235 | |
| Potassium chloride | Roth | 6781.1 | |
| ProLong Glass Antifade Moutant | Invitrogen | P36980 | |
| Qualitative filter paper | VWR | 516-0813 | |
| Rock Inhibitior | Merck | SCM075 | |
| Rotenone | Sigma | 83-794 | |
| S100β | Abcam | Ab11178 | 1:600 |
| SB-431542 | Cayman Chemical Company | 13031 | |
| Scalpel blades | Heinz Herenz Hamburq | 1110918 | |
| SMI312 | Biolegend | 837904 | 1:500 |
| Sodium bicarbonate | Merck/Sigma | 31437-1kg-M | |
| Sodium chloride | Roth | 3957 | |
| Sodium dihydrogen phosphate | Applichem | 131965 | |
| Sodium Pyruvate | Gibco | 11360070 | |
| SOX2 | Santa Cruz Biotechnology | Sc-17320 | 1:100 |
| StemPro Accutase Cell Dissociation Reagent | Gibco/StemPro | A1110501 | Reagent A |
| Super Glue Gel | UHU | 63261 | adhesive gel |
| SuperFrost Plus | VWR | 631-0108 | |
| Syringe for single usage (1 mL) | BD Plastipak | 300015 | |
| TB2 Thermoblock | Biometra | ||
| TC Plate 24 Well | Sarstedt | 83.3922 | |
| TC Plate 6 Well | Sarstedt | 83.392 | |
| TGFbeta3 | Miltenyi Biotec | 130-094-007 | |
| Tissue Culture Hood | ThermoFisher | 51032711 | |
| TOM20 | Santa Cruz Biotechnology | SC-11415 | 1:200 |
| Triton-X | Merck | X100-5ML | |
| UltraPure 0.5M EDTA | Invitrogen | 15575020 | |
| Vibratome Microm HM 650 V | Thermo Scientific | Production terminated, any other adjustable microtome is fine, too. | |
| Vibratome Wilkinson Classic Razor Blade | Wilkinson Sword | 70517470 | |
| Whatman Benchkote | Merck/Sigma | 28418852 | |
| Wnt Antagonist I | EMD Millipore Corp | 3378738 | |
| XF 96 extracellular flux analyser | Seahorse Bioscience | 100737-101 | |
| XF Assay DMEM Medium | Seahorse Bioscience | 103680-100 | |
| XF Calibrant Solution | Seahorse Bioscience | 100840-000 | |
| XFe96 FluxPak (96-well microplate) | Seahorse Bioscience | 102416-100 |
Riferimenti
- Koopman, W. J., Willems, P. H., Smeitink, J. A. Monogenic mitochondrial disorders. New England Journal of Medicine. 366 (12), 1132-1141 (2012).
- Gorman, G. S., et al. Mitochondrial diseases. Nature Review Disease Primers. 2, 16080 (2016).
- Vafai, S. B., Mootha, V. K. Mitochondrial disorders as windows into an ancient organelle. Nature. 491 (7424), 374-383 (2012).
- Carelli, V., Chan, D. C. Mitochondrial DNA: impacting central and peripheral nervous systems. Neuron. 84 (6), 1126-1142 (2014).
- Russell, O. M., Gorman, G. S., Lightowlers, R. N., Turnbull, D. M. Mitochondrial diseases: hope for the future. Cell. 181 (1), 168-188 (2020).
- Weissig, V. Drug development for the therapy of mitochondrial diseases. Trends in Molecular Medicine. 26 (1), 40-57 (2020).
- Tyynismaa, H., Suomalainen, A. Mouse models of mitochondrial DNA defects and their relevance for human disease. EMBO Reports. 10 (2), 137-143 (2009).
- Ma, H., et al. Metabolic rescue in pluripotent cells from patients with mtDNA disease. Nature. 524 (7564), 234-238 (2015).
- Galera-Monge, T., et al. Mitochondrial dysfunction and calcium dysregulation in Leigh syndrome induced pluripotent stem cell derived neurons. International Journal of Molecular Science. 21 (9), 3191 (2020).
- Zheng, X., et al. Alleviation of neuronal energy deficiency by mTOR inhibition as a treatment for mitochondria-related neurodegeneration. Elife. 5, 13378 (2016).
- Lorenz, C., et al. Human iPSC-derived neural progenitors are an effective drug discovery model for neurological mtDNA disorders. Cell Stem Cell. 20 (5), 659-674 (2017).
- Inak, G., et al. Concise review: induced pluripotent stem cell-based drug discovery for mitochondrial disease. Stem Cells. 35 (7), 1655-1662 (2017).
- Chiaradia, I., Lancaster, M. A. Brain organoids for the study of human neurobiology at the interface of in vitro and in vivo. Nature Neuroscience. 23 (12), 1496-1508 (2020).
- Lancaster, M. A., Knoblich, J. A. Generation of cerebral organoids from human pluripotent stem cells. Nature Protocol. 9 (10), 2329-2340 (2014).
- Liput, M., et al. Tools and approaches for analyzing the role of mitochondria in health, development and disease using human cerebral organoids. Developmental Neurobiology. , (2021).
- Winanto, K. Z. J., Soh, B. S., Fan, Y., Ng, S. Y. Organoid cultures of MELAS neural cells reveal hyperactive Notch signaling that impacts neurodevelopment. Cell Death and Disease. 11 (3), 182 (2020).
- Romero-Morales, A. I., et al. Human iPSC-derived cerebral organoids model features of Leigh Syndrome and reveal abnormal corticogenesis. bioRxiv. , (2020).
- Inak, G., et al. Defective metabolic programming impairs early neuronal morphogenesis in neural cultures and an organoid model of Leigh syndrome. Nature Communications. 12 (1), 1929 (2021).
- Falk, M. J. Neurodevelopmental manifestations of mitochondrial disease. Journal of Developmental & Behavioral Pediatrics. 31 (7), 610-621 (2010).
- Velasco, S., et al. Individual brain organoids reproducibly form cell diversity of the human cerebral cortex. Nature. 570 (7762), 523-527 (2019).
- Pfiffer, V., Prigione, A. Assessing the bioenergetic profile of human pluripotent stem cells. Methods in Molecular Biology. 1264, 279-288 (2015).
- Ludikhuize, M. C., Meerlo, M., Burgering, B. M. T., Colman, R. M. J. Protocol to profile the bioenergetics of organoids using Seahorse. STAR Protocols. 2 (1), 100386 (2021).
- Menacho, C., Prigione, A. Tackling mitochondrial diversity in brain function: from animal models to human brain organoids. International Journal of Biochemestry & Cell Biology. 123, 105760 (2020).
- Del Dosso, A., Urenda, J. P., Nguyen, T., Quadrato, G. Upgrading the physiological relevance of human brain organoids. Neuron. 107 (6), 1014-1028 (2020).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon