Method Article
Génération d’organoïdes cérébraux humains pour la modélisation des maladies mitochondriales
Dans cet article
Résumé
Nous décrivons un protocole détaillé pour la génération d’organoïdes cérébraux dérivés de cellules souches pluripotentes induites par l’homme et leur utilisation dans la modélisation des maladies mitochondriales.
Résumé
Les maladies mitochondriales représentent la plus grande classe d’erreurs innées du métabolisme et sont actuellement incurables. Ces maladies provoquent des anomalies neurodéveloppementales dont les mécanismes sous-jacents restent à élucider. Un obstacle majeur est le manque de modèles efficaces récapitulant la déficience neuronale précoce observée chez les patients. Les progrès de la technologie des cellules souches pluripotentes induites (CSPi) permettent la génération d’organoïdes cérébraux tridimensionnels (3D) qui peuvent être utilisés pour étudier l’impact des maladies sur le développement et l’organisation du système nerveux. Les chercheurs, y compris ces auteurs, ont récemment introduit des organoïdes cérébraux humains pour modéliser les troubles mitochondriaux. Cet article présente un protocole détaillé pour la génération robuste d’organoïdes cérébraux humains dérivés de l’iPSC et leur utilisation dans le profilage bioénergétique mitochondrial et les analyses d’imagerie. Ces expériences permettront l’utilisation d’organoïdes cérébraux pour étudier les dysfonctionnements métaboliques et développementaux et peuvent fournir des informations cruciales pour disséquer la pathologie neuronale des maladies mitochondriales.
Introduction
Les maladies mitochondriales représentent la plus grande classe d’erreurs innées du métabolisme1. Ils sont causés par des mutations génétiques perturbant différents processus mitochondriaux, y compris la phosphorylation oxydative (OXPHOS)2, l’assemblage de la chaîne respiratoire, la dynamique mitochondriale et la transcription ou la réplication de l’ADN mitochondrial3. Les tissus ayant des besoins énergétiques sont particulièrement affectés par le dysfonctionnement mitochondrial4. En conséquence, les patients atteints de maladies mitochondriales développent généralement des manifestations neurologiques précoces.
Il n’existe actuellement aucun traitement disponible pour les enfants atteints de maladies mitochondriales5. Un obstacle majeur au développement de médicaments pour les maladies mitochondriales est le manque de modèles efficaces récapitulant l’évolution de la maladie humaine6. Plusieurs des modèles animaux actuellement étudiés ne présentent pas les défauts neurologiques présents chez les patients7. Par conséquent, les mécanismes sous-jacents à la pathologie neuronale des maladies mitochondriales ne sont toujours pas entièrement compris.
Des études récentes ont généré des CSPi de patients atteints de maladies mitochondriales et ont utilisé ces cellules pour obtenir des cellules neuronales spécifiques au patient. Par exemple, des défauts génétiques associés à la maladie mitochondriale, le syndrome de Leigh, se sont avérés causer des aberrations dans la bioénergétique cellulaire8,9, la synthèse des protéines10 et l’homéostasie du calcium9,11. Ces rapports ont fourni d’importants indices mécanistes sur la déficience neuronale survenant dans les maladies mitochondriales, ouvrant la voie à la découverte de médicaments pour ces maladies incurables12.
Les cultures bidimensionnelles (2D), cependant, ne permettent pas d’étudier la complexité architecturale et l’organisation régionale des orgues 3D13. À cette fin, l’utilisation d’organoïdes cérébraux 3D dérivés d’iPSC spécifiques au patient14 peut permettre aux chercheurs d’obtenir des informations supplémentaires importantes et ainsi aider à disséquer l’impact des maladies mitochondriales sur le développement et la fonction du système nerveux15. Des études utilisant des organoïdes cérébraux dérivés de l’iPSC pour étudier les maladies mitochondriales commencent à découvrir les composants neurodéveloppementaux des maladies mitochondriales.
Les organoïdes de la moelle épinière porteurs de mutations associées à la maladie mitochondriale, à l’encéphalopathie mitochondriale, à l’acidose lactique et au syndrome des épisodes ressemblant à des accidents vasculaires cérébraux (MELAS) ont montré une neurogenèse défectueuse et une différenciation retardée des motoneurones16. Les organoïdes corticaux dérivés de patients atteints de la maladie mitochondriale, le syndrome de Leigh, ont montré une taille réduite, des défauts dans la génération de bourgeons épithéliaux neuronaux et une perte de l’architecture corticale17. Les organoïdes cérébraux de patients atteints du syndrome de Leigh ont montré que les défauts de la maladie s’initient au niveau des cellules progénitrices neurales, qui ne peuvent pas s’engager dans le métabolisme mitochondrial, provoquant une ramification neuronale aberrante et une morphogenèse18. Ainsi, les progéniteurs neuronaux peuvent représenter une cible thérapeutique cellulaire pour les maladies mitochondriales, et les stratégies favorisant leur fonction mitochondriale peuvent soutenir le développement fonctionnel du système nerveux.
L’utilisation d’organoïdes cérébraux pourrait aider à découvrir les composants neurodéveloppementaux des maladies mitochondriales. Les maladies mitochondriales sont principalement considérées comme une neurodégénérescence précoce5. Cependant, des défauts neurodéveloppementaux sont également présents chez les patients atteints de maladies mitochondriales, notamment un retard de développement et des troubles cognitifs19. Les organoïdes cérébraux spécifiques au patient peuvent aider à traiter ces aspects et à élucider comment les maladies mitochondriales peuvent avoir un impact sur le développement du cerveau humain. Le dysfonctionnement mitochondrial pourrait également jouer un rôle pathogénétique dans d’autres maladies neurologiques plus courantes, telles que la maladie d’Alzheimer, la maladie de Parkinson et la maladie de Huntington4. Par conséquent, élucider l’impact des défauts mitochondriaux dans le développement neurologique à l’aide d’organoïdes cérébraux pourrait également être déterminant pour l’étude de ces maladies. Cet article décrit un protocole détaillé pour générer des organoïdes cérébraux reproductibles qui peuvent être utilisés pour effectuer la modélisation des maladies mitochondriales.
Protocole
REMARQUE: L’utilisation de CSPi humaines peut nécessiter une approbation éthique. Les CSPi utilisées dans cette étude ont été dérivées de personnes témoins en bonne santé après approbation éthique locale (#2019-681). Toutes les procédures de culture cellulaire doivent être effectuées sous une hotte de culture cellulaire stérile, en désinfectant soigneusement tous les réactifs et consommables avant de les transférer sous le capot. Les CSPi humaines utilisées pour la différenciation devraient avoir un nombre de passage inférieur à 50 pour éviter les aberrations génomiques potentielles qui pourraient se produire lors d’une culture extensive. L’état pluripotent des cellules doit être validé avant la génération d’organoïdes, par exemple en surveillant l’expression de marqueurs associés à la pluripotence tels que NANOG ou OCT4. Des tests de mycoplasmes doivent être effectués chaque semaine pour s’assurer que les cultures sont exemptes de mycoplasmes.
1. Génération d’organoïdes cérébraux
- Culture des CSPi humaines
- Cultiver des CSPi humaines dans des conditions sans mangeoire dans un milieu iPSC (voir le tableau des matériaux) sur des plaques revêtues de 6 puits et les conserver dans un incubateur de culture tissulaire humidifié à 37 °C et 5 % de CO2.
REMARQUE: Le transfert des cellules nourricières peut entraver la différenciation organoïde. Passage des cellules au moins une fois dans des conditions sans mangeoire. - Passage des CSPi à 80% de confluence en utilisant un milieu de détachement sans enzyme dans des rapports allant de 1:4 à 1:12. Pour augmenter la survie cellulaire, ajoutez 10 μM d’inhibiteur de la protéine kinase rho-associée (ROCK) (Y27632) après chaque fractionnement.
- Cultiver des CSPi humaines dans des conditions sans mangeoire dans un milieu iPSC (voir le tableau des matériaux) sur des plaques revêtues de 6 puits et les conserver dans un incubateur de culture tissulaire humidifié à 37 °C et 5 % de CO2.
- Dissocier les CSPi (80 % de confluence) - Jour 0.
- Préparer le milieu de différenciation corticale I (CDMI) (tableau 1). Préchauffer le milieu CDMI à température ambiante (22-25 °C) avant de l’ajouter aux cellules.
- Lavez les puits contenant les CSPi avec une solution saline tamponnée au phosphate (PBS) pour éliminer les cellules mortes et les débris.
- Ajouter 500 μL de réactif préavertisSé A (table des matières) à chaque puits et incuber pendant 5 min à 37 °C. Vérifiez au microscope pour vous assurer du détachement des cellules.
- Ajouter 1 mL de milieu iPSC pour diluer le réactif A afin de neutraliser son activité.
- Utilisez une pipette de 1000 μL pour dissocier les cellules en les pipetant de haut en bas et transférez la suspension de la cellule dans un tube de centrifugeuse de 15 mL.
- Centrifuger doucement les iPSC à 125 x g pendant 5 min à température ambiante (22-25 °C).
- Aspirez soigneusement le surnageant pour éviter de perturber la pastille cellulaire.
- Remettez en suspension la pastille avec 1 mL de CDMI pour obtenir une suspension à cellule unique et comptez le nombre de cellules.
- Préparer le milieu d’ensemencement avec 9 000 cSPi par 100 μL dans cdMI complété par un inhibiteur de ROCK de 20 μM, un inhibiteur de la caténine WNT de 3 μM (IWR1) et 5 μM de SB431542.
- Ajouter 100 μL de milieu d’ensemencement par puits à une plaque de fond en V de 96 puits.
- Conservez la plaque dans un incubateur de culture tissulaire humidifié à 37 °C et 5 % de CO2.
- Génération de neurosphère
- Le jour 1, observez que des agrégats cellulaires ronds (neurosphères) avec des bordures lisses définies se forment. Notez les cellules mortes autour des agrégats. Continuer à cultiver dans l’incubateur à 37 °C et 5% de CO2.
- Le jour 3, agitez la plaque en tapotant trois fois sur les côtés pour détacher les cellules mortes.
- Ajouter 100 μL de CDMI complété par un inhibiteur de ROCK de 20 μM, 3 μM IWR1 et 5 μM SB431542 à chaque puits.
- Remettre la plaque dans l’incubateur à 37 °C et 5 % de CO2.
- Le jour 6, retirez soigneusement 80 μL du milieu surnageant de chaque puits. Évitez de toucher le fond du puits.
- Ajouter 100 μL de CDMI complété par 3 μM IWR1 et 5 μM SB431542 à chaque puits. Remettre la plaque dans l’incubateur à 37 °C et 5 % de CO2.
- Répétez les étapes 5 et 6 tous les 3 jours jusqu’au jour 18.
- Transfert de neurosphères
- Le jour 18, préparer le milieu de différenciation corticale II (CDMII) (tableau 1) et ajouter 10 mL à une plaque de culture cellulaire à ultra-faible fixation de 100 mm.
- Utilisez une pipette de 200 μL avec la pointe coupée pour transférer les neurosphères rondes de la plaque de 96 puits à la plaque de culture cellulaire à très faible fixation de 100 mm.
REMARQUE: Soyez doux pour éviter d’endommager les neurosphères en vous assurant que l’ouverture de la pointe est assez large et que les agrégats ne sont pas aspirés trop rapidement. - Retirer 5 mL de milieu de la plaque contenant les neurosphères et ajouter 5 mL de CDMII frais.
REMARQUE: Cette procédure aide à réduire la quantité de milieu CDMI qui peut avoir été transférée par le transfert de neurosphères. - Placez la plaque sur un agitateur orbital à 70 tr/min à l’intérieur d’un incubateur de culture tissulaire humidifié à 37 °C et 5 % de CO2.
REMARQUE: Inspectez visuellement les neurosphères le lendemain. Augmentez la vitesse du shaker orbital si les neurosphères sont agglomérées ou attachées au fond de la plaque. - Tous les 3 jours, aspirez soigneusement le milieu surnageant et remplacez-le par du CDMII frais. Laissez une petite quantité du milieu pour empêcher les neurosphères de se dessécher.
- Le jour 35, préparez le milieu de différenciation corticale III (CDMIII) (tableau 1).
REMARQUE: Le composant de la matrice doit être dissous dans le CDMIII froid. - Aspirer le milieu de la plaque et ajouter 10 mL de CDMIII froid.
REMARQUE: Il est plus efficace d’utiliser un milieu froid afin que le composant de la matrice puisse recouvrir les organoïdes sans former d’amas. - Après avoir changé le milieu, replacez la plaque sur un agitateur orbital à 70 tr/min à l’intérieur d’un incubateur de culture tissulaire humidifié à 37 °C et 5 % de CO2.
- Changez le milieu tous les 3-5 jours en fonction du taux de croissance, comme indiqué par la couleur du milieu.
- Le jour 70, préparez le milieu de différenciation corticale IV (CDMIV) (tableau 1). Utilisez le milieu CDMIV jusqu’à ce que l’âge souhaité des organoïdes soit atteint. Pendant cette période, maintenez la plaque sur un agitateur orbital réglé à 70 tr/min à l’intérieur d’un incubateur de culture tissulaire humidifié (37 °C et 5 % de CO2).
- Changez le milieu tous les 3-5 jours, en fonction du taux de croissance.
2. Immunocoloration des organoïdes cérébraux
- Préparation des tissus
- Préparez une solution de paraformaldéhyde (PFA) à 4 % et placez-la sous une hotte de sécurité.
REMARQUE: Portez de l’équipement de sécurité personnelle lorsque vous manipulez du PFA. - Recueillez les organoïdes cérébraux et transférez-les doucement avec une pipette Pasteur en plastique de 3 mL à pointe émoussée dans une plaque de 6 puits remplie de PFA.
REMARQUE: Utilisez des organoïdes de plus de 40 jours pour permettre la visualisation de structures avec une complexité cellulaire plus élevée. - Conserver les organoïdes dans la solution de PFA pendant 1 h à température ambiante.
- Retirez soigneusement le PFA avec une pipette Pasteur en plastique de 3 mL et lavez les organoïdes fixes trois fois à l’aide de PBS.
- Conserver les organoïdes fixes à 4 °C dans du PBS jusqu’à nouvel usage.
- Préparez une solution de paraformaldéhyde (PFA) à 4 % et placez-la sous une hotte de sécurité.
- Préparation de tranches organoïdes cérébrales
- Préparer une solution de gélose à 3% et chauffer lentement jusqu’à ce qu’elle soit liquéfiée.
- Placez le moule (l’extrémité coupée d’une seringue de 10 mL) sur un morceau de papier filtre absorbant (côté lisse vers le haut). Placez une gouttelette de gélose dessus.
- Retirez rapidement un seul organoïde de la plaque de 6 puits avec une spatule et retirez l’excès de PBS avec du papier filtre.
REMARQUE: Veillez à ne pas toucher l’organoïde directement avec du papier filtre. - Placez l’organoïde sur la gouttelette d’agar.
- Répétez cette procédure avec jusqu’à trois organoïdes.
REMARQUE: Travaillez rapidement pour éviter la solidification de la gélose au cours de cette étape. - Remplissez le moule avec de la gélose jusqu’à ce que tous les organoïdes soient complètement recouverts.
- Attendez que la gélose commence à se solidifier, puis transférez doucement tout le moule contenant les organoïdes, y compris le papier filtre absorbant, sur un élément de refroidissement.
REMARQUE: Si un élément de refroidissement n’est pas disponible, conservez les organoïdes pendant quelques minutes dans un réfrigérateur à 4 ° C. - En attendant, préparez-vous à la procédure de tranchage: placez une lame de rasoir (nettoyée à l’acétone et lavée à l’eau double distillée) dans le support du vibratome, montez-la sur le bain et remplissez-la de PBS.
- Retirez le moule de la gélose (solidifiée) et utilisez un scalpel pour le couper afin de former un cube.
- Fixez le cube de gélose contenant les organoïdes sur la plaque porteuse du vibratome avec du gel adhésif (voir le tableau des matériaux) et placez-le dans le bain contenant du PBS.
- Ajustez le vibratome (voir le tableau des matériaux) pour couper des tranches à une épaisseur de 150 μm.
REMARQUE: Les réglages du vibratome (angle, amplitude, fréquence et vitesse appropriés de la lame) peuvent être similaires à ceux utilisés pour trancher le tissu cérébral fixe dérivé d’animaux postnatals précoces. Cependant, les réglages idéaux dépendent fortement du type de vibratome et doivent être déterminés dans un premier temps pour éviter la distorsion ou même l’arrachage du tissu lors de la coupe. - Commencez la procédure de coupe. Utilisez une pipette en verre ou une spatule pour transférer doucement chaque tranche fraîchement coupée dans une assiette de 24 puits remplie de PBS.
- Conserver la plaque contenant les tranches à 4 °C (jusqu’à quelques jours) jusqu’à la suite du traitement.
- Transférez les tranches hors de la plaque avec une pipette en verre ou une spatule sur des lames de microscope. Utilisez un minimum de 2 tranches par diapositive.
- Retirez soigneusement la gélose et l’excès de PBS avec une seringue.
- Laissez sécher les tranches jusqu’à ce qu’elles adhèrent aux lames.
REMARQUE: Bien que les lames de microscope puissent être stockées dans des chambres en plastique remplies de PBS à 4 ° C, elles doivent être colorées dès que possible après la procédure de tranchage.
- Coloration immunohistochimique
- Préparez la solution bloquante (Tableau 1).
- Utilisez un stylo PAP pour dessiner une bordure hydrophobe autour des tranches sur la diapositive afin de garder toutes les solutions sur les diapositives.
- Ajouter soigneusement la solution bloquante sur la lame et incuber pendant 1 h à température ambiante (22-25 °C). Pour éviter de détruire le tissu, n’ajoutez pas la solution directement sur les tranches.
- Aspirer la solution bloquante et appliquer l’anticorps primaire désiré dilué dans la solution bloquante.
- Incuber la lame pendant la nuit dans une chambre humidifiée à 4 °C.
- Rincez la lame trois fois avec 1x PBS pendant 10 minutes chacun.
- Incuber les tranches avec l’anticorps secondaire spécifique dilué dans la solution bloquante et effectuer une coloration Hoechst (1:2 500) pendant 1 h à température ambiante dans l’obscurité.
REMARQUE: N’oubliez pas d’effectuer des contrôles négatifs pour confirmer qu’il n’y a pas de liaison non spécifique ou d’auto-fluorescence. - Rincez trois fois avec 1x PBS pendant 10 minutes chacun dans l’obscurité.
- Ajoutez une goutte de support de montage à la tranche, placez un couvercle sur le bord de la goutte et posez lentement le couvercle vers le bas vers la tranche pour éviter les bulles d’air.
- Laissez la glissière reposer pendant la nuit à température ambiante. Appliquez du vernis à ongles sur la bordure du couvercle pour sceller davantage la diapositive. Pour un stockage à long terme, conserver à 4 °C.
- Documentation de la coloration
- Pour numériser de grandes images pour l’assemblage, utilisez un microscope à grand champ vertical motorisé équipé (voir la table des matériaux pour plus de détails) d’un objectif de haute qualité; Ensemble de filtres DAPI (par exemple, excitation (EX): 340-380 nm, miroir dichroïque (DM): 400 nm, filtre barrière (BA): 435-485 nm); ensemble de filtres à isothiocyanate de fluorescéine (p. ex. EX : 465-495 nm, DM : 505 nm, BA : 515-555 nm); Ensemble de filtres Alexa594 (par exemple, ET 575/40; T 600 LPXR; HC 623/24); appareil photo numérique; logiciel d’acquisition haute performance permettant des points de suture et des opérations de pile automatisés.
- Pour la gestion des images, utilisez un programme de traitement d’image capable de générer des fichiers tif 8 bits, de recadrer des points, d’ajuster le contraste et la luminosité, de fusionner les couches (par exemple, bleu, vert et rouge) et d’ajouter des barres d’échelle.
- Pour numériser des détails, utilisez un microscope à balayage laser confocal motorisé équipé d’un objectif de haute qualité, un laser UV (EX: 408 nm), un laser Argon (EX: 488 nm), un laser Hélium-Néon (EX: 543), un logiciel d’imagerie pour un microscope confocal.
- Pour la gestion des détails de l’image, utilisez un programme de traitement d’image capable de générer des projections d’intensité maximale de piles z confocales (par exemple, des sections optiques de 0,6 μm chacune), de générer des fichiers tif 8 bits, d’ajuster le contraste et la luminosité, de fusionner les canaux (par exemple, bleu, vert et rouge), d’ajouter des barres d’échelle.
- Utilisez un éditeur graphique pour organiser les figures.
3. Profilage bioénergétique des organoïdes cérébraux
- Préparation d’organoïdes pour le profilage bioénergétique
- Préparez la solution de papaïne et de DNase en suivant le protocole du fabricant.
- Transférer 3 à 5 organoïdes dans une plaque de 6 puits. Lavez-les deux fois avec du PBS préavertissé.
- Ajouter 2 mL de solution de papaïne activée préavertissée contenant de la DNase. À l’aide d’une lame, coupez les organoïdes en petits morceaux.
- Placez la plaque sur un agitateur orbital réglé à 27 tr/min à l’intérieur d’un incubateur de culture cellulaire (à 37 °C, 5 % de CO2) et incubez pendant 15 à 20 min.
REMARQUE: Le temps d’incubation dépend du stade organoïde. Les organoïdes à un stade précoce peuvent être utilisés tels quels. Pour les organoïdes de plus de 3 mois, il est recommandé de couper les organoïdes en 2-3 morceaux avant la dissociation et d’incuber les morceaux à une vitesse de basculement fixée à 27 tr/min pendant 15-20 min à 37 °C. Cette procédure peut aider à éliminer le tissu nécrotique qui peut être présent dans les organoïdes à un stade avancé. - Prélever les tissus digérés dans un tube de 15 mL et ajouter 5 mL de milieu de culture organoïde CDMIV (tableau 1).
- Triturez le tissu avec une pipette en plastique de 10 mL en pipetant de haut en bas 10 à 15 fois. Laissez le tissu non dissocié se déposer au fond du tube.
- Transférez soigneusement la suspension cellulaire dans un tube de 15 mL, en évitant tout morceau de tissu non dissocié. Filtrer la solution à l’aide d’une crépine cellulaire de 40 μm (p. ex., tubes à fond rond en polystyrène avec capuchons de crépine cellulaire).
- Abattez les cellules par centrifugation à 300 x g pendant 5 min à température ambiante.
- Évaluez le nombre de cellules et la qualité à l’aide du bleu trypan.
- Plaquez le nombre souhaité (~ 20 000/puits) de cellules sur des microplaques revêtues de 96 puits. Changer le milieu 6-8 h après le placage en milieu neuronal (tableau 1).
- Incuber la microplaque enduite de 96 puits dans un incubateur à CO2 (37 °C, 5 % de CO2) pendant 4 jours.
- Profilage bioénergétique
- Le jour 3, après avoir replaqué les cellules dissociées, ajoutez 200 μL de solution d’étalonnage dans chaque puits de la partie inférieure de la microplaque de 96 puits et placez la cartouche de capteur verte supérieure sur la microplaque hydratée.
REMARQUE: Placez la cartouche de capteur sur le dessus de la microplaque dans la bonne orientation et assurez-vous que la solution d’étrier couvre tous les capteurs. - Incuber la microplaque hydratée de 96 puits dans un incubateur sans CO2 à 37 °C pendant la nuit.
- Allumez l’analyseur pour permettre à l’instrument de se stabiliser à 37 °C pendant la nuit.
- Le jour 4 après la replaquage, inspectez la culture organoïde dissociée sur la microplaque de 96 puits sous le microscope pour vous assurer que les cellules apparaissent comme une monocouche confluente.
- Préparer le milieu d’essai (tableau 1).
- Retirez le milieu neuronal de tous les puits avec une pipette sans toucher le fond du puits pour éviter d’endommager les cellules. Alternativement, inversez soigneusement toute la plaque, puis séchez-la sur du papier propre. Travaillez rapidement pour éviter la mort cellulaire.
- Lavez les cellules deux fois avec 200 μL de milieu d’essai préavertissé. Ajouter le milieu d’essai à un volume final de 180 μL par puits. Incuber la microplaque de 96 puits dans un incubateur sans CO2 à 37 °C pendant 1 h.
- Préparer des solutions de 10 μM d’inhibiteurs mitochondriaux dans le milieu d’essai. Notez que la concentration finale après injection est de 1 μM.
- Chargez la cartouche de capteur placée dans la microplaque hydratée avec 10x solutions d’inhibiteurs mitochondriaux.
- Ajouter 18 μL d’inhibiteur mitochondrial 1 dans le port A.
- Ajouter 19,8 μL d’inhibiteur mitochondrial 2 dans le port B.
- Ajouter 21,6 μL d’inhibiteur mitochondrial 2 dans le port C.
- Ajouter 23,4 μL d’inhibiteur mitochondrial 3 dans le port D.
- Placer la cartouche chargée dans la microplaque hydratée dans un incubateur sans CO2 à 37 °C jusqu’au début du test.
- Configurez un protocole d’exécution dans le logiciel de l’instrument (Tableau 2).
- Appuyez sur DÉMARRER. Prenez la cartouche chargée de l’incubateur sans CO2 et placez-la dans l’analyseur pour l’étalonnage.
REMARQUE: Assurez-vous que la plaque est insérée dans la bonne orientation et sans le couvercle. - Une fois l’étape d’étalonnage terminée, retirez la plaque d’étalonnage. Prenez la microplaque de 96 puits de l’incubateur sans CO2 et placez-la dans l’analyseur. Cliquez sur CONTINUER pour démarrer les mesures.
- Lorsque l’exécution est terminée, retirez la microplaque de culture cellulaire de 96 puits de l’analyseur et collectez le milieu de tous les puits sans perturber les cellules.
REMARQUE: Le milieu peut être conservé à -20 ° C et utilisé plus tard pour mesurer la quantité de lactate libérée par les cellules dans le milieu à l’aide d’un kit de dosage de lactate approprié. - Lavez les cellules avec 200 μL de 1x PBS dans chaque puits.
- Après avoir retiré le PBS, congeler la plaque à -20 °C.
REMARQUE: La plaque congelée peut être utilisée pour quantifier les cellules, les protéines ou l’ADN dans chaque puits de la microplaque. Cette quantification sera nécessaire pour normaliser les taux bioénergétiques obtenus. Suivez les instructions du fabricant pour les tests de quantification des cellules, des protéines ou de l’ADN.
- Le jour 3, après avoir replaqué les cellules dissociées, ajoutez 200 μL de solution d’étalonnage dans chaque puits de la partie inférieure de la microplaque de 96 puits et placez la cartouche de capteur verte supérieure sur la microplaque hydratée.
Résultats
Le protocole décrit ici facilite la génération robuste d’organoïdes ronds (Figure 1A). Les organoïdes générés contiennent des neurones matures qui peuvent être visualisés à l’aide de marqueurs protéiques spécifiques aux axones (SMI312) et aux dendrites (protéine 2 associée aux microtubules (MAP2)) (Figure 1B).. Les organoïdes matures contiennent non seulement des cellules neuronales (MAP2-positives), mais aussi des cellules gliales (par exemple, positives pour le marqueur astrocytaire S100 de la protéine B de liaison au calcium (S100ß)) (Figure 1B)).
En analysant les organoïdes cérébraux tranchés à l’aide de la microscopie confocale, il est possible d’identifier et de surveiller la distribution détaillée et l’organisation de différents types de cellules et de structures cellulaires. Cela pourrait fournir de nouvelles informations sur la façon dont les maladies mitochondriales pourraient affecter le développement du système nerveux. Par exemple, il est possible de surveiller les axones neuronaux (SMI312-positif) et les dendrites (MAP2-positif) (Figure 2A) ou l’apparition mutuelle de cellules neuronales (MAP2-positive) et de cellules gliales (S100ß-positive) (Figure 2A). Les images confocales peuvent également aider à étudier plus en détail la distribution et l’organisation des progéniteurs neuronaux ((région déterminante du sexe Y) box-2 (SOX2)-positif) en ce qui concerne les neurones (tubuline bêta-III (TUJ1)-positif) (Figure 2B). Enfin, les organoïdes cérébraux peuvent être colorés pour des marqueurs spécifiques aux mitochondries (tels que la protéine de la membrane mitochondriale externe, la translocase de la sous-unité de membrane externe 20 kDa (TOM20)) (Figure 2C)).
Le protocole décrit permet aux chercheurs d’effectuer un profilage bioénergétique des organoïdes cérébraux. En utilisant cette procédure, il est possible de mesurer à la fois le métabolisme mitochondrial en utilisant le taux de consommation d’oxygène (OCR) (Figure 2D) et le métabolisme glycolytique en utilisant le taux d’acidification extracellulaire (ECAR) (Figure 2E). Le profilage bioénergétique permet de surveiller comment les cellules peuvent modifier leurs profils OCR et ECAR en réponse à une administration séquentielle d’inhibiteurs mitochondriaux.
Tout d’abord, l’inhibiteur de l’ATP synthase, l’olligomycine, peut être appliqué. L’oligomycine provoque une baisse du profil OCR (Figure 2D) et, par conséquent, identifie l’OCR nécessaire à la production d’ATP. Lors du traitement à l’oligomycine, il peut également y avoir une augmentation compensatoire de l’ECAR (Figure 2E), ce qui suggère que les cellules peuvent réguler à la hausse la glycolyse pour prévenir le stress métabolique causé par la réduction du métabolisme mitochondrial. La double application ultérieure de l’ionophore de protons, le cyanure de carbonyle-p-trifluorométhoxyphénylhydrazon (FCCP), provoque la perte du potentiel de la membrane mitochondriale. Comme les molécules d’oxygène sont maintenant libres de se déplacer, cela provoque une augmentation rapide de l’OCR (Figure 2D).
Ces changements dans le profil OCR identifient la capacité respiratoire maximale des cellules. L’administration finale de roténone plus antimycine A provoque un blocage du transport d’électrons et, par conséquent, une forte diminution de l’OCR (Figure 2D). L’ECAR peut présenter une fluctuation après le traitement par FCCP et roténone plus antimycine A (Figure 2E), en fonction de la capacité glycolytique résiduelle des cellules. Les profils OCR et ECAR peuvent être radicalement modifiés chez les organoïdes cérébraux dérivés de patients mitochondriaux.
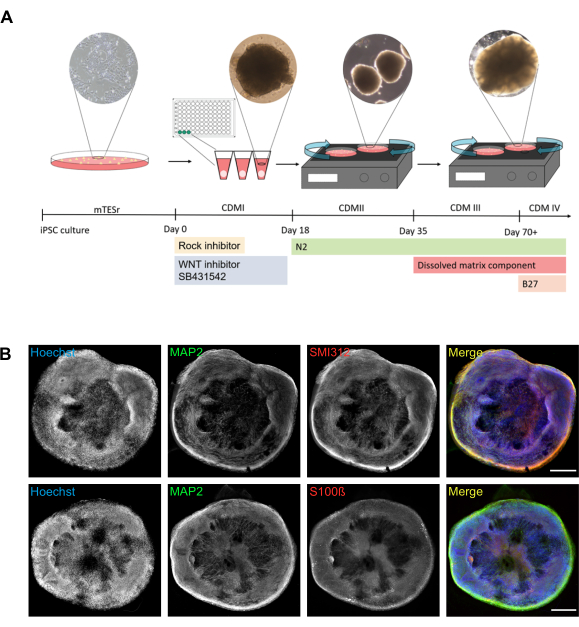
Figure 1: Génération d’organoïdes cérébraux à partir de CSPi humaines. (A) Représentation schématique du protocole utilisé pour produire des organoïdes cérébraux avec les images de transmission correspondantes. Le jour 0 correspond à la dissociation des CSPi et à l’ensemencement dans une plaque de 96 puits avec fond en V à l’aide de CDMI complété par un inhibiteur de ROCK, un inhibiteur de WNT et SB431542. Au jour 18, les neurosphères sont transférées des plaques de 96 puits vers des boîtes de culture cellulaire de 100 mm avec CDMII complété par N2. À partir de ce moment, les cultures sont positionnées sur un agitateur orbital. Au jour 35, le milieu passe de CDMII à CDMIII, qui contient également une composante de matrice dissoute (tableau 1). À partir du jour 70, le CDMIII passe au CDMIV complété par le B27. Une image représentative de la neurosphère a été prise au jour 12 à l’aide d’une caméra de microscope avec un grossissement de 10x. Une première image organoïde a été prise au jour 22 à l’aide d’une caméra de microscope à grossissement 4x. Une image organoïde mature a été prise au jour 40 à l’aide d’une caméra de microscope avec un grossissement 4x. (B) La structure globale et l’organisation cellulaire des organoïdes cérébraux peuvent être visualisées à l’aide de la microscopie à grand champ. Des images représentatives à grand champ cousu sont montrées pour visualiser les relations entre les dendrites (MAP2-positives) et les axones (SMI312-positif), et entre les cellules neuronales (MAP2-positives) et les astrocytes présumés (S100ß-positif). Les cellules ont été contre-colorées avec Hoechst pour révéler les noyaux. Toutes les images ont été prises à l’aide d’organoïdes cérébraux âgés de 78 jours. La colonne de droite montre la superposition de trois canaux (fusion). Barres d’échelle = 500 μm. Abréviations : iPSC = cellules souches pluripotentes induites; MDP = Milieu de différenciation corticale; ROCK = Rho kinase; MAP2 = protéine 2 associée aux microtubules; S100ß= S100 protéine B liant le calcium. Veuillez cliquer ici pour voir une version plus grande de cette figure.

Figure 2 : Visualisation et profilage bioénergétique des organoïdes cérébraux pour la modélisation des maladies mitochondriales. L’organisation détaillée et l’architecture des organoïdes peuvent être analysées à l’aide de la microscopie confocale. Toutes les images ont été prises à l’aide d’organoïdes cérébraux âgés de 78 jours et contre-colorées avec Hoechst pour révéler les noyaux. La colonne de droite montre la superposition de trois canaux (fusion). Barres d’échelle = 50 μm. (A) Projections représentatives à foyer étendu (44-48 plans optiques, 0,6 μm chacun) abordant l’interaction entre les dendrites (MAP2-positives, pointes de flèches) et les axones (SMI312-positif, flèches), et entre les cellules neuronales (MAP2-positives, pointes de flèches) et les astrocytes présumés (S100ß-positif, flèches). (B) Projections représentatives à foyer étendu (14 à 31 plans optiques, 0,6 μm chacun) montrant la distribution des neurones (TUJ1 positif, pointes de flèche) par rapport aux progéniteurs neuronaux (SOX2 positifs, flèches). (C) Projections représentatives à foyer étendu (20 plans optiques, 0,6 μm chacun) montrant la distribution dans les neurones (TUJ1-positif, pointes de flèche) des mitochondries (visualisées à l’aide d’anticorps contre la protéine de membrane mitochondriale externe TOM20, flèches). (D) La respiration mitochondriale des organoïdes cérébraux peut être surveillée en fonction du profil de l’OCR après administration séquentielle de différents inhibiteurs mitochondriaux (voir le texte pour plus de détails). (E) L’activité glycolytique des organoïdes cérébraux peut être surveillée sur la base de l’ECAR lors de l’administration séquentielle d’inhibiteurs mitochondriaux (voir le texte pour plus de détails). Pour le profilage bioénergétique, environ 10 à 15 organoïdes cérébraux ont été dissociés pour obtenir suffisamment de cellules pour la replaque sur la microplaque de 96 puits. Les barres indiquent les SEM sur la base des résultats obtenus dans deux expériences indépendantes. Abréviations : MAP2 = protéine 2 associée aux microtubules ; S100ß = S100 protéine B liant le calcium; TUJ1 = tubuline bêta-III; SOX2 = (région déterminante du sexe Y) case 2; TOM20 = translocase de la membrane externe 20 kDa sous-unité; OCR = taux de consommation d’oxygène; ECAR = taux d’acidification extracellulaire; Oligome. = oligomycine; FCCP = cyanure de carbonyle-p-trifluorométhoxyphénylhydrazon; R = roténone; Anta = antimycine A; SEM = erreur-type des moyennes. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Composition des médias | |||
| CDMI (Jour 0-18) | Conc. final. | ||
| Glasgow-MEM | Gibco | 11710-035 | [1:1] |
| Remplacement du sérum Knockout (KSR) | Gibco | 10828010 | 20% |
| MEM-NEAA (solution d’acides aminés non essentiels MEM) | Gibco | 11140-050 | 0,1 mM |
| Sodium Pyruvate | Gibco | 11360070 | 1 mM |
| 2-mercaptéthanol | Gibco | 31350-010 | 0,1 mM |
| Pénicilline et streptomycine | Gibco | 15140-122 | 100 U/mL et 100 μg/mL |
| CDMII (Jour 18-35) | Conc. final. | ||
| DMEM/F12 | Gibco | 31330038 | [1:1] |
| Glutamax | Gibco | 35050-061 | 2 mM |
| Supplément N-2 (100x) | Gibco | 17502-048 | 1% |
| Concentré lipidique chimiquement défini | Gibco | 11905031 | 1% |
| Pénicilline et streptomycine | Gibco | 15140-122 | 100 U/mL et 100 μg/mL |
| MDPIII (Jour 35-70) | Conc. final. | ||
| DMEM/F12 | Gibco | 31330038 | [1:1] |
| Glutamax | Gibco | 35050-061 | 2 mM |
| Supplément N-2 (100x) | Gibco | 17502-048 | 1% |
| Concentré lipidique chimiquement défini | Gibco | 11905031 | 1% |
| Pénicilline et streptomycine | Gibco | 15140-122 | 100 U/mL et 100 μg/mL |
| Sérum fœtal bovin (FBS) | Gibco | 10270-106 | 10% |
| Héparine | Merck | H3149-25KU | 5 μg/mL |
| Matrigel | Corning | 356231 | 1% |
| CDMIV(Jour 70+) | Conc. final. | ||
| DMEM/F12 | Gibco | 31330038 | [1:1] |
| Glutamax | Gibco | 35050-061 | 2 mM |
| Supplément N-2 (100x) | Gibco | 17502-048 | 1% |
| Concentré lipidique chimiquement défini | Gibco | 11905031 | 1% |
| Pénicilline et streptomycine | Gibco | 15140-122 | 100 U/mL et 100 μg/mL |
| Sérum fœtal bovin (FBS) | Gibco | 10270-106 | 10% |
| Héparine | Merck | H3149-25KU | 5 μg/mL |
| Matrigel | Corning | 356231 | 2% |
| Supplément de B-27 avec vitamine A 50x | Gibco | 17504044 | 2% |
| Milieu neuronal | Conc. final. | ||
| DMEM/F12 | Gibco | 31330038 | [1:1] |
| Supplément N-2 | Gibco | 17502048 | [1x] |
| Supplément de B-27 avec vitamine A 50x | Gibco | 17504044 | [1x] |
| Acide L-ascorbique | Sigma Aldrich | N° A92902 | [200 μM] |
| db-cAMP (dibutyryl adénosine monophosphate cyclique) | Sigma Aldrich | D0627 | 500 μM |
| BDNF (facteur neutrotrophique dérivé du cerveau) | MACS Miltenyi | 130-093-811 | [10 ng/mL] |
| GDNF (facteur neurotrophique dérivé de la lignée cellulaire gliale) | MACS Miltenyi | 130-096-290 | [10 ng/mL] |
| TGF-β3 humain (facteur de croissance transformant-bêta3) | MACS Miltenyi | 130-094-007 | [1 ng/mL] |
| Milieu d’essai | Conc. final. | ||
| Seahorse XF DMEM moyen | Bioscience de l’hippocampe | 103680-100 | 500 mL |
| Sodium Pyruvate | Gibco | 11360070 | 1 mM |
| L-Glutamine | Lonza | BEBP17-605E | 2 mM |
| Glucose | Sigma Aldrich | 50-99-7 | 10 mM |
| Solution de blocage | Conc. final. | ||
| PBS-Interpolation | [1:1] 0,1 % Préadolescent | ||
| Sérum d’âne | Sigma Aldrich | D9663 | 10% |
| Triton-X | Merck | X100-5ML | 1% |
Tableau 1 : Détails des milieux et des solutions utilisés pour la génération d’organoïdes.
| Initialisation | Ligne de base (X3) | Injection d’oligomycine (X3) | Injection FCCP (X3) | Injection FCCP (X3) | Injection anti-A + pourriture (X3) |
| Calibrer | Mélanger | Mélanger | Mélanger | Mélanger | Mélanger |
| (04:00) | (04:00) | (04:00) | (04:00) | (04:00) | |
| Equilibrer (12:00) | Attendre | Attendre | Attendre | Attendre | Attendre |
| (02:00) | (02:00) | (02:00) | (02:00) | (02:00) | |
| Mesure (03:00) | Mesure (03:00) | Mesure (03:00) | Mesurer | Mesure (03:00) | |
| (03:00) |
Tableau 2 : Configuration du protocole pour le profilage bioénergétique. Description des étapes et de leur durée en minutes à l’aide du logiciel Seahorse Wave Desktop. Abréviations : FCCP = carbonyl cyanide-p-trifluoromethoxyphenylhydrazon; Pourriture = roténone; Anti A = Antimycine A.
Discussion
Cet article décrit la génération reproductible d’organoïdes cérébraux humains dérivés de l’iPSC et leur utilisation pour la modélisation des maladies mitochondriales. Le protocole décrit ici est modifié sur la base d’un travail précédemment publié20. Un avantage majeur du protocole actuel est qu’il ne nécessite pas l’intégration manuelle de chaque organoïde dans une matrice d’échafaudage. En fait, la solution matricielle est simplement dissoute dans le milieu de culture cellulaire. De plus, il n’est pas nécessaire d’utiliser des bioréacteurs coûteux, car les organoïdes peuvent être cultivés dans des plaques de culture tissulaire standard à 6 puits positionnées sur un agitateur orbital à l’intérieur de l’incubateur. Cette procédure permet également la culture parallèle de plusieurs plaques contenant différents organoïdes dérivés de différentes lignées individuelles, augmentant ainsi le débit des expériences et permettant le suivi des différences potentielles émergeant dans les profils de croissance de différents organoïdes. Nous avons testé ce protocole en utilisant différents CSPi dérivés de témoins sains et de personnes touchées par des maladies mitochondriales, avec des résultats cohérents.
Pour la modélisation des maladies mitochondriales, il est essentiel d’utiliser différents marqueurs pour visualiser la morphologie et l’organisation du réseau mitochondrial. Cette procédure permet d’étudier si le nombre, la morphologie ou la distribution mitochondriale peuvent être modifiés dans les organoïdes cérébraux dérivés de patients atteints de maladies mitochondriales. La présence et l’organisation de progéniteurs neuronaux dans les organoïdes cérébraux pourraient être d’une importance cruciale pour la modélisation des troubles mitochondriaux. Nous avons récemment découvert que les mutations à l’origine de la maladie mitochondriale, le syndrome de Leigh, perturbent l’architecture cellulaire et la distribution des cellules progénitrices neurales dans les organoïdes cérébraux dérivés du patient18.
Pour effectuer un profilage bioénergétique, nous avons adapté une méthode décrite précédemment pour évaluer la bioénergétique des cellules souches pluripotentes21. Un protocole récent décrivait comment effectuer un profilage bioénergétique d’organoïdes dérivés de l’intestin grêle de souris, du côlon humain et de tumeurs colorectales22. Cependant, ces organoïdes sont assez petits par rapport aux organoïdes cérébraux, et par conséquent, un protocole différent, tel que celui rapporté ici, est nécessaire pour les organoïdes cérébraux. Nous avons récemment utilisé ce protocole pour évaluer le profil bioénergétique des organoïdes cérébraux humains porteurs de mutations du gène surféite locus protein 1 (SURF1) qui cause la maladie mitochondriale grave, le syndrome de Leigh18. Nous avons constaté que le profil OCR est particulièrement affecté chez les organoïdes du syndrome de Leigh, comme le montre une diminution significative du taux d’OCR basal, du taux de production d’ATP et du taux de respiration maximal18.
En conclusion, nous présentons ici un protocole détaillé pour la génération robuste d’organoïdes cérébraux humains et décrivons comment effectuer des expériences qui seraient importantes pour l’étude des mécanismes pathologiques sous-jacents aux maladies mitochondriales. Les organoïdes cérébraux humains peuvent également être d’une importance cruciale pour élucider la diversité mitochondriale dans le cerveau humain et son rôle dans la santé et les maladies humaines23. Il est important de préciser que les organoïdes cérébraux générés avec les protocoles actuellement disponibles, y compris celui décrit ici, ont encore des limites. Il s’agit, par exemple, de l’absence de vascularisation et de l’absence de population de microglies24. Ces aspects doivent être pris en considération pour interpréter correctement les résultats.
Par exemple, l’absence de vascularisation et de microglie pourrait limiter les mécanismes compensatoires qui peuvent être en place in vivo. Les organoïdes cérébraux dérivés du patient pourraient donc présenter des défauts plus forts que ceux observés chez les patients17,18. De plus, malgré une reproductibilité générale de ce protocole20, une hétérogénéité ligne à ligne peut être observée. À cette fin, lors de la réalisation d’études de modélisation de la maladie, il est toujours important de quantifier systématiquement l’uniformité des organoïdes du contrôle et du patient en évaluant les modèles de morphologie (taille, couche) et la distribution des marqueurs moléculaires entre différents organoïdes.
Enfin, il n’est pas possible de générer des organoïdes cérébraux à partir d’une seule CSPi, ce qui limite la faisabilité d’un dépistage génétique à grande échelle avec CRISPR/Cas9. Compte tenu du rythme de la recherche, il est probable que certaines des limites actuelles du protocole décrit ici seront bientôt surmontées. Des protocoles optimisés seront disponibles. Ces modèles 3D de maladies mitochondriales permettront, espérons-le, la découverte éventuelle de thérapies réalisables pour les maladies mitochondriales, qui sont préjudiciables, et pour les maladies incurables avec des besoins médicaux hautement non satisfaits.
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt financier ou non financier concurrent.
Remerciements
Nous remercions Miriam Bünning pour son soutien technique. Nous reconnaissons le soutien de la Deutsche Forschungsgemeinschaft (DFG) (PR1527/5-1 à A.P.), de Spark et de l’Institut de la santé de Berlin (BIH) (FONDS DE VALIDATION DE LA BOSNIE-HERZÉGOVINE à A.P.), de la United Mitochondrial Disease Foundation (UMDF) (Leigh Syndrome International Consortium Grant to A.P.), de l’hôpital universitaire de Düsseldorf (Forschungskommission UKD à A.P.) et du ministère fédéral allemand de l’Éducation et de la Recherche (BMBF) (e: Subvention bio jeune chercheur AZ 031L0211 à A.P.). Les travaux en laboratoire du C.R.R. ont été soutenus par la DFG (FOR 2795 « Synapses sous stress », Ro 2327/13-1).
matériels
| Name | Company | Catalog Number | Comments |
| 2-mercaptoethanol | Gibco | 31350-010 | |
| Affinity Designer | Serif (Europe) Ltd | Layout software; Vector graphics editor | |
| Alexa Fluor 488 donkey anti-guinea pig | Sigma Aldrich | SAB4600033-250UL | 1:300 |
| Alexa Fluor 488 donkey anti-mouse | Thermo Fisher Scientific | A-31571 | 1:300 |
| Antimycin A | Sigma Aldrich | 1397-94-0 | |
| Anti-β-Tubulin III (TUJ-1) | Sigma Aldrich | T8578 | 1:2000 |
| Argon Laser | Melles Griot | Any other Laser, e.g., diode lasers emitting 488 is fine, too | |
| Ascorbic acid | Sigma | A92902 | |
| B-27 with Vitamin A | Gibco | 17504044 | |
| Bacto Agar | Becton Dickinson | 3% in PBS, store solution at -20 °C | |
| BDNF | Miltenyi Biotec | 130-093-0811 | |
| cAMP | Sigma | D0627 | |
| Cell Star cell culture 6 well plate | Greiner-Bio-One | 657160 | |
| Chemically Defined Lipid Concentrate | Gibco | 11905031 | |
| Confocal laser scanning microscope C1 | Nikon Microscope Solutions | Modular confocal microscope system | |
| Corning Matrigel Growth Factor Reduced (GFR) Basement membrane matrix, Phenol Red-free, LDEV-free | Corning | 356231 | Matrix component |
| CyQUANT Cell Proliferation Assay Kit | Thermo Fisher | C7026 | |
| DMEM/F12 | ThermoFisher | 31330038 | |
| DMSO | Sigma | D2660-100ML | |
| Donkey anti-goat Cy3 | Merck Millipore | AP180C | 1:300 |
| Donkey anti-mouse Cy3 | Merck Millipore | AP192C | 1:300 |
| Donkey anti-rabbit Cy3 | Merck Millipore | AP182C | 1:300 |
| DPBS | Gibco | 14190250 | |
| DS-Q1Mc camera | Nikon Microscope Solutions | ||
| Eclipse 90i upright widefield microscope | Nikon Microscope Solutions | ||
| Eclipse E 600FN upright microscope | Nikon Microscope Solutions | ||
| Eclipse Ts2 Inverted Microscope | Nikon Microsope Solutions | ||
| EZ-C1 Silver Version 3.91 | Nikon Microscope Solutions | Imaging software for confocal microscope | |
| FCCP | Sigma Aldrich | 370-86-5 | |
| Fetal Bovine Serum | Gibco | 10270-106 | |
| GDNF | Miltenyi Biotec | 130-096-291 | |
| Glasgow MEM | Gibco | 11710-035 | |
| Glass Pasteur pipette | Brand | 747715 | Inverted |
| Glutamax | Gibco | 35050-061 | |
| Helium-Neon Laser | Melles Griot | Every other Laser, e.g., diode lasers emitting 594 is fine, too | |
| Heparin | Merck | H3149-25KU | |
| HERACell 240i CO2 Incubator | Thermo Scientific | 51026331 | |
| Hoechst 33342 | Invitrogen | H3570 | 1:2500 |
| Image J 1.53c | Wayne Rasband National Institute of Health | Image processing Software | |
| Injekt Solo 10 mL/ Luer | Braun | 4606108V | |
| Knockout Serum Replacement | Gibco | 10828010 | |
| Laser (407 nm) | Coherent | Any other Laser, e.g., diode lasers emitting 407 is fine, too | |
| Map2 | Synaptic Systems | No. 188004 | 1:1000 |
| Maxisafe 2030i | |||
| MEM NEAA | Gibco | 11140-050 | |
| mTeSR Plus | Stemcell Technology | 85850 | iPSC medium |
| Multifuge X3R Centrifuge | Thermo Scientific | 10325804 | |
| MycoAlert Mycoplasma Detection Kit | Lonza | # LT07-218 | |
| N2 Supplement | Gibco | 17502-048 | |
| Needle for single usage (23G x 1” TW) | Neoject | 10016 | |
| NIS-Elements Aadvanced Research 3.2 | Nikon | Imaging software | |
| Oligomycin A | Sigma Aldrich | 75351 | |
| Orbital Shaker Heidolph Unimax 1010 | Heidolph | 543-12310-00 | |
| PAP Pen | Sigma | Z377821-1EA | To draw hydrophobic barrier on slides. |
| Papain Dissociation System kit | Worthington | LK003150 | |
| Paraformaldehyde | Merck | 818715 | 4% in PBS, store solution at -20 °C |
| Pasteur pipette 7mL | VWR | 612-1681 | Graduated up to 3 mL |
| Penicillin-Streptomycin | Gibco | 15140-122 | |
| Plan Apo VC 20x / 0.75 air DIC N2 ∞/0.17 WD 1.0 | Nikon Microscope Solutions | Dry Microscope Objective | |
| Plan Apo VC 60x / 1.40 oil DIC N2 ∞/0.17 WD 0.13 | Nikon Microscope Solutions | Oil Immersion Microscope Objective | |
| Polystyrene Petri dish (100 mm) | Greiner Bio-One | 664161 | |
| Polystyrene round-bottom tube with cell-strainer cap (5 mL) | Falcon | 352235 | |
| Potassium chloride | Roth | 6781.1 | |
| ProLong Glass Antifade Moutant | Invitrogen | P36980 | |
| Qualitative filter paper | VWR | 516-0813 | |
| Rock Inhibitior | Merck | SCM075 | |
| Rotenone | Sigma | 83-794 | |
| S100β | Abcam | Ab11178 | 1:600 |
| SB-431542 | Cayman Chemical Company | 13031 | |
| Scalpel blades | Heinz Herenz Hamburq | 1110918 | |
| SMI312 | Biolegend | 837904 | 1:500 |
| Sodium bicarbonate | Merck/Sigma | 31437-1kg-M | |
| Sodium chloride | Roth | 3957 | |
| Sodium dihydrogen phosphate | Applichem | 131965 | |
| Sodium Pyruvate | Gibco | 11360070 | |
| SOX2 | Santa Cruz Biotechnology | Sc-17320 | 1:100 |
| StemPro Accutase Cell Dissociation Reagent | Gibco/StemPro | A1110501 | Reagent A |
| Super Glue Gel | UHU | 63261 | adhesive gel |
| SuperFrost Plus | VWR | 631-0108 | |
| Syringe for single usage (1 mL) | BD Plastipak | 300015 | |
| TB2 Thermoblock | Biometra | ||
| TC Plate 24 Well | Sarstedt | 83.3922 | |
| TC Plate 6 Well | Sarstedt | 83.392 | |
| TGFbeta3 | Miltenyi Biotec | 130-094-007 | |
| Tissue Culture Hood | ThermoFisher | 51032711 | |
| TOM20 | Santa Cruz Biotechnology | SC-11415 | 1:200 |
| Triton-X | Merck | X100-5ML | |
| UltraPure 0.5M EDTA | Invitrogen | 15575020 | |
| Vibratome Microm HM 650 V | Thermo Scientific | Production terminated, any other adjustable microtome is fine, too. | |
| Vibratome Wilkinson Classic Razor Blade | Wilkinson Sword | 70517470 | |
| Whatman Benchkote | Merck/Sigma | 28418852 | |
| Wnt Antagonist I | EMD Millipore Corp | 3378738 | |
| XF 96 extracellular flux analyser | Seahorse Bioscience | 100737-101 | |
| XF Assay DMEM Medium | Seahorse Bioscience | 103680-100 | |
| XF Calibrant Solution | Seahorse Bioscience | 100840-000 | |
| XFe96 FluxPak (96-well microplate) | Seahorse Bioscience | 102416-100 |
Références
- Koopman, W. J., Willems, P. H., Smeitink, J. A. Monogenic mitochondrial disorders. New England Journal of Medicine. 366 (12), 1132-1141 (2012).
- Gorman, G. S., et al. Mitochondrial diseases. Nature Review Disease Primers. 2, 16080 (2016).
- Vafai, S. B., Mootha, V. K. Mitochondrial disorders as windows into an ancient organelle. Nature. 491 (7424), 374-383 (2012).
- Carelli, V., Chan, D. C. Mitochondrial DNA: impacting central and peripheral nervous systems. Neuron. 84 (6), 1126-1142 (2014).
- Russell, O. M., Gorman, G. S., Lightowlers, R. N., Turnbull, D. M. Mitochondrial diseases: hope for the future. Cell. 181 (1), 168-188 (2020).
- Weissig, V. Drug development for the therapy of mitochondrial diseases. Trends in Molecular Medicine. 26 (1), 40-57 (2020).
- Tyynismaa, H., Suomalainen, A. Mouse models of mitochondrial DNA defects and their relevance for human disease. EMBO Reports. 10 (2), 137-143 (2009).
- Ma, H., et al. Metabolic rescue in pluripotent cells from patients with mtDNA disease. Nature. 524 (7564), 234-238 (2015).
- Galera-Monge, T., et al. Mitochondrial dysfunction and calcium dysregulation in Leigh syndrome induced pluripotent stem cell derived neurons. International Journal of Molecular Science. 21 (9), 3191 (2020).
- Zheng, X., et al. Alleviation of neuronal energy deficiency by mTOR inhibition as a treatment for mitochondria-related neurodegeneration. Elife. 5, 13378 (2016).
- Lorenz, C., et al. Human iPSC-derived neural progenitors are an effective drug discovery model for neurological mtDNA disorders. Cell Stem Cell. 20 (5), 659-674 (2017).
- Inak, G., et al. Concise review: induced pluripotent stem cell-based drug discovery for mitochondrial disease. Stem Cells. 35 (7), 1655-1662 (2017).
- Chiaradia, I., Lancaster, M. A. Brain organoids for the study of human neurobiology at the interface of in vitro and in vivo. Nature Neuroscience. 23 (12), 1496-1508 (2020).
- Lancaster, M. A., Knoblich, J. A. Generation of cerebral organoids from human pluripotent stem cells. Nature Protocol. 9 (10), 2329-2340 (2014).
- Liput, M., et al. Tools and approaches for analyzing the role of mitochondria in health, development and disease using human cerebral organoids. Developmental Neurobiology. , (2021).
- Winanto, K. Z. J., Soh, B. S., Fan, Y., Ng, S. Y. Organoid cultures of MELAS neural cells reveal hyperactive Notch signaling that impacts neurodevelopment. Cell Death and Disease. 11 (3), 182 (2020).
- Romero-Morales, A. I., et al. Human iPSC-derived cerebral organoids model features of Leigh Syndrome and reveal abnormal corticogenesis. bioRxiv. , (2020).
- Inak, G., et al. Defective metabolic programming impairs early neuronal morphogenesis in neural cultures and an organoid model of Leigh syndrome. Nature Communications. 12 (1), 1929 (2021).
- Falk, M. J. Neurodevelopmental manifestations of mitochondrial disease. Journal of Developmental & Behavioral Pediatrics. 31 (7), 610-621 (2010).
- Velasco, S., et al. Individual brain organoids reproducibly form cell diversity of the human cerebral cortex. Nature. 570 (7762), 523-527 (2019).
- Pfiffer, V., Prigione, A. Assessing the bioenergetic profile of human pluripotent stem cells. Methods in Molecular Biology. 1264, 279-288 (2015).
- Ludikhuize, M. C., Meerlo, M., Burgering, B. M. T., Colman, R. M. J. Protocol to profile the bioenergetics of organoids using Seahorse. STAR Protocols. 2 (1), 100386 (2021).
- Menacho, C., Prigione, A. Tackling mitochondrial diversity in brain function: from animal models to human brain organoids. International Journal of Biochemestry & Cell Biology. 123, 105760 (2020).
- Del Dosso, A., Urenda, J. P., Nguyen, T., Quadrato, G. Upgrading the physiological relevance of human brain organoids. Neuron. 107 (6), 1014-1028 (2020).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon