Method Article
Istituzione di Cancer Stem colture cellulari da umana convenzionale osteosarcoma
In questo articolo
Riepilogo
La presenza di cellule staminali tumorali (CSC) in sarcomi ossei è stato recentemente collegato a loro patogenesi. In questo articolo, vi presentiamo l'isolamento di CSC da colture cellulari primarie ottenute da biopsie umane di osteosarcoma convenzionale (OS) con la capacità di CSC di crescere in condizioni non aderenti.
Abstract
Gli attuali miglioramenti nella terapia contro osteosarcoma (OS) hanno prolungato la vita dei pazienti affetti da cancro, ma il tasso di sopravvivenza di cinque anni rimane scarsa quando si è verificato metastasi. L'(CSC) teoria Cancer Stem Cell ritiene che ci sia un sottogruppo di cellule tumorali all'interno del tumore che hanno caratteristiche staminali-simili, compresa la capacità di mantenere il tumore e di resistere chemioterapia multifarmaco. Pertanto, una migliore comprensione della biologia del sistema operativo e la patogenesi è necessaria al fine di promuovere lo sviluppo di terapie mirate per sradicare questo particolare sottoinsieme e per ridurre la morbilità e la mortalità tra i pazienti. Isolando CSC, che stabilisce colture cellulari di CSC, e studiare la loro biologia sono importanti passi per migliorare la nostra comprensione della biologia del sistema operativo e la patogenesi. La creazione di OS-CSC di derivazione umana da biopsie di OS è stato reso possibile utilizzando diversi metodi, tra cui la capacità di creare colture di cellule staminali 3-dimensionale sotto nonadherecondizioni NT. In queste condizioni, CSC possono creare colonie galleggianti sferici formate da cellule staminali derivate; queste colonie sono denominate "sfere cellulari". Qui, si descrive un metodo per stabilire le culture CSC da colture cellulari primarie di OS convenzionale ottenuti da biopsie del sistema operativo. Descriviamo chiaramente i vari passaggi necessari per isolare e caratterizzare CSC.
Introduzione
I sarcomi sono un gruppo eterogeneo di tumori rari del tessuto connettivo maligni provenienti prevalentemente dal mesoderma embrionale 1. I diversi tipi includono sarcomi ossei e sarcomi dei tessuti molli. sarcomi ossei, un gruppo di tumori primari relativamente poco diffusa, costituiti da diversi sottotipi, tra osteosarcoma (OS). OS, uno dei tumori più comuni primari dell'osso, è un tumore maligno mesenchimali che esibisce vasta clinica, istologica e eterogeneità molecolare 2, 3. Sfortunatamente, OS verifica prevalentemente nei bambini e nei giovani adulti 4, 5 e rappresenta il 60% i sottotipi istologici comuni di sarcoma osseo nell'infanzia 6, 7. OS colpisce di solito le aree scheletrici, che sono caratterizzati da una crescita ossea rapida (ad esempio, la metafisi delle ossa lunghe). Tra i istologicamente differenti sottotipi di OS, OS convenzionale, chiamato anche midollare o OS centrale, ha un alto grado di malignità e sha contingentere del 80% 8. Questo 80% è composto da 60% OS convenzionale osteoblastica, 10% OS chondroblastic, e il 10% OS fibroblastico 6, 8-10. Altri sottotipi OS includono anaplastico, telangectasico, gigante ricco di cellule e piccoli OS cellule. Nonostante i progressi della chirurgia e chemioterapia combinata nella gestione del sistema operativo, il risultato rimane scarsa, con un tasso di sopravvivenza a lungo termine del 65-70% nei pazienti senza metastasi 11, 12. Ricorrenze lontane si verificano di frequente come metastasi polmonari o, meno frequentemente, come le metastasi alle ossa lontane e recidive locali 13. Le metastasi sono spesso resistenti ai trattamenti convenzionali. Questa resistenza è la ragione per cui la sopravvivenza libera da malattia a 10 anni è di circa il 30% nei pazienti con malattia metastatica alla diagnosi 14, 15.
Come con tessuto normale, tessuto del cancro è composto da un insieme eterogeneo di tipi cellulari. Le cellule all'interno del tumore sembrano corrispondere a diversi stadi di sviluppo. All'interno di ogni ntessuto ormal risiede una sottopopolazione di cellule con la capacità di selfrenew, fornendo così progenitori e cellule mature per omeostasi tissutale. Analogamente, il cancro è composto di una popolazione eterogenea di cellule simili a diversi stadi di sviluppo, con differenti gradi di proliferazione e potenziale oncogeno. Un sottoinsieme di queste cellule tumorali, definito cellule staminali tumorali (CSC), costituisce una riserva di Portante in cellule con la capacità esclusiva di selfrenew e mantenere il potenziale maligno dei tumori, generando così le diverse linee cellulari che costituiscono la massa tumorale 16. Nel 1990, gli studi sulla leucemia mieloide acuta fornito la prima prova convincente per l'esistenza di CSC sottopopolazioni 17, 18. CSC da allora sono stati isolati da un gran numero di tumori solidi 19, diventando così uno degli argomenti più studiati nella ricerca sul cancro. CSC può infatti derivare da cellule staminali normali da mutazioni nei geni che rendono il normalele cellule staminali cancerose 20-23. Mutazioni multiple trasformando e le interazioni con il microambiente potrebbe anche contribuire a progenitori sane e cellule mature che acquisiscono la capacità selfrenewal e l'immortalità che caratterizzano CSC. Ci sono diverse ipotesi su questa trasformazione. Progenitori sane, le cellule mature sane e cellule tumorali, possono dedifferentiate alle cellule staminali, ottenendo un fenotipo staminali come attivando geni selfrenewal-associata 24-28. Nonostante i numerosi studi recenti, le origini del CSC sono ancora da scoprire.
Una caratteristica particolare del CSC è che la loro capacità di resistere alla approccio multi-terapia, che consiste di chirurgia e chemioterapia combinata con diversi farmaci. Recenti studi hanno dimostrato che la CSC può anche acquisire resistenza agli agenti chemioterapici citotossici. Le possibili spiegazioni per questa resistenza sono la sovraespressione di ATP-binding cassette (ABC) multidrug transporter (vale a dire,MDR1 e BCRP1), sovraespressione di chemioterapia metabolizzare enzimi come l'aldeide deidrogenasi 1 (ALDH1), e / o cambiamenti nella cinetica del ciclo cellulare 30-33. La conseguenza diretta di tutti questi concetti che sono stati descritti finora è che una terapia del cancro sarebbe efficace solo se la sottopopolazione CSC sono stati completamente eliminato, mentre recidiva locale o metastasi a distanza potrebbero verificarsi se anche un solo CSC sopravvissuto.
La scoperta di CSC nei sarcomi umani 34, in particolare OS 35, o in qualsiasi altro tumori ossei e dei tessuti molli, ha una grande importanza clinica in quanto offre una possibile spiegazione del perché molti trattamenti sembrano essere efficaci inizialmente, ma i pazienti in seguito ricaduta. Pertanto, la speranza per il futuro battaglia contro OS convenzionale è quello di trovare nuove e specifiche terapie mirate basata sullo sviluppo di farmaci innovativi diretti a OS-CSC grazie alla caratterizzazione molecolare di questo subpopolazione e allo studio della biologia CSC.
Nel 1992, Reynolds e colleghi, che stavano indagando se un sottogruppo di cellule staminali era presente nel cervello dei mammiferi adulti, sviluppato un metodo per isolare le cellule sospettate di essere cellule 36, 37 stem-like. Questo metodo si basa sulla capacità particolare di queste cellule a formare colonie sferiche se coltivate in condizioni non aderenti. Tecniche simili sono state impiegate da Gibbs e colleghi nel 2005 per studiare una sottopopolazione di cellule staminali simil-in sarcomi dell'osso 38. Per isolare e caratterizzare OS-CSC da colture cellulari primarie di diversi tipi di sistema operativo convenzionale, abbiamo deciso di adattare questa tecnica per linee di cellule del sistema operativo.
Qui, descriviamo questo metodo adattato del dosaggio formazione ambito, denominato "assay sarcosphere", che può essere utilizzato per isolare OS-CSC da linee cellulari primarie derivate finite da biopsie umane di OS convenzionale. Noi descriviamo anche tutte le tecniche used per convalidare la radice come CSC fenotipo delle linee cellulari isolate da questo test: 1) valutazione dell'espressione dei geni che caratterizzano pluripotenti cellule staminali embrionali (ESC) e del gene CD133, che è un marker di CSC; 2) (CFU) saggio formanti colonia unità; 3) valutazione della capacità di queste cellule di differenziarsi in osteoblasti e adipociti sotto opportune condizioni di differenziazione; 4) studio dei marcatori di superficie delle cellule staminali mesenchimali (MSC) (vale a dire, CD44, CD105 e Stro-1) per immunofluorescenza e mediante analisi di citometria di flusso; 5) valutazione dell'attività ALDH di queste cellule.
Protocollo
Tutti sperimentazione utilizzando tessuti umani qui descritti è stato approvato dal comitato etico locale (Rif. N. 141/12). Il consenso informato per la raccolta di campioni di tessuto e per l'utilizzo e la conservazione dei campioni è stato ottenuto da donatori a AOUC.
1. Preparazione per la Cultura
- Preparare terreno di coltura di crescita (GM) con l'aggiunta del 10% fetale siero bovino (FBS), 100 UI / ml di penicillina e 100 mg / ml di streptomicina per mezzo di F12 di Ham modificata di Coon. Filtrare e sterilizzare GM usando un filtro di 0,22 micron. Conservare GM fino a 1 mese a 4 ° C.
- Preparare media collagenasi (CM) aggiungendo 20% FBS, 100 IU / ml di penicillina, 100 ug / ml di streptomicina e 3 mg / ml di collagenasi di tipo II e medio F12 di Ham modificata di Coon. Filtrare e sterilizzare CM usando un filtro di 0,22 micron. Conservare CM fino a 1 mese a -20 ° C.
- Preparare mezzo per la cella di congelamento (FM) aggiungendo 40% FBS, 100 IU / ml di penicillina, 100 ug / ml di streptomicina e 6.5% Dimetilsolfossido (DMSO) per mezzo di F12 di Ham modificata di Coon. Filtro e sterilizzare FM utilizzando un filtro di 0,22 micron. Conservare FM fino a 1 mese a 4 ° C.
- Preparare mezzo per CFU assay (CFUM) con l'aggiunta del 20% FBS, 100 UI / ml di penicillina e 100 mg / ml di streptomicina per mezzo di F12 di Ham modificata di Coon. Filtro e sterilizzare CFUM utilizzando un filtro di 0,22 micron. Conservare CFUM fino a 1 mese a 4 ° C.
- Preparare tripsina sciogliendo 400 mg tripsina, 200 mg EDTA e 1.000 mg D (+) - glucosio anidro a 1.000 ml di Dulbecco Phosphate-Buffered Saline (DPBS) senza Ca 2+ e Mg 2+.
NOTA: Dividere fresco tripsina-EDTA in 50 ml aliquote nel 25 cm 2 palloni utilizzando un filtro 0,22 micron e congelare a -20 ° C per un massimo di 3 mesi. Conservare scongelato aliquote sterilizzate per filtrazione a 4 ° C senza perdita di attività. - Preparare il terreno di coltura di cellule staminali (SCGM) con l'aggiunta del 10% FBS, 100 UI / ml di penicillina, 100 ug / ml streptomycin, e 10 ng / ml bFGF (25 mg / ml di magazzino) per mezzo di F12 di Ham modificata di Coon. Filtrare SCGM utilizzando un filtro di 0,22 micron e conservare SCGM fino a 2 settimane a 4 ° C.
- Preparare 2% metilcellulosa (MC) sciogliendo MC in ultrapura H 2 O a 4 ° C per 3 d. Quando MC è completamente sciolto, autoclave e conservare a 4 ° C.
NOTA: Dopo MC è sterilizzato, diventa solido. Per portare MC allo stato liquido, conservare a 4 ° C. - Preparare mezzo di crescita sarcosphere (SGM). Preparare questa nuova media (non più di 2 settimane prima dell'uso) con l'aggiunta di 100 UI / ml di penicillina, 100 ug / ml umana bFGF (25 mg / ml di magazzino), 20 nM progesterone (10 micron magazzino), 100 micron putrescina, 30 nm selenite di sodio (30 micron magazzino), 25 mg / ml di transferrina (25 mg / ml di magazzino), 20 mg / insulina ml (20 mg / ml di magazzino), e 10 ng / mL EGF umano (10 mg / ml magazzino) a 2X Coon di modifica medio F12 di Ham. Filtrare e sterilizzare SGM con un fil 0,22 micronter. Memorizzare fino a 2 settimane a 4 ° C.
- Preparare medio osteogenico (OM) con l'aggiunta di 10% FBS (origine sudamericana), 100 UI / ml di penicillina, 100 ug / ml di streptomicina, 10 nM desametasone (100 micron magazzino), 0.2 mM di sodio L-ascorbile-2-fosfato (1 m stock), 10 mM β-glicerofosfato (5 mg / ml di magazzino) e 1 mg / ml calceina (200 mcg / magazzino ml) per mezzo di F12 di Ham modificata di Coon. Filtro e sterilizzare OM con un filtro 0,22 micron. Conservare a 4 ° C.
NOTA: Le soluzioni desametasone azionari Conservare sotto azoto liquido per mantenere la loro attività. Preparare OM fresco ogni 2 settimane per mantenere l'attività del desametasone per mantenere il potenziale di differenziazione del mezzo. - Preparare tampone di lisi degli eritrociti (ELB) sciogliendo 1,66 mg NH 4 Cl, 0,2 mg K 2 HPO 4 e 0.007 mg di EDTA in 200 ml di acqua distillata (DH 2 O). Filtrare e sterilizzare ELB usando un filtro di 0,22 micron. Conservare a 4 °C.
- Preparare medio adipogenico (AM) con l'aggiunta di 10% FBS (Sud America origine), 100 UI / ml di penicillina, 100 ug / ml di streptomicina, 1 mM dexametasone (1 mM magazzino), 1 mM bovina insulina (10 mM magazzino), 0.5 mM isobutylmethylxanthine (IBMX) (500 mm magazzino), e 100 micron indometacina (200 mm) per mezzo di F12 di Ham modificata di Coon. Filtrare e sterilizzare sto usando un filtro di 0,22 micron. Conservare a 4 ° C.
NOTA: Le soluzioni desametasone azionari Conservare sotto azoto liquido per mantenere la loro attività. Preparare AM fresco ogni 2 settimane per mantenere l'attività del desametasone per mantenere il potenziale di differenziazione del mezzo. - Preparare 2% di sieroalbumina bovina (BSA) in DPBS (BSA / DPBS). Sciogliere 10 g di BSA in 500 ml DPBS e preparare la soluzione di riserva da aliquotting 50 ml di soluzione in 50 ml provette coniche. Conservare a -20 ° C.
- Preparare una soluzione di paraformaldeide al 4% (PFA) in DPBS (PFA / DPBS). In un chCappuccio emical, diluire paraformaldeide in DPBS e preparare la soluzione di riserva da aliquotting 50 ml di soluzione in 50 ml provette coniche. Conservare a 4 ° C.
2. Stabilire colture cellulari primarie OS e OS linee cellulari finiti (OSA)
NOTA: colture di cellule del sistema operativo primario sono stati preparati da campioni freschi di biopsie OS convenzionali raccolti presso la "Unità Ortopedia Oncologica e Ricostruttiva", AOUC Careggi, Firenze. Tutte le biopsie, che sono stati ottenuti mediante aspirazione ago o la resezione chirurgica di una piccola porzione del tumore (Figura 1A, B), sono stati immediatamente posti in terreno di coltura addizionato con 100 IU / ml di penicillina e 100 ug / ml di streptomicina (pH 7,4) e trasportati al laboratorio dove sono stati elaborati. Tutte le operazioni descritte sono state condotte in condizioni asettiche utilizzando una cappa a flusso laminare.
- L'isolamento delle cellule del sistema operativo
- Posizionare la biopsia in un100 millimetri Petri con un piccolo volume di GM. Con una lancetta sterile e pinzette Perry, tritare i campioni di tessuto OS tagliandoli a pezzi il più piccolo possibile (0,5-1 mm) (Figura 2A, B).
- Coprire i frammenti di tessuto con 10 ml CM per la digestione enzimatica (Figura 2B) e incubare per 3 ore a 37 ° C, 5% CO 2 incubatore. Rimuovere delicatamente la sospensione di frammenti con una pipetta e trasferirlo in una provetta conica da 15 ml per centrifugazione immediata (400 xg per 5 min) a sedimentare frammenti.
NOTA: Dopo centrifugazione, se le biopsie sono ricchi di eritrociti, un deposito rossa composto di eritrociti può essere visto sopra i frammenti. Pertanto, prima di procedere alla dispersione meccanica, trattare il campione con il tampone di lisi degli eritrociti. Eliminare il surnatante tramite aspirazione e aggiungere 5 ml ELB. - Sospendere frammenti pellet per 1 minuto e centrifugare la sospensione a 400 xg per 2min. Rimuovere il surnatante tramite aspirazione. Aggiungere 5 ml di GM e meccanicamente disperdere i frammenti con una pipetta 10 ml sierologica (formato di apertura, a 1,5 mm Ø interno) 10 - 20 volte.
- Centrifugare la sospensione a 400 xg per 5 min. Rimuovere il surnatante tramite aspirazione, sospendere il pellet cellulare con 10 ml GM, e poi trasferire la sospensione cellulare risultante in da 100 mm piastra di Petri. Incubare la piastra di Petri a 37 ° C, 5% CO 2 incubatore e sostituire il GM fresca completa GM ogni 3 d.
NOTA: Molto spesso, i campioni di tessuto del sistema operativo ottenuti con agoaspirato sono molto piccole e contiene frammenti di ossa, che non vengono digeriti dalla digestione enzimatica con collagenasi. Pertanto, dopo il surnatante è stato rimosso, tentare di separare le cellule dal tessuto osseo schiacciando frammenti pellet con una pipetta.
- Subculture
NOTA: Per stabilire una OSA, quando le cellule raggiungono confluenza approssimativa, rimuoverli dalla theiR recipiente di coltura, diluire, e mettere in un piatto fresco per consentire un'ulteriore crescita. Questa procedura sottocultura si ottiene utilizzando la procedura enzimatica definito tripsinizzazione.- Rimuovere il supporto per aspirazione. Per dissociare il monostrato cellulare, aggiungere 2 - 3 ml di tripsina a temperatura ambiente (max 18 - 20 ° C), agitare delicatamente una volta, e rimuovere immediatamente la tripsina per aspirazione. Ripetere due volte. Incubare le cellule a 37 ° C per 3 - 4 minuti fino a quando iniziano a staccarsi.
NOTA: Buona tripsinizzazione può essere visto dall'utente esperto come piccoli fori che formano nel monostrato leggermente opaco quando il piatto si svolge alla luce. Questa dissociazione può essere monitorata utilizzando un microscopio invertito; questo approccio è raccomandato per principianti. - Arrestare la reazione tripsina con l'aggiunta di 10 ml di GM e lavare bene il piatto con una pipetta di staccare tutte le cellule. Trasferire 1 ml della sospensione ad un nuovo 100 millimetri piastra di Petri e aggiungere 9 ml di GM (1/10 scissione delle cellule).
NOTA: La sospensione cellulare rimanente può essere utilizzata per l'espansione delle cellule (trasferimento ad un altro piatto 100 millimetri Petri), possono essere crioconservati (vedi paragrafo 2.3), o può essere contato e placcato in piatti sperimentali o pozzetti per saggi di proliferazione o altri scopi (vedi sotto).
- Rimuovere il supporto per aspirazione. Per dissociare il monostrato cellulare, aggiungere 2 - 3 ml di tripsina a temperatura ambiente (max 18 - 20 ° C), agitare delicatamente una volta, e rimuovere immediatamente la tripsina per aspirazione. Ripetere due volte. Incubare le cellule a 37 ° C per 3 - 4 minuti fino a quando iniziano a staccarsi.
- Crioconservazione di colture primarie del sistema operativo e OSA
NOTA: Congelare un confluente 100 millimetri capsula di Petri in 4-5 cryovials. Il processo di crioconservazione deve essere eseguita rapidamente perché DMSO, che protegge le membrane cellulari durante il congelamento, è tossico per le cellule a temperature non congelamento.- Dissociano colture cellulari dal monostrato da tripsinizzazione (vedi Sezione 2.2), le cellule pellet per centrifugazione a 400 xg per 5 minuti, rimuovere il surnatante tramite aspirazione, e quindi sospendere rapidamente il pellet cellulare in FM.
- Aliquotare 1 ml di questa sospensione per flaconcino criogenico. Porre immediatamente le fiale piene in un congelatore orizzontale con una giacca di 2-propanolo e conservare immediatamente a -80 ° CO / N. Trasferimento cryovials di azoto liquido il giorno seguente per la conservazione a lungo termine.
- Linee cellulari di sbrinamento del sistema operativo e colture primarie
- linee cellulari di sbrinamento del sistema operativo da stoccaggio azoto liquido, cryovials scongelamento rapidamente mettendoli in un bagno d'acqua di laboratorio a 37 ° C. Trasferire il contenuto dei cryovials in un tubo da 15 ml e aggiungere circa 10 ml di GM (vedere la sezione 2.3 per rimuovere DMSO). Rimuovere il surnatante tramite aspirazione e di sospendere il pellet cellulare in 10 ml di GM. Piatto sospensione di cellule in un piatto 100 millimetri Petri e incubare a 37 ° C, 5% CO 2 incubatore.
3. Sarcosphere Assay per isolare OS-CSC
NOTA: Questo esperimento viene eseguito su OSA. La durata di questo esperimento è legata alla capacità delle cellule di formare queste colonie sfera (sarcospheres), e l'intervallo di tempo è 7, 14, 21, e 28 d.
- Estabdell'organico di test sarcosphere
- Preparare la camera emocitometro e tutti i reagenti di anticipo.
- Rimuovere il supporto per aspirazione e dissociare le cellule dal monostrato da tripsinizzazione (vedi Sezione 2.2). Mescolare la sospensione cellulare a fondo, pipettaggio per disperdere eventuali grumi, e raccogliere 10 microlitri utilizzando un puntale 20 l.
- Trasferire la sospensione cellulare 10 microlitri subito al bordo della camera dell'emocitometro, espellere la sospensione, e permettono di trarre sotto il vetrino per capillarità.
- Osservare la camera dell'emocitometro in contrasto di fase. Selezionare un obiettivo 10X e mettere a fuoco le linee della griglia nella camera. Contare le cellule che si trovano in questa zona 1 millimetro 2 utilizzando le suddivisioni (legato anche da tre linee parallele) e linee griglia singoli come un aiuto per il conteggio.
- Misurare la concentrazione e applicare la seguente equazione: 1 ml = n * 10 4 / z (n: numero intero di cellule in tutta la contatipiazze 1 mm 2; z: il numero di caselle contati 1 mm 2). Calcolare il volume della sospensione cellulare necessari alla piastra un totale di 240.000 cellule suddiviso in 40.000 cellule per ciascun pozzetto nella piastra da 6 pozzetti fissaggio ultra-bassa.
NOTA: Assicurarsi di placcare il numero corretto di celle placcatura cellule in un pozzo in più. Pertanto, la quantità totale di cellule da placcare è 280.000 cellule. - Preparare 35 ml SGM aggiungendo 50% del 2% MC (SGM-MC) in un pallone a 2 25 cm. Preparare il volume totale di SGM considerando 5 ml in più per placcare un pozzetto aggiuntivo.
- Aggiungere il volume calcolato di sospensione cellulare al SGM-MC preparata nel pallone e mescolare delicatamente con una pipetta per disperdere eventuali grumi. Piastra le cellule in un 6-pozzetti attacco ultra-bassa e utilizzare un microscopio invertito per osservare come appaiono le cellule dopo essere stato placcato.
- Incubare le cellule a 37 ° C, 5% CO 2 incubatore. Ogni 3 d, aggiunge fraliquote ESH di bFGF e EGF a ciascun pozzetto per rinfrescare la concentrazione dei fattori di crescita. Aggiungere 2 microlitri bFGF e 5 microlitri EGF a mantenere sia alla concentrazione finale di 10 ng / ml in ciascun pozzetto.
- Isolamento di sarcospheres
NOTA: Monitorare il buon andamento del dosaggio sarcosphere a 7, 14, 21, e 28 d per decidere quando è necessario isolare i sarcospheres che formavano.- Preparare tutti i reagenti e attrezzature nel cappa a flusso laminare (Figura 3A, B). Assemblare l'unità di filtrazione disponendo un filtro di rete del diametro di 25 mm e una maglia 40 micron in un supporto filtro a membrana; sterilizzare in autoclave (Figura 4A-E).
- Per ciascun pozzetto, trasferire il terreno contenente le sarcospheres ad una siringa con un supporto filtro a membrana sterile utilizzando una pipetta sterile con una punta 1.000 microlitri. Dopo aver rimosso completamente il terreno contenente le sarcospheres, lavare il pozzo da Adding 5 ml di GM per essere sicuri di trasferire tutte le sfere ad una siringa. Usate il microscopio per verificare se tutti i sarcospheres sono stati recuperati. In caso contrario, procedere ripetendo questo passaggio.
- Filtrare la sospensione con un supporto filtro a membrana solo per gravità senza pressione per evitare di danneggiare le sfere ed evitare aventi sarcospheres attraversano attraverso il filtro, evitando così la perdita di sfere. Rimuovere le bolle d'aria da delicatamente con una pipetta di vetro sterile Pasteur.
NOTA: Talvolta la filtrazione è bloccato dalla presenza di una bolla d'aria nel portafiltro membrana. - Lavare l'unità di filtrazione aggiungendo 10 ml GM alla siringa e lasciandolo filtro senza pressione per essere sicuri di eliminare le singole celle. Smontare l'unità filtro dalla siringa e metterlo in una capsula di Petri.
- Rimuovere il filtro a rete dal supporto filtro a membrana con una pinzetta Perry e lavarlo scuotendolo delicatamente con le pinzette in 60 millimetriCapsula di Petri per liberare i sarcospheres dal groviglio della membrana. Quindi, posizionare la membrana in un pozzo e confermare mediante osservazione microscopica che non ci sono più sfere aggrovigliati nella membrana. Se le sfere sono ancora aggrovigliati nella membrana, procedere con un altro lavaggio come descritto al punto 3.2.4.
- Osservare le sarcospheres rilasciati utilizzando un microscopio. Incubare le sarcospheres rilasciate in un 37 ° C, 5% CO 2 incubatore. Ripetere tutti i passaggi per ogni pozzetto di un 6-pozzetti attacco ultra-bassa.
4. OS-CSC Lines
NOTA: OS-CSC si ottengono dalle sarcospheres che mostrano l'espansione aderente con la reintroduzione e reculturing queste cellule in un monostrato dopo che sono placcati in piccoli 60 millimetri di Petri non è più in condizioni di fissaggio ultra-bassi.
- Culture OS-CSC
- Per consentire la crescita, le cellule sottocultura quando raggiungono circa il 90% di confluenza in un 60piatto mm Petri. Ripetere il passaggio 2.2.1 per stabilire culture OS-CSC.
- Smettere tripsinizzazione con l'aggiunta di 4 ml SCGM e lavare bene il piatto, con una pipetta di staccare tutte le cellule. Trasferire la sospensione cellulare ad un nuovo 100 millimetri piastra di Petri e aggiungere 6 ml SCGM. Quando OS-CSC raggiungere il 90% di confluenza, sottocultura ripetendo sezione 2.2.
NOTA: Per l'analisi in vitro per caratterizzare il fenotipo di cellule staminali-come isolato OS-CSC, le cellule possono essere placcati in diversi tipi di piastre. - Conserva le linee stabilite OS-CSC di crioconservazione (ripetere tutti i passi della sezione 2.3). Sbrinare il crioconservati OS-CSC ripetendo tutte le fasi del paragrafo 2.4.
5. In vitro A nalisi di caratterizzare OS-CSC:
- Preparazione delle cellule per immunofluorescenza
NOTA: Le cellule vengono fissate in paraformaldeide quando raggiungono il giusto grado di confluenza in ciascun pozzetto di una 24 pozzetti plaTE. Il grado di confluenza è correlato al tipo di esperimenti.- Piastra OS-CSC in un 24-pozzetti per studiare le cellule staminali (MSC) marcatori di superficie mesenchimali mediante immunofluorescenza. Fissare le cellule in ciascun pozzetto quando raggiungono 50 - 60% di confluenza. Rimuovere il SCGM mediante aspirazione e lavare due volte in un DPBS 24-pozzetti.
- Sotto una cappa chimica, aggiungere 500 microlitri 4% PFA / DPBS in ciascun pozzetto. Incubare a temperatura ambiente per 10 min. Rimuovere la PFA / DPBS e lavare 3x con ultrapura dH 2 O. Lasciare che il 24-pozzetti per asciugare nella cappa.
- Immunofluorescenza per i marcatori MSC
NOTA: Immunofluorescenza del OS-CSC fissato nel 4% PFA / DPBS può essere utilizzato per indagare il fenotipo MSC-come OS-CSC utilizzando anticorpi diretti contro CD44, Stro-1 e CD105. Il seguente metodo è utilizzato abitualmente dagli autori.- In una cappa chimica, permeabilize cellule che sono state fissate in 4% PFA / DPBS con l'aggiunta di 500 microlitri 0.2% Triton X-100 / DPBS in ciascun pozzetto. Incubare le cellule a 37 ° C per 30 min. lavare delicatamente le cellule 3x con DPBS. Aggiungere 300 microlitri RNasi diluito 1 / 1.000 con 2% BSA / DPBS alle cellule e incubare a 37 ° C per 30 min.
- lavare delicatamente le cellule 3x con il 2% BSA / DPBS. Macchiare le cellule per i marcatori MSC. Aggiungere l'anticorpo primario solo ai pozzetti scelti come controlli positivi per ciascun anticorpo.
- Aggiungere 300 microlitri anti-CD105 diluiti 1/10 con il 2% BSA / DPBS, 300 microlitri anti-CD44 diluito 1/10 con il 2% BSA / DPBS, 300 microlitri anti-Stro-1 diluito 1/10 con il 2% BSA / DPBS e solo 200 microlitri 2% BSA / DPBS rispettivi pozzetti scelti come controlli negativi per ogni anticorpo.
- Incubare le cellule in un ambiente umido a 4 ° CO / N. Lavare i pozzetti 3 volte con DPBS e poi due volte con 2% BSA / DPBS.
- Rivela anticorpi primari con l'aggiunta di specifici anticorpi secondari.
- Aggiungere 300 microlitri anti-coniglio IgG (asino anti-IgG di coniglio [H + L]) diluito 1/100con il 2% BSA / DPBS in tutti i pozzetti scelta come controllo positivo e negativo per CD44 e CD105. Aggiungere 300 microlitri FITC anti-topo IG (FITC-coniglio anti-topo IgG [H + L]) diluito 1/100 con il 2% BSA / DPBS in tutti i pozzetti scelta come controllo positivo e negativo per Stro-1. Incubare le cellule al buio a temperatura ambiente per 60 min. Lavare i pozzetti 6x con DPBS.
- Stain MSC cellule marcatore-positivi per actina citoscheletro con l'aggiunta di 300 microlitri falloidina diluito 1/100 con il 2% BSA / DPBS in tutti i pozzetti macchiato per MSC marcatore immunofluorescenza.
- Incubare le cellule per 40 minuti a temperatura ambiente. Lavare i pozzetti 3 volte con DPBS e poi lavare due volte con ultrapura dH 2 O. Procedere alla controcolorazione dei nuclei dopo la colorazione di immunofluorescenza descritto sopra. Preparare propidio soluzione di ioduro di 10 -5 M in DPBS (stock 1.5 x 10 -3 M) in una cappa aspirante.
- Aggiungere 200 soluzione propidio ioduro microlitri di ciascun pozzetto colorati come descritto sopra. Incubare la cellas per 2 - 3 min a RT. Lavare i pozzetti due volte con ultrapura dH 2 O.
NOTA: ripetere tutte le fasi di sezioni 5.1 - 5.2 sulla linea cellulare HCT8, una linea principale continua differenziata cancro del colon cella che viene utilizzato come controllo negativo.
- Aggiungere 200 soluzione propidio ioduro microlitri di ciascun pozzetto colorati come descritto sopra. Incubare la cellas per 2 - 3 min a RT. Lavare i pozzetti due volte con ultrapura dH 2 O.
- Saggio differenziazione osteogenico
NOTA: osteogenico differenziazione ha una durata di 20 d.- Piatto OS-CSC in piastre da 24 pozzetti ad una densità cellulare di 1 x 10 4 cellule / cm 2 in SCCGM. Lasciare le cellule di crescere in SCGM fino raggiungere l'80 - 90% di confluenza in ciascun pozzetto.
- Inizia la differenziazione osteogenica cambiando la SCGM di OM. Permettono alle cellule di crescere in OM e rinfrescare media ogni 3 - 4 d. Arrestare il dosaggio differenziazione osteogenico a 10 d per valutare la presenza di fosfatasi alcalina (ALP).
- Fissare le cellule in 4% PFA / DPBS (vedere paragrafo 5.1). Valutare il fenotipo osteoblastico dalla colorazione citochimica per ALP e per HAutilizzando Alizarina Rossa S colorazione.
- ALP colorazione citochimica
NOTA: Preparare la miscela colorante in una cappa chimica immediatamente prima di iniziare la colorazione.- Disciogliere 40 mg Fast Blue BB o Fast Red Violet LB sale in 50 ml di Tris-HCl, pH 9 (soluzione A). Disciogliere 5 mg naftolo-AS-MX sale fosfato di sodio in 1 ml di DMSO (Soluzione B). Aggiungere Soluzione B interamente alla soluzione A e mescolare bene, ottenendo cellule Soluzione C. Lavare due volte con DPBS.
- Aggiungere 500 ml - 1 ml di soluzione C in ogni pozzetto e incubare a 37 ° C e 5% di CO 2. Monitorare il corso di macchiare ogni 10 min osservando le cellule al microscopio.
- Arrestare il colorazione quando le cellule ALP-positivi diventano intensamente colorati (blu con il Fast Blue BB sale o rosso con Fast Red Violet LB sale), che di solito si verifica entro 30 minuti. Lavare le cellule 3x con ultrapura dH 2 O per rimuovere tutti i residui di soluzione C. Se un precipitato del I macchias presenti, lavare le cellule rapidamente una volta con etanolo assoluto.
- Procedere alla controcolorazione dei nuclei dopo la colorazione ALP come descritto nei passi 5.2.5 e 5.2.5.1.
- Alizarina Rossa S colorazione citochimica
NOTA: Preparare la miscela colorante prima di iniziare l'esperimento.- Preparare il 2% Alizarina Red S (2 g Alizarina Rossa S in 100 ml di H 2 O ultrapura). Aggiungere 2,5% NH 3 al 2% Alizarina Rossa S per raggiungere pH 6,0. Conservare 2% soluzione Alizarina Rossa S a 4 ° C. Lavare le cellule una volta con DPBS.
- Aggiungere Alizarina Rossa S alle cellule per pochi secondi.
- Lavare i pozzetti con ultrapura dH 2 O per controllare il grado di colorazione; Se i depositi di HA non sono intensamente colorate, ripetere il punto 5.3.5.2. Fermare la colorazione quando i depositi HA diventano intensamente di colore rosso, che di solito si verifica entro pochi minuti. Lavare i pozzetti con ultrapura dH 2 O.
- differenziazione adipogenico
NOTA: Assay durata dipende linee OS-CSC. Il test può durare 14-30 d.- Piatto OS-CSC in piastre da 24 pozzetti ad una densità cellulare di 1 x 10 4 cellule / cm 2 in SCGM. Lasciare le cellule di crescere in SCGM fino raggiungere l'80 - 90% di confluenza in ciascun pozzetto. Avviare la differenziazione adipogenico cambiando la SCGM AM. Permettono alle cellule di crescere in AM e aggiornare la AM due volte a settimana.
- Arrestare il saggio di differenziazione adipogenico quando vescicole lipidiche sono visibili. Valutare il fenotipo adipogenico da Oil Red O colorazione e da ematossilina controcolorazione per nuclei.
- Ematossilina di contrasto per i nuclei
NOTA: Preparare la miscela colorante prima di iniziare l'esperimento.- Preparare la soluzione ematossilina 5%, chiamato Emallume Carazzi 39. Conservare la soluzione a 4 ° C. Aggiungere Emallume Carazzi solo per 2 minuti. Lavare pozzi con ULTrapure dH 2 O.
- saggio CFU
NOTA: Questo esperimento deve essere eseguito in triplicato.- Piastra OS-CSC in 100 mm piastre di Petri ad una densità cellulare di 450 cellule / cm 2 in CFUM. Incubare le cellule a 37 ° C, 5% CO 2 incubatore per 4 settimane. Aggiornare CFUM due volte a settimana.
- Colorare il CFU con blu di toluidina. Contare le colonie colorate utilizzando un microscopio invertito. Calcolo efficienza CFU secondo la seguente formula: (numero di colonie formate / numero di cellule seminate) * 100.
- Analisi delle attività ALDH
NOTA: ALDH attività è stata valutata utilizzando un Colorimetrico Assay Kit ALDH attività sulle due linee OS-CSC e su una linea di fibroblasti finita, che è stato utilizzato come controllo negativo. Questo kit quantifica l'attività enzimatica ALDH dalla lettura dell'assorbanza a 450 nm. Tutti i test sono stati eseguiti in triplicato.- Dissociarsi colture cellulari dal monostrato da tripsinizzazione (vedi Sezione 2.2). Agglomerare cellule per centrifugazione a 400 xg per 5 min. Seguire protocollo del produttore.
- Analisi citofluorimetrica:
NOTA: sospensioni di cellule singole sono necessari per la colorazione ottimale dei campioni per citometria a flusso. Una sospensione singola cella deve essere preparato per ciascun anticorpo da testare. Dissociarsi colture cellulari dal monostrato da tripsinizzazione (vedi Sezione 2.2).- Posizionare la sospensione cellulare in un tubo conico, eseguire una conta cellulare utilizzando il Bürker Conteggio sezione, centrifugare le cellule a 400 xg e risospendere in un volume adeguato di tampone di separazione per ottenere una sospensione cellulare ad una concentrazione cellulare finale di 1 x 10 5 cellule / ml.
- Centrifugare la sospensione cellulare per 5 minuti a 4 ° C. Eliminare il surnatante e lavare il pellet con tampone di separazione. Ripetere questa operazione due volte.
- cellule Stain per MSMarcatori C (vale a dire, CD44, CD105 e Stro-1) 40.
- Analizzare sospensioni cellulari colorate positivamente in un citometro di flusso. Analizzare i campioni marcati entro 1 d. Incubare questi campioni a 4 ° C fino all'analisi.
- RT-PCR
- Estrazione e isolamento di RNA
- Aggiungere 1 ml di reagente di lisi ai cellulari campioni congelati confezionati di OS-CSC e lisare le cellule direttamente nel tubo pipettando il pellet su e giù parecchie volte.
- Centrifugare i campioni a 12.000 xg per 1 min a 4 ° C. Rimuovere delicatamente il surnatante. Trasferire il surnatante in una nuova provetta, aggiungere 200 microlitri cloroformio e tappare la provetta in modo sicuro. Agitare la provetta vigorosamente per 15 sec e incubare il campione per 5 minuti a temperatura ambiente.
- Centrifugare i campioni a 12.000 xg per 15 min a 4 ° C. La miscela separa in tre diverse fasi. L'RNA è esclusivamente nella fase acquosa superiore incolore.
- essere particularly attenzione, rimuovere solo la fase acquosa e trasferire questa fase in un nuovo tubo per procedere con isolamento dell'RNA. Aggiungere 500 microlitri isopropanolo al tubo contenente la fase acquosa. Agitare la provetta delicatamente a mano. Incubare il campione a temperatura ambiente per 10 min. Centrifugare il campione a 12.000 xg per 10 min a 4 ° C.
NOTA: Dopo che il campione viene centrifugato, è possibile vedere l'RNA, che forma un pellet simile a gel sul lato e sul fondo della provetta. - Rimuovere il surnatante dal tubo, facendo attenzione a lasciare il pellet di RNA sul fondo. Aggiungere 1 ml di 75% di etanolo al tubo. Vortex la provetta a mano per pochi secondi, quindi si centrifuga il tubo a 7500 xg per 5 minuti a 4 ° C.
- Eliminare l'etanolo e asciugare il pellet di RNA. Quando il pellet è asciutto, risospendere il pellet di RNA in acqua RNase-free (10 - 50 mL) pipettando la soluzione su e giù parecchie volte.
- Determinare la resa e la purezza del RNA da measurante l'assorbanza a 260 nm e 280 nm utilizzando uno spettrofotometro. Valutare l'integrità del RNA totale su gel di agarosio standard di 1%. Conservare l'RNA a -80 ° C.
- Reazione a catena della polimerasi inversa
- Sintetizzare cDNA prima Strand da 500 campioni ng di RNA utilizzando un kit di trascrizione inversa. campioni di RNA Scongelare su ghiaccio e scongelare le soluzioni necessarie inclusi nel kit a temperatura ambiente. Procedere per sintetizzare cDNA seguenti protocollo del produttore.
- Semi-quantitativa della trascrittasi inversa-reazione a catena della polimerasi (RT-PCR)
- Eseguire tutte PCR utilizzando 1 ml di cDNA per ogni campione come modello in un volume di reazione finale di 24 ml. Utilizzare le sequenze di primer elencati nella tabella 1 per l'amplificazione di Nanog, Oct 3/4, SOX2 e CD133 geni.
- Separare i prodotti di RT-PCR da 1,8% elettroforesi su gel di agarosio e macchia con bromuro di etidio. Fotografia sotto illum UVinazione.
- Estrazione e isolamento di RNA
Risultati
Campioni OS ottenuti tramite aspirazione ago o resezione chirurgica di una piccola porzione del tumore (Figura 1A, B) permettono l'isolamento di un solo OSA se trattata appunto, come descritto nella sezione Protocollo (Figura 2A, B). Purtroppo, il numero di cellule isolate da biopsie è basso, con un campo di potenza da 30 - 50%. L'uscita dipende dal tipo e la dimensione delle biopsie (Figura 5A, B). Queste cellule devono essere trattati con precisione. Di conseguenza, circa un mese è necessario per le colture primarie di raggiungere confluenza in da 100 mm piastra di Petri. Trascorso questo tempo, OSA sono ottenuti da due campioni del sistema operativo ha segnato OSA5 e OSA6 (Figura 6A, B). Quindi, è necessario sottocultura linea cellulare primaria per ottenere un adeguato numero di cellule per eseguire l'analisi e caratterizzazione crioconservare cella lines. Al 3 ° passaggio di sottocultura, quando entrambe le linee cellulari primarie OSA raggiungano la confluenza, essi sono placcati in 6 pozzetti ultra-basso di attacco per il dosaggio sarcosphere. Questo tipo di piastra è usata perché ci permette di mantenere le cellule in uno stato sospeso, per evitare che le cellule staminali da differenziazione attaccamento-mediata, per evitare che le cellule ancoraggio-dipendenti dalla divisione, e, infine, per ridurre l'attaccamento al substrato. Quindi, il loro uso ci permette di creare una condizione di stress per le cellule tumorali, che è necessaria per la selezione di CSC. A 24 ore dopo l'inizio del test, le cellule appaiono isolate l'una dall'altra (Figura 7). Dopo 7 d di monitorare la progressione del saggio, piccole colonie sferiche hanno iniziato a formarsi e sono visibili (Figura 8). Al 28 d, diversi grandi colonie sferiche che si sono formati in ogni pozzetto si possono osservare (Figura 9A, B). Dopo le sarcospheres have stato in coltura per 28 d, queste colonie sferici di grandi dimensioni possono essere isolati. La figura 10 mostra la procedura per isolare sarcospheres da 6 pozzetti piastre di fissaggio ultra-basse e in condizioni reculturing aderenti. La figura 11 mostra le colonie sferiche galleggianti dopo l'isolamento. Le grandi colonie sferiche placcate in normali piastre di fissaggio mostrano espansione aderente dopo l'isolamento (Figura 12A, B). Le cellule che si espandono dalle singole sarcospheres sono probabilmente le cellule tumorali con cellule simil-staminali fenotipo. Quindi, dopo l'isolamento, OS-CSC erano probabilmente ottenuti. Queste cellule sono chiamate OSA5-CSC e OSA6-CSC (Figura 13A, B).
A questo punto, è necessario procedere alla caratterizzazione del cell-like fenotipo staminali per le due linee OS-CSC ottenute, come descritto sopra. Le analisi per la caratterizzazione del fenotipo wer cellule staminali simil-e eseguita al 4 ° passaggio di sottocultura dopo le sarcospheres per ogni linea OS-CSC sono stati isolati. Le due linee di cellule, OSA5-CSC e OSA6-CSC, hanno mostrato una forte positività per i marcatori di superficie MSC (CD105 e CD44) (Figura 14A, B e Figura 15A, B), mentre hanno mostrato positività moderata per il marcatore di superficie MSC Stro- 1 (Figura 16A, B). Le nostre osservazioni sono state confermate dai risultati negativi ottenuti con la linea commerciale e differenziato delle cellule del cancro del colon HCT8 (Figura 14C, 15C Figura, Figura 16C). È stata osservata una totale mancanza di colorazione specifica e non specifica per questi marcatori di superficie nella linea cellulare HCT8.
Per valutare il fenotipo MSC dei due OS-CSC, abbiamo anche analisi eseguite citometria a flusso. Entrambe le linee di OS-CSC hanno espresso alti livelli di CD44 e CD105. Tuttavia, le cellule in entrambe le linee cellulari, solo 1,14% espresso Stro-1. Pertanto, questa riSult ha confermato la presenza moderata di Stro-1 come dimostrato da immunofluorescenza. Al contrario, il 99.62% del OSA5-CSC espresso CD44 e 87.38% di queste cellule CD105 espresso; 99,88% del OSA6-CSC espresso CD44 e 95.79% di queste cellule CD105 espresso. Inoltre, entrambe le linee cellulari sono CD45-.
Abbiamo valutato l'espressione di 3 marcatori ESC (Nanog, Oct 3/4, Sox2) e del gene CD133, un altro marcatore CSC, mediante RT-PCR. Abbiamo notato che tutti questi geni sono stati espressi in entrambe le linee OS-CSC (Figura 17). I saggi di differenziazione adipogenici e osteogeniche hanno mostrato la capacità di entrambe le linee OSA-CSC isolati di differenziarsi in osteoblasti (Figura 18A - D e la Figura 19A - D) e in adipociti (Figura 20A - D).
Inoltre, il CFU un ssay (Figura 21) ha mostrato un buon tasso di efficienza clonogenica, con il 13% per OSA5-CSC e il 14% per OSA6-CSC. Diversi studi recenti hanno dimostrato che alti livelli di attività ALDH sono caratteristici di vari tipi di cancro. Questo parametro può essere utilizzato come marcatore di cellule staminali del cancro ed è correlato ad una prognosi infausta. Il saggio di attività ALDH ha mostrato che entrambe le linee OS-CSC hanno alti livelli di attività ALDH (Figura 22), mentre l'attività ALDH è stata osservata al limite inferiore quantificabile nella linea di fibroblasti che è stato utilizzato come controllo negativo in questo saggio.

Figura 1. Esempi di OS biopsia campioni. (A). campione bioptico ottenuto da ago aspirato. (B). campione bioptico ottenuto da resezione chirurgica di una parte del tumore.884 / 53884fig1large.jpg "target =" _ blank "> Clicca qui per vedere una versione più grande di questa figura.

Figura 2. disaggregazione meccanica di un campione di sistema operativo. (A) La frammentazione di un campione con una pinzetta Perry e una lancetta. (B) Frammenti sospesi a CM (indicato dalla freccia). Clicca qui per vedere una versione più grande di questa figura.
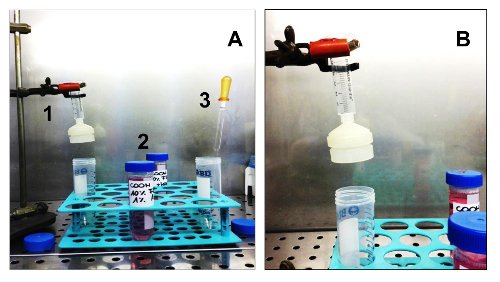
Figura 3. Attrezzature e materiali di consumo necessari per isolare Sarcospheres. (A). Tutte le attrezzature necessarie per l'isolamento delle cellule. 1. Una siringa sterile con un supporto filtro a membrana sterile; 2. Due diversi media: GM unND SCGM; 3. sterile vetro pipetta Pasteur. (B) Particolare della siringa montata su un supporto, con il supporto del filtro a membrana. Cliccate qui per vedere una versione più grande di questa figura.

Figura 4. Unità di filtrazione. Diversi componenti necessari per assemblare l'unità di filtrazione (A) (il filtro a rete è indicato dalla freccia). Osservazione contrasto di fase dei 40 micron maglie (una maglia è indicato dalla freccia) del filtro a rete (B). ingrandimento originale: 10X. L'unità di filtrazione assemblata (C - D). Unità di filtrazione sterilizzato (E). Clicca qui per visualizzare un grande version di questa figura.
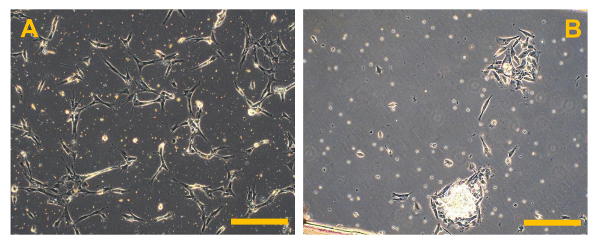
Figura 5. colture cellulari primarie di OS convenzionali. Osservazione contrasto di fase di colture cellulari primarie di alta qualità del sistema operativo. In (A), diversi frammenti ossei luminosi sono visibili, mentre in (B), diversi piccoli eritrociti arrotondati e galleggianti sono presenti. ingrandimento originale: 10X. Dimensione Bar:. 100 micron Clicca qui per vedere una versione più grande di questa figura.
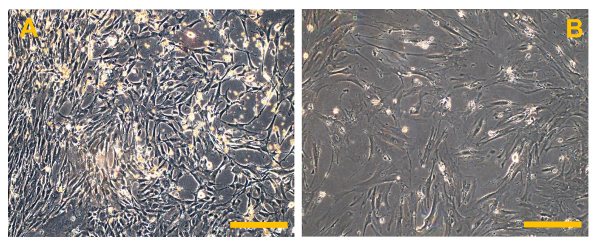
Figura 6. convenzionale osteosarcoma linee cellulari finiti (OSA). (A) OSA5 e (B) OSA6. Osservazione in contrasto di fase.ingrandimento originale: 10X. Dimensione Bar:. 100 micron Clicca qui per vedere una versione più grande di questa figura.
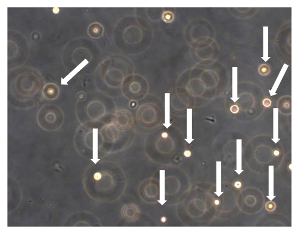
Figura 7. Sarcosphere Assay di OSA5 e OSA6. Dopo 24 ore dall'inizio del test, le cellule sono state galleggiando e isolate tra loro (cellule sono indicati da frecce). Osservazione in contrasto di fase. Ingrandimento originale:. 20X Clicca qui per vedere una versione più grande di questa figura.
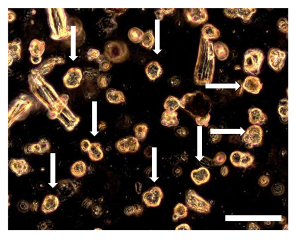
Figura 8. Sarcosphere Saggio di OSA5 e OSA6 alle 7 del D. 7 d nel saggio, diversi piccoli SphericaL colonie circondate da singole cellule potrebbero essere osservati. I sarcospheres (alcuni di questi sarcospheres sono indicati con frecce) appaiono galleggiante nel mezzo o leggermente stabilì nel fondo del pozzo. Osservazione in contrasto di fase. ingrandimento originale: 20X. Dimensione Bar:. 100 micron Clicca qui per vedere una versione più grande di questa figura.
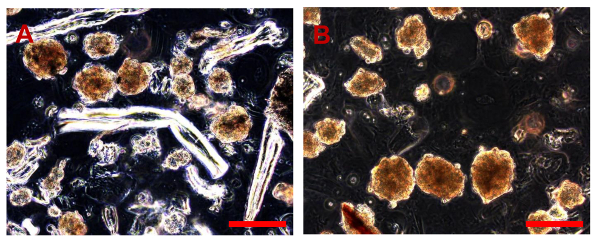
Figura 9. Sarcosphere Assay di OSA5 e OSA6 a 28 D. Dopo 28 d, diversi grandi sarcospheres ambra si osservano in ogni pozzetto delle piastre per ciascuna linea cellulare OSA, OSA5 (A) e OSA6 (B). Dimensione Bar: 100 micron. Cliccate qui per vedere una versione più grande di questa figura.

Figura 10. Passaggi per l'isolamento di Sarcospheres i passaggi per sarcospheres isolamento da 6 pozzetti ultra-bassi di attaccamento e la loro reculturing in condizioni aderenti sono visualizzabili (a) tutte le attrezzature necessarie per l'isolamento..: 1. Un 6-ben ultra-low piatto con sarcospheres formate in ogni pozzetto, 2. siringa con il supporto del filtro rete, 3. pipetta, 4. 1.000 ml punte sterili, 5. 2 sterili pinzette Perry, 6. 2 diversi terreni di coltura , 7. sterile pipetta Pasteur 8. piastre di Petri. (B) Raccolta di sarcospheres. Il mezzo contenuta in ciascun pozzetto viene raccolto con una pipetta con una sterile, punta 1.000 ml. (C) Raccogliere la sospensione nella siringa. La sospensione raccolto viene trasferito alla siringa per avviare il processo di filtraggio naturale utilizzando laSupporto filtro a membrana. (D) filtrazione naturale. (E) smontaggio del portafiltro membrana dalla siringa. Dopo tutto la sospensione viene filtrata, il portafiltro rete viene smontato e messo in una piastra di Petri; (F) Smontaggio del supporto del filtro a membrana. Sarcospheres sono contenute nei pori del filtro netto nel portafiltro rete, quindi devono essere liberate utilizzando le pinzette Perry. (G, H) La rimozione del sarcospheres dal filtro a membrana. Cliccate qui per vedere una versione più grande di questa figura.
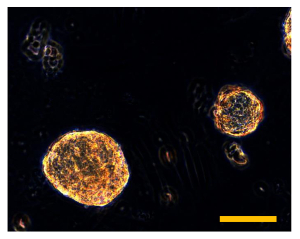
Figura 11. Isolamento Sarcosphere. Isolati sarcospheres galleggiano nel mezzo del piatto 60 millimetri Petri. Osservazione in contrasto di fase.ingrandimento originale: 40X. Dimensione Bar:. 100 micron Clicca qui per vedere una versione più grande di questa figura.
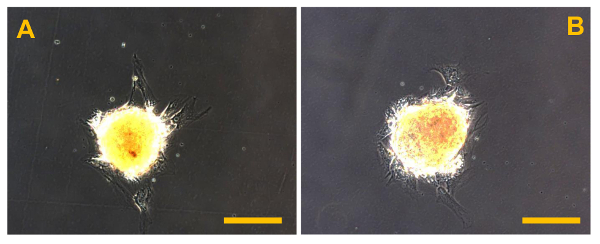
Figura 12. Sarcosphere Dopo isolamento. Sarcospheres da OSA5 (A) e OSA6 (B) linee cellulari così inizio dell'espansione aderenti seguenti reintroduzione e reculturing in un monostrato in condizioni aderenti a 48 ore dopo l'isolamento. Osservazione in contrasto di fase. ingrandimento originale: 20X. Dimensione Bar:. 100 micron Clicca qui per vedere una versione più grande di questa figura.
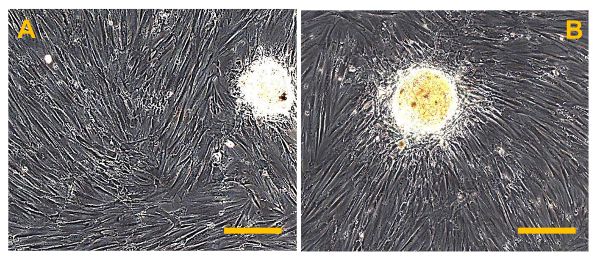
Figura 13. Sarcospheres a 7 D dopo l'isolamento. Sarcospheres da OSA5 (A) e linee cellulari OSA6 (B) hanno mostrato espansione aderente seguente reintroduzione e reculturing in un monostrato in condizioni aderenti alle 7 d dopo l'isolamento. Osservazione in contrasto di fase. ingrandimento originale: 20X. Dimensione Bar:. 100 micron Clicca qui per vedere una versione più grande di questa figura.
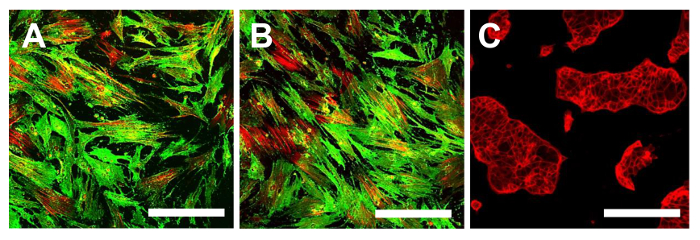
Figura 14. immunofluorescenza colorazione per CD105. Colorazione immunofluorescenza per CD105 nelle linee OSA-CSC OSA5-CSC (A) e OSA6-CSC (B) e nella linea cellulare continua HCT8 (C), che è stato utilizzato come controllo negativo. LSCM colore convenzionale: Verde per CD105 e rosso per citoscheletro. ingrandimento originale: 10X. Dimensione Bar:. 100 micron Clicca qui per vedere una versione più grande di questa figura.
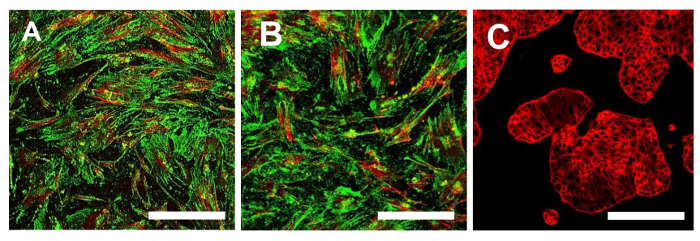
Figura 15. immunofluorescenza colorazione per CD44. Colorazione immunofluorescenza per CD44 nelle linee OSA-CSC OSA5-CSC (A) e OSA6-CSC (B) e nella linea cellulare continua HCT8 (C), che è stato utilizzato come controllo negativo. LSCM nei colori tradizionali: verde per CD44 e rosso per il citoscheletro. ingrandimento originale: 10X. Dimensione Bar:. 100 micron Clicca qui per vedere una versione più grande di questa figura.
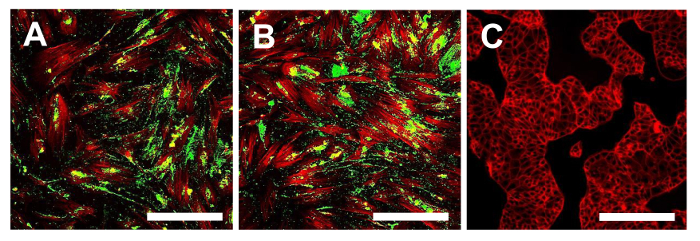
Figura 16. Immunofluorescenza di Stro1. Colorazione immunofluorescenza per Stro-1 nelle linee OSA-CSC OSA5-CSC (A) e OSA6-CSC (B) e nella linea cellulare continua HCT8 (C), che è stato usato come un fatto negativo controllo. LSCM nei colori tradizionali: verde per Stro-1 e rosso per citoscheletro. ingrandimento originale: 10X. Dimensione Bar:. 100 micron Clicca qui per vedere una versione più grande di questa figura.
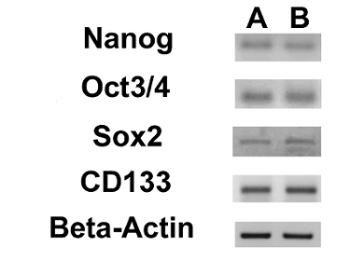
Figura 17. Espressione del nucleare CES marcatori e del CD133 Gene. RT-PCR che mostra l'espressione di Nanog, Oct 3/4, Sox2 e CD133 in OSA5-CSC (A) e in OSA6-CSC ( B). Clicca qui per vedere una versione più grande di questa figura.
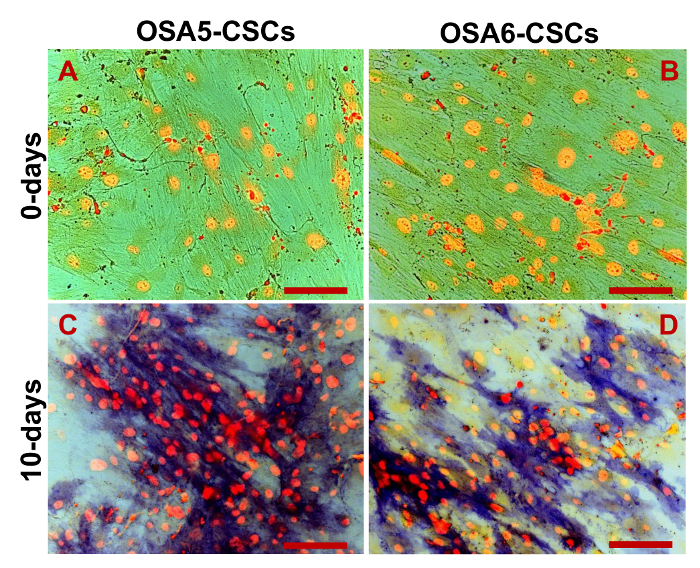
Figura 18. osteogenico differenziazione Assay -. ALP differenziazione osteogenica a 0 d (A, B) e dopo 10 d (C, D) dell'induzione come determinato mediante colorazione citochimica per ALP utilizzando Fast Blue BB. In blu, ALP + cellule; in rosso, il nucleo di contrasto con ioduro di propidio. osservazione composito in campo chiaro e in fluorescenza. ingrandimento originale: 20X. Dimensione Bar:. 100 micron Clicca qui per vedere una versione più grande di questa figura.

Figura 19. osteogenico differenziazione Assay -. HA differenziazione osteogenica a 0 d (A, B) e dopo 20 d (C, D) dell'induzione come determinato mediante colorazione citochimica per idrossiapatite (HA) con Alizarina Red S. Le cellule sono contrapposti, depositi blu / grigio, e il sgranate di HA sono macchiati di rosso. Osservazione in contrasto di fase. ingrandimento originale: 40X. Dimensione Bar:. 100 micron Clicca qui per vedere una versione più grande di questa figura.
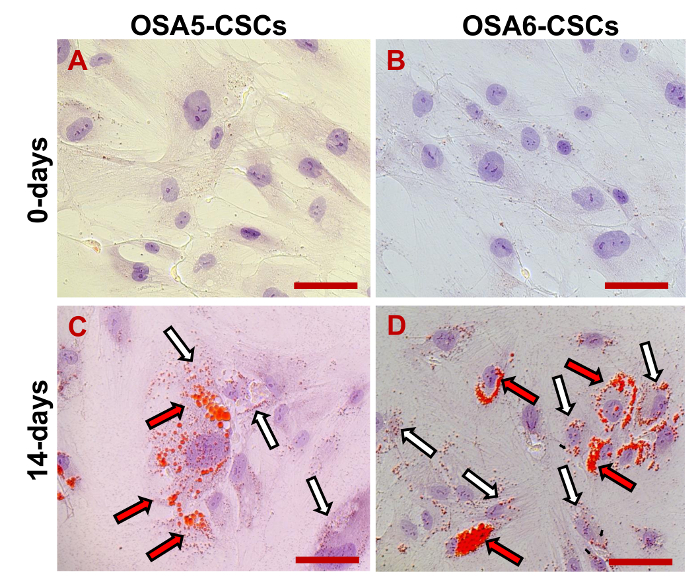
Figura 20. adipogenico Differenziazione Assay. Differenziazione adipogenico a 0 d (A, B) e dopo 14 d (C, D) di induzione determinato cytochcolorazione emical con Oil Red O. In rosso, le vescicole lipidiche (vescicole grandi sono indicati dalle frecce nere / rosse, le vescicole più piccole sono indicati dalle frecce nere / bianco); in blu / viola, i nuclei contrastate da ematossilina. L'osservazione in campo chiaro. ingrandimento originale: 40X. Dimensione Bar:. 100 micron Clicca qui per vedere una versione più grande di questa figura.
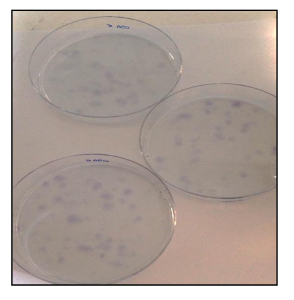
Figura 21. CFU Assay. Saggio CFU di linee di OSA-CSC colorate con blu di toluidina. Cliccate qui per vedere una versione più grande di questa figura.
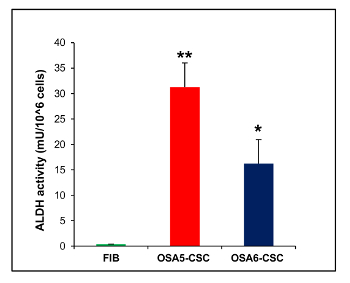 /> Figura 22. ALDH Activity Assay. Il saggio colorimetrico ALDH rilevato alti livelli di attività ALDH nelle due linee di OS-CSC, OSA5-CSC e OSA6-CSC, mentre il saggio rilevati l'assenza di questa attività nella linea di cellule differenziate finita dei fibroblasti, FIB. Le barre di errore: SD. **: P <0,001 vs FIB; *:. P <0.01 vs FIB Cliccate qui per vedere una versione più grande di questa figura.
/> Figura 22. ALDH Activity Assay. Il saggio colorimetrico ALDH rilevato alti livelli di attività ALDH nelle due linee di OS-CSC, OSA5-CSC e OSA6-CSC, mentre il saggio rilevati l'assenza di questa attività nella linea di cellule differenziate finita dei fibroblasti, FIB. Le barre di errore: SD. **: P <0,001 vs FIB; *:. P <0.01 vs FIB Cliccate qui per vedere una versione più grande di questa figura.
| Gene | oligonucleotidi | Sequence (5¹-3¹) | Dimensione Amplicon (bp) | Tₐ (° C) | |
| Nanog | Forward Primer Reverse Primer | 87 | 60 | ||
| Ottobre 3/2 | Forward Primer Reverse Primer | GGGAGGAGCTAGGGAAAGA TCCTTCCTTAGTGAATGAAGAACT | 77 | 60 | |
| Sox2 | Forward Primer Reverse Primer | TGCAGTACAACTCCATGACC GGACTTGACCACCGAACC | 125 | 55 | |
| CD133 | Forward Primer Reverse Primer | CCAGAAGCCGGGTCATAAAT ATTCACTCAAGGCACCATCC | 127 | 56 | |
| BP, paia di basi di dimensioni amplicone; Tₐ, ricotturatemperatura | |||||
Tabella 1. Elenco dettagliato di primer sequenze per Nanog, Oct 3/4, Sox2 e CD133 con la dimensione Amplicon e la ricottura temperatura
Discussione
CSC hanno diverse proprietà che permettono l'identificazione di questo particolare sottoinsieme cellulare nella massa tumorale. Sulla base di queste caratteristiche, come la resistenza acquisita citotossica agenti chemioterapici per la sovraespressione di ATP-binding cassette multidrug trasportatori di efflusso 28, 32, 33, o per la upregulation dell'espressione di enzimi detossificanti come ALDH 32, per l'espressione di un particolare marcatore di superficie, come CD133, CD44, CD34, CD90, e altri 30, 34, 35, 41, diversi metodi per isolare CSC sono stati sviluppati 42-44. Una di queste tecniche è il saggio formazione sfera, che si basa sulla capacità di CSC di crescere in condizioni non aderenti.
La capacità delle cellule staminali dei tessuti e CSC per formare sfere è stato descritto in studi sulla identificazione delle cellule staminali neurali da Reynolds et al. 37. Successivamente, Gibbs et al. 38 usEd questi studi per iniziare a isolare CSC da tumori solidi, in particolare, dai sarcomi ossei. Abbiamo deciso di utilizzare il metodo di dosaggio formazione sfera illustrato da Gibbs et al. Isolare CSC da linee cellulari ottenute da biopsie OSA OS convenzionali. Abbiamo adattato il metodo originale per migliorare i risultati di questo test e facilitare la sua riproducibilità per altre linee cellulari di cancro. Con riferimento alla costituzione del saggio formazione sfera, abbiamo verificato che placcatura 40.000 cellule / pozzetto è una buona pratica per mantenere le cellule in isolamento all'inizio del saggio. Questo accorgimento è molto importante per evitare la possibilità che le colonie sferiche provengono da aggregazione cellulare e non dalla capacità particolare ed esclusiva di un singolo CSC di crescere in condizioni non aderenti e formare una colonia sferica. Questa capacità è un punto particolarmente critico di questo test.
Abbiamo inoltre attestato che per ottenere una buona velocità di formazione sfera, È sufficiente aggiornare aliquote di fattori di crescita ogni 3 d e non tutti i giorni come descritto nel metodo originale. In questo studio, abbiamo anche stabilito e ampiamente descritto un buon metodo per isolare le colonie sferiche che si sono formate quando coltivate in condizioni non aderenti. Questo passaggio è fondamentale in questo saggio perché è molto importante cercare di isolare il maggior numero di sfere possibile che si formano in ciascun pozzetto senza danneggiarle. E 'anche importante isolare solo le sfere e non le singole cellule, che possono rimanere in sospensione per la durata del test. Per superare questi punti critici, abbiamo sviluppato un particolare metodo di isolamento, che, come sopra indicato, ha dato buoni risultati per isolamento CSC. Ovviamente, c'è la possibilità che non tutte le sfere che formano possono essere recuperati, ma la percentuale di perdita è molto bassa. In effetti, abbiamo anche la possibilità di utilizzare un filtro con 40 micron pori per isolare sfere dopo diventano grandi (moduloed circa 100 - 200 cellule).
Questo isolamento arresta formazione sfera, ma permette alle singole celle, parte del residuo metilcellulosa, e le piccole sfere da filtrare. Tale eliminazione viene eseguita mediante filtrazione completa come descritto nel protocollo.
Inoltre, la selezione delle più grandi colonie sferiche attraverso i 40 micron maglia con la conseguente perdita delle colonie sferiche più piccole permette di selezionare le CSC con la più alta capacità di formare colonie sferiche e con maggiore stemness. Tutte queste modifiche sono state eseguite per migliorare l'analisi e per aiutare i ricercatori che studiano CSC di comprendere e riprodurre la fase più critica del metodo originale del saggio formazione sfera.
Tra gli studi in materia di metodi in vitro per la CSC isolamento, questo studio ha lo scopo di mostrare come questo saggio formazione sfera adattato potrebbe essere un buon metodo per isolare CSC dalinee cellulari OSA. Gli adattamenti del metodo originale e la tecnica dettagliata isolamento descritti migliorare la sua efficacia. In breve tempo, buon numero di CSC può essere ottenuto e utilizzato per diversi esperimenti. Pertanto, è possibile confermare rapidamente i fenotipi staminali-simili e, in particolare, per studiare il fenotipo doppio stelo-like che caratterizza OS-CSC. Così, questo test modificato potrebbe essere una buona tecnica per isolare CSC e studiare la loro biologia. In futuro, questo metodo, con ulteriori adattamenti, può anche essere utilizzata per isolare CSC da altre linee cellulari tumorali finiti ottenuti da biopsie di tumori solidi rare.
La possibilità di isolare CSC da tumori solidi rare come OS, non solo permette il miglioramento degli studi su questo particolare cancro, ma si estende anche a studi di diversi tipi di cancro per sviluppare metodi migliori per il loro isolamento e futuri studi di biologia questo importante sottoinsieme cellulare. Pertanto, come abbiamohanno fatto in questo studio, è importante migliorare i metodi di isolamento CSC attraverso lo studio di CSC biologia, con l'obiettivo finale di trovare bersagli molecolari e di sviluppare una terapia antitumorale molto specifico diretto contro questo particolare sottoinsieme cellulare, che è probabilmente responsabile il mantenimento del tumore primario, lo sviluppo della sua ricorrenza, e l'origine di metastasi in diversi organi. Lo studio di CSC biologia è importante anche per la ricerca di terapie che potrebbero essere incisivo nella cura di tumori, come il sistema operativo, per i quali il tasso di sopravvivenza dopo il trattamento neoadiuvante rimane molto povera.
Divulgazioni
The authors declare that they have no competing financial interest.
Riconoscimenti
This study was supported by ITT (Istituto Toscano Tumori) Grant Proposal 2010.
Materiali
| Name | Company | Catalog Number | Comments |
| Dulbecco's Phosphate Buffered Saline with Ca2+ and Mg2+ (DPBS) | LONZA | BE17-513F | _ |
| Dulbecco's Phosphate Buffered Saline without Ca2+ and Mg2+ (DPBS) | LONZA | BE17-512F | _ |
| Porcine Trypsin 1:250 | BD Difco | 215310 | Solvent: DPBS. Stock concentration: Powder |
| Ethylenediamine tetraacetic acid disodium salt dihydrate(EDTA) | Sigma-Aldrich | E4884 | Solvent: DPBS. Stock concentration: Powder |
| Collagenase from Clostridium histolyticum | Sigma-Aldrich | C0130 | Solvent: Buffer Solution, pH 7.4. Stock concentration: Powder |
| Dimethyl sulphoxide (DMSO) | BDH Chemicals-VWR | 10323 | _ |
| Nutrient Mixture F-12 Ham | Sigma-Aldrich | F6636 | Solvent: Ultrapure dH2O. Stock concentration: Powder |
| 2-Phospho-L-ascorbic acid trisodium salt | Sigma-Aldrich | 49752 | Solvent: DPBS. Stock concentration: 5 mg/mL |
| β-Glycerol phosphate disodium salt pentahydrate | Sigma-Aldrich | 50020 | Solvent: DPBS. Stock concentration: 1 M |
| Insulin. Human Recombinant | Sigma-Aldrich | 91077 | Solvent: NaOH 0.1 M. Stock concentration: 10 mM |
| 3-Isobutyl-1-methylxanthine | Sigma-Aldrich | I5879 | Solvent: DMSO. Stock concentration: 500 mM |
| Indomethacin | Sigma-Aldrich | I7378 | Solvent: DMSO. Stock concentration: 200 mM |
| Dexamethasone | Sigma-Aldrich | D4902 | Solvent: DMSO. Stock concentration: 1 mM / 100 µM. Store in liquid nitrogen to maintain the biological activity |
| Fetal Bovine Serum (FBS) | Sigma-Aldrich | F7524 | _ |
| Fetal Bovine Serum South America | EUROCLONE | ECS0180L | _ |
| Penicillin-Streptomycin (PEN-STREP) 10,000 U/mL | LONZA | DE17-602E | _ |
| Methyl cellulose | Sigma-Aldrich | 274429 | Solvent: Ultrapure dH2O. Stock concentration: 2% |
| Putresceine dihydrochloride | Sigma-Aldrich | P5780 | Solvent: Ultrapure dH2O. Stock concentration: Powder |
| apo-Transferrin | Sigma-Aldrich | T-1147 | Solvent: DPBS. Stock concentration: 25 mg/mL |
| Human Epidermal Growth Factor (EFGF) | Sigma-Aldrich | E5036 | Solvent: DPBS pH 7.4. Stock concentration: 10 µg/mL |
| Fibroblast Growth Factor-Basic Human | Sigma-Aldrich | F0291 | Solvent: DPBS + 0.2% BSA. Stock concentration: 25 µg/mL |
| Selenous Acid | Sigma-Aldrich | 211176 | Solvent: DPBS. Stock concentration: 30 mM |
| Progesterone | Sigma-Aldrich | P8783 | Solvent: ETOH. Stock concentration: 10 mM |
| Toluidine Blue O | Sigma-Aldrich | 198161 | Solvent: Ultrapure dH2O. Stock concentration: Powder |
| Oil Red O | ICN Biochemicals | 155984 | Solvent: 2-Propanol. Stock concentration: Powder |
| Naphtol AS-MX Phosphate Disodium Salt | Sigma-Aldrich | N5000 | Solvent: DMSO. Stock concentration: Powder |
| Fast Blue BB Salt | Sigma-Aldrich | F3378 | Solvent: Tris HCL, pH 9.0. Stock concentration: Powder |
| Fast Red Violet LB Salt | Sigma-Aldrich | F3381 | Solvent: Tris HCL, pH 9.1. Stock concentration: Powder |
| Bovine Serum Albumin, Fraction V (BSA) | Sigma-Aldrich | A-4503 | Solvent: DPBS. Stock concentration: 2% |
| Alizarin Red S | ICN Biochemicals | 100375 | Solvent: Ultrapure dH2O. Stock concentration: Powder |
| Formaldehyde solution | Sigma-Aldrich | 533998 | 4% |
| Triton-100X | MERCK | 11869 | Solvent: DPBS. Stock concentration: 0.2%. Danger - Use only under chemical hood |
| Calcein | MERCK | 2315 | Solvent: DPBS . Stock concentration: 200 µg/mL |
| 2-Propanol | MERCK | 109634 | Danger - Use only under chemical hood |
| Ab-CD105 (Mouse monoclonal [SN6] to CD105 (FITC) | Abcam | ab11415 | Liquid. Application: Flow Cytometry (Flow Cyt) |
| Ab-CD44 (Mouse monoclonal [F10-44-2] to CD44 (PE/Cy7®) ) | Abcam | ab46793 | Liquid. Application: Flow Cytometry (Flow Cyt) |
| Ab-CD45 (Mouse monoclonal [MEM-28] to CD45 (PerCP)) | Abcam | ab65952 | Liquid. Application: Flow Cytometry (Flow Cyt) |
| Ab-CD105 (Human CD105 Purified Antibody) | Invitrogen | MHCD10500 | Solvent: DPBS. Stock concentration: Lyophilized. Application: Immunofluorescence staining (IF) |
| Ab-CD44 (Anti-CD44 Antibody) | Abcam | EPR1013Y(ab51037) | Solvent: DPBS. Stock concentration: Lyophilized. Application: Immunofluorescence staining (IF) |
| Ab-Stro-1 (Mouse anti-STRO-1) | Invitrogen | 398401 | Solvent: DPBS. Stock concentration: Lyophilized. Application: Flow Cytometry (Flow Cyt) Immunofluorescence staining (IF) |
| Alexa Fluor 488 (Anti-Rabbit IgG (Alexa Fluor 488 Donkey Anti-Rabbit IgG (H+L)) | Invitrogen | A-21206 | Solvent: DPBS. Stock concentration: Lyophilized. Application: Immunofluorescence staining (IF) |
| FITC Anti-Mouse IG (FITC-Rabbit Anti Mouse IgG (H+L)) | Invitrogen | 61-6511 | Solvent: DPBS. Stock concentration: Lyophilized. Application: Flow Cytometry (Flow Cyt) Immunofluorescence staining (IF) |
| Alexa Fluor 635 Phalloidin | Invitrogen | A34054 | Solvent: DPBS. Stock concentration: Lyophilized. Application: Immunofluorescence staining (IF) |
| AutoMACS™Running Buffer MACS Separation Buffer | Miltenyi | 130,091,221 | Liquid. Store at 4 °C |
| QIAzol®Lysis Reagent | QIAGEN | 79306 | Danger - Use only under chemical hood |
| QUANTITECT® Reverse Transcription Kit | QIAGEN | 205314 | _ |
| Chlorophorm | Sigma-Aldrich | C2432 | Liquid. Danger - Use only under chemical hood |
| Laminar flow hood | GELAIRE | BSB6A | _ |
| Chemical hood | ARREDI TECNICI Villa | Modello DYNAMICA | _ |
| Centrifuge | EPPENDORF | 5415R | _ |
| Laser Scanning Confocal Microscopy LSM 5109 Meta | ZEISS | _ | _ |
| iCycler PCR Thermalcycler | BIORAD | _ | _ |
| CyFlow®SPACE | (PARTEC) | _ | _ |
| Inverted Micrposcope Axiovert 200M | ZEISS | _ | _ |
| Freezing container , | Nalgene | _ | _ |
| Original Pipet-Aid | pbiBrand | _ | _ |
| Micropipettes | EPPENDORF | _ | _ |
| Glass Pasteur Pipette | SIGMA | _ | _ |
| VICTOR3™ | PERKIN ELMER | _ | _ |
| Conical tubes (15 and 50 mL) | BD FALCON | 352096 (for 15 mL) 352070 (for 50 mL) | _ |
| 24-Well Clear Flat Bottom TC-Treated Multiwell-Cell-Culture-Plate | BD FALCON | 353047 | _ |
| 6-Well Clear Flat Bottom Ultra Low Attachment Multiple-Well-Plates | CORNING | 3471 | _ |
| Serological pipettes (5 and 10 mL) | BD FALCON | 357543 (for 5 mL) 357551 (for 10mL) | _ |
| Syringe (5mL) | B|BRAUN | 4617053V | _ |
| Petri dish 100X20 mm | BD FALCON | 353003 | _ |
| Röhren Tubes (3.5 mL, 55x12mm, PS) | SARSTEDT | 55,484 | _ |
| Petri dish 60X15 mm | BD FALCON | 353004 | _ |
| Cryovials 1.5 mL | NALGENE | 5000-1020 | _ |
| Cell-Culture Flasks 25 cm² | BD FALCON | 353014 | _ |
| Nylon Net Filter, Hydrophilic | MERCK | NY4104700 | _ |
| Swinnex Filter Holder | MERCK | SX0002500 | _ |
| Perry tweezer | _ | _ | _ |
| Lancet | _ | _ | _ |
| Dounce | _ | _ | _ |
Riferimenti
- Reddick, R. L., Michelitch, H. J., Levine, A. M., Triche, T. J. Osteogenic sarcoma: a study of the ultrastructure. Cancer. 45 (1), 64-71 (1980).
- Gatta, G., et al. Childhood cancer survival trends in Europe: a EUROCARE Working Group study. J. Clin. Oncol. 23 (16), 3742-3751 (2005).
- Olstad, O. K., et al. Molecular heterogeneity in human osteosarcoma demonstrated by enriched mRNAs isolated by directional tag PCR subtraction cloning. Anticancer. Res. 23 (3B), 2201-2216 (2003).
- Geller, D. S., Gorlick, R. Osteosarcoma: a review of diagnosis, management, and treatment strategies. Clin Adv Hematol Oncol. 8 (10), 705-718 (2010).
- Tang, N., Song, W. X., Luo, J., Haydon, R. C., He, T. C. Osteosarcoma development and stem cell differentiation. Clin. Orthop. Relat. Res. 466 (9), 2114-2130 (2008).
- Kempf-Bielack, B., et al. Osteosarcoma relapse after combined modality therapy: an analysis of unselected patients in the Cooperative Osteosarcoma Study Group (COSS). J. Clin. Oncol. 23 (3), 559-568 (2005).
- Meyers, P. A., et al. Osteosarcoma: a randomized, prospective trial of the addition of ifosfamide and/or muramyl tripeptide to cisplatin, doxorubicin, and high-dose methotrexate. J. Clin. Oncol. 23 (9), 2004-2011 (2005).
- Gorlick, R., et al. Biology of childhood osteogenic sarcoma and potential targets for therapeutic development: meeting summary. Clin. Cancer Res. 9 (15), 5442-5453 (2003).
- Hayden, J. B., Hoang, B. H. Osteosarcoma: basic science and clinical implications. Orthop. Clin. North Am. 37 (1), 1-7 (2006).
- Thomas, D., Kansara, M. Epigenetic modifications in osteogenic differentiation and transformation. J. Cell. Biochem. 98 (4), 757-769 (2006).
- Araki, N., et al. Involvement of the retinoblastoma gene in primary osteosarcomas and other bone and soft-tissue tumors. Clin. Orthop. Relat. Res. 270 (270), 271-277 (1991).
- Chou, A. J., Gorlick, R. Chemotherapy resistance in osteosarcoma: current challenges and future directions. Expert. Rev. Anticancer Ther. 6 (7), 1075-1085 (2006).
- Arndt, C. A. S., Crist, W. M. Common musculoskeletal tumorsof childhood and adolescence. N. Engl. J. Med. 341 (5), 342-352 (1999).
- Longhi, A., Errani, C., De Paolis, M., Mercuri, M., Bacci, G. Primary bone osteosarcoma in the pediatric age: state of the art. Cancer. Treat. Rev. 32 (6), 423-436 (2006).
- Bacci, G., et al. Long-term outcome for patients with nonmetastatic osteosarcoma of the extremity treated at the istituto ortopedico rizzoli according to the istituto ortopedico rizzoli/osteosarcoma-2 protocol: an updated report. J. Clin. Oncol. 18 (24), 4016-4027 (2000).
- Clarke, M. F., et al. Cancer stem cells-perspectives on current status and future directions: AACR workshop on cancer stem cells. Cancer. Res. 66 (19), 9339-9344 (2006).
- Lapidot, T., et al. A cell initiating human acute myeloid leukaemia after transplantation into SCID mice. Nature. 367 (6464), 645-648 (1994).
- Bonnet, D., Dick, J. E. Human acute myeloid leukemia is organized as a hierarchy that originates from a primitive hematopoietic cell. Nat. Med. 3 (7), 730-737 (1997).
- Visvader, J. E., Lindeman, G. J. Cancer stem cells in solid tumors: accumulating evidence and unresolved questions. Nat. Rev. Cancer. 8 (10), 755-768 (2008).
- Bapat, S. A. Evolution of cancer stem cells. Semin. Cancer Biol. 17 (3), 204-213 (2007).
- Rubio, D., et al. Spontaneous human adult stem cell transformation. Cancer. Res. 65 (8), 3035-3039 (2005).
- Burns, J. S., et al. Tumorigenic heterogeneity in cancer stem cells evolved from long-term cultures of telomerase-immortalized human mesenchymal stem cells. Cancer. Res. 65 (8), 3126-3135 (2005).
- Zhang, M., Rosen, J. M. Stem cells in the etiology and treatment of cancer. Curr. Opin. Genet. Dev. 16 (1), 60-64 (2006).
- Li, Y., et al. Evidence that transgenes encoding components of the Wnt signaling pathway preferentially induce mammary cancers from progenitor cells. Proc. Natl Acad. Sci. USA. 100 (26), 15853-15858 (2003).
- Sell, S. Cellular origin of cancer: dedifferentiation or stem cell maturation arrest?. Environ Health Perspect. 101 (Suppl 5), 15-26 (1993).
- Wernig, M., et al. In vitro reprogramming of fibroblasts into a pluripotent ES-cell-like state. Nature. 448 (7151), 318-324 (2007).
- Niwa, H., Miyazaki, J., Smith, A. G. Quantitative expression of oct-3/4 defines differentiation, dedifferentiation or self-renewal of ES cells. Nat. Genet. 24 (4), 372-376 (2000).
- Hochedlinger, K., Jaenisch, R. Nuclear reprogramming and pluripotency. Nature. 441 (7097), 1061-1067 (2006).
- Dean, M., Fojo, T., Bates, S. Tumor stem cells and drug resistance. Nat. Rev. Cancer. 5 (4), 275-284 (2005).
- Ma, S., Lee, T. K., Zheng, B. J., Chan, K. W., Guan, X. Y. CD133+ HCC cancer stem cells confer chemoresistance by preferential expression of the Akt/PKB survival pathway. Oncogene. 27 (12), 1749-1758 (2008).
- Wu, C., et al. Side population cells isolated from mesenchymal neoplasms have tumor initiating potential. Cancer. Res. 67 (17), 8216-8222 (2007).
- Ma, I., Allan, A. L. The role of human aldehyde dehydrogenase in normal and cancer stem cells. Stem. Cell. Rev. 7 (2), 292-306 (2011).
- Awad, O., et al. High ALDH activity identifies chemotherapy-resistant Ewing's sarcoma stem cells that retain sensitivity to EWS-FLI1 inhibition. PLoS One. 5 (11), e13943 (2010).
- Fujii, H., et al. Sphere-forming stem-like cell populations with drug resistance in human sarcoma cell lines. Int. J. Oncol. 34 (5), 1381-1386 (2009).
- Di Fiore, R., et al. Genetic and molecular characterization of the human osteosarcoma 3AB-OS cancer stem cell line: a possible model for studying osteosarcoma origin and stemness. J. Cell. Physiol. 228 (6), 1189-1201 (2013).
- Reynolds, B. A., Tetzlaff, W., Weiss, S. A multipotent EGF-responsive striatal embryonicprogenitor cell produces neurons and astrocytes. J. Neurosci. 12 (11), 4565-4574 (1992).
- Reynolds, B. A., Weiss, S. Generation of neurons and astrocytes from isolated cells of the adult mammalian central nervous system. Science. 255 (5052), 1707-1710 (1992).
- Gibbs, C. P., et al. Stem-like cells in bone sarcomas: implications for tumorigenesis. Neoplasia. 7 (11), 967-976 (2005).
- Beccari, N., Mazzi, V. . Manuale di tecnica microscopic. Casa Editrice Dr. Francesco Vallardi Società Editrice Libraria. , 99-100 (1966).
- Majumdar, M. K., Thiede, M. A., Mosca, J. D., Moorman, M., Gerson, S. L. Phenotypic and functional comparison of cultures of marrow-derived mesenchymal stem cells (MSCs) and stromal cells. J. Cell. Physiol. 176 (1), 57-66 (1998).
- Tirino, V., et al. Human primary bone sarcomas contain CD133+ cancer stem cells displaying high tumorigenicity in vivo. FASEB J. 25 (6), 2022-2030 (2011).
- Tirino, V., et al. Cancer stem cells in solid tumors: an overview and new approaches for their isolation and characterization. FASEB J. 27 (1), 13-24 (2013).
- Martins-Neves, S. R., et al. Therapeutic implications of an enriched cancer stem-like cell population in a human osteosarcoma cell line. BMC Cancer. 12 (1), 139 (2012).
- Tang, Q. L., et al. Enrichment of osteosarcoma stem cells by chemotherapy. Chin. J. Cancer. 30 (6), 426-432 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon