Method Article
Dithranol come Matrix Assisted Laser per Matrix desorbimento / ionizzazione Imaging su una trasformata di Fourier Ionorisonanza Ciclotronica Mass Spectrometer
In questo articolo
Riepilogo
Dithranol (DT; 1,8-diidrossi-9 ,10-dihydroanthracen-9-one) è stato precedentemente segnalato come una matrice MALDI per l'imaging tessuto di piccole molecole; protocolli per l'uso di DT per l'imaging MALDI di lipidi endogeni sul superficie di sezioni di tessuto di positivo-ion MALDI-MS su uno strumento quadrupolo-FTICR altissima risoluzione sono forniti qui.
Abstract
Spettrometria di massa di imaging (MSI) determina la localizzazione e distribuzione pattern spaziali di composti sulla superficie di una sezione di tessuto, principalmente utilizzando MALDI (matrice desorbimento laser assistita / ionizzazione) basati tecniche analitiche. Sono necessarie nuove matrici per piccola molecola MSI, che possono migliorare l'analisi di basso peso molecolare (MW) composti,. Queste matrici dovrebbero fornire maggiori segnali di analiti riducendo segnali di fondo MALDI. Inoltre, l'uso di strumenti ad altissima risoluzione, come trasformata di Fourier ciclotrone ionico (FTICR) spettrometri di massa, ha la capacità di risolvere segnali analita da segnali a matrice, e questo può parzialmente superare molti problemi associati con lo sfondo proveniente dalla MALDI matrice. La riduzione delle intensità dei cluster matrice metastabili di FTICR MS può anche contribuire a superare alcuni dei disturbi associati con picchi matrice su altri strumenti. Ad alta risoluzionestrumenti quali gli spettrometri di massa FTICR sono vantaggiosi in quanto possono produrre modelli di distribuzione di molti composti contemporaneamente pur fornendo fiducia in identificazioni chimici. Dithranol (DT; 1,8-diidrossi-9 ,10-dihydroanthracen-9-one) è stato precedentemente segnalato come una matrice MALDI per imaging dei tessuti. In questo lavoro, un protocollo per l'uso di DT per MALDI imaging lipidi endogeni dalle superfici di sezioni di tessuto di mammifero, da positivo ioni MALDI-MS, su un ibrido quadrupolo altissima risoluzione sono stati inseriti strumento FTICR.
Introduzione
Spettrometria di massa di imaging (MSI) è una tecnica analitica per la determinazione della localizzazione e distribuzione pattern spaziali di composti sulla superficie di una sezione di tessuto 1,2. Matrix desorbimento laser assistita / ionizzazione (MALDI) MSI per l'analisi di peptidi e proteine è stato usato per più di un decennio e ci sono stati grandi miglioramenti nei metodi di preparazione del campione, sensibilità di rilevamento, risoluzione spaziale, riproducibilità ed elaborazione dati 3,4. Combinando le informazioni risultanti dalle sezioni istologicamente colorate ed esperimenti MSI, patologi sono in grado di correlare le distribuzioni di composti specifici con le caratteristiche fisiopatologicamente interessanti 5.
I modelli di distribuzione di piccole molecole, tra cui farmaci esogeni 6,7 e loro metaboliti 8-10 sono stati interrogati tramite MALDI-MS tissutale 11. I lipidi sono forse il cla più ampiamente studiatass di composti con immagini MALDI, sia nelle SM 12-17 e MS / MS 18 modalità. L'uso di MALDI MSI per l'imaging piccola molecola è stata limitata da diversi fattori: 1) matrici MALDI sono essi stessi piccole molecole (tipicamente m / z <500), che generano segnali di ioni abbondanti. Questi segnali abbondanti possono sopprimere la ionizzazione di analiti piccole molecole e interferire con la loro rilevazione 19,20. Rivestimento senza solventi matrice 21, matrice sublimazione 22, e la matrice preverniciato MALDI MS 23, tra gli altri, sono stati sviluppati per migliorare l'MSI di piccole molecole.
Le nuove matrici che possono migliorare l'analisi di composti a basso MW sono di grande interesse per la piccola molecola MSI. Queste matrici dovrebbero fornire maggiori segnali di analiti con i segnali a matrice diminuito. Nella modalità positiva di litio, 2,5-diidrossibenzoico acido (DHB) e α-ciano-4-idrossicinnamico acido (CHCA) sono le due matrici MALDI MS comunemente utilizzati per MSI 24 . La matrice ideale sarebbe formare piccoli cristalli, in modo da preservare la localizzazione spaziale degli analiti. DHB tende a formare cristalli più grandi, quindi applicando la matrice mediante sublimazione è stata sviluppata per superare parzialmente questo problema, e ha permesso l'uso di questa matrice per l'imaging sensibile dei fosfolipidi 22,25. 9-Aminoacridine è stato usato per MSI di analiti protici in modalità positiva di litio e 26 nucleotidi e fosfolipidi nella modalità 26-29 di ioni negativi. 2-mercaptobenzotiazolo è stato trovato che invia rilevamento MALDI efficiente di lipidi 30, ed è stato utilizzato per l'imaging di cervello di topo gangliosidi 31. La risoluzione ultra della trasformata di Fourier ioni ciclotrone risonanza (FTICR) spettrometri di massa possono in qualche modo alleviare questo problema, risolvendo i segnali di analiti dai segnali matrice 32. Un altro vantaggio dell'uso di FTICR-MS è che le intensità dei cluster matrice metastabili sono riduzioneed 33, che riduce anche queste interferenze 27.
L'uso di ditranolo (DT; 1,8-diidrossi-9 ,10-dihydroanthracen-9-one) come matrice MALDI per imaging dei tessuti è stato precedentemente riportato 34. In questo lavoro attuale, un protocollo dettagliato è fornito per l'uso del DT per il MSI di lipidi endogeni sulle superfici delle sezioni di tessuto lente bovina, in modalità positiva di litio.
Protocollo
1. Tissue Sezioni
- Flash-congelare i campioni di emissione, una volta raccolte, utilizzando azoto liquido, li spediremo in ghiaccio secco (se richiesto spedizione), e conservarli a -80 ° C fino al sezionamento dei tessuti. (Se si usano campioni commerciali, garantiscono che i campioni sono preparati in questo modo.)
- Tagliare gli organi a una dimensione gestibile per adattarsi al bersaglio MALDI. Tagliare le parti indesiderate dell'organo. Per questo studio qui descritto, lenti di vitello erano decapsulated utilizzando una procedura precedentemente descritta 35 prima sezionamento tessuto.
- Togliere interi organi da -80 ° C freezer e fissarli su una fase di taglio tessuto criogenico. Per fissare un obiettivo di vitello, mettere una o due gocce d'acqua sul palco tessuto taglio di un criostato. Introdurre rapidamente la lente in acqua prima che si solidifica. In alternativa, la temperatura ottimale di taglio (OCT) composti possono anche essere utilizzati per fissare un tessuto sulla fase di taglio. Se si utilizzano i composti ottobre, un miniMal quantità OCT deve essere applicato e si deve prestare attenzione a garantire che le sezioni di tessuto taglio non saranno contaminati con composti dei PTOM che possono interferire con ionizzazione e rilevazione degli analiti 5,36,37.
- Lasciare la temperatura all'interno del criostato equilibrare a -18 ° C. Temperature più fredde o più calde possono essere utilizzati per i tessuti più morbidi o duri, rispettivamente. Poi tagliare il tessuto equatoriale in 20 micron fette spesse. Utilizzare 10-15 micron fette spesse per la maggior parte dei tessuti, tuttavia, a causa della natura fragile del tessuto lente bovina, sono stati usati 20 micron fette spesse. Per tessuto lente bovina, scartare le prime sezioni di tessuto e utilizzare solo le fette che sono vicini o al piano equatoriale.
- Se una lente tessuto oculare viene esposta, utilizzare 1,5 ml di acido formico (98% in purezza, grado LC-MS) per prewet la superficie di un ossido di indio e stagno (ITO) rivestite vetrino.
- Trasferire accuratamente le sezioni di tessuto alla superficie del ITO-covetro ated vetrino microscopico all'interno del criostato. La sezione di tessuto rapidamente disgelo e diventerà saldamente apposte sulla superficie di scorrimento. Solitamente, sezioni di tessuto multipli possono essere montati su uno stesso vetrino ITO rivestite in questo modo.
- Liofilizzare il vetrino per 15 minuti prima dell'applicazione matrice MALDI.
- Per le prove matrice, sciogliere le singole matrici in opportuni solventi. Individuare manualmente 1 ml di ciascuna soluzione di matrice sulla sezione di tessuto. Inoltre, individuare uno standard di calibrazione piccola molecola sul tessuto per la verifica della sensibilità MALDI.
- Aggiungere tre marchi insegnamento al vetrino ITO ricoperto da scrivere sulla superficie non conduttiva del vetrino ITO rivestite con una penna correzione fluido. Prendete un'immagine ottica della diapositiva tessuto utilizzando uno scanner piano e salvarla in un formato appropriato come tiff o jpg.
2. Matrix Coating
2.1. Automated Matrix Coating
- Apsoluzioni matrice strato, che contengono acetonitrile o acetonitrile / acqua miscelata come solventi, automaticamente verso le superfici delle sezioni di tessuto utilizzando un Bruker ImagePrep o una simile spruzzatore matrice elettronico.
- Coprire i bordi della superficie frontale del vetrino ITO rivestite con nastro in modo che la matrice non rivestire i bordi della diapositiva. Questo assicura che i marchi insegnamento sulla superficie opposta possono essere usate per l'allineamento scorrevole tessuto. Non coprire i bordi del vetrino con matrice in cui sono utilizzati come punti di contatto per mantenere la conducibilità elettrica della slitta ITO rivestite.
- Rivestire il vetrino utilizzando venti cicli di rivestimento matrice (2-sec spruzzo, 30 sec incubazione, e 60 sec tempo di asciugatura per ogni ciclo).
2.2. Manuale Matrix Coating
- Se solventi organici (ad esempio cloroformio ed acetato di etile) che sono compatibili con i materiali di produzione del polverizzatore matrice elettronico sono requirosso, utilizzare un aerografo spruzzatore pneumatico assistita per applicare la matrice. Aggiungere la soluzione di matrice preparata al serbatoio del solvente della pistola aerografo e applicare una leggera corrente di azoto pressurizzato per innescare lo spray.
- Coprire i bordi della superficie frontale dei vetrini ITO rivestite con nastro in modo che la matrice non rivestire i bordi della diapositiva. Questo assicura che i marchi insegnamento sulla superficie opposta possono essere usate per l'allineamento scorrevole tessuto. Non coprire i bordi del vetrino con matrice in cui sono utilizzati come punti di contatto per mantenere la conducibilità elettrica della slitta ITO rivestite.
- Dopo che sono stati osservati spray stabili e sottili, spruzzare manualmente la matrice in modo che esso completamente ricopre la sezione di tessuto. Applicare la quantità minima di soluzione di matrice richiesta di bagnare appena la superficie durante ogni ciclo per evitare possibili delocalizzazione analita. In generale, utilizzare circa 10 cicli di spruzzo matrice per rivestire una sezione di tessuto, il numero di cicli èdipendenti dal tipo di tessuto e composizione della matrice.
3. MALDI MS
- Preparare una soluzione di massa diluendo il "ES sintonia Mix" soluzione standard di un fattore 1:200 a 60:40 isopropanolo: acqua (contenente 0,1% di acido formico nella miscela finale).
- Introdurre 2 ml / min della soluzione diluita "ES sintonia Mix" in dual-mode ionizzazione elettrospray (ESI) / sorgente di ioni MALDI sulla massa spettrometro FTICR, dal lato ESI.
- Usare lo strumento FTICR in modalità ESI positiva-ione, con rilevamento a banda larga e una dimensione di acquisizione dati di 1,024 kb / sec. I parametri tipici ESI sono capillare tensione elettrospray, 3.900 V; spruzzare tensione scudo, 3.600 V; gas nebulizzatore (N 2) flusso, 2 L / min; gas secco (N 2) flusso e temperatura, 4 L / min e 200 ° C; skimmer 1 tensione, 15 V, il tempo di volo (TOF), 0.009 sec; gas di collisione (Ar) il flusso, 0,4 L / s; sorgente di tempo di accumulo di ioni, 0.1 sec;e la cella di collisione tempo di accumulo di ioni, 0,2 sec. Regolare i parametri di funzionamento FTICR al fine di massimizzare la sensibilità analitica nell'intervallo massa da m / z 200 a 1.400, pur mantenendo una buona dominio del tempo libero-induzione decadimento (FID) segnali. In genere, i parametri di funzionamento ICR sono tensione sidekick, 8 V; Sidekick tensione di offset, 8 V; eccitazione amplificazione di 10; dell'eccitazione tempo di impulso, 0,01-,015 sec; davanti tensione di placca trappola, 1,5 V; trappola di nuovo tensione di placca, 1,6 V e analizzatore tensione ingresso, -4 V. Dopo una serie di parametri di funzionamento FTICR è stata determinata, acquisire spettri di massa ESI e calibrare lo strumento utilizzando masse di riferimento delle mescole standard nella soluzione "ES sintonia Mix".
- Per sintonizzare lo strumento per il funzionamento MALDI, sciogliere diverse aliquote 1 pl di una terfenadina mista e soluzione standard reserpina nella soluzione matrice ad una concentrazione di 1 mM ciascuno e individuare queste soluzioni direttamente su uno dei tissue sezioni (cioè una sezione di tessuto test) che è stato montato su un vetrino ITO rivestite. Posizionare il vetrino ITO rivestite in un adattatore per slitta tessuto (cioè un particolare obiettivo MALDI) e caricare l'adattatore dal lato MALDI nella duplice sorgente ionica ESI / MALDI. Ottimizzare i parametri operativi MALDI appropriati per la potenza del laser e il numero di colpi laser per accumulo segnale MALDI per ogni scansione massa, ecc parametri di funzionamento MALDI tipici sono: colpi laser, 50, e una tensione MALDI piatto di 300 V.
- Dopo la messa a punto, calibrazione, e ottimizzando lo strumento per esperimenti MALDI-MSI, allineare la posizione fisica di una sezione di tessuto per essere ripreso con la sua immagine ottica registrata all'interno del software di imaging. Utilizzare i tre marchi "correzione-fluido", precedentemente messi sul lato opposto della superficie di scorrimento ITO rivestite (passo 1,9), per questo allineamento utilizzando un metodo di triangolazione tre punti.
- Eseguire un ESI simultanea e MALOperazione di DI in modo che ogni spettro di massa contiene i picchi di massa di riferimento della soluzione "ES sintonia Mix" per il post-acquisizione calibrazione massa interna. Ciò comporterà la misura della massa più accurata durante MALDI MS. Per fare questo, prima attenuare il segnale ESI diminuendo la tensione capillare finché i segnali MALDI dominano spettri mentre i segnali calibranti ESI sono ancora abbastanza elevata per la calibratura massa interna.
- Successivamente, impostare un metodo rastering automatizzato per irraggiamento laser. Definire le regioni di tessuto per essere ripreso e impostare la dimensione del passo raster laser appropriato. Si noti che le dimensioni di passo più piccolo raster forniscono immagini di risoluzione dei tessuti superiori, ma richiedono un tempo significativamente più lungo di massa spettrale acquisizione e più spazio di archiviazione dei dati. Il numero di pixel dell'immagine dipende dal passo raster laser da impostare e le dimensioni del tessuto. Per una tipica lente bovino che ha una dimensione 1 cm2 tessuto, un tessuto immagine è solitamente composto di ca. 5.000 pixel, se viene utilizzato un passo raster laser di 200 micron su uno strumento FTICR. Utilizzare una analisi "posto a caso", perché ciò impedisce polarizzazione basata sulla posizione a causa dell'attenuazione del segnale graduale durante l'esperimento.
4. Analisi dei dati
- Calibrare gli spettri di massa MALDI utilizzando calibrazione interna per il confronto iniziale e selezionare i picchi di MS / MS. De-isotopi e scegliere le cime monoisotopico come precedentemente descritto, utilizzando uno script VBA personalizzato 38.
- Esportare il conseguente liste di picco monoisotopico e inserire i valori m / z misurate nel METLIN 39 e / o le HMDB 40 database metaboloma per corrispondenza di massa con le voci di libreria. Si consideri la (M + H) +, (M + Na) + e (M + K) + ioni durante le ricerche di database, con un errore massa ammissibile di ± 1 ppm.
- Generare immagini MALDI per tutte le entità lipidi rilevati su tutta la sezione di tessuto con imagsoftware e-analysis, con ampiezza di filtro massa di 1 ppm all'apice del picco.
- Una volta che le immagini sono state generate per tutti i valori m / z che corrispondono voci del database, generare immagini per tutte le altre vette, nonché a cercare modelli di distribuzione unici che possono essere studiati più tardi.
5. La conferma delle identità dei lipidi Imaged
- Confermare l'identità dei lipidi alti abbondanza, che hanno caratteristiche frammenti di ioni che possono essere rilevati utilizzando lo strumento FTICR (ad es 184,073 per fosfolipidi), da MALDI-MS/MS. Eseguire MALDI-MS/MS utilizzando collisione indotta dissociazione (CID) direttamente sul tessuto.
- Per tali specie lipidiche che non può essere confermata direttamente MALDI-MS/MS, utilizzare un sistema UPLC accoppiato ad uno spettrometro di massa q-TOF 34.
- Sezionare manualmente aliquote contenenti ~ 10 mg di tessuto dalla zona in cui le specie di interesse è stato localizzato. Posizionare questi aliquote di tessuto in 2provette per centrifuga ml.
- Omogeneizzare ciascuna aliquota tessuto in 250 ml di acqua, utilizzando un mulino miscelatore con due cinque millimetri sfere metalliche in acciaio inox.
- Aggiungere 1 ml di soluzione di cloroformio-metanolo (1:3, v / v) e miscelare i tubi. Successivamente, centrifugare le provette con una microcentrifuga a 12.000 xg per 10 min.
- Raccogliere il surnatante e asciugarle in un concentratore di velocità rotativo vuoto.
- Sciogliere i residui in 100 ml di isopropanolo 30:70: acqua. Iniettare una aliquota di 10 microlitri nella colonna UPLC per la separazione con gradiente di eluizione.
- Utilizzare le condizioni cromatografiche per on-line LC-MS/MS lipidi, che sono stati pubblicati in precedenza 34.
- Generare una corrente di ioni estratto (XIC) cromatogrammi utilizzando i valori teorici m / z, con una finestra di ± 50 ppm intorno masse teoriche.
- Se composti autentici per tali lipidi sono disponibili, corrispondere i tempi di ritenzione dei composti autentici con quelle del corrispondentecorrispondente picchi XIC dai campioni di tessuto. Se i composti sono uguali, i tempi di ritenzione e spettri MS / MS devono corrispondere.
- Se un composto autentico non è disponibile, utilizzare il modello di frammentazione del lipide rilevato per abbinare uno spettro standard di MS / MS da un database metabolome come METLIN o HMDB. Utilizzare interpretazione spettrale massa de novo di determinare una possibile struttura per il lipide.
Risultati
I campioni di tessuto che sono sezionate e montate disgelo sui vetrini ITO rivestite devono essere intatti, senza strappi visibile. Per molti tessuti, montaggio su un vetrino ITO rivestito disgelo tessuto diretta è accettabile. Per alcuni tessuti specifici come obiettivo bovini, ampia lacerazione del tessuto è spesso visto durante il montaggio disgelo diretta viene utilizzato (Figura 1a). Prerivestimento del vetrino ITO con etanolo o acido formico (Figura 1b) aiuta a mantenere l'integrità delle sezioni di tessuto durante il montaggio tessuto.
Sia la scelta della matrice e la selezione del solvente sono fattori importanti che influenzano la qualità degli spettri MALDI. Quando un adeguato spettro MALDI MS viene acquisita dalla sezione di tessuto, lo spettro di massa è generalmente fitto con segnali lipidici nel area di rilevamento massa (Figura 2a). Una matrice ed un solvente devono essere scelti in modo che abbiano polaritàont-size: 14.399999618530273px; line-height:. 28px; "> simile agli analiti di interesse, perché il processo MALDI richiede una soluzione in fase solida dell'analita nei cristalli matrice Generalmente le migliori intensità di segnale analita provengono dall'uso di matrici MALDI con solubilità simile a quello degli analiti desiderati 41,42. figura 2a mostra un esempio di spettro prodotto con un solvente efficiente matrice (70% ACN con 0,01% TFA), e la Figura 2b mostra una scarsa scelta di matrice e solvente (70% MeOH con 0,01% TFA) per ditranolo.
Uno dei vantaggi di un dual-mode ionizzazione elettrospray (ESI) / sorgente di ioni MALDI è che permette l'aggiunta di segnali calibranti ESI acquisendo simultaneamente spettri MALDI senza interferire con il processo di ablazione. Questi segnali calibranti ESI consentono calibrazione massa interna per fornire un'elevata accuratezza di massa con errore di massa <0,5 ppm 38. Come l'ESI segnale della soluzione standard "ES sintonia Mix" può essere un ordine di grandezza più forte dei segnali MALDI degli analiti dal tessuto, i segnali calibranti ESI-derivati devono essere attenuati. Segnali Calibrant dovrebbero essere visibili e di intensità sufficiente per la calibrazione degli spettri, ma non dovrebbero dominare gli spettri.
Una volta che l'insieme di spettri di massa da un esperimento MALDI-MSI è stata acquisita l'immagine per ciascuna delle ioni rilevati può essere generato, con ogni pixel rappresenta un punto di irradiazione laser dalla superficie di una sezione di tessuto. Combinando tutti i singoli pixel con differenti intensità di ioni attraverso la sezione di tessuto da un esperimento MALDI MSI riflette la ionizzazione degli analiti bersaglio all'interno del tessuto 1. Questo può, a sua volta, fornire informazioni sulle concentrazioni relative degli analiti in diverse porzioni della sezione di tessuto (Figura 3b). Si deve prestare attenzione nella lavorazione delDati da molti fattori possono influenzare ciò che si vede e come i dati vengono interpretati. Nella maggior parte degli esperimenti, i dati vengono normalizzati per la corrente ionica totale (TIC) all'interno di ciascuno spettro. Senza questa normalizzazione, zone con una migliore analita-matrice di co-cristallizzazione (ovvero i cosiddetti "hot spot") potrebbero causare segnali più forti per gli analiti e questo sarebbe falsare i dati, fornendo informazioni che potrebbero non correlare bene con le reali concentrazioni relative di gli analiti (figure 3c-3d).
Preparazione del tessuto può anche cambiare radicalmente l'immagine che viene generato. Se il campione è "troppo umido" (cioè troppo solvente è stato applicato), poi gli analiti delocalizzare sul tessuto e molte delle informazioni spaziali verranno persi (Figura 3f). Il metodo di acquisizione dati è anche importante nell'immagine finale ottenuto. Come esperimenti MALDI su sezioni di tessuto non trattati sono intrinsecamente "dirty & #34;, la sensibilità dello strumento può diminuire nel tempo. Per gli esperimenti brevi questa diminuzione non può essere evidente, tuttavia, può essere un problema per esperimenti più lunghi o campioni particolarmente sporchi. Se i dati sono acquisiti linearmente all'interno del campione questo può portare ad una distorsione localizzazione come saranno analizzati specifiche regioni della sezione di tessuto dopo la sensibilità dello strumento è diminuito. Pertanto, con macchie casuali per tutte le acquisizioni di dati è raccomandato. Anche se questo metodo richiede più tempo, aiuta a eliminare o minimizzare questa tendenza nei dati.
Come mostrato nella precedente carta 34, rispetto al CHCA e DHB, DT abilitato il rilevamento di specie lipidiche addizionali, mentre i lipidi rilevati con CHCA e DBH potrebbero ancora essere rilevati.
 >
>
Figura 1. Montaggio e taglio tessuti. Immagini ottiche comparative dei due sezioni di tessuto lenti vitello bovina (spessore 20 micron), senza prewetting acido formico (a), e con prewetting acido formico (b), montate sui vetrini ITO rivestite.
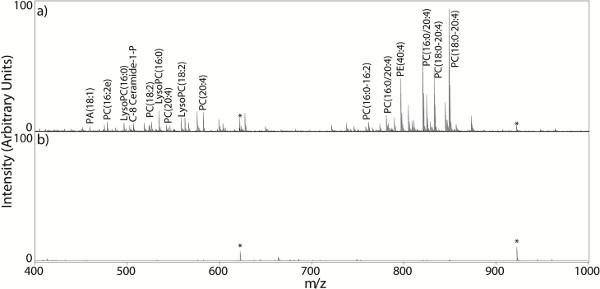
Figura 2. . Mass Spectra MALDI MS spettri acquisiti direttamente da una sezione di tessuto: a) un ideale spettro densamente popolato massa con segnali lipidici (70% ACN con 0,01% TFA), b) uno spettro di massa generato da una sezione di tessuto rivestito con una scarsa scelta di solvente (70% MeOH con il 0,01% TFA). Clicca qui per ingrandire la figura .
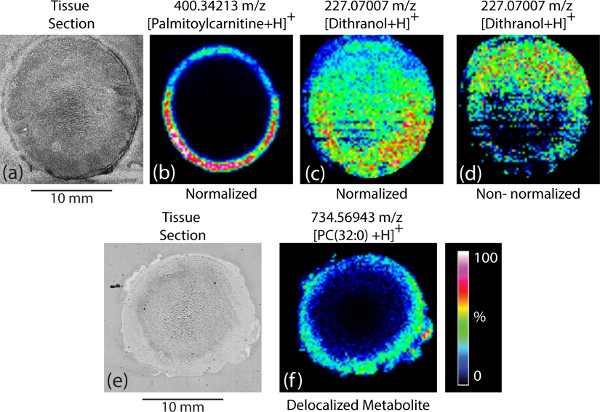
Figura 3. Immagini MALDI MSI immagini rappresentativa MALDI MSI: a) una sezione obiettivo bovina tessuto; b) un'immagine MSI della stessa sezione di tessuto; c) un'immagine MSI che mostra uno ione di sfondo; d) una mappa ione non normalizzata dello stesso ione e). una sezione di tessuto lente bovini e f) una mappa di ioni che mostra un analita parzialmente delocalizzato a causa di overwetting.
Discussione
Le considerazioni più importanti per il successo MALDI MSI sono: 1) la preparazione dei tessuti; 2) scelta della matrice; 3) l'applicazione della matrice e 4) l'interpretazione dei dati e l'analisi. Quando il campione e la matrice sono opportunamente preparate, l'acquisizione dati MS è automatizzato. L'analisi dei dati da questo tipo di esperimento è piuttosto costosa.
Preparazione dei tessuti adeguato è fondamentale per il successo delle sperimentazioni MALDI MSI. La sorgente del tessuto e la movimentazione possono avere un grande impatto sull'analisi finale. I campioni devono essere immediatamente lampeggiare congelati in azoto liquido e conservati a -80 ° C, e non devono essere conservati per un periodo di tempo prolungato, come alcuni metaboliti possono essere instabile anche a -80 ° C.
Per molti tessuti di mammiferi, 10-15 micron fette spesse di tessuto sono stati raccomandati per MALDI MSI. In questi esperimenti, lenti di vitello sono stati tagliati equatoriale in 20 micron di spessore s tessutolices seguenti lente decapsulation utilizzando una procedura precedentemente descritta 35. Le sezioni di tessuto lente vitello bovino spessi sono stati usati perché è stato trovato difficile mantenere l'integrità di una sezione di tessuto lente sia durante il taglio del tessuto e tessuto durante il montaggio. La lente è un tessuto sfericamente simmetrica lungo il suo piano equatoriale, in modo che solo fette, prossimi alla, o presso, il piano equatoriale sono stati raccolti.
A causa della difficoltà nel mantenere l'integrità del tessuto durante il taglio del tessuto e di montaggio, prewetting utilizzando un solvente come etanolo (per le proteine e peptidi 35) o acido formico (per lipidi) può essere utilizzato. Va anche osservato che, per le proteine e di imaging peptide, i campioni sono spesso lavati con un solvente organico per rimuovere piccole molecole compresi lipidi e OCT utilizzati per il montaggio, tuttavia, una fase di lavaggio deve essere fatto solo con solventi che non si dissolvono l' analiti di interesse, e deve essere evitato per la SMAanalisi ll molecola.
La scelta della matrice è fondamentale anche per tutti gli esperimenti MALDI, tuttavia, esecuzione in tessuto matrice può non essere la stessa prestazione matrice con uno standard purificata. Ad esempio, a potenza laser minimo, DT generato abbondanti segnali di fondo DT-legati dalle macchie matrice senza tessuto e questi segnali sono stati assegnati come oligomeri DT ei corrispondenti addotti di sodio e di potassio, tuttavia, sul tessuto, molti di questi segnali erano non osservato, indicando che il test di differenti matrici sul campione specifico di scelta può essere importante nella scelta della matrice appropriata per una data analisi MALDI MSI. DT è raramente utilizzato per l'analisi dei lipidi a causa della bassa alta riportato generato dalla matrice, tuttavia, in confronto con DHB e CHCA per la profilatura in tessuto di lipidi tramite MALDI-MS FTICR, DT ha prodotto risultati favorevoli. Così questa matrice potrebbe essere una matrice potenzialmente utile per MALDI imaging dei tessuti che utilizza questo strumento 34.
Efficiente co-cristallizzazione della matrice e l'analita è un prerequisito per l'alta sensibilità di analisi MALDI-MS. Così la solubilità in fase solida di un analita nella matrice è importante nel processo MALDI 41,42. Matrici MALDI con solubilità simili a quelle degli analiti desiderati sono stati segnalati per produrre forti intensità di segnale analita. Poiché DT è un acido organico debole, così come un composto organico molto idrofobico, basato sulla classica teoria Brønsted-Lowry neutralizzazione acido-base 43 e la teoria di solubilità, si prevede di favorire la ionizzazione di carica positiva e meno polare composti. Infatti, abbiamo trovato che i lipidi polari prevalere i composti identificati nella modalità positiva ioni quando DT è stato utilizzato come matrice.
I solventi utilizzati per la preparazione di una soluzione di matrice svolgono un ruolo importante in analisi dei tessuti diretto tramite MALDI-MS. Il pH è stato implicato comefattore importante dell'efficacia di una matrice, e TFA è un additivo comune usato con CHCA e DHB. Tuttavia, con DT, l'aggiunta di un modificatore di acido o base aveva poco effetto sui dati risultanti. A causa della idrofobicità di DT e la sua limitata solubilità in solventi polari, si raccomanda un solvente organico lipofilo. Nell'analizzare lipidi, una miscela di cloroformio-metanolo (2:1, v: v) con acido formico 1%, che è un solvente organico tipico per estrazione dei lipidi, è stato utilizzato. Abbiamo ipotizzato che questo fornisce una migliore co-cristallizzazione di lipidi con DT. La natura lipofila del solvente può anche impedire la solubilizzazione di altri composti quali proteine e sali, come dimostrato nel precedente liquido superficie di aspirazione analisi di spettrometria di massa (MS) LESA-esperimenti 44. Ciò permetterebbe di migliorare la cristallizzazione della matrice e lipidi con meno contaminanti. Il sistema solvente che viene selezionato per analisi dovrebbe massimizzare la solubilità della matrice e la desianaliti rossi (lipidi) riducendo al minimo la solubilità di contaminanti indesiderati (sali e proteine / peptidi).
Il rivestimento della matrice sulla superficie del tessuto deve essere il più uniforme possibile. Per massimizzare la risoluzione spaziale dell'immagine, la dimensione dei cristalli dovrebbe essere il più piccolo possibile 45. Utilizzando uno spruzzatore matrice elettronica, la matrice può essere rivestita in modo uniforme e riproducibile con una piccola dimensione dei cristalli. Questo è il metodo preferito nel nostro laboratorio. E 'molto preferisce applicazione manuale in quanto riduce le dimensioni spot, omogeneità e riproducibilità. Tuttavia, molti dei solventi che sono utili per MSI di piccole molecole non sono compatibili con i materiali utilizzati nella produzione della matrice spruzzatore automatico. Anche se non ancora implementata nel nostro laboratorio, applicazione basata matrice sublimazione è stato raccomandato per l'analisi dei lipidi 22. Questo metodo fornisce una migliore (cioè ridotto) dimensione dello spot, omogeneità e riproduzionebilità e questo probabilmente dovrebbe essere il metodo di scelta quando la matrice selezionata è amabile a questo metodo.
Per il rivestimento di matrici contenenti solventi incompatibili con lo spruzzatore matrice automatizzato (compresi cloroformio), un metodo alternativo di rivestimento matrice è di usare una pistola aerografo. Per queste soluzioni matrice, abbiamo utilizzato uno spruzzatore aerografo assistita pneumaticamente. Sebbene l'uso della pistola spazzola di aria non è un metodo ideale, può essere l'unico metodo che può essere utilizzato per solventi e matrici che sono incompatibili con gli altri metodi, e con la formazione e esperienza può generare rivestimento matrice molto uniforme. Quando si applica manualmente la soluzione di matrice, solo la quantità minima di liquido deve essere applicato durante ogni ciclo di bagnare appena la superficie del tessuto. Troppo liquido potrebbe potenzialmente delocalizzare analiti a causa dei solventi organici. Ci sono problemi di riproducibilità con applicazione manuale ed esperienza nel rivestimento diapositiva con questo metodo is essenziale per il successo. A causa della natura manuale dell'applicazione matrice pistola aerografo, bisogna fare attenzione per garantire che venga fatto un rivestimento uniforme; un rivestimento disomogeneo può portare a dati inclinati, che è rappresentativo del rivestimento matrice e non la localizzazione analita.
Nel condurre esperimenti MALDI MSI, identificazione iniziale degli analiti rilevati è di solito basato sulla metabolome database di ricerca delle masse misurate precisi che sono di solito disponibili solo quando uno strumento ad alta risoluzione viene utilizzato 38. La doppia sorgente di ioni ESI / MALDI sulla spettrometro di massa Apex-Qe 12-Tesla ibrido quadrupolo-FTICR consente l'aggiunta di segnali ESI-generati per l'uso come i picchi calibrante massa interne a ciascun spettro di massa MALDI, senza intaccare il desorbimento MALDI / processo di ionizzazione. L'uso di taratura massa interna è cruciale per la precisione di misura di alta massa MALDI-FTICR. Calibrazione esterna non può tenere pienamente contoeffetti di carica spaziale all'interno della cellula ICR 46-48. La messa a punto e calibrazione del FTICR è fondamentale per il successo. Su questo tipo di strumento un parametro chiamato "tempo di volo" (TOF), che è il tempo necessario per uno ione di viaggiare dalla cella di collisione all'analizzatore (la cella ICR) è uno dei più importanti definito dall'utente impostazioni che influenzano la sensibilità di rilevazione di uno strumento FTICR. All'interno di un determinato intervallo di massa (cioè m / z 200-1,400), un TOF minore favorisce la rilevazione dei minori ioni m / z e TOF maggiore favorisce la rilevazione di maggiori ioni m / z. Così, per il rilevamento ad alta sensibilità di entrambi gli ioni basse e alte m / z nell'intervallo di massa di rilevamento, un valore TOF di 9 msec è desiderabile.
Per un esperimento pratico, un trade-off deve essere fatta tra dimensioni ragionevoli acquisizione dati, risoluzione di massa, e il tempo speso per l'acquisizione dei dati di imaging MS. Per un esperimento FTICR MS, la dimensione del file di dati acquisiti e la risolu massasulla dipendono dalla dimensione di acquisizione dati del decadimento di induzione libera segnale (FID). Una maggiore dimensione acquisizione dati comporterà risoluzione massa maggiore e una maggiore dimensione di file di dati. Tuttavia, una maggiore dimensione di acquisizione dati provoca anche un MS velocità di scansione più lenta. Come un trade-off, è consigliabile una taglia acquisizione di dati di 1,024 kb / sec essere utilizzato sul FTICR. Una dimensione di acquisizione dati inferiore e una corrispondente risoluzione massa inferiore non consentono la separazione di alcune specie ioniche isobariche.
Il laser raster passo deve essere scelto in modo che sia abbastanza piccolo per fornire una buona risoluzione pixel per immagini MS degli analiti di interesse, tuttavia, una dimensione molto piccola passo raster può rendere il file di dati ingestibile considerando un file di dati di imaging MS è composto da migliaia di spettri di massa MALDI. Data la relativamente grandi dimensioni delle lenti bovini, ca. 1,2 -1,5 cm di diametro, abbiamo utilizzato un passo raster di 250 micron. Utilizzando un dato acqu 1.024 kb / secdimensioni isition e un micron raster grandezza 250 step, il nostro set di dati campione era di circa 60 Gb. Durante l'acquisizione dati, un'analisi posto a caso deve essere utilizzato, come questo impedisce polarizzazione basata sulla posizione a causa dell'attenuazione del segnale graduale, tuttavia, posto a caso richiede tempo significativamente più lungo per acquisire i dati.
Poiché i dataset MALDI MSI acquisite sul nostro strumento FTICR MS includono i dati di massa accurati, il estratta m / z può essere ricercato sui database metaboloma come METLIN e / o HMDB. Utilizzando una finestra di ± 1 ppm le assegnazioni iniziali di molti segnali di ioni a metaboliti sono di alta fiducia. Tuttavia, poiché molte specie hanno la stessa formula chimica, la conferma della ID deve spesso essere fatto utilizzando mezzi alternativi. Così, gli esperimenti MS / MS, e il confronto del MS / MS spettri con i dati precedentemente pubblicati devono essere eseguite per un'identificazione sicura. Spettri MALDI-MS/MS volte può essere acquisito direttamente sullo strumento FTICR, ma per MAmolecole lipidiche ny, le abbondanze e / o l'efficienza di ionizzazione non sono sufficienti per ottenere utili dati MS / MS, e sono necessari arricchimento e purificazione prima di LC-MS/MS. In queste analisi, la maggior parte dei lipidi osservati erano fosfolipidi polari, e sono stati assegnati come PC, PES, SMS, PS, PG, PA, e fosfati di ceramide (CerPs); altre molecole lipidiche identificati includono lipidi steroli, acilcarnitine, nonché di gliceridi. Sulla base delle proprietà del solvente e matrice, questo è prevedibile. La MS / MS spettri di PC contengono un picco significativo a m / z 184,073, che è stato attribuito al gruppo di testa polare PC, fosfocolina, nonché ulteriori informazioni strutturalmente importante, che può dare l'identificazione univoca delle molecole. Inoltre, utilizzando questo metodo, molti lipidi steroli vengono rilevati, tuttavia, la maggior parte non può essere inequivocabilmente assegnato a identità uniche, anche con i dati MS / MS. Forti addotti potassio generalmente predominano gli spettri, ma protonato e sodiated addotti possono essere rilevati.
Poiché il processo di MS MALDI è solo in grado di fornire informazioni relativa abbondanza sulla base dell'efficienza di ionizzazione locale all'interno di ogni pixel, la cura deve essere presa quando si interpretano i dati MALDI MSI senza isotopo stabile standard interni marcati 49. Inoltre, per la fiducia nei risultati della localizzazione, conferma di eventuali distribuzioni identificati deve essere eseguita con metodi alternativi. LC-MS/MS Esibirsi su campioni di tessuto sezionati per la conferma è raccomandato.
Sulla base delle masse accurate misurati nelle formule chimiche low range di massa può essere generato per un determinato m / z; entro un errore massa 1 ppm e consentendo un numero illimitato di C, H, O e N, e un massimo di 2 S e P 2 spesso solo una singola composizione elementare è possibile. Questo m / z può essere cercato anche contro il HMDB ei database METLIN, che possono produrre potenziali composti candidati. Purtroppo,sul FTICR MS il cut-off basso-massa è di ca. 130 Da, che può rendere difficile svolgere direttamente MS / MS. Così, conferma utilizzando un altro sistema è spesso richiesto.
Esperimenti LC-MS/MS Q-TOF sono comunemente condotti su campioni di tessuto che sono stati dissezionati manualmente da zone specifiche del tessuto di interesse. Utilizzando il metodo di estrazione dei lipidi descritto, XICs possono essere generati per i composti target e MS / MS possono essere acquistati per la conferma. In entrambi i casi rispetto ad una norma autentica o de novo delucidazione strutturale è necessaria prima che ci possa essere un incarico chimico fiducioso.
Dithranol è stato usato per esplorare modelli di distribuzione dei lipidi in lente di vitello e testato anche sul ratto fegato, cuore e tessuti renali 34. MALDI MSI può essere utilizzato per la diagnosi degli stati di malattia umana ed è già in uso per analisi patologiche 50. Con lo sviluppo di più robusto e rapido tecnologie per MALDI MSI, la localizzazione spaziale dei composti specifici possono essere informazioni utili per un patologo. Una volta che un analita può essere ordinariamente ripreso con un metodo basato MALDI-MSI, può essere usato per scopi diagnostici. Infatti, l'imaging tessuto potrà essere eseguita in ambiente ospedaliero, con uno strumento situato accanto alla sala operatoria, dove, come è stato già dimostrato 51, potrebbe essere utilizzata per determinare con precisione i margini di tumori.
Divulgazioni
Non abbiamo nulla da rivelare.
Riconoscimenti
Gli autori vorrebbero riconoscere Genome Canada e Genome British Columbia per il finanziamento della piattaforma e supporto. Ringraziamo anche il Dott. Carol E. Parker per la revisione critica del manoscritto assistenza e la modifica. CHL grazie anche British Columbia Proteomica di rete per il supporto.
Materiali
| Name | Company | Catalog Number | Comments |
| Rat Liver | Pel-Freez Biologicals | 56023-2 | |
| Bovine Calf Lens | Pel-Freez Biologicals | 57114-2 | Sample should be decapsulated29 before use |
| Dithranol (DT) | Sigma-Aldrich | 10608 | MALDI Matrix |
| α-Cyano-4-hydroxy-cinnamic Acid (CHCA) | Sigma-Aldrich | 70990 | MALDI Matrix |
| 2,5-Dihydroxybenzoic Acid (DHB) | Sigma-Aldrich | 85707 | MALDI Matrix |
| Reserpine | Sigma-Aldrich | 83580 | |
| Terfenadine | Sigma-Aldrich | T9652 | |
| Formic Acid | Sigma-Aldrich | 14265 | |
| Ammonium Formate | Sigma-Aldrich | 14266 | |
| Ammonium Hydroxide | Sigma-Aldrich | 320145 | |
| Trifluoroacetic Acid (TFA) | Sigma-Aldrich | 302031 | |
| Water | Sigma-Aldrich | 39253 | |
| Methanol | Sigma-Aldrich | 34860 | |
| Acetonitrile | Sigma-Aldrich | 34967 | |
| Ethyl Acetate | Sigma-Aldrich | 34972 | |
| Isopropanol | Sigma-Aldrich | 34965 | |
| Chloroform | Sigma-Aldrich | 366927 | |
| Acetone | Sigma-Aldrich | 34850 | |
| Ethanol | Commercial Alcohols | 95% | |
| ES Tuning Mix | Agilent Technologies | G2431A | |
| ITO Coated Glass Slides | Hudson Surface Technology | PSI1207000 | Ensure that samples are placed on the electrically conductive side |
| Wite-Out Shake-N-Squeeze Correction Pen | Bic | WOSQP11 | |
| Airbrush Sprayer | Iwata | Eclipse HP-CS | |
| ImagePrep | Bruker | 249500-LS | |
| MALDI adapter | Bruker | 235380 |
Riferimenti
- Chaurand, P., Stoeckli, M., Caprioli, R. M. Direct Profiling of Proteins in Biological Tissue Sections by MALDI Mass Spectrometry. Anal. Chem. 71, 5263-5270 (1999).
- Caprioli, R. M., Farmer, T. B., Gile, J. Molecular Imaging of Biological Samples. Localization of Peptides and Proteins Using MALDI-TOF MS. Anal. Chem. 69, 4751-4760 (1997).
- Amstalden van Hove, E. R., Smith, D. F., Heeren, R. M. A. A concise review of mass spectrometry imaging. J. Chromatogr. A. 1217, 3946-3954 (2010).
- Norris, J. L., Caprioli, R. M. Analysis of Tissue Specimens by Matrix-Assisted Laser Desorption/Ionization Imaging Mass Spectrometry in Biological and Clinical Research. Chem. Rev. Feb 11, (2013).
- Walch, A., Rauser, S., Deininger, S. -. O., Höfler, H. MALDI imaging mass spectrometry for direct tissue analysis: a new frontier for molecular histology. Histochem. Cell Biol. 130, 421-434 (2008).
- Hsieh, Y., et al. Matrix-assisted laser desorption/ionization imaging mass spectrometry for direct measurement of clozapine in rat brain tissue. Rapid Commun. Mass Spectrom. 20, 965-972 (2006).
- Trim, P. J., et al. Matrix-assisted laser desorption/ionization-ion mobility separation-mass spectrometry imaging of vinblastine in whole body tissue sections. Anal. Chem. 80, 8628-8634 (2008).
- Khatib-Shahidi, S., Andersson, M., Herman, J. L., Gillespie, T. A., Caprioli, R. M. Direct molecular analysis of whole-body animal tissue sections by imaging MALDI mass spectrometry. Anal. Chem. 78, 6448-6456 (2006).
- Atkinson, S. J., Loadman, P. M., Sutton, C., Patterson, L. H., Clench, M. R. Examination of the distribution of the bioreductive drug AQ4N and its active metabolite AQ4 in solid tumours by imaging matrix-assisted laser desorption/ionisation mass spectrometry. Rapid Commun. Mass Spectrom. 21, 1271-1276 (2007).
- Drexler, D. M., et al. Utility of imaging mass spectrometry (IMS) by matrix-assisted laser desorption ionization (MALDI) on an ion trap mass spectrometer in the analysis of drugs and metabolites in biological tissues. J. Pharmacol. Toxicol. Methods. 55, 279-288 (2007).
- Prideaux, B., Stoeckli, M. Mass spectrometry imaging for drug distribution studies. J. Proteomics. 75, 4999-5013 (2012).
- Sugiura, Y., Setou, M. Imaging Mass Spectrometry for Visualization of Drug and Endogenous Metabolite Distribution: Toward In Situ Pharmacometabolomes. J. Neuroimmune Pharmacol. 5, 31-43 (2009).
- Garrett, T. J., Yost, R. A. Analysis of intact tissue by intermediate-pressure MALDI on a linear ion trap mass spectrometer. Anal. Chem. 78, 2465-2469 (2006).
- Woods, A. S., Jackson, S. N. Brain tissue lipidomics: direct probing using matrix-assisted laser desorption/ionization mass spectrometry. AAPS J. 8, 391-395 (2006).
- Cha, S., Yeung, E. S. Colloidal graphite-assisted laser desorption/ionization mass spectrometry and MSn of small molecules. 1. Imaging of cerebrosides directly from rat brain tissue. Anal. Chem. 79, 2373-2385 (2007).
- Burnum, K. E., et al. Spatial and temporal alterations of phospholipids determined by mass spectrometry during mouse embryo implantation. J. Lipid Res. 50, 2290-2298 (2009).
- Veloso, A., et al. Anatomical distribution of lipids in human brain cortex by imaging mass spectrometry. J. Am. Soc. Mass Spectrom. 22, 329-338 (2011).
- Tanaka, H., et al. Distribution of phospholipid molecular species in autogenous access grafts for hemodialysis analyzed using imaging mass spectrometry. Anal. Bioanalyt. Chem. 400, 1873-1880 (2011).
- Lou, X., van Dongen, J. L., Vekemans, J. A., Meijer, E. W. Matrix suppression and analyte suppression effects of quaternary ammonium salts in matrix-assisted laser desorption/ionization time-of-flight mass spectrometry: an investigation of suppression mechanism. Rapid Comm. Mass Spectrom. 23, 3077-3082 (2009).
- Knochenmuss, R., Karbach, V., Wiesli, U., Breuker, K., Zenobi, R. The matrix suppression effect in matrix-assisted laser desorption/ionization: application to negative ions and further characteristics. Rapid Commun. Mass Spectrom. 12, 529-534 (1998).
- Puolitaival, S. M., Burnum, K. E., Cornett, D. S., Caprioli, R. M. Solvent-free matrix dry-coating for MALDI imaging of phospholipids. J. Am. Soc. Mass Spectrom. 19, 882-886 (2008).
- Hankin, J. A., Barkley, R. M., Murphy, R. C. Sublimation as a Method of Matrix Application for Mass Spectrometric Imaging. J. Am. Soc. Mass Spectrom. 19, 1646-1652 (2007).
- Grove, K. J., Frappier, S. L., Caprioli, R. M. Matrix pre-coated MALDI MS targets for small molecule imaging in tissues. J. Am. Soc. Mass Spectrom. 22, 192-195 (2011).
- Fuchs, B., Süss, R., Schiller, J. An update of MALDI-TOF mass spectrometry in lipid research. Prog. Lipid Res. 49, 450-475 (2010).
- Murphy, R. C., Hankin, J. A., Barkley, R. M., Zemski Berry, K. A. MALDI imaging of lipids after matrix sublimation/deposition. Biochim. Biophys. Acta. 1811, 970-975 (2011).
- Vermillion-Salsbury, R. L., Hercules, D. M. 9-Aminoacridine as a matrix for negative mode matrix-assisted laser desorption/ionization. Rapid Commun. Mass Spectrom. 16, 1575-1581 (2002).
- Hu, C., et al. Analytical strategies in lipidomics and applications in disease biomarker discovery. J. Chromatogr. B Analyt. Technol. Biomed. Life Sci. 877, 2836-2846 (2009).
- Miura, D., et al. Ultrahighly sensitive in situ metabolomic imaging for visualizing spatiotemporal metabolic behaviors. Anal. Chem. 82, 9789-9796 (2010).
- Cerruti, C. D., Benabdellah, F., Laprevote, O., Touboul, D., Brunelle, A. MALDI Imaging and Structural Analysis of Rat Brain Lipid Negative Ions with 9-Aminoacridine Matrix. Anal. Chem. 84, 2164-2171 (2012).
- Astigarraga, E., et al. Profiling and Imaging of Lipids on Brain and Liver Tissue by Matrix-Assisted Laser Desorption/Ionization Mass Spectrometry Using 2-Mercaptobenzothiazole as a Matrix. Anal. Chem. 80, 9105-9114 (2008).
- Whitehead, S. N., et al. Imaging mass spectrometry detection of gangliosides species in the mouse brain following transient focal cerebral ischemia and long-term recovery. PloS one. 6, e20808 (2011).
- Cornett, D. S., Frappier, S. L., Caprioli, R. M. MALDI-FTICR imaging mass spectrometry of drugs and metabolites in tissue. Anal. Chem. 80, 5648-5653 (2008).
- Deininger, S. O., et al. Normalization in MALDI-TOF imaging datasets of proteins: practical considerations. Anal. Bioanalyt. Chem. 401, 167-181 (2011).
- Le, C. H., Han, J., Borchers, C. H. Dithranol as a MALDI matrix for tissue imaging of lipids by Fourier transform ion cyclotron resonance mass spectrometry. Anal. Chem. 84, 8391-8398 (2012).
- Han, J., Schey, K. L. MALDI Tissue Imaging of Ocular Lens α-Crystallin. Invest. Ophthalmol. Vis. Sci. 47, 2990-2996 (2006).
- Schwartz, S. A., Reyzer, M. L., Caprioli, R. M. Direct tissue analysis using matrix-assisted laser desorption/ionization mass spectrometry: practical aspects of sample preparation. J. Mass Spectrom. 38, 699-708 (2003).
- Chen, Y., et al. Imaging MALDI mass spectrometry of sphingolipids using an oscillating capillary nebulizer matrix application system. Meth. Mol. Biology. 656, 131-146 (2010).
- Han, J., et al. Towards high throughput metabolomics using ultrahigh field Fourier transform ion cyclotron resonance mass spectrometry. Metabolomics. 4, 128-140 (2008).
- Smith, C. A., et al. METLIN: a metabolite mass spectral database. Ther. Drug Monit. 27, 747-751 (2005).
- Wishart, D. S., et al. HMDB: a knowledgebase for the human metabolome. Nucleic Acids Res. 37, D603-D610 (2009).
- Hoteling, A. J., Erb, W. J., Tyson, R. J., Owens, K. G. Exploring the importance of the relative solubility of matrix and analyte in MALDI sample preparation using HPLC. Anal. Chem. 76, 5157-5164 (2004).
- Hoteling, A. J., Mourey, T. H., Owens, K. G. Importance of solubility in the sample preparation of poly(ethylene terephthalate. for MALDI TOFMS. Anal. Chem. 77, 750-756 (2005).
- Shroff, R., Rulísek, L., Doubsky, J., Svatos, A. Acid-base-driven matrix-assisted mass spectrometry for targeted metabolomics. Proc. Nat. Acad. Sci. U.S.A. 106, 10092-10096 (2009).
- Eikel, D., et al. Liquid extraction surface analysis mass spectrometry (LESA-MS) as a novel profiling tool for drug distribution and metabolism analysis: the terfenadine example. Rapid Comm. Mass Spectrom. 25, 3587-3596 (2011).
- Sadeghi, M., Vertes, A. Crystallite size dependence of volatilization in matrix-assisted laser desorption ionization. Appl. Surf. Sci. 127 - 129, 226-234 (1998).
- O'Connor, P. B., Costello, C. E. Internal Calibration on Adjacent Samples (InCAS) with Fourier Transform Mass Spectrometry. Anal. Chem. 72, 5881-5885 (2000).
- Jing, L., Amster, I. J. An improved calibration method for the matrix-assisted laser desorption/ionization-Fourier transform ion cyclotron resononance analysis of 15N-metabolically- labeled proteome digests using a mass difference approach. Eur. J. Mass Spectrom. 18, 269-277 (2012).
- Zhang, L. -. K., Rempel, D., Pramanik, B. N., Gross, M. L. Accurate mass measurements by Fourier transform mass spectrometry. Mass Spectrom. Rev. 24, 286-309 (2005).
- Clemis, E. J., et al. Quantitation of spatially-localized proteins in tissue samples using MALDI-MRM imaging. Anal. Chem. 84, 3514-3522 (2012).
- Schwamborn, K., Caprioli, R. M. Molecular imaging by mass spectrometry--looking beyond classical histology. Nat. Rev. Cancer. 10, 639-646 (2010).
- Oppenheimer, S. R., Mi, D., Sanders, M. E., Caprioli, R. M. Molecular analysis of tumor margins by MALDI mass spectrometry in renal carcinoma. J. Proteome Res. 9, 2182-2190 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon