Method Article
le développement et l’application de tests biophysiques pour l’évaluation de la formation de complexes ternaires induite par la protéolyse ciblant les chimères (PROTACS)
Dans cet article
Résumé
Nous décrivons ici des protocoles pour la caractérisation biophysique de la formation de complexes ternaires induite par protéolyse ciblant les chimères (PROTACS) qui impliquent les ubiquitine ligases Von Hippel-Lindau, E3 ligase (VHL) et Cereblon (CRBN). Les méthodes biophysiques illustrées ici comprennent la résonance plasmonique de surface (SPR), l’interférométrie de biocouche (BLI) et la calorimétrie de titrage isotherme (ITC).
Résumé
Les ligases E3 et les protéines ciblées pour la dégradation peuvent être induites à former des complexes par des molécules hétérobifonctionnelles dans un processus en plusieurs étapes. La cinétique et la thermodynamique des interactions impliquées contribuent à l’efficacité de l’ubiquitination et à la dégradation de la protéine qui en résulte. Les techniques biophysiques telles que la résonance plasmonique de surface (SPR), l’interférométrie des biocouches (BLI) et la calorimétrie de titrage isotherme (ITC) fournissent des informations précieuses qui peuvent être utilisées dans l’optimisation de ces interactions. À l’aide de deux systèmes modèles, une boîte à outils de dosage biophysique pour comprendre la coopérativité de la formation de complexes ternaires et l’impact de « l’effet crochet » sur la cinétique de liaison a été établie. Dans un cas, une molécule de protéolyse ciblant la chimère (PROTAC) qui induisait la formation d’un complexe ternaire entre Brd4BD2 et VHL a été évaluée. La molécule hétérobifonctionnelle, MZ1, a des affinités nM à la fois pour la protéine Brd4BD2 (SPR K D = 1 nM, ITC K D = 4 nM) et pour le complexe VHL (SPR K D = 29 nM, ITC K D = 66 nM). Pour ce système, des tests SPR, BLI et ITC robustes ont été développés qui ont reproduit les résultats publiés démontrant la coopérativité de la formation de complexes ternaires. Dans l’autre cas, une molécule induisant des complexes ternaires entre une protéine de 46,0 kDa, PPM1D, et le cerveau [CRBN (319-442)] a été étudiée. La molécule hétérobifonctionnelle, BRD-5110, a un SPR KD = 1 nM pour PPM1D mais une liaison beaucoup plus faible contre le complexe CRBN tronqué (319-442) (SPR KD= ~ 3 μM). Dans ce cas, la liaison pour le CRBN dans le SPR n’était pas saturable, ce qui a entraîné un « effet d’hameçon ». Les exigences en matière de débit et de réactifs pour le SPR, le BLI et l’ITC ont été évaluées, et des recommandations générales pour leur application aux projets PROTAC ont été fournies.
Introduction
La polyubiquitination des protéines dans la cellule est un processus étroitement régulé qui implique des enzymes de la famille des ubiquitines ligases 1,2. Les enzymes terminales de la voie sont les ubiquitine ligases E3 qui attachent de manière covalente les molécules d’ubiquitine à leurs partenaires de liaison aux protéines3. La polyubiquitination de ces partenaires de liaison aux protéines les cible pour la dégradation protéolytique par le protéasome4. Ce système fait partie du processus d’homéostasie des protéines qui a été exploité thérapeutiquement pour induire la dégradation des protéines impliquées dans la maladie5. Les petites molécules qui induisent l’interaction entre les ligases de l’ubiquitine E3, telles que la ligase E3 de Von Hippel-Lindau (VHL) ou le cerveau (CRBN), sont généralement composées d’une ogive de liaison à la ligase E3 reliée par un liant flexible à une ogive qui se lie à la protéine ciblée pour la dégradation. Ces molécules hétérobifonctionnelles sont communément appelées chimères ciblant la protéolyse ou PROTACS6.
Le développement de PROTACS consiste à évaluer la capacité des molécules à induire la dégradation des protéines dans les cellules. De nombreux systèmes de dosage cellulaire ont été développés pour surveiller l’interaction induite entre la protéine cible et les composants de la ligase E3, tels que VHL ou CRBN, lors du traitement des cellules avec une molécule PROTAC. L’un de ces tests cellulaires, le système nanoluc-Halotag7, implique une ligase E3 fusionnée à l’accepteur Halotag et une protéine cible marquée avec un donneur nanoluc. La formation d’un complexe ternaire rapproche le donneur nanoluc et l’accepteur Halotag, ce qui permet le transfert d’énergie du donneur à l’accepteur, ce qui entraîne l’émission de lumière. Les variations de ce système peuvent être utilisées pour évaluer la perméabilité cellulaire des molécules PROTACS8 ou les changements dans le niveau relatif d’ubiquitination de la protéine cible9. Bien que ces systèmes cellulaires soient essentiels à l’optimisation de PROTACS, la formation de complexes entre les ligases E3 et les protéines ciblées pour la dégradation est un processus en plusieurs étapes10,11. La cinétique et la thermodynamique des interactions binaires et ternaires impliquées contribuent à l’ubiquitination de l’efficacité et à la dégradation de la protéine12,13,14 qui en résulte.
On y décrit des protocoles qui peuvent être adaptés pour la caractérisation biophysique de la formation de complexes ternaires induite par PROTACS à l’aide de la résonance plasmonique de surface (SPR), de l’interférométrie de biocouche (BLI) et de la calorimétrie de titrage isotherme (ITC). Les protocoles SPR et ITC pour la molécule MZ1 PROTAC qui induit la formation de complexes ternaires entre Brd4BD2 et VHL dérivés des rapports de littérature13,15 et décrits ici ont permis de récapituler les résultats rapportés avec quelques modifications des procédures rapportées, qui seront discutées. Une description d’un test BLI utilisé pour évaluer la formation de complexes ternaires entre MZI, Brd4BD2 et VHL est incluse dans ce rapport. Les mesures d’affinité de BLI étaient cohérentes avec celles observées dans le SPR et l’ITC. Un protocole précédemment publié dans lequel un test SPR a été développé pour évaluer la formation de complexes ternaires induite par PROTAC entre PPM1D, une protéine phosphatase Ser/Thr dont l’expression est induite de manière dépendante de p5316, et CRBN est également décrit. Dans ce cas, la molécule PROTAC a une affinité nanomolaire pour PPM1D mais seulement une affinité micromolaire pour CRBN. Dans ce cas, la liaison de la molécule PROTAC au CRBN n’est pas saturable, ce qui entraîne l'« effet crochet » couramment observé. L’effet d’hameçon est une propriété de trois systèmes corporels dans lesquels il y a deux espèces qui peuvent former un complexe hétérotrimérique lorsque les deux sont liées à une molécule de pontage (Figure 1)17. L’effet d’hameçon est observé lorsque l’espèce pontière est en concentration excessive par rapport aux deux autres espèces. L’état résultant est un état dans lequel les interactions binaires surpassent les interactions ternaires. Les systèmes où l’effet d’hameçon est observé nécessitent des considérations spécifiques sur la conception expérimentale discutées dans le présent rapport. Des concepts généraux et des exigences en réactifs pour l’évaluation de l’utilisation de tests biophysiques pour l’évaluation de la formation de complexes ternaires induite par PROTAC sont fournis.
Protocole
Toutes les protéines ont été surexprimées dans E. coli avec un bon rendement et une bonne pureté (>80%) selon les protocoles de la littérature18. La biotinylation a été réalisée à l’aide d’une réaction catalysée par BirA18. Toutes les petites molécules ont été préparées à 1 mM de solutions mères dans du DMSO à 100 %. Les procédures décrites dans le présent document ne nécessitent pas d’équipement de sécurité ou de précautions de laboratoire spécialisés. L’équipement de protection individuelle (EPI) standard de laboratoire doit être utilisé (c’est-à-dire une blouse de laboratoire, des lunettes de sécurité et des gants).
Les protéines utilisées dans cette étude sont énumérées ci-dessous :
VHL : complexe VHL(53-213)/ElonginB (1-104)/ElonginC(17-112) biotinylé avec Avi-tag à l’extrémité C de l’ElonginB.
Brd4BD2 : Brd4BD2 non étiqueté (333-460)
CRBN : CRBN biotinylé(319-442) avec Avi-tag à l’extrémité N
PPM1D : PPM1D(1-420) non marqué ou double marqué His8 à l’extrémité N
Les petites molécules utilisées dans cette étude sont énumérées ci-dessous :
MZ1 (MW = 1002,6 Da) : PROTAC qui se lie à VHL et Brd4BD2
BRD-2512 (MW = 841,4 Da) : CRBN KD ~3 μM, ne se lie pas à PPM1D
BRD-5110 (MW = 872,0 Da) : CRBN K D ~3 μM, PPM1D KD = 1-2 nM
BRD-4761 (MW = 476,6 Da) : ne se lie pas à CRBN, PPM1D KD = 1-2 nM
1. Méthode 1 : ITC (calorimétrie de titrage isotherme)
REMARQUE : Les titrages sont effectués à l’aide d’un micro-calorimètre avec auto-injection.
- Préparation du tampon : Préparer 3 L de tampon contenant 20 mM HEPES, 150 mM de NaCl, 1 mM de TCEP, pH 7,5.
- Dialyse : Dialyser VHL et Brd4BD2 (~ 500 μL à 150 μM chacun) contre 1 L du tampon préparé à l’étape 1.1, 3 fois à 4 °C, pendant 4 h, 2 h et environ 16 h, respectivement. Conservez le tampon après la dernière dialyse pour l’utiliser dans les étapes suivantes.
- Préparez les échantillons sur une plaque de 96 puits munie d’un couvercle en plastique.
- Pour chaque titrage, préparez 400 μL de solution pour la cellule, 125 μL pour la seringue et 400 μL de tampon pour le nettoyage. Ajouter des échantillons à trois puits consécutifs sur la plaque. Étant donné que le composé est préparé à 1 mM dans du DMSO à 100 %, ajoutez le même pourcentage de DMSO aux solutions protéiques pour assurer le tampon correspondant dans la cellule et la seringue. Ajoutez 2 % de DMSO aux solutions finales.
- Échantillon pour le titrage de la VHL en MZ1 : Préparer une solution cellulaire contenant 392 μL de tampon, 4 μL de MZ1 à 1 mM (concentration finale de 10 μM) et 4 μL de DMSO à 100 %. Préparer une solution de seringue contenant 122,5 μL de VHL à 85,7 μM, 2,5 μL de DMSO à 100 % (concentration finale de 84 μM).
- Échantillons pour le titrage de la VHL dans le tampon (les données seront utilisées pour la soustraction de fond des données générées à partir de l’échantillon 1.3.1.1) : Préparer une solution cellulaire contenant 392 μL de tampon, 8 μL de DMSO à 100 %. Préparer une solution de seringue contenant 122,5 μL de VHL à 85,7 μM (concentration finale de 84 μM) et ajouter 2,5 μL de DMSO à 100 %.
- Échantillons pour le titrage du VHL en MZ1 et Brd4 BD2 : Préparer une solution cellulaire contenant 392 μL de 17,1 μM Brd4BD2 (concentration finale de 16,8 μM), 3,36 μL de MZ1 à 1 mM (concentration finale de 8,4 μM) et 4,64 μL de DMSO à 100 %. Préparer une solution de seringue contenant 122,5 μL de VHL à 85,7 μM (concentration finale de 84 μM) et 2,5 μL de DMSO à 100 %.
- Echantillons pour le titrage de la VHL en Brd4 BD2 (bruit de fond du 1.3.1.3) : Préparer une solution cellulaire contenant 392 μL de Brd4BD2 à 17,1 μM (concentration finale de 16,8 μM) et 8 μL de DMSO à 100 %. Préparer une solution de seringue contenant 122,5 μL de VHL à 85,7 μM (concentration finale de 84 μM) et 2,5 μL de DMSO à 100 %.
- Pour chaque titrage, préparez 400 μL de solution pour la cellule, 125 μL pour la seringue et 400 μL de tampon pour le nettoyage. Ajouter des échantillons à trois puits consécutifs sur la plaque. Étant donné que le composé est préparé à 1 mM dans du DMSO à 100 %, ajoutez le même pourcentage de DMSO aux solutions protéiques pour assurer le tampon correspondant dans la cellule et la seringue. Ajoutez 2 % de DMSO aux solutions finales.
- Exécutez les quatre titrages sur le micro-calorimètre. Chacune consiste en 19 injections de solution de seringue de 2 μL à raison de 2 μL/s à des intervalles de temps de 120 s. Faites une première injection de protéines (0,4 μL) et jetez-la lors de l’analyse des données. Effectuer toutes les expériences à 25 °C, en remuant à 600 tr/min.
- Analyse des données : Ajuster les données à un modèle à site de liaison unique pour obtenir la stœchiométrie (n), la constante de dissociation (KD) et l’enthalpie de liaison (ΔH) à l’aide du logiciel d’analyse fourni par le fabricant (Figure 2).
2. Méthode 2 : BLI (interférométrie des biocouches)
- Réalisez des expériences BLI à l’aide de capteurs enrobés de streptavidine (SA) à température ambiante (RT) avec un taux d’acquisition de 5 Hz. Au cours de l’expérience BLI automatisée, assurez-vous que les capteurs sont stationnaires à l’intérieur d’une seule colonne et se déplacent entre les différentes colonnes d’une plaque noire à fond plat de 96 puits avec un volume de puits maximal de 392 μL. Remplissez chaque puits de la plaque utilisée dans l’expérience avec 200 μL de solution.
REMARQUE : Le BLI n’est utile que pour détecter les interactions protéine-protéine (c’est-à-dire la formation de complexes ternaires). Il n’est pas assez sensible pour détecter les interactions entre les protéines et les petites molécules.- Préparation du tampon : Préparer 100 mL de tampon contenant 20 mM HEPES, 150 mM de NaCl, 1 mM de TCEP, 0,05 % de P20, pH 7,5.
- Optimisation de l’étape d’immobilisation : Chargez le capteur de test à 1-3 nm, comme recommandé par le fabricant. Une charge de ~1,0 nm est utilisée pour les procédures décrites ici. Pour ce faire, plongez le capteur dans une solution contenant du VHL à 1,5 μg/mL pendant 80 s.
- Effectuez des mesures cinétiques BLI en appliquant sept capteurs SA en utilisant l’ordre suivant :
- Pour la première phase de référence, plongez dans le tampon pendant 60 s.
- Pour la phase d’immobilisation, tremper dans une solution de VHL à 1,5 μg/mL pendant 80 s.
- Pour la deuxième phase de référence, plongez dans le tampon pendant 60 s.
- Pour la phase d’association, plonger dans une solution d’une concentration fixe de Brd4BD2 à 2 μM, d’une concentration fixe de DMSO à 2 % et de concentrations variables de MZ1 à 100 nM, 50 nM, 25 nM, 12,5 nM, 6,3 nM, 3,1 nM et 0 nM (capteur de référence) pendant 300 s.
- Pour la phase de dissociation, plonger dans le tampon pendant 600 s.
- Effectuez l’analyse des données à l’aide du logiciel du fabricant. k on, k off et kd sont signalés pour l’ajustement des données (Figure 3).
3. Méthode 3 : SPR (résonance plasmonique de surface)
REMARQUE : Toutes les expériences SPR sont effectuées à l’aide de puces de capteur enrobées de streptavidine (SA) à RT. Bien que la puce NTA soit utilisée pour la détection entre les protéines et les petites molécules, elle doit être utilisée avec prudence lorsqu’elle est appliquée au complexe ternaire, car un bruit de fond beaucoup plus élevé que celui de la puce SA est observé, peut-être en raison d’interactions électrostatiques entre la surface de la puce chargée et la protéine dans l’analyte.
- SPR pour l’interaction VHL-MZ1
- Préparation du tampon.
- Préparez un tampon de 1 L contenant 20 mM HEPES, 150 mM de NaCl, 1 mM de TCEP, 0,005 % de P20, pH 7,5 et passez-le dans une unité de filtration de 0,2 μm.
- Retirez 20 mL de tampon pour une utilisation ultérieure (tampon sans DMSO) et remplissez-le avec 20 mL de DMSO (2 % de DMSO dans le tampon final en cours d’exécution). Maintenir le DMSO à 2 % dans tous les échantillons décrits ci-dessous (étapes 3.1 et 3.2)
- Activer la puce SA en suivant le protocole du fabricant et immobiliser le VHL à ~2000 RU (injecter une solution protéique de 5 μg/mL à 5 μL/min).
- Préparer MZ1 en 3 fois, 8 points de dilution en série avec la concentration maximale à 10 μM sur une plaque de polypropylène translucide à fond conique de 384 puits d’un volume maximal de 130 μL. Recouvrez la plaque de feuilles de plastique transparentes auto-adhésives compatibles avec les microplaques en polypropylène.
- Préparez la concentration maximale avec le tampon sans DMSO pour assurer la concentration finale de 2 % de DMSO. À 147 μL de tampon sans DMSO, ajoutez 1,5 μL de stock MZ1 à 1 mM et 1,5 μL de DMSO 100 %.
- Préparez la dilution en série 3 fois avec le tampon de fonctionnement contenant 2 % de DMSO. À 100 μL du tampon courant, ajouter 50 μL de solution à la concentration maximale préparée à l’étape 3.1.3.1 et bien mélanger pour préparer la 2econcentration la plus élevée.
- Transférez ensuite 50 μL de la solution préparée à l’étape 3.1.3.2 dans les 100 μL suivants de tampon courant, mélangez bien pour préparer la 3ème concentration la plus élevée, et ainsi de suite.
- Exécutez SPR à l’aide de la configuration multi-cycles : Mode : Haute performance ; Temps de contact : 120 s ; Temps de dissociation : 300 s ; débit : 50 μL/min.
- Effectuer l’analyse des données à l’aide du logiciel d’évaluation fourni par le fabricant de l’instrument. L’analyse en régime permanent a indiqué que KD = 26 (± 3) nM et Rmax de ~91 % (± 5 %) de liaison ont été obtenus (Figure 4A)
- Préparation du tampon.
- SPR pour VHL : MZ1 : Complexe ternaire Brd4BD2
- Préparez la mémoire tampon comme décrit à l’étape 3.1.1.
- Activez la puce SA en suivant le protocole du fabricant et immobilisez VHL à ~100 RU. Injecter une solution protéique de 0,5 μg/mL VHL à un débit de 5 μL/min et un temps de contact compris entre 1 et 5 min jusqu’à ce qu’une densité de surface de ~100 RU soit atteinte.
- Préparer les échantillons pour deux injections à cycle unique en 5 points et 5 fois dans une plaque à fond rond en polystyrène transparent de 96 puits d’un volume maximal de 323 μL et la recouvrir de feuilles de plastique transparentes autoadhésives compatibles avec les microplaques de polystyrène, comme décrit aux étapes 3.2.3.1 à 3.2.3.2.
- Contrôle négatif : l’analyte ne contient que Brd4BD2 .
- Préparer la concentration maximale à 25 μM dans un tampon de fonctionnement d’un volume de 200 μL (#A5)
- Ajouter 160 μL de Brd4BD2 à 2 μM dans le tampon de fonctionnement aux 4 puits suivants à gauche (#A1-A4)
- Transférez 40 μL de A5 sur A4. Bien mélanger.
- Transférez 40 μL de A4 au format A3. Bien mélanger. Continuez ainsi jusqu’à A1.
- Formation d’un complexe ternaire : l’analyte contient MZ1 et Brd4BD2.
- Préparer la concentration maximale en ajoutant 4 μL de MZ1 à 20 μM dans une solution de DMSO à 100 % à 196 μL contenant 25,5 μM de Brd4BD2 dans un tampon sans DMSO (#B5). Les concentrations finales sont de 25 μM Brd4BD2, 100 nM de MZ1 et 2 % de DMSO.
- Ajouter 160 μL de Brd4BD2 à 2 μM dans le tampon de fonctionnement aux 4 puits suivants à gauche (#B1-B4)
- Transférer 40 μL de B5 à B4. Bien mélanger.
- Transférer 40 μL de B4 à B3. Bien mélanger. Continuez ainsi jusqu’à B1.
- Contrôle négatif : l’analyte ne contient que Brd4BD2 .
- Exécutez SPR à l’aide de la configuration à cycle unique : Temps de contact : 100 s ; Temps de dissociation : 720 s ; débit : 50 μL/min. Appliquez trois injections de tampon avant chaque échantillon pour assurer un fond stable.
- Analyse des données : Appliquer la troisième injection du tampon comme arrière-plan pour la soustraction. En tant que témoin négatif, lorsque MZ1 n’est pas présent, la réponse SPR entre VHL et Brd4BD2 est négligeable. Lorsque MZ1 est présent, l’ajustement cinétique indique que l’interaction entre VHL et le complexe MZ1-Brd4BD2 a k on = 7,9 (± 1,5) *107 /M/s, koff = 0,014 s-1 et KD = 1 nM. (Graphique 4B)
- SPR pour les interactions CRBN et petites molécules
- Préparation du tampon.
- Préparez un tampon de 1 L contenant 50 mM de Tris, 100 mM de NaCl, 1 mM de MgCl 2, 0,5 mM de TCEP, 0,005 % de P20, pH 7,5 et passez-le dans une unité de filtration de0,2 μm.
- Retirez 20 mL de tampon et remplissez 20 mL de DMSO à 100 % (2 % de DMSO dans le tampon de fonctionnement final). Il est important de maintenir le DMSO à 2 % dans tous les échantillons décrits ci-dessous (étapes 3.3, 3.4 et 3.5).
- Activez la puce SA en suivant le protocole du fabricant et immobilisez CRBN à ~800 RU.
- Préparer les composés en dilution en série 3 fois et 6 points sur une plaque à 384 puits avec une concentration maximale de 30 μM, comme décrit au point 3.1.3.
- Exécutez SPR à l’aide de la configuration multicycle : Mode : Haute performance, temps de contact : 60 s, temps de dissociation : 90 s ; débit : 50 μL/min. Pour ce système, trois injections de tampon suffisent pour obtenir un bruit de fond stable. Pour d’autres systèmes de dosage, il peut être nécessaire d’effectuer des injections de tampon supplémentaires.
- Effectuez l’analyse des données à l’aide du logiciel du fabricant.
REMARQUE : L’analyse en régime permanent indique que le KD du BRD-2512 et du BRD-5110 est d’environ 3 μM. Cependant, la liaison n’a atteint que ~ 70% de liaisonR max à la concentration maximale. La faible affinité et la forme du sensorgramme indiquent que l’insolubilité du composé à des concentrations élevées est susceptible de se produire. Ainsi, le KD réel pourrait être supérieur à 3 μM. BRD-4761, qui ne se lie pas au CRBN, a été inclus comme témoin négatif.
- Préparation du tampon.
- SPR pour les interactions PPM1D et petites molécules
- Préparez la mémoire tampon comme décrit à l’étape 3.3.1. Utilisez une puce de capteur d’acide nitriloacétique (NTA).
- Appliquer un cycle unique car l’ogive PPM1D a un k lent activé et kdésactivé. Régénérez la puce NTA en utilisant la configuration par défaut du fabricant et immobilisez PPM1D à ~ 1000 RU après chaque injection de composé (injecter une solution protéique de 5 μg/mL à 5 μL/min).
- Préparer les composés en dilution en série 3 fois et en 5 points avec la concentration maximale à 400 nM comme décrit au point 3.1.3.
- Exécutez SPR à l’aide de la configuration à cycle unique : Mode : haute performance ; Temps de contact : 90 s ; Temps de dissociation : 600 s ; débit : 50 μL/min. Appliquez trois injections de tampon avant chaque composé pour assurer un fond stable.
- Analyse des données. Appliquez la troisième injection du tampon comme arrière-plan pour la soustraction. En tant que témoin négatif, BRD-2512 ne se lie pas à PPM1D. Pour le BRD-4761 et le BRD-5110, l’ajustement cinétique indiquait que le KD pour les deux était de 1 à 2 nM.
- SPR pour CRBN :PROTAC : Complexe ternaire PPM1D
- Préparez la mémoire tampon comme décrit à l’étape 3.3.1.
- Activer la puce SA en suivant le protocole du fabricant et immobiliser le CRBN à ~35 RU (injecter une solution protéique de 0,5 μg/mL à 5 μL/min).
- Préparer trois composés en dilution série 3 fois et 6 points avec une concentration maximale de 30 μM tout en maintenant [PPM1D] à 1 μM dans tous les échantillons, y compris les blancs, en utilisant la méthode décrite à l’étape 3.2.3.
- Exécutez SPR à l’aide de la configuration multi-cycles : Mode : Haute performance ; Temps de contact : 60 s ; Temps de dissociation : 90 s ; débit : 50 μL/min.
- Effectuez l’analyse des données à l’aide du logiciel du fabricant.
REMARQUE : Alors que le BRD-2512 et le BRD-4761 ne présentent pas de réponse ou une réponse négligeable, le BRD-5110 montre clairement « l’effet d’accrochage » à l’état stationnaire avec une cinétique marche/arrêt rapide (Figure 5A-C). La réponse expérimentale de liaison de BRD-5110 (Figure 5C) se situe entre la réponse prédite de la simulation en supposant que K D (CRBN, cpd) est de 3 ou 10 μM (Figure 5E), ce qui suggère que le K D ternaire et le KD binaire sont très similaires. Il n’y a pas de coopérativité apparente de PPM1D : BRD-5110 : formation du complexe CRBN.
Résultats
La caractérisation du complexe binaire VHL : MZ1 et du complexe ternaire VHL : MZ1 : Brd4BD2 peut être trouvée dans la Figure 2 (ITC), la Figure 3 (BLI) et la Figure 4 (SPR) en utilisant un tampon très similaire. Le KD extrait des tests orthogonaux est cohérent. La coopérativité peut être calculée par K D (binaire) / KD (ternaire), ce qui est très positif (15 de l’ITC ou 26 de la SPR).
La caractérisation du système CRBN :PROTAC : PPM1D a été réalisée par SPR (Figure 5A-D). Le CRBN a été immobilisé à ~35 RU pour faciliter l’observation de la formation du complexe ternaire. La liaison de PROTAC seule a donné lieu à un signal de <2 RU, ce qui est inférieur au bruit. PPM1D dans l’analyte donne un signal de fond élevé sur la surface de la puce SA, et la concentration la plus élevée qui peut être appliquée est d’environ 1 μM. Cette valeur est inférieure au KD entre le CRBN et son ogive ≥3 μM), on s’attend donc à un « effet d’hameçon ». Le SPR est suffisamment sensible pour le détecter, ce qui est en bon accord avec la simulation (Figure 5E). La simulation a été réalisée en utilisant les équilibres non coopératifs de la littérature19 combinés au calcul classique du SPR [Responsemax = (Response Ligand × MassImmobilization)/Mass Ligand]. Étant donné que le KD entre le CRBN et le composé n’est pas déterminé avec précision en raison de l’insolubilité du composé à haute concentration, la simulation a été effectuée à l’aide de quatre KD hypothétiques : 1 μM, 3 μM, 10 μM ou 30 μM. Les résultats expérimentaux se situent entre les courbes simulées de 3 μM et 10 μM, ce qui est presque identique au KD dans le système binaire, ce qui suggère qu’il n’y a pas de coopérativité.

Figure 1 : Illustration de trois scénarios de liaison et définition de différents KD. (A) Systèmes classiques à deux composants. (B) Système à trois composants dans lequel une extrémité de PROTAC peut être saturée, de sorte qu’il peut être évalué comme un système à deux composants. (C) Système à trois composants dans lequel l’on observe un « effet crochet ». Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Résultats de l’ITC. Titrage de VHL en MZ1 (à gauche) ou MZ1 :Brd4BD2 complexe (à droite). Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Résultats de l’BLI. MZ1 intervient dans la formation du complexe ternaire VHL : MZ1 : Brd4BD2. (A) Données brutes. (B) Soustraction des signaux de fond où [MZ1] = 0. (C) Ajustement cinétique de B pour extraire k on, koff et KD. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Résultats du SPR. (A) Liaison de MZ1 à VHL. (B) Complexe binaire MZ1 :Brd4BD2 se liant à VHL. Veuillez cliquer ici pour voir une version agrandie de cette figure.
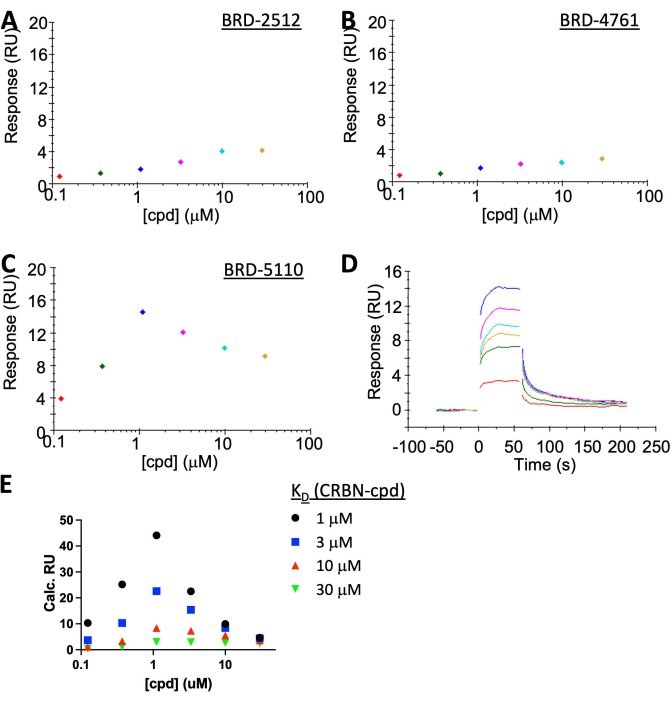
Figure 5 : Résultats SPR montrant « l’effet crochet » d’un PPM1D-PROTAC représentatif. Le CRBN a été immobilisé sur la surface de la puce SA tandis que le [PPM1D] a été maintenu à 1 μM dans l’analyte dans tous les cas. (A) BRD-2512, un composé qui ne se lie qu’au CRBN, ne donne presque aucune réponse. (B) BRD-4761, un composé qui ne se lie qu’à PPM1D, ne donne pas non plus de réponse. (C,D) BRD-5110, un PROTAC avec l’ogive de CRBN dans BRD-2512 et l’ogive de PPM1D dans BRD-4761, a induit la formation du complexe ternaire. (E) Une simulation des résultats SPR en supposant que le KD entre le CRBN et le composé est de 1 μM (noir), 3 μM (bleu), 10 μM (rouge) ou 30 μM (vert). La courbe BRD-2512 est comprise entre 3 μM et 10 μM, ce qui est très proche de la binaire KD mesurée, suggérant qu’il n’y a pas de coopérativité (coopérativité = 1). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
La caractérisation biophysique des interactions binaires et ternaires entre les molécules PROTAC et leurs partenaires de liaison aux protéines peut fournir des informations uniques et complémentaires par rapport aux systèmes cellulaires largement utilisés. Comprendre l’affinité entre chaque ogive d’une molécule PROTAC et ses partenaires de liaison aux protéines peut aider à orienter les efforts de chimie médicinale vers l’optimisation de ces interactions. Des structures cristallines de complexes PROTAC ternaires publiées précédemment ont révélé que les atomes de la région de liaison peuvent former des interactions avec l’un ou les deux partenaires de liaison protéique16,20. La détermination expérimentale de la coopérativité de la formation de complexes ternaires peut soutenir l’optimisation des linkers.
Ce rapport décrit l’utilisation de trois techniques biophysiques différentes qui peuvent fournir des informations sur les affinités de liaison entre les molécules PROTAC et leurs partenaires de liaison aux protéines. La méthode 1 détaille le dispositif expérimental de calorimétrie de titrage isotherme (ITC) pour la molécule PROTAC, MZ1, le complexe ligase VHL E3 et le bromodomaine Brd4BD2. Les résultats de l’ITC ont montré desK D de 59 nM pour l’interaction binaire entre MZ1 et VHL et de 4 nM pour l’interaction ternaire entre VHL et MZ1 et Brd4BD2 pré-mélangés. Les affinités étaient cohérentes avec celles observées dans SPR (liaison VHL immobilisée à MZ1 K D = 26 nM, liaison VHL immobilisée à MZ1 et Brd4BD2 K D=1 nM pré-mélangés) et BLI (KD = 2,8 nM). Bien que les résultats de l’ITC KD pour la liaison VHL à MZ1 soient cohérents avec les valeurs rapportées16, la stœchiométrie obtenue est différente. Une explication possible de ce résultat est la faible solubilité de MZ1 dans le tampon à base de HEPES utilisé dans le protocole décrit ici, alors que les résultats de la littérature ont été générés à l’aide d’un tampon à base de Bis-tris. Les auteurs ont préféré utiliser les mêmes composants de tampon pour SPR, ITC et BLI.
La méthode 2 décrit le dispositif expérimental pour l’analyse BLI de l’interaction de VHL immobilisé, d’une concentration fixe de Brd4BD2 et de concentrations variables de MZ1. En raison des limites de sensibilité de la technique, des valeursK D, k on et koff pour la formation de complexes ternaires ont pu être générées, mais pas pour l’interaction binaire entre MZ1 et les protéines.
La méthode 3 décrit plusieurs dosages SPR. Le SPR est plus sensible que le BLI et peut être appliqué pour observer à la fois les interactions protéine-petite molécule (binaire) et protéine-protéine (ternaire). Dans ce dernier cas, les signaux de fond doivent être surveillés de près, car la protéine contenue dans l’analyte pourrait donner des signaux élevés et instables. Le SPR est très sensible aux réactifs à indice de réfraction élevé, notamment le DMSO, le glycérol et les détergents. Si la protéine est stockée dans le tampon contenant du glycérol ou du détergent, le tampon en cours d’exécution doit contenir des concentrations correspondantes de ces composants. Alternativement, l’application d’une chromatographie d’exclusion de taille les élimine complètement avant toute expérience SPR. Il faut veiller à ce que les concentrations de DMSO correspondent étroitement aux échantillons de tampon et d’analyte. Les corrections de solvant DMSO sont effectuées conformément aux instructions du fabricant.
La méthode de l’étape 3.1 décrit le test SPR pour l’interaction binaire VHL-MZ1. La méthode 3.2 décrit le dosage SPR pour le complexe ternaire VHL : MZ1 : Brd4 BD2 où le VHL est immobilisé et l’analyte est soit Brd4 BD2 seul, soit le complexe MZ1 :Brd4BD2. Dans ce système, l’interaction entre Brd4BD2 et VHL est négligeable. La formation du complexe ternaire est très coopérative (ɑ = 26). Le taux de décalage pour la formation de complexes ternaires est de 0,014 s-1, ce qui nécessite l’utilisation d’une cinétique à cycle unique. Les résultats de l’ITC montrent également une formation de complexes ternaires hautement coopératifs (ɑ=15). Les méthodes SPR des étapes 3.3, 3.4 et 3.5 décrivent des tests permettant d’évaluer la formation d’un complexe entre CRBN et PPM1D induite par la présence d’une molécule PROTAC, BRD-5110. La molécule PROTAC a une faible affinité pour le CRBN (K D ~3 μM) et une forte affinité pour le PPM1D (KD = 1-2 nM). En conséquence, la faible liaison au CRBN n’est pas saturée et se traduit par un « effet crochet » observé. Bien qu’il soit possible d’augmenter la solubilité du ligand en augmentant la concentration de DMSO utilisée dans l’expérience, il est important dans ces cas de surveiller attentivement la stabilité des protéines qui peuvent être affectées négativement par des concentrations élevées de DMSO. De plus, le DMSO a une chaleur de dissolution élevée qui peut obscurcir la chaleur de liaison des ligands aux protéines. Il faut veiller à faire correspondre les concentrations de DMSO de la solution dans la seringue et de la solution dans la cellule. Les auteurs recommandent la dialyse des deux solutions contre la même préparation tampon.
Des recommandations et des lignes directrices générales sont fournies sur la base des expériences réalisées et rapportées ici. Lorsque les affinités des interactions binaires entre les molécules PROTAC et leurs partenaires de liaison aux protéines sont fortes (KD <1 μM), SPR fournit des affinités fiables et reproductibles ainsi que des informations précieuses sur la coopérativité de la formation de complexes ternaires. Lorsque les affinités de l’interaction binaire entre l’un des partenaires de liaison protéique et la molécule PROTAC sont faibles (KD >1 μM), la configuration du test devra être modifiée. Dans ces cas, l’utilisation de simulations moléculaires où les constantes de liaison sont fixes et où les concentrations de ligand et d’analyte sont variées peut être utile pour guider la conception des essais et interpréter les résultats expérimentaux. Les tests ITC fournissent des informations importantes sur la stœchiométrie de la liaison, mais nécessitent beaucoup plus de réactifs protéiques et composés que le SPR et le BLI. De plus, la solubilité de la molécule PROTAC peut être limitante pour les expériences ITC. Le BLI a un débit plus élevé que l’ITC et nécessite moins de réactifs protéiques et composés. Cependant, en raison des limitations de sensibilité, le BLI ne peut être utilisé que pour évaluer la formation de complexes ternaires et non les interactions binaires entre les molécules PROTAC et leurs partenaires de liaison aux protéines. Il est recommandé d’utiliser le SPR pour les tests de routine des tests de liaison PROTAC binaires et ternaires et des tests BLI et ITC utilisés pour la validation orthogonale des résultats du SPR.
Déclarations de divulgation
Les auteurs n’ont pas d’intérêts financiers concurrents ou d’autres conflits d’intérêts.
Remerciements
Ce travail a été soutenu par un prix d’innovation et de développement technologique du Center for the Development of Therapeutics du Broad Institute du MIT et de Harvard. Les auteurs tiennent à remercier les membres de l’équipe de la haute direction et du comité d’examen pour leur soutien à ce travail.
matériels
| Name | Company | Catalog Number | Comments |
| 96-plate | Greiner | 655076 | flat-bottom, black plates used In BLI experiments |
| 96-well plate | Nunc | 73520-120 | Plate use for ITC sample preparation |
| 96-well plate | Greiner | 650101 | Plate used to prepare samples for SPR experiments |
| Auto iTC200 micro-calorimeter | Malvern Panalytical | Instrument used to perform ITC experiments. Product discontinued. | |
| Biacore S200 | Cytiva | 29136649 | Instrument used to perform SPR experiments |
| MZ1 | ProbeChem | PC-60099 | PROTAC that binds to VHL and Brd4BD2 |
| NTA sensor chip | Cytiva | BR100532 | SPR chip used to perform SPR experiments involving PPM1D |
| Octet Red-384 | Sartorius | Instrument used to perform BLI experiments. Product discontinued. | |
| Plate cover | Malvern | PQA0001 | Cover for Nunc 96-well plate (73520-120) |
| Plate cover | Cytiva | 28975816 | Plate cover for Greiner plate (650101) |
| Series S SA sensor chip | Cytiva | BR100531 | SPR chip used to perform SPR experiments involving MZ1:VHL:BRD4 |
| Streptavidin (SA) Dip and Read Biosensors | Sartorius | 18-509 | Coated sensors used in BLI experiments |
Références
- Balaji, V., Hoppe, T. Regulation of E3 ubiquitin ligases by homotypic and heterotypic assembly. F1000Research. 9, (2020).
- Song, L., Luo, Z. -. Q. Post-translational regulation of ubiquitin signaling. Journal of Cell Biology. 218 (6), 1776-1786 (2019).
- Yang, Q., Zhao, J., Chen, D., Wang, Y. E3 ubiquitin ligases: styles, structures and functions. Molecular Biomedicine. 2 (1), 23 (2021).
- Grice, G. L., Nathan, J. A. The recognition of ubiquitinated proteins by the proteasome. Cellular and Molecular Life Sciences: CMLS. 73 (18), 3497-3506 (2016).
- Chirnomas, D., Hornberger, K. R., Crews, C. M. Protein degraders enter the clinic - a new approach to cancer therapy. Nature Reviews Clinical Oncology. 20 (4), 265-278 (2023).
- Toure, M., Crews, C. M. Small-molecule PROTACS: New approaches to protein degradation. Angewandte Chemie (International ed. In English). 55 (6), 1966-1973 (2016).
- Ottis, P., Toure, M., Cromm, P. M., Ko, E., Gustafson, J. L., Crews, C. M. Assessing different E3 ligases for small molecule induced protein ubiquitination and degradation. ACS Chemical Biology. 12 (10), 2570-2578 (2017).
- Riching, K. M., et al. Quantitative live-cell kinetic degradation and mechanistic profiling of PROTAC mode of action. ACS Chemical Biology. 13 (9), 2758-2770 (2018).
- Nabet, B., et al. The dTAG system for immediate and target-specific protein degradation. Nature Chemical Biology. 14 (5), 431-441 (2018).
- Paiva, S. -. L., Crews, C. M. Targeted protein degradation: elements of PROTAC design. Current Opinion in Chemical Biology. 50, 111-119 (2019).
- Hershko, A., Ciechanover, A. The ubiquitin system. Annual Review of Biochemistry. 67, 425-479 (1998).
- Chan, K. -. H., Zengerle, M., Testa, A., Ciulli, A. Impact of target warhead and linkage vector on inducing protein degradation: Comparison of bromodomain and extra-terminal (BET) degraders derived from triazolodiazepine (JQ1) and tetrahydroquinoline (I-BET726) BET inhibitor scaffolds. Journal of Medicinal Chemistry. 61 (2), 504-513 (2018).
- Roy, M. J., et al. SPR-measured dissociation kinetics of PROTAC ternary complexes influence target degradation rate. ACS Chemical Biology. 14 (3), 361-368 (2019).
- Pierce, N. W., Kleiger, G., Shan, S., Deshaies, R. J. Detection of sequential polyubiquitylation on a millisecond timescale. Nature. 462 (7273), 615-619 (2009).
- Gadd, M. S., et al. Structural basis of PROTAC cooperative recognition for selective protein degradation. Nature Chemical Biology. 13 (5), 514-521 (2017).
- Nahta, R., Castellino, R. C. Phosphatase magnesium-dependent 1 δ (PPM1D), serine/threonine protein phosphatase and novel pharmacological target in cancer. Biochemical Pharmacology. 184, 114362 (2021).
- Douglass, E. F., Miller, C. J., Sparer, G., Shapiro, H., Spiegel, D. A. A comprehensive mathematical model for three-body binding equilibria. Journal of the American Chemical Society. 135 (16), 6092-6099 (2013).
- Zorba, A., et al. Delineating the role of cooperativity in the design of potent PROTACs for BTK. Proceedings of the National Academy of Sciences. 115 (31), E7285-E7292 (2018).
- Fairhead, M., Howarth, M. Site-specific biotinylation of purified proteins using BirA. Methods in Molecular Biology. 1266, 171-184 (2015).
- Nowak, R. P., et al. Plasticity in binding confers selectivity in ligand-induced protein degradation. Nature Chemical Biology. 14 (7), 706-714 (2018).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon