Method Article
Desarrollo y Aplicación de Ensayos Biofísicos para la Evaluación de la Formación de Complejos Ternarios Inducida por Quimeras Dirigidas a la Proteólisis (PROTACS)
En este artículo
Resumen
En este trabajo se describen protocolos para la caracterización biofísica de la formación de complejos ternarios inducidos por quimeras dirigidas a proteólisis (PROTACS) que involucran a las ubiquitinas ligasa Von Hippel-Lindau E3 ligasa (VHL) y Cereblon (CRBN). Los métodos biofísicos que se ilustran en este documento incluyen la resonancia de plasmones de superficie (SPR), la interferometría de biocapas (BLI) y la calorimetría de titulación isotérmica (ITC).
Resumen
Las ligasas E3 y las proteínas destinadas a la degradación pueden ser inducidas a formar complejos por moléculas heterobifuncionales en un proceso de varios pasos. La cinética y la termodinámica de las interacciones involucradas contribuyen a la eficiencia de la ubiquitinación y la degradación resultante de la proteína. Las técnicas biofísicas como la resonancia de plasmones de superficie (SPR), la interferometría de biocapas (BLI) y la calorimetría de titulación isotérmica (ITC) proporcionan información valiosa que se puede utilizar en la optimización de esas interacciones. Utilizando dos sistemas modelo, se estableció un conjunto de herramientas de ensayo biofísico para comprender la cooperatividad de la formación de complejos ternarios y el impacto del "efecto gancho" en la cinética de unión. En un caso, se evaluó una molécula de proteólisis dirigida a quimera (PROTAC) que indujo la formación de complejos ternarios entre Brd4BD2 y VHL. La molécula heterobifuncional, MZ1, tiene afinidades nM tanto por la proteína Brd4BD2 (SPR K D = 1 nM, ITC K D = 4 nM) como por el complejo BVS (SPR KD = 29 nM, ITC KD = 66 nM). Para este sistema, se desarrollaron ensayos robustos de SPR, BLI e ITC que reprodujeron los resultados publicados que demuestran la cooperatividad de la formación de complejos ternarios. En el otro caso, se estudió una molécula que indujo complejos ternarios entre una proteína de 46,0 kDa, PPM1D, y cereblon [CRBN (319-442)]. La molécula heterobifuncional, BRD-5110, tiene una SPR K D = 1 nM para PPM1D pero una unión mucho más débil contra el complejo CRBN truncado (319-442) (SPR KD = ~ 3 μM). En ese caso, la unión de CRBN en SPR no era saturable, lo que daba lugar a un "efecto gancho". Se evaluaron los requisitos de rendimiento y reactivos para SPR, BLI e ITC, y se proporcionaron recomendaciones generales para su aplicación a los proyectos PROTAC.
Introducción
La poliubiquitinación de proteínas en la célula es un proceso estrechamente regulado que involucra enzimas de la familia de la ubiquitina ligasa 1,2. Las enzimas terminales en la vía son las ligasas de ubiquitina E3 que unen covalentemente las moléculas de ubiquitina a sus socios de unión a proteínas3. La poliubiquitinación de esos socios de unión a proteínas se dirige a ellos para la degradación proteolítica por el proteasoma4. Este sistema forma parte del proceso de homeostasis de proteínas que se ha aprovechado terapéuticamente para inducir la degradación de proteínas implicadas en la enfermedad5. Las moléculas pequeñas que inducen la interacción entre las ligasas de ubiquitina E3, como la ligasa E3 de Von Hippel-Lindau (VHL) o el cereblon (CRBN), suelen estar compuestas por una ojiva de unión a la ligasa E3 conectada por un enlazador flexible a una ojiva que se une a la proteína que se destina a la degradación. Estas moléculas heterobifuncionales se conocen comúnmente como quimeras dirigidas a la proteólisis o PROTACS6.
El desarrollo de PROTACS implica la evaluación de la capacidad de las moléculas para inducir la degradación de proteínas en las células. Se han desarrollado muchos sistemas de ensayo celular que monitorizan la interacción inducida entre la proteína diana y los componentes de la ligasa E3, como VHL o CRBN, tras el tratamiento de las células con una molécula PROTAC. Uno de estos ensayos celulares, el sistema nanoluc-Halotag7, involucra una ligasa E3 fusionada con el aceptor de Halotag y una proteína diana marcada con un donante de nanoluc. La formación de complejos ternarios acerca el donante de nanoluc y el aceptor de Halotag, lo que permite la transferencia de energía del donante al aceptor, lo que resulta en la emisión de luz. Las variaciones de este sistema se pueden utilizar para evaluar la permeabilidad celular de las moléculas PROTACS8 o los cambios en el nivel relativo de ubiquitinación de la proteína diana9. Si bien estos sistemas celulares son esenciales para impulsar la optimización de PROTACS, la formación de complejos entre las ligasas E3 y las proteínas destinadas a la degradación es un proceso de varios pasos10,11. La cinética y termodinámica de las interacciones binarias y ternarias involucradas contribuyen a la eficiencia, ubiquitinación y la degradación resultante de la proteína12,13,14.
En este trabajo se describen los protocolos que pueden adaptarse para la caracterización biofísica de la formación de complejos ternarios inducidos por PROTACS utilizando resonancia de plasmón de superficie (SPR), interferometría de biocapas (BLI) y calorimetría de titulación isotérmica (ITC). Los protocolos SPR e ITC para la molécula MZ1 PROTAC que induce la formación de complejos ternarios entre Brd4BD2 y VHL derivados de los reportes de la literatura13,15 y aquí descritos fueron capaces de recapitular los resultados reportados con alguna modificación de los procedimientos reportados, los cuales serán discutidos. En este informe se incluye una descripción de un ensayo BLI utilizado para evaluar la formación de complejos ternarios entre MZI, Brd4BD2 y VHL. Las mediciones de afinidad de BLI fueron consistentes con las observadas en SPR e ITC. También se describe un protocolo previamente publicado en el que se desarrolló un ensayo SPR para evaluar la formación de complejos ternarios inducidos por PROTAC entre PPM1D, una proteína fosfatasa Ser/Thr cuya expresión es inducida de forma dependiente de p5316, y CRBN. En este caso, la molécula PROTAC tiene una afinidad nanomolar por PPM1D, pero solo una afinidad micromolar por CRBN. En este caso, la unión de la molécula PROTAC a CRBN no es saturable, lo que da lugar al "efecto gancho" comúnmente observado. El efecto gancho es una propiedad de tres sistemas corporales en los que hay dos especies que pueden formar un complejo heterotrimérico cuando ambas están unidas a una molécula puente (Figura 1)17. El efecto gancho se observa cuando la especie puente está en exceso de concentración en relación con las otras dos especies. El estado resultante es aquel en el que las interacciones binarias superan a las interacciones ternarias. Los sistemas en los que se observa el efecto gancho requieren consideraciones específicas de diseño experimental que se analizan en este informe. Se proporcionan conceptos generales y requisitos de reactivos para evaluar la utilización de ensayos biofísicos para la evaluación de la formación de complejos ternarios inducidos por PROTAC.
Protocolo
Todas las proteínas fueron sobreexpresadas en E. coli con buen rendimiento y pureza (>80%) siguiendo los protocolos de la literatura18. La biotinilación se llevó a cabo mediante una reacción catalizada por BirA18. Todas las moléculas pequeñas se prepararon en soluciones madre de 1 mM en 100% DMSO. Los procedimientos descritos en este documento no requieren equipo de seguridad de laboratorio especializado ni precauciones. Se debe usar equipo de protección personal (EPP) estándar de laboratorio (es decir, bata de laboratorio, gafas de seguridad y guantes).
Las proteínas aplicadas en este estudio se enumeran a continuación:
VHL: complejo VHL(53-213)/ElonginB (1-104)/ElonginC(17-112) biotinilado con Avi-tag en el extremo C-terminal de ElonginB.
Brd4BD2: Brd4BD2 sin etiquetar(333-460)
CRBN: CRBN biotinilado (319-442) con Avi-tag en el extremo N
PPM1D: PPM1D(1-420) no marcado o doble marcado con His8 en el extremo N
Las moléculas pequeñas aplicadas en este estudio se enumeran a continuación:
MZ1 (MW = 1002,6 Da): PROTAC que se une a BVS y Brd4BD2
BRD-2512 (MW = 841,4 Da): CRBN K D ~3 μM, no se une a PPM1D
BRD-5110 (MW = 872,0 Da): CRBN K D ~3 μM, PPM1D KD = 1-2 nM
BRD-4761 (MW = 476,6 Da): no se une a CRBN, PPM1D KD = 1-2 nM
1. Método 1: ITC (calorimetría de titulación isotérmica)
NOTA: Las valoraciones se realizan mediante un microcalorímetro con autoinyección.
- Preparación del tampón: Preparar 3 L de tampón que contenga 20 mM de HEPES, 150 mM de NaCl, 1 mM de TCEP, pH 7,5.
- Diálisis: Diálisis de VHL y Brd4BD2 (~ 500 μL a 150 μM cada uno) contra 1 L del tampón preparado en el paso 1.1, 3 veces a 4 °C, durante 4 h, 2 h y aproximadamente 16 h, respectivamente. Guarde el tampón después de la última diálisis para usarlo en los pasos posteriores.
- Prepare las muestras en una placa de 96 pocillos con una cubierta de plástico.
- Para cada valoración, prepare 400 μl de solución para la célula, 125 μl para la jeringa y 400 μl de tampón para la limpieza. Agregue muestras a tres pocillos consecutivos en la placa. Dado que el material compuesto se prepara a 1 mM en DMSO al 100%, agregue el mismo porcentaje de DMSO a las soluciones proteicas para garantizar el tampón correspondiente en la célula y la jeringa. Agregue un 2% de DMSO a las soluciones finales.
- Muestra para la valoración de la enfermedad de VHL en MZ1: Prepare una solución celular que contenga 392 μL de tampón, 4 μL de MZ1 a 1 mM (concentración final de 10 μM) y 4 μL de DMSO al 100 %. Prepare una solución de jeringa que contenga 122,5 μL de VHL a 85,7 μM, 2,5 μL de DMSO al 100% (concentración final de 84 μM).
- Muestras para la valoración de la BVS en el tampón (los datos se utilizarán para la sustracción de fondo de los datos generados a partir de la muestra 1.3.1.1): Preparar una solución celular que contenga 392 μL tampón, 8 μL de DMSO al 100%. Prepare una solución de jeringa que contenga 122,5 μL de VHL a 85,7 μM (concentración final de 84 μM) y agregue 2,5 μL de DMSO al 100%.
- Muestras para la valoración de la enfermedad VHL en MZ1 y Brd4 BD2: Prepare una solución celular que contenga 392 μL de 17,1 μM de Brd4BD2 (concentración final de 16,8 μM), 3,36 μL de MZ1 a 1 mM (concentración final de 8,4 μM) y 4,64 μL de DMSO al 100 %. Prepare una solución de jeringa que contenga 122,5 μl de VHL a 85,7 μM (concentración final de 84 μM) y 2,5 μl de DMSO al 100 %.
- Muestras para la valoración de la enfermedad VHL en Brd4 BD2 (antecedentes de 1.3.1.3): Prepare una solución celular que contenga 392 μL de 17,1 μM de Brd4BD2 (concentración final de 16,8 μM) y 8 μL de DMSO al 100%. Prepare una solución de jeringa que contenga 122,5 μl de VHL a 85,7 μM (concentración final de 84 μM) y 2,5 μl de DMSO al 100 %.
- Para cada valoración, prepare 400 μl de solución para la célula, 125 μl para la jeringa y 400 μl de tampón para la limpieza. Agregue muestras a tres pocillos consecutivos en la placa. Dado que el material compuesto se prepara a 1 mM en DMSO al 100%, agregue el mismo porcentaje de DMSO a las soluciones proteicas para garantizar el tampón correspondiente en la célula y la jeringa. Agregue un 2% de DMSO a las soluciones finales.
- Ejecute las cuatro valoraciones en el microcalorímetro. Cada una consta de 19 inyecciones de solución de jeringa de 2 μL a una velocidad de 2 μL/s a intervalos de tiempo de 120 s. Realice una inyección inicial de proteína (0,4 μL) y deséchela durante el análisis de los datos. Realice todos los experimentos a 25 °C, agitando a 600 rpm.
- Análisis de los datos: Ajuste los datos a un modelo de un solo sitio de unión para obtener la estequiometría (n), la constante de disociación (KD) y la entalpía de unión (ΔH) utilizando el software de análisis proporcionado por el fabricante (Figura 2).
2. Método 2: BLI (interferometría de biocapas)
- Realizar experimentos BLI utilizando sensores recubiertos de estreptavidina (SA) a temperatura ambiente (RT) con una tasa de adquisición de 5 Hz. Durante el experimento BLI automatizado, asegúrese de que los sensores estén estacionarios dentro de una sola columna y se muevan entre las diferentes columnas de una placa negra de fondo plano de 96 pocillos con un volumen máximo de pocillos de 392 μL. Llene cada pocillo de la placa utilizada en el experimento con 200 μL de solución.
NOTA: BLI solo es útil para detectar interacciones proteína-proteína (es decir, formación de complejos ternarios). No es lo suficientemente sensible como para detectar interacciones entre proteínas y moléculas pequeñas.- Preparación del tampón: Preparar 100 mL de tampón que contenga 20 mM de HEPES, 150 mM de NaCl, 1 mM de TCEP, 0,05% de P20, pH 7,5.
- Optimización para la etapa de inmovilización: Cargue el sensor de prueba a 1-3 nm, según lo recomendado por el fabricante. Se utiliza una carga de ~1,0 nm para los procedimientos descritos aquí. Para lograr esto, sumerja el sensor en una solución que contenga VHL a 1,5 μg/mL durante 80 s.
- Realice mediciones cinéticas BLI aplicando siete sensores SA utilizando la siguiente secuencia:
- Para la primera fase de referencia, sumérjase en el tampón durante 60 s.
- Para la fase de inmovilización, sumergir en una solución de VHL a 1,5 μg/mL durante 80 s.
- Para la segunda fase de referencia, sumérjase en el tampón durante 60 s.
- Para la fase de asociación, sumergir en una solución una concentración fija de Brd4BD2 a 2 μM, una concentración fija de DMSO al 2% y concentraciones variables de MZ1 a 100 nM, 50 nM, 25 nM, 12,5 nM, 6,3 nM, 3,1 nM y 0 nM (sensor de referencia) durante 300 s.
- Para la fase de disociación, sumérjase en el tampón durante 600 s.
- Realice análisis de datos utilizando el software del fabricante. k on, k off y kd se informan para el ajuste de datos (Figura 3).
3. Método 3: SPR (resonancia de plasmón de superficie)
NOTA: Todos los experimentos de SPR se llevan a cabo utilizando chips sensores recubiertos de estreptavidina (SA) en RT. Aunque el chip NTA se utiliza para la detección entre proteínas y moléculas pequeñas, debe usarse con precaución cuando se aplica al complejo ternario, ya que se observa un fondo mucho más alto que el chip SA, posiblemente debido a las interacciones electrostáticas entre la superficie del chip cargado y la proteína en el analito.
- SPR para la interacción BVS-MZ1
- Preparación del tampón.
- Prepare un tampón de 1 L que contenga 20 mM de HEPES, 150 mM de NaCl, 1 mM de TCEP, 0,005% de P20, pH7,5 y páselo a través de una unidad de filtro de 0,2 μm.
- Retire 20 ml de tampón para uso futuro (tampón sin DMSO) y rellene con 20 ml de DMSO (2 % de DMSO en el tampón de ejecución final). Mantenga el DMSO al 2% en todas las muestras que se describen a continuación (pasos 3.1 y 3.2)
- Activar el chip SA siguiendo el protocolo del fabricante e inmovilizar la VHL a ~2000 RU (inyectar solución proteica de 5 μg/mL a 5 μL/min).
- Prepare MZ1 en una dilución seriada de 3 veces y 8 puntos con una concentración superior de 10 μM en una placa de polipropileno translúcida de fondo cónico de 384 pocillos con un volumen máximo de 130 μL. Cubra la placa con láminas de plástico transparentes autoadhesivas compatibles con microplacas de polipropileno.
- Prepare la concentración máxima con el tampón libre de DMSO para garantizar el 2% de DMSO en la concentración final. A 147 μL de tampón libre de DMSO, agregue 1,5 μL de material MZ1 a 1 mM y 1,5 μL de DMSO al 100%.
- Prepare la dilución en serie de 3 veces con el tampón de funcionamiento que contiene un 2% de DMSO. A 100 μL del tampón corriente, añadir 50 μL de solución a la concentración máxima preparada en el paso 3.1.3.1, y mezclar bien para preparar la2ª concentración más alta.
- A continuación, transfiera 50 μL de la solución preparada en el paso 3.1.3.2 a los siguientes 100 μL de tampón corriente, mezcle bien para preparar la3ª concentración más alta, y así sucesivamente.
- Ejecute SPR utilizando la configuración de varios ciclos: Modo: Alto rendimiento; tiempo de contacto: 120 s; tiempo de disociación: 300 s; caudal: 50 μL/min.
- Realice el análisis de datos utilizando el software de evaluación proporcionado por el fabricante del instrumento. El análisis en estado estacionario indicó que KD = 26 (± 3) nM y Rmax de ~91% (± 5%) de unión lograda (Figura 4A)
- Preparación del tampón.
- SPR para BVS: MZ1: complejo ternario Brd4BD2
- Prepare el búfer como se describe en el paso 3.1.1.
- Active el chip SA siguiendo el protocolo del fabricante e inmovilice la VHL a ~100 RU. Inyectar una solución proteica de 0,5 μg/mL de VHL a un caudal de 5 μL/min y un tiempo de contacto entre 1-5 min hasta alcanzar una densidad superficial de ~100 RU.
- Prepare las muestras para dos inyecciones de ciclo único de 5 puntos y 5 veces en una placa inferior redonda de poliestireno transparente de 96 pocillos con un volumen máximo de 323 μL y cúbrala con láminas de plástico transparentes autoadhesivas que sean compatibles con las microplacas de poliestireno como se describe en los pasos 3.2.3.1-3.2.3.2.
- Control negativo: el analito contiene Brd4BD2 solamente.
- Preparar la concentración máxima a 25 μM en tampón de funcionamiento con un volumen de 200 μL (#A5)
- Agregue 160 μL de Brd4BD2 a 2 μM en el tampón de funcionamiento a los siguientes 4 pocillos de la izquierda (#A1-A4)
- Transfiera 40 μL de A5 a A4. Mezclar bien.
- Transfiera 40 μL de A4 a A3. Mezclar bien. Siga haciéndolo hasta A1.
- Formación de complejos ternarios: el analito contiene MZ1 y Brd4BD2.
- Prepare la concentración máxima añadiendo 4 μL de MZ1 a 20 μM en una solución de DMSO al 100% a 196 μL que contengan 25,5 μM de Brd4BD2 en tampón libre de DMSO (#B5). Las concentraciones finales son 25 μM Brd4BD2, 100 nM MZ1 y 2% DMSO.
- Agregue 160 μL de Brd4BD2 a 2 μM en el tampón de funcionamiento a los siguientes 4 pocillos de la izquierda (#B1-B4)
- Transfiera 40 μL de B5 a B4. Mezclar bien.
- Transfiera 40 μL de B4 a B3. Mezclar bien. Sigue haciéndolo hasta B1.
- Control negativo: el analito contiene Brd4BD2 solamente.
- Ejecute SPR utilizando la configuración de ciclo único: Tiempo de contacto: 100 s; tiempo de disociación: 720 s; caudal: 50 μL/min. Aplique tres inyecciones de tampón antes de cada muestra para garantizar un fondo estable.
- Análisis de datos: Aplique la tercera inyección del búfer como fondo para la resta. Como control negativo, cuando MZ1 no está presente, la respuesta de SPR entre VHL y Brd4BD2 es despreciable. Cuando MZ1 está presente, el ajuste cinético indica que la interacción entre VHL y el complejo MZ1-Brd4BD2 tiene k on = 7,9 (± 1,5) *107 /M/s, koff = 0,014 s-1 y KD = 1 nM. (Figura 4B)
- SPR para CRBN e interacciones de moléculas pequeñas
- Preparación del tampón.
- Prepare un tampón de 1 L que contenga 50 mM de Tris, 100 mM de NaCl, 1 mM de MgCl 2, 0,5 mM de TCEP, 0,005% de P20, pH 7,5 y páselo por una unidad de filtro de0,2 μm.
- Retire 20 ml de tampón y rellene 20 ml de DMSO al 100 % (2 % de DMSO en el tampón de ejecución final). Es importante mantener el DMSO al 2% en todas las muestras que se describen a continuación (pasos 3.3, 3.4 y 3.5).
- Active el chip SA siguiendo el protocolo del fabricante e inmovilice CRBN a ~800 RU.
- Preparar los compuestos en una dilución seriada de 3 veces y 6 pt en una placa de 384 pocillos con una concentración máxima de 30 μM, como se describe en el punto 3.1.3.
- Ejecute SPR utilizando la configuración de ciclo múltiple: Modo: Alto rendimiento, tiempo de contacto: 60 s, tiempo de disociación: 90 s; caudal: 50 μL/min. Para este sistema, tres inyecciones de tampón son suficientes para obtener un fondo estable. Para otros sistemas de ensayo, puede ser necesario realizar inyecciones de tampón adicionales.
- Realice análisis de datos utilizando el software del fabricante.
NOTA: El análisis de estado estacionario indica que KD de BRD-2512 y BRD-5110 son de alrededor de 3 μM. Sin embargo, la unión solo alcanzó ~ 70%de unión máxima de R en la concentración superior. Tanto la afinidad débil como la forma del sensorgrama indican que es probable que ocurra insolubilidad de compuestos a altas concentraciones. Por lo tanto, el KD real podría ser superior a 3 μM. BRD-4761, que no se une a CRBN, se incluyó como control negativo.
- Preparación del tampón.
- SPR para PPM1D e interacciones de moléculas pequeñas
- Prepare el búfer como se describe en el paso 3.3.1. Utilice un chip sensor de ácido nitriloacético (NTA).
- Aplique un solo ciclo porque la ojiva PPM1D tiene un encendido y apagado k lentos. Regenere el chip NTA utilizando la configuración predeterminada del fabricante e inmovilice PPM1D a ~ 1000 RU después de cada inyección de compuesto (inyecte una solución de proteína de 5 μg/ml a 5 μl/min).
- Preparar los compuestos en una dilución seriada de 3 veces y 5 puntos con la concentración máxima a 400 nM, como se describe en el punto 3.1.3.
- Ejecute SPR utilizando la configuración de un solo ciclo: Modo: alto rendimiento; Tiempo de contacto: 90 s; tiempo de disociación: 600 s; caudal: 50 μL/min. Aplique tres inyecciones de tampón antes de cada compuesto para garantizar un fondo estable.
- Análisis de datos. Aplique la tercera inyección del búfer como fondo para la resta. Como control negativo, BRD-2512 no muestra unión a PPM1D. Para BRD-4761 y BRD-5110, el ajuste cinético indicó que el KD para ambos era de 1-2 nM.
- SPR para CRBN:PROTAC: Complejo ternario PPM1D
- Prepare el búfer como se describe en el paso 3.3.1.
- Activar el chip SA siguiendo el protocolo del fabricante e inmovilizar CRBN a ~35 RU (inyectar una solución proteica de 0,5 μg/mL a 5 μL/min).
- Preparar tres compuestos en una dilución seriada de 3 veces y 6 puntos con una concentración máxima de 30 μM manteniendo [PPM1D] a 1 μM en todas las muestras, incluidos los blancos, utilizando el método descrito en el paso 3.2.3.
- Ejecute SPR utilizando la configuración de varios ciclos: Modo: Alto rendimiento; Tiempo de contacto: 60 s; tiempo de disociación: 90 s; caudal: 50 μL/min.
- Realice análisis de datos utilizando el software del fabricante.
NOTA: Mientras que BRD-2512 y BRD-4761 muestran una respuesta nula o insignificante, BRD-5110 muestra claramente el "efecto gancho" en el estado estacionario con una cinética rápida de encendido/apagado (Figura 5A-C). La respuesta de unión experimental de BRD-5110 (Figura 5C) se encuentra entre la respuesta predicha de la simulación cuando se asume que K D (CRBN, cpd) es de 3 o 10 μM (Figura 5E), lo que sugiere que el K D ternario y el KD binario son muy similares. No hay cooperación aparente de PPM1D: BRD-5110: formación del complejo CRBN.
Resultados
La caracterización del complejo binario BVS: MZ1 y del complejo ternario BVS: MZ1: Brd4BD2 se puede encontrar en la Figura 2 (ITC), la Figura 3 (BLI) y la Figura 4 (SPR) utilizando un tampón muy similar. El KD extraído de los ensayos ortogonales es consistente. La cooperatividad se puede calcular por K D (binario) / KD (ternario), que es altamente positivo (15 de ITC o 26 de SPR).
La caracterización del sistema CRBN:PROTAC: PPM1D se realizó mediante SPR (Figura 5A-D). El CRBN se inmovilizó a ~35 RU's para facilitar la observación de la formación del complejo ternario. La unión de PROTAC por sí sola dio como resultado una señal de <2 RU que está por debajo del ruido. PPM1D en el analito proporciona una alta señal de fondo en la superficie del chip SA, y la concentración más alta que se puede aplicar es de alrededor de 1 μM. Este valor es inferior al KD entre CRBN y su ojiva ≥3 μM), por lo que se espera un "efecto gancho". SPR es lo suficientemente sensible como para detectarlo, lo que concuerda bien con la simulación (Figura 5E). La simulación se realizó utilizando los equilibrios no cooperativos de la literatura19 combinados con el cálculo clásico de SPR [Response max = (ResponseLigand × MassImmobilization)/Mass Ligand]. Dado que el KD entre CRBN y el compuesto no se determina con precisión debido a la insolubilidad del compuesto a alta concentración, la simulación se realizó utilizando cuatro KD supuestos: 1 μM, 3 μM, 10 μM o 30 μM. Los resultados experimentales se situaron entre las curvas simuladas de 3 μM y 10 μM, que es casi idéntica a la KD en el sistema binario, lo que sugiere que no hay cooperatividad.

Figura 1: Ilustración de tres escenarios de enlace y definición de diferentes KD. (A) Sistemas clásicos de dos componentes. (B) Sistema de tres componentes en el que un extremo de PROTAC puede saturarse, por lo tanto, puede evaluarse como un sistema de dos componentes. (C) Sistema de tres componentes en el que se observa el "efecto gancho". Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Resultados del ITC. Valoración de la VHL en el complejo MZ1 (izquierda) o MZ1:Brd4BD2 (derecha). Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Resultados de BLI. MZ1 media la formación del complejo ternario BVS: MZ1: Brd4BD2. (A) Datos brutos. (B) Resta de señales de fondo donde [MZ1] = 0. (C) Ajuste cinético de B para extraer k on, koff y KD. Haga clic aquí para ver una versión más grande de esta figura.

Figura 4: Resultados de la SPR . (A) Unión de MZ1 a la BVS. (B) MZ1:Brd4BD2 binario de enlace a BVS. Haga clic aquí para ver una versión más grande de esta figura.
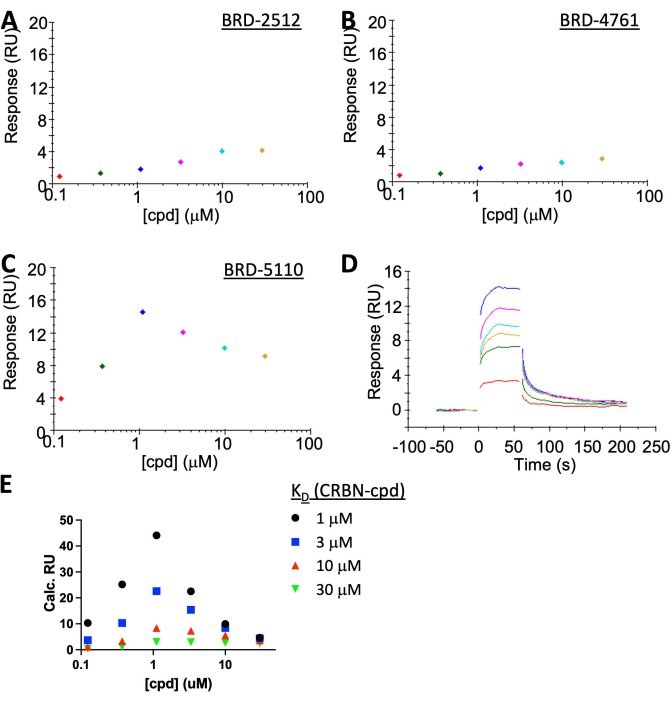
Figura 5: Resultados de SPR que muestran el "efecto gancho" de un PPM1D-PROTAC representativo. La CRBN se inmovilizó en la superficie del chip SA, mientras que [PPM1D] se mantuvo a 1 μM en el analito para todos los casos. (A) BRD-2512, un compuesto que solo se une a CRBN, casi no da respuesta. (B) BRD-4761, un compuesto que solo se une a PPM1D, tampoco da respuesta. (C,D) BRD-5110, un PROTAC con la ojiva de CRBN en BRD-2512 y la ojiva de PPM1D en BRD-4761, indujo la formación del complejo ternario. (E) Una simulación de los resultados de SPR asumiendo que el KD entre CRBN y el compuesto es 1 μM (negro), 3 μM (azul), 10 μM (rojo) o 30 μM (verde). La curva BRD-2512 está entre 3 μM y 10 μM, que está muy cerca de la binaria KD medida, lo que sugiere que no hay cooperatividad (cooperatividad = 1). Haga clic aquí para ver una versión más grande de esta figura.
Discusión
La caracterización biofísica de las interacciones binarias y ternarias entre las moléculas PROTAC y sus socios de unión a proteínas puede proporcionar información única y complementaria en relación con los sistemas celulares ampliamente utilizados. Comprender la afinidad entre cada ojiva de una molécula PROTAC y sus socios de unión a proteínas puede ayudar a guiar los esfuerzos de química médica hacia la optimización de esas interacciones. Las estructuras cristalinas de los complejos ternarios PROTAC publicadas anteriormente han revelado que los átomos en la región enlazadora pueden formar interacciones con uno o ambos de los socios de unión a proteínas16,20. La determinación experimental de la cooperatividad de la formación de complejos ternarios puede apoyar la optimización del enlazador.
En este informe se describe la utilización de tres técnicas biofísicas diferentes que pueden proporcionar información sobre las afinidades de unión entre las moléculas de PROTAC y sus socios de unión a proteínas. En el Método 1 se detalla la configuración experimental de la calorimetría de titulación isotérmica (ITC) para la molécula PROTAC, MZ1, el complejo VHL E3 ligasa y el bromodominio Brd4BD2. Los resultados del ITC mostraron KD de 59 nM para la interacción binaria entre MZ1 y BVS y de 4 nM para la interacción ternaria entre BVS y MZ1 premezclado y Brd4BD2. Las afinidades fueron consistentes con las observadas en SPR (unión inmovilizada de la VHL a MZ1 KD = 26 nM, inmovilización de la unión de la VHL a MZ1 premezclada y Brd4BD2 K D=1 nM) y BLI (KD= 2,8 nM). Si bien los resultados de ITC KD para la unión de VHL a MZ1 son consistentes con los valores reportados16, la estequiometría obtenida es diferente. Una posible explicación para este resultado es la escasa solubilidad de MZ1 en el tampón basado en HEPES utilizado en el protocolo descrito aquí, mientras que los resultados de la literatura se generaron utilizando un tampón basado en Bis-tris. Los autores prefirieron utilizar los mismos componentes de búfer en SPR, ITC y BLI.
El método 2 describe la configuración experimental para el análisis BLI de la interacción de la enfermedad de VHL inmovilizada, una concentración fija de Brd4BD2 y concentraciones variables de MZ1. Debido a las limitaciones de sensibilidad de la técnica, se pudieron generar valores de KD, k on yk off para la formación de complejos ternarios, pero no para la interacción binaria entre MZ1 y las proteínas.
El método 3 describe múltiples ensayos de SPR. La SPR es más sensible que la BLI y se puede aplicar para observar tanto las interacciones proteína-molécula pequeña (binaria) como proteína-proteína (ternaria). En este último caso, las señales de fondo deben controlarse cuidadosamente, ya que la proteína del analito podría dar señales altas e inestables. SPR es muy sensible a los reactivos con un alto índice de refracción, incluidos el DMSO, el glicerol y los detergentes. Si la proteína se almacena en el tampón que contiene glicerol o detergente, el tampón de funcionamiento debe contener concentraciones equivalentes de esos componentes. Alternativamente, la aplicación de cromatografía de exclusión por tamaño los elimina por completo antes de cualquier experimento de SPR. Se debe tener cuidado de hacer coincidir estrechamente las concentraciones de DMSO entre las muestras de tampón y analito. Las correcciones del disolvente DMSO se realizan de acuerdo con las instrucciones del fabricante.
El método del paso 3.1 describe el ensayo SPR para la interacción binaria VHL-MZ1. El método 3.2 describe el ensayo SPR para el complejo ternario BVS: MZ1:Brd4 BD2 donde la VHL está inmovilizada y el analito es Brd4 BD2 solo o el complejo MZ1:Brd4BD2. En este sistema, la interacción entre Brd4BD2 y VHL es insignificante. La formación del complejo ternario es altamente cooperativa (ɑ = 26). La tasa de desconexión para la formación de complejos ternarios es de 0,014 s-1, lo que requiere el uso de cinética de ciclo único. Los resultados del ITC también muestran una formación de complejos ternarios altamente cooperativos (ɑ=15). Los métodos SPR en los pasos 3.3, 3.4 y 3.5 describen ensayos para evaluar la formación de un complejo entre CRBN y PPM1D inducido por la presencia de una molécula de PROTAC, BRD-5110. La molécula PROTAC tiene una afinidad débil por CRBN (K D ~3 μM) y una fuerte afinidad por PPM1D (KD = 1-2 nM). Como resultado, la unión débil a CRBN no está saturada y da lugar a un "efecto gancho" observado. Si bien es posible aumentar la solubilidad del ligando aumentando la concentración de DMSO utilizada en el experimento, es importante en esos casos monitorear cuidadosamente la estabilidad de las proteínas que pueden verse afectadas negativamente por altas concentraciones de DMSO. Además, el DMSO tiene un alto calor de disolución que puede oscurecer el calor de unión de los ligandos a la proteína. Se debe tener cuidado de hacer coincidir las concentraciones de DMSO de la solución en la jeringa y la solución en la célula. Los autores recomiendan la diálisis de las dos soluciones frente a la misma preparación tampón.
Se proporcionan recomendaciones y directrices generales basadas en los experimentos realizados y reportados aquí. Cuando las afinidades de las interacciones binarias entre las moléculas PROTAC y sus socios de unión a proteínas son fuertes (KD <1 μM), SPR proporciona afinidades confiables y reproducibles junto con información valiosa sobre la cooperatividad de la formación de complejos ternarios. Cuando las afinidades de la interacción binaria entre uno de los socios de unión a proteínas y la molécula PROTAC son débiles (KD >1 μM), será necesario modificar la configuración del ensayo. En esos casos, el uso de simulaciones moleculares en las que las constantes de unión son fijas y las concentraciones de ligando y analito son variadas puede ser valioso para guiar el diseño del ensayo y la interpretación de los resultados experimentales. Los ensayos ITC proporcionan información importante sobre la estequiometría de la unión, pero requieren significativamente más reactivos proteicos y compuestos en relación con SPR y BLI. Además, la solubilidad de la molécula PROTAC puede ser limitante para los experimentos de ITC. BLI tiene un mayor rendimiento que ITC y requiere menos proteínas y reactivos compuestos. Sin embargo, debido a las limitaciones de sensibilidad, BLI solo se puede utilizar para evaluar la formación de complejos ternarios y no las interacciones binarias entre las moléculas de PROTAC y sus socios de unión a proteínas. Se recomienda que la SPR se utilice para las pruebas rutinarias de los ensayos de unión a PROTAC binarios y ternarios y los ensayos BLI e ITC utilizados para la validación ortogonal de los resultados de la SPR.
Divulgaciones
Los autores no tienen intereses financieros contrapuestos ni otros conflictos de intereses.
Agradecimientos
Este trabajo fue apoyado por un premio de Innovación y Desarrollo Tecnológico del Centro para el Desarrollo de Terapias del Instituto Broad del MIT y Harvard. Los autores desean agradecer a los miembros del equipo directivo superior y al comité de revisión por su apoyo a este trabajo.
Materiales
| Name | Company | Catalog Number | Comments |
| 96-plate | Greiner | 655076 | flat-bottom, black plates used In BLI experiments |
| 96-well plate | Nunc | 73520-120 | Plate use for ITC sample preparation |
| 96-well plate | Greiner | 650101 | Plate used to prepare samples for SPR experiments |
| Auto iTC200 micro-calorimeter | Malvern Panalytical | Instrument used to perform ITC experiments. Product discontinued. | |
| Biacore S200 | Cytiva | 29136649 | Instrument used to perform SPR experiments |
| MZ1 | ProbeChem | PC-60099 | PROTAC that binds to VHL and Brd4BD2 |
| NTA sensor chip | Cytiva | BR100532 | SPR chip used to perform SPR experiments involving PPM1D |
| Octet Red-384 | Sartorius | Instrument used to perform BLI experiments. Product discontinued. | |
| Plate cover | Malvern | PQA0001 | Cover for Nunc 96-well plate (73520-120) |
| Plate cover | Cytiva | 28975816 | Plate cover for Greiner plate (650101) |
| Series S SA sensor chip | Cytiva | BR100531 | SPR chip used to perform SPR experiments involving MZ1:VHL:BRD4 |
| Streptavidin (SA) Dip and Read Biosensors | Sartorius | 18-509 | Coated sensors used in BLI experiments |
Referencias
- Balaji, V., Hoppe, T. Regulation of E3 ubiquitin ligases by homotypic and heterotypic assembly. F1000Research. 9, (2020).
- Song, L., Luo, Z. -. Q. Post-translational regulation of ubiquitin signaling. Journal of Cell Biology. 218 (6), 1776-1786 (2019).
- Yang, Q., Zhao, J., Chen, D., Wang, Y. E3 ubiquitin ligases: styles, structures and functions. Molecular Biomedicine. 2 (1), 23 (2021).
- Grice, G. L., Nathan, J. A. The recognition of ubiquitinated proteins by the proteasome. Cellular and Molecular Life Sciences: CMLS. 73 (18), 3497-3506 (2016).
- Chirnomas, D., Hornberger, K. R., Crews, C. M. Protein degraders enter the clinic - a new approach to cancer therapy. Nature Reviews Clinical Oncology. 20 (4), 265-278 (2023).
- Toure, M., Crews, C. M. Small-molecule PROTACS: New approaches to protein degradation. Angewandte Chemie (International ed. In English). 55 (6), 1966-1973 (2016).
- Ottis, P., Toure, M., Cromm, P. M., Ko, E., Gustafson, J. L., Crews, C. M. Assessing different E3 ligases for small molecule induced protein ubiquitination and degradation. ACS Chemical Biology. 12 (10), 2570-2578 (2017).
- Riching, K. M., et al. Quantitative live-cell kinetic degradation and mechanistic profiling of PROTAC mode of action. ACS Chemical Biology. 13 (9), 2758-2770 (2018).
- Nabet, B., et al. The dTAG system for immediate and target-specific protein degradation. Nature Chemical Biology. 14 (5), 431-441 (2018).
- Paiva, S. -. L., Crews, C. M. Targeted protein degradation: elements of PROTAC design. Current Opinion in Chemical Biology. 50, 111-119 (2019).
- Hershko, A., Ciechanover, A. The ubiquitin system. Annual Review of Biochemistry. 67, 425-479 (1998).
- Chan, K. -. H., Zengerle, M., Testa, A., Ciulli, A. Impact of target warhead and linkage vector on inducing protein degradation: Comparison of bromodomain and extra-terminal (BET) degraders derived from triazolodiazepine (JQ1) and tetrahydroquinoline (I-BET726) BET inhibitor scaffolds. Journal of Medicinal Chemistry. 61 (2), 504-513 (2018).
- Roy, M. J., et al. SPR-measured dissociation kinetics of PROTAC ternary complexes influence target degradation rate. ACS Chemical Biology. 14 (3), 361-368 (2019).
- Pierce, N. W., Kleiger, G., Shan, S., Deshaies, R. J. Detection of sequential polyubiquitylation on a millisecond timescale. Nature. 462 (7273), 615-619 (2009).
- Gadd, M. S., et al. Structural basis of PROTAC cooperative recognition for selective protein degradation. Nature Chemical Biology. 13 (5), 514-521 (2017).
- Nahta, R., Castellino, R. C. Phosphatase magnesium-dependent 1 δ (PPM1D), serine/threonine protein phosphatase and novel pharmacological target in cancer. Biochemical Pharmacology. 184, 114362 (2021).
- Douglass, E. F., Miller, C. J., Sparer, G., Shapiro, H., Spiegel, D. A. A comprehensive mathematical model for three-body binding equilibria. Journal of the American Chemical Society. 135 (16), 6092-6099 (2013).
- Zorba, A., et al. Delineating the role of cooperativity in the design of potent PROTACs for BTK. Proceedings of the National Academy of Sciences. 115 (31), E7285-E7292 (2018).
- Fairhead, M., Howarth, M. Site-specific biotinylation of purified proteins using BirA. Methods in Molecular Biology. 1266, 171-184 (2015).
- Nowak, R. P., et al. Plasticity in binding confers selectivity in ligand-induced protein degradation. Nature Chemical Biology. 14 (7), 706-714 (2018).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados