Method Article
Utilisation de co-culture pour détecter les interactions interspécifiques par voie chimique
Dans cet article
Résumé
Les bactéries produisent des composés sécrétés qui ont le potentiel d'affecter la physiologie de leurs voisins microbiennes. Nous décrivons ici un écran de co-culture qui permet la détection de ces médiation chimiquement interspécifiques interactions en mélangeant les microbes du sol avec des souches rapporteurs fluorescents transcription de Bacillus subtilis sur milieu solide.
Résumé
Dans la nature, les bactéries existent rarement dans l'isolement, ils sont plutôt entourés d'un large éventail d'autres micro-organismes qui modifient l'environnement local en sécrétant des métabolites. Ces métabolites ont le potentiel de moduler la physiologie et la différenciation de leurs voisins et microbiennes sont des facteurs susceptibles importants dans la création et le maintien de communautés microbiennes complexes. Nous avons développé un écran de co-culture à base de fluorescence pour identifier ces interactions microbiennes médiées par voie chimique. L'écran consiste à combiner une souche rapporteur de transcription fluorescent avec des microbes de l'environnement sur milieux solides et en permettant aux colonies de se développer en co-culture. Le rapporteur transcriptionnel fluorescent est conçu de telle sorte que la souche bactérienne choisie est fluorescent lorsqu'il exprime un phénotype particulier d'intérêt (à savoir la formation de biofilms, la sporulation, la production de facteurs de virulence, etc.) Le criblage est réalisée dans des conditions de croissance WHEre ce phénotype n'est pas exprimé (et donc de la souche rapporteuse est typiquement non-fluorescent). Quand un microbe environnement sécrète un métabolite qui active ce phénotype, il diffuse à travers l'agar-agar et active la construction de rapporteur fluorescent. Cela permet au-métabolite produisant induire microbe devant être détectée: ce sont les colonies non fluorescentes plus proximales des colonies fluorescentes. Ainsi, cet écran permet l'identification des microbes de l'environnement qui produisent des métabolites diffusibles qui activent une réponse physiologique particulier dans une souche de journaliste. Cette publication explique comment: a) sélectionner des conditions de dépistage de co-culture appropriées, b) préparer le journaliste et les microbes de l'environnement pour le dépistage, c) effectuer l'écran de co-culture, d) isoler putatif organismes induisant, et e) de confirmer leur activité dans un écran secondaire. Nous avons développé cette méthode pour dépister les organismes du sol qui activent biofilm matrice-production dans Bacillus subtilis
Introduction
Nous sommes intéressés à comprendre comment les métabolites que les bactéries sécrètent affectent la physiologie et le développement des microbes voisins. De nombreux métabolites ont été caractérisés pour leurs effets bioactifs sur d'autres microbes. Deux exemples bien décrits comprennent les antibiotiques, qui inhibent la croissance d'autres micro-organismes et des molécules de détection de quorum, qui altèrent l'expression génique globale d'autres microbes. Cependant, les bactéries produisent de nombreuses autres petites molécules de produits naturels qui n'ont pas de bioactivité connus 1. Nous émettons l'hypothèse que les bactéries ont évolué et conservé la capacité de produire certains de ces métabolites car ils leur permettent de moduler la physiologie cellulaire de leurs voisins microbiennes dans les communautés microbiennes complexes qui existent dans la plupart des bactéries.
Les types de cellules de Bacillus subtilis
Nous avons concentré nos études sur les interactions microbiennes médiation chimique qui impliquent Bacilsubtilis UGB. Ce n'est pas seulement en raison de son statut de bactérie modèle à Gram positif et les outils génétiques résultantes disponibles pour sa manipulation, mais aussi en raison de sa capacité à se différencier en différents types cellulaires caractérisés. Des exemples comprennent des cellules qui sont: la natation, produisant la matrice extracellulaire qui est requise pour la formation de biofilm robuste; compétentes pour absorber l'ADN à partir de l'environnement, et la sporulation, entre autres deux. Chacun de ces types de cellules exprime un régulon de transcription caractéristique qui les rend physiologiquement et / ou physiquement distinct de leurs frères et sœurs génétiquement identiques. Dans de nombreuses conditions de croissance, plusieurs types de cellules coexistent en tant que sous-populations différentes au sein d'une seule colonie de B. cellules subtilis 3. Bien que de nombreuses espèces de bactéries peuvent présenter analogue type de cellule hétérogénéité, ce phénomène a été particulièrement bien étudié dans B. subtilis.
En particulier, les gènes qui sont epuegulated à l'intérieur de chacun de ceux-ci spécifique B. types de cellules subtilis ont été identifiés. L'identification de ces gènes régulés à la hausse est essentiel pour le travail décrit ici parce que beaucoup de ces phénotypes microbiennes d'intérêt sont difficiles ou impossibles à observer directement. Par exemple, nous ne pouvons pas détecter visuellement un trait comme la natation sur de solides (1,5%) des plaques d'agar, même si une sous-population de B. cellules subtilis produisent des flagelles dans ces conditions 3. Un autre exemple est biofilm matrice-production. la production de la matrice peut être visualisée par la morphologie des colonies (comme il en résulte colonies ridées macroscopiquement), mais seulement sur certains milieu de culture, et seulement après plusieurs jours de croissance 4. Cependant, en connaissant les gènes qui sont régulés à la hausse au cours de la différenciation, on peut construire reporters transcriptionnels qui agissent comme des marqueurs de différenciation cellulaire dans ces types cellulaires.
constructions Reporter
Ces fluorescent transcriptional reporters sont constitués par les promoteurs pour des gènes spécifiques du type cellulaire entraînant la production d'un gène rapporteur, par exemple une protéine fluorescente. Les exemples incluent P hag-YFP (pour la natation cellules), P Tapa-YFP (pour les cellules de la matrice produisant biofilm), et P SSPB-YFP (pour la sporulation des cellules), où P x indique la région du promoteur pour le gène x. Ces constructions rapporteurs sont intégrés dans un locus sur le chromosome neutre (figure 1 et le voir ci-dessous) de sorte que la régulation native du phénotype est laissée intacte. Cependant, maintenant, quand une cellule exprime ce phénotype, il exprime aussi une protéine fluorescente. Ceci permet d'obtenir une lecture facile à visualiser de l'activation du comportement particulier phénotypique, ce qui nous permet de cribler des microbes qui activent cette réponse physiologique. Bien que de tels rapporteurs sont couramment utilisés en microbiologie, ils n'ont pas été largement appliquée dans des écrans à identifinteractions métaboliques entre les microbes y avant cette méthode a été décrite 5.
Il ya un certain nombre de considérations importantes dans la conception et la construction de-spécifique de type cellules souches rapporteurs. Nous avons utilisé journalistes fluorescents exclusivement transcription, bien que d'autres types de constructions sont certainement possibles. Nous déconseillons l'utilisation de fusions traductionnelles comme marqueurs de différenciation de type de cellule dans l'écran, cependant, pour deux raisons: 1) le désir de quitter le spécifique de type cellulaire protéine native imperturbable, et 2) la reconnaissance que diffuse, cellulaire large fluorescence sera plus facile à détecter que les points lacrymaux localisée dans les cellules (commun avec fusions traductionnelles).
sélection d'un gène rapporteur
Après avoir décidé d'utiliser la transcription en-lue, le gène rapporteur doit être sélectionnée (par exemple LacZ, fluorescence, ou la luciférase). LacZ a l'avantage de nécessiter le moins spécialeized matériel de détection, mais il ya un risque beaucoup plus élevé de faux positifs parmi les microbes de l'environnement. Dans nos mains, le niveau du lac organismes + parmi les microbes du sol de fond était prohibitif (>> 10% des microbes du sol étaient bleues (Lac +) sur des plaques X-gal; données non présentées). Il est possible que la concentration en titrant de X-gal dans le milieu, ce qui pourrait être optimisé pour permettre l'utilisation d'un rapporteur X-gal, mais nous n'avons pas cherché ce produit. Luciférase offre une grande sensibilité de détection et est le journaliste le plus orthogonal: il n'y a presque aucune chance de microbes de l'environnement étant intrinsèquement luminescent. Cependant, nous avons constaté qu'il est difficile d'identifier les instruments à notre institution qui permettait la détection de luminescence dans des boîtes de Pétri entières, comme la plupart ont été conçus pour numériser les régions ne localisées dans des plaques multi-puits. Il pourrait également y avoir des complications dans la visualisation des colonies luminescents d'une manière qui a également permis à la physique simultanée estolation des organismes induisant. Tout en utilisant des fiduciaires peut avoir rendu cela possible, nous la place choisi d'utiliser journalistes transcription fluorescentes, qui ont été prouvée à travailler en B. subtilis, à condition sensibilité adéquate des taux de détection et de faux positifs faibles entre les organismes du sol, et autorisés à utiliser des instruments facilement disponibles à la fois pour la visualisation et les procédures d'isolement.
sélection de fluorophore
Le fluorophore spécifique choisi dépendra de vos espèces bactériennes, le milieu de croissance agar que vous utilisez, et le filtre de fluorescence particulier définit dont vous disposez. Avec nos instruments, nous avons constaté que les deux B. subtilis eux-mêmes et l'agar ils ont été cultivés sur exposé moins de fluorescence de fond quand YFP (protéine fluorescente jaune) filtres ont été utilisés, ce qui rend ce journaliste supérieure à la GFP (protéine fluorescente verte) dans nos mains colonies. L'usage des codons des protéines fluorescentes sontsouvent optimisé pour les eucaryotes, il est donc important de choisir un fluorophore soit connu de la littérature de travailler dans vos espèces bactériennes, ou pour tester explicitement l'aide d'un promoteur constitutif. Un grand nombre de constante évolution variantes de protéines fluorescentes sont actuellement disponibles 6, qui ont été examinés dans un certain nombre de sources 7,8, dont certains prévoient explicitement des conseils sur le choix d'une protéine fluorescente correspondant à votre expérience 9.
sélection de promoteur
La sélection d'un promoteur dépendra en grande partie sur le type de cellule ou phénotype d'intérêt. Pour les organismes tels que B. subtilis, certains gènes rapporteurs spécifiques du type cellulaire ont été établies dans la littérature. Pour d'autres souches bactériennes, en examinant microarray ou données transcription sera nécessaire de fournir des informations sur les gènes qui sont fortement régulés à la hausse dans les conditions où votre intérêt i cellulaires manifesté. Une étude récente a catalogué la transcription de B. subtilis moins de 104 différentes conditions de croissance à l'aide de puces à ADN de carrelage 10. Ce document fournit des informations complètes sur les gènes sont fortement régulés à la hausse dans des conditions différentes, ce qui est inestimable pour les phénotypes moins bien caractérisés.
Plutôt que de cartographier les régions promotrices précises pour chaque gène d'intérêt, nous utilisons généralement tout simplement la séquence 200-500 pb en amont du gène en tant que promoteur. La longueur de la séquence exacte dépend du contexte génomique: les régions plus courtes sont utilisés lorsque cela est nécessaire pour éviter d'inclure des régions codantes en amont du cadre de lecture ouvert voisin.
Loci et l'intégration neutre
Comment maintenir la construction reporter dans votre souche bactérienne devient la dernière question de la conception d'une souche de transcription rapporteur fluorescent. Chez les bactéries, les gènes d'intérêt sont souvent maintenussur des plasmides à l'aide de la sélection antibiotique. Cependant, il peut ne pas être possible d'utiliser des antibiotiques au cours de la co-culture sans tuer les microbes environnementaux. Si plasmides sont maintenus de façon stable dans vos espèces bactériennes, il peut être possible cultiver vos bactéries contenant un journaliste porté par un plasmide en présence d'antibiotiques pour préparer votre rapporteur pour le dépistage, puis éliminer les antibiotiques au cours de la co-culture elle-même dans l'espoir que le plasmide être maintenue suffisamment pour permettre la fluorescence. Toutefois, si les plasmides sont facilement perdus dans votre bactérie, ou sont perdus dans des conditions de stress, ce ne sera pas une option viable. Dans de nombreux cas, la meilleure solution consiste à intégrer la construction de rapporteur sur le chromosome bactérien, ce qui permet le maintien stable du rapporteur, même en l'absence de sélection. Pour l'intégration de ne pas perturber l'expression normale ou d'un règlement de votre gène d'intérêt, nous vous recommandons d'intégrer dans un site ectopique sur le chromosome que can agir comme un «lieu neutre». Dans B. subtilis ces sites d'intégration sont des gènes qui - lorsqu'ils sont mutés - transmettre un phénotype dans certains médias minimale (permettant intégrants à être identifiés sans sélection antibiotique), mais ne modifient pas la croissance ou le taux de sporulation dans les médias riches, et inclure ces gènes que amyE, lacA, thrC, pyrD, GLTA, et Saca (transport de la capacité à utiliser l'amidon, β-galactosides, la thréonine, l'uracile, le glutamate et le saccharose, respectivement) 11-13.
Bien que l'intégration de ces gènes ont été utilisés de manière fiable pendant de nombreuses années à B. subtilis (en particulier au amyE et lacA), la même connaissance peut ne pas être disponible pour des gènes dans de nombreuses autres espèces bactériennes. L'utilisation de sites de fixation du phage sont une bonne alternative pour les sites d'intégration chromosomiques neutres: de nombreuses spécifiques à l'espèce 14 à 16, ainsi que les sites d'intégration généraux, tels que le site de fixation de Tn7 (att) ont Tn7été identifié et utilisé pour les insertions de gènes chez de nombreuses espèces bactériennes 17,18.
Microbes de l'environnement
Nous utilisons le sol comme une source directe des microbes de l'environnement pour l'écran de co-culture. Le sol contient une grande diversité de microbes, et bon nombre de ces organismes sont riche source de produits naturels. En utilisant des suspensions liquides de sol placés directement sur des plaques avec notre souche rapporteur transcriptionnel fluorescente (sans isolement préalable des bactéries du sol), nous simplifions grandement l'approche expérimentale. Le sol peut être utilisé soit immédiatement après la récolte, ou être congelé à -80 ° C pour une utilisation future. Utilisation immédiate a l'avantage une plus grande diversité de microbes peut potentiellement être cultivé, y compris ceux qui ne survivra pas bien la congélation. Il présente l'inconvénient que la concentration des organismes du sol cultivable partir de ces échantillons est inconnue, ce qui augmente le nombre de plaques d'écran qui doit être utilisé. Delayed utilisation présente l'avantage que les cfu / ml pour chaque source dans le sol peuvent être déterminés à l'avance, ce qui permet un certain nombre de colonies optimisé pour être cultivée sur chaque plaque d'écran. Cependant, elle nécessite que les organismes du sol soient capables de survivre à la congélation.
Notez que la diversification de la piscine de l'inducteur en cours d'examen (c'est à dire les sources de sol) semble être plus efficace dans l'identification de nouveaux interspécifiques interactions que le dépistage en profondeur sur le même sol: une plus grande diversité phylogénétique a été observée dans les résultats identifiés dans notre écran matrice induction sources de sol supplémentaires ont été examinées plutôt que le dépistage des mêmes sources plus approfondie du sol (EA queue et R. Kolter, Harvard Medical School, résultats non publiés).
Vue d'ensemble
L'approche que nous décrivons ici est simple en termes de ses exigences techniques. Il s'agit de: 1) la construction d'une transcription rapporteur fluorescent dans B. subtilis ou uned'autres espèces bactériennes d'intérêt, 2) l'identification des conditions dans lesquelles cette rapporteur n'est pas activé, 3) la préparation d'aliquotes de cette souche rapporteuse et les organismes à cribler (dans notre sol de cas, mais d'autres sources peuvent être utilisées à la place), 4) le mélange de ceux-ci deux ensembles de microbes sur des milieux solides, 5) l'identification et l'isolement des organismes induisant putatif, et 6) qui confirme que ces micro-organismes ne activent en effet ce phénotype dans un écran secondaire. Une fois identifiés, ces organismes et de leurs métabolites nous fournissent des outils chimiques pour moduler le comportement des bactéries, pour étudier la physiologie bactérienne et interactions microbiennes, et d'agir éventuellement en tant que nouveaux échafaudages pour des composés thérapeutiques futures.
Protocole
Une. Sélectionner un gène rapporteur et un rapporteur fluorescent Construire transcriptionnelle
Pour B. subtilis:
- Voir l'article JoVE en référence 19 pour un protocole décrivant la construction de journalistes transcription fluorescentes dans Bacillus subtilis.
Pour d'autres espèces bactériennes:
- Identifier un gène qui est régulé à la hausse au cours de la réponse physiologique d'intérêt. Ceci peut être basé sur la littérature existante ou analyse transcriptionnelle du microbe dans des conditions particulières.
- Construire un journaliste de la transcription de ce gène fluorescent pour agir comme un proxy pour le changement dans le phénotype. Cette construction doit comprendre le promoteur de ce gène régulée à la hausse entraînant la production d'une protéine fluorescente approprié (voir la figure 1).
- Intégrer cette construction dans un lieu neutre sur le chromosome. Cela garantit que reg natiflation du gène d'intérêt n'est pas perturbé, et évite la nécessité de mécanismes de sélection de plasmide (par exemple les antibiotiques) qui pourraient interférer avec la croissance des microbes de l'environnement.
2. Déterminer les conditions de co-culture
Pour B. subtilis P Tapa-YFP journaliste:
- Utilisez 0.1x LB, Lennox (1 g de tryptone, 0,5 g d'extrait de levure, 0,5 g de NaCl par litre) moyen pour ce journaliste, puisque B. subtilis matrice-production est minime sur Luria Broth 20. Ce milieu permet B. colonies subtilis à croître à submillimétrique mais la taille observable, tout en permettant à divers taxons de terre pour cultiver 5.
- Inclure tampon 100 mM MOPS pour minimiser les changements de pH potentiels.
Pour d'autres espèces bactériennes:
- Utilisez les données publiées transcription ou tester empiriquement diverses conditions de culture pour identifier celui où le microbe se développe mais le activation du rapporteur fluorescent est négligeable (pour permettre son activation à détecter lorsque la souche rapporteur est cultivé dans la co-culture avec des microbes induisant.)
- Utilisez un milieu à faible teneur en éléments nutritifs (par rapport à traditionnellement riches milieux microbiologiques) lors de la sélection des microbes de l'environnement de milieux oligotrophes (comme le sol), depuis de nombreuses bactéries oligotrophes ne poussent pas lorsqu'ils sont présentés avec des conditions élevées d'éléments nutritifs 21. Un milieu nutritif faible permet également de réduire la taille des colonies, ce qui augmente le débit de l'écran.
- Sélectionnez un milieu à faible fluorescence de fond et une bonne clarté optique.
- Optimiser la température de croissance pour permettre à la fois la souche rapporteur et les microbes de l'environnement de croître en même temps.
- Envisager l'ajout d'un agent tampon. L'utilisation de la mémoire tampon dans les plaques réduira la possibilité de détecter des changements de pH physiologiques à médiation par 22, à moins que ces interactions sont d'intérêt.
3. Préparer Reporter aliquotes
Pour B. subtilis P Tapa-YFP journaliste:
- Streak souche rapporteuse de -80 ° C stock congelé sur une plaque LB frais à l'aide d'un cure-dent stérile ou bâton applicateur.
- Cultiver une nuit à 30 ° C.
- Effectuer des dilutions en série dans une culture liquide pour réduire au minimum la fluorescence de fond résultant de la croissance sur un milieu solide:
- Inoculer une culture liquide de 5 ml de LB et de croître en secouant à 37 ° C.
- Lorsque la culture atteint une DO600 ~ 0,6, diluer dans 5 ml LB frais jusqu'à une DO 600 de 0,02.
- Croître à 37 ° C en agitant de nouveau jusqu'à ce que les cultures atteint OD 600 ~ 0,6.
- Répétez dilutions de croissance de série un total de 3x.
- Laissez culture finale de dilution en série pousser à OD 600 ~ 0,4.
- Ajouter du glycérol à 15-20%.
- Aliquote 50-200 ul dans 0,5 ml microtubes de geler et à -80 ° C.
- Mamanke aliquotes équivalentes pour la souche parentale non fluorescent du reporter (ils seront tenus au cours du dépistage secondaire).
Pour d'autres espèces bactériennes:
- Utiliser des cellules qui ont une faible fluorescence de fond (par exemple sont cultivées dans des conditions où il existe peu d'expression à partir du promoteur utilisé dans la reporter).
- Faire et congeler des aliquotes contenant ufc / ml connus (unités formatrices de colonies par ml de formation) de la souche rapporteur de sorte qu'un nombre approprié de colonies peut être cultivé sur chaque plaque d'écran de co-culture (voir la section ci-dessus pour plus de détails).
- Faire aliquotes équivalentes pour la souche parentale non fluorescent (ils seront tenus au cours du dépistage secondaire).
4. Obtenir des échantillons de sol
- Recueillir le sol dans des tubes coniques stériles ou des sacs stériles aide d'une spatule, en rejetant les 0,5 premiers centimètres du sol de surface exposée.
- Ajouter une solution saline stérile (0,85% de NaCl) à un rapport de 10 ml pour 1 gdu sol pour faire une suspension de sol.
- Sélectionnez une méthode pour déloger les bactéries de particules de sol: a) l'utilisation immédiate de l'échantillon frais ou b) utilisation différée après la congélation de l'échantillon.
- Pour une utilisation immédiate, vortex suspension pendant 1 min.
- Pour une utilisation différée, mélanger suspension de sol dans un mélangeur pour trois cycles 1 min, placer le bol sur la glace pendant 1 min silences entre les cycles de mélange.
- Laissez suspension de sol se contenter de ~ 1 min.
- Déplacez couche aqueuse supérieure dans un nouveau tube.
- Ajouter du glycérol à une concentration finale de 15 à 20%.
- Aliquote 50-200 ul dans 0,5 ml microtubes de geler et à -80 ° C.
5. Déterminer ufc / ml de Frozen Reporter Et aliquotes de sol
- Décongeler un sol et journaliste aliquote congelé. Les microbes du sol peuvent être décongelés sur la glace. Parce que B. subtilis lyse à 4 ° C, ces aliquotes doivent être décongelés rapidement à la température ambiante afin de minimiser le temps passé à basse température.
- Faire deux seria répliquéel dilutions (à 10 -8) à 0,1 x LB ou un autre tampon isotonique.
- Plate cinq spots ul de chaque dilution en série sur des plaques d'agar-agar du même milieu qui sera utilisé pour le dépistage de la co-culture.
- Croître à RT (ou la température qui sera utilise pour le dépistage).
- Le lendemain, compter le nombre de colonies dans chaque endroit et calculer ufc / ml de chacune des aliquotes congelées.
6. Confirmez aliquotes concentrations pour les plaques de l'écran de propagation
- Décongeler une aliquote congelé comme dans l'étape 5.1.
- Diluer à 1 x 10 5, 2,5 x 10 5, 5 x 10 5, 1 x 10 6 et 2,5 x 10 7 cfu / ml.
- Ajouter 50 place pi de chaque dilution au centre des assiettes individuelles. Ceux-ci devraient donner des plaques à l'optimum calculé de 25 000 colonies par plaque, ainsi que 2 - et 5 - fois plus et moins, ce qui permet la meilleure dilution réelle à déterminer.
- Ajouter environ 20 verre stérile (3 mm) perles deles tapotant doucement sur la plaque. Perles offrent une répartition plus égale des colonies sur la plaque d'un épandeur de verre courbé fait.
- Étaler cellules en gardant plaques sur la paillasse et les secouer d'avant en arrière, en les faisant tourner pendant que vous travaillez, jusqu'à ce que le liquide soit absorbé. Ne pas continuer secouer les perles une fois que la plaque est sec, sinon il va commencer à tuer les bactéries.
- Retournez sur plaques et jeter des perles dans le bécher de déchets contenant de l'éthanol.
- Laissez les plaques se pour le moment de votre test (par exemple, 24 heures) à la température correcte pour votre test (par exemple 24 ° C / RT).
- L'utilisation d'un stéréoscope dissection, compter le nombre de colonies dans deux ou plusieurs champs de vision.
- Calculer le nombre de colonies par région, et déterminer le nombre de colonies se trouvent sur chaque plaque.
- Réglez dilutions futures si nécessaire. Le nombre réel de colonies n'est pas aussi important que d'avoir le même nombre sur chaque plaque d'écran comparable.
7. Préparer coculture plaques
- Aliquote dégel de journaliste (et aliquote de sol si gelé) comme dans l'étape 5.1.
- Diluer journaliste (à des concentrations optimisées en section 6) en 0,1 x LB ou autre peu de nutriments, solution isotonique.
- Pour un sol gelé: Diluer à concentration optimisée à l'article 6 de 0,1 x LB ou autre peu de nutriments, solution isotonique.
- Pour les sols frais: Faire des dilutions de suspension de sol frais, s'appuyant sur la prédiction que les ufc / ml de la suspension de sol peuvent varier de 10 -4 à 10 -9 ufc / ml.
- Place 50 pi de sol et journaliste dilutions donnant sur le centre de la plaque de l'écran de co-culture. En outre, le sol de la plaque seule et journaliste seul comme témoins.
- Étaler à l'aide de billes de verre comme décrit dans l'étape 6.4 à 6.6.
- Si il ya des conditions de croissance connus qui activent votre rapporteur fluorescent, série ou la plaque de votre journaliste dans ces conditions et poussent à utiliser comme un contrôle positif lors du dépistage.
- Incuber à 24 ° C pendant 24 à 28 h (ou en fonction de votre journaliste / test)
8. co-culture de l'écran Plaques pour fluorescence
- En utilisant un éclairage en fond clair, concentrez votre stéréoscope de sorte que les colonies sont tranchantes.
- Regardez les plaques de sol que pour déterminer si votre échantillon de sol a autofluorescentes colonies. Si oui, ce sol peut entraîner un taux élevé de faux positifs (avec une augmentation concomitante de dépistage secondaire).
- Utiliser un ratio élevé de journaliste: sol dans vos assiettes de co-culture pour augmenter les chances que l'inducteur sera entouré de plusieurs colonies de journaliste, réduisant ainsi le risque qu'ils seront détectés comme faux positifs (figure 2).
- Utilisation d'une protéine fluorescente différente (une qui émet dans un canal différent).
- Déterminer le calendrier de dosage. Pour la plupart des nouveaux journalistes, le moment de l'induction potentielle est inconnue, et doit donc être déterminé de manière empirique.
- Commencer le dépistage de la fluorescence dès que les colonies deviennent visibles avec le stéréoscope de dissection (grossissement 30X ~) et continuent d'examiner les plaques périodiquement jusqu'à ce que la croissance a cessé et / ou la fluorescence de fond devient trop élevé.
- Une fois la fenêtre de temps d'induction potentiel est déterminé pour un journaliste en particulier, il devrait être similaire pour toutes les plaques de coculture contenant ce journaliste, ce qui simplifie la surveillance des plaques d'écran. Pour l'B. journaliste subtilis P Tapa-YFP, le moment approprié pour examiner les plaques est entre 24-28 heures après l'étalement des cellules, car la B. subtilis P SSPB-YFP journaliste, il est entre 26-32 heures de croissance.
- Maximisez votre sensibilité de fluorescence:
- Assurez-vous que la fluorescence de dissection étendue est dans une pièce sombre ou entouré de rideaux occultants. Induction sera probablement moins intense qu'un FP produite constitutivement et exige une plus grande sensibilitétivité à détecter.
- Prévoyez du temps pour la lampe à fluorescence pour stabiliser et vos yeux de s'adapter à l'obscurité avant de tenter de détecter la fluorescence de vos plaques de co-culture (au moins 1-2 min).
- L'utilisation d'un contrôle positif (si vous en avez un), veiller à ce que le grossissement que vous utilisez vous permet de détecter la fluorescence. Le grossissement est généralement préférable si votre champ de vision est d'environ 30-50x le diamètre de votre colonie typique (200-400X).
- Éteignez la lumière brillante et ouvrir l'obturateur pour votre fluorescence.
- Après vos yeux se sont adaptés à l'obscurité, se déplacer lentement la plaque d'avant en arrière au sein de votre champ de vision, à la recherche de points lumineux.
- Commencez par le haut de la plaque et d'utiliser un motif en zigzag pour déplacer le côté à l'autre plaque que vous vous déplacez vers le bas de la plaque.
- Pratique à déplacer la plaque en fond clair pour avoir une idée de la lenteur à se déplacer à la plaque, et pour être sûr que vous couvrez l'ensemble surzone face.
- Déplacer la plaque assez lentement que les colonies ne deviennent pas floue. Il est préférable de sur-échantillonner la surface plutôt que dans les zones de justesse.
- Après un balayage complet, tourner la plaque à 90 ° et la répétition. Les yeux humains sont remarquablement bien à détecter, même faible fluorescence grâce à cette méthode.
- Si vous détectez fluorescence, arrêter de bouger la plaque et revenir en arrière et trouver la zone fluorescente.
- Tourner le fond clair lentement, de déterminer si la fluorescence est associée à une colonie bactérienne (et non détritus du sol autofluorescente ou composants de médias). Si c'est le cas, les colonies non fluorescentes proximales à la colonie fluorescent sont des organismes induisant putatifs.
9. Isoler putatifs induisant organismes
- Une fois induites colonies (fluorescentes) ont été identifiés, isoler les colonies sécrétant les composés inducteurs.
- Si une concentration suffisamment élevée de colonies rapporteurs sont de plus en plus sur la plaque(> 0,5:1 journaliste: ufc de sol), les colonies inducteurs putatifs seront entourés par de multiples colonies fluorescentes (à nouveau, voir la figure 2).
- Dans les cas où la complexité de la croissance de la co-culture, il est ambigu colonie qui est l'inducteur, d'isoler de multiples colonies potentielles inducteurs pour le test ultérieur dans l'écran secondaire.
- Localiser les colonies que vous voulez prendre dans le centre du champ de vue.
- Si elles sont à proximité du bord de la plaque, tournez la plaque de sorte que la lèvre de la plaque est loin de votre main dominante (si vous êtes droitier, placez la lèvre de la plaque sur la gauche). Cela vous permet d'approcher les colonies à un angle faible, ce qui améliore la précision de votre cueillette.
- Placez des plaques fraîches de série les organismes des induisant putatifs à proximité, ainsi que d'un récipient de déchets à jeter les embouts utilisés dans.
- En sortant de la lampe à fluorescence, tourner la lumière lentementsur, de sorte que vous serez en mesure d'identifier (par la forme et la position), la colonie fluorescent et les organismes induisant putatifs environnantes. Vous devrez peut-être revenir en arrière avec la lumière à quelques reprises pour être en mesure d'identifier les colonies Vous voulez choisir quand il n'ya pas de fluorescence et seule lueur d'espoir.
- Utilisez une tige de verre (200 mm de long de diamètre x 5 mm) pour ramasser un, rond 200 pi pointe gel chargement stérile et maintenez-le comme un crayon. C'est l'outil de cueillette que vous allez utiliser pour isoler des colonies individuelles.
- Paume de votre main extérieure contre la scène pour le stabiliser sur la surface de travail. Placez l'autre main sur le côté intérieur, le pouce de votre main pour stabiliser votre outil de cueillette.
- Si nécessaire, retournez à nouveau entre fluorescence et en fond clair vue d'identifier les colonies que vous souhaitez prendre.
- Garder la pointe de la pipette au-dessus de la surface de la plaque, le déplacer dans votre champ de vision et centrez-dessus de la colonie que vous souhaitez prendre. La pointe sera de mise au point.
- Utilisation du bord externe de la main (ce qui est en appui sur la platine du microscope ou de la surface de travail), faire pivoter la pointe de la pipette vers le bas lentement à la colonie pour être ramassé. Touchez-le très légèrement, en essayant de minimiser le nombre de colonies d'autres les contacts de pointe.
- Sans tourner l'outil de cueillette, série sur une section d'une nouvelle plaque. Étendre avec un toucher doux pour éviter rayer la gélose. Parce que le nombre de cellules transférées par ce procédé est assez faible (les colonies sont typiquement beaucoup plus petite que manipulée), une strie unique en continu donnera lieu à des colonies isolées.
- Répétez ce processus avec d'autres organismes inducteurs putatifs.
- Incuber les plaques à 24 ° C (ou la température de votre test).
10. Streak putatifs induisant organismes pour obtenir des colonies isolées simples
- L'utilisation d'un stéréoscope, déterminer - en fonction de la structure de la colonie ou la morphologie - s'il existe des types de colonies différentes contenues dans chacun de vos ind ramassé putatiforganismes ucing.
- Consulter les colonies en utilisant un stade où vous pouvez éclairer les colonies de fois au-dessus ainsi que d'en bas pour détecter des différences de colonies potentielles.
- Restreak chaque morphotype différent sur une nouvelle plaque et incuber jusqu'à grandi.
- Restreak une fois de plus et laisser croître. Si différents morphotypes persistent, continuer à restreak à la pureté.
11. Retester putatifs induisant organismes dans l'écran secondaire
- Retester tous les organismes induisant putatifs dans un écran secondaire pour déterminer qui activent la transcription rapporteur fluorescent. L'écran secondaire est constitué d'une pelouse de microcolonies de la souche de la transcription rapporteur fluorescent, avec des plaques de contrôle, sur laquelle les organismes induisant putatifs sont patchés ou repérés.
- Mettre en place trois plaques identiques: l'un contenant une pelouse microcolonie de la souche de la transcription rapporteur fluorescent (à la même concentration d'uns a été utilisé lors du dépistage de co-culture), l'un contenant une pelouse microcolonie de la souche mère de type sauvage sans un journaliste de fluorescence et un contenant pas de pelouse.
- Marquer la partie supérieure de l'arrière de chaque plaque afin de déterminer l'orientation de la plaque.
- Ajouter des marqueurs de position pour les patches / taches; jusqu'à dix organismes induisant putatifs peuvent être testés sur chaque jeu de plaques (figure 3).
- Aliquotes de pelouse dégel (rapporteurs et de déformation de parent non fluorescent) comme dans l'étape 5.1.
- Étaler 50 pi de 5 x 10 5 UFC / ml dilution sur des plaques de gazon (ou d'autres dilutions optimisés) en utilisant des billes stériles comme dans l'étape 6.4.
- Laissez plaques sec.
- Patch ou repérer putatif organismes induisant:
- Sélectionnez patch si une approche plus facile et plus rapide est souhaitée, et il est acceptable d'avoir un nombre moins précis de cellules déposées. Pour patcher:
- Touchez un cure-dent stérile de la colonie à l'épreuve - ne pas ramasser toutes les cellules.
- Patch (faire une petite série) sur la plaque vierge.
- Répétez patch avec un cure-dent frais sur la plaque de journaliste.
- Répétez patch avec un cure-dent frais sur la plaque de contrôle.
- Sélectionnez taches si une approche quantitative et reproductible est souhaitée. Longues permet le nombre de cellules déposées à être normalisé (voir référence 5 pour plus de détails), et permet la puissance relative des différents organismes inducteurs à comparer. Pour repérer:
- Remettre en suspension les organismes induisant putatif dans 1 ml de milieu liquide dans une cuvette en plastique stérile.
- Prenez la DO 600 des remises en suspension.
- En utilisant la formule X = 250 ÷ (OD 600 à 0,5), ajouter le volume X à chaque remise en suspension de 500 pi de milieu liquide pour obtenir une solution avec une DO600 de 0,5. Cette méthode simplifie les étapes de pipetage nécessaires lorsque vous effectuez plusieurs dilutions de normaliser OD c'est parce que vous pouvez utiliser le même volume (500 pi) pour l'ensemble de vos diluants.
- Spot 1 pi de chaque remise en suspension de DO normalisée de chacune des trois plaques.
- Sélectionnez patch si une approche plus facile et plus rapide est souhaitée, et il est acceptable d'avoir un nombre moins précis de cellules déposées. Pour patcher:
- Laissez croître à 24 ° C pendant 24-28 heures (ou selon votre rapporteur / test).
- Utilisez la dissection microscope à fluorescence pour identifier les organismes induisant putatifs qui activent votre souche fluorescente rapporteur mais pas votre souche de contrôle parental. Ces isolats sont vos résultats positifs - les microbes de l'environnement qui sécrètent des composés qui induisent le phénotype d'intérêt.
Résultats
Cet écran est utilisé pour identifier les organismes du sol sécrétant des composés qui modifient la physiologie de B. subtilis. Les résultats décrits ici sont axées sur le type de cellule-matrice produisant de B. subtilis, qui produit la protéine et des exopolysaccharides qui sont nécessaires pour la formation de biofilm robuste dans cette bactérie. Nous avons choisi le promoteur de l'opéron Tapa-sipW-tasa pour notre construction rapporteur fluorescent (P Tapa-YFP). Cet opéron code pour le composant de structure de la protéine de la matrice et est régulée à la hausse au cours de la production de matrice du biofilm 23. Notre reporter matrice (figure 1) a été construit comme décrit précédemment 19.
Des travaux antérieurs ont montré que B. subtilis matrice produit, en réponse à la quorum-sensing-like molécule surfactine auto-produite, ainsi que les métabolites purifiées produites par d'autres bactéries du sol 20. Nous nous sommes intéressés dans l'expansion de laétudes soi pour enquêter plus largement les microbes du sol qui font métabolites capables d'induire la production de matrice dans B. subtilis. Nous avons choisi d'utiliser diluée LB pour la croissance, car ce milieu était déjà connu pour conduire à une mauvaise production de la matrice 20, nous fournissant une condition de la croissance où notre souche rapporteur était non fluorescent. Nous avons ensuite optimisé le nombre de colonies appropriées pour le dépistage dans ces conditions de croissance. Afin d'optimiser chaque plaque de l'écran, il est nécessaire de déterminer le nombre de colonies se développer à partir des sols et des rapporteurs aliquotes congelées et ce que la concentration appropriée des colonies et des conditions nutritives sont. Idéalement, nous voulons que chaque plaque de co-culture pour contenir un nombre équivalent de sol et journaliste colonies (soit un ratio de 1:1 de journaliste: sol) et d'être étroitement espacés, des colonies individuelles. Ce ratio élevé de colonies rapporteurs augmente la probabilité qu'un évacuateur activer plusieurs colonies rapporteurs environnantes. Avoir ac multiplecolonies inducteurs tivé entourant un présumé confiance inducteur colonie augmente dans l'identification de l'organisme induisant réelle (figure 2). La teneur en éléments nutritifs contrôle le degré de formation de croissance / de colonies alors que la dilution de l'inoculum détermine si les colonies résultantes sont dispersées de manière appropriée. Sur un 10 cm de diamètre boîte de Petri standard avec support faible teneur en nutriments, nous avons constaté que près de 25 000 colonies total par plaque (50 pi de 5 x 10 5 UFC / ml dilution) à condition que le meilleur séparation de B. subtilis colonies sur milieu LB 0,1 x MOPS (figure 4).
Bien que le cfu / ml calculée à partir des dilutions en série fournit une concentration approximative de bactéries dans les aliquotes, il est nécessaire de veiller à ce que la concentration des colonies obtenues est appropriée quand une plaque entière est étalé avec des cellules. L'ufc / plaque calculée et réelle ufc / plaque ne sont pas toujours identiques (Figure 4 ). Placage pelouses de la colonie de concentrations équivalentes est important de permettre aux différentes souches de reporter à comparer (sinon, les différences dans la disponibilité des nutriments peuvent modifier leur état physiologique et interférer avec les résultats).
Après la préparation des aliquotes de la journaliste et le sol, nous les mixé sur des plaques d'écran de co-culture et les avons examinés pour fluorescence à l'aide d'un stéréoscope (figure 5). Nous avons également plaqué contrôles qui ont été inoculées avec seulement sol ou de la B. subtilis souche P rapporteur Tapa-YFP. B. subtilis produit matrice du biofilm (fluorescence) en réponse à de nombreux microbes du sol comme on le voit par les colonies fluorescentes dans l'image de la co-culture sur la figure 5. Pour les sols que nous avons examinés, nous avons eu un taux de succès élevé pour le journaliste P Tapa-YFP. Comme décrit dans la référence 5, entre 12-67% des isolats (à partir de six échantillons de sol différents) ont la capacité de induCE fluorescence dans la souche rapporteur P Tapa-YFP. Ceci est en contraste à nos résultats non publiés de écrans analogues utilisant la sporulation (P SSPB-YFP) et compétence (P COMG-YFP) journalistes. Après de longues dépistage (> 200 000 colonies pour chaque journaliste), seuls deux organismes ont été identifiés qui induisent la sporulation, alors qu'aucun ont été identifiés qui induisent compétence. Ainsi, les taux de réussite pour différents types de cellules sont très variables et peuvent être difficiles à prévoir à l'avance.
Nous avons ensuite choisi colonies inducteurs putatifs individuels. Les colonies sur les plaques d'écran de co-culture sont assez petites sur le support pauvres en nutriments, nous recommandons (diamètre submillimétrique). Néanmoins, il est possible de choisir avec précision et de très petites colonies isoler la main (figure 6) à partir d'une plaque d'écran de coculture compliqué. La méthode manuelle que nous utilisons est simple et ne nécessite ni outils spécialisés, ni flamme stérilisation. Ces colonies inducteurs putatifs sont ensuite réamorcée à l'isolement. Parce que les plaques de coculture sont entassés avec des colonies, il n'est pas rare - même avec très prudent technique de prélèvement - d'avoir plus d'un organisme en croissance de chaque échantillon inducteur putatif. Un examen attentif doit permettre l'isolement des colonies morphologiquement distinctes. Tous les organismes induisant putatifs sont ensuite testés dans un écran secondaire. Résultats positifs et négatifs à la fois le patch et la méthode de point sont présentés dans la figure 3. Compte tenu de leur croissance dense, notre capacité à collecter physiquement induire colonies des plaques de coculture a été assez bonne, avec environ 50% des colonies examinées dans l'écran secondaire étant vrais positifs. Autres résultats de cet écran ainsi que le travail de suivi qui s'en dégagent ont été décrits précédemment 5.
/ Ftp_upload/50863/50863fig1.jpg "/>
Figure 1. Fluorescent transcription construction de rapporteur. L'ovale bleu représente une cellule bactérienne et la ligne pointillée représente son chromosome. Cet exemple montre un reporter fluorescent de la transcription pour la production de matrice. Le locus natif reste intact (P matrice-matrice, où "P" et la flèche indique la région promotrice), tandis que la construction de rapporteur (P matrice-YFP) est inséré dans le reste du chromosome dans un locus neutre.
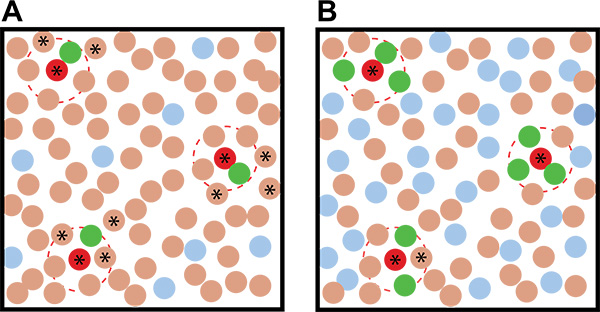
Figure 2. Exemples idéalisés des résultats de dépistage de co-culture avec différents rapports de la journaliste: les microbes de l'environnement. A) L'utilisation d'un faible journaliste: rapport de microbe de l'environnement conduit à plus d'ambiguïté dans l'identification des organismes induisant putatifs que lorsque B) Une grande journaliste: rapport de microbe de l'environnement est utilisé. Les cercles bruns représentent les organismes du sol, les cercles rouges représentent induisant les organismes du sol, les cercles bleus représentent les colonies de rapporteurs non induites, et les colonies vertes représentent colonies rapporteurs induits. Les lignes rouges en pointillés indiquent le rayon du métabolite d'induction de l'action. Les étoiles indiquent les colonies non fluorescentes qui - en fonction de leur proximité avec les colonies fluorescentes - sont des organismes induisant putatifs et devraient être ramassés et testés à nouveau dans l'écran secondaire.
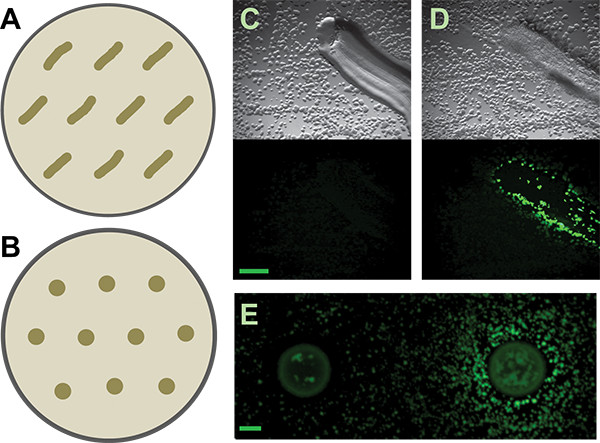
Figure 3. Écran secondaire. A et B) Schéma de la façon de distribuer patché ou repéré isolats sur des plaques d'écran secondaires, respectivement, pour la B. subtilis matrice journaliste. Espacement plus généreux peut être nécessaire pour d'autres journalistes ou isolats induisant, depse terminant à la capacité de diffusion de leurs métabolites actifs. C et D) Les résultats représentatifs du sol patché isolats qui sont négatifs et positifs, respectivement, pour induire la B. subtilis P Tapa-YFP-reporter. Les panneaux supérieurs sont les images en fond clair; panneaux inférieurs sont les images de fluorescence. La barre d'échelle est de 1 mm. E) négatif et les résultats positifs de sol tacheté isolats de la même journaliste. La barre d'échelle est de 2 mm.

Figure 4. Détermination de la concentration de microcolonie. La distribution et la taille des colonies dépendront à la fois sur les concentrations de nutriments et de cellules. A) Les différences dans la croissance de B. subtilis sur 0,01 x LB (rangée supérieure) contre 0.08x LB (rangée du bas). Cellules de 0,01 x LB ne font pas dans microcolonies, tandis que ttuyau sur 0.08x LB faire. (Notez que pour nos écrans, nous avons augmenté les niveaux de nutriments légèrement de ceux indiqué ici:. Du 0.08x LB à 0,1 x LB) Ces images sont de 1 points de pi de séquentielles 1:05 dilutions à connu ufc / ml. En extrapolant à partir de ces concentrations, pour obtenir des distributions similaires de colonies à travers une plaque de 10 cm Petri exigerait placage (de gauche à droite): 3.200.000; 640 000 et 128 000 au total ufc par boîte. Cependant, les résultats de repérage dans la distribution inégale des cellules (elles sont concentrées au niveau des bords de tache) par rapport à la propagation des cellules sur toute la plaque. Ainsi, une fois la concentration en éléments nutritifs est choisi, il est important d'examiner les plaques réparties avec une variété de concentrations. La barre d'échelle est de 0,1 mm B) Ces panneaux présentent les résultats de la propagation (de gauche à droite) 50000;. 25000 et 5000 ufc total par plaque sur des plaques LB 0.08x. A partir de ces images, nous avons sélectionné 25 000 comme notre numéro de cible de ufc / plaque. La barre d'échelle est de 0,1 mm.

Figure 5. Co-culture de B. subtilis P Tapa-YFP en mélange avec les organismes du sol. Superposition de fond clair et l'image de la fluorescence à partir d'une plaque d'écran de co-culture contenant le B. subtilis P Tapa-YFP matrice reporter mélangé avec les organismes du sol. Arrowhead indique inducteur putatif entouré de microcolonies fluorescence rapporteurs. La barre d'échelle est de 1 mm.
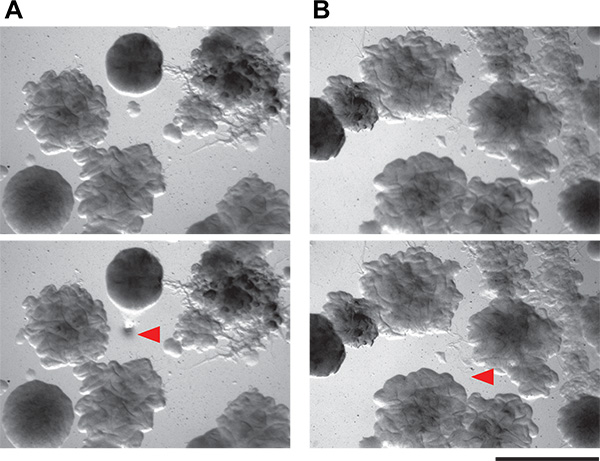
Figure 6. Démonstration de la faisabilité d'isoler minuscules colonies bactériennes à partir de plaques de coculture. A et B) Ces panneaux présentent deux champs de vision des plaques de gélose contenant les communautés microbiennes complexes à partir du sol. Colonies aussi petites que 0,1 mm peuvent être isolés en utilisant la technique de prélèvement décriteici. Top panneaux sont le champ de vision avant la cueillette de la colonie, et le panneau inférieur sont les mêmes champs de vision après la cueillette colonie. Flèches rouges indiquent où les cellules ont été enlevés.
Discussion
Une des limites inhérentes à ce protocole est qu'il repose sur la cultivabilité d'organismes microbiens. Comme il a été bien documenté 24, plus la vie microbienne sur la planète ne peut pas (encore) être cultivé dans les conditions de culture explorées à ce jour. Ainsi, un grand nombre d'interactions entre les espèces microbiennes qui se produisent dans les milieux naturels ne soit pas détecté en utilisant cette approche. Cependant, depuis notre désir est d'identifier non seulement l'existence de ces interactions, mais aussi étudier les mécanismes et les molécules impliquées dans les médiateurs, la capacité de cultiver ces microbes est une nécessité. Même au sein des espèces cultivables, ce domaine a été mal explorée, ce qui l'approche décrite ici une précieuse contribution en tant que méthode pour identifier les interactions médiatisées chimiquement entre les microbes. En outre, bien que ce protocole a été optimisé pour le dépistage de la matrice-induction de Bacillus subtilis, on peut théoriquement être appliqué à uny reporter fluorescent transcription chez les autres espèces bactériennes.
Une autre limitation liée de cette approche est que cet écran (par définition) nécessite la co-culture. Dans les milieux naturels, les microbes avec des taux de croissance peuvent encore coexister dans la proximité spatiale tout en exploitant des niches écologiques. Ces interactions microbiennes passent inaperçus par notre écran de co-culture, cependant, qui ne fera que permettre la croissance des microbes de l'environnement avec les besoins en nutriments et des taux similaires à ceux de l'espèce rapporteurs de croissance. Les modifications qui seraient séparer la croissance des organismes de potentiel d'induction de la croissance de la souche rapporteuse sont certainement possibles. Nous avons aussi prévu que la croissance des hyphes de champignons - communs dans le sol - pourrait entraîner des difficultés dans l'écran de co-culture. Bien que le délai très court de notre écran avec B. subtilis signifiait que quelques champignons ont été détectés, l'addition de composés antifongiques dans le milieu de croissance pourrait minimize cette préoccupation.
La possibilité de sélectionner un phénotype et le gène approprié pour la construction de rapporteur fluorescent devrait pas être difficile, compte tenu de la richesse du séquençage et de la transcription des données déjà disponibles ou faciles à obtenir pour de nombreuses espèces bactériennes. Cependant, une difficulté avec l'approche décrite ici est la nécessité d'identifier les conditions de croissance qui réduisent la fluorescence de votre souche rapporteuse de fond, permettant la détection de la fluorescence induction. L'identification de ces conditions doit souvent être fait de manière empirique, bien que les données de transcription peuvent aider la recherche (par exemple, les données de carrelage de puces à ADN disponibles pour la croissance de B. subtilis permettant l'identification des conditions où les gènes d'intérêt sont mal exprimés 10). Pour certains journalistes cette recherche empirique peut être difficile, en partie parce que l'expression de nombreux phénotypes bactériens est hétérogène. En d'autres termes, il est rare de trouver conditions dans lequel aucune des cellules dans la population expriment Phénotype X. Ainsi, en fonction du nombre de cellules au sein de cette sous-population et la force de l'expression des gènes, il peut être difficile d'identifier les conditions qui fournissent suffisamment faible fluorescence de fond pour permettre à induction à détecter . Une alternative à cette recherche empirique des conditions idéales de dépistage peut être au niveau de la journaliste par mutagenèse dirigée d'expression "tune". En agissant sur la région du promoteur et / ou un site de liaison ribosomique de la construction de rapporteur, le niveau de fluorescence de fond pourrait être diminué. Cela pourrait étendre l'utilité de cet écran en permettant même avec certains gènes activation constitutive à examiner pour l'induction.
Une fois les organismes induisant ont été identifiées et confirmées dans un écran secondaire, ils peuvent être identifiés par séquençage phylogénétiquement leur gène ARNr 16S. Il est également possible de quantifier l'ampleur de la fluorescence utilisant OD place 600 normalisée dans l'écran secondaire 5. Cela peut fournir des informations sur les membres de la communauté produisent des composés qui affectent votre souche de journaliste et dans quelle mesure. Par conséquent, cela peut conduire à des hypothèses sur lesquelles les interactions microbiennes peuvent se produire dans les milieux naturels et la possibilité d'explorer la coévolution potentiel de ces organismes producteurs et répondre. D'autres orientations futures comprennent l'élucidation de la structure de la molécule sécrétée elle-même, la détermination du mécanisme (s) par lequel l'organisme répondant détecte ce composé, et de l'utiliser comme un outil de produit chimique à moduler les phénotypes bactériens.
Même avec les considérations exposées ci-dessus, la méthode décrite ici est une contribution importante. Il évite le travail nécessaire à l'assemblage d'une bibliothèque de microbes de l'environnement, mais permet leur séparation et d'isolement en utilisant des milieux solides. La force de cet écran de co-culture est queelle propose un procédé conceptuellement et techniquement simple pour cribler des milliers d'espèces microbiennes afin d'identifier ceux qui sécrètent des composés bioactifs d'intérêt tout en étant applicable à de nombreuses espèces de bactéries et de phénotypes.
Déclarations de divulgation
Les auteurs déclarent qu'ils n'ont aucun intérêt financier concurrents.
Remerciements
L'auteur remercie Roberto Kolter (Harvard Medical School) pour ses conseils et son aide lors de l'élaboration de cet écran de co-culture inestimable. Elle remercie également Matthew Powers pour lire le manuscrit pour plus de clarté, et Chia-yi Cheng pour l'aide à l'obtention de la figure 6.
matériels
| Name | Company | Catalog Number | Comments |
| Spectrophotometer | Any spectrophotomer capable of measuing OD600 absorbance values. | ||
| Luria broth, Lennox | VWR | 80017-484 | Alternative media sources may be necessary. |
| Glass beads, 3 mm | VWR | 26396-508 | |
| Gel loading tips, round | VWR | 29442-666 | |
| Glass rods | VWR | 59060-069 | |
| Fluorescence dissecting stereoscope | Zeiss | N/A | The author used a Zeiss Stemi SV6 dissection stereoscope with an EXFO X-cite 120 fluorescent light source, a long-pass YFP filter cube, an achromat 0.63X objective, 10X eyepieces, and an Axio HRC HR digital camera. Most screening was done with the focusing mount at 2.0-3.2X. Any dissecting stereoscope with fluorescence capabilities is fine, provided you have the correct filters for the FP you are using. It is best if there is a shutter that allows you to easily switch between brightfield and fluorescense, as well as a stage that allows illumination from above and below. If you want to capture images, an attached camera is also necessary. |
Références
- Berdy, J. Bioactive microbial metabolites. J. Antibiot. 58, 1-26 (2005).
- Lopez, D., Vlamakis, H., Kolter, R. Generation of multiple cell types in Bacillus subtilis. FEMS Microbiol. Rev. 33, 152-163 (2009).
- Vlamakis, H., Aguilar, C., Losick, R., Kolter, R. Control of cell fate by the formation of an architecturally complex bacterial community. Genes Dev. 22, 945-953 (2008).
- Branda, S. S., Gonzalez-Pastor, J. E., Ben-Yehuda, S., Losick, R., Kolter, R. Fruiting body formation by Bacillus subtilis. Proc. Natl. Acad. Sci. U.S.A. 98, 11621-11626 (2001).
- Shank, E. A., et al. Interspecies interactions that result in Bacillus subtilis forming biofilms are mediated mainly by members of its own genus. Proc. Natl. Acad. Sci. U.S.A. 108, 1236-1243 (2011).
- Piston, D. W., Patterson, G. H., Lippincott-Schwartz, J., Claxton, N. S., Davidson, M. W. Introduction to Fluorescent Proteins. , (2013).
- Chudakov, D. M., Matz, M. V., Lukyanov, S., Lukyanov, K. A. Fluorescent proteins and their applications in imaging living cells and tissues. Physiol. Rev. 90, 1103-1163 (2010).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. J. Cell Sci. 120, 4247-4260 (2007).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat. Methods. 2, 905-909 (2005).
- Nicolas, P., et al. Condition-dependent transcriptome reveals high-level regulatory architecture in Bacillus subtilis. Science. 335, 1103-1106 (2012).
- Middleton, R., Hofmeister, A. New shuttle vectors for ectopic insertion of genes into Bacillus subtilis. Plasmid. 51, 238-245 (2004).
- Shimotsu, H., Henner, D. J. Construction of a single-copy integration vector and its use in analysis of regulation of the trp operon of Bacillus subtilis. Gene. 43, 85-94 (1986).
- Guerout-Fleury, A. M., Frandsen, N., Stragier, P. Plasmids for ectopic integration in Bacillus subtilis. Gene. 180, 57-61 (1996).
- Semsey, S., Blaha, B., Koles, K., Orosz, L., Papp, P. P. Site-specific integrative elements of rhizobiophage 16-3 can integrate into proline tRNA (CGG) genes in different bacterial genera. J. Bacteriol. 184, 177-182 (2002).
- Charpentier, E., et al. Novel cassette-based shuttle vector system for gram-positive bacteria. Appl. Environ. Microbiol. 70, 6076-6085 (2004).
- Yang, H. Y., Kim, Y. W., Chang, H. I. Construction of an integration-proficient vector based on the site-specific recombination mechanism of enterococcal temperate phage phiFC1. J. Bacteriol. 184, 1859-1864 (2002).
- Choi, K. H., Schweizer, H. P. mini-Tn7 insertion in bacteria with single attTn7 sites: example Pseudomonas aeruginosa. Nat. Protoc. 1, 153-161 (2006).
- Craig, N. L. Tn7: a target site-specific transposon. Mol. Microbiol. 5, 2569-2573 (1991).
- Garcia-Betancur, J. C., Yepes, A., Schneider, J., Lopez, D. Single-cell analysis of Bacillus subtilis biofilms using fluorescence microscopy and flow cytometry. J. Vis. Exp. (60), e3796(2012).
- Lopez, D., Fischbach, M. A., Chu, F., Losick, R., Kolter, R. Structurally diverse natural products that cause potassium leakage trigger multicellularity in Bacillus subtilis. Proc. Natl. Acad. Sci. U.S.A. 106, 280-285 (2009).
- Vartoukian, S. R., Palmer, R. M., Wade, W. G. Strategies for culture of 'unculturable' bacteria. FEMS Microbiol. Lett. 309, 1-7 (2010).
- Romano, J. D., Kolter, R. Pseudomonas-Saccharomyces interactions: influence of fungal metabolism on bacterial physiology and survival. J. Bacteriol. 187, 940-948 (2005).
- Branda, S. S., Chu, F., Kearns, D. B., Losick, R., Kolter, R. A major protein component of the Bacillus subtilis biofilm matrix. Mol. Microbiol. 59, 1229-1238 (2006).
- Zengler, K., et al. Cultivating the uncultured. Proc. Natl. Acad. Sci. U.S.A. 99, 15681-15686 (2002).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon