Method Article
Usando Coculture detectar químicamente mediadas por interacciones interespecíficas
En este artículo
Resumen
Las bacterias producen compuestos secretados que tienen el potencial de afectar a la fisiología de sus vecinos microbianos. Aquí se describe una pantalla de co-cultivo que permite la detección de este tipo de interespecies mediadas químicamente interacciones mediante la mezcla de los microbios del suelo con cepas reportero transcripcional fluorescentes de Bacillus subtilis en medios sólidos.
Resumen
En la naturaleza, rara vez existen bacterias en forma aislada, sino que están en vez rodeados por una gran variedad de otros microorganismos que alteran el medio ambiente local mediante la secreción de metabolitos. Estos metabolitos tienen el potencial de modular la fisiología y la diferenciación de sus vecinos microbianos y son factores importantes que puedan en el establecimiento y mantenimiento de las comunidades microbianas complejas. Hemos desarrollado una pantalla de cocultivo basado en la fluorescencia para identificar tales interacciones microbianas mediadas químicamente. La pantalla se combina una transcripción cepa fluorescente reportero con microbios ambientales en medios sólidos y permitiendo que las colonias crezcan en cocultivo. El reportero transcripcional fluorescente está diseñado de manera que la cepa bacteriana elegida emite fluorescencia cuando se está expresando un fenotipo particular de interés (es decir, la formación de biopelículas, la esporulación, la producción de factor de virulencia, etc.) El cribado se realiza en condiciones de crecimiento where este fenotipo no se expresa (y por lo tanto la cepa reportero es típicamente no fluorescente). Cuando un microbio ambiental segrega un metabolito que activa este fenotipo, que se difunde a través del agar y activa el constructo indicador fluorescente. Esto permite que el productor de metabolitos de inducción de microbio a detectar: son las colonias no fluorescentes más proximales a las colonias fluorescentes. Por lo tanto, esta pantalla permite la identificación de los microbios ambientales que producen metabolitos difusibles que activan una respuesta fisiológica particular, en una cepa reportero. Esta publicación explica cómo: a) seleccionar las condiciones de selección adecuadas co-cultivo, b) preparará el reportero y microbios ambientales para la detección, c) realizar la pantalla de co-cultivo, d) aislar organismos putativo inducir, y e) confirmar su actividad en una pantalla secundaria. Hemos desarrollado este método para la detección de organismos del suelo que activan biopelícula matriz de producción en Bacillus subtilis
Introducción
Estamos interesados en la comprensión de cómo los metabolitos que las bacterias secretan afectan la fisiología y el desarrollo de los microbios vecinos. Muchos metabolitos se han caracterizado por sus efectos bioactivos sobre otros microbios. Dos ejemplos bien descritos incluyen antibióticos, que inhiben el crecimiento de otros microbios, y las moléculas de detección de quórum, que alteran la expresión génica global de otros microbios. Sin embargo, las bacterias producen muchos otros pequeños productos naturales de molécula que no tienen bioactividades conocidos 1. Nuestra hipótesis es que las bacterias han evolucionado y conservado la capacidad de producir algunos de estos metabolitos, ya que les permite modular la fisiología celular de sus vecinos microbianas en las complejas comunidades microbianas dentro de la cual existen la mayoría de las bacterias.
Tipos de células de Bacillus subtilis
Nos hemos centrado nuestros estudios sobre las interacciones microbianas mediadas químicamente que involucran Bacilsubtilis lus. Esto no es sólo debido a su estatus como modelo la bacteria Gram-positivas y las herramientas genéticas resultantes disponibles para su manipulación, pero también por su capacidad de diferenciarse en tipos de células caracterizadas. Algunos ejemplos son las células que son: natación, que producen la matriz extracelular que se requiere para la formación de biopelículas robusta; competentes para tomar ADN del medio ambiente, y en esporulación, entre otros 2. Cada uno de estos tipos de células expresa un regulón transcripcional característica que los hace fisiológicamente y / o físicamente distinto de sus hermanos genéticamente idénticos. Bajo muchas condiciones de crecimiento, múltiples tipos de células coexisten como diversas subpoblaciones dentro de una sola colonia de B. subtilis células 3. Aunque muchas especies de bacterias pueden exhibir análoga tipo celular heterogeneidad, este fenómeno ha sido particularmente bien estudiado en B. subtilis.
En particular, los genes que son UPRegulated dentro de cada uno de ellos específico B. Se han identificado los tipos de células subtilis. La identificación de estos genes regulados positivamente es esencial para el trabajo que se describe aquí, porque muchos de estos fenotipos microbianas de interés son difíciles o imposibles de observar directamente. Por ejemplo, no se puede detectar visualmente un rasgo como la natación (1.5%) placas de agar sólido, a pesar de que una subpoblación de B. subtilis células producen flagelos en esas condiciones 3. Otro ejemplo es la matriz-la producción de biopelículas. Producción de la matriz puede ser visualizado por morfología de las colonias (tal como resulta en colonias macroscópicamente arrugadas), pero sólo en cierta medio de crecimiento, y sólo después de varios días de crecimiento 4. Sin embargo, sabiendo que son upregulated genes durante la diferenciación, podemos construir reporteros transcripcionales que actúan como marcadores para la diferenciación celular en estos tipos de células.
Reportero construcciones
Estos fluorescente treporteros ranscriptional consisten de los promotores de genes específicos de tipo celular de conducir la producción de un gen reportero, por ejemplo una proteína fluorescente. Los ejemplos incluyen P HAG-YFP (para células natación), P tapa-YFP (para células de la matriz de producción de biofilm), y P SSPB-YFP (por esporulación de las células), donde P x indica la región del promotor para el gen x. Estas construcciones indicadoras se integran en un locus neutro en el cromosoma (Figura 1 y ver más abajo) de manera que la regulación natural de la fenotipo se deja intacta. Sin embargo, ahora, cuando una célula expresa este fenotipo, sino que también expresa una proteína fluorescente. Esto proporciona una lectura de la activación de comportamiento fenotípico particular, puede visualizar fácilmente, lo que nos permite para detectar microbios que activan la respuesta fisiológica. Aunque tales reporteros son comúnmente utilizados en microbiología, que no se han aplicado ampliamente en pantallas a identifinteracciones metabólicas Y entre los microbios antes de este método fue descrito 5.
Hay una serie de consideraciones importantes en el diseño y construcción de reportero cepas celulares específicos del tipo. Hemos utilizado los reporteros fluorescentes exclusivamente de la transcripción, aunque otros tipos de construcciones son ciertamente posibles. Desaconsejamos el uso de fusiones de traducción como marcadores para el tipo de célula diferenciación en nuestra pantalla, sin embargo, por dos razones: 1) el deseo de abandonar el tipo de células específicas proteína nativa no perturbada, y 2) el reconocimiento de que un difuso, de células- amplia de fluorescencia será más fácil de detectar que puntos lagrimales localizado dentro de las células (común con fusiones de traducción).
La selección de genes reportero
Después de tomar la decisión de usar la transcripción como un dispositivo de lectura, el gen reportero debe ser seleccionado (por ejemplo, LacZ, fluorescencia, o luciferasa). LacZ tiene la ventaja de necesitar menos especialzado equipos para detectar, pero hay una probabilidad mucho mayor de falsos positivos entre los microbios ambientales. En nuestras manos, el nivel de fondo de LAC + organismos entre los microbios del suelo era prohibitivo (>> 10% de los microbios del suelo eran azules (Lac +) en placas X-gal; datos no presentados). Es posible que mediante la valoración de la concentración de X-gal en el medio, esto puede ser optimizado para permitir el uso de un reportero de X-gal, aunque no se intentó esto. Luciferasa ofrece una alta sensibilidad de detección y es el reportero más ortogonal: no hay casi ninguna posibilidad de microbios ambientales ser inherentemente luminiscente. Sin embargo, encontramos que hace difícil identificar la instrumentación en nuestra institución que permitió la detección de luminiscencia a través de la totalidad de las placas de Petri, ya que la mayoría fueron diseñados para explorar regiones localizadas sólo en placas de múltiples pocillos. También puede haber complicaciones en la visualización de colonias luminiscentes de una manera que también permitió la física simultánea esolation de organismos que inducen. Durante el uso de los fiduciarios puede haber hecho esto posible, en lugar elegido para utilizar marcadores transcripcionales fluorescentes, que se ha comprobado que funcionan en B. subtilis, a condición de sensibilidad adecuada de detección y bajas tasas de falsos positivos entre los organismos del suelo, y les permite usar de la instrumentación fácilmente disponible tanto para la visualización y procedimientos de aislamiento.
Selección Fluoróforo
El fluoróforo específico seleccionado dependerá de sus especies bacterianas, el medio de cultivo de agar que está usando, y el filtro de fluorescencia especial establece que tiene disponible. Con nuestro instrumentación, se encontró que tanto la B. subtilis colonias ellos mismos y el agar que se cultivaron en exhibieron menos fluorescencia de fondo cuando se utilizaron YFP (proteína fluorescente de color amarillo) filtros, por lo que el reportero superior a GFP (proteína fluorescente verde) en nuestras manos. El uso de codones de proteínas fluorescentes estánfrecuencia optimizada para eucariotas, por lo que es importante seleccionar un fluoróforo o bien conocidos por la literatura para trabajar en sus especies de bacterias, o para probar de forma explícita el uso de un promotor constitutivo. Un gran número de las siempre cambiantes variantes de la proteína fluorescente están actualmente disponibles 6, que se han revisado en varias fuentes 7,8, algunas de las cuales prevé explícitamente orientación sobre la elección de una proteína fluorescente apropiado para su experimento 9.
Selección del promotor
La selección de un promotor dependerá en gran medida del tipo de célula o fenotipo de interés. Para los organismos tales como B. subtilis, algunos genes indicadores específicos de tipo celular se han establecido en la literatura. Para otras cepas bacterianas, el examen de microarrays o datos de la transcripción será necesario para proporcionar información acerca de qué genes están muy upregulated bajo las condiciones en las que el tipo de célula de interés is manifestada. Un estudio reciente catalogado la transcripción de B. subtilis 104 bajo condiciones de crecimiento diferentes con suelo de baldosas microarrays 10. Este documento proporciona información completa acerca de qué genes están altamente regulados por incremento en diferentes condiciones, que tiene un valor incalculable para los fenotipos menos bien caracterizados.
En lugar de la cartografía de las regiones precisas de promotor para cada gen de interés, por lo general simplemente usamos la secuencia de 200-500 pb aguas arriba del gen como el promotor. La duración exacta depende de la secuencia genómica contexto: las regiones más cortas se utilizan cuando es necesario para evitar la inclusión de las regiones de codificación de aguas arriba de la vecina marcos de lectura abierta.
Loci y la integración Neutro
¿Cómo mantener el reportero construir en su cepa bacteriana se convierte en la última pregunta en el diseño de una cepa reportero transcripcional fluorescente. En las bacterias, los genes de interés se mantienen con frecuenciaen plásmidos utilizando la selección de antibióticos. Sin embargo, puede que no sea posible utilizar antibióticos durante cocultivo sin matar los microbios ambientales. Si plásmidos se mantienen establemente en sus especies de bacterias, puede ser posible que aumenten sus bacterias que contiene un reportero plásmidos cargo en presencia de antibióticos para preparar su reportero en los controles, y luego eliminar los antibióticos durante la propia co-cultivo con la esperanza de que el plásmido se ser suficientemente mantenido para permitir la fluorescencia. Sin embargo, si los plásmidos se pierden fácilmente en su bacteria, o se pierden en condiciones de estrés, esto no será una opción viable. En muchos casos, la mejor solución será integrar el constructo indicador en el cromosoma bacteriano, que permite el mantenimiento estable del reportero, incluso en ausencia de selección. Para que la integración de no interrumpir la expresión normal o regulación de su gen de interés, se recomienda integrar en un sitio ectópico en el cromosoma que can actuar como un "locus neutro." En B. subtilis estos sitios de integración son los genes que - cuando muta - transmitir un fenotipo en ciertos medios mínimos (que permite a los integrantes pueden identificar sin selección de antibióticos), y sin embargo no alteran el crecimiento o las tasas de esporulación en rich media, e incluyen genes tales como amyE, lacA, thrC, pyrD, gltA, y sacA (transporte de la capacidad de utilizar almidón, β-galactósidos, treonina, uracilo, glutamato, y sacarosa, respectivamente) 11-13.
Mientras que la integración en estos genes se han utilizado de forma fiable durante muchos años en B. subtilis (particularmente en amyE y lacA), un conocimiento similar, puede no estar disponible para los genes en otras especies bacterianas. El uso de los sitios de unión de fagos son grandes alternativas para los sitios de integración cromosómica neutros: muchas especies específicas de 14 a 16, así como los sitios de integración generales, tales como el sitio de unión Tn7 (Tn7 att) tienenhan identificado y utilizado para las inserciones de genes en muchas especies bacterianas 17,18.
Microbios ambientales
Utilizamos el suelo como fuente directa de microorganismos ambientales para nuestra pantalla de co-cultivo. El suelo contiene una gran diversidad de microbios, y muchos de estos organismos son rica fuente de productos naturales. Mediante el uso de suspensiones líquidas de suelos colocados directamente sobre las placas con nuestra cepa fluorescente reportero transcripcional (sin aislamiento previo de las bacterias del suelo), simplificamos en gran medida el enfoque experimental. El suelo o bien se puede utilizar inmediatamente después de la cosecha, o se congeló a -80 ° C para su uso futuro. El uso inmediato tiene la ventaja de que una mayor diversidad de microbios potencialmente se puede cultivar, incluidos los que no va a sobrevivir la congelación también. Tiene la desventaja de que se desconoce la concentración de los organismos del suelo cultivables partir de estas muestras, lo que aumenta el número de placas de pantalla que se debe utilizar. Deluso ayed tiene la ventaja de que las ufc / ml para cada fuente de suelo se pueden determinar de antemano, lo que permite un número optimizado de colonias que se cultiva en cada placa pantalla. Sin embargo, requiere que los organismos del suelo sean capaces de sobrevivir a la congelación.
Tenga en cuenta que la diversificación de la piscina inductor siendo examinada (es decir, las fuentes de suelo) parece ser más eficaz en la identificación de nuevas interacciones entre especies que el cribado en profundidad sobre el mismo suelo: una mayor diversidad filogenética se observó en los accesos identificados en nuestra pantalla de matriz de inducción como fuentes de suelos además se examinaron en lugar de detección de las mismas fuentes de suelos más a fondo (EA Shank y R. Kolter, Escuela de Medicina de Harvard, resultados no publicados).
Visión de conjunto
El enfoque que describimos aquí es sencillo en cuanto a sus requisitos técnicos. Se trata de: 1) la construcción de un reportero transcripcional fluorescente en B. subtilis o unaotras especies bacterianas de interés, 2) la identificación de condiciones bajo las cuales no se activa este reportero, 3) la preparación de partes alícuotas de esta cepa reportero y organismos que se proyectarán (en nuestro caso del suelo, pero otras fuentes podrían ser utilizados en su lugar), 4) la mezcla de estos dos conjuntos de microbios en medios sólidos, 5) identificar y aislar los organismos putativo inducir, y 6) confirmando que estos organismos en efecto, activan este fenotipo en una pantalla secundaria. Una vez identificados, estos organismos y sus metabolitos nos proporcionan herramientas químicas para modular el comportamiento bacteriano, para estudiar la fisiología bacteriana y las interacciones microbianas y actuar potencialmente como nuevos andamios para compuestos terapéuticos futuros.
Protocolo
1. Seleccionar un gen informador y Construir un reportero fluorescente transcripcional
Para B. subtilis:
- Consulte el artículo de Jove en referencia 19 para un protocolo que describe la construcción de marcadores transcripcionales fluorescentes en Bacillus subtilis.
Para otras especies bacterianas:
- Identificar un gen que se regula positivamente durante la respuesta fisiológica de interés. Esto puede basarse en la bibliografía existente o análisis transcripcional del microbio en condiciones particulares.
- Construir un reportero transcripcional fluorescente para este gen para actuar como un proxy para el cambio en el fenotipo. Esta construcción debe incluir el promotor de este gen upregulated conducir la producción de una proteína fluorescente apropiada (véase la Figura 1).
- Integrar este constructo en un lugar neutral en el cromosoma. Esto asegura que reg nativaulación del gen de interés no se interrumpe, y evita la necesidad de mecanismos de selección de plásmido (por ejemplo, antibióticos) que podrían interferir con el crecimiento de microbios ambientales.
2. Determinar cocultivo Condiciones
Para B. subtilis P reportero-Tapa YFP:
- Uso 0.1x LB, Lennox (1 g de triptona, 0,5 g de extracto de levadura, 0,5 g de NaCl por litro) medio de este reportero, ya que B. matriz-producción subtilis es mínimo en caldo Luria 20. Este medio permite B. colonias subtilis crezcan hasta submilimétricas pero el tamaño observable, al tiempo que permite diversos taxones de suelo para crecer 5.
- Incluya tampón MOPS 100 mM para reducir al mínimo los posibles cambios de pH.
Para otras especies bacterianas:
- Utilice los datos publicados de la transcripción o empíricamente probar varias condiciones de cultivo para identificar una donde el microbio crece pero el activation del indicador fluorescente es insignificante (para permitir su activación a detectar cuando el reportero cepa se cultiva en cocultivo con los microbios que inducen.)
- Utilice un medio con bajo contenido de nutrientes (en relación con tradicionalmente ricos medios microbiológicos) cuando el cribado microbios ambientales de ambientes oligotróficos (tales como el suelo), ya que muchas bacterias oligotróficas no crecen cuando se presenta con condiciones de alto de nutrientes 21. Un medio bajo en nutrientes también reduce el tamaño de la colonia, aumentando el rendimiento de la pantalla.
- Seleccione un medio con baja fluorescencia de fondo y una buena claridad óptica.
- Optimizar la temperatura de crecimiento para permitir que tanto la cepa reportero y microbios ambientales que crecen simultáneamente.
- Considere la posibilidad de la adición de un agente tampón. El uso de tampón en las placas reducirá la posibilidad de detectar cambios de pH mediada en la fisiología 22, a menos que tales interacciones son de interés.
3. Preparar alícuotas Reporter
Para B. subtilis P reportero-Tapa YFP:
- Racha cepa reportera de -80 ° C madre congelada en una placa de LB fresco con un palillo estéril o aplicador.
- Haga crecer durante la noche a 30 ° C.
- Realizar diluciones en serie en cultivo líquido para minimizar la fluorescencia de fondo que surge de crecimiento en un medio sólido:
- Inocular un cultivo líquido de 5 ml de LB y crecer con agitación a 37 ° C.
- Cuando el cultivo alcanzó una DO600 ~ 0,6, diluir en 5 ml de LB a un OD 600 de 0,02.
- Crece a 37 ° C de nuevo con agitación hasta que los cultivos llega a OD 600 ~ 0,6.
- Repita diluciones seriadas de crecimiento de un total de 3 veces.
- Deje de cultivo final de dilución en serie crecer a OD 600 ~ 0,4.
- Añadir glicerol al 15-20%.
- Alícuota 50-200 l en 0,5 ml tubos de microcentrífuga y congelar a -80 ° C.
- Mamáke alícuotas equivalentes para la cepa parental no fluorescente del reportero (que se requerirán durante el cribado secundario).
Para otras especies bacterianas:
- Utilizar las células que tienen una baja fluorescencia de fondo (es decir, se cultivan en condiciones donde hay poca expresión del promotor utilizado en el reportero).
- Marca y congelar alícuotas que contienen conocidos ufc / ml (unidades formadoras de colonias por ml) que forman de la cepa reportera de manera que un número adecuado de colonias se puede cultivar en cada placa de la pantalla co-cultivo (véase la sección anterior para más detalles).
- Hacer alícuotas equivalentes para la cepa parental no fluorescente (serán necesarios durante la investigación secundaria).
4. Obtener muestras de suelo
- Recoge del suelo en tubos cónicos estériles o bolsas estériles usando una espátula, desechando los 0,5 cm del suelo de la superficie expuesta.
- Añadir solución salina estéril (NaCl al 0,85%) en una proporción de 10 ml por 1 gde suelo para hacer una suspensión del suelo.
- Seleccione un método para desalojar las bacterias de las partículas del suelo: a) el uso inmediato de la muestra fresca o b) la utilización retardada después de la congelación de la muestra.
- Para uso inmediato, suspensión de vórtice durante 1 min.
- Para el uso retardado, mezclar suspensión del suelo en la licuadora para tres ciclos de 1 min, colocando el vaso de la licuadora en hielo durante 1 min descansa entre los ciclos de mezclado.
- Deje lechada suelo conformarse ~ 1 min.
- Mueva capa acuosa superior a un tubo nuevo.
- Añadir glicerol a una concentración final de 15-20%.
- Alícuota 50-200 l en 0,5 ml tubos de microcentrífuga y congelar a -80 ° C.
5. Determinar ufc / ml de Frozen reportero y alícuotas de suelo
- Descongelar un suelo y el reportero congelada alícuota. Los microbios del suelo se pueden descongelar en hielo. Debido a B. subtilis lisis a 4 ° C, esas alícuotas deben descongelarse rápidamente a temperatura ambiente para reducir al mínimo el tiempo dedicado a baja temperatura.
- Hacer dos seria réplical (diluciones a 10 -8) en 0,1 x LB u otro tampón isotónica.
- Placa 5 puntos mu l de cada dilución en serie en placas de agar del mismo medio que se usará para el cribado de cocultivo.
- Crecer a TA (o la temperatura que va a utilizar para la selección).
- El día siguiente, contar el número de colonias en cada punto y calcular ufc / ml de cada una de las alícuotas congeladas.
6. Confirmar alícuotas Concentraciones para placas de pantalla Spread
- Descongelar una alícuota congelada como en el paso 5.1.
- Diluir hasta 1 x 10 5, 2,5 x 10 5, 5 x 10 5, 1 x 10 6, y 2,5 x 10 7 ufc / ml.
- Añadir 50 l lugar de cada dilución a centro de las placas individuales. Estos deberían producir placas con el óptimo calculado de 25.000 colonias por placa, así como 2 - y 5 - veces más y menos, lo que permite la mejor dilución real a determinar.
- Añadir aproximadamente 20 vidrio estéril (3 mm) de cuentas porgolpeando suavemente sobre la placa. Perlas proporcionan una distribución más uniforme de las colonias a través de la placa de un esparcidor de vidrio doblado hace.
- Difundir las células, manteniendo platos en la mesa de trabajo y moviendo hacia atrás y adelante, haciéndolos girar a medida que trabaja, hasta que el líquido se haya absorbido. No continúe sacudir las cuentas una vez que la placa está seca, de lo contrario comenzará a matar a las bacterias.
- Voltear platos una y descartar los granos en el vaso de residuos que contiene etanol.
- Deje placas crecen durante el tiempo de su ensayo (por ejemplo, 24 horas) a la temperatura correcta para su ensayo (por ejemplo, 24 ° C / RT).
- El uso de un estereoscopio de disección, contar el número de colonias en dos o más campos de visión.
- Calcular el número de colonias por área, y determinar el número de colonias son en cada placa.
- Ajuste diluciones futuros según sea necesario. El número real de colonias no es tan importante como tener el mismo número en cada placa de pantalla comparables.
7. Preparar las placas de cocultivo
- Alícuota Descongele reportero (y alícuota del suelo si congelado) como en el paso 5.1.
- Diluir reportero (a concentraciones optimizadas en la sección 6) en 0,1 x LB u otro bajo en nutrientes, solución isotónica.
- Para el suelo congelado: Se diluye hasta la concentración óptima en el apartado 6 en 0.1x LB u otro bajo en nutrientes, solución isotónica.
- Por tierra fresca: Hacer diluciones de lodo fresco suelo, basadas en la predicción de que las ufc / ml de la suspensión de suelo podrían variar de 10 -4 a 10 -9 ufc / ml.
- Punto 50 l de diluciones de suelo y reportero en el centro de la placa de la pantalla co-cultivo. Además, el suelo de placa solo y reportero solos como controles.
- Extender el uso de cuentas de vidrio como se describe en el paso 6.4 a 6.6.
- Si se dan las condiciones de crecimiento conocidos que activan su indicador fluorescente, veta o la placa de su reportero en estas condiciones y crecen para usar como un control positivo en el cribado.
- Se incuba a 24 ° C durante 24 - 28 horas (o según corresponda a su reportero / ensayo)
8. Las placas de cocultivo pantalla para fluorescencia
- El uso de iluminación de campo claro, enfoque su estereoscopio para que las colonias son nítidas.
- Mira las placas de suelo sólo para determinar si su muestra de suelo ha autofluorescentes colonias. Si es así, este suelo puede dar lugar a una alta tasa de falsos positivos (con un aumento concomitante en el cribado secundario).
- Utilice una alta proporción de reportero: suelo en sus placas de cocultivo para aumentar la probabilidad de que un inductor estará rodeado de múltiples colonias reportero, reduciendo la posibilidad de que sean detectados como falsos positivos (Figura 2).
- Utilice una proteína fluorescente diferente (uno que emite en un canal diferente).
- Determinar el momento del ensayo. Para la mayoría de los nuevos reporteros, el momento de la inducción potencial es desconocido, y por lo tanto debe ser determinada empíricamente.
- Comience la detección de fluorescencia, tan pronto como las colonias se vuelven claramente visibles con el estereoscopio de disección (ampliación ~ 30X) y continuar el examen de las placas de manera periódica hasta que haya finalizado el crecimiento y / o la fluorescencia de fondo se vuelve demasiado alto.
- Una vez que la ventana de tiempo del potencial de inducción se determina para un reportero particular, debe ser similar para todas las placas de cocultivo contienen reportero que, simplificar el control de las placas pantalla. Para el B. reportero subtilis P tapa-YFP, el momento oportuno para examinar las placas es de entre 24 a 28 horas después de sembrar las células, porque el B. subtilis P reportero SSPB-YFP, es entre 26-32 horas de crecimiento.
- Maximizar la sensibilidad de la fluorescencia:
- Asegúrese de que su ámbito de aplicación es la disección de la fluorescencia en un cuarto oscuro o rodeado de cortinas opacas. La inducción es probable que sea menos intenso que un FP producido constitutivamente y requiere mayor sensibilidaddad de detectar.
- Dé tiempo para que la lámpara de fluorescencia para estabilizar y sus ojos se acostumbraran a la oscuridad antes de intentar detectar la fluorescencia de las placas de cocultivo (al menos 1-2 min).
- El uso de un control positivo (si tiene uno), asegúrese de que la ampliación que está utilizando le permite detectar la fluorescencia. El aumento es normalmente mejor si su campo de visión es de aproximadamente 30-50x el diámetro de la colonia típica (200-400X).
- Apague la luz brillante y abrir el obturador para su fluorescencia.
- Después de que sus ojos se han adaptado a la oscuridad, mueva lentamente la plancha hacia atrás y adelante a través de su campo de visión, en busca de puntos brillantes.
- Empezar desde la parte superior de la placa y el uso de un patrón de zig-zag para mover el lado a lado de la placa que se mueve hacia la parte inferior de la placa.
- Practique moviendo la placa de campo claro para tener una idea de cómo poco a poco para pasar a la placa, y para estar seguro de que usted está cubriendo todo el surenfrentar área.
- Mueva la placa con la suficiente lentitud que las colonias no se vuelven borrosas. Es mejor sobremuestrear la superficie en lugar de las zonas se pierda.
- Después de un barrido completo, gire la placa de 90 ° y repita. Los ojos humanos son muy buenos en la detección incluso de fluorescencia tenue a través de este método.
- En caso de detectar la fluorescencia, deje de mover la placa y volver atrás y encontrar el área fluorescente.
- Girando el campo claro en lentamente, determinar si la fluorescencia se asocia con una colonia bacteriana (y no detritos del suelo autofluorescente o componentes de medios). Si es así, las colonias no fluorescentes proximales a la colonia fluorescente son organismos que inducen putativo.
9. Aislar putativos Inductores de Organismos
- Una vez inducidos colonias (fluorescentes) han sido identificados, aislar aquellas colonias que secretan los compuestos que inducen.
- Si una concentración suficientemente alta de colonias reportero están creciendo en la placa(> 0,5:1 reportero: ufc suelo), las colonias de inducción putativos estarán rodeados de múltiples colonias fluorescentes (de nuevo, ver Figura 2).
- En los casos en que la complejidad del crecimiento cocultivo hace que sea ambigua que colonia es el inductor, aislar múltiples colonias potenciales de inducción para la posterior prueba en la pantalla secundaria.
- Localizar las colonias que quiere recoger en el centro del campo de visión.
- Si están cerca del borde de la placa, gire la placa de manera que el borde de la placa está lejos de su mano dominante (es decir, si usted es diestro, puso al borde de la placa a la izquierda). Esto le permite acercarse a las colonias en un ángulo bajo, lo que mejora la precisión de su cosecha.
- Coloque placas nuevas a raya los organismos que inducen supuestos a los alrededores, así como un vaso de precipitados de residuos para desechar puntas utilizadas en.
- Al salir de la lámpara de fluorescencia, gire la brillante luz lentamentesucesivamente, de modo que usted será capaz de identificar (por la forma y posición), la colonia fluorescente y los supuestos organismos que inducen circundantes. Puede que tenga que ir y venir con la luz unas cuantas veces para poder identificar las colonias que desea recoger cuando no hay fluorescencia y única luz brillante.
- Utilice una varilla de vidrio (largo x 5 mm de diámetro 200 mm) para recoger una, redondo 200 l consejo de carga de gel estéril y mantenerlo como un lápiz. Esta es la herramienta de recolección que va a utilizar para aislar colonias individuales.
- Descanse su palma exterior contra el escenario para estabilizarlo en la superficie de trabajo. Coloque la otra mano sobre el, lado del pulgar interior de su mano para estabilizar la herramienta de selección de valores.
- Si es necesario, dar la vuelta de nuevo entre las vistas de fluorescencia y campo claro para identificar las colonias que desea recoger.
- Manteniendo la punta de la pipeta por encima de la superficie de la placa, se mueven en su campo de visión y centrarla sobre la colonia que le gustaría recoger. La punta estará fuera de foco.
- Usando el borde exterior de su mano (que está descansando sobre la platina del microscopio o superficie de trabajo), girar la punta de la pipeta lentamente hacia abajo a la colonia para ser recogidos. Toca muy a la ligera, tratando de reducir al mínimo el número de otras colonias los contactos de la punta.
- Sin girar la herramienta de picking, consecutivas en una sección de una placa fresca. Corre con un toque suave para evitar la especulación del agar. Debido a que el número de células transferidas por este método es bastante pequeña (las colonias son mucho menores que típicamente manipulado), una sola raya continua dará lugar a colonias aisladas.
- Repita este proceso con otros organismos de inducción putativos.
- Incubar las placas a 24 ° C (o la temperatura de su ensayo).
10. Racha putativos Inductores Organismos para obtener colonias individuales aisladas
- El uso de un estereoscopio, determinar - sobre la base de la estructura de la colonia o la morfología - ya que hay diferentes tipos de colonias que aparecen en cada uno de su ind recogido putativoucing organismos.
- Mira las colonias utilizando una etapa donde se puede iluminar las colonias, tanto de arriba como de abajo para detectar posibles diferencias de colonias.
- Restreak cada morfotipo diferente en un plato fresco y se incuba hasta que creció.
- Restreak una vez más y dejar crecer. Si los diferentes morfotipos persisten, continúe restreak a la pureza.
11. Vuelva a probar putativos Inductores de Organismos en la pantalla secundaria
- Volver a probar todos los organismos que inducen putativos en una pantalla secundaria para determinar qué está activando el reportero transcripcional fluorescente. La pantalla secundaria se compone de un césped de microcolonias de la cepa reportero transcripcional fluorescente, junto con las placas de control, en el cual los organismos que inducen putativos están parcheados o manchados.
- Establecer tres placas idénticas: una que contiene un césped microcolonia de la cepa reportero transcripcional fluorescente (en la misma concentración de uns fue utilizado durante la investigación co-cultivo), uno que contiene un césped microcolonia de la cepa parental de tipo salvaje sin un reportero de fluorescencia, y uno que no contiene césped.
- Marque la parte superior de la parte posterior de cada placa para determinar la orientación de la placa.
- Agregar marcadores de posición para los parches / puntos; un máximo de diez organismos que inducen putativos se pueden probar en cada juego de placas (Figura 3).
- Alícuotas de césped Descongelar (reportero y deformación no fluorescente padre) como en el paso 5.1.
- Corre 50 l de una dilución 5 x 10 5 ufc / ml en placas de césped (o de otras diluciones optimizados) usando perlas estériles como en el paso 6.4.
- Deje placas secas.
- Parche o detectar organismos putativo inducir:
- Seleccionar parches si se desea un enfoque más sencillo y más rápido, y es aceptable tener un número menos precisa de las células depositadas. Para parchear:
- Toque un palillo estéril a la colonia a prueba - no recoger todas las células.
- Patch (hacer una pequeña raya) en la placa en blanco.
- Repita el parche con un palillo fresco sobre la placa indicadora.
- Repita el parche con un palillo fresco sobre la placa de control.
- Seleccionar manchado si se desea un enfoque cuantitativo y reproducible. Spotting permite que el número de células depositadas a normalizarse (véase la referencia 5 para obtener más detalles), y permite que la potencia relativa de los diferentes organismos de inducción para ser comparados. Para detectar:
- Resuspender putativo inducir organismos en 1 ml de medio líquido en una cubeta de plástico estéril.
- Tome el OD 600 de las resuspensiones.
- Uso de la fórmula X = 250 ÷ (OD 600 a 0,5), añadir el volumen X para cada resuspensión a 500 l de medio líquido para obtener una solución con un OD 600 de 0,5. Este método simplifica los pasos de pipeteado requeridos al realizar varias diluciones para normalizar OD de porque se puede utilizar el mismo volumen (500 l) para todas sus diluyentes.
- Punto 1 l de cada resuspensión-DO normalizada para cada una de las tres placas.
- Seleccionar parches si se desea un enfoque más sencillo y más rápido, y es aceptable tener un número menos precisa de las células depositadas. Para parchear:
- Deje crecer a 24 º C durante 24 a 28 horas (o según corresponda a su reportero / ensayo).
- Utilice el microscopio de disección fluorescente para identificar organismos que inducen supuestos que activan su cepa indicador fluorescente pero no su cepa de control parental. Estos aislados son sus éxitos positivos - los microbios ambientales que secretan compuestos que inducen el fenotipo de interés.
Resultados
Se utilizó este pantalla para identificar los organismos del suelo secretoras de compuestos que alteran la fisiología de B. subtilis. Los resultados descritos aquí se centran en el tipo de célula-matriz de la producción de B. subtilis, que produce la proteína y de exopolisacáridos que se requieren para la formación de biopelículas robusta en esta bacteria. Seleccionamos el promotor del operón tapa-sipW-TASA para nuestra construcción informadora fluorescente (P tapa-YFP). Este operón codifica el componente estructural de proteínas de la matriz y se regula positivamente durante la producción de matriz del biofilm 23. Nuestro reportero matriz (Figura 1) se construyó como se ha descrito previamente 19.
Trabajos anteriores han demostrado que B. subtilis produce matriz en respuesta a la percepción de quórum-surfactina como molécula de producción propia, así como metabolitos purificada producida por otras bacterias del suelo 20. Estábamos interesados en la ampliación de laestudios sí para investigar de manera más amplia que los microbios del suelo hacen metabolitos capaces de inducir la producción de matriz en B. subtilis. Hemos elegido para utilizar la dilución LB para el crecimiento, ya que este medio ya era conocido por llevar a la producción de la matriz pobres 20, que nos proporciona una condición de crecimiento donde nuestro reportero cepa era no fluorescente. A continuación, optimizado el número de colonias adecuadas para el cribado de bajo estas condiciones de crecimiento. Con el fin de optimizar cada placa de la pantalla, es necesario determinar el número de colonias crecen a partir de las alícuotas del suelo y del reportero congelados y lo que la concentración adecuada de colonias y condiciones de nutrientes son. Lo ideal es que queremos que cada placa de co-cultivo para contener un número equivalente de colonias de suelo y reportero (es decir, una proporción de 1:1 de reportero: suelo) y al estar estrechamente espaciadas, las colonias individuales. Esta alta proporción de colonias reportero aumenta la probabilidad de que un inductor activará múltiples colonias reportero circundantes. Tener múltiples accolonias inductor inactivados que rodean una confianza aumenta colonia inductor putativo en la localización del organismo reales de inducción (Figura 2). El contenido de nutrientes controla el grado de formación de crecimiento / colonia, mientras que la dilución del inóculo determina si las colonias resultantes se dispersan adecuadamente. En una placa de Petri de 10 cm de diámetro estándar con medio bajo en nutrientes, se encontró que aproximadamente 25.000 colonias total por placa (50 l de 5 x 10 5 ufc / ml de dilución), siempre la mejor separación de B. subtilis colonias en medio MOPS 0,1 X. LB (Figura 4).
Aunque el ufc calculado / ml de las diluciones en serie proporciona una concentración aproximada de bacterias en las partes alícuotas, es necesario asegurar que la concentración de las colonias resultantes es apropiado cuando una placa entera se propaga con células. El ufc / placa calculado y ufc / placa de concreto pueden no coincidir son idénticos (Figura 4 ). Plating céspedes de colonias de concentraciones equivalentes Es importante permitir que diferentes cepas reportero a comparar (en caso contrario, las diferencias en la disponibilidad de nutrientes pueden alterar su estado fisiológico e interferir con los resultados).
Después de preparar alícuotas de la periodista y el suelo, les mezclamos en placas de pantalla co-cultivo y los examinó por fluorescencia utilizando un estereoscopio (Figura 5). También chapada controles que sólo se inocularon con ya sea el suelo o el B. P subtilis cepa reportera tapa-YFP. B. subtilis produce matriz del biofilm (fluorescencia) en respuesta a numerosos microbios del suelo como se ve por las colonias fluorescentes en la imagen cocultivo en la Figura 5. Para los suelos que examinamos, tuvimos unas altas tasas de éxito para el reportero P tapa-YFP. Como se describe en la referencia 5, entre 12 a 67% de los aislados (de seis muestras de suelo distintos) tenían la capacidad de indufluorescencia ce en la cepa reportera P tapa-YFP. Esto está en contraste con nuestros resultados no publicados de pantallas análogas mediante la esporulación (P SSPB-YFP) y competencia (P ComG-YFP) a los periodistas. Después de una extensa proyección (> 200.000 colonias por cada reportero), sólo se identificaron dos organismos que inducen la esporulación, mientras que ninguno se identificó que inducen la competencia. Por lo tanto, las tasas de éxito para los diferentes tipos de células se ser muy variables y pueden ser difíciles de predecir de antemano.
Luego recogimos colonias inductores putativos individuales. Las colonias en las placas de cocultivo de pantalla son muy pequeñas en el medio bajo en nutrientes se recomienda (diámetro inferior al milímetro). Sin embargo, es posible recoger con precisión y aislar colonias muy pequeñas con la mano (Figura 6) desde el interior de una placa de pantalla complicado cocultivo. El método manual que utilizamos es simple y no requiere ni herramientas especiales ni la esterilización de la llama. Estas colonias inductor putativos son entonces restruck al aislamiento. Debido a que las placas de cocultivo están llenas de colonias, no es raro que - incluso con la técnica de picking muy cuidado - al tener más de un organismo en crecimiento de cada muestra inductor putativo. Un examen cuidadoso debe permitir el aislamiento de las colonias morfológicamente distintos. Todos los organismos que inducen putativos se ensayaron a continuación en una pantalla secundaria. Los resultados positivos y negativos de tanto el parche y el método de punto se muestran en la Figura 3. Teniendo en cuenta su crecimiento denso, nuestra capacidad de recolectar físicamente inducir colonias de las placas de cocultivo fue bastante bueno, con aproximadamente el 50% de las colonias examinadas en ser nuestra pantalla secundaria verdaderos positivos. Resultados adicionales de esta pantalla, así como el trabajo de seguimiento que surge de haber sido previamente descrito 5.
/ Ftp_upload/50863/50863fig1.jpg "/>
Figura 1. Transcripcional reportero fluorescente constructo. El óvalo azul representa una célula bacteriana y la línea discontinua representa su cromosoma. Este ejemplo muestra un reportero transcripcional fluorescente para la producción de la matriz. El locus nativo permanece intacta (matriz-matriz P, donde "P" y la flecha indica la región del promotor), mientras que el constructo indicador (P matriz-YFP) se inserta en otra parte en el cromosoma en un locus neutro.
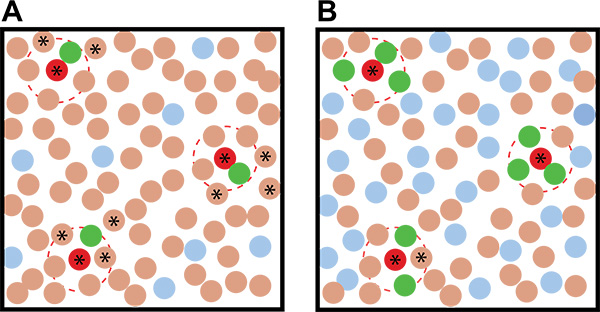
Figura 2. Ejemplos idealizados de cocultivo resultados del cribado con diferentes proporciones de la reportera: microbios ambientales. A) El uso de un reportero baja: proporción de microbios del medio ambiente conduce a una mayor ambigüedad en la identificación de supuestos organismos que inducen que cuando B) Un alto reportero: se utiliza la relación de microbios del medio ambiente. Los círculos marrones representan los organismos del suelo, los círculos rojos representan inductores de los organismos del suelo, los círculos azules representan las colonias reportero no inducidas, y las colonias verdes representan colonias reportero inducidos. Las líneas rojas punteadas indican el radio de acción del metabolito induce. Las estrellas indican colonias no fluorescentes que - en base a su proximidad a las colonias fluorescentes - son organismos que inducen supuestos y deben ser recogidos y ensayarse en la pantalla secundaria.
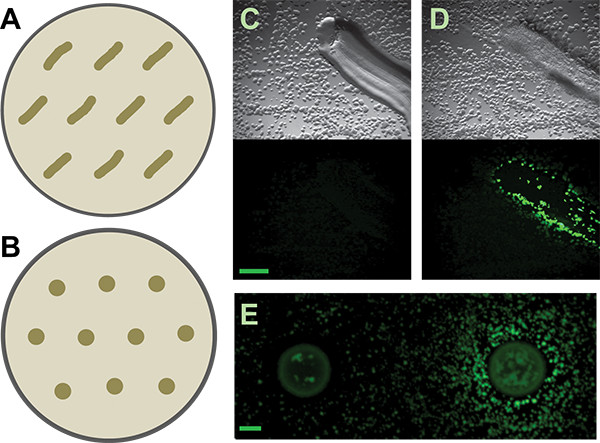
Figura 3. Pantalla secundaria. A y B) Esquema de la forma de distribuir parcheado o manchado aislamientos en placas de pantalla secundaria, respectivamente, para el B. reportero matriz subtilis. Más generosa separación puede ser necesaria para otros reporteros o aislamientos que inducen, depque termina en la capacidad de difusión de sus metabolitos activos. C y D) Los resultados representativos de suelos parcheado aislados que son negativos y positivos, respectivamente, para la inducción de la B. subtilis P tapa-YFP-reportero. Los paneles superiores son las imágenes de campo claro, paneles inferiores son las imágenes de fluorescencia. La barra de escala es de 1 mm. E) Negativo y los resultados positivos de suelo manchado aísla por el mismo periodista. La barra de escala es de 2 mm.

Figura 4. Determinación de la concentración microcolonia. La distribución y el tamaño de sus colonias dependerán tanto de las concentraciones de nutrientes y de la célula.) Diferencias A en el crecimiento de B. subtilis en 0.01x LB (fila superior) frente a 0.08x LB (fila inferior). Las células en 0.01x LB no se forman en microcolonias, mientras que tmanguera en 0.08x LB hacer. (Tenga en cuenta que para nuestras pantallas aumentamos los niveles de nutrientes ligeramente de los que se muestra aquí:. Desde 0.08x LB a 0.1x LB) Estas imágenes son de 1 mu l manchas de sucesivas diluciones 1:5 en conocido ufc / ml. La extrapolación de estas concentraciones, para obtener distribuciones similares de las colonias a través de una placa de Petri de 10 cm requeriría chapado (desde la izquierda): 3.200.000; 640.000 y 128.000 total de UFC por placa. Sin embargo, manchado resultados en la distribución desigual de las células (que se concentran en los bordes del punto) en comparación con la difusión de las células sobre toda la placa. Por lo tanto, una vez que se selecciona una concentración de nutrientes, es importante examinar las placas untadas con una variedad de concentraciones. La barra de escala es 0,1 mm B) Estos paneles muestran los resultados de la difusión (por la izquierda) 50.000;. 25000, y 5000 ufc total por placa en placas de LB 0.08x. A partir de estas imágenes, se seleccionaron 25.000 como nuestro número objetivo de ufc / placa. La barra de escala es de 0,1 mm.

Figura 5. Cocultivo de B. subtilis P tapa-YFP mezclado con los organismos del suelo. Superposición de imagen de fluorescencia de campo claro y de una placa de la pantalla co-cultivo que contiene el B. subtilis P reportero matriz Tapa YFP se mezcla con los organismos del suelo. Arrowhead indica inductor putativo rodeado de microcolonias reportero de fluorescencia. La barra de escala es de 1 mm.
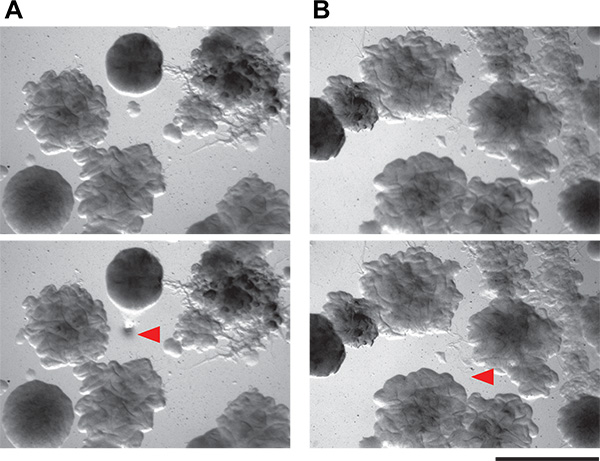
Figura 6. Demostración de la viabilidad de aislar colonias bacterianas diminutas de placas de cocultivo. A y B) Estos paneles muestran dos campos de visión de placas de agar que contienen las comunidades microbianas complejas desde el suelo. Colonias tan pequeños como 0,1 mm se pueden aislar usando la técnica de picking se describeaquí. Los paneles superiores son el campo de visión antes de la recolección de la colonia, y el panel inferior son los mismos campos de visión después de la recolección de la colonia. Puntas de flechas rojas indican donde se han eliminado las células.
Discusión
Una de las limitaciones inherentes de este protocolo es que se basa en la cultivabilidad de organismos microbianos. Como ha sido bien documentado 24, mayoría de la vida microbiana en el planeta no puede (aún) ser cultivado bajo las condiciones de cultivo explorados hasta la fecha. Por lo tanto, un gran número de interacciones entre especies microbianas que se están produciendo en los entornos naturales no sea detectada utilizando este enfoque. Sin embargo, ya que es nuestro deseo para identificar no sólo la existencia de tales interacciones, pero entonces también estudiar los mecanismos y moléculas que participan en la mediación de ellos, la capacidad para cultivar estos microbios es una necesidad. Incluso dentro de las especies cultivables, esta zona ha sido poco explorado, lo que hace el enfoque descrito aquí una valiosa contribución como un método para identificar las interacciones mediadas químicamente entre los microbios. Además, aunque este protocolo se ha optimizado para la detección de la matriz de la inducción de Bacillus subtilis, que puede teóricamente ser aplicada a unfluorescente reportero transcripcional y en cualquier otras especies bacterianas.
Otra limitación relacionada de este enfoque es que esta pantalla (por definición) requiere cocultivo. En ambientes naturales, los microbios con diferentes tasas de crecimiento todavía pueden coexistir en la proximidad espacial, mientras que la explotación de distintos nichos ambientales. Tales interacciones microbianas no sería detectada por nuestra pantalla de cocultivo, sin embargo, que sólo permite el crecimiento de microbios ambientales con las necesidades de nutrientes y las tasas de crecimiento similares a las de las especies reportero. Modificaciones que separar el crecimiento de los potenciales organismos inductores del crecimiento de la cepa reportero son ciertamente posibles. También prevé que el crecimiento de las hifas de los hongos - comunes en el suelo - podría causar dificultades en la pantalla de co-cultivo. Si bien a corto plazo de tiempo de nuestra pantalla con B. subtilis significaba que se detectaron pocos hongos, la adición de compuestos antifúngicos al medio de crecimiento podría meducirán al mínimo esta preocupación.
La capacidad para seleccionar un fenotipo y el gen apropiado para la construcción informadora fluorescente no debería ser difícil, teniendo en cuenta la riqueza de la secuenciación y los datos de la transcripción o bien ya disponibles o fácilmente obtenible para muchas especies bacterianas. Sin embargo, una dificultad con el enfoque descrito aquí es la necesidad de identificar las condiciones de crecimiento que minimizan la fluorescencia de fondo de la cepa reportera, que permite la detección de la inducción de fluorescencia. La identificación de estas condiciones a menudo debe hacerse empíricamente, aunque los datos de la transcripción pueden ayudar a esta búsqueda (por ejemplo los datos de microarrays suelo de baldosas disponibles para el crecimiento de B. subtilis permita la identificación de condiciones donde los genes de interés se expresa pobremente 10). Para algunos periodistas esta búsqueda empírica puede ser un reto, en parte debido a la expresión de muchos fenotipos bacterianos es heterogénea. En otras palabras, es raro encontrar a cONDICIONES en el que no hay células dentro de la población están expresando Fenotipo X. Por lo tanto, dependiendo del número de células dentro de esa subpoblación y la fuerza de la expresión génica, puede ser difícil identificar las condiciones que proporcionan suficientemente baja fluorescencia de fondo para permitir la inducción a detectar . Una alternativa a esta búsqueda empírica de las condiciones ideales de detección puede ser la de "afinar" los niveles de expresión del reportero mediante mutagénesis dirigida. Mediante la alteración de la región promotora y / o sitio de unión ribosómico del constructo reportero, los niveles de fluorescencia de fondo podrían ser disminuidas. Esto podría ampliar la utilidad de esta pantalla permite que incluso los genes con cierta activación constitutiva de ser examinados para la inducción.
Una vez que los organismos que inducen han sido identificado y confirmado en una pantalla secundaria, pueden ser identificados filogenéticamente por secuenciación de su gen 16S ARNr. También es posible cuantificar el grado de fluorescenciacencia utilizando OD punto 600-normalizado en la pantalla secundaria 5. Esto puede proporcionar información acerca de las cuales los miembros de la comunidad producen compuestos que afectan a su reportero cepa y en qué medida. En consecuencia, esto puede dar lugar a hipótesis sobre la cual las interacciones microbianas pueden estar ocurriendo en los entornos naturales y la capacidad de explorar la coevolución potencial de estos organismos que producen y responden. Otras direcciones futuras incluyen la aclaración de la estructura de la molécula secretada en sí, la determinación del mecanismo (s) por el cual el organismo que responde detecta este compuesto, y su uso como una herramienta química para modular fenotipos bacterianas.
Incluso con las consideraciones expuestas, el método descrito aquí es una contribución importante. Evita la mano de obra necesaria en el montaje de una biblioteca de microbios ambientales, sino que permite su separación física y el aislamiento mediante el uso de los medios sólidos. La fuerza de esta pantalla es que cocultivose proporciona un método conceptual y técnicamente sencillo de pantalla a través de miles de especies microbianas para identificar aquellos que secretan compuestos bioactivos de interés mientras que ser aplicable a muchas especies bacterianas y fenotipos.
Divulgaciones
Los autores declaran que no tienen intereses financieros en competencia.
Agradecimientos
El autor agradece a Roberto Kolter (Harvard Medical School) por su asesoramiento y asistencia durante el desarrollo de esta pantalla cocultivo invaluable. Agradece también a Mateo Poderes por leer el manuscrito para mayor claridad, y Chia-yi Cheng para obtener ayuda con la obtención de la figura 6.
Materiales
| Name | Company | Catalog Number | Comments |
| Spectrophotometer | Any spectrophotomer capable of measuing OD600 absorbance values. | ||
| Luria broth, Lennox | VWR | 80017-484 | Alternative media sources may be necessary. |
| Glass beads, 3 mm | VWR | 26396-508 | |
| Gel loading tips, round | VWR | 29442-666 | |
| Glass rods | VWR | 59060-069 | |
| Fluorescence dissecting stereoscope | Zeiss | N/A | The author used a Zeiss Stemi SV6 dissection stereoscope with an EXFO X-cite 120 fluorescent light source, a long-pass YFP filter cube, an achromat 0.63X objective, 10X eyepieces, and an Axio HRC HR digital camera. Most screening was done with the focusing mount at 2.0-3.2X. Any dissecting stereoscope with fluorescence capabilities is fine, provided you have the correct filters for the FP you are using. It is best if there is a shutter that allows you to easily switch between brightfield and fluorescense, as well as a stage that allows illumination from above and below. If you want to capture images, an attached camera is also necessary. |
Referencias
- Berdy, J. Bioactive microbial metabolites. J. Antibiot. 58, 1-26 (2005).
- Lopez, D., Vlamakis, H., Kolter, R. Generation of multiple cell types in Bacillus subtilis. FEMS Microbiol. Rev. 33, 152-163 (2009).
- Vlamakis, H., Aguilar, C., Losick, R., Kolter, R. Control of cell fate by the formation of an architecturally complex bacterial community. Genes Dev. 22, 945-953 (2008).
- Branda, S. S., Gonzalez-Pastor, J. E., Ben-Yehuda, S., Losick, R., Kolter, R. Fruiting body formation by Bacillus subtilis. Proc. Natl. Acad. Sci. U.S.A. 98, 11621-11626 (2001).
- Shank, E. A., et al. Interspecies interactions that result in Bacillus subtilis forming biofilms are mediated mainly by members of its own genus. Proc. Natl. Acad. Sci. U.S.A. 108, 1236-1243 (2011).
- Piston, D. W., Patterson, G. H., Lippincott-Schwartz, J., Claxton, N. S., Davidson, M. W. . Introduction to Fluorescent Proteins. , (2013).
- Chudakov, D. M., Matz, M. V., Lukyanov, S., Lukyanov, K. A. Fluorescent proteins and their applications in imaging living cells and tissues. Physiol. Rev. 90, 1103-1163 (2010).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. J. Cell Sci. 120, 4247-4260 (2007).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat. Methods. 2, 905-909 (2005).
- Nicolas, P., et al. Condition-dependent transcriptome reveals high-level regulatory architecture in Bacillus subtilis. Science. 335, 1103-1106 (2012).
- Middleton, R., Hofmeister, A. New shuttle vectors for ectopic insertion of genes into Bacillus subtilis. Plasmid. 51, 238-245 (2004).
- Shimotsu, H., Henner, D. J. Construction of a single-copy integration vector and its use in analysis of regulation of the trp operon of Bacillus subtilis. Gene. 43, 85-94 (1986).
- Guerout-Fleury, A. M., Frandsen, N., Stragier, P. Plasmids for ectopic integration in Bacillus subtilis. Gene. 180, 57-61 (1996).
- Semsey, S., Blaha, B., Koles, K., Orosz, L., Papp, P. P. Site-specific integrative elements of rhizobiophage 16-3 can integrate into proline tRNA (CGG) genes in different bacterial genera. J. Bacteriol. 184, 177-182 (2002).
- Charpentier, E., et al. Novel cassette-based shuttle vector system for gram-positive bacteria. Appl. Environ. Microbiol. 70, 6076-6085 (2004).
- Yang, H. Y., Kim, Y. W., Chang, H. I. Construction of an integration-proficient vector based on the site-specific recombination mechanism of enterococcal temperate phage phiFC1. J. Bacteriol. 184, 1859-1864 (2002).
- Choi, K. H., Schweizer, H. P. mini-Tn7 insertion in bacteria with single attTn7 sites: example Pseudomonas aeruginosa. Nat. Protoc. 1, 153-161 (2006).
- Craig, N. L. Tn7: a target site-specific transposon. Mol. Microbiol. 5, 2569-2573 (1991).
- Garcia-Betancur, J. C., Yepes, A., Schneider, J., Lopez, D. Single-cell analysis of Bacillus subtilis biofilms using fluorescence microscopy and flow cytometry. J. Vis. Exp. (60), e3796 (2012).
- Lopez, D., Fischbach, M. A., Chu, F., Losick, R., Kolter, R. Structurally diverse natural products that cause potassium leakage trigger multicellularity in Bacillus subtilis. Proc. Natl. Acad. Sci. U.S.A. 106, 280-285 (2009).
- Vartoukian, S. R., Palmer, R. M., Wade, W. G. Strategies for culture of 'unculturable' bacteria. FEMS Microbiol. Lett. 309, 1-7 (2010).
- Romano, J. D., Kolter, R. Pseudomonas-Saccharomyces interactions: influence of fungal metabolism on bacterial physiology and survival. J. Bacteriol. 187, 940-948 (2005).
- Branda, S. S., Chu, F., Kearns, D. B., Losick, R., Kolter, R. A major protein component of the Bacillus subtilis biofilm matrix. Mol. Microbiol. 59, 1229-1238 (2006).
- Zengler, K., et al. Cultivating the uncultured. Proc. Natl. Acad. Sci. U.S.A. 99, 15681-15686 (2002).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados