Method Article
Mit Cokultur zur Erkennung chemisch vermittelte Interspezies-Interaktionen
In diesem Artikel
Zusammenfassung
Bakterien produzieren sezernierten Verbindungen, die das Potenzial, die Physiologie der Mikroorganismen ihre Nachbarn beeinflussen. Hier beschreiben wir eine Co-Kultur Bildschirm, der Nachweis solcher chemisch vermittelte Inter Wechselwirkungen durch Mischen von Bodenmikroben mit fluoreszierenden Reporter-Transkriptions Stämmen von Bacillus subtilis auf festen Medien ermöglicht.
Zusammenfassung
In der Natur, Bakterien existieren selten isoliert, sondern werden durch eine Vielfalt von anderen Mikroorganismen, die die lokale Umwelt zu verändern durch Sekretion von Metaboliten umgeben. Diese Metaboliten haben das Potenzial, die Physiologie und die Differenzierung ihrer mikrobielle Nachbarn modulieren und sind wahrscheinlich wichtige Faktoren bei der Errichtung und Instandhaltung von komplexen mikrobiellen Gemeinschaften. Wir haben eine Fluoreszenz-basierten Kokultur Bildschirm, um solche chemisch vermittelte mikrobielle Wechselwirkungen identifizieren. Der Bildschirm beinhaltet die Kombination eines fluoreszierenden Reporterstamm mit Transkriptionsumwelt Mikroben auf festen Medien und so die Kolonien in Co-Kultur wachsen. Die Leuchtstofftranskriptions Reporter ist so konzipiert, dass die gewählte Bakterienstamm fluoresziert, wenn es einen bestimmten Phänotyp von Interesse exprimieren (dh die Biofilmbildung, Sporenbildung, Virulenzfaktor Produktion, etc.) Screening ist unter Wachstumsbedingungen durchgeführt wheWieder dieser Phänotyp nicht exprimiert wird (und damit der Reporter-Stamm ist in der Regel nicht fluoreszierend). Wenn ein Umwelt Mikrobe sondert ein Metabolit, der diesen Phänotyp aktiviert, diffundiert es durch den Agar und aktiviert die fluoreszierenden Reporter-Konstrukt. Dies ermöglicht die Induzierung Metaboliten produzierenden Mikroben nachzuweisenden: sie sind nicht-fluoreszierenden Kolonien meisten proximal zu den fluoreszierenden Kolonien. Somit ermöglicht dieser Bildschirm die Ermittlung von Umwelt Mikroben, die diffusionsfähige Metaboliten, die eine bestimmte physiologische Reaktion in einem Reporterstamm aktivieren produzieren. Diese Publikation beschreibt, wie: a) wählen Sie die entsprechende Co-Kultur Screeningbedingungen, b) bereiten die Reporter und Umwelt Mikroben für Screening, c) führen die Co-Kultur-Bildschirm, d) zu isolieren mutmaßlichen Induktion Organismen, und e) bestätigen ihre Tätigkeit in einem zweiten Bildschirm. Wir haben diese Methode, um für Bodenorganismen, die Biofilmmatrix-Produktion in Bacillus subtilis aktivieren Bildschirm
Einleitung
Wir interessieren uns für das Verständnis, wie die Metaboliten, die Bakterien absondern, beeinflussen die Physiologie und die Entwicklung der Nachbar Mikroben. Viele Metaboliten wurden für ihre bioaktiven Effekte auf andere Mikroben gekennzeichnet. Zwei gut beschriebenen Beispiele sind Antibiotika, die das Wachstum anderer Mikroorganismen hemmen und Quorum Sensing-Moleküle, die die globale Genexpression von anderen Mikroben zu ändern. Allerdings Bakterien produzieren viele andere kleine Molekül natürlichen Produkten, die keine bekannten Bioaktivitäten 1 haben. Wir vermuten, dass Bakterien entwickelt haben und erhalten die Möglichkeit, einige dieser Stoffwechselprodukte, weil sie es ihnen ermöglichen, die zelluläre Physiologie ihrer mikrobielle Nachbarn in den komplexen mikrobiellen Gemeinschaften, in dem die meisten Bakterien existieren modulieren.
Bacillus subtilis Zelltypen
Wir haben unsere Studien über chemisch vermittelte mikrobielle Interaktionen, die bacil beinhalten konzentriertlus subtilis. Dies ist nicht nur wegen ihres Status als den gram-positiven Bakterium Modell und den daraus resultierenden genetischen Werkzeuge für seine Betätigung zur Verfügung, sondern auch wegen seiner Fähigkeit, gekennzeichnet Zelltypen. Beispiele sind Zellen, die sind: Schwimmen, Herstellung der extrazellulären Matrix, die für robuste Biofilmbildung erforderlich ist; zuständigen aufnehmen DNA aus der Umgebung, und sporenbildenden, unter anderen zwei. Jeder dieser Zelltypen exprimiert ein Merkmal Transkriptions-Regulon, das sie physiologisch und / oder physikalisch von den genetisch identischen Geschwistern macht. Unter vielen Wachstumsbedingungen, mehreren Zelltypen existieren verschiedene Subpopulationen innerhalb einer einzigen Kolonie von B. subtilis-Zellen 3. Obwohl viele Arten von Bakterien können analog Zelltyp Heterogenität aufweisen, dieses Phänomen wurde besonders gut in B. studierte subtilis.
Insbesondere sind Gene, die UPRInnerhalb jeder dieser spezifischen B egulated subtilis Zelltypen identifiziert worden. Identifizieren solcher hochregulierte Gene ist für die hier beschriebene Arbeit, weil viele dieser mikrobiellen Phänotypen von Interesse schwierig oder unmöglich direkt zu beobachten sind. Zum Beispiel können wir nicht visuell ein Merkmal zu erkennen, wie Schwimmen auf solide (1,5%)-Agar-Platten, auch wenn eine Subpopulation von B. subtilis-Zellen produzieren Geißeln unter diesen Bedingungen 3. Ein weiteres Beispiel ist Biofilmmatrix-Produktion. Matrixproduktion durch Koloniemorphologie sichtbar gemacht werden (wie es in makroskopisch faltige Kolonien führt), sondern nur auf bestimmte Wachstumsmedium, und erst nach mehreren Tagen des Wachstums 4. Doch wir wissen, welche Gene während der Differenzierung hochreguliert, können wir Transkriptions Reportern, die als Marker für zelluläre Differenzierung in diesen Zelltypen wirken zu konstruieren.
Reporter-Konstrukte
Diese Fluoreszenz transcriptional Reporter aus den Promotoren für die Zelltyp-spezifische Gene Antreiben der Herstellung eines Reportergens, zum Beispiel ein fluoreszierendes Protein ist. Beispiele umfassen P hag-YFP (zum Schwimmen Zellen), P Tapa-YFP (für Biofilm-Matrix-produzierenden Zellen) und P SSPB-YFP (für sporen Zellen), wobei P x die Promotorregion für die Gen-x. Diese Reporterkonstrukte werden in einem neutralen Locus auf Chromosom integriert (Fig. 1 und siehe unten), so daß das native Regelung der Phänotyp bleibt intakt. Aber jetzt, wenn eine Zelle diesen Phänotyp exprimiert, es drückt auch ein fluoreszierendes Protein. Dies stellt eine leicht visualisiert Auslesen der Aktivierung bestimmter phänotypisches Verhalten, so dass wir für diese Mikroben, die physiologische Reaktion zu aktivieren screenen. Obwohl solche Journalisten werden häufig in der Mikrobiologie eingesetzt, sie wurden nicht weitgehend Bildschirme identif angewendety metabolische Wechselwirkungen zwischen Mikroben vor dieser Methode wurde 5 beschrieben.
Es gibt eine Anzahl von wichtigen Überlegungen bei der Gestaltung und Konstruktion von Zelltyp-spezifischen Reporterstämmen. Wir haben ausschließlich Transkriptions fluoreszierenden Reportern verwendet, obwohl andere Typen von Konstrukten sind sicherlich möglich. Wir raten von der Verwendung der translationalen Fusionen als Marker für Zelltyp Differenzierung in unserem Bildschirm, jedoch aus zwei Gründen: 1) der Wunsch, die nativen Zelltyp-spezifischen Protein unbeeindruckt lassen, und 2) die Erkenntnis, dass eine diffuse, Zell- breite Fluoreszenz wird leichter zu erkennen als in den Zellen lokalisiert puncta (gemeinsam mit translationale Fusionen).
Reporter-Gen-Auswahl
Nach der Entscheidung, die Transkription als Auslese verwenden, muss der Reporter-Gen ausgewählt werden (zB LacZ, Fluoreszenz oder Luciferase). LacZ hat den Vorteil, benötigen mindestens die SonderIzed Ausrüstung, um zu erkennen, aber es gibt eine viel höhere Wahrscheinlichkeit von Fehlalarmen unter Umwelt Mikroben. In unseren Händen, der Hintergrundpegel von Lac + Organismen unter Bodenmikroben war unerschwinglich hoch (>> 10% der Bodenmikroben waren blau (Lac +) auf der X-Gal-Platten, Daten nicht gezeigt). Es ist möglich, dass durch Titrieren der Konzentration von X-gal in dem Medium, das könnte optimiert werden, um die Verwendung einer X-Gal-Reporter ermöglichen, obwohl wir nicht versucht dieses. Luciferase eine hohe Nachweisempfindlichkeit und ist die orthogonale Reporter: Es gibt nahezu keine Chance Umweltmikroben inhärent Leucht. Allerdings fanden wir es schwierig, Instrumentierung an unserer Hochschule, die Lumineszenzdetektion erlaubt über die gesamte Petrischalen zu identifizieren, da die meisten wurden entwickelt, um nur lokalisierte Bereiche in Multi-Well-Platten zu scannen. Es könnte auch Komplikationen bei der Visualisierung Leucht Kolonien in einer Weise, die auch erlaubt die gleichzeitige körperliche ist seinBildung von Ol-Organismen zu induzieren. Bei der Verwendung von Treuhändern kann dies möglich gemacht haben, haben wir entschieden, statt Transkriptions fluoreszierenden Reporter, die bewiesen wurden in B. arbeiten, verwenden subtilis, sofern ausreichende Nachweisempfindlichkeit und niedrige False-Positive-Raten unter den Bodenorganismen und erlaubt, von leicht verfügbaren Instrumente sowohl für die Visualisierung und Isolierungsverfahren zu verwenden.
Fluorophor-Auswahl
Die spezifische Fluorophor ausgewählt werden auf Ihrem Bakterienarten ab, die Agar-Nährmedium Sie verwenden, und die insbesondere Fluoreszenzfilter setzt Sie zur Verfügung haben. Mit unserer Messtechnik, haben wir festgestellt, dass sowohl die B. subtilis Kolonien selbst und die sie auf Agar zeigten weniger Hintergrundfluoreszenz gewachsen, wenn YFP (gelb fluoreszierendes Protein)-Filter verwendet wurden, so dass Reporter überlegen GFP (green fluorescent protein) in unseren Händen. Die Codon-Verwendung von fluoreszierenden Proteinen sindhäufig für Eukaryoten optimiert, so dass es wichtig ist, eine Fluorophor entweder aus der Literatur bekannt, in Ihrem Bakterienarten zu arbeiten, oder sie explizit über einen konstitutiven Promotor testen auswählen. Eine große Anzahl von sich ständig weiterentwickelnden fluoreszierenden Protein-Varianten sind derzeit verfügbar 6, die in einer Reihe von Quellen, 7,8, von denen einige Leitlinien ausdrücklich vor, auf die Auswahl eines geeigneten Fluoreszenzprotein für Ihr Experiment 9 wurde bewertet haben.
Promoter Auswahl
Die Auswahl eines Promotors wird weitgehend auf Zelltyp oder Phänotyp von Interesse ab. Für Organismen wie B. subtilis wurden einige Zelltyp-spezifische Reportergene in der Literatur etabliert. Für andere Bakterienstämme, werden untersucht Mikroarray-oder Transkriptions Daten erforderlich sein, Informationen darüber, welche Gene stark unter den Bedingungen hochreguliert werden bieten, wo Ihr Zelltyp von Interesse is manifestierten. Eine aktuelle Studie katalogisiert die Transkription von B. subtilis unter 104 verschiedenen Wachstumsbedingungen mit 10 Fliesen-Microarrays. Dieses Papier bietet umfassende Informationen darüber, welche Gene sind stark unter verschiedenen Bedingungen hochreguliert, die von unschätzbarem Wert für weniger gut charakterisierten Phänotypen ist.
Anstatt genaue Kartierung Promotorregionen für jedes Gen von Interesse, wir verwenden in der Regel einfach die Sequenz 200-500 bp stromaufwärts des Gens als Promotor. Der genaue Ablauf Länge ist abhängig von der genomischen Kontext: kürzere Regionen verwendet werden, wenn notwendig, um zu vermeiden, einschließlich der vor-kodierenden Regionen aus Nachbar offene Leserahmen.
Neutral Loci und Integration
Wie pflegen Sie die Reporter Konstrukt in Ihrem Bakterienstamm wird die letzte Frage bei der Gestaltung eines fluoreszierenden Transkriptions-Reporter-Stamm. In Bakterien sind die Gene von Interesse häufig beibehaltenauf Plasmiden mit antibiotischer Selektion. Jedoch kann es nicht möglich sein, während der Co-Kultur ohne Antibiotika töten die Umweltmikroben verwendet werden. Wenn Plasmide stabil in Ihrer Bakterienarten gehalten, kann es möglich sein, das Wachstum Ihres Bakterien, die ein Plasmid-borne Reporter in Gegenwart von Antibiotika, um Ihre Reporter für das Screening vorbereiten und dann zu beseitigen Antibiotika während der Co-Kultur selbst, in der Hoffnung, dass das Plasmid wird ausreichend aufrechterhalten werden, um Fluoreszenz zu ermöglichen. Allerdings, wenn Plasmide sind leicht in Ihre Bakterium verloren, oder werden unter Stressbedingungen verloren, wird dies nicht eine sinnvolle Option sein. In vielen Fällen ist die beste Lösung, um das Reporterkonstrukt auf dem bakteriellen Chromosom, das die stabile Aufrechterhaltung des Reporter auch in Abwesenheit von Selektion ermöglicht integrieren. Um für die Integration, um die normale Expression oder Regulation des Gens von Interesse nicht zu stören, empfehlen wir, in einer Eileiter Website integrieren auf dem Chromosom, dass CAn als handeln "neutralen Ort". In B. subtilis diese Integrationsstellen sind Gene, die - wenn sie mutiert sind - vermitteln einen Phänotyp in bestimmten Minimalmedien (so dass Integranten ohne Antibiotika-Selektion identifiziert werden kann), aber nicht Wachstum oder Sporenbildung Raten in Rich-Media verändern und umfassen solche Gene als amyE, lacA, thrC pyrD, gltA und sacA (Fördern der Fähigkeit, Stärke zu nutzen, β-Galactosidasen, Threonin, Uracil, Glutamat und Saccharose, jeweils) 11-13.
Während die Integration in diesen Genen haben viele Jahre zuverlässig in B verwendet wurde subtilis (insbesondere bei amyE und lacA) können ähnliche Wissen nicht für Gene, die in vielen anderen Bakterienarten zur Verfügung. Der Einsatz von Phagen-Bindungsstellen sind gute Alternativen für neutrale chromosomalen Integrationsstellen: Viele Spezies-spezifische 14-16, sowie allgemeine Integrationsstellen wie dem Tn7 Befestigungsstelle (att Tn7) habenidentifiziert worden, und in vielen Bakterienarten 17,18 für die Gen-Insertionen verwendet.
Umweltmikroben
Wir verwenden Boden als direkte Quelle von Umweltmikroben für unsere Co-Kultur-Bildschirm. Der Boden enthält eine große Vielfalt an Mikroorganismen, und viele dieser Organismen sind reich an natürlichen Produkten. Durch die Verwendung von flüssigen Suspensionen von Boden direkt auf Platten mit unseren Transkriptions fluoreszierenden Reporter-Stamm (ohne vorherige Isolierung von Bakterien aus dem Boden) gelegt, wir vereinfachen den experimentellen Ansatz. Der Boden kann entweder unmittelbar nach der Ernte verwendet werden oder bei -80 ° C für die zukünftige Verwendung eingefroren werden. Sofortige Anwendung hat den Vorteil, dass eine größere Vielfalt von Mikroben kann möglicherweise aufgewachsen werden, einschließlich derjenigen, die nicht überleben Einfrieren gut. Es hat den Nachteil, dass die Konzentration der Anbaubodenorganismen aus diesen Proben nicht bekannt ist, die Anzahl von Siebplatten, die verwendet werden müssen. Delayed Verwendung hat den Vorteil, dass die KBE / ml für jeden Boden Quelle kann im Voraus bestimmt werden, so dass eine optimierte Anzahl der Kolonien, die auf jeder Bildschirmplatte angebaut werden. Allerdings erfordert es, dass die Bodenorganismen überlebensfähig Gefrierpunkt.
Beachten Sie, dass die Diversifizierung der Induktor Pool wird (dh den Bodenquellen) untersucht zu sein scheint, bei der Identifizierung neuer Inter Wechselwirkungen als eingehende Screening auf demselben Boden effektiver: mehr phylogenetischen Vielfalt wurde in den in unserer Matrix-Bildschirm, wie Induktions identifizierten Hits beobachtet zusätzlichen Bodenquellen wurden anstatt Screening der gleichen Erde Quellen gründlicher (EA Shank und R. Kolter, Harvard Medical School, unveröffentlichte Ergebnisse) untersucht.
Überblick
Der Ansatz, den wir hier beschreiben, ist einfach in seiner technischen Anforderungen. Es beinhaltet: 1) Konstruktion eines fluoreszierenden Reporter-Transkriptions in B. subtilis oderandere Bakterienspezies von Interesse, 2) Identifizieren Bedingungen, unter denen der Reporter nicht aktiviert ist, 3) Herstellung von Aliquots dieser Reporter-Stamm und Organismen gescreent werden (in diesem Fall Erde, aber andere Quellen könnten stattdessen verwendet werden), 4) Mischen derselben zwei Sätze von Mikroben auf festen Medien, 5) Identifizierung und Isolierung von mutmaßlichen Induktion Organismen, und 6), die bestätigt, dass diese Organismen tatsächlich diesen Phänotyp in einem sekundären Bildschirm zu aktivieren. Einmal identifiziert, diese Organismen und deren Stoffwechsel uns mit chemischen Methoden, um bakterielle Verhalten modulieren, um bakteriellen Physiologie und mikrobielle Wechselwirkungen zu untersuchen und möglicherweise handeln als neue Gerüste für zukünftige therapeutische Verbindungen.
Protokoll
1. Wählen Sie ein Reporter Gene und Konstruieren Sie eine Leuchtstofftranskriptions Reporter
Für B. subtilis:
- Siehe den Artikel in JoVE Referenz 19 für ein Protokoll beschreibt die Konstruktion von fluoreszierenden Reporter-Transkriptions in Bacillus subtilis.
Für andere Bakterienarten:
- Identifizieren ein Gen, das bei der physiologischen Reaktion von Interesse hochreguliert wird. Dies kann auf bestehende Literatur oder Transkriptionsanalyse der Mikrobe unter bestimmten Bedingungen basieren.
- Konstruieren Sie einen fluoreszierenden Reporter-Transkriptions für dieses Gen als Proxy für die Änderung im Phänotyp zu handeln. Dieses Konstrukt sollte den Promotor des Gens hochreguliert fahren die Produktion eines geeigneten Fluoreszenzprotein (siehe Abbildung 1) umfassen.
- Integration dieses Konstrukts in einer neutralen Locus auf dem Chromosom. Dies gewährleistet, dass einheimische reglation des Gens von Interesse nicht gestört wird, und vermeidet die Notwendigkeit für das Plasmid Selektionsmechanismen (z. B. Antibiotika), das das Wachstum von Mikroben Umwelt stören.
2. Bestimmen Sie Cokultur AGB
Für B. subtilis P Tapa-YFP-Reporter:
- Verwenden 0.1x LB, Lennox (1 g Trypton, 0,5 g Hefeextrakt, 0,5 g NaCl pro Liter) Medium für diese Reporter, da B. subtilis Matrix-Produktion ist minimal auf Luria Broth 20. Dieses Medium ermöglicht B. subtilis-Kolonien auf Submillimeter aber beobachtbare Größe zu wachsen, während gleichzeitig diverse Taxa aus dem Boden zu 5 wachsen.
- Fügen Sie 100 mM MOPS-Puffer, um mögliche pH-Änderungen zu minimieren.
Für andere Bakterienarten:
- Verwenden veröffentlicht Transkriptions Daten oder empirisch testen verschiedene Kulturbedingungen eine, wo die Mikrobe wächst zu identifizieren, sondern die activation der fluoreszierenden Reporter vernachlässigbar ist (damit dessen Aktivierung detektiert werden, wenn das Reporter-Stamm wird in Kokultur mit Induktion Mikroben gezüchtet.)
- Verwenden Sie ein Medium mit geringer Nährstoffgehalt (bezogen auf die traditionell reich mikrobiologischen Medien) beim Screening Umwelt Mikroben aus nährstoffarmen Umgebungen (wie der Boden), da viele oligotrophen Bakterien nicht wachsen, wenn mit hoher Nährstoffbedingungen 21 vorgestellt. Eine niedrige Nährmedium reduziert auch Koloniegröße, die Erhöhung der Durchsatzleistung der Bildschirm.
- Wählen Sie ein Medium mit geringer Hintergrundfluoreszenz und gute optische Klarheit.
- Optimieren Wachstumstemperatur, damit sowohl der Reporter-Stamm-und Umwelt Mikroben, gleichzeitig wachsen.
- Betrachten wir die Zugabe eines Puffermittels. Die Verwendung des Puffers in den Platten wird die Möglichkeit der Erfassung pH-vermittelte Veränderungen in der Physiologie 22 zu verringern, es sei denn, solche Wechselwirkungen von Interesse.
3. Bereiten Reporter Aliquots
Für B. subtilis P Tapa-YFP-Reporter:
- Streak Reporterstamm von -80 ° C eingefroren Lager auf eine frische LB-Platte mit einem sterilen Zahnstocher oder Applikator-Stick.
- Wachsen über Nacht bei 30 ° C
- Führen serielle Verdünnungen in Flüssigkultur Hintergrundfluoreszenz, die sich aus dem Wachstum auf einem festen Medium zu minimieren:
- Impfen eine 5 ml Flüssigkultur von LB und wachsen mit bei 37 ° C schüttelnd
- Wenn die Kultur eine OD 600 ~ 0,6 erreicht, in 5 ml frischem LB verdünnt auf eine OD 600 von 0,02.
- Wachsen bei 37 ° C unter Schütteln bis wieder Kulturen erreicht OD 600 ~ 0,6.
- Wiederholen serielle Verdünnungen Wachstum insgesamt 3x.
- Lassen endgültige Verdünnungs Kultur wachsen, um OD 600 ~ 0,4.
- In Glycerin zu 15-20%.
- Aliquot 50-200 ul in 0,5 ml Mikrozentrifugenröhrchen übertragen und bei -80 ° C.
- Make gleichwertige Teilmengen für den nicht-fluoreszierenden Elternstamm des Reporters (wird sie während sekundäre Screening erforderlich sein).
Für andere Bakterienarten:
- Verwenden Zellen, die eine geringe Hintergrundfluoreszenz (dh unter Bedingungen, wo es wenig Expression vom Promotor im Reporter gewöhnt).
- Stellen und frieren Aliquots, die bekannte KBE / ml (koloniebildende Einheiten pro ml) der Reporterstamm, so dass eine entsprechende Anzahl von Kolonien können auf jedem Bildschirm Kokultur Platte angebaut werden (siehe Abschnitt oben für Details).
- Gleichwertige Teilmengen für den nicht-fluoreszierenden Elternstamm (sie werden während der sekundäre Screening erforderlich sein).
4. Erhalten Bodenproben
- Sammeln Boden in sterile konische Röhrchen oder sterile Beutel mit einem Spatel, Verwerfen der oberen 0,5 cm von freigelegten Oberfläche Boden.
- Hinzufügen steriler Kochsalzlösung (0,85% NaCl) in einem Verhältnis von 10 ml pro 1 gvon Boden, um eine Bodenschlamm zu machen.
- Wählen Sie eine Methode, um Bakterien aus Bodenpartikel zu entfernen: a) sofortige Verwendung von frischen Probe oder b) verzögert Verwendung nach dem Einfrieren der Probe.
- Für den sofortigen Einsatz, Vortex-Slurry für 1 min.
- Für die verzögerte Einsatz, mischen Bodenschlamm in den Mixer für drei Zyklen 1 min, indem Sie den Mixbecher auf Eis für 1 min ruht zwischen Mischzyklen.
- Lassen Bodenschlamm für ~ 1 min absetzen.
- Bewegen obere wässrige Schicht in ein frisches Röhrchen.
- Hinzufügen Glycerin bis zu einer Endkonzentration von 15-20%.
- Aliquot 50-200 ul in 0,5 ml Mikrozentrifugenröhrchen übertragen und bei -80 ° C.
5. Bestimmen Sie KBE / ml von Frozen Reporter und Boden Aliquots
- Tauen Sie eine gefrorene Boden und Reporter aliquoten. Bodenmikroben auf Eis aufgetaut werden. Weil B. subtilis Lyse bei 4 ° C, sollten diese Aliquots schnell bei Raumtemperatur aufgetaut werden, um die Zeit bei niedriger Temperatur verbracht zu minimieren.
- Machen Sie zwei Wiederholungs serial Verdünnungen (bis 10 -8) in 0,1 LB oder andere isotonische Puffer.
- Platte 5 ul Flecken jeder seriellen Verdünnung auf Agar-Platten mit dem gleichen Medium, das für die Co-Kultur-Screening verwendet werden.
- Wachsen bei RT (oder die Temperatur, die mit Hilfe wird für das Screening).
- Am nächsten Tag, zählen die Anzahl der Kolonien in jedem Ort und berechnen KBE / ml von jeder der gefrorenen Aliquots.
6. Bestätigen Aliquot Konzentrationen für Spread-Screen-Platten
- Tauen Sie eine gefrorene aliquoten wie in Schritt 5.1.
- Verdünnt auf 1 × 10 5, 2,5 × 10 5, 5 x 10 5, 1 x 10 6 und 2,5 x 10 7 KBE / ml.
- In 50 ul Stelle jeder Verdünnung bis zur Mitte der einzelnen Platten. Diese sollten Platten mit der berechneten optimalen 25.000 Kolonien pro Platte sowie zwei ergeben - und 5 - fach mehr und weniger, so dass die beste tatsächliche Verdünnungs bestimmt werden.
- In etwa 20 sterile Glas (3 mm) Perlen durchleichtes Klopfen sie auf der Platte. Perlen bieten eine gleichmäßigere Verteilung der Kolonien über die Platte als eine gebogene Glastreuer tut.
- Verbreiten Zellen, indem sie Platten auf dem Labortisch und sie hin und her schütteln, Drehen auf, wie Sie arbeiten, bis die Flüssigkeit aufgesogen ist. Machen Sie nicht weiter erschüttern die Perlen einmal die Platte trocken ist, sonst wird es anfangen, um die Bakterien zu töten.
- Flip Platten über und entsorgen Perlen in Abfallbecherglas Ethanol enthält.
- Lassen Platten wachsen für die Zeit Ihres Assay (z. B. 24 h) bei der richtigen Temperatur für Ihr Assay (z. B. 24 ° C / RT).
- Mit einem Sezieren Stereoskop, zählen die Anzahl der Kolonien in zwei oder mehr Sichtfelder.
- Berechnen der Anzahl von Kolonien pro Flächen, und bestimmen, wie viele Kolonien auf jeder Platte.
- Passen Zukunft Verdünnungen wie nötig. Die tatsächliche Anzahl der Kolonien ist nicht so wichtig wie die gleiche Anzahl auf jedem Bildschirm Platte vergleichbar.
7.. Bereiten Cokultur Platten
- Tauwetter Reporter Aliquot (und Boden, wenn Aliquot eingefroren), wie in Schritt 5.1.
- Verdünnen Reporter (Konzentrationen in Abschnitt 6 optimiert) in 0,1 LB oder andere nährstoffarmen, isotonischen Lösung.
- Für gefrorenen Boden: Verdünnen der Konzentration in Abschnitt 6 in 0,1 LB oder andere nährstoffarmen, isotonischen Lösung optimiert.
- Für frische Erde: Stellen Verdünnungen von frischem Boden Gülle, basierend auf der Vorhersage, dass die KBE / ml der Bodenschlamm konnte von 10 -4 bis -9 10 KBE / ml liegen.
- Punkt 50 ul von Boden-und Reporter-Verdünnungen in die Mitte der Kokultur Bildschirmplatte. Auch Bodenplatte allein und Reporter allein als Kontrollen.
- Verbreiten Sie mit Glasperlen, wie in Schritt 6,4-6,6 beschrieben.
- Wenn es bekannte Wachstumsbedingungen, die Ihre fluoreszierenden Reporter, streifen aktivieren oder platten Ihre Reporter unter diesen Bedingungen wachsen, um als eine positive Kontrolle beim Screening zu verwenden.
- Inkubation bei 24 ° C für 24 bis 28 Stunden (oder entsprechend Ihrer Reporter / Test)
8. Screen-Co-Kultur-Platten für Fluoreszenz
- Mit Hellfeldbeleuchtung, konzentrieren Sie Ihre Stereoskop, so dass die Kolonien sind scharf.
- Schauen Sie sich die Boden-Platten nur um festzustellen, ob Ihre Bodenprobe hat Kolonien autofluoreszierenden. Wenn ja, kann dieser Boden in einer hohen Rate von False Positives (mit einem gleichzeitigen Anstieg der sekundären Screening) führen.
- Verwenden Sie ein hohes Verhältnis von Reporter: Boden in Ihrem Co-Kultur-Platten, die Chance, dass ein Induktor wird durch mehrere Reporter Kolonien umgeben werden, was die Chance, dass sie als falsch-positive nachgewiesen werden (Abbildung 2) zu erhöhen.
- Verwenden Sie einen anderen fluoreszierende Protein (eine, die in einem anderen Kanal sendet).
- Bestimmen Sie den Test Timing. Für die meisten neuen Reportern, den Zeitpunkt der potentiellen Induktion ist bekannt und muß daher empirisch bestimmt werden.
- Beginnen Screening auf Fluoreszenz sobald Kolonien deutlich sichtbar mit dem Sezieren Stereoskop (~ Vergrßerung 30X) und weiterhin in regelmäßigen Abständen die Prüfung der Platten, bis das Wachstum aufgehört hat, und / oder die Hintergrundfluoreszenz zu hoch.
- Sobald das Zeitfenster der möglichen Induktion für eine bestimmte Reporter festgestellt, sollte es ähnlich für alle Co-Kultur-Platten, dass Reporter, die Vereinfachung der Überwachung der Rasterplatten. Für die B. subtilis P Tapa-YFP-Reporter, ist die geeignete Zeit, um die Platten zu untersuchen zwischen 24-28 Stunden nach dem Ausstreichen der Zellen, für den B. subtilis P SSPB YFP-Reporter, ist es zwischen 26 bis 32 Stunden des Wachstums.
- Maximieren Sie Ihre Fluoreszenz-Empfindlichkeit:
- Stellen Sie sicher, Fluoreszenz-Binokular ist in einem dunklen Raum oder durch lichtundurchlässige Vorhänge umgeben. Induktion wird wahrscheinlich weniger intensiv als einer konstitutiv produziert FP und erfordert größere Sensivität zu erkennen.
- Nehmen Sie sich Zeit für die Fluoreszenzlampe zu stabilisieren und die Augen an die Dunkelheit, bevor Sie versuchen, um die Fluoreszenz von Ihrem Co-Kultur-Platten (mindestens 1-2 min) zu erkennen einzustellen.
- Mit einer positiven Kontrolle (falls Sie eine haben), stellen Sie sicher, dass Sie mit der Vergrößerung können Sie Fluoreszenz zu erkennen. Die Vergrößerung ist in der Regel am besten, wenn Ihr Blickfeld ist ungefähr 30-50x typische Koloniedurchmesser (200-400X).
- Schalten Sie das helle Licht, und öffnen Sie den Auslöser für Ihre Fluoreszenz.
- Nachdem deine Augen an die Dunkelheit gewöhnt, langsam bewegen die Platte hin und her über Ihr Blickfeld, auf der Suche nach hellen Flecken.
- Starten Sie von der Oberseite der Platte und mit einem Zick-Zack-Muster, um die Platte von Seite zu Seite zu bewegen, wie Sie auf der Unterseite der Platte zu bewegen.
- Praxis Bewegen der Platte im Hell, um ein Gefühl dafür, wie langsam zu bewegen, um die Platte zu bekommen, und um sicher zu sein, dass Sie für die gesamte sur werdenGesichtsbereich.
- Bewegen Sie die Platte langsam genug, dass die Kolonien nicht verschwommen. Es ist besser, die Oberfläche nicht verpassen Bereichen überabzutasten.
- Nach einer vollen Schwung, drehen Sie die Platte um 90 ° und wiederholen. Menschliche Augen sind erstaunlich gut bei der Erkennung auch schwache Fluoreszenz durch diese Methode.
- Wenn Sie Fluoreszenz zu erkennen, die die Platte zu stoppen und gehen Sie zurück und finden Sie die Leuchtstoffbereich.
- Drehen des Hell langsam, zu bestimmen, ob die Fluoreszenz mit einer Bakterienkolonie (und nicht autofluoreszenten Boden Detritus oder Medienkomponenten) zugeordnet ist. Wenn dem so ist, werden die nicht-fluoreszierenden Kolonien nahe der Fluoreszenz Kolonie sind mutmaßlichen Induktion Organismen.
9. Isolieren Mutmaßliche Inducing Organismen
- Sobald induziert (Leuchtstoff)-Kolonien wurden identifiziert, isolieren diese sekretierenden Kolonien induzierenden Verbindungen.
- Wenn eine ausreichend hohe Konzentration von Reporter-Kolonien auf der Platte wachsenden(> 0,5:1 Reporter: Boden KBE), werden die vermeintlichen Induktions Kolonien von mehreren fluoreszierenden Kolonien umgeben sein (wieder, siehe Abbildung 2).
- In Fällen, in denen die Komplexität der Kokultur Wachstum macht es eindeutig die Kolonie der Induktor, isolieren mehrere potenzielle Induktions Kolonien für die anschließende Prüfung in der sekundären Bildschirm.
- Lokalisieren Sie die Kolonien in der Mitte des Sichtfeldes holen möchten.
- Wenn sie nahe an den Rand der Platte sind, drehen Sie die Platte, so dass die Lippe der Platte weg von Ihrer dominanten Hand (dh, wenn Sie Rechtshänder sind, legen Sie die Lippe der Platte auf der linken Seite). Dies ermöglicht Ihnen, die Kolonien in einem niedrigen Winkel, die die Genauigkeit der Kommissionierung verbessert nähern.
- Legen Sie frische Platten zu Streifen die mutmaßlichen Induktion Organismen auf, um in der Nähe, als auch als Abfallbecherglas verwendet Tipps in verwerfen.
- Verlassen der Fluoreszenzlampe auf, langsam das helle Lichtauf, so dass Sie in der Lage sein zu erkennen (von Form und Position), die Leuchtstoff Kolonie und die umliegenden mutmaßlichen Induktion Organismen. Sie müssen möglicherweise mit dem Licht ein paar mal hin und her gehen zu können, um die Kolonien Sie zu holen, wenn es keine Fluoreszenz und nur helles Licht wollen zu identifizieren.
- Verwenden Sie einen Glasstab (200 mm lang x 5 mm Durchmesser) zu holen eine sterile, rund 200 ul Gel-loading Spitze und halten Sie ihn wie einen Bleistift. Dies ist die Kommissionierung Werkzeug, das Sie verwenden, um einzelne Kolonien zu isolieren.
- Gönnen Sie Ihren äußeren Handfläche gegen die Bühne, um sie auf der Arbeitsfläche zu stabilisieren. Zeigen Sie mit der anderen Hand auf der inneren Seite der Daumen der Hand zu stabilisieren Ihre Auswahlwerkzeug.
- Wenn nötig, wieder Flip zwischen Fluoreszenz-und Hellblick, um die Kolonien zu holen Sie wollen zu identifizieren.
- Halten Sie die Pipettenspitze über der Plattenoberfläche, verschieben Sie sie in Ihr Blickfeld und zentrieren sie über der Kolonie Sie gerne abholen. Die Spitze wird unscharf.
- Mit der Außenkante der Hand (die auf den Mikroskoptisch oder Arbeitsfläche ruht), drehen Sie die Pipettenspitze langsam in die Kolonie abgeholt werden. Berühren Sie es sehr leicht, zu versuchen, zu minimieren, wie viele anderen Kolonien die Spitze Kontakten.
- Ohne Drehen des Auswahlwerkzeug, Streifen auf einen Abschnitt von einem frischen Teller. Verbreiten Sie mit einem weichen Note zu vermeiden das Fugenhobeln Agar. Da die Anzahl der Zellen, die durch dieses Verfahren überführt ist ziemlich klein (die Kolonien sind viel kleiner als normalerweise betätigt), wird eine einzelne kontinuierliche Streifen in isolierte Kolonien führen.
- Wiederholen Sie diesen Vorgang mit anderen mutmaßlichen Induktionsorganismen.
- Platten bei 24 ° C (oder die Temperatur des Assay).
10. Streak Mutmaßliche Inducing Organismen auf isolierte Einzelkolonien erhalten
- Mit einem Stereoskop, bestimmen - basierend auf Koloniestruktur oder Morphologie - ob es verschiedene in jedem Ihrer vermeintlichen abgeholt ind enthalten Kolonietypenucing Organismen.
- Schauen Sie sich die Kolonien mit einer Bühne, wo man die Kolonien sowohl von oben als auch von unten zu beleuchten, um mögliche Kolonie Unterschiede zu erkennen.
- Restreak jedes andere Morphotyp auf eine frische Platte und Inkubation bis gewachsen.
- Restreak ein mehr Zeit und wachsen lassen. Wenn verschiedene Morphotypen bestehen bleiben, weiterhin auf Reinheit restreak.
11. Retest Mutmaßliche Inducing Organismen in Sekundärbildschirm
- Retest alle vermeintlichen Induktion Organismen in einem sekundären Bildschirm, um festzustellen, welche die Aktivierung des Transkriptions fluoreszierenden Reporter werden. Der sekundäre Bildschirm besteht aus einem Rasen von der fluoreszierenden Mikrokolonien Transkriptions-Reporter-Stamm, zusammen mit Steuerplatten, auf die vermeintlichen Induktion Organismen gepatcht oder gefleckt.
- Ein, die eine Mikrokolonie Rasen des Transkriptions fluoreszierenden Reporterstamm (in der gleichen Konzentration ein: drei identische Platten Sets wurde in Co-Kultur-Screening verwendet), eine mit einer Mikrokolonie Rasen des Wildtyp-Elternstamm ohne Fluoreszenz-Reporter, und eine, die keinen Rasen.
- Markieren Sie die Spitze der Rückseite jeder Platte, die Platte Ausrichtung zu bestimmen.
- Hinzufügen Positionsmarkierungen für die Patches / Flecken, bis zu zehn mutmaßlichen induzierende Organismen können auf jeden Satz von Platten (3) getestet werden.
- Tauwetter Rasen Aliquots (nicht-fluoreszierenden Reporter und Elternstamm), wie in Schritt 5.1.
- Verbreiten 50 ul einer 5 x 10 5 cfu / ml Verdünnung auf Rasen Platten (oder anderen optimierten Verdünnungen) mit sterilen Perlen, wie in Schritt 6.4.
- Lassen Platten trocken.
- Patch-oder Punkt mutmaßlichen Induktion Organismen:
- Patching wählen, wenn ein schneller und einfacher Ansatz ist erwünscht, und es ist akzeptabel, eine weniger genaue Anzahl der Zellen abgelagert haben. Um zu patchen:
- Tippen Sie auf einen sterilen Zahnstocher in die Kolonie zu Test - nicht abholen alle Zellen.
- Patch (einen kleinen Streifen) auf den leeren Teller.
- Wiederholen Patch mit frischen Zahnstocher auf Reporter Platte.
- Wiederholen Patch mit frischen Zahnstocher auf Steuerplatte.
- Wählen Spotting, wenn eine quantitative und reproduzierbare Vorgehensweise gewünscht wird. Spotting kann die Anzahl der abgelegten Zellen normalisiert werden (siehe Bezugs 5 für nähere Details), und erlaubt die relative Wirksamkeit der verschiedenen induzierende Organismen, die verglichen werden. Zu erkennen:
- Resuspendieren putative induzieren Organismen in 1 ml flüssigem Medium in einem sterilen Plastik Küvette.
- Nehmen Sie die OD 600 der Resuspensionen.
- Mit Hilfe der Formel X = 250 ÷ (OD 600 bis 0,5), fügen Sie die Lautstärke für jeden X Aufwirbelung zu 500 ul der flüssigen Medium, um eine Lösung mit einer OD 600 von 0,5 erhalten. Diese Methode vereinfacht die erforderlichen Schritte bei der Durchführung Pipettieren mehrere Verdünnungen zu normalisieren OD, weil Sie das gleiche Volumen (50 verwenden0 ul) für alle Verdünnungsmittel.
- Spot 1 ul jeder OD normierten Resuspension zu jeder der drei Platten.
- Patching wählen, wenn ein schneller und einfacher Ansatz ist erwünscht, und es ist akzeptabel, eine weniger genaue Anzahl der Zellen abgelagert haben. Um zu patchen:
- Lassen Sie wachsen bei 24 ° C für 24-28 h (oder entsprechend Ihrer Reporter / Test).
- Verwenden Sie die Leuchtstoff Binokular zu induzieren mutmaßlichen Organismen, die Ihre fluoreszierenden Reporter-Stamm aber nicht Ihre elterliche Kontrollstamm aktivieren identifizieren. Diese Isolate sind Ihre positive Treffer - die Umwelt Mikroben, die Verbindungen, die Ihre Phänotyp von Interesse zu induzieren absondern.
Ergebnisse
Dieser Bildschirm wurde verwendet, um Bodenorganismen zu identifizieren sezer Verbindungen, die die Physiologie von B. ändern subtilis. Die hier beschriebenen Ergebnisse konzentrieren sich auf die Matrix-produzierenden Zelltyp von B. subtilis, die das Protein und Exopolysaccharid, die für robuste Biofilmbildung in diesem Bakterium benötigt werden produziert. Wir haben uns für den Promotor des Tapa-sipW Tasa-Operon für unsere fluoreszierenden Reporter-Konstrukt (P Tapa-YFP). Dieses Operon kodiert das Protein strukturelle Komponente der Matrix und während der Biofilmmatrixproduktion 23 hochreguliert. Unsere Matrix Reporter (Fig. 1) wurde konstruiert, wie zuvor beschrieben 19.
Frühere Arbeiten haben gezeigt, dass B. subtilis produziert Matrix in Reaktion auf die selbst produzierten Quorum-sensing-ähnliches Molekül Surfactin sowie gereinigt Metaboliten durch andere Bodenbakterien 20 erzeugt. Wir waren in den Ausbau derse Studien, die untersuchen, welche breiter Bodenmikroben machen Metaboliten induzieren kann Matrixproduktion in B. subtilis. Wir entschieden, verdünnten LB für Wachstum zu verwenden, da dieses Medium bereits bekannt, schlechte Matrixproduktion 20 führen, die uns mit einer Wachstumsbedingung, wo unsere Reporter-Stamm war nicht fluoreszierend. Dann optimiert die Anzahl der Kolonien für das Screening unter diesen Wachstumsbedingungen geeignet. Um die einzelnen Bildschirmplatte zu optimieren, ist es notwendig, festzustellen, wie viele Kolonien wachsen aus den gefrorenen Boden und Reporter-Aliquots und was die entsprechenden Konzentration von Kolonien und Nährstoffbedingungen. Im Idealfall wollen wir jedes Kokultur Platte, eine entsprechende Anzahl von Boden-und Reporter-Kolonien (dh ein 1:1-Verhältnis von Reporter: Boden) enthalten und eng angeordnet werden, einzelne Kolonien. Dieses hohe Verhältnis von Reporter Kolonien erhöht die Wahrscheinlichkeit, dass ein Induktor wird mehrere umliegende Reporter Kolonien zu aktivieren. Nachdem mehrere acviert Induktor Kolonien eine mutmaßliche Induktor Kolonie steigt das Vertrauen in die Ermittlung der tatsächlichen Induktions Organismus (Abbildung 2) umgibt. Der Nährstoffgehalt steuert das Ausmaß des Wachstums / Koloniebildung während der Verdünnung des Inokulums ermittelt, ob die resultierenden Kolonien werden in geeigneter Weise dispergiert. Auf einem Standard-Durchmesser von 10 cm Petrischale mit niedrigen Nährmedium, fanden wir, dass insgesamt etwa 25.000 Kolonien pro Platte (50 ul einer 5 x 10 5 KBE / ml Verdünnung) lieferte die beste Trennung von B. subtilis-Kolonien auf LB 0,1 x MOPS-Medium (Abbildung 4).
Obwohl die berechnete cfu / ml der Verdünnungen stellt eine ungefähre Konzentration von Bakterien in den Aliquots, ist es notwendig sicherzustellen, dass die Konzentration der resultierenden Kolonien geeignet ist, wenn eine gesamte Platte mit Zellen verteilt. Die berechnete KBE / Platte und tatsächlichen KBE / Platte nicht immer identisch sind (Abbildung 4 ). Plating Kolonie Rasen des äquivalenten Konzentrationen ist wichtig, damit unterschiedliche Reporterstämmen verglichen werden (sonst können Unterschiede in der Nährstoffverfügbarkeit ihrer physiologischen Zustand zu ändern und nicht mit den Ergebnissen).
Nach der Vorbereitung Aliquots der Reporter und Boden, vermischt wir sie auf Kokultur Bildschirm Platten und untersuchte sie für die Fluoreszenz mit einem Stereoskop (Abbildung 5). Wir vergoldet auch Steuerelemente, die entweder nur mit Erde oder der B. geimpft wurden subtilis P Tapa-YFP Reporterstamm. B. subtilis produziert Biofilmmatrix (Fluoreszenz) in Reaktion auf zahlreiche Mikroben aus dem Boden, wie durch die fluoreszierende Kolonien in der Kokultur Bild 5 ersichtlich. Für die Böden untersuchten wir, wir hatten eine hohe Trefferquote für die P Tapa-YFP Reporter. Wie in Referenz 5, zwischen 12-67% der Isolate (aus sechs verschiedenen Bodenproben) beschrieben hatte die Fähigkeit, induce Fluoreszenz in der P Tapa-YFP Reporterstamm. Dies steht im Gegensatz zu unserem unveröffentlichte Ergebnisse aus analogen Bildschirmen mit der Sporenbildung (P SSPB-YFP) und Kompetenz (P ComG-YFP) Reportern. Nach umfangreichen Screening (> 200.000 Kolonien für jeden Reporter) wurden nur zwei Organismen festgestellt, dass die Sporenbildung zu induzieren, während keiner identifiziert wurden, dass die Zuständigkeit zu induzieren. Somit sind die Trefferraten für verschiedene Zelltypen sind sehr variabel und kann schwierig sein, vorherzusagen.
Wir haben dann abgeholt einzelnen vermeintlichen Induktions Kolonien. Die Kolonien auf den Bildschirm Kokultur Platten sind recht klein, auf der Nieder Nährmedium empfehlen wir (Submillimeter-Durchmesser). Dennoch ist es möglich, genau zu nehmen und zu isolieren, sehr kleine Kolonien von Hand (Abbildung 6) aus einer komplizierten Kokultur Bildschirmplatte. Die manuelle Methode, die wir verwenden, ist einfach und erfordert weder Spezialwerkzeuge noch Abflammen. Diese vermeintliche Induktor Kolonien werden dann restruck zu Isolation. Da die Co-Kultur-Platten werden mit Kolonien überfüllt, ist es nicht ungewöhnlich - auch bei sehr vorsichtig Kommissioniertechnik - mehr als ein Organismus wächst aus jedem vermeintlichen Induktor Probe haben. Sorgfältige Prüfung sollte Isolierung von morphologisch unterschiedliche Kolonien zu ermöglichen. Alle mutmaßlichen Organismen induziert werden dann in einem zweiten Bildschirm getestet. Positive und negative Ergebnisse sowohl von der Patch-und Spot-Verfahren sind in Fig. 3 gezeigt. In Anbetracht ihrer dichten Wachstum, unsere Fähigkeit, physisch sammeln Induktion Kolonien von den Co-Kultur-Platten war ziemlich gut, mit etwa 50% der in unserem zweiten Bildschirm wahr Positive Kolonien untersucht. Weitere Ergebnisse von diesem Bildschirm sowie Follow-up-Arbeit, die sich aus es waren zuvor 5 beschrieben.
/ Ftp_upload/50863/50863fig1.jpg "/>
Fig. 1 ist. Fluoreszierende Transkriptionsreporterkonstrukt. Das blaue Oval entspricht einer Bakterienzelle und die gestrichelte Linie stellt seinem Chromosom. Dieses Beispiel zeigt eine Leuchtstoff Transkriptions-Reporter für die Produktion von Matrix. Die native Locus intakt bleibt (P Matrix-Matrix, wobei "P" und der Pfeil die Promotor-Region), während das Reporterkonstrukt (P-Matrix-YFP) ist an anderer Stelle in das Chromosom in einer neutralen Stelle eingefügt.
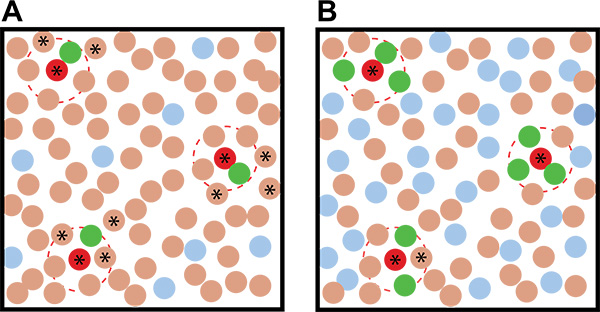
2. Idealisierte Beispiele für Co-Kultur Screening-Ergebnisse mit unterschiedlichen Verhältnissen des Reporters: Umwelt Mikroben. A) Die Verwendung eines Low-Reporter: Umwelt Mikrobe Verhältnis führt zu mehr Unklarheit bei der Identifizierung von mutmaßlichen Induktion Organismen, als wenn B) Eine hohe Reporter: Umwelt Mikrobe Verhältnis verwendet. Die braunen Kreise stellen Bodenorganismen, die roten Kreise stellen Induktion Bodenorganismen, die blauen Kreise stellen nicht-induzierten Reporter Kolonien, und die grünen Kolonien repräsentieren induzierten Reporter Kolonien. Die gestrichelten roten Linien zeigen den Aktionsradius des Induktions Metaboliten. Sterne zeigen, nicht fluoreszierenden Kolonien, die - auf ihrer Nähe zu den fluoreszierenden Kolonien Basis - sind mutmaßliche Induktion Organismen und sollte abgeholt und in den sekundären Bildschirm erneut getestet werden.
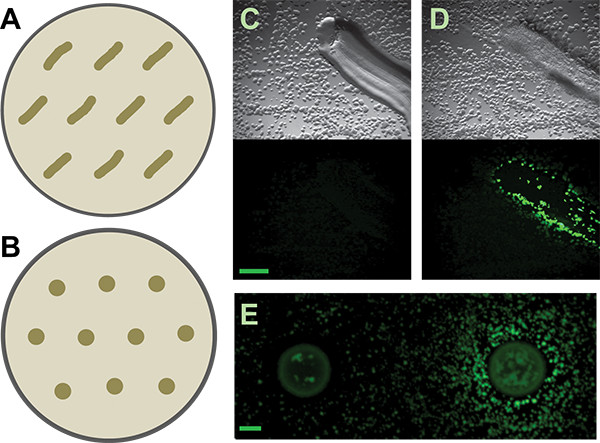
3. Sekundäre Bildschirm. A und B) Schematische Darstellung, wie verteilen gepatcht oder gefleckte isoliert auf sekundären Bildschirm Platten, jeweils für die B. subtilis Matrix Reporter. Weitere großzügige Abstand kann für andere Reporter oder Induktion Isolate, dep erforderlichendend mit dem Diffusionsvermögen der aktiven Metaboliten. C und D) Repräsentative Ergebnisse aus Patched Bodenisolaten, die negativ bzw. positiv, zur Induktion der B sind subtilis P Tapa-YFP-Reporter. Top-Panels sind die Hell Bilder; unteren Platten sind die Fluoreszenzbilder. Maßstab ist 1 mm. E) Negative und positive Ergebnisse aus spotted Bodenisolaten für die gleiche Reporter. Maßstab ist 2 mm.

4. Bestimmung der Mikrokolonie-Konzentration. Die Verteilung und Größe der Kolonien sowohl auf Nährstoff-und Zellkonzentrationen abhängen. A) Unterschiede im Wachstum von B. subtilis auf 0,01 x LB (obere Reihe) gegen 0.08x LB (untere Reihe). Cells auf 0,01 x LB nicht in Mikrokolonien bilden, während tSchlauch auf 0.08x LB zu tun. (Beachten Sie, dass für unsere Bildschirme wir den Nährstoffgehalt erhöhte sich leicht von den hier gezeigten:. Von 0.08x auf 0,1 x LB LB) Diese Bilder sind von 1 ul-Spots von sequentiellen 01.05 Verdünnungen an bekannten KBE / ml. Extrapoliert man diesen Konzentrationen ähnliche Verteilungen der Kolonien über eine 10 cm Petrischale bekommen würde Beschichtung (von links) erfordern: 3.200.000; 640.000 und 128.000 KBE pro Platte insgesamt. Allerdings, Schmierblutungen zu einer ungleichmäßigen Verteilung der Zellen (die an den Kanten Ort konzentriert sind) im Vergleich zu verbreiten Zellen über die gesamte Platte. Somit kann, sobald eine Nährstoffkonzentration gewählt wird, ist es wichtig zu Platten mit einer Vielzahl von Konzentrationen verteilt untersuchen. Maßstabsbalken ist 0,1 mm B) Diese Tafeln zeigen die Ergebnisse der Verbreitung (von links) 50.000;. 25.000, und 5000 insgesamt KBE pro Platte auf 0.08x LB-Platten. Aus diesen Bildern, 25.000 wählten wir als unser Ziel von KBE / Platte. Maßstab beträgt 0,1 mm.

5. Cokultur von B. subtilis P Tapa-YFP mit Bodenorganismen gemischt. Lagerung von Hellfeld-und Fluoreszenzbild von einer Co-Kultur-Bildschirm Schild mit B. subtilis P Tapa-YFP Matrix Reporter mit Bodenorganismen gemischt. Pfeilspitze zeigt mutmaßlichen Induktor durch Fluoreszenz-Reporter Mikrokolonien umgeben. Maßstab ist 1 mm.
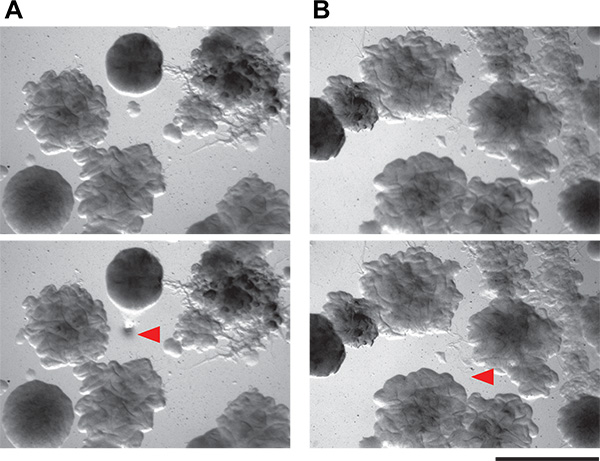
6. Demonstration der Machbarkeit der Isolierung von winzigen Bakterienkolonien von Co-Kultur-Platten. A und B) Diese Platten zeigen zwei Sichtfelder der Agarplatten mit komplexen Mikrobengemeinschaften aus dem Boden. Kolonien so klein wie 0,1 mm können mit dem beschriebenen Verfahren isoliert werden Kommissionierunghier. Top-Panels sind das Blickfeld vor Kolonie Kommissionierung und Bodenplatte sind die gleichen Sichtfelder nach Kolonie Kommissionierung. Rote Pfeilspitzen zeigen, wo Zellen entfernt wurden.
Diskussion
Eine der inhärenten Beschränkungen des Protokolls ist, dass es auf der Kultivierbarkeit von mikrobiellen Organismen beruht. Wie gut dokumentiert 24, die meisten mikrobiellen Lebens auf dem Planeten kann (noch) nicht unter den Kulturbedingungen bis heute erforscht angebaut werden. So wird eine große Anzahl von Interaktionen zwischen Mikrobenarten, die in natürlicher Umgebung auftreten, unerkannt mit diesem Ansatz. Da unser Wunsch ist es, die Existenz solcher Wechselwirkungen nicht nur zu identifizieren, dann aber auch die Mechanismen und Moleküle bei der Vermittlung von ihnen beteiligt zu studieren, ist die Fähigkeit, diese Mikroben pflegen eine Notwendigkeit. Selbst innerhalb der Anbauarten, hat sich dieser Bereich wenig erforscht worden, so dass die hier beschriebene Ansatz einen wertvollen Beitrag als Methode zur chemisch vermittelte Wechselwirkungen zwischen Mikroben zu identifizieren. Zusätzlich kann, obwohl dieses Protokoll wurde optimiert, um für die Matrix-Induktion von Bacillus subtilis zu screenen, kann theoretisch auf eine angewandt werdeny Transkriptions fluoreszierenden Reporter in anderen Bakterienarten.
Ein weiterer Zusammenhang Einschränkung dieses Ansatzes ist, dass dieser Bildschirm (per Definition) erfordert Co-Kultur. In natürlichen Umgebungen kann Mikroben mit unterschiedlichen Wachstumsraten noch in räumlicher Nähe nebeneinander unter Ausnutzung verschiedener Umwelt Nischen. Solche mikrobiellen Wechselwirkungen würden unentdeckt von unserem Co-Kultur-Bildschirm, aber, die nur das Wachstum der Mikroben Umwelt mit Nährstoffbedarf und Wachstum ähnlich denen der Reporter-Spezies Raten ermöglicht. Modifikationen, die das Wachstum der potentiellen induzierende Organismen aus dem Wachstum des Reporterstamm trennen wäre sicherlich möglich sind. Wir erwarten, dass die Hyphen-Wachstum von Pilzen - in Boden gemeinsam - vielleicht Schwierigkeiten in der Co-Kultur-Bildschirm führen. Während der kurzen Zeitskala von unserem Bildschirm mit B. subtilis bedeutete, daß einige Pilze nachgewiesen wurden, indem Antipilzverbindungen zum Wachstumsmedium konnte minimize dieses Anliegen.
Die Fähigkeit, einen entsprechenden Phänotyp und Gen für die fluoreszierenden Reporter-Konstrukt wählen sollte nicht schwierig sein, angesichts der Fülle von Sequenzierungs-und Transkriptions-Daten entweder bereits vorhanden oder leicht erhältlich für viele Bakterienarten. Besteht eine Schwierigkeit mit dem hier beschriebenen Ansatz jedoch die Notwendigkeit, die Wachstumsbedingungen, die die Hintergrund-Fluoreszenz des Reporterstamm zu minimieren, was die Detektion von Fluoreszenz-Induktions identifizieren. Die Identifizierung dieser Bedingungen müssen oft empirisch durchgeführt werden, obwohl Transkriptions Daten können diese Suche (beispielsweise die Fliesen Microarray-Daten für das Wachstum von B. subtilis vorhanden ermöglicht Identifikation von Bedingungen, in denen die Gene von Interesse sind schlecht ausgedrückt 10) zu unterstützen. Für einige Reporter kann diese empirische Suche schwierig sein, zum Teil, weil der Ausdruck vieler bakterieller Phänotypen ist heterogen. In anderen Worten, ist es selten, c findenie Bedingungen, in denen keine Zellen innerhalb der Population sind Phänotyp exprimieren X. Somit kann in Abhängigkeit von der Anzahl der Zellen innerhalb dieser Untergruppe und der Stärke der Genexpression, kann es schwierig sein, Bedingungen zu schaffen, die ausreichend geringe Hintergrundfluoreszenz zu ermöglichen Induktions nachzuweisenden identifizieren . Eine Alternative zu dieser empirischen Suche nach idealen Screeningbedingungen können zu "tunen" die Expressionsniveaus des Reporters mit Mutagenese sein. Durch Veränderung der Promotor-Region und / oder die ribosomale Bindungsstelle des Reporter-Konstrukt, könnten die Hintergrundfluoreszenz Niveaus verringert werden. Dies könnte den Nutzen dieser Bildschirm, indem es auch Gene, die mit einem gewissen konstitutiven Aktivierung für Induktion untersucht werden erweitern.
Nach Induktion Organismen wurden identifiziert und in einem sekundären Bildschirm bestätigt, können sie phylogenetisch durch Sequenzierung ihrer 16S rRNA-Gen identifiziert werden. Es ist auch möglich, das Ausmaß der Fluoreszenz zu quantifizierenzenz mit OD 600-normierte Ort in den sekundären Bildschirm 5. Dies können Informationen über die Mitglieder der Gemeinschaft erzeugen Verbindungen, die Ihre Reporterstamm beeinflussen und in welchem Umfang. Folglich kann dies zu Hypothesen über die mikrobielle Wechselwirkungen können in natürlicher Umgebung und die Möglichkeit, das Potenzial dieser Koevolution Herstellung und Reaktion auftretenden Organismen erforschen führen werden. Andere zukünftige Richtungen umfassen Aufklärung der Struktur des Moleküls selbst sezerniert, die Bestimmung der Mechanismus (en), durch die der Organismus reagiert erfasst diese Verbindung und Verwendung als chemischer bei der Bekämpfung bakterieller Phänotypen zu modulieren.
Selbst mit den oben beschriebenen Überlegungen ist das hier beschriebene Verfahren einen wichtigen Beitrag. Es vermeidet die Arbeit in der Montage eine Bibliothek von Umwelt Mikroben beteiligt, aber ermöglicht die räumliche Trennung und Isolierung von mit festen Medien. Die Stärke dieser Kokultur Bildschirm ist, dasses bietet eine konzeptionell und technisch einfache Methode, um durch Tausende von Mikrobenarten zu screenen, um diejenigen, die bioaktiven Verbindungen von Interesse absondern, während sie auf viele Bakterienarten und Phänotypen zu identifizieren.
Offenlegungen
Die Autoren erklären, dass sie keine finanziellen Interessen konkurrieren.
Danksagungen
Der Autor dankt Roberto Kolter (Harvard Medical School) für seine wertvolle Ratschläge und Unterstützung bei der Entwicklung dieser Kokultur Bildschirm. Sie hat auch dank Matthew Powers zum Lesen des Manuskripts für Klarheit und Chia Cheng-yi um Unterstützung bei der Erlangung Abbildung 6.
Materialien
| Name | Company | Catalog Number | Comments |
| Spectrophotometer | Any spectrophotomer capable of measuing OD600 absorbance values. | ||
| Luria broth, Lennox | VWR | 80017-484 | Alternative media sources may be necessary. |
| Glass beads, 3 mm | VWR | 26396-508 | |
| Gel loading tips, round | VWR | 29442-666 | |
| Glass rods | VWR | 59060-069 | |
| Fluorescence dissecting stereoscope | Zeiss | N/A | The author used a Zeiss Stemi SV6 dissection stereoscope with an EXFO X-cite 120 fluorescent light source, a long-pass YFP filter cube, an achromat 0.63X objective, 10X eyepieces, and an Axio HRC HR digital camera. Most screening was done with the focusing mount at 2.0-3.2X. Any dissecting stereoscope with fluorescence capabilities is fine, provided you have the correct filters for the FP you are using. It is best if there is a shutter that allows you to easily switch between brightfield and fluorescense, as well as a stage that allows illumination from above and below. If you want to capture images, an attached camera is also necessary. |
Referenzen
- Berdy, J. Bioactive microbial metabolites. J. Antibiot. 58, 1-26 (2005).
- Lopez, D., Vlamakis, H., Kolter, R. Generation of multiple cell types in Bacillus subtilis. FEMS Microbiol. Rev. 33, 152-163 (2009).
- Vlamakis, H., Aguilar, C., Losick, R., Kolter, R. Control of cell fate by the formation of an architecturally complex bacterial community. Genes Dev. 22, 945-953 (2008).
- Branda, S. S., Gonzalez-Pastor, J. E., Ben-Yehuda, S., Losick, R., Kolter, R. Fruiting body formation by Bacillus subtilis. Proc. Natl. Acad. Sci. U.S.A. 98, 11621-11626 (2001).
- Shank, E. A., et al. Interspecies interactions that result in Bacillus subtilis forming biofilms are mediated mainly by members of its own genus. Proc. Natl. Acad. Sci. U.S.A. 108, 1236-1243 (2011).
- Piston, D. W., Patterson, G. H., Lippincott-Schwartz, J., Claxton, N. S., Davidson, M. W. . Introduction to Fluorescent Proteins. , (2013).
- Chudakov, D. M., Matz, M. V., Lukyanov, S., Lukyanov, K. A. Fluorescent proteins and their applications in imaging living cells and tissues. Physiol. Rev. 90, 1103-1163 (2010).
- Shaner, N. C., Patterson, G. H., Davidson, M. W. Advances in fluorescent protein technology. J. Cell Sci. 120, 4247-4260 (2007).
- Shaner, N. C., Steinbach, P. A., Tsien, R. Y. A guide to choosing fluorescent proteins. Nat. Methods. 2, 905-909 (2005).
- Nicolas, P., et al. Condition-dependent transcriptome reveals high-level regulatory architecture in Bacillus subtilis. Science. 335, 1103-1106 (2012).
- Middleton, R., Hofmeister, A. New shuttle vectors for ectopic insertion of genes into Bacillus subtilis. Plasmid. 51, 238-245 (2004).
- Shimotsu, H., Henner, D. J. Construction of a single-copy integration vector and its use in analysis of regulation of the trp operon of Bacillus subtilis. Gene. 43, 85-94 (1986).
- Guerout-Fleury, A. M., Frandsen, N., Stragier, P. Plasmids for ectopic integration in Bacillus subtilis. Gene. 180, 57-61 (1996).
- Semsey, S., Blaha, B., Koles, K., Orosz, L., Papp, P. P. Site-specific integrative elements of rhizobiophage 16-3 can integrate into proline tRNA (CGG) genes in different bacterial genera. J. Bacteriol. 184, 177-182 (2002).
- Charpentier, E., et al. Novel cassette-based shuttle vector system for gram-positive bacteria. Appl. Environ. Microbiol. 70, 6076-6085 (2004).
- Yang, H. Y., Kim, Y. W., Chang, H. I. Construction of an integration-proficient vector based on the site-specific recombination mechanism of enterococcal temperate phage phiFC1. J. Bacteriol. 184, 1859-1864 (2002).
- Choi, K. H., Schweizer, H. P. mini-Tn7 insertion in bacteria with single attTn7 sites: example Pseudomonas aeruginosa. Nat. Protoc. 1, 153-161 (2006).
- Craig, N. L. Tn7: a target site-specific transposon. Mol. Microbiol. 5, 2569-2573 (1991).
- Garcia-Betancur, J. C., Yepes, A., Schneider, J., Lopez, D. Single-cell analysis of Bacillus subtilis biofilms using fluorescence microscopy and flow cytometry. J. Vis. Exp. (60), e3796 (2012).
- Lopez, D., Fischbach, M. A., Chu, F., Losick, R., Kolter, R. Structurally diverse natural products that cause potassium leakage trigger multicellularity in Bacillus subtilis. Proc. Natl. Acad. Sci. U.S.A. 106, 280-285 (2009).
- Vartoukian, S. R., Palmer, R. M., Wade, W. G. Strategies for culture of 'unculturable' bacteria. FEMS Microbiol. Lett. 309, 1-7 (2010).
- Romano, J. D., Kolter, R. Pseudomonas-Saccharomyces interactions: influence of fungal metabolism on bacterial physiology and survival. J. Bacteriol. 187, 940-948 (2005).
- Branda, S. S., Chu, F., Kearns, D. B., Losick, R., Kolter, R. A major protein component of the Bacillus subtilis biofilm matrix. Mol. Microbiol. 59, 1229-1238 (2006).
- Zengler, K., et al. Cultivating the uncultured. Proc. Natl. Acad. Sci. U.S.A. 99, 15681-15686 (2002).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten
Wir verwenden Cookies, um Ihre Erfahrung auf unserer Website zu verbessern.
Indem Sie unsere Website weiterhin nutzen oder auf „Weiter“ klicken, stimmen Sie zu, unsere Cookies zu akzeptieren.
