Method Article
Predicción rápida de la respuesta a fármacos in vivo mediante injertos de células de leucemia en embriones de pez cebra
En este artículo
Resumen
Este protocolo proporciona instrucciones paso a paso para generar y solucionar problemas de xenoinjertos de leucemia linfoblástica aguda (LLA) humana a partir de líneas celulares y material fresco del paciente en embriones de pez cebra inmunosuprimidos transitoriamente, junto con pautas para la evaluación de la respuesta a los medicamentos mediante citometría de flujo. La tubería experimental también se puede adaptar para tumores sólidos.
Resumen
El xenotrasplante de pez cebra es una técnica fundamental para investigar la patogénesis del cáncer humano y predecir las respuestas individuales a los fármacos. Este documento presenta un protocolo simplificado (ZefiX) para expandir muestras de pacientes de leucemia linfoblástica aguda precursora de células B primarias (BCP-ALL) o líneas celulares inmortalizadas en embriones de pez cebra inmunosuprimidos transitoriamente, utilizando citometría de flujo para el análisis de alta resolución de células individuales de las respuestas al tratamiento. En comparación con los injertos de tumores sólidos, las células de leucemia se benefician significativamente de la supresión de los factores diferenciadores de macrófagos y neutrófilos basada en oligonucleótidos antisentido morfolino durante el ensayo. El análisis por citometría de flujo de células de injerto disociadas permite una evaluación precisa del recuento de células, la tasa de proliferación y la vitalidad después del tratamiento por célula. Este enfoque se ha validado utilizando terapias dirigidas como venetoclax y dasatinib, con resultados de tratamiento comparados con los registros clínicos de muestras de pacientes relacionadas y controles de cultivo 2D tradicionales. En particular, el protocolo se completa en un plazo de 7 días, alineándose con los plazos de toma de decisiones clínicas. La metodología es adaptable para probar fármacos seleccionados en varios tipos de cáncer, incluidos los tumores sólidos, lo que permite estrategias terapéuticas personalizadas. Sin embargo, se deben considerar las limitaciones en el número de fármacos que se pueden evaluar, probablemente debido a las restricciones farmacocinéticas en los embriones de pez cebra.
Introducción
El xenotrasplante de pez cebra se ha convertido en un modelo in vivo crucial para comprender la patogénesis del cáncer y predecir las respuestas a los fármacos 1,2,3,4,5. Los modelos animales siguen siendo fundamentales para las pruebas preclínicas de fármacos, y el modelo de pez cebra ofrece ventajas significativas sobre otros sistemas in vivo, como el alto rendimiento y la rentabilidad 6,7,8. Este modelo también podría ayudar a las predicciones personalizadas de la respuesta al tratamiento, incluidas las terapias moleculares dirigidas y la terapia con células CAR-T 9,10,11,12.
La BCP-ALL puede beneficiarse especialmente del xenoinjerto de pez cebra, ya que la expansión de las células primarias de pacientes en cultivo sigue siendo un reto13. Existe una necesidad innegable de nuevos enfoques de tratamiento para la LLA. A pesar de una tasa de remisión alta de 80-85% en niños con LLA-BCP, las tasas de supervivencia a largo plazo para los pacientes con enfermedad recidivante o resistente al tratamiento oscilan solo entre aproximadamente 30%-60%14,15,16. En estos casos, las pruebas de fármacos que utilizan la cartera propuesta podrían integrarse en el entorno clínico para identificar la terapia óptima específica para el paciente14,15. Este enfoque personalizado puede ser crucial cuando se trata de múltiples resistencias a los medicamentos, reduciendo significativamente la carga del tratamiento para los pacientes al evitar medicamentos ineficaces o subóptimos con efectos secundarios graves.
Varias características hacen que el xenoinjerto de embriones de pez cebra sea un modelo adecuado. Las similitudes genéticas entre los seres humanos y el pez cebra (70% de homología genética y 84% de genes compartidos relacionados con enfermedades) respaldan los estudios de interacción gen-fármaco17. Por lo tanto, el uso de un embrión huésped transgénico puede revelar predisposiciones genéticas que afectan la susceptibilidad a los medicamentos18. Alternativamente, se pueden trasplantar células con modificaciones genéticas específicas para evaluar si la sensibilidad o resistencia al fármaco se alinea con los hallazgos in vitro . Los xenoinjertos de embriones de pez cebra también proporcionan información sobre los posibles efectos sistémicos de los fármacos. A pesar de que el desarrollo de los órganos en los embriones de 2-3 días de edad no está completamente maduro, los órganos están correctamente localizados y comparten parcialmente la composición celular con sus contrapartes adultas19.
Otras ventajas de este modelo incluyen que solo se necesitan unas pocas células cancerosas para el injerto, el mantenimiento de los embriones huéspedes es simple, ya que no se requiere alimentación dentro de los primeros 5 días de vida, y el éxito de la inyección se puede evaluar rápidamente debido a la transparencia y el tamaño de los embriones. Una característica única es que solo la inmunidad innata está activa en esta etapa de desarrollo, lo que facilita un injerto eficiente20. En el protocolo ZefiX descrito aquí (ver resumen en la Figura 1), la inmunodeficiencia se potencia aún más al suprimir el sistema inmune innato durante los primeros 4 días de vida utilizando oligonucleótidos estables antisentido Morpholino dirigidos a spi1 y csf3r, que bloquean la diferenciación de macrófagos y neutrófilos 21,22,23.
Este protocolo también difiere de los protocolos anteriores de xenotrasplante de pez cebra, que se desarrollaron principalmente para injertos de tumores sólidos y, por lo general, utilizan métodos de evaluación de la respuesta a medicamentos basados en imágenes de montaje completo. ZefiX está optimizado para células cancerosas líquidas, como las células BCP-ALL, y se ha utilizado con éxito para expandir material de paciente fresco o congelado21. ZefiX también se puede adaptar para las células cancerosas adherentes mediante la selección de enzimas apropiadas para la disociación de tejidos.
Otra ventaja importante es el análisis posterior mediante citometría de flujo, que ofrece varios beneficios: (i) un gran número de células de injerto se puede procesar rápidamente, lo que permite un análisis estadístico sólido a nivel de una sola célula, (ii) la tasa de proliferación y la viabilidad se pueden evaluar simultáneamente en células individuales, y (iii) los citómetros de flujo están comúnmente disponibles en entornos de investigación clínica, lo que permite la evaluación de la respuesta a los fármacos de las células de injerto a nivel de una sola célula en unas pocas horas. Para garantizar la reproducibilidad, este protocolo proporciona un canal estandarizado desde la preparación hasta el análisis de citometría de flujo, pasando por el trasplante, lo que permite la predicción de la respuesta al fármaco en las células de ALL en una semana.
Protocolo
Todos los experimentos con pez cebra cumplen con las directrices de los Institutos de Investigación de Medicina Experimental de la Charité-Universitätsmedizin de Berlín y con las autoridades oficiales. Todos los estudios involucraron embriones de pez cebra < 6 días después de la fertilización (dpf), eximiéndolos de la Ley de Protección de Animales. El pez cebra (Danio rerio) se crió y mantuvo en el animalario de Charité-Universitätsmedizin Berlin, Berlín, Alemania, de acuerdo con protocolos estándar. Se alojaron a 28 °C con un ciclo de luz de 14 h y 10 h de oscuridad. Para todos los experimentos se utilizaron peces de tipo salvaje de las cepas AB o TüLF.
NOTA: Establecer las condiciones óptimas de tratamiento para cada fármaco deseado antes de su aplicación con ZefiX incluye varios pasos necesarios. En primer lugar, determinar la concentración inhibitoria máxima media (IC50) de cada fármaco utilizando una línea celular adecuada dentro de un sistema de cultivo 2D convencional. Sobre la base de la experiencia previa, las concentraciones efectivas del fármaco para el tratamiento con ZefiX pueden ser de 5 a 50 veces mayores que las utilizadas en condiciones típicas de cultivo celular21,24. Antes de tratar los embriones injertados, es esencial evaluar la toxicidad dentro de los embriones huéspedes no trasplantados utilizando el rango de concentración establecido. Después de evaluar la toxicidad, exponga los embriones injertados en líneas celulares a una variedad de concentraciones de fármaco de alrededor de 50 veces el valor de IC50 determinado previamente en cultivo 2D. Si las células injertadas no muestran respuesta a dosis de hasta 100 veces el IC50, el fármaco puede considerarse ineficaz para ZefiX. Para mejorar potencialmente la eficacia, una opción es preacondicionar las células del injerto con el fármaco poco antes de su trasplante a los embriones25. Consulte la Tabla 1 para ver todas las soluciones utilizadas aquí.
1. Día 1: Preparación para el experimento
- Preparación del medio E3: Prepare 2 L de medio E3 esterilizado en autoclave para el mantenimiento del embrión.
- Preparación de oligonucleótidos (MO) antisentido morfolino: Prepare una solución madre de 50 μM que contenga ambos MO en un tubo de microcentrífuga de 1,5 mL utilizando agua libre de nucleasas. Almacene la solución madre a temperatura ambiente (RT). Prepare los MO para la inyección incubando la solución en un bloque calefactor a 65 °C durante 10 min.
NOTA: Los MOs se dirigen contra spi1 y csf3r para inhibir la diferenciación de macrófagos y células de neutrófilos, como se describe en las referencias 22,23,26. - Preparación de placas de inyección
- Para preparar 4-5 placas, disuelva el 1% de agarosa en medio E3 para crear una solución de 100 mL. Para las placas de trasplante, vierta ~ 20 ml de la solución en cada placa de Petri de 10 cm, asegurándose de que estén medio llenas. Revuelva para distribuir uniformemente el líquido.
- Para las placas de inyección de Morfolino, coloque el molde de inyección sobre la agarosa líquida en dos placas de Petri, asegurándose de que no se formen burbujas. Cubra los platos inclinando las tapas y déjelos en RT hasta que la agarosa se haya solidificado. Una vez solidificado, retire el molde y guarde las placas boca abajo a 4 °C en una bolsa de plástico sellada.
- Preparación de agujas de inyección
- Para inyecciones de Morfolino: Genere agujas a partir de capilares de 10 cm utilizando un extractor de agujas y puntas de ruptura para lograr un diámetro estimado de 10 μm (como se describe en27).
- Para el trasplante de células: Utilice agujas de inyección de extremo romo preextraídos disponibles en el mercado con un diámetro exterior de 20 μm.
- Cultivo de células de injerto: Células divididas (p. ej., Nalm6) con el objetivo de alcanzar una densidad objetivo del 70%-80% en el día 4 en un matraz de cultivo celular T175 con medio RPMI suplementado con 10% de FCS y 1% de P/S (RPMI-complete). Divida las células 3-4 veces antes de usarlas para garantizar una tasa de proliferación adecuada.
NOTA: Para la preparación de material de paciente fresco o fresco/congelado para trasplante, las instrucciones se pueden encontrar en el paso 4.3. A continuación, las células se preparan el día del trasplante. - Cría de peces cebra: Instale peces cebra de tipo salvaje en tanques de cría por la tarde, manteniendo a los machos y las hembras separados.
NOTA: Los peces cebra fueron criados y escenificados como se describe en la referencia28. Las referencias de tiempo (hpf o dpf) denotan horas o días después de la fertilización.
2. Día 2: Inyección de morfolino
- Complemente 500 mL de medio E3 esterilizado en autoclave con penicilina/estreptomicina al 1% (E3/P/S) y llene dos placas de Petri de 10 cm. Saca la placa de inyección de la nevera para precalentarla a RT.
- Apertura escalonada de la puerta de los tanques de cría para que la fertilización solo pueda tener lugar en una fracción de tanques a la vez (dependiendo de la velocidad de inyección) para garantizar microinyecciones oportunas de solución de morfolino en embriones de pez cebra en la etapa de una sola célula28,29. Comience abriendo una o dos compuertas dependiendo de la velocidad del inyector, cuando un lote de huevos se inyecta fácilmente, abra otra puerta o dos compuertas y así sucesivamente.
- Transfiera los óvulos fertilizados en lotes de 100 con la menor cantidad de líquido posible a la placa de inyección previamente preparada. Alinea los embriones en las ranuras de la placa.
- Inyecte 1 nL de una mezcla de 50 μM de ambos morfolinos en la célula o en el saco vitelino justo debajo de la célula durante la etapa de una célula29. Inyecte suficientes óvulos para los procedimientos posteriores (p. ej., para una placa de 96 pocillos, inyecte hasta 200 óvulos para tener suficiente respaldo para un posible abandono antes del trasplante).
- Transfiera los huevos inyectados a placas de Petri que contengan E3/P/S. Incube los huevos inyectados a 28 °C. Retenga los huevos no inyectados como peces de control para el análisis de citometría de flujo a 5 dpf. Almacene el medio E3/P/S restante a 4 °C.
3. Día 3: Descorionación
- Decorionación de embriones de pez cebra: Decorionar manualmente embriones cuando tienen más de 24 hpf utilizando dos pinzas de precisión29.
- Retire de los platos los embriones muertos que no muestren latidos ni movimiento y que parezcan opacos o con formas irregulares de la vajilla con una pipeta Pasteur.
- Pellizque el corion con pinzas de precisión para mantenerlo en su lugar. Pellizque justo al lado de la punta de las pinzas de precisión, sosteniendo el embrión en su lugar con las segundas pinzas, y separe con cuidado el corion para liberar el embrión
NOTA: Para descorionar embriones menores de 24 hpf, deben mantenerse en un plato recubierto de agarosa para evitar que se peguen al plástico. Es preferible la descorionación manual, ya que es más suave para los embriones. Un método alternativo de descoionación enzimática se describe en otro lugar30.
- Incubar los embriones descorionados a 28 °C durante la noche.
4. Día 4: Xenotrasplante y tratamiento farmacológico
- Preparación de los embriones huéspedes: Retire las placas de agarosa preparadas y el E3/P/S de la nevera y deje que alcancen RT. Examine los embriones para determinar la etapa de desarrollo adecuada a 48 hpf utilizando un microscopio estereoscópico. Incluya solo embriones debidamente estadificados y morfológicamente típicos en el flujo de trabajo, como se describe en otra parte29. Cuente todos los embriones sanos y planifique el tratamiento adicional. No mantener más de 100 embriones por placa de Petri de 10 cm a 28 °C para evitar velocidades de desarrollo desiguales causadas por la escasez de oxígeno.
NOTA: Siga el paso 4.2. para líneas celulares. Para material fresco/congelado, proceda directamente al Paso 4.3. - Preparación de líneas celulares
- Para preparar células BCP-ALL marcadas con fluorescencia para la citometría de flujo y el trasplante, lavar las células (desde el paso 1.5.) con 1x PBS: centrifugar a 350 x g durante 5 min, y resuspender en 20 mL de PBS.
- Cuente las células y transfiera 3 x 105 células sin teñir a un tubo FACS para el análisis de citometría de flujo del día 0 (0 días después de la inyección, ppp). Almacenar en hielo.
- Placa de 3 x 105 celdas en 3 mL de RPMI: completa en un pocillo de una placa de 6 pocillos y manténgala a 37 °C para un control de 3 ppp.
- Transfiera 1 x 107 celdas a un tubo de centrífuga de 15 mL para el etiquetado de CTV. Centrifugar a 350 x g durante 5 min en RT, verter el sobrenadante (usar una pipeta para el resto) y resuspender el pellet en 2,5 mL de PBS (RT) con 1 μL de solución madre de CTV.
- Incubar durante 5 min en la oscuridad a 37 °C, detener la reacción con 12,5 mL de RPMI-complete, luego incubar durante 10 min en la oscuridad a 37 °C.
- Centrifugar a 350 x g durante 5 min a RT y lavar una vez con 10 mL de RPMI-complete. Vuelva a centrifugar y vuelva a suspender en 10 mL de RPMI-complete.
- Filtrar las células con un filtro de 10 μm centrifugando a 350 x g durante 5 min. Vuelva a suspender el pellet, cuente las células y transfiera 3 x 105 células marcadas con CTV a un tubo FACS. Placa 3x 10^5 celdas con 3 mL de RPMI-completo en un pocillo de una placa de 6 pocillos y manténgalo a 37°C para un control de proliferación de 3 dpi. Almacene las células restantes en 1 mL de PBS en hielo hasta el trasplante.
- Preparación de material de paciente fresco/congelado
- Precaliente dos tubos de centrífuga de 15 mL con 10 mL de RPMI-complete cada uno a 37 °C. Descongele un vial que contenga 5-10 x 106 células en un baño de agua a 37 °C.
- Una vez que solo quede una pequeña cantidad de hielo, transfiera las celdas al tubo de centrífuga con el medio precalentado. Centrifugar a 350 x g durante 5 min a RT, desechar el sobrenadante y volver a suspender el pellet en el segundo tubo de RPMI-complete precalentado.
- Cuente las células viables con azul de tripán. Alícuota 1x 10^5 células en un tubo de centrífuga de 1,5 mL y centrifugar esta alícuota de células en un tubo FACS a 350 x g durante 5 min a RT, desechar el sobrenadante y resuspender el pellet en 300 μL de PBS.
- Almacene la alícuota resuspendida en hielo como un control sin tratar para el análisis de citometría de flujo.
NOTA: Si el tamaño de la muestra lo permite, mantenga un control sin tratar en cultivo para la medición de citometría de flujo el día 7. - Añadir 1 μL de la solución madre de CTV a 2,5 mL de PBS (RT) por cada 1 x 106 células. Ajuste el volumen de la solución madre de CTV si hay menos celdas disponibles.
- Incubar las células con CTV durante 5 min a 37 °C en la oscuridad, detener la reacción añadiendo 12,5 mL de RPMI completo precalentado (37 °C) e incubar durante 10 min en la oscuridad a 37 °C.
- Centrifugar las células a 350 x g durante 5 min a RT y lavar las células una vez con 10 mL de RPMI-complete. Centrifugar de nuevo a 350 x g durante 5 min a RT y resuspender las células en 10 mL de RPMI-completo.
- Filtre la suspensión de la célula en un tubo de centrífuga nuevo de 50 ml con un filtro de 10 μm y centrifugue a 350 x g durante 5 min.
- No deseche el sobrenadante. Vuelva a suspender el pellet y cuente las células. Transfiera 3 x 105 células marcadas con CTV a un tubo FACS.
- Centrifugar las células restantes a 350 x g durante 5 min a RT y desechar el sobrenadante. Vuelva a suspender las células restantes en 1 mL de PBS y almacene en hielo hasta su uso en el trasplante.
NOTA: Si quedan suficientes células CTV positivas, se pueden mantener en cultivo 2D en paralelo a los xenoinjertos para la comparación de la tasa de proliferación.
- Medición por citometría de flujo
NOTA: La citometría de flujo a 0 ppp debe ser realizada por una segunda persona para minimizar el tiempo que las células permanecen en hielo antes del trasplante. Antes de la primera medición por citometría de flujo utilizando el panel de tinción designado (CTV, CD19-Alexa488, APC-Annexin V y 7AAD), realice una prueba de compensación siguiendo las instrucciones del fabricante.- Prepare dos tubos FACS de la siguiente manera: Tubo 1: Células de control sin teñir; Tubo 2: Células teñidas con CTV, CD19, 7AAD y anexina.
NOTA: Tinción y etiquetas: CellTrace Violet (CTV) identifica las células de injerto marcadas frente a las células de peces huéspedes y evalúa la tasa de proliferación después de 3 días. El anticuerpo CD19, un marcador de la superficie celular, sirve como marcador adicional para identificar las células de injerto de LLA-BCP humana. Para otros tipos de cáncer, es posible que se requieran anticuerpos marcadores alternativos. La anexina V marca la apoptosis en etapa temprana para la evaluación de la viabilidad celular. 7AAD marca la apoptosis o necrosis en etapa tardía para la evaluación de la viabilidad celular. - Centrifugue los tubos (uno que contenga células teñidas con CTV y el otro que no esté teñido) a 350 x g durante 5 min en RT y deseche el sobrenadante del tubo 1. Vuelva a suspender el gránulo en 310 μL de tampón de unión a anexina (ABB).
- Tubo 2: Realice la tinción de anticuerpos y viabilidad como se describe a continuación.
NOTA: Este protocolo de tinción está optimizado para la tinción de células B CD19. En el caso de otros anticuerpos humanos utilizados para marcar diferentes tipos de células, es posible que sea necesario adaptar el protocolo.- Añada 98 μL de ABB al tubo 2 (que contiene células marcadas con CTV). Añadir 2 μL de anticuerpo CD19-Alexa488 (dilución 1:50) a los 98 μL de ABB añadidos. Mezclar bien.
- Incubar la mezcla a 4 °C durante 30 min y añadir 500 μL de ABB para detener la reacción. Centrifugar el tubo a 350 x g durante 5 min a 4 °C. Retire el sobrenadante y repita el paso de lavado.
- Vuelva a suspender el pellet celular en 100 μL de ABB y proceda a la tinción con 7AAD y APC-Anexina V. Realice la tinción de anexina V 7AAD y APC y la medición de citometría de flujo como se describe a continuación.
- Añada 5 μL de 7AAD y 5 μL de APC-anexina V a las células resuspendidas. Vórtice suavemente para mezclar. Incubar el tubo durante 15 minutos en la oscuridad a RT. Añadir 200 μL de ABB para alcanzar un volumen final de 310 μL.
- Realice el análisis de citometría de flujo el día del trasplante en el siguiente orden para evitar la contaminación cruzada: células de control sin teñir (tubo 1), células marcadas con CTV teñidas con Alexa488-CD19, APC-anexina V y 7AAD (tubo 2). Registre al menos 10.000 eventos por muestra para un análisis suficiente.
- Prepare dos tubos FACS de la siguiente manera: Tubo 1: Células de control sin teñir; Tubo 2: Células teñidas con CTV, CD19, 7AAD y anexina.
5. Trasplante
- Placa de trasplante: Prepare dos placas de Petri de 10 cm llenas de E3/P/S y colóquelas en la incubadora a 28 °C durante al menos 30 minutos para que se precalienten.
- Preparación de las células: Centrifugar las células a 350 x g durante 5 min en RT, desechar el sobrenadante y eliminar el líquido restante con una micropipeta. Agregue PBS para lograr un volumen final de 20 μL. Mantenga la suspensión celular concentrada en hielo.
NOTA: Las células no deben permanecer en hielo durante más de 2 horas durante el procedimiento de trasplante. - Aguja de trasplante y preparación del embrión huésped
- Transfiera 25-30 embriones a una de las placas de Petri precalentadas que contienen E3/P/S utilizando una pipeta Pasteur de vidrio para que sirva como control. Cargue 4 μL de la suspensión celular en la aguja de trasplante con una punta de microcargador.
NOTA: La carga debería realizarse sin problemas en 1-2 minutos. Si este no es el caso, agregue con cuidado más PBS a la suspensión de celdas. - Calibre la presión de inyección y la longitud del pulso ajustando el microinyector. Ajuste hasta que una inyección expulse aproximadamente 1.000 células ALL o 2 nL de suspensión celular (para 1x 107 células en 20 μL). Para estimar 1.000 células, expulse un volumen de la suspensión sobre una superficie de agarosa cubierta con medio. Cuente 100 células en un área pequeña, extrapola su distribución a la población total de células y estime el número total.
- Preparar 50 mL de E3 que contenga tricaína (concentración final: 80 mg/L). Transfiera los embriones del huésped a la solución de tricaína e incube durante al menos 2 minutos para garantizar una anestesia adecuada. El embrión está debidamente anestesiado cuando no se observa ninguna respuesta motora
NOTA: Se puede utilizar una punta de microcargador o pinzas para acercarse y/o tocar cuidadosamente el embrión. - Transfiera de 15 a 20 embriones descorionados a una placa de inyección recubierta de agarosa (consulte las instrucciones de preparación anteriores) utilizando la menor cantidad de líquido posible para evitar que los embriones se deslicen.
- Organice los embriones como se ilustra en la Figura 2A. Inyectar aproximadamente 1.000 células BCP-ALL positivas para CTV en la cavidad pericárdica. Introduzca la aguja en un ángulo de 45° con respecto a la dirección dorso-caudal, como se muestra en la Figura 2B para inyectar las células.
NOTA: Utilice agujas pre-tiradas de punta roma disponibles en el mercado con un diámetro de apertura de 20 μm, diseñadas específicamente para células leucémicas pequeñas (consulte la lista de materiales). Si la aguja se bloquea, recórtela según sea necesario, pero vuelva a calibrar el volumen de inyección y el recuento de células después. - Una vez inyectados los 15-20 embriones, transfiéralos a la placa de Petri precalentada llena de E3/P/S y manténgala a 28 °C.
- Repita los pasos hasta que se trasplanten aproximadamente 100 a 150 embriones para un ensayo de tratamiento con medicamentos, que debe incluir tres concentraciones de medicamento y un control.
- Incubar tanto los embriones trasplantados como los controles no trasplantados a 28 °C durante 1-3 h antes de iniciar el tratamiento farmacológico.
- Transfiera 25-30 embriones a una de las placas de Petri precalentadas que contienen E3/P/S utilizando una pipeta Pasteur de vidrio para que sirva como control. Cargue 4 μL de la suspensión celular en la aguja de trasplante con una punta de microcargador.
- Tratamiento farmacológico in vivo (placa de 96 pocillos)
- Mediante el uso de estereomicroscopía fluorescente, se examinan los embriones para confirmar el éxito del injerto (Figura 2C). Asegúrese de que la yema esté intacta, ya que el entorno de la yema podría ser tóxico para las células del injerto21. Deseche los embriones con células en la yema.
- Prepare 2,5 mL de una solución concentrada 2x para cada condición de medicamento que se va a probar en E3/P/S con DMSO al 0,5%. Además, prepare una solución de control de vehículos de 5 ml que contenga 0,5% de DMSO en E3/P/S.
- Añada 100 μL de E3/P/S + 0,5% de DMSO a cada pocillo de una placa de 96 pocillos. Este paso evita la transferencia involuntaria de cantidades mínimas de fármaco entre pocillos.
- Transfiera cuidadosamente un embrión injertado a cada pocillo. Utilice una pipeta Pasteur de vidrio para recoger cada embrión en la menor cantidad posible de medio E3.
- Deje que el embrión se hunda hasta el fondo de la punta de la pipeta inclinando suavemente la pipeta y libérela en el pocillo utilizando fuerzas capilares. Evite tocar el medio en el pocillo durante la transferencia.
- Agregue soluciones de fármaco a la placa: Llene 24 pocillos (2 filas) de la placa de 96 pocillos con 100 μL de la solución de control del vehículo o de una de las tres soluciones de fármaco concentradas 2x.
NOTA: Las concentraciones efectivas del fármaco deben determinarse en experimentos previos específicos para cada fármaco que se esté probando. - Mantener los embriones a 35 °C durante 72 h, incluidos los embriones no trasplantados, que servirán como controles para el análisis de citometría de flujo el día 7.
6. Día 7
- Disociación embrión/injerto
- Examinar la placa de 96 pocillos mediante microscopía estereoscópica para identificar y seleccionar embriones sanos de pez cebra. Agrupe aleatoriamente 10 embriones huéspedes sanos de cada afección en un tubo de microcentrífuga de 1,5 ml (lo ideal es que haya 2 tubos por afección).
- Extraer la mayor cantidad de líquido posible de cada tubo que contenga embriones y sacrificar los embriones por choque hipotérmico incubando los tubos en hielo durante 1 h.
- Agregue 500 μL de solución salina balanceada de Hank's (HBSS) sin calcio ni magnesio a cada tubo. Disociar mecánicamente los embriones e injertar células mediante trituración con una punta de micropipeta de 200 μL, pipeteando hacia arriba y hacia abajo aproximadamente 15 veces.
- Granular los fragmentos de tejido por centrifugación a 350 x g durante 5 min a RT. Mientras tanto, prepare tubos FACS con una tapa de filtro de filtro de malla fina de 35 μm que contenga 2 mL de PBS por condición (4 tubos en total).
- Vuelva a suspender cada gránulo en 500 μL de mezcla de enzimas (0,01 % de papaína, 0,1 % de dispasa II, 0,01 % de desoxirribonucleasa I y 12,4 mM de MgSO4 en HBSS sin calcio y magnesio) para la disociación enzimática. Incubar en RT durante 15 min.
- Durante la incubación, pipetee la mezcla hacia arriba y hacia abajo cada 5 minutos repetidamente usando la misma punta de pipeta para cada tubo individual para evitar la pérdida de tejido.
- Medición por citometría de flujo
- Transfiera las células disociadas a la tapa del filtro de malla fina de 35 μm de los tubos FACS y centrifugue a 350 x g durante 5 minutos.
- Durante la centrifugación, prepare una mezcla maestra para la tinción de la superficie de las células B CD19. Combine 98 μL de ABB con 2 μL de Alexa Fluor 488 anticuerpo CD19 humano para cada afección.
- Deseche el sobrenadante del pellet de células disociadas y vuelva a suspender el pellet en 100 μL de la mezcla maestra de tinción CD19.
- Realice el protocolo de tinción de anexina V CD19, 7AAD y APC para todas las muestras de ZefiX, incluidas las células 3 x 105 5 CTV positivas de cultivo celular paralelo, si están disponibles.
- Realice el análisis de citometría de flujo siguiendo el orden y registre el número de eventos descritos en la Tabla 2. Examine todas las muestras que contengan células huésped y de injerto de la forma más completa posible para evaluar el número total de células de injerto.
- Análisis de resultados mediante software comercial.
- Estrategia de compuerta para muestras de cultivo celular: Abra el software y cargue los archivos FCS en el espacio de trabajo. Cree un diagrama de puntos con CD19 y CTV para asegurarse de que la señal se superponga y para confirmar la presencia de las células cancerosas. A continuación, cree un diagrama de puntos con FSC-A (eje x) y SSC-A (eje y) para distinguir las celdas intactas (Q2) de los residuos (Q4; Figura 3A').
- Utilice la población de células intactas para crear una nueva gráfica con SSC-H (eje x) y SSC-A (eje y) para identificar celdas individuales (Figura 3A'').
- Cree otro diagrama de puntos con anexina V (eje x) y 7AAD (eje y) utilizando la población de celdas individuales (Figura 3A'''). Distingue las células en cuatro poblaciones: Células viables: Anexina V negativa, 7AAD negativa (Q4); Células apoptóticas precoces: Anexina V positiva, 7AAD negativa (Q3); Células apoptóticas/necróticas tardías: anexina V positiva, 7AAD positiva (Q2); Células necróticas: Anexina V negativa, 7AAD positiva (Q1).
- Repita estos pasos para muestras de 3 ppp (Figura 3B).
- Estrategia de activación para células de injerto y embrión huésped
- Separe las células de injerto humano de las células huésped mediante un diagrama de puntos con CTV (eje x) y CD19 (eje y). Identificar y aislar las células de injerto doble positivo CTV/CD19 (Figura 3C).
- Aplique la misma estrategia de compuerta descrita para las células de cultivo de 3 ppp (Figura 3B' - B''') a la población de células de injerto. Copie la estrategia de compuerta para mantener la coherencia.
- Combine todas las poblaciones de células negativas para anexina V y 7AAD en un histograma con valores de CTV en el eje x (Figura 3D). Calcule la media geométrica de las cinco muestras para determinar las tasas de proliferación.
- Cálculos para la evaluación de la respuesta al tratamiento
- Para determinar el número de divisiones celulares después de 3 días, use la fórmula:
n = logaritmo2 (I0/I)
Donde, I0 = Intensidad inicial de fluorescencia de CTV (media geométrica, 0 ppp), I = Intensidad de fluorescencia de CTV a las 72 h, n = Número de divisiones celulares.
Ejemplo: Células de paciente frescas congeladas divididas 2,6 veces en ZefiX log2(88317/14644) = 2,6
y 2,8x en el registro de cultivo 2D2(88317/12992) = 2,8 - Para determinar el número total de células de injerto por pez después de 3 días, divida el número total de células cancerosas (incluidas las células apoptóticas, pero excluyendo los desechos) por el número de peces agrupados en la muestra (por lo general, n = 10).
- Para determinar la viabilidad de las células de injerto CTV positivas después de 3 días, use la fórmula:
V = (C/100) x A
Donde: V = Viabilidad, C = fracción de células individuales intactas sin residuos (como porcentaje), A = Fracción de células negativas para la anexina V y 7AAD (como porcentaje).
- Para determinar el número de divisiones celulares después de 3 días, use la fórmula:
Resultados
Para una evaluación científica detallada del protocolo ZefiX, incluyendo el tratamiento con xenoinjertos y fármacos de muestras de células BCP-ALL primarias recién congeladas, consulte el manuscrito21 publicado anteriormente. La aprobación para el uso de muestras de pacientes en la investigación para pruebas preclínicas de medicamentos fue otorgada como parte de los estudios complementarios al ensayo ALL-REZ BFM 2002 (NCT00114348) y al registro y biobanco ALL-REZ BFM (EA2/055/12) por parte de los comités locales de ética de investigación médica, así como al ensayo internacional IntReALL SR 2010 (NCT01802814) por parte de la autoridad nacional. Se obtuvo el consentimiento informado de los pacientes y/o sus tutores a través del respectivo ensayo o registro en el que estaban inscritos.
La figura 2 ilustra un ejemplo de alineación de embriones en una placa de agarosa antes de la inyección, lo que ayuda a agilizar el proceso de inyección. La inyección debe realizarse en el ángulo descrito para dirigirse con precisión a la cavidad que rodea el corazón en desarrollo. Además, la Figura 2C proporciona una referencia de embriones 2 dpf inyectados con éxito que contienen células de injerto humano (azul), que se marcaron con CTV antes de la inyección. Se excluyeron los embriones con resultados de inyección diferentes a los mostrados en la Figura 2C , con especial atención a evitar la perforación del saco vitelino para garantizar la viabilidad de las células del injerto durante la incubación posterior de tres días.
Después del período de incubación de tres días, los embriones huéspedes se procesan en grupos de 10 para el análisis de citometría de flujo. Después de la disociación enzimática, las suspensiones celulares se tiñen con un anticuerpo anti-CD19 humano y dos marcadores de viabilidad: anexina V para las células apoptóticas tempranas y 7AAD para las células apoptóticas y necróticas tardías.
En la Figura 3 se presentan los datos de citometría de flujo de células BCP-ALL expandidas con ZefiX de un paciente con BCP-LLA. Los paneles A, A', A''y A''' muestran los datos recopilados a 0 ppp el día del trasplante. La Figura 3A muestra los valores de fluorescencia de CTV y CD19 para un total de 10.000 células como referencia para la estrategia de compuerta aplicada a las suspensiones de células huésped-injerto a 3 ppp (Figura 3C). En la Figura 3A', los desechos se excluyen a través de la compuerta regular de las celdas utilizando el área de dispersión directa (FSC-A) y el área de dispersión lateral (SSC-A). En la Figura 3A'', las celdas individuales se separan de los dobletes utilizando un gráfico de la altura del SSC (SSC-H) frente al SSC-A. Esta población unicelular se utiliza para la evaluación de la viabilidad en la Figura 3A''', donde las células viables (Q4) se distinguen de las células apoptóticas tempranas (Q3, valores más altos de anexina V) y las células apoptóticas o necróticas tardías (Q2, niveles más altos de 7AAD).
A modo de comparación, las células pacientes cultivadas en condiciones 2D convencionales también se analizan mediante citometría de flujo después de tres días (Figuras 3B, B', B'', B'''), siguiendo la misma estrategia de compuerta. La viabilidad de las células del paciente después de 72 h en cultivo 2D se calcula a partir de Q2 en la Figura 2B' y Q4 en la Figura 2B''': (95,0%/100)*61,9% = 58,8%.
En la Figura 3C, el material de partida es la suspensión celular de embriones huéspedes que contienen células de injerto. A diferencia de las mediciones in vitro, todas las células del tubo se analizan mediante citometría de flujo. Las células de injerto CD19 y CTV positivas se comprimen para separarlas de las células de pescado CD19 y CTV negativas. La población de células de injerto intactas se analiza con más detalle en la Figura 3C', donde se excluyen los residuos. A continuación, se evalúa la viabilidad de las células de un solo injerto utilizando la misma estrategia de compuerta que en las Figuras 3B.
Los resultados indican que el porcentaje de células individuales viables expandidas en embriones es del 95,2%, que es 1,6 veces mayor que la viabilidad de las células cultivadas en una placa (Figura 3B'''). Las tasas de división celular se calcularon in vivo e in vitro analizando la disminución de la intensidad de fluorescencia del CTV en cada población de 0 ppp a 3 ppp (Figura 3D). El número de divisiones celulares se determinó mediante la fórmula de la sección 6.4.1 y la media geométrica de cada curva CTV (Figura 3D). Las tasas de división calculadas (2,59 divisiones in vivo y 2,77 divisiones in vitro) sugieren que las células viables se dividen a un ritmo similar en ambas condiciones durante tres días.
Por último, el número medio de células de injerto intactas por embrión después de tres días se determinó dividiendo el número de células de injerto intactas (excluyendo los residuos, figura 3C') por el número de embriones agrupados en un tubo21.
En conclusión, las muestras frescas de BCP-ALL injertadas en embriones de pez cebra exhiben una mayor viabilidad después de tres días en comparación con el cultivo convencional en una placa y las células viables se dividen a una tasa comparable en ambas condiciones.
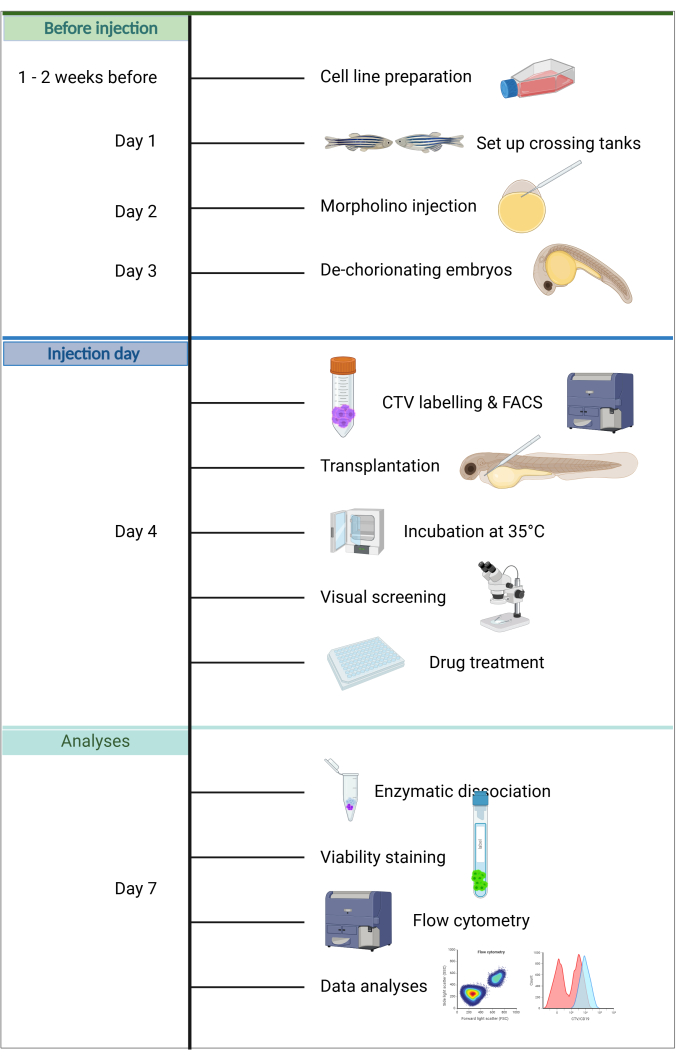
Figura 1. Flujo de trabajo de la canalización ALL-ZefiX. Creado en https://BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.
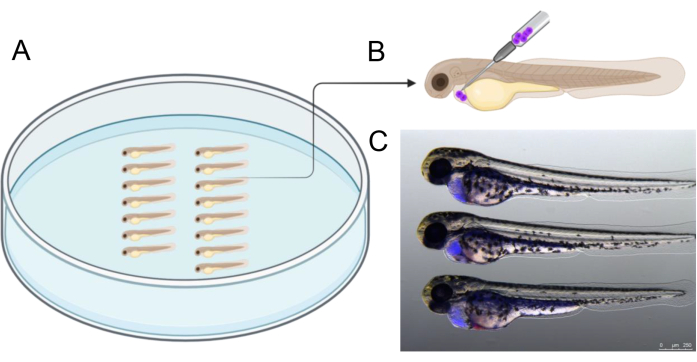
Figura 2: Disposición de la inyección. (A) La disposición de los embriones como se muestra facilita la inyección. Los embriones se pueden disponer con pinzas o con una punta de pipeta microcargadora de 20 μL que se cortó para tener una punta de 2,5 a 3 cm de longitud. (B) Representación esquemática del ángulo de inyección recomendado para injertar células en el pericardio de 2 embriones dpf. (C) Ayudas visuales para estimar correctamente la cantidad de células cancerosas trasplantadas. Esta imagen muestra un embrión de 48 hpf a las 3 h después de la inyección con células de cáncer humano previamente marcadas con CellTrace Violet). Creado en https://BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Estrategia de activación y análisis de citometría de flujo de una muestra fresca congelada derivada de un paciente de células blásticas BCP-ALL aisladas después de un cultivo 2D de injerto en embriones de pez cebra. (A, B) Las células de los pacientes se marcaron con CellTrace Violet (CTV) antes del cultivo. Las células se cultivaron en plástico de cultivo de tejidos a 37 °C durante 0 h (A) o 72 h (B) antes del análisis de citometría de flujo. (C) Células de pacientes marcadas con CTV y cultivadas a 35 °C durante 72 h como injertos en embriones de pez cebra huésped. Un grupo de 10 embriones se agrupó antes de la disociación de una sola célula para el análisis de citometría de flujo. Se aplicó la estrategia de compuerta en (B) y se identificó y cuantificó la fracción de células viables del injerto. Para ello, se separaron las células de injerto positivas para CTV (Q2) de las células de pez cebra autofluorescentes (Q1/4) para clasificar la población de células de injerto para su análisis. Se analizó la intensidad del marcaje de CTV en esta población de células de injerto. (D) Recuentos de células e intensidad media de las etiquetas de CTV a partir de la selección de células viables únicamente. Nótese el cambio de intensidad de CTV después de 3 días (3 ppp). Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Tabla de soluciones utilizadas. Haga clic aquí para descargar esta tabla.
Tabla 2: Muestras medidas por citometría de flujo. Haga clic aquí para descargar esta tabla.
Discusión
Los embriones de pez cebra se han convertido en un modelo de xenoinjerto cada vez más popular para el cribado de fármacos y la investigación del cáncer debido a su alta capacidad de rendimiento y rentabilidad. Estos xenoinjertos son prometedores como un pilar crítico de la medicina traslacional, ya que ayudan a la investigación preclínica y a la toma de decisiones 9,21. Sin embargo, los modelos de xenoinjertos de pez cebra para la expansión y el tratamiento de las células leucémicas humanas siguen estando infrarrepresentados en comparación con el extenso trabajo sobre injertos de tumores sólidos. Este protocolo ofrece una guía detallada para aprovechar los xenoinjertos de pez cebra en la investigación de la leucemia y, al mismo tiempo, se adapta para su uso en tumores sólidos.
Lograr un trasplante consistente de células cancerosas puede ser un desafío, lo que pone de relieve la necesidad de un análisis estandarizado y una mayor confiabilidad estadística. Este protocolo aborda estos problemas mediante la presentación de una canalización completa para la preparación, el trasplante y el análisis de citometría de flujo aguas abajo, junto con recomendaciones para la solución de problemas.
Inyección de morfolino para la inmunosupresión transitoria
Los embriones de pez cebra dependen de su sistema inmunitario innato durante los primeros días de desarrollo, lo que define el marco temporal de esta línea experimental20. Los macrófagos primitivos emergen alrededor de 12 hpf, y algunos se diferencian en neutrófilos en 33 hpf 20,31,32. Las células T entran en circulación aproximadamente 8 días después de la fecundación20,33. Los macrófagos y neutrófilos, como parte de la respuesta inmune innata, han sido implicados en la reducción de la supervivencia de las células BCP-ALL observada tres días después del trasplante en estudios previos21.
La inmunosupresión temporal mediada por morfolinos, dirigida a spi1 y csf3r, inhibe eficazmente la diferenciación de macrófagos y neutrófilos, lo que conduce a un mejor injerto de células BCP-ALL sin afectar la viabilidad del embrión21. Aunque este método no puede lograr un agotamiento permanente, ya que la eliminación completa de spi1 y csf3r es letal, sigue siendo el mejor enfoque para esta tubería.
La calibración de los volúmenes de inyección mediante una retícula y la administración precisa en el saco vitelino en la etapa de una célula garantizan inyecciones consistentes de Morpholino con altas tasas de supervivencia. Alternativas como las inyecciones de clodronato liposomal (Clodrosome) para la depleción de macrófagos se han mostrado prometedoras, pero requieren una mayor validación para esta línea34,35.
Preparación de células
Una población de células suficientemente densa y viable es fundamental para la expansión exitosa de BCP-ALL en este protocolo. CellTrace Violet (CTV) se utiliza para el etiquetado fluorescente para evaluar el éxito de la implantación a 0 ppp y para rastrear las tasas de proliferación a lo largo del experimento. A diferencia de otras etiquetas, el CTV no altera el comportamiento de las células, lo que permite un análisis preciso de la proliferación a nivel de una sola célula. Esto ofrece ventajas sobre la tinción de anticuerpos Ki-67, que solo captura las células durante la proliferación, pero no las células divididas que ya han salido del ciclo celular.
CTV también supera a CellTracker CM-DiI (DiI) en el reflejo de la viabilidad celular. El DiI y sus derivados son fluoróforos más estables, que a menudo persisten más allá de la muerte celular, lo que puede confundir los resultados experimentales2. Además, la inclusión de un anticuerpo específico de BCP-ALL contra CD19 en la citometría de flujo permite una identificación precisa de las células del injerto. Los anticuerpos específicos para humanos, como los anti-HLA, pueden servir como alternativas para otros tipos de célulascancerosas 36.
Trasplante de células cancerosas
El injerto consistente requiere una dilución y concentración óptimas de la suspensión celular. La suspensión debe mantener una densidad suficiente y evitar la viscosidad que perjudique la inyección. Este protocolo prioriza la inyección en la cavidad pericárdica o espacio perivitelino (PVS) sobre el saco vitelino, ya que estos sitios ofrecen una mejor vascularización y menos condiciones hipóxicas37. El trasplante de saco vitelino, aunque accesible, a menudo resulta en altas tasas de mortalidad y poca viabilidad celular21.
La obstrucción de la aguja debido a las micropartículas sigue siendo un desafío de procedimiento. El filtrado de la suspensión celular y la recalibración de los volúmenes de inyección después de recortar las agujas bloqueadas son pasos esenciales. Solo los embriones con pericardio densamente lleno deben ser utilizados para tratamientos farmacológicos posteriores21,36.
La temperatura de incubación sugerida de 35 °C equilibra la temperatura natural de las células cancerosas humanas (37 °C) y la temperatura estándar de la carcasa del pez cebra (28 °C)21. Los embriones de pez cebra se adaptan a esta temperatura con deformaciones mínimas en el desarrollo, y el entorno mejora la proliferación y la supervivencia de las células frescas derivadas de los pacientes38.
Tratamiento farmacológico
Se desarrollaron modelos de xenoinjertos de pez cebra para facilitar el cribado de fármacos de alto rendimiento. Sin embargo, el tratamiento farmacológico sigue siendo uno de los aspectos más desafiantes del ensayo ZefiX. Muchos fármacos estándar y terapias dirigidas no llegan eficazmente a las células de injerto in vivo. También podría requerir la prueba de un panel más grande de concentraciones de drogas. Ejemplos exitosos, como venetoclax y dasatinib, requieren concentraciones significativamente más altas que en los ensayos de cultivo 2D convencionales21.
Por otra parte, el tratamiento previo de las células in vitro antes del trasplante también permite estudiar ciertos efectos sistémicos y localizados. Por ejemplo, este enfoque puede ser adecuado para tratamientos basados en virus adenoasociados (AAV) en el glioblastoma39.
Si se observan efectos del tratamiento farmacológico in vitro pero no in vivo utilizando esta tubería, una alternativa podría ser, por ejemplo, el trasplante a la etapa de células 1k (3 hpf) o la etapa de blástula y el inicio del tratamiento farmacológico a las 24 hpf40,41. Esto podría permitir que los fármacos lleguen a las células del injerto que no tienen éxito en embriones de 48 h de edad o la coinyección de células y fármacos al mismo tiempo25.
Análisis de disociación y citometría de flujo
La disociación del tejido es fundamental para analizar el número total de células del injerto e interpretar de forma fiable los resultados experimentales. Una combinación de disociación mecánica y enzimática garantiza una suspensión unicelular de alta calidad al tiempo que mantiene la integridad de las proteínas de la superficie celular. Es posible que sea necesario ajustar las condiciones de disociación (por ejemplo, la composición de la enzima, el pipeteo o el uso de un homogeneizador Dounce) para diferentes tipos de cáncer.
Las muestras deben filtrarse para evitar la obstrucción del citómetro de flujo, y las proteínas pegajosas o los lípidos pueden mitigarse con EDTA o deyeming de embriones antes de la disociación.
Resumen
El protocolo ZefiX proporciona una línea experimental rápida y rentable para la investigación preclínica del cáncer, los estudios de resistencia a los medicamentos y las evaluaciones personalizadas de tratamientos. Si bien los modelos de xenoinjerto de pez cebra tienen limitaciones y no pueden adaptarse a todos los tipos de fármacos, este protocolo estandarizado permite la expansión in vivo de células y líneas celulares frescas de leucemia en pacientes. Adaptable a otros tipos de cáncer, ofrece una herramienta prometedora para la predicción rápida y personalizada de la respuesta a los fármacos dentro del marco temporal de la toma de decisiones clínicas.
Divulgaciones
Todos los autores declaran no tener conflictos de intereses.
Agradecimientos
Este trabajo ha contado con el apoyo de la Deutsche Forschungsgemeinschaft (DFG) dentro del Centro de Investigación Colaborativa CRC1588, proyecto número 493872418 y la Dr. Kleist Stiftung, Berlín, así como por la Deutsche José Carreras Leukämie Stiftung (R03/2016), la Berliner Krebsgesellschaft (HEFF201633KK) y el Consorcio Alemán del Cáncer (DKTK, Joint Funding Call 2016). Agradecemos a Julia Köppke y Mareike Wolff por su lectura crítica del manuscrito.
Materiales
| Name | Company | Catalog Number | Comments |
| Petri dish (10 cm) | Greiner | P7237 | |
| 7-AAD viability staining solution | Invitrogen | 00-6993-50 | |
| Agarose (LE, analytic grade) | Biozym | 840004 | |
| Air pressure injector | Narishige | IM400 | with external gas supply |
| Alexa Fluor 488 anti-human CD19 antibody | Biolegend | 302219 | |
| Annexin binding buffer | Biolegend | 422201 | Or see solutions for preparation |
| APC annexin V | Biolegend | 640941 | |
| Capillaries (10 cm, OD 1.0 mm, with filaments) | WPIINC | TW100F-4 | 1.0 OD; 0.75 ID |
| Cell culture flask (T-175) | Sarstedt | 83,39,12,002 | |
| CellTrace Violet | Invitrogen | C34557 | |
| Dimethyl sulphoxide (DMSO) | Roth | A994.1 | |
| Dispase II | Sigma Aldrich | D4693-1g | |
| DNase I | AppliChem GmbH | A3778 | |
| Eppendorf tubes (1.5 ml) | Eppendorf | 30120086 | |
| FACS tube (Polystyrene round botton Tube with Cell strainer Cap, 5 ml) | Falcon | 352235 | |
| Falcon tubes (50 ml) | Falcon | 352070 | |
| Fetal calf serum (FCS) | Sigma Aldrich | C8056 | |
| Fine mesh filter (10 µm) | PluriStrainer | 435001050 | |
| Fine mesh filter (20 µm) | PluriStrainer | 431002040 | |
| Flow cytometer | Becton Dickinson | BD LSRFortessa X-20 | |
| Fluorescent stereomicroscope | Leica | ||
| Fluorescent stereomicroscope with camera | Leica | M165 FC | Camera: DFC7000 T |
| Hank’s Balanced Salt Solution (HBSS, Calcium and Magnesium free ) | Sigma Aldrich | 88284 | |
| Injection mold (Zebrafish MI/Transplant KIT) | World Precision Instruments | Z-MOLDS | |
| Injection needles (without filament) | Biomedical instruments | VZIPbl-20-10-55 | Zebrafish injection pipette, blunt, OD: 20μm ± 1, TL:~10mm, PL: 55mm, Glass: BM100T-10P |
| Macro-centrifuge | Eppendorf | ||
| Micro-centrifuge | |||
| Morpholino (csf3r) | Gene Tools LLC | csf3r (GAAGCACAAGCGA GACGGATGCCA) | |
| Morpholino (spi1) | Gene Tools LLC | spi1(GATATACTGATAC TCCATTGGTGGT) | |
| Papain | Sigma Aldrich | P3125 | |
| Penicillin-Streptomycin (Penstrep; 10.000 U/ml) | Gibco | 15140122 | |
| Plates (4-well) | Greiner Bio one | 657160 | |
| Plates (96-well) | Greiner Bio one | 657180 | |
| Roswell Park Memorial Institute (RPMI) 1640 Medium | Gibco | 21875-034 | |
| Tricaine (MS-222) | Sigma Aldrich | E10521-50G | Ethy-3 aminobenzoate methanesulfenate |
Referencias
- Fontana, C. M., Van Doan, H. Zebrafish xenograft as a tool for the study of colorectal cancer: a review. Cell Death Dis. 15, 1-12 (2024).
- Sturtzel, C., et al. Refined high-content imaging-based phenotypic drug screening in zebrafish xenografts. NPJ Precis Oncol. 7 (1), 1-16 (2023).
- Al-Hamaly, M. A., Turner, L. T., Rivera-Martinez, A., Rodriguez, A., Blackburn, J. S. Zebrafish cancer avatars: A translational platform for analyzing tumor heterogeneity and predicting patient outcomes. Int J Mol Sci. 24, 2288(2023).
- Gamble, J. T., Elson, D. J., Greenwood, J. A., Tanguay, R. L., Kolluri, S. K. The zebrafish xenograft models for investigating cancer and cancer therapeutics. Biology. 10 (4), 252(2021).
- Costa, B., Estrada, M. F., Mendes, R. V., Fior, R. Zebrafish avatars towards personalized medicine-A comparative review between avatar models. Cells. 9 (2), 293(2020).
- Wang, W., et al. Progress in building clinically relevant patient-derived tumor xenograft models for cancer research. Animal Model Exp Med. 6 (5), 381-398 (2023).
- Xiao, J., Glasgow, E., Agarwal, S. Zebrafish xenografts for drug discovery and personalized medicine. Trend Cancer. 6 (7), 569-579 (2020).
- Fazio, M., Ablain, J., Chuan, Y., Langenau, D. M., Zon, L. I. Zebrafish patient avatars in cancer biology and precision cancer therapy. Nat Rev Cancer. 20 (5), 263-273 (2020).
- Costa, B., et al. Zebrafish avatar-test forecasts clinical response to chemotherapy in patients with colorectal cancer. Nat Comm. 15 (1), 4771(2024).
- Grissenberger, S., et al. Chapter 8 - Preclinical testing of CAR T cells in zebrafish xenografts. Method Cell Biol. 167, 133-147 (2022).
- Yan, C., et al. Single-cell imaging of T cell immunotherapy responses in vivo. J Exp Med. 218 (10), 20210314(2021).
- Pascoal, S., et al. A preclinical embryonic zebrafish xenograft model to investigate CAR T cells in vivo. Cancers. 12 (3), 567(2020).
- Pal, D., et al. Long-term in vitro maintenance of clonal abundance and leukaemia-initiating potential in acute lymphoblastic leukaemia. Leukemia. 30 (8), 1691-1700 (2016).
- Beneduce, G., et al. Blinatumomab in children and adolescents with relapsed/refractory B cell precursor acute lymphoblastic leukemia: A real-life multicenter retrospective study in seven AIEOP (Associazione Italiana di Ematologia e Oncologia Pediatrica) Centers. Cancers. 14 (2), 426(2022).
- Xie, J., et al. Short-course blinatumomab for refractory/relapse precursor B acute lymphoblastic leukemia in children. Front Pediatr. 11, 1187607(2023).
- Mengxuan, S., Fen, Z., Runming, J. Novel treatments for pediatric relapsed or refractory acute B-cell lineage lymphoblastic leukemia: Precision medicine era. Front Pediatr. 10, 923419(2022).
- Howe, K. The zebrafish reference genome sequence and its relationship to the human genome. Nature. 496 (7446), 498-503 (2013).
- Lee, H. C., Lin, C. Y., Tsai, H. J. Zebrafish, an in vivo platform to screen drugs and proteins for biomedical use. Pharmaceuticals. 14 (6), 500(2021).
- Santoriello, C., Zon, L. I. Hooked! Modeling human disease in zebrafish. J Clin Investigation. 122 (7), 2337-2343 (2012).
- Miao, K. Z., Kim, G. Y., Meara, G. K., Qin, X., Feng, H. Tipping the scales with zebrafish to understand adaptive tumor immunity. Front Cell Dev Biol. 9, 660969(2021).
- Gauert, A., et al. Fast, in vivo model for drug-response prediction in patients with B-cell precursor acute lymphoblastic leukemia. Cancers. 12 (7), 1883(2020).
- Ellett, F., Pase, L., Hayman, J. W., Andrianopoulos, A., Lieschke, G. J. Mpeg1 promoter transgenes direct macrophage-lineage expression in zebrafish. Blood. 117 (4), e49-e56 (2011).
- Pase, L., et al. Neutrophil-delivered myeloperoxidase dampens the hydrogen peroxide burst after tissue wounding in zebrafish. Current Biol. 22 (19), 1818-1824 (2012).
- Wijk, R. C. V., et al. Mechanistic and quantitative understanding of pharmacokinetics in Zebrafish larvae through nanoscale blood sampling and metabolite modeling of paracetamol. J Pharmacol Exp Ther. 371 (1), 15-24 (2019).
- Lázaro-Navarro, J., et al. Inhibiting casein kinase 2 sensitizes acute lymphoblastic leukemia cells to venetoclax via MCL1 degradation. Blood Advances. 5 (24), 5501(2021).
- Rhodes, J., et al. Interplay of pu.1 and gata1 determines myelo-erythroid progenitor cell fate in zebrafish. Developmental Cell. 8 (1), 97-108 (2005).
- Zakaria, Z. Z., Eisa-Beygi, S., Benslimane, F. M., Ramchandran, R., Yalcin, H. C. Design and microinjection of Morpholino antisense oligonucleotides and mRNA into zebrafish embryos to elucidate specific gene function in heart dvelopment. J Vis Exp. (186), e63324(2022).
- ZFIN: Zebrafish Book: Contents. , https://zfin.org/zf_info/zfbook/cont.html (2025).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Dev Dyn. 203 (3), 253-310 (1995).
- Hasegawa, E. H., Gist H Farr, I. I. I., Maves, L. Comparison of pronase versus manual dechorionation of zebrafish embryos for small molecule treatments. J Dev Biol. 11 (2), 16(2023).
- Wattrus, S. J., Zon, L. I. Stem cell safe harbor: the hematopoietic stem cell niche in zebrafish. Blood Adv. 2 (21), 3063-3069 (2018).
- Harvie, E. A., Huttenlocher, A. Neutrophils in host defense: new insights from zebrafish. J Leukocyte Biol. 98 (4), 523-537 (2015).
- Page, D. M., et al. An evolutionarily conserved program of B-cell development and activation in zebrafish. Blood. 122 (8), e1-e11 (2013).
- Nguyen-Chi, M., et al. TNF signaling and macrophages govern fin regeneration in zebrafish larvae. Cell Death Dis. 8 (8), e2979-e2979 (2017).
- Rosowski, E. E. Determining macrophage versus neutrophil contributions to innate immunity using larval zebrafish. Dis Models Mech. 13 (1), dmm041889(2020).
- Rebelo de Almeida, C., et al. Zebrafish xenografts as a fast screening platform for bevacizumab cancer therapy. Comm Biol. 3 (1), 1-13 (2020).
- Pringle, E. S., et al. The zebrafish xenograft platform-A novel tool for modeling KSHV-associated diseases. Viruses. 12 (1), 12(2020).
- Pype, C., et al. Incubation at 32.5 °C and above causes malformations in the zebrafish embryo. Reprod Toxicol. 56, 56-63 (2015).
- Xu, X., et al. Adeno-associated virus (AAV)-based gene therapy for glioblastoma. Cancer Cell Int. 21 (1), 76(2021).
- Siebert, J., et al. Rhabdomyosarcoma xenotransplants in zebrafish embryos. Pediat Blood Cancer. 70 (1), e30053(2023).
- van Bree, N., et al. Development of an orthotopic medulloblastoma zebrafish model for rapid drug testing. Neuro-Oncol. noae210, (2024).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados