Method Article
Aplicación de la interferencia de ARN en el nematodo de la madera de pino, Bursaphelenchus xylophilus
En este artículo
Resumen
Aquí, presentamos un método detallado de remojo de interferencia de ARN en Bursaphelenchus xylophilus para facilitar el estudio de las funciones de los genes.
Resumen
El nematodo de la madera del pino, Bursaphelenchus xylophilus, es una de las especies invasoras más destructivas del mundo, causando el marchitamiento y eventual muerte de los pinos. A pesar del reconocimiento de su importancia económica y ambiental, hasta ahora ha sido imposible estudiar las funciones genéticas detalladas de los nematodos parásitos de plantas (PPN) utilizando la genética directa convencional y los métodos transgénicos. Sin embargo, como tecnología de genética inversa, la interferencia de ARN (ARNi) facilita el estudio de los genes funcionales de los nematodos, incluido B. xylophilus.
Este documento describe un nuevo protocolo para el ARNi del gen ppm-1 en B. xylophilus, que se ha informado que desempeña un papel crucial en el desarrollo y la reproducción de otros nematodos patógenos. Para el ARNi, el promotor T7 se unió al terminal 5' del fragmento objetivo mediante la reacción en cadena de la polimerasa (PCR), y el ARN bicatenario (dsRNA) se sintetizó mediante transcripción in vitro . Posteriormente, la administración de dsRNA se logró empapando los nematodos en una solución de dsRNA mezclada con neuroestimulantes sintéticos. Los juveniles sincronizados de B. xylophilus (aproximadamente 20.000 individuos) se lavaron y remojaron en dsRNA (0,8 μg/mL) en el tampón de remojo durante 24 h en la oscuridad a 25 °C.
La misma cantidad de nematodos se colocó en un tampón de remojo sin dsRNA como control. Mientras tanto, otra cantidad idéntica de nematodos se colocó en un tampón de remojo con el gen dsRNA de la proteína fluorescente verde (gfp) como control. Después del remojo, el nivel de expresión de las transcripciones objetivo se determinó mediante PCR cuantitativa en tiempo real. Los efectos del ARNi se confirmaron mediante la observación microscópica de los fenotipos y una comparación del tamaño corporal de los adultos entre los grupos. El protocolo actual puede ayudar a avanzar en la investigación para comprender mejor las funciones de los genes de B. xylophilus y otros nematodos parásitos hacia el desarrollo de estrategias de control a través de la ingeniería genética.
Introducción
Los nematodos parásitos de plantas (PPN) son una amenaza continua para la seguridad alimentaria y los ecosistemas forestales. Causan un estimado de 100 mil millones de dólares en pérdidas económicas cada año1, los más problemáticos de los cuales son principalmente nematodos del nudo de la raíz, nematodos del quiste y nematodos de la madera del pino. El nematodo de la madera del pino, Bursaphelenchus xylophilus, es un nematodo endoparásito migratorio, que es el patógeno causal de la enfermedad del marchitamiento del pino2. Ha causado un gran daño a los bosques de pinos en todo el mundo3. Usando la terminología de Van Megen et al.4, B. xylophilus es un miembro de Parasitaphelenchidae y pertenece al clado 10, mientras que la mayoría de los otros parásitos principales de plantas pertenecen al clado 12.
Como parásito de plantas independiente y recientemente evolucionado, B. xylophilus es un modelo atractivo para estudios comparativos. Hasta la fecha, ha habido una investigación sustancial sobre los nematodos del nudo de la raíz y los nematodos del quiste pertenecientes al clado 12, que son endoparásitos sedentarios obligados y son algunos de los nematodos más intensamente estudiados. Sin embargo, realizar más investigaciones en esta importante área conlleva un gran desafío: la función de los genes del parasitismo es un cuello de botella de la investigación. Los estudios funcionales generalmente incluyen expresión ectópica y experimentos de knockdown/out, pero se basan en protocolos efectivos de transformación genética para el nematodo. Como resultado, la genética inversa en PPNs se basa casi exclusivamente en el silenciamiento génico por RNAi.
El ARNi, un mecanismo ampliamente presente en las células eucariotas, silencia la expresión génica mediante la introducción de ARN bicatenario (dsRNA)5. Hasta la fecha, el mecanismo de silenciamiento génico posttranscripcional inducido por dsRNA se ha encontrado en todos los eucariotas estudiados, y la tecnología de ARNi, como herramienta de investigación genómica funcional y otras aplicaciones, se ha desarrollado rápidamente en muchos organismos. Desde el descubrimiento de la maquinaria de ARNi en Caenorhabditis elegans en 19986, las técnicas de ARNi se han convertido en métodos eficaces para identificar la función génica de los nematodos y se proponen como una nueva forma de controlar eficazmente los nematodos patógenos7.
El ARNi es técnicamente fácil: empapar los juveniles en dsRNA puede ser suficiente; Sin embargo, la eficacia y reproducibilidad de este enfoque varían ampliamente con las especies de nematodos y el gen diana8. El silenciamiento de 20 genes implicados en las vías de ARNi del nematodo del nudo radicular, Meloidogyne incognita, fue investigado utilizando dsRNAs largos como desencadenantes, resultando en diversas respuestas, incluyendo un aumento y ningún cambio en la expresión de algunos genes9. Estos resultados muestran que los genes diana pueden responder a la eliminación de ARNi de manera diferente, lo que requiere una evaluación exhaustiva de su idoneidad como objetivos para el control de nematodos a través de ARNi. Sin embargo, actualmente hay una escasez de investigación sobre la biología del desarrollo y la reproducción de B. xylophilus.
Como continuación del trabajo anterior10,11,12,13, describimos aquí un protocolo para aplicar RNAi para estudiar la función del gen ppm-1 de B. xylophilus, incluida la síntesis de dsRNA, el remojo de neuroestimulantes sintéticos y la detección cuantitativa de la reacción en cadena de la polimerasa (qPCR). El conocimiento obtenido de este enfoque experimental probablemente contribuirá notablemente a comprender los sistemas biológicos básicos y prevenir la enfermedad del marchitamiento del pino.
Protocolo
El estudio fue aprobado por el consejo de experimentación animal de la Universidad Agrícola y Forestal de Zhejiang. El aislado de B. xylophilus NXY61 fue extraído originalmente de un Pinus massoniana enfermo en el área de Ningbo de la provincia de Zhejiang, China11.
1. Clonación de genes
NOTA: Consulte la Tabla de materiales para obtener detalles sobre los cebadores utilizados en este protocolo.
- Recolectar nematodos.
- Cultivar la cepa de B. xylophilus en el micelio de Botrytis cinerea en placas de agar dextrosa de patata (PDA) a 25 °C durante 3-5 días.
- Recoge los nematodos usando el método del embudo de Bellman14.
- Coloque un tubo de goma sujeto debajo de un embudo y coloque dos capas de papel de filtro en la boca del embudo. Transfiera los cultivos de hongos al embudo y agregue agua para sumergir la estera fúngica. Espere 2 h, luego recoja los nematodos.
- Extraiga el ARN total de los nematodos utilizando un reactivo de extracción de ARN total (consulte la Tabla de materiales)11 de acuerdo con los siguientes pasos.
- Añadir 500 μL de reactivo de extracción y 100 μL de perlas magnéticas a un tubo de centrífuga de 2 ml. Aspirar 20 μL de los nematodos y transferir la muestra a un molinillo para moler a 9.000 × g durante 30 s. Incubar durante 5 min y luego centrifugar durante 10 min a 12.000 × g y 4 °C.
- Transfiera el sobrenadante a un nuevo tubo de centrífuga. Agregue 100 μL de cloroformo, tape el tubo y mezcle invirtiendo el tubo varias veces. Incubar durante 3 min y luego centrifugar durante 10 min a 12.000 × g a 4 °C.
- Transfiera el sobrenadante a un nuevo tubo de centrífuga. Añadir 250 μL de alcohol isopropílico y vortex vigorosamente. Centrifugar a 12.000 × g durante 10 min.
- Deseche el sobrenadante. Agregue 500 μL de etanol al 75% para lavar el ARN y luego vortice la muestra. Centrifugar durante 5 min a 12.000 × g y 4 °C.
- Seque al aire el pellet de ARN durante 5 min.
- Resuspender el pellet en 30 μL de agua libre de RNasa.
- Calcule la concentración de ARN utilizando la fórmula: A260 × dilución × 40 = μg ARN/ml. Calcule la relación A260/A280.
NOTA: Una proporción de ~2 se considera pura.
- Realizar la transcripción inversa de ARN de buena calidad para obtener la plantilla de ADNc.
- Diseñar y utilizar un par de cebadores específicos, ppm-1-F/R (ver la Tabla de materiales), para amplificar la secuencia codificante parcial del gen Bx-ppm-1 en B. xylophilus (número de acceso GenBank QTZ96795).
- Clone las secuencias del gen ppm-1 en el vector pGEM-Teasy que contiene el promotor T7 siguiendo un protocolo de clonación estándar11.
- Configure las reacciones de PCR de la siguiente manera: 2 μL de ADNc, 25 μL de 2x Ex Taq Polymerase Premix, 2 μL de cada imprimación (10 pmol/l) y agua destilada estéril hasta un volumen final de 50 μL.
- Realice el procedimiento de amplificación de la siguiente manera: 5 min a 94 °C; seguido de 35 ciclos de 30 s a 94 °C, 30 s a 55 °C y 1 min a 72 °C; y un paso final de extensión a 72 °C durante 5 min.
- Clone los productos amplificados en un vector pGEM-T Easy para la secuenciación.
2. Síntesis de dsRNA
- Preparar la plantilla de ADN para la síntesis de dsRNA utilizando PCR con cebadores diseñados para agregar sitios promotores T7 en ambos extremos. Agregue la secuencia del promotor T7 al extremo 5' de los cebadores.
- Utilizar el plásmido que contiene el fragmento del gen ppm-1 (894 pb) como plantilla para la PCR y recuperar el fragmento que contiene el promotor T711. Utilice el procedimiento y el sistema de PCR descritos anteriormente.
- Utilice un kit de transcripción in vitro para sintetizar dsRNA11.
- Descongele los reactivos congelados en hielo.
- Agregue 2 μL de tampón de reacción 10x, 2 μL de mezcla enzimática y 1 μg de ADN a un tubo de centrífuga. Agregue agua libre de nucleasas para producir una reacción estándar de 4 μL. Luego, mezcle volúmenes iguales de las cuatro soluciones de ribonucleótidos (ATP, CTP, GTP y UTP) y agregue 8 μL de la mezcla al tubo. Mezclar bien e incubar a 37 °C durante 4 h.
- Añadir 1 μL de DNasa, mezclar bien e incubar durante 15 min a 37 °C.
- Detenga la reacción y agregue 30 μL de agua libre de nucleasa y 30 μL de solución de precipitación de LiCl para precipitar el ARN. Homogeneizar. Incubar a -20 °C durante la noche.
- Centrifugar durante 15 min a 12.000 × g y 4 °C. Deseche el sobrenadante.
- Agregue 1 ml de etanol al 75% para lavar el ARN. Vortex la muestra y centrifugarla durante 10 min a 12.000 × g y 4 °C.
- Seque al aire el pellet de ARN durante 3 min.
- Resuspender el pellet en 30 μL de agua libre de RNasa.
- Analice la calidad del dsRNA utilizando un espectrofotómetro. Pipetear 1 μL de la muestra de dsRNA en el pedestal de medición y ajustar la longitud de onda a 340 nm. Visualice los productos en un gel de agarosa al 1,0%.
3. ARNi por remojo
- Mezclar 4 μL de 5x tampón de remojo (0,05% gelatina, 5,5 mM KH 2 PO 4,2,1mM NaCl, 4,7 mM NH4 Cl, 3 mM espermidina) con el dsRNA yddH2Opara producir un volumen total de 20 μL y una concentración final de ARN de 0,8 μg/mL.
- Adquirir larvas J2.
- Recoger los nematodos de los cultivos fúngicos y transferirlos a una placa de Petri de vidrio de 6 cm de diámetro. Agregue 10 ml de agua al plato para asegurarse de que los nematodos puedan nadar libremente. Mantenga los nematodos en el plato durante 30 minutos y espere a que los huevos se adhieran al fondo.
- Retire el agua y los nematodos con cuidado, asegurándose de no molestar a los huevos. Repita los pasos hasta que se eliminen todas las larvas y adultos, dejando solo los huevos en el plato.
- Incubar los huevos recolectados durante 24 h en la oscuridad a 25 °C para obtener larvas J2. Recolectar las larvas J2, colocarlas en un tubo y lavarlas tres veces conddH2Opara el experimento RNAi15.
- Transfiera las larvas J2 al tubo de 2 ml que contiene la solución de dsRNA y agregue la solución de resorcinol (envuelta en papel de estaño y disuelta en agua) para producir una concentración final de 1.0%. Incubar las larvas con centrifugación a 15 × g en una mesa agitadora durante 24 h a 25 °C para asegurarse de que las larvas absorben eficazmente el dsRNA.
- Remoje la misma cantidad de nematodos en el tampón de remojo sin la sonda dsRNA o con una sonda GFP dsRNA como control. Utilice el gen GFP (gfp, M62653.1) como control no endógeno y sintetice el dsRNA de gfp utilizando cebadores específicos del gen T7-GFP-F/R.
4. Detección de qPCR
- Limpie las larvas J2 conddH2O, incluidas aquellas con interferencia del gen diana, interferencia del gen GFP y el grupo de control no perturbado. Extraiga el ARN total de cada grupo utilizando el método descrito anteriormente.
- Inicie la qPCR.
- Diseñe los cebadores q-ppm-1-F/R utilizando el software deseado (consulte la Tabla de materiales).
- Configure la reacción de qPCR en 12 μL que contengan 1 μL de ADNc, 6 μL de premezcla fluorescente, 0,4 μL de cada cebador (10 pmol/l) y agua destilada estéril.
- Realizar qPCR de la siguiente manera: 2 min a 95 °C; seguido de 40 ciclos de 10 s a 95 °C, 30 s a 55 °C y 1 min a 72 °C.
- Utilice el gen actb (número de acceso GenBank EU100952) y el gen tbb-2 (número de acceso GenBank MT769316), u otros genes, como genes de referencia interna para evaluar los cambios en el nivel de expresión génica después de RNAi15.
- De acuerdo con el valor del umbral del ciclo (Ct) y la curva de disolución, utilice el método 2-ΔΔCt para estimar el nivel de expresión relativa del gen objetivo y verificar la eficiencia de interferencia.
- Reste el valor de Ct del gen de referencia interno de cada muestra del valor de Ct del gen diana para obtener el valor de ΔCt. Luego, reste el valor ΔCt del grupo de interferencia del valor ΔCt del grupo de control para obtener el valor ΔΔCt.
NOTA: Un valor ΔΔCt mayor que 0 indica que la interferencia es efectiva.
- Reste el valor de Ct del gen de referencia interno de cada muestra del valor de Ct del gen diana para obtener el valor de ΔCt. Luego, reste el valor ΔCt del grupo de interferencia del valor ΔCt del grupo de control para obtener el valor ΔΔCt.
5. Evaluar la longitud corporal de los nematodos adultos siguiendo el ARNi
- Después del RNAi, cultivar las larvas J2 hasta la edad adulta en céspedes de B. cinerea en placas de PDA durante 60 h a 25 °C15.
- Recoja a los adultos utilizando el método del embudo de Bellman (consulte el paso 1.1.2.1)14.
- Adquiera imágenes de los nematodos adultos bajo un microscopio y use el software ImageJ (u otro software de medición) para medir la longitud del cuerpo.
- Mida la longitud con ImageJ seleccionando Analizar | Establecer escala. Si se conoce la distancia, introduzca el valor de longitud de la línea recta dibujada. Introduzca la unidad de longitud.
- Marque la casilla de verificación Global (use este estándar para todas las imágenes) y haga clic en Aceptar para seleccionar la longitud que se va a medir con una línea recta en la imagen que se va a medir.
- Utilice el comando Ctrl+M (medición) para mostrar los resultados y registrarlos en la ventana de resultados .
- Tomar medidas de 50 nematodos machos y 50 hembras para el análisis estadístico12.
- Analice los datos calculando la media y la desviación estándar para cada muestra. Comparar las medias de las muestras de los diferentes grupos utilizando la prueba t de Student.
Resultados
Análisis de la expresión de ppm-1 de B. xylophilus después de RNAi
El nivel de expresión relativo del gen ppm-1 de B. xylophilus empapado con GFP dsRNA y el empapado con el gen diana dsRNA fue de 0,92 y 0,52, respectivamente (el nivel de expresión génica ppm-1 del grupo control tratado conddH2O se estableció en 1) (Figura 1). Por lo tanto, el dsRNA exógeno no tiene ningún efecto sobre la expresión de ppm-1 de B. xylophilus; sin embargo, ppm-1 dsRNA puede inhibir eficazmente la expresión del gen diana.
Efecto de la expresión de ppm-1 en el crecimiento y desarrollo de B. xylophilus
Después del ARNi, el tamaño de los adultos disminuyó notablemente (Figura 2), lo que resultó en el fenotipo mutante SMA (tamaño corporal pequeño). Aunque los individuos tratados con ARNi desarrollaron hasta la madurez sexual, su longitud corporal fue sustancialmente menor que la de los adultos normales. Específicamente, después del RNAi en nematodos en etapa J2, la longitud corporal media de hembras y machos fue de 544.61 μm y 526.24 μm, respectivamente. En contraste, la longitud corporal media de hembras y machos en el grupo control fue de 971,86 μm y 912,31 μm, respectivamente (Figura 3), representando diferencias significativas (P = 0,0322).
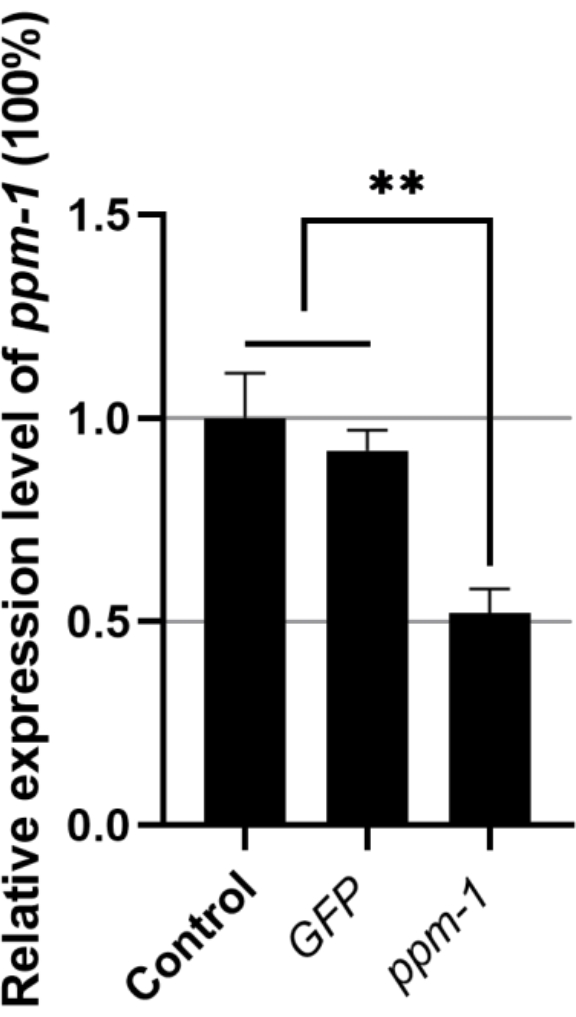
Figura 1: Expresión del gen ppm-1 siguiendo el RNAi de Bursaphelenchus xylophilus. **P < 0,001. Abreviaturas: RNAi = ARN de interferencia; GFP = proteína verde fluorescente. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Reducción de la longitud corporal de adultos después de la interferencia de ppm-1 en Bursaphelenchus xylophilus. Imágenes de un macho adulto (A) y una hembra (C) del grupo RNAi. Imágenes de un macho adulto (B) y una hembra (D) del grupo control. Barras de escala = 50 μm. Abreviatura: RNAi = ARN de interferencia. Haga clic aquí para ver una versión más grande de esta figura.
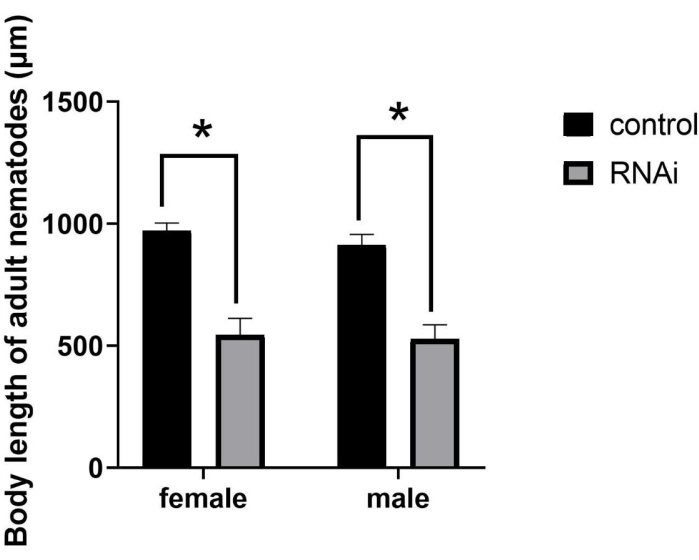
Figura 3: Cuantificación y análisis estadístico de la longitud corporal de adultos de Bursaphelenchus xylophilus tras RNAi del gen ppm-1 . (*P = 0,0322). Abreviatura: RNAi = ARN de interferencia. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Aunque la historia de vida y el entorno parasitario de B. xylophilus son diferentes de los de otros nematodos, ha habido investigaciones limitadas sobre la patogénesis molecular de este patógeno vegetal. A pesar de los grandes avances realizados en la aplicación de la tecnología de edición del genoma CRISPR/Cas9 en C. elegans y otros nematodos, hasta la fecha sólo se ha publicado la tecnología RNAi aplicada a B. xylophilus 17. El ARNi es una de las herramientas más poderosas disponibles para estudiar la función génica de los nematodos y ha sido ampliamente utilizada en investigaciones que aclaran la función del gen B. xylophilus, las vías de transducción de señales y la terapia génica18,19,20. En contraste con los enfoques de eliminación de genes utilizados en modelos de invertebrados, la eliminación mediada por RNAi de genes objetivo en nematodos permite la evaluación rápida de la función génica.
Hay tres formas de conducir el ARNi en C. elegans: inyección 6, remojo21 y alimentación6. Debido a que las larvas J2 solo se alimentan después de la infección, el método de remojo se usa típicamente para el ARNi de los nematodos parásitos de plantas. El paso clave en el método de remojo es agregar un agente nervioso para estimular a los nematodos a alimentarse. Urwin utilizó por primera vez la octopamina para estimular la J2 de dos especies de nematodos del quiste oral, Globodera palida y Heterodera glycines, lo que resultó en la absorción de dsRNA de la solución de remojo22. El mismo método se ha utilizado con éxito para inducir a J2 del nematodo del nudo de la raíz Meloidogyne incognita a absorber dsRNA 23. El resorcinol también puede inducir la captación de dsRNA por J2 de M. incognita y puede ser más eficaz que la octopamina para este nematodo24. Además, la adición de espermidina al tampón de remojo con un tiempo de incubación prolongado mejoró la eficiencia del ARNi en nematodos25. Después de 24 h de remojo, dsRNA entra efectivamente en B. xylophilus, silenciando así el gen ppm-1.
Este protocolo, por lo tanto, proporciona una referencia para el estudio futuro del ARNi de otros nematodos parásitos de plantas con comportamiento de fagocitosis. Además, el efecto de suspensión de la mesa de agitación juega un papel importante para maximizar la ventaja del método de remojo. Este método puede permitir el tratamiento simultáneo de una gran cantidad de nematodos. El ARNi es más fácil de operar cuando se centra en los genes diana para los que no se pueden obtener fácilmente mutantes y para evaluar los efectos en diferentes puntos del desarrollo porque los nematodos pueden empaparse en cualquier etapa de la vida. Por lo tanto, el ARNi se puede utilizar para estudiar la función de un gen específico en una etapa específica del desarrollo. Wang et al. analizaron la influencia de MAPK en la fecundidad de B. xylophilus y su importante papel en el crecimiento y desarrollo de nematodos utilizando el remojo de dsRNA26. Qiu et al. encontraron que la velocidad de migración y la fecundidad de B. xylophilus en pinos disminuyó después de la regulación negativa del gen Bxpel1, lo que indica que este gen es un factor patogénico importante de B. xylophilus27.
Sin embargo, no todo el ARNi en los nematodos funciona a través del dsRNA. Dulovic y Streit aplicaron pequeños ARN interferentes (siRNAs) en lugar de los dsRNAs más largos para interferir con éxito con Strongyloides ratti28. La limitación del uso de dsRNA para RNAi en Strongyloides puede estar relacionada con la ausencia de genes como rsd-6, sid-1 o sid-2 que se sabe que están involucrados en la absorción de dsRNA. El ARNi también se asocia con las desventajas de un efecto posicional y un knockout temporal e incompleto y tiene una efectividad limitada para algunos genes y tipos de células (como las neuronas)29. Sin embargo, hasta que se logre un gran avance en la tecnología transgénica de B. xylophilus , el ARNi representa una estrategia de investigación relativamente efectiva.
El ARNi ha demostrado un gran potencial para la protección de cultivos. Usando la tecnología de ARNi, se han criado papas transgénicas capaces de producir dsRNA que se dirige a los genes de nematodos de nudo de raíz para producir resistencia completa a los nematodos del nudo de la raíz30. La expresión de dsRNAs que se dirigen a genes de insectos puede proporcionar protección a los cultivos en ausencia de insecticidas químicos y ofrece la ventaja adicional de no producir proteínas extrañas, con un número casi ilimitado de genes diana31. Por lo tanto, la investigación acelerada en el campo del ARNi aplicado para el control de plagas proporcionará una mejor bioseguridad para el control de nematodos de plantas que los métodos transgénicos u otros métodos de control químico.
En conclusión, este protocolo describe la preparación de dsRNA del gen ppm-1 para lograr RNAi empapando directamente las larvas de B. xylophilus en solución de dsRNA. El efecto de interferencia se confirmó en base a una reducción significativa en el tamaño corporal de las larvas después de que se convirtieron en adultos en comparación con el del control, lo que demuestra que el gen ppm-1 ejerce efectos sobre el crecimiento y desarrollo de B. xylophilus. Este estudio proporciona una base teórica para revelar aún más la función de ppm-1 con valor práctico adicional para guiar el control biológico de este parásito vegetal. Se cree que con la mayor divulgación y mejora del mecanismo de la tecnología RNAi, su aplicación en el campo del control de B. xylophilus tendrá amplias perspectivas.
Divulgaciones
No se declararon conflictos de intereses.
Agradecimientos
Esta investigación fue financiada por la Fundación Nacional de Ciencias Naturales de China (31870637, 31200487) y financiada conjuntamente por el Plan de Investigación Clave de Zhejiang (2019C02024, LGN22C160004).
Materiales
| Name | Company | Catalog Number | Comments |
| Baermann funnel | n/a | n/a | to isolate nematodes |
| Beacon Designer 7.9 | Shanghai kangyusheng information technology co. | n/a | to design qPCR primers |
| Botrytis cinerea | n/a | n/a | as food for nematodes |
| Bursaphelenchus xylophilus | n/a | n/a | its number was NXY61 and was it was originally extracted from diseased Pinus massoniana in Ningbo, Zhejiang province, China. |
| constant temperature incubator | Shanghai Jing Hong Laboratory Instrument Co. | H1703544 | to cultur nematodes |
| Electrophoresis apparatus | Bio-Rad Laboratories | 1704466 | to achieve electrophoretic analysis |
| Ethanol, 75% | Sinopharm Chemical Reagent Co. | 80176961 | to extract RNA |
| Ex Taq Polymerase Premix | Takara Bio Inc. | RR030A | for PCR |
| Ex Taq Polymerase Premix | Takara Bio Inc. | RR390A | for PCR |
| Gel imager | LongGene Scientific Instruments Co. | LG2020 | to make nucleic acid bands visible |
| GraphPad Prism 8 | GraphPad Prism | n/a | to analyze the data and make figurs |
| High Speed Centrifuge | Hangzhou Allsheng Instruments Co. | AS0813000 | centrifug |
| High-flux tissue grinder | Bertin | to extract RNA | |
| ImageJ software | National Institutes of Health | n/a | to measure the body lengths |
| isopropyl alcohol | Shanghai Aladdin Biochemical Technology Co. | L1909022 | to extract RNA |
| Leica DM4B microscope | Leica Microsystems Inc. | to observe nematodes | |
| magnetic beads | Aoran science technology co. | 150010C | to extract RNA |
| MEGAscript T7 High Yield Transcription Kit | Thermo Fisher Scientific Inc. | AM1333 | to synthesize dsRNA in vitro |
| NanoDrop ND-2000 spectrophotometer | Thermo Fisher Scientific Inc. | NanoDrop 2000/2000C | to analyze the quality of the dsRNA |
| PCR Amplifier | Bio-Rad Life Medical Products Co. | 1851148 | to amplify nucleic acid sequence |
| Petri dishes | n/a | n/a | to cultur nematodes |
| pGEM-T Easy vector | Promega Corporation | A1360 | for cloning |
| Potato Dextrose Agar (Medium) | n/a | n/a | to cultur Botrytis cinerea |
| Prime Script RT reagent Kit with gDNA Eraser | Takara Bio Inc. | RR047B | to synthetic cDNA |
| Primer Premier 5.0 | PREMIER Biosoft | n/a | to design PCR primers |
| primers:ppm-1-F/R | Tsingke Biotechnology Co. | n/a | F: 5'-GATGCGAAGTTGCCAATCATTCT -3'; R: 5'- CCAGATCCAGTCCACCATACACC -3 |
| q-ppm-1-F/R | Tsingke Biotechnology Co. | n/a | F: 5'-CATCCGAATGGCAATACAG-3'; R: 5'-ACTATCCTCAGCGTTAGC-3' |
| Real-time thermal cycler qTOWER 2.2 | Analytique Jena Instruments (Beijing) Co. | for qPCR | |
| shaking table | Shanghai Zhicheng analytical instrument manufacturing co. | to soak nematodes | |
| stereoscopic microscope | Chongqing Optec Instrument Co. | 1814120 | to observe nematodes |
| T7-GFP-F/R | Tsingke Biotechnology Co. | n/a | F: 5'-TAATACGACTCACTATAGGGAAA GGAGAAGAACTTTTCAC-3'; R: 5'-TAATACGACTCACTATAGGGCTG TTACAAACTCAAGAAGG-3' |
| T7 promoter | Tsingke Biotechnology Co. | n/a | TAATACGACTCACTATAGGG |
| Takara MiniBEST Agarose Gel DNA Extraction Kit | Takara Bio Inc. | 9762 | to recover DNA |
| TaKaRa TB Green Premix Ex Taq (Tli RNaseH Plus) | Takara Bio Inc. | RR820A | for qPCR |
| trichloroethane | Shanghai LingFeng Chemical Reagent Co. | to extract RNA | |
| TRIzol Reagent | Thermo Fisher Scientific Inc. | 15596026 | total RNA extraction reagent,to extract RNA |
Referencias
- Nicol, J. M., Jones, J., Gheysen, G., Fenoll, C., et al. Current nematode threats to world agriculture. Genomics and Molecular Genetics of Plant-Nematode Interactions. , 21-43 (2011).
- Jones, J. T., et al. Top 10 plant-parasitic nematodes in molecular plant pathology. Molecular Plant Pathology. 14 (9), 946-961 (2013).
- Kikuchi, T., et al. Genomic insights into the origin of parasitism in the emerging plant pathogen Bursaphelenchus xylophilus. PLoS Pathogens. 7 (9), 1002219 (2011).
- Megen, H. V., et al. A phylogenetic tree of nematodes based on about 1200 full-length small subunit ribosomal DNA sequences. Nematology. 11 (6), 927-950 (2009).
- Niu, J. H., Jian, H., Xu, J. M., Guo, Y. D., Liu, Q. RNAi technology extends its reach: Engineering plant resistance against harmful eukaryotes. African Journal of Biotechnology. 9 (45), 7573-7582 (2010).
- Fire, A., et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 91 (6669), 806-811 (1998).
- Shahid, M., Imran, A., Mazhar, H., Yusuf, Z., Rob, W. B. Engineering novel traits in plants through RNA interference. Trends in Plant Science. 11 (11), 559-565 (2006).
- Marmonier, A., et al. In vitro acquisition of specific small interfering RNAs inhibits the expression of some target genes in the plant ectoparasite Xiphinema index. International Journal of Molecular Sciences. 20 (13), 3266 (2019).
- Iqbal, S., Fosu-Nyarko, J., Jones, M. G. K. Attempt to silence genes of the RNAi pathways of the root-knot nematode, Meloidogyne incognita results in diverse responses including increase and no change in expression of some genes. Frontiers in Plant Science. 11, 328 (2020).
- Zhou, L. F., et al. Molecular characterization and functional analysis of akt-1 in pinewood nematode, Bursaphelenchus xylophilus. Forest Pathology. 51 (1), 12647 (2021).
- Zhou, L. F., et al. The role of mab-3 in spermatogenesis and ontogenesis of pinewood nematode, Bursaphelenchus xylophilus. Pest Management Science. 77 (1), 138-147 (2021).
- Tang, J., et al. Bxy-fuca encoding α-L-fucosidase plays crucial roles in development and reproduction of the pathogenic pinewood nematode, Bursaphelenchus xylophilus. Pest Management Science. 76 (1), 205-214 (2020).
- Wang, J. H., et al. Molecular characterization and functional analysis of daf-8 in the pinewood nematode, Bursaphelenchus xylophilus. Journal of Forestry Research. , (2021).
- Viglierchio, D. R., Schmitt, R. V. On the methodology of nematode extraction from field samples: Baermann funnel modifications. Journal of Nematology. 15 (3), 438-444 (1983).
- Zhu, N., et al. Observation and quantification of mating behavior in the pinewood nematode, Bursaphelenchus xylophilus. Journal of Visualized Experiments: JoVE. (118), e54842 (2016).
- Zhou, L. F., Chen, F. M., Ye, J. R., Pan, H. Y. Selection of reliable reference genes for RT-qPCR analysis of Bursaphelenchus mucronatus gene expression from different habitats and developmental stages. Frontiers in Genetics. 9, 269-279 (2018).
- Wang, M., et al. Double-stranded RNA-mediated interference of dumpy genes in Bursaphelenchus xylophilus by feeding on filamentous fungal transformants. International Journal for Parasitology. 46 (5-6), 351-360 (2016).
- Ma, H. B., Lu, Q., Liang, J., Zhang, X. Y. Functional analysis of the cellulose gene of the pine wood nematode, Bursaphelenchus xylophilus, using RNA interference. Genetics and Molecular Research: GMR. 10 (3), 1931-1941 (2011).
- Cheng, X. Y., Dai, S. M., Xiao, L., Xie, B. Y. Influence of cellulase gene knock down by dsRNA interference on the development and reproduction of the pine wood nematode, Bursaphelenchus xylophilus. Nematology. 12 (12), 225-233 (2010).
- Xue, Q., Wu, X. Q., Zhang, W. J., Deng, L. N., Wu, M. M. Cathepsin L-like cysteine proteinase genes are associated with the development and pathogenicity of pine wood nematode, Bursaphelenchus xylophilus. International Journal of Molecular Sciences. 20 (1), 215 (2019).
- Tabara, H., Grishok, A., Mello, C. C. RNAi in C. elegans: Soaking in the genome sequence. Science. 282 (5388), 430-431 (1998).
- Urwin, P. E., Lilley, C. J., Atkinson, H. J. Ingestion of double-stranded RNA by pre parasitic juvenile cyst nematodes leads to RNA interference. Molecular Plant-Microbe Interactions: MPMI. 15 (8), 747-752 (2002).
- Bakhetia, M., Charlton, W., Atkinson, H. J., McPherson, M. J. RNA interference of dual oxidase in the plant nematode Meloidogyne incognita. Molecular Plant-Microbe Interactions: MPMI. 18 (10), 1099-1106 (2005).
- Rosso, M. N., Dubrana, M. P., Cimbolini, N., Jaubert, S., Abad, P. Application of RNA interference to root-knot nematode genes encoding esophageal gland proteins. Molecular Plant-Microbe Interactions: MPMI. 18 (7), 615-620 (2005).
- Chen, Q., Rehman, S., Smant, G., Jones, J. T. Functional analysis of pathogenicity proteins of the potato cyst nematode Globodera rostochiensis using RNAi. Molecular Plant-Microbe Interactions: MPMI. 18 (7), 621-625 (2005).
- Wang, D. D., Li, Y., Li, J., Xie, B. Y., Chen, G. H. Molecular clone and its RNAi interference effect analysis of mapk gene in Bursaphelenchus xylophilus ( in Chinese). Acta Phytopathologica Sinica. 46 (5), 662-669 (2016).
- Qiu, X., Wu, X., Huang, L., Ye, J. R. Influence of Bxpel1 gene silencing by dsRNA interference on the development and pathogenicity of the pine wood nematode, Bursaphelenchus xylophilus. International Journal of Molecular Sciences. 17 (1), 125 (2016).
- Dulovic, A., Streit, A. RNAi-mediated knockdown of daf-12 in the model parasitic nematode Strongyloides ratti. PLoS Pathogens. 15 (3), 1007705 (2019).
- Li, L., Zhao, H., Cui, Y., Wei, H., Li, M. Research progress of gene editing technology. Life Science Research. 21 (3), 268-274 (2017).
- Bindhya, C. Y., Karuppannan, V., Kuppuswamy, S. Host-generated double stranded RNA induces RNAi in plant-parasitic nematodes and protects the host from infection. Molecular and Biochemical Parasitology. 148 (2), 219-222 (2006).
- Jiang, Z., Sher, A. K., David, G. H., Ralph, B. Next-generation insect-resistant plants: RNAi-mediated crop protection. Trends in Biotechnology. 35 (9), 871-882 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados