Method Article
Anwendung von RNA-Interferenz im Kiefernnematoden, Bursaphelenchus xylophilus
In diesem Artikel
Zusammenfassung
Hier stellen wir eine detaillierte Einweichmethode der RNA-Interferenz in Bursaphelenchus xylophilus vor, um die Untersuchung von Genfunktionen zu erleichtern.
Zusammenfassung
Der Kiefernfadenwurm, Bursaphelenchus xylophilus, ist eine der zerstörerischsten invasiven Arten weltweit, die das Welken und schließlich das Absterben von Kiefern verursacht. Trotz der Anerkennung ihrer ökonomischen und ökologischen Bedeutung war es bisher unmöglich, die detaillierten Genfunktionen von pflanzenparasitischen Nematoden (PPNs) mit konventioneller Vorwärtsgenetik und transgenen Methoden zu untersuchen. Als umgekehrte genetische Technologie erleichtert die RNA-Interferenz (RNAi) jedoch die Untersuchung der funktionellen Gene von Nematoden, einschließlich B. xylophilus.
Dieser Artikel skizziert ein neues Protokoll für RNAi des ppm-1-Gens in B. xylophilus, von dem berichtet wurde, dass es eine entscheidende Rolle bei der Entwicklung und Reproduktion anderer pathogener Nematoden spielt. Für RNAi wurde der T7-Promotor durch Polymerase-Kettenreaktion (PCR) mit dem 5′-Terminal des Zielfragments verknüpft, und doppelsträngige RNA (dsRNA) wurde durch In-vitro-Transkription synthetisiert. Anschließend wurde die dsRNA-Abgabe erreicht, indem die Nematoden in einer dsRNA-Lösung mit synthetischen Neurostimulanzien eingeweicht wurden. Synchronisierte Jungtiere von B. xylophilus (ca. 20.000 Individuen) wurden gewaschen und im Einweichpuffer für 24 h im Dunkeln bei 25 °C in dsRNA (0,8 μg/ml) eingeweicht.
Die gleiche Menge an Nematoden wurde in einen Einweichpuffer ohne dsRNA als Kontrolle gegeben. In der Zwischenzeit wurde eine weitere identische Menge von Nematoden in einen Einweichpuffer mit dem grün fluoreszierenden Protein (gfp) -Gen dsRNA als Kontrolle gegeben. Nach dem Einweichen wurde das Expressionsniveau der Zieltranskripte mittels quantitativer real-time PCR bestimmt. Die Wirkung von RNAi wurde dann durch mikroskopische Beobachtung der Phänotypen und einen Vergleich der Körpergröße der Erwachsenen unter den Gruppen bestätigt. Das aktuelle Protokoll kann dazu beitragen, die Forschung voranzutreiben, um die Funktionen der Gene von B. xylophilus und anderen parasitären Nematoden besser zu verstehen, um Kontrollstrategien durch Gentechnik zu entwickeln.
Einleitung
Pflanzenparasitäre Nematoden (PPNs) sind eine anhaltende Bedrohung für die Ernährungssicherheit und die Waldökosysteme. Sie verursachen jedes Jahr schätzungsweise 100 Milliarden USD an wirtschaftlichen Verlusten1, von denen die problematischsten vor allem Wurzelknotennematoden, Zystennematoden und Kiefernnematoden sind. Der Kiefernfadenwurm, Bursaphelenchus xylophilus, ist ein wandernder, endoparasitischer Fadenwurm, der der ursächliche Erreger der Kiefernwelkeist 2. Es hat den Kiefernwäldern weltweit großen Schaden zugefügt3. Nach der Terminologie von Van Megen et al.4 ist B. xylophilus ein Mitglied der Parasitaphelenchidae und gehört zur Klade 10, während die meisten anderen wichtigen Pflanzenparasiten zur Klade 12 gehören.
Als eigenständiger und neu entwickelter Pflanzenparasit ist B. xylophilus ein attraktives Modell für vergleichende Studien. Bis heute gibt es umfangreiche Forschungen über Wurzelknotennematoden und Zystennematoden der Klade 12, die obligate, sitzende Endoparasiten sind und zu den am intensivsten untersuchten Nematoden gehören. Die weitere Forschung auf diesem wichtigen Gebiet ist jedoch mit einer großen Herausforderung verbunden: Die Funktion von Parasitengenen ist ein Forschungsengpass. Funktionelle Studien umfassen im Allgemeinen ektopische Expressions- und Knockdown- / Out-Experimente, stützen sich jedoch auf effektive genetische Transformationsprotokolle für den Nematoden. Infolgedessen beruht die umgekehrte Genetik in PPNs fast ausschließlich auf Gen-Silencing durch RNAi.
RNAi, ein Mechanismus, der in eukaryotischen Zellen weit verbreitet ist, bringt die Genexpression zum Schweigen, indem er doppelsträngige RNA (dsRNA) einführt5. Bis heute wurde der durch dsRNA induzierte posttranskriptionelle Gen-Silencing-Mechanismus in allen untersuchten Eukaryoten gefunden, und die RNAi-Technologie als Werkzeug der funktionellen Genomforschung und anderer Anwendungen hat sich in vielen Organismen schnell entwickelt. Seit der Entdeckung der RNAi-Maschinerie in Caenorhabditis elegans im Jahr 19986 haben sich RNAi-Techniken zu wirksamen Methoden zur Identifizierung der Genfunktion von Nematoden entwickelt und werden als neuer Weg zur wirksamen Kontrolle pathogener Nematoden vorgeschlagen7.
RNAi ist technisch das Einweichen der Jungtiere in dsRNA kann ausreichen; Die Wirksamkeit und Reproduzierbarkeit dieses Ansatzes variiert jedoch stark mit der Nematodenspezies und dem Zielgen8. Die Stummschaltung von 20 Genen, die an den RNAi-Signalwegen des Wurzelknotennematoden, Meloidogyne incognita, beteiligt sind, wurde unter Verwendung langer dsRNAs als Auslöser untersucht, was zu verschiedenen Reaktionen führte, einschließlich einer Zunahme und keiner Veränderung der Expression einiger Gene9. Diese Ergebnisse zeigen, dass Zielgene unterschiedlich auf RNAi-Knockdown reagieren können, was eine umfassende Bewertung ihrer Eignung als Ziele für die Nematodenkontrolle über RNAi erfordert. Derzeit gibt es jedoch einen Mangel an Forschung zur Entwicklungs- und Fortpflanzungsbiologie von B. xylophilus.
Als Fortsetzung der früheren Arbeit10,11,12,13 beschreiben wir hier ein Protokoll zur Anwendung von RNAi zur Untersuchung der Funktion des ppm-1-Gens von B. xylophilus, einschließlich der Synthese von dsRNA, des Einweichens synthetischer Neurostimulanzien und des quantitativen Polymerase-Kettenreaktionsnachweises (qPCR). Die Erkenntnisse aus diesem experimentellen Ansatz werden wahrscheinlich deutlich zum Verständnis grundlegender biologischer Systeme und zur Vorbeugung von Kiefernwelke beitragen.
Protokoll
Die Studie wurde vom Rat für Tierversuche der Zhejiang Agricultural & Forestry University genehmigt. Das B. xylophilus-Isolat NXY61 wurde ursprünglich aus einem erkrankten Pinus massoniana in der Region Ningbo in der Provinz Zhejiang, Chinaextrahiert 11.
1. Klonen von Genen
HINWEIS: Weitere Informationen zu den in diesem Protokoll verwendeten Grundierungen finden Sie in der Materialtabelle .
- Sammle Nematoden.
- Kultur des B. xylophilus-Stammes auf den Myzelien von Botrytis cinerea auf Kartoffel-Dextrose-Agar (PDA)-Platten bei 25 °C für 3-5 Tage.
- Sammeln Sie die Nematoden mit der Bellman-Trichtermethode14.
- Legen Sie einen eingespannten Gummischlauch unter einen Trichter und legen Sie zwei Schichten Filterpapier in den Mund des Trichters. Die Pilzkulturen in den Trichter geben und Wasser hinzufügen, um die Pilzmatte einzutauchen. Warten Sie 2 Stunden und sammeln Sie dann die Nematoden.
- Extrahieren Sie die gesamte RNA aus den Nematoden mit einem Gesamt-RNA-Extraktionsreagenz (siehe Materialtabelle)11 gemäß den folgenden Schritten.
- Geben Sie 500 μL Extraktionsreagenz und 100 μL Magnetperlen in ein 2 ml Zentrifugenröhrchen. 20 μL der Nematoden aspirieren und die Probe zum Mahlen bei 9.000 × g für 30 s in ein Mahlwerk geben. 5 min inkubieren und dann 10 min bei 12.000 × g und 4 °C zentrifugieren.
- Den Überstand in ein neues Zentrifugenröhrchen überführen. Fügen Sie 100 μL Chloroform hinzu, verschließen Sie das Röhrchen und mischen Sie, indem Sie das Röhrchen mehrmals umdrehen. 3 min inkubieren und dann 10 min bei 12.000 × g bei 4 °C zentrifugieren.
- Den Überstand in ein neues Zentrifugenröhrchen überführen. Fügen Sie 250 μL Isopropylalkohol hinzu und wirbeln Sie kräftig. Zentrifugieren bei 12.000 × g für 10 min.
- Verwerfen Sie den Überstand. Fügen Sie 500 μL 75% Ethanol hinzu, um die RNA zu waschen und dann die Probe zu wirbeln. Zentrifugieren Sie es für 5 min bei 12.000 × g und 4 °C.
- Trocknen Sie das RNA-Pellet 5 min an der Luft.
- Resuspendieren Sie das Pellet in 30 μL RNase-freiem Wasser.
- Berechnen Sie die RNA-Konzentration mit der Formel: A260 × Verdünnung × 40 = μg RNA/ml. Berechnen Sie das A260/A280-Verhältnis.
HINWEIS: Ein Verhältnis von ~2 gilt als rein.
- Führen Sie eine umgekehrte Transkription von RNA guter Qualität durch, um die cDNA-Vorlage zu erhalten.
- Entwerfen und verwenden Sie ein Paar spezifischer Primer, ppm-1-F/R (siehe Materialtabelle), um die teilweise kodierende Sequenz des Bx-ppm-1-Gens in B. xylophilus (GenBank-Zugangsnummer QTZ96795) zu amplifizieren.
- Klonen Sie die ppm-1-Gensequenzen in den pGEM-Teasy-Vektor, der den T7-Promotor enthält, nach einem Standard-Klonierungsprotokoll11.
- Stellen Sie die PCR-Reaktionen wie folgt ein: 2 μL cDNA, 25 μL 2x Ex Taq Polymerase Premix, 2 μL jedes Primers (10 pmol/l) und steriles destilliertes Wasser auf ein Endvolumen von 50 μL.
- Führen Sie den Verstärkungsvorgang wie folgt durch: 5 min bei 94 °C; gefolgt von 35 Zyklen von 30 s bei 94 °C, 30 s bei 55 °C und 1 min bei 72 °C; und eine letzte Verlängerungsstufe bei 72 °C für 5 min.
- Klonen Sie die amplifizierten Produkte in einen pGEM-T Easy Vektor für die Sequenzierung.
2. Synthese von dsRNA
- Bereiten Sie die DNA-Vorlage für die dsRNA-Synthese unter Verwendung der PCR mit Primern vor, die entwickelt wurden, um T7-Promotorstellen an beiden Enden hinzuzufügen. Fügen Sie die T7-Promotorsequenz zum 5'-Ende der Primer hinzu.
- Verwenden Sie das Plasmid, das das ppm-1-Genfragment (894 bp) enthält, als Vorlage für die PCR und gewinnen Sie das Fragment, das den T7-Promotor11 enthält. Verwenden Sie das oben beschriebene PCR-Verfahren und -System.
- Verwenden Sie ein In-vitro-Transkriptionskit , um dsRNA11 zu synthetisieren.
- Die gefrorenen Reagenzien auf Eis auftauen.
- Geben Sie 2 μL 10x Reaktionspuffer, 2 μL Enzymmischung und 1 μg DNA in ein Zentrifugenröhrchen. Fügen Sie nukleasefreies Wasser hinzu, um eine Standardreaktion von 4 μL zu erzeugen. Dann mischen Sie gleiche Volumina der vier Ribonukleotidlösungen (ATP, CTP, GTP und UTP) zusammen und geben 8 μL der Mischung in das Röhrchen. Gründlich mischen und 4 h bei 37 °C inkubieren.
- 1 μL DNase zugeben, gut mischen und 15 min bei 37 °C inkubieren.
- Stoppen Sie die Reaktion und fügen Sie 30 μL nukleasefreies Wasser und 30 μL LiCl-Fällungslösung hinzu, um die RNA auszufällen. Gründlich mischen. Über Nacht bei -20 °C inkubieren.
- 15 min bei 12.000 × g und 4 °C zentrifugieren. Verwerfen Sie den Überstand.
- Fügen Sie 1 ml 75% Ethanol hinzu, um die RNA zu waschen. Wirbeln Sie die Probe und zentrifugieren Sie sie für 10 min bei 12.000 × g und 4 °C.
- Trocknen Sie das RNA-Pellet 3 min an der Luft.
- Resuspendieren Sie das Pellet in 30 μL RNase-freiem Wasser.
- Analysieren Sie die Qualität der dsRNA mit einem Spektralphotometer. Pipettieren Sie 1 μL der dsRNA-Probe auf den Messsockel und stellen Sie die Wellenlänge auf 340 nm ein. Visualisieren Sie die Produkte auf einem 1,0% igen Agarose-Gel.
3. RNAi durch Einweichen
- Mischen Sie 4 μL 5x Einweichpuffer (0,05% Gelatine, 5,5 mM KH 2 PO 4, 2,1 mM NaCl, 4,7 mM NH4 Cl, 3 mM Spermidin) mit der dsRNA und ddH2O, um ein Gesamtvolumen von 20 μL undeine endgültige RNA-Konzentration von 0,8 μg / ml zu erhalten.
- Erwerben Sie J2-Larven.
- Sammeln Sie die Nematoden aus den Pilzkulturen und geben Sie sie in eine Glas-Petrischale von 6 cm Durchmesser. Fügen Sie der Schale 10 ml Wasser hinzu, damit die Nematoden frei schwimmen können. Halten Sie die Nematoden 30 Minuten in der Schale und warten Sie, bis die Eier am Boden haften.
- Entfernen Sie das Wasser und die Nematoden vorsichtig und achten Sie darauf, die Eier nicht zu stören. Wiederholen Sie die Schritte, bis alle Larven und Erwachsenen entfernt sind und nur die Eier in der Schale bleiben.
- Die gesammelten Eier 24 h im Dunkeln bei 25 °C ausbrüten, um J2-Larven zu erhalten. Sammeln Sie die J2-Larven, legen Sie sie in ein Röhrchen und waschen Sie sie dreimal mitddH2Ofür das RNAi-Experiment15.
- Die J2-Larven werden in das 2-ml-Röhrchen mit der dsRNA-Lösung überführt und Resorcinlösung (in Alufolie eingewickelt und in Wasser gelöst) hinzugefügt, um eine Endkonzentration von 1,0% zu erhalten. Die Larven mit Zentrifugation bei 15 × g auf einem Schütteltisch für 24 h bei 25 °C inkubieren, um sicherzustellen, dass die Larven die dsRNA effektiv aufnehmen.
- Weichen Sie die gleiche Menge Nematoden im Einweichpuffer ohne die dsRNA-Sonde oder mit einer GFP-dsRNA-Sonde als Kontrolle ein. Verwenden Sie das GFP-Gen (gfp, M62653.1) als nicht-endogene Kontrolle und synthetisieren Sie die dsRNA von gfp mit genspezifischen Primern T7-GFP-F/R.
4. qPCR-Nachweis
- Reinigen Sie die J2-Larven mitddH2O, einschließlich derjenigen mit Zielgeninterferenz, GFP-Geninterferenz und der ungestörten Kontrollgruppe. Extrahieren Sie die gesamten RNAs aus jeder Gruppe mit der oben beschriebenen Methode.
- Starten Sie die qPCR.
- Entwerfen Sie die q-ppm-1-F/R-Primer mit der gewünschten Software (siehe Materialtabelle).
- Die qPCR-Reaktion wird in 12 μL mit 1 μL cDNA, 6 μL fluoreszierender Vormischung, 0,4 μL jedes Primers (10 pmol/l) und sterilem destilliertem Wasser eingerichtet.
- Führen Sie die qPCR wie folgt durch: 2 min bei 95 °C; gefolgt von 40 Zyklen von 10 s bei 95 °C, 30 s bei 55 °C und 1 min bei 72 °C.
- Verwenden Sie das actb-Gen (GenBank-Zugangsnummer EU100952) und das tbb-2-Gen (GenBank-Zugangsnummer MT769316) oder andere Gene als interne Referenzgene, um die Veränderungen des Genexpressionsniveaus nach RNAi15 zu bewerten.
- Verwenden Sie entsprechend dem Zyklusschwellenwert (Ct) und der Auflösungskurve die 2-ΔΔCt-Methode , um das relative Expressionsniveau des Zielgens abzuschätzen und die Interferenzeffizienz zu überprüfen.
- Subtrahieren Sie den Ct-Wert des internen Referenzgens jeder Probe vom Ct-Wert des Zielgens, um den ΔCt-Wert zu erhalten. Subtrahieren Sie dann den ΔCt-Wert der Interferenzgruppe vom ΔCt-Wert der Kontrollgruppe, um den ΔΔCt-Wert zu erhalten.
HINWEIS: Ein ΔΔCt-Wert größer als 0 zeigt an, dass die Störung wirksam ist.
- Subtrahieren Sie den Ct-Wert des internen Referenzgens jeder Probe vom Ct-Wert des Zielgens, um den ΔCt-Wert zu erhalten. Subtrahieren Sie dann den ΔCt-Wert der Interferenzgruppe vom ΔCt-Wert der Kontrollgruppe, um den ΔΔCt-Wert zu erhalten.
5. Bewerten Sie die Körperlänge von Nematoden, die RNAi folgen
- Nach RNAi kultivieren Sie die J2-Larven bis ins Erwachsenenalter auf B. cinerea-Rasenflächen in PDA-Platten für 60 h bei 25 °C15.
- Sammeln Sie die Erwachsenen mit der Bellman-Trichtermethode (siehe Schritt 1.1.2.1)14.
- Nehmen Sie Bilder der erwachsenen Nematoden unter einem Mikroskop auf und verwenden Sie die ImageJ-Software (oder eine andere Messsoftware), um die Körperlängen zu messen.
- Messen Sie die Länge mit ImageJ, indem Sie Analyze | Legen Sie die Skalierung fest. Wenn der Abstand bekannt ist, geben Sie den Längenwert der gezeichneten geraden Linie ein. Geben Sie die Längeneinheit ein.
- Aktivieren Sie das Kontrollkästchen Global (verwenden Sie diesen Standard für alle Bilder), und klicken Sie auf OK , um die zu messende Länge mit einer geraden Linie auf dem zu messenden Bild auszuwählen.
- Verwenden Sie den Befehl Strg+M (Messung), um die Ergebnisse anzuzeigen und im Ergebnisfenster festzuhalten.
- Nehmen Sie Messungen von 50 männlichen und 50 weiblichen Nematoden für statistische Analysenvor 12.
- Analysieren Sie die Daten, indem Sie den Mittelwert und die Standardabweichung für jede Stichprobe berechnen. Vergleichen Sie die Mittelwerte der Stichproben aus den verschiedenen Gruppen mit dem t-Test des Schülers.
Ergebnisse
Analyse der ppm-1-Expression von B. xylophilus nach RNAi
Das relative Expressionsniveau des ppm-1-Gens von B. xylophilus, das mit GFP dsRNA getränkt und mit Zielgen dsRNA getränkt war, betrug 0,92 bzw. 0,52 (das ppm-1-Genexpressionsniveau der ddH2 O-behandelten Kontrollgruppe wurde auf 1 festgelegt) (Abbildung 1). Daher hat exogene dsRNA keinen Einfluss auf die ppm-1-Expression von B. xylophilus; ppm-1 dsRNA kann jedoch die Expression des Zielgens wirksam hemmen.
Wirkung der ppm-1-Expression auf Wachstum und Entwicklung von B. xylophilus
Nach RNAi nahm die Größe der Erwachsenen deutlich ab (Abbildung 2), was zum mutierten SMA-Phänotyp (Small Body Size) führte. Obwohl sich RNAi-behandelte Personen zur Geschlechtsreife entwickelten, war ihre Körperlänge wesentlich kleiner als bei normalen Erwachsenen. Insbesondere nach RNAi in Nematoden im J2-Stadium betrug die mittlere Körperlänge von Weibchen und Männchen 544,61 μm bzw. 526,24 μm. Im Gegensatz dazu betrug die mittlere Körperlänge von Weibchen und Männchen in der Kontrollgruppe 971,86 μm bzw. 912,31 μm (Abbildung 3), was signifikante Unterschiede darstellt (P = 0,0322).
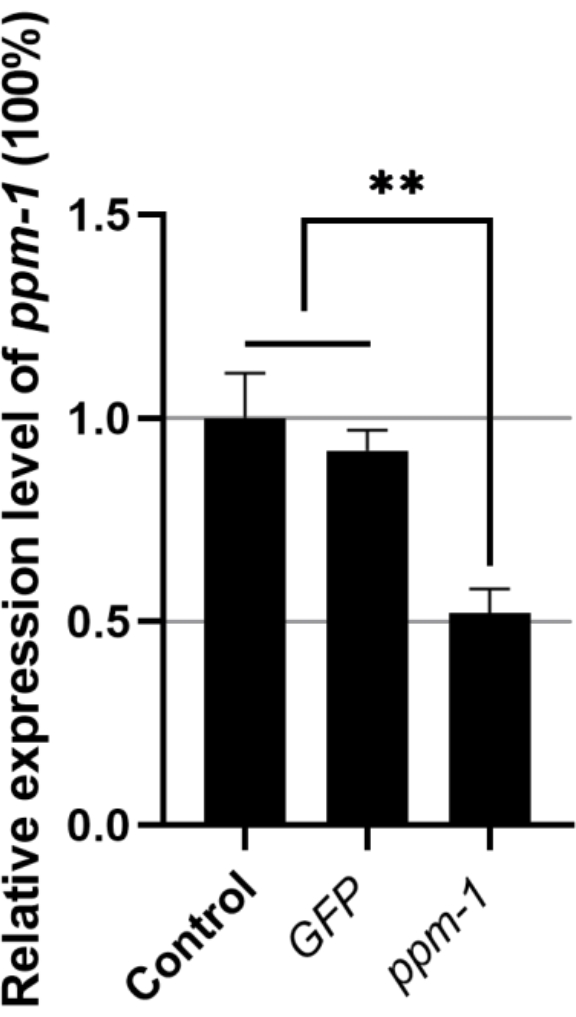
Abbildung 1: Expression des ppm-1-Gens nach RNAi von Bursaphelenchus xylophilus. **P < 0,001. Abkürzungen: RNAi = RNA-Interferenz; GFP = grün fluoreszierendes Protein. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.

Abbildung 2: Körperlängenreduktion von Erwachsenen nach der Interferenz von ppm-1 in Bursaphelenchus xylophilus. Bilder eines erwachsenen Mannes (A) und einer Frau (C) der RNAi-Gruppe. Bilder eines erwachsenen Mannes (B) und einer Frau (D) der Kontrollgruppe. Maßstabsbalken = 50 μm. Abkürzung: RNAi = RNA-Interferenz. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Abbildung 3: Quantifizierung und statistische Analyse der Körperlänge von Erwachsenen von Bursaphelenchus xylophilus nach RNAi des ppm-1-Gens . (*P = 0,0322). Abkürzung: RNAi = RNA-Interferenz. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Diskussion
Obwohl sich die Lebensgeschichte und die parasitäre Umgebung von B. xylophilus von denen anderer Nematoden unterscheiden, gibt es nur begrenzte Forschung über die molekulare Pathogenese dieses Pflanzenpathogens. Trotz großer Fortschritte bei der Anwendung der CRISPR/Cas9-Genom-Editing-Technologie bei C. elegans und anderen Nematoden wurde bisher nur die RNAi-Technologie veröffentlicht, die auf B. xylophilus angewendet wurde17. RNAi ist eines der leistungsfähigsten verfügbaren Werkzeuge zur Untersuchung der Genfunktion von Nematoden und wurde häufig in der Forschung zur Aufklärung der B. xylophilus-Genfunktion, Signaltransduktionswege und Gentherapie eingesetzt18,19,20. Im Gegensatz zu den Gen-Knockout-Ansätzen, die in wirbellosen Modellen verwendet werden, ermöglicht der RNAi-vermittelte Knockdown von Zielgenen in Nematoden die schnelle Bewertung der Genfunktion.
Es gibt drei Möglichkeiten, RNAi in C. elegans durchzuführen: Injektion 6, Einweichen21 und Fütterung6. Da sich J2-Larven erst nach einer Infektion ernähren, wird die Einweichmethode typischerweise für die RNAi von pflanzenparasitischen Nematoden verwendet. Der wichtigste Schritt bei der Einweichmethode besteht darin, ein Nervengift hinzuzufügen, um die Nematoden zum Fressen anzuregen. Urwin verwendete zunächst Octopamin, um das J2 von zwei oralen Zystennematodenarten, Globodera pallida und Heterodera-Glycinen, zu stimulieren, was zur Aufnahme von dsRNA aus der Einweichlösungführte 22. Die gleiche Methode wurde erfolgreich verwendet, um J2 des Wurzelknotennematoden Meloidogyne incognita zur Absorption von dsRNA 23 zu induzieren. Resorcinol kann auch die Aufnahme von dsRNA durch J2 von M. incognita induzieren und kann für diesen Nematoden24 wirksamer sein als Octopamin. Darüber hinaus verbesserte die Zugabe von Spermidin zum Einweichpuffer mit einer verlängerten Inkubationszeit die Effizienz von RNAi in Nematoden25. Nach 24 Stunden Einweichen dringt dsRNA effektiv in B. xylophilus ein und bringt dadurch das ppm-1-Gen zum Schweigen.
Dieses Protokoll liefert daher eine Referenz für die zukünftige Untersuchung der RNAi anderer pflanzenparasitärer Nematoden mit Phagozytoseverhalten. Darüber hinaus spielt die Suspensionswirkung des Schütteltisches eine wichtige Rolle, um den Vorteil der Einweichmethode zu maximieren. Diese Methode kann die gleichzeitige Behandlung einer großen Anzahl von Nematoden ermöglichen. RNAi ist einfacher zu operieren, wenn man sich auf die Zielgene konzentriert, für die Mutanten nicht leicht gewonnen werden können, und um die Auswirkungen an verschiedenen Entwicklungspunkten zu bewerten, da Nematoden in jedem Lebensstadium eingeweicht werden können. So kann RNAi verwendet werden, um die Funktion eines bestimmten Gens in einem bestimmten Entwicklungsstadium zu untersuchen. Wang et al. analysierten den Einfluss von MAPK auf die Fruchtbarkeit von B. xylophilus und seine wichtige Rolle beim Wachstum und der Entwicklung von Nematoden mittels dsRNA-Einweichen26. Qiu et al. fanden heraus, dass die Migrationsgeschwindigkeit und Fruchtbarkeit von B. xylophilus in Kiefern nach einer Herunterregulierung des Bxpel1-Gens abnahm, was darauf hindeutet, dass dieses Gen ein wichtiger pathogener Faktor von B. xylophilus27 ist.
Allerdings funktioniert nicht jede RNAi in Nematoden durch dsRNA. Dulovic und Streit verwendeten kleine interferierende RNAs (siRNAs) anstelle der längeren dsRNAs, um erfolgreich mit Strongyloides ratti28 zu interferieren. Die Einschränkung der Verwendung von dsRNA für RNAi in Strongyloides kann mit dem Fehlen von Genen wie rsd-6, sid-1 oder sid-2 zusammenhängen, von denen bekannt ist, dass sie an der Aufnahme von dsRNA beteiligt sind. RNAi ist auch mit den Nachteilen eines Positionseffekts und eines vorübergehenden und unvollständigen Knockouts verbunden und hat für einige Gene und Zelltypen (wie Neuronen) eine begrenzte Wirksamkeit29. Bis jedoch ein Durchbruch in der transgenen Technologie von B. xylophilus erreicht ist, stellt RNAi eine relativ effektive Forschungsstrategie dar.
RNAi hat ein großes Potenzial für den Pflanzenschutz gezeigt. Unter Verwendung der RNAi-Technologie wurden transgene Kartoffeln, die in der Lage sind, dsRNA zu produzieren, die auf Wurzelknotennematodengene abzielt, so gezüchtet, dass sie eine vollständige Resistenz gegen Wurzelknotennematodenerzeugen 30. Die Expression von dsRNAs, die auf Insektengene abzielen, kann in Abwesenheit chemischer Insektizide Pflanzenschutz bieten und bietet den zusätzlichen Vorteil, dass mit einer nahezu unbegrenzten Anzahl von Zielgenen keine Fremdproteine produziertwerden 31. Daher wird eine beschleunigte Forschung auf dem Gebiet der angewandten RNAi zur Schädlingsbekämpfung eine bessere Biosicherheit für die Pflanzennematodenbekämpfung bieten als transgene Methoden oder andere chemische Bekämpfungsmethoden.
Zusammenfassend beschreibt dieses Protokoll die Herstellung von dsRNA des ppm-1-Gens zur Erzielung von RNAi durch direktes Einweichen der Larven von B. xylophilus in dsRNA-Lösung. Der Interferenzeffekt wurde aufgrund einer signifikanten Verringerung der Körpergröße der Larven bestätigt, nachdem sie sich zu Erwachsenen entwickelt hatten, verglichen mit der der Kontrolle, was zeigt, dass das ppm-1-Gen Auswirkungen auf das Wachstum und die Entwicklung von B. xylophilus ausübt. Diese Studie liefert eine theoretische Grundlage, um die Funktion von ppm-1 mit zusätzlichem praktischen Wert bei der biologischen Kontrolle dieses Pflanzenparasiten weiter aufzudecken. Es wird angenommen, dass mit der weiteren Offenlegung und Verbesserung des RNAi-Technologiemechanismus seine Anwendung auf dem Gebiet der B. xylophilus-Kontrolle breite Perspektiven haben wird.
Offenlegungen
Interessenkonflikte wurden nicht erklärt.
Danksagungen
Diese Forschung wurde von der National Natural Science Foundation of China (31870637, 31200487) finanziert und gemeinsam durch den Zhejiang Key Research Plan (2019C02024, LGN22C160004) finanziert.
Materialien
| Name | Company | Catalog Number | Comments |
| Baermann funnel | n/a | n/a | to isolate nematodes |
| Beacon Designer 7.9 | Shanghai kangyusheng information technology co. | n/a | to design qPCR primers |
| Botrytis cinerea | n/a | n/a | as food for nematodes |
| Bursaphelenchus xylophilus | n/a | n/a | its number was NXY61 and was it was originally extracted from diseased Pinus massoniana in Ningbo, Zhejiang province, China. |
| constant temperature incubator | Shanghai Jing Hong Laboratory Instrument Co. | H1703544 | to cultur nematodes |
| Electrophoresis apparatus | Bio-Rad Laboratories | 1704466 | to achieve electrophoretic analysis |
| Ethanol, 75% | Sinopharm Chemical Reagent Co. | 80176961 | to extract RNA |
| Ex Taq Polymerase Premix | Takara Bio Inc. | RR030A | for PCR |
| Ex Taq Polymerase Premix | Takara Bio Inc. | RR390A | for PCR |
| Gel imager | LongGene Scientific Instruments Co. | LG2020 | to make nucleic acid bands visible |
| GraphPad Prism 8 | GraphPad Prism | n/a | to analyze the data and make figurs |
| High Speed Centrifuge | Hangzhou Allsheng Instruments Co. | AS0813000 | centrifug |
| High-flux tissue grinder | Bertin | to extract RNA | |
| ImageJ software | National Institutes of Health | n/a | to measure the body lengths |
| isopropyl alcohol | Shanghai Aladdin Biochemical Technology Co. | L1909022 | to extract RNA |
| Leica DM4B microscope | Leica Microsystems Inc. | to observe nematodes | |
| magnetic beads | Aoran science technology co. | 150010C | to extract RNA |
| MEGAscript T7 High Yield Transcription Kit | Thermo Fisher Scientific Inc. | AM1333 | to synthesize dsRNA in vitro |
| NanoDrop ND-2000 spectrophotometer | Thermo Fisher Scientific Inc. | NanoDrop 2000/2000C | to analyze the quality of the dsRNA |
| PCR Amplifier | Bio-Rad Life Medical Products Co. | 1851148 | to amplify nucleic acid sequence |
| Petri dishes | n/a | n/a | to cultur nematodes |
| pGEM-T Easy vector | Promega Corporation | A1360 | for cloning |
| Potato Dextrose Agar (Medium) | n/a | n/a | to cultur Botrytis cinerea |
| Prime Script RT reagent Kit with gDNA Eraser | Takara Bio Inc. | RR047B | to synthetic cDNA |
| Primer Premier 5.0 | PREMIER Biosoft | n/a | to design PCR primers |
| primers:ppm-1-F/R | Tsingke Biotechnology Co. | n/a | F: 5'-GATGCGAAGTTGCCAATCATTCT -3'; R: 5'- CCAGATCCAGTCCACCATACACC -3 |
| q-ppm-1-F/R | Tsingke Biotechnology Co. | n/a | F: 5'-CATCCGAATGGCAATACAG-3'; R: 5'-ACTATCCTCAGCGTTAGC-3' |
| Real-time thermal cycler qTOWER 2.2 | Analytique Jena Instruments (Beijing) Co. | for qPCR | |
| shaking table | Shanghai Zhicheng analytical instrument manufacturing co. | to soak nematodes | |
| stereoscopic microscope | Chongqing Optec Instrument Co. | 1814120 | to observe nematodes |
| T7-GFP-F/R | Tsingke Biotechnology Co. | n/a | F: 5'-TAATACGACTCACTATAGGGAAA GGAGAAGAACTTTTCAC-3'; R: 5'-TAATACGACTCACTATAGGGCTG TTACAAACTCAAGAAGG-3' |
| T7 promoter | Tsingke Biotechnology Co. | n/a | TAATACGACTCACTATAGGG |
| Takara MiniBEST Agarose Gel DNA Extraction Kit | Takara Bio Inc. | 9762 | to recover DNA |
| TaKaRa TB Green Premix Ex Taq (Tli RNaseH Plus) | Takara Bio Inc. | RR820A | for qPCR |
| trichloroethane | Shanghai LingFeng Chemical Reagent Co. | to extract RNA | |
| TRIzol Reagent | Thermo Fisher Scientific Inc. | 15596026 | total RNA extraction reagent,to extract RNA |
Referenzen
- Nicol, J. M., Jones, J., Gheysen, G., Fenoll, C., et al. Current nematode threats to world agriculture. Genomics and Molecular Genetics of Plant-Nematode Interactions. , 21-43 (2011).
- Jones, J. T., et al. Top 10 plant-parasitic nematodes in molecular plant pathology. Molecular Plant Pathology. 14 (9), 946-961 (2013).
- Kikuchi, T., et al. Genomic insights into the origin of parasitism in the emerging plant pathogen Bursaphelenchus xylophilus. PLoS Pathogens. 7 (9), 1002219 (2011).
- Megen, H. V., et al. A phylogenetic tree of nematodes based on about 1200 full-length small subunit ribosomal DNA sequences. Nematology. 11 (6), 927-950 (2009).
- Niu, J. H., Jian, H., Xu, J. M., Guo, Y. D., Liu, Q. RNAi technology extends its reach: Engineering plant resistance against harmful eukaryotes. African Journal of Biotechnology. 9 (45), 7573-7582 (2010).
- Fire, A., et al. Potent and specific genetic interference by double-stranded RNA in Caenorhabditis elegans. Nature. 91 (6669), 806-811 (1998).
- Shahid, M., Imran, A., Mazhar, H., Yusuf, Z., Rob, W. B. Engineering novel traits in plants through RNA interference. Trends in Plant Science. 11 (11), 559-565 (2006).
- Marmonier, A., et al. In vitro acquisition of specific small interfering RNAs inhibits the expression of some target genes in the plant ectoparasite Xiphinema index. International Journal of Molecular Sciences. 20 (13), 3266 (2019).
- Iqbal, S., Fosu-Nyarko, J., Jones, M. G. K. Attempt to silence genes of the RNAi pathways of the root-knot nematode, Meloidogyne incognita results in diverse responses including increase and no change in expression of some genes. Frontiers in Plant Science. 11, 328 (2020).
- Zhou, L. F., et al. Molecular characterization and functional analysis of akt-1 in pinewood nematode, Bursaphelenchus xylophilus. Forest Pathology. 51 (1), 12647 (2021).
- Zhou, L. F., et al. The role of mab-3 in spermatogenesis and ontogenesis of pinewood nematode, Bursaphelenchus xylophilus. Pest Management Science. 77 (1), 138-147 (2021).
- Tang, J., et al. Bxy-fuca encoding α-L-fucosidase plays crucial roles in development and reproduction of the pathogenic pinewood nematode, Bursaphelenchus xylophilus. Pest Management Science. 76 (1), 205-214 (2020).
- Wang, J. H., et al. Molecular characterization and functional analysis of daf-8 in the pinewood nematode, Bursaphelenchus xylophilus. Journal of Forestry Research. , (2021).
- Viglierchio, D. R., Schmitt, R. V. On the methodology of nematode extraction from field samples: Baermann funnel modifications. Journal of Nematology. 15 (3), 438-444 (1983).
- Zhu, N., et al. Observation and quantification of mating behavior in the pinewood nematode, Bursaphelenchus xylophilus. Journal of Visualized Experiments: JoVE. (118), e54842 (2016).
- Zhou, L. F., Chen, F. M., Ye, J. R., Pan, H. Y. Selection of reliable reference genes for RT-qPCR analysis of Bursaphelenchus mucronatus gene expression from different habitats and developmental stages. Frontiers in Genetics. 9, 269-279 (2018).
- Wang, M., et al. Double-stranded RNA-mediated interference of dumpy genes in Bursaphelenchus xylophilus by feeding on filamentous fungal transformants. International Journal for Parasitology. 46 (5-6), 351-360 (2016).
- Ma, H. B., Lu, Q., Liang, J., Zhang, X. Y. Functional analysis of the cellulose gene of the pine wood nematode, Bursaphelenchus xylophilus, using RNA interference. Genetics and Molecular Research: GMR. 10 (3), 1931-1941 (2011).
- Cheng, X. Y., Dai, S. M., Xiao, L., Xie, B. Y. Influence of cellulase gene knock down by dsRNA interference on the development and reproduction of the pine wood nematode, Bursaphelenchus xylophilus. Nematology. 12 (12), 225-233 (2010).
- Xue, Q., Wu, X. Q., Zhang, W. J., Deng, L. N., Wu, M. M. Cathepsin L-like cysteine proteinase genes are associated with the development and pathogenicity of pine wood nematode, Bursaphelenchus xylophilus. International Journal of Molecular Sciences. 20 (1), 215 (2019).
- Tabara, H., Grishok, A., Mello, C. C. RNAi in C. elegans: Soaking in the genome sequence. Science. 282 (5388), 430-431 (1998).
- Urwin, P. E., Lilley, C. J., Atkinson, H. J. Ingestion of double-stranded RNA by pre parasitic juvenile cyst nematodes leads to RNA interference. Molecular Plant-Microbe Interactions: MPMI. 15 (8), 747-752 (2002).
- Bakhetia, M., Charlton, W., Atkinson, H. J., McPherson, M. J. RNA interference of dual oxidase in the plant nematode Meloidogyne incognita. Molecular Plant-Microbe Interactions: MPMI. 18 (10), 1099-1106 (2005).
- Rosso, M. N., Dubrana, M. P., Cimbolini, N., Jaubert, S., Abad, P. Application of RNA interference to root-knot nematode genes encoding esophageal gland proteins. Molecular Plant-Microbe Interactions: MPMI. 18 (7), 615-620 (2005).
- Chen, Q., Rehman, S., Smant, G., Jones, J. T. Functional analysis of pathogenicity proteins of the potato cyst nematode Globodera rostochiensis using RNAi. Molecular Plant-Microbe Interactions: MPMI. 18 (7), 621-625 (2005).
- Wang, D. D., Li, Y., Li, J., Xie, B. Y., Chen, G. H. Molecular clone and its RNAi interference effect analysis of mapk gene in Bursaphelenchus xylophilus ( in Chinese). Acta Phytopathologica Sinica. 46 (5), 662-669 (2016).
- Qiu, X., Wu, X., Huang, L., Ye, J. R. Influence of Bxpel1 gene silencing by dsRNA interference on the development and pathogenicity of the pine wood nematode, Bursaphelenchus xylophilus. International Journal of Molecular Sciences. 17 (1), 125 (2016).
- Dulovic, A., Streit, A. RNAi-mediated knockdown of daf-12 in the model parasitic nematode Strongyloides ratti. PLoS Pathogens. 15 (3), 1007705 (2019).
- Li, L., Zhao, H., Cui, Y., Wei, H., Li, M. Research progress of gene editing technology. Life Science Research. 21 (3), 268-274 (2017).
- Bindhya, C. Y., Karuppannan, V., Kuppuswamy, S. Host-generated double stranded RNA induces RNAi in plant-parasitic nematodes and protects the host from infection. Molecular and Biochemical Parasitology. 148 (2), 219-222 (2006).
- Jiang, Z., Sher, A. K., David, G. H., Ralph, B. Next-generation insect-resistant plants: RNAi-mediated crop protection. Trends in Biotechnology. 35 (9), 871-882 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten