Method Article
Los ensayos ACT1-CUP1 determinan las sensibilidades específicas del sustrato de los mutantes espliceosomales en levaduras en ciernes
En este artículo
Resumen
El ensayo ACT1-CUP1, un ensayo de crecimiento de cobre, proporciona una lectura rápida del empalme del ARN mensajero precursor (pre-ARNm) y el impacto que los factores de empalme mutante tienen en la función espliceosomal. Este estudio proporciona un protocolo y destaca la personalización posible para abordar la cuestión de empalme de interés.
Resumen
Las mutaciones introducidas en el espliceosoma o su sustrato han contribuido significativamente a nuestra comprensión de las complejidades de la función espliceosomal. Ya sea relacionada con la enfermedad o seleccionada funcionalmente, muchas de estas mutaciones se han estudiado utilizando ensayos de crecimiento en el organismo modelo Saccharomyces cerevisiae (levadura). El ensayo de crecimiento de cobre específico de empalme, o ensayo ACT1-CUP1, proporciona un análisis exhaustivo de la mutación a nivel fenotípico. El ensayo ACT1-CUP1 utiliza reporteros que confieren tolerancia al cobre cuando se empalma correctamente. Por lo tanto, en presencia de cobre, los cambios en la viabilidad de la levadura se correlacionan con los cambios en la producción de ARNm a través del empalme. En un experimento típico, el espliceosoma de levadura es desafiado con diferentes reporteros de empalme sin consenso y la mutación del factor de empalme de interés para detectar cualquier impacto sinérgico o antitético en el empalme. Aquí se da una descripción completa de la preparación de la placa de cobre, el recubrimiento de las células de levadura y la evaluación de los datos. Se describe una selección de experimentos complementarios, destacando la versatilidad de los reporteros ACT1-CUP1. El ensayo ACT1-CUP1 es una herramienta útil en la caja de herramientas de empalme gracias a la lectura directa de los efectos mutacionales y las posibilidades comparativas del uso continuo en el campo.
Introducción
El espliceosoma es una gran máquina biológica que cataliza la eliminación de intrones, regiones no codificantes en el ARN mensajero precursor (pre-ARNm)1,2. La caracterización del efecto de un mutante de un solo punto en 1 de las casi 100 proteínas y 5 ARN no codificantes es a menudo ambigua cuando se estudia la proteína o el ARN de forma aislada. El cambio en la función del componente mutado se puede evaluar mejor in vivo en el contexto del espliceosoma completo y funcional.
El ensayo de crecimiento de cobre descrito aquí es un indicador rápido de la eficiencia de empalme en Saccharomyces cerevisiae o levadura en ciernes. Desarrollado por C.F. Lesser y C. Guthrie y publicado en 1993, este ensayo combina la facilidad de trabajar con un organismo modelo simple y la lectura directa de la viabilidad celular3. La viabilidad se correlaciona con qué tan bien los espliceosomas en estas células pueden reconocer y empalmar la transcripción del reportero.
Este ensayo de crecimiento de cobre se denomina más comúnmente ensayo ACT1-CUP1. El nombre ACT1-CUP1 se origina a partir de los dos genes fusionados para crear un reportero de eficiencia de empalme. ACT1 es el gen de la actina de la levadura, que está altamente expresado y tiene un intrón empalmado eficientemente 4,5. Cup1p es un quelante de cobre que secuestra cobre en la célula para evitar la interferencia con las funciones celulares regulares 6,7,8. El reportero ACT1-CUP1 contiene estos genes en secuencia tal que CUP1 está en el marco de lectura adecuado solo si se produce un empalme previo al ARNm del intrón de ACT1 (Figura 1). La proteína de fusión resultante contiene los primeros 21 aminoácidos de actina y la proteína Cup1p de longitud completa, lo que aumenta la viabilidad de la levadura en un ambiente rico en cobre3. Por lo tanto, un aumento en la cantidad de empalme del reportero da como resultado una mayor concentración de Cup1p y una mayor resistencia al cobre (Figura 1). En comparación con otros genes reporteros, CUP1 afecta la viabilidad celular incluso a niveles bajos, tiene un amplio rango de sensibilidad y puede usarse para seleccionar directamente mutaciones de empalme 3,6,7. Además, CUP1 no es esencial para el crecimiento estándar de la levadura y, por lo tanto, la homeostasis celular no se ve afectada durante la configuración de este ensayo. Complementario a los ensayos de deleción o crecimiento de temperatura, ACT1-CUP1 proporciona información sobre los efectos sobre el empalme en condiciones óptimas de crecimiento de levadura.
El espliceosoma reconoce su sustrato a través de tres secuencias intrónicas, a saber, el sitio de empalme de 5' (5' SS), el sitio de ramificación (BS) y el sitio de empalme de 3' (3' SS). Se han generado numerosos reporteros ACT1-CUP1 que contienen secuencias no consensuadas en estos sitios. Una selección de los reporteros ACT1-CUP1 más comunes se muestra en la Figura 1 y la Tabla 1. Como el espliceosoma interactúa con cada sitio de empalme de forma única en diferentes puntos del ciclo de empalme, la robustez del espliceosoma se puede probar en diferentes pasos en función de qué informador sin consenso se utiliza. Los reporteros sin consenso se nombran por la posición mutada dentro del intrón y la base a la que fue mutado. Por ejemplo, A3c es un reportero con una mutación en el SS 5', específicamente la posición 3 de la adenosina de consenso a una citosina. Este reportero interactuará fuertemente con las mutaciones del espliceosoma que afectan la selección y el uso de 5' SS. En su estudio inicial, Lesser y Guthrie determinaron qué mutaciones 5' SS inhibían el empalme3. Más tarde, el mismo año, Burgess y Guthrie publicaron reporteros sin consenso en los tres sitios de empalme en una pantalla supresora de mutaciones en la ATPasa Prp16p9. Comparando el consenso con los reporteros sin consenso, el ensayo ACT1-CUP1 ha sido una clave importante para comprender la robustez y selectividad del espliceosoma de levadura e inferir la función de los espliceosomas de otros eucariotas.
A medida que los reporteros ACT1-CUP1 sin consenso sensibilizan al espliceosoma a una mayor perturbación, el impacto de una sola mutación del factor de empalme puede caracterizarse a través de los reporteros que impacta positiva o negativamente. Esto se ha aplicado al empalme de preguntas de investigación de varias maneras. En primer lugar, el ensayo ACT1-CUP1 puede y ha sido utilizado como una pantalla genética para mutaciones en los factores de empalme. Por ejemplo, Prp8p, la proteína de empalme más grande, sirve como una plataforma sobre la cual el núcleo de ARN del espliceosoma cataliza la reacción de empalme. Esto se dedujo, en parte, a través de cómo los mutantes Prp8p mejoraron o redujeron el empalme de diferentes reporteros ACT1-CUP1 10,11,12,13,14,15,16,17. Otros componentes proteicos del espliceosoma también se han investigado utilizando ACT1-CUP1, incluyendo Hsh155p, Cwc2p, Cef1p y Ecm2p 18,19,20,21,22,23,24,25. Los umbrales energéticos para Prp16p y otras cuatro ATPasas involucradas en la transición espliceosomal también se han estudiado con este ensayo 9,26,27,28,29,30. Los ARN nucleares pequeños (snRNAs) también se han estudiado ampliamente utilizando ACT1-CUP1 para identificar las secuencias de pre-ARNm que coordinan y los cambios en la estructura secundaria que sufren los snRNAs durante el empalme 3,31,32,33,34,35,36,37.
El ensayo ACT1-CUP1 requiere una cepa de levadura donde todas las copias del gen CUP1 han sido eliminadas. Como CUP1 puede tener un alto número de copias 6,38, la preparación de una cepa knock-out completa puede requerir múltiples rondas o un cribado extenso. Como resultado, las cepas de levadura cup1Δ a menudo se han compartido entre laboratorios, al igual que los reporteros.
Si se evalúan mutaciones en un factor de empalme a partir de una copia plásmida, el gen de tipo salvaje para este factor debe ser eliminado. Además, el fondo de levadura debe permitir la selección de al menos dos plásmidos, uno que contenga un reportero ACT1-CUP1, históricamente en un plásmido de selección de nutrientes de leucina, y otro que contenga una mutación o perturbación en la maquinaria de empalme que se estudiará (Figura 2). Por lo general, en un solo ensayo, varias cepas de levadura, cada una con la perturbación de empalme de consulta (QSP) y un reportero diferente, probarán el impacto de la consulta en el empalme.
Las variables independientes en el ensayo ACT1-CUP1 permiten al investigador evaluar la gravedad de un QSP. Estas variables independientes son la concentración de cobre y la selección de múltiples reporteros de empalme sin consenso. En primer lugar, como las cepas de levadura se cultivan en placas que contienen un rango de concentraciones de cobre (Figura 2), la configuración del ensayo incluye la selección del gradiente de concentraciones utilizadas. Los estudios pueden utilizar un gradiente de concentración de cobre de curso para obtener una lectura inicial de la viabilidad y luego repetir el ensayo con un gradiente más fino para identificar diferencias sutiles de viabilidad. La segunda variable es la amplia gama de reporteros ACT1-CUP1 posibles de probar (Figura 1 y Tabla 1). Si el QSP afecta la viabilidad de la levadura de manera diferente en presencia de un informador sin consenso versus de tipo salvaje, se puede concluir que el QSP afecta un paso en el empalme o una región del espliceosoma importante durante el reconocimiento o procesamiento de esa región del intrón.
La caja de herramientas de levadura es extensa, y el ensayo ACT1-CUP1 es una parte integral de la investigación de empalme. El ensayo ACT1-CUP1 a menudo se realiza junto con un análisis genético, estructural y / o bioquímico más profundo sobre el impacto de un QSP. Como estos estudios más detallados generalmente tienen un procedimiento más largo y / o un precio más alto, un enfoque frecuente es la detección de mutantes interesantes con ACT1-CUP1 primero.
Aquí se proporciona un protocolo de ensayo ACT1-CUP1, incluida la preparación de la placa de cobre. Este ensayo proporciona a los investigadores una respuesta inicial al efecto de un QSP en el empalme y qué regiones intrónicas se ven más afectadas por la perturbación.
Protocolo
1. Construcción de la cepa de levadura
- Generar u obtener una cepa de S. cerevisiae cuyo fondo incluya leu2 y cup1Δ. Para generar este fondo, utilice el método de levadura bien establecido que emplea acetato de litio y ADN monocatenario39.
NOTA: Las cepas de levadura haploide pueden contener una, dos o más copias de CUP1 6,38. Consulte la información genómica de la cepa de levadura seleccionada al diseñar cebadores knock-out para flanquear la(s) ubicación(es) del gen CUP1. - Realizar una transformación de levadura para incorporar el QSP ya sea a través de la incorporación genómica o en un plásmido. Utilizar un protocolo bien establecido como los descritos en investigaciones previas40,41,42.
- Realizar una transformación de levadura con la(s) cepa(s) resultante(s) del paso 1.2. para agregar el plásmido reportero ACT1-CUP1 deseado.
NOTA: Las células deben mantenerse en placas y medios de leucina (-Leu) para asegurar la retención de los plásmidos reporteros ACT1-CUP1 después de esta transformación. - Realice los pasos 1.2. y 1.3. para cada QSP y cada plásmido reportero ACT1-CUP1 que se vaya a probar, incluidas las cepas de control.
2. Preparación de la placa de cobre
- Seleccione un rango de concentración de cobre que se adapte a los reporteros que se someterán a la prueba (consulte la Tabla 1 para la letalidad de los reporteros de uso frecuente).
NOTA: Un ejemplo de un rango de concentración de cobre integral es 30 concentraciones diferentes de cobre de 0 mM, 0.025 mM, 0.05 mM, 0.075 mM, 0.1 mM, 0.15 mM, 0.2 mM, 0.25 mM, 0.3 mM, 0.35 mM, 0.4 mM, 0.45 mM, 0.5 mM, 0.6 mM, 0.7 mM, 0.8 mM, 0.9 mM, 1.0 mM, 1.1 mM, 1.2 mM, 1.3 mM, 1,4 mM, 1,5 mM, 1,6 mM, 1,7 mM, 1,8 mM, 1,9 mM, 2,0 mM, 2,25 mM y 2,5 mM Cu2+. - Hacer una solución madre de 1 M CuSO4 y filtro estéril a través de un filtro estéril PES (polietersulfona) de 0,22 μm.
- Por placa de cobre deseada, prepare una dilución de 2 ml del stock de CuSO4 en agua estéril.
NOTA: Como la placa con 0 mM Cu 2+ siempre será analizada y fotografiada como referencia, se recomienda hacer dos placas de 0 mM Cu2+, una al principio y otra al final del paso de recubrimiento (paso 3.4.).- Calcule la cantidad de existencias para la concentración final deseada de cobre en 40 ml del volumen de la placa (Tabla suplementaria 1).
- Agregue la cantidad calculada de agua estéril y 1 caldo de CuSO4 M a un tubo estéril de 2 ml.
- Placas de vertido para el ensayo ACT1-CUP1.
NOTA: Una alternativa al protocolo a continuación es combinar inicialmente el medio y el agar en un recipiente grande y alícuota después de esterilizarlos en autoclave en recipientes más pequeños para lograr diferentes concentraciones de cobre para cada placa. Cualquiera que sea el método que se tome, es importante asegurarse de que la concentración del medio sea consistente entre todas las placas a pesar de que cada una tenga una concentración de cobre diferente.- Etiquete cada plato vacío para ser vertido con la concentración final de cobre que contendrá. Prepare al menos una placa cuadrada por concentración de cobre para ser probada.
- Etiquete una botella de 100 ml por concentración de cobre a analizar.
- A cada botella, agregue 790 mg de agar (2% p/v de agar) y una barra de agitación.
- En un vaso de precipitados grande, combine el medio de crecimiento -Leu para que se viertan todas las placas de cobre. Por placa a hacer, disuelva 265 mg de base de nitrógeno de levadura (YNB) y 64 mg de mezcla de abandono menos leucina (y cualquier otro nutriente que pueda ser necesario para mantener el plásmido QSP en las células) en 34 ml de agua desionizada.
- Agregue 34 ml de la solución de medios de cultivo -Leu a cada botella de 100 ml preparada y tapa con papel de aluminio. Etiquete la lámina con la concentración de cobre prevista.
- Autoclave para esterilizar y disolver el agar utilizando el ciclo líquido recomendado para el autoclave.
- Tan pronto como sea posible, agregue 4 ml de glucosa al 20% p/v (filtrada estéril) a cada botella.
- Haga coincidir las etiquetas y agregue las diluciones de 2 ml de CuSO4 a su botella prevista.
NOTA: Como se pueden hacer decenas de placas de cobre al mismo tiempo, cada una con una concentración diferente, etiquetar todas las botellas, tubos y placas claramente con la concentración de cobre prevista evitará confusiones durante el vertido de la placa. - Use una placa de agitación para mezclar durante ~ 30 s y vierta o pipete 35 ml en la placa etiquetada, evitando burbujas. Dejar enfriar antes de almacenar o usar.
NOTA: Con frecuencia, las placas se fabrican 1 día o 2 días antes del ensayo y se almacenan a 4 °C hasta unas horas antes de su uso. Las placas deben estar a temperatura ambiente (RT) antes de que comience el recubrimiento (paso 3.4.).
3. Ensayo ACT1-CUP1
- Raye las cepas deseadas en placas de -Leu.
NOTA: Si se trabaja con crioexistencias, se debe tener cuidado para garantizar que las células se reactiven lo suficiente del almacenamiento antes del recubrimiento. Un procedimiento recomendado para esto es salir del stock criogénico y dejar que crezca durante 3-5 días a 30 ° C. Luego, retome una pequeña muestra y déjela crecer durante otros 2-3 días a 30 ° C. - Cultive cultivos nocturnos en 10 ml de medios.
- Preparar los medios de cultivo -Leu utilizando las mismas proporciones que se describen en el paso 2.4.2. Por 10 ml de medio, agregue 66 mg de base de nitrógeno de levadura (YNB) y 16 mg de mezcla de abandono menos leucina a 9 ml de agua desionizada. Pasar a través de un filtro estéril PES de 0,22 μm.
- Por cepa de levadura, añadir 9 ml de medios de cultivo -Leu y 1 ml de glucosa al 20% p/v (filtrada estéril) a un tubo cónico estéril de 50 ml.
- Usando un palo estéril o una punta de pipeta, reúna una pequeña muestra de levadura (~ 1 mm redonda) e inocule el medio.
- Agitar todos los cultivos nocturnos a 180 rpm y 30 °C.
NOTA: Si está disponible, se pueden usar rotadores en lugar de un agitador.
- Diluir las cepas a un OD600 0.5 ± 0.05 en 10% de glicerol.
- Por cepa, añadir 100 μL de cultivo a una cubeta que contenga 900 μL de agua.
- Mida el OD600 con un espectrofotómetro.
- Calcular la dilución requerida para estar en un OD600 de 0.5 en un volumen final de 2 mL.
- Diluir cada cepa a OD600 0.5 en glicerol al 10% (estéril).
- Vuelva a medir el OD600 para confirmar que la densidad celular está dentro del rango deseado de 0,5 ± 0,05.
- Coloque las tensiones en las placas de cobre.
NOTA: Se puede utilizar una variedad de métodos para chapar las cepas, incluido el pipeteo manual de volúmenes de 5-10 μL, el uso de un pipetor repetido o multicanal, o el estampado con un replicador de pines. Este último método se describe a continuación, aunque la mayoría de los pasos serán similares independientemente del método.- Establezca un lugar de trabajo estéril y un quemador Bunsen encendido.
- Para un replicador de 48 pines, pipetear 200 μL de cada cepa diluida en un pocillo separado de una placa de 96 pocillos. Llene los espacios vacíos en la cuadrícula de 6 x 8 con 200 μL de glicerol al 10% (estéril).
NOTA: Un ejemplo de un esquema de recubrimiento para nueve cepas de levadura se encuentra en la Tabla Suplementaria 2. - Sumerja el replicador en una placa poco profunda de etanol al 95% (v / v) y llama para esterilizar. Deje enfriar durante al menos 2 minutos después de que la llama se extinga para evitar que el calor choque las células.
- Coloque cuatro placas cerca del quemador y retire las tapas.
- Sumerja el replicador en la placa de 96 pocillos y levántelo en un movimiento rápido.
- Coloque suavemente sobre el plato y balancee ligeramente hacia adelante y hacia atrás para facilitar una buena transferencia.
- Levante en un movimiento rápido y colóquelo exactamente en la misma orientación en la placa de 96 pocillos.
- Repita para hasta otros tres platos. Repita el proceso de sumergir el replicador en etanol, flamear para esterilizar y esperar a enfriar cada cuatro placas.
- Después de que se haya plateado una placa, muévala con un movimiento suave hacia un lado, pero aún dentro del paraguas de esterilización de la llama.
NOTA: Se recomienda hacer las diluciones de levadura y el recubrimiento cerca de una llama. Las placas pueden contaminarse fácilmente durante el secado. - Deje que las placas se sequen completamente antes de colocar las tapas, generalmente 3-5 min.
- Incubar las placas durante 3 días a 30 °C.
4. Recopilación y análisis de datos
- Retire las placas de la incubadora e inspecciónelas visualmente.
- Grabe las imágenes de las placas con una cámara disponible u otro sistema de imágenes digitales.
- Registrar (o puntuar) para cada cepa se observa el último crecimiento visible de la concentración de cobre.
NOTA: Las células son capaces de empalmar y permanecer viables hasta esa concentración. Para mantener la consistencia, utilice siempre el mismo método, ya sea a simple vista o a partir de las imágenes de la placa, para marcar la última concentración de cobre viable. Las colonias muy pequeñas a veces son visibles a simple vista, pero no en la imagen. La diferencia entre la inspección visual directa o la grabación de imágenes es pequeña, generalmente un paso en el gradiente. Como las imágenes de las colonias se utilizan a menudo en las publicaciones, se recomienda puntuar por las imágenes. - Combine datos de múltiples ensayos ACT1-CUP1 para la misma cepa para sacar conclusiones sobre cómo el QSP afecta el empalme.
NOTA: Las cifras de publicación suelen mostrar imágenes de las colonias de levadura a una concentración de 0 mM Cu2+ , la última concentración viable de cobre y la concentración posterior donde la colonia ha muerto. Los datos también se pueden mostrar como un gráfico de barras con barras de error para la desviación estándar entre réplicas. No es necesario normalizar los datos, pero sí establecerán la viabilidad del control del factor de empalme WT en 1 y compararán el efecto de la(s) mutación(es) introducida(s).
Resultados
Los ensayos de crecimiento, como ACT1-CUP1, requieren la evaluación visual y comparativa de múltiples colonias. Aquí, cada cepa se cultivó hasta la saturación durante la noche, se diluyó a un OD600 de 0.5 y se chapó en 20 placas que contienen un rango de concentraciones de cobre de 0 mM a 1.1 mMCuSO4 (Figura 3). Este rango es menor que el enumerado en el protocolo, ya que permitió la evaluación completa del impacto de los QSP y los reporteros ACT1-CUP1 utilizados y descritos a continuación. Las placas fueron fotografiadas y puntuadas (Figura 3 y Tabla Suplementaria 3).
Para este experimento representativo, el fondo de levadura incluye un gen ADE2 alterado, lo que hace que las colonias sean de diferentes tonos de rojo (Figura 3A, B). Esta interrupción común de la levadura en la vía de producción de adenosina causa una acumulación de un precursor de pigmentación roja para la adenosina. Por lo tanto, el color rojo es un indicador de la madurez de la colonia de levadura (es decir, la cantidad y la edad de las células presentes). Para los propósitos de un ensayo ACT1-CUP1, el color rojo puede servir como un indicador de especies fúngicas contaminantes si son de color blanco o amarillo. El color puede ser una confirmación secundaria de la tolerancia al cobre, ya que las colonias más viables serán de un tono rojo más profundo.
Al chapar con un replicador de pines, hay varias aberraciones comunes. Primero, es posible crear colonias que tienen forma ovalada si el replicador se levanta mientras se mueve hacia la izquierda o hacia la derecha (Figura 3B, flecha naranja). Además, las microcolonias también pueden formarse a partir de pequeñas gotas de solución celular si el replicador de pines se introduce en ángulo o si se agita por encima de la placa (Figura 3B, flecha roja). A menudo inocuas, ocasionalmente, las microcolonias pueden mezclarse con una colonia estampada, y esto impide la interpretación de esa colonia. Los problemas adicionales de recubrimiento incluyen un tiempo de espera insuficiente después de la esterilización para que los pines del replicador se enfríen y un contacto insuficiente entre la placa y los pines, de modo que se produzca una transferencia deficiente de los medios de cultivo. En ambos casos, pocas células, si es que hay alguna, crecerán en la placa, incluso para las cepas de control que contienen el reportero de tipo salvaje. Al igual que con cualquier ensayo de crecimiento, independientemente del método de recubrimiento, es importante realizar el ACT1-CUP1 por triplicado para confirmar que los resultados son consistentes y repetibles. Idealmente, estas diferentes réplicas deben realizarse en placas de cobre preparadas en diferentes momentos para garantizar la reproducibilidad.
Para estos experimentos representativos, el reportero de A3c fue seleccionado para resaltar el impacto de una secuencia no consensuada en la tolerancia al cobre en ausencia de perturbaciones adicionales del espliceosoma. A3c reduce significativamente la cantidad de ARNm CUP1 producido, ya que perturba la capacidad del espliceosoma para reconocer y utilizar el 5' SS15. La levadura con el reportero A3c sobrevivió a 0.15 mM Cu2+, mientras que las células reporteras de tipo salvaje mantuvieron la viabilidad hasta el final del rango de concentraciones de cobre probadas (Figura 3C). El reportero de tipo salvaje que contiene células crece hasta 2.5 mM Cu2+ sin un impacto en la viabilidad (datos no mostrados).
El snRNA U6 es un componente catalítico esencial del espliceosoma. Múltiples estudios han utilizado ACT1-CUP1 para estudiar el efecto que las mutaciones pueden tener sobre este ARN 16,32,34. Duplicado para este estudio, se estudiaron tres secuencias de snRNA U6, a saber, la secuencia de tipo salvaje (WT), la posición 57 sustituida de uridina a una citosina (U57c) y la posición 57 sustituida de uridina a adenosina (U57a) (Figura 3 y Figura 4). Combinado con reporteros ACT1-CUP1 que impactan los pasos catalíticos en el empalme, se determinó que U57c favorece el primer paso catalítico, y U57a favorece la progresión al segundo paso16,32.
Para configurar el ensayo ACT1-CUP1, se creó una cepa con la copia genómica del snRNA U6 eliminada y las secuencias mutadas o de tipo salvaje del snRNA U6 incluidas en los plásmidos. Como el snRNA U6 es un componente esencial de la célula, se utilizaron tres transformaciones separadas para eliminar primero el snRNA genómico U6 mientras se mantenía la viabilidad celular, posteriormente introducir snRNAs U6 mutados en plásmidos y, finalmente, agregar los reporteros ACT1-CUP1. Para la primera transformación, se utilizó una cepa de levadura cup1Δ en el knock-out de la copia genómica de U6 snRNA mientras se agregaba simultáneamente WT U6 snRNA en un plásmido marcador de selección URA para mantener la viabilidad. Una transformación posterior agregó snRNA U6 mutado o de tipo salvaje en un plásmido marcador de selección TRP, seleccionando contra el marcador URA a través de la selección 5-FOA. Por lo tanto, se generaron tres cepas de levadura cup1Δ , cada una con una de las secuencias de snRNA U6, a saber, WT, U57a y U57c. Una transformación final de levadura para cada cepa agregó uno de los tres plásmidos reporteros ACT1-CUP1 diferentes que se utilizarán en este experimento. Los reporteros seleccionados fueron el reportero de tipo salvaje, A3c, y una mutación de la adenosina a guanina (BS-G). Se generaron un total de nueve cepas para este experimento, cada una con una secuencia de snRNA U6 y un informador ACT1-CUP1 (Tabla suplementaria 4 y Tabla complementaria 5).
Los resultados mostraron que las diferentes bases en la posición 57 del snRNA U6 tienen impactos únicos en el espliceosoma en combinación con una mutación 5' SS o BS (Figura 4). Tanto los reporteros A3c como BS-G inhiben principalmente el empalme estabilizando la conformación del primer paso catalítico14,15. Por lo tanto, U57c es una mutación aditiva que disminuye la tolerancia al cobre en combinación con cualquiera de estos reporteros (Figura 4B). En contraste, U57a aumenta la tolerancia al cobre porque promueve la progresión al segundo paso (Figura 4B)16,32. La disminución de la tolerancia al cobre de la cepa reportera BS-G con U57a en comparación con U6 snRNA WT destaca un probable impacto secundario de BS-G en el segundo paso del empalme16.
Estos resultados también resaltan la naturaleza cualitativa de este ensayo y por qué se deben probar las secuencias de consulta y reportero de tipo salvaje. Si bien el patrón general de aumento o disminución de la tolerancia al cobre es válido para estas mutaciones de ARNn U6 en comparación con el ARNn U6 de tipo salvaje, la concentración exacta de cobre a la que sobreviven las células puede diferir entre los estudios (Tabla 1) y difirió entre estos datos representativos y otros resultados publicados. Esto probablemente se deba a los antecedentes de la cepa que contiene la deleción de CUP1 , pero también puede deberse a la salud general de las cepas antes del recubrimiento (es decir, qué tan pronto después de la generación de la cepa o restreak de crioterapia se realizó el ensayo), las diferencias en cómo se preparan las placas y la variabilidad entre las diferentes incubadoras. Una varianza similar fue observada en Mayerle et al. para las mutaciones de consulta Prp8 en la literatura43. Por lo tanto, la comparación de las tendencias de tolerancia al cobre se puede realizar para diferentes ensayos ACT1-CUP1, pero la comparación numérica de las concentraciones de cobre debe hacerse solo dentro del mismo laboratorio y, a veces, con el mismo conjunto de placas de cobre.
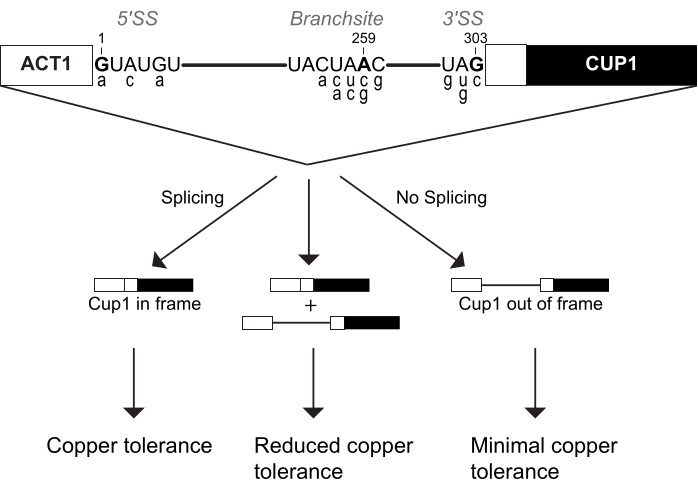
Figura 1: El diseño del reportero ACT1-CUP1. La concentración de reportero empalmado se correlaciona directamente con la tolerancia al cobre de levadura. El diagrama del reportero incluye los tres sitios de empalme del sitio de empalme de 5' (5' SS), sitio de ramificación (BS) y sitio de empalme de 3' (3' SS). Se muestran las secuencias de consenso de levadura para estos sitios, con aquellos a los que se dirige la escisión indicados en negrita y numerados según su ubicación en el intrón. Las secuencias de reporteros ACT1-CUP1 sin consenso comúnmente utilizadas se muestran en minúsculas debajo de sus correspondientes ubicaciones de secuencias de consenso y se enumeran en la Tabla 1. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Flujo de trabajo del ensayo ACT1-CUP1. Para este ensayo, una cepa de levadura debe ser cup1Δ leu2 y contener el plásmido reportero ACT1-CUP1 deseado y el gen QSP, etiquetado Gen en la figura. El gen QSP debe insertarse genómicamente o en un plásmido. El protocolo que utiliza un replicador de pines implica cuatro pasos para preparar las células de levadura. El paso 1 es hacer crecer las células hasta la saturación. El paso 2 es medir el OD 600 del cultivo y diluir a un OD600 de 0.5. El paso 3 es distribuir sobre una placa de 96 pocillos. El paso 4 es planchar en placas que contengan concentraciones crecientes de cobre (indicado con color azul creciente). El paso 1, el paso 2 y el paso 4 serían idénticos para el pipeteo manual. Una vez emplatadas, las placas se incuban durante 3 días a 30 °C y luego se marcan para determinar su viabilidad. Haga clic aquí para ver una versión más grande de esta figura.

Figura 3: Vistas representativas de los datos de ACT1-CUP1. Para este experimento, los espliceosomas de levadura son desafiados con dos mutaciones diferentes de U6 snRNA y probados en presencia de tres reporteros sin consenso. Tres réplicas de este ensayo se presentan en las mismas placas con fines de demostración. Se recomienda para este ensayo hacer réplicas en placas separadas y en días separados. (A) Se muestra un ensayo completo con un gradiente de cobre de 0 mM a 1,1 mM sobre 20 placas. A medida que aumenta la concentración de cobre, las cepas con un empalme más bajo muestran una confluencia disminuida hasta el punto en que la concentración de cobre se vuelve letal. El fondo de levadura era ade2 y, por lo tanto, las colonias maduras son de color rojo. (B) Comparación de las mismas placas a 0 mM y 0,1 mM CuSO4 fotografiadas con una cámara de mano versus un sistema de imágenes digitales. Este es un ejemplo de cómo un número de reporteros no consensuados pueden ser comparados lado a lado y con el reportero de tipo salvaje. Las observaciones comunes en las placas incluyen una gota espuria de medios de cultivo que cayeron del replicador de pines (flecha roja) y un ligero ovalamiento de las colonias debido al deslizamiento del pasador a lo largo de la superficie de la placa o al movimiento de la placa antes de que la gota de cultivo se haya secado lo suficiente (flecha naranja). (C) Ejemplo del efecto del reportero A3c sobre la viabilidad celular en comparación con el reportero de tipo salvaje. Las imágenes como las que se muestran en (B) se recortan y alinean para resaltar las diferencias de crecimiento en diferentes concentraciones de cobre. La tolerancia al cobre de la cepa reportera A3c disminuye a 0,15 mM Cu2+ en comparación con la viabilidad de la cepa reportera de tipo salvaje hasta el final del rango probado. Haga clic aquí para ver una versión más grande de esta figura.
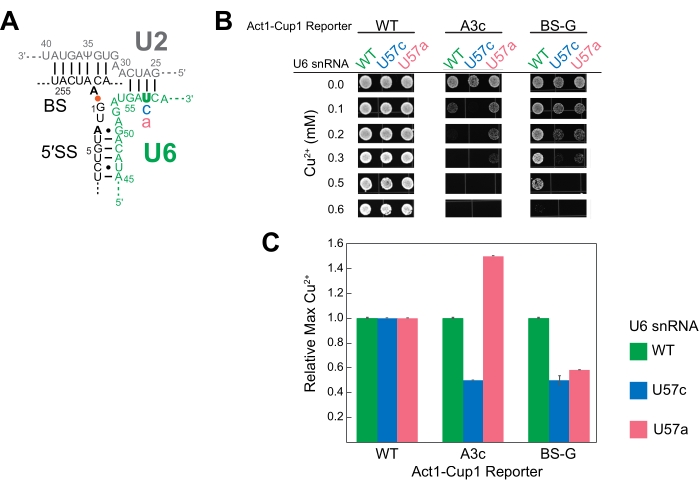
Figura 4: Resultados representativos de ACT1-CUP1 monitoreando el empalme en levaduras con mutaciones del componente de empalme . (A) Esquema de los componentes de ARN del sitio activo inmediatamente después del primer paso catalítico. Los snRNAs U2 y U6 son dúplex, acercando los 5' SS y BS del intrón. El enlace intrónico entre A259 (BS) y G1 (5' SS) se indica con el punto naranja. Las ubicaciones de las sustituciones de secuencia no consensuadas en el reportero ACT1-CUP1 probado en este experimento se indican en negrita negra. La posición mutada en U6 snRNA (U57) está en negrita verde, y las bases sustituidas están en azul o rosa. (B) Una posibilidad para la presentación de datos de ACT1-CUP1 en una publicación incluye varias imágenes de colonias de concentraciones relevantes de Cu2+ . El reportero de WT sobrevivió más allá de la concentración de cobre probada para las tres cepas de snRNA U6 consultadas. (C) Un gráfico de barras que compara los efectos por informador y por mutante U6 snRNA. La normalización se realiza para cada reportero ACT1-CUP1 estableciendo la tolerancia al cobre del snRNA U6 WT en 1 y calculando la relación para las mutaciones U57c y U57a. Las barras de error representan la desviación estándar de tres réplicas. Haga clic aquí para ver una versión más grande de esta figura.
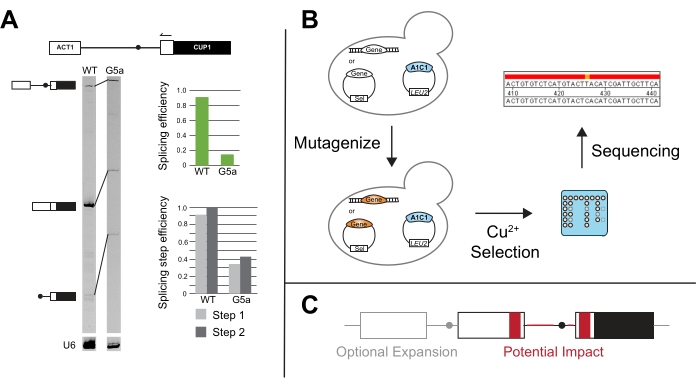
Figura 5: Métodos adicionales para complementar los resultados de ACT1-CUP1. (A) La extensión del cebador se realiza a través de un recocido de cebador en el exón 3' ACT1 y luego se alarga en el intrón. En este ejemplo, la imprimación está marcada al final con colorante IR700, y la extensión de la imprimación se realizó como se describe en 20,21. La extensión del cebador de U6 snRNA se realiza en la misma reacción para servir como control de carga. El gel desnaturalizante de 19:1 bis/acrilamida al 7% resuelve los productos de pre-ARNm, ARNm y lariat después de la extensión del cebador utilizando un dispositivo de imagen de gel infrarrojo cercano como se describe en van der Feltz et al.22. La intensidad de las diferentes bandas se puede utilizar para medir la eficiencia general del empalme, así como para distinguir las diferencias de empalme que se producen en el primer o segundo paso, como se cuantifica con ImageJ44 u otro software de cuantificación de banda de gel. Tanto la eficiencia de empalme como la eficiencia del paso de empalme se calculan como se describe en Query y Konarska14. (B) Las pantallas mutacionales con reporteros ACT1-CUP1 pueden utilizar la selección de cobre para identificar mutantes que afectan el empalme. Los genes de interés se pueden secuenciar en las cepas resultantes para determinar si la mutación ocurrió en ese gen. (C) Se pueden hacer alteraciones al informador fuera de los sitios de empalme para monitorear los cambios de empalme en relación con la estructura del intronte, las secuencias exónicas, el número de exones o la exportación nuclear de ARN no empalmado. Las regiones en gris son regiones de expansión opcionales para incluir en un reportero, y las áreas en rojo son regiones en las que la secuencia y los cambios estructurales podrían probarse para determinar su impacto potencial en el empalme utilizando el ensayo ACT1-CUP1. Haga clic aquí para ver una versión más grande de esta figura.
| Nombre del reportero | Región intrónica | Secuencia | Última concentración viable de Cu2+ (mM) | |
| WT | 5' | GUAUGU | > 2.5 | |
| G1a | 5ʹ | un UAUGU | 0,0133 o 0,0532 | |
| A3c | 5ʹ | GUcUGU | 0.15^,18 o 0.216 | |
| G5a | 5ʹ | GUAUaU | 0,303 ó 0,259 | |
| WT | Sucursal | UACUAAC | > 2.5 | |
| C256a | Sucursal | UAaUAAC | 0,1526 ó 0,1832 | |
| U257c/a | Sucursal | UACc/dAAC | 0,226 o 0,332 ó 0,545 | 0,059,32 |
| A258c/u | Sucursal | UACUc/uAC | 0,826 | 1,045 ó 1,646 |
| BS-C | Sucursal | UACUAcC | 0,153 o 0,1832,56 ó 0,2 26 | |
| BS-G | Sucursal | UACUAgC | 0,0516,32 o 0,625 ó 0,846 | |
| C260g | Sucursal | UACUAAg | 0,826 | |
| WT | 3' | UAG | > 2.5 | |
| U301g | 3ʹ | G AG | 0,1518 | |
| A302g/u | 3' | Ug/uG | 0,0139 | 0,07516 o 0,1824 |
| G303c | 3ʹ | UAc | 0,0532 | |
Tabla 1: Lista de reporteros comunes y la letalidad de la concentración de Cu2+ reportada. Hay más de 100 citas de Lesser y Guthrie3. Si bien se utilizó una pequeña selección de estas citas para crear esta tabla, resaltan la tendencia general de diferencias leves a moderadas entre los estudios en las concentraciones de viabilidad de cobre informadas. En ACT1-CUP1, es importante comparar la proteína o ARN de tipo salvaje con los mutantes, todos con el mismo fondo de cepa de levadura utilizando las concentraciones publicadas como guía, pero anticipando que las concentraciones observadas pueden diferir. Datos recopilados de la Figura 3 (^) y múltiples publicaciones anotadas por las siguientes citas 3,9,16,18,24,25,26,32,45,46.
Tabla suplementaria 1: Contenido de una sola placa de cobre y un ejemplo de cálculos realizados para lograr diluciones de cobre de 0 mM a 2,5 mM a partir de una matriz de CuSO4 de 1 M. Haga clic aquí para descargar esta tabla.
Tabla complementaria 2: Ejemplo de un esquema de recubrimiento para el replicador de 48 pines. Haga clic aquí para descargar esta tabla.
Tabla suplementaria 3: Ejemplo de viabilidad obtenida de placas de cobre incubadas durante 3 días a 30°C. Haga clic aquí para descargar esta tabla.
Tabla complementaria 4: Lista de cepas de levadura utilizadas para generar los datos representativos. Haga clic aquí para descargar esta tabla.
Tabla complementaria 5: Lista de plásmidos utilizados para generar los datos representativos. Plásmidos de ARNn U6 generados y publicados en investigaciones previas22,47. Haga clic aquí para descargar esta tabla.
Discusión
ACT1-CUP1 es un ensayo de crecimiento, y se debe tener cuidado para garantizar que las diferencias de crecimiento observadas solo puedan atribuirse a defectos de empalme. Todas las cepas deben manipularse de manera similar antes del enchapado, incluyendo tener una longitud y un tipo similares de crecimiento y condiciones de almacenamiento. Si se utilizan cepas sensibles a la temperatura, los ensayos ACT1-CUP1 solo deben realizarse en condiciones en las que esas cepas crezcan de manera comparable al tipo silvestre. Relacionadamente, para el componente QSP, se recomienda tener fondos de levadura idénticos y niveles de expresión de genes QSP para no nublar la interpretación de los resultados. Al considerar el número de QSP y reporteros a utilizar, no se recomienda probar más de 30 cepas a la vez con el método del replicador de pines. Con el tiempo adicional para realizar cada paso, las células se asentarán y los resultados del ensayo serán inconsistentes debido a la disminución de la viabilidad celular.
Además de las limitaciones que ACT1-CUP1 tiene inherentemente como ensayo de crecimiento, un QSP puede tener un impacto más complejo en el empalme que el que se puede resolver con este método. Como el empalme es un proceso de varios pasos y los factores se reciclan, el fenotipo observado puede dar lugar a que la perturbación afecte a más de un paso de empalme. Esto es cierto incluso para los análisis posteriores que pueden seguir a ACT1-CUP1, algunos de los cuales se describen a continuación. Los datos resaltarán el paso más perturbado del proceso, aunque otros pasos pueden verse afectados por la función alterada del factor de empalme.
La extensión de la imprimación de los reporteros ACT1-CUP1 se utilizó por primera vez en el artículo original de Lesser y Guthrie y a menudo se realiza si los resultados de ACT1-CUP1 muestran un defecto de crecimiento3. Este ensayo se dirige al informador y utiliza la longitud de los productos de PCR para determinar las cantidades relativas de informador sin empalmar, parcialmente empalmado y completamente empalmado presente (Figura 5A). La eficiencia general del empalme y si el defecto afecta más fuertemente al primer o segundo paso catalítico se calculan tomando las proporciones de las cantidades del producto de PCR14. Por ejemplo, el reportero 5' SS G5a ha reducido el empalme en comparación con el reportero de tipo salvaje, pero sus eficiencias de primer y segundo paso siguen un patrón similar al tipo salvaje (Figura 5A). Esto apunta hacia un defecto previo al primer paso catalítico, posiblemente en el ensamblaje del espliceosoma, porque ambos pasos se ven afectados de manera similar31.
Se han desarrollado nuevos ensayos a partir del ensayo canónico ACT1-CUP1, como la detección de mutantes que mejoran el empalme en presencia de otros mutantes del factor de empalme y/o secuencias de empalme sin consenso (Figura 5B). Por ejemplo, la exposición de levaduras que contenían el reportero ACT1-CUP1 a UV y luego la selección en presencia de cobre produjo mutantes Prp8 y Hsh155 que mejoraron el empalme de secuencias no consensuadas14,19.
Ampliando más allá del estudio del efecto de los sitios de empalme y los factores constitutivos de empalme constitutivo, la dependencia del crecimiento de CUP1 se ha utilizado para estudiar el empalme de intrón múltiple, el control de la exportación nuclear y el impacto de UTR y otras secuencias periféricas en el empalme (Figura 5C). Algunos de estos estudios han creado reporteros con CUP1 y otros genes de levadura que contienen intrón que pueden tener patrones de empalme más complejos para estudiar el efecto sobre la estructura secundaria intrónica y el empalme de transcripción multiintrón 48,49,50,51,52. El vínculo entre el empalme y la exportación nuclear se estudió teniendo transcripciones empalmadas o no empalmadas que codifican CUP1 en el marco53,54. La longitud de la pista de pirimidina y la dependencia de la distancia de la selección del sitio de empalme debido a factores específicos también se ha probado 20,55,56,57. Estos ejemplos y muchos otros resaltan la versatilidad de ACT1-CUP1 y otros ensayos de crecimiento CUP1.
El ensayo ACT1-CUP1 vincula las perturbaciones a un ciclo de reacción complejo con un fenotipo de crecimiento directo con un amplio rango de sensibilidad. Este ensayo heredado ha sido utilizado por múltiples laboratorios para sentar las bases de la comprensión del ciclo de empalme. Más recientemente, ACT1-CUP1 se ha utilizado para responder preguntas que surgen de la gran cantidad de datos estructurales ahora disponibles21,22,25. Los estudios estructurales de los espliceosomas unidos a secuencias sin consenso podrían emparejarse con los resultados de ACT1-CUP1 para interpretar cómo se correlacionan la estructura alterada y la función alterada. ACT1-CUP1 es una primera prueba ideal para empalmar mutaciones que pueden complementar un análisis más complejo.
Divulgaciones
El autor no tiene nada que revelar.
Agradecimientos
Gracias a Aaron Hoskins y a los miembros del laboratorio Hoskins de la Universidad de Wisconsin-Madison por el uso de cepas y equipos de levadura en la generación de las figuras 3-5. Gracias a Harpreet Kaur y Xingyang Fu por sus perspicaces comentarios sobre el manuscrito. Gracias a los estudiantes, el personal y la facultad de apoyo de la Universidad del Noroeste durante la escritura, edición y filmación de este documento. Gracias a Isabelle Marasigan por su ayuda en la filmación de este método.
Materiales
| Name | Company | Catalog Number | Comments |
| 1.5 mL sterile microcentrifuge tubes | Fisher Scientific | 05-408-129 | Or comparable item from a different manufacturer. |
| 2 mL sterile microcentrifuge tubes | Fisher Scientific | 05-408-138 | Or comparable item from a different manufacturer. |
| 50 mL sterile centrifuge tubes | Fisher Scientific | 07-201-332 | Or comparable item from a different manufacturer. |
| 96-well round bottom microplate | Fisher Scientific | 07-200-760 | Or comparable item from a different manufacturer. |
| 190 proof ethanol | Fisher Scientific | 22-032-600 | Or comparable item from a different manufacturer. |
| 500 mL Filter System (0.22 µm) | CellTreat Scientific Products | 229707 | Or comparable item from a different manufacturer. |
| Agar | Fisher Scientific | BP1423-500 | Any molecular grade agar will work. |
| Autoclave | Tuttnauer | 3870EA | Or comparable item from a different manufacturer. |
| Bunsen burner | Humboldt | PN6200.1 | Or comparable item from a different manufacturer. |
| Cell Density Meter | VWR | 490005-906 | Or other spectral device that can measure absorbance at 595 nm. |
| Copper sulfate Pentahydrate | Fisher Scientific | LC134051 | Or comparable item from a different manufacturer. |
| Digital imaging system | Cytiva | 29399481 | ImageQuant 4000 (used for Figure 3), Amersham ImageQuant 800, or comparable item from a different manufacturer. |
| Dropout mix (-Leu) | USBiological Life Sciences | D9525 | Use the appropriate drop out mix for your experiment. It is possible you will be using a yeast nutrient marker for your query perturbation also. In that case, the drop out mix should be for that marker and Leu |
| D-Glucose | Fisher Scientific | AAA1682836 | Or comparable item from a different manufacturer. |
| Gel band quantifying software | Cytiva | 29-0006-05 | ImageQuant TL v8.1 (used for figure 5A) or comparable item from a different manufacturer. |
| Hand held camera | Nikon | D3500 | Or comparable item from a different manufacturer. |
| Near infra-red gel imaging device | Cytiva | 29238583 | Amersham Typhoon NIR (used for Figure 5a) or comparable item from a different manufacturer. |
| Laboratory grade clamp | Fisher Scientific | 05-769-7Q | Or comparable item from a different manufacturer. |
| Laboratory grade stand and clamp | Fisher Scientific | 12-000-101 | Or comparable item from a different manufacturer. |
| Magnetic stir bars | Fisher Scientific | 14-513-51 | Or comparable item from a different manufacturer. |
| Pin replicator | VP Scientific | VP 407AH | |
| Semi-micro disposable cuvettes | VWR | 97000-590 | Or comparable item from a different manufacturer. |
| Shaker | JEIO Tech | IST-3075 | Or comparable item from a different manufacturer. |
| Spectrophotometer | Biowave | 80-3000-45 | Or any spectophotometer that can measure the absorbance at 600 nm. |
| Square plates | VWR | 102091-156 | Circular plates may also be used though are more challenging if using a pin replicator. |
| Stir plate | Fisher Scientific | 11-520-16S | Or comparable item from a different manufacturer. |
| Yeast nitrogen base | USBiological Life Sciences | Y2025 | Or comparable item from a different manufacturer. |
Referencias
- Wahl, M. C., Will, C. L., Luhrmann, R. The spliceosome: Design principles of a dynamic RNP machine. Cell. 136 (4), 701-718 (2009).
- Wilkinson, M. E., Charenton, C., Nagai, K. RNA splicing by the spliceosome. Annual Review of Biochemistry. 89, 359-388 (2020).
- Lesser, C. F., Guthrie, C. Mutational analysis of pre-mRNA splicing in Saccharomyces cerevisiae using a sensitive new reporter gene, CUP1. Genetics. 133 (4), 851-863 (1993).
- Ng, R., Abelson, J. Isolation and sequence of the gene for actin in Saccharomyces cerevisiae. Proceedings of the National Academy of Sciences of the United States of America. 77 (7), 3912-3916 (1980).
- Gallwitz, D., Sures, I. Structure of a split yeast gene: Complete nucleotide sequence of the actin gene in I. Proceedings of the National Academy of Sciences of the United States of America. 77 (5), 2546-2550 (1980).
- Fogel, S., Welch, J. W., Cathala, G., Karin, M. Gene amplification in yeast: CUP1 copy number regulates copper resistance. Current Genetics. 7 (5), 347-355 (1983).
- Hamer, D. H., Thiele, D. J., Lemontt, J. E. Function and autoregulation of yeast copperthionein. Science. 228 (4700), 685-690 (1985).
- Winge, D. R., Nielson, K. B., Gray, W. R., Hamer, D. H. Yeast metallothionein. Sequence and metal-binding properties. Journal of Biological Chemistry. 260 (27), 14464-14470 (1985).
- Burgess, S. M., Guthrie, C. A mechanism to enhance mRNA splicing fidelity: The RNA-dependent ATPase Prp16 governs usage of a discard pathway for aberrant lariat intermediates. Cell. 73 (7), 1377-1391 (1993).
- Collins, C. A., Guthrie, C. Allele-specific genetic interactions between Prp8 and RNA active site residues suggest a function for Prp8 at the catalytic core of the spliceosome. Genes & Development. 13 (15), 1970-1982 (1999).
- Siatecka, M., Reyes, J. L., Konarska, M. M. Functional interactions of Prp8 with both splice sites at the spliceosomal catalytic center. Genes & Development. 13 (15), 1983-1993 (1999).
- Umen, J. G., Guthrie, C. Mutagenesis of the yeast gene PRP8 reveals domains governing the specificity and fidelity of 3' splice site selection. Genetics. 143 (2), 723-739 (1996).
- Grainger, R. J., Beggs, J. D. Prp8 protein: At the heart of the spliceosome. RNA. 11 (5), 533-557 (2005).
- Query, C. C., Konarska, M. M. Suppression of multiple substrate mutations by spliceosomal prp8 alleles suggests functional correlations with ribosomal ambiguity mutants. Molecular Cell. 14 (3), 343-354 (2004).
- Konarska, M. M., Vilardell, J., Query, C. C. Repositioning of the reaction intermediate within the catalytic center of the spliceosome. Molecular Cell. 21 (4), 543-553 (2006).
- Liu, L., Query, C. C., Konarska, M. M. Opposing classes of prp8 alleles modulate the transition between the catalytic steps of pre-mRNA splicing. Nature Structural and Molecular Biology. 14 (6), 519-526 (2007).
- MacRae, A. J., et al. Prp8 positioning of U5 snRNA is linked to 5' splice site recognition. RNA. 24 (6), 769-777 (2018).
- Query, C. C., Konarska, M. M. CEF1/CDC5 alleles modulate transitions between catalytic conformations of the spliceosome. RNA. 18 (5), 1001-1013 (2012).
- Tang, Q., et al. SF3B1/Hsh155 HEAT motif mutations affect interaction with the spliceosomal ATPase Prp5, resulting in altered branch site selectivity in pre-mRNA splicing. Genes & Development. 30 (24), 2710-2723 (2016).
- Carrocci, T. J., Zoerner, D. M., Paulson, J. C., Hoskins, A. A. SF3b1 mutations associated with myelodysplastic syndromes alter the fidelity of branchsite selection in yeast. Nucleic Acids Research. 45 (8), 4837-4852 (2017).
- Kaur, H., Groubert, B., Paulson, J. C., McMillan, S., Hoskins, A. A. Impact of cancer-associated mutations in Hsh155/SF3b1 HEAT repeats 9-12 on pre-mRNA splicing in Saccharomyces cerevisiae. PLoS One. 15 (4), 0229315(2020).
- vander Feltz, C., et al. Saccharomyces cerevisiae Ecm2 modulates the catalytic steps of pre-mRNA splicing. RNA. 27 (5), 591-603 (2021).
- Carrocci, T. J., Paulson, J. C., Hoskins, A. A. Functional analysis of Hsh155/SF3b1 interactions with the U2 snRNA/branch site duplex. RNA. 24 (8), 1028-1040 (2018).
- Hogg, R., de Almeida, R. A., Ruckshanthi, J. P., O'Keefe, R. T. Remodeling of U2-U6 snRNA helix I during pre-mRNA splicing by Prp16 and the NineTeen Complex protein Cwc2. Nucleic Acids Research. 42 (12), 8008-8023 (2014).
- Hansen, S. R., Nikolai, B. J., Spreacker, P. J., Carrocci, T. J., Hoskins, A. A. Chemical inhibition of pre-mRNA splicing in living Saccharomyces cerevisiae. Cell Chemical Biology. 26 (3), 443-448 (2019).
- Xu, Y. Z., Query, C. C. Competition between the ATPase Prp5 and branch region-U2 snRNA pairing modulates the fidelity of spliceosome assembly. Molecular Cell. 28 (5), 838-849 (2007).
- Staley, J. P., Guthrie, C. An RNA switch at the 5' splice site requires ATP and the DEAD box protein Prp28p. Molecular Cell. 3 (1), 55-64 (1999).
- Bousquet-Antonelli, C., Presutti, C., Tollervey, D. Identification of a regulated pathway for nuclear pre-mRNA turnover. Cell. 102 (6), 765-775 (2000).
- Villa, T., Guthrie, C. The Isy1p component of the NineTeen complex interacts with the ATPase Prp16p to regulate the fidelity of pre-mRNA splicing. Genes & Development. 19 (16), 1894-1904 (2005).
- Mayas, R. M., Maita, H., Staley, J. P. Exon ligation is proofread by the DExD/H-box ATPase Prp22p. Nature Structure and Molecular Biology. 13 (6), 482-490 (2006).
- Lesser, C. F., Guthrie, C. Mutations in U6 snRNA that alter splice site specificity: Implications for the active site. Science. 262 (5142), 1982-1988 (1993).
- McPheeters, D. S. Interactions of the yeast U6 RNA with the pre-mRNA branch site. RNA. 2 (11), 1110-1123 (1996).
- Perriman, R. J., Ares, M. Rearrangement of competing U2 RNA helices within the spliceosome promotes multiple steps in splicing. Genes & Development. 21 (7), 811-820 (2007).
- Mefford, M. A., Staley, J. P. Evidence that U2/U6 helix I promotes both catalytic steps of pre-mRNA splicing and rearranges in between these steps. RNA. 15 (7), 1386-1397 (2009).
- Hilliker, A. K., Mefford, M. A., Staley, J. P. U2 toggles iteratively between the stem IIa and stem IIc conformations to promote pre-mRNA splicing. Genes & Development. 21 (7), 821-834 (2007).
- Wu, G., et al. Pseudouridines in U2 snRNA stimulate the ATPase activity of Prp5 during spliceosome assembly. EMBO Journal. 35 (6), 654-667 (2016).
- Crotti, L. B., Bacikova, D., Horowitz, D. S. The Prp18 protein stabilizes the interaction of both exons with the U5 snRNA during the second step of pre-mRNA splicing. Genes & Development. 21 (10), 1204-1216 (2007).
- Fogel, S., Welch, J. W. Tandem gene amplification mediates copper resistance in yeast. Proceedings of the National Academy of Sciences of the United States of America. 79 (17), 5342-5346 (1982).
- Gardner, J. M., Jaspersen, S. L. Manipulating the yeast genome: Deletion, mutation, and tagging by PCR. Methods Molecular Biology. 1205, 45-78 (2014).
- JoVE. Yeast Transformation and Cloning. In Biology I: yeast, Drosophila and C. Elegant. JoVE Science Education Database. , Cambridge, MA. (2021).
- Gietz, R. D., Woods, R. A. Yeast transformation by the LiAc/SS Carrier DNA/PEG method. Methods Molecular Biology. 313, 107-120 (2006).
- Gietz, R. D., Woods, R. A. Genetic transformation of yeast. Biotechniques. 30 (4), 816(2001).
- Mayerle, M., et al. Structural toggle in the RNaseH domain of Prp8 helps balance splicing fidelity and catalytic efficiency. Proceedings of the National Academy of Sciences of the United States of America. 114 (18), 4739-4744 (2017).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Beier, D. H., et al. Dynamics of the DEAD-box ATPase Prp5 RecA-like domains provide a conformational switch during spliceosome assembly. Nucleic Acids Research. 47 (20), 10842-10851 (2019).
- vander Feltz, C., DeHaven, A. C., Hoskins, A. A. Stress-induced pseudouridylation alters the structural equilibrium of yeast U2 snRNA Stem II. Journal of Molecular Biology. 430 (4), 524-536 (2018).
- Rodgers, M. L., Didychuk, A. L., Butcher, S. E., Brow, D. A., Hoskins, A. A. A multi-step model for facilitated unwinding of the yeast U4/U6 RNA duplex. Nucleic Acids Research. 44 (22), 10912-10928 (2016).
- Stutz, F., Rosbash, M. A functional interaction between Rev and yeast pre-mRNA is related to splicing complex formation. EMBO Journal. 13 (17), 4096-4104 (1994).
- Libri, D., Lescure, A., Rosbash, M. Splicing enhancement in the yeast rp51b intron. RNA. 6 (3), 352-368 (2000).
- Libri, D., Stutz, F., McCarthy, T., Rosbash, M. RNA structural patterns and splicing: Molecular basis for an RNA-based enhancer. RNA. 1 (4), 425-436 (1995).
- Howe, K. J., Kane, C. M., Ares, M. Perturbation of transcription elongation influences the fidelity of internal exon inclusion in Saccharomyces cerevisiae. RNA. 9 (8), 993-1006 (2003).
- Cuenca-Bono, B., et al. SUS1 introns are required for efficient mRNA nuclear export in yeast. Nucleic Acids Research. 39 (19), 8599-8611 (2011).
- Scherrer, F. W., Spingola, M. A subset of Mer1p-dependent introns requires Bud13p for splicing activation and nuclear retention. RNA. 12 (7), 1361-1372 (2006).
- Hálová, M., et al. Nineteen complex-related factor Prp45 is required for the early stages of cotranscriptional spliceosome assembly. RNA. 23 (10), 1512-1524 (2017).
- Umen, J. G., Guthrie, C. A novel role for a U5 snRNP protein in 3' splice site selection. Genes & Development. 9 (7), 855-868 (1995).
- Crotti, L. B., Horowitz, D. S. Exon sequences at the splice junctions affect splicing fidelity and alternative splicing. Proceedings of the National Academy of Sciences of the United States of America. 106 (45), 18954-18959 (2009).
- Perriman, R., Ares, M. Invariant U2 snRNA nucleotides form a stem loop to recognize the intron early in splicing. Molecular Cell. 38 (3), 416-427 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados