Method Article
Aufnahme fluoreszierender markierter kleiner extrazellulärer Vesikel in vitro und im Rückenmark
In diesem Artikel
Zusammenfassung
Wir beschreiben ein Protokoll, um aus Makrophagen gewonnene kleine extrazelluläre Vesikel mit PKH-Farbstoffen zu kennzeichnen und ihre Aufnahme in vitro und im Rückenmark nach intrathekaler Entbindung zu beobachten.
Zusammenfassung
Kleine extrazelluläre Vesikel (sEVs) sind 50-150 nm Vesikel, die von allen Zellen abgesondert werden und in Körperflüssigkeiten vorhanden sind. sEVs übertragen Biomoleküle wie RNA, Proteine und Lipide von Donor- zu Akzeptorzellen und sind damit wichtige Signalvermittler zwischen Zellen. Im zentralen Nervensystem (ZNS) können sEVs interzelluläre Signalübertragungen vermitteln, einschließlich Neuroimmuninteraktionen. sEV-Funktionen können untersucht werden, indem die Aufnahme von markierten sEVs in Empfängerzellen sowohl in vitro als auch in vivoverfolgt wird. Dieser Artikel beschreibt die Markierung von sEVs aus den konditionierten Medien von RAW 264.7-Makrophagenzellen unter Verwendung eines PKH-Membranfarbstoffs. Es zeigt die Aufnahme unterschiedlicher Konzentrationen markierter sEVs zu mehreren Zeitpunkten durch Neuro-2a-Zellen und primäre Astrozyten in vitro. Ebenfalls gezeigt wird die Aufnahme von sEVs, die intrathekal in Rückenmarksneuronen, Astrozyten und Mikroglia der Maus abgegeben werden, die durch konfokale Mikroskopie visualisiert werden. Die repräsentativen Ergebnisse zeigen zeitabhängige Variationen in der Aufnahme von sEVs durch verschiedene Zellen, was dazu beitragen kann, eine erfolgreiche sEVs-Abgabe in das Rückenmark zu bestätigen.
Einleitung
Kleine extrazelluläre Vesikel (sEVs) sind nanogroße, membranbasierte Vesikel mit einem Größenbereich von 50-150 nm. Sie stammen aus multi-vesikulären Körpern (MVBs) und werden bei der Fusion der MVBs mit der Plasmamembran aus Zellen freigesetzt. sEVs enthalten miRNAs, mRNAs, Proteine und bioaktive Lipide, und diese Moleküle werden in Form von Zell-zu-Zell-Kommunikation zwischen Zellen übertragen. sEVs können von Empfängerzellen durch eine Vielzahl von endozytischen Wegen internalisiert werden, und diese Erfassung von sEVs durch Empfängerzellen wird durch die Erkennung von Oberflächenmolekülen sowohl auf EVs als auch auf den Zielzellenvermittelt 1.
sEVs haben aufgrund ihrer Fähigkeit, molekulare und phänotypische Veränderungen in Akzeptorzellen auszulösen, ihres Nutzens als therapeutisches Mittel und ihres Potenzials als Träger für Frachtmoleküle oder pharmakologische Wirkstoffe an Interesse gewonnen. Aufgrund ihrer geringen Größe kann die Bildgebung und Verfolgung von sEVs eine Herausforderung darstellen, insbesondere für In-vivo-Studien und klinische Umgebungen. Daher wurden viele Methoden entwickelt, um sEVs zu kennzeichnen und abbilden, um ihre Bioverteilung und Verfolgung in vitro und in vivo2zu unterstützen.
Die gebräuchlichste Technik zur Untersuchung der sEV-Bioverteilung und der Wechselwirkungen mit Zielzellen bestehtdarin,sie mit fluoreszierenden Farbstoffmolekülen3,4,5,6,7zukennzeichnen. EVs wurden zunächst mit Zellmembranfarbstoffen markiert, die üblicherweise zur Abbildung von Zellen verwendet wurden. Diese fluoreszierenden Farbstoffe färben im Allgemeinen die Lipiddoppelschicht oder Proteine von Interesse auf sEVs. Mehrere lipophile Farbstoffe zeigen ein starkes fluoreszierendes Signal, wenn sie in das Zytosol eingebaut werden, darunter DiR (1,1′-Dioctadecyl-3,3,3′,3′-tetramethylindotricarbocyaniniodid), DiL (1 ,1′-Dioctadecyl-3, 3,3′, 3′-Tetramethylindocarbocyaninperchlorat) und DiD (1 ,1′-Dioctadecyl-3, 3,3′,3′-Tetramethylindocarbocyanin-4-Chlorbenzolsulfonatsalz)8,9,10,11.
Andere lipophile Farbstoffe, wie PKH67 und PKH26, haben eine hoch fluoreszierende polare Kopfgruppe und einen langen aliphatischen Kohlenwasserstoffschwanz, der sich leicht in jede Lipidstruktur einfüg und zu einer langfristigen Farbstoffretention und stabilen Fluoreszenz führt12. PKH-Farbstoffe können auch EVs kennzeichnen, was die Untersuchung der EV-Eigenschaften in vivo13ermöglicht. Viele andere Farbstoffe wurden verwendet, um Exosomen mittels Fluoreszenzmikroskopie und Durchflusszytometrie zu beobachten, einschließlich Lipidmarkierungsfarbstoffe14 und zelldurchlässige Farbstoffe wie Carboxyfluoresceindiacetat-Succinimidylester (CFDA-SE)15,16 und Calceinacetoxymethyl (AM)-Ester17.
Untersuchungen des sEV-vermittelten Crosstalks zwischen verschiedenen Zellen im ZNS haben wichtige Erkenntnisse über die Pathogenese neuroinflammatoratorischer und neurodegenerativer Erkrankungen geliefert18. Zum Beispiel können sEVs aus Neuronen Beta-Amyloid-Peptide und phosphorylierte Tau-Proteine verbreiten und bei der Pathogenese der Alzheimer-Krankheithelfen 19. Darüber hinaus enthalten EVs, die aus Erythrozyten gewonnen werden, große Mengen an Alpha-Synuclein und können die Blut-Hirn-Schranke überwinden und zur Parkinson-Pathologie beitragen20. Die Fähigkeit von sEVs, physiologische Barrieren21 zu überwinden und ihre Biomoleküle auf Zielzellen zu übertragen, macht sie zu bequemen Werkzeugen, um therapeutische Medikamente an das ZNS22zu liefern.
Die Visualisierung der sEV-Aufnahme durch unzählige ZNS-Zellen im Rückenmark ermöglicht sowohl mechanistische Studien als auch die Bewertung des therapeutischen Nutzens exogen verabreichter sEVs aus verschiedenen zellulären Quellen. Dieser Artikel beschreibt die Methodik zur Markierung von sEVs, die von Makrophagen abgeleitet sind, und zur Abbildung ihrer Aufnahme in vitro und in vivo im Lendenwirbelmark durch Neuronen, Mikroglia und Astrozyten, um die sEV-Abgabe durch Visualisierung qualitativ zu bestätigen.
Protokoll
HINWEIS: Alle Verfahren wurden in Übereinstimmung mit dem NIH-Leitfaden für die Pflege und Verwendung von Labortieren durchgeführt und vom Institutional Animal Care & Use Committee des Drexel University College of Medicine genehmigt. Zeitschwangere CD-1-Mäuse wurden für die astrozytäre Kultur verwendet, und alle Dämme wurden 15 Tage nach der Imprägnierung erhalten. Zehn bis zwölf Wochen alte C57BL/6-Mäuse wurden für In-vivo-Aufnahmeexperimente verwendet.
1. Isolierung von sEVs aus RAW 264.7 Makrophagenzellen
- Kultur RAW 264,7 Zellen in 75 cm2 Kolben in DMEM Exosomen-erschöpftem Medium mit 10% Exosomen-depleted fetal bovine serum (FBS) und 1% Penicillin-Streptomycin (Pen-Streptokokken) für 24-48 h.
- Sammeln Sie 300 ml konditioniertes Medium und Zentrifuge bei 300 × g für 10 min bei 4 °C.
- Überstand und Zentrifuge bei 2.000 × g für 20 min bei 4 °C sammeln.
- Den Überstand in Zentrifugenröhrchen überführen, 35 min bei 12.000 × g bei 4 °C zentrifugieren.
- Sammeln Sie den Überstand und filtern Sie durch einen 0,22 μm Spritzenfilter.
- Übergabe in Ultrazentrifugenröhrchen und Zentrifuge für 80 min bei 110.000 × g bei 4 °C.
- Lagern Sie den Überstand (exosomenarmes Medium), resuspendieren Sie das Pellet in 2 mL 1x phosphatgepufferter Kochsalzlösung (PBS) und zentrifugieren Sie für 1 h bei 110.000 × g bei 4 °C.
- Resuspendieren Sie das Pellet in 100 μL PBS zur weiteren Charakterisierung mittels Nanopartikel-Tracking-Analyse (NTA) und Transmissionselektronenmikroskopie (TEM) oder im RipA-Puffer (Radioimmunoprecipitation Assay) für Western Blotting.
2. Charakterisierung von sEVs
- Nanopartikel-Tracking-Analyse (NTA)
HINWEIS: Die Größenverteilung und Partikelanzahl/Konzentration der gereinigten sEVs aus RAW 264.7-Zellen wurden mit NTA gemessen.- Verdünnen Sie die sEVs in gefiltertes PBS, um 20-60 Vesikel pro Sichtfeld für eine optimale Verfolgung zu erhalten.
- Die verdünnte Probe wird mit einer Spritzenpumpe mit konstantem Durchfluss in eine Durchflusszelle einführt.
- Nehmen Sie 3-5 Videos von jeweils 30 s auf. Stellen Sie die Verschlusszeit und Verstärkung ein und fokussieren Sie die Kameraeinstellungen manuell, damit die maximale Anzahl von Vesikeln sichtbar ist und verfolgt und analysiert werden kann.
- Führen Sie die Proben zwischen den einzelnen Aufzeichnungen auf, um Replikationsmessungen durchzuführen. Optimieren Sie die NTA-Einstellungen nach der Erfassung und halten Sie die Einstellungen zwischen den Samples konstant.
- Analysieren Sie jedes Video mit der NTA-Software, um die durchschnittliche Größe und Konzentration der Vesikel zu erhalten.
- Führen Sie alle NTA-Messungen mit identischen Systemeinstellungen durch, um Konsistenz zu erhalten.
- Westlicher Schandfleck
- Quantifizieren Sie die Gesamtproteinmengen in sEVs, Zelllysaten und exosomenarmen Medien mit einem Protein-Assay-Kit gemäß den Anweisungen des Herstellers.
- Zur Zelllysatpräparation die RAW 264,7 Zellen in 75 cm2 Kolben bis zu 80-90% konfluent züchtigen. Lösen Sie die Zellen mit 0,25% Trypsin für 10-15 min, neutralisieren Sie das Trypsin mit Nährmedien und pelletieren Sie die Zellen, indem Sie bei 400 × g für 5 min drehen. Resuspendieren Sie die Zellen in frischem Wachstumsmedium.
- Zählen Sie die Zellen mit einem Hämozytometer und übertragen Sie 1 × 106 Zellen in ein anderes Röhrchen. Waschen Sie die Zellen zweimal mit PBS unter den gleichen Zentrifugationsbedingungen wie oben und fügen Sie dem Zellpellet aus dem letzten Spin 50 μL Lysepuffer (RIPA-Puffer mit Protease-Inhibitor-Cocktail hinzugefügt) hinzu.
- Wirbeln Sie die Zellen vor und halten Sie sie für 20 Minuten auf Eis. Das Gemisch bei 10.000 × g für 30 min bei 4 °C zentrifugieren, den Überstand (d.h. das Lysat) in frischen Mikrozentrifugenröhrchen sammeln und bis zur Verwendung bei −80 °C aufbewahren.
- Konzentrieren Sie 2 ml exosomenarme Medien auf 100 μL mit 3 kDa-cutoff-Zentrifugalfiltern, bevor Sie die Proteinmenge quantifizieren. Mischen Sie die sEVs mit Lysepuffer im Verhältnis 1: 1, Wirbel für 30 s und inkubieren Sie auf Eis für 15 Minuten, um die Menge an Protein zu quantifizieren.
- Mischen Sie gleiche Mengen an Protein (2 μg) der sEVs, RAW 264,7 Zelllysat und Exosomen-erschöpfte Medien mit reduzierendem Probenpuffer.
- Denaturieren Sie die Proben bei 95 °C für 5 min, halten Sie sie 5 min auf Eis und drehen Sie sie für 2 min bei 10.000 × g. Laden Sie die Proben auf ein 12% Tris-Glycin-Proteingel und lassen Sie das Gel bei 125 V für 45 minuten laufen.
- Übertragen Sie das Protein auf eine Polyvinylidendifluorid (PVDF) -Membran bei 25 V für 2 h.
- Nach dem Transfer die PVDF-Membranen mit Sperrpuffer (siehe Materialtabelle)für 1 h bei Raumtemperatur blockieren.
- Inkubieren Sie den Blot mit primären Antikörpern auf einem Shaker über Nacht bei 4 °C.
HINWEIS: Primäre Antikörper waren Anti-CD81 (1:1.000), Anti-Alpha-1,3/1,6-Mannosyltransferase (ALG-2)-interagierendes Protein X (Alix) (1:1.000), Anti-Calnexin (1:1.000) und Anti-Glyceraldehyd-3-Phosphat-Dehydrogenase (GAPDH) (1:1.000). - Waschen Sie die Blots 3 x 15 min mit 1x Tris-gepufferter Kochsalzlösung, 0,1% Tween 20 (TBST) und inkubieren Sie bei Raumtemperatur mit Ziegen-Anti-Maus-IgG-Meerrettichperoxidase (HRP)- oder Esel-Anti-Kaninchen-IgG-HRP-konjugierten sekundären Antikörpern (1:10.000) für 1 h auf dem Shaker.
- Waschen Sie die Blots 3 x 15 min mit 1x TBST und detektieren Sie die Proteine mit einem HRP-Substrat.
- Analysieren Sie die Blots durch verbesserte Chemilumineszenz mit einem Western Blot Imager.
- Transmissionselektronenmikroskopie (TEM)
- Fixieren Sie sEVs, indem Sie sie in 2% Paraformaldehyd (PFA) in 0,1 M Phosphatpuffer (PB) resusdieren; Wirbel für 2 x 15 s.
- Legen Sie einen Tropfen 10 μL sEV-Suspension auf sauberen Parafilm. Schweben Sie das kohlenstoffbeschichtete Formvar-Gitter auf dem Tropfen mit der beschichteten Seite zur Aufhängung hin. Lassen Sie die Membranen in trockener Umgebung 20 Minuten einziehen.
- Legen Sie die Gitter (Membranseite nach unten) auf einen Tropfen PB, um sie 3 x 2 min zu waschen.
- Die Gitter für 5 min auf 50 μL 1% Glutaraldehyd übertragen.
- Waschen Sie die Gitter mit 100 μL destilliertem Wasser für 8 x 2 min.
- Kontrastieren Sie die Probe, indem Sie die Gitter für 2 min auf einen Tropfen von 1% Uranylacetat legen.
- Betten Sie die Probe mit 50 μL 0,2% Uranylacetat mit 2% Methylcelluloselösung für 10 min auf eine mit Parafilm bedeckte Eisschale ein.
- Verwenden Sie Edelstahlschlaufen, um die Gitter zu halten und überschüssige Flüssigkeit mit Filterpapier zu entfernen.
- Trocknen Sie das Gitter für 10 Minuten an der Luft, während Sie sich noch auf der Schleife beflüchte.
- Unter einem Transmissionselektronenmikroskop bei 80 kV beobachten.
3. Kennzeichnung von sEVs
- Verdünnen Sie 20 μg sEVs in 1 ml Verdünnungsmittelpuffer oder das gleiche Volumen PBS in 1 ml Verdünnungsmittelpuffer zur Farbstoffkontrolle.
- 3 μL PKH67- oder PKH26-Farbstoff in 1 ml Verdünnungspuffer verdünnen und durch Pipettieren mischen.
- Verdünnter PKH-Farbstoff zu den verdünnten sEVs geben und durch Pipettieren mischen. 5 min im Dunkeln bei Raumtemperatur inkubieren. Für eine Farbstoffkontrolle mischen Sie den verdünnten Farbstoff mit verdünnten PBS aus Schritt 3.1.
- 2 ml 1% Rinderserumalbumin (BSA) in PBS in das Röhrchen mit dem Farbstoff und der sEV-Mischung und in das Farbstoffkontrollröhrchen geben, um überschüssigen Farbstoff zu absorbieren.
- Zentrifuge für 1 h bei 110.000 × g bei 4 °C. Entsorgen Sie den Überstand, resuspendieren Sie das Pellet in 2 mL PBS und zentrifugieren Sie für 1 h bei 110.000 × g bei 4 °C. Wiederholen Sie die Wäsche mit PBS und drehen Sie die markierten sEVs oder die Farbstoffkontrolle in einem gleichen PbS-Volumen wieder auf.
- Quantifizieren Sie die Menge an Gesamtprotein mit der Bradford-Methode.
4. Aufnahme von sEVs durch Neuro-2a-Zellen
- Legen Sie 18-mm-Coverlips in eine 12-Well-Platte und eine Platte 10 ×10 4 Neuro-2a-Zellen in jede Vertiefung in insgesamt 1 ml vollständiges DMEM-Medium mit 10% FBS und 1% Pen-Strep.
- Ändern Sie das Medium in dmEM-exosomenarmes Medium, wenn die Zellkonfluenz 80-90% beträgt. Fügen Sie 1, 5 oder 10 μg markierte sEVs in jeder Vertiefung für 1, 4 und 24 h für die dosis- und zeitabhängige Aufnahme hinzu oder fügen Sie ein gleiches Volumen an Farbstoffkontrolle hinzu.
5. Primäre astrozytäre Kulturen
- Betäuben Sie 4 postnatale Welpen 4 Tage nach der Geburt, indem Sie eine Hypothermie induzieren.
- Sammeln Sie die Gehirne in einer 60-mm-Petrischale, die eiskalte Hank's Balanced Salt Solution (HBSS) enthält, ergänzt mit 10 mM 4-(2-Hydroxyethyl)-1-piperazinethansulfonsäure (HEPES).
- Sezieren Sie beide kortikalen Lappen und entfernen Sie die Meningen. Die Gewebe mit einer sterilisierten Klinge zerhacken.
- Das Gewebe wird in ein 15 ml konisches Röhrchen mit Papain/Desoxyribonuklease I Dissoziationspuffer gegeben und 20 min bei 37 °C inkubiert. Alle 5 Min. schwenken.
HINWEIS: Für 4 Mauskortikale werden 9 ml 7,5 U/ml Papain in HBSS bei 37 °C für mindestens 30 min aktiviert, durch einen 0,22 μm Spritzenfilter filtriert und mit Desoxyribonuklease I bis zu einer Endkonzentration von 0,1 mg/ml gemischt. - Saugen Sie den Überstand an und fügen Sie 5 ml vollständiges DMEM hinzu, um die Enzymaktivität zu inaktivieren. Vorsichtig triturieren, um das Gewebe mit einer 5 mL serologischen Glaspipette und einer flammpolierten Pasteur-Pipette zu dissoziieren.
- Die Zellsuspension durch ein 40 μm Zellsieb geben und die Zellen bei 250 × g für 5 min bei 4 °C zentrifugieren. Saugen Sie das Medium an und säen Sie die Zellen in 10 ml vollständigem DMEM in einem 75 cm2 Kolben aus. Ersetzen Sie das überstehende Medium 4 h nach der Beschichtung durch 15 mL frisches DMEM-Medium.
- Nach 14 Tagen in vitroden Kolben in einen Orbitalschüttler geben, um die Mikroglia und Oligodendrozyten bei 320 U / min für 6 h zu lösen.
- Trypsinisieren Sie die verbleibenden Astrozyten mit 5 ml des Zelldissoziationsenzyms (Materialtabelle) für 10 min bei 37 °C. Fügen Sie 5 ml vollständiges DMEM hinzu, um die enzymatische Wirkung zu inaktivieren, und pelletieren Sie die Zellen bei 250 × g für 5 min bei 4 °C.
- Stellen Sie die Zellen in vollständigem DMEM wieder auf. 5 × 104 Zellen auf 12 mm #1,5 Coverlips in einer 24-Well-Platte.
6. Aufnahme von sEVs durch Astrozyten
- Wenn Astrozyten 80-90% Konfluenz erreichen, wechseln Sie das Medium in DMEM-Exosom-erschöpftes Medium.
- Fügen Sie 1 μg unmarkierte, markierte sEVs, ein gleiches Volumen an Farbstoffkontrolle oder PBS zu den Zellen hinzu. Verwenden Sie die Zellen zur Färbung 1 h und 24 h nach der sEV-Behandlung.
7. Immunfluoreszenz
- Spülen Sie die Zellen mit PBS 3x aus und fixieren Sie sie mit 4% PFA in PB für 10 min bei Raumtemperatur.
- Waschen Sie die festen Zellen 3 x 5 min mit PB und permeabilisieren Sie sie mit 0,1% Triton X-100 in PB für 10-15 min und waschen Sie sie mit PB 3 x 5 min.
- Blockieren Sie die Zellen mit 5% normalem Ziegenserum (NGS) in PB für 1 h bei Raumtemperatur.
- Inkubieren Sie die Zellen mit primären Antikörpern: Mikrotubuli-assoziiertes Protein 2 (MAP2A, 1:500) für Neuro-2a-Zellen oder Gliafibrillary Acidic Protein (GFAP, 1:500) für primäre Astrozyten in frischen 5% NGS/PB über Nacht bei 4 °C mit sanftem Schütteln.
- 3 x 10 min mit PB waschen und fluorophorkonjugierte sekundäre Antikörper (Goat Anti-Mouse IgG1, Alexa Fluor 594; oder Goat Anti-Mouse IgG H&L, Alexa Fluor 488) in 5% NGS geben und 2 h bei Raumtemperatur auf einer Wippe inkubieren.
- 3 x 10 min mit PB waschen und mit 1 μg/ml Kernfleck 4',6-Diamidino-2-phenylindol (DAPI) 10 min bei Raumtemperatur inkubieren. Waschen Sie die Zellen erneut 3x mit PB.
- Montieren Sie die Coverlips auf #1 Dias mit einem Antifade-Montagemedium. Lassen Sie sie über Nacht im Dunkeln trocknen und lagern Sie die vorbereiteten Glasobjektträger bei 4 °C bis zur Bildgebung auf einem konfokalen Mikroskop.
8. In-vivo-Aufnahme von sEVs
- Intrathekale Injektion von 5 μg unmarkierten oder markierten sEVs, die in 10 μL PBS resuspendiert wurden, oder gleiches Volumen (10 μL) farbstoffkontrolle (wie in Abschnitt 3 hergestellt) in C57BL/6-Mäuse.
- Nach 6 und 18 Stunden nach der Injektion von sEVs betäuben Sie Mäuse tief durch intraperitoneale Injektion von 100 mg / kg Körpergewicht Ketamin und 10 mg / kg Körpergewicht Xylazin.
- Führen Sie eine intrakardiale Perfusion von Mäusen mit 0,9% Kochsalzlösung durch, um Blut auszuspülen, gefolgt von frisch zubereitetem eiskaltem 4% PFA / PB.
- Sezieren Sie das Rückenmark und fixieren Sie es in 4% PFA/PB bei 4 °C für 24 h. Kryoschutz das Gewebe in 30% Saccharose in PB bei 4 °C für 24 h oder bis das Gewebe sinkt. Lagern Sie das Gewebe bei 4 °C bis zur Immunhistochemie.
9. Immunhistochemie
- L4-L5 Rückenmark in O.C.T Compound einbetten. Auf Trockeneis einfrieren, bis es vollständig erstarrt ist.
- Durchschneiden Sie das Gewebe bei 30 μm (Querschnitt für das Rückenmark) mit einem Kryostaten und sammeln Sie die Abschnitte in einer 24-Well-Platte, die PB enthält. Waschen Sie die Abschnitte 3 x 5 min mit 0,3% Triton in PB.
- Blockieren Sie unspezifische Bindungsstellen mit 5% NGS in 0,3% Triton/PB für 2 h bei Raumtemperatur.
- Verdünnte primäre Antikörper: Anti-MAP2A (1:500), GFAP (1:1.000), Iba1 für Mikroglia (1:2.000) mit 5% NGS in 0,3% Triton/PB und inkubieren sie die Abschnitte über Nacht bei 4 °C auf einem Shaker.
- Waschen Sie die Abschnitte 3 x 5 min mit 0,3% Triton/PB und fügen Sie sekundäre Antikörper (Donkey Anti-Rabbit IgG Alexa Fluor 488, 1:500 oder Goat Anti-Mouse IgG Alexa Fluor 488, 1:500) in 5% NGS/PB für 2 h bei Raumtemperatur hinzu.
- 3 x 5 min mit PB waschen und die Abschnitte in 1 μg/ml DAPI für 10 min bei Raumtemperatur inkubieren. Waschen Sie die Abschnitte 3 x 5 min mit PB.
- Montieren Sie die Schnitte auf einem sauberen Klebeobjektträger (Table of Materials) mit einem feinen Pinsel unter einem Lichtmikroskop.
- Befeuchten Sie den Abdeckrlip mit einem Montagemedium. Über Nacht im Dunkeln bei Raumtemperatur aushärten.
- Bild unter einem konfokalen Mikroskop mit den jeweiligen Lasern.
Ergebnisse
Nach der Isolierung von sEVs aus RAW 264.7-konditionierten Medien durch Zentrifugation wurde NTA verwendet, um die Konzentration und Größenverteilung der gereinigten sEVs zu bestimmen. Die durchschnittliche mittlere Größe von RAW 264,7-abgeleiteten sEVs betrug 140 nm und die Spitzenpartikelgröße betrug 121,8 nm, was bestätigt, dass die meisten nachweisbaren Partikel in der Lichtstreumessung in den Größenbereich von Exosomen oder sEVs bei 50-150 nm fielen (Abbildung 1A). Wie in den minimalen Informationen für Studien mit extrazellulären Vesikeln 2018 (MISEV2018)23vorgeschlagen, analysierten wir eine Reihe von Proteinen, die vorhanden sein sollten oder aus verschiedenen EV-Populationen ausgeschlossen werden sollten. Western Blotting von sEVs, Zelllysat und exo-depleted Media zeigte, dass sEV-abgeleitete Proteinproben die sEV-Markerproteine Alix, CD81 und GAPDH enthielten. Die Zelllysatfraktion wurde mit dem endoplasmatischen Retikulum-residenten Protein Calnexin angereichert, das in den sEVs fehlte. Somit diente Calnexin als negativer Marker für zelluläre Kontamination (Abbildung 1B).
Als nächstes führten wir Dosis-Wirkungs- und Zeitverlaufsexperimente für die sEV-Aufnahme in vitro durch. Neuro-2a-Zellen wurden mit einer Einzeldosis von 1 μg PKH67-markierten sEVs für 1, 4 und 24 h inkubiert, woraufhin die Aufnahme verschiedener Konzentrationen von sEVs (1, 5 und 10 μg) nach 1 h untersucht wurde. Die Ergebnisse der NTA zeigten, dass 1 μg Protein im Durchschnitt ~1 x 109 Partikeln entsprach. Parallel dazu wurden PBS, unmarkierte sEVs und Farbstoff-alone-Kontrollen getestet. Wir beobachteten, dass die Aufnahme von sEVs bei 1 h (Abbildung 2A) und für die 1, 5 und 10 μg sEVs (Abbildung 2B) auftrat. Fluoreszenz konnte bei 4 h für 5 und 10 μg sEVs (Abbildung 2C) nach der Inkubation nachgewiesen werden. Als nächstes wurde die Aufnahme von PKH26-markierten sEVs durch primäre Astrozyten untersucht (Abbildung 3). Maximale Fluoreszenz aus der sEV-Aufnahme in primären kortikalen Astrozyten trat nach 24 h auf. Nicht markierte sEVs zeigten keine Fluoreszenz, was zeigt, dass die sEV-Autofluoreszenz nicht signifikant zu Falschalarmen beiträgt (Ergänzende Abbildung S1A).
Als nächstes wurden markierte sEVs intrathekal in Mäuse injiziert, um die Abgabe und Aufnahme von sEVs durch verschiedene Zellen im Rückenmark mittels Immunhistochemie und konfokaler Mikroskopie zu beurteilen. Wir haben MAP2 als neuronalen Marker, GFAP als astrozytischen Marker und IBA1 als mikroglialen Marker gefärbt. Neuronen (Abbildung 4), Astrozyten (Abbildung 5) und Mikrogliazellen (Abbildung 6) nahmen alle PKH26-markierte sEVs auf, und eine maximale sEV-Fluoreszenz wurde 6 Stunden nach der Injektion beobachtet. Während die sEVs nicht immer mit den zellulären Markern kolokalisierten, beobachteten wir keine differentielle Aufnahme durch ZNS-Zellen. Die intrathekale Injektion mit 5 μg unmarkiertem RAW 264,7 sEVs oder Farbstoffkontrolle zeigte keine signifikante Fluoreszenz (Ergänzende Abbildung S1B). Fluoreszierende Signale wurden in den Meningen beobachtet, sowohl 6 h als auch 18 h nach der Injektion von sEVs (Ergänzende Abbildung S1C).

Abbildung 1: Charakterisierung von gereinigten RAW 264.7 sEVs. (A) Größe und Konzentration von sEVs wurden mit NanoSight NS300 bestimmt. Die Partikel wurden verfolgt und basierend auf der Brownschen Bewegung und dem Diffusionskoeffizienten dimensioniert. Die Größenverteilung von sEVs wird in nm dargestellt. Die Konzentration von sEVs wurde als Partikel/ml ausgedrückt. (B) Western Blot von Proteinen, die aus gereinigten sEVs, Zelllysat und exosomenarmen Medien unter Verwendung der sEV-Marker ALIX, GAPDH und CD81 gewonnen werden. Der endoplasmatische Retikulumproteinmarker Calnexin dient als Kontrolle zur Überwachung der zellulären Kontamination in sEV-Präparaten. (C) Die Transmissionselektronenmikroskopie zeigte die Größe und Morphologie von sEVs. Maßstabsbalken = 100 nm. Abkürzungen: sEVs = kleine extrazelluläre Vesikel; ALIX = Alpha-1,3/1,6-Mannosyltransferase (ALG-2)-interagierendes Protein X; GAPDH = Glyceraldehyd-3-phosphat-Dehydrogenase; CD81 = Differenzierungscluster 81. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
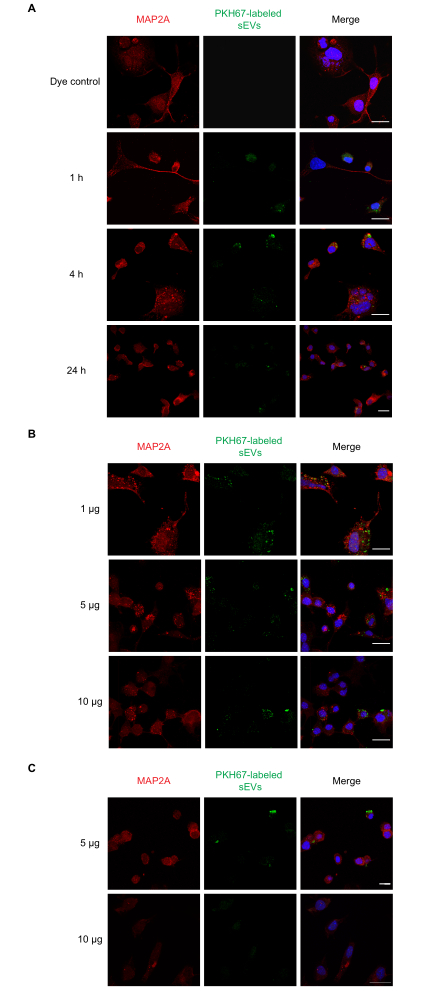
Abbildung 2: Aufnahme von markierten RAW 264.7 sEVs durch Neuro-2a-Zellen. (A) PKH67-markierte sEVs (1 μg) wurden den kultivierten Neuro-2a-Zellen für 1, 4 oder 24 h zugesetzt. (B) PKH67-markierte sEVs (1, 5 oder 10 μg) wurden Neuro-2a-Zellen für 1 h zugesetzt. (C) PKH67-markierte sEVs (5 oder 10 μg) wurden Neuro-2a-Zellen für 4 h zugesetzt. sEV-Aufnahme wurde in allen Dosierungsgruppen mit konfokaler Mikroskopie beobachtet. Negative Kontrollgruppen, die mit PKH-Farbstoff allein behandelt wurden, zeigten keine sEV-Färbung (Ergänzende Abbildung S1). Neuro-2a-Zellen wurden mit MAP2A immungefärbt (untersucht mit Alexa Fluor 594, rot dargestellt), während Zellkerne mit DAPI (blau dargestellt) und sEVs mit PKH67 (grün dargestellt) gefärbt wurden. Maßstabsleiste = 50 μm. Abkürzungen: sEVs = kleine extrazelluläre Vesikel; MAP2A = Mikrotubuli-assoziiertes Protein 2A; DAPI = 4′,6-Diamidino-2-phenylindol. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 3: Aufnahme von PKH26-markierten RAW 264,7 sEVs durch primäre kortikale Astrozyten der Maus. Ein μg sEVs wurde mit PKH26-Farbstoff markiert und dem primären Astrozytenkulturmedium zugesetzt. Die Aufnahme von sEVs wurde nach 1 und 24 h nach der Addition mit einem konfokalen Laserscanning-Mikroskop beobachtet. Astrozyten wurden mit GFAP gefärbt (untersucht mit Alexa Fluor 488, rot dargestellt), während Zellkerne mit DAPI (blau dargestellt) und sEVs zuvor mit PKH26 (grün dargestellt) gefärbt wurden. Skalenstab = 20 μm. PKH26-Farbstoff allein diente als Negativkontrolle für die sEV-Färbung. Abkürzungen: sEVs = kleine extrazelluläre Vesikel; GFAP = glial fibrillary acidic protein; DAPI = 4′,6-Diamidino-2-phenylindol. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Aufnahme von RAW 264,7 sEVsin Neuronen. PKH26-markierte sEVs wurden intrathekal in Mäuse injiziert; 6 und 18 h später wurden die Mäuse mit 4% PFA durchblutet, und das Rückenmark wurde isoliert und bei 30 μm durchtrennen. Rückenmarksabschnitte wurden mit einem Zellmarker (untersucht mit Alexa Fluor 488, rot dargestellt) und DAPI-Kerngegenfleck (blau dargestellt) immungefärbt, während sEVs zuvor mit PKH26 (grün dargestellt) markiert wurden. Rückenmarksabschnitte wurden immungefärbt, damit MAP2A die Neuronen sichtbar machen konnte (rot). Die konfokale Mikroskopie zeigt sEVs in MAP2A-positiven Neuronen zu verschiedenen Zeitpunkten. Die Negativkontrolle, PKH26-Farbstoff-allein-Gruppe, zeigte keine sEV-Färbung. Maßstabsleiste = 20 μm. Abkürzungen: sEVs = kleine extrazelluläre Vesikel; PFA = Paraformaldehyd; MAP2A = Mikrotubuli-assoziiertes Protein 2A; DAPI = 4′,6-Diamidino-2-phenylindol. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 5: Aufnahme von RAW 264,7 sEVs in Astrozyten. PKH26-markierte sEVs wurden intrathekal in Mäuse injiziert; 6 und 18 h später wurden die Mäuse mit 4% PFA durchblutet, und das Rückenmark wurde isoliert und bei 30 μm durchtrennen. Rückenmarksabschnitte wurden mit einem Zellmarker (untersucht mit Alexa Fluor 488, rot dargestellt) und DAPI-Kerngegenfleck (blau dargestellt) immungefärbt, während sEVs zuvor mit PKH26 (grün dargestellt) markiert wurden. Rückenmarksabschnitte wurden immungefärbt, um gfAP die Astrozyten zu visualisieren (rot). Die konfokale Mikroskopie zeigt sEVs in GFAP-positiven Astrozyten zu verschiedenen Zeitpunkten. Die Negativkontrolle, PKH26-Farbstoff-allein-Gruppe, zeigte keine sEV-Färbung. Maßstabsleiste = 20 μm. Abkürzungen: sEVs = kleine extrazelluläre Vesikel; PFA = Paraformaldehyd; GFAP = glial fibrillary acidic protein; DAPI = 4′,6-Diamidino-2-phenylindol. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
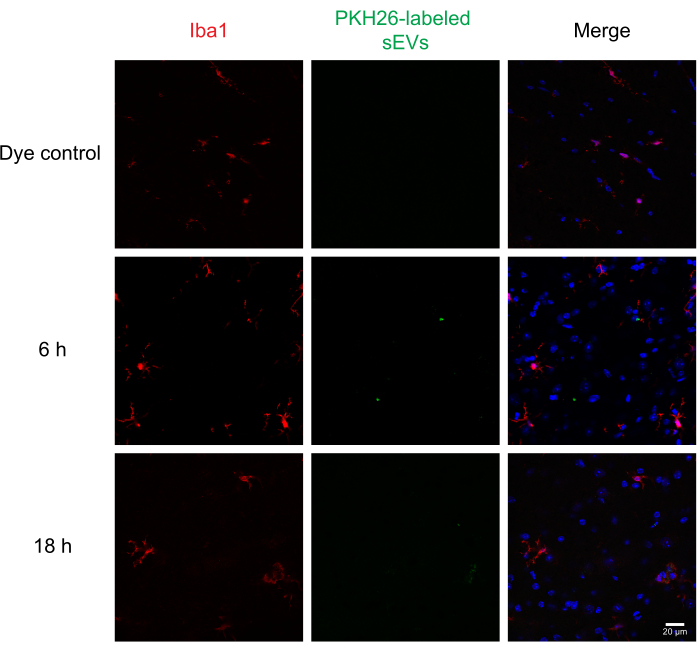
Abbildung 6: Aufnahme von RAW 264,7 sEVs in Mikroglia. PKH26-markierte sEVs wurden intrathekal in Mäuse injiziert; 6 und 18 h später wurden die Mäuse mit 4% PFA durchblutet, und das Rückenmark wurde isoliert und bei 30 μm durchtrennen. Rückenmarksabschnitte wurden mit einem Zellmarker (untersucht mit Alexa Fluor 488, rot dargestellt) und DAPI-Kerngegenfleck (blau dargestellt) immungefärbt, während sEVs zuvor mit PKH26 (grün dargestellt) markiert wurden. Rückenmarksabschnitte wurden für IBA1 immungefärbt, um die Mikroglia (rot) sichtbar zu machen. Die konfokale Mikroskopie zeigt sEVs in IBA1-positiven Mikroglia zu verschiedenen Zeitpunkten. Die Negativkontrolle, PKH26-Farbstoff-allein-Gruppe, zeigte keine sEV-Färbung. Maßstabsleiste = 20 μm. Abkürzungen: sEVs = kleine extrazelluläre Vesikel; PFA = Paraformaldehyd; IBA1 = ionisiertes calciumbindendes Adaptermolekül 1; DAPI = 4′,6-Diamidino-2-phenylindol. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung S1: Aufnahme von markierten RAW 264,7 sEVs durch primäre kortikale Astrozyten der Maus und im Rückenmark. (A) Kontrollen für die Aufnahme von PKH26-markierten RAW 264,7 sEVs durch primäre kortikale Astrozyten der Maus. Ein μg unmarkierte sEVs, die in PBS resuspendiert wurden, oder ein gleiches Volumen PBS wurde parallel zum Kulturmedium der Astrozyten zugegeben. Bei 1 h wurde keine Fluoreszenz für PBS und die unmarkierte Kontrolle mit einem konfokalen Laserscanning-Mikroskop beobachtet. Astrozyten wurden mit GFAP gefärbt (untersucht mit Alexa Fluor 488, rot dargestellt), während die Kerne mit DAPI (blau) gegengefärbt wurden und unbeschriftete sEVs unter demselben Alexa Fluor 546-Kanal wie PKH26-markierte sEVs visualisiert wurden. Skalenleiste = 50 μm. (B) Kontrollen für die Aufnahme von PKH26-markierten RAW 264,7 sEVs durch das Rückenmark der Maus in vivo. Fünf μg unmarkierte sEVs oder Farbstoffkontrolle wurden intrathekal in Mäuse injiziert. Auch hier wurden keine fluoreszierenden Signale für unmarkierte sEVs oder farbstofffreie Kontrolle mit einem konfokalen Laserscanning-Mikroskop beobachtet. Astrozyten wurden mit GFAP gefärbt (untersucht mit Alexa Fluor 488, rot dargestellt), während Kerne mit DAPI (blau) gegengefärbt wurden und unbeschriftete sEVs unter demselben Alexa Fluor 546-Kanal wie PKH26-markierte sEVs visualisiert wurden. Maßstabsbalken = 50 μm. (C)Repräsentative Bilder zeigen das Vorhandensein von RAW 264,7 sEVs in Mauswirbelhäuten 6 h und 18 h nach intrathekaler Entbindung. Fünf μg sEVs wurden mit PKH26-Farbstoff markiert (grün dargestellt), und die Kerne wurden mit DAPI (blau dargestellt) gegengefärbt. Sternchen weisen auf die Arteria spinalis an. Maßstabsleiste = 50 μm. Abkürzungen: sEVs = kleine extrazelluläre Vesikel; PBS = phosphatgepufferte Kochsalzlösung; GFAP = glial fibrillary acidic protein; DAPI = 4′,6-Diamidino-2-phenylindol. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
In diesem Protokoll zeigten wir die Markierung von sEVs mit PKH-Farbstoffen und die Visualisierung ihrer Aufnahme im Rückenmark. PKH lipophile Fluoreszenzfarbstoffe werden häufig zur Markierung von Zellen durch Durchflusszytometrie und Fluoreszenzmikroskopie3,5,6,12,24,25verwendet. Aufgrund ihrer relativ langen Halbwertszeit und geringen Zytotoxizität können PKH-Farbstoffe für eine Vielzahl von In-vivo- und In-vitro-Zell-Tracking-Studien verwendet werden26,27. Obwohl eine ausgezeichnete Membranretention und biochemische Stabilität von Vorteil sind, kann die Interkalation von fluoreszierenden Sonden mit Lipoprotein-Verunreinigungen, die mit sEVs gereinigt wurden, die Interpretation von sEV-Internalisierungs- und Funktionsstudien beeinträchtigen. Daher sind die Reinigung und Kennzeichnung von sEVs kritische Schritte im Protokoll, da die Persistenz der Farbstoffe mit Verunreinigungen zu einer Fehlinterpretation der In-vivo-Verteilung führen kann28. Die Einbeziehung von Kontrollen ist entscheidend, um falsch-positive Fluoreszenzsignale aufgrund der unspezifischen Markierung von Partikeln und der langen Halbwertszeit dieser Farbstoffe zu vermeiden.
Aggregation und Mizellenbildung von lipophilen Farbstoffen können ebenfalls falsche Signale liefern. Wir haben das Problem des freien oder ungebundenen Farbstoffs angegangen, indem wir eine Farbstoff-allein-Kontrolle einführten und die EV-Aufnahme zu früheren Zeitpunkten visualisierten. Eine wichtige Einschränkung für die PKH-Markierung ist, dass bei der PKH26-Farbstoffmarkierung von sEVs zahlreiche PKH26-Nanopartikel gebildet werden. Obwohl nicht in diesem Protokoll enthalten, wird berichtet, dass PKH26-Nanopartikel durch einen Saccharosegradienten29entfernt werden können. Eine weitere Studie untersuchte die Wirkung der PKH-Kennzeichnung auf die Größe von sEVs durch NTA und berichtete über eine Größenzunahme nach der PKH-Kennzeichnung30. Dennoch dienen PKH-Farbstoffe als pragmatischer und wertvoller Tracer, um zu zeigen, wo sich die sEVs durchquert haben. Eine weitere Einschränkung dieser Studie ist, dass wir sEVs nicht quantifiziert haben, da sich dieses Protokoll auf die Bestätigung der zellulären Aufnahme nach intrathekaler Verabreichung konzentriert. Neuartige Cyanin-basierte Membransonden wurden kürzlich für die hochempfindliche Fluoreszenzbildgebung von sEVs entwickelt, ohne die Größe zu verändern oder Artefakte wie die Bildung von PKH-Nanopartikeln31zu erzeugen, und werden zweifellos zukünftige Markierungsstudien verbessern.
Obwohl Makrophagen eine wichtige Rolle bei der Neuroinflammation spielen, üben sie auch neuroprotektive Funktionen aus, indem sie ihre Ladung über Exosomenabgeben 32. Unsere Studien zeigen, dass markierte Makrophagen-abgeleitete sEVs nach intrathekaler Verabreichung von Neuro-2a-Zellen, primären Astrozyten und im Lendenwirbelmark aufgenommen werden. Die Ergebnisse deuten darauf hin, dass eine längere Inkubationszeit zu einer geringeren sEV-Signalintensität führen kann, was auf den Abbau von sEVs oder die Zellteilung durch Neuro-2a-Zellen in Kultur33,34zurückzuführen ist. Obwohl der Durchsatz niedrig ist, kann dieses Protokoll zur Visualisierung markierter sEVs im Rückenmark für erste Validierungsstudien verwendet werden, die die sEV-Aufnahme bestätigen, bevor die funktionellen Auswirkungen von intrathekal abgegebenen sEVs untersucht werden. Da wir im Allgemeinen eine ähnliche sEV-Aufnahme in mehreren ZNS-Zelltypen beobachtet haben, scheint der Aufnahmeprozess nicht selektiv zu sein. Wenn Autofluoreszenz ein Problem in der Bildgebung ist, können unmarkierte sEVs als zusätzliche Kontrolle verwendet werden, um die sEV-Autofluoreszenz während der Bildgebung von Geweben und Kulturen zu negieren. Obwohl die Dosis und der Verabreichungsweg von sEVs das Muster der Bioverteilung beeinflussen können11,ist dieses Protokoll nicht für die quantitative Analyse der sEV-Aufnahme optimiert. Zur Untersuchung von sEVs werden verschiedene Ansätze und verschiedene Bildgebungsstrategien eingesetzt, die kontinuierlich für das In-vivo-Tracking von sEVs2verfeinert und optimiert werden.
Dieses Protokoll soll nur ein Ansatz sein, um die sEV-Aufnahme zu bestätigen. Wie bei allen Protokollen kann die Kreuzvalidierung mit multimodalen Ansätzen von Vorteil sein. Insbesondere kann die Aufnahme von sEVs durch die Untersuchung des biomolekularen Frachttransfers zu Empfängerzellen und -geweben bestätigt werden. Wenn der Forscher die miRNA-Zusammensetzung der gelieferten sEVs kennt, wäre ein alternativer Ansatz zur Bestätigung des sEV-Transfers, auf miRNA-Veränderungen in den Empfängerzellen zu überprüfen oder die Veränderungen der Expressionsniveaus der Zielgene für die übertragenen miRNAs zu bestimmen. PBS-behandelte Proben können als Kontrolle für diesen Ansatz verwendet werden. Insgesamt unterstützen diese Ergebnisse das Konzept, dass makrophagenbasierte sEVs von ZNS-Zellen in vitro und in vivoaufgenommen werden. Dieses Protokoll kann verwendet werden, um die Rolle von sEVs bei Wirbelsäulenerkrankungen, Schmerzen und Entzündungen zu untersuchen und festzustellen, ob sEVs als zelluläre Vehikel für die Abgabe von therapeutischen kleinen Molekülen, RNA und Proteinen entwickelt werden können.
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Studie wurde durch Zuschüsse von NIH NINDS R01NS102836 und dem Pennsylvania Department of Health Commonwealth Universal Research Enhancement (CURE) unterstützt, die an Seena K. Ajit vergeben wurden. Wir danken Dr. Bradley Nash für die kritische Lektüre des Manuskripts.
Materialien
| Name | Company | Catalog Number | Comments |
| Amicon Ultra 0.5 mL centrifugal filters | MilliporeSigma | Z677094 | |
| Anti-Alix Antibody | Abcam | ab186429 | 1:1000 |
| Anti-Calnexin Antibody | Abcam | Ab10286 | 1:1000 |
| Anti-CD81 Antibody | Santa Cruz Biotechnology | sc-166029 | 1:1000 |
| Anti-GAPDH Monoclonal Antibody (14C10) | Cell Signaling Technology | 2118 | 1:1000 |
| Anti-Glial Fibrillary Acidic Protein Antibody | Sigma-Aldrich | MAB360 | 1:500 for IF; 1:1000 for IHC |
| Anti-Iba1 Antibody | Wako | 019-19741 | 1:2000 |
| Anti-MAP2A Antibody | Sigma-Aldrich | MAB378 | 1:500 |
| Bovine Serum Albumin (BSA) | VWR | 0332 | |
| Cell Strainer, 40 μm | VWR | 15-1040-1 | |
| Centrifuge Tubes | Thermo Scientific | 3118-0050 | 12,000 x g |
| Coverslip, 12-mm, #1.5 | Electron Microscopy Sciences | 72230-01 | |
| Coverslip, 18-mm, #1.5 | Electron Microscopy Sciences | 72222-01 | |
| DAPI | Sigma-Aldrich | D9542-1MG | 1 µg/mL |
| DC Protein Assay | Bio-Rad | 500-0116 | |
| Deoxyribonuclease I (DNAse I) | MilliporeSigma | D4513-1VL | |
| Donkey Anti-Rabbit IgG H&L (HRP) | Abcam | ab16284 | 1:10000 |
| Donkey Anti-Rabbit IgG H&L, Alexa Fluor 488 | Invitrogen | A-21206 | 1:500 |
| Double Frosted Microscope Slides, #1 | Thermo Scientific | 12-552-5 | |
| DPBS without Calcium and Magnesium | Corning | 21-031-CV | |
| Dulbecco's Modified Eagle Medium (DMEM) | Corning | 10-013-CV | |
| Exosome-Depleted Fetal Bovine Serum | Gibco | A27208-01 | |
| Fetal Bovine Serum (FBS) | Corning | 35-011-CV | |
| FluorChem M imaging system | ProteinSimple | ||
| FV3000 Confocal Microscope | Olympus | ||
| Goat Anti-Mouse IgG H&L (HRP) | Abcam | ab6789 | 1:10000 |
| Goat Anti-Mouse IgG H&L, Alexa Fluor 488 | Invitrogen | A-11001 | 1:500 |
| Goat Anti-Mouse IgG1, Alexa Fluor 594 | Invitrogen | A-21125 | 1:500 |
| Hank's Balanced Salt Solution (HBSS) | VWR | 02-0121 | |
| HEPES | Gibco | 15630080 | |
| HRP Substrate | Thermo Scientific | 34094 | |
| Intercept blocking buffer, TBS | LI-COR Biosciences | 927-60001 | |
| Laemmli SDS Sample Buffer | Alfa Aesar | AAJ61337AC | |
| Micro Cover Glass, #1 | VWR | 48404-454 | |
| Microm HM550 | Thermo Scientific | ||
| NanoSight NS300 system | Malvern Panalytical | ||
| NanoSight NTA 3.2 software | Malvern Panalytical | ||
| Neuro-2a Cell Line | ATCC | CCL-131 | |
| Normal Goat Serum | Vector Laboratories | S-1000 | |
| O.C.T Compound | Sakura Finetek | 4583 | |
| Papain | Worthington Biochemical Corporation | NC9597281 | |
| Paraformaldehyde | Electron Microscopy Sciences | 19210 | |
| Penicillin-Streptomycin | Gibco | 15140122 | |
| PKH26 | Sigma-Aldrich | MINI26-1KT | |
| PKH67 | Sigma-Aldrich | MINI67-1KT | |
| Protease Inhibitor Cocktail | Thermo Scientific | 1862209 | |
| PVDF Transfer Membrane | MDI | SVFX8302XXXX101 | |
| RAW 267.4 Cell Line | ATCC | TIB-71 | |
| RIPA Buffer | Sigma-Aldrich | R0278 | |
| Sodium Chloride | AMRESCO | 0241-2.5KG | |
| Superfrost Plus Gold Slides | Thermo Scientific | 15-188-48 | adhesive slides |
| T-75 Flasks | Corning | 431464U | |
| Tecnai 12 Digital Transmission Electron Microscope | FEI Company | ||
| TEM Grids | Electron Microscopy Sciences | FSF300-cu | |
| Tris-Glycine Protein Gel, 12% | Invitrogen | XP00120BOX | |
| Tris-Glycine SDS Running Buffer | Invitrogen | LC26755 | |
| Tris-Glycine Transfer Buffer | Invitrogen | LC3675 | |
| TrypLE Express | Gibco | 12605028 | cell dissociation enzyme |
| Triton X-100 | Acros Organics | 327371000 | |
| Trypsin, 0.25% | Corning | 25-053-CL | |
| Tween 20 | Sigma-Aldrich | P1379 | |
| Ultracentrifuge Tubes | Beckman | 344058 | 110,000 x g |
Referenzen
- Mulcahy, L. A., Pink, R. C., Carter, D. R. F. Routes and mechanisms of extracellular vesicle uptake. Journal of Extracellular Vesicles. 3 (1), 24641(2014).
- Betzer, O., et al. Advances in imaging strategies for in vivo tracking of exosomes. Wiley Interdisciplinary Reviews. Nanomedicine and Nanobiotechnology. 12 (2), 1594(2020).
- Dehghani, M., Gaborski, T. R. Fluorescent labeling of extracellular vesicles. Methods in Enzymology. 645, 15-42 (2020).
- González, M. I., et al. Covalently labeled fluorescent exosomes for in vitro and in vivo applications. Biomedicines. 9 (1), 81(2021).
- Chuo, S. T. -Y., Chien, J. C. -Y., Lai, C. P. -K. Imaging extracellular vesicles: current and emerging methods. Journal of Biomedical Science. 25 (1), 91(2018).
- vander Vlist, E. J., Nolte-'tHoen, E. N. M., Stoorvogel, W., Arkesteijn, G. J. A., Wauben, M. H. M. Fluorescent labeling of nano-sized vesicles released by cells and subsequent quantitative and qualitative analysis by high-resolution flow cytometry. Nature Protocols. 7 (7), 1311-1326 (2012).
- Hoshino, A., et al. Tumour exosome integrins determine organotropic metastasis. Nature. 527 (7578), 329-335 (2015).
- Heinrich, L., et al. Confocal laser scanning microscopy using dialkylcarbocyanine dyes for cell tracing in hard and soft biomaterials. Journal of Biomedical Materials Research. Part B, Applied Biomaterials. 81 (1), 153-161 (2007).
- Haney, M. J., et al. Exosomes as drug delivery vehicles for Parkinson's disease therapy. Journal of Controlled Release. 207, 18-30 (2015).
- Grange, C., et al. Biodistribution of mesenchymal stem cell-derived extracellular vesicles in a model of acute kidney injury monitored by optical imaging. International Journal of Molecular Medicine. 33 (5), 1055-1063 (2014).
- Wiklander, O. P., et al. Extracellular vesicle in vivo biodistribution is determined by cell source, route of administration and targeting. Journal of Extracellular Vesicles. 4, 26316(2015).
- Lai, C. P., et al. Visualization and tracking of tumour extracellular vesicle delivery and RNA translation using multiplexed reporters. Nature Communications. 6 (1), 7029(2015).
- Deddens, J. C., et al. Circulating extracellular vesicles contain miRNAs and are released as early biomarkers for cardiac injury. Journal of Cardiovascular Translational Research. 9 (4), 291-301 (2016).
- Montecalvo, A., et al. Mechanism of transfer of functional microRNAs between mouse dendritic cells via exosomes. Blood. 119 (3), 756-766 (2012).
- Escrevente, C., Keller, S., Altevogt, P., Costa, J. Interaction and uptake of exosomes by ovarian cancer cells. BMC Cancer. 11, 108(2011).
- Cho, E., et al. Comparison of exosomes and ferritin protein nanocages for the delivery of membrane protein therapeutics. Journal of Controlled Release. 279, 326-335 (2018).
- Mantel, P. Y., et al. Malaria-infected erythrocyte-derived microvesicles mediate cellular communication within the parasite population and with the host immune system. Cell Host & Microbe. 13 (5), 521-534 (2013).
- Porro, C., Trotta, T., Panaro, M. A. Microvesicles in the brain: Biomarker, messenger or mediator. Journal of Neuroimmunology. 288, 70-78 (2015).
- De Toro, J., Herschlik, L., Waldner, C., Mongini, C. Emerging roles of exosomes in normal and pathological conditions: new insights for diagnosis and therapeutic applications. Frontiers in Immunology. 6, 203(2015).
- Matsumoto, J., et al. Transmission of alpha-synuclein-containing erythrocyte-derived extracellular vesicles across the blood-brain barrier via adsorptive mediated transcytosis: another mechanism for initiation and progression of Parkinson's disease. Acta Neuropathologica Communications. 5 (1), 71(2017).
- Matsumoto, J., Stewart, T., Banks, W. A., Zhang, J. The transport mechanism of extracellular vesicles at the blood-brain barrier. Current Pharmaceutical Design. 23 (40), 6206-6214 (2017).
- Shaimardanova, A., et al. Extracellular vesicles in the diagnosis and treatment of central nervous system diseases. Neural Regeneration Research. 15 (4), 586-596 (2020).
- Théry, C., et al. Minimal information for studies of extracellular vesicles 2018 (MISEV2018): a position statement of the International Society for Extracellular Vesicles and update of the MISEV2014 guidelines. Journal of Extracellular Vesicles. 7 (1), 1535750(2018).
- Hoen, E. N. M. N. -t, et al. Quantitative and qualitative flow cytometric analysis of nanosized cell-derived membrane vesicles. Nanomedicine: Nanotechnology, Biology and Medicine. 8 (5), 712-720 (2012).
- Gangadaran, P., Hong, C. M., Ahn, B. -C. An update on in vivo imaging of extracellular vesicles as drug delivery vehicles. Frontiers in Pharmacology. 9, 169(2018).
- Teare, G. F., Horan, P. K., Slezak, S. E., Smith, C., Hay, J. B. Long-term tracking of lymphocytes in vivo: the migration of PKH-labeled lymphocytes. Cellular Immunology. 134 (1), 157-170 (1991).
- Kuffler, D. P. Long-term survival and sprouting in culture by motoneurons isolated from the spinal cord of adult frogs. Journal of Comparative Neurology. 302 (4), 729-738 (1990).
- Takov, K., Yellon, D. M., Davidson, S. M. Confounding factors in vesicle uptake studies using fluorescent lipophilic membrane dyes. Journal of Extracellular Vesicles. 6 (1), 1388731(2017).
- Pužar Dominkuš, P., et al. PKH26 labeling of extracellular vesicles: Characterization and cellular internalization of contaminating PKH26 nanoparticles. Biochimica et Biophysica Acta. Biomembranes. 1860 (6), 1350-1361 (2018).
- Dehghani, M., Gulvin, S. M., Flax, J., Gaborski, T. R. Systematic evaluation of PKH labelling on extracellular vesicle size by nanoparticle tracking analysis. Scientific Reports. 10 (1), 9533(2020).
- Shimomura, T., et al. New lipophilic fluorescent dyes for labeling extracellular vesicles: characterization and monitoring of cellular uptake. Bioconjugate Chemistry. 32 (4), 680-684 (2021).
- Yuan, D., et al. Macrophage exosomes as natural nanocarriers for protein delivery to inflamed brain. Biomaterials. 142, 1-12 (2017).
- Polanco, J. C., Li, C., Durisic, N., Sullivan, R., Götz, J. Exosomes taken up by neurons hijack the endosomal pathway to spread to interconnected neurons. Acta Neuropathologica Communications. 6 (1), 10(2018).
- Jurgielewicz, B. J., Yao, Y., Stice, S. L. Kinetics and specificity of HEK293T extracellular vesicle uptake using imaging flow cytometry. Nanoscale Research Letters. 15 (1), 170(2020).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten