Method Article
THP-1 Hücrelerinde Elektroporasyon Tabanlı CRISPR-Cas9 Aracılı Gen Nakavt ve Tek Hücreli Klon İzolasyonu
Bu Makalede
Özet
THP-1 hücre hattı, biyoloji ile ilgili çeşitli araştırma alanlarında insan monositlerinin / makrofajlarının işlevlerini araştırmak için bir model olarak yaygın olarak kullanılmaktadır. Bu makale, sağlam ve tekrarlanabilir fenotipik verilerin üretilmesini sağlayan verimli CRISPR-Cas9 tabanlı mühendislik ve tek hücreli klon izolasyonu için bir protokolü açıklamaktadır.
Özet
İnsan akut monositik lösemi (AML) THP-1 hücre hattı, insan immün yetmezlik virüsü (HIV) gibi önemli insan patojenleri ile etkileşimleri de dahil olmak üzere, insan monositinden türetilmiş makrofajların işlevlerini incelemek için bir model olarak yaygın olarak kullanılmaktadır. Miyeloid kökenli diğer ölümsüzleştirilmiş hücre dizileriyle karşılaştırıldığında, THP-1 hücreleri birçok sağlam enflamatuar sinyal yolunu korur ve forbol-12-miristat 13-asetat (PMA) ile tedavi edildiğinde makrofajlara farklılaşma yeteneği de dahil olmak üzere birincil monositlerinkine daha çok benzeyen fenotipik özellikler gösterir. Hedeflenen gen nakavt (KO) yoluyla THP-1 hücrelerini tasarlamak için CRISPR-Cas9 teknolojisinin kullanılması, virüs-konak etkileşimleri de dahil olmak üzere bağışıklıkla ilgili mekanizmaları daha iyi karakterize etmek için güçlü bir yaklaşım sağlar. Bu makale, önceden monte edilmiş Cas9:sgRNA ribonükleoproteinlerini hücre çekirdeğine iletmek için elektroporasyon kullanan verimli CRISPR-Cas9 tabanlı mühendislik için bir protokolü açıklamaktadır. Aynı lokusu biraz farklı pozisyonlarda hedefleyen çoklu sgRNA'ların kullanılması, büyük DNA fragmanlarının silinmesine neden olur, böylece T7 endonükleaz I testi tarafından değerlendirildiği gibi düzenleme verimliliğini artırır. Genetik düzeyde CRISPR-Cas9 aracılı düzenleme, Sanger dizilemesi ve ardından CRISPR Düzenlemelerinin Çıkarımı (ICE) analizi ile doğrulandı. Protein tükenmesi, fonksiyonel bir test ile birlikte immünoblotlama ile doğrulandı. Bu protokol kullanılarak, hedeflenen lokusta %100'e kadar indels ve protein ekspresyonunda %95'in üzerinde bir azalma elde edildi. Yüksek düzenleme verimliliği, seyreltmeyi sınırlayarak tek hücreli klonları izole etmeyi kolaylaştırır.
Giriş
THP-1, akut lösemiden (AML) muzdarip bir hastadan izole edilen ve primer monositlerinkine çok benzeyen fenotipik özellikler gösteren bir insan monosit türevi hücre hattıdır1. Fenotip olarak hem sınırlı yaşam süresi hem de donörler arası / donör içi değişkenliği göstermeyen ve bölünmeyen primer monosit türevli makrofajlarla karşılaştırıldığında, THP-1 hücreleri neredeyse sonsuza kadar kültürlenebilir ve sonuçların tekrarlanabilirliğini destekleyen daha homojen bir davranışa sahip olabilir 2,3,4,5,6. Özellikle, THP-1 hücreleri, forbol-12-miristat 13-asetat (PMA) ile makrofaj benzeri bir fenotipe doğru farklılaşabilir, bu da onları monositlerin / makrofajların enflamatuar sinyalleretepkilerini araştırmak için yaygın olarak kullanılan bir in vitro model haline getirir 7,8,9,10,11,12,13 veya HIV 14 dahil olmak üzere klinik olarak ilgili insan patojenlerinin neden olduğu enfeksiyon,15,16. THP-1 hücrelerini genetik olarak tasarlama olasılığı, biyoloji ile ilgili birçok araştırma alanında ilgi çekicidir.
Kümelenmiş Düzenli Aralıklı Kısa Palindromik Tekrarlar-CRISPR ilişkili protein 9 (CRISPR-Cas9), bir genetik mühendisliği aracı olarak yeniden programlanmış, istilacı viral genomları parçalamak için RNA güdümlü nükleaz üzerinde geçiş yapan prokaryotik adaptif bir bağışıklık sistemidir17. Genom düzenleme süreci üç adımda ilerler: tanıma, bölünme ve onarım. Tek kılavuzlu bir RNA (sgRNA), Cas9 nükleazını 20 bp kılavuz dizisi ile baz eşleşmesi yoluyla belirli bir genomik lokusa alır. 20 bp genomik hedef dizinin doğrudan 3'ü olan bir Protospacer Bitişik Motif (PAM) dizisinin varlığı, 17 ve 18 (PAM'ın 3-bp 5'i) arasındaki her iki DNA zincirinde Cas9 aracılı çözülmeyi ve bölünmeyi tetikler. Elde edilen çift iplikli kopma (DSB), iki ana onarım yolu tarafından işlenir. Hasarlı lokus ile homoloji taşıyan bir onarım şablonunun yokluğunda, hataya eğilimli Homolog Olmayan Uç Birleştirme (NHEJ) yolu, rastgele nükleotid eklemeleri ve/veya delesyonları (indels) başlatacak ve potansiyel olarak çerçeve kayması mutasyonlarına ve/veya erken sonlandırma kodonlarının (PTC) eklenmesine yol açacaktır. Buna karşılık, PTC içeren mRNA'lar, anlamsız aracılı mRNA bozunması (NMD) yolu tarafından bozunma ile hedeflenir ve sonuçta protein ekspresyonu/işlevinibozar 18,19,20. Alternatif olarak, şablona bağımlı Homoloji Yönelimli Onarım (HDR) yolu DSB'yi çalıştırabilir ve aslına uygun olarak onarabilir. Bu mekanizma, knock-in'ler ve baz ikameleri dahil olmak üzere hassas gen düzenlemesi elde etmek için kullanılmıştır. Hücre döngüsü durumunun, DSB onarım yolunun seçimini etkileyen önemli bir faktör olduğunu belirtmekte fayda var. Gerçekten de, NHEJ hücre döngüsünün tüm aşamalarında aktiftir, HDR ise esas olarak S / G2 fazları21 ile sınırlıdır.
THP-1 hücreleri süspansiyon halinde büyür ve plazmid DNA ile transfekte edilmesi herkesin bildiği gibi zordur, bu da muhtemelen canlılıklarını ve / veya farklılaşma kapasitelerini de değiştiren bir prosedürdür22,23. Hem Cas9'u hem de sgRNA'yı kodlayan HIV-1 bazlı lentiviral vektörlerle transdüksiyon, genellikle ilgilenilen bir geni nakavt etmek (KO) için kullanılır24. Cas9/sgRNA kasetinin hücresel genoma entegrasyonu, uzun süreli ekspresyon ve verimli KO sağlar, ancak aynı zamanda hedef dışı etkilerin kalıcı bir kaynağıdır25. Alternatif olarak, önceden monte edilmiş Cas9:sgRNA ribonükleoproteinleri (RNP'ler), elektrik darbelerinin uygulanması üzerine hem plazma hem de nükleer zarlarda geçici gözenek oluşumunu içeren bir yöntem olan elektroporasyon ile verilir. Hücre canlılığını korumak, bu yaklaşımı gerçekleştirirken önemli bir zorluktur.
Burada, verimli CRISPR-Cas9 tabanlı düzenleme elde etmek için bir protokol oluşturmak için bir araç olarak hizmet etmek üzere GFP'yi (THP-1_GFP) kararlı bir şekilde ifade eden bir THP-1 hücre hattı üretildi. Aynı anda üç sgRNA kullanarak EGFP genini inaktive etmek için bir strateji tasarlandıktan sonra (çoklu kılavuz yaklaşımı), bir okuma olarak GFP ekspresyonu kullanılarak çeşitli elektroporasyon koşulları arasında KO etkinliği belirlendi. Hücre proliferasyonu paralel olarak izlendi. Gen düzenleme, hem bir T7 endonükleaz I (T7EI) testi hem de Sanger dizilimi ile doğrulandı, ardından CRISPR Düzenlemelerinin Çıkarımı (ICE) algoritması26 ile analiz edildi. THP-1 hücrelerinin elektroporasyondan sonra normal büyüme oranlarını geri kazanmasıyla %95'e varan GFP ekspresyonu azalması sağlayan parametreler, endojen bir geni (SAMHD1) inaktive etmek ve tek hücreli THP-1 klonları üretmek için başarıyla kullanıldı.
Protokol
Bu çalışmada kullanılan reaktiflerin ve ekipmanların detayları Malzeme Tablosunda listelenmiştir.
1. CRISPOR ile kılavuz tasarımı (Şekil 1.1)
NOT: SnapGene Viewer yazılımı, düzenleme hedef bölgesine ve ilgilenilen gen içindeki PCR primer hibridizasyonunun konumuna açıklama eklemek için 4, 7 ve 10. adımlarda kullanılabilir.
- Ensembl web sitesine (www.ensembl.org) gidin. Arama kutusuna bir tür seçin ve ilgilendiğiniz genin adını girin. Git'e tıklayın. Gene karşılık gelen sonucu seçin (transkript değil).
- Transkript tablosunu göster'e tıklayın ve ardından Biotype sütununda altın etiketli Protein kodlamasına karşılık gelen Transkript Kimliğini seçin. Transkript sayfasına girdikten sonra, soldaki menüden Exons'a tıklayın.
- Aşağı kaydırın ve Sırayı indir'e tıklayın. Dosya biçiminin MOSTA olduğundan emin olun. Ayarlar - Dahil Edilen Diziler'de, Genomik dizi dışındaki her şeyin seçimini kaldırın.
Transkript kutusunun her iki ucundaki Yan sırayla "500" sayısını girin. Önizleme'ye tıklayın, tüm diziyi seçin (başlık olmadan yalnızca nükleotidler) ve kopyalayın (Ctrl+C). - SnapGene Viewer'ı açın ve Yeni > DNA dosyasına tıklayın. Sırayı (Ctrl+V) Sıra oluştur kutusuna yapıştırın. Ortak özellikleri algıla seçeneğinin işaretini kaldırın ve Topoloji'de Doğrusal'ı seçin.
Dosyayı yeniden adlandırın ve Oluştur'a tıklayın. Soldaki menüde Enzimleri göster (ilk simge) seçimini kaldırın. Pencerenin alt kısmında, Sıra sekmesini seçin.
NOT: Bu adım, ekzonlar, intronlar ve 500 bp yan diziler (isteğe bağlı) dahil olmak üzere tüm gen dizisinin alınmasına izin verir. Bu son bilgi, ilk ekzon içinde yer alan bir hedef bölgenin amplifikasyonu için PCR primerleri tasarlamak için kullanışlıdır. - Ensembl web sitesine geri dönün, dosya önizlemesinde yukarı kaydırın ve Geri'ye tıklayın. Şimdi, Dosya biçimini RTF olarak değiştirin. Ayarlar - Dahil Edilen Diziler'de, Ekzonlar dışındaki her şeyin seçimini kaldırın. Varyasyonları göster bölümünde Hayır'ı seçin. Sayfanın üst kısmındaki İndir'e tıklayın.
- Ekzon dizisini içeren ve ilk ATG'den başlayarak kodlama dizisini mavi renkte gösteren indirilen dosyayı (Word ile) açın. CRISPR-Cas9 tarafından yönlendirilen düzenleme tarafından hedeflenecek eksonu seçin (öneriler için aşağıya bakın), seçin ve kopyalayın (Ctrl + C).
- Hedeflenen ekson, proteinin işlevsel olarak önemli bir alanını kodlayan erken bir ekzon veya bir ekzondur. 3' UTR'ye yakın geç bir eksonda bir PTC'nin kurulmasının muhtemelen NMD'yi ortaya çıkarmayacağını ve bunun da C-terminal olarak kesilmiş bir proteinin ekspresyonuna yol açacağını belirtmekte fayda var. Tersine, bir PTC'nin doğal başlatma bölgesine proksimalde erken bir ekzon sokulması, gayri meşru çeviri riski (ITL, diğer adıyla alternatif çeviri başlatma (ATI)) riski ile ilişkilidir ve bu, ilk ATG kodonunun akış aşağısındaki bir çerçeve içi çeviri başlatma bölgelerinde (TIS) başlayan N-terminal olarak kesilmiş bir proteinin beklenmedik ekspresyonunu verir. Bu ikinci riski azaltmak için, ATGpr27 (atgpr.dbcls.jp) ve/veya NetStart 1.028 (services.healthtech.dtu.dk/services/NetStart-1.0/) kullanılarak alternatif TIS oluşumunun değerlendirilmesi tavsiye edilir.
- Hedef ekzonun bir kodlama dizisi içerdiğinden emin olun. Bununla birlikte, silinen parçaya dahil etmek için ilk ATG'nin yukarı akışında 5'UTR bölgesi ile bir sgRNA tavlaması seçmek yararlı olabilir.
- İdeal olarak, hedeflenen ekson, genin tüm protein kodlayan transkript varyantları için ortak olmalıdır. İlgilenilen geni arayarak NCBI'nin Genom Veri Görüntüleyicisi'nde (www.ncbi.nlm.nih.gov/gdv/) durumun böyle olup olmadığını kontrol edin.
Görüntüleme penceresindeki gen adına (yeşil renkte) tıklamak, transkript varyantlarını (mor renkte) gösterecektir. Ekzonlar bir dikdörtgen ile temsil edilir.
- SnapGene Viewer'da, ekzon dizisini aramak için Ctrl+F, Ctrl+V ve ardından Enter tuşlarına basın. Yeni bir özellik eklemek için Ctrl+T tuşlarına basın, adlandırın, türü "ekzon" olarak değiştirin ve Tamam'a tıklayın.
- CRISPOR web sitesine (http://crispor.gi.ucsc.edu/) gidin ve ekzon dizisini Adım 1'e yapıştırın. İlk olarak, Adım 2'de bir referans genomu seçin ve ardından Adım 3'te hedeflenecek PAM tipini, tipik olarak SpCas9 için 20bp-NGG seçin. GÖNDER'e tıklayın.
- Aralarında 150 bp'ye kadar olacak şekilde iki sgRNA seçin, ardından aralarında üçüncü bir sgRNA seçin. İşte sgRNA seçimi için bazı yönergeler:
- MIT özgüllük skoru, hedef dışı etkilerle ilgilidir. Daha yüksek bir puan, daha az potansiyel hedef dışı olduğunu gösterir. Sağ sütun, konumlarıyla birlikte (bir ekson, bir intron veya bir intergenik bölgede) en olasıdan en az olasıya doğru sıralanmış, tahmin edilen üç hedef dışı siteyi görüntüler. Tahmin edilen hedef dışı sitelerin tam listesine tümünü göster'e tıklayarak erişilebilir. Mümkünse, MIT puanı >80 olan sgRNA'ları seçin ve 0, 1 veya 2 uyumsuzluk için hedef dışı olmayanlara öncelik verin. Ek olarak, fenotipi etkileme potansiyeli en yüksek olan bir eksonda hedef dışı olan sgRNA'lardan kaçınılmalıdır.
- Tahmin edilen etkinlik için Doench '16 puanına bakın. Yüksek bir Doench '16 puanının, sgRNA'nın etkili olma olasılığının daha yüksek olduğunu gösterdiğini unutmayın. Gerçek etkinlik deneysel olarak belirlenmelidir. Bu nedenle, birlikte kullanılması amaçlanmamış olsalar bile, birkaç sgRNA'nın seçilmesi her zaman yararlıdır.
- Çok yüksek veya çok düşük bir GC içeriği ve belirli motifler, sgRNA verimliliğine zarar verebilir ve bundan kaçınılmalıdır. Bu parametreler CRISPOR tarafından vurgulanır.
- sgRNA dizisini ve ilişkili PAM dizisini SnapGene Viewer'daki gen dizisine eklemek için adım 1.7'yi tekrarlayın. Aynı zamanda, oligonükleotid siparişi verirken gereken 5'-3' oryantasyonunu korumak için sgRNA dizisini (PAM olmadan) bir metne veya Excel dosyasına yapıştırın.
- Hedef bölgenin PCR tabanlı amplifikasyonu için, onu çevreleyen birkaç primer tasarlayın. Amplikon boyutu 800 ile 1000 bp arasında olmalıdır. Bu protokol için primerleri tasarlamak için PrimerQuest kullanıldı (https://eu.idtdna.com/pages/tools/primerquest). Alternatif olarak, CRISPOR, hedeflenen genomik bölgeyi ve hedef dışı bölgeleri çoğaltmak için bir primer listesi sağlar. Potansiyel hedef dışı sitelerin tam listesini görüntüledikten sonra (adım 1.9.1), sağ alt köşedeki Hedef dışı primerler'e tıklayın.
2. Elektroporasyon için reaktif ve hücre hazırlığı (Şekil 1.2)
- Kuyucukları %20 ısıyla inaktive edilmiş (56 ° C, 30 dakika), filtrelenmiş (0.20 μm) fetal sığır serumu (FBS) ile desteklenmiş 500 μL RPMI 1640 ortamı ile doldurarak elektroporasyondan sonra hücreleri geri kazanmak için 24 oyuklu bir kültür plakası hazırlayın. Antibiyotik eklemeyin. Plakayı 37 ° C'de ve% 5 CO2'de 24 saat boyunca elektroporasyona kadar nemlendirilmiş bir inkübatörde tutun.
- sgRNA'lar kuru olarak gönderilirse, bunları TE tamponu (10 mM Tris, 1 mM EDTA, pH 8.0) ile 100 μM'lik bir nihai konsantrasyona (yani 1 nmol sgRNA başına 10 μL TE tamponu) rehidre edin. 30 saniye boyunca vorteks, tam rehidrasyona izin vermek için gece boyunca 4 °C'de inkübe edin ve kısa bir pipet homojenizasyonundan sonra sgRNA stok çözeltisini -20 °C'de saklayın. Son hacme bağlı olarak, birden fazla donma-çözülme döngüsünden kaçınmak için alikotlar yapın.
- 100 μM stok çözeltisini nükleaz içermeyen suda seyrelterek 30 μM'lik bir nihai konsantrasyonda çalışan bir sgRNA çözeltisi hazırlayın. 30 saniye boyunca vorteksleyin ve oda sıcaklığında 5 dakika inkübe edin.
- Cas9:sgRNA RNP'lerini, üç 30 μM sgRNA'nın her birinin 1 μL'sini ve 0.5 μL 20 μM Cas9 çözeltisini 3.5 μL resüspansiyon tamponu R'de aynı anda seyrelterek 1:9 molar oranda birleştirin, elektroporasyon kitinde bulunur (bir deneysel koşul için 7 μL'lik nihai hacim; buna göre ölçeklendirin). Kısa bir süre vorteks yapın ve oda sıcaklığında 5 dakika inkübe edin.
- Bu arada, 0,5 μL 20 μM Cas9'dan 6,5 μL resüspansiyon tamponu R. Vortex'e kısaca girerek düzenlenmemiş bir kontrol hazırlayın ve oda sıcaklığında 5 dakika inkübe edin.
- Elektroporasyon koşulu başına 12 μL'lik bir nihai hacim için tüm numunelere 5 μL yeniden süspansiyon tamponu R ekleyin.
- THP-1 hücrelerini elektroporasyon için hazırlayın.
- Tripan mavisi dışlama testi ile konsantrasyonu ve canlılığı değerlendirmek. Hücreleri %0.4'lük bir tripan mavisi boyama solüsyonunda 1: 2 oranında seyreltin. 30 sn inkübasyondan sonra, iyice homojenize edin ve Neubauer tarafından geliştirilmiş ızgara stiline sahip bir sayma odasının duvarına 10 μL ekleyin. Üç büyük kare sayın ve hücre konsantrasyonunu elde etmek için sayıyı 100'e bölün (x106 hücre / mL).
NOT: Hücrelerin sağlığı, elektroporasyona karşı hassasiyetlerini etkiler. Hücre kültürünün en uygun koşullarda gerçekleştirilmesine özen gösterilmelidir. İnkübatörün dışındaki süre sınırlı olmalı ve tüm reaktifler ve çözeltiler önceden hazırlanmalı ve ısıtılmalıdır. - Her koşul için 0,2 x 106 hücreye eşdeğer bir hacim toplayın ve santrifüjleyin (336 x g, 5 dk, 20 °C).
- Süpernatanı bir pipet kullanarak aspire edin ve peleti 500 μL PBS'de yeniden süspanse edin. Tekrar santrifüjleyin (336 x g, 5 dk, 20 °C).
- Süpernatanı bir pipet kullanarak dikkatlice aspire edin ve THP-1 hücre peletini 12 μL RNP çözeltisi ile yeniden süspanse edin (adım 2.6).
- Tripan mavisi dışlama testi ile konsantrasyonu ve canlılığı değerlendirmek. Hücreleri %0.4'lük bir tripan mavisi boyama solüsyonunda 1: 2 oranında seyreltin. 30 sn inkübasyondan sonra, iyice homojenize edin ve Neubauer tarafından geliştirilmiş ızgara stiline sahip bir sayma odasının duvarına 10 μL ekleyin. Üç büyük kare sayın ve hücre konsantrasyonunu elde etmek için sayıyı 100'e bölün (x106 hücre / mL).
3. Elektroporasyon sistemi kurulumu ve nükleofeksiyon (Şekil 1.3)
- Pipet istasyonunu bir biyogüvenlik kabininin altına yerleştirin, desteğe bir elektroporasyon tüpü yerleştirin ve elektroporasyon kitinde bulunan 3 mL tampon E ekleyin.
- Elektroporasyon cihazını açtıktan sonra, aşağıdaki elektroporasyon parametrelerini ayarlamak için dokunmatik ekranı kullanın: Voltaj = 1 500 V, Süre = 10 ms, Sayı = 3.
- Elektroporasyon pipetini bir uçla donatın ve 10 μL RNP/THP-1 solüsyonunu aspire edin (adım 2.7.4). Pipeti elektroporasyon tüpüne yerleştirin ve elektroporasyon cihazı ekranında Başlat düğmesine basın. Tamamlandı mesajının görünmesini bekleyin ve pipeti tüpten çıkarın.
- Hücreleri önceden ısıtılmış 24 oyuklu plakanın bir kuyusuna aktarın ve nazikçe homojenize edin. Plakayı nemlendirilmiş inkübatöre geri koyun ve 72 saat boyunca rahatsız edilmeden dinlenmelerine izin verin.
NOT: RNP/THP-1 süspansiyonunu pipetlerken herhangi bir kabarcık yapmamaya dikkat edin, çünkü bunlar elektroporasyon prosedürüne müdahale edecektir. Görünür bir elektrik arkı olmadığında bir hata mesajı görüntülenirse, elektroporasyon pipetini tüpten çıkarın ve tekrar Başlat'a basın. Bununla birlikte, kısa bir parlak kıvılcım şeklinde bir elektrik arkı gözlenirse, kabarcıkların varlığına işaret edebilir. Elektroporasyon prosedürü, bir hata mesajı olmasa bile büyük olasılıkla başarısız olacaktır.
4. Elektroporasyondan 72 saat sonra THP-1 geri kazanımı (Şekil 1.4)
- Elektroporasyondan 72 saat sonra konsantrasyonu değerlendirmek için hücreleri sayın (adım 2.7.1).
- Yeterli hücre varsa (ör.≥0.6 x 106 hücre / mL), bunları en az% 20 FBS ve% 1 Penisilin-Streptomisin ile desteklenmiş RPMI ile bir faktör 2 seyreltin ve konsantrasyonu 0.3-0.5 x 10mL başına 6 hücre arasına getirin. Aksi takdirde, hücrenin 72 saat daha iyileşmesine izin verin.
- KO doğrulaması için yeterli olana kadar hücreleri geçirin ve yükseltin. Bu arada, tek hücreli klonların izolasyonu başlatılabilir (adım 7).
5. T7EI uyumsuzluk testi ile gen düzenleme doğrulaması (Şekil 1.5)
NOT: T7EI'nin 1 bp'den büyük uyumsuzlukları tanıdığı göz önüne alındığında, tahlil düzenleme verimliliğini hafife alabilir. Bu nedenle, T7EI testi, uygun şekilde modifiye edilmedikçe homozigot hücre popülasyonlarını (yani tek hücreli klonları) taramak için yararlı değildir (adım 5.7).
- Hücre konsantrasyonunu değerlendirin (adım 2.7.1) ve 1.5 mL'lik bir tüpte 0.1 x 106 hücreye eşdeğer bir hacim çekin. Santrifüj (336 x g, 5 dk, 20 °C), süpernatanı aspire edin ve peleti 500 μL PBS'de yeniden süspanse edin. Tekrar santrifüjleyin ve bir pipet kullanarak, peleti rahatsız etmeden mümkün olduğunca fazla süpernatan aspire edin. Numuneyi hızlı bir şekilde dondurun ve -20 °C'de saklayın.
- PCR amplifikasyonu için bir matris görevi görmesi için genomik DNA'yı çıkarın.
- Peletleri 50 μL DNA ekstraksiyon solüsyonu ile yeniden süspanse edin, homojenize edin ve tüm hacmi 0.2 mL'lik bir PCR tüpüne aktarın. Kısaca vorteks ve santrifüj (3 saniye boyunca darbe).
- Tüpü bir termal döngüleyiciye yerleştirin ve 65 °C'de 15 dakika, ardından 98 °C'de 10 dakika ısıtın.
- Ekstrakte edilen DNA'yı 90 μL ultra saf su ile seyreltin. Kısaca vorteks ve santrifüj (5.000 x g, 3 s).
- PCR astarını (Malzeme Tablosuna bakınız) 10 μM'lik (yani 10 pmol/μL) nihai konsantrasyon için ultra saf suda seyreltin.
- PCR karışımını (aşağıdaki NOT'u izleyerek) 0.2 mL'lik bir PCR tüpünde hazırlayın (bir koşul için son hacim = 50 μL). Genellikle, en az iki koşul olacaktır: KO ve yalnızca Cas9 ile düzenlenmemiş kontrol.
NOT: Saflaştırılmış genomik DNA: 10 μL; İleri ve geri astar (10 μM): her biri 2,5 μL, nihai konsantrasyon 500 nM; Reaksiyon Tamponu (5x): 10 μL. Eklemeden önce iyice sarın. dNTP'yi karıştırın (her birinden 25 mM): 0.6 μL, her dNTP için 0.3 mM'lik nihai konsantrasyon. DNA polimeraz: 0.5 μL; Ultra saf su: 23,9 μL. Kısa bir süre vorteks ve santrifüj (3 saniye boyunca darbe). - Tüpleri termal döngüleyiciye yerleştirin ve PCR programını aşağıdaki ayarlarla çalıştırın:
NOT: 95 ° C'de 5 dakika boyunca bir döngü, ardından 30 döngü [20 s için 98 ° C (denatürasyon adımı), 15 s için X °C (tavlama adımı), 72 s için 45 ° C (uzama adımı)], ardından 72 ° C'de 2 dakika boyunca bir son döngü. Amplifikasyonun sonunda, tüpleri, vorteks ve santrifüjü kısa bir süre çıkarın (3 saniye boyunca darbe). Tavlama sıcaklığı (X), astarların erime sıcaklığı (Tm) eksi 5 °C'dir. - Poliklonal düzenlenmiş bir popülasyon için: 0.2 mL'lik yeni bir tüpte, 19.5 μL'lik bir son hacim için 17.5 μL PCR amplikonu ve 2 μL NEBuffer 2 (10x) ekleyin. Kısaca girdap ve santrifüjleyin (3 saniye boyunca darbe). Alternatif olarak, tek hücreli klonları taramak için, hem düzenlenmiş hem de düzenlenmemiş kontrol hücrelerinden PCR ürünlerinin 1: 1'ini karıştırın (adım 5.6) (Ek Şekil 1).
- Heterodubleks oluşum için, tüpleri termal döngüleyiciye yerleştirin ve aşağıdaki programı çalıştırın: 95 °C'de 10 dakika boyunca bir döngü, biri 95 ila 85 °C arasında -2 °C/s'lik bir rampa ile, biri 85 ila 25 °C arasında -0,3 °C/s'lik bir rampa ile ve HOLD'da 10 °C'de bir son soğutma döngüsü.
- Heterodubleks oluşumundan sonra, tüpe 0.5 μL T7EI çözeltisi ekleyin. 37 °C'de 30 dakika inkübe edin.
- % 1.2 agaroz jeli hazırlayın:
- Agarozu bir cam şişede tartın ve uygun hacimde 1x TAE tamponu (40 mM Tris-asetat, 1 mM EDTA, pH 8.3) ekleyin. Kapağı sıkıca vidalamamaya dikkat ederek bir mikrodalgaya koyun. Agaroz kristalleri tamamen çözülene kadar birkaç kez ısıtın.
NOT: Gerekirse, çözeltiyi karıştırın. Kaynamasına izin vermeyin, aksi takdirde agaroz konsantrasyonunu değiştirerek hacim azalır. - 1/20.000 oranında seyreltilmiş DNA jel boyasını ekleyin ve iyice karıştırın.
- Agaroz çözeltisini bir kalıba dökün, bir tarak ekleyin ve oda sıcaklığında katılaşmasına izin verin.
- Agarozu bir cam şişede tartın ve uygun hacimde 1x TAE tamponu (40 mM Tris-asetat, 1 mM EDTA, pH 8.3) ekleyin. Kapağı sıkıca vidalamamaya dikkat ederek bir mikrodalgaya koyun. Agaroz kristalleri tamamen çözülene kadar birkaç kez ısıtın.
- 5 μL T7EI-sindirim ürününü (adım 5.9) veya sindirilmemiş PCR ürününü 5 μL su ve 2 μL 6x DNA yükleme boyası ile karıştırarak jel elektroforezi için numuneler hazırlayın. Numuneleri ve boyut merdivenini yükleyin ve 45 dakika boyunca 80 V'ta hareket ettirin (zaman, DNA parçalarının beklenen boyutuna bağlı olarak uyarlanabilir). Migrasyon bittikten sonra, uygun bir görüntüleme sistemi ile jelin bir görüntüsünü elde edin.
6. Sanger dizileme analizi ile gen düzenleme doğrulaması (Şekil 1.6)
- Genetik modifikasyonu karakterize etmek için PCR ürünlerini saflaştırın (adım 5.4) ve Sanger dizilemesinden sonra sonuçları ICE aracı (https://www.synthego.com/products/bioinformatics/crispr-analysis) ile analiz edin.
- Modifikasyonlara rağmen bir proteinin üretilip üretilemeyeceğini kontrol edin.
- ICE sonuçlarını indirin ve contribs.txt dosyasını açın.
- Düzenlenen dizileri WT muadili ile karşılaştırın. İndelleri haritalayın ve hedeflenen genomik bölgede ortaya çıktıklarını doğrulayın. Boyutlarını değerlendirin. İç uzunluk üçün katı değilse, bir çerçeve kayması meydana gelecek ve muhtemelen bir PTC tanıtılacaktır. Mutasyona uğramış mRNA'nın NMD aracılı bozunmaya uğraması bekleniyor.
7. Seyreltmeyi sınırlayarak tek hücreli klon izolasyonu (Şekil 1.7)
NOT: Tek hücreli klonların izolasyonu zorunlu değildir. Bununla birlikte, bunu yapmayı seçerseniz, birden fazla klonu karakterize etmek ve fenotiplerini orijinal poliklonal popülasyonla karşılaştırmak önemlidir.
- Bir hücre süspansiyonu örneği alın, kültür ortamı ile 1/2 oranında seyreltin ve konsantrasyonu değerlendirin (adım 2.7.1). Seyreltme, sayım doğruluğunu artırır.
- 7 hücre / mL konsantrasyona ulaşmak için 2 ila 3 seri seyreltme adımı gerçekleştirin. Yuvarlak tabanlı 96 oyuklu bir plakada (yani, oyuk başına 0.7 hücre) oyuk başına 100 μL hücre süspansiyonu dağıtın. Bir hücre içeren kuyucukları tanımlamak için mikroskopta plakaları gözlemlemeden önce hücrenin birkaç saat boşalmasına izin verin.
- Düzenli olarak koloni oluşumunu ve büyümesini izleyin ve gerektiğinde hücreleri daha büyük bir kültür plakasına veya şişeye aktarın.
8. THP-1 KO SAMHD1 hücrelerinin HIV-1 kısıtlama testi ile fonksiyonel karakterizasyonu
- % 10 FBS,% 1 Penisilin-Streptomisin (tam RPMI) ile desteklenmiş ve farklılaşma için 300 ng / mL PMA içeren 300 μL RPMI'de oyuk başına 0.25 x10 6 hücreli 24 oyuklu bir kültür plakasında tohum THP-1. Plakayı 24 saat boyunca nemlendirilmiş bir inkübatörde (37 °C,% 5 CO2) tutun.
- Ortamı PMA içermeyen tam bir RPMI ortamıyla değiştirin ve plakayı 24 saat daha inkübatöre geri koyun.
- Bir vakum pompası kullanarak tüm kültür ortamını aspire edin. 250 μL VSVg psödotipli HIV-1-GFP viral stok içeren çözelti ekleyin (MOI = 0.5 IFU/hücre). Enfekte olmayan bir kontrol ekleyin. Enfeksiyonu senkronize etmek için plakayı 2 saat boyunca 4 ° C'ye yerleştirin.
- Hücreleri RPMI ile bir kez yıkayın. Her oyuğa 500 μL tam RPMI ekleyin ve standart koşullarda 48 saat inkübe edin (zaman ayarlanabilir).
- Kültür ortamını çıkardıktan sonra, hücreleri bir kez soğuk 1x PBS ile yıkayın. Ardından, her oyuğa 100 μL% 0.05 Tripsin-EDTA ekleyin. Enzimi inaktive etmek için 200 μL tam RPMI eklemeden önce plakayı hücreler tamamen ayrılana kadar (5 dakika yeterli olmalıdır) 37 ° C'de bir inkübatöre yerleştirin. Her oyuktan 250 μL'yi 96 oyuklu yuvarlak tabanlı bir plakaya aktarın (akış sitometresinin kültür plakalarını kabul ettiğinden emin olun).
- Plakayı santrifüjleyin (757 x g, 3 dk, yavaşlama = 6, 20 °C) ve çok kanallı bir pipet kullanarak süpernatanı aspire edin. Peleti 100 μL %4 PFA ile yeniden süspanse edin ve 4 °C'de 10 dakika inkübe edin. 100 μL soğuk 1X PBS ekleyin.
- Akış sitometrisi ile GFP pozitif hücreler tarafından temsil edilen enfeksiyon oranını analiz edin (önerilen bir geçit stratejisi için Ek Şekil 2'ye bakınız).
Sonuçlar
GFP raportör proteinini (THP-1_GFP) stabil bir şekilde eksprese eden bir THP-1 hücre hattı oluşturuldu (Şekil 2A) ve verimli bir CRISPR-Cas9 aracılı gen baskısı için bir protokol oluşturmak için bir araç olarak kullanıldı. Bu amaçla, EGFP genini hedefleyen 3 sgRNA, CRISPOR web aracı29 (Şekil 2B) ile tasarlandı ve bunlar aynı anda Cas9 ile 9:1'lik bir molar oranda kompleks haline getirildi ve farklı ayarlar kullanılarak elektroporasyon yoluyla hücrelere verilmeden önce RNP'ler oluşturdu. Daha sonra, hücreler ya tek başına% 20 FBS içeren RPMI ortamında büyütüldü (Şekil 2C, üst panel) ya da şartlandırılmış ortamla 1: 1 oranında seyreltildi (Şekil 2C, alt panel). EGFP KO verimliliğinin bir okuması olarak hücre proliferasyonu ve GFP ekspresyonu zaman içinde izlendi. Çeşitli koşullar için, GFP pozitif hücrelerin yüzdesi keskin bir şekilde azaldı ve elektroporasyondan (pe) sonraki 7. günde %>90'lık bir azalmaya ulaştı. 3. günde, muhtemelen elektroporasyonun hücre canlılığı üzerindeki olumsuz etkisine bağlı olarak hücre sayısı yarı yarıya azalmıştı. Bununla birlikte, hücre sayısı tekrar yükseldi ve iki katına çıkma süresi 7 pe gününe kadar normal bir orana döndü (Şekil 2C, üst panel). Şartlandırılmış ortamın kullanımı, deneysel koşullarımız altında hücre geri kazanımını desteklemedi (Şekil 2C, alt panel). Bu sonuçlara dayanarak, takip deneyleri için 1500 V'luk üç adet 10 ms'lik darbe seçildi.
CRISPR-Cas9 aracılı EGFP KO daha sonra genomik düzeyde karakterize edildi. Bunun için, THP-1_GFP hücrelerinin genomik DNA'sı ekstrakte edildi, Cas9: sgRNA RNP'leri (düzenlenmiş) veya tek başına Cas9 (düzenlenmemiş) ile elektropore edildi ve hedef bölgeyi içeren 891 bp'lik bir bölgeyi PCR-amplifiye etmek için bir şablon olarak kullanıldı. EGFP'den yoksun ebeveyn THP-1 hücreleri de negatif kontrol olarak dahil edildi. Daha sonra, düzenlenmiş THP-1_GFP hücrelerinin PCR ürünü, T7EI testi ile uyumsuzluk tespiti için hazırlandı, ardından DNA fragmanlarının agaroz jel elektroforezi ile ayrılması izledi. WT ampliconunun beklenen boyutuna karşılık gelen ~ 900 bp bandı, düzenlenmemiş THP-1_GFP hücreleri için kolayca görselleştirildi, ancak ebeveyn THP-1 kontrolü için değil (Şekil 2D, satır 2 ve 3'ü karşılaştırın). Düzenlenmiş THP-1_GFP hücreleri söz konusu olduğunda, ~ 900 bp bandı tespit edilemez hale geldi ve boyutu, CRISPR düzenlemesine bağlı olarak ~ 75 bp'lik bir parçanın kaybıyla eşleşen daha hızlı göç eden bir bantla değiştirildi (Şekil 2D, satır 4). Bu bantlama modeli, T7EI sindirimi üzerine değiştirildi, daha küçük DNA parçalarının ortaya çıkmasına neden oldu ve hedef bölgenin başarılı bir şekilde düzenlenmesini daha da doğruladı (Şekil 2D, satır 5). Düzenleme verimliliği ve düzenlenen popülasyonun spesifik genotipleri hakkında bilgi edinmek için, PCR amplikonu Sanger yöntemi kullanılarak dizilendi, ardından ICE aracı26 ile analiz edildi. Düzenlenmemiş kontrol numunesi paralel olarak işlendi. Düzenlenen örnekteki WT olmayan dizilerin yüzdesine ve muhtemelen işlevsel bir KO'ya yol açan indellerin oranına karşılık gelen %100'lük indel ve KO skorlarını aldık (Şekil 2E, solda). İndel boyutu spektrumunun ve frekansının ayrıntılı analizi, 62 bp veya 76 bp delesyon taşıyan ve toplam dizilerin sırasıyla% 57 veya% 37'sini oluşturan iki baskın popülasyonun varlığını gösterdi (Şekil 2E, sağda). Bu bulgular, sgRNA #1'in, sgRNA #2 veya #3 ile kombinasyon halinde, hedef gende eşzamanlı DSB'yi tetiklediğini göstermektedir (Şekil 2B).
Bu protokol daha sonra endojen SAMHD1 kodlayan geni inaktive etmek için uygulandı (Şekil 3A). SAMHD1 KO THP-1 poliklonal hücre hattının karakterizasyonu, düzenlenmemiş THP-1 hücrelerininkiyle eşleşen zaman içinde hücre proliferasyonunun izlenmesini içeriyordu (Şekil 3B). Gen düzenlemenin sonucu, hedef dizinin PCR tabanlı amplifikasyonu ile değerlendirildi, bu da SAMHD1 KO hücreleri için yalnızca Cas9 kontrolüne kıyasla daha küçük bir bant ortaya çıkardı (Şekil 3C, satır 2 ve 4'ü karşılaştırın), bu da bir DNA dizisi kaybının göstergesidir. Bu gözlemler, SAMHD1 KO hücreleri için bölünme ürünlerinin görünümünü gösteren, ancak düzenlenmemiş hücreler olan T7EI testi ile doğrulandı (Şekil 3C, satır 3 ve 5'i karşılaştırın). Sanger sıralama verilerinin ICE aracıyla analizi, sırasıyla %100 ve %97'lik indel ve KO skorları döndürdü. SAMHD1 lokusu içindeki ana modifikasyonlar, dizilerin sırasıyla% 46,% 13 ve% 10'unda 170 bp, 93 bp veya 104 bp dizi delesyonlarıydı (Şekil 3D). Ham hücre lizatındaki protein ekspresyon seviyeleri de immünoblotlama ile değerlendirildi. Endojen SAMHD1'in boyutuyla tutarlı ~ 70 kDa bandı, düzenlemeden sonra neredeyse tespit edilemez hale gelir ve tahmini% >97'lik bir azalmaya karşılık gelir (Şekil 3E). Son olarak, SAMHD1 KO THP-1 poliklonal hücre hatlarının fenotipik karakterizasyonu, HIV-1 enfeksiyonuna duyarlılığın test edilmesiyle de değerlendirildi. İyi bilinen antiviral rolü30,31 ile uyumlu olarak, SAMHD1 lokusunun inaktivasyonuna artmış bir HIV-1 enfeksiyon oranı eşlik etmiştir (Şekil 3F).
Paralel olarak, seyreltmeyi sınırlayarak tek hücreli klonlar üretildi. SAMHD1 KO klonlarından genomik hedef bölgenin amplifikasyonu, düzenlenmemiş kontrol hücrelerine kıyasla daha yüksek elektroforetik hareketliliğe sahip PCR ürünleri verdi (Şekil 4A). Sanger dizileme verilerinin ICE tabanlı analizi, biri (klon A2) hariç seçilen tüm tek hücreli klonlar için %100'lük bir indel skoru döndürdü (Şekil 4B, solda). Genomik düzeyde düzenleme sonuçlarının karakterizasyonu, ilk ATG'nin 28 pb aşağısında bulunan 98 bp'lik bir delesyon ile klon A3 için tek bir dizi döndürdü ve böylece çerçeve dışı bir değişikliğe yol açtı (Şekil 4B, sağ). Diğer klonlar, sınırlayıcı seyreltme adımında farklı düzenleme olaylarına ve/veya kuyu başına birden fazla hücreye maruz kalmış alellerin varlığını gösteren üç (klon B1) veya daha fazla dizi içeriyordu (Şekil 4B, sağ). Özellikle, A4 ve A7 klonlarının dizilerinin %11'i, ilk ATG'nin yukarısında indeller taşır ve kodlama dizisini değiştirmeden bırakır. Ayrıca, klon A7 dizilerinin %1'i, ilk ATG'nin aşağı akışında 123 bp'lik bir 9-bp fragmanından yoksundu ve bu da 41 amino asidin çerçeve içi silinmesine neden oldu. Bu bulgularla tutarlı olarak, fonksiyonel çalışmalar, bozulmuş bir SAMHD1 lokusu (A3, A8, B1, B3) olan ve dolayısıyla SAMHD1 ekspresyonu olmayan klonların, HIV-1 enfeksiyonuna yüksek derecede izin verdiğini ortaya koymuştur (Şekil 4C). Tersine, SAMHD1'in sağlam olduğu A4 ve A7 klonları, ebeveyn veya düzenlenmemiş kontrol THP-1 klonları kadar refrakterdi (Şekil 4C). Sonuç olarak, klon A3'teki SAMHD1 lokusunun başarılı bir şekilde düzenlenmesi doğrulandı.
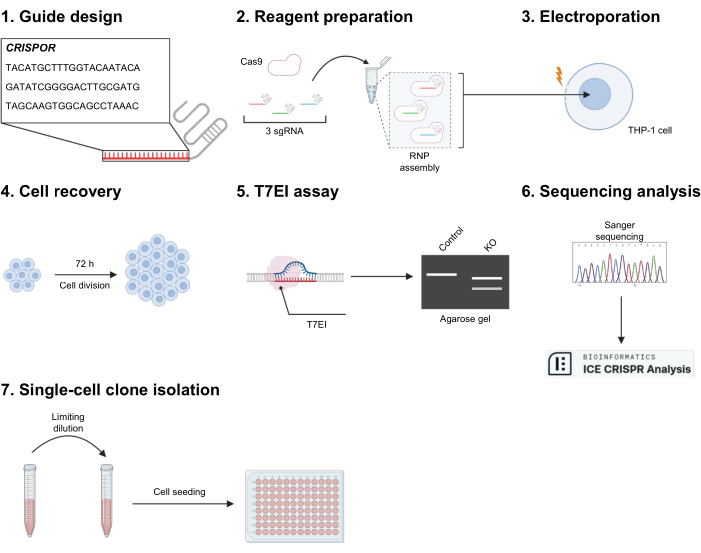
Şekil 1: Deneysel taslak. (1) CRISPOR, hedeflenen eksona karşı sgRNA dizileri oluşturmak için kullanılır. Bunlardan üçü, yüksek hedef içi etkinlik ve zayıf hedef dışı tahmine göre seçilir. (2) Üç gRNA, ribonükleoproteini (RNP) birleştirmek için Cas9 ile karıştırılır, bu da üç farklı RNP'nin bir karışımı ile sonuçlanır. (3) Hücreler, RNP'lerin girişine izin vermek için elektroporasyona tabi tutulur. (4) Hücreler 24 oyuklu bir kültür plakasına aktarılır ve en az 72 saat dinlenmeye bırakılır. (5) Gen düzenleme ilk olarak bir T7EI sindirim testi ile kalitatif olarak test edilir, ve (6) Sanger dizilimi ve ICE analizi ile daha fazla karakterize edilen doğrulanmış koşullar için. (7) Gerekirse, klonal popülasyonlar üretmek için seyreltme sınırlandıktan sonra hücreler tohumlanabilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
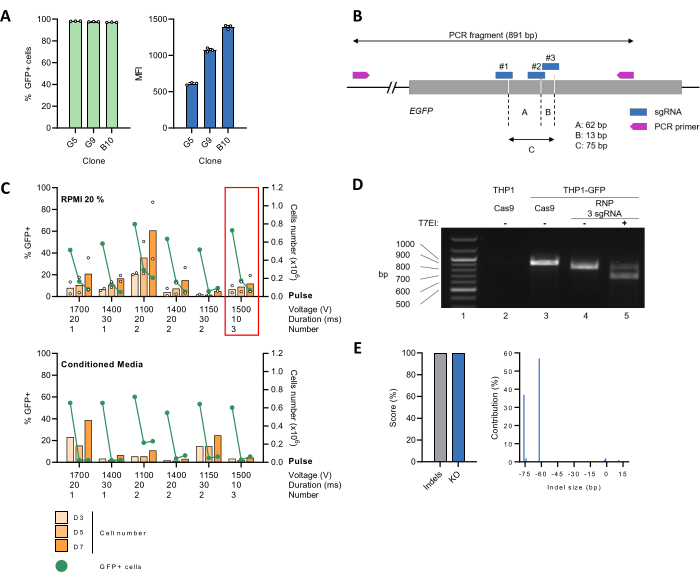
Şekil 2: THP-1 hücrelerinde EGFP KO için elektroporasyon ayarlarının ayarlanması ve gen düzenleme doğrulaması. (A) THP-1 hücreleri, EGFP raportör genini kodlayan lentiviral vektörlerle dönüştürüldü. Tek hücreli klonlar, seyreltmenin sınırlandırılması ve ardından 30 gün boyunca kültürde genleşme ile elde edildi. Akış sitometrisi ile yapılan analiz, neredeyse tüm hücre popülasyonunun GFP'yi eksprese ettiğini doğruladı. Entegre EGFP kopya sayısının göstergesi olan GFP pozitif popülasyonun medyan floresan yoğunluğu (MFI), birkaç hücre klonu arasında değişmiştir. Takip çalışmaları için Clone G5 seçildi. (B) Hem sgRNA hem de primerler için bağlanma bölgeleri dahil olmak üzere EGFP geninin şematik gösterimi. Cas9 kesim yerleri kesikli çizgilerle gösterilir. (C) Farklılaşmamış THP-1_GFP (2 x 105) hücreleri, belirtilen ayarlarla elektroporasyondan önce önceden monte edilmiş Cas9: sgRNA RNP'leri ile karıştırıldı. Daha sonra hücreler, tek başına 500 μL önceden ısıtılmış% 20 FBS içeren RPMI ortamı içeren 24 oyuklu bir plakaya transfer edildi veya 48 saatlik bir THP-1 hücre kültürü tarafından üretilen şartlandırılmış ortam ile 1: 1 oranında seyreltildi. Proliferasyon (turuncu çubuklar) ve GFP pozitif hücrelerin yüzdesi (yeşil çizgi) zaman içinde izlendi. Veriler, n = 2 biyolojik kopyanın ortalamasını temsil eder. Kırmızı çerçeve, sonraki deneyler için seçilen koşulu vurgular. (D) Düzenlenmiş (RNP 3 sgRNA) veya düzenlenmemiş (Cas9) THP-1_GFP hücrelerinin genomik DNA'sı ekstrakte edildi ve hedef lokusu çevreleyen bölge PCR ile amplifiye edildi. Negatif kontrol olarak ebeveyn THP-1 hücreleri kullanıldı. Amplikonlar, bir heterodupleks oluşumuna yol açan bir denatürasyon/yeniden doğuş döngüsüne tabi tutuldu. Daha sonra, T7EI testi ile uyumsuzluklar tespit edildi. DNA fragmanları %1.2 agaroz jel elektroforezi ile ayrıldı. (E) Saflaştırılmış PCR ürünleri Sanger yöntemi ile dizilendi, ardından ICE algoritması ile analiz edildi. Indel ve KO skorları, örnekteki WT olmayan dizilerin yüzdesini ve sırasıyla çerçeve kayması veya 21+ bp indel olan hücrelerin oranını gösterir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
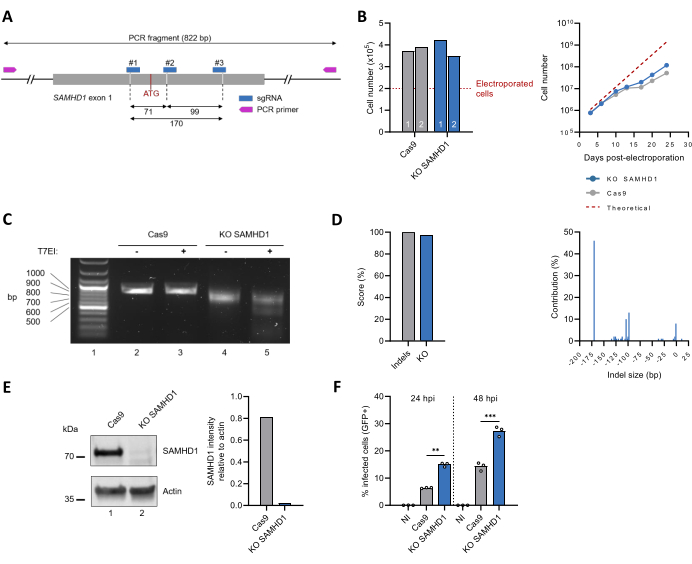
Şekil 3: Bir poliklonal SAMHD1 KO THP-1 hücre popülasyonunun üretimi ve karakterizasyonu. (A) sgRNA ve primerler için bağlanma bölgeleri dahil olmak üzere SAMHD1 ekzon 1'in şematik gösterimi. Cas9 kesim yerleri kesikli çizgilerle gösterilir ve ilk ATG kodonu kırmızı ile gösterilir. (B) Elektroporasyondan üç gün sonra, canlı hücreler (n = 2), Tripan mavisi dışlama testi (soldaki diyagram) kullanılarak ölçüldü. Hücre büyümesi 25 gün boyunca izlendi (sağ diyagram). Kesikli çizgi, 49 saatlik tahmini iki katına çıkma süresine dayanan teorik hücre sayısına karşılık gelir. (C) Şekil 2D'de gösterildiği gibi genomik düzeyde düzenleme sonuçları değerlendirildi. (D) CRISPR düzenlemesini takiben genotipler, Sanger dizilerinin ICE tabanlı analizi ile tanımlanmış ve ölçülmüştür. (E) Hem düzenlenmiş hem de düzenlenmemiş THP-1 hücrelerinin ham lizatında bulunan protein, SDS-PAGE jeli üzerinde migrasyon ile ayrıldı ve daha sonra SAMHD1'e karşı antikorlar kullanılarak immünoblotlama ile görselleştirildi. Yükleme kontrolü olarak aktin kullanıldı. SAMHD1 ve Aktin bantlarının yoğunluğu, ImageJ yazılımı ile dansitometri ile ölçüldü. (F) Düzenlenmemiş (Cas9) ve KO SAMHD1 THP-1 hücreleri, PMA (300 ng / mL, 24 saat) ile tedavi ile farklılaştırıldı ve daha sonra bir raportör olarak GFP'yi ifade eden bir VSVg psödotipli HIV-1 virüsü ile sorgulandı. GFP pozitif hücrelerin oranı, enfeksiyondan 24 saat ve 48 saat sonra (hpi) akış sitometrisi ile puanlandı. NI: enfekte değil. **P < 0,01; P < 0.001, Welch düzeltmesi ile eşleşmemiş t-testi ile. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 4: SAMHD1 KO tek hücreli klonların üretimi ve karakterizasyonu. (A) Seçilen SAMHD1 KO tek hücreli klonlar için düzenleme sonucu, agaroz jel elektroforezi ile hedef bölgeyi içeren bölgenin PCR amplifikasyonu ile üretilen DNA fragmanlarının hareketliliğinin izlenmesiyle değerlendirildi. (B) Sanger dizileme verileri, Şekil 2E'deki gibi ICE aracı ile analiz edildi. Sağdaki grafikteki çubuklar, her hücre popülasyonundaki indel çeşitliliğini temsil eder. Yeşil çubuk, baskın diziyi ve ilişkili silme işleminin toplam boyutunu temsil eder. Parantez içindeki sayılar, sürekli olmayan boşlukların boyutunu gösterir. (C) Düzenlenmemiş ve SAMHD1 KO tek hücreli klonların HIV-1 enfeksiyonuna izin verilebilirliği Şekil 3F'deki gibi test edildi ve elektroporasyonlu olmayan ebeveyn THP-1 hücreleri veya düzenlenmemiş hücre klonları (Cas9) ile karşılaştırıldı. Enfeksiyon oranı 48 hpi olarak puanlandı. *P < 0.05; **P < 0,01; P < 0.001; P < 0.0001; ns P≥ 0.05 Brown-Forsythe ve Welch ANOVA ile Dunnett'in çoklu karşılaştırma testi ile. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Ek Şekil 1: Uyarlanmış T7EI testi ile KO tek hücreli klonlar için düzenleme sonucunun değerlendirilmesi. KO tek hücreli klonların veya düzenlenmemiş hücrelerin genomik DNA'sı saflaştırıldı ve hedef lokusu çevreleyen bölge PCR ile amplifiye edildi. Her bir tek hücreli klonun amplikonları, düzenlenmemiş kontrol hücrelerininkilerle 1: 1 oranında karıştırıldı. Heterodubleks oluşumu ve T7EI sindiriminden sonra, ürün agaroz jel elektroforezi ile analiz edildi. Düzenlenmiş klonlar birden fazla sindirim parçası üretirken (klonlar A, B, C, D ve E), düzenlenmemiş olanlar tek bir WT bandı gösterir (klonlar F ve G). Bu dosyayı indirmek için lütfen buraya tıklayın.
Ek Şekil 2: HIV-1 ile enfekte PMA ile tedavi edilen THP-1 hücrelerinin yüzdesini ölçmek için akış sitometrisi geçit stratejisi. Yan saçılma alanına (SSC-A) karşı ileri saçılma alanı (FSC-A) kullanılarak, kalıntıları ortadan kaldırmak ve PMA ile muamele edilmiş THP-1 hücrelerini seçmek için bir ilk kapı tanımlanır. Daha sonra, bir SSC-H grafiğine karşı bir SSC-A ile tek hücreler seçilir. Son olarak, FSC-H'ye karşı GFP-A ile GFP pozitif hücreler tespit edilir. Bu dosyayı indirmek için lütfen buraya tıklayın.
Tartışmalar
Burada, THP-1 hücre hattının başarılı bir CRISPR aracılı düzenlemesini elde etmek için bir protokol açıklanmaktadır. Yaklaşım, önceden monte edilmiş sgRNA/Cas9 RNP'lerin elektroporasyon/nükleofeksiyon yoluyla transferine dayanır. Bu strateji, sgRNA/Cas9 kasetinin lentiviral aracılı entegrasyonu üzerine potansiyel olarak ortaya çıkan ve nükleazın kalıcı ekspresyonunu sağlayan hedef dışı etkileri sınırlamak için seçilmiştir. İlgili geni hedefleyen çoklu sgRNA'lar, güvenilir ve verimli düzenleme elde etmek için seçildi, bu da genomik indeller oluşturma olasılığını artırarak protein ekspresyonu ve fonksiyonel KO32 kaybına yol açtı. Aşırı hücre ölümünü önlerken sgRNA/Cas9 RNP'lerin verimli bir şekilde iletilmesini sağlamak için gerekli olan hücre zarının optimum geçirgenliğini elde etmek için elektroporasyon ayarlarına özel dikkat gösterildi33. Bu bağlamda, çok önemli bir parametre, elektroporasyondan önceki hücre kültürünün canlılığıdır (yani, düşük geçiş sayısı).
CRISPR-Cas9 aracılı düzenleme, Sanger diziliminden önce ön seçim için birden fazla numunenin uygun maliyetli ve hızlı bir değerlendirmesini sağlayan T7EI uyumsuzluk tespit testi ile genetik düzeyde doğrulandı. Bu yöntem başlangıçta, tam uzunlukta amplikonların ve bölünme ürünlerininyoğunluğunu karşılaştırarak tek bir sgRNA kullanarak düzenleme verimliliğini tahmin etmek için geliştirilmiştir 34,35. Dikkate değer bir şekilde, çoklu sgRNA kullanıldığında üretilen büyük indeller (>70 bp), standart jel elektroforezi ile düzenlenmiş ve düzenlenmemiş hücrelerden hedeflenen lokusların PCR amplifikasyonu ile elde edilen DNA fragmanlarının hareketliliği karşılaştırılarak tespit edilebilir. Bununla birlikte, T7EI testi, bir poliklonal veya uyarlanmış protokolü kullanarak bir monoklonal hücre popülasyonunda küçük indelleri (>1bp) barındıran dizilerin varlığını tespit etmek için yararlı olabilir.
Kodlama dizisini kesintiye uğratan hedef içi çerçeve dışı mutasyonların varlığı, tüm hücre popülasyonunun genomunun ekstrakte edildiği ve PCR amplifikasyonu için bir matris olarak kullanıldığı basit bir yaklaşımla doğrulandı. Daha sonra, saflaştırılmış amplikonlar toplu olarak Sanger dizilimine tabi tutulur, ardından ICE aracı ile biyoinformatik analiz yapılır. Genel olarak, bu yöntem, PCR ürünlerinin bir plazmide alt klonlanması ve ligasyon ürünlerinin, ayrı ayrı dizilenen tek hücreli kolonileri izole etmek için bakterilere dönüştürülmesi ihtiyacının üstesinden gelir.
Hedeflenen lokusun inaktivasyonu doğrulandıktan sonra, belirli bir fenotipi ilgilenilen proteinin eksikliğine güvenle atfetmek ve hedef dışı ve aynı zamanda mRNA yapısını ve/veya işlevini etkileyebilecek hedef dışı değişikliklerden kaynaklanan beklenmedik etkileri dışlamak için düzenlenmiş hücre hattının kapsamlı bir karakterizasyonu gereklidir.
Protein ekspresyonunun yokluğu, farklı epitopları tanıyan muhtemelen iki antikor kullanılarak immünoblotlama ve / veya immünofloresan ile doğrulanmalıdır. Gerçekten de, kanonik ATG kodonuna yakın erken bir eksonda bir PTC'nin sokulması, N-terminal olarak kesilmiş protein varyantları36 veren ITL olaylarının riski ile ilişkilidir. Dikkat çekici bir şekilde, Tuladhar ve ark. ticari veya kurum içi kökenli CRISPR ile düzenlenmiş hücrelerin önemli bir kısmında (~% 50) anormal protein türlerinin tespit edildiğini bildirmiştir18,19. Yeni protein ürünlerinin ortaya çıkması, indel içeren ekzonun atlanması veya kanonik ATG kodonunun alternatif TIS aşağı akışında protein sentezinin başlamasına neden olan iç ribozom giriş bölgelerinin (IRES) ortaya çıkması gibi çeşitli ve potansiyel olarak hücre tipine özgü süreçlere atfedildi. ITL'nin belirli koşullar altında NMD'den bir kaçışla birleştiğine dair ipuçları da vardır37,38. Benzer şekilde, geç bir ekzonu hedefleyen genom düzenleme stratejileri, gen ekspresyonunun20'nin etkisiz ablasyonunun altında yatan mutasyona uğramış mRNA'nın NMD'sini tetiklemede başarısız olabilir. Son olarak, Cas9 birden fazla sgRNA tarafından yönlendirilirse, inversiyonlar veya büyük DNA fragmanı delesyonları39 dahil olmak üzere karmaşık kromozomal yeniden düzenlemeler ortaya çıkabilir, bu da özellikle düzenleyici elemanlar hedeflendiğinde genomun yapısını ve ekspresyonunu değiştirebilir40.
İstenmeyen genom düzenleme sonuçlarının etkisini daha da azaltmak için, protein ekspresyonu kaybı ile takip eden fenotip (yani antiviral aktivite) arasındaki ilişkiyi doğrulamak için fonksiyonel çalışmalara ihtiyaç vardır. Düzenlenmiş hücreler alt klonlanırsa, birden fazla klonun kendi aralarında ve poliklonal popülasyon ile davranışları karşılaştırılmalıdır. Düzenlenen hücrelerin fenotipini bir WT fenotipine tersine çevirmek için gen ekspresyonunun geri yüklenmesinden oluşan kurtarma deneylerinin yapılması da özgüllüğü güçlendirecek ve olası hedef dışı olayları dışlayacaktır.
Bu protokol THP-1 hücrelerinin düzenlenmesi için tasarlanmış olsa da, CRISPR-Cas9 KO iş akışı için diğer hücre hatlarına uyarlanabilen genel bir yapı sağlar. Elektroporasyon parametrelerinin ayarlanması gerekebilir ve 1800 V'tan daha yüksek bir voltaj uygulanırsa, üretici talimatlarına göre bir yeniden süspansiyon tamponu T kullanılmalıdır. Dikkate alınması gereken bir diğer değişken, RNP montajı sırasında Cas9 ve sgRNA arasındaki konsantrasyon oranıdır. 1:9 oranı bu durumda iyi çalışsa da, onu değiştirmek diğer durumlarda düzenleme verimliliğini artırabilir. Son olarak, burada faydalı olmasa da, elektroporasyondan sonra hücre geri kazanımı için şartlandırılmış ortamın kullanımı test edilmelidir, çünkü etki farklı hücre hatlarında farklı olabilir.
Açıklamalar
Tüm yazarların çıkar çatışması yoktur.
Teşekkürler
JP Concordet'e (MNHN, U1154/UMR7196, Paris), G. Bossis'e (IGMM, Montpellier) ve D. Schlüter'e (Hannover Tıp Fakültesi, Almanya) protokolleri paylaştıkları ve tartıştıkları için minnettarız. Bu proje, Avrupa Birliği'nin Horizon 2020 araştırma ve yenilik programından (AZ'ye 101017572 No'lu hibe sözleşmesi) ve ANRS'den (AZ'ye hibe ECTZ162721) fon almıştır. Enfeksiyon Hastalıkları Modeli ve Yenilikçi Tedaviler (IDMIT) araştırma altyapısı, referans ANR_11_INSB_0008 altında "programme investissement d'avenir (PIA)" tarafından desteklenmektedir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filter | ClearLine | 146560 | _ |
| 0.4 % trypan blue | Beckman Coulter | 383200 | _ |
| 1.5 mL tube | Eppendorf | 3810X | _ |
| 24-well plate | Corning | 353047 | _ |
| 6x TriTrack DNA Loading Dye | Thermo scientific | R1161 | _ |
| 75 cm² Culture Flask Vented Cap | Corning | 353136 | _ |
| 8-Strip PCR Tubes with Caps | Life technologies | AM12230 | _ |
| 96-well plates Flat bottom | Corning | 353072 | _ |
| 96-well plates Round bottom | Corning | 353077 | _ |
| Agarose | Euromedex | D5 | _ |
| ATGpr | _ | _ | https://atgpr.dbcls.jp/ |
| ChemiDoc Imaging System | BIO-RAD | 12003153 | _ |
| Counting slide | NanoEntek | DHC-N04 | _ |
| CRISPOR | _ | _ | http://crispor.gi.ucsc.edu/ |
| DPBS | Gibco | 14190094 | _ |
| Ensembl | EMBL-EBI | _ | https://www.ensembl.org/index.html |
| Fetal Bovine Serum | Sigma-Aldrich | F7524 | _ |
| FlowJo | BD Life Sciences | v10.10 | _ |
| GeneRuler 100 bp Plus DNA Ladder | Thermo scientific | SM0323 | _ |
| Genome Data Viewer | NCBI | _ | https://www.ncbi.nlm.nih.gov/gdv/ |
| GraphPad Prism | Dotmatics | _ | Version 9.3.1 |
| Herculase II Fusion DNA Polymerases | Agilent | 600679 | _ |
| ICE CRISPR Analysis Tool | Synthego | _ | https://www.synthego.com/products/bioinformatics/crispr-analysis |
| Image Lab Touch | BIO-RAD | _ | Version 2.4.0.03 |
| NEBuffer 2 | New England Biolabs | B7002S | Included with T7EI M0302S |
| Neon Kit, 10 µL | Invitrogen | MPK1025K | Electroporation kit containing tips, tubes, buffer R and E |
| Neon Transfection System | Invitrogen | MPK5000 | _ |
| NetStart 1.0 | _ | _ | https://services.healthtech.dtu.dk/services/NetStart-1.0/ |
| Nuclease-free Water | Synthego | _ | _ |
| PCR primer (EGFP) | Eurofins | _ | Fw : GGAATGCAAGGTCTGTTGAATG ; Rev : CACCTTGATGCCGTTCTTCT |
| PCR primer (SAMHD1) | Eurofins | _ | Fw : CGGGATTGATTTGAGGACGA ; Rev : GGGTGGCAAGTTAGTGAAGA |
| Penicillin-streptomycin (10,000 U/mL) | Gibco | 15140122 | _ |
| PFA | Electron Microscopy Sciences | 15714 | _ |
| PMA | Sigma-Aldrich | P8139 | _ |
| PrimerQuest | IDT | _ | https://eu.idtdna.com/pages/tools/primerquest |
| QIAquick PCR Purification Kit | Qiagen | 28104 | _ |
| QuickExtract DNA Extraction Solution | Biosearch Technologies | QE09050 | _ |
| RPMI 1640, GlutaMAX | Gibco | 61870010 | _ |
| SnapGene Viewer | Dotmatics | _ | Version 7 |
| SpCas9 2NLS Nuclease | Synthego | _ | _ |
| SYBR Safe DNA Gel Stain | Invitrogen | S33102 | _ |
| Synthetic sgRNA (EGFP) | Synthego | _ | #1 : CGCGCCGAGGUGAAGUUCGA ; #2 : UUCAAGUCCGCCAUGCCCGA ; #3 : CAACUACAAGACCCGCGCCG |
| Synthetic sgRNA (SAMHD1) | Synthego | _ | #1 : AUCGCAACGGGGACGCUUGG ; #2 : GCAGUCAAGAACCUCGGCGC ; #3 : CCAUCCCGACUACAAGACAU |
| Syringe Plastipak Luer Lock | BD | 301229 | _ |
| T100 Thermal Cycler | BIO-RAD | 1861096 | _ |
| T7 endonuclease I | New England Biolabs | M0302S | _ |
| TAE buffer UltraPure, 10x | Invitrogen | 15558026 | 400 mM Tris-Acetate, 10 mM EDTA |
| THP-1 cells | ATCC | TIB-202 | _ |
| Trypsin-EDTA (0,05 %) | Gibco | 25300054 | _ |
| ZE5 Cell Analyzer | BIO-RAD | 12014135 | _ |
Referanslar
- Tsuchiya, S., et al. Establishment and characterization of a human acute monocytic leukemia cell line (THP-1). Int J Cancer. 26 (2), 171-176 (1980).
- Danis, V. A., Millington, M., Hyland, V. J., Grennan, D. Cytokine production by normal human monocytes: inter-subject variation and relationship to an IL-1 receptor antagonist (IL-lRa) gene polymorphism. Clin Exp Immunol. 99, (1995).
- Bol, S. M., et al. Donor variation in in vitro HIV-1 susceptibility of monocyte-derived macrophages. Virology. 390 (2), 205-211 (2009).
- Appleby, L. J., et al. Sources of heterogeneity in human monocyte subsets. Immunol Lett. 152 (1), 32-41 (2013).
- O'Neill, M. B., et al. Single-cell and bulk RNA-sequencing reveal differences in monocyte susceptibility to Influenza A virus infection between Africans and Europeans. Front Immunol. 12, 768189 (2021).
- Wen, Y., et al. Comparability study of monocyte-derived dendritic cells, primary monocytes, and THP1 cells for innate immune responses. J Immunol Methods. 498, 113147 (2021).
- Inagaki, Y., et al. Interferon-g-induced apoptosis and activation of THP-1 macrophages. Life Sci. 71 (21), 2499-2508 (2002).
- Boonkaewwan, C., Toskulkao, C., Vongsakul, M. Anti-inflammatory and immunomodulatory activities of stevioside and its metabolite steviol on THP-1 cells. J Agric Food Chem. 54 (3), 785-789 (2006).
- Chanput, W., et al. β-Glucans are involved in immune-modulation of THP-1 macrophages. Mol Nutr Food Res. 56 (5), 822-833 (2012).
- Cui, J., et al. USP3 inhibits type I interferon signaling by deubiquitinating RIG-I-like receptors. Cell Res. 24 (4), 400-416 (2014).
- Chen, S., et al. SAMHD1 suppresses innate immune responses to viral infections and inflammatory stimuli by inhibiting the NF-κB and interferon pathways. Proc Natl Acad Sci USA. 115 (16), E3798-E3807 (2018).
- Pradhananga, S., Spalinskas, R., Poujade, F. A., Eriksson, P., Sahlén, P. Promoter anchored interaction landscape of THP-1 macrophages captures early immune response processes. Cell Immunol. 355, 104148 (2020).
- Mezzasoma, L., Talesa, V. N., Romani, R., Bellezza, I. Anp and BNP exert anti-inflammatory action via npr-1/cgmp axis by interfering with canonical, non-canonical, and alternative routes of inflammasome activation in human THP1 cells. Int J Mol Sci. 22 (1), 1-17 (2021).
- Martinat, C., et al. SUMOylation of SAMHD1 at Lysine 595 is required for HIV-1 restriction in non-cycling cells. Nat Commun. 12 (1), 4582 (2021).
- Rensen, E., et al. Clustering and reverse transcription of HIV-1 genomes in nuclear niches of macrophages. EMBO J. 40 (1), e105247 (2021).
- Ikeda, T., et al. APOBEC3 degradation is the primary function of HIV-1 Vif determining virion infectivity in the myeloid cell line THP-1. mBio. 14 (4), e0078223 (2023).
- Jinek, M., et al. A programmable dual RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337, 816-821 (2012).
- Kurosaki, T., Maquat, L. E. Nonsense-mediated mRNA decay in humans at a glance. J Cell Sci. 129 (3), 461-467 (2016).
- Tuladhar, R., et al. CRISPR-Cas9-based mutagenesis frequently provokes on-target mRNA misregulation. Nat Commun. 10 (1), 4056 (2019).
- Embree, C. M., Abu-Alhasan, R., Singh, G. Features and factors that dictate if terminating ribosomes cause or counteract nonsense-mediated mRNA decay. J Biol Chem. 298 (11), 102592 (2022).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Schnoor, M., et al. Efficient non-viral transfection of THP-1 cells. J Immunol Methods. 344 (2), 109-115 (2009).
- Tang, X., et al. A method for high transfection efficiency in THP-1 suspension cells without PMA treatment. Anal Biochem. 544, 93-97 (2018).
- Ji, W., Zhang, L., Liu, X. Protocol using lentivirus to establish THP-1 suspension cell lines for immunostaining and confocal microscopy. STAR Protoc. 4 (1), 102032 (2023).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, (2023).
- . Synthego Performance Analysis, ICE Analysis Available from: https://www.synthego.com/help/citing-ice (2019)
- Salamov, A. A., Nishikawa, T., Swindells, M. B. Assessing protein coding region integrity in cDNA sequencing projects. Bioinformatics. 14, 384-390 (1998).
- Pedersen, A. G., Nielsen, H. Neural network prediction of translation initiation sites in eukaryotes: Perspectives for EST and genome analysis. Proc Int Conf Intell Syst Mol Biol. 5, 226-233 (1997).
- Concordet, J. P., Haeussler, M. CRISPOR: Intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46 (W1), W242-W245 (2018).
- Laguette, N., et al. SAMHD1 is the dendritic- and myeloid-cell-specific HIV-1 restriction factor counteracted by Vpx. Nature. 474 (7353), 654-657 (2011).
- Hrecka, K., et al. Vpx relieves inhibition of HIV-1 infection of macrophages mediated by the SAMHD1 protein. Nature. 474 (7353), 658-661 (2011).
- Seki, A., Rutz, S. Optimized RNP transfection for highly efficient CRI SPR/Cas9-mediated gene knockout in primary T cells. J Exp Med. 215 (3), 985-997 (2018).
- Batista Napotnik, T., Polajžer, T., Miklavčič, D. Cell death due to electroporation - A review. Bioelectrochemistry. 141, 107871 (2021).
- Perretta-Tejedor, N., Freke, G., Seda, M., Long, D. A., Jenkins, D. Generating mutant renal cell lines using CRISPR technologies. Methods Mol Biol. 2067, 323-340 (2020).
- Sentmanat, M. F., Peters, S. T., Florian, C. P., Connelly, J. P., Pruett-Miller, S. M. A survey of validation strategies for CRISPR-Cas9 editing. Sci Rep. 8 (1), 888 (2018).
- Makino, S., Fukumura, R., Gondo, Y. Illegitimate translation causes unexpected gene expression from on-target out-of-frame alleles created by CRISPR-Cas9. Sci Rep. 6, 39608 (2016).
- Bowling, A., et al. Downstream alternate start site allows N-terminal nonsense variants to escape NMD and results in functional recovery by readthrough and modulator combination. J Pers Med. 12 (9), 1448 (2022).
- Inácios, &. #. 1. 9. 4. ;., et al. Nonsense mutations in close proximity to the initiation codon fail to trigger full nonsense-mediated mRNA decay. J Biol Chem. 279 (31), 32170-32180 (2004).
- Li, J., et al. Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9. J Mol Cell Biol. 7 (4), 284-298 (2015).
- Guo, Y., et al. CRISPR inversion of CTCF sites alters genome topology and enhancer/promoter function. Cell. 162 (4), 900-910 (2015).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır