Method Article
Elektroporationsbasierter CRISPR-Cas9-vermittelter Gen-Knockout in THP-1-Zellen und Einzelzell-Klonisolierung
In diesem Artikel
Zusammenfassung
Die THP-1-Zelllinie wird häufig als Modell verwendet, um die Funktionen menschlicher Monozyten/Makrophagen in verschiedenen biologiebezogenen Forschungsbereichen zu untersuchen. Dieser Artikel beschreibt ein Protokoll für effizientes CRISPR-Cas9-basiertes Engineering und Einzelzell-Klonisolierung, das die Produktion robuster und reproduzierbarer phänotypischer Daten ermöglicht.
Zusammenfassung
Die THP-1-Zelllinie der humanen akuten monozytären Leukämie (AML) wird häufig als Modell verwendet, um die Funktionen von humanen Monozyten-abgeleiteten Makrophagen zu untersuchen, einschließlich ihres Zusammenspiels mit bedeutenden humanen Krankheitserregern wie dem humanen Immundefizienzvirus (HIV). Im Vergleich zu anderen immortalisierten Zelllinien myeloischen Ursprungs behalten THP-1-Zellen viele intakte Entzündungssignalwege bei und weisen phänotypische Merkmale auf, die denen primärer Monozyten ähnlicher sind, einschließlich der Fähigkeit, sich bei Behandlung mit Phorbol-12-Myristat-13-acetat (PMA) in Makrophagen zu differenzieren. Der Einsatz der CRISPR-Cas9-Technologie zur Entwicklung von THP-1-Zellen durch gezielten Gen-Knockout (KO) bietet einen leistungsstarken Ansatz zur besseren Charakterisierung immunbezogener Mechanismen, einschließlich Virus-Wirt-Interaktionen. Dieser Artikel beschreibt ein Protokoll für effizientes CRISPR-Cas9-basiertes Engineering unter Verwendung von Elektroporation, um vorassemblierte Cas9:sgRNA-Ribonukleoproteine in den Zellkern zu bringen. Die Verwendung mehrerer sgRNAs, die auf denselben Locus an leicht unterschiedlichen Positionen abzielen, führt zur Deletion großer DNA-Fragmente, wodurch die Editierungseffizienz erhöht wird, wie mit dem T7-Endonuklease-I-Assay bewertet wurde. Die CRISPR-Cas9-vermittelte Editierung auf genetischer Ebene wurde durch Sanger-Sequenzierung mit anschließender Inference of CRISPR Edits (ICE)-Analyse validiert. Die Proteinverarmung wurde durch Immunblotting in Verbindung mit einem funktionellen Assay bestätigt. Mit diesem Protokoll wurden bis zu 100% Indels im Ziellocus und eine Abnahme der Proteinexpression um über 95% erreicht. Die hohe Editiereffizienz macht es bequem, einzellige Klone zu isolieren, indem die Verdünnung begrenzt wird.
Einleitung
THP-1 ist eine von humanen Monozyten abgeleitete Zelllinie, die von einem Patienten mit akuter Leukämie (AML) isoliert wurde und phänotypische Merkmale aufweist, die denen der primären Monozytensehr ähnlich sind 1. Im Vergleich zu primären Monozyten-abgeleiteten Makrophagen, die sich nicht teilen und sowohl eine begrenzte Lebensdauer als auch eine Variabilität des Phänotyps zwischen und innerhalb der Donoren aufweisen, können THP-1-Zellen praktisch unbegrenzt kultiviert werden und haben ein homogeneres Verhalten, das die Reproduzierbarkeit der Ergebnisse begünstigt 2,3,4,5,6 . Bemerkenswert ist, dass THP-1-Zellen in Richtung eines Makrophagen-ähnlichen Phänotyps mit Phorbol-12-Myristat-13-acetat (PMA) differenziert werden können, was sie zu einem weit verbreiteten In-vitro-Modell macht, um die Reaktionen von Monozyten/Makrophagen auf Entzündungssignale 7,8,9,10,11,12,13 oder eine Infektion durch klinisch relevante humane Krankheitserreger, einschließlich HIV 14, zu untersuchen,15,16. Die Möglichkeit, THP-1-Zellen gentechnisch zu verändern, ist in vielen biologischen Forschungsbereichen von Interesse.
Das CRISPR-assoziierte Protein 9 (CRISPR-Cas9) ist ein prokaryotisches adaptives Immunsystem, das auf RNA-gesteuerte Nuklease zurückgreift, um eindringende virale Genome abzubauen, das als gentechnisches Werkzeug umprogrammiert wurde17. Der Prozess der Genom-Editierung verläuft in drei Schritten: Erkennen, Spalten und Reparieren. Eine Single-Guide-RNA (sgRNA) rekrutiert die Cas9-Nuklease durch Basenpaarung mit ihrer 20-bp-Guide-Sequenz an einen spezifischen genomischen Locus. Das Vorhandensein einer Protospacer Adjacent Motif (PAM)-Sequenz direkt 3' der genomischen 20-bp-Zielsequenz löst die Cas9-vermittelte Abwicklung und Spaltung an beiden DNA-Strängen zwischen den Positionen 17 und 18 (3-bp 5' der PAM) aus. Der resultierende Doppelstrangbruch (DSB) wird über zwei Hauptreparaturwege verarbeitet. In Ermangelung einer Reparaturschablone, die eine Homologie mit dem beschädigten Locus aufweist, führt der fehleranfällige Non-Homologus End Joining (NHEJ)-Weg zufällige Nukleotidinsertionen und/oder -deletionen (Indels) ein, was möglicherweise zu Frameshift-Mutationen und/oder der Einführung von Codons für vorzeitige Termination (PTC) führt. Im Gegenzug werden PTC-haltige mRNAs durch den Nonsense-vermittelten mRNA-Zerfallsweg (NMD) abgebaut, wodurch letztendlich die Proteinexpression/-funktion gestört wird 18,19,20. Alternativ kann der vorlagenabhängige HDR-Pfad (Homology-Directed Repair) den DSB betreiben und originalgetreu reparieren. Dieser Mechanismus wurde genutzt, um eine präzise Geneditierung zu erreichen, einschließlich Knock-Ins und Basensubstitutionen. Es ist erwähnenswert, dass der Zellzyklusstatus ein wichtiger Faktor ist, der die Wahl des DSB-Reparaturwegs beeinflusst. Tatsächlich ist NHEJ in allen Stadien des Zellzyklus aktiv, während HDR hauptsächlich auf die S/G2-Phasen beschränkt ist21.
THP-1-Zellen wachsen in Suspension und sind notorisch schwer mit Plasmid-DNA zu transfizieren, ein Verfahren, das möglicherweise auch ihre Lebensfähigkeit und/oder Differenzierungsfähigkeit verändert22,23. Die Transduktion mit HIV-1-basierten lentiviralen Vektoren, die sowohl für Cas9 als auch für die sgRNA kodieren, wird häufig eingesetzt, um ein Gen von Interesse auszuschalten (KO)24. Die Integration der Cas9/sgRNA-Kassette in das zelluläre Genom gewährleistet eine verlängerte Expression und eine effiziente KO, ist aber auch eine anhaltende Quelle für Off-Target-Effekte25. Alternativ werden die vorassemblierten Cas9:sgRNA-Ribonukleoproteine (RNPs) durch Elektroporation abgegeben, eine Methode, bei der durch Anlegen elektrischer Impulse vorübergehend Poren sowohl in der Plasma- als auch in der Kernmembran gebildet werden. Die Erhaltung der Lebensfähigkeit der Zellen ist eine wichtige Herausforderung bei diesem Ansatz.
In dieser Arbeit wurde eine THP-1-Zelllinie hergestellt, die GFP (THP-1_GFP) stabil exprimiert, um als Werkzeug zur Etablierung eines Protokolls für eine effiziente CRISPR-Cas9-basierte Editierung zu dienen. Nach der Entwicklung einer Strategie zur Inaktivierung des EGFP-Gens mit drei sgRNAs gleichzeitig (Multi-Guide-Ansatz) wurde die KO-Effizienz unter verschiedenen Elektroporationsbedingungen unter Verwendung der GFP-Expression als Auslesung bestimmt. Parallel dazu wurde die Zellproliferation überwacht. Die Geneditierung wurde sowohl durch einen T7-Endonuklease I (T7EI)-Assay als auch durch eine Sanger-Sequenzierung bestätigt, gefolgt von einer Analyse mit dem Inference of CRISPR Edits (ICE)-Algorithmus26. Parameter, die zu einer Verringerung der GFP-Expression um bis zu 95 % führten, wobei THP-1-Zellen nach der Elektroporation normale Wachstumsraten wiedererlangten, wurden erfolgreich eingesetzt, um ein endogenes Gen (SAMHD1) zu inaktivieren und einzellige THP-1-Klone zu produzieren.
Protokoll
Die Einzelheiten zu den Reagenzien und der Ausrüstung, die in dieser Studie verwendet wurden, sind in der Materialtabelle aufgeführt.
1. Führungsaufbau mit CRISPOR (Abbildung 1.1)
HINWEIS: Die SnapGene Viewer-Software kann in den Schritten 4, 7 und 10 verwendet werden, um die Editierungszielstelle und die Position der PCR-Primer-Hybridisierung innerhalb des interessierenden Gens zu annotieren.
- Rufen Sie die Ensembl-Website auf (www.ensembl.org). Wählen Sie im Suchfeld eine Spezies aus und geben Sie den Namen des interessierenden Gens ein. Klicken Sie auf Los. Wählen Sie das Ergebnis aus, das dem Gen entspricht (kein Transkript).
- Klicken Sie auf Transkripttabelle anzeigen und wählen Sie dann die Transkript-ID aus, die der goldmarkierten Proteincodierung in der Spalte Biotyp entspricht. Sobald Sie sich auf der Transkriptseite befinden, klicken Sie im linken Menü auf Exons .
- Scrollen Sie nach unten und klicken Sie auf Sequenz herunterladen. Stellen Sie sicher, dass das Dateiformat FACHA ist. Deaktivieren Sie unter Einstellungen - Eingeschlossene Sequenzen alles außer der genomischen Sequenz.
Geben Sie die Zahl "500" in Flankierende Sequenz an einem der Enden des Transkriptfelds ein. Klicken Sie auf Vorschau, wählen Sie die gesamte Sequenz aus (nur die Nukleotide ohne Header) und kopieren Sie sie (Strg+C). - Öffnen Sie SnapGene Viewer und klicken Sie auf Neu > DNA-Datei. Fügen Sie die Sequenz (Strg+V) in das Feld Sequenz erstellen ein. Deaktivieren Sie Allgemeine Features erkennen, und wählen Sie In Topologie die Option Linear aus.
Benennen Sie die Datei um und klicken Sie auf Erstellen. Deaktivieren Sie im linken Menü die Option Enzyme anzeigen (erstes Symbol). Wählen Sie am unteren Rand des Fensters die Registerkarte Sequenz aus.
HINWEIS: Dieser Schritt ermöglicht den Abruf der gesamten Gensequenz, einschließlich Exons, Introns und flankierenden Sequenzen mit 500 bp (optional). Diese letztgenannte Information ist nützlich für die Entwicklung von PCR-Primern für die Amplifikation einer Zielstelle, die sich innerhalb des ersten Exons befindet. - Scrollen Sie auf der Ensembl-Website in der Dateivorschau nach oben und klicken Sie auf Zurück. Ändern Sie nun das Dateiformat in RTF. Deaktivieren Sie unter Einstellungen - Eingeschlossene Sequenzen alles außer Exons. Wählen Sie unter Varianten anzeigen die Option Nein aus. Klicken Sie oben auf der Seite auf Download .
- Öffnen Sie die heruntergeladene Datei (mit Word), die die Sequenz der Exons enthält und die Codierungssequenz in Blau anzeigt, beginnend mit dem anfänglichen ATG. Wählen Sie das Exon aus, auf das die CRISPR-Cas9-gerichtete Bearbeitung abzielen soll (Empfehlungen siehe unten), wählen Sie es aus und kopieren Sie es (Strg+C).
- Das anvisierte Exon ist entweder ein frühes Exon oder ein Exon, das für eine funktionell wichtige Domäne des Proteins kodiert. Es ist erwähnenswert, dass die Etablierung eines PTC in einem späten Exon in der Nähe der 3'-UTR wahrscheinlich nicht in der Lage sein wird, NMD auszulösen, was zur Expression eines C-terminalen verkürzten Proteins führt. Umgekehrt ist die Einführung eines PTC in einem frühen Exon proximal der nativen Initiationsstelle mit dem Risiko einer illegitimen Translation (ITL, auch bekannt als alternative Translationsinitiation (ATI)) verbunden, die zur unerwarteten Expression eines N-terminalen verkürzten Proteins führt, das an einer In-Frame-Translationsinitiationsstelle (TIS) stromabwärts des ersten ATG-Codons beginnt. Um dieses letztgenannte Risiko zu mindern, wird empfohlen, das Auftreten alternativer TIS mit ATGpr27 (atgpr.dbcls.jp) und/oder NetStart 1.028 (services.healthtech.dtu.dk/services/NetStart-1.0/) zu bewerten.
- Stellen Sie sicher, dass das Ziel-Exon eine Codierungssequenz enthält. Es könnte sich jedoch als nützlich erweisen, ein sgRNA-Annealing mit der 5'UTR-Region stromaufwärts des ursprünglichen ATG zu wählen, um es in das deletierte Fragment aufzunehmen.
- Im Idealfall sollte das zielgerichtete Exon allen proteinkodierenden Transkriptvarianten des Gens gemeinsam sein. Überprüfen Sie, ob dies auf dem Genome Data Viewer (www.ncbi.nlm.nih.gov/gdv/) des NCBI der Fall ist, indem Sie nach dem interessierenden Gen suchen.
Ein Klick auf den Gennamen (in grün) im Anzeigefenster zeigt die Transkriptvarianten (in lila) an. Exons werden durch ein Rechteck dargestellt.
- Drücken Sie in SnapGene Viewer STRG+F, STRG+V und dann die EINGABETASTE , um nach der Sequenz des Exons zu suchen. Drücken Sie Strg+T , um eine neue Funktion hinzuzufügen, benennen Sie sie, ändern Sie den Typ in "exon" und klicken Sie auf OK.
- Gehen Sie auf die CRISPOR-Website (http://crispor.gi.ucsc.edu/) und fügen Sie die Exon-Sequenz in Schritt 1 ein. Wählen Sie zunächst in Schritt 2 ein Referenzgenom aus und dann in Schritt 3 den Typ des PAM, auf den abgezielt werden soll, in der Regel 20bp-NGG für SpCas9. Klicken Sie auf SENDEN.
- Wählen Sie zwei sgRNAs aus, so dass sie bis zu 150 bp voneinander entfernt sind, und wählen Sie dann eine dritte sgRNA dazwischen aus. Hier sind einige Richtlinien für die sgRNA-Auswahl:
- Der MIT-Spezifitätswert hängt mit Off-Target-Effekten zusammen. Eine höhere Punktzahl weist auf weniger potenzielle Off-Targets hin. In der rechten Spalte werden drei vorhergesagte Off-Target-Stellen angezeigt, die von der wahrscheinlichsten bis zur unwahrscheinlichsten eingestuft sind, zusammen mit ihren Positionen (in einem Exon, einem Intron oder einer intergenen Region). Die vollständige Liste der vorhergesagten Off-Target-Websites kann durch Klicken auf Alle anzeigen aufgerufen werden. Wenn möglich, wählen Sie sgRNAs mit einem MIT-Score >80 aus und priorisieren Sie diejenigen ohne Off-Targets für 0, 1 oder 2 Diskrepanzen. Darüber hinaus sollten sgRNAs mit Off-Targets in einem Exon, die das größte Potenzial zur Beeinflussung des Phänotyps haben, vermieden werden.
- Für die vorhergesagte Wirksamkeit beziehen Sie sich auf den Doench '16 Score. Beachten Sie, dass ein hoher Doench '16-Score lediglich darauf hinweist, dass die sgRNA mit größerer Wahrscheinlichkeit wirksam ist. Die tatsächliche Wirksamkeit muss experimentell ermittelt werden. Aus diesem Grund ist es immer sinnvoll, mehrere sgRNAs auszuwählen, auch wenn sie nicht für die gemeinsame Verwendung gedacht sind.
- Ein entweder zu hoher oder zu niedriger GC-Gehalt sowie bestimmte Motive können sich nachteilig auf die sgRNA-Effizienz auswirken und sollten vermieden werden. Diese Parameter werden von CRISPOR hervorgehoben.
- Wiederholen Sie Schritt 1.7, um die sgRNA-Sequenz und die zugehörige PAM-Sequenz zur Gensequenz im SnapGene Viewer hinzuzufügen. Fügen Sie gleichzeitig die sgRNA-Sequenz (ohne PAM) in eine Text- oder Excel-Datei ein, um die bei der Bestellung des Oligonukleotids erforderliche 5'-3'-Ausrichtung beizubehalten.
- Für die PCR-basierte Amplifikation der Zielstelle entwerfen Sie einige Primer, die sie umgeben. Die Amplikongröße sollte zwischen 800 und 1000 bp liegen. PrimerQuest wurde verwendet, um die Primer für dieses Protokoll zu entwerfen (https://eu.idtdna.com/pages/tools/primerquest). Alternativ bietet CRISPOR eine Liste von Primern zur Amplifikation der genomischen Zielregion sowie der Off-Target-Stellen. Nachdem Sie die vollständige Liste der potenziellen Off-Target-Sites (Schritt 1.9.1) angezeigt haben, klicken Sie auf Off-Target-Primer in der unteren rechten Ecke.
2. Reagenz- und Zellvorbereitung für die Elektroporation (Abbildung 1.2)
- Bereiten Sie eine 24-Well-Kulturplatte vor, um Zellen nach der Elektroporation zu gewinnen, indem Sie die Wells mit 500 μl RPMI 1640-Medium füllen, das mit 20 % hitzeinaktiviertem (56 °C, 30 min), gefiltertem (0,20 μm) fötalem Rinderserum (FBS) ergänzt wird. Fügen Sie keine Antibiotika hinzu. Bewahren Sie die Platte in einem befeuchteten Inkubator bei 37 °C und 5 % CO2 24 h bis zur Elektroporation auf.
- Wenn die sgRNAs trocken versendet werden, rehydrieren Sie sie mit TE-Puffer (10 mM Tris, 1 mM EDTA, pH 8,0) auf eine Endkonzentration von 100 μM (d. h. 10 μl TE-Puffer pro 1 nmol sgRNA). 30 s lang vortexen, über Nacht bei 4 °C inkubieren, um eine vollständige Rehydrierung zu ermöglichen, und nach einer kurzen Pipettenhomogenisierung die sgRNA-Stammlösung bei -20 °C lagern. Je nach Endvolumen sind Aliquote zu erstellen, um mehrfache Gefrier-Tau-Zyklen zu vermeiden.
- Bereiten Sie eine Arbeits-sgRNA-Lösung mit einer Endkonzentration von 30 μM her, indem Sie die 100 μM-Stammlösung in nukleasefreiem Wasser verdünnen. 30 s vortexen und 5 min bei Raumtemperatur inkubieren.
- Assemblieren Sie die Cas9:sgRNA-RNPs in einem molaren Verhältnis von 1:9, indem Sie gleichzeitig 1 μl jeder der drei 30 μM sgRNAs und 0,5 μl 20 μM Cas9-Lösungen in 3,5 μl Resuspensionspuffer R verdünnen, der im Elektroporationskit enthalten ist (endgültiges Volumen von 7 μl, für eine Versuchsbedingung; entsprechend skalieren). Kurz vortexen und 5 min bei Raumtemperatur inkubieren.
- In der Zwischenzeit wird eine uneditierte Kontrolle hergestellt, indem 0,5 μl 20 μM Cas9 zu 6,5 μl Resuspensionspuffer R. Vortex gegeben und 5 min bei Raumtemperatur inkubiert werden.
- Geben Sie 5 μl Resuspensionspuffer R zu allen Proben, um ein Endvolumen von 12 μl pro Elektroporationsbedingung zu erhalten.
- Bereiten Sie die THP-1-Zellen für die Elektroporation vor.
- Zur Beurteilung der Konzentration und Lebensfähigkeit durch den Trypanblau-Ausschlusstest. Verdünnen Sie die Zellen 1:2 in einer 0,4%igen Trypanblau-Färbelösung. Nach 30 s Inkubation gut homogenisieren und 10 μl in eine Wand einer Zählkammer mit einem von Neubauer verbesserten Gitterstil geben. Zählen Sie drei große Quadrate und teilen Sie die Zählung durch 100, um die Zellkonzentration (x106 Zellen/ml) zu erhalten.
HINWEIS: Die Gesundheit der Zellen beeinflusst ihre Empfindlichkeit gegenüber Elektroporation. Es sollte darauf geachtet werden, dass die Zellkultur unter optimalen Bedingungen durchgeführt wird. Die Zeit außerhalb des Inkubators muss begrenzt werden, und alle Reagenzien und Lösungen sollten im Voraus vorbereitet und aufgewärmt werden. - Für jede Bedingung ein Volumen entnehmen, das 0,2 x 106 Zellen entspricht, und zentrifugieren (336 x g, 5 min, 20 °C).
- Der Überstand wird mit einer Pipette aspiriert und das Pellet in 500 μl PBS resuspendiert. Erneut zentrifugieren (336 x g, 5 min, 20 °C).
- Der Überstand wird vorsichtig mit einer Pipette aspiriert und das THP-1-Zellpellet mit den 12 μl RNP-Lösung resuspendiert (Schritt 2.6).
- Zur Beurteilung der Konzentration und Lebensfähigkeit durch den Trypanblau-Ausschlusstest. Verdünnen Sie die Zellen 1:2 in einer 0,4%igen Trypanblau-Färbelösung. Nach 30 s Inkubation gut homogenisieren und 10 μl in eine Wand einer Zählkammer mit einem von Neubauer verbesserten Gitterstil geben. Zählen Sie drei große Quadrate und teilen Sie die Zählung durch 100, um die Zellkonzentration (x106 Zellen/ml) zu erhalten.
3. Aufbau des Elektroporationssystems und Nukleofektion (Abbildung 1.3)
- Stellen Sie die Pipettenstation unter eine Biosicherheitswerkstatt, setzen Sie einen Elektroporationsschlauch in die Halterung ein und fügen Sie 3 ml Puffer E hinzu, der im Elektroporationskit enthalten ist.
- Stellen Sie nach dem Einschalten des Elektroporationsgeräts über den Touchscreen die folgenden Elektroporationsparameter ein: Spannung = 1 500 V, Dauer = 10 ms, Zahl = 3.
- Die Elektroporationspipette mit einer Spitze ausstatten und 10 μl der RNP/THP-1-Lösung aspirieren (Schritt 2.7.4). Führen Sie die Pipette in den Elektroporationsschlauch ein und drücken Sie auf dem Bildschirm des Elektroporationsgeräts auf Start . Warten Sie, bis die Meldung Abgeschlossen angezeigt wird, und entfernen Sie die Pipette aus dem Röhrchen.
- Übertragen Sie die Zellen in eine Vertiefung der vorgeheizten 24-Well-Platte und homogenisieren Sie sie vorsichtig. Stellen Sie die Platte wieder in den befeuchteten Brutschrank und lassen Sie sie 72 h ungestört ruhen.
HINWEIS: Achten Sie darauf, beim Pipettieren der RNP/THP-1-Suspension keine Blasen zu bilden, da diese den Elektroporationsvorgang beeinträchtigen. Wenn eine Fehlermeldung angezeigt wird, weil kein Lichtbogen sichtbar ist, entfernen Sie die Elektroporationspipette aus dem Röhrchen und drücken Sie erneut Start . Wenn jedoch ein Lichtbogen in Form eines kurzen hellen Funkens beobachtet wird, kann dies auf das Vorhandensein von Blasen hinweisen. Das Elektroporationsverfahren wird wahrscheinlich fehlschlagen, auch wenn keine Fehlermeldung angezeigt wird.
4. THP-1-Wiederfindung 72 h nach der Elektroporation (Abbildung 1.4)
- Zählen Sie die Zellen zur Bestimmung der Konzentration (Schritt 2.7.1) 72 h nach der Elektroporation.
- Wenn genügend Zellen vorhanden sind (d. h. ≥0,6 x 106 Zellen/ml), verdünnen Sie sie mindestens um den Faktor 2 mit RPMI, ergänzt mit 20 % FBS und 1 % Penicillin-Streptomycin, und bringen Sie die Konzentration zwischen 0,3 und 0,5 x 106 Zellen pro ml. Andernfalls lassen Sie die Zelle weitere 72 Stunden erholen.
- Passage und Amplifikation der Zellen, bis genügend Zellen für die KO-Validierung vorhanden sind. In der Zwischenzeit kann die Isolierung von einzelligen Klonen eingeleitet werden (Schritt 7).
5. Validierung der Geneditierung durch T7EI-Mismatch-Assay (Abbildung 1.5)
HINWEIS: Der Assay könnte die Editierungseffizienz unterschätzen, da T7EI Fehlanpassungen von mehr als 1 bp erkennt. Daher ist der T7EI-Assay nicht für das Screening homozygoter Zellpopulationen (d. h. Einzelzellklone) geeignet, es sei denn, er wird entsprechend modifiziert (Schritt 5.7).
- Bestimmen Sie die Zellkonzentration (Schritt 2.7.1) und entnehmen Sie ein Volumen, das 0,1 x 106 Zellen in einem 1,5-ml-Röhrchen entspricht. Zentrifugieren (336 x g, 5 min, 20 °C), den Überstand aspirieren und das Pellet in 500 μl PBS resuspendieren. Erneut zentrifugieren und mit einer Pipette so viel Überstand wie möglich absaugen, ohne das Pellet zu stören. Die Probe wird eingefroren und bei -20 °C gelagert.
- Extrahieren Sie die genomische DNA, um sie als Matrix für die PCR-Amplifikation zu verwenden.
- Resuspendieren Sie das Pellet mit 50 μl DNA-Extraktionslösung, homogenisieren Sie es und übertragen Sie das gesamte Volumen in ein 0,2-ml-PCR-Röhrchen. Vortexen und kurz zentrifugieren (3 s pulsieren).
- Legen Sie das Rohr in einen Thermocycler und erhitzen Sie es 15 Minuten lang auf 65 °C, gefolgt von 98 °C für 10 Minuten.
- Verdünnen Sie die extrahierte DNA mit 90 μl Reinstwasser. Vortexen und kurz zentrifugieren (5.000 x g, 3 s).
- Verdünnen Sie den PCR-Primer (siehe Materialtabelle) in Reinstwasser auf eine Endkonzentration von 10 μM (d. h. 10 pmol/μL).
- Bereiten Sie die PCR-Mischung (gemäß der nachstehenden Anmerkung) in einem 0,2-ml-PCR-Röhrchen vor (Endvolumen = 50 μl, für eine Bedingung). Normalerweise gibt es mindestens zwei Bedingungen: das KO und das unbearbeitete Steuerelement nur mit Cas9.
HINWEIS: Gereinigte genomische DNA: 10 μl; Vorwärts- und Rückwärtsprimer (10 μM): je 2,5 μL, die Endkonzentration beträgt 500 nM; Reaktionspuffer (5x): 10 μL. Vor der Zugabe gut vortexen. Mischen Sie dNTP (je 25 mM): 0,6 μl, Endkonzentration von 0,3 mM für jedes dNTP. DNA-Polymerase: 0,5 μL; Reinstwasser: 23,9 μL. Vortex und kurz zentrifugieren (3 s pulsieren). - Legen Sie die Röhrchen in den Thermocycler und führen Sie das PCR-Programm mit den folgenden Einstellungen aus:
HINWEIS: Ein Zyklus bei 95 °C für 5 min, gefolgt von 30 Zyklen [98 °C für 20 s (Denaturierungsschritt), X °C für 15 s (Glühschritt), 72 °C für 45 s (Dehnungsschritt)], dann ein letzter Zyklus bei 72 °C für 2 min. Entfernen Sie am Ende der Amplifikation die Röhrchen, den Wirbel und zentrifugieren Sie kurz (Impuls für 3 s). Die Glühtemperatur (X) ist die Schmelztemperatur (Tm) der Primer minus 5 °C. - Für eine polyklonale editierte Population: In ein neues 0,2-ml-Röhrchen 17,5 μl des PCR-Amplikons und 2 μl NEBuffer 2 (10x) für ein Endvolumen von 19,5 μl geben. Vortex und kurz zentrifugieren (Puls für 3 s). Alternativ können zum Screening von Einzelzellklone 1:1 der PCR-Produkte sowohl aus den editierten als auch aus den uneditierten Kontrollzellen gemischt werden (Schritt 5.6) (Ergänzende Abbildung 1).
- Für die Heteroduplex-Bildung legen Sie die Rohre in den Thermocycler und führen das folgende Programm durch: einen Zyklus bei 95 °C für 10 min, einen mit einer Rampe von -2 °C/s von 95 auf 85 °C, einen mit einer Rampe von -0,3 °C/s von 85 auf 25 °C und einen abschließenden Kühlzyklus bei 10 °C in der WARTESCHLEIFE.
- Nach der Heteroduplex-Bildung werden 0,5 μl T7EI-Lösung in das Röhrchen gegeben. Bei 37 °C 30 min inkubieren.
- Bereiten Sie ein 1,2%iges Agarose-Gel vor:
- Wiegen Sie die Agarose in einer Glasflasche und fügen Sie das entsprechende Volumen von 1x TAE-Puffer (40 mM Tris-Acetat, 1 mM EDTA, pH 8,3) hinzu. Stellen Sie es in die Mikrowelle und achten Sie darauf, die Kappe nicht fest zu schrauben. Mehrmals erhitzen, bis sich die Agarosekristalle vollständig aufgelöst haben.
HINWEIS: Falls erforderlich, mischen Sie die Lösung. Lassen Sie es nicht kochen, da sonst das Volumen abnimmt und sich die Agarosekonzentration verändert. - Fügen Sie den DNA-Gel-Fleck mit 1/20.000 verdünnt hinzu und mischen Sie ihn gut.
- Gießen Sie die Agaroselösung in eine Form, fügen Sie einen Kamm hinzu und lassen Sie sie bei Raumtemperatur erstarren.
- Wiegen Sie die Agarose in einer Glasflasche und fügen Sie das entsprechende Volumen von 1x TAE-Puffer (40 mM Tris-Acetat, 1 mM EDTA, pH 8,3) hinzu. Stellen Sie es in die Mikrowelle und achten Sie darauf, die Kappe nicht fest zu schrauben. Mehrmals erhitzen, bis sich die Agarosekristalle vollständig aufgelöst haben.
- Bereiten Sie die Proben für die Gelelektrophorese vor, indem Sie 5 μl der T7EI-Aufschlussprodukte (Schritt 5.9) oder das unverdaute PCR-Produkt mit 5 μl Wasser und 2 μl 6x DNA-Beladungsfarbstoff mischen. Laden Sie die Proben- und Größenleiter und migrieren Sie 45 Minuten lang bei 80 V (die Zeit kann je nach erwarteter Größe der DNA-Fragmente angepasst werden). Sobald die Migration abgeschlossen ist, nehmen Sie ein Bild des Gels mit einem geeigneten Bildgebungssystem auf.
6. Validierung der Geneditierung durch Sanger-Sequenzierungsanalyse (Abbildung 1.6)
- Um die genetische Veränderung zu charakterisieren, reinigen Sie die PCR-Produkte (Schritt 5.4) und analysieren Sie nach der Sanger-Sequenzierung die Ergebnisse mit dem ICE-Tool (https://www.synthego.com/products/bioinformatics/crispr-analysis).
- Kontrollieren Sie, ob ein Protein trotz der Modifikationen hergestellt werden kann.
- Laden Sie die ICE-Ergebnisse herunter und öffnen Sie die contribs.txt Datei.
- Vergleichen Sie die bearbeiteten Sequenzen mit dem WT-Gegenstück. Kartieren Sie die Indels und stellen Sie sicher, dass sie in der gewünschten genomischen Region entstanden sind. Beurteilen Sie ihre Größe. Wenn die Indellänge kein Vielfaches von drei ist, kommt es zu einer Frameshift, und es wird möglicherweise ein PTC eingeführt. Die mutierte mRNA wird voraussichtlich einen NMD-vermittelten Abbau durchlaufen.
7. Isolierung eines Einzelzellklons durch Begrenzung der Verdünnung (Abbildung 1.7)
HINWEIS: Die Isolierung von einzelligen Klonen ist nicht obligatorisch. Wenn Sie sich jedoch dafür entscheiden, ist es wichtig, mehrere Klone zu charakterisieren und ihren Phänotyp mit der ursprünglichen polyklonalen Population zu vergleichen.
- Es wird eine Probe der Zellsuspension entnommen, sie zu 1/2 mit Kulturmedium verdünnt und die Konzentration bestimmt (Schritt 2.7.1). Die Verdünnung erhöht die Zählgenauigkeit.
- Führen Sie 2 bis 3 serielle Verdünnungsschritte durch, um eine Konzentration von 7 Zellen/ml zu erreichen. Geben Sie 100 μl der Zellsuspension pro Vertiefung in eine 96-Well-Platte mit rundem Boden ab (d. h. 0,7 Zellen pro Vertiefung). Lassen Sie die Zelle einige Stunden dekantieren, bevor Sie die Platten am Mikroskop betrachten, um Vertiefungen zu identifizieren, die eine Zelle enthalten.
- Überwachen Sie regelmäßig die Koloniebildung und das Wachstum und übertragen Sie die Zellen bei Bedarf in eine größere Kulturplatte oder einen größeren Kolben.
8. Funktionelle Charakterisierung von THP-1 KO SAMHD1 Zellen mittels HIV-1 Restriktionsassay
- THP-1 in einer 24-Well-Kulturplatte mit 0,25 x 106 Zellen pro Well in 300 μl RPMI, ergänzt mit 10 % FBS, 1 % Penicillin-Streptomycin (vollständiges RPMI) und mit 300 ng/ml PMA zur Differenzierung. Bewahren Sie die Platte 24 h lang in einem befeuchteten Inkubator (37 °C, 5 % CO2) auf.
- Ersetzen Sie das Medium durch ein PMA-freies RPMI-Vollmedium und stellen Sie die Platte für weitere 24 h wieder in den Inkubator.
- Saugen Sie mit einer Vakuumpumpe das gesamte Kulturmedium ab. 250 μl VSVg-pseudotypisierte HIV-1-GFP-Virus-Stammlösung (MOI = 0,5 IFU/Zelle) zugeben. Schließen Sie ein nicht infiziertes Steuerelement ein. Stellen Sie die Platte 2 Stunden lang bei 4 °C auf, um die Infektion zu synchronisieren.
- Waschen Sie die Zellen einmal mit RPMI. Geben Sie 500 μl vollständiges RPMI in jede Vertiefung und inkubieren Sie 48 Stunden lang unter Standardbedingungen (die Zeit kann angepasst werden).
- Nach der Entnahme des Nährmediums die Zellen einmal mit kaltem 1x PBS waschen. Geben Sie dann 100 μl 0,05 % Trypsin-EDTA in jede Vertiefung. Stellen Sie die Platte bei 37 °C in einen Inkubator, bis sich die Zellen vollständig gelöst haben (5 Minuten sollten ausreichen), bevor Sie 200 μl vollständiges RPMI hinzufügen, um das Enzym zu inaktivieren. Übertragen Sie 250 μl aus jeder Vertiefung in eine 96-Well-Platte mit rundem Boden (stellen Sie sicher, dass das Durchflusszytometer Kulturplatten akzeptiert).
- Die Platte zentrifugieren (757 x g, 3 min, Verzögerung = 6, 20 °C) und den Überstand mit einer Mehrkanalpipette ansaugen. Das Pellet mit 100 μl 4 % PFA resuspendieren und 10 min bei 4 °C inkubieren. Fügen Sie 100 μl kaltes 1X PBS hinzu.
- Analysieren Sie die Infektionsrate, dargestellt durch GFP-positive Zellen, mittels Durchflusszytometrie (siehe Ergänzende Abbildung 2 für eine vorgeschlagene Gating-Strategie).
Ergebnisse
Es wurde eine THP-1-Zelllinie erzeugt, die das GFP-Reporterprotein (THP-1_GFP) stabil exprimiert (Abbildung 2A) und als Werkzeug zur Etablierung eines Protokolls für eine effiziente CRISPR-Cas9-vermittelte Genedition verwendet. Zu diesem Zweck wurden mit dem CRISPOR-Web-Tool29 (Abbildung 2B) 3 sgRNAs, die auf das EGFP-Gen abzielen, mit dem CRISPOR-Web-Tool 29 (Abbildung 2B) entwickelt, die gleichzeitig mit Cas9 in einem molaren Verhältnis von 9:1 komplexiert wurden, um RNPs zu bilden, bevor sie durch Elektroporation unter verschiedenen Einstellungen in die Zellen abgegeben wurden. Als nächstes wurden die Zellen entweder in 20 % FBS-haltigem RPMI-Medium allein gezüchtet (Abbildung 2C, oberes Bild) oder 1:1 mit konditioniertem Medium verdünnt (Abbildung 2C, unteres Bild). Die Zellproliferation und die GFP-Expression, als Auslese der EGFP-KO-Effizienz, wurden über die Zeit überwacht. Bei mehreren Erkrankungen ging der Anteil der GFP-positiven Zellen stark zurück und erreichte am 7. Tag nach der Elektroporation (PE) eine Reduktion von >90 %. An Tag 3 hatte sich die Anzahl der Zellen halbiert, was wahrscheinlich auf die negativen Auswirkungen der Elektroporation auf die Lebensfähigkeit der Zellen zurückzuführen ist. Die Zellzahl stieg jedoch wieder an, und die Verdopplungszeit kehrte an Tag 7 pe zu einer normalen Rate zurück (Abbildung 2C, oberes Bild). Die Verwendung von konditionierten Medien begünstigte die Zellrückgewinnung unter unseren experimentellen Bedingungen nicht (Abbildung 2C, unteres Bild). Basierend auf diesen Ergebnissen wurden drei 10 ms Pulse von 1500 V für Folgeexperimente ausgewählt.
Anschließend wurde der CRISPR-Cas9-vermittelte EGFP KO auf genomischer Ebene charakterisiert. Zu diesem Zweck wurde die genomische DNA von THP-1_GFP-Zellen extrahiert, entweder mit den Cas9:sgRNA-RNPs (editiert) oder Cas9 allein (uneditiert) elektroporiert und als Template für die PCR-Amplifikation einer 891 bp-Region verwendet, die die Zielstelle enthält. Elterliche THP-1-Zellen ohne EGFP wurden ebenfalls als Negativkontrolle eingeschlossen. Als nächstes wurde das PCR-Produkt von editierten THP-1_GFP-Zellen für den Mismatch-Nachweis durch den T7EI-Assay vorbereitet, gefolgt von der Trennung der DNA-Fragmente durch Agarose-Gelelektrophorese. Eine ~900 bp-Bande, die der erwarteten Größe des WT-Amplikons entspricht, konnte für uneditierte THP-1_GFP-Zellen gut sichtbar gemacht werden, nicht jedoch für die elterliche THP-1-Kontrolle (Abbildung 2D, vergleiche Zeilen 2 und 3). Im Fall der editierten THP-1_GFP-Zellen wurde die ~900 bp-Bande nicht mehr nachweisbar und durch eine schneller migrierende Bande ersetzt, deren Größe dem Verlust eines ~75 bp-Fragments infolge der CRISPR-Editierung entsprach (Abbildung 2D, Zeile 4). Dieses Bandenmuster wurde beim T7EI-Verdau verändert, was zum Auftreten kleinerer DNA-Fragmente führte, was die erfolgreiche Editierung der Zielstelle weiter bestätigt (Abbildung 2D, Zeile 5). Um einen Einblick in die Editierungseffizienz und die spezifischen Genotypen der editierten Population zu erhalten, wurde das PCR-Amplikon mit der Sanger-Methode sequenziert, gefolgt von einer Analyse mit dem ICE-Tool26. Die uneditierte Kontrollstichprobe wurde parallel verarbeitet. Wir erhielten Indel- und KO-Scores von 100%, was dem Prozentsatz der Nicht-WT-Sequenzen in der editierten Stichprobe bzw. dem Anteil der Indels, die wahrscheinlich zu einem funktionellen KO führen, entspricht (Abbildung 2E, links). Eine detaillierte Analyse des Indel-Größenspektrums und der Häufigkeit zeigte das Vorhandensein von zwei dominanten Populationen mit einer 62-bp- oder 76-bp-Deletion, die 57 % bzw. 37 % der Gesamtsequenzen ausmachten (Abbildung 2E, rechts). Diese Ergebnisse deuten darauf hin, dass sgRNA #1 in Kombination mit sgRNA #2 oder #3 gleichzeitig DSB im Zielgen auslöste (Abbildung 2B).
Dieses Protokoll wurde dann angewendet, um das endogene SAMHD1-kodierende Gen zu inaktivieren (Abbildung 3A). Die Charakterisierung der polyklonalen SAMHD1 KO THP-1-Zelllinie umfasste die Überwachung der Zellproliferation im Laufe der Zeit, die der von uneditierten THP-1-Zellen entsprach (Abbildung 3B). Das Ergebnis der Geneditierung wurde durch PCR-basierte Amplifikation der Zielsequenz bewertet, die eine kleinere Bande für die SAMHD1-KO-Zellen im Vergleich zur reinen Cas9-Kontrolle aufzeigte (Abbildung 3C, vergleiche Zeilen 2 und 4), was auf einen DNA-Sequenzverlust hinweist. Diese Beobachtungen wurden durch den T7EI-Assay bestätigt, der das Auftreten von Spaltprodukten für die SAMHD1 KO-Zellen, aber nicht für uneditierte Zellen zeigte (Abbildung 3C, vergleiche Zeilen 3 und 5). Die Analyse der Sanger-Sequenzierungsdaten mit dem ICE-Tool ergab Indel- und KO-Werte von 100 % bzw. 97 %. Die wichtigsten Modifikationen innerhalb des SAMHD1-Locus waren Sequenzdeletionen von 170 bp, 93 bp oder 104 bp in 46 %, 13 % bzw. 10 % der Sequenzen (Abbildung 3D). Die Proteinexpressionsniveaus im Rohzelllysat wurden ebenfalls durch Immunblotting bestimmt. Die ~70 kDa-Bande, die mit der Größe des endogenen SAMHD1 übereinstimmt, ist nach der Editierung praktisch nicht mehr nachweisbar, was einer geschätzten Reduktion von >97 % entspricht (Abbildung 3E). Schließlich wurde auch die phänotypische Charakterisierung der polyklonalen SAMHD1 KO THP-1 Zelllinien durch Testen der Anfälligkeit für eine HIV-1-Infektion untersucht. In Übereinstimmung mit seiner bekannten antiviralen Rolle30,31 ging die Inaktivierung des SAMHD1-Locus mit einer erhöhten HIV-1-Infektionsrate einher (Abbildung 3F).
Parallel dazu wurden einzellige Klone durch Begrenzung der Verdünnung erzeugt. Die Amplifikation der genomischen Zielstelle aus den SAMHD1 KO-Klonen führte zu PCR-Produkten mit erhöhter elektrophoretischer Mobilität im Vergleich zu denen von uneditierten Kontrollzellen (Abbildung 4A). Die ICE-basierte Analyse der Sanger-Sequenzierungsdaten ergab einen Indel-Score von 100% für alle ausgewählten Einzelzellklone bis auf einen (Klon A2) (Abbildung 4B, links). Die Charakterisierung der Editierungsergebnisse auf genomischer Ebene ergab eine einzelne Sequenz für Klon A3 mit einer 98-bp-Deletion, die sich 28 pb stromabwärts des ursprünglichen ATG befand, was zu einer Änderung außerhalb des Rahmens führte (Abbildung 4B, rechts). Die anderen Klone enthielten drei (Klon B1) oder mehr Sequenzen, was auf das Vorhandensein von Allelen hinweist, die unterschiedliche Editierungsereignisse durchlaufen haben und/oder mehr als eine Zelle pro Vertiefung beim begrenzenden Verdünnungsschritt durchlaufen haben (Abbildung 4B, rechts). Bemerkenswert ist, dass 11% der Sequenzen der Klone A4 und A7 Indels stromaufwärts des ursprünglichen ATG tragen, so dass die kodierende Sequenz unverändert bleibt. Darüber hinaus fehlte 1% der Klon-A7-Sequenzen ein 123-bp-Fragment 9-bp stromabwärts des ursprünglichen ATG, was zu einer In-Frame-Deletion von 41 Aminosäuren führte. In Übereinstimmung mit diesen Ergebnissen zeigten funktionelle Studien, dass Klone mit einem gestörten SAMHD1-Locus (A3, A8, B1, B3), wodurch die SAMHD1-Expression fehlte, eine hohe Permissive für eine Infektion mit HIV-1 aufwiesen (Abbildung 4C). Umgekehrt waren die Klone A4 und A7, bei denen SAMHD1 intakt ist, genauso refraktär wie elterliche oder uneditierte THP-1-Klone (Abbildung 4C). Zusammenfassend konnte die erfolgreiche Editierung des SAMHD1-Locus im Klon A3 bestätigt werden.
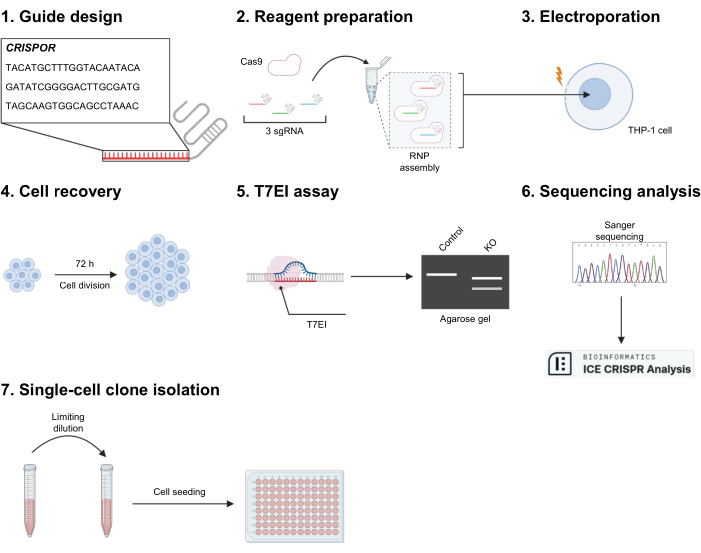
Abbildung 1: Experimenteller Abriss. (1) CRISPOR wird verwendet, um sgRNA-Sequenzen gegen das Zielexon zu erzeugen. Drei von ihnen werden nach hoher On-Target-Wirksamkeit und schwacher Off-Target-Vorhersage ausgewählt. (2) Die drei gRNAs werden mit Cas9 gemischt, um das Ribonukleoprotein (RNP) zusammenzusetzen, was zu einer Mischung aus drei verschiedenen RNPs führt. (3) Die Zellen werden elektroporiert, um den Eintritt der RNPs zu ermöglichen. (4) Die Zellen werden auf eine 24-Well-Kulturplatte überführt und mindestens 72 Stunden ruhen gelassen. (5) Die Geneditierung wird zunächst durch einen T7EI-Verdauungsassay qualitativ getestet. und (6) für die validierten Bedingungen, die durch Sanger-Sequenzierung und ICE-Analyse weiter charakterisiert werden. (7) Falls erforderlich, können Zellen nach Begrenzung der Verdünnung ausgesät werden, um klonale Populationen zu erzeugen. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
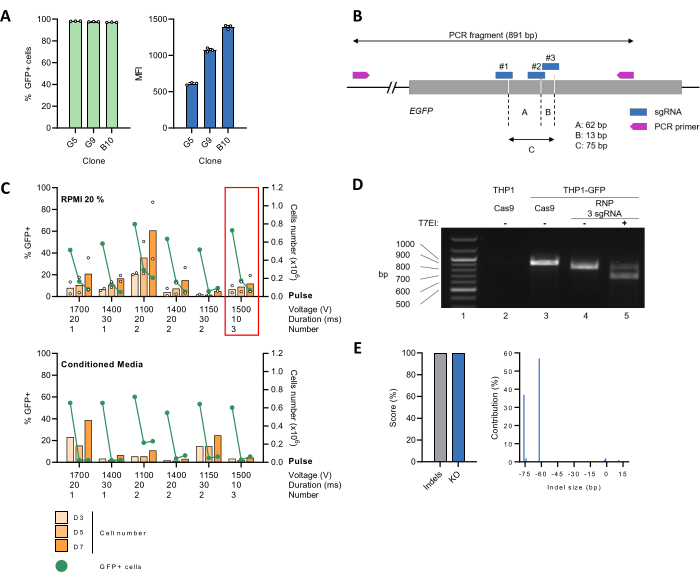
Abbildung 2: Einrichtung der Elektroporationseinstellungen für EGFP KO in THP-1-Zellen und Validierung der Geneditierung. (A) THP-1-Zellen wurden mit lentiviralen Vektoren transduziert, die für das EGFP-Reportergen kodieren. Einzelzellklone wurden durch Begrenzung der Verdünnung und anschließender Expansion in Kultur für 30 Tage erhalten. Die Analyse mittels Durchflusszytometrie bestätigte, dass praktisch die gesamte Zellpopulation GFP exprimierte. Die mediane Fluoreszenzintensität (MFI) der GFP-positiven Population, die auf die integrierte EGFP-Kopienzahl hinweist, variierte zwischen mehreren Zellklonen. Klon G5 wurde für Folgestudien ausgewählt. (B) Schematische Darstellung des EGFP-Gens , einschließlich der Bindungsstellen sowohl für sgRNA als auch für Primer. Die Cas9-Schnittstellen sind durch die gestrichelten Linien gekennzeichnet. (C) Undifferenzierte THP-1_GFP (2 x 10,5) Zellen wurden vor der Elektroporation mit den angegebenen Einstellungen mit vorassemblierten Cas9:sgRNA RNPs gemischt. Als nächstes wurden die Zellen in eine 24-Well-Platte mit 500 μl vorgewärmtem 20 % FBS-haltigem RPMI-Medium allein übertragen oder 1:1 mit konditioniertem Medium verdünnt, das durch eine 48-h-THP-1-Zellkultur hergestellt wurde. Die Proliferation (orangefarbene Balken) und der Prozentsatz der GFP-positiven Zellen (grüne Linie) wurden über die Zeit überwacht. Die Daten stellen den Mittelwert für n = 2 biologische Replikate dar. Der rote Rahmen markiert die Bedingung, die für nachfolgende Experimente ausgewählt wurde. (D) Die genomische DNA von editierten (RNP 3 sgRNA) oder uneditierten (Cas9) THP-1_GFP-Zellen wurde extrahiert und die Region um den Ziellocus mittels PCR amplifiziert. Als Negativkontrolle wurden elterliche THP-1-Zellen verwendet. Die Amplikons durchliefen einen Denaturierungs-/Renaturierungszyklus, der zur Bildung eines Heteroduplex führte. Als nächstes wurden Diskrepanzen mit dem T7EI-Assay nachgewiesen. DNA-Fragmente wurden durch 1,2% Agarose-Gelelektrophorese getrennt. (E) Aufgereinigte PCR-Produkte wurden nach der Sanger-Methode sequenziert, gefolgt von einer Analyse mit dem ICE-Algorithmus. Die Indel- und KO-Scores geben den Prozentsatz der Nicht-WT-Sequenzen in der Stichprobe und den Anteil der Zellen mit einem Frameshift bzw. 21+ bp Indel an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
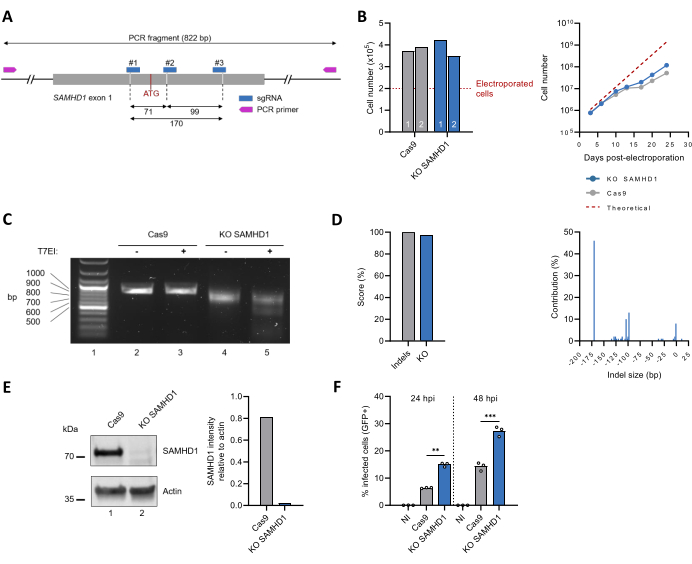
Abbildung 3: Herstellung und Charakterisierung einer polyklonalen SAMHD1 KO THP-1 Zellpopulation. (A) Schematische Darstellung des SAMHD1 Exons 1, einschließlich der Bindungsstellen für sgRNA und Primer. Die Cas9-Schnittstellen sind durch die gestrichelten Linien gekennzeichnet, und das anfängliche ATG-Codon ist rot dargestellt. (B) Drei Tage nach der Elektroporation wurden lebensfähige Zellen (n = 2) mit dem Trypanblau-Ausschlusstest quantifiziert (linkes Diagramm). Das Zellwachstum wurde über 25 Tage überwacht (rechtes Diagramm). Die gestrichelte Linie entspricht der theoretischen Zellzahl, die auf einer geschätzten Verdopplungszeit von 49 Stunden basiert. (C) Die Editierungsergebnisse auf genomischer Ebene wurden bewertet, wie in Abbildung 2D gezeigt. (D) Genotypen nach CRISPR-Editierung wurden durch ICE-basierte Analyse von Sanger-Sequenzen identifiziert und quantifiziert. (E) Das Protein, das im Rohlysat sowohl von editierten als auch von uneditierten THP-1-Zellen enthalten war, wurde durch Migration auf SDS-PAGE-Gel abgetrennt und anschließend durch Immunblotting mit Antikörpern gegen SAMHD1 sichtbar gemacht. Als Ladesteuerung wurde Aktin verwendet. Die Intensität der SAMHD1- und Aktin-Banden wurde durch Densitometrie mit der ImageJ-Software quantifiziert. (F) Unbearbeitete (Cas9) und KO SAMHD1 THP-1-Zellen wurden durch Behandlung mit PMA (300 ng/ml, 24 h) differenziert und anschließend mit einem VSVg-pseudotypisierten HIV-1-Virus, das GFP exprimiert, als Reporter herausgefordert. Der Anteil der GFP-positiven Zellen wurde mittels Durchflusszytometrie 24 h und 48 h nach der Infektion (hpi) bewertet. NI: nicht infiziert. **P < 0,01; P < 0,001 durch ungepaarten t-Test mit Welch-Korrektur. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.

Abbildung 4: Produktion und Charakterisierung von SAMHD1 KO Einzelzellklonen. (A) Das Editierungsergebnis für ausgewählte SAMHD1 KO Einzelzellklone wurde durch Überwachung der Mobilität der DNA-Fragmente bewertet, die durch PCR-Amplifikation der Region, die die Zielstelle enthält, mittels Agarosegelelektrophorese erzeugt wurden. (B) Die Sanger-Sequenzierungsdaten wurden mit dem ICE-Tool analysiert, wie in Abbildung 2E zu sehen. Die Balken in der rechten Grafik stellen die Vielfalt der Indels in jeder Zellpopulation dar. Der grüne Balken stellt die dominante Sequenz und die Gesamtgröße der zugehörigen Deletion dar. Zahlen in Klammern geben die Größe der nicht kontinuierlichen Lücken an. (C) Die Durchlässigkeit von uneditierten und SAMHD1-KO-Einzelzellklone für eine HIV-1-Infektion wurde wie in Abbildung 3F getestet und mit der von nicht elektroporierten elterlichen THP-1-Zellen oder uneditierten Zellklonen (Cas9) verglichen. Die Infektionsrate wurde mit 48 hpi bewertet. *P < 0,05; **P < 0,01; P < 0,001; P < 0,0001; ns P≥ 0,05 von Brown-Forsythe und Welch ANOVA mit Dunnetts Mehrfachvergleichstest. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung 1: Bewertung des Editierungsergebnisses für KO-Einzelzellklone mittels adaptiertem T7EI-Test. Die genomische DNA von KO-Einzelzellklonen oder uneditierten Zellen wurde aufgereinigt und die Region um den Ziellocus mittels PCR amplifiziert. Die Amplikons jedes Einzelzellklons wurden im Verhältnis 1:1 mit denen der uneditierten Kontrollzellen gemischt. Nach Heteroduplex-Bildung und T7EI-Aufschluss wurde das Produkt mittels Agarose-Gelelektrophorese analysiert. Die editierten Klone produzieren mehrere Verdauungsfragmente (Klone A, B, C, D und E), während die nicht editierten Klone eine einzelne WT-Bande aufweisen (Klone F und G). Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Abbildung 2: Durchflusszytometrische Gating-Strategie zur Quantifizierung des Prozentsatzes der HIV-1-infizierten PMA-behandelten THP-1-Zellen. Unter Verwendung der vorderen Streufläche (FSC-A) gegen die seitliche Streufläche (SSC-A) wird ein erstes Gate definiert, um Ablagerungen zu eliminieren und die PMA-behandelten THP-1-Zellen auszuwählen. Als nächstes werden einzelne Zellen mit einem SSC-A gegen ein SSC-H-Diagramm ausgewählt. Schließlich werden GFP-positive Zellen mit GFP-A gegen FSC-H nachgewiesen. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
In dieser Arbeit wird ein Protokoll beschrieben, um eine erfolgreiche CRISPR-vermittelte Editierung der THP-1-Zelllinie zu erhalten. Der Ansatz beruht auf dem Transfer von vorassemblierten sgRNA/Cas9 RNPs durch Elektroporation/Nukleofektion. Diese Strategie wurde gewählt, um die Off-Target-Effekte zu begrenzen, die möglicherweise bei der lentiviralen Integration der sgRNA/Cas9-Kassette auftreten und eine persistente Expression der Nuklease ermöglichen. Mehrere sgRNAs, die auf das interessierende Gen abzielen, wurden ausgewählt, um eine zuverlässige und effiziente Editierung zu erreichen, was die Wahrscheinlichkeit erhöht, genomische Indels zu erzeugen, was zu einem Verlust der Proteinexpression und des funktionellen KO32 führt. Besonderes Augenmerk wurde auf die Elektroporationseinstellungen gelegt, um eine optimale Permeabilisierung der Zellmembran zu erreichen, die erforderlich war, um eine effiziente Abgabe der sgRNA/Cas9-RNPs zu gewährleisten und gleichzeitig einen übermäßigen Zelltod zu verhindern33. Ein entscheidender Parameter ist in diesem Zusammenhang die Vitalität der Zellkultur vor der Elektroporation (d.h. niedrige Passagezahl).
Die CRISPR-Cas9-vermittelte Editierung wurde auf genetischer Ebene mit dem T7EI Mismatch Detection Assay validiert, der eine kostengünstige und schnelle Bewertung mehrerer Proben für die Vorselektion vor der Sanger-Sequenzierung ermöglicht. Diese Methode wurde ursprünglich entwickelt, um die Editierungseffizienz anhand einer einzelnen sgRNA abzuschätzen, indem die Intensität von Amplikons und Spaltprodukten in voller Länge verglichenwurde 34,35. Bemerkenswert ist, dass die großen Indels (>70 bp), die bei Verwendung mehrerer sgRNA erzeugt werden, durch einen Vergleich der Beweglichkeit der DNA-Fragmente nachgewiesen werden können, die durch PCR-Amplifikation der Zielloci aus editierten und uneditierten Zellen durch Standard-Gelelektrophorese erhalten wurden. Nichtsdestotrotz könnte der T7EI-Assay nützlich sein, um das Vorhandensein von Sequenzen zu bestimmen, die kleine Indels (>1bp) entweder in einer polyklonalen oder, unter Verwendung des angepassten Protokolls, in einer monoklonalen Zellpopulation enthalten.
Das Vorhandensein von On-Target-Out-of-Frame-Mutationen, die die kodierende Sequenz unterbrechen, wurde durch einen einfachen Ansatz bestätigt, bei dem das Genom der gesamten Zellpopulation extrahiert und als Matrix für die PCR-Amplifikation verwendet wird. Als nächstes werden die gereinigten Amplikons einer Sanger-Sequenzierung in großen Mengen unterzogen, gefolgt von einer bioinformatischen Analyse mit dem ICE-Tool. Insgesamt überwindet diese Methode die Notwendigkeit, die PCR-Produkte in ein Plasmid zu subklonen und die Ligationsprodukte in Bakterien umzuwandeln, um Einzelzellkolonien zu isolieren, die einzeln sequenziert werden.
Sobald die Inaktivierung des Ziellocus validiert ist, ist eine gründliche Charakterisierung der editierten Zelllinie erforderlich, um einen bestimmten Phänotyp mit Sicherheit auf das Fehlen des interessierenden Proteins zurückzuführen und unerwartete Effekte auszuschließen, die sich aus Off-Target-, aber auch On-Target-Veränderungen ergeben, die sich auf die mRNA-Struktur und/oder -Funktion auswirken könnten.
Das Fehlen einer Proteinexpression sollte durch Immunblotting und/oder Immunfluoreszenz unter Verwendung von möglicherweise zwei Antikörpern, die unterschiedliche Epitope erkennen, bestätigt werden. Tatsächlich ist die Einführung eines PTC in einem frühen Exon in der Nähe des kanonischen ATG-Codons mit dem Risiko von ITL-Ereignissen verbunden, die N-terminale verkürzte Proteinvarianten hervorbringen36. Bemerkenswert ist, dass Tuladhar et al. über den Nachweis aberranter Proteinspezies in einem signifikanten Anteil (~50%) von CRISPR-editierten Zellen kommerziellen oder hauseigenen Ursprungs berichteten 18,19. Das Vorkommen neuartiger Proteinprodukte wurde auf verschiedene und potenziell zelltypspezifische Prozesse zurückgeführt, wie z.B. das Überspringen des Indel-haltigen Exons oder das Auftauchen von internen Ribosomen-Eintrittsstellen (IRES), die die Initiierung der Proteinsynthese an alternativen TIS stromabwärts des kanonischen ATG-Codons verursachen. Es gibt auch Hinweise darauf, dass ITL unter bestimmten Umständen mit einer Flucht aus NMD gekoppelt ist37,38. In ähnlicher Weise könnten Genom-Editing-Strategien, die auf ein spätes Exon abzielen, die NMD der mutierten mRNA nicht auslösen, die einer ineffektiven Ablation der Genexpression zugrunde liegt20. Wenn Cas9 schließlich von mehreren sgRNAs gesteuert wird, können komplexe chromosomale Umlagerungen auftreten, einschließlich Inversionen oder Deletionen großer DNA-Fragmente39, die die Struktur und die Expression des Genoms verändern könnten, insbesondere wenn regulatorische Elemente angegriffen werden40.
Um die Auswirkungen unbeabsichtigter Genom-Editing-Ergebnisse weiter zu mildern, sind funktionelle Studien erforderlich, um den Zusammenhang zwischen dem Verlust der Proteinexpression und dem daraus resultierenden Phänotyp (d. h. der antiviralen Aktivität) zu bestätigen. Wenn editierte Zellen subkloniert sind, sollte das Verhalten mehrerer Klone untereinander und mit der polyklonalen Population verglichen werden. Die Durchführung von Rettungsexperimenten, die darin bestehen, die Genexpression wiederherzustellen, um den Phänotyp der editierten Zellen in einen WT-Phänotyp umzukehren, wird auch die Spezifität stärken und mögliche Off-Target-Ereignisse ausschließen.
Obwohl dieses Protokoll für die Editierung von THP-1-Zellen konzipiert wurde, bietet es eine allgemeine Struktur für den CRISPR-Cas9-KO-Workflow, die an andere Zelllinien angepasst werden kann. Es kann erforderlich sein, die Elektroporationsparameter anzupassen, und wenn eine Spannung über 1800 V angelegt wird, sollte gemäß den Anweisungen des Herstellers ein Resuspensionspuffer T verwendet werden. Eine weitere zu berücksichtigende Variable ist das Konzentrationsverhältnis zwischen Cas9 und sgRNA während der RNP-Assemblierung. Obwohl das Verhältnis 1:9 in diesem Fall gut funktioniert hat, kann eine Änderung die Bearbeitungseffizienz unter anderen Umständen verbessern. Schließlich sollte die Verwendung von konditionierten Medien zur Zellrückgewinnung nach der Elektroporation getestet werden, da die Wirkung bei verschiedenen Zelllinien unterschiedlich sein könnte.
Offenlegungen
Alle Autoren haben keine Interessenkonflikte.
Danksagungen
Wir danken JP Concordet (MNHN, U1154/UMR7196, Paris), G. Bossis (IGMM, Montpellier) und D. Schlüter (Medizinische Hochschule Hannover, Deutschland) für den Austausch von Protokollen und die Diskussion. Dieses Projekt wurde durch das Forschungs- und Innovationsprogramm Horizon 2020 der Europäischen Union (Grant Agreement No 101017572 to AZ) und ANRS (Grant ECTZ162721 to AZ) gefördert. Die Forschungsinfrastruktur Infectious Disease Model and Innovative Therapies (IDMIT) wird durch das "program investissement d'avenir (PIA)" unter der Referenz ANR_11_INSB_0008 unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filter | ClearLine | 146560 | _ |
| 0.4 % trypan blue | Beckman Coulter | 383200 | _ |
| 1.5 mL tube | Eppendorf | 3810X | _ |
| 24-well plate | Corning | 353047 | _ |
| 6x TriTrack DNA Loading Dye | Thermo scientific | R1161 | _ |
| 75 cm² Culture Flask Vented Cap | Corning | 353136 | _ |
| 8-Strip PCR Tubes with Caps | Life technologies | AM12230 | _ |
| 96-well plates Flat bottom | Corning | 353072 | _ |
| 96-well plates Round bottom | Corning | 353077 | _ |
| Agarose | Euromedex | D5 | _ |
| ATGpr | _ | _ | https://atgpr.dbcls.jp/ |
| ChemiDoc Imaging System | BIO-RAD | 12003153 | _ |
| Counting slide | NanoEntek | DHC-N04 | _ |
| CRISPOR | _ | _ | http://crispor.gi.ucsc.edu/ |
| DPBS | Gibco | 14190094 | _ |
| Ensembl | EMBL-EBI | _ | https://www.ensembl.org/index.html |
| Fetal Bovine Serum | Sigma-Aldrich | F7524 | _ |
| FlowJo | BD Life Sciences | v10.10 | _ |
| GeneRuler 100 bp Plus DNA Ladder | Thermo scientific | SM0323 | _ |
| Genome Data Viewer | NCBI | _ | https://www.ncbi.nlm.nih.gov/gdv/ |
| GraphPad Prism | Dotmatics | _ | Version 9.3.1 |
| Herculase II Fusion DNA Polymerases | Agilent | 600679 | _ |
| ICE CRISPR Analysis Tool | Synthego | _ | https://www.synthego.com/products/bioinformatics/crispr-analysis |
| Image Lab Touch | BIO-RAD | _ | Version 2.4.0.03 |
| NEBuffer 2 | New England Biolabs | B7002S | Included with T7EI M0302S |
| Neon Kit, 10 µL | Invitrogen | MPK1025K | Electroporation kit containing tips, tubes, buffer R and E |
| Neon Transfection System | Invitrogen | MPK5000 | _ |
| NetStart 1.0 | _ | _ | https://services.healthtech.dtu.dk/services/NetStart-1.0/ |
| Nuclease-free Water | Synthego | _ | _ |
| PCR primer (EGFP) | Eurofins | _ | Fw : GGAATGCAAGGTCTGTTGAATG ; Rev : CACCTTGATGCCGTTCTTCT |
| PCR primer (SAMHD1) | Eurofins | _ | Fw : CGGGATTGATTTGAGGACGA ; Rev : GGGTGGCAAGTTAGTGAAGA |
| Penicillin-streptomycin (10,000 U/mL) | Gibco | 15140122 | _ |
| PFA | Electron Microscopy Sciences | 15714 | _ |
| PMA | Sigma-Aldrich | P8139 | _ |
| PrimerQuest | IDT | _ | https://eu.idtdna.com/pages/tools/primerquest |
| QIAquick PCR Purification Kit | Qiagen | 28104 | _ |
| QuickExtract DNA Extraction Solution | Biosearch Technologies | QE09050 | _ |
| RPMI 1640, GlutaMAX | Gibco | 61870010 | _ |
| SnapGene Viewer | Dotmatics | _ | Version 7 |
| SpCas9 2NLS Nuclease | Synthego | _ | _ |
| SYBR Safe DNA Gel Stain | Invitrogen | S33102 | _ |
| Synthetic sgRNA (EGFP) | Synthego | _ | #1 : CGCGCCGAGGUGAAGUUCGA ; #2 : UUCAAGUCCGCCAUGCCCGA ; #3 : CAACUACAAGACCCGCGCCG |
| Synthetic sgRNA (SAMHD1) | Synthego | _ | #1 : AUCGCAACGGGGACGCUUGG ; #2 : GCAGUCAAGAACCUCGGCGC ; #3 : CCAUCCCGACUACAAGACAU |
| Syringe Plastipak Luer Lock | BD | 301229 | _ |
| T100 Thermal Cycler | BIO-RAD | 1861096 | _ |
| T7 endonuclease I | New England Biolabs | M0302S | _ |
| TAE buffer UltraPure, 10x | Invitrogen | 15558026 | 400 mM Tris-Acetate, 10 mM EDTA |
| THP-1 cells | ATCC | TIB-202 | _ |
| Trypsin-EDTA (0,05 %) | Gibco | 25300054 | _ |
| ZE5 Cell Analyzer | BIO-RAD | 12014135 | _ |
Referenzen
- Tsuchiya, S., et al. Establishment and characterization of a human acute monocytic leukemia cell line (THP-1). Int J Cancer. 26 (2), 171-176 (1980).
- Danis, V. A., Millington, M., Hyland, V. J., Grennan, D. Cytokine production by normal human monocytes: inter-subject variation and relationship to an IL-1 receptor antagonist (IL-lRa) gene polymorphism. Clin Exp Immunol. 99, (1995).
- Bol, S. M., et al. Donor variation in in vitro HIV-1 susceptibility of monocyte-derived macrophages. Virology. 390 (2), 205-211 (2009).
- Appleby, L. J., et al. Sources of heterogeneity in human monocyte subsets. Immunol Lett. 152 (1), 32-41 (2013).
- O'Neill, M. B., et al. Single-cell and bulk RNA-sequencing reveal differences in monocyte susceptibility to Influenza A virus infection between Africans and Europeans. Front Immunol. 12, 768189 (2021).
- Wen, Y., et al. Comparability study of monocyte-derived dendritic cells, primary monocytes, and THP1 cells for innate immune responses. J Immunol Methods. 498, 113147 (2021).
- Inagaki, Y., et al. Interferon-g-induced apoptosis and activation of THP-1 macrophages. Life Sci. 71 (21), 2499-2508 (2002).
- Boonkaewwan, C., Toskulkao, C., Vongsakul, M. Anti-inflammatory and immunomodulatory activities of stevioside and its metabolite steviol on THP-1 cells. J Agric Food Chem. 54 (3), 785-789 (2006).
- Chanput, W., et al. β-Glucans are involved in immune-modulation of THP-1 macrophages. Mol Nutr Food Res. 56 (5), 822-833 (2012).
- Cui, J., et al. USP3 inhibits type I interferon signaling by deubiquitinating RIG-I-like receptors. Cell Res. 24 (4), 400-416 (2014).
- Chen, S., et al. SAMHD1 suppresses innate immune responses to viral infections and inflammatory stimuli by inhibiting the NF-κB and interferon pathways. Proc Natl Acad Sci USA. 115 (16), E3798-E3807 (2018).
- Pradhananga, S., Spalinskas, R., Poujade, F. A., Eriksson, P., Sahlén, P. Promoter anchored interaction landscape of THP-1 macrophages captures early immune response processes. Cell Immunol. 355, 104148 (2020).
- Mezzasoma, L., Talesa, V. N., Romani, R., Bellezza, I. Anp and BNP exert anti-inflammatory action via npr-1/cgmp axis by interfering with canonical, non-canonical, and alternative routes of inflammasome activation in human THP1 cells. Int J Mol Sci. 22 (1), 1-17 (2021).
- Martinat, C., et al. SUMOylation of SAMHD1 at Lysine 595 is required for HIV-1 restriction in non-cycling cells. Nat Commun. 12 (1), 4582 (2021).
- Rensen, E., et al. Clustering and reverse transcription of HIV-1 genomes in nuclear niches of macrophages. EMBO J. 40 (1), e105247 (2021).
- Ikeda, T., et al. APOBEC3 degradation is the primary function of HIV-1 Vif determining virion infectivity in the myeloid cell line THP-1. mBio. 14 (4), e0078223 (2023).
- Jinek, M., et al. A programmable dual RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337, 816-821 (2012).
- Kurosaki, T., Maquat, L. E. Nonsense-mediated mRNA decay in humans at a glance. J Cell Sci. 129 (3), 461-467 (2016).
- Tuladhar, R., et al. CRISPR-Cas9-based mutagenesis frequently provokes on-target mRNA misregulation. Nat Commun. 10 (1), 4056 (2019).
- Embree, C. M., Abu-Alhasan, R., Singh, G. Features and factors that dictate if terminating ribosomes cause or counteract nonsense-mediated mRNA decay. J Biol Chem. 298 (11), 102592 (2022).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Schnoor, M., et al. Efficient non-viral transfection of THP-1 cells. J Immunol Methods. 344 (2), 109-115 (2009).
- Tang, X., et al. A method for high transfection efficiency in THP-1 suspension cells without PMA treatment. Anal Biochem. 544, 93-97 (2018).
- Ji, W., Zhang, L., Liu, X. Protocol using lentivirus to establish THP-1 suspension cell lines for immunostaining and confocal microscopy. STAR Protoc. 4 (1), 102032 (2023).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, (2023).
- . Synthego Performance Analysis, ICE Analysis Available from: https://www.synthego.com/help/citing-ice (2019)
- Salamov, A. A., Nishikawa, T., Swindells, M. B. Assessing protein coding region integrity in cDNA sequencing projects. Bioinformatics. 14, 384-390 (1998).
- Pedersen, A. G., Nielsen, H. Neural network prediction of translation initiation sites in eukaryotes: Perspectives for EST and genome analysis. Proc Int Conf Intell Syst Mol Biol. 5, 226-233 (1997).
- Concordet, J. P., Haeussler, M. CRISPOR: Intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46 (W1), W242-W245 (2018).
- Laguette, N., et al. SAMHD1 is the dendritic- and myeloid-cell-specific HIV-1 restriction factor counteracted by Vpx. Nature. 474 (7353), 654-657 (2011).
- Hrecka, K., et al. Vpx relieves inhibition of HIV-1 infection of macrophages mediated by the SAMHD1 protein. Nature. 474 (7353), 658-661 (2011).
- Seki, A., Rutz, S. Optimized RNP transfection for highly efficient CRI SPR/Cas9-mediated gene knockout in primary T cells. J Exp Med. 215 (3), 985-997 (2018).
- Batista Napotnik, T., Polajžer, T., Miklavčič, D. Cell death due to electroporation - A review. Bioelectrochemistry. 141, 107871 (2021).
- Perretta-Tejedor, N., Freke, G., Seda, M., Long, D. A., Jenkins, D. Generating mutant renal cell lines using CRISPR technologies. Methods Mol Biol. 2067, 323-340 (2020).
- Sentmanat, M. F., Peters, S. T., Florian, C. P., Connelly, J. P., Pruett-Miller, S. M. A survey of validation strategies for CRISPR-Cas9 editing. Sci Rep. 8 (1), 888 (2018).
- Makino, S., Fukumura, R., Gondo, Y. Illegitimate translation causes unexpected gene expression from on-target out-of-frame alleles created by CRISPR-Cas9. Sci Rep. 6, 39608 (2016).
- Bowling, A., et al. Downstream alternate start site allows N-terminal nonsense variants to escape NMD and results in functional recovery by readthrough and modulator combination. J Pers Med. 12 (9), 1448 (2022).
- Inácios, &. #. 1. 9. 4. ;., et al. Nonsense mutations in close proximity to the initiation codon fail to trigger full nonsense-mediated mRNA decay. J Biol Chem. 279 (31), 32170-32180 (2004).
- Li, J., et al. Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9. J Mol Cell Biol. 7 (4), 284-298 (2015).
- Guo, Y., et al. CRISPR inversion of CTCF sites alters genome topology and enhancer/promoter function. Cell. 162 (4), 900-910 (2015).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten