Method Article
נוקאאוט גנים בתיווך CRISPR-Cas9 מבוסס אלקטרופורציה בתאי THP-1 ובידוד שיבוט של תא בודד
In This Article
Summary
קו התאים THP-1 נמצא בשימוש נרחב כמודל לחקירת התפקודים של מונוציטים/מקרופאגים אנושיים בתחומי מחקר שונים הקשורים לביולוגיה. מאמר זה מתאר פרוטוקול עבור הנדסה יעילה מבוססת CRISPR-Cas9 ובידוד שיבוטים של תא יחיד, המאפשר ייצור של נתונים פנוטיפיים חסונים וניתנים לשחזור.
Abstract
קו תאי THP-1 של לוקמיה מונוציטית חריפה אנושית (AML) נמצא בשימוש נרחב כמודל לחקר הפונקציות של מקרופאגים אנושיים שמקורם במונוציטים, כולל יחסי הגומלין שלהם עם פתוגנים אנושיים משמעותיים כגון נגיף הכשל החיסוני האנושי (HIV). בהשוואה לקווי תאים אימורטליים אחרים ממקור מיאלואידי, תאי THP-1 שומרים על מסלולי איתות דלקתיים שלמים רבים ומציגים מאפיינים פנוטיפיים הדומים יותר לאלה של מונוציטים ראשוניים, כולל היכולת להתמיין למקרופאגים כאשר הם מטופלים בפורבול-12-מיריסטאט 13-אצטט (PMA). השימוש בטכנולוגיית CRISPR-Cas9 להנדסת תאי THP-1 באמצעות נוקאאוט גן ממוקד (KO) מספק גישה רבת עוצמה לאפיון טוב יותר של מנגנונים הקשורים למערכת החיסון, כולל אינטראקציות בין וירוס למארח. מאמר זה מתאר פרוטוקול להנדסה יעילה מבוססת CRISPR-Cas9 המשתמשת באלקטרופורציה כדי להעביר ריבונוקלאופרוטאינים Cas9:sgRNA שהורכבו מראש לגרעין התא. שימוש במספר sgRNAs המכוונים לאותו לוקוס במיקומים מעט שונים מביא למחיקה של שברי DNA גדולים, ובכך מגביר את יעילות העריכה, כפי שהוערך על ידי בדיקת T7 endonuclease I. עריכה בתיווך CRISPR-Cas9 ברמה הגנטית אומתה על ידי ריצוף סנגר ואחריו ניתוח הסקה של עריכות CRISPR (ICE). דלדול החלבון אושר על ידי אימונובלוטינג יחד עם בדיקה פונקציונלית. באמצעות פרוטוקול זה הושגו עד 100% אינדל בלוקוס הממוקד וירידה של למעלה מ-95% בביטוי החלבון. יעילות העריכה הגבוהה מקלה על בידוד שיבוטים של תא בודד על ידי הגבלת הדילול.
Introduction
THP-1 הוא קו תאים אנושי שמקורו במונוציטים שבודד מחולה הסובל מלוקמיה חריפה (AML), המציג מאפיינים פנוטיפיים הדומים מאוד לאלה של מונוציטים ראשוניים1. בהשוואה למקרופאגים ראשוניים שמקורם במונוציטים, שאינם מתחלקים ומציגים הן תוחלת חיים מוגבלת והן שונות בין/תוך-תורם בפנוטיפ, ניתן לתרבת תאי THP-1 כמעט לנצח ויש להם התנהגות הומוגנית יותר המעדיפה שחזור תוצאות 2,3,4,5,6 . יש לציין כי ניתן להתמיין בתאי THP-1 לכיוון פנוטיפ דמוי מקרופאגים עם phorbol-12-myristate 13-acetate (PMA), מה שהופך אותם למודל מבחנה בשימוש נרחב לחקירת התגובות של מונוציטים/מקרופאגים לאותות דלקתיים 7,8,9,10,11,12,13 או זיהום על ידי פתוגנים אנושיים רלוונטיים מבחינה קלינית, כולל HIV 14,15,16. האפשרות להנדס גנטית תאי THP-1 מעניינת בתחומי מחקר רבים הקשורים לביולוגיה.
Clustered Regular Interspaced Short Palindromic Repeats-CRISPR associated protein 9 (CRISPR-Cas9) הוא מערכת חיסון אדפטיבית פרוקריוטית המבוססת על נוקלאז מונחה RNA כדי לפרק גנומים נגיפיים פולשים, אשר תוכנתה מחדש ככלי להנדסה גנטית17. תהליך עריכת הגנום מתנהל בשלושה שלבים: זיהוי, מחשוף ותיקון. RNA מדריך יחיד (sgRNA) מגייס את נוקלאז Cas9 למיקום גנומי ספציפי באמצעות זיווג בסיסים עם רצף המדריך של 20 bp. נוכחותו של רצף Protospacer Adjacent Motif (PAM) ישירות 3' מרצף המטרה הגנומי של 20 bp מפעילה את ההתפרקות והפיצול בתיווך Cas9 בשני גדילי ה-DNA בין מיקומים 17 ו-18 (3-bp 5' של ה-PAM). שבירת הגדיל הכפול (DSB) המתקבלת מעובדת על ידי שני מסלולי תיקון עיקריים. בהיעדר תבנית תיקון הנושאת הומולוגיה עם הלוקוס הפגום, מסלול הצטרפות הקצה הלא-הומולוגי (NHEJ) הנוטה לשגיאות יציג הכנסות ו/או מחיקות אקראיות של נוקלאוטידים (indels), מה שעלול להוביל למוטציות בשינוי מסגרת ו/או הכנסת קודוני סיום מוקדמים (PTC). בתורו, mRNAs המכילים PTC ממוקדים על ידי פירוק על ידי מסלול דעיכת mRNA בתיווך שטויות (NMD), ובסופו של דבר משבשים את ביטוי/תפקוד החלבון 18,19,20. לחלופין, מסלול התיקון המונחה הומולוגיה (HDR) התלוי בתבנית יכול לפעול ולתקן נאמנה את ה-DSB. מנגנון זה נרתם להשגת עריכה גנטית מדויקת, כולל דפיקות והחלפות בסיסים. ראוי לציין כי מצב מחזור התא הוא גורם חשוב המשפיע על בחירת מסלול תיקון DSB. ואכן, NHEJ פעיל בכל שלבי מחזור התא, בעוד ש-HDR מוגבל בעיקר לשלבי S/G221.
תאי THP-1 גדלים בתרחיף וידועים לשמצה כקשים להעברה עם DNA פלסמיד, הליך שאולי גם משנה את הכדאיות ו/או יכולת ההתמיינות שלהם22,23. התמרה עם וקטורים לנטי-ויראליים מבוססי HIV-1 המקודדים הן את Cas9 והן את ה-sgRNA משמשת לעתים קרובות כדי להפיל (KO) גן מעניין24. שילוב קלטת Cas9/sgRNA בגנום התא מבטיח ביטוי ממושך ו-KO יעיל, אך הוא גם מקור מתמשך להשפעות מחוץ למטרה25. לחלופין, הריבונוקלאופרוטאינים Cas9:sgRNA (RNPs) שהורכבו מראש מועברים על ידי אלקטרופורציה, שיטה הכוללת היווצרות זמנית של נקבוביות הן בפלזמה והן בממברנות הגרעין בעת הפעלת דחפים חשמליים. שימור כדאיות התאים הוא אתגר חשוב בעת נקיטת גישה זו.
כאן, קו תאים THP-1 המבטא ביציבות GFP (THP-1_GFP) הופק כדי לשמש ככלי להקמת פרוטוקול להשגת עריכה יעילה מבוססת CRISPR-Cas9. לאחר תכנון אסטרטגיה להשבתת הגן EGFP באמצעות שלושה sgRNAs בו זמנית (גישה מרובת מדריכים), נקבעה יעילות KO בין מספר תנאי אלקטרופורציה באמצעות ביטוי GFP כקריאה. התפשטות התאים נוטרה במקביל. עריכת גנים אושרה הן על ידי בדיקת T7 endonuclease I (T7EI) והן על ידי ריצוף Sanger, ואחריו ניתוח עם אלגוריתם Inference of CRISPR Edits (ICE)26. פרמטרים שהניבו ירידה של עד 95% בביטוי GFP, כאשר תאי THP-1 התאוששו משיעורי גדילה תקינים לאחר אלקטרופורציה, הופעלו בהצלחה כדי לנטרל גן אנדוגני (SAMHD1) ולייצר שיבוטי THP-1 חד-תאיים.
Protocol
פרטי הריאגנטים והציוד המשמש במחקר זה מפורטים בטבלת החומרים.
1. עיצוב מדריך עם CRISPOR (איור 1.1)
הערה: ניתן להשתמש בתוכנת SnapGene Viewer בשלבים 4, 7 ו-10 כדי להוסיף הערות לאתר יעד העריכה ולמיקום הכלאת פריימר ה-PCR בתוך הגן המבוקש.
- היכנסו לאתר האנסמבל (www.ensembl.org). בתיבה חיפוש , בחר מין והזן את שם הגן המעניין. לחץ על Go. בחר את התוצאה המתאימה לגן (לא לתעתיק).
- לחץ על הצג טבלת תמלול ולאחר מכן בחר את מזהה התמלול המתאים לקידוד החלבון עם תווית הזהב בעמודה ביוטייפ . לאחר דף התמליל, לחץ על Exons בתפריט השמאלי.
- גלול מטה ולחץ על הורד רצף. ודא שפורמט הקובץ הוא FASTA. בהגדרות - רצפים כלולים, בטל את הבחירה בכל הפריטים מלבד רצף גנומי.
הזן את המספר "500" ברצף האגפים בכל אחד מקצוות תיבת התמלול . לחץ על תצוגה מקדימה, בחר את כל הרצף (רק את הנוקלאוטידים ללא הכותרת), והעתק אותו (Ctrl+C). - פתח את SnapGene Viewer ולחץ על קובץ DNA > חדש. הדבק את הרצף (Ctrl+V) בתיבה צור רצף . בטל את הסימון של זהה תכונות נפוצות ובחר ליניארי בטופולוגיה.
שנה את שם הקובץ ולחץ על צור. בתפריט השמאלי, בטל את הבחירה באפשרות הצג אנזימים (סמל ראשון). בחלק התחתון של החלון, בחר את הכרטיסיה רצף .
הערה: שלב זה מאפשר אחזור של רצף הגנים השלם, כולל אקסונים, אינטרונים ורצפי אגף של 500 bp (אופציונלי). מידע אחרון זה שימושי לתכנון פריימרים PCR להגברה של אתר יעד הממוקם בתוך האקסון הראשון. - בחזרה לאתר Ensembl, גלול למעלה בתצוגה המקדימה של הקובץ ולחץ על הקודם. כעת, שנה את פורמט הקובץ ל-RTF. בהגדרות - רצפים כלולים, בטל את הבחירה בכל הפריטים מלבד Exons. בהצג משתנים, בחר לא. לחץ על הורד בראש העמוד.
- פתח את הקובץ שהורדת (עם Word), המכיל את רצף האקסונים ומציג את רצף הקידוד בכחול, החל מה-ATG הראשוני. בחר את האקסון שימוקד על ידי העריכה המכוונת CRISPR-Cas9 (ראה להלן להמלצות), בחר אותו והעתק אותו (Ctrl+C).
- האקסון הממוקד הוא אקסון מוקדם או אקסון המקודד תחום חשוב מבחינה תפקודית של החלבון. ראוי לציין כי הקמת PTC באקסון מאוחר קרוב ל-3' UTR ככל הנראה לא תצליח לעורר NMD, מה שיוביל לביטוי של חלבון קטום C-terminally. לעומת זאת, הכנסת PTC באקסון מוקדם קרוב לאתר ההתחלה המקורי קשורה לסיכון של תרגום לא לגיטימי (ITL, הידוע גם בשם התחלת תרגום חלופי (ATI)), המניב ביטוי בלתי צפוי של חלבון קטום N-terminally שמתחיל באתרי התחלת תרגום בתוך המסגרת (TIS) במורד הזרם של קודון ה-ATG הראשון. כדי להפחית את הסיכון האחרון, מומלץ להעריך את התרחשותם של TIS חלופיים באמצעות ATGpr27 (atgpr.dbcls.jp) ו/או NetStart 1.028 (services.healthtech.dtu.dk/services/NetStart-1.0/).
- ודא שאקסון היעד מכיל רצף קידוד. עם זאת, זה עשוי להיות שימושי לבחור חישול sgRNA עם אזור 5'UTR במעלה הזרם של ה-ATG הראשוני כדי לכלול אותו בקטע שנמחק.
- באופן אידיאלי, האקסון הממוקד צריך להיות משותף לכל גרסאות התעתיק המקודדות לחלבון של הגן. בדוק אם זה המקרה במציג נתוני הגנום של NCBI (www.ncbi.nlm.nih.gov/gdv/) על ידי חיפוש הגן המעניין.
לחיצה על שם הגן (בירוק) בחלון התצוגה תציג את גרסאות התעתיק (בסגול). אקסונים מיוצגים על ידי מלבן.
- ב- SnapGene Viewer, הקש Ctrl+F, Ctrl+V ולאחר מכן Enter כדי לחפש את רצף האקסון. לחץ על Ctrl+T כדי להוסיף תכונה חדשה, תן לה שם, שנה את הסוג ל"אקסון" ולחץ על אישור.
- עבור לאתר CRISPOR (http://crispor.gi.ucsc.edu/) והדבק את רצף האקסון בשלב 1. ראשית, בחר גנום ייחוס בשלב 2 ולאחר מכן את סוג ה-PAM שיש למקד בשלב 3, בדרך כלל 20bp-NGG עבור SpCas9. לחץ על שלח.
- בחר שני sgRNA כך שהם יופרדו עד 150 bp, ולאחר מכן בחר sgRNA שלישי ביניהם. להלן מספר הנחיות לבחירת sgRNA:
- ציון הספציפיות של MIT קשור להשפעות מחוץ למטרה. ציון גבוה יותר מצביע על פחות שחקנים פוטנציאליים מחוץ למטרה. העמודה הימנית מציגה שלושה אתרים חזויים מחוץ למטרה המדורגים מהסבירות הגבוהה ביותר לסבירות הנמוכה ביותר, יחד עם מיקומם (באקסון, אינטרון או אזור בין-גני). ניתן לגשת לרשימה המלאה של אתרים צפויים מחוץ ליעד על ידי לחיצה על הצג הכל. במידת האפשר, בחר sgRNAs עם ציון MIT >80, ותעדוף את אלה ללא מטרות עבור 0, 1 או 2 אי התאמות. בנוסף, יש להימנע מ-sgRNAs עם מטרות מחוץ לאקסון, שיש להם את הפוטנציאל הגדול ביותר להשפיע על הפנוטיפ.
- ליעילות חזויה, עיין בציון Doench '16. שימו לב שציון גבוה של Doench '16 פשוט מצביע על כך שה-sgRNA סביר יותר להיות יעיל. יש לקבוע את היעילות בפועל בניסוי. מסיבה זו, תמיד כדאי לבחור מספר sgRNAs, גם אם הם אינם מיועדים לשימוש יחד.
- תכולת GC גבוהה מדי או נמוכה מדי, כמו גם מוטיבים מסוימים, עלולים להזיק ליעילות sgRNA ויש להימנע מהם. פרמטרים אלה מודגשים על ידי CRISPOR.
- חזור על שלב 1.7 כדי להוסיף את רצף ה-sgRNA ואת רצף ה-PAM המשויך לרצף הגנים ב-SnapGene Viewer. במקביל, הדבק את רצף ה-sgRNA (ללא ה-PAM) בקובץ טקסט או Excel כדי לשמור על הכיוון 5'-3' הנדרש בעת הזמנת האוליגונוקלאוטיד.
- להגברה מבוססת PCR של אתר היעד, תכנן כמה פריימרים המקיפים אותו. גודל האמפליקון צריך להיות בין 800 ל-1000 bp. PrimerQuest שימש לעיצוב הפריימרים לפרוטוקול זה (https://eu.idtdna.com/pages/tools/primerquest). לחלופין, CRISPOR מספק רשימה של פריימרים כדי להגביר את האזור הגנומי הממוקד כמו גם את האתרים מחוץ למטרה. לאחר הצגת הרשימה המלאה של אתרים פוטנציאליים מחוץ ליעד (שלב 1.9.1), לחץ על פריימרים מחוץ למטרה בפינה השמאלית התחתונה.
2. ריאגנטים והכנת תאים לאלקטרופורציה (איור 1.2)
- הכן צלחת תרבית של 24 בארות לשחזור תאים לאחר אלקטרופורציה על ידי מילוי הבארות ב-500 מיקרוליטר של מדיום RPMI 1640 בתוספת 20% מומת בחום (56 מעלות צלזיוס, 30 דקות), מסונן (0.20 מיקרומטר) סרום בקר עוברי (FBS). אין להוסיף אנטיביוטיקה. שמור את הצלחת בחממה לחה בטמפרטורה של 37 מעלות צלזיוס ו-5% CO2 למשך 24 שעות עד לאלקטרופורציה.
- אם ה-sgRNAs נשלחים יבשים, יש להחזיר אותם ללחות עם מאגר TE (10 מ"מ Tris, 1 מ"מ EDTA, pH 8.0) לריכוז סופי של 100 מיקרומטר (כלומר 10 מיקרוליטר של מאגר TE לכל 1 ננומול של sgRNA). מערבולת למשך 30 שניות, דגירה בטמפרטורה של 4 מעלות צלזיוס למשך הלילה כדי לאפשר התייבשות מלאה, ולאחר הומוגניזציה קצרה של פיפטה, אחסן את תמיסת מלאי ה-sgRNA ב-20 מעלות צלזיוס. בהתאם לנפח הסופי, בצע ציטוטים כדי למנוע מחזורי הקפאה-הפשרה מרובים.
- הכן תמיסת sgRNA עובדת בריכוז סופי של 30 מיקרומטר על ידי דילול תמיסת המלאי של 100 מיקרומטר במים נטולי נוקלאז. מערבולת למשך 30 שניות ודגירה 5 דקות בטמפרטורת החדר.
- הרכיבו את ה-Cas9:sgRNA RNPs ביחס מולארי של 1:9 על ידי דילול בו-זמני של 1 μL של כל אחד משלושת ה-30 μM sgRNAs ו-0.5 μL של תמיסות Cas9 של 20 μM ב-3.5 μL של מאגר השעיה מחדש R, הכלול בערכת האלקטרופורציה (נפח סופי של 7 μL, עבור תנאי ניסוי אחד; קנה מידה בהתאם). מערבולת קצרה ודוגרת במשך 5 דקות בטמפרטורת החדר.
- בינתיים, הכינו בקרה לא ערוכה על ידי הוספת 0.5 מיקרוליטר של 20 מיקרומטר Cas9 ל-6.5 מיקרוליטר של מאגר השעיה מחדש R. מערבולת לזמן קצר ודגרו למשך 5 דקות בטמפרטורת החדר.
- הוסף 5 מיקרוליטר של מאגר השעיה R לכל הדגימות לנפח סופי של 12 מיקרוליטר לכל מצב אלקטרופורציה.
- הכן את תאי THP-1 לאלקטרופורציה.
- להעריך ריכוז וכדאיות על ידי מבחן אי הכללה כחול טריפאן. יש לדלל את התאים ביחס של 1:2 בתמיסת צביעה כחולה של 0.4%. לאחר דגירה של 30 שניות, הומוגניזציה היטב והוסף 10 מיקרוליטר בקיר של תא ספירה עם סגנון רשת משופר של נויבאואר. ספרו שלושה ריבועים גדולים וחלקו את הספירה ב-100 כדי לקבל את ריכוז התא (x106 תאים/מ"ל).
הערה: בריאות התאים משפיעה על רגישותם לאלקטרופורציה. יש להקפיד לבצע את תרבית התאים בתנאים אופטימליים. יש להגביל את הזמן מחוץ לחממה, ולהכין ולחמם את כל הריאגנטים והפתרונות מראש. - עבור כל תנאי, אסוף נפח שווה ערך ל-0.2 x 106 תאים וצנטריפוגה (336 x גרם, 5 דקות, 20 מעלות צלזיוס).
- שאפו את הסופרנטנט באמצעות פיפטה והשעו מחדש את הגלולה ב-500 מיקרוליטר של PBS. שוב צנטריפוגה (336 x גרם, 5 דקות, 20 מעלות צלזיוס).
- שאפו את הסופרנטנט בזהירות באמצעות פיפטה והשעו מחדש את כדורית התא THP-1 עם 12 מיקרוליטר של תמיסת RNP (שלב 2.6).
- להעריך ריכוז וכדאיות על ידי מבחן אי הכללה כחול טריפאן. יש לדלל את התאים ביחס של 1:2 בתמיסת צביעה כחולה של 0.4%. לאחר דגירה של 30 שניות, הומוגניזציה היטב והוסף 10 מיקרוליטר בקיר של תא ספירה עם סגנון רשת משופר של נויבאואר. ספרו שלושה ריבועים גדולים וחלקו את הספירה ב-100 כדי לקבל את ריכוז התא (x106 תאים/מ"ל).
3. הקמת מערכת אלקטרופורציה ונוקלאופקציה (איור 1.3)
- הנח את תחנת הפיפטה מתחת לארון בטיחות ביולוגית, הכניס צינור אלקטרופורציה לתמיכה, והוסף 3 מ"ל של מאגר E, הכלול בערכת האלקטרופורציה.
- לאחר הפעלת מכשיר האלקטרופורציה, השתמש במסך המגע כדי להגדיר את פרמטרי האלקטרופורציה הבאים: מתח = 1 500 וולט, משך = 10 אלפיות השנייה, מספר = 3.
- ציידו את פיפטת האלקטרופורציה בקצה ושאבו 10 מיקרוליטר מתמיסת RNP/THP-1 (שלב 2.7.4). הכנס את הפיפטה לתוך צינור האלקטרופורציה ולחץ על התחל במסך מכשיר האלקטרופורציה. המתן להופעת ההודעה הושלם והסר את הפיפטה מהצינור.
- העבירו את התאים לבאר של צלחת 24 הבארות שחוממה מראש והומוגניזציה בעדינות. החזירו את הצלחת לאינקובטור הלח ותנו להם לנוח ללא הפרעה במשך 72 שעות.
הערה: הקפד לא ליצור בועות בעת פיפטינג של מתלה RNP/THP-1, מכיוון שהן יפריעו להליך האלקטרופורציה. אם מופיעה הודעת שגיאה בהיעדר קשת חשמלית גלויה, הסר את פיפטת האלקטרופורציה מהצינור ולחץ שוב על התחל . עם זאת, אם נצפתה קשת חשמלית בצורת ניצוץ בהיר קצר, זה עשוי להצביע על נוכחות של בועות. הליך האלקטרופורציה צפוי להיכשל, גם בהיעדר הודעת שגיאה.
4. התאוששות THP-1 72 שעות לאחר אלקטרופורציה (איור 1.4)
- ספרו את התאים כדי להעריך את הריכוז (שלב 2.7.1) 72 שעות לאחר האלקטרופורציה.
- אם יש מספיק תאים (כלומר, ≥0.6 x 106 תאים/מ"ל), יש לדלל אותם לפחות פקטור 2 עם RPMI בתוספת 20% FBS ו-1% פניצילין-סטרפטומיצין ולהביא את הריכוז בין 0.3-0.5 x 106 תאים למ"ל. אחרת, אפשר לתא להתאושש למשך 72 שעות נוספות.
- מעבר והגברה של התאים עד שיהיה מספיק לאימות KO. בינתיים, ניתן להתחיל בבידוד של שיבוטים חד-תאיים (שלב 7).
5. אימות עריכת גנים על ידי בדיקת אי התאמה T7EI (איור 1.5)
הערה: הבדיקה עלולה להמעיט ביעילות העריכה בהתחשב בכך ש-T7EI מזהה אי-התאמות גדולות מ-1 bp. לפיכך, בדיקת T7EI אינה שימושית לסינון אוכלוסיות תאים הומוזיגוטיות (כלומר, שיבוטים של תא בודד) אלא אם כן היא שונה כראוי (שלב 5.7).
- העריכו את ריכוז התאים (שלב 2.7.1) ומשכו נפח שווה ערך ל-0.1 x 106 תאים בצינור של 1.5 מ"ל. צנטריפוגה (336 x גרם, 5 דקות, 20 מעלות צלזיוס), שואבים את הסופרנטנט ומשהים מחדש את הגלולה ב-500 מיקרוליטר של PBS. שוב צנטריפוגה ובעזרת פיפטה, שאפו כמה שיותר סופרנטנט מבלי להפריע לגלולה. הקפיאו את הדגימה בהצמדה ואחסנו בטמפרטורה של -20 מעלות צלזיוס.
- חלץ את ה-DNA הגנומי כדי לשמש מטריצה להגברת PCR.
- השעו מחדש את הגלולה עם 50 מיקרוליטר של תמיסת מיצוי DNA, הומוגניזציה והעבירו את כל הנפח בצינור PCR של 0.2 מ"ל. מערבולת וצנטריפוגה בקצרה (דופק למשך 3 שניות).
- הנח את הצינור במחזור תרמי וחמם אותו בחום של 65 מעלות צלזיוס למשך 15 דקות, ולאחר מכן 98 מעלות צלזיוס למשך 10 דקות.
- יש לדלל את ה-DNA המופק ב-90 מיקרוליטר של מים טהורים במיוחד. מערבולת וצנטריפוגה בקצרה (5,000 x גרם, 3 שניות).
- לדלל את פריימר ה-PCR (ראה טבלת חומרים) במים טהורים במיוחד לריכוז סופי של 10 מיקרומטר (כלומר, 10 pmol/μL).
- הכן את תערובת ה-PCR (בהתאם להערה למטה) בצינור PCR של 0.2 מ"ל (נפח סופי = 50 מיקרוליטר, למצב אחד). בדרך כלל, יהיו לפחות שני תנאים: ה- KO והפקד הלא ערוך עם Cas9 בלבד.
הערה: DNA גנומי מטוהר: 10 מיקרוליטר; פריימר קדימה ואחורה (10 מיקרומטר): 2.5 מיקרוליטר כל אחד, הריכוז הסופי של 500 ננומטר; מאגר תגובה (5x): 10 מיקרוליטר מערבולת הרבה לפני ההוספה. מערבבים dNTP (25 מ"מ מכל אחד): 0.6 מיקרוליטר, ריכוז סופי של 0.3 מ"מ לכל dNTP. DNA פולימראז: 0.5 מיקרוליטר; מים טהורים במיוחד: 23.9 מיקרוליטר מערבולת וצנטריפוגה לזמן קצר (דופק למשך 3 שניות). - הנח את הצינורות במחזור התרמי והפעל את תוכנית ה-PCR עם ההגדרות הבאות:
הערה: מחזור אחד ב-95 מעלות צלזיוס למשך 5 דקות, ואחריו 30 מחזורים [98 מעלות צלזיוס למשך 20 שניות (שלב דנטורציה), X מעלות צלזיוס למשך 15 שניות (שלב חישול), 72 מעלות צלזיוס למשך 45 שניות (שלב התארכות)], ואז מחזור אחרון אחד ב-72 מעלות צלזיוס למשך 2 דקות. בתום ההגברה, הסר את הצינורות, המערבולת והצנטריפוגה לזמן קצר (דופק למשך 3 שניות). טמפרטורת החישול (X) היא טמפרטורת ההיתוך (Tm) של הפריימרים מינוס 5 מעלות צלזיוס. - לאוכלוסייה ערוכה רב-שבטית: בשפופרת חדשה של 0.2 מ"ל, הוסף 17.5 מיקרוליטר של אמפליקון PCR ו-2 מיקרוליטר של NEBuffer 2 (פי 10) לנפח סופי של 19.5 מיקרוליטר. מערבולת וצנטריפוגה בקצרה (דופק למשך 3 שניות). לחלופין, כדי לסנן שיבוטים של תא בודד, ערבב 1:1 מתוצרי ה-PCR הן מתאי הבקרה הערוכים והן מתאי הבקרה הלא ערוכים (שלב 5.6) (איור משלים 1).
- עבור היווצרות ההטרו-דופלקס, הנח את הצינורות במחזור התרמי והפעל את התוכנית הבאה: מחזור אחד ב-95 מעלות צלזיוס למשך 10 דקות, אחד עם רמפה של -2 מעלות צלזיוס לשנייה מ-95 עד 85 מעלות צלזיוס, אחד עם רמפה של -0.3 מעלות צלזיוס לשנייה מ-85 עד 25 מעלות צלזיוס ומחזור קירור סופי אחד ב-10 מעלות צלזיוס ב-HOLD.
- לאחר היווצרות ההטרודופלקס, הוסף 0.5 מיקרוליטר של תמיסת T7EI לתוך הצינור. יש לדגור בטמפרטורה של 37 מעלות צלזיוס למשך 30 דקות.
- הכינו ג'ל אגרוז 1.2%:
- שקלו את האגרוז בבקבוק זכוכית והוסיפו את הנפח המתאים של מאגר TAE 1x (40 מ"מ טריס-אצטט, 1 מ"מ EDTA, pH 8.3). הכניסו אותו למיקרוגל, שימו לב לא להבריג את המכסה בחוזקה. מחממים מספר פעמים עד שגבישי האגרוז מומסים לחלוטין.
הערה: במידת הצורך, מערבבים את התמיסה. אל תאפשר לו לרתוח, אחרת הנפח יקטן, וישנה את ריכוז האגרוז. - מוסיפים את כתם ג'ל ה- DNA המדולל ב -1/20,000 ומערבבים היטב.
- יוצקים את תמיסת האגרוז לתבנית, מוסיפים מסרק ומניחים להתמצק בטמפרטורת החדר.
- שקלו את האגרוז בבקבוק זכוכית והוסיפו את הנפח המתאים של מאגר TAE 1x (40 מ"מ טריס-אצטט, 1 מ"מ EDTA, pH 8.3). הכניסו אותו למיקרוגל, שימו לב לא להבריג את המכסה בחוזקה. מחממים מספר פעמים עד שגבישי האגרוז מומסים לחלוטין.
- הכינו דגימות לאלקטרופורזה של ג'ל על ידי ערבוב של 5 מיקרוליטר של מוצרי העיכול T7EI (שלב 5.9) או מוצר ה-PCR הלא מעוכל עם 5 מיקרוליטר מים ו-2 מיקרוליטר של צבע טעינת DNA פי 6. טען את הדגימות ואת סולם הגודל והעבר ב-80 וולט למשך 45 דקות (ניתן להתאים את הזמן בהתאם לגודל הצפוי של שברי ה-DNA). לאחר סיום הנדידה, רכשו תמונה של הג'ל עם מערכת הדמיה מתאימה.
6. אימות עריכת גנים על ידי ניתוח ריצוף סנגר (איור 1.6)
- כדי לאפיין את השינוי הגנטי, יש לטהר את תוצרי ה-PCR (שלב 5.4) ולאחר ריצוף סנגר, לנתח את התוצאות באמצעות כלי ICE (https://www.synthego.com/products/bioinformatics/crispr-analysis).
- לשלוט אם ניתן לייצר חלבון למרות השינויים.
- הורד את תוצאות ה-ICE ופתח את קובץ contribs.txt.
- השווה את הרצפים הערוכים עם המקביל של WT. מפה את האינדלים וודא שהם נוצרו באזור הגנומי הממוקד. העריכו את גודלם. אם אורך האינדל אינו כפולה של שלוש, יתרחש שינוי מסגרת, וייתכן שיוצג PTC. ה-mRNA המוטנטי צפוי לעבור פירוק בתיווך NMD.
7. בידוד שיבוט של תא בודד על ידי הגבלת דילול (איור 1.7)
הערה: בידוד של שיבוטים חד-תאיים אינו חובה. עם זאת, אם בוחרים לעשות זאת, חשוב לאפיין שיבוטים מרובים ולהשוות את הפנוטיפ שלהם לאוכלוסייה הפוליקלונית המקורית.
- קח דגימה של תרחיף תאים, דלל אותה 1/2 עם מדיום תרבית והעריך את הריכוז (שלב 2.7.1). דילול מגדיל את דיוק הספירה.
- בצע 2 עד 3 שלבי דילול סדרתי כדי להגיע לריכוז של 7 תאים/מ"ל. הוצא 100 מיקרוליטר של תרחיף התאים לבאר בצלחת עגולה תחתונה של 96 בארות (כלומר, 0.7 תאים לבאר). הניחו לתא להתרוקן במשך כמה שעות לפני שאתם מתבוננים בלוחות במיקרוסקופ כדי לזהות בארות המכילות תא אחד.
- עקוב באופן קבוע אחר היווצרות וצמיחה של מושבה והעביר תאים לצלחת תרבית גדולה יותר או לבקבוק בעת הצורך.
8. אפיון פונקציונלי של תאי THP-1 KO SAMHD1 על ידי בדיקת הגבלת HIV-1
- זרע THP-1 בצלחת תרבית של 24 בארות עם 0.25 x 106 תאים לבאר ב-300 מיקרוליטר של RPMI בתוספת 10% FBS, 1% פניצילין-סטרפטומיצין (RPMI מלא) ומכיל 300 ננוגרם/מ"ל PMA להתמיינות. שמור את הצלחת באינקובטור לח (37 מעלות צלזיוס, 5% CO2) למשך 24 שעות.
- החלף את המדיום במדיום RPMI שלם ללא PMA והחזיר את הצלחת לחממה למשך 24 שעות נוספות.
- בעזרת משאבת ואקום, שאפו את כל מדיום התרבות. הוסף 250 מיקרוליטר של תמיסה המכילה מלאי ויראלי HIV-1-GFP פסאודוטיפ VSVg (MOI = 0.5 IFU/תא). כלול בקרה שאינה נגועה. הנח את הצלחת בטמפרטורה של 4 מעלות צלזיוס למשך שעתיים כדי לסנכרן את הזיהום.
- שטפו את התאים פעם אחת עם RPMI. הוסף 500 מיקרוליטר של RPMI שלם לכל באר ודגר במשך 48 שעות בתנאים סטנדרטיים (ניתן לכוונן את הזמן).
- לאחר הסרת מדיום התרבית, יש לשטוף את התאים פעם אחת עם PBS קר 1x. לאחר מכן, הוסף 100 מיקרוליטר של 0.05% טריפסין-EDTA בכל באר. מניחים את הצלחת באינקובטור בטמפרטורה של 37 מעלות צלזיוס עד שהתאים מנותקים לחלוטין (5 דקות אמורות להספיק) לפני הוספת 200 מיקרוליטר של RPMI מלא כדי לנטרל את האנזים. העבירו 250 מיקרוליטר מכל באר לצלחת תחתונה עגולה של 96 בארות (ודא שציטומטר הזרימה מקבל לוחות תרבית).
- צנטריפוגה את הצלחת (757 x גרם, 3 דקות, האטה = 6, 20 מעלות צלזיוס) ושואבים את הסופרנטנט באמצעות פיפטה רב-ערוצית. השעו מחדש את הגלולה עם 100 מיקרוליטר של 4% PFA ודגרו בטמפרטורה של 4 מעלות צלזיוס למשך 10 דקות. הוסף 100 מיקרוליטר PBS קר 1X.
- לנתח את שיעור ההדבקה, המיוצג על ידי תאים חיוביים ל-GFP, על ידי ציטומטריית זרימה (ראה איור משלים 2 לאסטרטגיית שער מוצעת).
תוצאות
קו תאים THP-1 נוצר ביציבות המבטא את חלבון מדווח GFP (THP-1_GFP) (איור 2A) ושימש ככלי להקמת פרוטוקול למהדורת גנים יעילה בתיווך CRISPR-Cas9. למטרה זו, 3 sgRNA המכוונים לגן EGFP תוכננו עם כלי האינטרנט CRISPOR29 (איור 2B), אשר הורכבו בו זמנית עם Cas9 ביחס מולארי של 9:1 ליצירת RNPs לפני העברתם לתאים על ידי אלקטרופורציה באמצעות הגדרות שונות. לאחר מכן, תאים גודלו במדיום RPMI המכיל 20% FBS בלבד (איור 2C, פאנל עליון) או מדוללים ביחס של 1:1 עם מדיה מותנית (איור 2C, פאנל תחתון). התפשטות תאים וביטוי GFP, כקריאה של יעילות EGFP KO, נוטרו לאורך זמן. במספר מצבים, אחוז התאים החיוביים ל-GFP ירד בחדות, והגיע לירידה של >90% ביום השביעי לאחר האלקטרופורציה (pe). ביום השלישי, מספר התאים ירד בחצי, ככל הנראה בגלל ההשפעה השלילית של אלקטרופורציה על כדאיות התאים. עם זאת, מספר התאים עלה שוב, וזמן ההכפלה חזר לקצב נורמלי ביום 7 pe (איור 2C, פאנל עליון). השימוש במדיה מותנית לא העדיף התאוששות תאים בתנאי הניסוי שלנו (איור 2C, פאנל תחתון). על סמך תוצאות אלה, נבחרו שלושה פולסים של 10 אלפיות השנייה של 1500 וולט לניסויי מעקב.
לאחר מכן אופיין ה-EGFP KO בתיווך CRISPR-Cas9 ברמה הגנומית. לשם כך, ה-DNA הגנומי של תאי THP-1_GFP חולץ, עבר אלקטרופוטציה עם Cas9:sgRNA RNPs (ערוך) או Cas9 לבד (לא נערך), ושימש כתבנית להגברת PCR של אזור 891 bp המכיל את אתר היעד. תאי THP-1 הוריים חסרי EGFP נכללו גם הם כביקורת שלילית. לאחר מכן, תוצר ה-PCR של תאי THP-1_GFP ערוכים הוכן לזיהוי אי התאמה על ידי בדיקת T7EI, ואחריו הפרדת שברי ה-DNA על ידי אלקטרופורזה של ג'ל אגרוז. פס של ~900 bp המתאים לגודל הצפוי של אמפליקון WT הוצג בקלות עבור תאי THP-1_GFP לא ערוכים אך לא עבור בקרת THP-1 הורית (איור 2D, השווה שורות 2 ו-3). במקרה של תאי THP-1_GFP ערוכים, פס ~900 bp הפך לבלתי ניתן לגילוי והוחלף בפס נדידה מהיר יותר, שגודלו תאם לאובדן של מקטע ~75 bp כתוצאה מעריכת CRISPR (איור 2D, שורה 4). דפוס הרצועות הזה השתנה עם עיכול T7EI, מה שגרם להופעת מקטעי דנ"א קטנים יותר, מה שאישר עוד יותר את העריכה המוצלחת של אתר המטרה (איור 2D, שורה 5). כדי לקבל תובנה לגבי יעילות העריכה והגנוטיפים הספציפיים של האוכלוסייה הערוכה, רצף אמפליקון ה-PCR בשיטת סנגר, ואחריו ניתוח עם כלי ICE26. דגימת הבקרה הלא ערוכה עובדה במקביל. אחזרנו ציוני אינדל ו-KO של 100%, המתאימים לאחוז הרצפים שאינם WT במדגם הערוך ולשיעור האינדלים שסביר להניח שמובילים ל-KO פונקציונלי, בהתאמה (איור 2E, משמאל). ניתוח מפורט של הספקטרום והתדירות של גודל האינדל הראה נוכחות של שתי אוכלוסיות דומיננטיות הנושאות מחיקה של 62 נקודות בסיס או 76 נקודות בסיס ומהוות 57% או 37% מכלל הרצפים, בהתאמה (איור 2E, מימין). ממצאים אלה מצביעים על כך ש-sgRNA #1, בשילוב עם sgRNA #2 או #3, הפעיל DSB במקביל בגן המטרה (איור 2B).
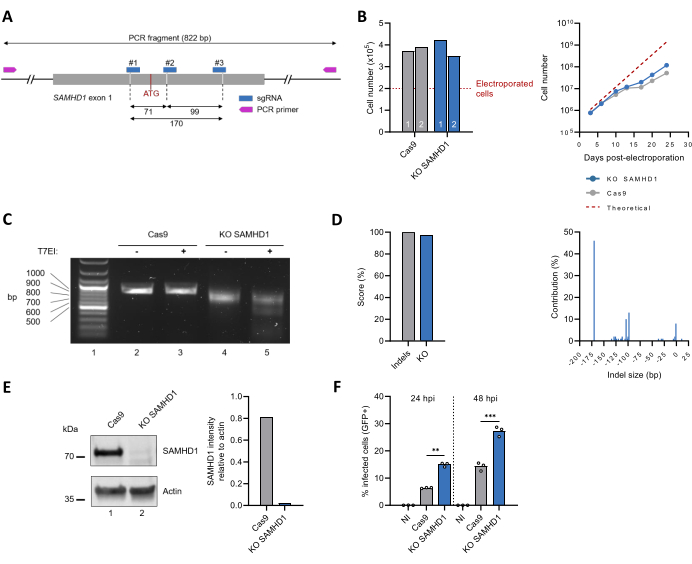
פרוטוקול זה יושם לאחר מכן כדי לנטרל את הגן האנדוגני המקודד ל-SAMHD1 (איור 3A). אפיון קו התאים הפוליקלונלי SAMHD1 KO THP-1 כלל ניטור התפשטות תאים לאורך זמן, שתאם לזה של תאי THP-1 לא ערוכים (איור 3B). תוצאת עריכת הגנים הוערכה על ידי הגברה מבוססת PCR של רצף המטרה, אשר חשפה פס קטן יותר עבור תאי SAMHD1 KO בהשוואה לביקורת Cas9 בלבד (איור 3C, השווה שורות 2 ו-4), מה שמעיד על אובדן רצף DNA. תצפיות אלה אושרו על ידי מבחן T7EI, המראה את הופעתם של תוצרי מחשוף עבור תאי SAMHD1 KO, אך לא ללא עריכה (איור 3C, השווה שורות 3 ו-5). ניתוח נתוני ריצוף סנגר עם כלי ICE החזיר ציוני אינדל ו-KO של 100% ו-97%, בהתאמה. השינויים העיקריים בלוקוס SAMHD1 היו מחיקות רצף של 170 bp, 93 bp או 104 bp ב-46%, 13% ו-10% מהרצפים, בהתאמה (איור 3D). רמות ביטוי החלבון בליזאט התא הגולמי הוערכו גם על ידי אימונובלוטינג. פס ה-~70 kDa התואם את הגודל של SAMHD1 אנדוגני הופך כמעט בלתי ניתן לזיהוי לאחר העריכה, המקביל להפחתה משוערת של >97% (איור 3E). לבסוף, אפיון פנוטיפי של קווי התאים הפוליקלונליים SAMHD1 KO THP-1 הוערך גם על ידי בדיקת הרגישות לזיהום ב-HIV-1. בהסכמה עם תפקידו האנטי-ויראלי הידוע30,31, השבתת לוקוס SAMHD1 לוותה בשיעור זיהום מוגבר ב-HIV-1 (איור 3F).
במקביל, נוצרו שיבוטים של תא בודד על ידי דילול מוגבל. הגברה של אתר המטרה הגנומי משיבוטי SAMHD1 KO הניבה תוצרי PCR עם ניידות אלקטרופורטית מוגברת בהשוואה לאלו של תאי בקרה לא ערוכים (איור 4A). ניתוח מבוסס ICE של נתוני ריצוף סנגר החזיר ציון אינדל של 100% עבור כל שיבוטי התא הבודד שנבחרו מלבד אחד (שיבוט A2) (איור 4B, משמאל). אפיון תוצאות העריכה ברמה הגנומית החזיר רצף יחיד לשיבוט A3 עם מחיקה של 98 bp הממוקם 28 pb במורד הזרם של ה-ATG הראשוני, ובכך הוביל לשינוי מחוץ למסגרת (איור 4B, מימין). השיבוטים האחרים הכילו שלושה (שיבוט B1) או יותר, המעידים על נוכחות של אללים שעברו אירועי עריכה שונים ו/או יותר מתא אחד לבאר בשלב הדילול המגביל (איור 4B, מימין). יש לציין כי 11% מרצפי השיבוטים A4 ו-A7 נושאים אינדליס במעלה הזרם של ה-ATG הראשוני, מה שמשאיר את רצף הקידוד ללא שינוי. יתר על כן, ל-1% מרצפי השיבוט A7 חסר שבר של 123 bp 9-bp במורד הזרם של ה-ATG הראשוני, וכתוצאה מכך מחיקה בתוך המסגרת של 41 חומצות אמינו. בהתאם לממצאים אלה, מחקרים פונקציונליים גילו כי שיבוטים עם מיקום SAMHD1 משובש (A3, A8, B1, B3), ובכך חסרים ביטוי SAMHD1, היו מתירניים מאוד להדבקה ב-HIV-1 (איור 4C). לעומת זאת, שיבוטים A4 ו-A7, שבהם SAMHD1 שלם, היו עמידים כמו שיבוטי THP-1 הוריים או לא ערוכים (איור 4C). לסיכום, אושרה העריכה המוצלחת של לוקוס SAMHD1 בשיבוט A3.
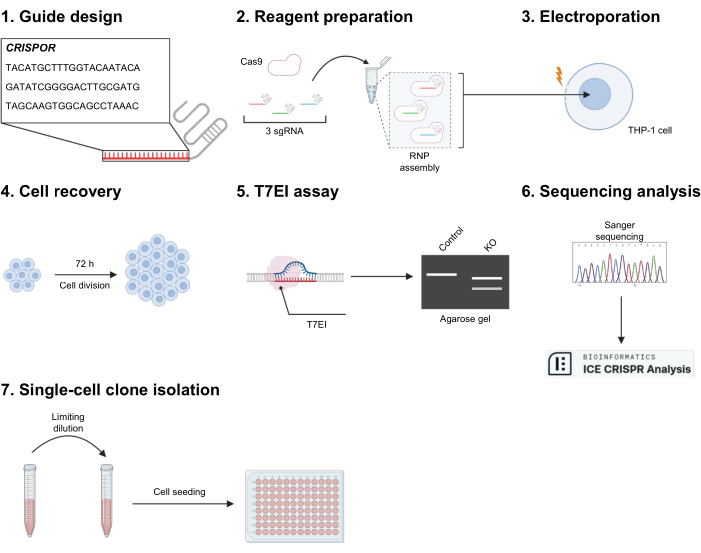
איור 1: מתאר ניסוי. (1) CRISPOR משמש ליצירת רצפי sgRNA כנגד האקסון הממוקד. שלושה מהם נבחרים על פי יעילות גבוהה על המטרה וחיזוי חלש מחוץ למטרה. (2) שלושת ה-gRNAs מעורבבים עם Cas9 כדי להרכיב את הריבונוקלאופרוטאין (RNP), וכתוצאה מכך נוצר ערבוב של שלושה RNPs שונים. (3) התאים עוברים אלקטרופורטציה כדי לאפשר כניסה של ה-RNPs. (4) התאים מועברים לצלחת תרבית של 24 בארות ומניחים להם לנוח לפחות 72 שעות. (5) עריכת גנים נבדקת תחילה באופן איכותי על ידי בדיקת עיכול T7EI, ו-(6) עבור התנאים המאומתים, המאופיינים עוד יותר על ידי ריצוף סנגר וניתוח ICE. (7) במידת הצורך, ניתן לזרוע תאים לאחר הגבלת הדילול כדי לייצר אוכלוסיות משובטות. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
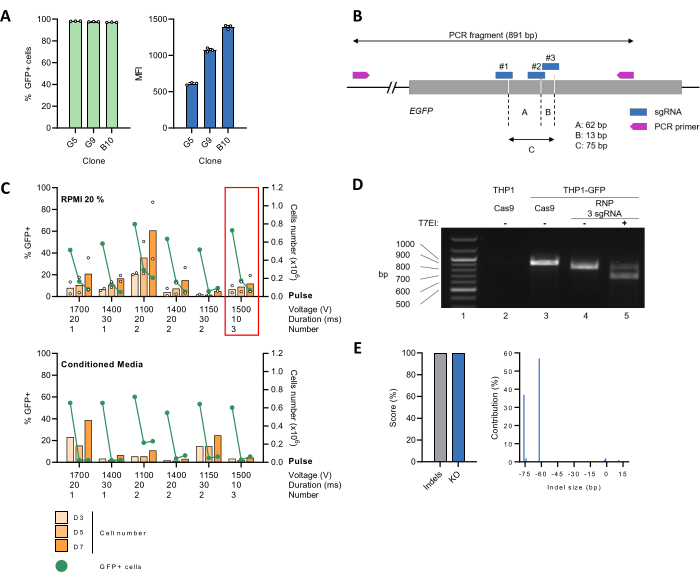
איור 2: הגדרת הגדרות אלקטרופורציה עבור EGFP KO בתאי THP-1 ואימות עריכת גנים. (A) תאי THP-1 הומרו עם וקטורים לנטי-ויראליים המקודדים את הגן המדווח EGFP. שיבוטים של תא בודד הושגו על ידי הגבלת דילול ואחריו הרחבה בתרבית למשך 30 יום. ניתוח על ידי ציטומטריית זרימה אישר שכמעט כל אוכלוסיית התאים ביטאה GFP. עוצמת הקרינה החציונית (MFI) של האוכלוסייה החיובית ל-GFP, המעידה על מספר העותק המשולב של EGFP, השתנתה בין מספר שיבוטי תאים. Clone G5 נבחר למחקרי המשך. (B) ייצוג סכמטי של הגן EGFP, כולל אתרי הקישור הן עבור sgRNA והן עבור פריימרים. אתרי החיתוך Cas9 מסומנים על ידי הקווים המקווקוים. (C) תאי THP-1_GFP לא מובחנים (2 x 105) עורבבו עם Cas9:sgRNA RNPs שהורכבו מראש לפני אלקטרופורציה עם ההגדרות שצוינו. לאחר מכן, התאים הועברו בצלחת של 24 בארות המכילה 500 מיקרוליטר של מדיום RPMI המכיל 20% FBS מחומם מראש לבד או מדולל 1:1 עם מדיום מותנה המיוצר על ידי תרבית תאים THP-1 של 48 שעות. התפשטות (פסים כתומים) ואחוז התאים החיוביים ל-GFP (קו ירוק) נוטרו לאורך זמן. הנתונים מייצגים את הממוצע עבור n = 2 שכפולים ביולוגיים. המסגרת האדומה מדגישה את המצב שנבחר לניסויים הבאים. (D) ה-DNA הגנומי של תאי THP-1_GFP ערוכים (RNP 3 sgRNA) או לא ערוכים (Cas9) חולץ, והאזור המקיף את מוקד המטרה הוגבר על ידי PCR. תאי THP-1 הוריים שימשו כביקורת שלילית. האמפליקונים עברו מחזור דנטורציה/רנטורציה, מה שהוביל להיווצרות הטרודופלקס. לאחר מכן, זוהו אי התאמות על ידי בדיקת T7EI. שברי DNA הופרדו על ידי אלקטרופורזה של 1.2% ג'ל אגרוז. (E) תוצרי PCR מטוהרים רוצפו בשיטת Sanger, ואחריהם ניתוח עם אלגוריתם ICE. ציוני Indel ו-KO מציינים את אחוז הרצפים שאינם WT במדגם, ואת שיעור התאים עם שינוי מסגרת או 21+ bp indel, בהתאמה. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
איור 3: ייצור ואפיון של אוכלוסיית תאי SAMHD1 KO THP-1 רב-שבטית. (A) ייצוג סכמטי של אקסון 1 SAMHD1, כולל אתרי הקישור ל-sgRNA ופריימרים. אתרי החיתוך של Cas9 מסומנים על ידי הקווים המקווקוים, וקידון ה-ATG הראשוני מוצג באדום. (B) שלושה ימים אחרי האלקטרופורציה, תאים ברי קיימא (n = 2) כומתו באמצעות מבחן ההדרה הכחולה של טריפן (תרשים משמאל). צמיחת התאים נוטרה במשך 25 יום (תרשים מימין). הקו המקווקו מתאים למספר התא התיאורטי המבוסס על זמן הכפלה משוער של 49 שעות. (C) תוצאות העריכה ברמה הגנומית הוערכו, כפי שמוצג באיור 2D. (D) גנוטיפים בעקבות עריכת CRISPR זוהו וכומתו על ידי ניתוח מבוסס ICE של רצפי Sanger. (E) החלבון הכלול בליזאט הגולמי של תאי THP-1 ערוכים ולא ערוכים הופרד על ידי נדידה על ג'ל SDS-PAGE, ולאחר מכן, הודגם על ידי אימונובלוטינג באמצעות נוגדנים נגד SAMHD1. אקטין שימש כבקרת הטעינה. העוצמה של רצועות SAMHD1 ו-Actin כומתה על ידי צפיפות עם תוכנת ImageJ. (F) תאי THP-1 לא ערוכים (Cas9) ו-KO SAMHD1 התמיינו על ידי טיפול ב-PMA (300 ננוגרם/מ"ל, 24 שעות) ולאחר מכן אותגרו עם נגיף HIV-1 פסאודוטיפ VSVg המבטא GFP כמדווח. שיעור התאים החיוביים ל-GFP נמדד על ידי זרימה ציטומטרית ב-24 שעות ו-48 שעות לאחר ההדבקה (hpi). NI: לא נגוע. **P < 0.01; P < 0.001 על ידי מבחן t לא מזווג עם התיקון של וולש. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.

איור 4: ייצור ואפיון של שיבוטים חד-תאיים SAMHD1 KO. (A) תוצאת העריכה עבור שיבוטים חד-תאיים נבחרים של SAMHD1 KO הוערכה על ידי ניטור הניידות של מקטעי ה-DNA המיוצרים על ידי הגברת PCR של האזור המכיל את אתר המטרה על ידי אלקטרופורזה של ג'ל אגרוז. (B) נתוני ריצוף סנגר נותחו באמצעות כלי ICE, כמו באיור 2E. העמודות בגרף הימני מייצגות את מגוון האינדלים בכל אוכלוסיית תאים. הפס הירוק מייצג את הרצף הדומיננטי ואת הגודל הכולל של המחיקה הקשורה אליו. מספרים בסוגריים מציינים את גודל הרווחים הלא רציפים. (C) המתירנות של שיבוטים חד-תאיים לא ערוכים ו-SAMHD1 KO לזיהום HIV-1 נבדקה כמו באיור 3F, והושוותה לזו של תאי THP-1 הוריים לא חשמליים או שיבוטי תאים לא ערוכים (Cas9). שיעור ההדבקה הוערך ב-48 hpi. *P < 0.05; **P < 0.01; P < 0.001; P < 0.0001; ns P≥ 0.05 על ידי בראון-פורסיית' ו-Welch ANOVA עם מבחן ההשוואות המרובות של דנט. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
איור משלים 1: הערכת תוצאת העריכה עבור שיבוטים חד-תאיים של KO על ידי מבחן T7EI מותאם. ה-DNA הגנומי של שיבוטים חד-תאיים של KO או תאים לא ערוכים טוהר, והאזור המקיף את מיקום המטרה הוגבר על ידי PCR. האמפליקונים של כל שיבוט של תא בודד עורבבו ביחס של 1:1 עם אלה של תאי ביקורת לא ערוכים. לאחר היווצרות הטרודופלקס ועיכול T7EI, המוצר נותח על ידי אלקטרופורזה של ג'ל אגרוז. השיבוטים הערוכים מייצרים מקטעי עיכול מרובים (שיבוטים A, B, C, D ו-E), בעוד שהלא ערוכים מציגים פס WT יחיד (שיבוטים F ו-G). אנא לחץ כאן להורדת קובץ זה.
איור משלים 2: אסטרטגיית שער זרימה ציטומטרית לכימות אחוז תאי THP-1 נגועים ב-PMA שטופלו ב-HIV-1. באמצעות אזור הפיזור הקדמי (FSC-A) כנגד אזור הפיזור הצדדי (SSC-A), מוגדר שער ראשון לסילוק פסולת ובחירת תאי THP-1 שטופלו ב-PMA. לאחר מכן, תאים בודדים נבחרים עם SSC-A כנגד עלילת SSC-H. לבסוף, תאים חיוביים ל-GFP מזוהים עם GFP-A כנגד FSC-H. אנא לחץ כאן להורדת קובץ זה.
Discussion
כאן, מתואר פרוטוקול להשגת עריכה מוצלחת בתיווך CRISPR של קו התאים THP-1. הגישה מסתמכת על העברה של sgRNA/Cas9 RNPs שהורכבו מראש על ידי אלקטרופורציה/נוקלאופציה. אסטרטגיה זו נבחרה כדי להגביל את ההשפעות מחוץ למטרה שעלולות להתעורר עם אינטגרציה מתווכת לנטיוויראלית של קלטת sgRNA/Cas9, המניבה ביטוי מתמשך של הנוקלאז. מספר sgRNAs המכוונים לגן המעניין נבחרו כדי להשיג עריכה אמינה ויעילה, מה שמגדיל את הסבירות ליצירת אינדל גנומי, מה שמוביל לאובדן ביטוי חלבון ו-KO32 פונקציונלי. תשומת לב מיוחדת הוקדשה להגדרות האלקטרופורציה כדי להשיג חדירות אופטימלית של קרום התא, מה שנדרש כדי להבטיח אספקה יעילה של ה-RNPs sgRNA/Cas9 תוך מניעת מוות מוגזם של תאים33. בהקשר זה, פרמטר מכריע הוא המרץ של תרבית התא לפני האלקטרופורציה (כלומר, מספר מעבר נמוך).
העריכה בתיווך CRISPR-Cas9 אומתה ברמה הגנטית עם בדיקת זיהוי אי התאמה T7EI, המספקת הערכה חסכונית ומהירה של דגימות מרובות לבחירה מוקדמת לפני ריצוף סנגר. שיטה זו פותחה בתחילה כדי להעריך את יעילות העריכה באמצעות sgRNA יחיד על ידי השוואת העוצמה של אמפליקונים באורך מלא ומוצרי מחשוף34,35. ראוי לציין כי ניתן לזהות את האינדלים הגדולים (>70 bp) המיוצרים בעת שימוש ב-sgRNA מרובים על ידי השוואת הניידות של שברי ה-DNA המתקבלים על ידי הגברת PCR של המיקומים הממוקדים מתאים ערוכים לעומת לא ערוכים על ידי אלקטרופורזה ג'ל סטנדרטית. עם זאת, בדיקת T7EI עשויה להיות שימושית כדי לוודא את נוכחותם של רצפים המכילים אינדל קטן (>1bp) באוכלוסיית תאים רב-שבטיים או, באמצעות הפרוטוקול המותאם, באוכלוסיית תאים חד-שבטיים.
נוכחותן של מוטציות מחוץ למסגרת על המטרה, הקוטעות את רצף הקידוד, אושרה על ידי גישה פשוטה, שבה הגנום של כל אוכלוסיית התאים מופק ומשמש כמטריצה להגברת PCR. לאחר מכן, אמפליקונים מטוהרים עוברים ריצוף סנגר בכמויות גדולות, ואחריו ניתוח ביואינפורמטיקה עם כלי ICE. בסך הכל, שיטה זו מתגברת על הצורך בשיבוט תוצרי ה-PCR לפלסמיד והפיכת תוצרי הקשירה לחיידקים כדי לבודד מושבות חד-תאיות, המרוצפות בנפרד.
לאחר אימות ההשבתה של הלוקוס הממוקד, נדרש אפיון יסודי של קו התאים הערוך כדי לייחס בביטחון פנוטיפ נתון להיעדר החלבון המעניין ולשלול השפעות בלתי צפויות הנובעות משינויים מחוץ למטרה, אך גם על המטרה שעלולים להשפיע על מבנה ו/או תפקוד ה-mRNA.
יש לאשר את היעדר ביטוי החלבון על ידי אימונובלוטינג ו/או אימונופלואורסצנציה באמצעות שני נוגדנים המזהים אפיטופים שונים. ואכן, הכנסת PTC באקסון מוקדם קרוב לקודון ATG הקנוני קשורה לסיכון לאירועי ITL, המניבים גרסאות חלבון קטומות N-terminally36. יש לציין כי Tuladhar et al. דיווחו על זיהוי של מיני חלבונים חריגים בחלק משמעותי (~50%) מהתאים שנערכו ב-CRISPR ממקור מסחרי או פנימי18,19. התרחשותם של מוצרי חלבון חדשים יוחסה לתהליכים מגוונים ופוטנציאליים ספציפיים לסוג התא כגון דילוג על האקסון המכיל אינדל או הופעת אתרי כניסת ריבוזום פנימיים (IRES) הגורמים להתחלת סינתזת חלבון ב-TIS חלופי במורד הזרם של קודון ה-ATG הקנוני. ישנם גם רמזים לכך ש-ITL מלווה בבריחה מ-NMD בנסיבות מסוימות37,38. באופן דומה, אסטרטגיות עריכת גנום המכוונות לאקסון מאוחר עשויות להיכשל בהפעלת NMD של ה-mRNA המוטנטי העומד בבסיס אבלציה לא יעילה של ביטוי גנים20. לבסוף, אם Cas9 מונחה על ידי מספר sgRNAs, עלולים להיווצר סידורים כרומוזומליים מורכבים, כולל היפוכים או מחיקות גדולות של מקטעי DNA39, שעלולים לשנות את המבנה והביטוי של הגנום, במיוחד כאשר אלמנטים רגולטוריים מכוונים40.
כדי להפחית עוד יותר את ההשפעה של תוצאות לא מכוונות של עריכת גנום, יש צורך במחקרים פונקציונליים כדי לאשר את הקשר בין אובדן ביטוי החלבון לבין הפנוטיפ שנוצר כתוצאה מכך (כלומר, פעילות אנטי-ויראלית). אם תאים ערוכים משובטים, יש להשוות את ההתנהגות של שיבוטים מרובים בינם לבין עצמם ועם האוכלוסייה הרב-שבטית. ביצוע ניסויי הצלה, המורכבים משחזור ביטוי גנים כדי להפוך את הפנוטיפ של התאים הערוכים לפנוטיפ WT, יחזק גם את הספציפיות וימנע אירועים אפשריים מחוץ למטרה.
למרות שפרוטוקול זה תוכנן לעריכה של תאי THP-1, הוא מספק מבנה כללי לזרימת העבודה של CRISPR-Cas9 KO שניתן להתאים לקווי תאים אחרים. ייתכן שתידרש התאמת פרמטרי האלקטרופורציה, ואם מופעל מתח גבוה מ-1800 וולט, יש להשתמש במאגר מתלה T בהתאם להוראות היצרן. משתנה נוסף שיש לקחת בחשבון הוא יחס הריכוז בין Cas9 ל-sgRNA במהלך הרכבת RNP. למרות שהיחס של 1:9 עבד היטב במקרה זה, שינוי שלו עשוי לשפר את יעילות העריכה בנסיבות אחרות. לבסוף, למרות שאינו מועיל כאן, יש לבדוק את השימוש במדיה מותנית לשחזור תאים לאחר אלקטרופורציה, מכיוון שההשפעה יכולה להיות שונה עם קווי תאים שונים.
Disclosures
לכל המחברים אין ניגודי אינטרסים.
Acknowledgements
אנו אסירי תודה לג'יי.פי קונקורדה (MNHN, U1154/UMR7196, פריז), ג. בוסיס (IGMM, מונפלייה) וד. שלוטר (בית הספר לרפואה של הנובר, גרמניה) על שיתוף הפרוטוקולים ועל הדיון. פרויקט זה קיבל מימון מתוכנית המחקר והחדשנות Horizon 2020 של האיחוד האירופי (הסכם מענק מס' 101017572 ל-AZ) ו-ANRS (מענק ECTZ162721 ל-AZ). מודל המחלות הזיהומיות ותשתית המחקר של טיפולים חדשניים (IDMIT) נתמכת על ידי "תוכנית השקעה ד'אווניר (PIA)" תחת הפניה ANR_11_INSB_0008.
Materials
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filter | ClearLine | 146560 | _ |
| 0.4 % trypan blue | Beckman Coulter | 383200 | _ |
| 1.5 mL tube | Eppendorf | 3810X | _ |
| 24-well plate | Corning | 353047 | _ |
| 6x TriTrack DNA Loading Dye | Thermo scientific | R1161 | _ |
| 75 cm² Culture Flask Vented Cap | Corning | 353136 | _ |
| 8-Strip PCR Tubes with Caps | Life technologies | AM12230 | _ |
| 96-well plates Flat bottom | Corning | 353072 | _ |
| 96-well plates Round bottom | Corning | 353077 | _ |
| Agarose | Euromedex | D5 | _ |
| ATGpr | _ | _ | https://atgpr.dbcls.jp/ |
| ChemiDoc Imaging System | BIO-RAD | 12003153 | _ |
| Counting slide | NanoEntek | DHC-N04 | _ |
| CRISPOR | _ | _ | http://crispor.gi.ucsc.edu/ |
| DPBS | Gibco | 14190094 | _ |
| Ensembl | EMBL-EBI | _ | https://www.ensembl.org/index.html |
| Fetal Bovine Serum | Sigma-Aldrich | F7524 | _ |
| FlowJo | BD Life Sciences | v10.10 | _ |
| GeneRuler 100 bp Plus DNA Ladder | Thermo scientific | SM0323 | _ |
| Genome Data Viewer | NCBI | _ | https://www.ncbi.nlm.nih.gov/gdv/ |
| GraphPad Prism | Dotmatics | _ | Version 9.3.1 |
| Herculase II Fusion DNA Polymerases | Agilent | 600679 | _ |
| ICE CRISPR Analysis Tool | Synthego | _ | https://www.synthego.com/products/bioinformatics/crispr-analysis |
| Image Lab Touch | BIO-RAD | _ | Version 2.4.0.03 |
| NEBuffer 2 | New England Biolabs | B7002S | Included with T7EI M0302S |
| Neon Kit, 10 µL | Invitrogen | MPK1025K | Electroporation kit containing tips, tubes, buffer R and E |
| Neon Transfection System | Invitrogen | MPK5000 | _ |
| NetStart 1.0 | _ | _ | https://services.healthtech.dtu.dk/services/NetStart-1.0/ |
| Nuclease-free Water | Synthego | _ | _ |
| PCR primer (EGFP) | Eurofins | _ | Fw : GGAATGCAAGGTCTGTTGAATG ; Rev : CACCTTGATGCCGTTCTTCT |
| PCR primer (SAMHD1) | Eurofins | _ | Fw : CGGGATTGATTTGAGGACGA ; Rev : GGGTGGCAAGTTAGTGAAGA |
| Penicillin-streptomycin (10,000 U/mL) | Gibco | 15140122 | _ |
| PFA | Electron Microscopy Sciences | 15714 | _ |
| PMA | Sigma-Aldrich | P8139 | _ |
| PrimerQuest | IDT | _ | https://eu.idtdna.com/pages/tools/primerquest |
| QIAquick PCR Purification Kit | Qiagen | 28104 | _ |
| QuickExtract DNA Extraction Solution | Biosearch Technologies | QE09050 | _ |
| RPMI 1640, GlutaMAX | Gibco | 61870010 | _ |
| SnapGene Viewer | Dotmatics | _ | Version 7 |
| SpCas9 2NLS Nuclease | Synthego | _ | _ |
| SYBR Safe DNA Gel Stain | Invitrogen | S33102 | _ |
| Synthetic sgRNA (EGFP) | Synthego | _ | #1 : CGCGCCGAGGUGAAGUUCGA ; #2 : UUCAAGUCCGCCAUGCCCGA ; #3 : CAACUACAAGACCCGCGCCG |
| Synthetic sgRNA (SAMHD1) | Synthego | _ | #1 : AUCGCAACGGGGACGCUUGG ; #2 : GCAGUCAAGAACCUCGGCGC ; #3 : CCAUCCCGACUACAAGACAU |
| Syringe Plastipak Luer Lock | BD | 301229 | _ |
| T100 Thermal Cycler | BIO-RAD | 1861096 | _ |
| T7 endonuclease I | New England Biolabs | M0302S | _ |
| TAE buffer UltraPure, 10x | Invitrogen | 15558026 | 400 mM Tris-Acetate, 10 mM EDTA |
| THP-1 cells | ATCC | TIB-202 | _ |
| Trypsin-EDTA (0,05 %) | Gibco | 25300054 | _ |
| ZE5 Cell Analyzer | BIO-RAD | 12014135 | _ |
References
- Tsuchiya, S., et al. Establishment and characterization of a human acute monocytic leukemia cell line (THP-1). Int J Cancer. 26 (2), 171-176 (1980).
- Danis, V. A., Millington, M., Hyland, V. J., Grennan, D. Cytokine production by normal human monocytes: inter-subject variation and relationship to an IL-1 receptor antagonist (IL-lRa) gene polymorphism. Clin Exp Immunol. 99, (1995).
- Bol, S. M., et al. Donor variation in in vitro HIV-1 susceptibility of monocyte-derived macrophages. Virology. 390 (2), 205-211 (2009).
- Appleby, L. J., et al. Sources of heterogeneity in human monocyte subsets. Immunol Lett. 152 (1), 32-41 (2013).
- O'Neill, M. B., et al. Single-cell and bulk RNA-sequencing reveal differences in monocyte susceptibility to Influenza A virus infection between Africans and Europeans. Front Immunol. 12, 768189 (2021).
- Wen, Y., et al. Comparability study of monocyte-derived dendritic cells, primary monocytes, and THP1 cells for innate immune responses. J Immunol Methods. 498, 113147 (2021).
- Inagaki, Y., et al. Interferon-g-induced apoptosis and activation of THP-1 macrophages. Life Sci. 71 (21), 2499-2508 (2002).
- Boonkaewwan, C., Toskulkao, C., Vongsakul, M. Anti-inflammatory and immunomodulatory activities of stevioside and its metabolite steviol on THP-1 cells. J Agric Food Chem. 54 (3), 785-789 (2006).
- Chanput, W., et al. β-Glucans are involved in immune-modulation of THP-1 macrophages. Mol Nutr Food Res. 56 (5), 822-833 (2012).
- Cui, J., et al. USP3 inhibits type I interferon signaling by deubiquitinating RIG-I-like receptors. Cell Res. 24 (4), 400-416 (2014).
- Chen, S., et al. SAMHD1 suppresses innate immune responses to viral infections and inflammatory stimuli by inhibiting the NF-κB and interferon pathways. Proc Natl Acad Sci USA. 115 (16), E3798-E3807 (2018).
- Pradhananga, S., Spalinskas, R., Poujade, F. A., Eriksson, P., Sahlén, P. Promoter anchored interaction landscape of THP-1 macrophages captures early immune response processes. Cell Immunol. 355, 104148 (2020).
- Mezzasoma, L., Talesa, V. N., Romani, R., Bellezza, I. Anp and BNP exert anti-inflammatory action via npr-1/cgmp axis by interfering with canonical, non-canonical, and alternative routes of inflammasome activation in human THP1 cells. Int J Mol Sci. 22 (1), 1-17 (2021).
- Martinat, C., et al. SUMOylation of SAMHD1 at Lysine 595 is required for HIV-1 restriction in non-cycling cells. Nat Commun. 12 (1), 4582 (2021).
- Rensen, E., et al. Clustering and reverse transcription of HIV-1 genomes in nuclear niches of macrophages. EMBO J. 40 (1), e105247 (2021).
- Ikeda, T., et al. APOBEC3 degradation is the primary function of HIV-1 Vif determining virion infectivity in the myeloid cell line THP-1. mBio. 14 (4), e0078223 (2023).
- Jinek, M., et al. A programmable dual RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337, 816-821 (2012).
- Kurosaki, T., Maquat, L. E. Nonsense-mediated mRNA decay in humans at a glance. J Cell Sci. 129 (3), 461-467 (2016).
- Tuladhar, R., et al. CRISPR-Cas9-based mutagenesis frequently provokes on-target mRNA misregulation. Nat Commun. 10 (1), 4056 (2019).
- Embree, C. M., Abu-Alhasan, R., Singh, G. Features and factors that dictate if terminating ribosomes cause or counteract nonsense-mediated mRNA decay. J Biol Chem. 298 (11), 102592 (2022).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Schnoor, M., et al. Efficient non-viral transfection of THP-1 cells. J Immunol Methods. 344 (2), 109-115 (2009).
- Tang, X., et al. A method for high transfection efficiency in THP-1 suspension cells without PMA treatment. Anal Biochem. 544, 93-97 (2018).
- Ji, W., Zhang, L., Liu, X. Protocol using lentivirus to establish THP-1 suspension cell lines for immunostaining and confocal microscopy. STAR Protoc. 4 (1), 102032 (2023).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, (2023).
- . Synthego Performance Analysis, ICE Analysis Available from: https://www.synthego.com/help/citing-ice (2019)
- Salamov, A. A., Nishikawa, T., Swindells, M. B. Assessing protein coding region integrity in cDNA sequencing projects. Bioinformatics. 14, 384-390 (1998).
- Pedersen, A. G., Nielsen, H. Neural network prediction of translation initiation sites in eukaryotes: Perspectives for EST and genome analysis. Proc Int Conf Intell Syst Mol Biol. 5, 226-233 (1997).
- Concordet, J. P., Haeussler, M. CRISPOR: Intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46 (W1), W242-W245 (2018).
- Laguette, N., et al. SAMHD1 is the dendritic- and myeloid-cell-specific HIV-1 restriction factor counteracted by Vpx. Nature. 474 (7353), 654-657 (2011).
- Hrecka, K., et al. Vpx relieves inhibition of HIV-1 infection of macrophages mediated by the SAMHD1 protein. Nature. 474 (7353), 658-661 (2011).
- Seki, A., Rutz, S. Optimized RNP transfection for highly efficient CRI SPR/Cas9-mediated gene knockout in primary T cells. J Exp Med. 215 (3), 985-997 (2018).
- Batista Napotnik, T., Polajžer, T., Miklavčič, D. Cell death due to electroporation - A review. Bioelectrochemistry. 141, 107871 (2021).
- Perretta-Tejedor, N., Freke, G., Seda, M., Long, D. A., Jenkins, D. Generating mutant renal cell lines using CRISPR technologies. Methods Mol Biol. 2067, 323-340 (2020).
- Sentmanat, M. F., Peters, S. T., Florian, C. P., Connelly, J. P., Pruett-Miller, S. M. A survey of validation strategies for CRISPR-Cas9 editing. Sci Rep. 8 (1), 888 (2018).
- Makino, S., Fukumura, R., Gondo, Y. Illegitimate translation causes unexpected gene expression from on-target out-of-frame alleles created by CRISPR-Cas9. Sci Rep. 6, 39608 (2016).
- Bowling, A., et al. Downstream alternate start site allows N-terminal nonsense variants to escape NMD and results in functional recovery by readthrough and modulator combination. J Pers Med. 12 (9), 1448 (2022).
- Inácios, &. #. 1. 9. 4. ;., et al. Nonsense mutations in close proximity to the initiation codon fail to trigger full nonsense-mediated mRNA decay. J Biol Chem. 279 (31), 32170-32180 (2004).
- Li, J., et al. Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9. J Mol Cell Biol. 7 (4), 284-298 (2015).
- Guo, Y., et al. CRISPR inversion of CTCF sites alters genome topology and enhancer/promoter function. Cell. 162 (4), 900-910 (2015).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved