Method Article
THP-1 细胞中基于电穿孔的 CRISPR-Cas9 介导的基因敲除和单细胞克隆分离
摘要
THP-1 细胞系被广泛用作研究人类单核细胞/巨噬细胞在各种生物学相关研究领域功能的模型。本文介绍了一种基于 CRISPR-Cas9 的高效工程和单细胞克隆分离方案,能够产生可靠且可重复的表型数据。
摘要
人急性单核细胞白血病 (AML) THP-1 细胞系被广泛用作研究人单核细胞衍生巨噬细胞功能的模型,包括它们与人类免疫缺陷病毒 (HIV) 等重要人类病原体的相互作用。与其他来源于骨髓的永生化细胞系相比,THP-1 细胞保留了许多完整的炎症信号通路,并显示出更接近原代单核细胞的表型特征,包括在用佛波醇-12-肉豆蔻酸酯 13-乙酸酯 (PMA) 处理时分化为巨噬细胞的能力。使用 CRISPR-Cas9 技术通过靶向基因敲除 (KO) 来改造 THP-1 细胞,为更好地表征免疫相关机制(包括病毒与宿主相互作用)提供了一种强大的方法。本文介绍了一种基于高效 CRISPR-Cas9 的工程方案,使用电穿孔将预组装的 Cas9:sgRNA 核糖核蛋白递送到细胞核中。如T7核酸内切酶I测定所评估的那样,使用多个sgRNA靶向略有不同位置的同一位点会导致大DNA片段的缺失,从而提高编辑效率。通过 Sanger 测序验证了 CRISPR-Cas9 介导的基因水平编辑,然后进行了 CRISPR 编辑推断 (ICE) 分析。通过免疫印迹结合功能测定来确认蛋白质耗竭。使用该方案,在目标基因座中实现了高达 100% 的插入缺失和超过 95% 的蛋白质表达降低。高编辑效率使得通过限制稀释来方便地分离单细胞克隆。
引言
THP-1 是从急性白血病 (AML) 患者中分离的人单核细胞衍生细胞系,其表型特征与原代单核细胞非常相似1。与原代单核细胞衍生的巨噬细胞相比,原代单核细胞衍生的巨噬细胞不会分裂,并且表现出有限的寿命和供体间/供体内表型的变异性,THP-1 细胞几乎可以永久培养,并且具有更均匀的行为,有利于结果的可重复性 2,3,4,5,6.值得注意的是,THP-1 细胞可以通过佛波醇-12-肉豆蔻酸酯 13-乙酸酯 (PMA) 分化为巨噬细胞样表型,使其成为一种广泛使用的体外模型,用于研究单核细胞/巨噬细胞对炎症信号的反应 7,8,9,10,11,12,13 或临床相关人类病原体(包括 HIV14)感染 14,15,16。对 THP-1 细胞进行基因工程改造的可能性在许多生物学相关研究领域都引起了人们的兴趣。
成簇的规则间隔短回文重复序列-CRISPR 相关蛋白 9 (CRISPR-Cas9) 是一种原核适应性免疫系统,在 RNA 引导的核酸酶上传递以降解入侵的病毒基因组,该基因组已被重新编程为基因工程工具17。基因组编辑的过程分三个步骤进行:识别、切割和修复。单向导 RNA (sgRNA) 通过与 20 bp 向导序列的碱基配对,将 Cas9 核酸酶募集到特定的基因组位点。20 bp 基因组靶序列的 3' 直接存在原始间隔区相邻基序 (PAM) 序列会触发 Cas9 介导的 17 和 18 位之间两条 DNA 链(PAM 的 3 bp 5')的解旋和切割。由此产生的双链断裂 (DSB) 由两条主要修复途径处理。在没有与受损基因座具有同源性的修复模板的情况下,易出错的非同源末端连接 (NHEJ) 通路将引入随机核苷酸插入和/或缺失 (插入缺失),可能导致移码突变和/或引入过早终止密码子 (PTC)。反过来,含 PTC 的 mRNA 通过无义介导的 mRNA 衰变 (NMD) 途径降解成为靶标,最终破坏蛋白质表达/功能 18,19,20。或者,模板依赖性同源定向修复 (HDR) 通路可以作并忠实修复 DSB。这种机制已被用于实现精确的基因编辑,包括敲入和碱基替换。值得注意的是,细胞周期状态是影响 DSB 修复途径选择的重要因素。事实上,NHEJ 在细胞周期的所有阶段都具有活性,而 HDR 主要局限于 S/G2 期21。
THP-1 细胞在悬浮液中生长,众所周知,很难用质粒 DNA 转染,这一过程也可能改变其活力和/或分化能力22,23。使用编码 Cas9 和 sgRNA 的基于 HIV-1 的慢病毒载体进行转导通常用于敲除 (KO) 目标基因24。将 Cas9/sgRNA 盒整合到细胞基因组中可确保延长表达和高效 KO,但也是脱靶效应的持续来源25。或者,预组装的 Cas9:sgRNA 核糖核蛋白 (RNP) 通过电穿孔递送,该方法涉及在施加电脉冲时在质膜和核膜中临时形成孔。在采用这种方法时,保持细胞活力是一个重要的挑战。
在这里,生产了稳定表达 GFP (THP-1_GFP) 的 THP-1 细胞系,作为建立实现基于 CRISPR-Cas9 的高效编辑方案的工具。在设计了同时使用三个 sgRNA 灭活 EGFP 基因的策略(多向导方法)后,使用 GFP 表达作为读数确定了几种电穿孔条件下的 KO 效率。同时监测细胞增殖。通过 T7 核酸内切酶 I (T7EI) 测定和 Sanger 测序确认基因编辑,然后使用 CRISPR 编辑推断 (ICE) 算法进行分析26。随着 THP-1 细胞在电穿孔后恢复正常生长速率,导致高达 95% GFP 表达降低的参数被成功用于灭活内源基因 (SAMHD1) 并产生单细胞 THP-1 克隆。
研究方案
本研究中使用的试剂和设备的详细信息列在 材料表中。
1. 使用 CRISPOR 进行指导设计(图 1.1)
注:SnapGene Viewer 软件可用于步骤 4、7 和 10 中,以注释编辑靶位点和目标基因内 PCR 引物杂交的位置。
- 转到 Ensembl 网站 (www.ensembl.org)。在 Search 框中,选择一个物种并输入目标基因的名称。点击 Go。选择与基因对应的结果(不是转录本)。
- 单击 Show transcript table(显示转录表),然后选择与 Biotype 列中的金色标记 Protein coding(蛋白质编码)相对应的转录本 ID。进入成绩单页面后,单击左侧菜单中的 Exons。
- 向下滚动并单击 Download sequence (下载序列)。确保 File format 为 FASTA。在 Settings - Included Sequences (设置 - 包含的序列) 中,取消选择除 Genomic sequence 之外的所有内容。
在 脚本框的两端的 Flanking sequence 中输入数字 “500”。单击 Preview,选择整个序列(仅选择没有标题的核苷酸),然后复制它 (Ctrl+C)。 - 打开 SnapGene 查看器并单击 New > DNA file(新建 DNA 文件)。将序列 (Ctrl+V) 粘贴到 Create a sequence 框中。取消选中 Detect common features ,然后选择 Linear in Topology。
重命名文件并单击 Create。在左侧菜单中,取消选择 Show enzymes (第一个图标)。在窗口底部,选择 Sequence 选项卡。
注:此步骤允许检索完整的基因序列,包括外显子、内含子和 500 bp 侧翼序列(可选)。后一种信息可用于设计用于扩增位于第一个外显子内的靶位点的 PCR 引物。 - 返回 Ensembl 网站,在文件预览上向上滚动,然后单击 Back(返回)。现在,将 文件格式 更改为 RTF。在 设置 - 包含的序列 中,取消选择除 Exons 之外的所有内容。在 Show variants (显示变体) 中,选择 No (否)。单击页面顶部的 Download(下载 )。
- 打开下载的文件(带有 Word),其中包含外显子序列并以蓝色显示编码序列,从初始 ATG 开始。选择要由 CRISPR-Cas9 定向编辑靶向的外显子(请参阅下面的建议),选择它,然后复制它 (Ctrl+C)。
- 目标外显子是早期外显子或编码蛋白质功能重要结构域的外显子。值得注意的是,在靠近 3' UTR 的晚期外显子中建立 PTC 可能无法引发 NMD,从而导致 C 端截短蛋白的表达。相反,在天然起始位点近端的早期外显子中引入 PTC 与非法翻译(ITL,又名选择性翻译起始 (ATI))的风险有关,从而产生 N 末端截短蛋白的意外表达,该蛋白从第一个 ATG 密码子下游的框内翻译起始位点 (TIS) 开始。为了减轻后一种风险,建议使用 ATGpr27 (atgpr.dbcls.jp) 和/或 NetStart 1.028 (services.healthtech.dtu.dk/services/NetStart-1.0/) 评估替代 TIS 的发生。
- 确保目标外显子包含编码序列。然而,选择初始 ATG 上游 5'UTR 区的 sgRNA 退火以将其包含在缺失的片段中可能证明是有用的。
- 理想情况下,靶向外显子应该是该基因的所有蛋白质编码转录本变体共有的。通过搜索目标基因,在 NCBI 的 Genome Data Viewer (www.ncbi.nlm.nih.gov/gdv/) 上检查是否是这种情况。
单击显示窗口中的基因名称(绿色)将显示转录本变体(紫色)。外显子由矩形表示。
- 在 SnapGene 查看器中,按 Ctrl+F、Ctrl+V,然后按 Enter 键搜索外显子序列。按 Ctrl+T 添加新特征,为其命名,将类型更改为“exon”,然后单击 “确定”。
- 转到 CRISPOR 网站 (http://crispor.gi.ucsc.edu/) 并粘贴步骤 1 中的外显子序列。首先,在 第 2 步 中选择一个参考基因组,然后在 第 3 步中选择要靶向的 PAM 类型,通常为 SpCas9 的 20 bp-NGG。单击 SUBMIT(提交)。
- 选择两个 sgRNA,使它们之间最多相隔 150 bp,然后在两者之间选择第三个 sgRNA。以下是 sgRNA 选择的一些指南:
- MIT 特异性评分与脱靶效应相关。分数越高表示潜在的脱靶越少。右列显示三个预测的脱靶位点,从最不可能到最不可能排序,以及它们的位置(在外显子、内含子或基因间区域)。单击 Show all 可访问预测的脱靶站点的完整列表。如果可能,选择 MIT 评分为 >80 的 sgRNA,优先考虑那些没有脱靶的 0、1 或 2 错配的 sgRNA。此外,应避免使用外显子中具有脱靶的 sgRNA,这些 sgRNA 最有可能影响表型。
- 有关预测疗效,请参阅 Doench '16 评分。请注意,高 Doench '16 分数仅表明 sgRNA 更有可能有效。实际疗效必须通过实验确定。因此,选择多个 sgRNA 总是有用的,即使它们不打算一起使用。
- GC 含量过高或过低以及某些基序都可能对 sgRNA 效率有害,应避免使用。CRISPOR 突出显示了这些参数。
- 重复步骤 1.7 将 sgRNA 序列和相关的 PAM 序列添加到 SnapGene Viewer 中的基因序列中。同时,将 sgRNA 序列(不含 PAM)粘贴到文本或 Excel 文件中,以保留订购寡核苷酸时所需的 5'-3' 方向。
- 对于靶位点的基于 PCR 的扩增,请围绕它设计几个引物。扩增子大小应在 800 到 1000 bp 之间。PrimerQuest 用于设计该方案的引物 (https://eu.idtdna.com/pages/tools/primerquest)。或者,CRISPOR 提供了一系列引物来扩增目标基因组区域以及脱靶位点。显示潜在脱靶位点的完整列表(步骤 1.9.1)后,单击右下角的脱靶引物 。
2. 用于电穿孔的试剂和细胞制备(图 1.2)
- 准备一个 24 孔培养板,通过在电穿孔后用 500 μL 补充有 20% 热灭活(56 °C,30 分钟)、过滤 (0.20 μm) 胎牛血清 (FBS) 的 RPMI 1640 培养基填充孔来回收细胞。不要添加抗生素。将板保存在 37 °C 和 5% CO2 的加湿培养箱中 24 小时,直至电穿孔。
- 如果 sgRNA 是干燥运输的,则用 TE 缓冲液(10 mM Tris、1 mM EDTA,pH 8.0)再水化至终浓度为 100 μM(即 每 1 nmol sgRNA 10 μL TE 缓冲液)。涡旋30秒,在4°C下孵育过夜以完全再水化,并在短暂的移液器匀浆后,将sgRNA储备液储存在-20°C。 根据最终体积,分装以避免多次冻融循环。
- 通过在无核酸酶的水中稀释 100 μM 储备液,制备终浓度为 30 μM 的工作 sgRNA 溶液。涡旋 30 秒,并在室温下孵育 5 分钟。
- 通过在电穿孔试剂盒中包含的 3.5 μL 重悬缓冲液 R 中同时稀释 1 μL 三个 30 μM sgRNA 和 0.5 μL 20 μM Cas9 溶液,以 1:9 的摩尔比组装 Cas9:sgRNA RNP(最终体积为 7 μL,对于一个实验条件;相应地缩放)。短暂涡旋并在室温下孵育 5 分钟。
- 同时,通过将 0.5 μL 的 20 μM Cas9 添加到 6.5 μL 的重悬缓冲液 R. Vortex 中短暂漩涡并在室温下孵育 5 分钟来制备未经编辑的对照。
- 向所有样品中加入 5 μL 重悬缓冲液 R,每种电穿孔条件的最终体积为 12 μL。
- 准备用于电穿孔的 THP-1 细胞。
- 通过台盼蓝排除试验评估浓度和活力。在 0.4% 台盼蓝染色溶液中以 1:2 稀释细胞。孵育 30 秒后,充分匀浆,并在计数室的壁中加入 10 μL,采用 Neubauer 改进的网格样式。计数三个大方块,将计数除以 100 得到细胞浓度 (x106 个细胞/mL)。
注意:细胞的健康状况会影响它们对电穿孔的敏感性。应注意在最佳条件下进行细胞培养。必须限制在培养箱外的时间,并且应提前准备好所有试剂和溶液并预热。 - 对于每种条件,收集相当于 0.2 x 106 个细胞的体积并离心(336 x g ,5 分钟,20 °C)。
- 使用移液管吸出上清液,并将沉淀重悬于 500 μL PBS 中。再次离心(336 x g ,5 分钟,20 °C)。
- 使用移液管小心吸出上清液,并用 12 μL RNP 溶液重悬 THP-1 细胞沉淀(步骤 2.6)。
- 通过台盼蓝排除试验评估浓度和活力。在 0.4% 台盼蓝染色溶液中以 1:2 稀释细胞。孵育 30 秒后,充分匀浆,并在计数室的壁中加入 10 μL,采用 Neubauer 改进的网格样式。计数三个大方块,将计数除以 100 得到细胞浓度 (x106 个细胞/mL)。
3. 电穿孔系统设置和成核转染(图 1.3)
- 将移液器站放在生物安全柜下,将电穿孔管放在支架中,并加入 3 mL 缓冲液 E,包含在电穿孔试剂盒中。
- 打开电穿孔设备后,使用触摸屏设置以下电穿孔参数: 电压 = 1 500 V,持续时间 = 10 ms,数字 = 3。
- 为电穿孔移液器配备吸头并吸出 10 μL 的 RNP/THP-1 溶液(步骤 2.7.4)。将移液器插入电穿孔管中,然后按电穿孔设备屏幕上的 Start(开始 )。等待消息 Complete 出现,然后从试管中取出移液器。
- 将细胞转移到预热的 24 孔板的孔中,并轻轻匀浆。将板放回加湿的培养箱中,让它们不受干扰地静置 72 小时。
注意:移液 RNP/THP-1 悬液时注意不要产生任何气泡,因为它们会干扰电穿孔程序。如果在没有可见电弧的情况下出现错误消息,请从试管中取出电穿孔移液器,然后再次按下 Start(开始 )。但是,如果观察到短暂明亮火花形式的电弧,则可能表明存在气泡。即使没有错误消息,电穿孔程序也可能会失败。
4. THP-1 回收率电穿孔后 72 小时(图 1.4)
- 电穿孔后 72 小时对细胞进行计数以评估浓度(步骤 2.7.1)。
- 如果有足够的细胞(即 ≥0.6 x 106 个细胞/mL),用补充有 20% FBS 和 1% 青霉素-链霉素的 RPMI 将它们稀释至少 2 倍,并使浓度在 0.3-0.5 x 106 个细胞/mL 之间。否则,让细胞再恢复 72 小时。
- 传代并扩增细胞,直到有足够的细胞进行 KO 验证。同时,可以开始分离单细胞克隆(步骤 7)。
5. 通过 T7EI 错配测定进行基因编辑验证(图 1.5)
注意:鉴于 T7EI 识别大于 1 bp 的错配,该测定可能低估了编辑效率。因此,除非经过适当修饰,否则 T7EI 测定对于筛选纯合细胞群(即单细胞克隆)没有用(步骤 5.7)。
- 评估细胞浓度(步骤 2.7.1)并在 1.5 mL 试管中取出相当于 0.1 x 106 个细胞的体积。离心(336 x g,5 分钟,20 °C),吸出上清液,并将沉淀重悬于 500 μL PBS 中。再次离心,并使用移液管吸出尽可能多的上清液,而不会干扰沉淀。快速冷冻样品并储存在 -20 °C。
- 提取基因组 DNA 作为 PCR 扩增的基质。
- 用 50 μL DNA 提取溶液重悬沉淀,匀浆,并在 0.2 mL PCR 管中转移整个体积。涡旋并短暂离心(脉冲 3 秒)。
- 将试管放入热循环仪中,在 65 °C 下加热 15 分钟,然后在 98 °C 下加热 10 分钟。
- 用 90 μL 超纯水稀释提取的 DNA。涡旋并短暂离心(5,000 x g ,3 s)。
- 用超纯水稀释 PCR 引物(参见 材料表), 终浓度为 10 μM(即 10 pmol/μL)。
- 在 0.2 mL PCR 管中制备 PCR 混合物(按照下面的注释)(对于一种条件,最终体积 = 50 μL)。通常,至少有两个条件:KO 和仅使用 Cas9 的未编辑控件。
注:纯化的基因组 DNA:10 μL;正向和反向引物 (10 μM):各 2.5 μL,终浓度为 500 nM;反应缓冲液 (5x):10 μL。添加前充分涡旋。混合 dNTP(每种 25 mM):0.6 μL,每种 dNTP 的最终浓度为 0.3 mM。DNA 聚合酶:0.5 μL;超纯水:23.9 μL。涡旋并短暂离心(脉冲 3 秒)。 - 将试管放入热循环仪中,并使用以下设置运行 PCR 程序:
注:一个循环在 95 °C 下 5 分钟,然后是 30 个循环 [98 °C 20 秒(变性步骤),X °C 15 秒(退火步骤),72 °C 45 秒(伸长步骤)],然后在 72 °C 下最后一个循环 2 分钟。扩增结束时,取出试管,涡旋并短暂离心(脉冲 3 秒)。退火温度 (X) 是引物的熔解温度 (Tm) 减去 5 °C。 - 对于多克隆编辑群体:在新的 0.2 mL 试管中,加入 17.5 μL PCR 扩增子和 2 μL NEBuffer 2 (10x),最终体积为 19.5 μL。涡旋并短暂离心(脉冲 3 秒)。或者,为了筛选单细胞克隆,将来自编辑和未编辑对照细胞的 PCR 产物以 1:1 的比例混合(步骤 5.6)(补充图 1)。
- 对于异源双链体形成,将试管放入热循环仪中并运行以下程序:一个在 95 °C 下循环 10 分钟,一个在 95 °C 至 85 °C 下以 -2 °C/s 的斜坡,一个从 85 °C 到 25 °C 的斜坡为 -0.3 °C/s,并在 10 °C 下进行一次最终冷却循环 HOLD。
- 异源双链体形成后,向试管中加入 0.5 μL T7EI 溶液。在 37 °C 下孵育 30 分钟。
- 准备 1.2% 琼脂糖凝胶:
- 在玻璃瓶中称量琼脂糖,并加入适当体积的 1x TAE 缓冲液(40 mM Tris-乙酸,1 mM EDTA,pH 8.3)。将其放入微波炉中,注意不要拧紧盖子。加热数次,直到琼脂糖晶体完全溶解。
注意:如果需要,混合溶液。不要让它沸腾,否则体积会减少,从而改变琼脂糖浓度。 - 加入以 1/20,000 稀释的 DNA 凝胶染料并充分混合。
- 将琼脂糖溶液倒入模具中,加入梳子,在室温下凝固。
- 在玻璃瓶中称量琼脂糖,并加入适当体积的 1x TAE 缓冲液(40 mM Tris-乙酸,1 mM EDTA,pH 8.3)。将其放入微波炉中,注意不要拧紧盖子。加热数次,直到琼脂糖晶体完全溶解。
- 通过将 5 μL 的 T7EI 消化产物(步骤 5.9)或未消化的 PCR 产物与 5 μL 水和 2 μL 的 6x DNA 上样染料混合来制备用于凝胶电泳的样品。加载样品和分子量标准,并在 80 V 下迁移 45 分钟(时间可根据 DNA 片段的预期大小进行调整)。迁移结束后,使用适当的成像系统获取凝胶的图像。
6. 通过 Sanger 测序分析进行基因编辑验证(图 1.6)
- 为了表征基因修饰,纯化 PCR 产物(步骤 5.4),并在 Sanger 测序后,使用 ICE 工具 (https://www.synthego.com/products/bioinformatics/crispr-analysis) 分析结果。
- 控制在修饰后是否可以生产蛋白质。
- 下载 ICE 结果并打开 contribs.txt 文件。
- 将编辑后的序列与 WT 对应序列进行比较。绘制插入缺失并验证它们是否出现在目标基因组区域。评估他们的规模。如果插入缺失长度不是 3 的倍数,则会发生移码,并且可能会引入 PTC。预计突变的 mRNA 将经历 NMD 介导的降解。
7. 通过有限稀释分离单细胞克隆(图 1.7)
注:单细胞克隆的分离不是强制性的。然而,如果选择这样做,则重要的是表征多个克隆并将其表型与原始多克隆群体进行比较。
- 取细胞悬液样品,用培养基稀释 1/2,并评估浓度(步骤 2.7.1)。稀释可提高计数准确性。
- 执行 2 至 3 个连续稀释步骤,以达到 7 个细胞/mL 的浓度。在圆底 96 孔板中每孔分配 100 μL 细胞悬液(即每孔 0.7 个细胞)。让细胞倒出几个小时,然后在显微镜下观察板以识别含有一个细胞的孔。
- 定期监测集落形成和生长,并在需要时将细胞转移到更大的培养板或培养瓶中。
8. 通过 HIV-1 限制性试验对 THP-1 KO SAMHD1 细胞进行功能表征
- 将 THP-1 接种在 24 孔培养板中,每孔 0.25 x 106 个细胞,溶于 300 μL 补充有 10% FBS、1% 青霉素-链霉素(完全 RPMI)并含有 300 ng/mL PMA 的 RPMI 中用于分化。将板保存在加湿培养箱(37°C,5% CO2 )中 24 小时。
- 用不含 PMA 的完全 RPMI 培养基替换培养基,并将板放回培养箱中再放置 24 小时。
- 使用真空泵吸出所有培养基。加入 250 μL VSVg 假型 HIV-1-GFP 病毒原液(MOI = 0.5 IFU/细胞)。包括未感染的对照。将板在 4 °C 下放置 2 小时以同步感染。
- 用 RPMI 洗涤细胞一次。向每个孔中加入 500 μL 完全 RPMI,并在标准条件下孵育 48 小时(时间可以调整)。
- 去除培养基后,用冷的 1x PBS 洗涤细胞一次。然后,在每个孔中加入 100 μL 0.05% 胰蛋白酶-EDTA。将板置于 37 °C 的培养箱中,直到细胞完全分离(5 分钟就足够了),然后加入 200 μL 完全 RPMI 以灭活酶。将 250 μL 从每个孔转移到 96 孔圆底板中(确保流式细胞仪接受培养板)。
- 离心板(757 x g ,3 分钟,减速 = 6,20 °C)并使用多通道移液器吸出上清液。用 100 μL 4% PFA 重悬沉淀,并在 4 °C 下孵育 10 分钟。加入 100 μL 冷的 1X PBS。
- 通过流式细胞术分析以 GFP 阳性细胞表示的感染率(有关建议的门控策略,请参见 补充图 2 )。
结果
稳定产生表达 GFP 报告蛋白 (THP-1_GFP) 的 THP-1 细胞系 (图 2A),并用作建立高效 CRISPR-Cas9 介导基因编辑方案的工具。为此,使用 CRISPOR 网络工具29 设计了靶向 EGFP 基因的 3 个 sgRNA(图 2B),它们以 9:1 的摩尔比同时与 Cas9 复合形成 RNP,然后使用不同的设置通过电穿孔递送到细胞中。接下来,细胞在单独的含有 20% FBS 的 RPMI 培养基中生长(图 2C,上图)或用条件培养基以 1:1 稀释(图 2C,下图)。随着时间的推移监测细胞增殖和 GFP 表达,作为 EGFP KO 效率的读数。在几种情况下,GFP 阳性细胞的百分比急剧下降,在电穿孔 (pe) 后第 7 天达到 >90%。在第 3 天 pe,细胞数量减半,可能是由于电穿孔对细胞活力的负面影响。然而,细胞数量再次增加,到第 7 天 pe 时倍增时间恢复到正常速率(图 2C,上图)。在我们的实验条件下,使用条件培养基不利于细胞回收(图 2C,下图)。基于这些结果,选择了三个 1500 V 的 10 ms 脉冲进行后续实验。
然后在基因组水平上表征 CRISPR-Cas9 介导的 EGFP KO。为此,提取 THP-1_GFP 细胞的基因组 DNA,用 Cas9:sgRNA RNP(编辑)或单独的 Cas9(未编辑)进行电穿孔,并用作模板对包含靶位点的 891 bp 区域进行 PCR 扩增。缺乏 EGFP 的亲本 THP-1 细胞也作为阴性对照。接下来,制备编辑后的 THP-1_GFP 细胞的 PCR 产物,用于通过 T7EI 测定进行错配检测,然后通过琼脂糖凝胶电泳分离 DNA 片段。对于未编辑的 THP-1_GFP 细胞,对应于 WT 扩增子预期大小的 ~900 bp 条带很容易可视化,但对于亲本 THP-1 对照则不然(图 2D,比较第 2 行和第 3 行)。在编辑的 THP-1_GFP 细胞的情况下,~900 bp 条带变得无法检测到,并被迁移速度更快的条带所取代,其大小与 CRISPR 编辑导致的 ~75 bp 片段丢失相匹配(图 2D,第 4 行)。这种条带模式在 T7EI 消化时发生改变,导致出现更小的 DNA 片段,进一步证实了靶位点的成功编辑(图 2D,第 5 行)。为了深入了解编辑效率和编辑群体的特定基因型,使用 Sanger 方法对 PCR 扩增子进行测序,然后使用 ICE 工具26 进行分析。未编辑的对照样品并行处理。我们检索到 100% 的插入缺失和 KO 评分,分别对应于编辑样本中非 WT 序列的百分比和可能导致功能性 KO 的插入缺失的比例(图 2E,左)。对插入缺失大小谱和频率的详细分析表明,存在两个携带 62 bp 或 76 bp 缺失的优势群体,分别占总序列的 57% 或 37%(图 2E,右)。这些发现表明,sgRNA #1 与 sgRNA #2 或 #3 结合,触发了靶基因中的并发 DSB(图 2B)。
然后应用该方案灭活内源性 SAMHD1 编码基因(图 3A)。SAMHD1 KO THP-1 多克隆细胞系的表征包括监测细胞增殖随时间的变化,这与未编辑的 THP-1 细胞相匹配(图 3B)。通过基于 PCR 的靶序列扩增来评估基因编辑的结果,与仅 Cas9 对照相比,SAMHD1 KO 细胞的条带更小(图 3C,比较第 2 行和第 4 行),表明 DNA 序列丢失。T7EI 测定证实了这些观察结果,显示 SAMHD1 KO 细胞的切割产物出现,但并非未经编辑的细胞(图 3C,比较第 3 行和第 5 行)。使用 ICE 工具对 Sanger 测序数据进行分析,得到的插入缺失和 KO 评分分别为 100% 和 97%。SAMHD1 基因座内的主要修饰分别为 46%、13% 和 10% 的序列中的 170 bp、93 bp 或 104 bp 序列缺失(图 3D)。还通过免疫印迹评估粗细胞裂解物中的蛋白表达水平。与内源性 SAMHD1 大小一致的 ~70 kDa 条带在编辑后变得几乎无法检测到,相当于估计减少 >97%(图 3E)。最后,还通过检测 HIV-1 感染的易感性来评估 SAMHD1 KO THP-1 多克隆细胞系的表型特征。与其众所周知的抗病毒作用30,31 一致,SAMHD1 基因座的失活伴随着 HIV-1 感染率的增加(图 3F)。
同时,通过有限稀释生成单细胞克隆。与未编辑的对照细胞相比,从 SAMHD1 KO 克隆扩增基因组靶位点产生的 PCR 产物具有更高的电泳迁移率(图 4A)。基于 ICE 的 Sanger 测序数据分析显示,除一个(克隆 A2)外,所有选定的单细胞克隆的插入缺失评分为 100%(图 4B,左)。在基因组水平上对编辑结果进行表征,返回克隆 A3 的单个序列,其中 98 bp 缺失位于初始 ATG 下游 28 pb 处,从而导致帧外变化(图 4B,右)。其他克隆包含三个(克隆 B1)或更多序列,表明在有限稀释步骤中存在经历不同编辑事件的等位基因和/或每孔多个细胞(图 4B,右)。值得注意的是,克隆 A4 和 A7 序列中有 11% 在初始 ATG 的上游带有插入缺失,编码序列保持不变。此外,1% 的克隆 A7 序列在初始 ATG 下游 9 bp 处缺乏 123 bp 片段,导致框内缺失 41 个氨基酸。与这些发现一致,功能研究表明,具有 SAMHD1 基因座破坏的克隆 (A3、A8、B1、B3),因此缺乏 SAMHD1 表达,对 HIV-1 感染非常容易(图 4C)。相反, SAMHD1 完整的克隆 A4 和 A7 与亲本或未编辑的对照 THP-1 克隆一样难治(图 4C)。综上所述,证实了克隆 A3 中 SAMHD1 基因座的成功编辑。
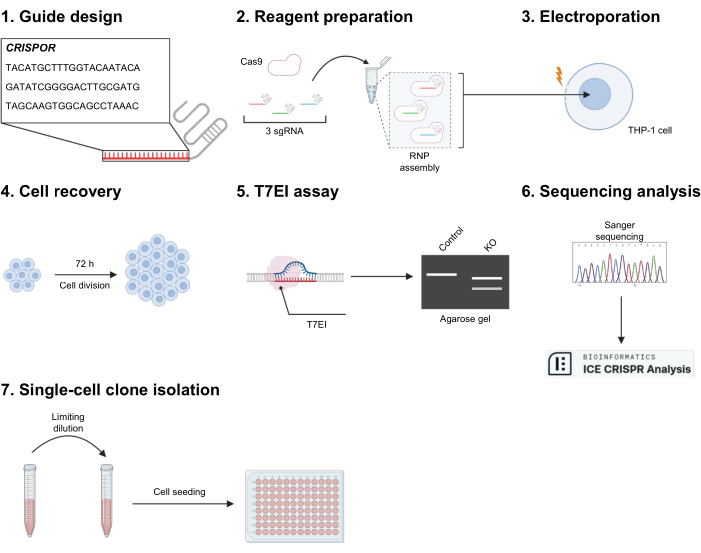
图 1:实验大纲。 (1) CRISPOR 用于生成针对目标外显子的 sgRNA 序列。其中 3 个是根据高目标效能和弱脱靶预测选择的。(2) 将三种 gRNA 与 Cas9 混合以组装核糖核蛋白 (RNP),从而产生三种不同 RNP 的混合物。(3) 对细胞进行电穿孔以允许 RNP 进入。(4) 将细胞转移到 24 孔培养板中,静置至少 72 小时。(5) 首先通过 T7EI 消化测定定性测试基因编辑, (6) 对于经过验证的条件,通过 Sanger 测序和 ICE 分析进一步表征。(7) 如有必要,可以在有限稀释后接种细胞以产生克隆群。 请单击此处查看此图的较大版本。
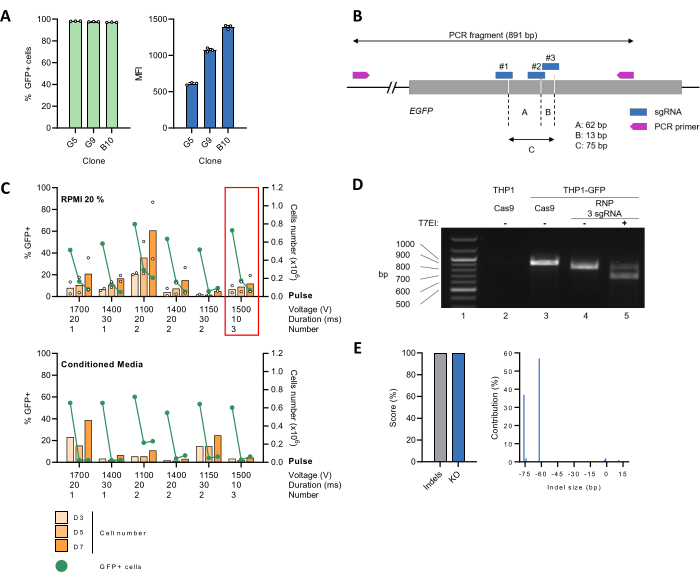
图 2:THP-1 细胞中 EGFP KO 的电穿孔设置和基因编辑验证。 (A) 用编码 EGFP 报告基因的慢病毒载体转导 THP-1 细胞。通过有限稀释然后在培养物中扩增 30 天获得单细胞克隆。流式细胞术分析证实,几乎整个细胞群都表达 GFP。GFP 阳性群体的中位荧光强度 (MFI),指示整合的 EGFP 拷贝数, 在几个细胞克隆中有所不同。选择克隆 G5 进行后续研究。(B) EGFP 基因的示意图,包括 sgRNA 和引物的结合位点。Cas9 切割位点由虚线表示。(C) 未分化的 THP-1_GFP (2 x 105) 细胞与预组装的 Cas9:sgRNA RNP 混合,然后按指定设置进行电穿孔。接下来,将细胞转移到含有 500 μL 预热的含 20% FBS 的 RPMI 培养基的 24 孔板中,或用 48 小时 THP-1 细胞培养物产生的条件培养基以 1:1 稀释。随着时间的推移监测增殖 (橙色条) 和 GFP 阳性细胞的百分比 (绿线)。数据表示 n = 2 个生物学重复的平均值。红框突出显示为后续实验选择的条件。(D) 提取编辑 (RNP 3 sgRNA) 或未编辑 (Cas9) THP-1_GFP 细胞的基因组 DNA,并通过 PCR 扩增靶基因座周围的区域。亲本 THP-1 细胞用作阴性对照。扩增子经历变性/复性循环,导致异源双链体的形成。接下来,通过 T7EI 测定检测错配。通过 1.2% 琼脂糖凝胶电泳分离 DNA 片段。(E) 纯化的 PCR 产物通过 Sanger 方法测序,然后使用 ICE 算法进行分析。Indel 和 KO 评分分别表示样品中非 WT 序列的百分比,以及具有移码或 21+ bp 插入缺失的细胞比例。 请单击此处查看此图的较大版本。
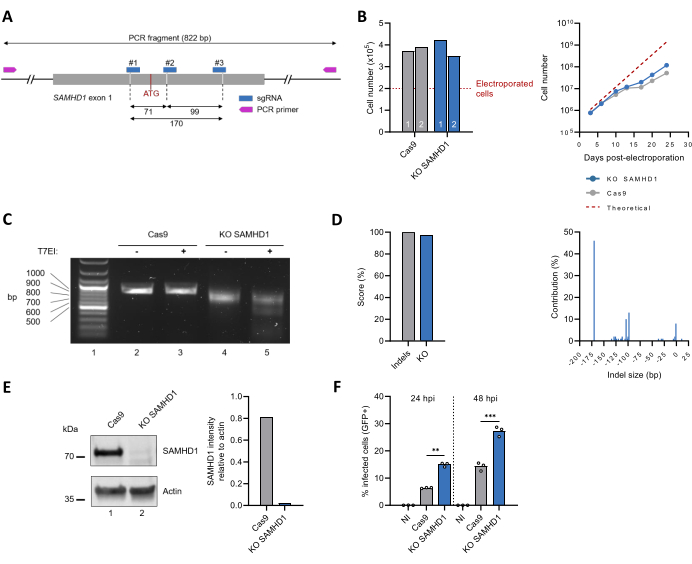
图 3:多克隆 SAMHD1 KO THP-1 细胞群的生产和表征。 (A) SAMHD1 外显子 1 的示意图,包括 sgRNA 和引物的结合位点。Cas9 切割位点由虚线表示,初始 ATG 密码子以红色显示。(B) 电穿孔后 3 天,使用台盼蓝排阻试验对活细胞 (n = 2) 进行定量(左图)。监测细胞生长 25 天(右图)。虚线对应于基于估计倍增时间为 49 小时的理论细胞数。(C) 评估了基因组水平的编辑结果,如图 2D 所示。(D) 通过基于 ICE 的 Sanger 序列分析来鉴定和定量 CRISPR 编辑后的基因型。(E) 通过在 SDS-PAGE 凝胶上迁移分离已编辑和未编辑的 THP-1 细胞的粗裂解物中包含的蛋白质,然后使用针对 SAMHD1 的抗体通过免疫印迹进行可视化。肌动蛋白用作负载对照。SAMHD1 和肌动蛋白条带的强度通过使用 ImageJ 软件的光密度测定进行量化。(F) 未编辑的 (Cas9) 和 KO SAMHD1 THP-1 细胞通过 PMA (300 ng/mL,24 h) 处理分化,然后用表达 GFP 作为报告基因的 VSVg 假型 HIV-1 病毒攻击。在感染后 24 h 和 48 h (hpi) 通过流式细胞术对 GFP 阳性细胞的比例进行评分。NI: 未感染。**P < 0.01;P < 0.001 通过未配对的 t 检验 和 Welch 校正。 请单击此处查看此图的较大版本。

图 4:SAMHD1 KO 单细胞克隆的生产和表征。 (A) 通过监测琼脂糖凝胶电泳对包含靶位点的区域进行 PCR 扩增产生的 DNA 片段的迁移率,评估所选 SAMHD1 KO 单细胞克隆的编辑结果。(B) 使用 ICE 工具分析 Sanger 测序数据,如图 2E 所示。右图中的条形表示每个细胞群中插入缺失的多样性。绿色条表示显性序列和相关缺失的总大小。括号中的数字表示非连续间隙的大小。(C) 如图 3F 所示,测试了未编辑和 SAMHD1 KO 单细胞克隆对 HIV-1 感染的允许性,并与未电穿孔的亲本 THP-1 细胞或未编辑的细胞克隆 (Cas9) 进行了比较。感染率评分为 48 hpi。*P < 0.05;**P < 0.01;P < 0.001;P < 0.0001;Brown-Forsythe 和 Welch 方差分析的 ns P≥ 0.05 与 Dunnett 多重比较检验。 请单击此处查看此图的较大版本。
补充图 1:通过适应性 T7EI 测试评估 KO 单细胞克隆的编辑结果。 纯化 KO 单细胞克隆或未编辑细胞的基因组 DNA,并通过 PCR 扩增靶位周围的区域。将每个单细胞克隆的扩增子与未编辑的对照细胞的扩增子以 1:1 的比例混合。异源双链体形成和 T7EI 消化后,通过琼脂糖凝胶电泳分析产物。编辑后的克隆产生多个消化片段(克隆 A、B、C、D 和 E),而未编辑的克隆显示单个 WT 条带(克隆 F 和 G)。 请点击此处下载此文件。
补充图 2:流式细胞术门控策略,用于量化 HIV-1 感染的 PMA 处理的 THP-1 细胞的百分比。 使用前向散射区 (FSC-A) 对抗侧向散射区 (SSC-A),定义第一个门以消除碎片并选择 PMA 处理的 THP-1 细胞。接下来,根据 SSC-H 图使用 SSC-A 选择单个细胞。最后,用 GFP-A 检测针对 FSC-H 的 GFP 阳性细胞。 请点击此处下载此文件。
讨论
这里描述了一种方案,以获得成功的 THP-1 细胞系的 CRISPR 介导的编辑。该方法依赖于通过电穿孔/成核转染转移预组装的 sgRNA/Cas9 RNP。选择该策略是为了限制慢病毒介导的 sgRNA/Cas9 盒整合时可能出现的脱靶效应,从而产生核酸酶的持续表达。选择靶向目标基因的多个 sgRNA 以实现可靠和高效的编辑,这增加了产生基因组插入缺失的可能性,从而导致蛋白质表达和功能 KO32 的丢失。特别注意电穿孔设置以实现细胞膜的最佳透化,这是确保 sgRNA/Cas9 RNP 有效递送同时防止细胞过度死亡所必需的33。在这方面,一个关键参数是电穿孔前细胞培养物的活力(即低传代数)。
使用 T7EI 错配检测测定在基因水平上验证了 CRISPR-Cas9 介导的编辑,该检测提供了对多个样本的成本效益和快速评估,以便在 Sanger 测序之前进行预选。该方法最初是为了通过比较全长扩增子和切割产物的强度来估计使用单个 sgRNA 的编辑效率34,35。值得注意的是,使用多个 sgRNA 时产生的大插入缺失 (>70 bp) 可以通过比较通过标准凝胶电泳对编辑细胞和未编辑细胞的靶基因座进行 PCR 扩增获得的 DNA 片段的迁移率来检测。然而,T7EI 测定可能有助于确定在多克隆或使用改编方案的单克隆细胞群中是否存在含有小插入缺失 (>1bp) 的序列。
通过一种简单的方法证实了靶向框外突变的存在,这些突变会中断编码序列,其中提取整个细胞群的基因组并用作 PCR 扩增的基质。接下来,纯化的扩增子进行批量 Sanger 测序,然后使用 ICE 工具进行生物信息学分析。总体而言,该方法克服了将 PCR 产物亚克隆到质粒中并将连接产物转化为细菌以分离单细胞集落(单独测序)的需要。
一旦验证了目标基因座的失活,就需要对编辑后的细胞系进行彻底的表征,以自信地将给定的表型归因于缺乏感兴趣的蛋白质,并排除由脱靶引起的意外影响,但也排除可能影响 mRNA 结构和/或功能的脱靶变化。
应通过免疫印迹和/或免疫荧光来确认蛋白质表达的缺失,可能使用两种识别不同表位的抗体。事实上,在靠近经典 ATG 密码子的早期外显子中引入 PTC 与 ITL 事件的风险有关,ITL 事件会产生 N 末端截短的蛋白质变体36。值得注意的是,Tuladhar 等人报告了在很大一部分 (~50%) 的商业或内部来源的 CRISPR 编辑细胞中检测到异常蛋白质种类18,19。新型蛋白质产物的出现归因于多种多样且潜在的细胞类型特异性过程,例如跳过含有插入缺失的外显子或内部核糖体进入位点 (IRES) 的出现,导致蛋白质合成在经典 ATG 密码子下游的替代 TIS 开始。还有迹象表明,在某些情况下,ITL 与逃避 NMD 相伴而生37,38。同样,靶向晚期外显子的基因组编辑策略可能无法触发突变 mRNA 的 NMD,这是基因表达无效消融的基础20。最后,如果 Cas9 由多个 sgRNA 引导,则可能会出现复杂的染色体重排,包括倒位或大 DNA 片段缺失39,这可能会改变基因组的结构和表达,特别是当靶向调节元件时40。
为了进一步减轻意外基因组编辑结果的影响,需要进行功能研究来确认蛋白质表达缺失与随后的表型 (即抗病毒活性) 之间的关系。如果编辑的细胞是亚克隆的,则应比较多个克隆之间以及与多克隆群体的行为。进行挽救实验,包括恢复基因表达以将编辑细胞的表型逆转为 WT 表型,也将增强特异性并排除可能的脱靶事件。
尽管该方案是为编辑 THP-1 细胞而构思的,但它为 CRISPR-Cas9 KO 工作流程提供了一个通用结构,可以适应其他细胞系。可能需要调整电穿孔参数,如果施加高于 1800 V 的电压,则应根据制造商说明使用重悬缓冲液 T。另一个需要考虑的变量是 RNP 组装过程中 Cas9 和 sgRNA 之间的浓度比。尽管 1:9 的比例在这种情况下效果很好,但在其他情况下修改它可能会提高编辑效率。最后,虽然这里没有好处,但应测试在电穿孔后使用条件培养基进行细胞回收,因为不同细胞系的效果可能不同。
披露声明
所有作者都没有利益冲突。
致谢
我们感谢 JP Concordet(MNHN,U1154/UMR7196,巴黎)、G. Bossis(IGMM,蒙彼利埃)和 D. Schlüter(德国汉诺威医学院)分享方案和讨论。该项目已获得欧盟 Horizon 2020 研究和创新计划(AZ 的赠款协议第 101017572 号)和 ANRS(AZ 的赠款ECTZ162721)的资助。传染病模型和创新疗法 (IDMIT) 研究基础设施由参考 ANR_11_INSB_0008下的“未来计划投资 (PIA)”提供支持。
材料
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filter | ClearLine | 146560 | _ |
| 0.4 % trypan blue | Beckman Coulter | 383200 | _ |
| 1.5 mL tube | Eppendorf | 3810X | _ |
| 24-well plate | Corning | 353047 | _ |
| 6x TriTrack DNA Loading Dye | Thermo scientific | R1161 | _ |
| 75 cm² Culture Flask Vented Cap | Corning | 353136 | _ |
| 8-Strip PCR Tubes with Caps | Life technologies | AM12230 | _ |
| 96-well plates Flat bottom | Corning | 353072 | _ |
| 96-well plates Round bottom | Corning | 353077 | _ |
| Agarose | Euromedex | D5 | _ |
| ATGpr | _ | _ | https://atgpr.dbcls.jp/ |
| ChemiDoc Imaging System | BIO-RAD | 12003153 | _ |
| Counting slide | NanoEntek | DHC-N04 | _ |
| CRISPOR | _ | _ | http://crispor.gi.ucsc.edu/ |
| DPBS | Gibco | 14190094 | _ |
| Ensembl | EMBL-EBI | _ | https://www.ensembl.org/index.html |
| Fetal Bovine Serum | Sigma-Aldrich | F7524 | _ |
| FlowJo | BD Life Sciences | v10.10 | _ |
| GeneRuler 100 bp Plus DNA Ladder | Thermo scientific | SM0323 | _ |
| Genome Data Viewer | NCBI | _ | https://www.ncbi.nlm.nih.gov/gdv/ |
| GraphPad Prism | Dotmatics | _ | Version 9.3.1 |
| Herculase II Fusion DNA Polymerases | Agilent | 600679 | _ |
| ICE CRISPR Analysis Tool | Synthego | _ | https://www.synthego.com/products/bioinformatics/crispr-analysis |
| Image Lab Touch | BIO-RAD | _ | Version 2.4.0.03 |
| NEBuffer 2 | New England Biolabs | B7002S | Included with T7EI M0302S |
| Neon Kit, 10 µL | Invitrogen | MPK1025K | Electroporation kit containing tips, tubes, buffer R and E |
| Neon Transfection System | Invitrogen | MPK5000 | _ |
| NetStart 1.0 | _ | _ | https://services.healthtech.dtu.dk/services/NetStart-1.0/ |
| Nuclease-free Water | Synthego | _ | _ |
| PCR primer (EGFP) | Eurofins | _ | Fw : GGAATGCAAGGTCTGTTGAATG ; Rev : CACCTTGATGCCGTTCTTCT |
| PCR primer (SAMHD1) | Eurofins | _ | Fw : CGGGATTGATTTGAGGACGA ; Rev : GGGTGGCAAGTTAGTGAAGA |
| Penicillin-streptomycin (10,000 U/mL) | Gibco | 15140122 | _ |
| PFA | Electron Microscopy Sciences | 15714 | _ |
| PMA | Sigma-Aldrich | P8139 | _ |
| PrimerQuest | IDT | _ | https://eu.idtdna.com/pages/tools/primerquest |
| QIAquick PCR Purification Kit | Qiagen | 28104 | _ |
| QuickExtract DNA Extraction Solution | Biosearch Technologies | QE09050 | _ |
| RPMI 1640, GlutaMAX | Gibco | 61870010 | _ |
| SnapGene Viewer | Dotmatics | _ | Version 7 |
| SpCas9 2NLS Nuclease | Synthego | _ | _ |
| SYBR Safe DNA Gel Stain | Invitrogen | S33102 | _ |
| Synthetic sgRNA (EGFP) | Synthego | _ | #1 : CGCGCCGAGGUGAAGUUCGA ; #2 : UUCAAGUCCGCCAUGCCCGA ; #3 : CAACUACAAGACCCGCGCCG |
| Synthetic sgRNA (SAMHD1) | Synthego | _ | #1 : AUCGCAACGGGGACGCUUGG ; #2 : GCAGUCAAGAACCUCGGCGC ; #3 : CCAUCCCGACUACAAGACAU |
| Syringe Plastipak Luer Lock | BD | 301229 | _ |
| T100 Thermal Cycler | BIO-RAD | 1861096 | _ |
| T7 endonuclease I | New England Biolabs | M0302S | _ |
| TAE buffer UltraPure, 10x | Invitrogen | 15558026 | 400 mM Tris-Acetate, 10 mM EDTA |
| THP-1 cells | ATCC | TIB-202 | _ |
| Trypsin-EDTA (0,05 %) | Gibco | 25300054 | _ |
| ZE5 Cell Analyzer | BIO-RAD | 12014135 | _ |
参考文献
- Tsuchiya, S., et al. Establishment and characterization of a human acute monocytic leukemia cell line (THP-1). Int J Cancer. 26 (2), 171-176 (1980).
- Danis, V. A., Millington, M., Hyland, V. J., Grennan, D. Cytokine production by normal human monocytes: inter-subject variation and relationship to an IL-1 receptor antagonist (IL-lRa) gene polymorphism. Clin Exp Immunol. 99, (1995).
- Bol, S. M., et al. Donor variation in in vitro HIV-1 susceptibility of monocyte-derived macrophages. Virology. 390 (2), 205-211 (2009).
- Appleby, L. J., et al. Sources of heterogeneity in human monocyte subsets. Immunol Lett. 152 (1), 32-41 (2013).
- O'Neill, M. B., et al. Single-cell and bulk RNA-sequencing reveal differences in monocyte susceptibility to Influenza A virus infection between Africans and Europeans. Front Immunol. 12, 768189 (2021).
- Wen, Y., et al. Comparability study of monocyte-derived dendritic cells, primary monocytes, and THP1 cells for innate immune responses. J Immunol Methods. 498, 113147 (2021).
- Inagaki, Y., et al. Interferon-g-induced apoptosis and activation of THP-1 macrophages. Life Sci. 71 (21), 2499-2508 (2002).
- Boonkaewwan, C., Toskulkao, C., Vongsakul, M. Anti-inflammatory and immunomodulatory activities of stevioside and its metabolite steviol on THP-1 cells. J Agric Food Chem. 54 (3), 785-789 (2006).
- Chanput, W., et al. β-Glucans are involved in immune-modulation of THP-1 macrophages. Mol Nutr Food Res. 56 (5), 822-833 (2012).
- Cui, J., et al. USP3 inhibits type I interferon signaling by deubiquitinating RIG-I-like receptors. Cell Res. 24 (4), 400-416 (2014).
- Chen, S., et al. SAMHD1 suppresses innate immune responses to viral infections and inflammatory stimuli by inhibiting the NF-κB and interferon pathways. Proc Natl Acad Sci USA. 115 (16), E3798-E3807 (2018).
- Pradhananga, S., Spalinskas, R., Poujade, F. A., Eriksson, P., Sahlén, P. Promoter anchored interaction landscape of THP-1 macrophages captures early immune response processes. Cell Immunol. 355, 104148 (2020).
- Mezzasoma, L., Talesa, V. N., Romani, R., Bellezza, I. Anp and BNP exert anti-inflammatory action via npr-1/cgmp axis by interfering with canonical, non-canonical, and alternative routes of inflammasome activation in human THP1 cells. Int J Mol Sci. 22 (1), 1-17 (2021).
- Martinat, C., et al. SUMOylation of SAMHD1 at Lysine 595 is required for HIV-1 restriction in non-cycling cells. Nat Commun. 12 (1), 4582 (2021).
- Rensen, E., et al. Clustering and reverse transcription of HIV-1 genomes in nuclear niches of macrophages. EMBO J. 40 (1), e105247 (2021).
- Ikeda, T., et al. APOBEC3 degradation is the primary function of HIV-1 Vif determining virion infectivity in the myeloid cell line THP-1. mBio. 14 (4), e0078223 (2023).
- Jinek, M., et al. A programmable dual RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337, 816-821 (2012).
- Kurosaki, T., Maquat, L. E. Nonsense-mediated mRNA decay in humans at a glance. J Cell Sci. 129 (3), 461-467 (2016).
- Tuladhar, R., et al. CRISPR-Cas9-based mutagenesis frequently provokes on-target mRNA misregulation. Nat Commun. 10 (1), 4056 (2019).
- Embree, C. M., Abu-Alhasan, R., Singh, G. Features and factors that dictate if terminating ribosomes cause or counteract nonsense-mediated mRNA decay. J Biol Chem. 298 (11), 102592 (2022).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Schnoor, M., et al. Efficient non-viral transfection of THP-1 cells. J Immunol Methods. 344 (2), 109-115 (2009).
- Tang, X., et al. A method for high transfection efficiency in THP-1 suspension cells without PMA treatment. Anal Biochem. 544, 93-97 (2018).
- Ji, W., Zhang, L., Liu, X. Protocol using lentivirus to establish THP-1 suspension cell lines for immunostaining and confocal microscopy. STAR Protoc. 4 (1), 102032 (2023).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, (2023).
- . Synthego Performance Analysis, ICE Analysis Available from: https://www.synthego.com/help/citing-ice (2019)
- Salamov, A. A., Nishikawa, T., Swindells, M. B. Assessing protein coding region integrity in cDNA sequencing projects. Bioinformatics. 14, 384-390 (1998).
- Pedersen, A. G., Nielsen, H. Neural network prediction of translation initiation sites in eukaryotes: Perspectives for EST and genome analysis. Proc Int Conf Intell Syst Mol Biol. 5, 226-233 (1997).
- Concordet, J. P., Haeussler, M. CRISPOR: Intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46 (W1), W242-W245 (2018).
- Laguette, N., et al. SAMHD1 is the dendritic- and myeloid-cell-specific HIV-1 restriction factor counteracted by Vpx. Nature. 474 (7353), 654-657 (2011).
- Hrecka, K., et al. Vpx relieves inhibition of HIV-1 infection of macrophages mediated by the SAMHD1 protein. Nature. 474 (7353), 658-661 (2011).
- Seki, A., Rutz, S. Optimized RNP transfection for highly efficient CRI SPR/Cas9-mediated gene knockout in primary T cells. J Exp Med. 215 (3), 985-997 (2018).
- Batista Napotnik, T., Polajžer, T., Miklavčič, D. Cell death due to electroporation - A review. Bioelectrochemistry. 141, 107871 (2021).
- Perretta-Tejedor, N., Freke, G., Seda, M., Long, D. A., Jenkins, D. Generating mutant renal cell lines using CRISPR technologies. Methods Mol Biol. 2067, 323-340 (2020).
- Sentmanat, M. F., Peters, S. T., Florian, C. P., Connelly, J. P., Pruett-Miller, S. M. A survey of validation strategies for CRISPR-Cas9 editing. Sci Rep. 8 (1), 888 (2018).
- Makino, S., Fukumura, R., Gondo, Y. Illegitimate translation causes unexpected gene expression from on-target out-of-frame alleles created by CRISPR-Cas9. Sci Rep. 6, 39608 (2016).
- Bowling, A., et al. Downstream alternate start site allows N-terminal nonsense variants to escape NMD and results in functional recovery by readthrough and modulator combination. J Pers Med. 12 (9), 1448 (2022).
- Inácios, &. #. 1. 9. 4. ;., et al. Nonsense mutations in close proximity to the initiation codon fail to trigger full nonsense-mediated mRNA decay. J Biol Chem. 279 (31), 32170-32180 (2004).
- Li, J., et al. Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9. J Mol Cell Biol. 7 (4), 284-298 (2015).
- Guo, Y., et al. CRISPR inversion of CTCF sites alters genome topology and enhancer/promoter function. Cell. 162 (4), 900-910 (2015).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。