Method Article
Knockout genico mediato da CRISPR-Cas9 basato sull'elettroporazione in cellule THP-1 e isolamento di cloni a singola cellula
In questo articolo
Riepilogo
La linea cellulare THP-1 è ampiamente utilizzata come modello per studiare le funzioni dei monociti/macrofagi umani in varie aree di ricerca legate alla biologia. Questo articolo descrive un protocollo per l'ingegneria efficiente basata su CRISPR-Cas9 e l'isolamento di cloni di singole cellule, che consente la produzione di dati fenotipici robusti e riproducibili.
Abstract
La linea cellulare THP-1 per la leucemia monocitica acuta umana (LMA) è ampiamente utilizzata come modello per studiare le funzioni dei macrofagi derivati dai monociti umani, compresa la loro interazione con patogeni umani significativi come il virus dell'immunodeficienza umana (HIV). Rispetto ad altre linee cellulari immortalizzate di origine mieloide, le cellule THP-1 conservano molte vie di segnalazione infiammatoria intatte e mostrano caratteristiche fenotipiche che assomigliano più da vicino a quelle dei monociti primari, inclusa la capacità di differenziarsi in macrofagi quando trattate con forbolo-12-miristato 13-acetato (PMA). L'uso della tecnologia CRISPR-Cas9 per ingegnerizzare le cellule THP-1 attraverso il knockout genico mirato (KO) fornisce un approccio potente per caratterizzare meglio i meccanismi immuno-correlati, comprese le interazioni virus-ospite. Questo articolo descrive un protocollo per un'efficiente ingegneria basata su CRISPR-Cas9 che utilizza l'elettroporazione per fornire ribonucleoproteine Cas9:sgRNA preassemblate nel nucleo cellulare. L'utilizzo di più sgRNA che hanno come bersaglio lo stesso locus in posizioni leggermente diverse comporta la delezione di grandi frammenti di DNA, aumentando così l'efficienza dell'editing, come valutato dal saggio dell'endonucleasi T7 I. L'editing mediato da CRISPR-Cas9 a livello genetico è stato convalidato dal sequenziamento Sanger seguito dall'analisi Inference of CRISPR Edits (ICE). La deplezione delle proteine è stata confermata dall'immunoblotting accoppiato a un test funzionale. Utilizzando questo protocollo, sono stati raggiunti fino al 100% di indel nel locus target e una diminuzione di oltre il 95% dell'espressione proteica. L'elevata efficienza di editing rende conveniente isolare i cloni di singole cellule limitando la diluizione.
Introduzione
THP-1 è una linea cellulare umana derivata da monociti isolata da un paziente affetto da leucemia acuta (LMA), che mostra caratteristiche fenotipiche molto simili a quelle dei monociti primari1. Rispetto ai macrofagi primari derivati da monociti, che non si dividono e mostrano sia una durata di vita limitata che una variabilità inter-/intra-donatrice nel fenotipo, le cellule THP-1 possono essere coltivate praticamente all'infinito e hanno un comportamento più omogeneo che favorisce la riproducibilità dei risultati 2,3,4,5,6 . In particolare, le cellule THP-1 possono essere differenziate verso un fenotipo simile ai macrofagi con forbolo-12-miristato 13-acetato (PMA), rendendole un modello in vitro ampiamente utilizzato per studiare le risposte di monociti/macrofagi ai segnali infiammatori 7,8,9,10,11,12,13 o all'infezione da parte di patogeni umani clinicamente rilevanti, incluso l'HIV 14,15,16. La possibilità di ingegnerizzare geneticamente le cellule THP-1 è di interesse in molte aree di ricerca legate alla biologia.
La proteina 9 associata a CRISPR (CRISPR-Cas9) è un sistema immunitario adattativo procariotico che si basa su nucleasi guidata da RNA per degradare i genomi virali invasori, che è stato riprogrammato come strumento di ingegneria genetica17. Il processo di editing del genoma procede in tre fasi: riconoscimento, scissione e riparazione. Un RNA a guida singola (sgRNA) recluta la nucleasi Cas9 in uno specifico locus genomico attraverso l'accoppiamento di basi con la sua sequenza guida da 20 bp. La presenza di una sequenza di Protospacer Adjacent Motif (PAM) direttamente a 3' della sequenza bersaglio genomica a 20 bp innesca lo svolgimento e la scissione mediati da Cas9 su entrambi i filamenti di DNA tra le posizioni 17 e 18 (3-bp 5' del PAM). La rottura a doppio filamento (DSB) risultante viene elaborata da due principali percorsi di riparazione. In assenza di un modello di riparazione che presenti omologia con il locus danneggiato, la via di giunzione delle estremità non omologhe (NHEJ) soggetta a errori introdurrà inserzioni e/o delezioni nucleotidiche casuali (indel), portando potenzialmente a mutazioni frameshift e/o all'introduzione di codoni di terminazione prematura (PTC). A loro volta, gli mRNA contenenti PTC sono bersaglio della degradazione da parte della via di decadimento dell'mRNA (NMD) mediata da nonsenso, interrompendo in ultima analisi l'espressione/funzione della proteina 18,19,20. In alternativa, il percorso HDR (Homology-Directed Repair) dipendente dal modello può operare e riparare fedelmente il DSB. Questo meccanismo è stato sfruttato per ottenere un editing genetico preciso, compresi i knock-in e le sostituzioni di basi. Vale la pena notare che lo stato del ciclo cellulare è un fattore importante che influenza la scelta della via di riparazione del DSB. Infatti, l'NHEJ è attivo in tutte le fasi del ciclo cellulare, mentre l'HDR è principalmente limitato alle fasi S/G221.
Le cellule THP-1 crescono in sospensione e sono notoriamente difficili da trasfettare con il DNA plasmidico, una procedura che probabilmente altera anche la loro vitalità e/o capacità di differenziazione22,23. La trasduzione con vettori lentivirali basati su HIV-1 che codificano sia per Cas9 che per l'sgRNA è spesso impiegata per mettere fuori combattimento (KO) un gene di interesse24. L'integrazione della cassetta Cas9/sgRNA nel genoma cellulare garantisce un'espressione prolungata e un KO efficiente, ma è anche una fonte persistente di effetti off-target25. In alternativa, le ribonucleoproteine (RNP) pre-assemblate Cas9:sgRNA vengono veicolate mediante elettroporazione, un metodo che prevede la formazione temporanea di pori sia nel plasma che nelle membrane nucleari dopo l'applicazione di impulsi elettrici. Preservare la vitalità cellulare è una sfida importante quando si intraprende questo approccio.
Qui, è stata prodotta una linea cellulare THP-1 che esprime stabilmente GFP (THP-1_GFP) per fungere da strumento per stabilire un protocollo per ottenere un efficiente editing basato su CRISPR-Cas9. Dopo aver progettato una strategia per inattivare il gene EGFP utilizzando tre sgRNA contemporaneamente (approccio multi-guida), l'efficienza KO in diverse condizioni di elettroporazione è stata determinata utilizzando l'espressione di GFP come lettura. La proliferazione cellulare è stata monitorata in parallelo. L'editing genetico è stato confermato sia da un test dell'endonucleasi I T7 (T7EI) che dal sequenziamento Sanger, seguito dall'analisi con l'algoritmo Inference of CRISPR Edits (ICE)26. I parametri che hanno prodotto una diminuzione dell'espressione di GFP fino al 95%, con le cellule THP-1 che recuperano i normali tassi di crescita dopo l'elettroporazione, sono stati impiegati con successo per inattivare un gene endogeno (SAMHD1) e produrre cloni di THP-1 a singola cellula.
Protocollo
I dettagli dei reagenti e delle attrezzature utilizzate in questo studio sono elencati nella tabella dei materiali.
1. Guida la progettazione con CRISPOR (Figura 1.1)
NOTA: Il software SnapGene Viewer può essere utilizzato nei passaggi 4, 7 e 10 per annotare il sito di destinazione dell'editing e la posizione dell'ibridazione del primer PCR all'interno del gene di interesse.
- Vai al sito web di Ensembl (www.ensembl.org). Nella casella di ricerca , seleziona una specie e inserisci il nome del gene di interesse. Fare clic su Vai. Seleziona il risultato che corrisponde al gene (non una trascrizione).
- Fare clic su Mostra tabella di trascrizione e quindi selezionare l'ID trascrizione che corrisponde alla codifica delle proteine con etichetta oro nella colonna Biotipo . Una volta nella pagina della trascrizione, fai clic su Exons nel menu a sinistra.
- Scorri verso il basso e fai clic su Scarica sequenza. Assicurati che il formato del file sia FASTA. In Impostazioni - Sequenze incluse, deseleziona tutto tranne Sequenza genomica.
Immettere il numero "500" in Sequenza di fiancheggiamento a entrambe le estremità della casella di trascrizione . Fare clic su Anteprima, selezionare l'intera sequenza (solo i nucleotidi senza l'intestazione) e copiarla (Ctrl+C). - Apri SnapGene Viewer e fai clic su Nuovo file > DNA. Incollare la sequenza (CTRL+V) nella casella Crea una sequenza . Deselezionare Rileva funzioni comuni e selezionare Lineare in Topologia.
Rinomina il file e fai clic su Crea. Nel menu a sinistra, deseleziona Mostra enzimi (prima icona). Nella parte inferiore della finestra, seleziona la scheda Sequenza .
NOTA: Questo passaggio consente il recupero dell'intera sequenza genica, inclusi esoni, introni e sequenze fiancheggianti da 500 bp (opzionale). Quest'ultima informazione è utile per la progettazione di primer PCR per l'amplificazione di un sito bersaglio situato all'interno del primo esone. - Torna al sito Web di Ensembl, scorri verso l'alto l'anteprima del file e fai clic su Indietro. Ora, cambia il formato del file in RTF. In Impostazioni - Sequenze incluse, deselezionare tutto tranne gli esoni. In Mostra varianti, seleziona No. Fare clic su Download nella parte superiore della pagina.
- Apri il file scaricato (con Word), che contiene la sequenza degli esoni e mostra la sequenza di codifica in blu, a partire dall'ATG iniziale. Scegliere l'esone da prendere di mira con la modifica diretta da CRISPR-Cas9 (vedere di seguito per le raccomandazioni), selezionarlo e copiarlo (Ctrl+C).
- L'esone bersaglio è un esone precoce o un esone che codifica per un dominio funzionalmente importante della proteina. Vale la pena notare che l'instaurazione di un PTC in un esone tardivo vicino al 3' UTR probabilmente non riuscirà a suscitare NMD, portando all'espressione di una proteina troncata C-terminale. Al contrario, l'introduzione di un PTC in un esone precoce prossimale al sito di iniziazione nativo è associata a un rischio di traduzione illegittima (ITL, noto anche come ATI)), producendo l'espressione inaspettata di una proteina troncata N-terminale che inizia in un sito di inizio della traduzione in-frame (TIS) a valle del primo codone ATG. Per mitigare quest'ultimo rischio, si consiglia di valutare l'insorgenza di TIS alternativi utilizzando ATGpr27 (atgpr.dbcls.jp) e/o NetStart 1.028 (services.healthtech.dtu.dk/services/NetStart-1.0/).
- Assicurarsi che l'esone di destinazione contenga una sequenza di codifica. Tuttavia, potrebbe rivelarsi utile scegliere una ricottura di sgRNA con la regione 5'UTR a monte dell'ATG iniziale per includerla nel frammento eliminato.
- Idealmente, l'esone bersaglio dovrebbe essere comune a tutte le varianti di trascrizione codificanti proteine del gene. Controlla se questo è il caso sul Genome Data Viewer (www.ncbi.nlm.nih.gov/gdv/) dell'NCBI cercando il gene di interesse.
Cliccando sul nome del gene (in verde) nella finestra del display verranno mostrate le varianti del trascritto (in viola). Gli esoni sono rappresentati da un rettangolo.
- In SnapGene Viewer, premere Ctrl+F, Ctrl+V, quindi Invio per cercare la sequenza dell'esone. Premi Ctrl+T per aggiungere una nuova funzionalità, assegnale un nome, cambia il tipo in "exon" e fai clic su OK.
- Vai al sito Web CRISPOR (http://crispor.gi.ucsc.edu/) e incolla la sequenza dell'esone nel passaggio 1. Innanzitutto, selezionare un genoma di riferimento nel passaggio 2 e quindi il tipo di PAM da scegliere come bersaglio nel passaggio 3, in genere 20bp-NGG per SpCas9. Fare clic su INVIA.
- Seleziona due sgRNA in modo che siano separati fino a 150 bp, quindi seleziona un terzo sgRNA intermedio. Ecco alcune linee guida per la selezione degli sgRNA:
- Il punteggio di specificità del MIT è correlato agli effetti fuori bersaglio. Un punteggio più alto indica un minor numero di potenziali fuori target. La colonna di destra mostra tre siti fuori bersaglio previsti classificati dal più al meno probabile, insieme alle loro posizioni (in un esone, un introne o una regione intergenica). È possibile accedere all'elenco completo dei siti fuori target previsti facendo clic su Mostra tutto. Se possibile, seleziona gli sgRNA con un punteggio MIT >80, dando la priorità a quelli senza fuori bersaglio per 0, 1 o 2 discrepanze. Inoltre, gli sgRNA con off-target in un esone, che hanno il maggior potenziale di influenzare il fenotipo, dovrebbero essere evitati.
- Per l'efficacia prevista, fare riferimento al punteggio Doench '16. Si noti che un punteggio Doench '16 elevato indica semplicemente che l'sgRNA ha maggiori probabilità di essere efficace. L'efficacia effettiva deve essere determinata sperimentalmente. Per questo motivo, è sempre utile selezionare più sgRNA, anche se non sono destinati ad essere utilizzati insieme.
- Un contenuto di GC troppo alto o troppo basso, così come alcuni motivi, possono essere dannosi per l'efficienza dell'sgRNA e dovrebbero essere evitati. Questi parametri sono evidenziati da CRISPOR.
- Ripetere il passaggio 1.7 per aggiungere la sequenza sgRNA e la sequenza PAM associata alla sequenza genica in SnapGene Viewer. Allo stesso tempo, incolla la sequenza di sgRNA (senza il PAM) in un file di testo o Excel per mantenere l'orientamento 5'-3' richiesto quando si ordina l'oligonucleotide.
- Per l'amplificazione basata sulla PCR del sito target, progettare un paio di primer che lo circondano. La dimensione dell'amplicone dovrebbe essere compresa tra 800 e 1000 bp. PrimerQuest è stato utilizzato per progettare i primer per questo protocollo (https://eu.idtdna.com/pages/tools/primerquest). In alternativa, CRISPOR fornisce un elenco di primer per amplificare la regione genomica mirata e i siti fuori bersaglio. Dopo aver visualizzato l'elenco completo dei potenziali siti fuori bersaglio (passaggio 1.9.1), fai clic su Primer fuori bersaglio nell'angolo in basso a destra.
2. Preparazione del reagente e della cellula per l'elettroporazione (Figura 1.2)
- Preparare una piastra di coltura a 24 pozzetti per recuperare le cellule dopo l'elettroporazione riempiendo i pozzetti con 500 μl di terreno RPMI 1640 integrato con il 20% di siero fetale bovino (FBS) inattivato a caldo (56 °C, 30 min) filtrato (0,20 μm). Non aggiungere antibiotici. Conservare la piastra in un'incubatrice umidificata a 37 °C e 5% di CO2 per 24 ore fino all'elettroporazione.
- Se gli sgRNA vengono spediti secchi, reidratarli con tampone TE (10 mM Tris, 1 mM EDTA, pH 8,0) a una concentrazione finale di 100 μM (cioè 10 μL di tampone TE per 1 nmol di sgRNA). Agitare per 30 s, incubare a 4 °C durante la notte per consentire la completa reidratazione e, dopo una breve omogeneizzazione con pipetta, conservare la soluzione madre di sgRNA a -20 °C. A seconda del volume finale, fare delle aliquote per evitare più cicli di gelo-scongelamento.
- Preparare una soluzione di sgRNA funzionante a una concentrazione finale di 30 μM diluendo la soluzione madre da 100 μM in acqua priva di nucleasi. Agitare per 30 s e incubare 5 minuti a temperatura ambiente.
- Assemblare gli RNP Cas9:sgRNA con un rapporto molare 1:9 diluendo simultaneamente 1 μL di ciascuno dei tre sgRNA da 30 μM e 0,5 μL di soluzioni Cas9 da 20 μM in 3,5 μL di tampone di risospensione R, incluso nel kit di elettroporazione (volume finale di 7 μL, per una condizione sperimentale; scalare di conseguenza). Agitare brevemente e incubare per 5 minuti a temperatura ambiente.
- Nel frattempo, preparare un controllo non modificato aggiungendo 0,5 μL di 20 μM Cas9 a 6,5 μL di tampone di risospensione R. Vortex brevemente e incubare per 5 minuti a temperatura ambiente.
- Aggiungere 5 μl di tampone di risospensione R a tutti i campioni per un volume finale di 12 μl per condizione di elettroporazione.
- Preparare le celle THP-1 per l'elettroporazione.
- Valutare la concentrazione e la vitalità mediante test di esclusione del blu di tripano. Diluire le cellule 1:2 in una soluzione colorante di blu di tripano allo 0,4%. Dopo 30 s di incubazione, omogeneizzare bene e aggiungere 10 μl in una parete di una camera di conteggio con uno stile di griglia migliorato di Neubauer. Contare tre quadrati grandi e dividere il conteggio per 100 per ottenere la concentrazione cellulare (x106 cellule/mL).
NOTA: La salute delle cellule influenza la loro sensibilità all'elettroporazione. Bisogna fare attenzione a eseguire la coltura cellulare in condizioni ottimali. Il tempo all'esterno dell'incubatore deve essere limitato e tutti i reagenti e le soluzioni devono essere preparati e riscaldati in anticipo. - Per ogni condizione, raccogliere un volume equivalente a 0,2 x 106 celle e centrifugare (336 x g, 5 min, 20 °C).
- Aspirare il surnatante utilizzando una pipetta e risospendere il pellet in 500 μL di PBS. Centrifugare nuovamente (336 x g, 5 min, 20 °C).
- Aspirare con cautela il surnatante utilizzando una pipetta e risospendere il pellet cellulare THP-1 con i 12 μL di soluzione di RNP (passaggio 2.6).
- Valutare la concentrazione e la vitalità mediante test di esclusione del blu di tripano. Diluire le cellule 1:2 in una soluzione colorante di blu di tripano allo 0,4%. Dopo 30 s di incubazione, omogeneizzare bene e aggiungere 10 μl in una parete di una camera di conteggio con uno stile di griglia migliorato di Neubauer. Contare tre quadrati grandi e dividere il conteggio per 100 per ottenere la concentrazione cellulare (x106 cellule/mL).
3. Messa a punto e nucleofezione del sistema di elettroporazione (Figura 1.3)
- Posizionare la stazione di pipettatura sotto una cappa di biosicurezza, inserire una provetta per elettroporazione nel supporto e aggiungere 3 mL di tampone E, incluso nel kit di elettroporazione.
- Dopo aver acceso l'elettroporazione, utilizzare il touch screen per impostare i seguenti parametri di elettroporazione: Tensione = 1 500 V, Durata = 10 ms, Numero = 3.
- Dotare la pipetta per elettroporazione di un puntale e aspirare 10 μl della soluzione di RNP/THP-1 (passaggio 2.7.4). Inserire la pipetta nel tubo di elettroporazione e premere Start sullo schermo del dispositivo di elettroporazione. Attendere che venga visualizzato il messaggio Completato e rimuovere la pipetta dal tubo.
- Trasferire le cellule in un pozzetto della piastra preriscaldata a 24 pozzetti e omogeneizzare delicatamente. Rimettete le piastre nell'incubatrice umidificata e lasciatele riposare indisturbate per 72 h.
NOTA: Fare attenzione a non formare bolle durante il pipettaggio della sospensione RNP/THP-1, poiché interferirebbero con la procedura di elettroporazione. Se viene visualizzato un messaggio di errore in assenza di un arco elettrico visibile, rimuovere la pipetta per elettroporazione dal tubo e premere nuovamente Start . Tuttavia, se si osserva un arco elettrico sotto forma di una breve scintilla luminosa, potrebbe indicare la presenza di bolle. La procedura di elettroporazione probabilmente fallirà, anche in assenza di un messaggio di errore.
4. Recupero di THP-1 72 ore dopo l'elettroporazione (Figura 1.4)
- Contare le cellule per valutare la concentrazione (passaggio 2.7.1) 72 ore dopo l'elettroporazione.
- Se ci sono abbastanza cellule (cioè ≥0,6 x 106 cellule/mL), diluirle almeno un fattore 2 con RPMI integrato con il 20% di FBS e l'1% di penicillina-streptomicina e portare la concentrazione tra 0,3-0,5 x 106 cellule per mL. In caso contrario, lasciare che la cella si riprenda per altre 72 ore.
- Passaggio e amplificare le celle fino a quando non ce n'è abbastanza per la convalida del KO. Nel frattempo, può essere avviato l'isolamento di cloni a singola cellula (fase 7).
5. Validazione dell'editing genetico mediante saggio di disallineamento T7EI (Figura 1.5)
NOTA: Il test potrebbe sottostimare l'efficienza di editing dato che T7EI riconosce discrepanze superiori a 1 bp. Pertanto, il test T7EI non è utile per lo screening di popolazioni cellulari omozigoti (cioè cloni di singole cellule) a meno che non venga opportunamente modificato (passaggio 5.7).
- Valutare la concentrazione delle cellule (passaggio 2.7.1) e prelevare un volume equivalente a 0,1 x 106 cellule in una provetta da 1,5 mL. Centrifugare (336 x g, 5 min, 20 °C), aspirare il surnatante e risospendere il pellet in 500 μl di PBS. Centrifugare nuovamente e, utilizzando una pipetta, aspirare quanto più surnatante possibile senza disturbare il pellet. Congelare il campione e conservarlo a -20 °C.
- Estrarre il DNA genomico da utilizzare come matrice per l'amplificazione della PCR.
- Risospendere il pellet con 50 μL di soluzione di estrazione del DNA, omogeneizzare e trasferire l'intero volume in una provetta PCR da 0,2 mL. Agitare e centrifugare brevemente (pulsare per 3 s).
- Inserire la provetta in un termociclatore e riscaldarla a 65 °C per 15 minuti, seguita da 98 °C per 10 minuti.
- Diluire il DNA estratto con 90 μL di acqua ultrapura. Agitare e centrifugare brevemente (5.000 x g, 3 s).
- Diluire il primer PCR (vedere la Tabella dei materiali) in acqua ultrapura per una concentrazione finale di 10 μM (cioè 10 pmol/μL).
- Preparare la miscela per PCR (seguendo la NOTA di seguito) in una provetta per PCR da 0,2 mL (volume finale = 50 μL, per una condizione). Di solito, ci saranno almeno due condizioni: il KO e il controllo non modificato con il solo Cas9.
NOTA: DNA genomico purificato: 10 μL; Primer diretto e inverso (10 μM): 2,5 μL ciascuno, la concentrazione finale di 500 nM; Tampone di reazione (5x): 10 μL. Vortice bene prima dell'aggiunta. Miscela di dNTP (25 mM di ciascuno): 0,6 μL, concentrazione finale di 0,3 mM per ogni dNTP. DNA polimerasi: 0,5 μl; Acqua ultrapura: 23,9 μL. Vortice e centrifugare brevemente (pulsare per 3 s). - Posizionare le provette nel termociclatore ed eseguire il programma PCR con le seguenti impostazioni:
NOTA: Un ciclo a 95 °C per 5 minuti, seguito da 30 cicli [98 °C per 20 s (fase di denaturazione), X °C per 15 s (fase di ricottura), 72 °C per 45 s (fase di allungamento)], quindi un ciclo finale a 72 °C per 2 minuti. Al termine dell'amplificazione, rimuovere le provette, vorticare e centrifugare brevemente (pulsare per 3 s). La temperatura di ricottura (X) è la temperatura di fusione (Tm) degli inneschi meno 5 °C. - Per una popolazione policlonale modificata: in una nuova provetta da 0,2 mL, aggiungere 17,5 μL di amplicone PCR e 2 μL di NEBuffer 2 (10x) per un volume finale di 19,5 μL. Vortice e centrifugare brevemente (pulsare per 3 s). In alternativa, per lo screening di cloni di singole cellule, mescolare 1:1 dei prodotti PCR sia dalle cellule di controllo modificate che da quelle non modificate (passaggio 5.6) (Figura 1 supplementare).
- Per la formazione eteroduplex, posizionare le provette nel termociclatore ed eseguire il seguente programma: un ciclo a 95 °C per 10 min, uno con una rampa di -2 °C/s da 95 a 85 °C, uno con una rampa di -0,3 °C/s da 85 a 25 °C e un ciclo di raffreddamento finale a 10 °C in HOLD.
- Dopo la formazione eteroduplex, aggiungere 0,5 μl di soluzione T7EI nella provetta. Incubare a 37 °C per 30 min.
- Preparare un gel di agarosio all'1,2%:
- Pesare l'agarosio in un flacone di vetro e aggiungere il volume appropriato di 1x tampone TAE (40 mM di Tris-acetato, 1 mM di EDTA, pH 8,3). Mettetelo nel microonde facendo attenzione a non avvitare bene il tappo. Scaldare più volte fino a quando i cristalli di agarosio non saranno completamente sciolti.
NOTA: Se necessario, mescolare la soluzione. Non lasciarlo bollire, altrimenti il volume diminuirà, modificando la concentrazione di agarosio. - Aggiungere la colorazione in gel DNA diluita a 1/20.000 e mescolare bene.
- Versare la soluzione di agarosio in uno stampo, aggiungere un pettine e lasciare solidificare a temperatura ambiente.
- Pesare l'agarosio in un flacone di vetro e aggiungere il volume appropriato di 1x tampone TAE (40 mM di Tris-acetato, 1 mM di EDTA, pH 8,3). Mettetelo nel microonde facendo attenzione a non avvitare bene il tappo. Scaldare più volte fino a quando i cristalli di agarosio non saranno completamente sciolti.
- Preparare i campioni per l'elettroforesi su gel mescolando 5 μl di prodotti per la digestione T7EI (fase 5.9) o il prodotto per PCR non digerito con 5 μl di acqua e 2 μl di colorante caricante DNA 6x. Caricare i campioni e la scala dimensionale e migrare a 80 V per 45 minuti (il tempo può essere adattato a seconda delle dimensioni previste dei frammenti di DNA). Una volta terminata la migrazione, acquisire un'immagine del gel con un sistema di imaging appropriato.
6. Validazione dell'editing genetico mediante analisi di sequenziamento Sanger (Figura 1.6)
- Per caratterizzare la modificazione genetica, purificare i prodotti della PCR (step 5.4) e, dopo il sequenziamento Sanger, analizzare i risultati con lo strumento ICE (https://www.synthego.com/products/bioinformatics/crispr-analysis).
- Controlla se una proteina può essere prodotta nonostante le modifiche.
- Scarica i risultati ICE e apri il file contribs.txt.
- Confrontate le sequenze modificate con la controparte WT. Mappare gli indel e verificare che si siano originati nella regione genomica bersaglio. Valuta le loro dimensioni. Se la lunghezza dell'indel non è un multiplo di tre, si verificherà uno spostamento di fotogrammi e verrà eventualmente introdotto un PTC. L'mRNA mutato subirà una degradazione mediata da NMD.
7. Isolamento di cloni su una singola cellula limitando la diluizione (Figura 1.7)
NOTA: L'isolamento di cloni a singola cellula non è obbligatorio. Tuttavia, se si sceglie di farlo, è importante caratterizzare più cloni e confrontare il loro fenotipo con la popolazione policlonale originale.
- Prelevare un campione di sospensione cellulare, diluirlo 1/2 con terreno di coltura e valutare la concentrazione (passaggio 2.7.1). La diluizione aumenta la precisione del conteggio.
- Eseguire da 2 a 3 fasi di diluizione seriali per raggiungere una concentrazione di 7 cellule/mL. Erogare 100 μl di sospensione cellulare per pozzetto in una piastra a fondo tondo da 96 pozzetti (ovvero 0,7 celle per pozzetto). Lasciare decantare la cellula per alcune ore prima di osservare le piastre al microscopio per identificare i pozzetti contenenti una cellula.
- Monitora regolarmente la formazione e la crescita delle colonie e trasferisci le cellule in una piastra di coltura o in un pallone più grande, quando necessario.
8. Caratterizzazione funzionale di cellule THP-1 KO SAMHD1 mediante saggio di restrizione HIV-1
- Seminare THP-1 in una piastra di coltura a 24 pozzetti con 0,25 x 106 cellule per pozzetto in 300 μL di RPMI integrato con il 10% di FBS, l'1% di penicillina-streptomicina (RPMI completo) e contenente 300 ng/mL di PMA per la differenziazione. Conservare la piastra in un'incubatrice umidificata (37 °C, 5% CO2) per 24 ore.
- Sostituire il mezzo con un mezzo RPMI completo privo di PMA e rimettere la piastra nell'incubatrice per altre 24 ore.
- Utilizzando una pompa a vuoto, aspirare tutto il terreno di coltura. Aggiungere 250 μl di soluzione virale contenente stock per HIV-1-GFP pseudotipizzata VSVg (MOI = 0,5 IFU/cellula). Includi un controllo non infetto. Posizionare la piastra a 4 °C per 2 ore per sincronizzare l'infezione.
- Lavare le celle una volta con RPMI. Aggiungere 500 μl di RPMI completo a ciascun pozzetto e incubare per 48 ore in condizioni standard (il tempo può essere regolato).
- Dopo aver rimosso il terreno di coltura, lavare le cellule una volta con 1x PBS freddo. Quindi, aggiungere 100 μl di tripsina-EDTA allo 0,05% in ciascun pozzetto. Porre la piastra in un incubatore a 37 °C fino a quando le cellule non sono completamente staccate (5 minuti dovrebbero essere sufficienti) prima di aggiungere 200 μL di RPMI completo per inattivare l'enzima. Trasferire 250 μl da ciascun pozzetto in una piastra a fondo tondo da 96 pozzetti (assicurarsi che il citometro a flusso accetti piastre di coltura).
- Centrifugare la piastra (757 x g, 3 min, decelerazione = 6, 20 °C) e aspirare il surnatante utilizzando una pipetta multicanale. Risospendere il pellet con 100 μl di PFA al 4% e incubare a 4 °C per 10 minuti. Aggiungere 100 μl di 1X PBS freddo.
- Analizzare il tasso di infezione, rappresentato dalle cellule GFP-positive, mediante citometria a flusso (vedere la Figura 2 supplementare per una strategia di gating suggerita).
Risultati
Una linea cellulare THP-1 è stata generata esprimendo stabilmente la proteina reporter GFP (THP-1_GFP) (Figura 2A) e utilizzata come strumento per stabilire un protocollo per un'efficiente edizione genica mediata da CRISPR-Cas9. A questo scopo, 3 sgRNA mirati al gene EGFP sono stati progettati con lo strumento web CRISPOR29 (Figura 2B), che sono stati simultaneamente complessati con Cas9 a un rapporto molare di 9:1 per formare RNP prima della consegna nelle cellule mediante elettroporazione utilizzando impostazioni diverse. Successivamente, le cellule sono state coltivate in un solo terreno RPMI contenente FBS al 20% (Figura 2C, pannello superiore) o diluite 1:1 con terreno condizionato (Figura 2C, pannello inferiore). La proliferazione cellulare e l'espressione della GFP, come lettura dell'efficienza del KO per EGFP , sono state monitorate nel tempo. Per diverse condizioni, la percentuale di cellule GFP-positive è diminuita drasticamente, raggiungendo una riduzione del >90% il giorno 7 dopo l'elettroporazione (pe). Al giorno 3 a.e., il numero di cellule si era dimezzato, probabilmente a causa dell'impatto negativo dell'elettroporazione sulla vitalità cellulare. Tuttavia, il numero di cellule è aumentato di nuovo e il tempo di raddoppio è tornato a un ritmo normale entro il giorno 7 pe (Figura 2C, pannello superiore). L'uso di terreni condizionati non ha favorito il recupero cellulare nelle nostre condizioni sperimentali (Figura 2C, pannello inferiore). Sulla base di questi risultati, sono stati scelti tre impulsi di 10 ms di 1500 V per gli esperimenti di follow-up.
Il KO EGFP mediato da CRISPR-Cas9 è stato quindi caratterizzato a livello genomico. Per questo, il DNA genomico delle cellule THP-1_GFP è stato estratto, elettroporato con gli RNP Cas9:sgRNA (modificato) o Cas9 da solo (non modificato) e utilizzato come modello per amplificare con PCR una regione di 891 bp contenente il sito bersaglio. Anche le cellule parentali THP-1 prive di EGFP sono state incluse come controllo negativo. Successivamente, il prodotto PCR delle cellule THP-1_GFP modificate è stato preparato per il rilevamento del mismatch mediante il test T7EI, seguito dalla separazione dei frammenti di DNA mediante elettroforesi su gel di agarosio. Una banda di ~900 bp corrispondente alla dimensione attesa dell'amplicone WT è stata prontamente visualizzata per le cellule THP-1_GFP non modificate, ma non per il controllo THP-1 parentale (Figura 2D, confronta le linee 2 e 3). Nel caso delle cellule THP-1_GFP modificate, la banda di ~900 bp è diventata non rilevabile e sostituita da una a migrazione più rapida, la cui dimensione corrispondeva alla perdita di un frammento di ~75 bp conseguente all'editing CRISPR (Figura 2D, riga 4). Questo modello di bande è stato alterato dopo la digestione di T7EI, causando la comparsa di frammenti di DNA più piccoli, confermando ulteriormente il successo dell'editing del sito bersaglio (Figura 2D, riga 5). Per ottenere informazioni sull'efficienza dell'editing e sui genotipi specifici della popolazione modificata, l'amplicone PCR è stato sequenziato utilizzando il metodo Sanger, seguito dall'analisi con lo strumento ICE26. Il campione di controllo non modificato è stato elaborato in parallelo. Abbiamo recuperato punteggi indel e KO del 100%, corrispondenti rispettivamente alla percentuale di sequenze non-WT nel campione modificato e alla proporzione di indel che probabilmente portano a un KO funzionale (Figura 2E, a sinistra). Un'analisi dettagliata dello spettro delle dimensioni e della frequenza dell'indel ha mostrato la presenza di due popolazioni dominanti portatrici di una delezione di 62 bp o 76 bp e che rappresentano rispettivamente il 57% o il 37% delle sequenze totali (Figura 2E, a destra). Questi risultati suggeriscono che l'sgRNA #1, in combinazione con l'sgRNA #2 o #3, ha innescato DSB concomitanti nel gene bersaglio (Figura 2B).
Questo protocollo è stato poi applicato per inattivare il gene endogeno codificante SAMHD1 (Figura 3A). La caratterizzazione della linea cellulare policlonale SAMHD1 KO THP-1 ha incluso il monitoraggio della proliferazione cellulare nel tempo, che corrispondeva a quella delle cellule THP-1 non modificate (Figura 3B). L'esito dell'editing genetico è stato valutato mediante amplificazione basata su PCR della sequenza bersaglio, che ha rivelato una banda più piccola per le cellule KO SAMHD1 rispetto al controllo solo Cas9 (Figura 3C, confronta le linee 2 e 4), indicativo di una perdita di sequenza di DNA. Queste osservazioni sono state confermate dal test T7EI, che mostra l'aspetto di prodotti di scissione per le cellule SAMHD1 KO, ma non inedite (Figura 3C, confronta le linee 3 e 5). L'analisi dei dati di sequenziamento Sanger con lo strumento ICE ha restituito punteggi indel e KO rispettivamente del 100% e del 97%. Le principali modifiche all'interno del locus SAMHD1 sono state delezioni di sequenze di 170 bp, 93 bp o 104 bp rispettivamente nel 46%, 13% e 10% delle sequenze (Figura 3D). I livelli di espressione proteica nel lisato cellulare grezzo sono stati valutati anche mediante immunoblotting. La banda di ~70 kDa coerente con la dimensione di SAMHD1 endogena diventa praticamente non rilevabile dopo l'editing, corrispondente a una riduzione stimata del >97% (Figura 3E). Infine, è stata valutata anche la caratterizzazione fenotipica delle linee cellulari policlonali SAMHD1 KO THP-1 testando la suscettibilità all'infezione da HIV-1. In accordo con il suo ben noto ruolo antivirale 30,31, l'inattivazione del locus SAMHD1 è stata accompagnata da un aumento del tasso di infezione da HIV-1 (Figura 3F).
Parallelamente, sono stati generati cloni a singola cellula limitando la diluizione. L'amplificazione del sito bersaglio genomico dai cloni KO SAMHD1 ha prodotto prodotti PCR con una maggiore mobilità elettroforetica rispetto a quelli delle cellule di controllo non modificate (Figura 4A). L'analisi basata sull'ICE dei dati di sequenziamento Sanger ha restituito un punteggio indel del 100% per tutti i cloni a singola cellula selezionati tranne uno (clone A2) (Figura 4B, a sinistra). La caratterizzazione dei risultati dell'editing a livello genomico ha restituito una singola sequenza per il clone A3 con una delezione di 98 bp situata a 28 pb a valle dell'ATG iniziale, portando così a un cambiamento fuori dal fotogramma (Figura 4B, a destra). Gli altri cloni contenevano tre o più sequenze (clone B1), indicative della presenza di alleli che hanno subito diversi eventi di editing e/o più di una cellula per pozzetto nella fase di diluizione limite (Figura 4B, a destra). In particolare, l'11% delle sequenze di cloni A4 e A7 reca indels a monte dell'ATG iniziale, lasciando inalterata la sequenza codificante. Inoltre, l'1% delle sequenze di clone A7 mancava di un frammento di 123 bp a 9 bp a valle dell'ATG iniziale, con conseguente delezione in-frame di 41 amminoacidi. Coerentemente con questi risultati, gli studi funzionali hanno rivelato che i cloni con un locus SAMHD1 interrotto (A3, A8, B1, B3), quindi privi di espressione di SAMHD1, erano altamente permissivi all'infezione da HIV-1 (Figura 4C). Al contrario, i cloni A4 e A7, in cui SAMHD1 è intatto, erano refrattari quanto i cloni parentali o di controllo non modificati di THP-1 (Figura 4C). In conclusione, è stato confermato il successo dell'editing del locus SAMHD1 nel clone A3.
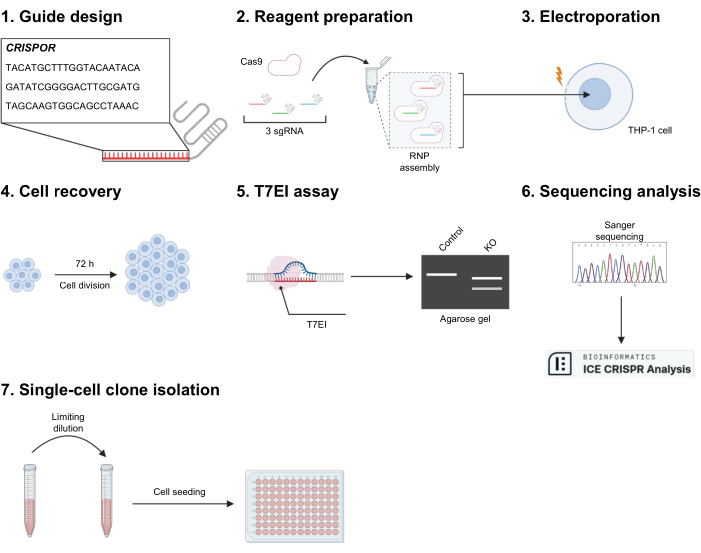
Figura 1: Cenni sperimentali. (1) CRISPOR viene utilizzato per generare sequenze di sgRNA contro l'esone bersaglio. Tre di essi sono selezionati in base all'elevata efficacia on-target e alla debole previsione off-target. (2) I tre gRNA vengono miscelati con Cas9 per assemblare la ribonucleoproteina (RNP), ottenendo un mix di tre diversi RNP. (3) Le cellule vengono elettroporate per consentire l'ingresso degli RNP. (4) Le cellule vengono trasferite in una piastra di coltura a 24 pozzetti e lasciate riposare per almeno 72 ore. (5) L'editing genetico viene prima testato qualitativamente mediante un saggio di digestione T7EI, e (6) per le condizioni convalidate, ulteriormente caratterizzate dal sequenziamento Sanger e dall'analisi ICE. (7) Se necessario, le cellule possono essere seminate dopo aver limitato la diluizione per produrre popolazioni clonali. Clicca qui per visualizzare una versione più grande di questa figura.
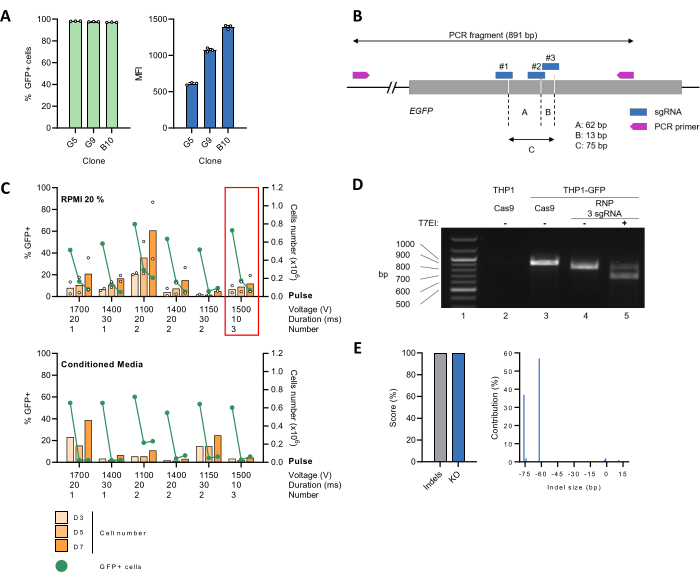
Figura 2: Impostazione delle impostazioni di elettroporazione per EGFP KO in cellule THP-1 e convalida dell'editing genetico. (A) Le cellule THP-1 sono state trasdotte con vettori lentivirali che codificano per il gene reporter EGFP . I cloni di singole cellule sono stati ottenuti limitando la diluizione seguita dall'espansione in coltura per 30 giorni. L'analisi mediante citometria a flusso ha confermato che praticamente l'intera popolazione cellulare esprimeva GFP. L'intensità mediana della fluorescenza (MFI) della popolazione GFP-positiva, indicativa del numero di copie EGFP integrato, variava tra diversi cloni cellulari. Il clone G5 è stato scelto per gli studi di follow-up. (B) Rappresentazione schematica del gene EGFP , inclusi i siti di legame sia per sgRNA che per primer. I siti di taglio Cas9 sono indicati dalle linee tratteggiate. (C) Le cellule THP-1_GFP indifferenziate (2 x 105) sono state miscelate con RNP di Cas9:sgRNA preassemblati prima dell'elettroporazione con le impostazioni indicate. Successivamente, le cellule sono state trasferite in una piastra a 24 pozzetti contenente 500 μl di terreno RPMI preriscaldato al 20% contenente FBS da solo o diluite 1:1 con terreno condizionato prodotto da una coltura cellulare THP-1 di 48 ore. La proliferazione (barre arancioni) e la percentuale di cellule GFP-positive (linea verde) sono state monitorate nel tempo. I dati rappresentano la media per n = 2 repliche biologiche. La cornice rossa evidenzia la condizione selezionata per gli esperimenti successivi. (D) È stato estratto il DNA genomico di cellule THP-1_GFP modificate (RNP 3 sgRNA) o non modificate (Cas9) e la regione circostante il locus bersaglio è stata amplificata mediante PCR. Le cellule THP-1 parentali sono state utilizzate come controllo negativo. Gli ampliconi hanno subito un ciclo di denaturazione/rinaturazione, che ha portato alla formazione di un eteroduplex. Successivamente, le discrepanze sono state rilevate dal test T7EI. I frammenti di DNA sono stati separati mediante elettroforesi su gel di agarosio all'1,2%. (E) I prodotti di PCR purificati sono stati sequenziati con il metodo Sanger, seguiti da analisi con l'algoritmo ICE. I punteggi Indel e KO indicano la percentuale di sequenze non-WT nel campione e la proporzione di cellule con un frameshift o 21+ bp indel, rispettivamente. Clicca qui per visualizzare una versione più grande di questa figura.
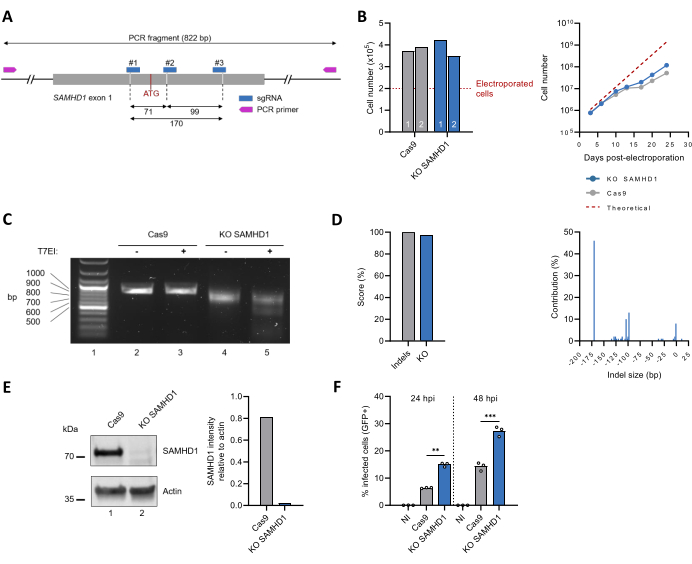
Figura 3: Produzione e caratterizzazione di una popolazione di cellule policlonali SAMHD1 KO THP-1. (A) Rappresentazione schematica dell'esone 1 di SAMHD1, inclusi i siti di legame per sgRNA e primer. I siti di taglio Cas9 sono indicati dalle linee tratteggiate e il codone ATG iniziale è mostrato in rosso. (B) Tre giorni dopo l'elettroporazione, le cellule vitali (n = 2) sono state quantificate utilizzando il test di esclusione del blu di tripano (diagramma a sinistra). La crescita cellulare è stata monitorata per 25 giorni (diagramma a destra). La linea tratteggiata corrisponde al numero teorico di cellule sulla base di un tempo di raddoppio stimato di 49 ore. (C) Sono stati valutati i risultati dell'editing a livello genomico, come mostrato nella Figura 2D. (D) I genotipi successivi all'editing CRISPR sono stati identificati e quantificati mediante analisi basate su ICE delle sequenze di Sanger. (E) La proteina contenuta nel lisato grezzo di cellule THP-1 modificate e non modificate è stata separata mediante migrazione su gel SDS-PAGE e, successivamente, visualizzata mediante immunoblotting utilizzando anticorpi contro SAMHD1. L'actina è stata utilizzata come controllo del carico. L'intensità delle bande SAMHD1 e actina è stata quantificata mediante densitometria con il software ImageJ. Le cellule THP-1 non modificate (Cas9) e KO SAMHD1 sono state differenziate mediante trattamento con PMA (300 ng/mL, 24 ore) e successivamente sottoposte a test con un virus HIV-1 pseudotipizzato VSVg che esprime GFP come reporter. La percentuale di cellule GFP-positive è stata valutata mediante citometria a flusso a 24 ore e 48 ore dopo l'infezione (hpi). NI: non infetto. **P < 0,01; P < 0,001 mediante test t non accoppiato con la correzione di Welch. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 4: Produzione e caratterizzazione di cloni a singola cellula SAMHD1 KO. (A) L'esito dell'editing per cloni a singola cellula SAMHD1 KO selezionati è stato valutato monitorando la mobilità dei frammenti di DNA prodotti dall'amplificazione PCR della regione contenente il sito bersaglio mediante elettroforesi su gel di agarosio. (B) I dati di sequenziamento Sanger sono stati analizzati con lo strumento ICE, come in Figura 2E. Le barre nel grafico a destra rappresentano la diversità degli indel in ciascuna popolazione cellulare. La barra verde rappresenta la sequenza dominante e la dimensione totale della delezione associata. I numeri tra parentesi indicano la dimensione degli spazi vuoti non continui. (C) La permissività dei cloni unicellulari non modificati e SAMHD1 KO per l'infezione da HIV-1 è stata testata come nella Figura 3F e confrontata con quella delle cellule parentali THP-1 non elettroporate o dei cloni cellulari non modificati (Cas9). Il tasso di infezione è stato valutato a 48 hpi. *P < 0,05; **P < 0,01; P < 0,001; P < 0,0001; ns P≥ 0,05 da Brown-Forsythe e Welch ANOVA con il test di confronto multiplo di Dunnett. Clicca qui per visualizzare una versione più grande di questa figura.
Figura 1 supplementare: Valutazione del risultato dell'editing per cloni di singole cellule KO mediante test T7EI adattato. Il DNA genomico di cloni di singole cellule KO o di cellule non modificate è stato purificato e la regione circostante il locus bersaglio è stata amplificata mediante PCR. Gli ampliconi di ciascun clone di singola cellula sono stati mescolati in un rapporto 1:1 con quelli delle cellule di controllo non modificate. Dopo la formazione di eteroduplex e la digestione di T7EI, il prodotto è stato analizzato mediante elettroforesi su gel di agarosio. I cloni modificati producono frammenti di digestione multipli (cloni A, B, C, D ed E), mentre quelli non modificati mostrano una singola banda WT (cloni F e G). Clicca qui per scaricare questo file.
Figura 2 supplementare: Strategia di gating della citometria a flusso per quantificare la percentuale di cellule THP-1 trattate con PMA infetta da HIV-1. Utilizzando l'area di dispersione diretta (FSC-A) contro l'area di dispersione laterale (SSC-A), viene definito un primo gate per eliminare i detriti e selezionare le celle THP-1 trattate con PMA. Successivamente, le singole celle vengono selezionate con un grafico SSC-A rispetto a un grafico SSC-H. Infine, le cellule GFP-positive vengono rilevate con GFP-A contro FSC-H. Clicca qui per scaricare questo file.
Discussione
Qui, viene descritto un protocollo per ottenere un editing mediato da CRISPR della linea cellulare THP-1. L'approccio si basa sul trasferimento di RNP sgRNA/Cas9 preassemblati mediante elettroporazione/nucleofezione. Questa strategia è stata scelta per limitare gli effetti off-target che potenzialmente insorgono in seguito all'integrazione lentivirale della cassetta sgRNA/Cas9, producendo un'espressione persistente della nucleasi. Sono stati selezionati più sgRNA che hanno come bersaglio il gene di interesse per ottenere un editing affidabile ed efficiente, che aumenta la probabilità di creare indel genomici, portando alla perdita dell'espressione proteica e al KO32 funzionale. Particolare attenzione è stata prestata alle impostazioni di elettroporazione per ottenere una permeabilizzazione ottimale della membrana cellulare, necessaria per garantire una consegna efficiente degli RNP sgRNA/Cas9 prevenendo al contempo un'eccessiva morte cellulare33. A questo proposito, un parametro cruciale è il vigore della coltura cellulare prima dell'elettroporazione (cioè un basso numero di passaggi).
L'editing mediato da CRISPR-Cas9 è stato convalidato a livello genetico con il test di rilevamento del mismatch T7EI, che fornisce una valutazione rapida ed economica di più campioni per la preselezione prima del sequenziamento Sanger. Questo metodo è stato inizialmente sviluppato per stimare l'efficienza di editing utilizzando un singolo sgRNA confrontando l'intensità degli ampliconi a lunghezza intera e dei prodotti di scissione34,35. Degno di nota, i grandi indel (>70 bp) prodotti quando si utilizzano più sgRNA possono essere rilevati confrontando la mobilità dei frammenti di DNA ottenuti mediante amplificazione PCR dei loci bersaglio da cellule modificate rispetto a quelle non modificate mediante elettroforesi su gel standard. Tuttavia, il test T7EI potrebbe essere utile per accertare la presenza di sequenze che ospitano piccoli indel (>1bp) in una popolazione cellulare policlonale o, utilizzando il protocollo adattato, in una popolazione di cellule monoclonali.
La presenza di mutazioni out-of-frame on-target, che interrompono la sequenza codificante, è stata confermata da un approccio semplice, in cui il genoma dell'intera popolazione cellulare viene estratto e utilizzato come matrice per l'amplificazione della PCR. Successivamente, gli ampliconi purificati vengono sottoposti a sequenziamento Sanger in massa, seguito da analisi bioinformatica con lo strumento ICE. Nel complesso, questo metodo supera la necessità di subclonare i prodotti della PCR in un plasmide e di trasformare i prodotti della legatura in batteri per isolare colonie di singole cellule, che vengono sequenziate individualmente.
Una volta convalidata l'inattivazione del locus target, è necessaria una caratterizzazione approfondita della linea cellulare modificata per attribuire con sicurezza un determinato fenotipo alla mancanza della proteina di interesse ed escludere effetti imprevisti derivanti da cambiamenti fuori bersaglio, ma anche su bersaglio che potrebbero influire sulla struttura e/o sulla funzione dell'mRNA.
L'assenza di espressione proteica deve essere confermata dall'immunoblotting e/o dall'immunofluorescenza utilizzando possibilmente due anticorpi che riconoscano epitopi diversi. Infatti, l'introduzione di un PTC in un esone precoce vicino al codone ATG canonico è associata al rischio di eventi ITL, che producono varianti proteiche troncate N-terminali36. Da notare, Tuladhar et al. hanno riportato il rilevamento di specie proteiche aberranti in una percentuale significativa (~50%) di cellule modificate con CRISPR di origine commerciale o interna18,19. La presenza di nuovi prodotti proteici è stata attribuita a processi diversi e potenzialmente specifici del tipo di cellula, come il salto dell'esone contenente indel o l'emergere di siti di ingresso dei ribosomi interni (IRES) che causano l'inizio della sintesi proteica a TIS alternativo a valle del codone ATG canonico. Ci sono anche indizi che l'ITL sia accoppiato con una fuga da NMD in determinate circostanze37,38. Allo stesso modo, le strategie di editing del genoma mirate a un esone tardivo potrebbero non riuscire a innescare la NMD dell'mRNA mutato alla base dell'ablazione inefficace dell'espressione genica20. Infine, se Cas9 è guidato da più sgRNA, potrebbero verificarsi complessi riarrangiamenti cromosomici, tra cui inversioni o grandi delezioni di frammenti di DNA39, che potrebbero alterare la struttura e l'espressione del genoma, in particolare quando gli elementi regolatori sono mirati40.
Per mitigare ulteriormente l'impatto degli esiti non intenzionali dell'editing del genoma, sono necessari studi funzionali per confermare la relazione tra la perdita di espressione proteica e il fenotipo conseguente (cioè l'attività antivirale). Se le cellule modificate vengono subclonate, il comportamento di più cloni tra loro e con la popolazione policlonale dovrebbe essere confrontato. L'esecuzione di esperimenti di salvataggio, che consistono nel ripristinare l'espressione genica per invertire il fenotipo delle cellule modificate in un fenotipo WT, rafforzerà anche la specificità ed escluderà possibili eventi fuori bersaglio.
Sebbene questo protocollo sia stato concepito per l'editing di cellule THP-1, fornisce una struttura generale per il flusso di lavoro KO di CRISPR-Cas9 che può essere adattata ad altre linee cellulari. Potrebbe essere necessario regolare i parametri di elettroporazione e, se viene applicata una tensione superiore a 1800 V, è necessario utilizzare un tampone di risospensione T, secondo le istruzioni del produttore. Un'altra variabile da considerare è il rapporto di concentrazione tra Cas9 e sgRNA durante l'assemblaggio dell'RNP. Sebbene il rapporto 1:9 abbia funzionato bene in questo caso, modificarlo può migliorare l'efficienza dell'editing in altre circostanze. Infine, anche se non è vantaggioso, l'uso di terreni condizionati per il recupero cellulare dopo l'elettroporazione dovrebbe essere testato, poiché l'effetto potrebbe essere diverso con diverse linee cellulari.
Divulgazioni
Tutti gli autori non hanno conflitti di interesse.
Riconoscimenti
Siamo grati a JP Concordet (MNHN, U1154/UMR7196, Parigi), G. Bossis (IGMM, Montpellier) e D. Schlüter (Hannover Medical School, Germania) per la condivisione dei protocolli e per la discussione. Questo progetto ha ricevuto finanziamenti dal programma di ricerca e innovazione Horizon 2020 dell'Unione Europea (accordo di sovvenzione n. 101017572 alla AZ) e ANRS (ECTZ162721 di sovvenzione alla AZ). L'infrastruttura di ricerca IDMIT (Infectious Disease Model and Innovative Therapies) è sostenuta dal "Programme investissement d'avenir (PIA)" di riferimento ANR_11_INSB_0008.
Materiali
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filter | ClearLine | 146560 | _ |
| 0.4 % trypan blue | Beckman Coulter | 383200 | _ |
| 1.5 mL tube | Eppendorf | 3810X | _ |
| 24-well plate | Corning | 353047 | _ |
| 6x TriTrack DNA Loading Dye | Thermo scientific | R1161 | _ |
| 75 cm² Culture Flask Vented Cap | Corning | 353136 | _ |
| 8-Strip PCR Tubes with Caps | Life technologies | AM12230 | _ |
| 96-well plates Flat bottom | Corning | 353072 | _ |
| 96-well plates Round bottom | Corning | 353077 | _ |
| Agarose | Euromedex | D5 | _ |
| ATGpr | _ | _ | https://atgpr.dbcls.jp/ |
| ChemiDoc Imaging System | BIO-RAD | 12003153 | _ |
| Counting slide | NanoEntek | DHC-N04 | _ |
| CRISPOR | _ | _ | http://crispor.gi.ucsc.edu/ |
| DPBS | Gibco | 14190094 | _ |
| Ensembl | EMBL-EBI | _ | https://www.ensembl.org/index.html |
| Fetal Bovine Serum | Sigma-Aldrich | F7524 | _ |
| FlowJo | BD Life Sciences | v10.10 | _ |
| GeneRuler 100 bp Plus DNA Ladder | Thermo scientific | SM0323 | _ |
| Genome Data Viewer | NCBI | _ | https://www.ncbi.nlm.nih.gov/gdv/ |
| GraphPad Prism | Dotmatics | _ | Version 9.3.1 |
| Herculase II Fusion DNA Polymerases | Agilent | 600679 | _ |
| ICE CRISPR Analysis Tool | Synthego | _ | https://www.synthego.com/products/bioinformatics/crispr-analysis |
| Image Lab Touch | BIO-RAD | _ | Version 2.4.0.03 |
| NEBuffer 2 | New England Biolabs | B7002S | Included with T7EI M0302S |
| Neon Kit, 10 µL | Invitrogen | MPK1025K | Electroporation kit containing tips, tubes, buffer R and E |
| Neon Transfection System | Invitrogen | MPK5000 | _ |
| NetStart 1.0 | _ | _ | https://services.healthtech.dtu.dk/services/NetStart-1.0/ |
| Nuclease-free Water | Synthego | _ | _ |
| PCR primer (EGFP) | Eurofins | _ | Fw : GGAATGCAAGGTCTGTTGAATG ; Rev : CACCTTGATGCCGTTCTTCT |
| PCR primer (SAMHD1) | Eurofins | _ | Fw : CGGGATTGATTTGAGGACGA ; Rev : GGGTGGCAAGTTAGTGAAGA |
| Penicillin-streptomycin (10,000 U/mL) | Gibco | 15140122 | _ |
| PFA | Electron Microscopy Sciences | 15714 | _ |
| PMA | Sigma-Aldrich | P8139 | _ |
| PrimerQuest | IDT | _ | https://eu.idtdna.com/pages/tools/primerquest |
| QIAquick PCR Purification Kit | Qiagen | 28104 | _ |
| QuickExtract DNA Extraction Solution | Biosearch Technologies | QE09050 | _ |
| RPMI 1640, GlutaMAX | Gibco | 61870010 | _ |
| SnapGene Viewer | Dotmatics | _ | Version 7 |
| SpCas9 2NLS Nuclease | Synthego | _ | _ |
| SYBR Safe DNA Gel Stain | Invitrogen | S33102 | _ |
| Synthetic sgRNA (EGFP) | Synthego | _ | #1 : CGCGCCGAGGUGAAGUUCGA ; #2 : UUCAAGUCCGCCAUGCCCGA ; #3 : CAACUACAAGACCCGCGCCG |
| Synthetic sgRNA (SAMHD1) | Synthego | _ | #1 : AUCGCAACGGGGACGCUUGG ; #2 : GCAGUCAAGAACCUCGGCGC ; #3 : CCAUCCCGACUACAAGACAU |
| Syringe Plastipak Luer Lock | BD | 301229 | _ |
| T100 Thermal Cycler | BIO-RAD | 1861096 | _ |
| T7 endonuclease I | New England Biolabs | M0302S | _ |
| TAE buffer UltraPure, 10x | Invitrogen | 15558026 | 400 mM Tris-Acetate, 10 mM EDTA |
| THP-1 cells | ATCC | TIB-202 | _ |
| Trypsin-EDTA (0,05 %) | Gibco | 25300054 | _ |
| ZE5 Cell Analyzer | BIO-RAD | 12014135 | _ |
Riferimenti
- Tsuchiya, S., et al. Establishment and characterization of a human acute monocytic leukemia cell line (THP-1). Int J Cancer. 26 (2), 171-176 (1980).
- Danis, V. A., Millington, M., Hyland, V. J., Grennan, D. Cytokine production by normal human monocytes: inter-subject variation and relationship to an IL-1 receptor antagonist (IL-lRa) gene polymorphism. Clin Exp Immunol. 99, (1995).
- Bol, S. M., et al. Donor variation in in vitro HIV-1 susceptibility of monocyte-derived macrophages. Virology. 390 (2), 205-211 (2009).
- Appleby, L. J., et al. Sources of heterogeneity in human monocyte subsets. Immunol Lett. 152 (1), 32-41 (2013).
- O'Neill, M. B., et al. Single-cell and bulk RNA-sequencing reveal differences in monocyte susceptibility to Influenza A virus infection between Africans and Europeans. Front Immunol. 12, 768189 (2021).
- Wen, Y., et al. Comparability study of monocyte-derived dendritic cells, primary monocytes, and THP1 cells for innate immune responses. J Immunol Methods. 498, 113147 (2021).
- Inagaki, Y., et al. Interferon-g-induced apoptosis and activation of THP-1 macrophages. Life Sci. 71 (21), 2499-2508 (2002).
- Boonkaewwan, C., Toskulkao, C., Vongsakul, M. Anti-inflammatory and immunomodulatory activities of stevioside and its metabolite steviol on THP-1 cells. J Agric Food Chem. 54 (3), 785-789 (2006).
- Chanput, W., et al. β-Glucans are involved in immune-modulation of THP-1 macrophages. Mol Nutr Food Res. 56 (5), 822-833 (2012).
- Cui, J., et al. USP3 inhibits type I interferon signaling by deubiquitinating RIG-I-like receptors. Cell Res. 24 (4), 400-416 (2014).
- Chen, S., et al. SAMHD1 suppresses innate immune responses to viral infections and inflammatory stimuli by inhibiting the NF-κB and interferon pathways. Proc Natl Acad Sci USA. 115 (16), E3798-E3807 (2018).
- Pradhananga, S., Spalinskas, R., Poujade, F. A., Eriksson, P., Sahlén, P. Promoter anchored interaction landscape of THP-1 macrophages captures early immune response processes. Cell Immunol. 355, 104148 (2020).
- Mezzasoma, L., Talesa, V. N., Romani, R., Bellezza, I. Anp and BNP exert anti-inflammatory action via npr-1/cgmp axis by interfering with canonical, non-canonical, and alternative routes of inflammasome activation in human THP1 cells. Int J Mol Sci. 22 (1), 1-17 (2021).
- Martinat, C., et al. SUMOylation of SAMHD1 at Lysine 595 is required for HIV-1 restriction in non-cycling cells. Nat Commun. 12 (1), 4582 (2021).
- Rensen, E., et al. Clustering and reverse transcription of HIV-1 genomes in nuclear niches of macrophages. EMBO J. 40 (1), e105247 (2021).
- Ikeda, T., et al. APOBEC3 degradation is the primary function of HIV-1 Vif determining virion infectivity in the myeloid cell line THP-1. mBio. 14 (4), e0078223 (2023).
- Jinek, M., et al. A programmable dual RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337, 816-821 (2012).
- Kurosaki, T., Maquat, L. E. Nonsense-mediated mRNA decay in humans at a glance. J Cell Sci. 129 (3), 461-467 (2016).
- Tuladhar, R., et al. CRISPR-Cas9-based mutagenesis frequently provokes on-target mRNA misregulation. Nat Commun. 10 (1), 4056 (2019).
- Embree, C. M., Abu-Alhasan, R., Singh, G. Features and factors that dictate if terminating ribosomes cause or counteract nonsense-mediated mRNA decay. J Biol Chem. 298 (11), 102592 (2022).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Schnoor, M., et al. Efficient non-viral transfection of THP-1 cells. J Immunol Methods. 344 (2), 109-115 (2009).
- Tang, X., et al. A method for high transfection efficiency in THP-1 suspension cells without PMA treatment. Anal Biochem. 544, 93-97 (2018).
- Ji, W., Zhang, L., Liu, X. Protocol using lentivirus to establish THP-1 suspension cell lines for immunostaining and confocal microscopy. STAR Protoc. 4 (1), 102032 (2023).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, (2023).
- . Synthego Performance Analysis, ICE Analysis Available from: https://www.synthego.com/help/citing-ice (2019)
- Salamov, A. A., Nishikawa, T., Swindells, M. B. Assessing protein coding region integrity in cDNA sequencing projects. Bioinformatics. 14, 384-390 (1998).
- Pedersen, A. G., Nielsen, H. Neural network prediction of translation initiation sites in eukaryotes: Perspectives for EST and genome analysis. Proc Int Conf Intell Syst Mol Biol. 5, 226-233 (1997).
- Concordet, J. P., Haeussler, M. CRISPOR: Intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46 (W1), W242-W245 (2018).
- Laguette, N., et al. SAMHD1 is the dendritic- and myeloid-cell-specific HIV-1 restriction factor counteracted by Vpx. Nature. 474 (7353), 654-657 (2011).
- Hrecka, K., et al. Vpx relieves inhibition of HIV-1 infection of macrophages mediated by the SAMHD1 protein. Nature. 474 (7353), 658-661 (2011).
- Seki, A., Rutz, S. Optimized RNP transfection for highly efficient CRI SPR/Cas9-mediated gene knockout in primary T cells. J Exp Med. 215 (3), 985-997 (2018).
- Batista Napotnik, T., Polajžer, T., Miklavčič, D. Cell death due to electroporation - A review. Bioelectrochemistry. 141, 107871 (2021).
- Perretta-Tejedor, N., Freke, G., Seda, M., Long, D. A., Jenkins, D. Generating mutant renal cell lines using CRISPR technologies. Methods Mol Biol. 2067, 323-340 (2020).
- Sentmanat, M. F., Peters, S. T., Florian, C. P., Connelly, J. P., Pruett-Miller, S. M. A survey of validation strategies for CRISPR-Cas9 editing. Sci Rep. 8 (1), 888 (2018).
- Makino, S., Fukumura, R., Gondo, Y. Illegitimate translation causes unexpected gene expression from on-target out-of-frame alleles created by CRISPR-Cas9. Sci Rep. 6, 39608 (2016).
- Bowling, A., et al. Downstream alternate start site allows N-terminal nonsense variants to escape NMD and results in functional recovery by readthrough and modulator combination. J Pers Med. 12 (9), 1448 (2022).
- Inácios, &. #. 1. 9. 4. ;., et al. Nonsense mutations in close proximity to the initiation codon fail to trigger full nonsense-mediated mRNA decay. J Biol Chem. 279 (31), 32170-32180 (2004).
- Li, J., et al. Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9. J Mol Cell Biol. 7 (4), 284-298 (2015).
- Guo, Y., et al. CRISPR inversion of CTCF sites alters genome topology and enhancer/promoter function. Cell. 162 (4), 900-910 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon