Method Article
THP-1 세포의 전기천공법 기반 CRISPR-Cas9 매개 유전자 녹아웃 및 단일 세포 클론 분리
요약
THP-1 세포주는 다양한 생물학 관련 연구 분야에서 인간 단핵구/대식세포의 기능을 조사하기 위한 모델로 널리 사용됩니다. 이 논문에서는 효율적인 CRISPR-Cas9 기반 엔지니어링 및 단일 세포 클론 분리를 위한 프로토콜에 대해 설명하여 강력하고 재현 가능한 표현형 데이터를 생성할 수 있습니다.
초록
인간 급성 단핵구 백혈병(AML) THP-1 세포주는 인간 면역결핍 바이러스(HIV)와 같은 중요한 인간 병원체와의 상호 작용을 포함하여 인간 단핵구 유래 대식세포의 기능을 연구하기 위한 모델로 널리 사용됩니다. 골수성 기원의 다른 불멸 세포주와 비교했을 때, THP-1 세포는 많은 온전한 염증 신호 경로를 유지하고 있으며, phorbol-12-myristate 13-acetate (PMA)로 처리했을 때 대식세포로 분화하는 능력을 포함하여 일차 단핵구와 더 유사한 표현형 특성을 나타냅니다. CRISPR-Cas9 기술을 사용하여 표적 유전자 녹아웃(KO)을 통해 THP-1 세포를 엔지니어링하면 바이러스-숙주 상호 작용을 포함한 면역 관련 메커니즘을 더 잘 특성화할 수 있는 강력한 접근 방식을 얻을 수 있습니다. 이 논문은 사전 조립된 Cas9:sgRNA 리보핵단백질을 세포핵으로 전달하기 위해 전기천공법을 사용하는 효율적인 CRISPR-Cas9 기반 엔지니어링을 위한 프로토콜에 대해 설명합니다. 약간 다른 위치에서 동일한 유전자 자리를 표적으로 하는 여러 sgRNA를 사용하면 큰 DNA 단편이 삭제되어 편집 효율성이 증가하며, 이는 T7 엔도뉴클레아제 I 분석법으로 평가됩니다. 유전자 수준에서의 CRISPR-Cas9 매개 편집은 Sanger 염기서열분석에 이어 ICE(Inference of CRISPR Edits) 분석으로 검증되었습니다. 단백질 고갈은 기능 분석법과 결합된 면역 블로팅에 의해 확인되었습니다. 이 프로토콜을 사용하여 표적 유전자 자리에서 최대 100% indels와 단백질 발현을 95% 이상 감소시켰습니다. 높은 편집 효율은 희석을 제한하여 단일 세포 클론을 편리하게 분리할 수 있습니다.
서문
THP-1은 급성 백혈병(AML)을 앓고 있는 환자로부터 분리된 인간 단핵구 유래 세포주로, 일차 단핵구1과 매우 유사한 표현형 특징을 나타냅니다. 표현형에서 제한된 수명과 공여체 간/공여자 간 변동성을 모두 분열하지 않고 나타내지 않는 1차 단핵구 유래 대식세포와 비교했을 때, THP-1 세포는 사실상 영원히 배양될 수 있으며 결과 재현성에 유리한 보다 균일한 행동을 할 수 있습니다 2,3,4,5,6. 특히, THP-1 세포는 phorbol-12-myristate 13-acetate (PMA)를 가진 대식세포와 같은 표현형으로 분화될 수 있으며, 이는 염증 신호 7,8,9,10,11,12,13 또는 HIV 14를 포함한 임상적으로 관련된 인간 병원체에 의한 감염에 대한 단핵구/대식세포의 반응을 조사하기 위해 널리 사용되는 체외 모델입니다,15,16. THP-1 세포를 유전적으로 조작할 수 있는 가능성은 많은 생물학 관련 연구 분야에서 관심을 끌고 있습니다.
CRISPR-Cas9(Clustered Regularly Interspaced Short Palindromic Repeats-CRISPR-Cas9)는 RNA 유도 뉴클레아제를 이용하여 침입하는 바이러스 게놈을 분해하는 원핵생물 적응 면역 체계로, 유전 공학 도구로 재프로그래밍되었습니다17. 게놈 편집 과정은 인식, 분열 및 복구의 세 단계로 진행됩니다. sgRNA(single-guide RNA)는 20-bp 가이드 염기서열과 염기쌍을 통해 Cas9 nuclease를 특정 게놈 자리로 모집합니다. 20-bp 게놈 표적 염기서열의 직접 3'에 Protospacer Adjacent Motif(PAM) 서열의 존재는 위치 17과 18(PAM의 3-bp 5') 사이의 두 DNA 가닥에서 Cas9 매개 풀림 및 절단을 유발합니다. 그 결과 발생하는 이중 가닥 절단(DSB)은 두 가지 주요 복구 경로에 의해 처리됩니다. 손상된 유전자 자리와 상동성을 지닌 복구 템플릿이 없는 경우, 오류가 발생하기 쉬운 NHEJ(Non-Homologous End Joining) 경로는 무작위 뉴클레오티드 삽입 및/또는 결실(indels)을 도입하여 잠재적으로 프레임 시프트 돌연변이 및/또는 조기 종결 코돈(PTC)의 도입으로 이어질 수 있습니다. 차례로, PTC 함유 mRNA는 nonsense-mediated mRNA decay (NMD) 경로에 의한 분해에 의해 표적화되어 궁극적으로 단백질 발현/기능을 방해합니다 18,19,20. 또는 템플릿 종속 HDR(Homology-Directed Repair) 경로가 작동하여 DSB를 충실하게 복구할 수 있습니다. 이 메커니즘은 knock-ins 및 base substitution을 포함한 정확한 유전자 편집을 달성하기 위해 활용되었습니다. 세포주기 상태가 DSB 복구 경로의 선택에 영향을 미치는 중요한 요소라는 점은 주목할 가치가 있습니다. 실제로, NHEJ는 세포주기의 모든 단계에서 활성화되는 반면, HDR은 주로 S/G2 단계21로 제한됩니다.
THP-1 세포는 현탁액에서 성장하며 플라스미드 DNA로 transfection하는 것이 매우 어렵기로 악명이 높으며, 이는 생존력 및/또는 분화 능력을 변경할 수도 있습니다22,23. Cas9 및 sgRNA를 모두 인코딩하는 HIV-1 기반 렌티바이러스 벡터를 사용한 형질도입은 종종 관심 유전자를 knockout(KO)하는 데 사용됩니다24. Cas9/sgRNA cassette를 세포 게놈에 통합하면 발현의 연장과 효율적인 KO가 보장되지만, off-target effect의 지속적인 원인이 되기도 합니다25. 또는 사전 조립된 Cas9:sgRNA 리보핵단백질(RNP)은 전기 충격을 가할 때 플라즈마와 핵막 모두에 기공을 임시로 형성하는 방법인 전기천공법을 통해 전달됩니다. 세포 생존율을 보존하는 것은 이 접근 방식을 수행할 때 중요한 과제입니다.
여기에서는 GFP를 안정적으로 발현하는 THP-1 세포주(THP-1_GFP)를 생산하여 효율적인 CRISPR-Cas9 기반 편집을 달성하기 위한 프로토콜을 확립하는 도구 역할을 수행하였습니다. 3개의 sgRNA를 동시에 사용하여 EGFP 유전자를 비활성화하는 전략을 설계한 후(multi-guide approach), GFP 발현을 판독값으로 사용하여 여러 전기천공 조건 중 KO 효율을 측정했습니다. 세포 증식은 병행하여 모니터링하였다. 유전자 편집은 T7 엔도뉴클레아제 I(T7EI) 분석과 Sanger 염기서열분석으로 확인한 후 ICE(Inference of CRISPR Edits) 알고리즘26을 사용한 분석을 통해 확인했습니다. THP-1 세포가 전기천공 후 정상적인 성장률을 회복하면서 최대 95%의 GFP 발현 감소를 이룬 매개변수는 내인성 유전자(SAMHD1)를 비활성화하고 단일 세포 THP-1 클론을 생산하는 데 성공적으로 사용되었습니다.
프로토콜
이 연구에 사용된 시약 및 장비의 세부 사항은 재료 표에 나열되어 있습니다.
1. CRISPOR를 사용한 가이드 설계(그림 1.1)
참고: SnapGene Viewer 소프트웨어는 4, 7 및 10단계에서 사용하여 편집 대상 부위와 관심 유전자 내 PCR 프라이머 혼성화 위치에 주석을 달 수 있습니다.
- Ensembl 웹 사이트(www.ensembl.org)로 이동합니다. 검색 상자에서 종을 선택하고 관심 있는 유전자의 이름을 입력합니다. 이동을 클릭합니다. 유전자에 해당하는 결과를 선택합니다(전사체 아님).
- Show transcript table을 클릭한 다음 Biotype 열에서 금색 라벨이 붙은 Protein coding에 해당하는 Transcript ID를 선택합니다. 성적 증명서 페이지에서 왼쪽 메뉴에서 Exons를 클릭합니다.
- 아래로 스크롤하여 다운로드 시퀀스를 클릭합니다. 파일 형식이 FASTA인지 확인합니다. Settings - Included Sequences에서 Genomic Sequence를 제외한 모든 항목을 선택 취소합니다.
스크립트 상자의 양쪽 끝에 있는 측면 순서로 숫자 "500"을 입력합니다. Preview를 클릭하고, 전체 염기서열(헤더가 없는 뉴클레오티드만)을 선택하고, 복사(Ctrl+C)합니다. - SnapGene 뷰어를 열고 새 > DNA 파일을 클릭합니다. 시퀀스(Ctrl+V)를 시퀀스 만들기 상자에 붙여넣습니다. Detect common features(공통 피쳐 탐지)를 선택 취소하고 Topology(토폴로지)에서 Linear(선형)를 선택합니다.
파일 이름을 바꾸고 만들기를 클릭합니다. 왼쪽 메뉴에서 Show enzymes(효소 표시 )(첫 번째 아이콘)를 선택 취소합니다. 창 아래쪽에서 시퀀스 탭을 선택합니다.
참고: 이 단계를 통해 엑손, 인트론 및 500bp 플랭크 염기서열(선택 사항)을 포함한 전체 유전자 염기서열을 검색할 수 있습니다. 이 후자의 정보는 첫 번째 엑손 내에 위치한 표적 부위의 증폭을 위한 PCR 프라이머를 설계하는 데 유용합니다. - Ensembl 웹 사이트로 돌아가서 파일 미리보기를 위로 스크롤하고 뒤로를 클릭합니다. 이제 파일 형식을 RTF로 변경합니다. Settings - Included Sequences에서 Exons를 제외한 모든 항목을 선택 취소합니다. 변형 표시에서 아니요를 선택합니다. 페이지 상단의 Download(다운로드 )를 클릭합니다.
- 엑손 서열이 포함되어 있고 초기 ATG부터 시작하여 코딩 서열을 파란색으로 표시하는 다운로드한 파일(Word 사용)을 엽니다. CRISPR-Cas9 지시 편집의 대상이 될 엑손을 선택하고(권장 사항은 아래 참조), 이를 선택한 다음 복사합니다(Ctrl+C).
- 표적 엑손은 초기 엑손(early exon) 또는 단백질의 기능적으로 중요한 도메인을 암호화하는 엑손(exon)입니다. 3' UTR에 가까운 후기 엑손에서 PTC를 확립하면 NMD를 유도하지 못할 가능성이 높으며, 이로 인해 C-말단으로 절단된 단백질이 발현된다는 점에 주목할 가치가 있습니다. 반대로, 네이티브 개시 부위에 근접한 초기 엑손(exon)에 PTC를 도입하는 것은 불법 번역(ITL, 일명 대체 번역 시작(ATI))의 위험과 관련이 있으며, 첫 번째 ATG 코돈의 다운스트림 프레임 내 번역 시작 부위(TIS)에서 시작하는 N-말단 절단 단백질의 예기치 않은 발현을 생성합니다. 후자의 위험을 완화하기 위해 ATGpr27 (atgpr.dbcls.jp) 및/또는 NetStart 1.028 (services.healthtech.dtu.dk/services/NetStart-1.0/)을 사용하여 대체 TIS의 발생을 평가하는 것이 좋습니다.
- 표적 엑손이 코딩 염기서열을 포함하고 있는지 확인합니다. 그러나 초기 ATG의 업스트림에 5'UTR 영역이 있는 sgRNA 어닐링을 선택하여 삭제된 단편에 포함하는 것이 유용할 수 있습니다.
- 이상적으로, 표적 엑손은 유전자의 모든 단백질 코딩 전사체 변이체에 공통적이어야 합니다. NCBI의 Genome Data Viewer(www.ncbi.nlm.nih.gov/gdv/)에서 관심 유전자를 검색하여 해당 사실을 확인합니다.
디스플레이 창에서 유전자 이름(녹색)을 클릭하면 전사체 변이체(자주색)가 표시됩니다. 엑손은 직사각형으로 표시됩니다.
- SnapGene Viewer에서 Ctrl+F, Ctrl+V, Enter 를 차례로 눌러 엑손의 염기서열을 검색합니다. Ctrl+T 를 눌러 새 기능을 추가하고, 이름을 지정하고, 유형을 "exon"으로 변경하고 확인을 클릭합니다.
- CRISPOR 웹사이트(http://crispor.gi.ucsc.edu/)로 이동하여 Step 1의 엑손 염기서열을 붙여넣습니다. 먼저 2단계에서 참조 게놈을 선택한 다음 3단계에서 표적으로 삼을 PAM 유형(일반적으로 SpCas9의 경우 20bp-NGG)을 선택합니다. SUBMIT(제출)을 클릭합니다.
- 최대 150 bp까지 분리되도록 두 개의 sgRNA를 선택한 다음 그 사이에 있는 세 번째 sgRNA를 선택합니다. 다음은 sgRNA 선택에 대한 몇 가지 지침입니다.
- MIT 특이성 점수는 off-target 효과와 관련이 있습니다. 점수가 높을수록 잠재적인 오프 타겟이 적다는 것을 나타냅니다. 오른쪽 열에는 가능성이 가장 높은 것부터 가장 낮은 것까지 순위가 매겨진 3개의 예측된 비표적 부위가 해당 위치(엑손, 인트론 또는 유전자 간 영역)와 함께 표시됩니다. 예측된 오프 타겟 사이트의 전체 목록은 모두 표시를 클릭하여 액세스할 수 있습니다. 가능한 경우 MIT 점수가 >80인 sgRNA를 선택하고 0, 1 또는 2 mismatch에 대해 off-target이 없는 sgRNA를 우선적으로 선택합니다. 또한, 표현형에 영향을 미칠 가능성이 가장 큰 엑손에서 off-target을 가진 sgRNA는 피해야 합니다.
- 예측된 효능은 Doench '16 점수를 참조하십시오. Doench '16 점수가 높다는 것은 단순히 sgRNA가 더 효과적일 가능성이 높다는 것을 나타냅니다. 실제 효능은 실험적으로 결정해야 합니다. 이러한 이유로 여러 sgRNA를 함께 사용할 의도가 없더라도 여러 sgRNA를 선택하는 것이 항상 유용합니다.
- 특정 motif뿐만 아니라 너무 높거나 너무 낮은 GC 함량은 sgRNA 효율성에 해로울 수 있으므로 피해야 합니다. 이러한 매개변수는 CRISPOR에 의해 강조 표시됩니다.
- 1.7단계를 반복하여 sgRNA 염기서열 및 관련 PAM 염기서열을 SnapGene Viewer의 유전자 염기서열에 추가합니다. 동시에 sgRNA 염기서열(PAM 제외)을 텍스트 또는 Excel 파일에 붙여넣어 올리고뉴클레오티드를 주문할 때 필요한 5'-3' 방향을 유지합니다.
- 표적 부위의 PCR 기반 증폭을 위해 이를 둘러싼 몇 개의 프라이머를 설계합니다. 앰플리콘 크기는 800에서 1000bp 사이여야 합니다. PrimerQuest는 이 프로토콜(https://eu.idtdna.com/pages/tools/primerquest)에 대한 프라이머를 설계하는 데 사용되었습니다. 또는 CRISPOR는 표적 유전체 영역과 off-target 부위를 증폭하기 위한 프라이머 목록을 제공합니다. 잠재적인 off-target site의 전체 목록을 표시한 후(단계 1.9.1), 오른쪽 하단 모서리에 있는 Off-target primer 를 클릭합니다.
2. 전기천공을 위한 시약 및 세포 준비(그림 1.2)
- 20% 열 불활성화(56°C, 30분), 여과(0.20μm) 소 태아 혈청(FBS)이 보충된 500μL의 RPMI 1640 배지로 웰을 채워 전기천공 후 세포를 회수할 수 있는 24웰 배양 플레이트를 준비합니다. 항생제를 첨가하지 마십시오. 플레이트를 37°C, 5% CO2 의 가습 인큐베이터에 전기천공까지 24시간 동안 보관하십시오.
- sgRNA가 건조된 상태로 배송되는 경우 TE 완충액(10mM Tris, 1mM EDTA, pH 8.0)으로 최종 농도 100μM(즉 , sgRNA 1nmol당 TE 완충액 10μL)로 재수화합니다. 30초 동안 Vortex를 배양하고 4°C에서 하룻밤 동안 배양하여 완전한 재수화를 허용하고 간단한 피펫 균질화 후 sgRNA 원액을 -20°C에서 보관합니다. 최종 부피에 따라 여러 번의 동결-해동 주기를 피하기 위해 부분 표본을 만듭니다.
- 100 μM 원액을 뉴클레아제가 없는 물에 희석하여 30 μM의 최종 농도에서 작동하는 sgRNA 용액을 준비합니다. 30초 동안 소용돌이치고 실온에서 5분 동안 배양합니다.
- 전기천공법 키트에 포함된 3.5 μL의 재현탁액 R에 3개의 30 μM sgRNA 각각과 0.5 μL의 20 μM Cas9 용액을 동시에 희석하여 Cas9:sgRNA RNP를 1:9 몰비로 조립합니다(하나의 실험 조건에 대한 최종 부피는 7 μL, 그에 따라 확장). 잠시 소용돌이치고 실온에서 5분 동안 배양합니다.
- 그 동안, 6.5μL의 재부유 완충액 R. Vortex에 0.5μL의 20μM Cas9를 간단히 첨가하여 편집되지 않은 대조군을 준비하고 실온에서 5분 동안 배양합니다.
- 전기천공법 조건당 12μL의 최종 부피를 얻기 위해 모든 샘플에 5μL의 재현탁 완충액 R을 추가합니다.
- 전기천공을 위해 THP-1 세포를 준비합니다.
- Trypan blue exclusion test에 의한 농도 및 생존력을 평가합니다. 세포를 0.4% 트리판 블루 염색 용액에 1:2로 희석합니다. 30초 배양 후 잘 균질화하고 Neubauer-improved grid 스타일로 계수 챔버의 벽에 10μL를 추가합니다. 세 개의 큰 사각형을 세고 개수를 100으로 나누어 세포 농도(x10,6 cells/mL)를 구합니다.
참고: 세포의 건강은 전기천공에 대한 민감도에 영향을 미칩니다. 최적의 조건에서 세포 배양을 수행하기 위해 주의를 기울여야 합니다. 인큐베이터 밖에서의 시간은 제한되어야 하며 모든 시약과 용액은 사전에 준비하고 예열해야 합니다. - 각 조건에 대해 0.2 x 106 세포 및 원심분리기(336 x g, 5분, 20°C)에 해당하는 부피를 수집합니다.
- 피펫을 사용하여 상층액을 흡입하고 펠릿을 500μL의 PBS에 재현탁합니다. 다시 원심분리기(336 x g, 5분, 20°C).
- 피펫을 사용하여 상층액을 조심스럽게 흡인하고 THP-1 세포 펠릿을 12μL의 RNP 용액으로 재현탁합니다(단계 2.6).
- Trypan blue exclusion test에 의한 농도 및 생존력을 평가합니다. 세포를 0.4% 트리판 블루 염색 용액에 1:2로 희석합니다. 30초 배양 후 잘 균질화하고 Neubauer-improved grid 스타일로 계수 챔버의 벽에 10μL를 추가합니다. 세 개의 큰 사각형을 세고 개수를 100으로 나누어 세포 농도(x10,6 cells/mL)를 구합니다.
3. Electroporation 시스템 설정 및 핵 절제 (그림 1.3)
- 피펫 스테이션을 생물 안전 캐비닛 아래에 놓고 지지대에 전기천공법 튜브를 넣으며 전기천공법 키트에 포함된 완충액 E 3mL를 추가합니다.
- electroporation 장치를 켠 후 터치 스크린을 사용하여 다음 electroporation 매개변수를 설정합니다. 전압 = 1 500V, 지속 시간 = 10ms, 숫자 = 3.
- 전기천공법 피펫에 팁을 장착하고 RNP/THP-1 용액 10μL를 흡입합니다(단계 2.7.4). 피펫을 electroporation tube에 삽입하고 electroporation device 화면에서 Start 를 누릅니다. 완료 메시지가 나타날 때까지 기다렸다가 튜브에서 피펫을 제거합니다.
- 예열된 24웰 플레이트의 웰로 세포를 옮기고 부드럽게 균질화합니다. 플레이트를 가습 인큐베이터에 다시 넣고 72시간 동안 방해받지 않고 그대로 두십시오.
참고: RNP/THP-1 현탁액을 피펫팅할 때 기포가 생기지 않도록 주의하십시오. 이는 전기천공법 절차를 방해할 수 있으므로 주의하십시오. 전기 아크가 보이지 않을 때 오류 메시지가 나타나면 튜브에서 전기천공법 피펫을 제거하고 시작을 다시 누릅니다. 그러나 짧은 밝은 불꽃 형태의 전기 아크가 관찰되면 기포가 있음을 나타낼 수 있습니다. electroporation 절차는 오류 메시지가 없는 경우에도 실패할 수 있습니다.
4. THP-1 회수 72시간 후 전기천공법(그림 1.4)
- 농도를 평가하기 위해 세포를 세십시오 (단계 2.7.1) electroporation 후 72 시간.
- 세포가 충분하면(즉, ≥0.6 x 106 cells/mL) 20% FBS와 1% 페니실린-스트렙토마이신이 보충된 RPMI로 factor2 이상으로 희석하고 농도를 mL당 0.3-0.5 x 106 cells 사이로 가져옵니다. 그렇지 않으면 세포가 72시간 더 회복되도록 합니다.
- KO 검증에 충분할 때까지 세포를 계대 계대 및 증폭합니다. 그 동안, 단일 세포 클론의 분리가 시작될 수 있습니다(7단계).
5. T7EI mismatch assay에 의한 유전자 편집 검증(그림 1.5)
참고: 이 분석은 T7EI가 1bp보다 큰 불일치를 인식한다는 점을 감안할 때 편집 효율성을 과소평가할 수 있습니다. 따라서 T7EI 분석은 적절하게 변형되지 않는 한 동형접합 세포 집단(즉, 단일 세포 클론)을 스크리닝하는 데 유용하지 않습니다(단계 5.7).
- 세포 농도를 평가하고(단계 2.7.1) 1.5mL 튜브에서 0.1 x 106 세포에 해당하는 부피를 회수합니다. 원심분리기(336 x g, 5분, 20°C)에서 상층액을 흡인하고 펠릿을 500μL의 PBS에 재현탁합니다. 다시 원심분리기를 하고 피펫을 사용하여 펠릿을 방해하지 않고 가능한 한 많은 상층액을 흡입합니다. 샘플을 스냅 동결하고 -20 °C에서 보관하십시오.
- PCR 증폭을 위한 매트릭스 역할을 할 게놈 DNA를 추출합니다.
- 50 μL의 DNA 추출 용액으로 펠릿을 재현탁하고, 균질화하고, 0.2 mL PCR 튜브에서 전체 부피를 옮깁니다. 와류와 원심분리기를 짧게 누릅니다(3초 동안 펄스).
- 튜브를 열 순환기에 넣고 65°C에서 15분 동안 가열한 다음 98°C에서 10분 동안 가열합니다.
- 추출된 DNA를 초순수 90μL로 희석합니다. 와류와 원심분리기를 짧게 (5,000 x g, 3 초).
- PCR 프라이머( 재료 표 참조) 를 초순수에 희석하여 최종 농도를 10μM(즉, 10pmol/μL)로 만듭니다.
- 0.2 mL PCR 튜브(최종 부피 = 50 μL, 한 가지 조건의 경우)에서 PCR 혼합물(아래 참고 참조)을 준비합니다. 일반적으로 적어도 두 가지 조건이 있습니다 : KO와 Cas9 만 사용하는 편집되지 않은 컨트롤.
참고 : 정제 된 게놈 DNA : 10 μL; 정방향 및 역방향 프라이머 (10 μM) : 각각 2.5 μL, 최종 농도 500 nM; 반응 완충액 (5x): 10 μL. 추가하기 전에 잘 와류. dNTP 혼합(각 25mM): 0.6μL, 각 dNTP에 대한 최종 농도 0.3mM. DNA 중합효소: 0.5 μL; 초순수: 23.9 μL. 와류 및 원심분리기를 짧게 (3초 동안 펄스). - 튜브를 열 순환기에 넣고 다음 설정으로 PCR 프로그램을 실행합니다.
참고: 95°C에서 5분 동안 1회 사이클, 30사이클[98°C에서 20초(변성 단계), 15초(어닐링 단계) 동안 X °C, 45초(연신율 단계)] 동안 72°C, 2분 동안 72°C에서 마지막 사이클. 증폭이 끝나면 튜브, 와류 및 원심 분리기를 잠시 제거합니다(3초 동안 펄스). 어닐링 온도(X)는 프라이머의 용융 온도(Tm)에서 5°C를 뺀 값입니다. - 다클론 편집 모집단의 경우: 새로운 0.2 mL 튜브에 17.5 μL의 PCR 앰플리콘과 2 μL의 NEBuffer 2(10x)를 추가하여 최종 부피 19.5 μL를 만듭니다. 와류 및 원심분리기를 간략하게 수행합니다(3초 동안 펄스). 대안적으로, 단일 세포 클론을 스크리닝하려면 편집된 대조 세포와 편집되지 않은 대조 세포 모두에서 PCR 산물을 1:1로 혼합합니다(단계 5.6)(보충 그림 1).
- 이중이중 형성을 위해 튜브를 열 순환기에 넣고 다음 프로그램을 실행합니다: 95°C에서 10분 동안 한 사이클, 95 - 85 °C에서 -2 °C/s의 램프, 85 - 25 °C에서 -0.3 °C/s의 램프, 10 °C에서 최종 냉각 사이클 1회.
- 이중이중 형성 후 튜브에 0.5μL의 T7EI 용액을 첨가합니다. 37 °C에서 30 분 동안 배양합니다.
- 1.2% 아가로스 젤을 준비합니다.
- 유리병에 아가로스의 무게를 측정하고 적절한 부피의 1x TAE 완충액(40mM 트리스-아세테이트, 1mM EDTA, pH 8.3)을 추가합니다. 캡을 단단히 조이지 않도록 주의하면서 전자레인지에 넣으십시오. 아가로스 결정이 완전히 용해될 때까지 여러 번 가열합니다.
알림: 필요한 경우 용액을 혼합합니다. 끓이지 마십시오. 그렇지 않으면 부피가 감소하여 아가로스 농도가 변합니다. - 1/20,000으로 희석한 DNA겔 염색을 넣고 잘 섞는다.
- 아가로스 용액을 틀에 붓고 빗을 넣고 실온에서 응고시킵니다.
- 유리병에 아가로스의 무게를 측정하고 적절한 부피의 1x TAE 완충액(40mM 트리스-아세테이트, 1mM EDTA, pH 8.3)을 추가합니다. 캡을 단단히 조이지 않도록 주의하면서 전자레인지에 넣으십시오. 아가로스 결정이 완전히 용해될 때까지 여러 번 가열합니다.
- 5 μL의 T7EI 분해 산물(5.9단계) 또는 분해되지 않은 PCR 산물을 5 μL의 물 및 2 μL의 6x DNA 로딩 염료와 혼합하여 겔 전기영동을 위한 샘플을 준비합니다. 샘플과 크기 ladder를 로드하고 80V에서 45분 동안 이동합니다(시간은 DNA 단편의 예상 크기에 따라 조정될 수 있음). 마이그레이션이 끝나면 적절한 이미징 시스템을 사용하여 겔의 이미지를 획득합니다.
6. Sanger 염기서열분석 분석을 통한 유전자 편집 검증 (그림 1.6)
- 유전자 변형을 특성화하려면 PCR 산물을 정제하고(5.4단계) Sanger 염기서열분석 후 ICE 도구(https://www.synthego.com/products/bioinformatics/crispr-analysis)로 결과를 분석합니다.
- 변형에도 불구하고 단백질이 생성될 수 있는지 여부를 제어합니다.
- ICE 결과를 다운로드하고 contribs.txt 파일을 엽니다.
- 편집된 염기서열을 WT 염기서열과 비교합니다. indels를 매핑하고 표적 게놈 영역에서 발생했는지 확인합니다. 그들의 크기를 평가하십시오. indel 길이가 3의 배수가 아닌 경우 프레임시프트가 발생하고 PTC가 도입될 수 있습니다. 돌연변이된 mRNA는 NMD 매개 분해를 겪을 것으로 예상됩니다.
7. 희석 제한에 의한 단일 세포 클론 분리(그림 1.7)
참고: 단일 세포 클론의 분리는 필수가 아닙니다. 그러나 그렇게 하기로 선택한 경우 여러 클론을 특성화하고 해당 표현형을 원래 다클론 집단과 비교하는 것이 중요합니다.
- 세포 현탁액 샘플을 채취하여 배양 배지로 1/2 희석하고 농도를 평가합니다(단계 2.7.1). 희석은 계수 정확도를 높입니다.
- 2-3개의 연속 희석 단계를 수행하여 7 cells/mL의 농도에 도달합니다. 둥근 바닥 96웰 플레이트에 웰당 100μL의 세포 현탁액을 분주합니다(즉, 웰당 0.7개 세포). 하나의 세포가 들어 있는 웰을 식별하기 위해 현미경에서 플레이트를 관찰하기 전에 몇 시간 동안 세포를 디캔팅하도록 합니다.
- 콜로니 형성 및 성장을 정기적으로 모니터링하고 필요한 경우 세포를 더 큰 배양 플레이트 또는 플라스크로 옮깁니다.
8. HIV-1 제한 분석에 의한 THP-1 KO SAMHD1 세포의 기능적 특성 분석
- 10% FBS, 1% 페니실린-스트렙토마이신(완전한 RPMI)이 보충되고 분화를 위해 300ng/mL PMA를 함유한 300μL의 RPMI에 웰당 0.25 x 106 개의 세포가 있는 24웰 배양 플레이트에 THP-1을 파종합니다. 플레이트를 가습 인큐베이터(37°C, 5% CO2)에 24시간 동안 보관하십시오.
- 배지를 PMA가 없는 완전한 RPMI 배지로 교체하고 플레이트를 인큐베이터에 다시 24시간 동안 넣습니다.
- 진공 펌프를 사용하여 모든 배양 배지를 흡인합니다. 250μL의 VSVg-pseudotyped HIV-1-GFP 바이러스 스톡 함유 용액(MOI = 0.5 IFU/cell)을 추가합니다. 감염되지 않은 컨트롤을 포함합니다. 플레이트를 4°C에서 2시간 동안 놓아 감염을 동기화합니다.
- RPMI로 세포를 한 번 씻습니다. 각 웰에 500μL의 완전한 RPMI를 추가하고 표준 조건에서 48시간 동안 배양합니다(시간 조정 가능).
- 배양액을 제거한 후 차가운 1x PBS로 세포를 한 번 세척합니다. 그런 다음 각 웰에 100μL의 0.05% 트립신-EDTA를 추가합니다. 세포가 완전히 분리될 때까지(5분이면 충분함) 플레이트를 37°C의 인큐베이터에 넣은 후 200μL의 완전한 RPMI를 추가하여 효소를 비활성화합니다. 각 웰에서 250 μL를 96웰 둥근 바닥 플레이트로 이동합니다(유세포 분석기가 배양 플레이트를 수용하는지 확인).
- 플레이트를 원심분리하고(757 x g, 3분, 감속 = 6, 20°C) 멀티채널 피펫을 사용하여 상층액을 흡입합니다. 펠릿을 100μL의 4% PFA로 재현탁하고 4°C에서 10분 동안 배양합니다. 100μL의 차가운 1X PBS를 추가합니다.
- 유세포 분석법(flow cytometry)을 통해 GFP 양성 세포로 대표되는 감염률을 분석합니다(제안된 게이팅 전략은 보충 그림 2 참조).
결과
GFP 리포터 단백질(THP-1_GFP)을 안정적으로 발현하는 THP-1 세포주를 생성하고(그림 2A), 효율적인 CRISPR-Cas9 매개 유전자 에디션을 위한 프로토콜을 확립하기 위한 도구로 사용되었습니다. 이를 위해 EGFP 유전자를 표적으로 하는 3개의 sgRNA를 CRISPOR 웹 도구29(그림 2B)로 설계했으며, 이는 9:1의 몰비로 Cas9와 동시에 복합체를 형성하여 RNP를 형성한 후 다른 설정을 사용하여 전기천공법을 통해 세포로 전달되었습니다. 다음으로, 세포를 20% FBS 함유 RPMI 배지 단독(그림 2C, 상단 패널)에서 성장시키거나 컨디셔닝 배지(그림 2C, 하단 패널)로 1:1로 희석했습니다. EGFP KO 효율의 판독값으로서의 세포 증식 및 GFP 발현은 시간이 지남에 따라 모니터링되었습니다. 여러 조건의 경우, GFP 양성 세포의 비율이 급격히 감소하여 전기천공법(PE) 후 7일째에 >90% 감소에 도달했습니다. 3일째 pe에서는 세포 수가 절반으로 줄었는데, 이는 전기천공법이 세포 생존율에 미치는 부정적인 영향 때문일 수 있습니다. 그러나 세포 수는 다시 증가했고, 7일째에는 배가시간이 정상 비율로 돌아왔습니다(그림 2C, 상단 패널). 컨디셔닝된 배지의 사용은 실험 조건에서 세포 회수에 유리하지 않았습니다(그림 2C, 하단 패널). 이러한 결과를 바탕으로 후속 실험을 위해 1500V의 10ms 펄스 3개를 선택했습니다.
그런 다음 CRISPR-Cas9 매개 EGFP KO를 게놈 수준에서 특성화했습니다. 이를 위해 THP-1_GFP 세포의 게놈 DNA를 추출하고 Cas9:sgRNA RNP(편집) 또는 Cas9 단독(편집되지 않음)으로 전기천공한 후 표적 부위를 포함하는 891bp 영역을 PCR 증폭하기 위한 템플릿으로 사용했습니다. EGFP 가 결핍된 부모 THP-1 세포도 음성 대조군으로 포함되었습니다. 다음으로, 편집된 THP-1_GFP 세포의 PCR 산물을 T7EI 분석법에 의한 불일치 검출을 위해 준비한 후, 아가로즈 겔 전기영동에 의한 DNA 단편의 분리를 진행하였다. WT 앰플리콘의 예상 크기에 해당하는 ~900 bp 밴드는 편집되지 않은 THP-1_GFP 세포에 대해서는 쉽게 시각화되었지만 부모 THP-1 대조군에 대해서는 시각화되지 않았습니다(그림 2D, 라인 2 및 3 비교). 편집된 THP-1_GFP 세포의 경우, ~900 bp 띠는 검출할 수 없게 되었고, 그 크기는 CRISPR 편집으로 인한 ~75 bp 단편의 손실과 일치하는 더 빠른 이동 띠로 대체되었습니다(그림 2D, 줄 4). 이 밴딩 패턴은 T7EI 분해 시 변경되어 더 작은 DNA 단편이 나타나게 하여 타겟 부위의 성공적인 편집을 더욱 확인했습니다(그림 2D, 라인 5). 편집 효율성과 편집된 모집단의 특정 유전자형에 대한 통찰력을 얻기 위해 Sanger 방법을 사용하여 PCR 앰플리콘을 염기서열분석한 다음 ICE 도구26을 사용하여 분석했습니다. 편집되지 않은 대조군 샘플은 병렬로 처리되었습니다. indel 및 KO 점수는 100%로 검색되었으며, 이는 편집된 샘플에서 non-WT 염기서열의 비율과 기능적 KO로 이어질 가능성이 있는 indel의 비율에 각각 해당합니다(그림 2E, 왼쪽). indel 크기 스펙트럼 및 빈도에 대한 상세한 분석은 62-bp 또는 76-bp 결실을 지니고 있으며 각각 전체 염기서열의 57% 또는 37%를 차지하는 두 개의 우성 집단의 존재를 보여주었습니다(그림 2E, 오른쪽). 이러한 결과는 sgRNA #1이 sgRNA #2 또는 #3과 함께 표적 유전자에서 동시 DSB를 유발했음을 시사합니다(그림 2B).
그런 다음 이 프로토콜을 적용하여 내인성 SAMHD1 코딩 유전자를 비활성화했습니다(그림 3A). SAMHD1 KO THP-1 다클론 세포주의 특성화에는 시간 경과에 따른 세포 증식 모니터링이 포함되었으며, 이는 편집되지 않은 THP-1 세포와 일치했습니다(그림 3B). 유전자 편집의 결과는 표적 염기서열의 PCR 기반 증폭으로 평가되었으며, 이는 Cas9 단독 대조군에 비해 SAMHD1 KO 세포의 띠가 더 작은 것으로 나타났으며(그림 3C, 2행과 4행 비교), 이는 DNA 염기서열 손실을 나타냅니다. 이러한 관찰은 T7EI 분석에 의해 확인되었으며, SAMHD1 KO에 대한 분할 산물의 출현을 보여주지만 편집되지 않은 세포는 아닙니다(그림 3C, 라인 3 및 5 비교). ICE 도구를 사용한 Sanger 염기서열분석 데이터를 분석한 결과, indel 및 KO 점수가 각각 100%와 97%로 반환되었습니다. SAMHD1 유전자 자리 내의 주요 변형은 각각 염기서열의 46%, 13% 및 10%에서 170bp, 93bp 또는 104bp 염기서열 결실이었습니다(그림 3D). 조세포 용해물에서의 단백질 발현 수준도 면역블로팅(immunoblotting)에 의해 평가되었다. 내인성 SAMHD1의 크기와 일치하는 ~70kDa 대역은 편집 후 거의 검출할 수 없게 되며, 이는 >97%의 추정 감소에 해당합니다(그림 3E). 마지막으로, HIV-1 감염에 대한 감수성을 테스트하여 SAMHD1 KO THP-1 다클론 세포주의 표현형 특성화도 평가했습니다. 잘 알려진 항바이러스 역할인30,31과 일치하여, SAMHD1 유전자 자리의 불활성화는 HIV-1 감염률 증가를 동반했습니다(그림 3F).
이와 동시에, 희석을 제한하여 단일 세포 클론을 생성하였다. SAMHD1 KO 클론에서 게놈 표적 부위를 증폭하면 편집되지 않은 대조 세포에 비해 전기영동 이동성이 증가된 PCR 산물을 얻을 수 있었습니다(그림 4A). Sanger 염기서열분석 데이터의 ICE 기반 분석은 선택된 모든 단일 세포 클론에 대해 100%의 indel score를 반환했지만 하나(클론 A2)는 하나였습니다(그림 4B, 왼쪽). 게놈 수준에서 편집 결과의 특성화는 초기 ATG의 28pb 다운스트림에 위치한 98-bp 결실과 함께 클론 A3에 대한 단일 염기서열을 반환하여 프레임 외 변경으로 이어졌습니다(그림 4B, 오른쪽). 다른 클론은 3개(클론 B1) 이상의 염기서열을 함유하고 있으며, 이는 제한 희석 단계에서 서로 다른 편집 이벤트 및/또는 웰당 하나 이상의 세포를 거친 대립유전자의 존재를 나타냅니다(그림 4B, 오른쪽). 특히, 클론 A4 및 A7의 염기서열 중 11%는 초기 ATG의 상류에 인델을 함유하고 있어 코딩 염기서열이 변경되지 않은 상태로 남아 있습니다. 더욱이, 클론 A7 염기서열의 1%는 초기 ATG의 9-bp 다운스트림에 123-bp 단편이 부족하여 41개의 아미노산이 프레임 내에서 결실되었습니다. 이러한 결과와 일관되게 기능적 연구는 SAMHD1 자리(A3, A8, B1, B3)가 파괴되어 SAMHD1 발현이 결여된 클론이 HIV-1 감염에 매우 관대하다는 것을 밝혔습니다(그림 4C). 반대로, SAMHD1 이 손상되지 않은 클론 A4 및 A7은 부모 또는 편집되지 않은 대조군 THP-1 클론과 마찬가지로 불응성이었습니다(그림 4C). 결론적으로, 클론 A3에서 SAMHD1 유전자 자리의 성공적인 편집이 확인되었습니다.
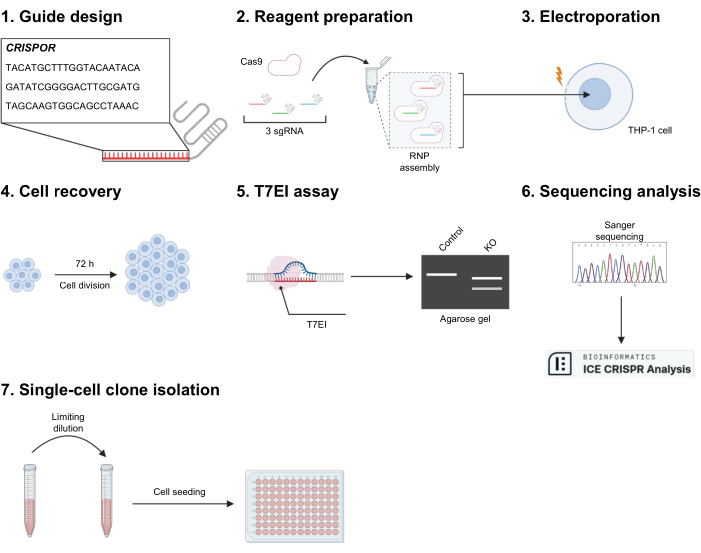
그림 1: 실험 개요. (1) CRPOR는 표적 엑손에 대한 sgRNA 염기서열을 생성하는 데 사용됩니다. 그 중 3개는 높은 on-target 효능과 약한 off-target 예측에 따라 선택됩니다. (2) 3개의 gRNA를 Cas9와 혼합하여 리보핵단백질(RNP)을 조립하여 3개의 서로 다른 RNP를 혼합합니다. (3) RNP가 들어갈 수 있도록 세포를 전기천공합니다. (4) 세포를 24웰 배양판으로 옮기고 최소 72시간 동안 휴지시킵니다. (5) 유전자 편집은 먼저 T7EI 분해 분석에 의해 정성적으로 테스트됩니다. 및 (6) Sanger 염기서열분석 및 ICE 분석으로 더욱 특징지어지는 검증된 조건의 경우. (7) 필요한 경우, 클론 집단을 생산하기 위해 희석을 제한 후 세포를 파종할 수 있습니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
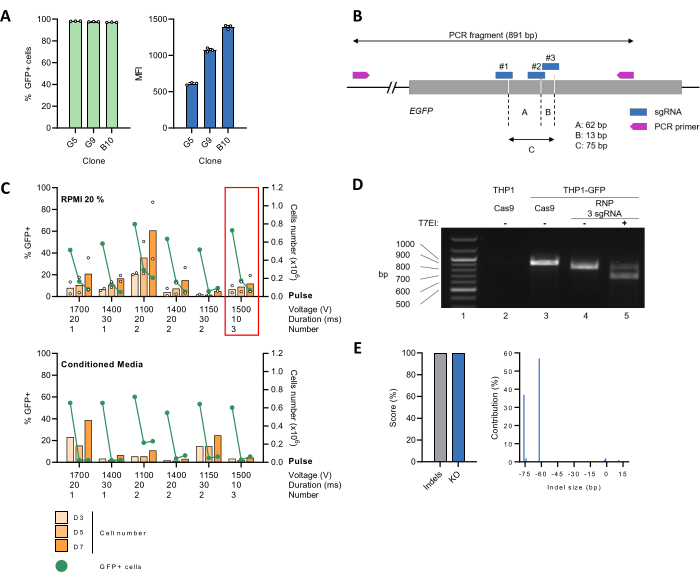
그림 2: THP-1 세포의 EGFP KO에 대한 전기천공(electroporation) 설정 및 유전자 편집 검증. (A) THP-1 세포는 EGFP 리포터 유전자를 암호화하는 렌티바이러스 벡터로 형질주입시켰다. 단세포 클론은 희석을 제한한 후 30일 동안 배양에서 팽창시켜 얻었습니다. 유세포 분석에 의한 분석은 거의 모든 세포 집단이 GFP를 발현하는 것을 확인했습니다. 통합 EGFP 복제 수를 나타내는 GFP 양성 집단의 중앙값 형광 강도(MFI)는 여러 세포 클론 간에 차이가 있었습니다. 후속 연구를 위해 클론 G5가 선택되었습니다. (B) sgRNA와 프라이머의 결합 부위를 포함한 EGFP 유전자의 개략적 표현. Cas9 절단 부위는 파선으로 표시됩니다. (C) 미분화 THP-1_GFP(2 x 105) 세포를 표시된 설정으로 전기천공법 전에 사전 조립된 Cas9:sgRNA RNP와 혼합했습니다. 다음으로, 500μL의 예가열된 20% FBS-함유 RPMI 배지를 단독으로 또는 48-h THP-1 세포 배양에 의해 생성된 컨디셔닝된 배지로 1:1로 희석한 24-웰 플레이트에서 세포를 옮겼습니다. 증식(주황색 막대) 및 GFP 양성 세포(녹색 선)의 비율은 시간이 지남에 따라 모니터링되었습니다. 데이터는 n = 2개의 생물학적 반복실험에 대한 평균을 나타냅니다. 빨간색 프레임은 후속 실험을 위해 선택한 조건을 강조 표시합니다. (D) 편집된(RNP 3 sgRNA) 또는 편집되지 않은(Cas9) THP-1_GFP 세포의 게놈 DNA를 추출하고, 타겟 유전자 좌위 주변 영역을 PCR로 증폭시켰다. 부모 THP-1 세포를 음성 대조군으로 사용했습니다. 앰플리콘은 변성/재생(denaturation/renaturation cycle)을 거쳐 이질이중(heteroduplex)의 형성으로 이어졌습니다. 다음으로, T7EI 분석에 의해 불일치가 검출되었습니다. DNA 단편은 1.2% 아가로스 겔 전기영동으로 분리하였다. (E) 정제된 PCR 산물을 Sanger 방법으로 염기서열분석한 후 ICE 알고리즘을 사용하여 분석했습니다. Indel 및 KO 점수는 샘플에서 non-WT 염기서열의 비율과 각각 frameshift 또는 21+ bp indel이 있는 세포의 비율을 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
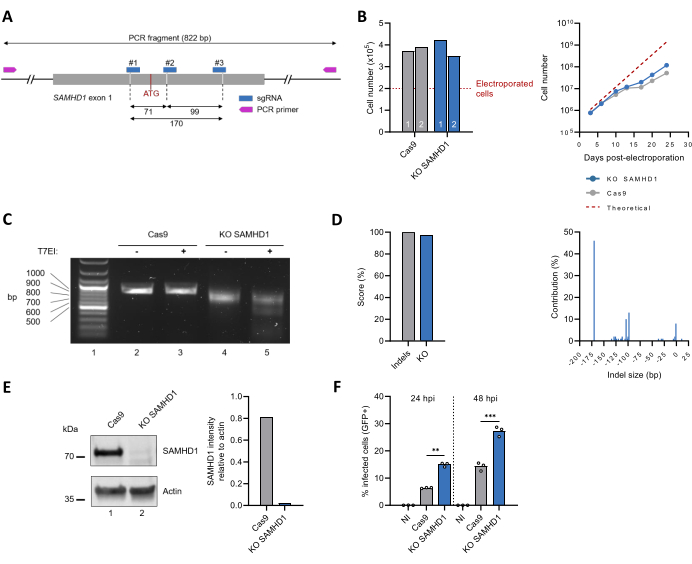
그림 3: 다클론 SAMHD1 KO THP-1 세포 집단의 생산 및 특성화. (A) sgRNA 및 프라이머의 결합 부위를 포함한 SAMHD1 엑손 1의 개략적 표현. Cas9 절단 부위는 점선으로 표시되며 초기 ATG 코돈은 빨간색으로 표시됩니다. (B) 전기천공법 3일 후, 생존 세포(n = 2)를 Trypan blue exclusion test(왼쪽 다이어그램)를 사용하여 정량화했습니다. 세포 성장은 25일 동안 모니터링되었습니다(오른쪽 다이어그램). 점선은 49시간의 예상 배가 시간을 기준으로 한 이론적 세포 수에 해당합니다. (C) 그림 2D와 같이 게놈 수준에서의 편집 결과를 평가했습니다. (D) CRISPR 편집에 따른 유전자형은 Sanger 염기서열의 ICE 기반 분석에 의해 식별되고 정량화되었습니다. (E) 편집된 THP-1 세포와 편집되지 않은 THP-1 세포의 조잡한 용해물에 포함된 단백질을 SDS-PAGE 겔에서 이동에 의해 분리하고, 다음으로 SAMHD1에 대한 항체를 사용하여 면역블로팅하여 시각화했습니다. Actin은 로딩 컨트롤로 사용되었습니다. SAMHD1 및 Actin 밴드의 강도는 ImageJ 소프트웨어를 사용한 밀도 측정으로 정량화되었습니다. (F) Unedited (Cas9) 및 KO SAMHD1 THP-1 세포를 PMA (300 ng/mL, 24 h)로 처리하여 분화한 후 GFP를 발현하는 VSVg-pseudotyped HIV-1 바이러스를 리포터로 투여하였다. GFP 양성 세포의 비율은 감염 후 24시간 및 48시간(hpi)에서 유세포 분석으로 점수화되었습니다. NI: 감염되지 않았습니다. **P < 0.01; P < 0.001은 Welch의 보정을 사용한 쌍체가 없는 t-검정 에 의한 것입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.

그림 4: SAMHD1 KO 단세포 클론의 생산 및 특성화. (A) 선택된 SAMHD1 KO 단세포 클론에 대한 편집 결과는 아가로스 겔 전기영동에 의한 표적 부위를 포함하는 영역의 PCR 증폭에 의해 생성된 DNA 단편의 이동성을 모니터링하여 평가되었습니다. (B) Sanger 염기서열분석 데이터는 그림 2E와 같이 ICE 도구로 분석되었습니다. 오른쪽 그래프의 막대는 각 세포 집단에서 indel의 다양성을 나타냅니다. 녹색 막대는 우세 염기서열과 관련 삭제의 총 크기를 나타냅니다. 괄호 안의 숫자는 비연속적인 간격의 크기를 나타냅니다. (C) HIV-1 감염에 대한 편집되지 않은 클론 및 SAMHD1 KO 단일 세포 클론의 허용성을 그림 3F와 같이 테스트하고, 전기천공되지 않은 부모 THP-1 세포 또는 편집되지 않은 세포 클론(Cas9)의 허용성을 비교했습니다. 감염률은 48hpi로 기록되었습니다. *P < 0.05; **P < 0.01; P < 0.001; P < 0.0001; ns P≥ 0.05 by Brown-Forsythe and Welch ANOVA with Dunnett의 다중 비교 검정. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
보충 그림 1: 적응된 T7EI 테스트에 의한 KO 단일 세포 클론의 편집 결과 평가. KO 단세포 클론 또는 편집되지 않은 세포의 게놈 DNA를 정제하고, 타겟 유전자 좌위를 둘러싼 영역을 PCR에 의해 증폭시켰다. 각 단세포 클론의 앰플리콘은 편집되지 않은 대조 세포의 앰플리콘과 1:1 비율로 혼합되었습니다. 헤테로듀플렉스 형성 및 T7EI 분해 후, 생성물은 아가로스 겔 전기영동에 의해 분석되었습니다. 편집된 클론은 여러 개의 분해 단편(클론 A, B, C, D 및 E)을 생성하는 반면, 편집되지 않은 클론은 단일 WT 밴드(클론 F 및 G)를 나타냅니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
보충 그림 2: HIV-1에 감염된 PMA로 처리된 THP-1 세포의 비율을 정량화하기 위한 유세포 분석 게이팅 전략. 측면 산란 영역(SSC-A)에 대한 전방 산란 영역(FSC-A)을 사용하여 파편을 제거하고 PMA 처리된 THP-1 세포를 선택하기 위해 첫 번째 게이트를 정의합니다. 다음으로, SSC-H 플롯에 대해 SSC-A를 사용하여 단일 셀을 선택합니다. 마지막으로, GFP 양성 세포는 FSC-H에 대해 GFP-A로 검출됩니다. 이 파일을 다운로드하려면 여기를 클릭하십시오.
토론
여기에서는 THP-1 세포주의 성공적인 CRISPR 매개 편집을 얻기 위한 프로토콜이 설명됩니다. 이 접근 방식은 전기천공법/핵 절편에 의한 사전 조립된 sgRNA/Cas9 RNP의 전달에 의존합니다. 이 전략은 sgRNA/Cas9 카세트의 렌티바이러스 매개 통합으로 인해 잠재적으로 발생하는 off-target 효과를 제한하여 nuclease의 지속적인 발현을 생성하기 위해 선택되었습니다. 신뢰할 수 있고 효율적인 편집을 달성하기 위해 관심 유전자를 표적으로 하는 여러 sgRNA를 선택했으며, 이는 게놈 인델 생성 가능성을 증가시켜 단백질 발현 및 기능적 KO32의 손실로 이어집니다. 세포막의 최적 투과화를 달성하기 위해 전기천공법 설정에 특별한 주의를 기울였으며, 이는 과도한 세포 사멸을 방지하면서 sgRNA/Cas9 RNP의 효율적인 전달을 보장하는 데 필요했습니다33. 이와 관련하여, 중요한 매개변수는 전기천공법 이전의 세포 배양의 활력(즉, 낮은 계대 수)입니다.
CRISPR-Cas9 매개 편집은 T7EI 불일치 검출 분석을 통해 유전자 수준에서 검증되었으며, 이는 Sanger 염기서열분석 전에 사전 선택을 위해 여러 샘플에 대한 비용 효율적이고 신속한 평가를 제공합니다. 이 방법은 처음에 전장 앰플리콘과 절단 산물의 강도를 비교하여 단일 sgRNA를 사용하여 편집 효율을 추정하기 위해 개발되었습니다34,35. 주목할 만한 것은, 여러 sgRNA를 사용할 때 생성되는 큰 indels(>70 bp)는 표준 겔 전기영동에 의해 편집된 세포와 편집되지 않은 세포에서 표적 유전자 자리의 PCR 증폭에 의해 얻어진 DNA 단편의 이동성을 비교함으로써 검출될 수 있다는 것입니다. 그럼에도 불구하고 T7EI 분석은 다클론 또는 적응된 프로토콜을 사용하여 단클론 세포 집단에서 작은 indels(>1bp)를 가진 염기서열의 존재를 확인하는 데 유용할 수 있습니다.
코딩 서열을 방해하는 온-타겟 아웃 오브 프레임 돌연변이의 존재는 전체 세포 집단의 게놈을 추출하여 PCR 증폭을 위한 매트릭스로 사용하는 간단한 접근 방식에 의해 확인되었습니다. 다음으로, 정제된 앰플리콘은 대량으로 Sanger 염기서열분석을 거친 후 ICE 도구를 사용한 생물정보학 분석을 수행합니다. 전반적으로 이 방법은 PCR 산물을 플라스미드로 subcloning하고 결찰 산물을 박테리아로 변환하여 개별적으로 염기서열분석되는 단세포 콜로니를 분리해야 할 필요성을 극복합니다.
표적 유전자 자리의 불활성화가 검증되면, 주어진 표현형이 관심 단백질의 결핍에 기인한다고 확신할 수 있게 하고, off-target에서 비롯된 예상치 못한 영향뿐만 아니라 mRNA 구조 및/또는 기능에 영향을 미칠 수 있는 on-target 변화로 인한 예상치 못한 영향을 배제하기 위해 편집된 세포주에 대한 철저한 특성화가 필요합니다.
단백질 발현의 부재는 서로 다른 항원결정기를 인식하는 두 개의 항체를 사용하여 면역 블로팅 및/또는 면역 형광을 통해 확인해야 합니다. 실제로, 표준 ATG 코돈에 가까운 초기 엑손에서 PTC의 도입은 N-말단으로 절단된 단백질 변이체를 생성하는 ITL 이벤트의 위험과 관련이 있습니다36. 주목할 점은, Tuladhar 등은 상용 또는 사내 기원의 CRISPR 편집 세포의 상당한 비율(~50%)에서 비정상적인 단백질 종의 검출을 보고했습니다18,19. 새로운 단백질 산물의 발생은 indel-함유 엑손의 건너뛰기 또는 내부 리보솜 진입 부위(IRES)의 출현과 같은 다양하고 잠재적으로 세포 유형 특이적 과정에 기인하며, 이는 표준 ATG 코돈의 하류에서 대체 TIS에서 단백질 합성을 시작합니다. 또한 ITL이 특정 상황에서 NMD로부터의 탈출과 연관되어 있다는 암시도 있다37,38. 이와 유사하게, 후기 엑손(late exon)을 표적으로 하는 게놈 편집 전략은 유전자 발현의 비효율적인 절제의 기저에 있는 돌연변이된 mRNA의 NMD를 유발하지 못할 수 있습니다20. 마지막으로, Cas9가 여러 sgRNA에 의해 유도되는 경우, 반전 또는 큰 DNA 단편 결실을 포함한 복잡한 염색체 재배열이 발생할 수 있습니다39, 이는 특히 조절 요소가 표적이 될 때 게놈의 구조와 발현을 변경할 수 있습니다40.
의도하지 않은 게놈 편집 결과의 영향을 더욱 완화하기 위해서는 단백질 발현 손실과 그에 따른 표현형(즉, 항바이러스 활성) 사이의 관계를 확인하기 위한 기능 연구가 필요합니다. 편집된 세포가 subclone된 경우, 여러 클론 간의 거동과 다클론 집단의 거동을 비교해야 합니다. 편집된 세포의 표현형을 WT 표현형으로 되돌리기 위해 유전자 발현을 복원하는 것으로 구성된 구조 실험을 수행하는 것도 특이성을 강화하고 가능한 off-target 이벤트를 배제할 것입니다.
이 프로토콜은 THP-1 세포의 편집을 위해 고안되었지만, 다른 세포주에 적용할 수 있는 CRISPR-Cas9 KO 워크플로우를 위한 일반적인 구조를 제공합니다. electroporation 매개 변수를 조정해야 할 수 있으며 1800V보다 높은 전압이 적용되는 경우 제조업체 지침에 따라 resuspension buffer T를 사용해야합니다. 고려해야 할 또 다른 변수는 RNP 조립 중 Cas9과 sgRNA 사이의 농도 비율입니다. 이 경우 1:9 비율이 잘 작동했지만 이를 수정하면 다른 상황에서는 편집 효율성이 향상될 수 있습니다. 마지막으로, 여기서는 유익하지 않지만, electroporation 후 세포 회수를 위한 컨디셔닝 배지의 사용은 세포주에 따라 효과가 다를 수 있으므로 테스트해야 합니다.
공개
모든 저자는 이해 상충이 없습니다.
감사의 말
프로토콜을 공유하고 토론해 주신 JP Concordet(MNHN, U1154/UMR7196, Paris), G. Bossis(IGMM, Montpellier), D. Schlüter(독일 하노버 의과대학)에게 감사드립니다. 이 프로젝트는 유럽 연합의 Horizon 2020 연구 및 혁신 프로그램(AZ에 대한 보조금 계약 101017572 없음) 및 ANRS(AZ에 대한 보조금 ECTZ162721)로부터 자금을 지원받았습니다. IDMIT(Infectious Disease Model and Innovative Therapies) 연구 인프라는 참조 ANR_11_INSB_0008에 따른 "PIA(Programme investissement d'avenir)"의 지원을 받습니다.
자료
| Name | Company | Catalog Number | Comments |
| 0.2 µm syringe filter | ClearLine | 146560 | _ |
| 0.4 % trypan blue | Beckman Coulter | 383200 | _ |
| 1.5 mL tube | Eppendorf | 3810X | _ |
| 24-well plate | Corning | 353047 | _ |
| 6x TriTrack DNA Loading Dye | Thermo scientific | R1161 | _ |
| 75 cm² Culture Flask Vented Cap | Corning | 353136 | _ |
| 8-Strip PCR Tubes with Caps | Life technologies | AM12230 | _ |
| 96-well plates Flat bottom | Corning | 353072 | _ |
| 96-well plates Round bottom | Corning | 353077 | _ |
| Agarose | Euromedex | D5 | _ |
| ATGpr | _ | _ | https://atgpr.dbcls.jp/ |
| ChemiDoc Imaging System | BIO-RAD | 12003153 | _ |
| Counting slide | NanoEntek | DHC-N04 | _ |
| CRISPOR | _ | _ | http://crispor.gi.ucsc.edu/ |
| DPBS | Gibco | 14190094 | _ |
| Ensembl | EMBL-EBI | _ | https://www.ensembl.org/index.html |
| Fetal Bovine Serum | Sigma-Aldrich | F7524 | _ |
| FlowJo | BD Life Sciences | v10.10 | _ |
| GeneRuler 100 bp Plus DNA Ladder | Thermo scientific | SM0323 | _ |
| Genome Data Viewer | NCBI | _ | https://www.ncbi.nlm.nih.gov/gdv/ |
| GraphPad Prism | Dotmatics | _ | Version 9.3.1 |
| Herculase II Fusion DNA Polymerases | Agilent | 600679 | _ |
| ICE CRISPR Analysis Tool | Synthego | _ | https://www.synthego.com/products/bioinformatics/crispr-analysis |
| Image Lab Touch | BIO-RAD | _ | Version 2.4.0.03 |
| NEBuffer 2 | New England Biolabs | B7002S | Included with T7EI M0302S |
| Neon Kit, 10 µL | Invitrogen | MPK1025K | Electroporation kit containing tips, tubes, buffer R and E |
| Neon Transfection System | Invitrogen | MPK5000 | _ |
| NetStart 1.0 | _ | _ | https://services.healthtech.dtu.dk/services/NetStart-1.0/ |
| Nuclease-free Water | Synthego | _ | _ |
| PCR primer (EGFP) | Eurofins | _ | Fw : GGAATGCAAGGTCTGTTGAATG ; Rev : CACCTTGATGCCGTTCTTCT |
| PCR primer (SAMHD1) | Eurofins | _ | Fw : CGGGATTGATTTGAGGACGA ; Rev : GGGTGGCAAGTTAGTGAAGA |
| Penicillin-streptomycin (10,000 U/mL) | Gibco | 15140122 | _ |
| PFA | Electron Microscopy Sciences | 15714 | _ |
| PMA | Sigma-Aldrich | P8139 | _ |
| PrimerQuest | IDT | _ | https://eu.idtdna.com/pages/tools/primerquest |
| QIAquick PCR Purification Kit | Qiagen | 28104 | _ |
| QuickExtract DNA Extraction Solution | Biosearch Technologies | QE09050 | _ |
| RPMI 1640, GlutaMAX | Gibco | 61870010 | _ |
| SnapGene Viewer | Dotmatics | _ | Version 7 |
| SpCas9 2NLS Nuclease | Synthego | _ | _ |
| SYBR Safe DNA Gel Stain | Invitrogen | S33102 | _ |
| Synthetic sgRNA (EGFP) | Synthego | _ | #1 : CGCGCCGAGGUGAAGUUCGA ; #2 : UUCAAGUCCGCCAUGCCCGA ; #3 : CAACUACAAGACCCGCGCCG |
| Synthetic sgRNA (SAMHD1) | Synthego | _ | #1 : AUCGCAACGGGGACGCUUGG ; #2 : GCAGUCAAGAACCUCGGCGC ; #3 : CCAUCCCGACUACAAGACAU |
| Syringe Plastipak Luer Lock | BD | 301229 | _ |
| T100 Thermal Cycler | BIO-RAD | 1861096 | _ |
| T7 endonuclease I | New England Biolabs | M0302S | _ |
| TAE buffer UltraPure, 10x | Invitrogen | 15558026 | 400 mM Tris-Acetate, 10 mM EDTA |
| THP-1 cells | ATCC | TIB-202 | _ |
| Trypsin-EDTA (0,05 %) | Gibco | 25300054 | _ |
| ZE5 Cell Analyzer | BIO-RAD | 12014135 | _ |
참고문헌
- Tsuchiya, S., et al. Establishment and characterization of a human acute monocytic leukemia cell line (THP-1). Int J Cancer. 26 (2), 171-176 (1980).
- Danis, V. A., Millington, M., Hyland, V. J., Grennan, D. Cytokine production by normal human monocytes: inter-subject variation and relationship to an IL-1 receptor antagonist (IL-lRa) gene polymorphism. Clin Exp Immunol. 99, (1995).
- Bol, S. M., et al. Donor variation in in vitro HIV-1 susceptibility of monocyte-derived macrophages. Virology. 390 (2), 205-211 (2009).
- Appleby, L. J., et al. Sources of heterogeneity in human monocyte subsets. Immunol Lett. 152 (1), 32-41 (2013).
- O'Neill, M. B., et al. Single-cell and bulk RNA-sequencing reveal differences in monocyte susceptibility to Influenza A virus infection between Africans and Europeans. Front Immunol. 12, 768189 (2021).
- Wen, Y., et al. Comparability study of monocyte-derived dendritic cells, primary monocytes, and THP1 cells for innate immune responses. J Immunol Methods. 498, 113147 (2021).
- Inagaki, Y., et al. Interferon-g-induced apoptosis and activation of THP-1 macrophages. Life Sci. 71 (21), 2499-2508 (2002).
- Boonkaewwan, C., Toskulkao, C., Vongsakul, M. Anti-inflammatory and immunomodulatory activities of stevioside and its metabolite steviol on THP-1 cells. J Agric Food Chem. 54 (3), 785-789 (2006).
- Chanput, W., et al. β-Glucans are involved in immune-modulation of THP-1 macrophages. Mol Nutr Food Res. 56 (5), 822-833 (2012).
- Cui, J., et al. USP3 inhibits type I interferon signaling by deubiquitinating RIG-I-like receptors. Cell Res. 24 (4), 400-416 (2014).
- Chen, S., et al. SAMHD1 suppresses innate immune responses to viral infections and inflammatory stimuli by inhibiting the NF-κB and interferon pathways. Proc Natl Acad Sci USA. 115 (16), E3798-E3807 (2018).
- Pradhananga, S., Spalinskas, R., Poujade, F. A., Eriksson, P., Sahlén, P. Promoter anchored interaction landscape of THP-1 macrophages captures early immune response processes. Cell Immunol. 355, 104148 (2020).
- Mezzasoma, L., Talesa, V. N., Romani, R., Bellezza, I. Anp and BNP exert anti-inflammatory action via npr-1/cgmp axis by interfering with canonical, non-canonical, and alternative routes of inflammasome activation in human THP1 cells. Int J Mol Sci. 22 (1), 1-17 (2021).
- Martinat, C., et al. SUMOylation of SAMHD1 at Lysine 595 is required for HIV-1 restriction in non-cycling cells. Nat Commun. 12 (1), 4582 (2021).
- Rensen, E., et al. Clustering and reverse transcription of HIV-1 genomes in nuclear niches of macrophages. EMBO J. 40 (1), e105247 (2021).
- Ikeda, T., et al. APOBEC3 degradation is the primary function of HIV-1 Vif determining virion infectivity in the myeloid cell line THP-1. mBio. 14 (4), e0078223 (2023).
- Jinek, M., et al. A programmable dual RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337, 816-821 (2012).
- Kurosaki, T., Maquat, L. E. Nonsense-mediated mRNA decay in humans at a glance. J Cell Sci. 129 (3), 461-467 (2016).
- Tuladhar, R., et al. CRISPR-Cas9-based mutagenesis frequently provokes on-target mRNA misregulation. Nat Commun. 10 (1), 4056 (2019).
- Embree, C. M., Abu-Alhasan, R., Singh, G. Features and factors that dictate if terminating ribosomes cause or counteract nonsense-mediated mRNA decay. J Biol Chem. 298 (11), 102592 (2022).
- Xue, C., Greene, E. C. DNA repair pathway choices in CRISPR-Cas9-mediated genome editing. Trends Genet. 37 (7), 639-656 (2021).
- Schnoor, M., et al. Efficient non-viral transfection of THP-1 cells. J Immunol Methods. 344 (2), 109-115 (2009).
- Tang, X., et al. A method for high transfection efficiency in THP-1 suspension cells without PMA treatment. Anal Biochem. 544, 93-97 (2018).
- Ji, W., Zhang, L., Liu, X. Protocol using lentivirus to establish THP-1 suspension cell lines for immunostaining and confocal microscopy. STAR Protoc. 4 (1), 102032 (2023).
- Guo, C., Ma, X., Gao, F., Guo, Y. Off-target effects in CRISPR/Cas9 gene editing. Front Bioeng Biotechnol. 11, (2023).
- . Synthego Performance Analysis, ICE Analysis Available from: https://www.synthego.com/help/citing-ice (2019)
- Salamov, A. A., Nishikawa, T., Swindells, M. B. Assessing protein coding region integrity in cDNA sequencing projects. Bioinformatics. 14, 384-390 (1998).
- Pedersen, A. G., Nielsen, H. Neural network prediction of translation initiation sites in eukaryotes: Perspectives for EST and genome analysis. Proc Int Conf Intell Syst Mol Biol. 5, 226-233 (1997).
- Concordet, J. P., Haeussler, M. CRISPOR: Intuitive guide selection for CRISPR/Cas9 genome editing experiments and screens. Nucleic Acids Res. 46 (W1), W242-W245 (2018).
- Laguette, N., et al. SAMHD1 is the dendritic- and myeloid-cell-specific HIV-1 restriction factor counteracted by Vpx. Nature. 474 (7353), 654-657 (2011).
- Hrecka, K., et al. Vpx relieves inhibition of HIV-1 infection of macrophages mediated by the SAMHD1 protein. Nature. 474 (7353), 658-661 (2011).
- Seki, A., Rutz, S. Optimized RNP transfection for highly efficient CRI SPR/Cas9-mediated gene knockout in primary T cells. J Exp Med. 215 (3), 985-997 (2018).
- Batista Napotnik, T., Polajžer, T., Miklavčič, D. Cell death due to electroporation - A review. Bioelectrochemistry. 141, 107871 (2021).
- Perretta-Tejedor, N., Freke, G., Seda, M., Long, D. A., Jenkins, D. Generating mutant renal cell lines using CRISPR technologies. Methods Mol Biol. 2067, 323-340 (2020).
- Sentmanat, M. F., Peters, S. T., Florian, C. P., Connelly, J. P., Pruett-Miller, S. M. A survey of validation strategies for CRISPR-Cas9 editing. Sci Rep. 8 (1), 888 (2018).
- Makino, S., Fukumura, R., Gondo, Y. Illegitimate translation causes unexpected gene expression from on-target out-of-frame alleles created by CRISPR-Cas9. Sci Rep. 6, 39608 (2016).
- Bowling, A., et al. Downstream alternate start site allows N-terminal nonsense variants to escape NMD and results in functional recovery by readthrough and modulator combination. J Pers Med. 12 (9), 1448 (2022).
- Inácios, &. #. 1. 9. 4. ;., et al. Nonsense mutations in close proximity to the initiation codon fail to trigger full nonsense-mediated mRNA decay. J Biol Chem. 279 (31), 32170-32180 (2004).
- Li, J., et al. Efficient inversions and duplications of mammalian regulatory DNA elements and gene clusters by CRISPR/Cas9. J Mol Cell Biol. 7 (4), 284-298 (2015).
- Guo, Y., et al. CRISPR inversion of CTCF sites alters genome topology and enhancer/promoter function. Cell. 162 (4), 900-910 (2015).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유