Method Article
Tek Parçacıklı İnterferometrik Yansıma Görüntüleme Bireysel Hücre Dışı Veziküllerin Karakterizasyonu ve Popülasyon Dinamiği
Bu Makalede
Özet
Bu protokol, hücre dışı vezikül (EV) boyutu, EV sayısı, EV fenotipi ve EV biyobelirteç kolokalizasyonunun çok seviyeli ve kapsamlı ölçümleri için tasarlanmış tek parçacıklı interferometrik yansıma görüntülemesi sunar.
Özet
Hücre dışı veziküller (EV'ler), çoğu hücre tarafından salgılanan bir lipid çift tabakasına sahip nanometre boyutunda veziküllerdir. EV'ler, protein, lipid, DNA ve RNA dahil olmak üzere çok sayıda farklı biyolojik molekül taşır ve çeşitli doku ve organlarda hücreden hücreye iletişimi kolaylaştırdığı varsayılır. Son zamanlarda, EV'ler çeşitli hastalıklar için tanı ve tedavi ajanları için biyobelirteçler olarak büyük ilgi görmüştür. EV karakterizasyonu için birçok yöntem geliştirilmiştir. Bununla birlikte, EV analizi için mevcut yöntemlerin hepsinin farklı sınırlamaları vardır. Bu nedenle, EV izolasyonu ve karakterizasyonu için verimli ve etkili yöntemler geliştirmek, olgunlaştıkça bu son teknoloji araştırma alanı için çok önemli adımlardan biri olmaya devam etmektedir. Burada, saflaştırılmamış biyolojik kaynaklardan EV'leri ve diğer metodolojilerle saflaştırılmış EV'leri tespit edebilen ve karakterize edebilen bir yöntem olarak tek parçacıklı interferometrik yansıma görüntüleme sensörünü (SP-IRIS) özetleyen ayrıntılı bir protokol sunuyoruz. Bu gelişmiş teknik, EV boyutu, EV sayısı, EV fenotipi ve biyobelirteç kolokalizasyonunun analizi için çok seviyeli ve kapsamlı ölçümler için kullanılabilir.
Giriş
Hücre dışı veziküller (EV'ler), kan, anne sütü, tükürük, idrar, safra, pankreas suyu ve beyin omurilik ve periton sıvıları dahil olmak üzere çok sayıda biyolojik sıvıdan izole edilebilen, hücresel kökenli nanometre boyutunda zar vezikülleridir. EV'lerin türetilmesi üç ana mekanizma ile gerçekleşir: apoptoz, multiveziküler cisimlerin plazma zarı ile füzyonu yoluyla salınması ve plazma zarınınkanaması 1. Donör hücre bileşenlerinin komşu veya uzak hücrelere ve dokulara EV transferine ilişkin kanıtlar, bu zarla çevrili paketlerin parakrin ve uzun mesafeli veya endokrin sinyal kaskadlarında önemli roller oynayabileceğini düşündürmektedir 1,2,3. EV'ler bir hücrenin fenotipinin anlık görüntüsünü sağlayabildiğinden, çeşitli hastalıkların tedavisi için tanı ve tedavi araçları olarak kullanılma potansiyelleri aktif bir araştırma alanı haline gelmiştir 4,5,6,7,8.
EV karakterizasyonuna yönelik birçok yöntem geliştirilmiştir 9,10,11,12,13. Bu yöntemlerin çoğu, EV'lerin popülasyonları hakkında öncelikle toplu olarak benzersiz ve değerli bilgiler sağlar. Bu tekniklerin bir alt kümesi, tek EV'ler içindeki veya üzerindeki maddelerle ilgili ayrıntılar sağlayabilirken, EV'leri tek EV düzeyinde karakterize etmek için sınırlamalar olabilir. Örneğin, immüno-elektron mikroskobu, tek EV'leri ve bileşimlerini anlamak için kullanılabilir, ancak bu teknik düşük verimlidir, popülasyon dinamiklerini tanımlamak için kullanılma kabiliyeti ciddi şekilde sınırlıdır ve önemli yöntemler geliştirme gerektirir14.
Son zamanlarda, ExoView platformu aracılığıyla tek parçacıklı interferometrik yansıma görüntüleme sensörü (SP-IRIS) tekniğinin geliştirilmesi ve ticarileştirilmesi, rutin ve basit bir otomatik veri toplama yöntemi kullanılarak bireysel EV karakterizasyonunun önünü açmıştır. Bu teknolojinin özü, tek biyolojik nanopartiküllerin interferometrik ölçümünü sağlayan 1 cm x 1 cm Si/SiO2 çift katmanlı çiptir. Çip, altı adede kadar farklı yakalama türünün çoğullanmış tespitine olanak tanıyan, ayrı ayrı işlevselleştirilmiş antikor noktalarından oluşan bir mikro dizi ile kaplanmıştır. Standart çipler, inkübasyon adımı sırasında yakalama için ortak tetraspanin işaretleyicilerini (CD81, CD63 ve CD9) içerir ve kullanıcı, tetraspaninlerden ayrı EV popülasyonlarını izole etmek için ek özel yakalama noktaları ekleyebilir. Kuluçka adımından sonra, her bir yakalama noktası, karşılık gelen işaretleyiciyi ifade eden birçok EV'yi kendisine bağlamıştır. Yakalanan bu EV'ler daha sonra basitçe yıkanabilir, kurutulabilir ve SP-IRIS15 aracılığıyla bir sayı ağırlıklı boyut dağılımı vermek için yakalama noktasına bağlı veziküllerin boyutunu 50-200 nm arasında ölçmek için okuyucuda taranabilir. Sistem ayrıca, yakalanan EV'lerin immüno-etiketlemesi için üç floresan algılama kanalı sunar ve hem SP-IRIS ölçümleri gibi boyutla sınırlı olmayan ortalama floresan yoğunluğunu hem de her floresan lekesi için kolokalizasyon özelliklerini sağlar. Bu, kullanıcının, EV başına dört farklı biyobelirteç (yakalama artı üç immünofloresan etiketi) ekranına dayalı olarak tek EV popülasyonlarını tanımlamasına olanak tanır. Sistem, immünofloresan ile yüzey proteinlerini ölçmenin ötesine geçebilir, çünkü isteğe bağlı bir kargo protokolü, kullanıcının yakalanan EV'lerin iç proteinlerini ve membranı kapsayan yüzey işaretleyicilerinin luminal epitoplarını araştırmasına ve ayrıca kullanıcının EV membran bütünlüğünü kontrol etmesine olanak tanır. Bu makalede, büyük EV popülasyonlarında tek bir EV seviyesinde dört adede kadar farklı biyobelirteç ile EV boyutu ve sayısı ile ilgili tutarlı veriler elde etmek için gerekli adımları özetleyen ayrıntılı bir protokol sunuyoruz. Bu teknik, hem işlenmemiş biyolojik sıvılarda hem de ultrasantrifüjleme, ultrafiltrasyon, çökeltme ajanları, immünoaffinite yakalama, mikroakışkanlar ve boyut dışlama kromatografisi gibi herhangi bir sayıda teknik kullanılarak izole edilen EV'lerde kullanılabilir.
Aşağıda açıklanan protokol, HEK 293 hücre kültürü ortamından ve yerleşik bir izolasyon yöntemi16 kullanılarak fare serumundan türetilen hücre dışı vezikülleri (EV) kullanır. Protokol, biyolojik sıvılardan izole edilen çok sayıda başka biyolojik sıvıya, hücre kültürü ortamına ve saflaştırılmış hücre dışı veziküllere uygulanmıştır. Bu protokol, Şekil 1'de gösterilen tipik bir deney için iş akışı ile iki günlük bir prosedüre bölünmüştür.

Şekil 1: Tahlil iş akışı. Boyut ve sayım, boyut sayımı ve yüzey boyama ve boyut sayımı ve kargo boyama arasında numune için tamamlanacak analiz türünü seçmek için tahlil iş akışı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Protokol
Serum örnekleri, Kansas Üniversitesi Tıp Merkezi (KUMC) protokolünde onaylanmış bir Kurumsal Hayvan Bakımı ve Kullanımı Komitelerine (IACUC) göre farelerden toplandı. Bu biyolojik örneklerin bu deneylerde kullanılması KUMC tarafından da onaylanmıştır.
1. Numune hazırlama (1. Gün)
- Nanopartikül izleme veya eşdeğer bir teknik kullanarak EV konsantrasyonunu belirleyin.
- Numuneyi inkübasyon çözeltisi ile 5 x 107-5 x 108 EVs/mL konsantrasyona seyreltin; minimum 50 μL gereklidir.
NOT: EV konsantrasyonunun bilinmediği numuneler için, 1 μg/mL'deki toplam protein ikame ölçü olarak kullanılabilir. Numunenin konsantrasyonunun düşük olması bekleniyorsa, yüklemeden önce İnkübasyon Çözeltisinde en az 1:1 seyreltme gerçekleştirin. - 24 oyuklu bir plakayı titreşimlerden ve kaba hareketlerden arındırılmış düz bir yüzeye yerleştirin.
NOT: Kontrastı artırmak için, plakanın altına beyaz bir kağıt yerleştirilebilir. - Kuyuları çevreleyen alanlara su ekleyin (Şekil 2).

Şekil 2: 24 kuyulu plaka yerleşimi. ddH2O'nun nerede kesileceği (mavi boya sadece görselleştirme amacıyla eklenmiştir) ve talaşların tutulacağı kuyuların yerleri gösterilir. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
2. Çiplerin hazırlanması ve ön taranması
- Cips içeren kapalı 48 kuyulu plakayı 4-8 °C buzdolabından çıkarın ve contayı açmadan önce oda sıcaklığına (~ 15 dakika) ulaşmalarını bekleyin.
NOT: Bu, talaşlar üzerinde lekelere zarar verebilecek yoğuşmayı önlemek için gereklidir. Talaşı paketten çıkardıktan sonra yüzeyde yoğuşma görülürse, diğerlerini çıkarmadan önce daha uzun süre bekleyin. - Ön tarama çalışmasına hazırlanmak üzere aynayı almak için 8. adıma geçin (Şekil 3).
NOT: Ön tarama verileri, numune ile inkübasyondan önce yakalama noktalarında tespit edilebilen herhangi bir parçacığı tanımlamak için kullanılacaktır, böylece analiz adımı sırasında çıkarılabilirler.

Şekil 3: Çipi makineye yüklemek için kullanılan aynanın görüntüsü. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
- Cımbız kullanarak, 48 oyuklu plakadan istenen sayıda talaşı (numune başına bir çip) çıkarın ve cipsleri ön tarama çalışması için doğrudan aynaya yükleyin. Deney için kullanılması amaçlanan tüm çipler adım 8'de açıklandığı gibi yüklendiğinde ve önceden tarandığında, adım 2.4'e geçin.
NOT: Talaşları tutarken, antikor yakalama noktaları bu bölgede olduğundan ve cımbızla dokunulduğunda zarar göreceğinden, merkezdeki karelere dokunmadığınızdan emin olun (Şekil 4).

Şekil 4: Talaş ve uygun talaş kullanımı. (A) Sarı noktalı çizgi, benekli antikorların konumunu veya çipin işlevsel tarafını gösterir. Çipli Kimlik Numarası ("58") satırının altında yer almaktadır. Şekil ayrıca uygun kullanımı da göstermektedir. (B) Çipin uygun olmayan şekilde kullanıldığını gösterir. (C) Çipin işlevsel olmayan tarafı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
- Önceden taranmış her bir çipi, işlevselleştirilmiş yüzeyi yukarı bakacak şekilde önceden hazırlanmış 24 oyuklu plakaya yerleştirin. İşlevselleştirilmiş yüzey, farklı numaralandırma, hizalama ızgaraları ve ortadaki üç kara kutu tarafından kolayca tanınır. İşlevselleştirilmemiş taraf, düz gümüş düzgün bir yüzeydir.
- Her bir çipin kuyu içinde ortalandığından emin olmak için ince uçlu cımbız kullanın (Şekil 5).
NOT: Talaşın kuyu içinde merkezlenmesi çok önemlidir, çünkü talaş kuyu duvarına temas ederse numune yüklemeden sonra fitillenebilir.

Şekil 5: Kuyuya uygun talaş yerleşiminin gösterilmesi. (A) Talaşlar, kuyunun kenarlarına hiçbir köşe değmeyecek şekilde kuyunun ortasına yerleştirilmelidir. (B) Köşelerin kuyu kenarlarına değdiği yerde talaşın yanlış yerleştirilmesinin tasviri. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
3. Çip ve numunenin yüklenmesi ve inkübasyonu
- Hazırlanan numunenin 35 μL'sini (1. adımdan itibaren) çipin üzerine pipetleyin.
NOT: Numunenin çip üzerinde eşit dağılımını engelleyebileceğinden veya çip üzerindeki antikor lekelerine zarar verebileceğinden, baloncuk eklememeye veya pipet ucuyla çipe dokunmamaya dikkat edin. Numune tüm talaş yüzeyine yayılmalıdır. Yükleme sırasında numunenin çipten fitillenmesi durumunda, ya kuyucuğa 250 μL'lik bir hacme kadar ek numune ekleyin ya da 250 μL'ye kadar inkübasyon solüsyonu ekleyin ve bu numune için değişen seyreltme faktörünü not edin. - Numunenin buharlaşmasını önlemek için kitte bulunan plaka filmini kullanarak plakayı kapatın.
- Numuneyi/çipi gece boyunca (~ 16 saat) kapalı plakada oda sıcaklığında titreşim veya hareket olmayan bir alanda inkübe edin.
NOT: Gece boyunca yapılan inkübasyondan sonra, kullanıcı araştırma sorusu için uygun Gün 2 seçeneğini seçecektir. Yalnızca EV boyutu ve sayısı isteniyorsa, 4. adıma (Gün 2) geçin. Çoklu yüzey işaretleyici analizi isteniyorsa, adım 5'e (Gün 2) atlayın.
4. EV boyutunun ve sayısının belirlenmesi (2. Gün)
- Bir çip içeren her bir oyuğun tarafına 1 mL A çözeltisi ekleyin, çözeltiyi doğrudan çip üzerine eklememeye veya çipi pipet ucuyla çizmemeye dikkat edin.
- Plakayı, oda sıcaklığında 3 dakika boyunca ~500 rpm'de dönen bir orbital çalkalayıcı üzerine yerleştirin.
NOT: Talaşlar plaka üzerinde tıkırdarsa, hızı hemen sıvı dönecek ancak tıkırtı olmayacak şekilde azaltın. - 750 μL sıvıyı çıkarın. Talaşın yanlışlıkla kurumasını önlemek için sıvının çıkarılması sırasında plakayı eğmekten kaçının.
- Adım 4.1'de açıklanan tekniği kullanarak 750 μL Çözelti B ekleyin ve oda sıcaklığında 3 dakika boyunca ~ 500 rpm'de çalkalayın.
- Çözelti B ile toplam üç yıkama için 4.3-4.4 adımlarını iki kez daha tekrarlayın.
- Son yıkamanın sonunda, 750 μL çözeltiyi çıkarın ve kuyuda 250 μL Çözelti B bırakın.
- 750 μL çift damıtılmış su (ddH2O) ekleyin ve oda sıcaklığında ~500 rpm'de 3 dakika çalkalayın.
- 10 cm'lik bir Petri kabını 50 mL ddH2O ile doldurun ve bir cımbız kullanarak her seferinde bir çipi kuyudan tabağa aktarın.
NOT: Çipi yatay olarak aktarmaya özen gösterin ve kurumadığından emin olun. ddH2O'yu değiştirmeden önce sekiz adede kadar talaş yıkanabilir. - ddH2O'da, bir cımbız kullanarak çipi kenarlarından tutun ve kalıntıları çıkarmak için tabağın içinde üç tur döndürün.
- Çipi sudan 45°'lik bir açıyla çıkarın ve çipi, çip kimliği yukarı bakacak şekilde emici kağıda yerleştirin (Şekil 6).
NOT: Çipler artık okunmaya hazırdır. SP-IRIS okuyucuda tarama yapmak için 8. adıma geçin.

Şekil 6: Çipi ddH2O suyundan 45°'lik bir açıyla çıkarmanın doğru yolu. (A) Üstten görünüm ve (B) çipin çıkarılacağı açıyı gösteren yandan görünüm. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
5. Antikor çözeltisinin hazırlanması (2. Gün)
- Uygun boyutta bir tüpe (0,5 ila 3 mL) çip başına 300 μL engelleme solüsyonu ekleyin.
- Bloke edici çözeltinin 300 μL'si başına 0.6 μL antikor ekleyin. Tüpe vurarak ve hızlı sıkarak hafifçe karıştırın.
NOT: İnsan CD9, CD81 ve CD63 veya murin CD9, CD81 ve CD63 için antikorlar uygun kitlere dahil edilmiştir. Diğer floresan konjuge antikorlar isteniyorsa, akış sitometrisi için yaptığınız gibi sabit bir numune yüklemesi ile 0.1 ila 10 μg / mL aralığında tipik bir titrasyon kullanarak optimal boyama konsantrasyonunu belirleyin.
6. İmmünofloresan boyama ile EV boyutunun, sayısının ve fenotiplemesinin belirlenmesi
- Bir çip içeren her bir kuyucuğun yanına 1 mL Çözelti A ekleyin, çözeltiyi doğrudan çip üzerine eklememeye veya çipi pipet ucuyla çizmemeye dikkat edin.
- Plakayı, oda sıcaklığında 3 dakika boyunca ~500 rpm'de dönen bir orbital çalkalayıcı üzerine yerleştirin.
NOT: Talaşlar plaka üzerinde tıkırdarsa, hızı hemen sıvı dönecek ancak tıkırtı olmayacak şekilde azaltın. - 750 μL sıvıyı çıkarın. Talaşın yanlışlıkla kurumasını önlemek için sıvının çıkarılması sırasında plakayı eğmekten kaçının.
- Adım 6.1'de açıklanan tekniği kullanarak 750 μL Çözelti A ekleyin ve oda sıcaklığında 3 dakika boyunca ~ 500 rpm'de çalkalayın.
- Çözelti A ile toplam üç yıkama için 6.3 ve 6.4 adımlarını iki kez tekrarlayın.
- Son yıkamanın sonunda, 750 μL çözeltiyi çıkarın ve kuyucukta 250 μL Çözelti A bırakın.
NOT: Kullanıcı Kargo Boyama isterse, bu noktada 7. adıma geçin. Değilse, adım 6.7'ye geçin - Kuyucuktaki her bir çipe 250 μL Antikor Çözeltisi (adım 5) ekleyin. Işıktan korumak için plakayı folyo ile örtün ve oda sıcaklığında orbital çalkalayıcı üzerinde 1 saat çalkalayın.
- 500 μL Çözelti A ekleyin, böylece oyuk başına toplam hacim ~1.000 μL olur.
- Hemen 750 μL'yi çıkarın ve 750 μL çözelti A ekleyin ve oda sıcaklığında 3 dakika orbital çalkalayıcı üzerinde ~500 rpm'de çalkalayın.
- Çözeltinin 750 μL'sini çıkarın ve 750 μL Çözelti B'yi ekleyin ve oda sıcaklığında 3 dakika boyunca ~ 500 rpm'de çalkalayın.
- Toplam üç yıkama için 6.10 adımını iki kez daha tekrarlayın.
- 750 μL çift damıtılmış su (ddH2O) ekleyin ve oda sıcaklığında ~500 rpm'de 3 dakika çalkalayın.
- 10 cm'lik bir Petri kabını 50 mL ddH2O ile doldurun ve bir cımbız kullanarak kuyudan bir çipi tabağa aktarın.
NOT: Çipi yatay olarak aktarmaya özen gösterin ve kurumadığından emin olun. Taze ddH2O ile değiştirilmeden önce sekiz adede kadar talaş yıkanabilir. - ddH2O'da, bir cımbız kullanarak çipi kenarlarından tutun ve kalıntıları gidermek için tabakta üç tur döndürün.
- Çipi 45°'lik bir açıyla sudan çıkarın ve çipi, çip kimliği yukarı bakacak şekilde emici kağıda yerleştirin (Şekil 6).
NOT: Çipler artık okunmaya hazırdır. Okuyucuda tarama için çiplerin nasıl ayarlanacağına ilişkin talimatlar için 8. adıma (Veri Toplama) geçin.
7. İsteğe bağlı kargo boyama
NOT: Bu protokol, iç ve yüzey işaretleyicilerinin aynı anda etiketlenmesine izin verir.
- Her kuyucukta 250 μL Çözelti A kalırken, her kuyucuğa 250 μL Çözelti C ekleyin.
- Hemen orbital çalkalayıcıya yerleştirin ve tam olarak 200 dakika boyunca ~10 rpm'ye ayarlayın.
NOT: Bu adımda ve aşağıdaki adım 7.8'de, hem zamanlama hem de daha yavaş sallama hızı kritik öneme sahiptir; Hızın kuyularda sadece yavaş dönme sağladığından emin olun ve bir zamanlayıcı kullanın. - Çözelti C ile 10 dakikalık inkübasyondan sonra, her bir oyuğa 500 μL Çözelti A ekleyin.
- Hemen 750 μL'yi çıkarın ve 750 μL Çözelti A ekleyin ve oda sıcaklığında 3 dakika orbital çalkalayıcı üzerinde ~500 rpm'de çalkalayın.
- Toplam üç yıkama için adım 7.4'ü iki kez daha tekrarlayın.
- Son yıkamanın sonunda, 750 μL çözeltiyi çıkarın ve ~ 250 μL çözeltiyi çip ile birlikte kuyuda bırakın.
- Her kuyucuğa 250 μL Çözelti D ekleyin.
- Hemen tam olarak 200 dakika boyunca ~10 rpm'ye ayarlanmış orbital çalkalayıcıya yerleştirin.
- Çözelti C ile 10 dakikalık inkübasyondan sonra, her bir oyuğa 500 μL Çözelti A ekleyin.
- Hemen 750 μL'yi çıkarın ve 750 μL çözelti A ekleyin ve oda sıcaklığında 3 dakika orbital çalkalayıcı üzerinde ~500 rpm'de çalkalayın.
- Toplam üç yıkama için 7.10 adımını iki kez tekrarlayın.
- Son yıkamadan sonra çözeltinin 750 μL'sini çıkarın ve boyama ve tahlil protokolünü tamamlamak için yukarıdaki adım 6.7'ye dönün.
8. Veri toplama
NOT: ExoView R100 kullanarak çiplerden veri toplama prosedürü otomatiktir ve kullanıcı girişi gerektirmez. Ayrıntılı talimatlar, Kullanım Kılavuzunda ve talaş taşıyıcının veya "mandren"in yüklenmesi ve veri toplama17 için ilgili videoda bulunabilir.
- Cihazın arkasındaki iki güç anahtarını kullanarak EV karakterizasyon platformunu açın ve ardından masaüstü simgesine çift tıklayarak tarayıcı yazılımını başlatın.
NOT: Tarayıcı yazılımı başlatıldığında, okuyucu otomatik olarak ana ekrana geçecek ve ardından kullanıcıdan "Çipleri yüklemek için kapıyı aç" uyarısı verecektir. - Gümüş tutamağı kaldırarak okuyucunun önündeki kapıyı açın. Bu, kullanıcının aynaya erişmesine ve yazılımda yeni bir tarama ayarlamasına izin vererek sahneyi çıkaracaktır.
- Kaydet klasörüne tıklayarak ve verilerin kaydedilmesi için istenen konumu seçerek kullanıcının verileri kaydetmek istediği konumu belirleyin.
NOT: Kaydetme konumunu deney adı gibi anlaşılır bir ad vermek ve ardından biri ön taramalar ve diğeri son taramalar için olmak üzere iki alt klasör oluşturmak genellikle yararlıdır. Bu, analiz sırasında verileri bulmayı ve eşleştirmeyi kolaylaştırır. - Çipleri taramak için, kontrol bilgisayarındaki çip dosyalarını bulun.
NOT: Çip dosyaları, kullanılan çip üzerindeki antikor nokta düzeninin haritalarıdır ve tarayıcının her çipte nerede tarama yapacağını bilmesini sağlar. Her kitte, içindeki çipler için chipfile'ların bulunduğu bir USB anahtarı vardır. Bunlar, kit teslim alındıktan sonra kontrol bilgisayarında kullanıcıların güvenilir bir şekilde bulabileceği bir yere kaydedilmelidir. - Çipleri, çiplerin üzerindeki numara ayna üzerindeki tutamağa bakacak şekilde aynaya yükleyin ve ardından bilgisayardaki Çip açılır menüsünden, çip dosyaları listesinden her bir çipi seçin ve tarayıcı yazılımındaki sanal aynada uygun eşleşen konuma yerleştirin. Tamamlandığında, kullanıcıdan Chuck'ı S'ye Yerleştirmesini isteyen bir ekran istemi görüntülenir.tage.
- Yüklü mandreni sahneye yerleştirin; ayna üzerindeki manyetik hizalama onu otomatik olarak sahnede doğru konuma taşıyacaktır.tage ve ardından Aynayı Yerleştir'in yanındaki Tamam'a tıklayıntage. Tarayıcı daha sonra otomatik veri toplama rutinine başlayacaktır.
NOT: Yazılım, her bir çipin tarama durumunu başarılı olarak bildirdiğinde veriler analiz edilmeye hazırdır.
9. Veri analizi
- Kontrol PC'sinin masaüstündeki ExoView Analyzer'a çift tıklayın. Yazılım önyüklendikten sonra, Prescan Data düğmesine tıklayın ve bölüm 8.3'te prescan veri kümesi için klasör konumunu seçin.
- Postscan Data düğmesine tıklayın ve bölüm 8.3'teki postscan veri kümesi için klasör konumunu seçin.
NOT: Her iki klasörde de veriler düzgün bir şekilde algılanırsa, en azından bazı yongalar için, yazılım Yonga dosyası konum düğmesinin yanında algılanan "X" sayıda yonga gösterecektir. Düzgün bir şekilde kaydedildiğinde, algılanan çipler taranan çip sayısıyla eşleşmelidir. - Veri yükleme alanının altındaki İleri'ye tıklayın.
- Meta tablo açılır menüsünde, her çipin bir hücresi olacaktır. İlgili kutuya tıklayarak ve bilgileri yazarak numune adlarını, seyreltme faktörlerini ve her bir algılama kanalında hangi işaretleyicilerin lekelendiğini girin; tamamlandığında, tekrar İleri'ye tıklayın.
NOT: Bu meta veriler, verilerle birlikte kaydedilecektir. - Sol üstteki QC düğmesinin altındaki Devre Dışı Bırak sekmesine tıklayarak kalite kontrolünü (QC) önceden oluşturun.
NOT: Bu özellik, belirli probların ve çiplerin analiz için kapatılmasına izin verir. Yazılım, verilerle ilgili iki farklı uyarı sağlayacaktır, biri belirli bir yakalamanın doymuş olduğunu gösterebilecek yüksek sayımlar için ve bir yakalama türünün kopyalarından birinin diğerlerinden farklı olduğunu tanımlayan yüksek Varyasyon Katsayısı (CV).- Yüksek CV için bir Uyarı Noktasına tıklayın, yazılım tarafından çağrılan noktaları inceleyin ve bariz bir fiziksel hasar olup olmadığına bakın; Hasar tespit edildiğinde, analizde o noktayı kapatmak için Nokta Numarasına tıklayın.
- Tüm uyarılar değerlendirilene kadar tekrarlayın ve ardından İleri'ye tıklayın.
- Sol üstteki QC düğmesinin altındaki Disable (Devre Dışı Bırak) sekmesinin yanında bulunan Cutoff (Kesme) sekmesine tıklayarak kesme analizini önceden oluşturun.
NOT: Bu, kontrol noktası floresan yanıtını bir grafikte sunacak ve kullanıcıya her renk kanalı (kırmızı/yeşil/mavi) için minimum ve maksimum olmak üzere iki ayar sunacaktır. Her bir floresan algılama kanalı için kesme değerinin ayarlanması, yapılması gereken tek veri ayarlamasıdır ve nispeten basittir. Daha da önemlisi, seçilen son kesim bir deney boyunca tutarlı olmalı ve belirtildiği gibi, kırmızı ve yeşil renkte 300-400 a.u. ve tipik deneylerde mavi için 400-700 a.u. Yazılım, bu aralıklar için de kullanıcı için renk kodlu kılavuzlara sahiptir.- O kanalın verilerini yüklemek ve mevcut kesintileri görüntülemek için Yeşil Renk sekmesine tıklayın. İzotip negatif kontrol noktası incelendiğinde, çok düşük floresan yoğunluklarına sahip algılama olayları görülebilir; Bu durumda, kullanıcılar genellikle yeşili 300-400 AU arasında ayarlamayı beklemelidir.
- Veri ekranının altındaki "Ort. % Inc" (kesmenin üzerinde yer alan kontrol noktasında tespit edilen parçacıkların ortalama %'si), o hücrede kırmızı veya sarı vurgulama ile gösterildiği gibi hiçbir uyarı kalmayana kadar algılama kanallarının her biri için minimumu artırın.
NOT: Maksimum değerin genellikle varsayılandan ayarlanması gerekmez, ancak parlak parçacıkları filtrelemek veya floresan algılamayı daha dar bir aralıkla sınırlamak için düşürülebilir. - İleri düğmesine tıklayın.
- Veri ekranının altındaki "Ort. % Inc" (kesmenin üzerinde yer alan kontrol noktasında tespit edilen parçacıkların ortalama %'si), o hücrede kırmızı veya sarı vurgulama ile gösterildiği gibi hiçbir uyarı kalmayana kadar algılama kanallarının her biri için minimumu artırın.
- Bu kanalın verilerini yüklemek ve mevcut kesintileri görüntülemek için Kırmızı Renk sekmesine tıklayın. Yine, izotip negatif kontrol noktası incelendiğinde, çok düşük floresan yoğunluklarına sahip algılama olayları görülebilir; Bu durumda, kullanıcılar genellikle kırmızıyı 300-400 AU arasında ayarlamayı beklemelidir. 9.6.1.1-9.6.1.3 adımlarında belirtilen prosedürlerin aynısını izleyin.
- Bu kanalın verilerini yüklemek ve mevcut kesintileri görüntülemek için Mavi Renk sekmesine tıklayın. Yine, izotip negatif kontrol noktasını inceleyerek, 9.6.1.1-9.6.1.3 adımlarında belirtildiği gibi aynı prosedürleri izleyin.
NOT: Mavi kanal, noktadaki antikorun, boş talaşlar için bile 400-700 au'luk biraz daha yüksek bir kesme gerektirecek yeterli seviyelerde otomatik floresan olması bakımından benzersizdir.
- O kanalın verilerini yüklemek ve mevcut kesintileri görüntülemek için Yeşil Renk sekmesine tıklayın. İzotip negatif kontrol noktası incelendiğinde, çok düşük floresan yoğunluklarına sahip algılama olayları görülebilir; Bu durumda, kullanıcılar genellikle yeşili 300-400 AU arasında ayarlamayı beklemelidir.
- Kesme analizinin tamamlanmasının ardından, İleri'ye tıklayın ve deney boyunca parçacık bağlanmasına genel bir bakış sağlayacak olan ısı haritası grafiği görünecektir. Kullanıcının verileri çizmek için kullanabileceği çeşitli görselleştirme araçları vardır. Bu çizimler ve ilişkili ham veriler, Çizim Raporu Ekle düğmesine tıklanarak bir rapora eklenebilir.
NOT: Isı haritası, belirli bir algılamanın tüm örneklerini, tüm yakalama görünümünü gösteren varsayılan veri ekranıdır. Varsayılan ekran, her bir yakalama noktasına bağlı benzersiz EV'leri ölçen toplamdır. - Raporu Dışa Aktar düğmesine tıklayın ve rapor için bir kaydetme konumu seçin.
NOT: Rapor bir tarayıcıda açılır. Rapora eklenmiş Filtrelenmiş Parçacık Listesi elektronik tablo dosyasını bulun; bu, EVTRACK'e gönderilecek bilgilerdir. - Ekranın üst kısmındaki açılır menülerde yer alan farklı Örnekler ve Yakalama türleri seçimlerini kullanarak rapor eklemeye devam edin. Görüntülenen veriler, sol üstteki kanal algılama düğmelerinin seçimi ile kontrol edilir. Algılama kanalını kapatmak (gri) veya açmak (renkli) için her bir renk düğmesine tıklayarak belirli bir fenotip/örnek seçimi/yakalama kanalı ayarlayın.
- İstenen grafikleri bir raporda toplamak için Rapora Çizim Ekle düğmesine tıklayın.
- Raporu Dışa Aktar'a tıklayın ve nihai rapor için kaydetme konumunu tanımlayın.
Sonuçlar
Şekil 7 (sol panel), çip üzerindeki CD63 noktasına bağlı ve sırasıyla yeşil, kırmızı ve mavi kanallarda CD81, CD63 ve CD9 için boyanmış HEK293 koşullu ortamdan türetilen EV'lerin üç renkli bir kompozit görüntüsünü göstermektedir. Şekil 7 (sağ üst panel), yakalanan EV'lerin her birinin, her kanalda değişen yoğunluklarda bir veya daha fazla rengin birlikte lokalizasyonunu görüntüleyebildiğini gösteren yakınlaştırılmış bir görüntüdür. Yakalanan EV'lerin boyanmasındaki fark, üç tetraspaninin tek veziküller üzerindeki ekspresyonunun heterojenliğini temsil eder ve veri analiz yazılımı ile ölçülebilir. Şekil 7 (sağ alt panel), 50 nm'nin üzerinde olan EV'lerin boyutlandırma histogramlarını gösterir. Boyutlandırma histogramı, CD63 noktasında yakalanan EV'lerin 50 nm'lik bir mod boyutuna sahip olduğunu gösterir. Spot görüntüye bağlı her EV için renk kolokalizasyonu, yoğunluğu ve boyutu, ölçümdeki temel verileri oluşturan fenotipik verileri temsil eder.

Şekil 7: Tam noktanın üç renkli görüntüsü, antiCD9-CF488, antiCD81-CF555, antiCD63-CF647 için bağlanmış ve boyanmış HEK293'ten türetilmiş EV'ler ile tam bir CD63 yakalama noktasını göstermektedir. Yakınlaştırılmış görüntü, floresan kolokalizasyon kullanılarak tek EV fenotiplerinin dijital tespitini gösterir. Bu çip için SP-IRIS'ten türetilen boyut dağılımı sağ altta gösterilmiştir. EV'ler, HEK293T hücrelerin şartlandırılmış ortamından toplandı. Hücreler% 80 birleşmeye kadar kültürlendi ve daha sonra PBS ile yıkandı ve EV tükenmiş FBS (% 10 hacim / hacim) içeren ortamda 48 saat boyunca kültürlendi. 48 saatin sonunda toplanan şartlandırılmış ortam daha sonra hücresel kalıntıları gidermek için 2.500 x g'da döndürüldü ve kalan süpernatant içeren EV'ler analiz edildi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Şekil 8 , bir kullanıcının karşılaşabileceği izotip kontrol yanıtlarının iki örneği için üç renkli bileşik görüntü ve yoğunluk histogramını göstermektedir. Üst panel (örnek örnek 1, EX1) nominal çalışmayı gösterir, izotip kontrol noktasının mavi (>500) ve kırmızı ve yeşil (>300 a.u.) otomatik floresan imzasından daha büyük floresan yoğunluğu ile hiçbir bağlanması yoktur, bu da kullanıcının düşük kesmeler ayarlamasına olanak tanır. Alt panel (numune spesifik olmayan 1, NS1), EV'lerin izotip kontrolüne ne zaman bağlandığını gösterir ve histogramda gösterildiği gibi yüksek yoğunluklu boyama ile sonuçlanır. Bu senaryoda, işaretleyiciye özgü noktalardaki veriler güvenilmezdir, çünkü kontrol noktasındaki EV'lerle aynı yoğunluk aralığını kapsar, aktif noktalardaki EV'lerin özel olarak orada işe alındığı çıkarımı artık geçerli değildir. Bu fenomen meydana geldiğinde, tipik olarak numunenin durumu ile ilgilidir.
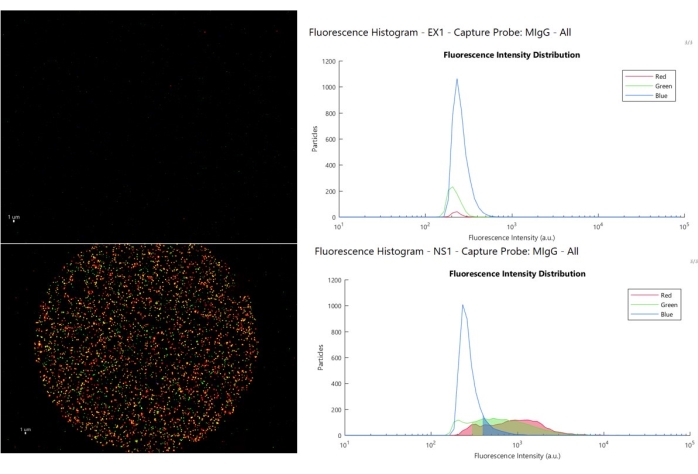
Şekil 8: Kesme analizi. Sol üstteki görüntü, nominal bir kontrol noktasını ve ilişkili floresan histogramları (sağ üst) gösterir, burada kesmenin üzerindeki parçacıklar gölgeli alanda gösterilir, sol alttaki görüntü, kontrol noktasına spesifik olmayan EV bağlanmasının bir örneğini sunar ve floresan histogramındaki gölgeli alanlar, yakalama noktasındaki pozitif EV'lerle aynı aralıkta, kesmenin üzerindeki alanlardır. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Şekil 9 , insan serumundan türetilmiş EV'lerin tek EV protein kolokalizasyonunu temsil etmektedir. EV'ler anti-CD63, CD9, CD81 ve mlgG yakalama antikoru kullanılarak yakalandı ve CD81 (yeşil), HSP72 (kırmızı) ve CD9 (mavi) için boyandı. Şekil 9 , CD81'in insan serumu türevli EV'lerde en çok biriken belirteç olduğunu göstermektedir. CD63 seviyesi ise CD81 ve CD9'a göre en düşük seviyededir. Anti-CD63, CD9 ve CD81 antikoru yakalanan EV'ler arasında, CD63 yakalanan EV'deki HSP72 oranı daha yüksekken, bu seviye anti-CD9 ve CD81 antikor yakalanan EV'ler arasında benzerdir. Tetraspanin belirteçlerini de taşıyan tüm HSP72 pozitif olmayan EV'ler arasında HSP72 ve CD63'ün spesifik lokalizasyonunun bu tespiti, tek parçacık interferometrisi ve floresansın benzersiz veri toplama yeteneğini göstermektedir.

Şekil 9: Tek-EV protein kolokalizasyonu. İnsanlardan türetilen EV'ler, anti-CD63, CD9, CD81 ve mlgG (izotip) yakalama antikoru kullanılarak yakalandı ve CD81 (yeşil), HSP72 (kırmızı) ve CD9 (mavi) için boyandı. EV popülasyonu, floresan modu kullanılarak görüntülendi. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tartışmalar
Mevcut EV karakterizasyon yöntemleri, büyük ölçüde, EV saflaştırma yöntemlerinin 9,10,11,12,13 mevcut deneysel sınırlamaları ile kısıtlanan saflaştırılmış EV'lere dayanmaktadır. Tek parçacıklı interferometrik yansıma görüntüleme (SP-IRIS), numune analizi için gereken saflaştırma adımlarını ortadan kaldırabilen ve böylece zamandan tasarruf sağlayan ve tipik EV iş akışlarıyla ilişkili maliyetleri azaltabilen etkili bir teknolojidir. Gerekli numune girişi genellikle çok küçüktür ve numunenin geri kalanını ek analitik yöntemler için kullanılabilir durumda bırakır.
Ayrıca, EV'leri antijene özgü bir şekilde ölçme yeteneği, temel EV araştırmalarının yanı sıra tanı ve tedavi uygulamalarında nadir olay tespiti için çok önemlidir. Bununla birlikte, hastalığa özgü EV'ler, dolaşımdaki diğer EV'lere ve kirleticilere göre kanda düşük konsantrasyonlarda bulunabilir 2,11,12. Protokol, sırasıyla 488 nm, 555 nm veya 647 nm'de uyarılma ile floresan etiketli antikorlar kullanılarak bu alt popülasyonların tespiti için diğer antikorları içerecek şekilde modifiye edilebilir. Bu durumda, ilgilenilen markörün tespitini optimize etmek için bir titrasyon serisinin kullanılabileceği pozitif ve negatif bir biyolojik kontrol geliştirilmelidir. Bu durumda, SP-IRIS tarafından kullanılan pozitif seçim yönteminin bu deney için zararlı olabileceğine dikkat edilmelidir, çünkü ilgilenilen işaretleyici tetraspanin aşağı çekme ile güçlü bir şekilde kolokalize olamaz ve bu da tespiti zorlaştırır. Bu durum ortaya çıkarsa, ilgilenilen işaretçiyi kullanarak çip üzerindeki EV'leri yakalamak ve böylece tetraspanin lekeleri üzerindeki pozitif seçim yanlılığının üstesinden gelmek avantajlıdır. SP-IRIS görüntülemenin genelleştirilmiş etiketleme yeteneği, belirli bir yakalama noktasında dört adede kadar işaretleyicinin algılanmasını mümkün kılar, bu da SP-IRIS ve uygun kontroller tarafından yeni belirteçlerin algılanmasının nasıl tasarlanacağı ve yorumlanacağı hakkında tartışmayı gerektirir. Bir çip için tek kontrol olarak İzotip kontrol noktasını kullanırken, kesme ayarı sırasında antikorun doğrudan izotip yakalama noktasına spesifik olmayan numune bağlanması olup olmadığını gerçekten düşünüyoruz. İzotip kontrolünde ve işaretleyiciye özgü noktada benzer bir kolokalizasyon imzası gözlemlersek, bu, EV'lerin kontrole özel olmayan bir şekilde yapıştığının ve protokolün incelenmesi gerektiğinin göstergesidir. İyi pozitif ve negatif kontrol numunelerinin seçilmesi, kullanıcıların spesifik olmayan bağlanmanın çip üzerindeki antikorlardan mı yoksa floresan olarak etiketlenmiş birincil antikorlardan mı olduğunu ayırt etmesine yardımcı olabilir. EV'lerin izotip kontrol noktasına bağlandığından şüpheleniliyorsa, negatif bir kontrol EV numunesi bir çip üzerinde sabit bir konsantrasyonda inkübe edilebilir ve ilgilenilen antikorun normal boyama konsantrasyonu ile boyanabilir. Kesme ayarları, bölüm 9 Veri Toplama, adım 9.6'da açıklandığı gibi yapılmalıdır. İlgilenilen işaretçi için noktalarda herhangi bir bağlama bulunmadığını onaylayın. Negatif EV numunesi üzerinde oluşturulan kesikler, deneydeki diğer numunelere uygulanabilir. Pozitif bir kontrol mevcut olduğunda, ek antikorlarla optimal boyama konsantrasyonunu belirlemek için bir titrasyon boyama testi yapmak yararlıdır.
EV'ler için SP-IRIS'te kullanılan spesifik algılamanın avantajı, teknik için sınırlamalar da sunabilir. Bölüm 3'teki inkübasyon adımı, doğası gereği, numunede bulunabilecek parçacıklar hakkında hiçbir bilgi sağlamayan, ancak yakalandıktan sonra yıkanacakları ve analiz sırasında gerçekleştirilmeyecekleri için belirli bir çip üzerindeki yakalama türlerine karşılık gelen belirteçleri göstermeyen parçacıklar hakkında hiçbir bilgi sağlamayan pozitif bir seçim yöntemidir. Tüm EV'lerde kabul edilmiş evrensel bir işaretleyici bulunmaması nedeniyle, kullanıcılar verilerini ölçülen bir popülasyon olarak gözden geçirmeli ve SP-IRIS yakalama verilerinin belirli bir numunedeki tüm EV'leri temsil edip etmediğine karar vermek için toplam partikül konsantrasyonunun ortogonal ölçümlerine karşı yakalanan partikül miktarını dikkate almalıdır.
SP-IRIS tekniği diğer biyolojik nanopartiküllere (örneğin, patojenik virüsler ve viral vektörler) uygulanmıştır18. SP-IRIS'i diğer biyolojik partiküllerin tespitine uygulamak için, çip üzerinde immüno-yakalamaya izin vermek için bir yüzey işaretleyicisine karşı bir yakalama antikoru veya probunun seçilmesi gerekir. Şu anda, 200 nm çapa kadar olan biyolojik parçacıklar SP-IRIS kullanılarak karakterize edilebilir. SP-IRIS tek algılama probu hassasiyetine sahip olduğundan, floresan okuması kullanılıyorsa çapta alt sınır yoktur. SP-IRIS çalışmalarının titizliğini ve tekrarlanabilirliğini sağlamak ve diğer EV verileriyle entegrasyonu kolaylaştırmak için EV Track platformunukullanmayı öneriyoruz 19. Toplam verinin yüklenmesi pratik değildir (gigabaytlarca) ve yalnızca ExoView yazılımını kullananlar tarafından erişilebilir. Bu nedenle, sorgulanan her numune ve bir yayının parçası için, ısı haritası ile kolayca dışa aktarılabilen Filtrelenmiş Parçacık Listesi dosyasının EV Track'e yüklenmesini öneriyoruz. Bu dosya, herhangi bir rakam veya sorunun doğrulanabileceği veya yanıtlanabileceği eksiksiz parçacık verilerine ve kesme ayarlarına sahiptir.
Sonuç olarak, SP-IRIS, tek bir EV boyutunun ve biyobelirteç ekranının rutin ölçümlerini yapmak için basit bir yol sağlayan etkili ve verimli bir EV karakterizasyonu yöntemi sunar. Bu yeni elde edilen veriler, EV yüklemesinin stokiyometrisi, nadir alt popülasyonların doğrudan tespiti hakkında gizli ayrıntıları ortaya çıkarabilir ve EV'lerin sağlık ve hastalıktaki önemli rolünü anlamamızda daha fazla ilerlemenin önünü açmaya yardımcı olabilir.
Açıklamalar
Clayton Deighan ve George Daaboul, NanoView Biosciences, Inc.'in çalışanları ve hissedarlarıdır.
Teşekkürler
Bu çalışma kısmen Kansas Üniversitesi Tıp Fakültesi Araştırma Ekipmanı ve Kaynak Tedarik Ödül Programı tarafından desteklenmiştir. PCG, LKC, FD ve AR, NIA R21 AG066488-01'den sağlanan fonlarla desteklendi.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 10-cm sterile Petri dish | Fisher | FB0875712 | |
| 15mL sterile tube | n/a | various | |
| 24-well cell culture plate, flat bottom | Fisher | 08-772-1 | |
| Blocking Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chipfiles | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chips | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chuck | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 250 ml | Fisher | 09-761-4 | |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 500 ml | Fisher | 09-761-10 | |
| Deionized (DI) water | Fisher | LC267404 | |
| EMS style tweezers with Carbon Fiber tips | Fisher | 50-193-0842 | |
| ExoView Human Tetraspanin Kit | NanoView Biosciences | EV-TETRA-C | Capture for hCD81, hCD9, hCD63, IgG Control + stains for hEV-A (hEV-CD63-647, hEV-CD81-555, hEV-CD9-488) 16 Chips per kit |
| ExoView R100 Imager | NanoView Biosciences | EV-R100 | Interferometric microscope including high specification camera including 3 color fluorescence and label free sizing and counting extracellular vesicles |
| Fluorescently labled huma CD9 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD63 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD81 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Incubation Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Orbital shaker or microplate shaker with digital settings capable of shaking at 500 rpm | n/a | various | |
| Plate Seal | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution A | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution B | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution C | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution D | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Square/flat tip tweezer | Fisher | 50-239-62 | |
| Straight strong point Boley style tweezers | Fisher | 16-100-124 | |
| Thermo Scientific Adhesive PCR Plate Seals | Fisher | AB-0558 |
Referanslar
- Maas, S. L. N., Breakefield, X. O., Weaver, A. M. Extracellular vesicles: Unique intercellular delivery vehicles. Trends in Cell Biology. 27 (3), 172-188 (2017).
- Shah, R., Patel, T., Freedman, J. E. Circulating extracellular vesicles in human disease. The New England Journal of Medicine. 379 (10), 958-966 (2018).
- Deng, F., Miller, J. A review on protein markers of exosome from different bio-resources and the antibodies used for characterization. Journal of Histotechnology. 42 (4), 226-239 (2019).
- Cohen, O., et al. ’Golden’ exosomes as delivery vehicles to target tumors and overcome intratumoral barriers: in vivo tracking in a model for head and neck cancer. Biomaterials Science. 9 (6), 2103-2114 (2021).
- Han, Y., et al. Overview and update on methods for cargo loading into extracellular vesicles. Processes (Basel). 9 (2), 356 (2021).
- Lee, M., Im, W., Kim, M. Exosomes as a potential messenger unit during heterochronic parabiosis for amelioration of Huntington's disease. Neurobiology of Disease. 155, 105374 (2021).
- Sun, B., et al. Characterization and biomarker analyses of post-COVID-19 complications and neurological manifestations. Cells. 10 (2), 386 (2021).
- Jiang, L., Gu, Y., Du, Y., Liu, J. Exosomes: Diagnostic biomarkers and therapeutic delivery vehicles for cancer. Molecular Pharmaceutics. 16 (8), 3333-3349 (2019).
- Bachurski, D., et al. Extracellular vesicle measurements with nanoparticle tracking analysis - An accuracy and repeatability comparison between NanoSight NS300 and ZetaView. Journal of Extracellular Vesicles. 8 (1), 1596016 (2019).
- Carmicheal, J., et al. Label-free characterization of exosome via surface enhanced Raman spectroscopy for the early detection of pancreatic cancer. Nanomedicine. 16, 88-96 (2019).
- Greening, D. W., Xu, R., Ji, H., Tauro, B. J., Simpson, R. J. A protocol for exosome isolation and characterization: evaluation of ultracentrifugation, density-gradient separation, and immunoaffinity capture methods. Methods in Molecular Biology. 1295, 179-209 (2015).
- Wu, Y., Deng, W., Klinke, D. J. Exosomes: improved methods to characterize their morphology, RNA content, and surface protein biomarkers. The Analyst. 140 (19), 6631-6642 (2015).
- van de Vlekkert, D., Qiu, X., Annunziata, I., d'Azzo, A. Isolation and characterization of exosomes from skeletal muscle fibroblasts. Journal of Visualized Experiments: JoVE. (159), (2020).
- Ayala-Mar, S., Donoso-Quezada, J., Gallo-Villanueva, R. C., Perez-Gonzalez, V. H., Gonzalez-Valdez, J. Recent advances and challenges in the recovery and purification of cellular exosomes. Electrophoresis. 40 (23-24), 3036-3049 (2019).
- Daaboul, G. G., et al. Digital detection of exosomes by interferometric imaging. Scientific Reports. 6, 37246 (2016).
- Pohler, K. G., et al. Circulating microRNA as candidates for early embryonic viability in cattle. NMolecular Reproduction and Development. 84 (8), 731-743 (2017).
- NanoView Biosciences. ExoView R100 User Guide. v240.4. NanoView Biosciences. , 202 (2021).
- Daaboul, G. G., et al. Enhanced light microscopy visualization of virus particles from Zika virus to filamentous ebolaviruses. PLoS One. 12, 0179728 (2017).
- ET Consortium. EV-TRACK: transparent reporting and centralizing knowledge in extracellular vesicle research. Nature Methods. 14, 228-232 (2017).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır