Method Article
Interferometrische Reflexionsbildgebung mit Einzelpartikeln, Charakterisierung einzelner extrazellulärer Vesikel und Populationsdynamik
In diesem Artikel
Zusammenfassung
Dieses Protokoll stellt eine interferometrische Einzelpartikel-Reflexionsbildgebung dar, die für die mehrstufige und umfassende Messung der extrazellulären Vesikelgröße (EV), der EV-Anzahl, des EV-Phänotyps und der EV-Biomarker-Kolokalisation konzipiert ist.
Zusammenfassung
Extrazelluläre Vesikel (EVs) sind nanometergroße Vesikel mit einer Lipiddoppelschicht, die von den meisten Zellen sezerniert werden. EVs tragen eine Vielzahl verschiedener biologischer Moleküle, darunter Proteine, Lipide, DNA und RNA, und sollen die Zell-zu-Zell-Kommunikation in verschiedenen Geweben und Organen erleichtern. In jüngster Zeit haben Elektrofahrzeuge als Biomarker für Diagnostika und Therapeutika für verschiedene Krankheiten große Aufmerksamkeit erregt. Für die Charakterisierung von Elektrofahrzeugen wurden viele Methoden entwickelt. Die derzeitigen Methoden zur EV-Analyse haben jedoch alle unterschiedliche Einschränkungen. Daher bleibt die Entwicklung effizienter und effektiver Methoden zur Isolierung und Charakterisierung von Elektrofahrzeugen einer der entscheidenden Schritte für dieses hochmoderne Forschungsfeld, während es reift. Hier stellen wir ein detailliertes Protokoll vor, das einen interferometrischen Reflexionssensor (SP-IRIS) für einzelne Partikel als eine Methode beschreibt, die in der Lage ist, EVs aus ungereinigten biologischen Quellen und gereinigte EVs mit anderen Methoden zu erkennen und zu charakterisieren. Diese fortschrittliche Technik kann für mehrstufige und umfassende Messungen zur Analyse der EV-Größe, der EV-Anzahl, des EV-Phänotyps und der Biomarker-Kolokalisation verwendet werden.
Einleitung
Extrazelluläre Vesikel (EVs) sind nanometergroße Membranvesikel zellären Ursprungs, die aus zahlreichen biologischen Flüssigkeiten isoliert werden können, darunter Blut, Muttermilch, Speichel, Urin, Galle, Pankreassaft sowie Liquor cerebrospinalis und Peritonealflüssigkeit. Die Ableitung von EVs erfolgt über drei Hauptmechanismen: Apoptose, Freisetzung durch Fusion von multivesikulären Körpern mit der Plasmamembran und Bläschen der Plasmamembran1. Hinweise auf einen EV-Transfer von Spenderzellbestandteilen auf benachbarte oder entfernte Zellen und Gewebe deuten darauf hin, dass diese membranumschlossenen Pakete eine wichtige Rolle in parakrinen sowie Langstrecken- oder endokrinen Signalkaskaden spielen können 1,2,3. Da EVs eine Momentaufnahme des Phänotyps einer Zelle liefern können, ist das Potenzial für ihren Einsatz als diagnostische und therapeutische Werkzeuge für die Behandlung verschiedener Krankheiten zu einem aktiven Forschungsgebiet geworden 4,5,6,7,8.
Viele Methoden zur Charakterisierung von EV wurden entwickelt 9,10,11,12,13. Die meisten dieser Methoden liefern einzigartige und wertvolle Informationen über die Populationen von Elektrofahrzeugen, hauptsächlich in großen Mengen. Während eine Teilmenge dieser Techniken Details über Substanzen in oder auf einzelnen EVs liefern kann, kann es Einschränkungen bei der Charakterisierung von EVs auf der Ebene einzelner EVs geben. Zum Beispiel kann die Immunelektronenmikroskopie verwendet werden, um einzelne EVs und ihre Zusammensetzung zu verstehen, aber diese Technik hat einen geringen Durchsatz, ist stark eingeschränkt in ihrer Fähigkeit, zur Beschreibung der Populationsdynamik verwendet zu werden, und erfordert eine erhebliche Methodenentwicklung14.
Vor kurzem hat die Entwicklung und Kommerzialisierung der Einzelpartikel-Interferometrie-Reflektiv-Imaging-Sensortechnik (SP-IRIS) über die ExoView-Plattform die individuelle EV-Charakterisierung mit einer routinemäßigen und einfachen automatisierten Datenerfassungsmethode eröffnet. Das Herzstück dieser Technologie ist der Chip, eine 1 cm x 1 cm große Si/SiO2-Doppelschicht , die die interferometrische Messung einzelner biologischer Nanopartikel ermöglicht. Der Chip ist mit einem Microarray aus einzelnen funktionalisierten Antikörper-Spots bestückt, was eine Multiplex-Detektion von bis zu sechs verschiedenen Capture-Typen ermöglicht. Standard-Chips enthalten die gängigen Tetraspanin-Marker (CD81, CD63 und CD9) für die Erfassung während des Inkubationsschritts, und der Benutzer kann zusätzliche benutzerdefinierte Fangpunkte hinzufügen, um unterschiedliche Populationen von EVs getrennt von den Tetraspaninen zu isolieren. Nach dem Inkubationsschritt hat jeder Capture-Spot viele EVs an sich gebunden, die den entsprechenden Marker ausdrücken. Diese eingefangenen EVs können dann einfach gewaschen, getrocknet und im Reader gescannt werden, um die Größe der an den Fangpunkt gebundenen Vesikel zwischen 50 und 200 nm zu quantifizieren und eine zahlengewichtete Größenverteilung über SP-IRIS15 zu erhalten. Das System bietet außerdem drei Fluoreszenzdetektionskanäle für die Immunmarkierung der eingefangenen EVs und liefert sowohl die mittlere Fluoreszenzintensität, die nicht durch die Größe begrenzt ist, wie z. B. SP-IRIS-Messungen, als auch Kolokalisationsaspekte für jede Fluoreszenzfärbung. Dies ermöglicht es dem Benutzer, Populationen einzelner EVs basierend auf der Anzeige von vier verschiedenen Biomarkern pro EV (Capture plus drei Immunfluoreszenzmarkierungen) zu definieren. Das System kann über die Messung von Oberflächenproteinen mit Immunfluoreszenz hinausgehen, da ein optionales Cargo-Protokoll es dem Benutzer ermöglicht, nach inneren Proteinen der eingefangenen EVs und luminalen Epitope von membranübergreifenden Oberflächenmarkern zu suchen und die Integrität der EV-Membran zu überprüfen. In diesem Artikel stellen wir ein detailliertes Protokoll zur Verfügung, in dem die Schritte beschrieben werden, die erforderlich sind, um konsistente Daten zur Größe und Anzahl von Elektrofahrzeugen zu erhalten, mit bis zu vier verschiedenen Biomarkern auf einer einzigen EV-Ebene bei großen Populationen von EVs. Diese Technik kann sowohl bei unverarbeiteten biologischen Flüssigkeiten als auch bei EVs angewendet werden, die mit einer Vielzahl von Techniken isoliert wurden, wie z. B. Ultrazentrifugation, Ultrafiltration, Fällungsmittel, Immunaffinitätserfassung, Mikrofluidik und Größenausschlusschromatographie.
Das im Folgenden beschriebene Protokoll verwendet extrazelluläre Vesikel (EV), die aus HEK 293-Zellkulturmedien und aus dem Mausserum unter Verwendung einer etablierten Isolierungsmethodegewonnen wurden 16. Das Protokoll wurde auf zahlreiche andere biologische Flüssigkeiten, Zellkulturmedien und gereinigte extrazelluläre Vesikel angewendet, die aus biologischen Flüssigkeiten isoliert wurden. Dieses Protokoll ist in ein zweitägiges Verfahren unterteilt, wobei der Arbeitsablauf für ein typisches Experiment in Abbildung 1 dargestellt ist.

Abbildung 1: Ablauf des Assays. Assay-Workflow zur Auswahl der Art der Analyse, die für die Probe durchgeführt werden soll, zwischen Größe und Anzahl, Größenzählung und Oberflächenfärbung sowie Größenzählung und Frachtfärbung. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Protokoll
Serumproben wurden von Mäusen gemäß einem genehmigten Protokoll des Institutional Animal Care and Use Committees (IACUC) am University of Kansas Medical Center (KUMC) entnommen. Die Verwendung dieser biologischen Proben in diesen Experimenten wurde auch von KUMC genehmigt.
1. Probenvorbereitung (Tag 1)
- Bestimmen Sie die EV-Konzentration mit Nanopartikel-Tracking oder einer gleichwertigen Technik.
- Die Probe wird mit der Inkubationslösung auf eine Konzentration von 5 x 107-5 x 108 EVs/ml verdünnt; Es werden mindestens 50 μl benötigt.
HINWEIS: Bei Proben, bei denen die EV-Konzentration unbekannt ist, kann ein Gesamtprotein von 1 μg/ml als Ersatzmaß verwendet werden. Wenn die Probe voraussichtlich eine niedrige Konzentration aufweist, führen Sie vor dem Laden mindestens eine 1:1-Verdünnung in der Inkubationslösung durch. - Stellen Sie eine 24-Well-Platte auf eine ebene Fläche, die frei von Vibrationen und schroffen Bewegungen ist.
HINWEIS: Um den Kontrast zu verbessern, kann ein weißes Blatt Papier unter die Platte gelegt werden. - Geben Sie Wasser in die Umgebung der Brunnen (Abbildung 2).

Abbildung 2: Layout der 24-Well-Platte. Die Positionen, an denen ddH2O aliquotiert werden soll (blauer Farbstoff wurde nur zu Visualisierungszwecken hinzugefügt) und die Vertiefungen, in denen die Chips gehalten werden, werden angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
2. Vorbereiten und Vorscannen von Chips
- Nehmen Sie die versiegelte 48-Well-Platte mit den Chips aus dem 4-8 °C Kühlschrank und lassen Sie sie auf Raumtemperatur (~15 min) kommen, bevor Sie die Versiegelung öffnen.
HINWEIS: Dies ist wichtig, um Kondensation auf den Spänen zu vermeiden, die die Flecken beschädigen kann. Wenn beim Entfernen des Chips aus der Packung Kondenswasser auf der Oberfläche beobachtet wird, warten Sie länger, bevor Sie andere entfernen. - Fahren Sie mit Schritt 8 fort, um das Spannfutter für den Pre-Scan-Lauf zu entnehmen (Abbildung 3).
HINWEIS: Die Pre-Scan-Daten werden verwendet, um Partikel zu identifizieren, die vor der Inkubation mit der Probe an den Fangstellen nachweisbar sind, damit sie während des Analyseschritts entfernt werden können.

Abbildung 3: Bild des Spannfutters, mit dem der Chip in die Maschine geladen wird. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Entfernen Sie mit einer Pinzette die gewünschte Anzahl von Chips (einen Chip pro Probe) von der 48-Well-Platte und laden Sie die Chips direkt in das Spannfutter für den Prescan-Lauf. Wenn alle Chips, die für das Experiment verwendet werden sollen, wie in Schritt 8 beschrieben eingelegt und vorgescannt wurden, fahren Sie mit Schritt 2.4 fort.
HINWEIS: Achten Sie beim Hantieren mit Chips darauf, die Quadrate in der Mitte nicht zu berühren, da sich die Antikörpereinfangpunkte in diesem Bereich befinden und beschädigt werden, wenn sie mit der Pinzette berührt werden (Abbildung 4).

Abbildung 4: Chip und ordnungsgemäße Handhabung des Chips. (A) Die gelbe gestrichelte Linie zeigt die Position der gefleckten Antikörper oder die funktionelle Seite des Chips. Die Chip-ID befindet sich unter der Zeile ("58"). Die Abbildung zeigt auch die richtige Handhabung. (B) Demonstriert eine unangemessene Handhabung des Chips. (C) Nicht funktionierende Seite des Chips. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
- Legen Sie jeden vorgescannten Chip mit der funktionalisierten Oberfläche nach oben in die vorbereitete 24-Well-Platte. Die funktionalisierte Oberfläche ist leicht an der eindeutigen Nummerierung, den Ausrichtungsrastern und den drei schwarzen Kästchen in der Mitte zu erkennen. Die nicht funktionalisierte Seite ist eine flache, silberne gleichmäßige Oberfläche.
- Stellen Sie mit einer Pinzette mit feiner Spitze sicher, dass jeder Chip in der Vertiefung zentriert ist (Abbildung 5).
HINWEIS: Die Zentrierung des Chips innerhalb der Vertiefung ist von entscheidender Bedeutung, da die Probe nach dem Laden abgerissen werden kann, wenn der Chip die Well-Wand berührt.

Abbildung 5: Demonstration der korrekten Platzierung der Späne in der Vertiefung. (A) Die Späne sollten in der Mitte der Vertiefung platziert werden, ohne dass Ecken die Seiten der Vertiefung berühren. (B) Darstellung einer unsachgemäßen Platzierung des Chips, wo die Ecken die Seiten der Vertiefung berühren. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
3. Beladung und Inkubation des Chips und der Probe
- Pipettieren Sie 35 μl der vorbereiteten Probe (aus Schritt 1) auf den Chip.
HINWEIS: Achten Sie darauf, keine Blasen hinzuzufügen oder den Chip mit der Pipettenspitze zu berühren, da dies die gleichmäßige Verteilung der Probe auf dem Chip verhindern oder die Antikörperflecken auf dem Chip beschädigen kann. Die Probe sollte sich über die gesamte Chipoberfläche verteilen. Falls sich die Probe während des Ladens vom Chip ablöst, geben Sie entweder eine zusätzliche Probe bis zu einem Volumen von 250 μl in die Vertiefung oder fügen Sie eine Inkubationslösung bis zu 250 μl hinzu und notieren Sie den geänderten Verdünnungsfaktor für diese Probe. - Versiegeln Sie die Platte mit der im Kit enthaltenen Plattenfolie, um eine Verdunstung der Probe zu verhindern.
- Inkubieren Sie die Probe/den Chip über Nacht (~16 h) in der versiegelten Platte bei Raumtemperatur an einem Ort, der frei von Vibrationen und Bewegung ist.
HINWEIS: Nach der Inkubation über Nacht wählt der Benutzer die entsprechende Option für Tag 2 für seine Forschungsfrage aus. Wenn nur die EV-Größe und -Anzahl gewünscht sind, fahren Sie mit Schritt 4 (Tag 2) fort. Wenn die Analyse mehrerer Oberflächenmarker gewünscht ist, fahren Sie mit Schritt 5 (Tag 2) fort.
4. Bestimmung der EV-Größe und -Anzahl (Tag 2)
- Geben Sie 1 ml Lösung A auf die Seite jeder Vertiefung, die einen Chip enthält, und achten Sie darauf, die Lösung nicht direkt auf den Chip zu geben oder den Chip mit der Pipettenspitze zu zerkratzen.
- Stellen Sie die Platte 3 Minuten lang bei Raumtemperatur mit ~500 U/min auf einen Orbitalschüttler.
HINWEIS: Wenn die Späne auf der Platte klappern, verringern Sie sofort die Geschwindigkeit, so dass die Flüssigkeit verwirbelt wird, aber nicht klappert. - 750 μl Flüssigkeit entfernen. Vermeiden Sie es, die Platte während der Flüssigkeitsentnahme zu kippen, um ein versehentliches Austrocknen des Späns zu verhindern.
- Fügen Sie 750 μl Lösung B mit der in Schritt 4.1 beschriebenen Technik hinzu und schütteln Sie sie 3 Minuten lang bei ~500 U/min bei Raumtemperatur.
- Wiederholen Sie die Schritte 4.3-4.4 noch zweimal für insgesamt drei Wäschen mit Lösung B.
- Entfernen Sie am Ende der letzten Wäsche 750 μl der Lösung und lassen Sie 250 μl Lösung B in der Vertiefung.
- 750 μl doppelt destilliertes Wasser (ddH2O) hinzufügen und bei ~500 U/min 3 min bei Raumtemperatur schütteln.
- Füllen Sie eine 10 cm große Petrischale mit 50 mL ddH2O und geben Sie jeweils einen Chip aus der Vertiefung in die Schale mit einer Pinzette.
HINWEIS: Achten Sie darauf, den Chip horizontal zu übertragen und sicherzustellen, dass er nicht austrocknet. Bis zu acht Späne können gewaschen werden, bevor der ddH2O ausgetauscht wird. - Halten Sie den Chip in der ddH2O mit einer Pinzette an den Rändern fest und schwenken Sie ihn drei Umdrehungen lang in der Schale, um den Schmutz zu entfernen.
- Nehmen Sie den Chip in einem Winkel von 45° aus dem Wasser und legen Sie den Chip mit der Chip-ID nach oben auf saugfähiges Papier (Abbildung 6).
HINWEIS: Die Chips können jetzt gelesen werden. Fahren Sie mit Schritt 8 fort, um das Scannen auf dem SP-IRIS-Lesegerät durchzuführen.

Abbildung 6: Korrekte Entnahme des Späns aus dem Wasser des ddH2O in einem Winkel von 45° . (A) Ansicht von oben und (B) Ansicht von der Seite, die den Winkel zeigt, in dem der Chip entfernt werden soll. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
5. Herstellung der Antikörperlösung (Tag 2)
- Geben Sie 300 μl Blockierungslösung pro Chip in ein Röhrchen geeigneter Größe (0,5 bis 3 ml).
- Fügen Sie 0,6 μl Antikörper pro 300 μl der Blockierungslösung hinzu. Vorsichtig mischen, indem Sie auf die Tube klopfen und schnell schleudern.
HINWEIS: Antikörper gegen humane CD9, CD81 und CD63 oder murine CD9, CD81 und CD63 sind in den entsprechenden Kits enthalten. Wenn andere fluoreszierend konjugierte Antikörper gewünscht werden, bestimmen Sie die optimale Färbekonzentration mit einer typischen Titration über einen Bereich von 0,1 bis 10 μg/ml bei konstanter Probenbeladung, wie Sie es bei der Durchflusszytometrie tun würden.
6. Bestimmung der EV-Größe, -Anzahl und -Phänotypisierung mit Immunfluoreszenzfärbung
- Geben Sie 1 ml Lösung A auf die Seite jeder Vertiefung, die einen Chip enthält, und achten Sie darauf, die Lösung nicht direkt auf den Chip zu geben oder den Chip mit der Pipettenspitze zu zerkratzen.
- Stellen Sie die Platte 3 Minuten lang bei Raumtemperatur mit ~500 U/min auf einen Orbitalschüttler.
HINWEIS: Wenn die Späne auf der Platte klappern, verringern Sie sofort die Geschwindigkeit, so dass die Flüssigkeit verwirbelt wird, aber nicht klappert. - Entfernen Sie 750 μL der Flüssigkeit. Vermeiden Sie es, die Platte während der Flüssigkeitsentnahme zu kippen, um ein versehentliches Austrocknen des Späns zu verhindern.
- Fügen Sie 750 μl Lösung A mit der in Schritt 6.1 beschriebenen Technik hinzu und schütteln Sie sie 3 Minuten lang bei Raumtemperatur bei ~500 U/min.
- Wiederholen Sie die Schritte 6.3 und 6.4 zweimal für insgesamt drei Wäschen mit Lösung A.
- Entfernen Sie am Ende der letzten Wäsche 750 μl der Lösung und lassen Sie 250 μl Lösung A in der Vertiefung.
HINWEIS: Wenn der Benutzer Ladungsflecken wünscht, fahren Sie an dieser Stelle mit Schritt 7 fort. Wenn nicht, fahren Sie mit Schritt 6.7 fort - Geben Sie 250 μl der Antikörperlösung (Schritt 5) in jeden Chip in der Vertiefung. Decken Sie die Platte zum Lichtschutz mit Folie ab und schütteln Sie sie 1 h lang auf dem Orbitalschüttler bei Raumtemperatur.
- Fügen Sie 500 μl Lösung A hinzu, sodass das Gesamtvolumen pro Vertiefung ~1.000 μl beträgt.
- Sofort 750 μl entfernen und 750 μl Lösung A hinzufügen und bei ~500 U/min auf dem Orbitalschüttler 3 Minuten bei Raumtemperatur schütteln.
- Entfernen Sie 750 μl der Lösung, fügen Sie 750 μl Lösung B hinzu und schütteln Sie sie 3 Minuten lang bei Raumtemperatur bei ~500 U/min.
- Wiederholen Sie Schritt 6.10 noch zweimal für insgesamt drei Wäschen.
- 750 μl doppelt destilliertes Wasser (ddH2O) hinzufügen und bei ~500 U/min 3 min bei Raumtemperatur schütteln.
- Füllen Sie eine 10 cm Petrischale mit 50 mL ddH2O und geben Sie einen Chip aus der Vertiefung mit einer Pinzette in die Schale.
HINWEIS: Achten Sie darauf, den Chip horizontal zu übertragen und sicherzustellen, dass er nicht austrocknet. Bis zu acht Chips können gewaschen werden, bevor sie gegen frische ddH2O ausgetauscht werden. - Halten Sie den Chip im ddH2O mit einer Pinzette an den Rändern fest und schwenken Sie ihn drei Umdrehungen lang in der Schale, um Schmutz zu entfernen.
- Entfernen Sie den Chip in einem Winkel von 45° aus dem Wasser und legen Sie den Chip mit der Chip-ID nach oben auf saugfähiges Papier (Abbildung 6).
HINWEIS: Die Chips können jetzt gelesen werden. Fahren Sie mit Schritt 8 (Datenerfassung) fort, um Anweisungen zum Einrichten der Chips für das Scannen auf dem Lesegerät zu erhalten.
7. Optionale Ladungsfärbung
HINWEIS: Dieses Protokoll ermöglicht die gleichzeitige Beschriftung von internen und Oberflächenmarkern.
- Wenn 250 μl Lösung A in jeder Vertiefung verbleiben, geben Sie 250 μl Lösung C in jede Vertiefung.
- Sofort auf den Orbitalschüttler setzen und für genau 10 min auf ~200 U/min einstellen.
HINWEIS: In diesem Schritt und Schritt 7.8 unten sind sowohl das Timing als auch die langsamere Schüttelgeschwindigkeit entscheidend. Stellen Sie sicher, dass die Geschwindigkeit nur ein langsames Verwirbeln in den Vertiefungen zulässt, und verwenden Sie einen Timer. - Nach 10 Minuten Inkubation mit Lösung C geben Sie 500 μl Lösung A in jede Vertiefung.
- Sofort 750 μl entfernen und 750 μl Lösung A hinzufügen und bei ~500 U/min auf dem Orbitalschüttler 3 Minuten bei Raumtemperatur schütteln.
- Wiederholen Sie Schritt 7.4 noch zweimal für insgesamt drei Wäschen.
- Entfernen Sie am Ende des letzten Waschgangs 750 μl der Lösung und lassen Sie ~250 μl der Lösung in der Vertiefung mit dem Chip.
- Geben Sie 250 μl Lösung D in jede Vertiefung.
- Sofort auf den auf ~200 U/min eingestellten Orbitalschüttler für genau 10 min stellen.
- Nach 10 Minuten Inkubation mit Lösung C geben Sie 500 μl Lösung A in jede Vertiefung.
- Sofort 750 μl entfernen und 750 μl Lösung A hinzufügen und bei ~500 U/min auf dem Orbitalschüttler 3 Minuten bei Raumtemperatur schütteln.
- Wiederholen Sie Schritt 7.10 zweimal für insgesamt drei Wäschen.
- Entfernen Sie 750 μl der Lösung nach dem letzten Waschen und kehren Sie zum Färben und Abschließen des Assay-Protokolls zu Schritt 6.7 zurück.
8. Datenerhebung
HINWEIS: Das Verfahren zum Sammeln von Daten von den Chips mit dem ExoView R100 ist automatisiert und erfordert keine Benutzereingaben. Detaillierte Anweisungen finden Sie in der Bedienungsanleitung und im entsprechenden Video zum Beladen des Chipträgers oder "Spannfutters" und zur Datenerfassung17.
- Schalten Sie die EV-Charakterisierungsplattform mit den beiden Netzschaltern auf der Rückseite des Geräts ein und starten Sie dann die Scanner-Software durch Doppelklicken auf das Desktop-Symbol.
HINWEIS: Wenn die Scanner-Software gestartet wird, wird das Lesegerät automatisch auf den Startbildschirm eingestellt und der Benutzer wird aufgefordert, die Tür zu öffnen, um Chips zu laden. - Öffnen Sie die Klappe an der Vorderseite des Lesegeräts, indem Sie den silbernen Griff anheben. Dadurch wird der Tisch ausgeworfen, sodass der Benutzer auf das Spannfutter zugreifen und einen neuen Scan in der Software einrichten kann.
- Identifizieren Sie den Speicherort, an dem der Benutzer die Daten speichern möchte, indem Sie auf den Ordner Speichern klicken und einen gewünschten Speicherort für die zu speichernden Daten auswählen.
HINWEIS: Es ist oft hilfreich, den Speicherort aussagekräftig zu benennen, z. B. den Namen des Experiments, und dann zwei Unterordner zu erstellen, einen für Vorscans und einen für Nachscans. Dies erleichtert das Auffinden und Abgleichen der Daten während der Analyse. - Um die Chips zu scannen, suchen Sie die Chipdateien auf dem Steuercomputer.
HINWEIS: Chipdateien sind Karten des Antikörper-Spot-Layouts auf dem verwendeten Chip und ermöglichen es dem Scanner zu wissen, wo auf jedem Chip gescannt werden muss. In jedem Kit befindet sich ein USB-Stick mit den Chipfiles für die Chips. Diese sollten nach Erhalt des Kits an einem Ort auf dem Steuercomputer gespeichert werden, an dem sie vom Benutzer zuverlässig gefunden werden können. - Laden Sie die Chips auf das Spannfutter, wobei die Nummer auf den Chips zum Griff am Spannfutter zeigt, und wählen Sie dann aus dem Dropdown-Menü "Chip" auf dem Computer jeden Chip aus der Liste der Chipdateien aus und platzieren Sie ihn an der entsprechenden Stelle im virtuellen Spannfutter in der Scannersoftware. Sobald der Vorgang abgeschlossen ist, wird eine Eingabeaufforderung auf dem Bildschirm angezeigt, in der der Benutzer aufgefordert wird, das Spannfutter auf der Bühne zu platzieren.
- Stellen Sie das beladene Spannfutter auf den Tisch. Die magnetische Ausrichtung am Spannfutter verschiebt es automatisch an die richtige Stelle auf der Bühne und klickt dann auf OK neben Spannfutter auf der Bühne platzieren. Der Scanner beginnt dann mit der automatischen Datenerfassungsroutine.
HINWEIS: Die Daten können analysiert werden, sobald die Software den Scan-Status jedes Chips als erfolgreich meldet.
9. Datenanalyse
- Doppelklicken Sie auf ExoView Analyzer auf dem Desktop des Steuerungs-PCs. Nachdem die Software hochgefahren ist, klicken Sie auf die Schaltfläche Prescan-Daten und wählen Sie den Speicherort für den Prescan-Datensatz in Abschnitt 8.3 aus.
- Klicken Sie auf die Schaltfläche Postscan-Daten und wählen Sie den Ordnerspeicherort für den Postscan-Datensatz in Abschnitt 8.3 aus.
HINWEIS: Wenn die Daten in beiden Ordnern ordnungsgemäß erkannt werden, zeigt die Software zumindest für einige der Chips die Anzahl der erkannten Chips "X" neben der Schaltfläche "Chipfile-Speicherort" an. Wenn sie ordnungsgemäß gespeichert werden, sollten die erkannten Chips mit der Anzahl der gescannten Chips übereinstimmen. - Klicken Sie auf Weiter am unteren Rand des Datenladebereichs.
- Im Dropdown-Menü der Meta-Tabelle hat jeder Chip eine Zelle. Geben Sie die Probennamen, die Verdünnungsfaktoren und die Marker ein, die in den einzelnen Detektionskanälen gefärbt werden, indem Sie in das entsprechende Feld klicken und die Informationen eingeben. Wenn Sie fertig sind, klicken Sie erneut auf Weiter .
HINWEIS: Diese Metadaten werden mit den Daten gespeichert. - Preform-Qualitätskontrolle (QC), indem Sie auf die Registerkarte Deaktivieren unter der Schaltfläche QC oben links klicken.
HINWEIS: Mit dieser Funktion können bestimmte Sonden und Chips für die Analyse ausgeschaltet werden. Die Software gibt zwei verschiedene Warnungen bezüglich der Daten aus: eine für hohe Anzahlen, die darauf hinweisen können, dass eine bestimmte Erfassung gesättigt ist, und eine hohe Variationskoeffizient (CV), die erkennt, wenn sich eine der Replikate eines Erfassungstyps von den anderen unterscheidet.- Klicken Sie auf einen Warnpunkt für einen hohen CV, untersuchen Sie die Stellen, die von der Software aufgerufen werden, und prüfen Sie, ob offensichtliche physische Schäden vorliegen. Wenn der Schaden identifiziert wurde, klicken Sie auf die Punktzahl , um diesen Punkt in der Analyse zu deaktivieren.
- Wiederholen Sie diesen Vorgang, bis alle Warnungen ausgewertet wurden, und klicken Sie dann auf Weiter.
- Preform-Cutoff-Analyse, indem Sie auf die Registerkarte Cutoff klicken, die sich neben der Registerkarte Deaktivieren unter der Schaltfläche QC oben links befindet.
HINWEIS: Dadurch wird die Fluoreszenzreaktion des Kontrollspots in einem Diagramm dargestellt und der Benutzer erhält zwei Einstellungen, ein Minimum und ein Maximum für jeden Farbkanal (Rot/Grün/Blau). Das Festlegen des Cutoff-Werts für jeden Fluoreszenzdetektionskanal ist die einzige Datenanpassung, die vorgenommen werden muss und relativ einfach ist. Wichtig ist, dass der endgültige Grenzwert, der gewählt wird, über ein Experiment hinweg konsistent sein sollte und, wie erwähnt, innerhalb der Faustregeln für 300-400 a.u. in Rot und Grün und 400-700 a.u. für Blau in typischen Experimenten liegen sollte; Die Software verfügt auch für diese Bereiche über farbcodierte Hilfslinien für den Benutzer.- Klicken Sie auf die Registerkarte Grüne Farbe , um die Daten für diesen Kanal zu laden und die aktuellen Cutoffs anzuzeigen. Bei der Untersuchung des Isotyp-Negativkontrollflecks kann es zu Detektionsereignissen mit sehr niedrigen Fluoreszenzintensitäten kommen; In diesem Fall sollten Benutzer in der Regel damit rechnen, das Grün zwischen 300 und 400 AE einzustellen.
- Erhöhen Sie den Mindestwert für jeden Nachweiskanal, bis der "Avg % Inc" (der durchschnittliche Prozentsatz der detektierten Partikel auf dem Kontrollfleck, der über dem Grenzwert liegt) unter der Datenanzeige keine Warnungen mehr aufweist, wie durch rote oder gelbe Hervorhebung in dieser Zelle angezeigt.
HINWEIS: Der Maximalwert muss in der Regel nicht von der Standardeinstellung angepasst werden, sondern kann abgesenkt werden, um helle Partikel herauszufiltern oder die Fluoreszenzdetektion auf einen engeren Bereich zu beschränken. - Klicken Sie auf die Schaltfläche Weiter .
- Erhöhen Sie den Mindestwert für jeden Nachweiskanal, bis der "Avg % Inc" (der durchschnittliche Prozentsatz der detektierten Partikel auf dem Kontrollfleck, der über dem Grenzwert liegt) unter der Datenanzeige keine Warnungen mehr aufweist, wie durch rote oder gelbe Hervorhebung in dieser Zelle angezeigt.
- Klicken Sie auf die Registerkarte Rote Farbe , um die Daten für diesen Kanal zu laden und die aktuellen Cutoffs anzuzeigen. Auch hier kann man bei der Untersuchung des Isotyp-Negativ-Kontrollflecks Detektionsereignisse mit sehr niedrigen Fluoreszenzintensitäten sehen; In diesem Fall sollten Benutzer in der Regel damit rechnen, den roten Wert zwischen 300 und 400 AE einzustellen. Befolgen Sie die gleichen Verfahren wie in den Schritten 9.6.1.1 bis 9.6.1.3.
- Klicken Sie auf die Registerkarte Blaue Farbe , um die Daten für diesen Kanal zu laden und die aktuellen Cutoffs anzuzeigen. Auch hier ist bei der Untersuchung des Isotyp-Negativkontrollflecks das gleiche Verfahren wie in den Schritten 9.6.1.1 bis 9.6.1.3 anzuwenden.
HINWEIS: Der blaue Kanal ist insofern einzigartig, als der Antikörper im Spot in ausreichendem Maße autofluoreszierend ist, um selbst für leere Chips einen etwas höheren Cutoff von 400-700 a.u. zu erfordern.
- Klicken Sie auf die Registerkarte Grüne Farbe , um die Daten für diesen Kanal zu laden und die aktuellen Cutoffs anzuzeigen. Bei der Untersuchung des Isotyp-Negativkontrollflecks kann es zu Detektionsereignissen mit sehr niedrigen Fluoreszenzintensitäten kommen; In diesem Fall sollten Benutzer in der Regel damit rechnen, das Grün zwischen 300 und 400 AE einzustellen.
- Klicken Sie nach Abschluss der Cutoff-Analyse auf Weiter und das Heatmap-Diagramm wird angezeigt, das einen Überblick über die Partikelbindung im gesamten Experiment gibt. Es gibt mehrere Visualisierungswerkzeuge, die der Benutzer zum Plotten der Daten verwenden kann. Diese Diagramme und die zugehörigen Rohdaten können zu einem Bericht hinzugefügt werden, indem Sie auf die Schaltfläche Diagrammbericht hinzufügen klicken.
HINWEIS: Die Heatmap ist die Standarddatenanzeige, die eine Ansicht aller Stichproben, aller Erfassungen einer bestimmten Erkennung anzeigt. Die Standardanzeige ist die Summe, die die eindeutigen EVs quantifiziert, die an jeden Aufnahmepunkt gebunden sind. - Klicken Sie auf die Schaltfläche Bericht exportieren und wählen Sie einen Speicherort für den Bericht aus.
HINWEIS: Der Bericht wird in einem Browser geöffnet. Suchen Sie die Tabellenkalkulationsdatei "Gefilterte Partikelliste", die an den Bericht angehängt ist. Dies sind die Informationen, die an EVTRACK übermittelt werden müssen. - Fahren Sie mit dem Hinzufügen von Berichten fort, indem Sie die verschiedenen Auswahlmöglichkeiten für Stichproben und Erfassungstypen in den Dropdown-Menüs oben auf dem Bildschirm verwenden. Die Anzeige der Daten wird mit der Auswahl der Schaltflächen zur Kanalerkennung oben links gesteuert. Legen Sie einen bestimmten Phänotyp-/Probenauswahl-/Aufnahmekanal fest, indem Sie auf die jeweilige Farbschaltfläche klicken, um diesen Erkennungskanal aus- (ausgegraut) oder ein- (farbig) zu schalten.
- Klicken Sie auf die Schaltfläche Diagramm zum Bericht hinzufügen , um die gewünschten Diagramme in einem Bericht zusammenzufassen.
- Klicken Sie auf Bericht exportieren und definieren Sie den Speicherort für den Abschlussbericht.
Ergebnisse
Abbildung 7 (linkes Bild) zeigt ein dreifarbiges Kompositbild von EVs, die von HEK293-konditionierten Medien abgeleitet wurden, die an den CD63-Spot auf dem Chip gebunden und in den folgenden Kanälen grün, rot und blau für CD81, CD63 und CD9 gefärbt wurden. Abbildung 7 (oben rechts) ist ein vergrößertes Bild, das zeigt, dass jeder der erfassten EVs eine Co-Lokalisierung einer oder mehrerer Farben mit unterschiedlicher Intensität in jedem Kanal anzeigen kann. Der Unterschied in der Färbung der eingefangenen EVs stellt die Heterogenität der Expression der drei Tetraspanine auf einzelnen Vesikeln dar und kann mit der Datenanalysesoftware quantifiziert werden. Abbildung 7 (unterer rechter Bereich) zeigt die Größenhistogramme der EVs, die über 50 nm liegen. Das Größenhistogramm zeigt, dass die erfassten EVs auf dem CD63-Spot eine Modengröße von 50 nm haben. Die Farbkolokalisierung, Intensität und Größe für jeden EV, der an das Spotbild gebunden ist, stellen die phänotypischen Daten dar, die die grundlegenden Daten bei der Messung bilden.

Abbildung 7: Das dreifarbige Bild des Vollflecks zeigt einen vollständigen CD63-Einfangfleck mit HEK293-abgeleiteten EVs, die für antiCD9-CF488, antiCD81-CF555 und antiCD63-CF647 gebunden und gefärbt sind. Das vergrößerte Bild zeigt die digitale Detektion einzelner EV-Phänotypen mittels Fluoreszenz-Kolokalisierung. Die von SP-IRIS abgeleitete Größenverteilung für diesen Chip ist unten rechts dargestellt. EVs wurden aus konditioniertem Medium HEK293T Zellen gesammelt. Die Zellen wurden auf 80 % Konfluenz kultiviert und dann mit PBS gewaschen und 48 Stunden lang in Medien kultiviert, die EV-abgereichertes FBS (10 % vol/vol) enthielten. Konditionierte Medien, die am Ende der 48 Stunden gesammelt wurden, wurden dann bei 2.500 x g heruntergeschleudert, um zelluläre Trümmer zu entfernen, und der verbleibende Überstand, der EVs enthielt, wurde analysiert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Abbildung 8 zeigt ein dreifarbiges Kompositbild und ein Intensitätshistogramm für zwei Beispiele für Typen von Isotypkontrollreaktionen, auf die ein Benutzer stoßen kann. Das obere Feld (Beispielbeispiel 1, EX1) zeigt den nominalen Betrieb, der Isotyp-Kontrollspot hat keine Bindung mit einer Fluoreszenzintensität, die größer ist als die Autofluoreszenzsignatur in Blau (>500) und (>300 a.u.) in Rot und Grün, so dass der Benutzer niedrige Grenzwerte einstellen kann. Das untere Feld (Probe unspezifisch 1, NS1) zeigt, wann EVs an die Isotypkontrolle binden, und führt zu einer Färbung mit hoher Intensität, wie das Histogramm zeigt. In diesem Szenario sind die Daten zu den markerspezifischen Spots unzuverlässig, da sie den gleichen Intensitätsbereich abdecken wie die EVs auf dem Kontrollspot, so dass die Schlussfolgerung, dass die EVs auf den aktiven Spots spezifisch dort rekrutiert werden, nicht mehr gilt. Wenn dieses Phänomen auftritt, hängt es in der Regel mit dem Zustand der Probe zusammen.
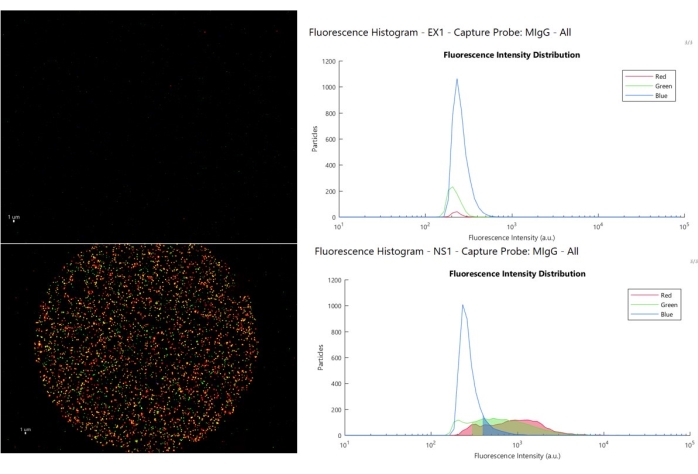
Abbildung 8: Cutoff-Analyse. Das obere linke Bild zeigt einen nominalen Kontrollfleck und die zugehörigen Fluoreszenzhistogramme (oben rechts), wobei die Partikel oberhalb des Grenzwerts im schattierten Bereich dargestellt sind, das untere linke Bild zeigt ein Beispiel für eine unspezifische EV-Bindung an den Kontrollpunkt, und die schattierten Bereiche im Fluoreszenzhistogramm sind diejenigen über dem Grenzwert, im gleichen Bereich wie positive EVs auf dem Erfassungspunkt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Abbildung 9 zeigt die Kolokalisation von aus humanem Serum gewonnenen EVs mit Einzel-EV-Proteinen. EVs wurden mit Anti-CD63-, CD9-, CD81- und mlgG-Capture-Antikörpern eingefangen und mit CD81 (grün), HSP72 (rot) und CD9 (blau) gefärbt. Abbildung 9 zeigt, dass CD81 der am stärksten akkumulierte Marker in aus humanem Serum gewonnenen EVs ist. Während der CD63-Spiegel im Vergleich zu CD81 und CD9 am niedrigsten ist. Bei den mit Anti-CD63-, CD9- und CD81-Antikörpern eingefangenen EVs ist das HSP72-Verhältnis in CD63-eingefangenen EVs höher, während dieses Niveau bei mit Anti-CD9- und CD81-Antikörpern eingefangenen EVs ähnlich ist. Dieser Nachweis der spezifischen Lokalisation von HSP72 und CD63 unter allen nicht-HSP72-positiven EVs, die auch die Tetraspanin-Marker tragen, zeigt die einzigartige Datenerfassungsfähigkeit der Einzelpartikelinterferometrie und Fluoreszenz.

Abbildung 9: Kolokalisation von Single-EV-Proteinen. EVs, die vom Menschen abgeleitet wurden, wurden mit Anti-CD63-, CD9-, CD81- und mlgG-Capture-Antikörpern (Isotyp) eingefangen und für CD81 (grün), HSP72 (rot) und CD9 (blau) gefärbt. Die EV-Population wurde im Fluoreszenzmodus abgebildet. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Diskussion
Aktuelle EV-Charakterisierungsmethoden stützen sich weitgehend auf gereinigte EVs, was durch die aktuellen experimentellen Einschränkungen der EV-Reinigungsmethodeneingeschränkt ist 9,10,11,12,13. Die interferometrische Einzelpartikel-Reflexionsbildgebung (SP-IRIS) ist eine effektive Technologie, die die für die Probenanalyse erforderlichen Aufreinigungsschritte eliminieren und somit Zeit sparen und Kosten senken kann, die mit typischen Arbeitsabläufen bei Elektrofahrzeugen verbunden sind. Der erforderliche Probeneinsatz ist in der Regel sehr gering, so dass der Rest der Probe für zusätzliche Analysemethoden zur Verfügung steht.
Darüber hinaus ist die Möglichkeit, EVs antigenspezifisch zu messen, sehr wichtig für die EV-Grundlagenforschung sowie für die Detektion seltener Ereignisse in diagnostischen und therapeutischen Anwendungen. Krankheitsspezifische EVs können jedoch im Vergleich zu anderen zirkulierenden EVs und Kontaminanten in geringen Konzentrationen im Blut vorhanden sein 2,11,12. Das Protokoll kann modifiziert werden, um andere Antikörper für den Nachweis dieser Subpopulationen unter Verwendung von fluoreszenzmarkierten Antikörpern mit Anregung bei 488 nm, 555 nm bzw. 647 nm aufzunehmen. In diesem Fall sollten eine positive und eine negative biologische Kontrolle entwickelt werden, gegen die eine Titrationsreihe verwendet werden kann, um den Nachweis des interessierenden Markers zu optimieren. In diesem Fall sollte beachtet werden, dass die von SP-IRIS verwendete positive Selektionsmethode für dieses Experiment nachteilig sein kann, da der interessierende Marker nicht stark mit dem Tetraspanin-Pulldown kolokalisieren konnte, was den Nachweis erschwerte. Wenn diese Situation eintritt, ist es vorteilhaft, EVs auf dem Chip mit dem Marker of Interest zu erfassen und so die positive Selektionsverzerrung auf den Tetraspanin-Spots zu überwinden. Die verallgemeinerte Markierungsfähigkeit der SP-IRIS-Bildgebung ermöglicht es, bis zu vier Marker an einem bestimmten Fangpunkt zu detektieren, was eine Diskussion darüber erforderlich macht, wie die Detektion neuartiger Marker mit SP-IRIS und geeignete Kontrollen gestaltet und interpretiert werden kann. Wenn wir den Isotyp-Kontrollspot als einzige Kontrolle für einen Chip verwenden, überlegen wir wirklich, ob es während der Cutoff-Einstellung eine unspezifische Probenbindung des Antikörpers direkt an den Isotyp-Capture-Spot gibt. Wenn wir eine ähnliche Kolokalisationssignatur auf der Isotypkontrolle und dem markerspezifischen Spot beobachten, ist dies ein Hinweis darauf, dass EVs unspezifisch an die Kontrolle gebunden sind und das Protokoll untersucht werden sollte. Die Auswahl guter positiver und negativer Kontrollproben kann den Anwendern helfen zu unterscheiden, ob die unspezifische Bindung von den Antikörpern auf dem Chip oder den fluoreszenzmarkierten Primärantikörpern stammt. Wenn der Verdacht besteht, dass EVs an den Isotyp-Kontrollfleck binden, kann eine negative Kontroll-EV-Probe mit einer festen Konzentration auf einem Chip inkubiert und mit der normalen Färbekonzentration des interessierenden Antikörpers gefärbt werden. Die Cutoff-Einstellungen sollten wie in Abschnitt 9 Datenerfassung, Schritt 9.6 beschrieben durchgeführt werden. Vergewissern Sie sich, dass an den Stellen für den Marker of Interest keine Bindung vorhanden ist. Die an der negativen EV-Probe festgelegten Cutoffs können auf den Rest der Proben im Experiment angewendet werden. Wann immer eine Positivkontrolle zur Verfügung steht, ist es sinnvoll, einen Titrationsfärbetest durchzuführen, um die optimale Färbekonzentration mit zusätzlichen Antikörpern zu bestimmen.
Der Vorteil der spezifischen Detektion, die in SP-IRIS für Elektrofahrzeuge verwendet wird, kann auch Einschränkungen für die Technik darstellen. Der Inkubationsschritt in Abschnitt 3 ist von Natur aus eine Positivselektionsmethode, die keine Informationen über Partikel liefert, die in der Probe vorhanden sein könnten, aber keine Marker aufweist, die den Einfangarten auf einem bestimmten Chip entsprechen, da sie nach der Einfangung weggespült und während der Analyse nicht realisiert werden. Aufgrund der Tatsache, dass es keinen akzeptierten universellen Marker gibt, der auf allen EVs vorhanden ist, sollten Benutzer ihre Daten als gemessene Grundgesamtheit überprüfen und die Menge der eingefangenen Partikel mit orthogonalen Maßen der Gesamtpartikelkonzentration vergleichen, um zu entscheiden, ob die SP-IRIS-Erfassungsdaten alle EVs in einer bestimmten Probe repräsentieren.
Die SP-IRIS-Technik wurde auf andere biologische Nanopartikel (z. B. pathogene Viren und virale Vektoren) angewendet18. Um SP-IRIS auf den Nachweis anderer biologischer Partikel anzuwenden, muss ein Capture-Antikörper oder eine Sonde gegen einen Oberflächenmarker ausgewählt werden, um den Immuncapture auf dem Chip zu ermöglichen. Derzeit können biologische Partikel bis zu einem Durchmesser von 200 nm mit SP-IRIS charakterisiert werden. Es gibt keine untere Durchmessergrenze, wenn die Fluoreszenzauslesung verwendet wird, da die SP-IRIS eine Empfindlichkeit von einer einzelnen Detektionssonde hat. Um die Genauigkeit und Reproduzierbarkeit von SP-IRIS-Studien zu gewährleisten und die Integration mit den anderen EV-Daten zu erleichtern, schlagen wir die Verwendung der EV Track-Plattform19 vor. Das Hochladen von Gesamtdaten ist nicht praktikabel (Gigabyte) und nur für Benutzer der ExoView-Software zugänglich. Wir schlagen daher vor, dass für jede abgefragte Probe und für jeden Teil einer Veröffentlichung die Datei mit der gefilterten Partikelliste, die einfach mit der Heatmap exportiert werden kann, in EV Track hochgeladen wird. Diese Datei enthält die vollständigen Partikeldaten und Cutoff-Einstellungen, anhand derer jede Zahl oder Frage überprüft oder beantwortet werden kann.
Zusammenfassend lässt sich sagen, dass SP-IRIS eine effektive und effiziente Methode zur Charakterisierung von EVs bietet, die eine einfache Möglichkeit bietet, routinemäßige Messungen der Einzel-EV-Größe und der Biomarker-Anzeige durchzuführen. Diese neu verfügbaren Daten können verborgene Details über die Stöchiometrie der EV-Belastung und die direkte Erkennung seltener Subpopulationen enthüllen und den Weg für weitere Fortschritte in unserem Verständnis der wichtigen Rolle von EVs bei Gesundheit und Krankheit ebnen.
Offenlegungen
Clayton Deighan und George Daaboul sind Mitarbeiter und Aktionäre von NanoView Biosciences Inc.
Danksagungen
Diese Arbeit wurde zum Teil vom University of Kansas School of Medicine Research Equipment and Resource Procurement Award Program gesponsert. PCG, LKC, FD und AR wurden mit Mitteln aus dem NIA R21 AG066488-01 unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| 10-cm sterile Petri dish | Fisher | FB0875712 | |
| 15mL sterile tube | n/a | various | |
| 24-well cell culture plate, flat bottom | Fisher | 08-772-1 | |
| Blocking Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chipfiles | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chips | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chuck | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 250 ml | Fisher | 09-761-4 | |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 500 ml | Fisher | 09-761-10 | |
| Deionized (DI) water | Fisher | LC267404 | |
| EMS style tweezers with Carbon Fiber tips | Fisher | 50-193-0842 | |
| ExoView Human Tetraspanin Kit | NanoView Biosciences | EV-TETRA-C | Capture for hCD81, hCD9, hCD63, IgG Control + stains for hEV-A (hEV-CD63-647, hEV-CD81-555, hEV-CD9-488) 16 Chips per kit |
| ExoView R100 Imager | NanoView Biosciences | EV-R100 | Interferometric microscope including high specification camera including 3 color fluorescence and label free sizing and counting extracellular vesicles |
| Fluorescently labled huma CD9 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD63 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD81 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Incubation Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Orbital shaker or microplate shaker with digital settings capable of shaking at 500 rpm | n/a | various | |
| Plate Seal | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution A | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution B | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution C | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution D | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Square/flat tip tweezer | Fisher | 50-239-62 | |
| Straight strong point Boley style tweezers | Fisher | 16-100-124 | |
| Thermo Scientific Adhesive PCR Plate Seals | Fisher | AB-0558 |
Referenzen
- Maas, S. L. N., Breakefield, X. O., Weaver, A. M. Extracellular vesicles: Unique intercellular delivery vehicles. Trends in Cell Biology. 27 (3), 172-188 (2017).
- Shah, R., Patel, T., Freedman, J. E. Circulating extracellular vesicles in human disease. The New England Journal of Medicine. 379 (10), 958-966 (2018).
- Deng, F., Miller, J. A review on protein markers of exosome from different bio-resources and the antibodies used for characterization. Journal of Histotechnology. 42 (4), 226-239 (2019).
- Cohen, O., et al. ’Golden’ exosomes as delivery vehicles to target tumors and overcome intratumoral barriers: in vivo tracking in a model for head and neck cancer. Biomaterials Science. 9 (6), 2103-2114 (2021).
- Han, Y., et al. Overview and update on methods for cargo loading into extracellular vesicles. Processes (Basel). 9 (2), 356 (2021).
- Lee, M., Im, W., Kim, M. Exosomes as a potential messenger unit during heterochronic parabiosis for amelioration of Huntington's disease. Neurobiology of Disease. 155, 105374 (2021).
- Sun, B., et al. Characterization and biomarker analyses of post-COVID-19 complications and neurological manifestations. Cells. 10 (2), 386 (2021).
- Jiang, L., Gu, Y., Du, Y., Liu, J. Exosomes: Diagnostic biomarkers and therapeutic delivery vehicles for cancer. Molecular Pharmaceutics. 16 (8), 3333-3349 (2019).
- Bachurski, D., et al. Extracellular vesicle measurements with nanoparticle tracking analysis - An accuracy and repeatability comparison between NanoSight NS300 and ZetaView. Journal of Extracellular Vesicles. 8 (1), 1596016 (2019).
- Carmicheal, J., et al. Label-free characterization of exosome via surface enhanced Raman spectroscopy for the early detection of pancreatic cancer. Nanomedicine. 16, 88-96 (2019).
- Greening, D. W., Xu, R., Ji, H., Tauro, B. J., Simpson, R. J. A protocol for exosome isolation and characterization: evaluation of ultracentrifugation, density-gradient separation, and immunoaffinity capture methods. Methods in Molecular Biology. 1295, 179-209 (2015).
- Wu, Y., Deng, W., Klinke, D. J. Exosomes: improved methods to characterize their morphology, RNA content, and surface protein biomarkers. The Analyst. 140 (19), 6631-6642 (2015).
- van de Vlekkert, D., Qiu, X., Annunziata, I., d'Azzo, A. Isolation and characterization of exosomes from skeletal muscle fibroblasts. Journal of Visualized Experiments: JoVE. (159), (2020).
- Ayala-Mar, S., Donoso-Quezada, J., Gallo-Villanueva, R. C., Perez-Gonzalez, V. H., Gonzalez-Valdez, J. Recent advances and challenges in the recovery and purification of cellular exosomes. Electrophoresis. 40 (23-24), 3036-3049 (2019).
- Daaboul, G. G., et al. Digital detection of exosomes by interferometric imaging. Scientific Reports. 6, 37246 (2016).
- Pohler, K. G., et al. Circulating microRNA as candidates for early embryonic viability in cattle. NMolecular Reproduction and Development. 84 (8), 731-743 (2017).
- NanoView Biosciences. ExoView R100 User Guide. v240.4. NanoView Biosciences. , 202 (2021).
- Daaboul, G. G., et al. Enhanced light microscopy visualization of virus particles from Zika virus to filamentous ebolaviruses. PLoS One. 12, 0179728 (2017).
- ET Consortium. EV-TRACK: transparent reporting and centralizing knowledge in extracellular vesicle research. Nature Methods. 14, 228-232 (2017).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten
Wir verwenden Cookies, um Ihre Erfahrung auf unserer Website zu verbessern.
Indem Sie unsere Website weiterhin nutzen oder auf „Weiter“ klicken, stimmen Sie zu, unsere Cookies zu akzeptieren.