Method Article
单个细胞外囊泡的单粒子干涉反射成像表征和种群动态
摘要
该协议提出了单粒子干涉反射成像,专为细胞外囊泡 (EV) 大小、EV 计数、EV 表型和 EV 生物标志物共定位的多级和综合测量而设计。
摘要
细胞外囊泡 (EV) 是纳米大小的囊泡,具有脂质双层,由大多数细胞分泌。EV 携带多种不同的生物分子,包括蛋白质、脂质、DNA 和 RNA,并被假定为促进不同组织和器官中的细胞间通讯。最近,电动汽车作为诊断的生物标志物和各种疾病的治疗剂引起了广泛关注。已经开发了许多用于 EV 表征的方法。但是,当前的 EV 分析方法都有不同的局限性。因此,随着这一尖端研究领域的成熟,开发高效的 EV 分离和表征方法仍然是其关键步骤之一。在这里,我们提供了一个详细的协议,概述了单粒子干涉反射成像传感器 (SP-IRIS),作为一种能够检测和表征来自未纯化生物来源的 EV 和通过其他方法纯化的 EV 的方法。这种先进的技术可用于多层次和全面的测量,以分析 EV 大小、EV 计数、EV 表型和生物标志物共定位。
引言
细胞外囊泡 (EV) 是纳米大小的细胞来源膜囊泡,可以从许多生物体液中分离出来,包括血液、母乳、唾液、尿液、胆汁、胰液以及脑脊液和腹膜液。EV 的衍生通过三种主要机制发生:细胞凋亡、通过多泡体与质膜融合释放以及质膜起泡1。供体细胞成分向邻近或远处细胞和组织的 EV 转移的证据表明,这些膜封闭的包装可能在旁分泌以及长距离或内分泌信号级联反应中发挥重要作用 1,2,3。由于 EV 可以提供细胞表型的快照,因此它们用作治疗各种疾病的诊断和治疗工具的潜力已成为一个活跃的研究领域 4,5,6,7,8。
已经开发了许多针对 EV 表征的方法 9,10,11,12,13。这些方法中的大多数都提供了有关主要批量 EV 种群的独特且有价值的信息。虽然这些技术的子集可以提供有关单个 EV 内部或上物质的详细信息,但在单个 EV 级别表征 EV 可能存在局限性。例如,免疫电子显微镜可用于了解单个 EV 及其组成,但该技术的通量低,用于描述种群动态的能力受到严重限制,并且需要大量方法开发14。
最近, 通过 ExoView 平台开发并商业化了单粒子干涉反射成像传感器 (SP-IRIS) 技术,使用常规和简单的自动数据收集方法开启了单个 EV 表征的大门。该技术的核心是芯片,即 1 cm x 1 cm Si/SiO2 双层,能够对单个生物纳米颗粒进行干涉测量。该芯片使用单个功能化抗体点的微阵列进行耕作,可对多达六种不同的捕获类型进行多重检测。标准芯片包括常见的跨膜四蛋白标记物(CD81、CD63 和 CD9),用于在孵育步骤中捕获,用户可以添加额外的定制捕获点,以分离与跨膜四蛋白分离的不同 EV 群体。孵育步骤后,每个捕获点都与其结合了许多 EV,这些 EV 表达相应的标记。然后,可以在阅读器中简单地清洗、干燥和扫描这些捕获的 EV,以量化 50-200 nm 之间与捕获点结合的囊泡的大小, 从而通过 SP-IRIS15 给出数字加权大小分布。该系统还提供三个荧光检测通道,用于对捕获的 EV 进行免疫标记,并提供平均荧光强度(不受大小(如 SP-IRIS 测量)和每个荧光染色剂的共定位方面。这允许用户根据每个 EV 的 4 个不同生物标志物(捕获加上 3 个免疫荧光标记)来定义单个 EV 的群体。该系统不仅可以使用免疫荧光测量表面蛋白,因为可选的货物方案允许用户探测捕获的 EV 的内部蛋白质和跨膜表面标志物的管腔表位,并允许用户检查 EV 膜的完整性。在本文中,我们提供了一个详细的协议,概述了获得有关 EV 大小和数量的一致数据所需的步骤,在大量 EV 群体的单个 EV 水平上最多有四种不同的生物标志物。该技术可用于未加工的生物液体和使用多种技术分离的 EV,例如超速离心、超滤、沉淀剂、免疫亲和捕获、微流体和尺寸排阻色谱。
下面描述的方案使用来自 HEK 293 细胞培养基和小鼠血清的细胞外囊泡 (EV),使用既定的分离方法16。该方案已应用于许多其他生物体液、细胞培养基和从生物体液中分离的纯化细胞外囊泡。该协议分为为期两天的程序,其工作流程如图 1 所示。

图 1:检测工作流程。 用于在大小和计数、大小计数和表面染色以及大小计数和货物染色之间选择要为样品完成的分析类型的分析工作流程。 请单击此处查看此图的较大版本。
研究方案
根据堪萨斯大学医学中心 (KUMC) 经批准的机构动物护理和使用委员会 (IACUC) 方案从小鼠中收集血清样本。在这些实验中使用这些生物样品也得到了 KUMC 的批准。
1. 样品制备(第 1 天)
- 使用纳米颗粒跟踪或等效技术确定 EV 浓度。
- 用孵育液将样品稀释至浓度为 5 x 107-5 x 108 EVs/mL;至少需要 50 μL。
注:对于 EV 浓度未知的样品,可使用 1 μg/mL 的总蛋白作为替代措施。如果预计样品浓度较低,请在上样前在孵育溶液中至少进行 1:1 稀释。 - 将 24 孔板放在平坦的表面上,避免振动和粗暴运动。
注意:为了增强对比度,可以在印版下方放置一张白纸。 - 向井周围的区域加水(图 2)。

图 2:24 孔板布局。 显示了分装 ddH2O 的位置(添加蓝色染料仅用于可视化目的)和将容纳芯片的孔。 请单击此处查看此图的较大版本。
2. 准备和预扫描芯片
- 从 4-8 °C 冰箱中取出含有芯片的密封 48 孔板,让它们达到室温(~15 分钟),然后再打开密封。
注意: 这对于避免芯片上出现冷凝水至关重要,这可能会损坏斑点。如果在从包装中取出芯片时观察到表面出现冷凝,请等待更长时间,然后再取出其他芯片。 - 继续执行步骤 8 以检索卡盘以准备预扫描运行(图 3)。
注:在与样品孵育之前,预扫描数据将用于识别在捕获点上可检测到的任何颗粒,以便在分析步骤中将其去除。

图 3:用于将芯片装入机床的卡盘图像。 请单击此处查看此图的较大版本。
- 使用镊子从 48 孔板中取出所需数量的芯片(每个样品一个芯片),然后将芯片直接加载到卡盘中进行预扫描运行。按照步骤 8 中的说明加载和预扫描所有用于实验的芯片后,请继续执行步骤 2.4。
注:处理芯片时,请确保不要触摸中心的方块,因为抗体捕获点位于该区域,如果被镊子触摸会损坏(图 4)。

图 4:芯片和正确的芯片处理。 (A) 黄色虚线表示斑点抗体的位置或芯片的功能侧。芯片 ID 位于行 (“58”) 下方。图还显示了正确的处理。(B) 证明芯片处理不当。(C) 芯片的非功能侧。 请单击此处查看此图的较大版本。
- 将每个预扫描的芯片和功能化表面朝上放入预先准备好的 24 孔板中。功能化的表面很容易通过不同的编号、对齐网格和中间的三个黑框来识别。非功能化的一面是平坦的银色均匀表面。
- 使用细尖镊子确保每个芯片都在孔内居中(图 5)。
注:将芯片在孔内居中至关重要,因为如果芯片接触孔壁,样品在加载后可能会被吸走。

图 5:在孔中正确放置芯片的演示。 (A) 芯片应设置在孔的中间,没有角落接触孔的侧面。(B) 描述芯片放置不当,即边角接触孔的侧面。 请单击此处查看此图的较大版本。
3. 芯片和样品的加载和孵育
- 将 35 μL 制备的样品(来自步骤 1)移液到芯片上。
注意:小心不要添加气泡或用移液器吸头接触芯片,因为这会阻止样品在芯片上均匀分布或损坏芯片上的抗体点。样品应分布在整个芯片表面。如果样品在加载过程中从芯片上吸出,则向孔中加入体积不超过 250 μL 的额外样品,或添加高达 250 μL 的孵育溶液,并注意该样品稀释因子的变化。 - 使用试剂盒中包含的板膜密封板,以防止样品蒸发。
- 将样品/芯片在室温下在密封板中无振动或移动的区域孵育过夜(~16 小时)。
注意:过夜孵育后,用户将为其研究问题选择合适的第 2 天选项。如果只需要 EV 大小和计数,请继续执行第 4 步(第 2 天)。如果需要多个表面标志物分析,请跳至第 5 步(第 2 天)。
4. 确定 EV 大小和数量(第 2 天)
- 在每个含有芯片的孔的一侧加入 1 mL 溶液 A,注意不要直接将溶液添加到芯片上或用移液器吸头刮擦芯片。
- 将板放在轨道振荡器上,在室温下以 ~500 rpm 旋转 3 分钟。
注意:如果木片在板上发出嘎嘎声,请立即降低速度,使液体旋转但没有嘎嘎声。 - 取出 750 μL 液体。在除液过程中避免倾斜板,以防止芯片意外干燥。
- 使用步骤 4.1 中描述的技术加入 750 μL 溶液 B,并在室温下以 ~500 rpm 摇动 3 分钟。
- 再重复步骤 4.3-4.4 两次,用溶液 B 总共洗涤 3 次。
- 在最后一次洗涤结束时,去除 750 μL 溶液,在孔中留下 250 μL 溶液 B。
- 加入 750 μL 双蒸水 (ddH2O),并在室温下以 ~500 rpm 摇动 3 分钟。
- 用 50 mL ddH2O 填充 10 cm 培养皿,并使用镊子一次将一个芯片从孔中转移到培养皿中。
注意: 注意水平转移芯片并确保其不会干燥。在更换 ddH2O 之前,最多可以清洗 8 个芯片。 - 在 ddH2O 中,使用镊子握住芯片的边缘,并在培养皿中旋转 3 圈以去除碎屑。
- 将芯片以 45° 角从水中取出,并将芯片放在吸水纸上,芯片 ID 朝上(图 6)。
注意:条带现在可以读取了。跳至步骤 8 以在 SP-IRIS 读取器上进行扫描。

图 6:以 45° 角从 ddH2O 水中去除芯片的正确方法。 (A) 顶部视图和 (B) 侧面视图,展示了去除芯片的角度。 请单击此处查看此图的较大版本。
5. 抗体溶液的制备(第 2 天)
- 将每个芯片 300 μL 封闭溶液添加到适当大小的试管 (0.5 至 3 mL) 中。
- 每 300 μL 封闭液添加 0.6 μL 抗体。轻敲试管并快速旋转,轻轻混合。
注:相应的试剂盒中包含针对人 CD9、CD81 和 CD63 或小鼠 CD9、CD81 和 CD63 的抗体。如果需要其他荧光偶联抗体,则使用 0.1 至 10 μg/mL 范围内的典型滴定来确定最佳染色浓度,并采用恒定的样品上样量,就像流式细胞术一样。
6. 通过免疫荧光染色确定 EV 大小、数量和表型
- 在每个含有芯片的孔的一侧加入 1 mL 溶液 A,注意不要直接将溶液添加到芯片上或用移液器吸头刮擦芯片。
- 将板放在轨道振荡器上,在室温下以 ~500 rpm 旋转 3 分钟。
注意:如果木片在板上发出嘎嘎声,请立即降低速度,使液体旋转但没有嘎嘎声。 - 取出 750 μL 液体。在除液过程中避免倾斜板,以防止芯片意外干燥。
- 使用步骤 6.1 中描述的技术加入 750 μL 溶液 A,并在室温下以 ~500 rpm 振荡 3 分钟。
- 重复步骤 6.3 和 6.4 两次,用溶液 A 总共洗涤 3 次。
- 在最后一次洗涤结束时,去除 750 μL 溶液,在孔中留下 250 μL 溶液 A。
注:如果用户需要货物染色,此时请继续执行步骤 7。如果没有,请继续执行步骤 6.7 - 向孔中的每个芯片中加入 250 μL 抗体溶液(步骤 5)。用箔纸盖住板以避光,并在室温下在轨道振荡器上摇动 1 小时。
- 加入 500 μL 溶液 A,使每孔总体积为 ~1,000 μL。
- 立即取出 750 μL 并加入 750 μL 溶液 A,并在室温下在轨道摇床上以 ~500 rpm 振荡 3 分钟。
- 取出 750 μL 溶液,加入 750 μL 溶液 B,在室温下以 ~500 rpm 振荡 3 分钟。
- 再重复步骤 6.10 两次,总共洗涤 3 次。
- 加入 750 μL 双蒸水 (ddH2O),并在室温下以 ~500 rpm 摇动 3 分钟。
- 用 50 mL ddH2O 填充 10 cm 培养皿,并使用镊子将一个芯片从孔中转移到培养皿中。
注意: 注意水平转移芯片并确保其不会干燥。在更换为新鲜的 ddH2O 之前,最多可以清洗 8 个芯片。 - 在 ddH2O 中,使用镊子握住芯片的边缘,并在培养皿中旋转 3 圈以去除碎屑。
- 以 45° 角从水中取出芯片,并将芯片放在吸水纸上,芯片 ID 朝上(图 6)。
注意:条带现在可以读取了。跳至步骤 8(数据收集),了解有关如何设置色标以在读取器上扫描的说明。
7. 可选货物染色
注意:该协议允许同时标记内部和表面标记物。
- 每个孔中剩余 250 μL 溶液 A 时,向每个孔中加入 250 μL 溶液 C。
- 立即放在轨道振荡器上,并设置为 ~200 rpm 精确 10 分钟。
注意:在此步骤和下面的步骤 7.8 中,时间和较慢的振荡速度都至关重要;确保速度仅在井中缓慢旋转并使用计时器。 - 与溶液 C 孵育 10 分钟后,向每个孔中加入 500 μL 溶液 A。
- 立即取出 750 μL 并加入 750 μL 溶液 A,并在室温下在轨道摇床上以 ~500 rpm 振荡 3 分钟。
- 再重复步骤 7.4 两次,总共洗涤 3 次。
- 在最后一次洗涤结束时,去除 750 μL 溶液,将 ~250 μL 溶液留在带有芯片的孔中。
- 向每个孔中加入 250 μL 溶液 D。
- 立即放在设置为 ~200 rpm 的轨道振荡器上精确 10 分钟。
- 与溶液 C 孵育 10 分钟后,向每个孔中加入 500 μL 溶液 A。
- 立即取出 750 μL 并加入 750 μL 溶液 A,并在室温下在轨道摇床上以 ~500 rpm 振荡 3 分钟。
- 重复步骤 7.10 两次,总共洗涤 3 次。
- 最后一次洗涤后取出 750 μL 溶液,然后返回上述步骤 6.7 进行染色并完成检测方案。
8. 数据收集
注意:使用 ExoView R100 从芯片收集数据的过程是自动化的,不需要用户输入。详细说明可以在用户指南和相应的视频中找到,用于加载芯片载体或“卡盘”和数据采集17。
- 使用仪器背面的两个电源开关打开 EV 表征平台的电源,然后双击桌面图标启动扫描仪软件。
注意:当扫描仪软件启动时,阅读器会自动设置到主屏幕,然后提示用户“打开门加载芯片”。 - 提起银色把手,打开读卡器前部的门。这将弹出载物台,允许用户访问卡盘并在软件中设置新的扫描。
- 通过单击 Save 文件夹并选择要保存数据的所需位置,确定用户希望保存数据的位置。
注意:将保存位置命名为有洞察力的名称(如实验名称)通常很有用,然后创建两个子文件夹,一个用于扫描前,一个用于扫描后。这使得在分析过程中查找和匹配数据变得更加容易。 - 要扫描芯片,请在控制计算机上找到芯片文件。
注:芯片文件是正在使用的芯片上抗体点布局的映射,允许扫描仪知道在每个芯片上扫描的位置。在每个套件中,都有一个 USB 密钥,其中包含用于芯片的芯片文件。收到套件后,应将这些材料保存到控制计算机上的某个位置,以便用户可以可靠地找到它们。 - 将芯片加载到卡盘上,芯片上的编号面向卡盘上的手柄,然后从计算机的芯片下拉菜单中,从芯片文件列表中选择每个芯片,并将其放置在扫描仪软件中虚拟卡盘中的适当匹配位置。完成后,将出现一个屏幕提示,要求用户将 Chuck 放在舞台上。
- 将加载好的卡盘放在载物台上;卡盘上的磁性对准会自动将其移动到舞台上的正确位置,然后单击 将卡盘放在舞台上 旁边的 OK 。然后,扫描程序将开始自动数据收集例程。
注意:一旦软件将每个芯片的扫描状态报告为成功,就可以对数据进行分析。
9. 数据分析
- 双击控制 PC 桌面上的 ExoView Analyzer 。软件启动后,单击 Prescan Data 按钮,然后在第 8.3 节中选择预扫描数据集的文件夹位置。
- 单击“ Postscan 数据 ”按钮,然后在第 8.3 节中选择 Postscan 数据集的文件夹位置。
注意:如果在两个文件夹中都正确检测到数据,则至少对于某些芯片,软件将在 Chipfile location(芯片文件位置)按钮旁边显示检测到的 “X” 个芯片。正确保存后,检测到的芯片应与扫描的芯片数量相匹配。 - 单击数据加载区域底部的 Next 。
- 在 meta table 下拉菜单中,每个条状标签都有一个单元格。通过单击相应的框并输入信息,输入样品名称、稀释因子和每个检测通道中正在染色的标记物;完成后,再次单击 Next 。
注意:此元数据将与数据一起保存。 - 通过单击左上角 QC 按钮下的 Disable 选项卡进行瓶坯质量控制 (QC)。
注意:此功能允许关闭特定的探针和芯片进行分析。该软件将提供有关数据的两种不同警告,一种是高计数,可能表明特定捕获已饱和,另一种是高变异系数 (CV),用于识别捕获类型的一个重复何时与其他重复不同。- 点击高 CV 的 Warning Spot ,检查软件调出的点,看看是否有明显的物理损伤;确定损坏后,单击 Spot 编号 以在分析中关闭该点。
- 重复此作,直到评估完所有警告,然后单击 Next(下一步)。
- 通过单击左上角 QC 按钮下 Disable 选项卡旁边的 Cutoff 选项卡进行预制截止分析。
注意:这将在图表中显示控制点荧光响应,并为用户提供两个设置,每个颜色通道(红/绿/蓝)的最小值和最大值。为每个荧光检测通道设置截止值是唯一需要进行的数据调整,并且相对简单。重要的是,选择的最终临界值在整个实验中应保持一致,并且如前所述,应符合经验法则,红色和绿色为 300-400 a.u.,蓝色为 400-700 a.u.;该软件也为用户提供了这些范围的颜色编码指南。- 单击 Green Color 选项卡以加载该通道的数据并显示当前截止值。检查同种型阴性对照点可能会看到荧光强度非常低的检测事件;在这种情况下,用户通常希望将绿色设置为 300-400 A.U. 之间。
- 增加每个检测通道的最小值,直到数据显示下的“Avg % Inc”(截止值上方包括的控制点上检测到的颗粒的平均百分比)没有警告,如该单元格中的红色或黄色突出显示所示。
注:最大值通常不需要从默认值进行调整,但可以降低该值以过滤掉明亮颗粒或将荧光检测限制在更窄的范围内。 - 点击 下一步 按钮。
- 增加每个检测通道的最小值,直到数据显示下的“Avg % Inc”(截止值上方包括的控制点上检测到的颗粒的平均百分比)没有警告,如该单元格中的红色或黄色突出显示所示。
- 单击 Red Color 选项卡以加载该通道的数据并显示当前截止值。同样,检查同种型阴性对照点,可能会看到荧光强度非常低的检测事件;在这种情况下,用户通常希望将红色设置为 300-400 A.U。按照步骤 9.6.1.1-9.6.1.3 中记下的相同过程进行作。
- 单击 Blue Color 选项卡以加载该通道的数据并显示当前截止值。同样,检查同种型阴性对照点,按照步骤 9.6.1.1-9.6.1.3 中记下的相同程序进行作。
注:蓝色通道的独特之处在于,斑点中的抗体在足够的水平下具有自发荧光,即使对于空白芯片,也需要略高的 400-700 a.u. 截止值。
- 单击 Green Color 选项卡以加载该通道的数据并显示当前截止值。检查同种型阴性对照点可能会看到荧光强度非常低的检测事件;在这种情况下,用户通常希望将绿色设置为 300-400 A.U. 之间。
- 完成截止分析后,单击 下一步 ,将出现热图,其中将概述整个实验中的粒子结合。用户可以使用多种可视化工具来绘制数据。这些绘图和关联的原始数据可以通过单击 Add Plot Report 按钮添加到报告中。
注:热图是默认数据显示,显示特定检测的所有样本、所有捕获视图。默认显示是量化绑定到每个捕获点的唯一 EV 的总数。 - 单击 Export Report 按钮并选择报告的保存位置。
注意:报告将在浏览器中打开。找到报告附带的 Filtered Particle List 电子表格文件;这是提交给 EVTRACK 的信息。 - 使用屏幕顶部下拉菜单中 Samples (样本) 和 Capture types (捕获类型) 的不同选择继续添加报告。通过选择左上角的通道检测按钮来控制显示的数据。通过单击每种颜色按钮来设置特定的表型/样本选择/捕获通道,以关闭(灰色)或打开(彩色)检测通道。
- 单击 Add Plot to Report 按钮,将所需的绘图聚合到报告中。
- 单击 Export Report 并定义最终报告的保存位置。
结果
图 7 (左图)显示了 EV 的三色合成图像,该图像来源于 HEK293 条件培养基,该培养基与芯片上的 CD63 点结合,并在以下通道中分别以绿色、红色和蓝色对 CD81、CD63 和 CD9 进行染色。 图 7 (右上图)是一个放大的图像,显示每个捕获的 EV 都可以在每个通道中显示一种或多种颜色的共定位,这些颜色的强度各不相同。捕获的 EV 的染色差异代表三种四跨膜蛋白在单个囊泡上表达的异质性,可以通过数据分析软件进行量化。 图 7 (右下图)显示了 50 nm 以上的 EV 的尺寸直方图。尺寸直方图显示,在 CD63 光斑上捕获的 EV 具有 50 nm 的模式大小。绑定到点图像的每个 EV 的颜色共定位、强度和大小代表表型数据,它构成了测量中的基本数据。

图 7:完整斑点的三色图像显示了一个完整的 CD63 捕获点,其中 HEK293 衍生的 EV 结合并针对抗 CD9-CF488、抗 CD81-CF555、抗 CD63-CF647 进行了染色。 缩放图像描述了使用荧光共定位对单个 EV 表型的数字检测。此芯片的 SP-IRIS 衍生尺寸分布如右下角所示。从 HEK293T 细胞的条件培养基中收集 EV。将细胞培养至 80% 汇合度,然后用 PBS 洗涤,并在含有 EV 耗尽的 FBS (10% vol/vol) 的培养基中培养 48 小时。然后将在 48 小时结束时收集的条件培养基以 2,500 x g 离心以去除细胞碎片,并分析剩余的含有 EV 的上清液。 请单击此处查看此图的较大版本。
图 8 显示了用户可能遇到的同种型对照反应类型的两个示例的三色合成图像和强度直方图。顶部面板(示例示例 1,EX1)显示了标称作,同型对照点没有结合,荧光强度大于蓝色 (>500) 和 (>300 a.u.) 的红色和绿色,允许用户设置低截止值。下图(样品非特异性 1,NS1)显示了 EV 何时与同种型对照结合并导致高强度染色,如直方图所示。在这种情况下,标记特异性点的数据不可靠,因为它覆盖的强度范围与控制点上的 EV 相同,因此活动点上的 EV 被专门招募在那里的推论不再成立。当这种现象发生时,它通常与样品的状态有关。
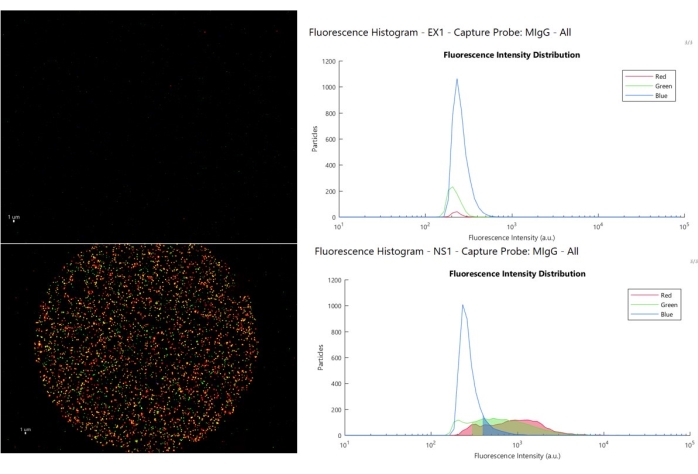
图 8:截止分析。 左上图显示了标称对照点和相关的荧光直方图(右上),其中高于截断值的颗粒显示在阴影区域中,左下角图像显示了非特异性 EV 与对照点结合的示例,荧光直方图中的阴影区域是高于截留点的区域,与捕获点上的阳性 EV 处于同一范围内。 请单击此处查看此图的较大版本。
图 9 表示人血清来源的 EV 的单 EV 蛋白共定位。使用抗 CD63、CD9、CD81 和 mlgG 捕获抗体捕获 EV,并对 CD81 (绿色) 、 HSP72 (红色) 和 CD9 (蓝色) 进行染色。 图 9 显示 CD81 是人血清衍生 EV 中积累最多的标志物。而 CD63 水平与 CD81 和 CD9 相比最低。在抗 CD63、CD9 和 CD81 抗体捕获的 EV 中,CD63 捕获的 EV 中的 HSP72 比率较高,而抗 CD9 和 CD81 抗体捕获的 EV 之间的 HSP72 比率相似。这种对 HSP72 和 CD63 在所有也携带四跨膜蛋白标志物的非 HSP72 阳性 EV 中的特异性定位的检测证明了单粒子干涉测量和荧光的独特数据收集能力。

图 9:单 EV 蛋白共定位。 使用抗 CD63、CD9、CD81 和 mlgG(同种型)捕获抗体捕获来自人的 EV,并对 CD81(绿色)、HSP72(红色)和 CD9(蓝色)进行染色。使用荧光模式对 EV 群体进行成像。 请单击此处查看此图的较大版本。
讨论
目前的 EV 表征方法主要依赖于纯化的 EV,这受到当前 EV 纯化方法实验局限性的限制 9,10,11,12,13。单粒子干涉反射成像 (SP-IRIS) 是一种有效的技术,可以消除样品分析所需的纯化步骤,从而节省时间并降低与典型 EV 工作流程相关的成本。所需的样品起始量通常非常小,因此样品的其余部分可用于其他分析方法。
此外,以抗原特异性方式测量 EV 的能力对于基础 EV 研究以及诊断和治疗应用中的罕见事件检测非常重要。然而,相对于其他循环 EV 和污染物,疾病特异性 EV 在血液中可能以低浓度存在 2,11,12。该方案可以修改为加入其他抗体,使用分别在 488 nm、555 nm 或 647 nm 激发的荧光标记抗体来检测这些亚群。在这种情况下,应开发阳性和阴性生物对照,据此,可以使用滴定系列来优化目标标记物的检测。在这种情况下,应该注意的是,SP-IRIS 采用的正选择方法可能对本实验有害,因为感兴趣的标记物不能与跨膜四蛋白下拉强烈共定位,从而使检测变得困难。如果出现这种情况,使用感兴趣的标记物在芯片上捕获 EV 是有利的,从而克服了 tetraspanin 斑点上的正选择偏差。SP-IRIS 成像的广义标记能力使得可以在特定捕获点上检测多达四个标记物,这需要讨论如何设计和解释 SP-IRIS 和适当控制对新标记物的检测。当使用同型对照点作为芯片的唯一对照时,我们实际上是在考虑在截留设置期间抗体是否存在非特异性样品结合直接与同型捕获点。如果我们在同种型对照和标记特异性点上观察到类似的共定位特征,则表明 EV 非特异性地粘附在对照上,应检查方案。选择良好的阳性和阴性对照样品可以帮助用户区分非特异性结合是来自芯片上的抗体还是荧光标记的一抗。如果怀疑 EV 与同型对照位点结合,则可以将阴性对照 EV 样品以固定浓度在芯片上孵育,并用目标抗体的正常染色浓度进行染色。应按照第 9 节数据收集步骤 9.6 中的说明执行截止设置。确认目标标记物的斑点上不存在结合。在负 EV 样品上建立的临界值可以应用于实验中的其余样品。只要有阳性对照可用,就进行滴定染色测试以确定使用其他抗体的最佳染色浓度是有用的。
SP-IRIS 对 EV 使用特异性检测的优势也可能给该技术带来局限性。第 3 节中的孵育步骤本质上是一种正向选择方法,它不提供样品中可能存在的颗粒的信息,但不显示与特定芯片上的捕获类型相对应的标记,因为它们在捕获后会被冲走,并且在分析过程中不会实现。由于所有 EV 上都不存在公认的通用标记,因此用户应将其数据作为测量群体进行审查,并根据总颗粒浓度的正交测量考虑捕获的颗粒数量,以确定 SP-IRIS 捕获数据是否代表特定样本中的所有 EV。
SP-IRIS 技术已应用于其他生物纳米颗粒(例如,病原病毒和病毒载体)18。为了将 SP-IRIS 应用于其他生物颗粒的检测,需要选择针对表面标志物的捕获抗体或探针,以便在芯片上进行免疫捕获。目前,可以使用 SP-IRIS 表征直径达 200 nm 的生物颗粒。如果使用荧光读数,则直径没有下限,因为 SP-IRIS 具有单个检测探针的灵敏度。为确保 SP-IRIS 研究的严谨性和可重复性并促进与其他 EV 数据的整合,我们建议使用 EV Track 平台19。上传总数据是不切实际的(千兆字节),只有使用 ExoView 软件的人才能访问。因此,我们建议对于每个被询问的样本和出版物的一部分,将可以通过热图轻松导出的过滤粒子列表文件上传到 EV Track。此文件具有完整的粒子数据和截止设置,从中可以验证或回答任何图形或问题。
总之,SP-IRIS 提供了一种有效的 EV 表征方法,提供了一种简单的方法来对单个 EV 尺寸和生物标志物显示进行常规测量。这些新可用的数据可以揭示 EV 负载化学计量的隐藏细节,直接检测稀有亚群,并有助于进一步推进我们对 EV 在健康和疾病中重要作用的理解。
披露声明
Clayton Deighan 和 George Daaboul 是 NanoView Biosciences Inc. 的员工和股东。
致谢
这项工作部分由堪萨斯大学医学院研究设备和资源采购奖励计划赞助。PCG、LKC、FD 和 AR 得到了 NIA R21 AG066488-01 的资金支持。
材料
| Name | Company | Catalog Number | Comments |
| 10-cm sterile Petri dish | Fisher | FB0875712 | |
| 15mL sterile tube | n/a | various | |
| 24-well cell culture plate, flat bottom | Fisher | 08-772-1 | |
| Blocking Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chipfiles | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chips | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chuck | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 250 ml | Fisher | 09-761-4 | |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 500 ml | Fisher | 09-761-10 | |
| Deionized (DI) water | Fisher | LC267404 | |
| EMS style tweezers with Carbon Fiber tips | Fisher | 50-193-0842 | |
| ExoView Human Tetraspanin Kit | NanoView Biosciences | EV-TETRA-C | Capture for hCD81, hCD9, hCD63, IgG Control + stains for hEV-A (hEV-CD63-647, hEV-CD81-555, hEV-CD9-488) 16 Chips per kit |
| ExoView R100 Imager | NanoView Biosciences | EV-R100 | Interferometric microscope including high specification camera including 3 color fluorescence and label free sizing and counting extracellular vesicles |
| Fluorescently labled huma CD9 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD63 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD81 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Incubation Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Orbital shaker or microplate shaker with digital settings capable of shaking at 500 rpm | n/a | various | |
| Plate Seal | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution A | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution B | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution C | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution D | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Square/flat tip tweezer | Fisher | 50-239-62 | |
| Straight strong point Boley style tweezers | Fisher | 16-100-124 | |
| Thermo Scientific Adhesive PCR Plate Seals | Fisher | AB-0558 |
参考文献
- Maas, S. L. N., Breakefield, X. O., Weaver, A. M. Extracellular vesicles: Unique intercellular delivery vehicles. Trends in Cell Biology. 27 (3), 172-188 (2017).
- Shah, R., Patel, T., Freedman, J. E. Circulating extracellular vesicles in human disease. The New England Journal of Medicine. 379 (10), 958-966 (2018).
- Deng, F., Miller, J. A review on protein markers of exosome from different bio-resources and the antibodies used for characterization. Journal of Histotechnology. 42 (4), 226-239 (2019).
- Cohen, O., et al. ’Golden’ exosomes as delivery vehicles to target tumors and overcome intratumoral barriers: in vivo tracking in a model for head and neck cancer. Biomaterials Science. 9 (6), 2103-2114 (2021).
- Han, Y., et al. Overview and update on methods for cargo loading into extracellular vesicles. Processes (Basel). 9 (2), 356 (2021).
- Lee, M., Im, W., Kim, M. Exosomes as a potential messenger unit during heterochronic parabiosis for amelioration of Huntington's disease. Neurobiology of Disease. 155, 105374 (2021).
- Sun, B., et al. Characterization and biomarker analyses of post-COVID-19 complications and neurological manifestations. Cells. 10 (2), 386 (2021).
- Jiang, L., Gu, Y., Du, Y., Liu, J. Exosomes: Diagnostic biomarkers and therapeutic delivery vehicles for cancer. Molecular Pharmaceutics. 16 (8), 3333-3349 (2019).
- Bachurski, D., et al. Extracellular vesicle measurements with nanoparticle tracking analysis - An accuracy and repeatability comparison between NanoSight NS300 and ZetaView. Journal of Extracellular Vesicles. 8 (1), 1596016 (2019).
- Carmicheal, J., et al. Label-free characterization of exosome via surface enhanced Raman spectroscopy for the early detection of pancreatic cancer. Nanomedicine. 16, 88-96 (2019).
- Greening, D. W., Xu, R., Ji, H., Tauro, B. J., Simpson, R. J. A protocol for exosome isolation and characterization: evaluation of ultracentrifugation, density-gradient separation, and immunoaffinity capture methods. Methods in Molecular Biology. 1295, 179-209 (2015).
- Wu, Y., Deng, W., Klinke, D. J. Exosomes: improved methods to characterize their morphology, RNA content, and surface protein biomarkers. The Analyst. 140 (19), 6631-6642 (2015).
- van de Vlekkert, D., Qiu, X., Annunziata, I., d'Azzo, A. Isolation and characterization of exosomes from skeletal muscle fibroblasts. Journal of Visualized Experiments: JoVE. (159), (2020).
- Ayala-Mar, S., Donoso-Quezada, J., Gallo-Villanueva, R. C., Perez-Gonzalez, V. H., Gonzalez-Valdez, J. Recent advances and challenges in the recovery and purification of cellular exosomes. Electrophoresis. 40 (23-24), 3036-3049 (2019).
- Daaboul, G. G., et al. Digital detection of exosomes by interferometric imaging. Scientific Reports. 6, 37246 (2016).
- Pohler, K. G., et al. Circulating microRNA as candidates for early embryonic viability in cattle. NMolecular Reproduction and Development. 84 (8), 731-743 (2017).
- NanoView Biosciences. ExoView R100 User Guide. v240.4. NanoView Biosciences. , 202 (2021).
- Daaboul, G. G., et al. Enhanced light microscopy visualization of virus particles from Zika virus to filamentous ebolaviruses. PLoS One. 12, 0179728 (2017).
- ET Consortium. EV-TRACK: transparent reporting and centralizing knowledge in extracellular vesicle research. Nature Methods. 14, 228-232 (2017).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可探索更多文章
This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。