Method Article
Imágenes interferométricas de reflectancia de una sola partícula: caracterización de vesículas extracelulares individuales y dinámica de poblaciones
En este artículo
Resumen
Este protocolo presenta imágenes de reflectancia interferométrica de una sola partícula que están diseñadas para las mediciones completas y multinivel del tamaño de las vesículas extracelulares (EV), el recuento de EV, el fenotipo de EV y la colocalización de biomarcadores EV.
Resumen
Las vesículas extracelulares (VE) son vesículas de tamaño nanométrico con una bicapa lipídica que son secretadas por la mayoría de las células. Los VE transportan una multitud de moléculas biológicas diferentes, incluidas proteínas, lípidos, ADN y ARN, y se postula que facilitan la comunicación de célula a célula en diversos tejidos y órganos. Recientemente, los VE han atraído una atención significativa como biomarcadores para el diagnóstico y agentes terapéuticos para diversas enfermedades. Se han desarrollado muchos métodos para la caracterización de vehículos eléctricos. Sin embargo, todos los métodos actuales para el análisis de EV tienen diferentes limitaciones. Por lo tanto, el desarrollo de métodos eficientes y efectivos para el aislamiento y la caracterización de vehículos eléctricos sigue siendo uno de los pasos cruciales para este campo de investigación de vanguardia a medida que madura. Aquí, proporcionamos un protocolo detallado que describe un sensor de imágenes de reflectancia interferométrica de una sola partícula (SP-IRIS), como un método que es capaz de detectar y caracterizar EV a partir de fuentes biológicas no purificadas y EV purificadas por otras metodologías. Esta técnica avanzada se puede utilizar para mediciones integrales y de varios niveles para el análisis del tamaño del EV, el recuento de EV, el fenotipo del EV y la colocalización de biomarcadores.
Introducción
Las vesículas extracelulares (VE) son vesículas de membrana de tamaño nanométrico de origen celular que se pueden aislar de numerosos fluidos biológicos, como sangre, leche materna, saliva, orina, bilis, jugo pancreático y fluidos cefalorraquídeos y peritoneales. La derivación de las VE se produce a través de tres mecanismos principales: apoptosis, liberación a través de la fusión de cuerpos multivesiculares con la membrana plasmática y formación de la membrana plasmática1. La evidencia de la transferencia EV de componentes de células donantes a células y tejidos vecinos o distantes sugiere que estos paquetes encerrados en membranas pueden desempeñar un papel importante en las cascadas de señalización paracrina, así como a larga distancia o endocrina 1,2,3. Debido a que los VE pueden proporcionar una instantánea del fenotipo de una célula, el potencial de su uso como herramientas diagnósticas y terapéuticas para el tratamiento de diversas enfermedades se ha convertido en un área activa de investigación 4,5,6,7,8.
Se han desarrollado muchos métodos dirigidos a la caracterización de VE 9,10,11,12,13. La mayoría de estos métodos proporcionan información única y valiosa sobre las poblaciones de vehículos eléctricos, principalmente a granel. Si bien un subconjunto de estas técnicas puede proporcionar detalles sobre las sustancias dentro o sobre los vehículos eléctricos individuales, puede haber limitaciones para caracterizar los vehículos eléctricos a nivel de un solo vehículo. Por ejemplo, la inmunomicroscopía electrónica se puede utilizar para comprender los VE individuales y su composición, pero esta técnica es de bajo rendimiento, su capacidad para ser utilizada para describir la dinámica de poblaciones y requiere el desarrollo de métodos significativos14.
Recientemente, el desarrollo y la comercialización de la técnica del sensor de imágenes de reflectancia interferométrica de una sola partícula (SP-IRIS), a través de la plataforma ExoView, ha abierto la caracterización individual de EV utilizando un método de recopilación de datos automatizado rutinario y sencillo. El núcleo de esta tecnología es el chip, una doble capa de Si/SiO2 de 1 cm x 1 cm, que permite la medición interferométrica de nanopartículas biológicas individuales. El chip está cultivado con un microarray de puntos de anticuerpos funcionalizados individuales, lo que permite la detección multiplexada de hasta seis tipos de captura diferentes. Los chips estándar incluyen los marcadores comunes de tetraspanina (CD81, CD63 y CD9) para la captura durante la etapa de incubación, y el usuario puede agregar puntos de captura personalizados adicionales para aislar distintas poblaciones de EV separadas de las tetraspaninas. Después de la etapa de incubación, cada punto de captura tiene unidos muchos EV que expresan el marcador correspondiente. Estos EV capturados se pueden lavar, secar y escanear simplemente en el lector para cuantificar el tamaño de las vesículas unidas al punto de captura entre 50 y 200 nm para dar una distribución de tamaño ponderada por número a través de SP-IRIS15. El sistema también ofrece tres canales de detección fluorescentes para el inmunomarcaje de los EV capturados, y proporciona tanto la intensidad fluorescente media, que no está limitada por el tamaño, como las mediciones de SP-IRIS, como los aspectos de colocalización para cada tinción fluorescente. Esto permite al usuario definir poblaciones de VE individuales en función de la visualización de cuatro biomarcadores diferentes por VE (captura más tres marcadores inmunofluorescentes). El sistema puede ir más allá de la medición de proteínas de superficie con inmunofluorescencia, ya que un protocolo de carga opcional permite al usuario sondear las proteínas internas de los EV capturados y los epítopos luminales de los marcadores de superficie que abarcan la membrana, así como permite al usuario comprobar la integridad de la membrana EV. En este artículo, proporcionamos un protocolo detallado que describe los pasos necesarios para obtener datos consistentes sobre el tamaño y el número de vehículos eléctricos, con hasta cuatro biomarcadores diferentes en un solo nivel de vehículo en grandes poblaciones de vehículos eléctricos. Esta técnica se puede utilizar tanto en fluidos biológicos no procesados como en VE aislados utilizando cualquier número de técnicas, como ultracentrifugación, ultrafiltración, agentes precipitantes, captura de inmunoafinidad, microfluídica y cromatografía de exclusión por tamaño.
El protocolo que se describe a continuación utiliza vesículas extracelulares (VE) derivadas de medios de cultivo celular HEK 293 y del suero de ratón utilizando un método de aislamiento establecido16. El protocolo se ha aplicado a muchos otros fluidos biológicos, medios de cultivo celular y vesículas extracelulares purificadas aisladas de fluidos biológicos. Este protocolo se divide en un procedimiento de dos días con el flujo de trabajo para un experimento típico que se muestra en la Figura 1.

Figura 1: Flujo de trabajo de ensayo. Flujo de trabajo de ensayo para elegir el tipo de análisis que se completará para la muestra entre el tamaño y el recuento, el recuento de tamaño y la tinción de la superficie, y el recuento de tamaño y la tinción de la carga. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
Se recolectaron muestras de suero de ratones de acuerdo con un protocolo aprobado por los Comités Institucionales de Cuidado y Uso de Animales (IACUC) en el Centro Médico de la Universidad de Kansas (KUMC). El uso de estas muestras biológicas en estos experimentos también fue aprobado por KUMC.
1. Preparación de la muestra (día 1)
- Determine la concentración de EV utilizando el seguimiento de nanopartículas o una técnica equivalente.
- Diluir la muestra con la solución de incubación hasta una concentración de 5 x 107-5 x 108 EV/mL; se necesita un mínimo de 50 μL.
NOTA: Para muestras en las que se desconoce la concentración de EV, se puede utilizar proteína total a 1 μg/mL como medida sustitutiva. Si se espera que la muestra tenga una concentración baja, realice al menos una dilución 1:1 en la solución de incubación antes de cargarla. - Coloque una placa de 24 pocillos sobre una superficie plana, libre de vibraciones y movimientos bruscos.
NOTA: Para mejorar el contraste, se puede colocar una hoja de papel blanca debajo de la placa. - Agregue agua a las áreas que rodean los pozos (Figura 2).

Figura 2: Diseño de placa de 24 pocillos. Se muestran las ubicaciones de dónde alicuota ddH2O (se agregó tinte azul solo con fines de visualización) y los pozos en los que se mantendrán las fichas. Haga clic aquí para ver una versión más grande de esta figura.
2. Preparación y preescaneo de chips
- Retire la placa sellada de 48 pocillos que contiene las patatas fritas de la nevera a 4-8 °C y deje que alcancen la temperatura ambiente (~15 min) antes de abrir el precinto.
NOTA: Esto es esencial para evitar la condensación en las virutas que puede dañar las manchas. Si se observa condensación en la superficie al retirar el chip del paquete, espere más tiempo antes de quitar otros. - Continúe con el paso 8 para recuperar el mandril y prepararse para la ejecución previa al escaneo (Figura 3).
NOTA: Los datos previos al escaneo se utilizarán para identificar cualquier partícula que sea detectable en los puntos de captura antes de la incubación con la muestra para que puedan eliminarse durante el paso de análisis.

Figura 3: Imagen del mandril utilizado para cargar el chip en la máquina. Haga clic aquí para ver una versión más grande de esta figura.
- Con unas pinzas, retire el número deseado de virutas (una viruta por muestra) de la placa de 48 pocillos y cargue las virutas directamente en el mandril para la ejecución de la exploración previa. Cuando todos los chips destinados a ser utilizados para el experimento hayan sido cargados y preescaneados como se describe en el paso 8, proceda al paso 2.4.
NOTA: Cuando manipule chips, asegúrese de no tocar los cuadrados del centro, ya que los puntos de captura de anticuerpos se encuentran en esta región y se dañarán si se tocan con la pinza (Figura 4).

Figura 4: Chip y manejo adecuado del chip. (A) La línea punteada amarilla indica la ubicación de los anticuerpos detectados, o el lado funcional del chip. El ID del chip se encuentra debajo de la línea ("58"). La figura también muestra el manejo adecuado. (B) Demuestra un manejo inadecuado del chip. (C) Lado no funcional del chip. Haga clic aquí para ver una versión más grande de esta figura.
- Coloque cada chip preescaneado con la superficie funcionalizada hacia arriba en la placa de 24 pocillos preparada previamente. La superficie funcionalizada se reconoce fácilmente por la numeración distintiva, las cuadrículas de alineación y las tres cajas negras en el medio. El lado no funcionalizado es una superficie plana y uniforme de plata.
- Use pinzas de punta fina para asegurarse de que cada viruta esté centrada dentro del pocillo (Figura 5).
NOTA: Centrar el chip dentro del pocillo es fundamental porque si el chip toca la pared del pocillo, la muestra puede desprenderse después de la carga.

Figura 5: Demostración de la colocación adecuada de las virutas en el pozo. (A) Las virutas deben colocarse en el centro del pozo, sin esquinas que toquen los lados del pozo. (B) Representación de la colocación incorrecta de la viruta, donde las esquinas tocan los lados del pozo. Haga clic aquí para ver una versión más grande de esta figura.
3. Carga e incubación del chip y la muestra
- Pipetee 35 μL de la muestra preparada (del paso 1) en el chip.
NOTA: Tenga cuidado de no añadir burbujas ni tocar el chip con la punta de la pipeta, ya que puede impedir la distribución uniforme de la muestra en el chip o dañar las manchas de anticuerpos en el chip. La muestra debe extenderse por toda la superficie de la viruta. En el caso de que la muestra se desprenda de la viruta durante la carga, agregue muestra adicional al pocillo hasta un volumen de 250 μL o agregue solución de incubación hasta 250 μL y observe el factor de dilución modificado para esa muestra. - Selle la placa, utilizando la película de placa incluida en el kit para evitar la evaporación de la muestra.
- Incubar la muestra/chip durante la noche (~16 h) en la placa sellada a temperatura ambiente en un área libre de vibraciones o movimiento.
NOTA: Después de la incubación nocturna, el usuario elegirá la opción apropiada del Día 2 para su pregunta de investigación. Si solo desea el tamaño y la cantidad de EV, continúe con el paso 4 (Día 2). Si desea el análisis de varios marcadores de superficie, vaya al paso 5 (Día 2).
4. Determinación del tamaño y la cantidad de vehículos eléctricos (Día 2)
- Añada 1 ml de solución A en el lateral de cada pocillo que contenga una viruta, teniendo cuidado de no añadir directamente la solución sobre la viruta ni rascar la viruta con la punta de la pipeta.
- Coloque la placa en un agitador orbital girando a ~500 rpm durante 3 minutos a temperatura ambiente.
NOTA: Si las virutas traquetean en el plato, disminuya inmediatamente la velocidad de modo que el líquido se arremoline pero no haya traqueteo. - Retire 750 μL de líquido. Evite inclinar la placa durante la extracción de líquido para evitar el secado accidental de la viruta.
- Agregue 750 μL de solución B usando la técnica descrita en el paso 4.1 y agite a ~500 rpm durante 3 min a temperatura ambiente.
- Repita los pasos 4.3-4.4 dos veces más para un total de tres lavados con la solución B.
- Al final del último lavado, retire 750 μL de la solución, dejando 250 μL de solución B en el pocillo.
- Añadir 750 μL de agua bidestilada (ddH2O) y agitar a ~500 rpm durante 3 min a temperatura ambiente.
- Llene una placa de Petri de 10 cm con 50 mL de ddH2O y transfiera una ficha a la vez desde el pocillo a la placa con una pinza.
NOTA: Tenga cuidado de transferir el chip horizontalmente y asegúrese de que no se seque. Se pueden lavar hasta ocho virutas antes de reemplazar el ddH2O. - En el ddH2O, sostenga el chip por los bordes con una pinza y gírelo en el plato durante tres revoluciones para eliminar los desechos.
- Retire el chip en un ángulo de 45° fuera del agua y colóquelo sobre papel absorbente con el ID del chip hacia arriba (Figura 6).
NOTA: Las fichas ya están listas para ser leídas. Vaya al paso 8 para escanear en el lector SP-IRIS.

Figura 6: Forma correcta de extraer la viruta del agua ddH2O en un ángulo de 45°. (A) Vista desde arriba y (B) Vista desde el lateral que muestra el ángulo en el que se debe quitar la viruta. Haga clic aquí para ver una versión más grande de esta figura.
5. Preparación de la solución de anticuerpos (Día 2)
- Agregue 300 μL de solución de bloqueo por chip en un tubo de tamaño adecuado (0,5 a 3 mL).
- Añadir 0,6 μL de anticuerpo por cada 300 μL de la solución bloqueante. Mezcle suavemente golpeando el tubo y gire rápidamente.
NOTA: Los anticuerpos para CD9, CD81 y CD63 humanos o CD9, CD81 y CD63 murinos están incluidos en los kits apropiados. Si se desean otros anticuerpos conjugados con fluorescencia, determine la concentración óptima de tinción utilizando una valoración típica en un rango de 0,1 a 10 μg/ml con una carga de muestra constante como lo haría para la citometría de flujo.
6. Determinación del tamaño, recuento y fenotipado de EV con tinción inmunofluorescente
- Añada 1 ml de solución A en el lado de cada pocillo que contenga una viruta, teniendo cuidado de no añadir directamente la solución sobre la viruta ni rayar la viruta con la punta de la pipeta.
- Coloque la placa en un agitador orbital girando a ~500 rpm durante 3 minutos a temperatura ambiente.
NOTA: Si las virutas traquetean en el plato, disminuya inmediatamente la velocidad de modo que el líquido se arremoline pero no haya traqueteo. - Retire 750 μL del líquido. Evite inclinar la placa durante la extracción de líquido para evitar el secado accidental de la viruta.
- Agregue 750 μL de solución A usando la técnica descrita en el paso 6.1 y agite a ~500 rpm durante 3 minutos a temperatura ambiente.
- Repita los pasos 6.3 y 6.4 dos veces para un total de tres lavados con la Solución A.
- Al final del último lavado, retire 750 μL de la solución, dejando 250 μL de solución A en el pocillo.
NOTA: Si el usuario desea la tinción de la carga, continúe con el paso 7 en este punto. Si no es así, continúe con el paso 6.7 - Añada 250 μL de la solución de anticuerpos (paso 5) a cada chip del pocillo. Cubra la placa con papel de aluminio para protegerla de la luz y agite durante 1 h en el agitador orbital a temperatura ambiente.
- Agregue 500 μL de solución A, por lo que el volumen total por pocillo es ~1,000 μL.
- Retire inmediatamente 750 μL y agregue 750 μL de solución A y agite a ~500 rpm en el agitador orbital durante 3 min a temperatura ambiente.
- Retire 750 μL de la solución y agregue 750 μL de la solución B y agite a ~ 500 rpm durante 3 minutos a temperatura ambiente.
- Repita el paso 6.10 dos veces más para un total de tres lavados.
- Añadir 750 μL de agua bidestilada (ddH2O) y agitar a ~500 rpm durante 3 min a temperatura ambiente.
- Llene una placa de Petri de 10 cm con 50 mL de ddH2O y transfiera una ficha del pocillo a la placa con una pinza.
NOTA: Tenga cuidado de transferir el chip horizontalmente y asegúrese de que no se seque. Se pueden lavar hasta ocho chips antes de cambiarlos por ddH2O frescos. - En el ddH2O, sujete el chip por los bordes con una pinza y gírelo en el plato durante tres revoluciones para eliminar los residuos.
- Retire la viruta en un ángulo de 45° fuera del agua y colóquela sobre papel absorbente con el ID de la viruta hacia arriba (Figura 6).
NOTA: Las fichas ya están listas para ser leídas. Vaya al paso 8 (Recopilación de datos) para obtener instrucciones sobre cómo configurar los chips para escanear en el lector.
7. Tinción de carga opcional
NOTA: Este protocolo permite el etiquetado simultáneo de marcadores internos y de superficie.
- Con 250 μL de solución A restantes en cada pocillo, agregue 250 μL de solución C en cada pocillo.
- Colóquelo inmediatamente en el agitador orbital y ajústelo a ~ 200 rpm durante exactamente 10 minutos.
NOTA: En este paso y en el paso 7.8 a continuación, tanto la sincronización como la velocidad de agitación más lenta son críticas; Asegúrese de que la velocidad solo dé un remolino lento en los pozos y use un temporizador. - Después de 10 minutos de incubación con la solución C, agregue 500 μL de solución A a cada pocillo.
- Retire inmediatamente 750 μL y agregue 750 μL de solución A y agite a ~500 rpm en el agitador orbital durante 3 min a temperatura ambiente.
- Repita el paso 7.4 dos veces más para un total de tres lavados.
- Al final del último lavado, retire 750 μL de la solución, dejando ~ 250 μL de la solución en el pocillo con el chip.
- Agregue 250 μL de solución D a cada pocillo.
- Colóquelo inmediatamente en el agitador orbital ajustado a ~ 200 rpm durante exactamente 10 minutos.
- Después de 10 minutos de incubación con la solución C, agregue 500 μL de solución A a cada pocillo.
- Retire inmediatamente 750 μL y agregue 750 μL de solución A y agite a ~500 rpm en el agitador orbital durante 3 min a temperatura ambiente.
- Repita el paso 7.10 dos veces para un total de tres lavados.
- Retire 750 μL de la solución después del último lavado y vuelva al paso 6.7 anterior para teñir y completar el protocolo de ensayo.
8. Recopilación de datos
NOTA: El procedimiento para recopilar datos de los chips con el ExoView R100 está automatizado y no requiere entradas del usuario. Las instrucciones detalladas se pueden encontrar en la Guía del usuario y en el vídeo correspondiente para cargar el portador de chips, o "mandril", y la adquisición de datos17.
- Encienda la plataforma de caracterización de vehículos eléctricos utilizando los dos interruptores de alimentación situados en la parte trasera del instrumento y, a continuación, inicie el software del escáner haciendo doble clic en el icono del escritorio.
NOTA: Cuando se inicie el software del escáner, el lector se configurará automáticamente en la pantalla de inicio y luego le pedirá al usuario que "Abra la puerta para cargar chips". - Abra la puerta de la parte frontal del lector levantando el mango plateado. Esto expulsará la etapa, lo que permitirá al usuario acceder al mandril y configurar un nuevo escaneo en el software.
- Identifique la ubicación donde el usuario desea guardar los datos haciendo clic en la carpeta Guardar y seleccionando la ubicación deseada para que se guarden los datos.
NOTA: A menudo es útil asignar un nombre a la ubicación de guardado de forma ingeniosa, como el nombre del experimento, y luego crear dos subcarpetas, una para los análisis previos y otra para los exámenes posteriores. Esto facilita la búsqueda y coincidencia de los datos durante el análisis. - Para escanear los chips, localice los archivos de chips en la computadora de control.
NOTA: Los archivos de chip son mapas de la disposición de las manchas de anticuerpos en el chip en uso y permiten que el escáner sepa dónde escanear en cada chip. En cada kit, hay una llave USB con los archivos de chip para los chips en su interior. Estos deben guardarse en una ubicación en la computadora de control al recibir el kit donde los usuarios puedan encontrarlos de manera confiable. - Cargue las fichas en el mandril con el número de las fichas frente al mango del mandril y, a continuación, en el menú desplegable Ficha del ordenador, seleccione cada ficha de la lista de archivos de chips y colóquela en la ubicación correspondiente adecuada en el mandril virtual del software del escáner. Una vez completado, aparecerá un mensaje en pantalla que le pedirá al usuario que coloque el mandril en el escenario.
- Coloque el mandril cargado en el escenario; la alineación magnética del mandril lo moverá automáticamente a la ubicación correcta en el escenario y, a continuación, haga clic en Aceptar junto a Colocar el mandril en el escenario. A continuación, el escáner comenzará la rutina automatizada de recopilación de datos.
NOTA: Los datos están listos para ser analizados una vez que el software informa que el estado de escaneo de cada chip se ha realizado correctamente.
9. Análisis de datos
- Haga doble clic en ExoView Analyzer en el escritorio de la PC de control. Después de que se inicie el software, haga clic en el botón Datos de escaneo previo y seleccione la ubicación de la carpeta para el conjunto de datos de escaneo previo en la sección 8.3.
- Haga clic en el botón Datos de postscan y seleccione la ubicación de la carpeta para el conjunto de datos postscan en la sección 8.3.
NOTA: Si los datos se detectan correctamente en ambas carpetas, para al menos algunos de los chips, el software mostrará "X" número de chips detectados junto al botón de ubicación del archivo de chip. Cuando se guardan correctamente, las fichas detectadas deben coincidir con el número de fichas escaneadas. - Haga clic en Siguiente en la parte inferior del área de carga de datos.
- En el menú desplegable de la tabla meta, cada ficha tendrá una celda. Ingrese los nombres de las muestras, los factores de dilución y los marcadores que se están tiñendo en cada canal de detección haciendo clic en el cuadro correspondiente y escribiendo la información; cuando haya terminado, vuelva a hacer clic en Siguiente .
NOTA: Estos metadatos se guardarán con los datos. - Control de calidad (QC) de la preforma haciendo clic en la pestaña Desactivar debajo del botón QC en la parte superior izquierda.
NOTA: Esta función permite apagar sondas y chips específicos para el análisis. El software proporcionará dos advertencias diferentes con respecto a los datos, una para recuentos altos que pueden indicar que una captura en particular está saturada, y un coeficiente de variación (CV) alto que identifica cuando una de las réplicas de un tipo de captura es diferente de las demás.- Haga clic en un punto de advertencia para un CV alto, examine los puntos que están siendo señalados por el software y vea si hay daños físicos obvios; cuando se identifique el daño, haga clic en el número de punto para desactivar ese punto en el análisis.
- Repita hasta que se hayan evaluado todas las advertencias y, a continuación, haga clic en Siguiente.
- Análisis de corte de preforma haciendo clic en la pestaña Corte ubicada junto a la pestaña Deshabilitar debajo del botón QC en la parte superior izquierda.
NOTA: Esto presentará la respuesta fluorescente del punto de control en un gráfico y ofrecerá al usuario dos configuraciones, un mínimo y un máximo para cada canal de color (rojo / verde / azul). Establecer el valor de corte para cada canal de detección fluorescente es el único ajuste de datos que debe realizarse y es relativamente simple. Es importante destacar que el límite final que se elija debe ser coherente en todo el experimento y, como se ha señalado, debe estar dentro de las reglas generales para 300-400 u.a. en rojo y verde, y 400-700 u.a. para azul en experimentos típicos; El software también tiene guías codificadas por colores para el usuario para estos rangos.- Haga clic en la pestaña Color verde para cargar los datos de ese canal y mostrar los cortes actuales. Al examinar el punto de control negativo del isotipo, se pueden ver eventos de detección con intensidades de fluorescencia muy bajas; En este caso, los usuarios normalmente deben esperar establecer el verde entre 300 y 400 U.A.
- Aumente el mínimo para cada uno de los canales de detección hasta que el "% promedio Inc" (el % promedio de las partículas detectadas en el punto de control incluido encima del límite) debajo de la pantalla de datos no tenga advertencias como lo indica el resaltado rojo o amarillo en esa celda.
NOTA: Por lo general, no es necesario ajustar el valor máximo del valor predeterminado, pero se puede reducir para filtrar las partículas brillantes o limitar la detección fluorescente a un rango más estrecho. - Haga clic en el botón Siguiente .
- Aumente el mínimo para cada uno de los canales de detección hasta que el "% promedio Inc" (el % promedio de las partículas detectadas en el punto de control incluido encima del límite) debajo de la pantalla de datos no tenga advertencias como lo indica el resaltado rojo o amarillo en esa celda.
- Haga clic en la pestaña Color rojo para cargar los datos de ese canal y mostrar los cortes actuales. Una vez más, al examinar el punto de control negativo del isotipo, se pueden ver eventos de detección con intensidades de fluorescencia muy bajas; En este caso, los usuarios normalmente deben esperar establecer el rojo entre 300 y 400 U.A. Siga los mismos procedimientos que se indican en los pasos 9.6.1.1-9.6.1.3.
- Haga clic en la pestaña Color azul para cargar los datos de ese canal y mostrar los cortes actuales. Nuevamente, examinando el punto de control negativo del isotipo, siga los mismos procedimientos que se indican en los pasos 9.6.1.1-9.6.1.3.
NOTA: El canal azul es único en el sentido de que el anticuerpo en la mancha es autofluorescente a niveles suficientes para requerir un corte ligeramente más alto de 400-700 u.a. incluso para chips en blanco.
- Haga clic en la pestaña Color verde para cargar los datos de ese canal y mostrar los cortes actuales. Al examinar el punto de control negativo del isotipo, se pueden ver eventos de detección con intensidades de fluorescencia muy bajas; En este caso, los usuarios normalmente deben esperar establecer el verde entre 300 y 400 U.A.
- Una vez finalizado el análisis de corte, haga clic en Siguiente y aparecerá el gráfico del mapa de calor, que proporcionará una visión general de la unión de partículas en todo el experimento. Hay varias herramientas de visualización que el usuario puede utilizar para trazar los datos. Estos gráficos y los datos brutos asociados se pueden agregar a un informe haciendo clic en el botón Agregar informe de trazado .
NOTA: El mapa de calor es la visualización de datos predeterminada que muestra todas las muestras, todas las capturas de una detección en particular. La visualización predeterminada es el total que cuantifica los EV únicos vinculados a cada punto de captura. - Haga clic en el botón Exportar informe y seleccione una ubicación de guardado para el informe.
NOTA: El informe se abrirá en un navegador. Busque el archivo de hoja de cálculo Lista de partículas filtradas adjunto al informe; esta es la información que se debe enviar a EVTRACK. - Continúe agregando informes utilizando las diferentes selecciones de Muestras y tipos de captura en los menús desplegables en la parte superior de la pantalla. Los datos mostrados se controlan con la selección de los botones de detección de canales en la parte superior izquierda. Establezca un fenotipo/selección de muestra/canal de captura en particular haciendo clic en cada botón de color para desactivar (atenuado) o activar (coloreado) ese canal de detección.
- Haga clic en el botón Agregar trazado al informe para agregar los gráficos deseados en un informe.
- Haga clic en Exportar informe y defina la ubicación de guardado para el informe final.
Resultados
La Figura 7 (panel izquierdo) muestra una imagen compuesta de tres colores de EV derivados de medios condicionados HEK293 unidos al punto CD63 en el chip y teñidos para CD81, CD63 y CD9 en los siguientes canales verde, rojo y azul, respectivamente. La Figura 7 (panel superior derecho) es una imagen ampliada que muestra que cada uno de los EV capturados puede mostrar la colocalización de uno o más colores con intensidades variables en cada canal. La diferencia en la tinción de las EV capturadas representa la heterogeneidad de la expresión de las tres tetraspaninas en vesículas individuales y puede cuantificarse mediante el software de análisis de datos. La Figura 7 (panel inferior derecho) muestra los histogramas de tamaño de los EV, que están por encima de 50 nm. El histograma de tamaño muestra que los EV capturados en el punto CD63 tienen un tamaño de modo de 50 nm. La colocalización del color, la intensidad y el tamaño de cada EV vinculado a la imagen puntual representan los datos fenotípicos, que constituyen los datos fundamentales de la medición.

Figura 7: La imagen tricolor del punto completo muestra un punto de captura de CD63 completo con EV derivados de HEK293 unidos y teñidos para antiCD9-CF488, antiCD81-CF555, antiCD63-CF647. La imagen ampliada muestra la detección digital de fenotipos individuales de EV mediante colocalización fluorescente. La distribución de tamaño derivada de SP-IRIS para este chip se muestra en la parte inferior derecha. Los EV se recolectaron a partir de un medio condicionado de HEK293T células. Las células se cultivaron hasta una confluencia del 80%, y luego se lavaron con PBS y se cultivaron durante 48 h en medios que contenían FBS empobrecido en EV (10% vol/vol). Los medios acondicionados recolectados al final de las 48 h se centrifugaron a 2.500 x g para eliminar los desechos celulares y se analizaron los sobrenadantes restantes que contenían EV. Haga clic aquí para ver una versión más grande de esta figura.
La Figura 8 muestra una imagen compuesta de tres colores y un histograma de intensidad para dos ejemplos de tipos de respuestas de control de isotipos que un usuario puede encontrar. El panel superior (ejemplo de ejemplo 1, EX1) muestra el funcionamiento nominal, el punto de control del isotipo no tiene unión con una intensidad fluorescente mayor que la firma autofluorescente en azul (>500) y (>300 u.a.) en rojo y verde, lo que permite al usuario establecer cortes bajos. El panel inferior (muestra inespecífica 1, NS1) muestra cuándo se unen los EV al control del isotipo y da lugar a una tinción de alta intensidad como se muestra en el histograma. En este escenario, los datos sobre los puntos específicos del marcador no son fiables, ya que cubren el mismo rango de intensidad que los VE en el punto de control, la inferencia de que los VE en los puntos activos se reclutan específicamente allí ya no se sostiene. Cuando se produce este fenómeno, suele estar relacionado con el estado de la muestra.
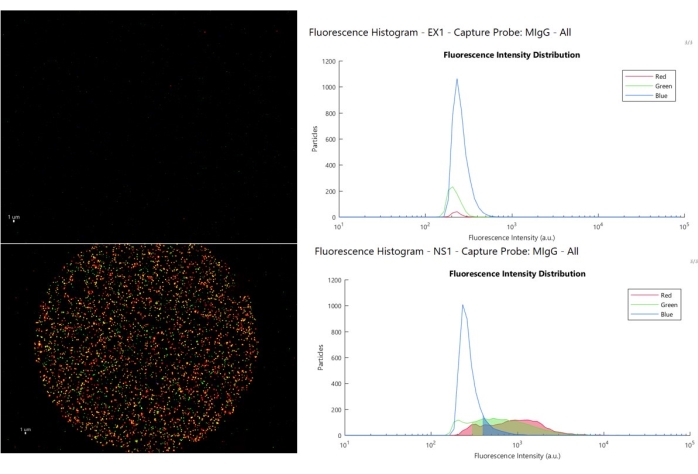
Figura 8: Análisis de corte. La imagen superior izquierda muestra un punto de control nominal y los histogramas fluorescentes asociados (arriba a la derecha), donde las partículas por encima del punto de corte se muestran en el área sombreada, la imagen inferior izquierda presenta un ejemplo de unión de EV inespecífica al punto de control, y las áreas sombreadas en el histograma fluorescente son las que están por encima del punto de corte, en el mismo rango que los EV positivos en el punto de captura. Haga clic aquí para ver una versión más grande de esta figura.
La figura 9 representa la colocalización de la proteína EV única de los VE derivados del suero humano. Los EV se capturaron con anticuerpos de captura anti-CD63, CD9, CD81 y mlgG y se tiñeron para CD81 (verde), HSP72 (rojo) y CD9 (azul). La Figura 9 muestra que CD81 es el marcador más acumulado en los VE derivados del suero humano. Mientras que, el nivel de CD63 es el más bajo en comparación con CD81 y CD9. Entre los VE capturados con anticuerpos anti-CD63, CD9 y CD81, la proporción de HSP72 en los VE capturados con CD63 es mayor, mientras que este nivel es similar entre los VE capturados con anticuerpos anti-CD9 y CD81. Esta detección de la localización específica de HSP72 y CD63 entre todos los EV no positivos para HSP72 que también llevan los marcadores de tetraspanina demuestra la capacidad única de recopilación de datos de la interferometría y la fluorescencia de una sola partícula.

Figura 9: Colocalización de proteínas Single-EV. Los VE derivados de humanos se capturaron utilizando anticuerpos de captura anti-CD63, CD9, CD81 y mlgG (isotipo) y se tiñeron para CD81 (verde), HSP72 (rojo) y CD9 (azul). La población de EV se fotografió utilizando el modo de fluorescencia. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Los métodos actuales de caracterización de VE se basan en gran medida en VE purificados, lo que está restringido por las limitaciones experimentales actuales de los métodos de purificación de VE 9,10,11,12,13. Las imágenes de reflectancia interferométrica de una sola partícula (SP-IRIS) son una tecnología eficaz que puede eliminar los pasos de purificación necesarios para el análisis de muestras y, por lo tanto, ahorrar tiempo y reducir los costos asociados con los flujos de trabajo típicos de EV. La entrada de muestra requerida es generalmente muy pequeña, dejando el resto de la muestra disponible para métodos analíticos adicionales.
Además, la capacidad de medir las VE de forma específica para el antígeno es muy importante para la investigación fundamental de las VE, así como para la detección de eventos raros en aplicaciones diagnósticas y terapéuticas. Sin embargo, las VE específicas de la enfermedad pueden estar presentes en bajas concentraciones en la sangre en relación con otras VE circulantes y contaminantes 2,11,12. El protocolo puede modificarse para incorporar otros anticuerpos para la detección de estas subpoblaciones utilizando anticuerpos marcados con fluorescencia con excitación a 488 nm, 555 nm o 647 nm respectivamente. En ese caso, se debe desarrollar un control biológico positivo y negativo contra el cual se pueda utilizar una serie de valoración para optimizar la detección del marcador de interés. Cabe señalar en este caso que el método de selección positiva empleado por SP-IRIS puede resultar perjudicial para este experimento porque el marcador de interés no pudo colocalizarse fuertemente con el tirón de la tetraspanina, lo que dificulta la detección. Si se presenta esta situación, es ventajoso capturar los EV en el chip utilizando el marcador de interés, superando así el sesgo de selección positiva en los puntos de tetraspanina. La capacidad de etiquetado generalizado de las imágenes SP-IRIS permite detectar hasta cuatro marcadores en un punto de captura en particular, lo que requiere un debate sobre cómo diseñar e interpretar la detección de nuevos marcadores por SP-IRIS y los controles adecuados. Cuando se utiliza el punto de control del isotipo como único control para un chip, realmente estamos considerando si hay una unión inespecífica de la muestra del anticuerpo directamente al punto de captura del isotipo durante la configuración de corte. Si observamos una firma de colocalización similar en el control del isotipo y en el punto específico del marcador, esto es indicativo de que los EV no están específicamente adheridos al control y el protocolo debe ser examinado. La selección de buenas muestras de control positivas y negativas puede ayudar a los usuarios a diferenciar si la unión inespecífica proviene de los anticuerpos en el chip o de los anticuerpos primarios marcados con fluorescencia. Si se sospecha que las EV se unen al punto de control del isotipo, se puede incubar una muestra de EV de control negativo a una concentración fija en un chip y teñirse con la concentración de tinción normal del anticuerpo de interés. La configuración de corte debe realizarse como se describe en la sección 9 Recopilación de datos, paso 9.6. Confirme que no hay ningún enlace en los puntos del marcador de interés. Los puntos de corte establecidos en la muestra EV negativa se pueden aplicar al resto de las muestras del experimento. Siempre que se disponga de un control positivo, es útil realizar una prueba de tinción de titulación para determinar la concentración óptima de tinción con anticuerpos adicionales.
La ventaja de la detección específica utilizada en SP-IRIS para EV también puede presentar limitaciones para la técnica. La etapa de incubación de la sección 3 es inherentemente un método de selección positiva, que no proporciona información sobre las partículas que podrían estar presentes en la muestra, pero no muestra los marcadores correspondientes a los tipos de captura en un chip específico, ya que se lavarán después de la captura y no se detectarán durante el análisis. Debido al hecho de que no existe un marcador universal aceptado presente en todos los VE, los usuarios deben revisar sus datos como una población medida y considerar la cantidad de partículas capturadas frente a medidas ortogonales de concentración total de partículas para decidir si los datos de captura de SP-IRIS representan todos los VE en una muestra en particular.
La técnica SP-IRIS se ha aplicado a otras nanopartículas biológicas (por ejemplo, virus patógenos y vectores virales)18. Para aplicar SP-IRIS a la detección de otras partículas biológicas, es necesario seleccionar un anticuerpo de captura o una sonda contra un marcador de superficie para permitir su inmunocaptura en el chip. Actualmente, las partículas biológicas de hasta 200 nm de diámetro se pueden caracterizar utilizando SP-IRIS. No hay un límite inferior en el diámetro si se utiliza la lectura de fluorescencia, ya que el SP-IRIS tiene una sensibilidad de sonda de detección única. Para garantizar el rigor y la reproducibilidad de los estudios SP-IRIS y facilitar la integración con los demás datos de vehículos eléctricos, proponemos utilizar la plataforma EV Track19. La carga de datos totales no es práctica (gigabytes) y solo es accesible para aquellos que usan el software ExoView. Por lo tanto, proponemos que para cada muestra interrogada y que forme parte de una publicación, el archivo de la lista de partículas filtradas que se puede exportar fácilmente con el mapa de calor se cargue en EV Track. Este archivo tiene los datos completos de partículas y la configuración de corte a partir de la cual se puede verificar o responder cualquier cifra o pregunta.
En conclusión, SP-IRIS ofrece un método eficaz y eficiente de caracterización de VE que proporciona una forma sencilla de realizar mediciones rutinarias de un solo tamaño de VE y visualización de biomarcadores. Estos nuevos datos disponibles pueden revelar detalles ocultos sobre la estequiometría de la carga de VE, la detección directa de subpoblaciones raras y ayudar a allanar el camino para nuevos avances en nuestra comprensión del importante papel de los VE en la salud y la enfermedad.
Divulgaciones
Clayton Deighan y George Daaboul son empleados y accionistas de NanoView Biosciences Inc.
Agradecimientos
Este trabajo fue patrocinado en parte por el Programa de Premios de Adquisición de Recursos y Equipos de Investigación de la Facultad de Medicina de la Universidad de Kansas. PCG, LKC, FD y AR fueron apoyados con fondos de NIA R21 AG066488-01.
Materiales
| Name | Company | Catalog Number | Comments |
| 10-cm sterile Petri dish | Fisher | FB0875712 | |
| 15mL sterile tube | n/a | various | |
| 24-well cell culture plate, flat bottom | Fisher | 08-772-1 | |
| Blocking Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chipfiles | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chips | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Chuck | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 250 ml | Fisher | 09-761-4 | |
| Corning Easy Grip Disposable Polystyrene Sterile Bottles 500 ml | Fisher | 09-761-10 | |
| Deionized (DI) water | Fisher | LC267404 | |
| EMS style tweezers with Carbon Fiber tips | Fisher | 50-193-0842 | |
| ExoView Human Tetraspanin Kit | NanoView Biosciences | EV-TETRA-C | Capture for hCD81, hCD9, hCD63, IgG Control + stains for hEV-A (hEV-CD63-647, hEV-CD81-555, hEV-CD9-488) 16 Chips per kit |
| ExoView R100 Imager | NanoView Biosciences | EV-R100 | Interferometric microscope including high specification camera including 3 color fluorescence and label free sizing and counting extracellular vesicles |
| Fluorescently labled huma CD9 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD63 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Fluorescently labled human CD81 IgG antibody | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Incubation Solution | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Orbital shaker or microplate shaker with digital settings capable of shaking at 500 rpm | n/a | various | |
| Plate Seal | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution A | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution B | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution C | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Solution D | NanoView Biosciences | EV-TETRA-C | Can be found in ExoView Human Tetraspanin Kit. |
| Square/flat tip tweezer | Fisher | 50-239-62 | |
| Straight strong point Boley style tweezers | Fisher | 16-100-124 | |
| Thermo Scientific Adhesive PCR Plate Seals | Fisher | AB-0558 |
Referencias
- Maas, S. L. N., Breakefield, X. O., Weaver, A. M. Extracellular vesicles: Unique intercellular delivery vehicles. Trends in Cell Biology. 27 (3), 172-188 (2017).
- Shah, R., Patel, T., Freedman, J. E. Circulating extracellular vesicles in human disease. The New England Journal of Medicine. 379 (10), 958-966 (2018).
- Deng, F., Miller, J. A review on protein markers of exosome from different bio-resources and the antibodies used for characterization. Journal of Histotechnology. 42 (4), 226-239 (2019).
- Cohen, O., et al. ’Golden’ exosomes as delivery vehicles to target tumors and overcome intratumoral barriers: in vivo tracking in a model for head and neck cancer. Biomaterials Science. 9 (6), 2103-2114 (2021).
- Han, Y., et al. Overview and update on methods for cargo loading into extracellular vesicles. Processes (Basel). 9 (2), 356 (2021).
- Lee, M., Im, W., Kim, M. Exosomes as a potential messenger unit during heterochronic parabiosis for amelioration of Huntington's disease. Neurobiology of Disease. 155, 105374 (2021).
- Sun, B., et al. Characterization and biomarker analyses of post-COVID-19 complications and neurological manifestations. Cells. 10 (2), 386 (2021).
- Jiang, L., Gu, Y., Du, Y., Liu, J. Exosomes: Diagnostic biomarkers and therapeutic delivery vehicles for cancer. Molecular Pharmaceutics. 16 (8), 3333-3349 (2019).
- Bachurski, D., et al. Extracellular vesicle measurements with nanoparticle tracking analysis - An accuracy and repeatability comparison between NanoSight NS300 and ZetaView. Journal of Extracellular Vesicles. 8 (1), 1596016 (2019).
- Carmicheal, J., et al. Label-free characterization of exosome via surface enhanced Raman spectroscopy for the early detection of pancreatic cancer. Nanomedicine. 16, 88-96 (2019).
- Greening, D. W., Xu, R., Ji, H., Tauro, B. J., Simpson, R. J. A protocol for exosome isolation and characterization: evaluation of ultracentrifugation, density-gradient separation, and immunoaffinity capture methods. Methods in Molecular Biology. 1295, 179-209 (2015).
- Wu, Y., Deng, W., Klinke, D. J. Exosomes: improved methods to characterize their morphology, RNA content, and surface protein biomarkers. The Analyst. 140 (19), 6631-6642 (2015).
- van de Vlekkert, D., Qiu, X., Annunziata, I., d'Azzo, A. Isolation and characterization of exosomes from skeletal muscle fibroblasts. Journal of Visualized Experiments: JoVE. (159), (2020).
- Ayala-Mar, S., Donoso-Quezada, J., Gallo-Villanueva, R. C., Perez-Gonzalez, V. H., Gonzalez-Valdez, J. Recent advances and challenges in the recovery and purification of cellular exosomes. Electrophoresis. 40 (23-24), 3036-3049 (2019).
- Daaboul, G. G., et al. Digital detection of exosomes by interferometric imaging. Scientific Reports. 6, 37246 (2016).
- Pohler, K. G., et al. Circulating microRNA as candidates for early embryonic viability in cattle. NMolecular Reproduction and Development. 84 (8), 731-743 (2017).
- NanoView Biosciences. ExoView R100 User Guide. v240.4. NanoView Biosciences. , 202 (2021).
- Daaboul, G. G., et al. Enhanced light microscopy visualization of virus particles from Zika virus to filamentous ebolaviruses. PLoS One. 12, 0179728 (2017).
- ET Consortium. EV-TRACK: transparent reporting and centralizing knowledge in extracellular vesicle research. Nature Methods. 14, 228-232 (2017).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados
Utilizamos cookies para mejorar su experiencia en nuestra página web.
Al continuar usando nuestro sitio web o al hacer clic en 'Continuar', está aceptando nuestras cookies.