Method Article
Dna İlkase tarafından DNA Dizisi Tanıma Yüksek-Throughput Primase Profilleme kullanarak
Bu Makalede
Özet
Protein bağlayıcı mikrodizi (PBM) deneyleri biyokimyasal tahlillerle birleşince, şablon DNA üzerinde RNA astarlarını sentezleyen bir enzim olan DNA primazının bağlayıcı ve katalitik özelliklerini birbirine bağlar. Yüksek iş itrifli primase profilleme (HTPP) olarak tanımlanan bu yöntem, çeşitli enzimlerin DNA bağlayıcı desenlerini ortaya çıkarmak için kullanılabilir.
Özet
DNA primazı, DNA replikasyonu sırasında DNA polimeraz tarafından gecikmeli iplikçikteki Okazaki parçalarının DNA sentezini başlatan kısa RNA astarları sentezler. Prokaryotik DnaG benzeri primazların DNA'ya bağlanması belirli bir trinükleotid tanıma dizisinde gerçekleşir. Okazaki parçalarının oluşumunda önemli bir adımdır. DNA primazlarının DNA tanıma dizilimini belirlemek için kullanılan konvansiyonel biyokimyasal araçlar sadece sınırlı bilgi sağlar. Yüksek iş gücü mikrodizi tabanlı bağlayıcı test ve ardışık biyokimyasal analizler kullanılarak, 1) spesifik bağlayıcı bağlamın (tanıma alanının yan dizileri) DNA primazının şablonuna bağlanma gücünü etkilediği gösterilmiştir. DNA, ve 2) DNA primase daha güçlü bağlanması daha uzun RNA astarları verir, enzimin daha yüksek prosesini gösterir. Bu yöntem PBM ve primaz aktivite testi birleştirir ve yüksek iş itfa primase profilleme (HTPP) olarak belirlenmiş ve dna primaz tarafından benzeri görülmemiş zaman ve ölçeklenebilirlik belirli dizi tanıma karakterizasyonu sağlar.
Giriş
HTPP, DNA primazının enzimatik aktivitesini etkileyen DNA şablonlarının belirli özelliklerini istatistiksel olarak belirlemek için biyokimyasal analizle birlikte DNA bağlayıcı mikrodizi teknolojisini(Şekil 1)kullanır. Bu nedenle HTPP, bu alanda bilgi sıçramasını kolaylaştıran teknolojik bir platform sağlamaktadır. Primaz tanıma sitelerini belirlemek için kullanılan klasik araçlar büyük miktarda veri verme yeteneğine sahip değildir, oysa HTPP bunu yapar.
PBM, transkripsiyon faktörlerinin DNA1,2'yebağlanma tercihlerini belirlemek için rutin olarak kullanılan bir tekniktir; ancak proteinlerin DNA'ya zayıf/geçici bağlanmasının saptanması için uygun değildir. Sekiz baz çiftinden oluşan tüm olası dizilere ortalama protein bağlama özgüllüğü hakkında bilgi sağlayan evrensel PBM'nin aksine, HTPP benzersiz dizi öğelerinden oluşan tek iplikli DNA şablonlarının kitaplığına dayanır. Bu tür DNA dizisi elemanları on binlerce kısa (birkaç on bp) genomik dizinin yanı sıra genomda bulunan ve farklı ortalama GC içeriğine sahip olan bazı DNA tekrarlayan dizi elementlerinde zenginleştirilmiş hesaplamalı olarak tasarlanmış DNA dizilerini içerir. . Böyle bir yüksek iş ilişkisi yaklaşımı, sistematik, nicel ve hipotez odaklı bir şekilde, ilkel bağlama ve enzimatik aktivitesi için önemli olan diziile ilgili özelliklerin belirlenmesine olanak sağlar3. Özellikle, primase-DNA bağlama tercihleri arasındaki önemli bağlantı, (spesifik üç nükleotit bağlama siteleri çevreleyen DNA dizileri tarafından modüle) ve primase prosesivite bu enzimatik sistem için tespit edilmiştir4.
Yeni teknoloji kapsamlı 5çalışılmıştırT7 DNA primase için bile primase tanıma siteleri anlayışımızı yeniden ziyaret etmek için uygulanmıştır. Özellikle, t7 DNA primase DNA tanıma siteleri gibi klasik kavramların yeniden incelenmesi (yaklaşık 40 yıl önce tespit edildi 6) protein-DNA bağlayıcı mikrodizi (PBM) ile ilgili özellikleri daha önce görülmemiş bir anlayış yol açmıştır bu tanıma sitelerinin yan sırası3. T7 DNA primazının (5'-GTC-3') üç nükleotid tanıma bölgesini çevreleyen sekansların rastgele olması beklenmektedir. Bunun yerine, TG açısından zengin yan dizilerin T7 DNA primazının daha uzun RNA astarlarını sentezleme şansını artırdığını ve proseste bir artış olduğunu gördük.
Proteinlerin DNA bağlayıcı özelliklerini in vitro incelemek için kullanılabilecek diğer yöntemler arasında elektroforetik hareketlilik değişim testi (EMSA)7, DNase I ayak izi8, yüzey plazmon rezonans (SPR)9ve Güneybatı blotting 10. Ancak bunlar, düşük iş verme yöntemleridir, sadece az sayıda DNA dizisinin araştırılması için geçerlidir. Buna ek olarak, bu tekniklerden bazılarının (örneğin, EMSA) hassasiyeti ve hassasiyeti düşüktür. Diğer taraftan, in vitro selection11, PBM'ye benzer şekilde, çok sayıda bağlama dizisinin tanımlanmasında kullanılabilecek bir tekniktir. Ancak, düşük yakınlık dizileri genellikle in vitro seçimi nin çoğu uygulamasında dışlanır; bu nedenle, bu yaklaşım tüm kullanılabilir diziler için karşılaştırmalı bağlama verileri elde etmek için uygun değildir. Evrensel PBM1,2 esas olarak prokaryotlar ve ökaryotlar dan transkripsiyon faktörlerin bağlayıcı özellikleri yanı sıra belirli faktörler (örneğin, bazı ligands varlığı, kofactors, vb) karakterize etmek için kullanılır bu etkileşimi etkiler12.
HTPP, ciltleme sırası bağlamı hakkında bilgi sağlamak için eşi görülmemiş yüksek iş letiye istatistiksel gücü yüksek hassasiyetle birleştirerek PBM uygulamasını DNA işleme enzimlerine genişletir. Bu tür veriler, mevcut diğer tekniklerin yukarıda belirtilen teknik sınırlamaları nedeniyle primases ve ilgili enzimler (DNA'ya zayıf/geçici bağlayıcılığı olan) için henüz elde edilmemiştir.
Protokol
1. Mikrodizi tasarımı
NOT: DNA probları, iki değişken yan bölge arasında yer alan T7 DNA primazının (GTC) tanıma bölgesinden oluşan özel 36 nükleotit dizilerini temsil eder ve ardından cam birslayt3'ebağlı sabit 24 nükleotit dizisi bulunur. 4 x 180.000 mikrodizi formatı kullandık, bu da her DNA dizisinin altı kopyada tespit ini sağladı, slaytta rastgele dağıtıldı.
-
Bilinen tanıma dizilerine sahip primases için DNA kitaplığı tasarımı
- Her dizinin 60 nükleotitten oluştuğundan emin olun. İlk 24 nükleotiti sabit tutun (cam kaydıraä a bayılacak). Değişken bölgeler aşağıdaki genel forma sahip olmalıdır: (N)16GTC(N)17; N istenilen nükleotiti temsil eder.
- Belirli bir bilimsel soruyu ele alacak şekilde yan bölgeyi tasarlayın (tasarımın bir örneği aşağıdaki metinde sunulmuştur). Yan bölgeler, yinelenen öğeler veya belirli simetri gibi belirli özellikleri içerecek şekilde tasarlanabilir.
NOT: İki veya üç spesifik nükleotitten oluşan farklı yan dizilerden oluşan sekiz kategori oluşturduk: T ve G (grup 1); T ve C (grup 2); C ve A (grup 3); A ve G (grup 4); G, C ve T (grup 5); C, T ve A (grup 6); G, A ve T (grup 7); G, A ve C (grup 8). Her grup için 2.000 farklı dizi tasarlandı. Her grup farklı türleri ve dizi tekrarları farklı sayılarda diziler alt grupları vardı. - Negatif kontrol dizileri kümesi (belirli bağlama sitesi eksik)4. Bu tür dizilerin varlığı, deneyde belirli bağlamanın oluşumunu doğrulamaya olanak tanır.
-
Bilinmeyen tanıma dizileri ile primases için DNA kitaplığı Tasarımı
- Tanıma sırası bilinmiyorsa, istenilen organizmanın (bakteri, mantar, vb.) genomundan dizileri (daha önce bahsedilen belirli özelliklere sahip olsun veya olmasın) seçerek DNA kitaplığını oluşturun.
-
Mikrodizi slayt tasarımı
- Ticari bir tedarikçiden özel slaytlar (örn. 4x180K, AMADID #78366) satın alın (daha fazla ayrıntı için Bkz. Malzemeler Tablosu). Her çoğaltmanın slaytta rasgele seçilmiş bir noktaya eklenmesi gereken altı çoğaltma halinde her sırayı sırala.
2. Primaz DNA bağlama deneyi
NOT: PBM'den bir gün önce (veya en az 2 saat önce) bloke çözeltisini hazırlayın [fosfat tamponsal salininde %2 w/v yağsız süt (PBS)] ve manyetik karıştırıcı üzerinde karıştırın. Kullanmadan önce, çözeltiyi 0,45 μM filtre ile filtreleyin. DNA iplikçiklerine bağlanan primase bağlanmayı saptamak için aşağıdaki sırada birkaç adım yapılmalıdır.
- Engelleme yordamı
- PBS'de %0,01 v/v Triton X-100 ile Coplin kavanozundaki mikrodizi slaytını önceden ıslayın (laboratuvar rotatöründe 125 rpm'de 5 dk).
- Conta kaydırasını (coverslip olarak da adlandırılır) su ve etanol ile kısa bir süre yıkayın ve basınçlı hava kullanarak kurulayın.
- Çelik hibridizasyon odasının (PBM odası) alt kısmını conta kaydırağı yukarı bakacak şekilde monte edin (ticari etiket yukarı bakacak şekilde). Montajla ilgili daha fazla bilgi için Malzeme Tablosu'nabakın.
- Pipet blokaj çözeltisi (%2 w/v skim süt PBS) her odaya.
- Coplin kavanozundaki mikrodizi slaytını çıkarın, ardından DNA olmayan tarafı kurulayın (DNA şirket etiketiyle aynı tarafta olmalıdır) ve ince bir silme kullanarak kenarlar. Mikrodiziyi yavaşça conta kaydırağına yerleştirin, kabarcıklardan kaçının (şirket etiketi aşağı bakacak şekilde). Hemen monte ve çelik hibridizasyon odası cihazları sıkın. Oda sıcaklığında 1 saat (RT) için kuluçka.
- 800 mL taze PBS ile bir boyama kabını ("PBS" çanak) doldurun. PBS'de ikinci bir boyama kabını ("WASH" çanak) 800 mL taze hazırlanmış %0,5 v/v Tween-20 ile doldurun.
- PBM odasını sökün ve mührü kırmamaya dikkat edin, mikrodizili slayt kapaklı "sandviçi" çıkarın. Slide ve coverslip arasına forceps yerleştirerek WASH çanak su altında sökmek.
- Su altında mikrodizi slayt çalkalayın ve hızlı bir Coplin kavanoza aktarın.
- PBS'de %0,1 v/v Tween-20 ile bir kez yıkayın (laboratuvar rotatöründe 125 rpm'de 5 dk).
- PBS'de %0,01 v/v Triton X-100 ile bir kez yıkayın (laboratuvar rotatöründe 125 rpm'de 2 dk).
- Kaydırağı hızlıca PBS içeren bir Coplin kavanozuna aktarın.
- Protein bağlanması
- PBM odasını daha önce açıklandığı gibi birleştirin (adım 2.1.2-2.1.3).
- 5 μM T7 DNA primaz içeren pipet protein bağlayıcı karışımı, 30 mM Tris-HCl pH 7.5, 6.5 mM MgCl2,30 mM K-glutamat, 6 mM dithiothreitol (DTT), 65 μM ribonükleozit trifosfat (rNTP), %2 w/v yağsız süt, 100 ng/μL büyükbaş serum albumin (BSA), 50 ng/μL somon DNA testisleri ve 0.02% v / v Triton X-100 (TX-100) conta slayt her odasına (kauçuk kenarlarıdokunmadan ve kabarcıklar tanıtmadan).
- Mikrodizikızmasını pbs kabına batırarak kısa bir süre durulayın, bu da fazla deterjanı slaytların yüzeyinden temizler. Slaytın DNA olmayan yüzeylerini ince bir silme ile silin.
- Bir odadan diğerine sızıntıyı önlemek için dikkatli olmak için, yavaş yavaş conta slayt üzerine mikrodizi slayt (aşağı bakan) indirin. Kabarcıkları tanıtmadan PBM oda cihazlarını hemen monte edin ve sıkın. Kabarcıklar oluşursa, çelik hazneye sert bir yüzeye hafifçe dokunarak çıkarılabilirler.
- Oda sıcaklığında 30 dakika (RT) kuluçka.
- Floresan antikor eki
- PBM odasını sökün ve mührü kırmamaya dikkat edin, mikrodizili slayt kapaklı "sandviçi" çıkarın. Wash kabında forceps kullanarak su altında sökülür. Su altında kaydırağı çalkalayın ve hızlı bir şekilde PBS içeren bir Coplin kavanoza aktarın.
- PBM odasını daha önce açıklandığı gibi conta kaydırağı yla birleştirin (2.1.2-2.1.3. adım).
- Alexa 488 konjuge antikor (bağlayıcı tampon 10 ng / μL) conta slayt ekleyin.
- Daha önce olduğu gibi PBS çanak daldırma tarafından slayt kısa bir süre durulayın, sonra yavaş yavaş PBS slayt kaldırmak, DNA olmayan yüzeyi silin ve conta slayt üzerine bakan aşağı yerleştirin.
- Foto-beyazlatma azaltmak için karanlıkta RT 30 dakika kuluçka (floresan prob ışığa duyarlıdır).
- Wash çanak içinde su altında coverslip kaldırarak, PBM odası ve coverslip daha önce olduğu gibi sökmek.
- Slaytları PBS kabına batırarak kısa bir süre durulayın. Slaytları basınçlı hava ile kurulayın. Tçalışmaya hazır olana kadar bir slayt kutusunda karanlıkta saklayın.
- Mikrodizi slaytını taramak
- 495 nm uyarma ve 519 nm emisyon ile mikrodizi tarayıcı (Malzeme Tablosubakın) kullanarak çip tarayın ve medyan floresan yoğunluğu toplamak.
3. Mikrodizi veri analizi
NOT: Tüm veri işleme MATLAB özel yazılı komut dosyaları kullanılarak yapıldı.
- Wilcoxon sıralama toplamı test p-değeri kullanarak ilk PBM veri işleme gerçekleştirin daha önce açıklandığı gibi4.
- Daha fazla analiz için her DNA dizisi için bağlayıcı yoğunluğun ortanca değerini kullanın.
- Daha sonra, tek yönlü ANOVA p-değerleri kullanarak, dna kütüphane tasarım bölümünde yukarıda açıklandığı gibi, DNA sondaları farklı gruplar için elde edilen primase-DNA bağlayıcı yoğunlukları gözlenen farklılıklarıistatistiksel önemi karşılaştırın3.
4. T7 DNA primazı ile katalize edilen şablon ait RNA sentezi
- Denaturing poliakrilamid jel hazırlanması
- 100 mL jel karışımı hazırlamak için, 62.5 mL%40 akrilamid-bisacrylamid (19:1), 42 g üre, 1.1 g Tris baz (2-Amino-2-(hidroksimetil)-1,3-propanediol), 0.55 g borik asit ve 0.4 mL 0.5 M etilediaminemineetik asit (EDTA çözeltisi) birleştirir.
- Karışımı 14 mL aliquots (16,5 cm x 26 cm x 0,03 cm bir jel için gerekli miktar) bölün ve 1 aya kadar 4 °C ışıktan korunun.
- Bir denaturing poliakrilamid jel hazırlamak için, 4 μL TEMED ve 40 μL 10% w / v amonyum persülfat 14 mL önceden hazırlanmış karışımı ekleyin, döküm ve RT en az 2 saat polimerize bırakmak. Jel 4 °C'de bir gün saklanabilir.
- Numune hazırlama
NOT: Reaktifleri, reaksiyon karışımını ve numuneleri buz üzerinde tutun.- On reaksiyon için gerekli reaksiyon karışımını hazırlamak için 11 μL 5x aktivite tamponu (200 mM Tris-HCl, pH = 7.5; 50 mM dithiothreitol, 250 mM potasyum glutamat, 50 mM MgCl2), 5.5 μL nükleotit tri-fosfat (NTPs) ve 5.5l radiolabeled ribonnucleottide [örneğin, ATP, (α-32P) 3000 Ci/mmol].
NOT: Tüm miktarlar, olası pipetleme hataları nedeniyle %10 artırılmıştır. Radyoetiketli ribonükleotidler radyoaktif sinyalin gücüne göre seyreltilmelidir (ilk seyreltme genellikle 1:10 veya daha fazladır). - Reaksiyon karışımının 2 μL'sini on PCR tüpüne aktarın.
- İlgili PCR tüpüne 1 μL 100 μM DNA şablonu ekleyin ve aşağı doğru döndürün.
- 2 μL 16 μM T7 DNA primaz ekleyin, aşağı spin, yavaşça karıştırın ve tekrar aşağı spin.
- 20 dakika RT'de reaksiyon uyguluyor.
- 5 μL'lik söndürme çözeltisi (%95 formamid, 20 mM EDTA, %0,05 w/v ksilen cyanol ve bromofenol mavisi) ekleyerek reaksiyonu durdurun ve aşağı doğru döndürün.
- On reaksiyon için gerekli reaksiyon karışımını hazırlamak için 11 μL 5x aktivite tamponu (200 mM Tris-HCl, pH = 7.5; 50 mM dithiothreitol, 250 mM potasyum glutamat, 50 mM MgCl2), 5.5 μL nükleotit tri-fosfat (NTPs) ve 5.5l radiolabeled ribonnucleottide [örneğin, ATP, (α-32P) 3000 Ci/mmol].
- RNA ürünlerinin elektroforez ile poliakrilamid jelden ayrılması ve müteakip sinyal tespiti
- Jeli 1 saat (10 mA, 600 V) 1x TBE tampon (90 mM Tris-borat, 2 mM EDTA) için önceden çalıştırın.
- Her numunenin 2 μL'ine kadar yükleyin ve 1x TBE tamponunda (90 mM Tris-borat, 2 mM EDTA) elektroforez (20 mA, 1100 V) gerçekleştirin.
- Jeli 80 °C'de 2 saat vakum altında kurutun.
- Fosfor görüntüleme plakasını, radyoaktif sinyalin gücüne bağlı olarak birkaç saat boyunca bir gece boyunca radyoaktif jele maruz bırakın.
- Fosforimager kullanarak sinyali (fotouyarılmış parlaklık) kaydedin (bkz. Malzeme Tablosu).
Sonuçlar
Primase bağlama alanlarının haritalanmasındaki bu teknolojik ilerleme, klasik aletler kullanılarak gözlemlemek imkansız olmasa da, zor olan DNA bağlama özelliklerinin elde edilmesine olanak sağlar. Daha da önemlisi, HTPP primase bağlayıcı sitelerin geleneksel anlayış yeniden ziyaret sağlar. Özellikle HTPP, bilinen 5'-GTC-3 tanıma dizilerine ek olarak bağlayıcı özellikleri ortaya çıkarır ve bu da T7 DNA primazının fonksiyonel aktivitelerinde değişikliklere yol açar. Yani, iki dizi grubu tanımlanmıştır: yanlarda T/G içeren güçlü bağlayıcı DNA dizileri ve yanlarda A/G içeren zayıf bağlayıcı diziler (güçlü bağlayıcı şablonlarda bulunan tüm timinler adeninlerle değiştirildi). Diziiçinde 5'-GTC-3' eksik olan DNA şablonlarına primas ebağlanmadı.
T/G açısından zengin yanlar gibi belirli özellikler içeren primase DNA tanıma siteleri, primase-DNA bağlanmasını 10 kata kadar artırdı ve şaşırtıcı bir şekilde yeni oluşan RNA'nın (üç kata kadar) uzunluğunu artırdı(Önceki yayında Şekil 2 3). Daha da önemlisi, HTPP DNA şablonunun dizilimi ile ilgili olarak astar uzunluğundaki değişkenliği gözlemlememizi ve ölçmemizi sağladı.
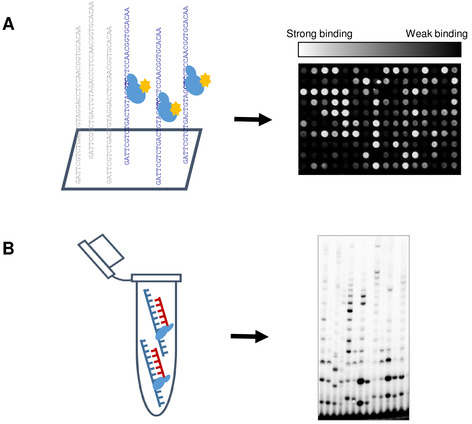
Şekil 1: Yüksek iş itimat primaz profilleme (HTPP) şematik gösterimi. (A) Slayt aktivite tampon (40 mM Tris-HCl, pH 7.5; 10 mM MgCl2, 50 mM K-glutamat, 10 mM DTT, 100 μM rNTPs) primase ile inkübe edildi. Sonra, Alexa 488-konjuge anti-onun floresan antikor protein etiket tanıtıldı. Hafif yıkama adımından sonra, slayt mikrodizi tarayıcı kullanılarak tarandı. Ortalama floresan sinyaline göre bağlayıcı afinite saptandı ve DNA dizileri buna göre gruplara ayrıldı. (B) Mikrodizi deneyinden elde edilen DNA bağlayıcı sonuçları T7 DNA primazının fonksiyonel özellikleriyle ilişkilendirmek için biyokimyasal tahliller yapıldı. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.

Şekil 2: PBM'den elde edilen iki grup DNA şablonu üzerinde T7 DNA primazının katalitik aktivitesinin karşılaştırılması (yanlarda T/G ile güçlü bağlanma ve yanlarda A/G ile zayıf bağlanma) elde edilir. (A) RNA astar oluşumu T7 DNA primazı tarafından katalize edilir. Reaksiyonlar, standart reaksiyon karışımında primas tanıma sırası (numaralı şeritler), 32P-α-ATP, ATP, CTP, UTP ve GTP ile oligonükleotidiçeriyordu. Bir grup DNA oligonükleotid (koyu mavi) yanlarda T/G bulunurken, tüm timinler ikinci grupta (açık mavi) adeninlerle değiştirildi. Kuluçkadan sonra radyoaktif RNA ürünleri elektroforez ile 7M üre içeren %25 poliakrilamid jel ile ayrılmış ve otoradyografi ile görselleştirilmiştir. (B) Bağıl uzunluk (her RNA astarını oluşturan ribonükleotid sayısı bilinmemektedir) ve iki grup DNA şablonu (panel A) üzerinde T7 DNA primazı ile sentezlenen RNA astarlarının miktarı bilinmemektedir. Çizimler, daha düşük afinite (yanlarda A/G içeren) şablonlara kıyasla daha yüksek yakınlıkla (yanlarda T/G içeren) primase bağlanan DNA şablonlarında daha uzun RNA astarlarının sentezlenmiş (artan prosesivite) olduğunu göstermektedir. (C) İki grup DNA şablonu üzerinde sentezlenen RNA astarlarının miktarının ölçülmesi. Sonuçlar, DNA bağlayıcı afinite ile T7 DNA primazı ile sentezlenen RNA miktarı arasında bir korelasyon olduğunu göstermektedir. AU (rasgele birim), sentezlenmiş RNA astarlarının miktarları ile doğrudan ilişkili olan radyoaktif sinyalin yoğunluğunun ölçüsüdür. Bu rakamın daha büyük bir sürümünü görüntülemek için lütfen buraya tıklayın.
Tartışmalar
PBM yöntemi transkripsiyon faktörlerinin bağlayıcı özelliklerini araştırmak için yaygın olarak kullanılmıştır ve düşük afinite ile DNA'ya bağlanan DNA primazgibi DNA işleme enzimlerine de uygulanabilir. Ancak, deneysel prosedürlerin bazı değişiklikler gereklidir. Mikrodizi deneyi birkaç adım içerir: DNA kütüphanesinin tasarımı, çipin hazırlanması, protein hedefinin bağlanması, floresan etiketleme ve tarama. Deterjan içeren solüsyonlar ile uzun yıkamalar zayıf/geçici bağlanma modu nedeniyle DNA şablonundan proteinin parçalanmasına neden olduğundan hafif yıkama adımları çok önemlidir. Diğer kritik adımlar arasında bağlayıcı koşullar (örn. tampon bileşimi, kofactors) ve her bir enzim için optimize edilmesi gereken kuluçka süreleri yer almaktadır.
Mikrodiziden elde edilen sonuçların biyokimyasal tahlillerde doğrulanması gerekmektedir. Biyokimyasal tahliller aynı zamanda DNA bağlayıcı afinite ve enzimaktivitesi/işitselliği arasındaki korelasyon gibi DNA işleme enzimlerinin ilginç özelliklerine de ışık verir. HTPP, DNA bağlanmasının T7 DNA primazının fonksiyonel özellikleri üzerindeki etkisini gözlemlememizi sağladı. Örneğin, primazıN DNA şablonu için daha yüksek bağlayıcı yakınlık göstermesi durumunda, daha uzun RNA astarlarının (artan prosesivite) oluşumunu katalize ettiği gözlenmiştir.
Genel olarak, bu makalede sunulan yöntem hızlı, güvenilirdir ve on binlerce farklı DNA dizisi üzerindeki bağlayıcı özellikleri aynı anda hipoteze dayalı tek bir deneyde test etme olanağı sağlar. Reaksiyon karışımına diğer proteinlerin veya metal kofaktörlerin eklenmesi primasların DNA bağlayıcı özellikleri üzerindeki etkilerini hızlı ve yüksek iş lenmeli bir şekilde araştırma olanağı sunar. Öte yandan, bu yöntemin uygulanması mikrodizi slaytlar nispeten yüksek fiyat ve primase aktivite tahlilleri için radyoaktif malzeme işleme gerekli güvenlik önlemleri ile sınırlıdır.
Farklı primases hem mikrodizi bağlama koşulları ve aktivite tahlilleri için tampon koşulları değişiklikler gerektirir dikkat etmek önemlidir. Örneğin, Mycobacterium tuberculosis primase reaksiyon tampon divalent manganez ile magnezyum ikame gerektirir. Uygun olmayan metal kofaktörleri veya uygun olmayan reaksiyon tamponları primaz aktivitesinin zayıfolmasına veya DNA bağlayıcılığının azalmasına yol açabilir.
Daha önce de belirtildiği gibi, primases zayıf yakınlık ile DNA şablonları bağlamak; bu nedenle, mikrodizi bağlama deneyi sırasında nazik yıkama adımları gereklidir. Aksi takdirde, floresan olarak etiketlenmiş antikor ilavesi üzerine floresan sinyal kaybına yol açan mikrodizi slayt dışarı yıkanmış olacak.
Özetlemek gerekirse, birçok prokaryotik primases DNA bağlama özellikleri (trinükleotid DNA tanıma dizisi de dahil olmak üzere) hala kötü anlaşılmıştır, çoğunlukla mevcut yöntemlerin teknik sınırlamalar nedeniyle. HTPP, DNA tanıma sitelerinin keşfi veya şablonla ilgili diğer faktörlerin araştırılması için hızlı ve verimli bir platformu temsil eder (örn. genel nükleotit bileşimi, GC içeriği, tekrarlayan DNA dizisi elemanlarının varlığı ve simetrisi) ssDNA bağlayıcı enzimlerin bağlayıcı afinitesini ve aktivitesini etkiler. Buna ek olarak, gelecekteki uygulamalar farklı proteinlerin veya kofaktörlerin DNA bağlayıcı afinitesi, tanıma şekilleri veya primases ve diğer DNA işleme enzimlerinin fonksiyonel aktivitesi üzerindeki etkilerine yönelik olabilir.
Açıklamalar
Yazarlar çıkar çatışması olmadığını beyan etmezler.
Teşekkürler
Bu araştırma İsRAİl Bİlİm VAKFI (hibe no. 1023/18) tarafından desteklenmiştir.
Malzemeler
| Name | Company | Catalog Number | Comments |
| 40% acrylamide-bisacrylamide (19:1) solution | Merck | 1006401000 | |
| 95% formamide | Sigma-Aldrich | F9037-100ML | |
| Alexa 488-conjugated anti-his antibody | Qiagen | 35310 | |
| Ammonuium persulfate (APS) | Sigma-Aldrich | A3678-100G | |
| ATP, [α-32P] – 3000 Ci/mmol | Perkin Elmer | NEG003H250UC | |
| Boric acid, granular | Glentham Life Sciences | GE4425 | |
| Bovine Serum Albumin (BSA) | Roche | 10735094001 | |
| Bromophenol blue | Sigma-Aldrich | B0126-25G | |
| Coplin jar | |||
| Dithiothreitol (DTT) | Sigma-Aldrich | D0632-25G | |
| DNA microarray | Agilent | 4x180K (AMADID #78366) https://www.agilent.com | |
| Ethylenediaminetetraacetic acid (EDTA) | Acros Organics | AC118430010 | |
| Fujifilm FLA-5100 phosphorimager | FUJIFILM Life Science | ||
| Glass slide staining rack | Thermo Scientific | 12869995 | If several slides are used |
| Lab rotator | Thermo Scientific | 88880025 | |
| Magnesium chloride | Sigma-Aldrich | 63064-500G | |
| Microarray Hybridization Chamber | Agilent | G2534A | https://www.agilent.com/cs/library/usermanuals/Public/G2534-90004_HybridizationChamber_User.pdf |
| Microarray scanner (GenePix 4400A) | Molecular Devices | ||
| Phosphate Buffered Saline (PBS) | Sigma-Aldrich | P4417-100TAB | |
| Potassium glutamate | Alfa Aesar | A172232 | |
| Ribonucleotide Solution Mix (rNTPs) | New England BioLabs | N0466S | |
| Salmon testes DNA | Sigma-Aldrich | D1626-1G | |
| Skim milk powder | Sigma-Aldrich | 70166-500G | |
| Staining dish | Thermo Scientific | 12657696 | |
| Tetramethylethylenediamine (TEMED) | Bio-Rad | 1610800 | |
| Tris base (2-Amino-2-(hydroxymethyl)-1,3-propanediol) | Sigma-Aldrich | 93362-500G | |
| Triton X-100 | Sigma-Aldrich | X100-500ML | |
| Tween-20 | Sigma-Aldrich | P9416-50ML | |
| Urea | Sigma-Aldrich | U6504-1KG | |
| Xylene cyanol | Alfa Aesar | B21530 |
Referanslar
- Berger, M. F., Bulyk, M. L. Protein binding microarrays (PBMs) for rapid, high-throughput characterization of the sequence specificities of DNA binding proteins. Methods in Molecular Biology. 338, 245-260 (2006).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nature Protocols. 4 (3), 393-411 (2009).
- Afek, A., et al. DNA Sequence Context Controls the Binding and Processivity of the T7 DNA Primase. iScience. 2, 141-147 (2018).
- Afek, A., Schipper, J. L., Horton, J., Gordan, R., Lukatsky, D. B. Protein-DNA binding in the absence of specific base-pair recognition. Proceedings of the Nationaly Academy of Sciences of the Unitet States of America. 111 (48), 17140-17145 (2014).
- Frick, D. N., Richardson, C. C. Interaction of bacteriophage T7 gene 4 primase with its template recognition site. Journal of Biological Chemistry. 274 (50), 35889-35898 (1999).
- Tabor, S., Richardson, C. C. Template recognition sequence for RNA primer synthesis by gene 4 protein of bacteriophage T7. Proceedings of the Nationaly Academy of Sciences of the Unitet States of America. 78 (1), 205-209 (1981).
- Fried, M., Crothers, D. M. Equilibria and kinetics of lac repressor-operator interactions by polyacrylamide gel electrophoresis. Nucleic Acids Research. 9 (23), 6505-6525 (1981).
- Galas, D. J., Schmitz, A. DNAse footprinting: a simple method for the detection of protein-DNA binding specificity. Nucleic Acids Research. 5 (9), 3157-3170 (1978).
- Jost, J. P., Munch, O., Andersson, T. Study of protein-DNA interactions by surface plasmon resonance (real time kinetics). Nucleic Acids Research. 19 (10), 2788(1991).
- Bowen, B., Steinberg, J., Laemmli, U. K., Weintraub, H. The detection of DNA-binding proteins by protein blotting. Nucleic Acids Research. 8 (1), 1-20 (1980).
- Oliphant, A. R., Brandl, C. J., Struhl, K. Defining the sequence specificity of DNA-binding proteins by selecting binding sites from random-sequence oligonucleotides: analysis of yeast GCN4 protein. Molecular Cell Biology. 9 (7), 2944-2949 (1989).
- Marmorstein, R., Fitzgerald, M. X. Modulation of DNA-binding domains for sequence-specific DNA recognition. Gene. 304, 1-12 (2003).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiThis article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır