Method Article
Reconocimiento de secuencia de ADN por ADN primas mediante perfiles de primas de alto rendimiento
En este artículo
Resumen
Los experimentos de microarray de unión a proteínas (PBM) combinados con ensayos bioquímicos vinculan las propiedades lácteas y catalíticas de la primasa de ADN, una enzima que sintetiza los imprimadores de ARN en el ADN de la plantilla. Este método, designado como perfilado de primas de alto rendimiento (HTPP), se puede utilizar para revelar patrones de unión al ADN de una variedad de enzimas.
Resumen
DNA primase sintetiza imprimaciones cortas de ARN que inician la síntesis de ADN de fragmentos de Okazaki en la hebra rezagada por ADN polimerasa durante la replicación del ADN. La unión de primas procariotas similares a DnaG al ADN se produce en una secuencia de reconocimiento de trinucleótidos específica. Es un paso fundamental en la formación de fragmentos de Okazaki. Las herramientas bioquímicas convencionales que se utilizan para determinar la secuencia de reconocimiento de ADN de la primasa del ADN proporcionan sólo información limitada. Utilizando un ensayo de unión basado en microarrays de alto rendimiento y análisis bioquímicos consecutivos, se ha demostrado que 1) el contexto de unión específico (secuencias de flanqueo del sitio de reconocimiento) influye en la fuerza de unión de la primasa de ADN a su plantilla EL ADN, y 2) la unión más fuerte de la primasa al ADN produce imprimaciones de ARN más largas, lo que indica una mayor procesatividad de la enzima. Este método combina el ensayo de actividad de PBM y primase y se designa como perfilado de primas de alto rendimiento (HTPP), y permite la caracterización del reconocimiento de secuencia específica por primas aganlos en un tiempo y escalabilidad sin precedentes.
Introducción
HTPP hace uso de la tecnología de microarray de unión al ADN combinada con el análisis bioquímico(Figura 1) para identificar estadísticamente características específicas de las plantillas de ADN que afectan a la actividad enzimática de la primasa del ADN. Por lo tanto, HTPP proporciona una plataforma tecnológica que facilita un salto de conocimiento en el campo. Las herramientas clásicas utilizadas para determinar sitios de reconocimiento de primas no tienen la capacidad de producir una gran cantidad de datos, mientras que HTPP sí.
PBM es una técnica utilizada rutinariamente para determinar las preferencias de unión de los factores de transcripción al ADN1,2; sin embargo, no es adecuado para la detección de la unión débil / transitoria de proteínas al ADN. A diferencia del PBM universal que proporciona información sobre la especificidad media de unión a proteínas a todas las secuencias posibles que consisten en ocho pares de base, HTPP se basa en la biblioteca de plantillas de ADN de una sola cadena que comprende elementos de secuencia únicos. Estos elementos de secuencia de ADN implican decenas de miles de secuencias genómicas cortas (pocas decenas de bp), así como secuencias de ADN diseñadas computacionalmente enriquecidas en ciertos elementos de secuencia repetitiva de ADN presentes en el genoma, que poseen diferentes contenidos promedio de GC . Este enfoque de alto rendimiento permite determinar, de forma sistemática, cuantitativa y basada en hipótesis, las propiedades relacionadas con la secuencia que son importantes para la unión de primas y su actividad enzimática3. En particular, se ha identificado el importante vínculo entre las preferencias de unión a primas-ADN (moduladas por secuencias de ADN que flanquean sitios específicos de unión a trinucleótidos) y la procesatividad de la primas para este sistema enzimático4.
La nueva tecnología se aplicó para revisar nuestra comprensión de los sitios de reconocimiento de primas, incluso para la primasa de ADN T7 que ha sido ampliamente estudiada5. Específicamente, el reexamen de conceptos clásicos, como los sitios de reconocimiento de ADN de la primasa tensa T7 (que se determinaron hace casi cuatro décadas 6) utilizando microarray de unión a proteínas y ADN (PBM) ha dado lugar a una visión sin precedentes de las características relacionadas con la secuencia de flanqueo de estos sitios de reconocimiento3. Se esperaba que las secuencias que flanquean el sitio de reconocimiento de trinucleótidos de t7 ADN primas (5'-GTC-3') sean aleatorios. En su lugar, encontramos que las secuencias de flanqueo ricas en TG aumentan las posibilidades de que la primasa de ADN T7 sintete imprimaciones de ARN más largas que indiquen un aumento en la procesabilidad.
Otros métodos que se pueden utilizar para estudiar las propiedades de unión al ADN de las proteínas in vitro incluyen el ensayo de cambio de movilidad electroforética (EMSA)7, DNase I huella8, resonancia de plán (SPR)9, y hincha del sudoeste 10. Sin embargo, estos son métodos de bajo rendimiento sólo aplicables a la investigación de un pequeño número de secuencias de ADN. Además, la precisión y sensibilidad de algunas de estas técnicas (por ejemplo, EMSA) es baja. Por otro lado, la selección in vitro11 es una técnica que, de forma similar a PBM, puede utilizarse para la identificación de numerosas secuencias de unión. Sin embargo, las secuencias de baja afinidad generalmente se excluyen en la mayoría de las aplicaciones de selección in vitro; por lo tanto, este enfoque no es adecuado para obtener datos vinculantes comparativos para todas las secuencias disponibles. El PBM1universal,2 se utiliza principalmente para caracterizar las especificidades vinculantes de los factores de transcripción de prokaryotes y eucariotas, así como factores específicos (por ejemplo, presencia de ciertos ligandos, cofactores, etc.) que pueden afectan a esta interacción12.
HTPP expande la aplicación PBM a las enzimas de procesamiento de ADN mediante la combinación de potencia estadística de alto rendimiento sin precedentes con alta precisión para proporcionar información sobre el contexto de secuencia de enlace. Estos datos aún no se han obtenido para los primas y enzimas relacionadas (que tienen una unión débil/transitoria al ADN) debido a las limitaciones técnicas antes mencionadas de otras técnicas disponibles.
Protocolo
1. Diseño de microarray
NOTA: Las sondas de ADN representan secuencias personalizadas de 36 nucleótidos, que consisten en el sitio de reconocimiento de la primasa de ADN T7 (GTC) situada entre dos regiones de flanqueo variables, seguidas de una secuencia constante de 24 nucleótidos aunada a una diapositiva de vidrio3. Usamos un formato de microarray de 4 x 180.000, que permitía la detección de cada secuencia de ADN en seis réplicas, distribuidas aleatoriamente en la diapositiva.
-
Diseño de la biblioteca de ADN para primas con secuencias de reconocimiento conocidas
- Asegúrese de que cada secuencia esté compuesta por 60 nucleótidos. Mantenga constantes los primeros 24 nucleótidos (que se fijarán al portaobjetos de vidrio). Las regiones variables deben tener la siguiente forma general: (N)16GTC(N)17; donde N representa cualquier nucleótido deseado.
- Diseñar la región de flanqueo para abordar una cuestión científica específica (un ejemplo del diseño se presenta en el siguiente texto). Las regiones de flanqueo pueden diseñarse para contener entidades específicas, como los elementos repetidos o la simetría específica.
NOTA: Hemos creado ocho categorías de diferentes secuencias de flanqueo compuestas por dos o tres nucleótidos específicos: T y G (grupo 1); T y C (grupo 2); C y A (grupo 3); A y G (grupo 4); G, C y T (grupo 5); C, T y A (grupo 6); G, A y T (grupo 7); G, A y C (grupo 8). Se diseñaron 2.000 secuencias diferentes para cada grupo. Cada grupo tenía subgrupos de secuencias con diferentes tipos y diferentes números de repeticiones de secuencia. - Incluir un conjunto de secuencias de control negativas (que carecen del sitio de enlace específico)4. La presencia de estas secuencias permite validar la aparición de un enlace específico en el experimento.
-
Diseño de la biblioteca de ADN para primas con secuencias de reconocimiento desconocidas
- Si se desconoce la secuencia de reconocimiento, cree la biblioteca de ADN seleccionando las secuencias (con o sin características específicas mencionadas anteriormente) del genoma del organismo deseado (bacterias, hongos, etc.).
-
Diseño de la diapositiva de microarray
- Comprar diapositivas personalizadas (por ejemplo, 4x180K, AMADID #78366) de un proveedor comercial (para obtener más detalles, consulte Tabla de materiales). Ordene cada secuencia en seis réplicas, donde cada réplica debe adjuntarse a un punto seleccionado aleatoriamente en la diapositiva.
2. Experimento de unión al ADN de primas
NOTA: El día anterior (o al menos 2 h antes) del PBM, prepare la solución de bloqueo [2% p/v de leche desnatada en solución salina tampón de fosfato (PBS)] y revuelva en un agitador magnético. Antes de su uso, filtre la solución con un filtro de 0,45 M. Para detectar la unión de primas a las hebras de ADN, se deben realizar varios pasos en el siguiente orden.
- Procedimiento de bloqueo
- Humedezca previamente la corredera de microarray en un frasco de Coplin con 0.01% v/v Triton X-100 en PBS (5 min a 125 rpm en un rotador de laboratorio).
- Lave brevemente el portaobjetos de la junta (también conocido como cubreobjetos) con agua y etanol y seque con aire comprimido.
- Montar la parte inferior de la cámara de hibridación de acero (cámara PBM) con la junta hacia arriba (etiqueta comercial hacia arriba). Para obtener más información sobre el ensamblaje, consulte la Tabla de materiales.
- Solución de bloqueo de pipetas (2% p/v de leche desnatada en PBS) en cada cámara.
- Retire la diapositiva de microarray del frasco de Coplin, luego seque el lado no ADN (el ADN debe estar en el mismo lado que la etiqueta de la empresa) y los bordes usando una toallita fina. Coloque lentamente el microarray en la corredera de la junta, evitando burbujas (etiqueta de la empresa hacia abajo). Montar y apretar inmediatamente el aparato de cámara de hibridación de acero. Incubar durante 1 h a temperatura ambiente (RT).
- Llene un plato de tinción (el plato "PBS") con 800 ml de PBS fresco. Llene un segundo plato de tinción (el plato "WASH") con 800 ml de 0,5% v/v Tween-20 recién preparadoen en PBS.
- Desenrosque la cámara PBM y retire el "sándwich" de la micromatriz, teniendo cuidado de no romper el sello. Desmontarlo bajo el agua en el plato WASH colocando fórceps entre el portaobjetos y el cubreobjetos.
- Agitar el tobogán de microarray bajo el agua y transferir rápidamente a un frasco de Coplin.
- Lavar una vez con 0,1% v/v Tween-20 en PBS (5 min a 125 rpm en un rotador de laboratorio).
- Lavar una vez con 0.01% v/v Triton X-100 en PBS (2 min a 125 rpm en un rotador de laboratorio).
- Transfiera rápidamente la diapositiva a un frasco de Coplin que contenga PBS.
- Unión a proteínas
- Montar la cámara PBM como se describió anteriormente (pasos 2.1.2–2.1.3).
- Mezcla de unión a proteínas de pipeta que contiene 5 -M T7 primas de ADN, 30 mM Tris-HCl pH 7,5, 6,5 mM MgCl2, 30 mM K-glutamato, 6 mM dithiothreitol (TDT), trifosfato de ribonucleósido de 65 m (rNTP), 2% con leche desnatada, albúmina sérica bovina de 100 ng/L (BSA), 50 ng/LL el salmón prueba el ADN, y 0.02% v/v Triton X-100 (TX-100) en cada cámara de la junta deslizante (sin tocar los lados de goma y sin introducir burbujas).
- Enjuague brevemente la diapositiva del microarray sumergiéndola en la placa PBS, que elimina el exceso de detergente de la superficie de los portaobjetos. Limpie las superficies que no son de ADN de la diapositiva con una toallita fina.
- Baje lentamente la corredera de microarray (hacia abajo) sobre la corredera de la junta, teniendo cuidado de evitar fugas de una cámara a otra. Montar y apretar inmediatamente el aparato de la cámara PBM sin introducir burbujas. Si las burbujas se forman, se pueden quitar tocando suavemente la cámara de acero contra una superficie dura.
- Incubar durante 30 min a temperatura ambiente (RT).
- Fijación de anticuerpos fluorescentes
- Desenrosque la cámara PBM y retire el "sándwich" de la micromatriz, teniendo cuidado de no romper el sello. Desmontar bajo el agua en el plato WASH usando fórceps. Agitar el portaobjetos bajo el agua y transferir rápidamente a un frasco de Coplin que contiene PBS.
- Montar la cámara PBM con la corredera de la junta como se describió anteriormente (pasos 2.1.2–2.1.3).
- Agregue el anticuerpo anti-his conjugado Alexa 488 (10 ng / L en el búfer de unión) a la diapositiva de la junta.
- Enjuague brevemente la diapositiva sumergiéndola en la placa PBS como antes, luego retire la diapositiva de PBS lentamente, limpie la superficie que no sea de ADN y colóquela hacia abajo sobre la diapositiva de la junta.
- Incubar 30 minutos a RT en la oscuridad (la sonda de fluorescencia es sensible a la luz) para reducir el fotoblanqueo.
- Desmontar la cámara PBM y cubrir se encuentra como antes, retirando la cubierta bajo el agua en el plato WASH.
- Enjuague brevemente las diapositivas sumergiéndolas en la placa PBS. Seque los portaobjetos con aire comprimido. Almacenar en la oscuridad en una caja de diapositivas hasta que esté listo para escanear.
- Escaneado de la diapositiva del microarray
- Escanee el chip utilizando un escáner de microarray (consulte la Tabla de Materiales)con excitación de 495 nm y una emisión de 519 nm y recoja la intensidad media de fluorescencia.
3. Análisis de datos de microarray
NOTA: Todo el procesamiento de datos se realizó utilizando scripts escritos personalizados en MATLAB.
- Realice el procesamiento de datos PBM inicial utilizando Wilcoxon rank sum test p-value como se describió anteriormente4.
- Utilice el valor medio de la intensidad de unión para cada secuencia de ADN para su posterior análisis.
- A continuación, utilizando los valores p de ANOVA unidireccionales, compare la significación estadística de las diferencias observadas en las intensidades de unión primas-ADN obtenidas para diferentes grupos de sondas de ADN, como se explica anteriormente en la sección3del diseño de la biblioteca de ADN.
4. Síntesis de ARN dirigida por plantillas catalizada por t7 ADN primase
- Preparación del gel de poliacrilamida desnaturalizante
- Para preparar 100 ml de mezcla de gel, Combinar 62,5 ml de 40% acrilamida-bisacrilamida (19:1), 42 g de urea, 1,1 g de base Tris (2-Amino-2-(hidroximetil)-1,3-propanediol), 0,55 g de ácido bórico y 0,4 ml de 0,5 M de ácido etilendiaminetetraacetic (EDTA).
- Divida la mezcla en alícuotas de 14 ml (la cantidad necesaria para un gel de 16,5 cm x 26 cm x 0,03 cm) y manténgala protegida de la luz a 4 oC durante un máximo de 1 mes.
- Para preparar un gel de poliacrilamida desnaturalizante, añadir 4 ml de TEMED y 40 ml de persulfato de amonio de 10% p/v a 14 ml de la mezcla previamente preparada, fundirlo y dejarlo polimerizar durante al menos 2 horas a RT. El gel se puede conservar durante un día a 4oC.
- Preparación de muestras
NOTA: Mantenga los reactivos, la mezcla de reacción y las muestras sobre hielo.- Para preparar la mezcla de reacción necesaria para diez reacciones, combine 11 ml de tampón de actividad de 5x (200 mM Tris-HCl, pH a 7,5; ditiothreitol de 50 mM, glutamato de potasio de 250 mM, 50 mM De MgCl2), 5,5 ml de mezcla de 2,5 mM de trifosfato de nucleótido (NTP) y 5,5 ol de ribonucleótido radiomarcado [p. ej., ATP, (-32P) 3000 Ci/mmol].
NOTA: Todas las cantidades se han aumentado en un 10% debido a posibles errores de pipeteo. Los ribonucleótidos radiomarcados deben diluirse de acuerdo con la fuerza de la señal radiactiva (la dilución inicial suele ser 1:10 o más). - Transfiera 2 l de la mezcla de reacción a diez tubos PCR.
- Añadir 1 l de plantilla de ADN de 100 m al tubo de PCR correspondiente y girar hacia abajo.
- Añadir 2 s de 16 m de primas de ADN T7, girar hacia abajo, mezclar suavemente y girar hacia abajo de nuevo.
- Incubar la reacción a RT durante 20 min.
- Detenga la reacción añadiendo 5 l de solución de temple (95% formamida, 20 mM EDTA, 0.05% p/v xileno ciano y bromofenol azul), mezclar y girar hacia abajo.
- Para preparar la mezcla de reacción necesaria para diez reacciones, combine 11 ml de tampón de actividad de 5x (200 mM Tris-HCl, pH a 7,5; ditiothreitol de 50 mM, glutamato de potasio de 250 mM, 50 mM De MgCl2), 5,5 ml de mezcla de 2,5 mM de trifosfato de nucleótido (NTP) y 5,5 ol de ribonucleótido radiomarcado [p. ej., ATP, (-32P) 3000 Ci/mmol].
- Separación de productos de ARN por electroforesis mediante gel de poliacrilamida desnaturalizante y posterior detección de señal
- Pre-ejecutar el gel durante 1 h (10 mA, 600 V) en 1 tampón TBE (90 mM Tris-borate, 2 mM EDTA).
- Cargue hasta 2 ml de cada muestra y realice electroforesis (20 mA, 1100 V) en 1 tampón TBE (90 mM de trisborato, 2 mM EDTA).
- Secar el gel durante 2 h a 80oC bajo el vacío.
- Exponga la placa de imágenes de fósforo a gel radiactivo durante unas horas hasta la noche, dependiendo de la fuerza de la señal radiactiva.
- Registre la señal (luminiscencia fotoestimulada) utilizando fosforimager (ver Tabla de Materiales).
Resultados
Este avance tecnológico para el mapeo de los sitios de unión de primas permite la obtención de propiedades de unión al ADN que son difíciles, si no imposibles, de observar utilizando herramientas clásicas. Más importante aún, HTPP permite la revisión de la comprensión tradicional de los sitios de encuadernación de primas. Específicamente, HTPP revela especificidades de unión además de conocidas 5 '-GTC-3' secuencias de reconocimiento, que conduce a cambios en las actividades funcionales de t7 PRIMAs de ADN. A saber, se identificaron dos grupos de secuencias: secuencias de ADN de unión fuerte que contenían T/G en los flancos y secuencias de unión débil que contenían A/G en los flancos (todas las timinas en las plantillas de unión fuerte fueron reemplazadas por adeninas). No se detectó la unión de primas a las plantillas de ADN que faltaban 5'-GTC-3' dentro de su secuencia.
Los sitios de reconocimiento de ADN de primas que contenían características específicas, como los flancos ricos en T/G, aumentaron la unión entre primas y ADN hasta 10 veces, y sorprendentemente también aumentaron la longitud del ARN recién formado (hasta triplicar)(Figura 2 en la publicación anterior 3). Es importante destacar que el HTPP nos permitió observar y cuantificar la variabilidad en la longitud de imprimación en relación con la secuencia de la plantilla de ADN.
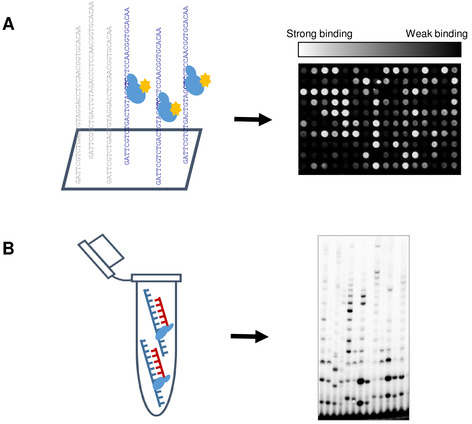
Figura 1: Representación esquemática de perfiles de primas de alto rendimiento (HTPP). (A) El portaobjetos se incuba con el primase en tampón de actividad (40 mM Tris-HCl, pH 7,5; 10 mM MgCl2, 50 mM de K-glutamato, 10 mM de TDT, 100 m de rNTPs). A continuación, Alexa 488-conjugado anticuerpo fluorescente se introdujo para etiquetar la proteína. Después del paso de lavado suave, la diapositiva fue escaneada con escáner de microarray. La afinidad de unión se determinó de acuerdo con la señal de fluorescencia mediana y las secuencias de ADN se dividieron en grupos en consecuencia. (B) Se realizaron ensayos bioquímicos para correlacionar los resultados de unión al ADN obtenidos del experimento de microarray, con las propiedades funcionales de la primasa de ADN T7. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Comparación de la actividad catalítica de la primasa de ADN T7 en dos grupos de plantillas de ADN (fuerte unión con T/G en los flancos y unión débil con A/G en los flancos) obtenida de PBM. (A) formación de imprimación de ARN catalizada por la primasa de ADN T7. Las reacciones contenían oligonucleótidos con la secuencia de reconocimiento de primas (carriles numerados), 32P-ATP, ATP, CTP, UTP y GTP en la mezcla de reacción estándar. Un grupo de oligonucleótidos de ADN (azul oscuro) contenía T/G en los flancos, mientras que todas las timinas fueron reemplazadas por adeninas en el segundo grupo (azul claro). Después de la incubación, los productos de ARN radiactivo fueron separados por electroforesis a través de un gel de poliacrilamida del 25% que contiene urea 7M y visualizados por autoradiografía. (B) Se desconoce la longitud relativa (número de ribonucleótidos que constituyen cada imprimación de ARN) y cantidad de imprimaciones de ARN sintetizadas por primasa de ADN T7 en dos grupos de plantillas de ADN (panel A). Las gráficas muestran que las imprimaciones de ARN más largas se sintetizan (mayor processividad) en plantillas de ADN que la primas se une con mayor afinidad (que contienen T/G en los flancos) en comparación con las plantillas que están unidas con menor afinidad (que contienen A/G en los flancos). (C) Cuantificación de la cantidad de imprimaciones de ARN sintetizadas en dos grupos de plantillas de ADN. Los resultados demuestran una correlación entre la afinidad de unión al ADN y la cantidad de ARN sintetizado por la primasa de ADN T7. La UA (unidad arbitraria) es la medida de la intensidad de la señal radiactiva que se correlaciona directamente con las cantidades de imprimaciones de ARN sintetizadas. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El método PBM se ha utilizado ampliamente para investigar las propiedades de unión de los factores de transcripción y también se puede aplicar a enzimas de procesamiento de ADN, como la primasa del ADN, que se unen al ADN con baja afinidad. Sin embargo, se requieren ciertas modificaciones de los procedimientos experimentales. El experimento de microarray implica varios pasos: diseño de la biblioteca de ADN, preparación del chip, unión de la diana proteica, etiquetado fluorescente y escaneo. Los pasos de lavado leves son críticos, ya que los lavados largos con soluciones que contienen detergentes causan disociación de la proteína de la plantilla de ADN debido al modo débil/transitorio de unión. Otros pasos críticos incluyen condiciones de unión (por ejemplo, composición de tampón, cofactores) y tiempos de incubación que deben optimizarse para cada enzima específica.
Los resultados obtenidos de microarray deben validarse en ensayos bioquímicos. Los ensayos bioquímicos también proporcionan información sobre características interesantes de las enzimas de procesamiento del ADN, como la correlación entre la afinidad de unión al ADN y la actividad/procesotividad enzimática. HTPP nos permitió observar el efecto de la unión al ADN en las propiedades funcionales de la primasa de ADN T7. Por ejemplo, se ha observado que si la primasa presenta una mayor afinidad de unión para la plantilla de ADN, cataliza la formación de imprimaciones de ARN más largas (mayor procesatividad).
En general, el método presentado en este artículo es rápido, confiable y proporciona la oportunidad de probar simultáneamente propiedades de unión en decenas de miles de secuencias de ADN diversas en un solo experimento de una manera basada en hipótesis. La adición de otras proteínas o cofactores metálicos a la mezcla de reacción ofrece la posibilidad de investigar su efecto sobre las propiedades de unión al ADN de las primas de una manera rápida y de alto rendimiento. Por otro lado, la aplicación de este método está limitada por un precio relativamente alto de los portaobjetos de microarray y las precauciones de seguridad necesarias al manipular material radiactivo para ensayos de actividad de primas.
Es importante tener en cuenta que diferentes primas requieren modificaciones tanto de las condiciones de unión de microarray como de las condiciones de amortiguación para los ensayos de actividad. Por ejemplo, la primasa de Mycobacterium tuberculosis requiere la sustitución de magnesio por manganeso divalente en el tampón de reacción. Los cofactores metálicos inapropiados o los tampones de reacción inapropiados pueden conducir a una mala actividad de la primasa o a una menor afinidad de unión al ADN.
Como se mencionó anteriormente, los primasse se unen a plantillas de ADN con una afinidad débil; por lo tanto, se requieren pasos de lavado suaves durante el experimento de unión a microarrays. De lo contrario, se lavarán desde el portaobjetos de microarray, lo que provocará la pérdida de la señal florescente tras la adición de anticuerpos etiquetados fluorescentemente.
En resumen, las propiedades de unión al ADN de muchos primas procaarios (incluida la secuencia de reconocimiento de ADN de trinucleótidos) todavía se entienden mal, sobre todo debido a las limitaciones técnicas de los métodos disponibles actualmente. HTPP representa una plataforma rápida y eficiente para el descubrimiento de los sitios de reconocimiento de ADN o investigaciones de otros factores relacionados con la plantilla (es decir, composición general de nucleótidos, contenido de GC, presencia de elementos de secuencia de ADN repetitivos y su simetría) que afectan la afinidad de unión y la actividad de las enzimas de unión ssDNA. Además, las aplicaciones futuras pueden orientarse hacia los efectos de diferentes proteínas o cofactores en la afinidad de unión al ADN, los patrones de reconocimiento o la actividad funcional de las primas y otras enzimas de procesamiento del ADN.
Divulgaciones
Los autores no declaran conflicto de intereses.
Agradecimientos
Esta investigación fue apoyada por la ISRAEL SCIENCE FOUNDATION (concesión No 1023/18).
Materiales
| Name | Company | Catalog Number | Comments |
| 40% acrylamide-bisacrylamide (19:1) solution | Merck | 1006401000 | |
| 95% formamide | Sigma-Aldrich | F9037-100ML | |
| Alexa 488-conjugated anti-his antibody | Qiagen | 35310 | |
| Ammonuium persulfate (APS) | Sigma-Aldrich | A3678-100G | |
| ATP, [α-32P] – 3000 Ci/mmol | Perkin Elmer | NEG003H250UC | |
| Boric acid, granular | Glentham Life Sciences | GE4425 | |
| Bovine Serum Albumin (BSA) | Roche | 10735094001 | |
| Bromophenol blue | Sigma-Aldrich | B0126-25G | |
| Coplin jar | |||
| Dithiothreitol (DTT) | Sigma-Aldrich | D0632-25G | |
| DNA microarray | Agilent | 4x180K (AMADID #78366) https://www.agilent.com | |
| Ethylenediaminetetraacetic acid (EDTA) | Acros Organics | AC118430010 | |
| Fujifilm FLA-5100 phosphorimager | FUJIFILM Life Science | ||
| Glass slide staining rack | Thermo Scientific | 12869995 | If several slides are used |
| Lab rotator | Thermo Scientific | 88880025 | |
| Magnesium chloride | Sigma-Aldrich | 63064-500G | |
| Microarray Hybridization Chamber | Agilent | G2534A | https://www.agilent.com/cs/library/usermanuals/Public/G2534-90004_HybridizationChamber_User.pdf |
| Microarray scanner (GenePix 4400A) | Molecular Devices | ||
| Phosphate Buffered Saline (PBS) | Sigma-Aldrich | P4417-100TAB | |
| Potassium glutamate | Alfa Aesar | A172232 | |
| Ribonucleotide Solution Mix (rNTPs) | New England BioLabs | N0466S | |
| Salmon testes DNA | Sigma-Aldrich | D1626-1G | |
| Skim milk powder | Sigma-Aldrich | 70166-500G | |
| Staining dish | Thermo Scientific | 12657696 | |
| Tetramethylethylenediamine (TEMED) | Bio-Rad | 1610800 | |
| Tris base (2-Amino-2-(hydroxymethyl)-1,3-propanediol) | Sigma-Aldrich | 93362-500G | |
| Triton X-100 | Sigma-Aldrich | X100-500ML | |
| Tween-20 | Sigma-Aldrich | P9416-50ML | |
| Urea | Sigma-Aldrich | U6504-1KG | |
| Xylene cyanol | Alfa Aesar | B21530 |
Referencias
- Berger, M. F., Bulyk, M. L. Protein binding microarrays (PBMs) for rapid, high-throughput characterization of the sequence specificities of DNA binding proteins. Methods in Molecular Biology. 338, 245-260 (2006).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nature Protocols. 4 (3), 393-411 (2009).
- Afek, A., et al. DNA Sequence Context Controls the Binding and Processivity of the T7 DNA Primase. iScience. 2, 141-147 (2018).
- Afek, A., Schipper, J. L., Horton, J., Gordan, R., Lukatsky, D. B. Protein-DNA binding in the absence of specific base-pair recognition. Proceedings of the Nationaly Academy of Sciences of the Unitet States of America. 111 (48), 17140-17145 (2014).
- Frick, D. N., Richardson, C. C. Interaction of bacteriophage T7 gene 4 primase with its template recognition site. Journal of Biological Chemistry. 274 (50), 35889-35898 (1999).
- Tabor, S., Richardson, C. C. Template recognition sequence for RNA primer synthesis by gene 4 protein of bacteriophage T7. Proceedings of the Nationaly Academy of Sciences of the Unitet States of America. 78 (1), 205-209 (1981).
- Fried, M., Crothers, D. M. Equilibria and kinetics of lac repressor-operator interactions by polyacrylamide gel electrophoresis. Nucleic Acids Research. 9 (23), 6505-6525 (1981).
- Galas, D. J., Schmitz, A. DNAse footprinting: a simple method for the detection of protein-DNA binding specificity. Nucleic Acids Research. 5 (9), 3157-3170 (1978).
- Jost, J. P., Munch, O., Andersson, T. Study of protein-DNA interactions by surface plasmon resonance (real time kinetics). Nucleic Acids Research. 19 (10), 2788(1991).
- Bowen, B., Steinberg, J., Laemmli, U. K., Weintraub, H. The detection of DNA-binding proteins by protein blotting. Nucleic Acids Research. 8 (1), 1-20 (1980).
- Oliphant, A. R., Brandl, C. J., Struhl, K. Defining the sequence specificity of DNA-binding proteins by selecting binding sites from random-sequence oligonucleotides: analysis of yeast GCN4 protein. Molecular Cell Biology. 9 (7), 2944-2949 (1989).
- Marmorstein, R., Fitzgerald, M. X. Modulation of DNA-binding domains for sequence-specific DNA recognition. Gene. 304, 1-12 (2003).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados