Method Article
ハイスループットプリマゼプロファイリングを用いたDNAプリマゼによるDNA配列認識
要約
タンパク質結合マイクロアレイ(PBM)実験は、生化学的アッセイと組み合わせることで、テンプレートDNA上でRNAプライマーを合成する酵素であるDNAプリマーゼの結合および触媒特性を連結します。この方法は、ハイスループットプリマゼプロファイリング(HTPP)として指定され、様々な酵素のDNA結合パターンを明らかにするために使用することができる。
要約
DNAプリマーゼは、DNA複製中にDNAポリメラーゼによって遅れ鎖上の岡崎断片のDNA合成を開始する短いRNAプライマーを合成します。DNAへの原核生物DnaG様プリマの結合は、特定のトリヌクレオチド認識配列で起こる。岡崎断片の形成において極めて重要な一歩です。DNAプリマゼのDNA認識配列を決定するために使用される従来の生化学的ツールは、限られた情報のみを提供します。ハイスループットマイクロアレイベースの結合アッセイと連続した生化学的分析を用いて、1)特定の結合コンテキスト(認識部位のフランク配列)がDNAプリマゼの結合強度に影響を及ぼすことが示された。DNA、および2)DNAへのプリマゼのより強い結合は、より長いRNAプライマーをもたらし、酵素のより高い処理性を示す。この方法は、PBMとプリマーゼ活性アッセイを組み合わせ、ハイスループットプリマーゼプロファイリング(HTPP)として指定されており、前例のない時間とスケーラビリティでDNAプリマーゼによる特異的配列認識の特性評価を可能にします。
概要
HTPPは、生化学的解析(図1)と組み合わせたDNA結合マイクロアレイ技術を利用して、DNAプリマゼの酵素活性に影響を与えるDNAテンプレートの特定の特徴を統計的に同定します。したがって、HTPPは、分野における知識の飛躍を促進する技術プラットフォームを提供します。霊長認識部位を決定するために使用される古典的なツールは、大量のデータを生成する機能を持っていませんが、HTPPはそうします。
PBMは、DNA1、2への転写因子の結合の好みを決定するために日常的に使用される技術である。しかし、DNAへのタンパク質の弱い/一過性結合の検出には適していません。8つの塩基対からなるすべての可能な配列に平均タンパク質結合特異性に関する情報を提供するユニバーサルPBMとは異なり、HTPPは、ユニークな配列要素を含む一本鎖DNAテンプレートのライブラリに基づいています。このようなDNA配列要素は、数万の短い(数十bp)ゲノム配列を含むだけでなく、異なる平均GC含有量を有するゲノム中に存在する特定のDNA反復配列要素に富む計算的に設計されたDNA配列を含む。.このようなハイスループットアプローチは、系統的、定量的、および仮説駆動的な方法で、プリマゼ結合およびその酵素活性3に重要な配列関連特性の決定を可能にする。特に、プリマーゼ-DNA結合の好みとの間の重要なリンクは、(特定の三ヌクレオチド結合部位を横切るDNA配列によって調節される)およびプリマゼ処理性がこの酵素系4について同定された。
この新技術は、広範囲に研究されているT7 DNAプリマゼについても、プリマゼ認識部位の理解を再検討するために適用された5.具体的には、タンパク質DNA結合マイクロアレイ(PBM)を用いてT7 DNAプリマーゼのDNA認識部位(ほぼ40年前に決定された)などの古典的な概念の再検討は、関連する特徴に関する前例のない洞察をもたらした。これらの認識サイト3の側面配列.T7 DNAプリマーゼ(5'-GTC-3')の三ヌクレオチド認識部位を横切る配列はランダムであると予想された。その代わりに、TGが豊富なフランク配列は、T7 DNAプリマーゼが処理性の増加を示すより長いRNAプライマーを合成する可能性を高めることがわかった。
インビトロでタンパク質のDNA結合特性を研究するために使用できる他の方法は、電気泳動性シフトアッセイ(EMSA)7、DNase Iフットプリント8、表面プラズモン共鳴(SPR)9、および南西部ブロッティングを含む10.しかし、これらは、少数のDNA配列を調査する場合にのみ適用可能な低スループット法である。さらに、これらの技術の一部の精度および感度(例えば、EMSA)は低い。一方、インビトロ選択11は、PBMと同様に、多数の結合配列の同定に使用することができる技術である。ただし、低アフィニティシーケンスは、通常、インビトロ選択のほとんどのアプリケーションで除外されます。したがって、この方法は、使用可能なすべてのシーケンスの比較バインディング データを取得するのに適していません。ユニバーサルPBM1、2は、主に原核生物および真核生物からの転写因子の結合特異性および特定の因子(例えば、特定のリガンドの存在、補因子など)を特徴付けるために使用される。この相互作用に影響を与える 12.
HTPPは、これまでにない高スループットの統計力と高精度を組み合わせることで、PBMアプリケーションをDNA処理酵素に拡大し、結合配列コンテキストに関する情報を提供します。このようなデータは、他の利用可能な技術の前述の技術的な制限のために、プリマセスおよび関連酵素(DNAに弱い/一過性結合を有する)についてまだ得られていない。
プロトコル
1. マイクロアレイの設計
注:DNAプローブは、2つの可変横面領域の間に位置するT7 DNAプリマーゼ(GTC)の認識部位からなるカスタム36ヌクレオチド配列を表し、続いてガラススライド3につながれた一定の24ヌクレオチド配列を示す。4 x 180,000 マイクロアレイ形式を使用し、スライド上でランダムに分散した 6 回の複製で各 DNA 配列をスポッティングすることを可能にしました。
-
既知の認識配列を持つプリマのDNAライブラリーの設計
- 各配列が60ヌクレオチドで構成されていることを確認します。最初の24ヌクレオチドを一定に保ちます(ガラススライドに取り付けられます)。可変領域は、次の一般的な形式を持つ必要があります: (N)16GTC(N)17;ここで、Nは任意の所望のヌクレオチドを表す。
- 特定の科学的な質問に対処するために側面領域を設計します (設計の例は、次のテキストで示されています)。側面領域は、繰り返し要素や特定の対称などの特定のフィーチャを含むように設計できます。
注:我々は、2つまたは3つの特定のヌクレオチドからなる異なる側面配列の8つのカテゴリを作成しました:TとG(グループ1)。T と C (グループ 2);C と A (グループ 3);A と G (グループ 4);G,CおよびT(グループ5);C, T および A (グループ 6);G,A,T(グループ7);G、AおよびC(グループ8)。2,000の異なるシーケンスは、グループごとに設計されました。各グループには、異なるタイプと異なるシーケンス繰り返し数を持つシーケンスのサブグループがあります。 - 負の制御シーケンスのセットを含める (特定のバインディング 部位を欠いている)4.このような配列の存在は、実験における特定の結合の発生を検証することを可能にする。
-
未知の認識配列を持つプリマのDNAライブラリーの設計
- 認識配列が不明な場合は、所望の生物(細菌、真菌など)のゲノムから配列(前述の特定の特徴の有無にかかわらず)を選択してDNAライブラリーを作成します。
-
マイクロアレイスライドの設計
- 商用サプライヤーからカスタムスライド(例えば、4x180K、AMADID#78366)を購入します(詳細は「材料の表」を参照)。各シーケンスを 6 つの反復で並べ替え、各レプリケートをスライド上のランダムに選択したスポットにアタッチする必要があります。
2. プリマセDNA結合実験
注:PBMの前日(または少なくとも2時間前)に、ブロッキング溶液[リン酸バッファー生理食液(PBS)中の2%w/vスキムミルク]を調べ、磁気攪拌機で攪拌する。使用する前に、0.45 μM フィルターでソリューションをフィルタリングします。DNA鎖に結合するプリマゼを検出するには、以下のいくつかの手順を実行する必要があります。
- ブロッキング手順
- PBSで0.01%v/vトリトンX-100(ラボローターで125rpmで5分)でコプリン瓶のマイクロアレイスライドを事前に濡らします。
- ガスケットスライド(カバースリップとも呼ばれる)を水とエタノールで簡単に洗浄し、圧縮空気を使用して乾燥させます。
- スチールハイブリダイゼーションチャンバ(PBMチャンバ)の底部を上向きにガスケットスライド(市販ラベル上向き)で組み立てます。アセンブリの詳細については、材料の表を参照してください。
- ピペットブロッキング溶液(PBSで2%w/vスキムミルク)を各チャンバーに入れます。
- コプリン瓶からマイクロアレイスライドを取り出し、非DNA側(DNAは会社のラベルと同じ側にある必要があります)とエッジを細かいワイプで乾燥させます。ガスケットスライドの上にマイクロアレイをゆっくりと置き、気泡(会社のラベルが下向き)を避けます。すぐに組み立て、鋼のハイブリダイゼーション室装置を締めます。室温(RT)で1時間インキュベートする。
- 1つの染色皿(「PBS」皿)を800 mLの新鮮なPBSで充填します。PBSで作りたての0.5%v/v Tween-20の800 mLで2番目の染色皿(「WASH」皿)を充填します。
- PBMチャンバを外し、マイクロアレイスライドカバースリップ「サンドイッチ」を取り外し、シールを壊さないのに注意してください。スライドとカバースリップの間に鉗子を置くことによってWASH皿の水中で分解する。
- マイクロアレイスライドを水中で振り、すぐにコプリン瓶に移します。
- PBSで0.1%v/v Tween-20で1回洗います(ラボローターで125rpmで5分)。
- PBSで0.01%v/vトリトンX-100で1回洗います(ラボロータで125rpmで2分)。
- スライドをPBSを含むコプリン瓶に素早く移します。
- タンパク質結合
- 前述の通りPBMチャンバを組み立てます(ステップ2.1.2~2.1.3)。
- 5 μM T7 DNAプリマーゼを含有するピペットタンパク質結合混合物、 30 mM トリス-HCl pH 7.5, 6.5 mM MgCl2,30 mM K-グルタミン酸, 6 mM ジチオトレトール (DTT), 65 μM リボヌクレオシド三リン酸 (rNTP), 2% w/v スキムミルク, 100 ng/μL ウシ腺血清 (BSA)サーモンはDNAを精巣し、0.02%v/vトリトンX-100(TX-100)をガスケットスライドの各チャンバに入れます(ゴム側に触れることなく、気泡を導入することなく)。
- マイクロアレイスライドをPBS皿に浸して短時間洗い流し、スライドの表面から余分な洗剤を取り除きます。スライドの非DNA表面を細かく拭き取ります。
- マイクロアレイスライド(下向き)をガスケットスライドにゆっくりと下げ、チャンバから別のチャンバへの漏れを防ぐように注意してください。気泡を導入することなく、すぐに組み立て、PBMチャンバー装置を締めます。気泡が形成される場合、それらは硬い表面に対して鋼室を穏やかに叩くことによって除去することができる。
- 室温(RT)で30分間インキュベートします。
- 蛍光抗体の付着
- PBMチャンバを外し、マイクロアレイスライドカバースリップ「サンドイッチ」を取り外し、シールを壊さないのに注意してください。鉗子を使用してWASH皿の水中で分解する。水中でスライドを振り、すぐにPBSを含むコプリン瓶に転送します。
- 前述のガスケットスライドでPBMチャンバを組み立てます(手順2.1.2~2.1.3)。
- Alexa 488結合抗彼抗体(結合バッファー内10ng/μL)をガスケットスライドに追加します。
- スライドを以前のようにPBS皿に浸して短時間すすいで、PBSからスライドをゆっくりと取り出し、非DNA表面を拭き取り、ガスケットスライドの上に下向きに置きます。
- 暗闇の中でRTで30分をインキュベートし(蛍光プローブは光感受性である)、光漂白を低減する。
- PBMチャンバーを分解し、以前のようにカバースリップし、WASH皿の水中でカバースリップを取り除きます。
- PBS皿に浸してスライドを簡単にすすいでください。スライドを圧縮空気で乾燥させます。スキャンの準備ができるまで、スライドボックスに暗闇の中に保存します。
- マイクロアレイスライドのスキャン
- 495 nmの励起と519 nmの発光でマイクロアレイスキャナ(材料の表を参照)を使用してチップをスキャンし、蛍光強度の中央値を収集します。
3. マイクロアレイデータ解析
注: すべてのデータ処理は、MATLAB のカスタム記述スクリプトを使用して実行されました。
- 前述の4.
- さらなる分析のために、各DNA配列の結合強度の中央値を使用します。
- 次に、一方通行のANOVAp値を用いて、DNAライブラリー設計セクション3で上述したように、DNAプローブの異なるグループに対して得られたプリマセDNA結合強度の観察された差の統計的有意性を比較する。
4. T7 DNAプリマゼにより触媒されたテンプレート指向RNA合成
- 脱窒ポリアクリルアミドゲルの調製
- ゲル混合物の100 mLを調製するには、40%アクリルアミドビサクリラム化物(19:1)の62.5mLを組み合わせ、尿素の42g、 トリス塩基の1.1g(2-アミノ-2-(ヒドロキシメチル)-1,3-プロパンジオール)、0.55gのホウ酸、0.5Mエチレンディアミンテトラセチン酸(EDTA)溶液の0.4 mL。
- 混合物を14 mLのアリコット(16.5 cm x 26 cm x 0.03 cmの1つのゲルに必要な量)に分割し、最大1ヶ月間4°Cで光から保護してください。
- 1つの回帰性ポリアクリルアミドゲルを調製するには、TEMEDの4μLと10%w/vツル酸アンモニウムの40 μLを以前に調製した混合物の14 mLに加え、それを鋳造し、RTで少なくとも2時間重合するように残します。ゲルは4°Cで1日保つことができる。
- サンプル調製
注:試薬、反応混合物、およびサンプルを氷の上に保管してください。- 10回の反応に必要な反応混合物を調製するには、5x活性バッファーの11 μL(200 mMトリス-HCl、pH = 7.5;50mMジチオスレイトール、250mMグルタミン酸カリウム、50mMMgCl2)、5.5μLの2.5mMMのヌクレオチド混合物(5.5mMM)を組み合わせて調製します。放射性標識リボヌクレオチド[例えば、ATP、(α-32P)3000 Ci/mmol]。
注:ピペッティングエラーの可能性があるため、すべての金額が10%増加しました。放射性標識リボヌクレオチドは、放射性信号の強さに応じて希釈されるべきである(初期希釈は通常1:10以上である)。 - 反応混合物の2μLを10個のPCRチューブに移す。
- 対応するPCRチューブに100 μM DNAテンプレートの1 μLを追加し、スピンダウンします。
- 16 μM T7 DNAプリマゼの2 μLを追加し、スピンダウンし、穏やかに混合し、再びスピンダウンします。
- RTで反応を20分間インキュベートします。
- 5 μLの消光溶液(95%ホルムアミド、20mM EDTA、0.05%w/vキシレンシアノールおよびブロモフェノールブルー)を加えて反応を停止し、混合し、スピンダウンする。
- 10回の反応に必要な反応混合物を調製するには、5x活性バッファーの11 μL(200 mMトリス-HCl、pH = 7.5;50mMジチオスレイトール、250mMグルタミン酸カリウム、50mMMgCl2)、5.5μLの2.5mMMのヌクレオチド混合物(5.5mMM)を組み合わせて調製します。放射性標識リボヌクレオチド[例えば、ATP、(α-32P)3000 Ci/mmol]。
- 脱電ポリアクリルアミドゲルとその後のシグナル検出による電気泳動によるRNA産物の分離
- 1x TBEバッファー(90 mMトリスボレート、2 mM EDTA)で1時間(10 mA、600V)のゲルを事前に実行します。
- 各サンプルの最大2 μLをロードし、1x TBEバッファ(90 mMトリスボレート、2 mM EDTA)で電気泳動(20 mA、1100 V)を実行します。
- 真空下で80°Cで2時間ゲルを乾燥させます。
- 蛍光体イメージングプレートを、放射性信号の強さに応じて、数時間から一晩の間、放射性ゲルにさらします。
- リンメージャーを用いて信号(光刺激発光)を記録する(材料の表を参照)。
結果
プリマゼ結合部位をマッピングするためのこの技術進歩により、古典的なツールを使用して観察することは不可能ではないにしても困難なDNA結合特性の取得を可能にします。さらに重要なことは、HTPPは、プリマゼ結合部位の伝統的な理解の再検討を可能にする。具体的には、HTPPは既知の5'-GTC-3'認識配列に加えて結合特異性を明らかにし、T7 DNAプリマゼの機能活性の変化につながる。すなわち、配列の2つのグループが同定された:側面にT/Gを含む強結合DNA配列と側面にA/Gを含む弱結合配列(強結合テンプレート内のすべてのチミンはアデニンに置き換えられた)。その配列内に5'-GTC-3'が欠けていたDNAテンプレートへのプリマゼ結合は検出されなかった。
T/Gリッチな側面などの特定の特徴を含むプリマゼDNA認識部位は、プリマセDNA結合を最大10倍まで増加させ、驚くべきことに新たに形成されたRNAの長さも増加しました(図2前の出版物)。3)重要なことに、HTPPはDNAテンプレートの配列に関連するプライマー長の変動を観察し、定量することができました。
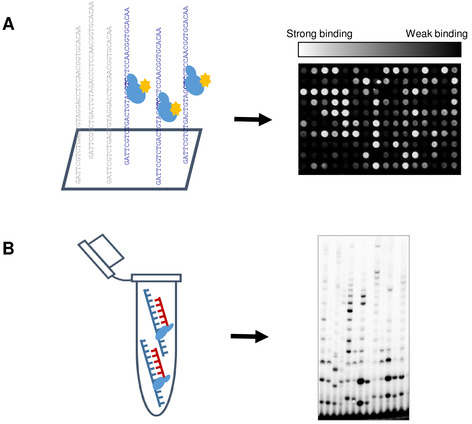
図 1: ハイスループット プリマゼ プロファイリング (HTPP) の概略表現。(A)スライドを活性バッファー内のプリマゼ(40 mMトリス-HCl、pH 7.5;10 mM MgCl2、50mM K-グルタミン酸、10mM DTT、100 μM rTPs)でインキュベートした。次に、Alexa 488-共役抗彼の蛍光抗体を導入し、タンパク質を標識した。軽度の洗浄工程の後、マイクロアレイスキャナを用いてスライドをスキャンした。結合親和性は、中央値蛍光シグナルに従って決定し、DNA配列はそれに応じてグループに分化した。(B)生化学アッセイは、マイクロアレイ実験から得られたDNA結合結果を、T7 DNAプリマゼの機能特性と相関させるために行った。この図のより大きなバージョンを表示するには、ここをクリックしてください。

図2:PBMから得られたDNAテンプレートの2つのグループ(側面におけるT/Gとの強い結合および側面におけるA/Gとの弱結合)に対するT7 DNAプリマゼの触媒活性の比較。(A)T7DNAプリマーゼにより触媒されたRNAプライマー形成。反応は、プリマゼ認識配列(番号付きレーン)を有するオリゴヌクレオチド、32P-α-ATP、ATP、CTP、UTP、およびGTPを標準反応混合物中に含有した。DNAオリゴヌクレオチド(濃い青色)の1つのグループは側面にT/Gを含み、すべてのチミンは第2群(水色)でアデニンに置き換えられた。インキュベーション後、放射性RNA産物を7M尿素を含む25%ポリアクリルアミドゲルを通して電気泳動で分離し、自動無線撮影で可視化した。(B)相対長(各RNAプライマーを構成するリボヌクレオチドの数は不明)と、DNAテンプレートの2群(パネルA)上のT7 DNAプリマーゼによって合成されたRNAプライマーの量。プロットは、より低い親和性(側面にA/Gを含む)と結合するより高い親和性(側面にT/Gを含む)とプリマーゼが結合するDNAテンプレート上で、より長いRNAプライマーが合成(処理性の増加)であることを示しています。(C)DNAテンプレートの2群に対する合成RNAプライマーの量の定量化。この結果は、T7 DNAプリマゼによるDNA結合親和性と合成RNAの量との相関関係を示す。AU(任意単位)は、合成されたRNAプライマーの量と直接相関する放射性信号の強度の尺度である。この図のより大きなバージョンを表示するには、ここをクリックしてください。
ディスカッション
このPBM法は転写因子の結合特性を調べるために広く用いられており、親和性の低いDNAに結合するDNAプリマーゼなどのDNA処理酵素にも適用できます。ただし、実験手順の特定の変更が必要です。マイクロアレイ実験には、DNAライブラリーの設計、チップの調製、タンパク質標的の結合、蛍光標識、およびスキャンといういくつかのステップが含まれます。洗剤を含む溶液を含む溶液を使用した長い洗浄は、結合の弱い/一時的なモードのためにDNAテンプレートからタンパク質の解離を引き起こすので、軽度の洗浄ステップが重要です。その他の重要なステップには、結合条件(例えば、緩衝組成物、共因子)および特定酵素ごとに最適化する必要があるインキュベーション時間が含まれる。
マイクロアレイから得られた結果は、生化学的アッセイで検証する必要があります。生化学アッセイはまた、DNA結合親和性と酵素活性/プロセス性との相関関係などのDNA処理酵素の興味深い特徴に関する洞察を提供します。HTPPにより、T7 DNAプリマゼの機能特性に対するDNA結合の効果を観察することができた。例えば、プリマーゼがDNAテンプレートに対して高い結合親和性を示す場合、より長いRNAプライマーの形成(処理性の増加)を触媒することが観察されている。
全体として、この記事で紹介する方法は高速で信頼性が高く、仮説主導の方法で単一の実験で数万の多様なDNA配列の結合特性を同時にテストする機会を提供します。反応混合物に他のタンパク質または金属対因子を加えて、プリマセスのDNA結合特性に対する影響を高速かつハイスループットの方法で調べることができます。一方、この方法の適用は、マイクロアレイスライドの比較的高い価格とプリマゼ活性アッセイの放射性物質を取り扱う際に必要な安全上の注意によって制限される。
異なるプリマセスは、活動アッセイのためのマイクロアレイ結合条件とバッファー条件の両方の変更を必要とすることに注意することが重要です。例えば、結核菌のプリマーゼは、反応バッファー内の二価マンガンとマグネシウムの置換を必要とする。不適切な金属共因子または不適切な反応バッファーは、不十分なプリマゼ活性またはDNA結合親和性の低下につながる可能性があります。
前述のように、プリマセスは親和性の弱いDNAテンプレートに結合します。したがって、マイクロアレイ結合実験では、穏やかな洗浄工程が必要です。さもなければ、それらはマイクロアレイスライドから洗い流され、蛍光標識抗体を添加すると蛍光シグナルの損失を引き起こす。
要約すると、多くの原核生物プリマ科(トリヌクレオチドDNA認識配列を含む)のDNA結合特性は、主に現在利用可能な方法の技術的な制限のために、まだ十分に理解されていません。HTPPは、DNA認識部位の発見または他のテンプレート関連因子の調査のための迅速かつ効率的なプラットフォームを表す(すなわち、全体的なヌクレオチド組成、GC含有量、反復的なDNA配列要素とその対称性)。ssDNA結合酵素の結合親和性および活性に影響を与える。さらに、将来の応用は、DNA結合親和性、認識パターン、またはプリマセスおよび他のDNA処理酵素の機能活性に対する異なるタンパク質または共因子の影響に向けられる可能性がある。
開示事項
著者は利益相反を宣言しない。
謝辞
本研究はイスラエル科学財団(助成第1023/18号)の支援を受けています。
資料
| Name | Company | Catalog Number | Comments |
| 40% acrylamide-bisacrylamide (19:1) solution | Merck | 1006401000 | |
| 95% formamide | Sigma-Aldrich | F9037-100ML | |
| Alexa 488-conjugated anti-his antibody | Qiagen | 35310 | |
| Ammonuium persulfate (APS) | Sigma-Aldrich | A3678-100G | |
| ATP, [α-32P] – 3000 Ci/mmol | Perkin Elmer | NEG003H250UC | |
| Boric acid, granular | Glentham Life Sciences | GE4425 | |
| Bovine Serum Albumin (BSA) | Roche | 10735094001 | |
| Bromophenol blue | Sigma-Aldrich | B0126-25G | |
| Coplin jar | |||
| Dithiothreitol (DTT) | Sigma-Aldrich | D0632-25G | |
| DNA microarray | Agilent | 4x180K (AMADID #78366) https://www.agilent.com | |
| Ethylenediaminetetraacetic acid (EDTA) | Acros Organics | AC118430010 | |
| Fujifilm FLA-5100 phosphorimager | FUJIFILM Life Science | ||
| Glass slide staining rack | Thermo Scientific | 12869995 | If several slides are used |
| Lab rotator | Thermo Scientific | 88880025 | |
| Magnesium chloride | Sigma-Aldrich | 63064-500G | |
| Microarray Hybridization Chamber | Agilent | G2534A | https://www.agilent.com/cs/library/usermanuals/Public/G2534-90004_HybridizationChamber_User.pdf |
| Microarray scanner (GenePix 4400A) | Molecular Devices | ||
| Phosphate Buffered Saline (PBS) | Sigma-Aldrich | P4417-100TAB | |
| Potassium glutamate | Alfa Aesar | A172232 | |
| Ribonucleotide Solution Mix (rNTPs) | New England BioLabs | N0466S | |
| Salmon testes DNA | Sigma-Aldrich | D1626-1G | |
| Skim milk powder | Sigma-Aldrich | 70166-500G | |
| Staining dish | Thermo Scientific | 12657696 | |
| Tetramethylethylenediamine (TEMED) | Bio-Rad | 1610800 | |
| Tris base (2-Amino-2-(hydroxymethyl)-1,3-propanediol) | Sigma-Aldrich | 93362-500G | |
| Triton X-100 | Sigma-Aldrich | X100-500ML | |
| Tween-20 | Sigma-Aldrich | P9416-50ML | |
| Urea | Sigma-Aldrich | U6504-1KG | |
| Xylene cyanol | Alfa Aesar | B21530 |
参考文献
- Berger, M. F., Bulyk, M. L. Protein binding microarrays (PBMs) for rapid, high-throughput characterization of the sequence specificities of DNA binding proteins. Methods in Molecular Biology. 338, 245-260 (2006).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nature Protocols. 4 (3), 393-411 (2009).
- Afek, A., et al. DNA Sequence Context Controls the Binding and Processivity of the T7 DNA Primase. iScience. 2, 141-147 (2018).
- Afek, A., Schipper, J. L., Horton, J., Gordan, R., Lukatsky, D. B. Protein-DNA binding in the absence of specific base-pair recognition. Proceedings of the Nationaly Academy of Sciences of the Unitet States of America. 111 (48), 17140-17145 (2014).
- Frick, D. N., Richardson, C. C. Interaction of bacteriophage T7 gene 4 primase with its template recognition site. Journal of Biological Chemistry. 274 (50), 35889-35898 (1999).
- Tabor, S., Richardson, C. C. Template recognition sequence for RNA primer synthesis by gene 4 protein of bacteriophage T7. Proceedings of the Nationaly Academy of Sciences of the Unitet States of America. 78 (1), 205-209 (1981).
- Fried, M., Crothers, D. M. Equilibria and kinetics of lac repressor-operator interactions by polyacrylamide gel electrophoresis. Nucleic Acids Research. 9 (23), 6505-6525 (1981).
- Galas, D. J., Schmitz, A. DNAse footprinting: a simple method for the detection of protein-DNA binding specificity. Nucleic Acids Research. 5 (9), 3157-3170 (1978).
- Jost, J. P., Munch, O., Andersson, T. Study of protein-DNA interactions by surface plasmon resonance (real time kinetics). Nucleic Acids Research. 19 (10), 2788(1991).
- Bowen, B., Steinberg, J., Laemmli, U. K., Weintraub, H. The detection of DNA-binding proteins by protein blotting. Nucleic Acids Research. 8 (1), 1-20 (1980).
- Oliphant, A. R., Brandl, C. J., Struhl, K. Defining the sequence specificity of DNA-binding proteins by selecting binding sites from random-sequence oligonucleotides: analysis of yeast GCN4 protein. Molecular Cell Biology. 9 (7), 2944-2949 (1989).
- Marmorstein, R., Fitzgerald, M. X. Modulation of DNA-binding domains for sequence-specific DNA recognition. Gene. 304, 1-12 (2003).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved