Method Article
Распознавание последовательности ДНК с помощью высокопроходимых профилирований primase
В этой статье
Резюме
Эксперименты по связыванию белков (PBM) в сочетании с биохимическими анализами связывают связывающие и каталитические свойства примаза ДНК, фермента, который синтезирует РНК-праймеры на шаблоне ДНК. Этот метод, обозначенный как высокопроизводительный профилирование примаза (HTPP), может быть использован для выявления ДНК-связывающих моделей различных ферментов.
Аннотация
Прима засовка ДНК синтезирует короткие РНК-праймеры, которые инициируют синтез фрагментов ДНК фрагментов Окадзаки на отстающей нити путем полимераза ДНК во время репликации ДНК. Привязка прокариотических примаз, похожих на днк, происходит в определенной последовательности распознавания тринуклеотидов. Это ключевой шаг в формировании фрагментов Окадзаки. Обычные биохимические инструменты, которые используются для определения последовательности распознавания ДНК примаза ДНК, предоставляют лишь ограниченную информацию. Использование высокой пропускной способности микроаррей на основе связывания анализа и последовательных биохимических анализов, было показано, что 1) конкретный обязательный контекст (фланговые последовательности сайта распознавания) влияет на силу прима ДНК к его шаблону ДНК, и 2) сильнее связывания примаза к ДНК дает больше РНК праймеров, что свидетельствует о более высокой processivity фермента. Этот метод сочетает в себе PBM и primase деятельности анализа и обозначен как высокой пропускной способности примаза профилирования (HTPP), и это позволяет характеристика конкретной последовательности распознавания примаза ДНК в беспрецедентное время и масштабируемости.
Введение
HTPP использует технологию связывания МИКРОаррей ДНК в сочетании с биохимическим анализом(рисунок 1) для статистическиго определения специфических особенностей шаблонов ДНК, влияющих на ферментативную активность примазы ДНК. Таким образом, HTPP обеспечивает технологическую платформу, которая облегчает скачок знаний в этой области. Классические инструменты, используемые для определения сайтов распознавания приносов, не имеют возможности давать огромное количество данных, в то время как HTPP делает.
PBM метод, обычно используемый для определения обязательных предпочтений транскрипционных факторов к ДНК1,2; однако, он не подходит для обнаружения слабого/переходного связывания белков к ДНК. В отличие от универсального PBM, который предоставляет информацию о средней специфичности связывания белка ко всем возможным последовательностям, состоящим из восьми базовых пар, HTPP основан на библиотеке одноцепочечных шаблонов ДНК, включающих уникальные элементы последовательности. Такие элементы последовательности ДНК включают десятки тысяч коротких (несколько десятков вт) геномных последовательностей, а также вычислительно разработанные последовательности ДНК, обогащенные определенными репетитными элементами последовательности ДНК, присутствующими в геноме, которые обладают разным средним содержанием GC . Такой высокопроизводительный подход позволяет определить, в систематическом, количественных и гипотезы инициативе образом, последовательности, связанные свойства, которые важны для примкассвязи и его ферментативной деятельности3. В частности, для этой ферментативной системы4была определена важная связь между примазно-ДНК связывающими предпочтениями (модулированными последовательностями ДНК, обрамляющими конкретные тринуклеотидные связывающие участки) и процессивой примазы.
Новая технология была применена, чтобы вернуться к нашему пониманию сайтов признания примаза даже для прима T7 ДНК, которая была широко изучена5. В частности, пересмотр классических концепций, таких как сайты распознавания ДНК примаза T7 DNA (которые были определены почти четыре десятилетия назад 6)с использованием белково-ДНК связывающего микроаррей (PBM) привело к беспрецедентному пониманию особенностей, связанных с фланговой последовательности этих сайтов распознавания3. Ожидалось, что последовательности фланговых трехнуклеотидных признания сайта прима задневок T7 (5'-GTC-3') будут случайными. Вместо этого, мы обнаружили, что TG богатых фланговых последовательностей увеличить шансы T7 ДНК прима синтезировать больше РНК праймеров, указывающих на увеличение процессоцивистии.
Другие методы, которые могут быть использованы для изучения ДНК-связывающих свойств белков in vitro включают электрофоретический перекос анализа (EMSA)7, DNase Iслед8, поверхностно-плазмон резонанс (SPR)9, и юго-западной blotting 10. Это, однако, методы низкой пропускной необходимости применимы только к исследованию небольшого числа последовательностей ДНК. Кроме того, точность и чувствительность некоторых из этих методов (например, EMSA) является низкой. С другой стороны, in vitro selection11 является методом, который, подобно PBM, может быть использован для идентификации многочисленных связывающих последовательностей. Тем не менее, низкие последовательности сродства, как правило, исключены в большинстве приложений в пробирке выбор; поэтому этот подход не подходит для получения сравнительных обязательных данных по всем имеющимся последовательностям. Универсальный PBM1,2 в основном используется для характеристики связывающей специфики транскрипционных факторов от прокариот и эукариот, а также специфические факторы (например, наличие определенных лигандов, кофакторов и т.д.), которые могут повлиять на это взаимодействие12.
HTPP расширяет применение PBM для ферментов обработки ДНК, сочетая беспрецедентную высокопроизводительную статистическую мощность с высокой точностью, чтобы предоставить информацию о контексте связывающей последовательности. Такие данные еще не получены по примазам и связанным с ними ферментам (которые имеют слабую/переходную связующую к ДНК) из-за вышеупомянутых технических ограничений других доступных методов.
протокол
1. Дизайн микроаррей
ПРИМЕЧАНИЕ: ДНК-зонды представляют обычай 36-нуклеотидных последовательностей, состоящий из места распознавания для T7 ДНК примазы (GTC), расположенных между двумя переменными фланговых регионах, а затем постоянная 24-нуклеотидной последовательности привязывается к стеклянной слайд3. Мы использовали формат microarray 4 x 180 000, который позволял обнаруживать каждую последовательность ДНК в шести репликациях, случайным образом распределенными на слайде.
-
Дизайн библиотеки ДНК для примас с известными последовательностями распознавания
- Убедитесь, что каждая последовательность состоит из 60 нуклеотидов. Держите первые 24 нуклеотидов постоянными (должны быть прикреплены к стеклянной горке). Переменные области должны иметь следующую общую форму: (N)16GTC (N)17; где N представляет любой желаемый нуклеотид.
- Дизайн фланговой области для решения конкретного научного вопроса (пример дизайна представлен в следующем тексте). Области фланга могут быть разработаны таким образом, чтобы содержать определенные объекты, такие как повторяющиеся элементы или специфическая симметрия.
ПРИМЕЧАНИЕ: Мы создали восемь категорий различных фланговых последовательностей, состоящих из двух или трех конкретных нуклеотидов: T и G (группа 1); T и C (группа 2); C и A (группа 3); A и G (группа 4); G, C и T (группа 5); C, T и A (группа 6); G, A и T (группа 7); G, A и C (группа 8). Для каждой группы было разработано 2000 различных последовательностей. Каждая группа имела подгруппы последовательностей с различными типами и различными числами повторов последовательности. - Включите набор отрицательных последовательностей управления (отсутствие конкретного сайта связывания)4. Наличие таких последовательностей позволяет проверить возникновение специфической привязки в эксперименте.
-
Дизайн библиотеки ДНК для примас с неизвестными последовательностями распознавания
- Если последовательность распознавания неизвестна, создайте библиотеку ДНК, выбрав последовательности (с упомянутыми ранее или без нее) из генома желаемого организма (бактерии, грибки и т.д.).
-
Дизайн слайда microarray
- Приобретите пользовательские слайды (например, 4x180K, AMADID #78366) у коммерческого поставщика (для получения более подробной информации см. Таблица материалов). Закажите каждую последовательность в шести репликациях, где каждая реплика должна быть прикреплена к случайно выбранному месту на слайде.
2. Primase ДНК связывания эксперимент
ПРИМЕЧАНИЕ: За день до (или по крайней мере 2 ч до) PBM, подготовить блокирующий раствор »2% w/v обезжиренное молоко в фосфатном буфере солевая (PBS)) и перемешать его на магнитном мешалке. Перед использованием отфильтруй терешения фильтром 0,45 мкм. Чтобы обнаружить привязку примя к нитей ДНК, следует выполнить несколько шагов в следующем порядке.
- Процедура блокировки
- Предварительно влажный microarray слайд в банку Коплин с 0,01% V / V Triton X-100 в PBS (5 мин при 125 об/мин на лабораторном ротаторе).
- Кратко промыть прокладку слайд (также называемый coverslip) с водой и этанолом и высушить с помощью сжатого воздуха.
- Соберите нижнюю часть камеры гибридизации стали (PBM камеры) с прокладкой слайд вверх (коммерческая этикетка вверх). Для получения более подробной информации о сборке, обратитесь к таблице материалов.
- Трубопровод блокирующий раствор (2% w/v обезжиренное молоко в PBS) в каждую камеру.
- Удалите microarray слайд из банка Коплин, а затем высушить не-ДНК стороне (ДНК должна быть на той же стороне, как этикетка компании) и края с помощью тонкой салфетки. Медленно поместите microarray на слайд прокладки, избегая пузырьков (компания этикетки вниз). Немедленно соберите и затяните стальной гибридизацию камерного аппарата. Инкубировать 1 ч при комнатной температуре (RT).
- Заполните одно окрашивание блюдо ("PBS" блюдо) с 800 мл свежего PBS. Заполните второе блюдо для окрашивания (блюдо "WASH") с 800 мл свежеприготовленного 0,5% v/v Tween-20 в PBS.
- Отвиньте камеру PBM и удалите microarray слайд-обложку "сэндвич", заботясь, чтобы не сломать печать. Разобрать его под водой в тарелке WASH, поместив щипчинки между горкой и крышкой.
- Встряхните microarray слайд под водой и быстро перейти к банку Коплин.
- Вымойте один раз с 0,1% v/v Tween-20 в PBS (5 мин при 125 об/мин на лабораторном ротаторе).
- Вымойте один раз с 0,01% V / V Triton X-100 в PBS (2 мин при 125 об/мин на лабораторном ротаторе).
- Быстро перенесите слайд в банку Coplin, содержащую PBS.
- Связывание белка
- Соберите камеру PBM, как описано ранее (шаги 2.1.2-2.1.3).
- Пипетка белка связывающая смесь, содержащая 5 мкм T7 ДНК примаза, 30 мМ Tris-HCl pH 7.5, 6.5 mM MgCl2,30 мМ K-глютамат, 6 мМ дитиотрейтол (DTT), 65 мкм ribonucleoside трифосфат (rNTP), 2% w/vskim milk, 100 нг/L зубчатый сывороточный альбом (BSA), 50 ng лосось яички ДНК, и 0,02% v/v Triton X-100 (TX-100) в каждой камере слайда прокладки (без касания резиновых сторон и без введения пузырьков).
- Промыть microarray слайд кратко, окунув его в блюдо PBS, который удаляет избыток моющего средства с поверхности слайдов. Протрите не-ДНК поверхностей слайда с тонкой салфеткой.
- Медленно опустите слайд microarray (лицом вниз) на слайд прокладки, стараясь предотвратить утечку из одной камеры в другую. Немедленно соберите и затяните аппарат PBM камеры без введения пузырей. Если пузырьки образуются, они могут быть удалены, мягко нажав стальной камеры на твердую поверхность.
- Инкубировать в течение 30 минут при комнатной температуре (RT).
- Флуоресцентные антитела крепления
- Отвиньте камеру PBM и удалите microarray слайд-обложку "сэндвич", заботясь, чтобы не сломать печать. Разобрать под водой в тарелке WASH с помощью щипков. Встряхните слайд под водой и быстро перенесите в банку Коплин, содержащую PBS.
- Соберите камеру PBM с слайдом прокладки, как описано ранее (шаги 2.1.2-2.1.3).
- Добавьте Alexa 488-конъюгированное анти-его антитело (10 нг/л в связывающем буфере) к слайду прокладки.
- Промыть слайд кратко, окунув его в блюдо PBS, как и раньше, а затем удалить слайд из PBS медленно, протрите не-ДНК поверхности и поместить его лицом вниз на прокладку слайд.
- Инкубировать 30 мин на RT в темноте (флуоресценция зонд светочувствительных), чтобы уменьшить фото-отбеливания.
- Разобрать камеру PBM и coverslip, как и раньше, удалив крышку под водой в тарелке WASH.
- Промыть слайды кратко, окунув их в блюдо PBS. Сухие горки со сжатым воздухом. Хранить в темноте в слайд-поле до готовности к сканированию.
- Сканирование слайда microarray
- Сканирование чипа с помощью сканера microarray (относится к таблице материалов)с возбуждением 495 нм и выбросом 519 нм и собирать среднюю интенсивность флуоресценции.
3. Анализ данных Microarray
ПРИМЕЧАНИЕ: Вся обработка данных была выполнена с использованием пользовательских письменных скриптов в MATLAB.
- Выполните первоначальную обработку данных PBM с помощью уилкоксона ранга сумма тест р-значение, как описано ранее4.
- Используйте медианное значение интенсивности связывания для каждой последовательности ДНК для дальнейшего анализа.
- Далее, используя односторонние aNOVA p-значения, сравните статистическую значимость наблюдаемых различий в прима-ДНК связывающей интенсивности, полученных для различных групп зондов ДНК, как указано выше в разделе дизайна библиотеки ДНК3.
4. Шаблон-направленный синтез РНК катализируется примаза ДНК T7
- Приготовление денатурного полиакриламидного геля
- Для приготовления 100 мл гелевой смеси смешайте 62,5 мл 40% акриламида-бисакриламидада (19:1), 42 г мочевины, 1,1 г основания Трис (2-амино-2--(гидроксиметил)-1,3-пропанедиол), 0,55 г борной кислоты и 0,4 мл 0,5 М этиленденаминтетраацетической кислоты (ЭДТА).
- Разделите смесь на 14 мл aliquots (количество, необходимое для одного геля 16,5 см х 26 см х 0,03 см) и держать их защищены от света при 4 градусах По Цельсию в течение 1 месяца.
- Чтобы подготовить один денатурный гель полиакриламид, добавьте 4 злte TEMED и 40 л 10% w/v амгармония persulfate к 14 мл ранее приготовленной смеси, отлить его, и оставить его полимеризировать, по крайней мере 2 ч на RT. Гель можно хранить в течение одного дня при 4 градусах Цельсия.
- Подготовка образцов
ПРИМЕЧАНИЕ: Храните реагенты, реакционную смесь и образцы на льду.- Для подготовки реакционной смеси, необходимой для десяти реакций, комбинируйте 11 qL 5x-буфер активности (200 мм Tris-HCl, рН 7,5; 50 ММ дитиотрейтол, 250 мм глутамата калия, 50 мМ МГКл2),5,5 мл л из 2,5 мМ смеси нуклеотида трифосфата (NTNt.phosp. радиомаркированный рибонуклеотид (например, АТФ, (З-32P) 3000 Ci/mmol.
ПРИМЕЧАНИЕ: Все суммы были увеличены на 10% из-за возможных ошибок трубач. Радиомаркированные рибонуклеотиды следует разбавлять в зависимости от силы радиоактивного сигнала (первоначальное разбавление обычно 1:10 или более). - Передача 2 ЗЛ реакционной смеси в десять пЦР-труб.
- Добавьте 1 юл из 100 шаблонов ДНК мКм в соответствующую трубку ПЦР и раскрутитесь вниз.
- Добавьте 2 зЛ из 16 прима ДНК T7, вращайтесь, аккуратно перемешайте и снова вращайтесь.
- Инкубировать реакцию на RT в течение 20 мин.
- Остановите реакцию, добавив 5 зЛ раствора для закалки (95% формамида, 20 мМ ЭДТА, 0,05% ж/в ксилена цианола и бромофенола синего), перемешайте и раскрутите вниз.
- Для подготовки реакционной смеси, необходимой для десяти реакций, комбинируйте 11 qL 5x-буфер активности (200 мм Tris-HCl, рН 7,5; 50 ММ дитиотрейтол, 250 мм глутамата калия, 50 мМ МГКл2),5,5 мл л из 2,5 мМ смеси нуклеотида трифосфата (NTNt.phosp. радиомаркированный рибонуклеотид (например, АТФ, (З-32P) 3000 Ci/mmol.
- Разделение РНК-продуктов электрофорезом через денатурирующий полиакриламидный гель и последующее обнаружение сигнала
- Предварительно запустить гель на 1 ч (10 мА, 600 В) в буфере 1x TBE (90 мм Трис-борат, 2 мМ EDTA).
- Загрузите до 2 кл/с каждого образца и выполните электрофорекс (20 мА, 1100 В) в буфере 1x TBE (90 мм Трис-борат, 2 мМ ЭДТА).
- Высушите гель в течение 2 ч при 80 градусах Цельсия под вакуумом.
- Выставить фосфорную пластину для радиоактивного геля в течение нескольких часов до ночи, в зависимости от силы радиоактивного сигнала.
- Запись сигнала (фотостимулированное люминесценция) с помощью фосфорайгера (см. Таблица материалов).
Результаты
Этот технологический прогресс для отображения привязки примоза позволяет получить свойства связывания ДНК, которые трудно, если не невозможно, наблюдать с помощью классических инструментов. Что еще более важно, HTPP позволяет пересмотреть традиционное понимание примяза обязательной сайтов. В частности, HTPP раскрывает обязательные особенности в дополнение к известным последовательностям распознавания 5'-GTC-3, что приводит к изменениям в функциональной деятельности примаза ДНК T7. А именно, были определены две группы последовательностей: сильно обязательные последовательности ДНК, которые содержали T/G на флангах, и слабосвязывающие последовательности, содержащие ся/Г на флангах (все тимины в сильно обязательных шаблонах были заменены аденинами). Не было обнаружено привязки примазы к шаблонам ДНК, которые отсутствовали 5'-GTC-3' в их последовательности.
Сайты распознавания днк примаза, содержащие специфические особенности, такие как T/G-богатые фланги, увеличенные прима-ДНК связывания до 10 раз, и удивительно также увеличили длину вновь сформированной РНК (до трех раз) (Рисунок 2 в предыдущей публикации 3), Важно, ЧТО HTPP позволило нам наблюдать и количественно изменчивость длины грунтовки по отношению к последовательности шаблона ДНК.
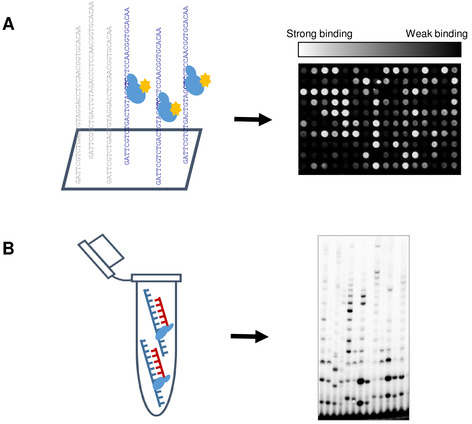
Рисунок 1: Схематическое представление профилирования примаза высокой пропускной силы (HTPP). ()Слайд был инкубирован с примазой в буфере активности (40 мм Tris-HCl, рН 7.5; 10 mM MgCl2, 50 мМ K-глутамат, 10 мм DTT, 100 мкм rNTPs). Далее, Alexa 488-конъюгированных анти-его флуоресцентные антитела был введен для обозначения белка. После мягкой стирки слайд был отсканирован с помощью сканера microarray. Связывание сродства было определено в соответствии со средним сигналом флуоресценции и последовательности ДНК были разделены на группы соответственно. (B) Биохимические анализы были выполнены для корреляции ДНК-обязательных результатов, полученных в результате эксперимента microarray, с функциональными свойствами примаза ДНК T7. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.

Рисунок 2: Сравнение каталитической активности примаза ДНК T7 на двух группах шаблонов ДНК (сильная связывание с Т/Г на флангах и слабая связывание с A/G на флангах), полученная из PBM. (A) ОБРАЗОВАНИЕ РНК грунтовки катализируется примаза ДНК T7. Реакции содержали олигонуклеотиды с последовательность распознавания примазы (число полос движения), 32P-я-AtP, ATP, CTP, UTP и GTP в стандартной смеси реакции. Одна группа ДНК-олигонуклеотидов (темно-синий) содержала На флангах Т/Г, в то время как все тимины были заменены аденинами во второй группе (светло-голубой). После инкубации радиоактивные РНК-продукты были разделены электрофорезом через 25% полиакриламидный гель, содержащий 7М мочевины и визуализированный с помощью авторрадиографии. (B) Относительная длина (количество рибонуклеотидов, составляющих каждый праймер РНК неизвестно) и количество РНК праймеры синтезируются T7 ДНК примазы на двух группах шаблонов ДНК (панель A). Сюжеты показывают, что более длинные РНК-праймеры синтезируются (повышенная процессуальность) на шаблонах ДНК, которые primase связывает с более высоким сродством (которые содержат T/G на флангах) по сравнению с шаблонами, которые связаны с более низкой сродством (которые содержат A/G на флангах). (C) Количественная оценка количества синтезированных РНК-праймеров на двух группах шаблонов ДНК. Результаты демонстрируют корреляцию между ДНК-связывающей близостью и количеством синтезированной РНК приманой ДНК T7. АС (произвольный блок) является мерой интенсивности радиоактивного сигнала, которая напрямую коррелирует с количеством синтезированных РНК-праймеров. Пожалуйста, нажмите здесь, чтобы просмотреть большую версию этой цифры.
Обсуждение
Метод PBM широко используется для исследования связывающих свойств транскрипционных факторов, а также может быть применен к ферментам обработки ДНК, таким как прима закваска ДНК, которые связываются с ДНК с низким сродством. Однако необходимы определенные изменения экспериментальных процедур. Эксперимент на микроарле включает в себя несколько этапов: проектирование библиотеки ДНК, приготовление чипа, связывание белковой мишени, флуоресцентную маркировку и сканирование. Мягкие шаги мытья критически, в виду того что длинние моет с разрешениями содержа моющие средства причиняют разъединение протеина от шаблона дна из-за слабого/переходного режима связывать. Другие критические шаги включают обязательные условия (например, состав буфера, кофакторы) и время инкубации, которые необходимо оптимизировать для каждого конкретного фермента.
Результаты, полученные с помощью микроаррей, должны быть проверены в биохимических анализах. Биохимические анализы также дают представление об интересных особенностях ферментов обработки ДНК, таких как корреляция между сродством связывания ДНК и ферментативной активностью/процессивой. HTPP позволил нам наблюдать влияние связывания ДНК на функциональные свойства примаза ДНК T7. Например, было отмечено, что если прима проявляет более высокую связующую сродство к шаблону ДНК, она катализует образование более длинных РНК-праймеров (повышенная процессуальность).
В целом, метод, представленный в этой статье, является быстрым, надежным и дает возможность одновременно проверить связывающие свойства на десятках тысяч различных последовательностей ДНК в одном эксперименте в основе гипотезы. Добавление других белков или металлических кофакторов в реакционную смесь дает возможность быстро, высокой пропускной связи исследовать их влияние на связывающие свойства примиз. С другой стороны, применение этого метода ограничено относительно высокой ценой слайдов микроаррей и мерами предосторожности, необходимыми при обработке радиоактивных материалов для анализов деятельности примаза.
Важно отметить, что различные прима требуют изменения как условий связывания микроаррей, так и условий буфера для анализов деятельности. Например, прима микобактерий туберкулеза требует замены магния с divalent марганца в буфере реакции. Неподходящие металлические кофакторы или ненадлежащие буферы реакции могут привести к плохой активности примазы или снижению сродства связывания ДНК.
Как упоминалось ранее, прима связаны с шаблонами ДНК со слабым сродством; поэтому, нежные шаги мытья необходимы во время эксперимента связывания microarray. В противном случае, они будут вымыты из microarray слайд, что приводит к потере флуоресцентного сигнала при добавлении флуоресцентно помечены антитела.
Подводя итог, можно сказать, что свойства связывания ДНК многих прокариотических примаза (включая последовательность распознавания ДНК тринуклеотида) до сих пор плохо изучены, главным образом из-за технических ограничений имеющихся в настоящее время методов. HTPP представляет собой быструю и эффективную платформу для открытия сайтов распознавания ДНК или исследований других факторов, связанных с шаблонами (т.е. общего состава нуклеотидов, содержания GC, наличия повторяющихся элементов последовательности ДНК и их симметрии) которые влияют на связывающее сродство и активность связывающих ферментов ssDNA. Кроме того, будущие приложения могут быть направлены на воздействие различных белков или кофакторов на сродство связывания ДНК, модели распознавания или функциональную активность примас и других ферментов обработки ДНК.
Раскрытие информации
Авторы не заявляют о конфликте интересов.
Благодарности
Это исследование было поддержано ISRAEL SCIENCE FOUNDATION (грант No 1023/18).
Материалы
| Name | Company | Catalog Number | Comments |
| 40% acrylamide-bisacrylamide (19:1) solution | Merck | 1006401000 | |
| 95% formamide | Sigma-Aldrich | F9037-100ML | |
| Alexa 488-conjugated anti-his antibody | Qiagen | 35310 | |
| Ammonuium persulfate (APS) | Sigma-Aldrich | A3678-100G | |
| ATP, [α-32P] – 3000 Ci/mmol | Perkin Elmer | NEG003H250UC | |
| Boric acid, granular | Glentham Life Sciences | GE4425 | |
| Bovine Serum Albumin (BSA) | Roche | 10735094001 | |
| Bromophenol blue | Sigma-Aldrich | B0126-25G | |
| Coplin jar | |||
| Dithiothreitol (DTT) | Sigma-Aldrich | D0632-25G | |
| DNA microarray | Agilent | 4x180K (AMADID #78366) https://www.agilent.com | |
| Ethylenediaminetetraacetic acid (EDTA) | Acros Organics | AC118430010 | |
| Fujifilm FLA-5100 phosphorimager | FUJIFILM Life Science | ||
| Glass slide staining rack | Thermo Scientific | 12869995 | If several slides are used |
| Lab rotator | Thermo Scientific | 88880025 | |
| Magnesium chloride | Sigma-Aldrich | 63064-500G | |
| Microarray Hybridization Chamber | Agilent | G2534A | https://www.agilent.com/cs/library/usermanuals/Public/G2534-90004_HybridizationChamber_User.pdf |
| Microarray scanner (GenePix 4400A) | Molecular Devices | ||
| Phosphate Buffered Saline (PBS) | Sigma-Aldrich | P4417-100TAB | |
| Potassium glutamate | Alfa Aesar | A172232 | |
| Ribonucleotide Solution Mix (rNTPs) | New England BioLabs | N0466S | |
| Salmon testes DNA | Sigma-Aldrich | D1626-1G | |
| Skim milk powder | Sigma-Aldrich | 70166-500G | |
| Staining dish | Thermo Scientific | 12657696 | |
| Tetramethylethylenediamine (TEMED) | Bio-Rad | 1610800 | |
| Tris base (2-Amino-2-(hydroxymethyl)-1,3-propanediol) | Sigma-Aldrich | 93362-500G | |
| Triton X-100 | Sigma-Aldrich | X100-500ML | |
| Tween-20 | Sigma-Aldrich | P9416-50ML | |
| Urea | Sigma-Aldrich | U6504-1KG | |
| Xylene cyanol | Alfa Aesar | B21530 |
Ссылки
- Berger, M. F., Bulyk, M. L. Protein binding microarrays (PBMs) for rapid, high-throughput characterization of the sequence specificities of DNA binding proteins. Methods in Molecular Biology. 338, 245-260 (2006).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nature Protocols. 4 (3), 393-411 (2009).
- Afek, A., et al. DNA Sequence Context Controls the Binding and Processivity of the T7 DNA Primase. iScience. 2, 141-147 (2018).
- Afek, A., Schipper, J. L., Horton, J., Gordan, R., Lukatsky, D. B. Protein-DNA binding in the absence of specific base-pair recognition. Proceedings of the Nationaly Academy of Sciences of the Unitet States of America. 111 (48), 17140-17145 (2014).
- Frick, D. N., Richardson, C. C. Interaction of bacteriophage T7 gene 4 primase with its template recognition site. Journal of Biological Chemistry. 274 (50), 35889-35898 (1999).
- Tabor, S., Richardson, C. C. Template recognition sequence for RNA primer synthesis by gene 4 protein of bacteriophage T7. Proceedings of the Nationaly Academy of Sciences of the Unitet States of America. 78 (1), 205-209 (1981).
- Fried, M., Crothers, D. M. Equilibria and kinetics of lac repressor-operator interactions by polyacrylamide gel electrophoresis. Nucleic Acids Research. 9 (23), 6505-6525 (1981).
- Galas, D. J., Schmitz, A. DNAse footprinting: a simple method for the detection of protein-DNA binding specificity. Nucleic Acids Research. 5 (9), 3157-3170 (1978).
- Jost, J. P., Munch, O., Andersson, T. Study of protein-DNA interactions by surface plasmon resonance (real time kinetics). Nucleic Acids Research. 19 (10), 2788(1991).
- Bowen, B., Steinberg, J., Laemmli, U. K., Weintraub, H. The detection of DNA-binding proteins by protein blotting. Nucleic Acids Research. 8 (1), 1-20 (1980).
- Oliphant, A. R., Brandl, C. J., Struhl, K. Defining the sequence specificity of DNA-binding proteins by selecting binding sites from random-sequence oligonucleotides: analysis of yeast GCN4 protein. Molecular Cell Biology. 9 (7), 2944-2949 (1989).
- Marmorstein, R., Fitzgerald, M. X. Modulation of DNA-binding domains for sequence-specific DNA recognition. Gene. 304, 1-12 (2003).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены