Method Article
DNA序列识别利用高通量原酶分析的DNAPrimase
摘要
蛋白质结合微阵列(PBM)实验与生化测定相结合,将DNA原蛋白酶的结合和催化特性联系起来,DNA原蛋白酶是一种在模板DNA上合成RNA引体的酶。该方法被指定为高通量原发性酶分析(HTPP),可用于揭示各种酶的DNA结合模式。
摘要
脱氧核糖核酸(DNA)原始酶合成短RNA引物,在DNA复制过程中通过DNA聚合酶启动Okazaki片段的DNA合成。原核DnaG样原蛋白酶与DNA结合发生在特定的三核苷酸识别序列中。这是冈崎碎片形成的关键一步。用于确定DNA原生酶DNA识别序列的传统生化工具只提供了有限的信息。使用高通量微阵列结合测定和连续生化分析,已经表明1)特定的结合背景(识别部位的粘结序列)影响DNA原发性酶与其模板的结合强度脱氧核糖核酸(DNA)和2)使原性酶与DNA结合得更强烈,从而产生更长的RNA引结,表明酶的工艺性更高。该方法结合了PBM和原发酶活性测定,被指定为高通量原发性酶分析(HTPP),允许在前所未有的时间和可扩展性下,通过DNA原发酶对特定序列识别进行表征。
引言
HTPP利用DNA结合微阵列技术与生化分析相结合(图1),从统计学上识别影响DNA原发酶酶酶活性的DNA模板的具体特征。因此,HTPP 提供了一个技术平台,有助于该领域的知识飞跃。用于确定原始酶识别站点的经典工具无法生成大量数据,而 HTPP 则无法生成。
PBM是一种常用的技术,用于确定转录因子与DNA1、2的结合偏好;然而,它不适合检测蛋白质与DNA的弱/瞬态结合。与向由八个碱基对组成的所有可能序列提供平均蛋白质结合特异性信息的通用 PBM 不同,HTPP 基于包含独特序列元素的单链 DNA 模板库。这种DNA序列元素涉及数以万计的短(几十bp)基因组序列,以及计算设计的DNA序列,这些序列丰富于基因组中某些DNA重复序列元素中,这些序列具有不同的平均GC含量.这种高通量方法允许以系统、定量和假设驱动的方式确定对原性酶结合及其酶活性3重要的序列相关属性。特别是,在这种酶系4中,已确定了原始酶-DNA结合偏好(由DNA序列在特定的三核苷酸结合位点两侧调节)和原始酶过程性之间的重要联系。
这项新技术被应用来重新审视我们对灵酶识别位点的理解,甚至对于T7DNA原性酶,已经被广泛研究5。具体来说,重新审查经典概念,如T7 DNA原发酶的DNA识别位点(这是近40年前确定6)使用蛋白质-DNA结合微阵列(PBM)已导致前所未有的洞察与这些识别站点的侧翼序列3。预计T7DNA原发酶(5'-GTC-3')三核苷酸识别位点两侧的序列将是随机的。相反,我们发现TG丰富的侧翼序列增加了T7 DNA原始酶合成较长RNA引注的机会,表明工艺性增加。
其他可用于研究体外蛋白质DNA结合特性的方法包括电泳移动转移测定(EMSA)7、DNase I足迹8、表面质子共振(SPR)9和西南印迹10.然而,这些低通量方法只适用于研究少量DNA序列。此外,其中一些技术(例如 EMSA)的精度和灵敏度较低。另一方面,体外选择11是一种技术,与PBM类似,可用于识别许多结合序列。然而,在大多数体外选择应用中,通常排除低亲和序列;因此,此方法不适合获取所有可用序列的比较绑定数据。通用PBM1,2主要用于描述原核生物和真核生物转录因子的结合特性,以及特定因素(例如,某些配体、辅助因子等)可能影响此交互12。
HTPP 将前所未有的高通量统计能力与高精度相结合,提供结合序列上下文的信息,将 PBM 应用扩展到 DNA 处理酶。由于上述其他可用技术的技术限制,尚未获得灵性酶和相关酶(与DNA具有弱/瞬态结合)的数据。
研究方案
1. 微阵列设计
注:DNA探针代表定制的36核苷酸序列,由位于两个可变侧翼区域之间的T7DNA原发酶(GTC)的识别位点组成,然后是与玻璃滑动3系成的恒定的24核苷酸序列。我们使用 4 x 180,000 微阵列格式,该格式允许在幻灯片上随机分布的六个复制中发现每个 DNA 序列。
-
具有已知识别序列的灵原体DNA库设计
- 确保每个序列由60个核苷酸组成。保持前 24 个核苷酸恒定(连接到玻璃滑块)。可变区域应具有以下一般形式:(N)16GTC(N)17;其中 N 表示任何所需的核苷酸。
- 设计侧翼区域以解决特定的科学问题(设计示例如下文本所示)。侧翼区域可以设计为包含特定特征,如重复元素或特定对称性。
注:我们创建了八类不同的齿面序列,由两个或三个特定的核苷酸组成:T和G(组1);T 和 C(第 2 组);C 和 A(第 3 组);A 和 G(第4组);G、C 和 T(第 5 组);C、T和A(第6组);G、A 和 T(第7组);G、A 和 C(组 8)。为每个组设计了 2,000 个不同的序列。每个组都有具有不同类型和不同序列重复数的序列子组。 - 包括一组负控制序列(缺少特定的绑定位点)4。此类序列的存在允许验证实验中特定绑定的发生。
-
具有未知识别序列的灵长类动物DNA库的设计
- 如果识别序列未知,则通过从所需生物体(细菌、真菌等)的基因组中选择序列(有或没有前面提到的特定特征)来创建 DNA 库。
-
微阵列幻灯片的设计
- 从商业供应商处购买定制幻灯片(例如 4x180K、AMADID #78366)(有关更多详细信息,请参阅材料表)。将每个序列按六个复制顺序排列,其中每个复制应附加到幻灯片上随机选择的点。
2. Primase DNA结合实验
注:前一天(或至少2小时前)PBM,准备阻滞溶液[2%无脱脂牛奶在磷酸盐缓冲盐水(PBS)],并在磁搅拌器上搅拌。使用前,使用 0.45 μM 过滤器过滤溶液。为了检测与DNA链结合的灵酶,应按以下顺序执行几个步骤。
- 阻塞过程
- 在 PBS 中用 0.01% v/v Triton X-100 预湿了科普林罐中的微阵列滑动(在实验室旋转器上,在 125 rpm 时为 5 分钟)。
- 用水和乙醇短暂清洗垫片滑轨(也称为盖玻片),并使用压缩空气干燥。
- 在垫片滑片朝上(商业标签朝上)下组装钢混合室(PBM 室)的底部。有关装配的更多详细信息,请参阅材料表。
- 移液阻滞溶液(PBS中2%的脱脂牛奶)进入每个腔室。
- 从 Coplin 罐中取出微阵列幻灯片,然后使用精细擦除擦干非 DNA 侧(DNA 应与公司标签位于同一侧)和边缘。慢慢将微阵列放在垫片上,避免气泡(公司标签朝下)。立即组装并拧紧钢混合室装置。在室温 (RT) 下孵育 1 小时。
- 用 800 mL 的新鲜 PBS 填充一个染色盘("PBS"盘)。在 PBS 中,用 800 mL 的新鲜准备的 0.5% v/v Tween-20 填充第二个染色盘("WASH"菜)。
- 拧下 PBM 造型室并拆下微阵列滑盖"三明治",注意不要折断密封件。通过在滑轨和盖玻片之间放置钳子,将其在 WASH 盘中的水下拆卸。
- 在水下摇动微阵列幻灯片,并迅速转移到科普林罐中。
- 在 PBS 中用 0.1% v/v Tween-20 洗涤一次(在实验室旋转器上,在 125 rpm 时 5 分钟)。
- 在 PBS 中用 0.01% v/v Triton X-100 洗涤一次(在实验室旋转器上,在 125 rpm 时 2 分钟)。
- 快速将幻灯片传输到包含 PBS 的科普林罐。
- 蛋白质结合
- 如前所述(步骤 2.1.2~2.1.3)组装PBM 造型室。
- 含有5μM T7 DNA原蛋白酶的移液器蛋白结合混合物, 30 mM Tris-HCl pH 7.5, 6.5 mM MgCl2, 30 mM K-谷氨酸, 6 mM 二硫磷醇 (DTT), 65 μM 肋核苷三磷酸酯 (rNTP), 2% w/v 脱脂牛奶, 100 纳克/μL 牛血清白蛋白 (BSA), 50 ng/μL鲑鱼睾号DNA,和0.02%v/v Triton X-100 (TX-100)进入垫片滑动的每个腔室(不接触橡胶侧,不引入气泡)。
- 将微阵列幻灯片浸入 PBS 盘中,从滑片表面去除多余的洗涤剂,从而短暂冲洗微阵列幻灯片。用精细的擦拭滑片的非 DNA 表面。
- 慢慢将微阵列滑轨(朝下)降至垫片滑轨上,小心防止从一个腔室泄漏到另一个腔室。立即组装并拧紧 PBM 造型室设备,而不会引入气泡。如果气泡确实形成,则可以通过轻轻敲击钢室对坚硬的表面来去除气泡。
- 在室温 (RT) 下孵育 30 分钟。
- 荧光抗体附件
- 拧下 PBM 造型室并拆下微阵列滑盖"三明治",注意不要折断密封件。使用钳子在 WASH 盘中在水下拆卸。在水下摇动幻灯片,并迅速转移到含有PBS的科普林罐。
- 按照前面所述(步骤 2.1.2_2.1.3)使用垫片滑动将 PBM 造型室组装。
- 将 Alexa 488 结合的抗其抗体(结合缓冲液中 10 纳克/μL)添加到垫片滑块中。
- 像以前那样在 PBS 盘中短暂冲洗幻灯片,然后缓慢地从 PBS 中取出幻灯片,擦拭非 DNA 表面并将其朝下放在垫片上。
- 在黑暗中在RT孵育30分钟(荧光探头对光敏感),以减少光漂白。
- 像以前那样拆卸 PBM 造型室和盖玻片,将外盖滑取在 WASH 盘中的水下。
- 将幻灯片浸入 PBS 盘中,请短暂冲洗幻灯片。用压缩空气擦干幻灯片。将在黑暗中存放在幻灯片盒中,直到准备好扫描。
- 扫描微阵列幻灯片
- 使用激发495nm和发射519nm的微阵列扫描仪扫描芯片(参见材料表),并收集荧光中值强度。
3. 微阵列数据分析
注: 所有数据处理都是使用 MATLAB 中的自定义写入脚本执行的。
- 使用 Wilcoxon 排名总和测试 p 值执行初始 PBM 数据处理,如前面4所述。
- 使用每个 DNA 序列的结合强度的中位数进行进一步分析。
- 接下来,使用单向ANOVA p值,比较不同DNA探针组获得的灵酶-DNA结合强度观测到的差异的统计显著性,如上文DNA库设计第3节所述。
4. 由T7DNA原代酶催化的模板导向RNA合成
- 变性聚丙烯酰胺凝胶的制备
- 要制备 100 mL 的凝胶混合物, 结合62.5 mL的40%丙烯酰胺-双环乙酰胺(19:1),42克尿素,1.1克三思基(2-Amino-2-(羟基)-1,3-丙二醇,0.55克硼酸,0.4 m L 0.5M乙烯四甲酸(EDTA)溶液。
- 将混合物分成14 mL等分(16.5厘米x26厘米x0.03厘米的凝胶所需的量),并在4°C下保护它们长达1个月。
- 要制备一种变性聚丙烯酰胺凝胶,在先前制备的混合物中加入4μL的TEMED和40μL的10%的10%的万伏硫酸铵,将其铸造,并在RT处进行至少2小时的聚合。凝胶可在4°C下保存一天。
- 样品制备
注:将试剂、反应混合物和样品保存在冰上。- 为了制备十种反应所需的反应混合物,将11μL的5倍活性缓冲液(200mM Tris-HCl,pHH = 7.5;50mM二硫磷醇,250 mM谷氨酸钾,50mMMgCl 2),5.5 μL的2.5 mM三磷酸三磷酸(NTPs)混合物,以及5.5 μL的聚磷剂(NTPs)和5.5μL放射性标记的核苷酸[例如,ATP,(+-32P)3000 Ci/mmol]。
注:由于可能的移液错误,所有量都增加了 10%。放射性标记的核苷酸应根据放射性信号的强度进行稀释(初始稀释通常为1:10或更多)。 - 将反应混合物的2 μL转移到10个PCR管中。
- 将1μL的100μMDNA模板加入相应的PCR管并旋转。
- 加入2 μL的16μM T7 DNA原生酶,向下旋转,轻轻混合,然后再次旋转。
- 在RT孵育反应20分钟。
- 加入5μL的淬火溶液(95%形式酰胺,20m EDTA,0.05%带二甲苯和溴酚蓝),混合并旋转,从而停止反应。
- 为了制备十种反应所需的反应混合物,将11μL的5倍活性缓冲液(200mM Tris-HCl,pHH = 7.5;50mM二硫磷醇,250 mM谷氨酸钾,50mMMgCl 2),5.5 μL的2.5 mM三磷酸三磷酸(NTPs)混合物,以及5.5 μL的聚磷剂(NTPs)和5.5μL放射性标记的核苷酸[例如,ATP,(+-32P)3000 Ci/mmol]。
- 通过变性聚丙烯酰胺凝胶和后续信号检测,通过电泳分离RNA产物
- 在 1x TBE 缓冲液(90 mM Tris-borate,2 mM EDTA)中预运行凝胶 1 小时(10 mA、600 V)。
- 每个样品的负载高达 2 μL,并在 1x TBE 缓冲液(90 mM Tris-borate,2 mM EDTA)中执行电泳(20 mA,1100 V)。
- 在真空下80°C干燥凝胶2小时。
- 根据放射性信号的强度,将磷成像板暴露在放射性凝胶中几个小时至一夜之间。
- 使用荧光仪记录信号(光刺激发光)(参见材料表)。
结果
这种绘制原始酶结合位点的技术进步使得获得DNA结合特性,即使不是不可能的话,也很难用经典工具观察。更重要的是,HTPP 能够重新审视对原始酶结合位点的传统理解。具体来说,HTPP揭示了结合特性,除了已知的5'-GTC-3'识别序列,这导致T7DNA原代酶的功能活动的变化。即,确定了两组序列:侧翼包含T/G的强结合DNA序列和侧翼中含有A/G的弱结合序列(强结合模板中的所有胸腺都被腺素取代)。未检测到在其序列中缺少 5'-GTC-3' 的 DNA 模板的灵性酶结合。
含有特定特征的灵酶DNA识别位点,如T/G富侧翼,将原发酶DNA结合增加至10倍,并出人意料地也增加了新形成的RNA的长度(高达三倍)(图2在以前的出版物中3.重要的是,HTPP使我们能够观察和量化与DNA模板序列相关的引种长度的变异性。
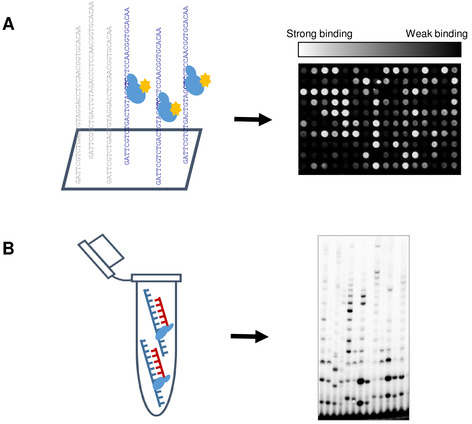
图 1:高通量原始酶分析 (HTPP) 的原理表示。(A) 幻灯片在活动缓冲液中孵育了原生酶(40 mM Tris-HCl,pH 7.5;10 mM MgCl2,50mM K-谷氨酸,10 mM DTT,100 μM rNTP)。接下来,Alexa 488 结合抗其荧光抗体被引入来标记该蛋白质。轻度洗涤步骤后,使用微阵列扫描仪扫描幻灯片。结合亲和力根据中值荧光信号确定,DNA序列相应地分为各组。(B) 进行生化测定,将微阵列实验获得的DNA结合结果与T7DNA原发酶的功能特性相关联。请点击此处查看此图的较大版本。

图2:从PBM获得的两组DNA模板(侧翼与T/G强结合,侧翼与A/G结合弱结合)的催化活性比较。(A) 由T7DNA原发酶催化的RNA引结形成.反应中含有寡核苷酸,在标准反应混合物中含有原发酶识别序列(编号通道)、32P-α-ATP、ATP、CTP、UTP 和 GTP。一组DNA寡核苷酸(深蓝色)在侧翼含有T/G,而第二组的所有胸腺都替换为腺苷(浅蓝色)。孵育后,放射性RNA产物通过含有7M尿素的25%聚丙烯酰胺凝胶进行电泳分离,并通过自光成像进行可视化。(B) 相对长度(构成每个RNA引体的核苷酸数量未知)和T7 DNA原始酶在两组DNA模板(面板A)上合成的RNA引基产物的数量。这些图显示,与与低亲和力结合的模板(在侧翼中包含 A/G)相比,在 DNA 模板上合成更长的 RNA 引种(增加过程性),这些原始酶结合的亲和力更高(侧翼中含有 T/G)。(C) 在两组DNA模板上定量合成RNA引注量。结果表明,T7DNA原位酶的DNA结合亲和力与合成RNA的量之间存在相关性。AU(任意单位)是放射性信号强度的测量,与合成RNA引体量直接相关。请点击此处查看此图的较大版本。
讨论
PBM方法已广泛用于研究转录因子的结合特性,还可用于DNA处理酶,如DNA原发酶,这些酶与低亲和力的DNA结合。然而,实验程序需要某些修改。微阵列实验涉及几个步骤:DNA库的设计、芯片的制备、蛋白质靶点结合、荧光标记和扫描。轻度洗涤步骤至关重要,因为长时间使用含有洗涤剂的溶液会导致蛋白质与DNA模板分离,因为结合的弱/瞬态模式。其他关键步骤包括结合条件(例如,缓冲物组合、辅助因子)和需要针对每种特定酶优化的孵育时间。
从微阵列获得的结果需要在生化测定中验证。生物化学测定还有助于深入了解DNA处理酶的有趣特征,如DNA结合亲和力和酶活性/过程性之间的相关性。HTPP使我们能够观察DNA结合对T7DNA原性酶功能特性的影响。例如,据观察,如果灵酶对DNA模板表现出更高的结合亲和力,它催化更长的RNA引物的形成(增加工艺性)。
总体而言,本文介绍的方法快速、可靠,并提供了以假设驱动的方式在单个实验中同时测试数万个不同DNA序列的绑定特性的机会。在反应混合物中加入其他蛋白质或金属辅助因子,可以快速、高通量的方式研究其对原性细胞DNA结合特性的影响。另一方面,由于微阵列滑片价格相对较高,以及处理用于刺激性酶活性测定的放射性物质时所需的安全预防措施,因此该方法的应用受到限制。
需要注意的是,不同的原样需要修改微阵列结合条件和活动测定的缓冲条件。例如,结核分枝杆菌的原发性酶需要在反应缓冲液中用二价锰替代镁。不适当的金属辅助因子或不适当的反应缓冲液可能导致原性酶活性差或DNA结合亲和力降低。
如前所述,灵原子与具有弱亲和力的DNA模板结合;因此,在微阵列结合实验中需要温和的洗涤步骤。否则,它们将从微阵列幻灯片中冲出,导致添加荧光标记抗体后失去荧光信号。
总之,许多原核原发原发素的DNA结合特性(包括三核苷酸DNA识别序列)仍然缺乏理解,这主要是由于目前可用方法的技术限制。HTPP 是发现 DNA 识别位点或调查其他模板相关因素(即整体核苷酸组成、GC 含量、重复DNA序列元素的存在及其对称性)的快速高效的平台影响ssDNA结合酶的结合亲和力和活性。此外,未来的应用可能针对不同蛋白质或辅助因子对DNA结合亲和力、识别模式或灵酶和其他DNA处理酶的功能活性的影响。
披露声明
提交人声明没有利益冲突。
致谢
这项研究得到了以色列科学基金会(第1023/18号赠款)的支持。
材料
| Name | Company | Catalog Number | Comments |
| 40% acrylamide-bisacrylamide (19:1) solution | Merck | 1006401000 | |
| 95% formamide | Sigma-Aldrich | F9037-100ML | |
| Alexa 488-conjugated anti-his antibody | Qiagen | 35310 | |
| Ammonuium persulfate (APS) | Sigma-Aldrich | A3678-100G | |
| ATP, [α-32P] – 3000 Ci/mmol | Perkin Elmer | NEG003H250UC | |
| Boric acid, granular | Glentham Life Sciences | GE4425 | |
| Bovine Serum Albumin (BSA) | Roche | 10735094001 | |
| Bromophenol blue | Sigma-Aldrich | B0126-25G | |
| Coplin jar | |||
| Dithiothreitol (DTT) | Sigma-Aldrich | D0632-25G | |
| DNA microarray | Agilent | 4x180K (AMADID #78366) https://www.agilent.com | |
| Ethylenediaminetetraacetic acid (EDTA) | Acros Organics | AC118430010 | |
| Fujifilm FLA-5100 phosphorimager | FUJIFILM Life Science | ||
| Glass slide staining rack | Thermo Scientific | 12869995 | If several slides are used |
| Lab rotator | Thermo Scientific | 88880025 | |
| Magnesium chloride | Sigma-Aldrich | 63064-500G | |
| Microarray Hybridization Chamber | Agilent | G2534A | https://www.agilent.com/cs/library/usermanuals/Public/G2534-90004_HybridizationChamber_User.pdf |
| Microarray scanner (GenePix 4400A) | Molecular Devices | ||
| Phosphate Buffered Saline (PBS) | Sigma-Aldrich | P4417-100TAB | |
| Potassium glutamate | Alfa Aesar | A172232 | |
| Ribonucleotide Solution Mix (rNTPs) | New England BioLabs | N0466S | |
| Salmon testes DNA | Sigma-Aldrich | D1626-1G | |
| Skim milk powder | Sigma-Aldrich | 70166-500G | |
| Staining dish | Thermo Scientific | 12657696 | |
| Tetramethylethylenediamine (TEMED) | Bio-Rad | 1610800 | |
| Tris base (2-Amino-2-(hydroxymethyl)-1,3-propanediol) | Sigma-Aldrich | 93362-500G | |
| Triton X-100 | Sigma-Aldrich | X100-500ML | |
| Tween-20 | Sigma-Aldrich | P9416-50ML | |
| Urea | Sigma-Aldrich | U6504-1KG | |
| Xylene cyanol | Alfa Aesar | B21530 |
参考文献
- Berger, M. F., Bulyk, M. L. Protein binding microarrays (PBMs) for rapid, high-throughput characterization of the sequence specificities of DNA binding proteins. Methods in Molecular Biology. 338, 245-260 (2006).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nature Protocols. 4 (3), 393-411 (2009).
- Afek, A., et al. DNA Sequence Context Controls the Binding and Processivity of the T7 DNA Primase. iScience. 2, 141-147 (2018).
- Afek, A., Schipper, J. L., Horton, J., Gordan, R., Lukatsky, D. B. Protein-DNA binding in the absence of specific base-pair recognition. Proceedings of the Nationaly Academy of Sciences of the Unitet States of America. 111 (48), 17140-17145 (2014).
- Frick, D. N., Richardson, C. C. Interaction of bacteriophage T7 gene 4 primase with its template recognition site. Journal of Biological Chemistry. 274 (50), 35889-35898 (1999).
- Tabor, S., Richardson, C. C. Template recognition sequence for RNA primer synthesis by gene 4 protein of bacteriophage T7. Proceedings of the Nationaly Academy of Sciences of the Unitet States of America. 78 (1), 205-209 (1981).
- Fried, M., Crothers, D. M. Equilibria and kinetics of lac repressor-operator interactions by polyacrylamide gel electrophoresis. Nucleic Acids Research. 9 (23), 6505-6525 (1981).
- Galas, D. J., Schmitz, A. DNAse footprinting: a simple method for the detection of protein-DNA binding specificity. Nucleic Acids Research. 5 (9), 3157-3170 (1978).
- Jost, J. P., Munch, O., Andersson, T. Study of protein-DNA interactions by surface plasmon resonance (real time kinetics). Nucleic Acids Research. 19 (10), 2788(1991).
- Bowen, B., Steinberg, J., Laemmli, U. K., Weintraub, H. The detection of DNA-binding proteins by protein blotting. Nucleic Acids Research. 8 (1), 1-20 (1980).
- Oliphant, A. R., Brandl, C. J., Struhl, K. Defining the sequence specificity of DNA-binding proteins by selecting binding sites from random-sequence oligonucleotides: analysis of yeast GCN4 protein. Molecular Cell Biology. 9 (7), 2944-2949 (1989).
- Marmorstein, R., Fitzgerald, M. X. Modulation of DNA-binding domains for sequence-specific DNA recognition. Gene. 304, 1-12 (2003).
转载和许可
请求许可使用此 JoVE 文章的文本或图形
请求许可This article has been published
Video Coming Soon
版权所属 © 2025 MyJoVE 公司版权所有,本公司不涉及任何医疗业务和医疗服务。