Method Article
זיהוי רצף דנ א על ידי Primase DNA שימוש בתפוקה גבוהה Primase פרופיל
In This Article
Summary
מיקרו-מערך מחייב חלבון (pbm) ניסויים בשילוב עם בחני יוכימיים מקשר את תכונות הכריכה והקטליטי של ה-dna primase, אנזים שעושה שילוב של ה-RNA התחל ב-dna תבנית. שיטה זו, המיועדת כprimase ליצירת פרופיל בתפוקה גבוהה (HTPP), יכולה לשמש לחשיפת דפוסי איגוד DNA של מגוון אנזימים.
Abstract
דנ א primase מגדלים קצרים RNA התחל ליזום סינתזה של דנ א של קטעי וקאזאקי על סטרנד בפיגור על ידי DNA פולימראז במהלך שכפול DNA. הכריכה של הprimases של prokaryotic בדומה ל-DNA מתרחשת ברצף זיהוי טריאוקלאוטית מסוים. זהו צעד מרכזי ביצירת קטעים של וקאזאקי. כלים ביוכימיים קונבנציונליים המשמשים כדי לקבוע את רצף זיהוי ה-DNA של primase DNA לספק רק מידע מוגבל. באמצעות שיטת האיגוד המבוסס על מיקרוarray עם תפוקה גבוהה וניתוחים ביוכימיים עוקבים, הוכח כי 1) ההקשר המחייב הספציפי (רצפי מעבר של אתר הזיהוי) משפיע על חוזק הכריכה של ה-DNA הprimase לתבנית שלו DNA, ו 2) מחייב חזק יותר של primase לתשואות ה-DNA התחל עוד RNA, המציין מעבד גבוה יותר של האנזים. שיטה זו משלבת PBM ו primase הפעילות והוא מיועד כprimase התפוקה גבוהה (HTPP), והוא מאפשר אפיון של זיהוי רצף ספציפי על ידי primase DNA בזמן ומדרגיות חסר תקדים.
Introduction
HTPP עושה שימוש בטכנולוגיה מיקרו מחייב DNA בשילוב עם ניתוח ביוכימיים (איור 1) כדי סטטיסטית לזהות תכונות ספציפיות של תבניות DNA המשפיעות על הפעילות האנזימטית של primase DNA. לכן, HTPP מספק פלטפורמה טכנולוגית המאפשרת קפיצת ידע בתחום. הכלים הקלאסיים המשמשים לקביעת אתרי זיהוי primase אין להם את היכולת להניב כמות מסיבית של נתונים, בעוד HTPP עושה.
Pbm היא טכניקה המשמשת באופן שגרתי כדי לקבוע את ההעדפות קשירה של גורמי תמלול ל-DNA1,2; עם זאת, הוא אינו מתאים לזיהוי של מחייב חלש/ארעי של חלבונים ל-DNA. בניגוד PBM אוניברסלי המספק מידע על ממוצע כריכת חלבון ספציפיות לכל הרצפים האפשריים המורכב של שמונה זוגות בסיס, HTPP מבוסס על הספריה של יחיד תקועים תבניות DNA הכוללת אלמנטים רצף ייחודי. אלה אלמנטים רצף DNA כרוך עשרות אלפים קצרים (עשרות מעטים של bp) רצפים גנומית, כמו גם רצפי DNA מעוצב מעוצבים מועשר ב-DNA מסוימים רצף החוזר רכיבים הנמצאים בגנום, אשר בעלי תוכן GC ממוצע שונים . גישה בעלת תפוקה גבוהה כזו מאפשרת נחישות, באופן שיטתי, כמותי ומונחה השערות, המאפיינים הקשורים ברצף החשובים לאיגוד הprimase ופעילותה האנזימטית3. בפרט, את הקשר החשוב בין primase-DNA העדפות איגוד, (מאופנן על ידי רצפי DNA האגפים מסוימים שלושה נוקלאוטיד אתרים מחייב) ו primase מעבד זוהה עבור מערכת זו אנזימטית4.
הטכנולוגיה החדשה הוחל לחזור על ההבנה שלנו של אתרי זיהוי primase אפילו עבור ה-DNA T7 primase כי כבר למד בהרחבה5. במיוחד, בחינת מחדש של מושגים קלאסיים, כגון אתרי זיהוי דנ א של T7 DNA primase (אשר נקבעו כמעט ארבעה עשורים לפני 6) באמצעות חלבון-dna המחייב מיקרוarray (pbm) הוביל תובנה חסרת תקדים לתוך תכונות הקשורות את הרצף האגף של אתרי ההוקרה האלה3. זה היה צפוי הרצפים החוצה אתר הכרה tri-נוקלאוטיד של T7 DNA primase (5 '-GTC-3 ') יהיה אקראי. במקום זאת, מצאנו כי רצפים של TG-עשיר האגפים להגדיל את הסיכויים של T7 DNA primase לסנתז עוד RNA התחל להצביע על עלייה בתהליך.
שיטות אחרות אשר ניתן להשתמש כדי ללמוד את המאפיינים של כריכת ה-DNA של חלבונים בתוך מבחנה כוללים את היכולת לנוע בתנועה אלקטרופלוטית (EMSA)7, dnase I footprinting דפסה8, משטח-פלמון תהודה (spr)9, ומדרום מערב בלוק 10. אלה הם, עם זאת, שיטות תפוקה נמוכה ישים רק כדי לחקור מספר קטן של רצפי DNA. בנוסף, הדיוק והרגישות של חלק מהשיטות האלה (למשל, EMSA) נמוך. מצד שני, בחירה מחוץ גופית11 היא טכניקה, בדומה pbm, ניתן להשתמש לזיהוי רצפים מחייב רבים. עם זאת, רצפי אהדה נמוכים בדרך כלל אינם נכללים ברוב היישומים של בחירת חוץ גופית; לפיכך, גישה זו אינה מתאימה לקבלת נתונים מחייבים השוואתיים עבור כל הרצפים הזמינים. ה-pbm האוניברסלי1,2 משמש בעיקר כדי לאפיין את הקשירה של גורמי שעתוק prokaryotes ו eukaryotes, כמו גם גורמים ספציפיים (למשל, נוכחות של ליגטים מסוימים, קופטים, וכו ') שעשויים ישפיע על האינטראקציה הזאת12.
HTPP מרחיב את היישום PBM לאנזימים עיבוד ה-DNA על ידי שילוב של עוצמה סטטיסטית בתפוקה גבוהה תקדים עם דיוק גבוהה כדי לספק מידע על ההקשר רצף כריכה. נתונים אלה עדיין לא הושגו עבור primases ואנזימים הקשורים (כי יש כריכה חלשה/ארעית ל-DNA) בשל מגבלות טכניות כאמור של טכניקות זמינות אחרות.
Protocol
1. עיצוב מיקרוarray
הערה: בדיקות דנ א מייצגות מותאם אישית 36-נוקלאוטיד רצפים, המורכב של אתר הזיהוי עבור T7 DNA primase (GTC) ממוקם בין שני אזורים משתנה האגפים, ואחריו רצף של 24-נוקלאוטיד קבוע קשור לשקופית זכוכית3. השתמשנו בתבנית מיקרוarray של 4 x 180,000, אשר אפשרה איתור של כל רצף דנ א בשש משכפל, שהופץ באופן אקראי על השקופית.
-
עיצוב ספריית ה-DNA עבור primases עם רצפי הוקרה ידועים
- ודא שכל רצף מורכב מ-60 נוקלאוטידים. שמור את הראשון 24 נוקלאוטידים קבוע (להיות מוצמד לשקופית הזכוכית). על אזורים משתנים להיות בעלי הטופס הכללי הבא: (N)16GTC (n)17; כאשר N מייצג את כל הנוקלאוטיד הרצוי.
- מעצבים את האזור האגף כדי לטפל בשאלה מדעית ספציפית (דוגמה לעיצוב מוצגת בטקסט הבא). האזורים האגפים עשויים להיות מיועדים להכיל תכונות ספציפיות, כגון רכיבי החזרה או סימטריה ספציפית.
הערה: יצרנו שמונה קטגוריות של רצפים שונים המורכבים משתיים או שלוש נוקלאוטידים מסוימים: T ו-G (קבוצה 1); T ו-C (קבוצה 2); C ו-A (קבוצה 3); A ו-G (קבוצה 4); G, C ו-T (קבוצה 5); C, T ו-A (קבוצה 6); G, A ו-T (קבוצה 7); G, A ו-C (קבוצה 8). 2,000 רצפים שונים עוצבו עבור כל קבוצה. כל קבוצה היו קבוצות משני של רצפים עם סוגים שונים ומספרים שונים של רצף חוזר. - כלול ערכה של רצפי בקרה שליליים (חסר אתר האיגוד הספציפי)4. הנוכחות של רצפים כאלה מאפשרת לאמת את ההתרחשות של איגוד ספציפי בניסוי.
-
עיצוב של ספריית DNA עבור primases עם רצפי הכרה לא ידוע
- אם רצף הזיהוי אינו ידוע, צור את ספריית ה-DNA על-ידי בחירת הרצפים (עם או בלי תכונות ספציפיות שהוזכרו קודם לכן) מן הגנום של האורגניזם הרצוי (חיידקים, פטריות, וכו ').
-
עיצוב שקופית מיקרוarray
- רכישת שקופיות מותאמות אישית (לדוגמה, 4x180K, AMADID #78366) מספק מסחרי (לפרטים נוספים ראו טבלת חומרים). סדר כל רצף בשישה משכפל, כאשר כל שכפול צריך להיות מוצמד לנקודה שנבחרה באופן אקראי בשקופית.
2. ניסוי מחייב DNA Primase
הערה: ביום שלפני (או לפחות 2 h לפני) PBM, להכין את פתרון חסימת [2% w/v חלב רזה בתמיסת פוספט מאגר (PBS)] ולערבב אותו על מערבב מגנטי. לפני השימוש, סנן את הפתרון באמצעות מסנן μM 0.45. כדי לזהות את הכריכה הprimase לגדילי ה-DNA, יש לבצע מספר צעדים בסדר הבא.
- חסימת פרוצדורה
- מראש השקופית מיקרוarray בצנצנת Coplin עם 0.01% v/v טריטון X-100 ב-PBS (5 דקות ב 125 rpm על מסובבי המעבדה).
- לשטוף בקצרה את שקופית gasket (המכונה גם coverslip) עם מים אתנול ויבש באמצעות אוויר דחוס.
- להרכיב חלק תחתון של הכלאה פלדה קאמרית (תא PBM) עם שקופית gasket פונה כלפי מעלה (תווית מסחרית פונה למעלה). לפרטים נוספים בנושא ההרכבה, עיין בטבלת החומרים.
- Pipet פתרון חסימה (2% w/v חלב רזה ב-PBS) לכל תא.
- הסר את שקופית המיקרו-מערך מצנצנת הקופלין, ולאחר מכן ייבש את הצד הלא-DNA (הדנ א אמור להיות באותו צד כמו התווית של החברה) והקצוות באמצעות ניגוב דק. הניחו באיטיות את המיקרו-מערך בשקופית האטם, הימנעות מבועות (תווית החברה פונה כלפי מטה). התאספו מיד ותהדקו. מכשירים קאמריים היברידיזציה דגירה של 1 h בטמפרטורת החדר (RT).
- ממלאים מנה אחת מכתים (המנה "PBS") עם 800 mL של PBS טרי. למלא את המנה השנייה מכתים (את "לשטוף" צלחת) עם 800 mL של טרי מוכן 0.5% v/v רצף-20 ב PBS.
- להוציא את החדר PBM ולהסיר את שקופיות microarray שקופית "סנדוויץ", מטפל לא לשבור את החותם. לפרק אותו מתחת למים בצלחת לשטוף ידי הצבת מלקחיים בין שקופית לבין coverslip.
- טלטל את שקופית המיקרו-מערך מתחת למים והעבר במהירות לצנצנת של קופלין.
- לשטוף פעם אחת עם 0.1% v/v רצף-20 ב-PBS (5 דקות ב 125 סל ד על מסובבי המעבדה).
- לשטוף פעם אחת עם 0.01% v/v טריטון X-100 ב-PBS (2 דקות ב 125 rpm על מסובבי המעבדה).
- העבר במהירות את השקופית לצנצנת של Coplin המכילה את ה-PBS.
- כריכת חלבונים
- הכנס את תא Pbm כפי שתואר קודם (שלבים 2.1.2 – 2.1.3).
- התערובת מחייבת חלבון פיפטה המכיל 5 μm T7 DNA primase, 30 mm טריס-HCl pH 7.5, 6.5 mm mgcl2, 30 מ"מ K-גלוטמט, 6 מ"מ מילימטר (dtt), 65 μm ribonucleoside צידה טריפוספט (rntp), 2% w/v חלב רזה, 100 ng/μl סרום שור (bsa), 50 ng/μl האשכים סלמון DNA, ו 0.02% v/v טריטון X-100 (TX-100) לתוך כל חדר של שקופית gasket (מבלי לגעת הצדדים גומי ומבלי להציג בועות).
- שטוף את שקופית המיקרו-מערך לזמן קצר על-ידי טבילה בה בצלחת ה-PBS, המסירה כביסה עודפת ממשטח השקופיות. נגב את המשטחים שאינם דנ א של השקופית עם מחיקה נאה.
- הנמך באיטיות את שקופית המיקרו-מערך (כלפי מטה) אל שקופית האטם, והיזהר ממניעת דליפה מתא אחד למשנהו. מיד להרכיב ולהדק את מנגנון הקאמרית PBM מבלי להציג בועות. אם בועות לעשות טופס, הם יכולים להיות מוסרים על ידי הקשה בעדינות על תא פלדה נגד משטח קשה.
- דגירה של 30 דקות בטמפרטורת החדר (RT).
- נוגדן פלורסנט
- להוציא את החדר PBM ולהסיר את שקופיות microarray שקופית "סנדוויץ", מטפל לא לשבור את החותם. לפרק מתחת למים בצלוחית לשטוף באמצעות מלקחיים. טלטל את השקופית מתחת למים והעבר במהירות לצנצנת Coplin המכילה את ה-PBS.
- להרכיב את חדר PBM עם שקופית gasket כמתואר בעבר (שלבים 2.1.2 – 2.1.3).
- הוסף אלקסה 488-מצודת נגד הנוגדן שלו (10 ng/μL במאגר הכריכה) לשקופית gasket.
- לשטוף את השקופית לזמן קצר על ידי טבילה אותה בצלחת PBS כמו לפני, ולאחר מכן להסיר את השקופית מ-PBS לאט, לנגב את משטח שאינו DNA ולמקם אותו כלפי מטה על שקופית gasket.
- דגירה 30 דקות ב RT בחושך (בדיקה הזריחה הוא רגיש קל) כדי להקטין את התמונה הלבנת.
- פרק את התא PBM ואת coverslip כמו קודם, הסרת שמיכות מתחת למים בצלחת לשטוף.
- שטוף את השקופיות לזמן קצר על-ידי טבילה בהן בצלחת ה-PBS. ייבש את השקופיות באוויר דחוס. אחסן באפלה בתיבה שקופית עד שהוא מוכן לסריקה.
- סריקת שקופית המיקרו-מערך
- סריקת השבב באמצעות סורק microarray (להתייחס לטבלה של חומרים) עם עירור של 495 ננומטר ופליטה של 519 nm ולאסוף את עוצמת הקרינה החציוני.
3. ניתוח נתונים במיקרו-מערך
הערה: כל עיבוד הנתונים בוצע באמצעות סקריפטים כתובים מותאמים אישית ב-MATLAB.
- בצע עיבוד נתונים הראשונית PBM באמצעות Wilcoxon דירוג סכום מבחן p-ערך כפי שמתואר בעבר4.
- השתמש בערך החציון של עוצמת האיגוד עבור כל רצף DNA לצורך ניתוח נוסף.
- לאחר מכן, באמצעות אחד הכיוון היחיד ANOVA p-ערכים, להשוות את המשמעות הסטטיסטית של ההבדלים הנצפים בעוצמות primase-DNA איגוד מושגת עבור קבוצות שונות של בדיקות דנ א, כפי שהוסבר לעיל ב העיצוב ספריית ה-DNA סעיף3.
4. תבנית בימוי RNA סינתזה מזרז על ידי T7 DNA primase
- הכנת בלמינל ג'ל פוליאקרילאמיד
- כדי להכין 100 ml של ג'ל תערובת, לשלב 62.5 ml של 40% אקרילאמיד-ביסאקרילאמיד (19:1), 42 g של אוריאה, 1.1 g של טריס בסיס (2-אמינית -2-(הידרוקסימתיל)-1, 3-propanediol), 0.55 g של חומצה חומצת, ו0.4 mL של 0.5 M ethylenediגנול חומצה (edta) הפתרון.
- חלק את התערובת לתוך 14 מ"ל (הסכום הדרוש עבור ג'ל אחד של 16.5 ס"מ x 26 ס"מ x 0.03 ס"מ) ולשמור אותם מוגנים מפני אור ב 4 ° c עד 1 חודש.
- כדי להכין 1 הרפתקאות אלקטרופורזה ג'ל, להוסיף 4 μl של temed ו-40 μl של 10% w/v אמוניום פרסולפט ל 14 מ ל של התערובת בעבר הוכנו, להטיל אותו, ולהשאיר אותו פולימלזציה עבור לפחות 2 h ב RT. הג ניתן לשמור ליום אחד ב -4 ° c.
- הכנה לדוגמא
הערה: שמור את הריאגנטים, תערובת התגובה, ודגימות על קרח.- כדי להכין את תערובת התגובה הנדרשת עבור עשרה תגובות לשלב 11 μL של מאגר פעילות 5x (200 mM טריס-HCl, pH = 7.5; 50 מ"מ dithiothreitol, 250 מילימטר אשלגן, 50 MgCl2), 5.5 μl של התערובת 2.5 mm של נוקלאוטיד tri-פוספט (ntps), ו 5.5 μl של מקור הקרינה [לדוגמה, ATP, (α-32P) 3000 Ci/ממול].
הערה: כל הסכומים הוגדלה ב-10% עקב שגיאות מסוימות אפשריות. בהתאם לעוצמת האות הרדיואקטיבית (הדילול הראשוני הוא בדרך כלל 1:10 או יותר). - העברה 2 μL של תערובת התגובה לעשר צינורות ה-PCR.
- הוסף 1 μL של 100 μM תבנית DNA לצינור ה-PCR המתאים והספין למטה.
- הוסף 2 μL של 16 μM T7 DNA primase, לסובב למטה, לערבב בעדינות, ו להסתחרר שוב.
- מודקון את התגובה ב RT עבור 20 דקות.
- להפסיק את התגובה על ידי הוספת 5 μl של הפתרון מקוצ'ינג (95% מתוך הטופס, 20 מ"מ edta, 0.05% w/v קסילן ו ברומרופנול כחול), לערבב, ו ספין למטה.
- כדי להכין את תערובת התגובה הנדרשת עבור עשרה תגובות לשלב 11 μL של מאגר פעילות 5x (200 mM טריס-HCl, pH = 7.5; 50 מ"מ dithiothreitol, 250 מילימטר אשלגן, 50 MgCl2), 5.5 μl של התערובת 2.5 mm של נוקלאוטיד tri-פוספט (ntps), ו 5.5 μl של מקור הקרינה [לדוגמה, ATP, (α-32P) 3000 Ci/ממול].
- הפרדת מוצרי RNA על ידי אלקטרופורזה באמצעות העברת ג'ל פוליאקרילמיד וזיהוי אותות שלאחר מכן
- Pre-הפעל את ג'ל עבור 1 h (10 mA, 600 V) במאגר 1x TBE (90 mM טריס-borate, 2 מ"מ EDTA).
- לטעון עד 2 μL של כל מדגם ולבצע אלקטרופורזה (20 mA, 1100 V) ב 1x מאגר TBE (90 mM טריס-borate, 2 מ"מ EDTA).
- מייבשים את הג ב-80 ° c בואקום.
- לחשוף את לוחית הדימות זרחן כדי ג'ל רדיואקטיבי למשך כמה שעות עד הלילה, בהתאם לחוזק של אות רדיואקטיבי.
- הקלט את האות (צילום מגורה) באמצעות פוספוריד (ראה טבלת חומרים).
תוצאות
מקדמה טכנולוגית זו למיפוי אתרי הכריכה הprimase מאפשרת קבלת תכונות מחייבות של דנ א, הקשות, אם לא בלתי אפשריות, להתבונן בכלים הקלאסיים. חשוב יותר, HTPP מאפשר המחודשת של ההבנה המסורתית של אתרי איגוד primase. באופן ספציפי, HTPP חושף את מחייב specificities בנוסף ידוע 5 '-GTC-3 ' רצפי זיהוי, אשר מוביל שינויים בפעילות תפקודית של T7 DNA primase. כלומר, שתי קבוצות של רצפים זוהו: חזק מחייב רצפי DNA כי הכיל T/G באגפים ו מחייב ברצפים חלשים כי הכיל A/G באגפים (כל thymines בתבניות חזקות מחייב הוחלפו על ידי אדנורינות). אין איגוד primase לתבניות DNA שחסרים 5 '-GTC-3 ' בתוך הרצף שלהם זוהה.
אתרי ה-DNA primase המכילים תכונות ספציפיות, כגון T/G-מטרים עשירים, גדל primase-DNA קשירה עד 10-קיפול, ובאופן מפתיע גם הגדיל את אורכו של RNA החדש שנוצר (עד שלשה) (איור 2 בפרסום הקודם 3). חשוב למדי, htpp איפשר לנו להתבונן ולכמת את השונות באורך פריימר ביחס לרצף של תבנית ה-DNA.
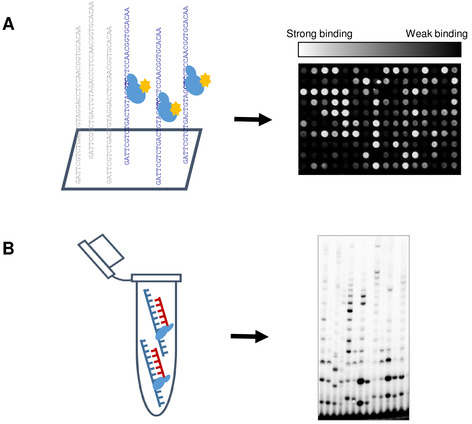
איור 1: ייצוג סכמטי של פרופיל primase תפוקה גבוהה (HTPP). (A) השקופית היה מודרט עם primase במאגר הפעילות (40 mm טריס-HCl, pH 7.5; 10 מ"מ MgCl2, 50 mM K-גלוטמט, 10 מ"מ Dtt, 100 Μm rntps). לאחר מכן, אלקסה 488-מצומדת נגד נוגדן הפלורסנט שלו הוצג כדי לתייג את החלבון. לאחר שלב הכביסה הקלה, השקופית נסרקת באמצעות סורק microarray. זיקה מחייבת נקבע על פי אות הזריחה החציוני ורצפי ה-DNA חולקו לקבוצות בהתאם. (ב) ביוכימיים שבוצעו כדי לתאם את התוצאות של ה-dna-מחייב שהתקבלו מניסוי microarray, עם המאפיינים הפונקציונליים של T7 DNA primase. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.

איור 2: השוואת פעילות קטליטי של T7 DNA primase על שתי קבוצות של תבניות dna (כריכה חזקה עם T/g באגפים ומחייב חלש עם A/g באגפים) שהתקבלו PBM. (א) RNA פריימר היווצרות מזרז על ידי T7 DNA primase. התגובות הכילו מחלת האודלונוסטאודים עם רצף זיהוי primase (נתיבים ממוספרים), 32P-α-ATP, ATP, CTP, utp, ו gtp בתערובת התגובה הסטנדרטית. קבוצה אחת של ה-DNA oligונודיאודים (כחול כהה) הכיל T/G באגפים, בעוד thymines וקשים הוחלפו באדביני בקבוצה השנייה (כחול בהיר). לאחר הדגירה, מוצרי ה-RNA הרדיואקטיבי הופרדו על-ידי אלקטרופורזה באמצעות ג'ל פוליאקרילמיד של 25% המכיל שתנן ודמיינו באמצעות האדיגרפיה האוטומטית. (ב) אורך יחסי (מספר של ribonucleotides המהווים כל פריימר RNA אינו ידוע) וכמות של התחל rna מסונתז על ידי T7 DNA primase על שתי קבוצות של תבניות DNA (פאנל A). המגרשים מראים כי עוד התחל RNA מסונתז (מוגבר מעבד) על תבניות DNA כי primase נקשר עם זיקה גבוהה יותר (המכילים T/G באגפים) לעומת התבניות הקשורות בזיקה נמוכה יותר (המכילים A/G באגפים). (ג) קוונפיקציה של כמות התחל של RNA מסונתז על שתי קבוצות של תבניות DNA. התוצאות להפגין מתאם בין הזיקה לאיגוד ה-DNA ואת כמות ה-RNA מסונתז על ידי T7 DNA primase. AU (יחידה שרירותית) הוא מדד של עוצמת האות רדיואקטיבי אשר מתואם ישירות עם כמויות של התחל RNA מסונתז. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
Discussion
שיטת PBM נעשה שימוש נרחב כדי לחקור את מאפייני הכריכה של שעתוק גורמים והוא יכול גם להיות מוחל על אנזימים עיבוד DNA, כגון primase DNA, כי לאגד DNA עם זיקה נמוכה. עם זאת, יש צורך בשינויים מסוימים בהליכים ניסיוניים. הניסוי המיקרו-מערך כרוך במספר שלבים: עיצוב ספריית ה-DNA, הכנת השבב, איגוד מטרת החלבון, תיוג פלורסנט וסריקה. שטיפת כביסה קלה הם קריטיים, מאז שוטף ארוך עם פתרונות המכילים דטרגנטים לגרום לדיסוציאציה של חלבון מתבנית ה-DNA בשל המצב חלש/ארעי של איגוד. שלבים קריטיים אחרים כוללים תנאי איגוד (לדוגמה, הרכב מאגר, קופנים) וזמני דגירה שצריכים להיות ממוטבים עבור כל אנזים ספציפי.
התוצאות המתקבלות ממיקרו-מערך צריכות להיות מאומתות בתוך הביוכימי. ביוכימיה ביוכימית גם לספק תובנה תכונות מעניינות של אנזימים עיבוד DNA כגון הקורלציה בין זיקה איגוד DNA ופעילות אנזימטית. HTPP איפשר לנו להתבונן בהשפעה של כריכת ה-DNA על תכונות פונקציונליות של T7 DNA primase. לדוגמה, זה כבר נצפתה כי אם primase מציג זיקה מחייב גבוה יותר עבור תבנית ה-DNA, זה מזרז את היווצרות של שעוד RNA התחל (מוגבר מעבד).
באופן כללי, השיטה המוצגת במאמר זה היא מהירה, אמינה ומספקת את ההזדמנות לבדוק בו מאפייני איגוד על עשרות אלפי רצפי DNA מגוונים בניסוי אחד בדרך מונחה השערות. תוספת של חלבונים אחרים או קופנים מתכת לתערובת התגובה מציעה את האפשרות לחקור את השפעתם על מאפייני איגוד ה-DNA של primases במהירות, תפוקה גבוהה באופן. מצד שני, היישום של שיטה זו מוגבל על-ידי מחיר גבוה יחסית של שקופיות microarray ואמצעי הזהירות הנדרשים בעת טיפול בחומר רדיואקטיבי עבור primase פעילות.
חשוב לציין שprimases שונים דורשים שינויים בתנאי האיגוד המיקרוarray ובתנאי האגירה עבור הפעילות. לדוגמה, primase של שחפת של פטרת מחייב החלפת מגנזיום עם מנגן דינטי במאגר התגובות. קופנים מתכת לא הולמים או מאגרי תגובה לא הולמים עלול לגרום לפעילות primase ירודה או ירידה הזיקה איגוד ה-DNA.
כפי שהוזכר קודם לכן, primases bind לתבניות DNA עם זיקה חלשה; לכן, נדרשים שטיפת כביסה עדינה במהלך ניסוי כריכת מיקרוarray. אחרת, הם יישטף החוצה מתוך השקופית microarray, מוביל לאובדן של האות הפלוראסלי בתוספת של נוגדן התווית באופן פלואורומיד.
כדי לסכם, את המאפיינים של איגוד ה-DNA של מprimases prokaryotic רבים (כולל רצף הזיהוי של ה-DNA trinucleotide) עדיין הבינו בצורה גרועה, בעיקר בשל המגבלות הטכניות של שיטות הזמינות כעת. HTPP מייצג פלטפורמה מהירה ויעילה לגילוי של אתרי זיהוי ה-DNA או חקירות של גורמים אחרים הקשורים לתבנית (כלומר, הרכב נוקלאוטיד הכולל, GC תוכן, נוכחות של החוזר אלמנטים רצף DNA וסימטריה שלהם) המשפיעות על הזיקה והפעילות המחייבים של אנזימי מאגד ssDNA. בנוסף, יישומים עתידיים עשויים להיות מכוונים לעבר ההשפעות של חלבונים שונים או קופנים על הזיקה איגוד ה-DNA, דפוסי הכרה, או פעילות פונקציונלית של primases ועוד אנזימים עיבוד DNA.
Disclosures
המחברים לא מצהירים. על ניגוד אינטרסים
Acknowledgements
מחקר זה נתמך על ידי הקרן הישראלית למדעים (גרנט no. 1023/18).
Materials
| Name | Company | Catalog Number | Comments |
| 40% acrylamide-bisacrylamide (19:1) solution | Merck | 1006401000 | |
| 95% formamide | Sigma-Aldrich | F9037-100ML | |
| Alexa 488-conjugated anti-his antibody | Qiagen | 35310 | |
| Ammonuium persulfate (APS) | Sigma-Aldrich | A3678-100G | |
| ATP, [α-32P] – 3000 Ci/mmol | Perkin Elmer | NEG003H250UC | |
| Boric acid, granular | Glentham Life Sciences | GE4425 | |
| Bovine Serum Albumin (BSA) | Roche | 10735094001 | |
| Bromophenol blue | Sigma-Aldrich | B0126-25G | |
| Coplin jar | |||
| Dithiothreitol (DTT) | Sigma-Aldrich | D0632-25G | |
| DNA microarray | Agilent | 4x180K (AMADID #78366) https://www.agilent.com | |
| Ethylenediaminetetraacetic acid (EDTA) | Acros Organics | AC118430010 | |
| Fujifilm FLA-5100 phosphorimager | FUJIFILM Life Science | ||
| Glass slide staining rack | Thermo Scientific | 12869995 | If several slides are used |
| Lab rotator | Thermo Scientific | 88880025 | |
| Magnesium chloride | Sigma-Aldrich | 63064-500G | |
| Microarray Hybridization Chamber | Agilent | G2534A | https://www.agilent.com/cs/library/usermanuals/Public/G2534-90004_HybridizationChamber_User.pdf |
| Microarray scanner (GenePix 4400A) | Molecular Devices | ||
| Phosphate Buffered Saline (PBS) | Sigma-Aldrich | P4417-100TAB | |
| Potassium glutamate | Alfa Aesar | A172232 | |
| Ribonucleotide Solution Mix (rNTPs) | New England BioLabs | N0466S | |
| Salmon testes DNA | Sigma-Aldrich | D1626-1G | |
| Skim milk powder | Sigma-Aldrich | 70166-500G | |
| Staining dish | Thermo Scientific | 12657696 | |
| Tetramethylethylenediamine (TEMED) | Bio-Rad | 1610800 | |
| Tris base (2-Amino-2-(hydroxymethyl)-1,3-propanediol) | Sigma-Aldrich | 93362-500G | |
| Triton X-100 | Sigma-Aldrich | X100-500ML | |
| Tween-20 | Sigma-Aldrich | P9416-50ML | |
| Urea | Sigma-Aldrich | U6504-1KG | |
| Xylene cyanol | Alfa Aesar | B21530 |
References
- Berger, M. F., Bulyk, M. L. Protein binding microarrays (PBMs) for rapid, high-throughput characterization of the sequence specificities of DNA binding proteins. Methods in Molecular Biology. 338, 245-260 (2006).
- Berger, M. F., Bulyk, M. L. Universal protein-binding microarrays for the comprehensive characterization of the DNA-binding specificities of transcription factors. Nature Protocols. 4 (3), 393-411 (2009).
- Afek, A., et al. DNA Sequence Context Controls the Binding and Processivity of the T7 DNA Primase. iScience. 2, 141-147 (2018).
- Afek, A., Schipper, J. L., Horton, J., Gordan, R., Lukatsky, D. B. Protein-DNA binding in the absence of specific base-pair recognition. Proceedings of the Nationaly Academy of Sciences of the Unitet States of America. 111 (48), 17140-17145 (2014).
- Frick, D. N., Richardson, C. C. Interaction of bacteriophage T7 gene 4 primase with its template recognition site. Journal of Biological Chemistry. 274 (50), 35889-35898 (1999).
- Tabor, S., Richardson, C. C. Template recognition sequence for RNA primer synthesis by gene 4 protein of bacteriophage T7. Proceedings of the Nationaly Academy of Sciences of the Unitet States of America. 78 (1), 205-209 (1981).
- Fried, M., Crothers, D. M. Equilibria and kinetics of lac repressor-operator interactions by polyacrylamide gel electrophoresis. Nucleic Acids Research. 9 (23), 6505-6525 (1981).
- Galas, D. J., Schmitz, A. DNAse footprinting: a simple method for the detection of protein-DNA binding specificity. Nucleic Acids Research. 5 (9), 3157-3170 (1978).
- Jost, J. P., Munch, O., Andersson, T. Study of protein-DNA interactions by surface plasmon resonance (real time kinetics). Nucleic Acids Research. 19 (10), 2788(1991).
- Bowen, B., Steinberg, J., Laemmli, U. K., Weintraub, H. The detection of DNA-binding proteins by protein blotting. Nucleic Acids Research. 8 (1), 1-20 (1980).
- Oliphant, A. R., Brandl, C. J., Struhl, K. Defining the sequence specificity of DNA-binding proteins by selecting binding sites from random-sequence oligonucleotides: analysis of yeast GCN4 protein. Molecular Cell Biology. 9 (7), 2944-2949 (1989).
- Marmorstein, R., Fitzgerald, M. X. Modulation of DNA-binding domains for sequence-specific DNA recognition. Gene. 304, 1-12 (2003).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved