Method Article
İzolasyon ve Zebra balığı Embriyolar gelen Tek Hücre Karakterizasyonu
Bu Makalede
Özet
This protocol describes a method for isolating single cells from zebrafish embryos, enriching for cells of interest, capturing zebrafish cells in microfluidic based single cell multiplex systems, and assessing gene expression from single cells.
Özet
The zebrafish (Danio rerio) is a powerful model organism to study vertebrate development. Though many aspects of zebrafish embryonic development have been described at the morphological level, little is known about the molecular basis of cellular changes that occur as the organism develops. With recent advancements in microfluidics and multiplexing technologies, it is now possible to characterize gene expression in single cells. This allows for investigation of heterogeneity between individual cells of specific cell populations to identify and classify cell subtypes, characterize intermediate states that occur during cell differentiation, and explore differential cellular responses to stimuli. This study describes a protocol to isolate viable, single cells from zebrafish embryos for high throughput multiplexing assays. This method may be rapidly applied to any zebrafish embryonic cell type with fluorescent markers. An extension of this method may also be used in combination with high throughput sequencing technologies to fully characterize the transcriptome of single cells. As proof of principle, the relative abundance of cardiac differentiation markers was assessed in isolated, single cells derived from nkx2.5 positive cardiac progenitors. By evaluation of gene expression at the single cell level and at a single time point, the data support a model in which cardiac progenitors coexist with differentiating progeny. The method and work flow described here is broadly applicable to the zebrafish research community, requiring only a labeled transgenic fish line and access to microfluidics technologies.
Giriş
hücre ve moleküler biyoloji en güncel çalışmalar nüfus ortalamasına dayanmaktadır. küçük popülasyonlar biyolojik süreçlerin ve hastalık sonucu önemli roller oynayabilir Bununla birlikte, önemli biyolojik olaylar bu geleneksel toplum tabanlı analizler maskelenmiş olabilir. Tek hücre düzeyinde heterojen popülasyonlar gen ifadesini anlama (ve vardır) ilgili biyolojik ve klinik Insights 1,2 yol açabilir. Embriyonik gelişim çalışmalarına ilgilendiren, daha büyük bir hücre popülasyonunda, progenitör hücreler, çoğunlukla az temsil edilmektedir zorlu sonuçta hücre kaderinin kararları 3 başlatmak gen ifadesinde ince değişiklikleri tespit etmek için yapım. Benzer şekilde, tek bir hücre tipi mikro-4 tepki olarak farklı ekspresyon profillerine sahip olabilir. Örneğin, (örneğin., Aort veya böbrek) aynı organ veya farklı organlarda yerleşik endotel hücreleri ortak Morp paylaşımı rağmen önemli heterojeniteye sergilerlerlitolojik ve fonksiyonel özellikleri 5. Buna ek olarak, aynı tümör doldurma kanser hücreleri, aynı zamanda, tek hücre düzeyinde 6 moleküler profiller ya da mutasyonlar farklı olabilir.
Modeli sistemlerinde, tek hücre transkriptomik başarılı yeni hücre popülasyonu tespit hücre farklılaşması sırasında meydana gelen ara durumları, özelliği ve 7,8,9 uyaranlara diferansiyel hücresel tepkileri ortaya çıkarmıştır. Bu tür anlayışlar, geleneksel toplum tabanlı çalışmalarda maskeli olurdu. Zebra balığı embriyolar kök, dede bir derece altında kullanılan kaynağıdır ve geliştirme sırasında tek hücre heterojenite ve hücresel kimliklerin moleküler düzenleme soruları keşfetmek için hücrelerin ayırt. Onların son derece basmakalıp, ex vivo geliştirme ve genetik manipülasyon kolaylığı onlara bu yaklaşımın 10,11 için mükemmel bir model sistem olun. Özellikle, tek hücre gen yorumlanması önemli bir sınırlamae ifade veri gelişimi sırasında yeni ara hücre devletlerin güvenilir kimlik doku koleksiyonu 9 çok dikkatli zamanlama gerektirir. Bu tutulan hücre arasındaki farklılıklar, yaşa bağlı hücre farklılaşması tarafından sunulan gen ekspresyonu, tek bir zaman noktasında bir doku içinde heterojen yerine heterojen temsil sağlamak için gereklidir. Fareler ile karşılaştırıldığında, zebra balığı embriyo gelişimi tam olarak embriyoların 12 çok sayıda senkronize edilebilir. Buna ek olarak, geniş bir kavrama boyutları ile, zebra balığı embriyolar kök ve projenitör hücre bol kaynağı olarak kullanılabilir.
Bu protokol, zebra balığı embriyoların hücreleri izole etmek ve QRT-PCR ile gen ekspresyon analizi için ticari olarak temin edilebilir, entegre Mikroakiskan devresi (IFC) çipi ve otoprep sistemi kullanılarak tek hücre yakalamak için bir yöntemi tarif etmektedir. Bu protokol, bütün dahil olmak üzere herhangi bir yüksek verimli çoğullama deneyleri hızla devredilecek olabilirHücresel heterojenite 13 daha kapsamlı analiz sağlar transcriptome sıralama. Ayrıca, geleneksel gen sentezleme tahlilleri için çeşitli avantajlar sağlar. Tek hücre izolasyon protokolü alt uygulamalar dahildir tehlikeye hücrelerinin oranını azaltır FACS sonrasında yüksek canlılığı, elde edilir. Bir IFC kullanarak, yakalanan hücreler doğrudan yakalama oranlarını değerlendirmek ve morfolojik hücre sağlığını değerlendirmek için görülebilir. Buna ek olarak, bu protokol mikroakışkan hücre yakalama teknolojileri sadece etiketli transgenik balık hattı ve erişimi gerektiren, Zebra balığı araştırma topluluğuna geniş çapta uygulanabilir.
Prensip kanıtı olarak, kalp progenitörlerinden edilen tek hücreler izole edilmiş ve bir IFC çip ele geçirilir ve daha sonra kalp farklılaşma markerlerinin nispi bolluk QRT-PCR ile ölçülmüştür. tek hücre düzeyinde gen ekspresyon analizi kalp atalarıdır kendi differe bir arada gösteriyorntiating soyu. Kalp atalarıdır tek hücreli profil elde edilen anlayış, geleneksel toplum tabanlı analizlerde maskelenmiş olabilir omurgalı gelişimi sırasında kalp progenitör hücreleri arasında gen ekspresyonu heterojenlikten, ışık tutabilir.
Protokol
Bu protokol, embriyolar üretme yaşarlar, ergin zebrabalıkları kullanılmasını gerektirir. embriyolar doku toplama hasat edilir. Deney yapmak için uygun etik inceleme kurulları onay almak için esastır.
1. Sahnelenen Embriyolar elde
- Deneyden bir gün önce, üreme için sağlıklı, yetişkin zebrafish hazırlamak. bir erkek ve bir üretim tankı açık bir bölücünün karşılıklı yanları üzerinde bir dişi yerleştirin.
- alt uygulama için yeterli embriyo üretimi için gerektiği kadar üreme tankları için 1.1 tekrarlayın. ilgi hücre tipinde floresan proteinleri ifade vahşi tip balık ve transgenik balıklar hem embriyolar elde edin.
NOT: Adımlar 2-8'de downstream uygulamalar için gerekli embriyo sayısı ilgi bir zaman noktasında ilgi hücrelerin göreceli bolluk bağlıdır. Bu hücre türüne göre değişebilir rağmen, 200 embriyolar 2,000-5,000 sıralı hücreler üretmek zaman cilgi arşın 24 hpf (saat sonra döllenme) de - Ertesi sabah, taze bir üreme tankına balık aktararak breading tankındaki suyu değiştirmek ve bölücü çıkarın. üreme teşvik etmek için bir açı tankı yatırın.
- sahnelenen embriyolar toplayın.
- Her 15 dk, taze üreme tankına yetişkinleri aktarılması ve çay süzgecinden geride yumurta geçerek embriyolar toplamak.
NOT: karşılaştırılabilir yoğunlukları ve sıcaklıklarda muhafaza zaman Zebra balığı embriyolar eş zamanlı olarak gelişir. - (1 L çift distile su içinde 0.21 g / L Anında okyanus tuzları) Yumurta Su ile yumurta durulayın ve bir petri transfer. hava sirkülasyonu ile 28.5 ° C'de nemli inkübatör petri aktarın.
- Her 15 dk, taze üreme tankına yetişkinleri aktarılması ve çay süzgecinden geride yumurta geçerek embriyolar toplamak.
- İki saat son toplanmasına, sıralama 10 cm petri kutularının içine çok hücreli embriyo döllenmiş ve çanak başına 50 embriyolar yoğunluğunu azaltır. Tek, 15 seçin embriyolaralt uygulama için toplama dakika zaman penceresi. 28.5 ° C embriyolar inkübe edin.
NOT: Örneğin, 08:30, 08:45, 09:00, 09:15, 09:30, 09:45, 10:00 ve 10:15 AM embriyolar toplamak. döllenmiş embriyo en fazla sayıda 9:00 toplanan pençelerinden ise zaman noktalarındaki karşılaştırıldığında, daha sonra aşağı uygulamalar için sadece bu embriyolar kullanın.
2. Tek Hücre Ayrışma için Kurma
- önce ilgi (18 hpf) zaman noktasına yaklaşık 30 dakika ince forseps ile el koryon embriyolar kaldırmak.
- Toplamak ve her durum için aşağıdaki etiket: İki 2 ml mikrosantrifüj tüpler, bir 40 mikron hücre süzgeç, bir 35 mm hücre kültürü çanak ve 35 mikron hücre süzgecinden ile tepesinde iki FACS tüpleri.
- Buz üzerinde aşağıdaki reaktifler Chill: Yumurta Su 1L çift distile su 0.21 g / L Anında Okyanus tuzları içeren; 55 mM NaCI, 1.8 mM KCI, 1.25 mM NaHCO 3 ihtiva eden tampon De-yolking; ve FACS Tampon Leibovitz L-15,% 5 ısı ile inaktive edilmiş fetal büyükbaş hayvan serumu ile takviye edilmiş ortam ihtiva etmektedir.
- RT Hücre Ayrılma Reaktif 1 numune başına 1 ml getirin. buz üzerinde örnek başına çözülme 1 ml Hücre Ayrılma Reaktif 2. Kullanmadan önce hemen oda sıcaklığına kadar getirin.
3. Tek Hücre Ayrılma
- Geniş delikli cam pipet kullanılarak, yumurta suyun minimum bir hacmi içinde 2 ml mikro santrifüj tüpüne 100-300 embriyolar aktarılır.
- 20 dakika boyunca buz içinde tüp 1 ml buz gibi soğuk yumurta su ile su değiştirilmesi ve daldırarak embriyolar öldürülür.
DİKKAT! (Ikincil ötanazi gibi hücre ayrışma ardından buz üzerinde ürpertici) bu ötenazi yöntemi için uygun kurumsal hayvan bakım gözetim komitesinden onay almak için çok önemlidir. Standart ötanazi tipik embriyolar da canlı hücreleri elde etmek için uygun değildir ki, soğutulmuş sonra ağartılmalarına nispeten 3 gün sonra gübreleme - 1 ml buz gibi soğuk Yumurta suyla embriyolar iki kez yıkayın. yıkamak, Yumurta Suyu ve Yumurta Su eklemek için bir P1000 kaldırmak için geniş bir delik cam pipet kullanın.
- P1000 ucu ile 8-12 kez 1 ml Deyolking Tampon ile embriyo su değiştirilmesi ve öğütülerek sarısı çıkarın veya sarısı çözülür ve kadar embriyoların sadece organları görebilir.
- 1 dakika boyunca 300 x g'de santrifüj ile doku toplanır. yavaşça doku pelet bozmadan süpernatant kaldırmak için bir pipet kullanın. 1'in ml Yumurta Su yeniden askıya.
- Tekrarlayın üç yıkar toplam adım 3.5, ancak son yıkamada, 1 ml RT Hücre Ayrılma Reaktif 1 yeniden askıya.
- yatay olarak yerleştirilmiş borular ile oda sıcaklığında 10 dakika boyunca hücre ayrışma Reaktif 1 inkübe edin. Her 2-3 dk hafifçe topaklanma önlemek için P1000 pipet ile çiğnemek.
NOT: ezerek toz haline getirme adımları sırasında hafifçe örnekleri taşıyınız. Bazen tek bir büyük yığın embriyo organları bir arapsaçı gelen tüp içinde oluşacaktır. Bu iradeNazik toz haline getirilerek destekli ek sindirim ile dağıtmak. VORTEKS DO NOT. - 3 dakika boyunca 300 x g'de santrifüj ile doku toplanır. 1 ml Hücre Ayrılma Reaktif 2 supernatant ve tekrar süspansiyon çıkarın.
- Oda sıcaklığında 5-15 dakika boyunca inkübe edin. yatay tüpleri yerleştirin. Her 2-3 dk hafifçe topaklanma önlemek için çiğnemek. Her 5 dk, sindirim ilerlemesini değerlendirmek.
- , Sindirim ilerlemesini değerlendirmek hücre kültürü çanak üzerine bir damlacık olarak 18 ul FACS Tampon ve pipet içine 2 ul süpernatant sulandırmak için.
- numune üzerinde bir lamel yerleştirin ve 10X ve 20X büyütmede bir doku kültürü mikroskop altında gözlemlemek.
NOT: Hazırlık tek hücreler, küçük kümeler, büyük kümeler, ve zaman zaman çoğunlukla-sağlam embriyo vücudun bir karışımı olarak görünmelidir. - Görme tek hücreleri ve küçük kümeler olan hazırlık oranını değerlendirmek.
NOT: Aşırı sindirim canlılığını azaltır; altında sindirim tek hücre verimi azaltacaktır.
- 5 dakika boyunca 300 x g'de santrifüj ile hücre hazırlama toplayın. Süpernatantı atın ve soğuk FACS Tampon 1 ml yeniden askıya.
- FACS Tampon ile 40 mikron hücre süzgeç ıslatın. 35 mm hücre kültürü çanak üzerine 40 mikron hücre süzgecinden hücre süspansiyonu geçirin. tampon sıralama 1 ml FACS hücre süzgeci bir kez yıkayın.
- 2 ml mikrosantrifüj tüp akış yoluyla aktarın. 5 dakika boyunca 300 x g'de santrifüj ile hücreler toplamak ve 100 ul FACS tampon içinde yeniden askıya.
- Bir hemasitometre kullanarak hücre verimi güvenin. Dahası seçim FACS makine için önerilen ml veya optimum konsantrasyon başına 5x10 6 hücre örnekleri sulandırmak. hemasitometre hücre verimi sayma yaparken isteğe bağlı, tripan mavisi ile counterstain ölü hücreler FACS sıralama öncesinde hücre hazırlık canlılığı teyit etmek.
NOT: Çok sıralamak için zaman aşırı miktarda alacak seyreltilmiş Hazırlıklar. çok c vardır Hazırlıklaroncentrated multimerler içine gelme eğilimindedir ve saf nüfus sıralamak için optimal bulunmaktadır. - Rezerv her numunenin% 10-20 olarak boyanmamış denetimlerini kullanmak için. ekleyerek diğer örnekler, leke ölü hücreler bir floresan ölü / canlı (L / D) ayrım Her numune içine boya. yıkama ETMEYİN.
Not: nükleik asitler, L / D boyası olarak kullanılabilir tehlikeye hücre zarları ve lekeleri hücreleri nüfuz herhangi bir flüoresan boya, florasan spektrumu ilgi seçimi ve fluorofor markalama hücrelerinin FACS makine ile uyumlu olması koşuluyla.- bazı hücreler FACS saflaştırma transit ölecek ve sıralı nüfus dışında tutulmalıdır olarak boya ekledikten sonra hazırlık yıkamayın.
- 20 ul FACS tamponu ile FACS tüp şapkalı 35 mikron hücre süzgeç ıslatın. süzgeç hücreleri ekleyin ve yerçekimi tarafından toplanır. Buz üzerinde muhafaza edin.
4. FACS Zenginleştirme
- için tek zenginleştirmek için FACS kullanınFloresan markörü eksprese eden canlı hücreler.
- yan dağılım genlikli (SSC-A) vs ileri dağılım (FSC-A) genlik bir dağılım arsa üzerinde enkaz hücreleri ayırt etmek için bir kapı olarak ayarlayın, hem doğrusal ölçeklendirme ile.
Dikkat balığı hücreler tipik olarak bir fare veya insan hücrelerine göre daha küçüktür. Bu hücreler ve döküntüler, arasındaki düşük bir bazal ayırma yansıtılır. - 4.1.1 set kapısından, tek hücre için zenginleştirmek ve ileri saçılım yüksekliği (FSC-H) ve düşük yan dağılım yüksekliği (SSC-H) bir dağılım grafiğini kullanarak multimerlerini, doğrusal ölçekleme hem dışlamak için bir kapı olarak ayarlayın.
NOT: orantısız yüksek FSC-H ve düşük SSC-H hücreler muhtemel multimerleri ve sıralama dışındadır. - tek renk kontrollerini kullanarak 4.1.2 gate seti, doğrusal ölçekleme ve kanal genliği ile FSA-A'nın bir dağılım grafiği kullanan tek canlı hücreleri dahil bir kapı set günlük ölçeklendirme ile L / D leke tespit etmek için kullanılır. L / D negatif hücreleri içerir.
- itibarenkapı, tek renk kontrollerini kullanarak, 4.1.3'de belirlenen günlük ölçekleme ile floresan protein hücre işaretleyici tespit etmek için kullanılan kanalın lineer ölçeklendirme ve genlik ile FSA-A'nın bir dağılım grafiği kullanılarak pozitif floresan ile tek canlı hücreleri dahil bir kapı set . Pozitif hücreler yer alır.
- Gerekirse L / D leke ve floresan protein spektrumları arasındaki girişim için hesap, herhangi bir tazminat kontrolleri ayarlayın.
- yan dağılım genlikli (SSC-A) vs ileri dağılım (FSC-A) genlik bir dağılım arsa üzerinde enkaz hücreleri ayırt etmek için bir kapı olarak ayarlayın, hem doğrusal ölçeklendirme ile.
- Adım 4.1 ayarlanan kapılarının her giren hücrelere ayarlayın sıralama mantığı.
- çift etiketli hücreleri kullanarak, hücre sıralama için yolluk doğrulayın.
NOT: Hücreler yerine enkaz, tek hücreler yerine multimer, L / D negatif ve floresan pozitif hücreler olacaktır sıralama için hücreler.
- çift etiketli hücreleri kullanarak, hücre sıralama için yolluk doğrulayın.
- Buz üzerinde bir mikrosantrifüj tüp içinde soğuk FACS Tampon 5 ul içine ilgi nüfus kriteri 2000-4000 hücreleri. hücre için mümkün olan en düşük basınçlar kullanmak, sıralama sırasında hücreler üzerinde kesme ve zorlanma en aza indirmek içinsıralayıcı.
NOT: FACS Tampon 5 ul damlacık FACS makinesinden çıkan hücreler için bir yastık görevi görür. şirketinden FACS tamponuna 2000-4000 hücrelerin toplanması öncesinde IFC fiş üzerine yüklemeden santrifüj ihtiyacını ortadan kaldırır. Hücreler pelet birbirine bağlı olmadığını santrifüj multimerlerinin oluşumuna yol açabilir, bu strateji, tavsiye edilir. - sonrası sıralama canlılığı analizi değerlendirin.
- L / D leke 1000 seyreltme: Transferi 1 ul tampon ve 1 sıralama 100 ul FACS ile taze bir FACS tüp hücreleri sıralanır. Adım 4.1 yolluk stratejisi ile FACS sıralayıcı aracılığıyla hücrelere geçerler. L oranını kullanın / negatiften pozitife D sıralı hücrelerin canlılığı tahmin etmek.
Mikroakiskan Chip üzerine 5. Yük Hücreleri
- hemasitometre kullanarak, konsantrasyon ve sıralanmış hücrelerinin boyutunu hem de ölçün.
- en az 10 ul sıralı hücreleri sulandırmak. 5 u bir kısım seyreltin, 5 ul tripan mavisi ile L hücreleri. hemasitometre üzerine yerleştirin ve lamel uygulayın.
- hemasitometre 4 x 4 ızgaraları tüm hücrelerin parlak bir alan görüntüleri toplamak. Canlı hücrelerinin sayısını ve canlı hücre / ml hesaplar. Canlı hücreler tripan mavisi kadar yapmayız.
Not: Tüm canlı hücre çapını ölçmek için herhangi bir standart Image Analysis yazılımının kullanılması. hücre boyutu, ortalama ve standart sapma hesaplayın ve ilgi hücre boyut aralığı için uygun olan bir IFC plaka seçin. Hücre boyutu aralığı bir IFC plaka hücre boyut aralığı straddles ise ilgi hücreleri tam kapsamlı yakalamak için birden fazla tabak kullanın.
- üreticinin talimatlarına göre IFC plakaya Yük hücrelerinin (Malzeme).
- 1x10 6 hücre / ml hücreleri sulandırmak ve üreticinin talimatlarına göre yüzdürme optimizasyon yapmak.
NOT: Yüzdürme optimizasyon hücreleri dibine çöker, ne de yükleme wel üstüne şamandıra ne sağlarIFC çip üzerine optimum yükleme l. hücrelere tampon oranı hücre tipine göre değişir, ancak genellikle 6 aralıkları olabilir: 4-7: Hücrelerin 3: tampon. - astar-IFC plakasına hücreleri ekleyin. Yük uyumlu Fluidics makinenin içine plaka ve Mikroakiskan devre boyunca ve yakalama şerit içine hücreleri itmek için bir hücre yükleme komut dosyasını çalıştırın.
- 1x10 6 hücre / ml hücreleri sulandırmak ve üreticinin talimatlarına göre yüzdürme optimizasyon yapmak.
- Hücreler IFC plaka üzerinde yakalama sitelerinde açılmış olduğunu doğrulamaktadır.
- Fluidics makineden IFC plakasını sökün. bir tabak adaptörü ile donatılmış bir mikroskop IFC plakasını monte edin.
- 10X büyütmede her yakalama site için parlak bir alan ve floresan anlık toplayın.
NOT: aydınlık yakalama süreleri lamba yoğunluğuna bağlı olarak, 10-50 ms arasında değişebilir. Floresan yakalama süreleri fluorofor parlaklığına bağlı olarak, 250-750 ms arasında değişebilir. fotohasar önlemek için hücrelerin ışığa maruz kalmaması.
6. cDNA Sentezi
- I'e göre yerinde hücre parçalama gerçekleştirinFC plaka üreticinin talimatlarına.
- IFC plaka üzerinde kuyu lizis tamponları ekleyin. üreticinin talimatlarına göre uyumlu fluidik makine ve çalışma hücre parçalama komut üzerine yük plakası.
- IFC plakası, üreticinin talimatlarına uygun olarak, ters transkripsiyon gerçekleştirin.
- IFC plakasına ters transkripsiyon ayraçlar eklenir. Uyumlu Fluidics makine üzerine yük plakası. Ters transkripsiyon komut dosyasını çalıştırın. Örnek döngü koşulları: 5 dakika 85 ° C'de daha sonra da 1 döngü, 10 dakika boyunca 25 ° C 'de 1 döngü, 1 saat süre ile 42 ° C' de 1 döngü.
- IFC plakası, üreticinin talimatlarına uygun olarak gene özel problar ile ön amplifikasyon gerçekleştirin.
Not: düşük kopya numaraları ile daha kesin nedeniyle, ara katkı boyalar ile kullanım için optimize florojenik etiketli problar yerine probları kullanımı.- Havuz astar ve son 180 nM her sulandırmak. IFC plakasına toplanmış primerler ekleyin. Uyumlu fluidi üzerine yük plakasıcs makinesi. Run amplifikasyon komut dosyası.
- Aşağıdaki çevrim koşulları ön amplifikasyon yapın: 1 döngü, 95 ° C'de 10 dakika, daha sonra da 18 döngü, 4 dakika 60 ° C'de, daha sonra 15 saniye boyunca 95 ° C 'de tavlama ve uzatma, denatüre edici ile. Mağaza hasat kadar 4 ° C 'de cDNA önceden büyütülmüş. kullanılıncaya kadar -20 ° C'de üreticinin talimatlarına ve saklamak için uygun mikroakışkan plakadan Hasat cDNA.
Tek Hedef QRT-PCR Tek Hücreleri 7. Select cDNA
- üreticinin talimatlarına uygun olarak 25 ul dilüsyon reaktif cDNA seyreltilir.
NOT: Yaklaşık son verim hücre başına ön amplifiye cDNA 28 ul. QRT-PCR bir hedef içermeyen bir kontrol olarak kullanılmak üzere bir seyreltici bir kısım ayırın.- parlak bir alan ve adım 5.3.2 kaydedilen floresan görüntülere değinen her yakalama sitesine puan. El ile saymak ve hücre sayısını kaydedin. Değerlendirmek ve her hücre grip olup olmadığını kayıtFlüoresan.
NOT: Her birey sitesi içerebilir 0,> 1 hücre birden çok hücre ve / veya tanımlanamayan enkaz ihtiva yakalama siteleri içeren 1 veya> 1 hücre. görüntüler yeterli kalitede ise, bir piksel değeri de floresan sinyal yoğunluğu ölçmek için atanabilir.
- parlak bir alan ve adım 5.3.2 kaydedilen floresan görüntülere değinen her yakalama sitesine puan. El ile saymak ve hücre sayısını kaydedin. Değerlendirmek ve her hücre grip olup olmadığını kayıtFlüoresan.
- Mah-PCR analizi için seçin örnekleri.
- Pozitif floresan ile tam 1 hücreyi ihtiva adım 7.2 tanımlanan yakalama sitelerinden cDNA örnekleri seçin. Her bir numuneden cDNA havuzunu 1 ul alikotları.
NOT: Bu ilgi genler seçilen hücrelerin herhangi ifade olup olmadığını belirlemek için kullanılan "Havuz" pozitif kontrol edilmesidir. - Bir negatif kontrol olarak tam olarak 0 hücreleri içeren en az bir yakalama sitesinden cDNA seçin. no-şablon kontrolü gibi adım 7.1.2 den seyreltici kullanın.
- Pozitif floresan ile tam 1 hücreyi ihtiva adım 7.2 tanımlanan yakalama sitelerinden cDNA örnekleri seçin. Her bir numuneden cDNA havuzunu 1 ul alikotları.
- Mah-PCR testi çalıştırın.
- Aşama 6.4.1 ön-yükseltme için kullanılan tek probları kullanın. triplicat tüm örnekleri yükleyine. 1 döngü, 50 ° C 2 dakika, 1 döngü, 10 dakika için 1, 60 ° C'de, sonra 95 ° C 15 saniye tavlama ve uzatma denatüre edici ile 40 döngü 95 ° C: 384 plaka üzerinde 10 ul reaksiyon için döngü koşulları min.
8. Veri Analizi
- Standart jel elektroforezi ile Mah-PCR ürünleri doğrulayın. Ürünü onaylamak beklenen molekül ağırlığında tek bir bant olduğunu (her sonda için değişir).
NOT: Bir sonda uygunsuz boyutta birden çok bantları veya tek bir bant üretirse, o zaman özgünlüğü sorgulanabilir ve bu analizden geni dışlar. - Herhangi bir anormallik 14 tüm amplifikasyon eğrileri ve hesabı inceleyin. Her bir numune / gen kombinasyonu 15 için ortalama BT değerini hesaplayın.
NOT: Pozitif kontroller CT değerleri tipik 10-30 arasında değişmektedir. Negatif kontroller CT değerleri tipik 35-40 (hiçbir amplifikasyon) arasında değişir. BT değerleri 30-35 derece düşük ifadesi olarak kabul edilirve dikkatli 14 ile yorumlanmalıdır. - Rapor verileri
- Numuneler CT = 10-30 düşersen, veriler de bir kontrol numunesine transkript bolluk katlık değişim olarak bildirilebilir.
Not: (- ΔΔCT) katlı değişim 2 ^ eşittir ACt ev bakıcısı geni ile ilgili numunenin ortalaması CT çıkarılarak bir ev bakıcısı geni ile ilişkili ve ΔΔCT numunesi ve bir pozitif kontrol 15 ile ACt farklılıklar olduğunu.
- Numuneler CT = 10-30 düşersen, veriler de bir kontrol numunesine transkript bolluk katlık değişim olarak bildirilebilir.
Sonuçlar
Prensip kanıtı olarak, gen ifadesi kalp gelişimi sırasında farklılaşma dinamikleri keşfetmeye değerlendirildi. zebrabalıkları kardiyak ataları Bunların, doğrusal kalp tüpü oluşturmak için sigorta ön yan levha mezoderm yer değiştiren hücrelerin mezodermal nüfusu ortaya çıkar. Füzyon öncesinde, kalp ataları kalp atalarıdır 16,17 erken belirli belirteç olduğu düşünülmektedir transkripsiyon faktörü Nkx2.5 (NK2 homeobox 5), ifade başlar. Burada, daha önce tarif edilen BAC transgenik balık Tg (Nkx2.5: ZsYellow) 18, Nkx2.5 kısaltılmıştır: ZsY 18 hücre gruplarının aşamasında, 18 saat sonra fertilizasyon (HPF) 12, tek hücre kalp farklılaşma belirteçleri incelemek için kullanılmıştır. Bu ZsYellow sinyali görsel olarak saptanabilir olduğu en erken zaman noktası oldu. Daha önce, bu transjenik soy için tarif edildiği gibi, ZsYellow olarak kardiyak progenitörlerin etiketler Biz28 hpf 19 de faringeal kemer endotel hücreleri doğuran bir kaç ekstra kardiyak hücreler olarak ll (Şekil 1A-B, veriler gösterilmemiştir). 18 hpf, Nkx2.5: ZsY embriyolar daha sonra ölü hücreleri hariç Sytox Blue ile lekelenmiş, bir tek hücre süspansiyonuna aktarılmış ayrıştırılmış. Canlı, ZsYellow pozitif Sytox Mavi negatif hücrelerin FACS 100 um meme (Şekil 1C-F) ile donatılmış bir MoFloXDP Sony SH800Z sıralayıcı kullanılarak kriteri. Kriteri nüfusu ve sonrası sıralama canlılığı saflığını değerlendirmek için kriteri hücrelerin bir tümböleni Sytox Blue ile lekelenmiş ve orijinal ayırma kapısı (Şekil 1G-J) giren etkinlikleri yüzdesi değerlendirildi.
Ayırma işleminden sonra hücreleri, üretici talimatlarına göre bir entegre mikro-akışkan devresi (IFC) yonga iş akışı (Şekil 2A) katılmıştır. tripan mavisi exclusio ile birlikte bir hemositometre,N hücre çapı, konsantrasyon ve canlılığını ölçmek için kullanılmıştır (Şekil 2B ve veriler gösterilmemiştir). (: 3.5 hücreleri: 6.5 tampon) Hücre canlılık optimize edildi üreticinin talimatlarına göre ve hücreler 5-10 mikron çapında hücreleri yakalamak için bir IFC yüklenmiştir. yakalama verimliliği, floresan ve / veya tüm yakalama sitelerinde görüntülendi parlak bir alan sinyallerini değerlendirmek için, FIJI bir döşeme fonksiyon Mikroakiskan plaka tek bir resim dikmek için kullanıldı. Her ele geçirme bölgesini de 0, 1 ya da> 1 tek tek hücreler (Şekil 2C-F) mevcuttur. FACS zenginleşmeden Beklendiği gibi, yakalanan hücreler ZsYellow (Şekil 2G) olarak ifade edilmiştir. Yakalama verimi Nkx2.5 kullanılarak 5 ayrı deneylerde% 90 aşıldı: ZsY hücreleri kriteri ve yakalama siteye en az% 70 tek bir hücre tarafından işgal edilmiştir (veriler gösterilmemiştir). Hücreler üreticinin talimatlarına göre, RNA ile izole edilmiş cDNA sentezlenir ve belirli hedef genlerinin çoğaltılmıştır, lize edildi.
Yukarıda protokolde tarif edildiği gibi hücre yakalama alanların bir alt düzeyinde QRT-PCR analizi için seçildi. Özel olarak, uzama faktörü 1a (efl1a), 20 ve gliseraldehid-3 fosfat (GAPDH) 21, her hücrede ifade edilmesi beklenen hazırlık geninin olarak tahlil edildi; GATA bağlayıcı protein 4 (gata4) 22 ve NK2 homeobox 5 (Nkx2.5) erken kalp progenitör belirteçler; İkinci kalp saha markeri 23,24 olarak ISL LIM homeobox 1 kutu 1 (isl1); ve miyozin hafif zincir (myl7) ve ventriküler miyozin ağır zincir (vmhc) 25 ventriküler kardiyomiyositlerde işaretlemek için. Gene özel problar 48 HPF embriyolar (Şekil 3) cDNA kullanılarak doğrulanmıştır. QRT-PCR 18 HPF embriyo, bir hedef içermeyen negatif kontrol 45 tek hücreler, bu genlerin göreceli gen ekspresyonu değerlendirmek için kullanılan ve bir havuz popülasyon pozitif kontrol containi edildi96 yakalama sitelerinden ng cDNA. 40 temsilci hücreleri ve kontroller için ham CT değerleri tüm gen ifadesi BT değerleri CT = 35.0 aştığından incelenen 46 hücrelerinin havuzundan, 6 hücreli analize dahil edilmemiştir, Özellikle Tablo 1'de gösterilmiştir ve bilinmeyen bu yüksek BT olsun değerler true, çok düşük ekspresyon seviyeleri ya da örnek bozulması atfedilebilir. Temizlik gen, ef1a CT değer aralığı bu yana, numuneler arasındaki gen ifadesini karşılaştırmak için çok geniş olduğunu ve birçok BT değerleri 30, CT değerleri bir ısı haritası (Şekil 3) olarak görüntülendi aştı. Örnekleri arasında karşılaştırıldığında, gen ekspresyonunda önemli bir heterojenlik gözlendi ve hücreler Tanım örneklerine göre Tip 1-5 (Şekil 3) gibi hücreler sınıflandırıldı.

Zebra balığı 1. Tek hücre izolasyonu Şekil Nkx2.5: 18 hpf ZsY pozitif hücreler temsilcisi Tg (Nkx2.5: ZsYellow) Whole montaj görüntülerini. embriyo başına (A) ZsYellow floresan veya (B) ile 18 hpf parlak bir alan görüntü ile birleşti. (CF) Temsilcisi FACS yolluk strateji ZsYellow pozitif hücrelerin ve zenginleştirmek üzere (GJ) (C, G) FSC / SSC boyut yolluk, (D, H) çifti ayrımcılık, (E, I) Live / Dead yolluk post-sıralama analizi ve (F, J) nüfus sıralanır. Ölçek çubuğu 100 mm. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
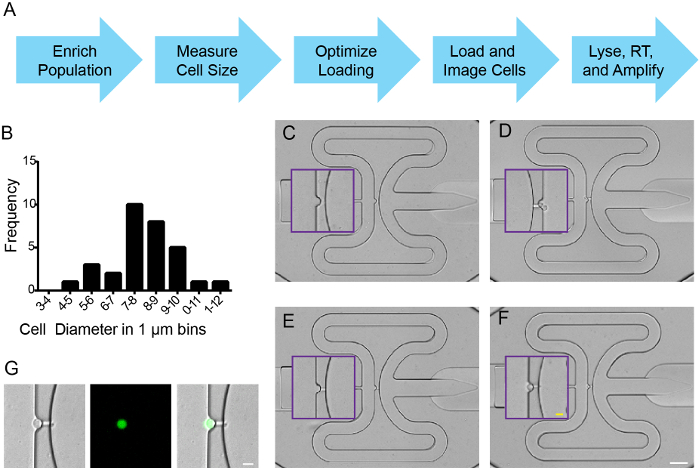
Şekil 2. Zebra balığı Nkx2.5 Tek hücre yakalama: ZsY ong> pozitif hücreler. (A) İş akışı. 18 hpf izole edilmiş embriyolar: Tg (ZsYellow Nkx2.5) için kriteri hücreleri için (b) hücre boyutu dağılımı. (C) Boş de olduğu (CF), IFC plaka Örnek hücre yakalama olaylar, (D) iki hücre, (E) "Kanal yakalama" ve (F) gibi fluidik kanal açılmış tek bir hücre yer alır içeriyorsa tek bir esir hücre. Mor kutular yakalama siteleri büyütülmüş görünümü için vaziyettedir işaretlemek. (G) aydınlık, ZsYellow Tek hücreli yakalama ve görüntüleri birleşti. (CF) Beyaz ölçek çubuğu 50 mikron olduğu; Sarı ölçek çubuğu 10 mm. (G) Ölçek çubuğu 10 mm. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
ŞEKIL 3 "src =" / files / ftp_upload / 53877 / 53877fig3.jpg "/>
Yakalama tek zebrabalıkları Nkx2.5 Şekil 3. Gen ifadesi analizi:. ZsY pozitif hücreler pozitif kontrollerde QRT-PCR ile gen ifadesi (2 gün-sonrası döllenme embriyo, tek hücre cDNA toplanmış), negatif kontrol (no-şablon ) ve 40 tek hücre (Cell 01-40). Key açıklandığı gibi Ham BT değerleri renk kodlu esas alınmıştır. Ef1a = uzama faktörü 1a, gata4 = GATA bağlayıcı protein 4, Nkx2.5 = NK2 homeobox 5 myl7 = miyozin hafif zincir 7, vmhc = ventriküler miyozin ağır zincir, GAPDH = gliseraldehid-3 fosfat ve isl1 = ISL LIM homeobox 1. Bu rakamın büyük halini görmek için lütfen buraya tıklayınız.
| Döngü Eşiği (BT) | ||||||||
| ef1a | gata4 | nkx25 | myl7 | vmhc | GAPDH | isl1 | ||
| Embriyo | 20.69 | 20.92 | 28.59 | 32.87 | 26.30 | 27.00 | 33.57 | |
| havuz | 26.4 | 22.4 | 27.7 | 27.4 | 24.5 | 29.2 | 40.0 | |
| NTC | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Tip 1 | Hücre 01 | 25.1 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 |
| Hücre 02 | 25.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre 03 | 26.3 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre 04 | 27.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre 05 | 28.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre 06 | 28.2 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre 07 | 29.3 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 400,0 | |
| Hücre 08 | 30.7 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre 09 | 34.3 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Tip 2 | Hücre 10 | 23.5 | 28.0 | 40.0 | 40.0 | 40.0 | 26.3 | 40.0 |
| Hücre 11 | 23.5 | 27.2 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre 12 | 23.7 | 17.9 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre 13 | 24.1 | 32.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre 14 | 24.9 | 27.5 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre 15 | 25.9 | 28.6 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre-16 | 26.0 | 28.1 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre 17 | 27.0 | 27.7 | 40.0 | 40.0 | 40.0 | 38.5 | 40.0 | |
| Hücre 18 | 27.0 | 27.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre-19 | 27.0 | 30.2 | 40.0 | 40.0 | 40.0 | 38.2 | 40.0 | |
| Hücre 20 | 27.0 | 28.2 | 40.0 | 40.0 | 400,0 | 39.2 | 40.0 | |
| Hücre 21 | 27.7 | 30.3 | 40.0 | 40.0 | 40.0 | 38.6 | 40.0 | |
| Hücre-22 | 30.4 | 26.2 | 40.0 | 40.0 | 40.0 | 38.3 | 40.0 | |
| Hücre 23 | 31.3 | 22.4 | 40.0 | 40.0 | 40.0 | 39.7 | 40.0 | |
| Hücre 24 | 31.5 | 28.8 | 40.0 | 40.0 | 40.0 | 39.5 | 40.0 | |
| Hücre 25 | 33.8 | 27.4 | 40.0 | 40.0 | 40.0 | 37.3 | 40.0 | |
| Hücre 26 | 40.0 | 27.4 | 40.0 | 40.0 | 40.0 | 36.6 | 40.0 | |
| Tip 3 | Hücre 27 | 24.7 | 19.9 | 28.8 | 40.0 | 40.0 | 40.0 | 40.0 |
| Hücre 28 | 24.8 | 28.4 | 26.3 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre 29 | 25.1 | 23.5 | 27.6 | 40.0 | 40.0 | 32.5 | 40.0 | |
| Hücre 30 | 26.3 | 21.4 | 27.5 | 40.0 | 40.0 | 39.9 | 40.0 | |
| Hücre-31 | 26.9 | 24.8 | 28.2 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre-32 | 27.0 | 22.5 | 23.8 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre-33 | 27.8 | 27.6 | 27.7 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Hücre-34 | 28.1 | 21.9 | 26.6 | 40.0 | 40.0 | 37.7 | 40.0 | |
| Hücre-35 | 29.3 | 26.5 | 28.1 | 40.0 | 40.0 | 38.3 | 40.0 | |
| Tip 4 | Hücre 36 | 24.8 | 20.3 | 26.0 | 27.4 | 26.6 | 40.0 | 40.0 |
| Hücre-37 | 28.7 | 22.3 | 25.9 | 28.9 | 22.9 | 38.5 | 40.0 | |
| Tip 5 | Hücre 38 | 25.5 | 20.0 | 29.3 | 23.0 | 19.9 | 25.9 | 40.0 |
| Hücre-39 | 25.9 | 21.8 | 28.6 | 25.1 | 21.7 | 25.7 | 40.0 | |
| Hücre 40 | 28.9 | 22.0 | 27.0 | 23.0 | 21.3 | 27.6 | 40.0 | |
Tablo 1. Ham CT değerleri.
Tartışmalar
Burada tarif edilen yöntem, bir kardiyak genlerin bir alt ekspresyonunu değerlendirmek için mikro-akışkan destekli tek hücre yakalama sisteminde kullanım için zebra balığı embriyolardan kalp progenitör hücrelerin bir popülasyonu ettirecek hücre tipi özel bir promoterin kontrolü altında bir floresan protein ifade kullanır hücreler. FACS lazer eksitasyon ve emisyon özellikleri tercih fluorofor (ler) ile uyumlu olduğu sürece, bu yöntem, herhangi bir flüoresan raportör hattı için kullanılabilir. Birçok zebrabalıkları muhabiri hatları varlığı zaten, ve yeni gazetecilere taşıyan transgenik balık az 3 ay içinde oluşturulabilir. Ek olarak, bu iş akışı, tek bir hücre-RNA sıralaması için tüm transcriptome cDNA üretmek üzere adapte edilebilir. Bunu yapmak için, 5-7 RNA sıralaması için uygun kitaplıkları oluşturmak için cDNA'nın hazırlanması için optimize kimyaları ile kullanım için biraz değiştirilmiş gerekir adımları tekrarlayın. Ancak, nüfus heterojenitesini doğrulamak için tavsiye edilirYüksek verimli sekanslama uygulamaları pursing önce burada tarif edilen yöntem kullanılarak.
Tarif edilen yönteme bazı sınırlamalar vardır dikkat etmek önemlidir. İlk olarak, entegre mikro-akışkan devresi (IFC) plakaların kullanımı özel, pahalı ekipman gerektirmektedir. Ancak, mikroakışkan cips geleneksel 96 kuyu ve tek hücrelere yönelik test 384 plaka biçimleri üzerinde önemli avantajlar sunmaktadır. mikrometre ölçek şeritli hücreleri ve reaktifler akan, tek hücreler doğrudan onlar sağlıklı tek hücre olduklarını teyit etmek için izlenebilir tek yakalama sitelerine konumlandırılmıştır. RNA ekstraksiyonu, ve cDNA sentezi, her bir hücre için, çok düşük hacimli kullanılarak IFC çip üzerinde yerinde oluşabilir. Entegre akışkan cips, bu yazının yazıldığı anda, tek bir kaynaktan aracılığıyla piyasada mevcuttur. ticari olmayan fabrikasyon çeşitli gruplar tarafından gerçekleştirilen olmasına rağmen, çoğu laboratuvar kapasitesinin dışındadır. 5-7 Mayıs Adımlarimalatlar diğer şirketlerin veya in-house üretilen platformlar için değiştirilmeleri gerekir. piyasada mevcut IFC plakaları 96 genler kapasiteli olsa İkincisi, seçilen genler zebrafish kullanılmak üzere onaylanmış florojenik etiketli gen probları durumu ile sınırlıdır. Üçüncü olarak, zebra balığı hücreleri, memeli hücrelerine göre tipik olarak daha düşüktür ve bu da küçük boyutlu işleme örnek sorunlar ortaya koymaktadır. Hücre çapı küçük bir değişiklik mevcut malzeme azaltma ve düşük tespit genleri eksprese olasılığını azaltarak, hücre hacmi büyük bir değişiklik anlamına gelir.
Bu protokol için birkaç ek hususlar vardır. Aşama 1 'de, bu kısa bir zaman dilimi içinde döllenmiş tek kullanım embriyolar önemlidir. sabit ve oksijen koşulları sınırlama olmadan sıcaklık tutarak, Zebra balığı embriyolar eş zamanlı olarak gelişir. Bu protokolün nihai okuma (tek hücre göreceli gen ifadesi) heteroge kaynağını ayırt edemeztek tek hücreler arasında neity. Bu nedenle, hücrelerarası heterojenite yorumlanması dikkatle sahnelenen embriyolar ve senkron gelişimi dayanır. adımlarda 2-3, ayrışma ve canlılık arasında bir tercih söz konusudur. 4. adım canlı hücreler için seçer, ve 3. adımı multimerlerini hariç yardımcı çeşitli filtreleme adımları içerir rağmen, aşırı sindirim FACS için kullanılabilir canlı malzemeyi azaltmak ve potansiyel sıralı hücre verimi azaltacaktır. Adımlar düşük canlılığı, sindirim süresini azaltmak toz haline getirme yoğunluğunu azaltarak, ve başlangıç maddesinde embriyo sayısını optimize dahil gidermek için. Çevrede (adım 4) hücre nüfusunun FACS zenginleştirme belki bu protokolde en kritik adımdır. Sıkı sıralama kriterleri sadece ilgi floresan proteini eksprese eden hücreler canlı ihtiva eden bir tek hücre hazırlama üretmek için gereklidir. Diğer nüfus hücreleri kirlenmesine hücrelerarası heterojenite yanlış temsil yol açabilir. özellikle,ilgi nüfusun FACS zenginleştirme bu protokolde gösterilen iki renk stratejisi ile sınırlı değildir. Daha karmaşık stratejiler daha rafine hücre popülasyonlarının üretmek ve prensip çalışmanın kanıtı bildirilen metodoloji uzatmak için hızlı bir yoldur edebilirsiniz. Adım 5'de, hücreler, tutulan hücre, hücre sağlığı ve floresans proteininin ifadesinin sayısı teyit etmek için gözlemlenebilir. Ama, hücre boyutu ve parlaklığına bağlı olarak, faiz flüorofor görsel olarak saptanabilir olmayacak ve güvenilir bir okuma değil olabilir. adımların 6-8 başarıyla tamamlanması üreticinin protokollerine dikkatli bağlılık gerektirir.
Prensip kanıtı olarak bilinen kardiyak belirteçlerin bir alt ekspresyon düzeyleri daha önce açıklanan BAC transgenik zebrafish hat Nkx2.5 türetilmiş tek değerlendirildi: ZsY bireysel Nkx2.5 kalp farklılaşma işaretlerini incelemek için: ZsY pozitif hücreler 18 saat görevde fertilizasyon (hpf). heterogenei araştırmak içinYakalanan Nkx2.5 ifade farklılaşma belirteçlerin ty: genlerin bir takımdır temizlik genler (ef1a, GAPDH), erken kalp şartname (gata4, Nkx2.5) 'de açık olduğu bilinen transkripsiyon faktörlerinin de dahil olmak üzere analiz edilmiştir ZsY ifade eden hücreler, bir ikinci kalp alan progenitör işaretleyici (isl1) ve bilinen genler daha sonra kalp farklılaşma (myl7, vmhc) sırasında açık olması. Her bir genin göreli bolluk döngüsü eşiği (BT) hesaplanarak QRT-PCR ile ölçülmüştür. Hesaplanan en düşük BT değeri 17.9 oldu ve en yüksek hayır amplifikasyon (Tablo 1) temsil eden, 40 oldu. > 35.0 CT değerleri tespit altında kabul edildi.
veri kümesinde 46 hücre, 6 dolayı herhangi genlerin başarısız amplifikasyon dışı bırakıldı. Geri kalan hücreler gata4, Nkx2.5, myl7 ve vmhc ve wer sentezlenmesi göre 5 tipe hücreleri alt sınıflandırılmıştırE grupları içinde ef1a değeriyle kriteri. GAPDH ilk muhtemel ev bakıcısı geni olarak dahil olmasına rağmen, sentezleme bir ev bakıcısı geni gibi uygunsuz hale hücreler boyunca oldukça değişken olmuştur. GAPDH ekspresyonu büyük olasılıkla daha iyi bu hücre tipinde kalp farklılaşma ile bağlantılı enerji metabolizması değişiklikleri temsil etmektedir. İlginç bir şekilde, (gata1 hücrelerinde 26) vardır ef1a un tespit edildiği bir hücre, ancak diğer bir gen tespit edilemez. Bu ef1a genellikle başarılı ve başarısız tek hücre, ters transkripsiyon reaksiyonları arasında ayırt etmek için yeterli bir ev bakıcısı geni ise, tüm uygulamalar için ideal olmayabilir göstermektedir. Hücrede 26 ef1a ifade eksikliği tek hücre transkripsiyonel dinamikleri nedeniyle olabilir. Son tek hücre çalışmaları, gen transkripsiyonu, hızlı ve önemsiz transkripsiyonel aktivitesini 26 evreleri arasında değişen, temelde stokastik olduğunu göstermektedir. TOnun transkripsiyonel patlama modeli tek hücre arasındaki niceliksel karşılaştırmalar için bir temizlik geninin kullanımının tamamen uygunsuz olabileceğini düşündürmektedir.
Tip 1 grubunda, ef1a tespit tek gendir. Bu hücre Nkx2.5 birini içerebilir: ZsY negatif hücrelerin veya hücre, arzulanan diğer genlerin çok düşük ekspresyonu ile. Hücre-26 hariç olmak üzere, 2 hücreleri Tip, bu genlerin bağıl ifade seviyelerinin hiçbir korelasyon hem ef1a ve gata4 ifade edilmiştir. Özellikle, Tip 1 ve Tip 2 hücrelerinde Nkx2.5 ekspresyonu nedeniyle optimal FACS sertlik, transkripsiyonel patlama transgen leakiness veya alt eşik ifade seviyeleri olabilir. Tip 3 hücreleri ef1a, gata4 ve NKX-2,5 fark edilebilir seviyelerde ifade edilmiştir. Bu hücreler, büyük olasılıkla kalp progenitör hücreler ve atriyal kardiyomiyositlerde ayırt içerebilir. Tip 4 hücreleri ef1a, gata4, Nkx2.5, myl7 edilebilir seviyelerde ifade/ em> ve vmhc ve ventriküler kardiyomiyositlere ayırt muhtemel hücrelerdir. Tip 5 hücreleri ef1a, gata4, Nkx2.5, myl7, vmhc ve GAPDH ifade edilmiştir. GAPDH tespit edilebilir seviyelere eklenmesi, bu hücreler farklı kardiyomiyositlerde bulunan geliştirilmiş glikolitik metabolizması kapasitesine sahip olduğunu göstermektedir. Bu Tg gelen ZsYellow pozitif hücreler için sıralama rağmen, bu ilginç oldu. (Nkx2.5: ZsYellow), embriyolar, 21/40 hücreleri bizim QRT-PCR analizinde Nkx2.5 alt eşik seviyeleri Bu transkripsiyonel patlama nedeniyle olabilir vardı veya endojen Nkx2.5 ve ZsYellow arasındaki transkript işleme farklılıkları yansıtıyor olabilir. Önemli bir şekilde, ikinci kalp alan işaretleyici isl1 bir hücrelerde tespit edilememiştir ya da cDNA eden yakalama tüm siteleri toplanan. Özetle, numuneler üzerinde karşılaştırarak, önemli heterojenite düşündüren, tek hücre düzeyinde belirgin olduğu 18 hpf, Nkx2.5: ZsY + celLS farklılaşma farklı aşamalarında kalp progenitörlerin gibi soy içerir.
Açıklamalar
The authors have nothing to disclose.
Teşekkürler
We thank Dr. C. Geoffrey Burns for fish stock. The authors are grateful to UNC Flow Cytometry Core Facility, UNC-CGIBD AAC core for resources enabling this project, and the ZAC facility for animal care. L.S. is supported by NIH T32 grant HL069768-13 (PI, Nobuyo Maeda). N.F. is supported by NSF Graduate Research Fellowship NSF-DGE-1144081. This study was supported by NIH P30DK034987 grant (to UNC Advanced Analytics Core), American Heart Association Scientist Development Grant 13SDG17060010 and Ellison Medical Foundation New Scholar Grant AG-NS-1064-13 (to Dr. Qian), and NIH R00 HL109079 grant (to Dr. Liu).
Malzemeler
| Name | Company | Catalog Number | Comments |
| Supplies | |||
| Dumont #5 forceps | Fine Science Tools | 11254-20 | For removing the chorion from embryos |
| Microcentrifuge tube 2 ml | GeneMate | C-3261-1 | |
| 40 um cell strainer | Biobasic | SP104151 | |
| 35 mm culture dish | Falcon | 351008 | |
| FACS tubes topped with 35 um cell strainer | Falcon | 352235 | |
| P1000 and tips | Rainin | 17005089 | |
| P20 and tips | Rainin | 17005091 | |
| IFC chip manufacturer's protocol | Fluidigm | 100-6117 | Version 100-6117 E1 was used in representative experiment |
| Wide bore pastuer glass pippette | VWR | 14673-010 | For transferring embryos |
| Adult wild type zebrafish | N/A | We used AB line | |
| Adult transgenic zebrafish | N/A | We used Tg(nkx2.5:ZsYellow) | |
| Name | Company | Catalog Number | Comments |
| Reagents for cell dissociation | |||
| Double distilled water | N/A | ||
| Instant Ocean Sea Salt | Instant Ocean | SS15-10 | |
| NaCl | FisherScientific | S271-3 | make stock in water and use for de-yolking buffer |
| KCl | Sigma Aldrich | P5405 | make stock in water and use for de-yolking buffer |
| NaHCO3 | Sigma Aldrich | S6014 | make stock in water and use for de-yolking buffer |
| Leibovitz's L-15 | Gibco | 21083-027 | |
| FBS | FisherScientific | 03-600-511 | Heat inactivate; any brand of FBS should be fine |
| Cell Dissociation Reagent 1 -TrypLE | Life Technologies | 12605-010 | Store at room temperature. |
| Cell Dissociation Reagent 2 - FACSmax | Genlantis | T200100 | Store -20; thaw on ice; bring to room temp before use |
| pronase (optional) | Sigma | P5147 | |
| L/D Dye - Sytox Blue | Life Technologies | S34857 | Any live/dead stain suitable for flow cytometry will work |
| Trypan Blue | Gibco | 15250-061 | |
| Name | Company | Catalog Number | Comments |
| Reagents for IFC plate use and qRT-PCR | |||
| Gene-specific probes | Probes will vary by experiment | ||
| TaqMan Probe ef1a | Life Technologies | Dr03432748_m1 | |
| TaqMan Probe gata4 | Life Technologies | Dr03443262_g1 | |
| TaqMan Probe nk2-5 | Life Technologies | Dr03074126_m1 | |
| TaqMan Probe myl7 | Life Technologies | Dr03105700_m1 | |
| TaqMan Probe vmhc | Life Technologies | Dr03431136_m1 | |
| TaqMan Probe isl1 | Life Technologies | Dr03425734_m1 | |
| TaqMan Gene Expression Master Mix | Life Technologies | 4369016 | |
| Reagents listed in IFC manufacturer's protocol | |||
| C1 Reagent Kit | Fluidigm | 100-5319 | Reagents for loading cells onto IFC plate |
| Ambion Single Cell-to-CTTM | Life Technologies | 4458237 | Reagents for reverse transcription and pre-amplification steps |
| Molecular Biology Quality Water | Corning | 46-000CM | |
| C1 IFC for PreAmp (5-10 um) | Fluidigm | 100-5757 | IFC plate for small cells |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| C1 AutoPrep machine | Fluidigm | 100-5477 | For IFC plate use |
| Hemocytometer | Sigma Aldrich | Z359629 | For counting cells and assessing cell size |
| Dissecting microscope | For removing embryos from chorion | ||
| Tissue culture microscope | For assessing single cell digestion | ||
| FACS machine | For isolating cells of interest |
Referanslar
- Speicher, M. R. Single-cell analysis: toward the clinic. Genome medicine. 5, (2013).
- Macaulay, I. C., Voet, T. Single cell genomics: advances and future perspectives. PLoS genetics. 10, e1004126(2014).
- Marco, E., et al. Bifurcation analysis of single-cell gene expression data reveals epigenetic landscape. Proceedings of the National Academy of Sciences of the United States of America. 111, 5643-5650 (2014).
- Yin, H., Marshall, D. Microfluidics for single cell analysis. Current opinion in biotechnology. 23, 110-119 (2012).
- Garlanda, C., Dejana, E. Heterogeneity of endothelial cells. Specific markers. Arteriosclerosis, thrombosis, and vascular biology. 17, 1193-1202 (1997).
- Diaz-Cano, S. J. Tumor heterogeneity: mechanisms and bases for a reliable application of molecular marker design. International journal of molecular sciences. 13, 1951-2011 (2012).
- Guo, G., et al. Mapping cellular hierarchy by single-cell analysis of the cell surface repertoire. Cell stem cell. 13, 492-505 (2013).
- Guo, G., et al. Resolution of cell fate decisions revealed by single-cell gene expression analysis from zygote to blastocyst. Developmental cell. 18, 675-685 (2010).
- Treutlein, B., et al. Reconstructing lineage hierarchies of the distal lung epithelium using single-cell RNA-seq. Nature. 509, 371-375 (2014).
- Kimmel, C. B. Genetics and early development of zebrafish. Trends in genetics : TIG. 5, 283-288 (1989).
- Liu, J., Stainier, D. Y. Zebrafish in the study of early cardiac development. Circulation research. 110, 870-874 (2012).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Developmental dynamics : an official publication of the American Association of Anatomists. 203, 253-310 (1995).
- Zhao, S., Fung-Leung, W. P., Bittner, A., Ngo, K., Liu, X. Comparison of RNA-Seq and microarray in transcriptome profiling of activated T cells. PloS one. 9, e78644(2014).
- McCall, M. N., McMurray, H. R., Land, H., Almudevar, A. On non-detects in qPCR data. Bioinformatics. 30, 2310-2316 (2014).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature. 3, 1101-1108 (2008).
- Lyons, I., et al. Myogenic and morphogenetic defects in the heart tubes of murine embryos lacking the homeo box gene Nkx2-5. Genes & development. 9, 1654-1666 (1995).
- Chen, J. N., Fishman, M. C. Zebrafish tinman homolog demarcates the heart field and initiates myocardial differentiation. Development. 122, 3809-3816 (1996).
- Zhou, Y., et al. Latent TGF-beta binding protein 3 identifies a second heart field in zebrafish. Nature. 474, 645-648 (2011).
- Paffett-Lugassy, N., et al. Heart field origin of great vessel precursors relies on nkx2.5-mediated vasculogenesis. Nature cell biology. 15, 1362-1369 (2013).
- McCurley, A. T., Callard, G. V. Characterization of housekeeping genes in zebrafish: male-female differences and effects of tissue type, developmental stage and chemical treatment. BMC molecular biology. 9, 102(2008).
- Tang, R., Dodd, A., Lai, D., McNabb, W. C., Love, D. R. Validation of zebrafish (Danio rerio) reference genes for quantitative real-time RT-PCR normalization. Acta biochimica et biophysica Sinica. 39, 384-390 (2007).
- Serbedzija, G. N., Chen, J. N., Fishman, M. C. Regulation in the heart field of zebrafish. Development. 125, 1095-1101 (1998).
- de Pater, E., et al. Distinct phases of cardiomyocyte differentiation regulate growth of the zebrafish heart. Development. 136, 1633-1641 (2009).
- Hami, D., Grimes, A. C., Tsai, H. J., Kirby, M. L. Zebrafish cardiac development requires a conserved secondary heart field. Development. 138, 2389-2398 (2011).
- Yelon, D., Horne, S. A., Stainier, D. Y. Restricted expression of cardiac myosin genes reveals regulated aspects of heart tube assembly in zebrafish. Developmental biology. 214, 23-37 (1999).
- Molina, N., et al. Stimulus-induced modulation of transcriptional bursting in a single mammalian gene. Proceedings of the National Academy of Sciences of the United States of America. 110, 20563-20568 (2013).
Yeniden Basımlar ve İzinler
Bu JoVE makalesinin metnini veya resimlerini yeniden kullanma izni talebi
Izin talebiDaha Fazla Makale Keşfet
This article has been published
Video Coming Soon
JoVE Hakkında
Telif Hakkı © 2020 MyJove Corporation. Tüm hakları saklıdır