Method Article
בידוד ואפיון של תאים בודדים מעוברים דג הזברה
In This Article
Summary
This protocol describes a method for isolating single cells from zebrafish embryos, enriching for cells of interest, capturing zebrafish cells in microfluidic based single cell multiplex systems, and assessing gene expression from single cells.
Abstract
The zebrafish (Danio rerio) is a powerful model organism to study vertebrate development. Though many aspects of zebrafish embryonic development have been described at the morphological level, little is known about the molecular basis of cellular changes that occur as the organism develops. With recent advancements in microfluidics and multiplexing technologies, it is now possible to characterize gene expression in single cells. This allows for investigation of heterogeneity between individual cells of specific cell populations to identify and classify cell subtypes, characterize intermediate states that occur during cell differentiation, and explore differential cellular responses to stimuli. This study describes a protocol to isolate viable, single cells from zebrafish embryos for high throughput multiplexing assays. This method may be rapidly applied to any zebrafish embryonic cell type with fluorescent markers. An extension of this method may also be used in combination with high throughput sequencing technologies to fully characterize the transcriptome of single cells. As proof of principle, the relative abundance of cardiac differentiation markers was assessed in isolated, single cells derived from nkx2.5 positive cardiac progenitors. By evaluation of gene expression at the single cell level and at a single time point, the data support a model in which cardiac progenitors coexist with differentiating progeny. The method and work flow described here is broadly applicable to the zebrafish research community, requiring only a labeled transgenic fish line and access to microfluidics technologies.
Introduction
רוב המחקרים הנוכחיים של ביולוגיה מולקולרית של תא מבוססים על ממוצעי אוכלוסייה. עם זאת, אירועים ביולוגיים חשובים עשויים להיות רעולי פנים על ידי ניתוחים מבוסס אוכלוסיה המסורתיים הללו, מאחר ואנשים קטין יכול לשחק תפקידים מרכזיים בתהליכים ביולוגיים ותוצאות המחלה. הבנת ביטוי גני אוכלוסיות הטרוגניות ברמת התא הבודדה יכולה (ויש לו) להוביל לתובנות ביולוגיים וקליניים רלוונטיים 1,2. דאגה ללימודי התפתחות עובריות, בתוך אוכלוסייה גדולה יותר של תאים, ובתאים הם בדרך כלל מיוצגים כראוי, מה שהופכים אותו מאתגר כדי לזהות שינויים עדינים ביטוי גנים שבסופו של דבר ליזום החלטות גורל תא 3. באופן דומה, סוג תא בודד יכול להיות ביטוי פרופילים שונים בתגובה במיקרו-סביבה של 4. לדוגמה, תאי אנדותל תושב אותו איבר או באיברים שונים (למשל., אבי העורקים או כליות) להפגין ההטרוגניות רבה, למרות שיתוף morp משותףתכונות hological ופונקציונליות 5. בנוסף, תאים סרטניים לאכלוס אותה הגידול יכולים להיות גם משתנים פרופילים או מוטציות מולקולריים ברמת התא הבודדה 6.
במערכות מודל, transcriptomics יחידים תאים זיהה בהצלחת אוכלוסיות תאים חדשות, המאופיינת מצבי ביניים המתרחשים במהלך התמיינות תאים, וגילה תגובות הסלולר הפרש לגירויי 7,8,9. תובנות אלה היו רעולי פנים במחקרים מבוסס אוכלוסיה קונבנציונאלי. עוברי דג זברה הם מקור מתחת מנוצל מאוד של גזע, אב, ובידול תאים לחקר שאלות ההטרוגניות תא בודד ורגולציה מולקולרית של זהויות הסלולר במהלך פיתוח. פיתוח וקלות מניפולציה גנטית סטריאוטיפית שלהם מאוד, לשעבר vivo לגרום להם מערכת מודל מצוינת עבור גישה זו 10,11. באופן ספציפי, מגבלה משמעותית לפרשנות של תא בודד genנתוני דואר ביטוי הוא כי זיהוי אמין של מדינות תא ביניים רומן במהלך פיתוח דורש תזמון מאוד זהיר של אוסף רקמות 9. הדבר נחוץ כדי להבטיח כי ההטרוגניות בין תאים שנתפסו מייצג ההטרוגניות בתוך רקמה בנקודת זמן אחת ולא הטרוגניות בביטוי גנים שהציג התמיינות תאים תלוי גיל. בהשוואה לעכברים, פיתוח עובר דג זברה יכול להיות מסונכרן בדיוק על פני מספר גדול של עובר 12. בנוסף, עם גדלי מצמד גדולים, עוברי דג זברה יכולים לשמש מקור שופע של תאי גזע מולידם.
פרוטוקול זה מתאר שיטה לבודד תאים מעוברים דג הזברה ללכוד תאים בודדים באמצעות מעגל מיקרופלואידיקה משולבת זמינים מסחרית (IFC) מערכת שבבים autoprep עבור ניתוח ביטוי גנים qRT-PCR. פרוטוקול זה יכול להיות להעברה מיד על כל מבחני ריבוב תפוקה גבוהה כולל כלרצף transcriptome המאפשר ניתוח מקיף יותר של ההטרוגניות הסלולר 13. הוא מציע גם מספר יתרונות מבחני ביטוי גנים מסורתיים. פרוטוקול בידוד תא הבודד מניב כדאיות גבוהה לאחר FACS, אשר מקטינה את חלקם של תאים נפגעים כלולי יישומים במורד זרם. באמצעות IFC, תאים שצולמו עשויים להיות שנצפו ישירות להעריך שיעורים ללכוד ולהעריך תא בריאות מורפולוגית. בנוסף, פרוטוקול זה הוא החלים רחב לקהילת מחקר דג הזברה, המחייב רק קו דג מהונדס שכותרתו וגישה לטכנולוגיות ללכוד תאי microfluidic.
כהוכחה עקרונית, תאים בודדים נגזרו אבות לב הבודדים שנתפסו על שבב IFC, ולאחר מכן את השפע היחסי של סמני בידול לב נמדד על ידי qRT-PCR. ניתוח התבטאות גנים ברמת התא הבודדה מוכיח כי אבות לב לדור בכפיפה אחת עם differe שלהםצאצאי ntiating. התובנה שנצבר פרופיל תא בודד של אבות לב עשוי לשפוך אור על ההטרוגניות דפוסי ביטוי גנים בין ובתאים לב במהלך ההתפתחות חוליות, אשר עשויים להיות רעולי פנים בניתוחים מבוסס אוכלוסיה מסורתית.
Protocol
פרוטוקול זה מחייב שימוש דג זברה לחיות, מבוגר לייצר עוברים. עוברים נקצרים לאיסוף רקמות. זה חיוני כדי לקבל את אישור ועדה בוחן אתיקה המתאימה לנהל את הניסוי הזה.
1. השג עובר מבוימים
- היום לפני הניסוי, להכין בריא, דג זברה מבוגרת לרבייה. מניחים זכר אחד ונקבה אחת משני צדי המתרס של מחיצה ברורה בתוך אקווריום רבייה.
- חזור על 1.1 לקערות רבייה רבות ככל הכרחיות ליצירת עובר מספיק עבור היישום במורד הזרם. השג עובר מן הדגים הוא סוג בר דגים מהונדסים המבטאים חלבוני ניאון בסוג התא של עניין.
הערה: מספר העוברים הדרושים יישומים במורד הזרם שלבים 2-8 תלוי השפע היחסי של תאים בעלי עניין בנקודת הזמן של עניין. אמנם זה עשוי להשתנות לפי סוג התא, 200 עוברים לייצר 2,000-5,000 תאים ממוינים כאשר גאמות של עניין מייצגים <1.0% של תאים הכולל ב 24 hpf (שעות לאחר ההפריה). - למחרת בבוקר, להחליף את המים במכל גידול החיות ידי העברת דגים לאקווריום רבייה טרי להסיר את המחיצה. הטה את הטנק בזווית לעודד רבייה.
- איסוף עובר מבוים.
- כל 15 דק ', לאסוף עובר על ידי העברה למבוגרי טנק רבייה טרי להעביר את הביצים אשר נשארות מאחור דרך מסננת תה.
הערה: עוברי דג הזברה לפתח סינכרוני כאשר שמרו על צפיפויות וטמפרטורות דומות. - יש לשטוף את הביצים עם ביצת מים (0.21 גר '/ ל מלחים אוקיינוס מיידי ב 1 ליטר מים מזוקקים פעמים) ולהעביר בצלחת פטרי. מעביר את צלחת פטרי חממה ולחה על 28.5 מעלות צלזיוס עם זרימת אוויר.
- כל 15 דק ', לאסוף עובר על ידי העברה למבוגרי טנק רבייה טרי להעביר את הביצים אשר נשארות מאחור דרך מסננת תה.
- שעתיים לאחר האוסף האחרון, מופרית מיון, עוברים רב-תאיים לתוך 10 ס"מ בצלחות פטרי ולהפחית צפיפות 50 עוברים לכל צלחת. עובר בחר מתוך אחת, 15חלון של אוסף זמן דק 'עבור יישום במורד הזרם. דגירה עוברת על 28.5 מעלות צלזיוס.
הערה: לדוגמה, לאסוף עוברים בשעה 8:30, 8:45, 9:00, 9:15, 9:30, 9:45, 10:00 ו 10:15. השוואת פני נקודות זמן, אם את המספר הגדול ביותר של עוברים מופרים הם מהציפורניים שנאספו בשעת 9:00, ולאחר מכן להשתמש רק עובר אלה עבור יישומים במורד זרם.
2. הגדרה עבור דיסוציאציה תא יחידה
- כ -30 דקות לפני נקודת הזמן של עניין (18 hpf) סרים עוברות מן הסיסי שלהם באופן ידני עם מלקחיים בסדר.
- אסוף ולתייג את המידע הבא לכל מצב: שני 2 מ"ל צינורות microcentrifuge, מסננת תא אחד 40 מיקרומטר, בצלחת תרבית תאים אחד 35 מ"מ, ושני צינורות FACS עקף עם מסננת תא 35 מיקרומטר.
- צ'יל החומרים הכימיים הבאים על קרח: מי ביצה המכילים מלחי אוקיינוס מיידי 0.21g / L ב 1 ליטר מים מזוקקים פעמים; דה-yolking מאגר המכיל 55 מ"מ NaCl, 1.8 מ"מ KCl, ו -1.25 מ"מ NaHCO 3; ו FACS מאגר המכיל L-15 של ליבוביץ תקשורת בתוספת 5% בסרום שור עובר חום מומת.
- תביאו 1 מ"ל לדגימה של מגיב סלולרי דיסוציאציה 1 עד RT. Cell ההפשרה 1 מ"ל דיסוציאציה מגיב 2 לדגימה על הקרח. מביאים RT מיד לפני השימוש.
3. דיסוציאציה תא יחיד
- בעזרת פיפטה זכוכית הנישאת רחבה, להעביר 100-300 עובר לצינור צנטריפוגות מיקרו 2 מיליליטר נפח מינימאלי של ביצת מים.
- להרדים העובר על ידי החלפת מים עם 1 מיליליטר קר כקרח ביצת מי הצללת צינורית קרח למשך 20 דקות.
זהירות! זה קריטי כדי לקבל את אישור ועדת ביקורת חית טיפול נאותה מוסדית שיטת המתת חסד זה (מצמרר על קרח ואחריו תא דיסוציאציה כמו המתת חסד משנית). המתת חסד רגיל בדרך כלל דורש כי עובריים <3 ימים לאחר הפריה הם מולבנים לאחר שהם מקוררים, אשר אינו מתאים לקבלת תאי קיימא. - לשטוף את העוברים פעמיים עם מים ביצה 1 מ"ל קר כקרח. כדי לשטוף, השתמש פיפטה זכוכית נישאת רחבה להסיר ביצת מים P1000 להוסיף ביצת מים.
- הסר את החלמון על ידי החלפת המים העובר עם 1 מיליליטר Deyolking הצפת triturating 8-12 פעמים עם טיפ P1000, או עד החלמון נמס ורק גופות העוברים גלויות.
- אסוף את הרקמה על ידי צנטריפוגה XG ב 300 דקות 1. השתמש פיפטה להסיר את supernatant בעדינות מבלי לשבש את גלולת הרקמות. Re- להשעות במים 1 מ"ל ביצה.
- 3.5 חזור על שלב עבור סכום כולל של שלושה שוטף, אבל על שטיפה של דבר, מחדש להשעות ב 1 מ"ל תא RT דיסוציאציה מגיב 1.
- מדגירים מגיבים סלולארי דיסוציאציה 1 במשך 10 דקות ב RT עם צינורות בצורה אופקית. כל 2-3 דקות, בעדינות triturate עם טפטפת P1000 כדי למנוע clumping.
הערה: ידית דגימות בעדינות במהלך שלבי טחינה דקים. מתישהו גוש יחיד, גדול יהווה בצינור מתוך סבך גופים עוברים. רצון זהלפזר בעיכול נוסף שנעזר טחינה דקה ועדינה. לא מערבולת. - אסוף רקמה על ידי צנטריפוגה XG ב 300 במשך 3 דקות. הסר supernatant ו resuspend ב 1 מ"ל תא דיסוציאציה מגיב 2.
- דגירה של 5-15 דקות ב RT. מניחים את הצינורות בצורה אופקית. כל 2-3 דקות, בעדינות triturate למנוע clumping. כל 5 דקות, להעריך את התקדמות עיכול.
- כדי להעריך את ההתקדמות העיכול, לדלל 2 supernatant μl לתוך 18 μl FACS הצפת פיפטה כמו טיפה על צלחת תרבית תאים.
- מניחים coverslip על מדגם ולבחון תחת מיקרוסקופ בתרבית רקמה בהגדלות 10X ו 20X.
הערה: ההכנה אמורה להופיע כמו שילוב של תאים בודדים, קבוצות קטנות, אשכולות גדולים, וגוף העובר מדי פעם בעיקר שלמים. - חזותי להעריך את השיעור בהכנה כי הוא תאים בודדים ואשכולות קטנים.
הערה: Over-עיכול יפחית כדאיות; תת עיכול יפחית תשואת תא בודדת.
- אסוף את הכנת התא על ידי צנטריפוגה XG ב 300 במשך 5 דקות. בטל supernatant מחדש להשעות ב 1 מ"ל קר חיץ FACS.
- להרטיב מסננת תא 40 מיקרומטר עם FACS חוצץ. לעבור את ההשעיה התא דרך מסננת תא 40 מיקרומטר על צלחת תרבית תאים 35 מ"מ. שטוף את זמן תא המסנן אחת עם 1 מיליליטר FACS מיון חיץ.
- העברת זרם דרך צינור 2 מ"ל microcentrifuge. איסוף תאים על ידי צנטריפוגה XG ב 300 במשך 5 דקות מחדש להשעות ב 100 μl FACS חוצץ.
- רוזן תשואת תא באמצעות hemocytometer. בהמשך לדלל דגימות 5x10 6 תאים לכל מיליליטר, או ריכוז אופטימלי מומלצים מכונת בחירת FACS. אופציונלי בספירת תאי התשואה על hemocytometer, תאים מתים counterstain עם trypan כחול כדי לאשר הכדאיות של התכשיר התא לפני מיון FACS.
הערה: תכשירים כי הם גם לדלל ייקח סכום עודף של זמן כדי למיין. תכשירים כי הם גם גoncentrated נוטים להתגודד לתוך multimers והם לא טובים למיון אוכלוסייה טהורה. - שמור 10-20% של כל דגימת שימוש באפליקציות כמו בלא כתם. לקבלת דוגמיות הנותרים, תאים מתים כתם על ידי הוספת חי ניאון / מת (L / D) אפליה לצבוע לתוך מדגם זה. אין לשטוף.
הערה: כל צבע פלואורסצנטי חודרת תאים עם ממברנות וכתמי תא שנפגעו חומצות גרעין עשויות לשמש לצבוע L / D, ובלבד ספקטרום הקרינה תואם מכונת FACS של תאי תיוג בחירת fluorophore של עניין.- אין לשטוף את ההכנה לאחר הוסיף לצבוע כמו כמה תאים ימותו העוברים טיהור FACS וצריכים מוצאים מהאוכלוסייה הממוינת.
- להרטיב את מסננת מיקרומטר תא 35 כתרים צינור FACS עם 20 חיץ μl FACS. הוספת תאים אל מסננת ולאסוף על ידי כוח הכבידה. אחסן על הקרח.
4. FACS העשרה
- השתמש FACS להעשיר עבור יחיד, תאי חיים המבטאים את בסמן פלורסנטי.
- גדר שער להבחין בין תאים מן ההריסות על מגרש פיזור של פיזור קדימה (FSC-A) משרעת לעומת משרעת פיזור בצד (SSC-A), שניהם עם דרוג ליניארית.
זהירות! תאי דג זברה הן בדרך כלל קטנה יותר מאשר עכבר או תאים אנושיים. הדבר בא לידי ביטוי בהפרדת בסיס תחתונה בין תאים ופסולת. - מהשער שנקבע 4.1.1, להגדיר שער להעשיר עבור תאים בודדים ואינם כוללים multimers באמצעות פיזור העלילה של גובה פיזור קדימה (FSC-H) וגובה פיזור נמוך בצד (SSC-H), שניהם עם דרוג ליניארית.
הערה: תאים עם גבוה FSC-H לא פרופורציונאלי והנמוך SSC-H הם multimers סביר ואינם נכללים מיון. - ממערך שער 4.1.2, באמצעות צבע שולט יחיד, להגדיר שער לכלול תאי חיים רק באמצעות פיזור עלילה של FSA-A עם דרוג ליניארית משרעת של הערוץ המשמש לאיתור L / כתם D עם דרוג יומן. כלול L / D תאים שליליים.
- מהשער שנקבע 4.1.3, באמצעות צבע שולט יחיד, להגדיר שער לכלול תאי חיים רק עם קרינה חיובית באמצעות פיזור עלילה של-A FSA עם דרוג משרעת ליניארי של הערוץ המשמש לאיתור סמן תא חלבון פלואורסצנטי עם דרוג יומן . כלול תאים חיוביים.
- גדר כל פקדי פיצוי, ואם יש צורך, לתת דין וחשבון על התערבות בין L / D כתם ספקטרום חלבון פלואורסצנטי.
- גדר שער להבחין בין תאים מן ההריסות על מגרש פיזור של פיזור קדימה (FSC-A) משרעת לעומת משרעת פיזור בצד (SSC-A), שניהם עם דרוג ליניארית.
- היגיון מעין סט לתאים מתחלקים כל השערים להגדיר בשלב 4.1.
- שימוש בתאי שכותרתו כפול, ודא gating עבור מיון התא.
הערה: תאים למיון יהיה תאים ולא פסולת, תאים בודדים ולא multimers, L / D שלילי קרינת תאים חיוביים.
- שימוש בתאי שכותרתו כפול, ודא gating עבור מיון התא.
- מיין 2,000-4,000 תאים מן האוכלוסייה עניין לתוך 5 μl של קר FACS חוצץ בצינור microcentrifuge על הקרח. כדי למזער גז ומתח על תאים במהלך מיון, השתמש לחצים הנמוכים ביותר אפשריים עבור התאמְמַיֵן.
הערה: אגל 5 μl של חיץ FACS משמש ככרית תאי יציאת מכונת FACS. איסוף 2,000-4,000 תאים ישירות לתוך FACS חוצץ מבטל את הצורך צנטריפוגה לפני הטעינה על שבבי IFC. מומלץ להשתמש באסטרטגיה זו משום צנטריפוגה יכול להוביל להיווצרות של multimers אם תאים לדבוק זה בזה גלולה. - להעריך את הניתוח הכדאי שלאחר המיון.
- ההעברה 1 μl מסודר תאים לצינור FACS טרי עם 100 μl FACS מיון חיץ 1: 1,000 דילול של כתם L / D. להעביר את התאים באמצעות סדרן FACS עם האסטרטגיה gating בשלב 4.1. השתמש שיעור L / D משלילי לחיובי להעריך כדאיות של תאים ממוינים.
5. תאי עומס על מיקרופלואידיקה צ'יפ
- באמצעות hemocytometer, למדוד גם את הריכוז וגודל תאים ממוינים.
- לדלל תאים ממוינים לפחות 10 μl. לדלל aliquot של 5 μ; תאי L עם 5 μl trypan כחולים. עומס על hemocytometer ולהחיל coverslip.
- איסוף תמונות שדה בהיר של כל התאים 4 x 4 רשתות של hemocytometer. ספירת תאים חיים ולחשב תאים / מ"ל חי. תאים חיים אינם תופסים trypan כחול.
הערה: השתמש בכל תוכנה לניתוח תמונה רגילה למדוד את הקוטר של כל תאי החיים. חשבת את הסטייה הממוצעת ורמת גודל התא, ובחר צלחת IFC מתאימה בטווח גודל תא של עניין. אם טווח תאי גודל בפישוק מגוון גודל תא צלחת IFC, השתמש הצלחות מרובות כדי ללכוד המגוון הרחב של תאים בעלי עניין.
- תאי עומס על צלחת IFC פי הוראות היצרן (ראה חומרים).
- לדלל התאים 1x10 6 תאים / מ"ל ולבצע אופטימיזציה הציפה לפי הוראות היצרן.
הערה: אופטימיזציה חוק ארכימדס מבטיחה כי התאים לא לשקוע לקרקעית ולא לצוף למעלה של wel טעינהl לטעינה אופטימלית על שבב IFC. יחס חיץ תאים עשויים להשתנות לפי סוג התא, אך בדרך כלל נע 6: 4-7: 3 של תאים: חיץ. - הוספת תאים לצלחת דרוכה-IFC. צלחת טען לתוך מכונת fluidics תואמת, ולהפעיל תסריט טעינת תא לדחוף תאים דרך מעגל מיקרופלואידיקה ולתוך נתיבים ללכוד.
- לדלל התאים 1x10 6 תאים / מ"ל ולבצע אופטימיזציה הציפה לפי הוראות היצרן.
- ודא תאים ונתקעו אתרים ללכוד בצלחת IFC.
- הסר את צלחת IFC ממכונה fluidics. הר צלחת IFC על מיקרוסקופ מצויד מתאם צלחת.
- אסוף תמונות של שדה קרינה בהירים עבור כל אתר לכיד בהגדלה 10X.
הערה: פעמים ללכוד Brightfield עשויים לנוע בין 10-50 מילישניות, תלוי בעוצמת המנורה. פעמים לכידות קרינה עשויות לנוע בין 250-750 מילישניות, תלויים בהיר fluorophore. הימנע overexposing תאים כדי למנוע נזקי.
6. סינתזה cDNA
- בצע תמוגה התא באתרו פי שאניצלחת FC הוראות היצרן.
- הוספת מאגרים תמוגה לבארות בצלחת IFC. צלחת לטעון על מכונת fluidics התואם תסריט תמוגה תא בטווח לפי הוראות יצרן.
- בצע שעתוק לאחור על פי הוראות יצרן צלחת IFC.
- הוספת ריאגנטים שעתוק לאחור לצלחת IFC. צלחת לטעון על מכונת fluidics תואם. הפעל סקריפט שעתוק לאחור. תנאי אופניים לדוגמא: 1 מחזור של 25 מעלות צלזיוס למשך 10 דקות, 1 מחזור ב 42 מעלות צלזיוס במשך שעה 1., ולאחר מכן 1 מחזור ב 85 מעלות צלזיוס למשך 5 דקות.
- בצע-הגברה מראש עם בדיקות גן ספציפי על פי הוראות יצרן צלחת IFC.
הערה: בשל לפרט יותר שלהם עם מספרים נמוכים להעתיק, להשתמש בדיקות fluorogenic שהכותרת ולא בדיקות מותאמות לשימוש עם צבעי intercalator.- פריימרים ברכה לדלל לכל 180 ננומטר סופי. להוסיף פריימרים ונקווה לצלחת IFC. צלחת טען על fluidi תואםמכונת cs. תסריט preamplification לרוץ.
- בצע-הגברה מראש עם תנאי המחזור הבאים: 1 מחזור ב 95 מעלות צלזיוס למשך 10 דקות, ואז 18 מחזורים עם denaturing ב 95 מעלות צלזיוס למשך 15 שניות ואז חישול וסיומת ב 60 מעלות צלזיוס למשך 4 דקות. חנות מראש מוגבר cDNA ב 4 ° C עד הקציר. קציר cDNA מהצלחת microfluidic בהתאם להוראות וחנות של היצרן ב -20C עד השימוש.
7. cDNA בחר תאים בודדים עבור יחיד יעד qRT-PCR
- לדלל cDNA ב -25 מגיב דילול μl פי הוראות היצרן.
הערה: תשואה סופית משוערת היא 28 μl של cDNA מראש מוגבר לכל תא. שמורת aliquot של diluent לשימוש כשלט ללא תבנית qRT-PCR.- בהתייחסו בשדה בהיר ותמונות ניאון רשמו בשלב 5.3.2, להבקיע כל אתר ללכוד. באופן ידני לספור ולהקליט את מספר התאים. להעריך ולהקליט אם כל תא הוא שפעתorescent.
הערה: כל אתר מסוים עשוי להכיל 0, 1, או> 1 התא, שם> תא 1 כולל אתרים ללכוד המכילים מספר תאים ו / או פסולת בלתי מזוהה. אם התמונות הן באיכות מספקת, ערך פיקסל עשוי גם להיות מוקצה לכמת את עוצמת האות פלואורסצנטי.
- בהתייחסו בשדה בהיר ותמונות ניאון רשמו בשלב 5.3.2, להבקיע כל אתר ללכוד. באופן ידני לספור ולהקליט את מספר התאים. להעריך ולהקליט אם כל תא הוא שפעתorescent.
- דגימות בחרו לניתוח qRT-PCR.
- בחר דוגמאות cDNA מאתר ללכוד שזוהו בשלב 7.2 המכילים תא 1 מדויק עם קרינה חיובית. בריכה 1 aliquots μl של cDNA מכל מדגם.
הערה: זהו "הפול" בקרה חיובית משמש כדי לקבוע אם הגנים של עניין באים לידי ביטוי בכל אחד מן התאים שנבחרו. - בחר cDNA מלפחות אתר ללכוד אחד שמכיל בדיוק 0 תאים כביקורת שלילית. השתמש diluent משלב 7.1.2 כביקורת ללא תבנית.
- בחר דוגמאות cDNA מאתר ללכוד שזוהו בשלב 7.2 המכילים תא 1 מדויק עם קרינה חיובית. בריכה 1 aliquots μl של cDNA מכל מדגם.
- הפעל assay qRT-PCR.
- השתמש בדיקות יחידות המשמשות הגברה מראש בשלב 6.4.1. טען את כל דגימות triplicatדואר. תנאי רכיבה על אופניים לתגובה 10 μl על צלחת גם 384: 1 מחזור 50 ° C 2 דקות, 1 מחזור 95 מעלות צלזיוס למשך 10 דקות, 40 מחזורים עם denaturing על 95 מעלות צלזיוס 15 שניות ואז חישול והסיומת ב 60 מעלות צלזיוס למשך 1 דקות.
ניתוח 8. נתונים
- אמת מוצרי qRT-PCR על ידי ג'ל אלקטרופורזה סטנדרטית. אשר המוצר היא להקה אחת ב משקל מולקולרי צפוי (משתנה עבור כל בדיקה).
הערה: אם בדיקה מייצרת להקות מרובות או להקה יחידה בגודל הולם, אז סגולי מוטל בספק, וזה כולל את הגן מניתוח. - בדוק את כל קימורי הגברת החשבון עבור בתופעות חריגות 14. חשב את הערך CT הממוצע עבור כל שילוב מדגם / גן 15.
הערה: ערכי CT עבור בקרות חיוביות בדרך כלל נעים בין 10-30. ערכי CT עבור פקדים שליליים בדרך כלל נעים בין 35-40 (לא הגברה). ערכי CT 30-35 נחשבים ביטוי נמוך מאודוצריך לפרש בזהירות 14. - נתונים דווח
- אם דגימות ליפול CT = 10-30, הנתונים עשויים להיות כמו כן דווח כי שינוי לקפל יחסית שפע תמליל מדגם שליטה.
הערה: מקפלים שינוי שווה 2 ^ (- ΔΔCT) שבו ΔCT הוא ביחס גן משק על ידי הפחתת CT הממוצע של המדגם מן גן משק ו ΔΔCT הוא ההבדלים ΔCT בין המדגם לבין כביקורת חיובית 15.
- אם דגימות ליפול CT = 10-30, הנתונים עשויים להיות כמו כן דווח כי שינוי לקפל יחסית שפע תמליל מדגם שליטה.
תוצאות
כהוכחה עקרונית, ביטוי גנים הוערך לחקור דינמיקת בידול במהלך התפתחות של לב. בשנת דג זברה, אבות לב נובעים אוכלוסיית mesodermal של תאי נודדים אל mesoderm הצלחת לרוחב הקדמי שבו הם פתיל כדי ליצור את צינור הלב ליניארי. לפני היתוך, אבות לב מתחילים לבטא את nkx2.5 גורם שעתוק (NK2 homeobox 5), אשר נחשב סמן ספציפי המוקדם של אבות לב 16,17. הנה, דג מהונדס BAC שתואר לעיל Tg (nkx2.5: ZsYellow) 18, מקוצר nkx2.5: ZsY שמש לבחון סמני בידול לב תאים בודדים ב 18 במת somite, 18 הפרית שעות שלאחר (hpf) 12. זו הייתה נקודת הזמן המוקדמת בו אות ZsYellow הייתה חזותי לזיהוי. כפי שתואר קודם לכן עבור קו מהונדס זה, ZsYellow תוויות אבות לב, כפי שאנוll כמו תאים עודפים-לב כמה שיוצרים אל בלוע קשת תאי אנדותל ב 28 hpf 19 (איור 1A-B, מידע לא מוצג). בגיל 18 hpf, nkx2.5: עובר ZsY היו ניתק לתוך השעית תא בודדת אז מוכתמת Sytox הכחול להוציא תאים מתים. Live, ZsYellow חיובי, תאים שלילי כחול Sytox היו FACS מיון או באמצעות סדרן SH800Z MoFloXDP או סוני מצויד 100 זרבובית מיקרומטר (איור 1 ג-ו). כדי להעריך טוהר כדאיות האוכלוסייה ופוסט מעין המסודרת, aliquot של תאים ממוינים הוכתם Sytox הכחול והעריך את אחוז האירועים שנפלו בתוך שער המיון המקורי (איור 1G-J).
לאחר המיון, תאים היו התקשרו מעגל microfluidic משולבים עבודת שבב (IFC) זרימה (איור 2 א) על פי הוראות היצרן. Hemocytometer, בשילוב עם exclusio trypan כחולn שימש למדידת קוטר התא, ריכוז, ואת הכדאיות (תרשים 2B, ו מידע לא מוצג). ציפת Cell הייתה מותאמת (6.5: 3.5 תאים: חיץ) על פי ההוראה של היצרן, ותאים הועמסו על גבי IFC ללכוד 5-10 מיקרומטר תאי קוטר. כדי להעריך ללכוד יעילות, קרינה ו / או אותות בשדה בהירים הם צלמו בכל האתרים ללכוד, פונקצית ריצוף בפיג'י שמשה לתפור תמונה אחת של צלחת מיקרופלואידיקה. כל אתר לכיד כלול 0, 1, או> 1 תאים בודדים (איור 2C-F). כצפוי מהעשרה FACS, תאים שנתפסו הביע ZsYellow (איור 2G). יעילות לכידת חריגה 90% ב 5 ניסויים בודדים באמצעות nkx2.5: ZsY מסודרים תאים, ולפחות 70% מהאתרים ללכוד נכבשו על ידי תא בודד (מידע לא מוצג). תאים היו lysed, RNA בודד, cDNA מסונתז גני המטרה ספציפיים היו מוגברים, והכל בהתאם להוראות היצרן.
לתת-קבוצה של אתרי ללכוד תאים נבחרו לניתוח qRT-PCR, כמתואר בפרוטוקול לעיל. באופן ספציפי, 1a גורם התארכות (efl1a) 20 ו glyceraldehyde-3 פוספט (GAPDH) 21 היו assayed כמו גני המשק צפויים להתבטא בכל תא; Gata חלבון מחייב 4 (gata4) 22 ו NK2 homeobox 5 (nkx2.5) כבר סמנים ובתאי לב; ISL LIM homeobox 1 תיבה 1 (isl1) כסמן שדה לב שני 23,24; שרשרת אור שרירן (myl7) ואת שרשרת כבדה חדרית שרירן (vmhc) 25 לציון cardiomyocytes חדרית. גן ספציפי בדיקות אומתו באמצעות cDNA מ 48 עוברי hpf (איור 3). qRT-PCR שימש להערכת ביטוי גנים היחסי של הגנים האלה ב -45 תאים בודדים מתוך 18 hpf עוברים, כביקורת שלילית ללא תבנית, וכן containi בקרה חיובית האוכלוסייה ונקווהng cDNA מכל 96 אתרי הצילום. ערכי הגלם CT עבור 40 תאים ובקרות נציגים מוצגים בטבלת 1. יש לציין, מהמאגר של 46 תאים שנבדק, 6 תאים לא נכללו בניתוח בגלל כל ערכי CT ביטוי הגנים חריגים CT = 35.0, וזה ידוע אם הגבוה הללו CT ערכים הם לייחס נכון, רמות ביטוי נמוכות מאוד או שפלה מדגמת. מאז המגוון של ערכי CT עבור גן המשק, ef1a, היה רחב מדי כדי להשוות ביטוי גנים בין דגימות, ורבי ערכי CT עלו על 30, ערכי CT היו דמיינו כמפה חום (איור 3). השוואת ברחבי דגימות, ההטרוגניות משמעותי בביטוי גנים נצפה, ותאים סווגו תאים כמו סוג 1-5 מבוסס על דפוס ביטוי (איור 3).

איור 1. בידוד תא יחיד של דג זברה nkx2.5: תאים חיוביים ZsY ב 18 hpf תמונות הר שלם של נציג Tg (nkx2.5: ZsYellow). העובר על 18 hpf עם (א) ZsYellow הקרינה לבד או (ב) התמזגה עם התמונה בשדה בהיר. (CF) האסטרטגיה gating נציג FACS להעשיר עבור תאים חיוביים ZsYellow ו (GJ) ניתוח שלאחר מין עם (C, G) FSC / gating גודל SSC, (D, H) כפיל אפליה, (E, I) gating Live / Dead , ו (F, J) מסודר אוכלוסייה. בר סולם הוא 100 מיקרומטר. לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
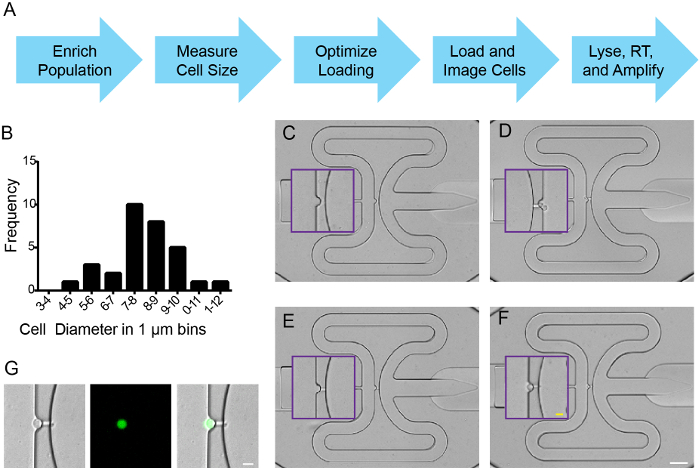
איור 2. לכידת תא יחידה של nkx2.5 דג זברה: ZsY אונג> תאים חיוביים. (א) זרימת עבודה. (ב) גודל תא הפצה עבור תאים ממוינים מן Tg (nkx2.5: ZsYellow) עוברים מבודדת 18 hpf. (CF) אירועי תא ללכוד נציג בצלחת IFC שם (C) הוא באר מים ריק (D) מכיל שני תאים, (E) יש תא בודד ונתקע ערוץ fluidics כמו "ללכוד ערוץ", ו- (F) הוא תא שנתפס יחיד. תיבות סגולות לסמן הבלעה עבור תצוגה מוגדלת של אתרים ללכוד. (G) לכידת תא יחיד עם brightfield, ZsYellow והתמזג תמונות. (CF) סרגל קנה מידה לבן הוא 50 מיקרומטר; סרגל קנה מידה צהוב הוא 10 מיקרומטר. (G) סרגל קנה מידה הוא 10 מיקרומטר. לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
igure 3 "src =" / files / ftp_upload / 53,877 / 53877fig3.jpg "/>
איור 3. ניתוח ביטוי גנים של nkx2.5 דג הזברה יחיד ללכוד:. ZsY תאים חיוביים ביטוי גנים על ידי qRT-PCR ב בקרות חיוביות (עוברים על 2 ימים-לאחר ההפריה, ונקווה cDNA של תאים בודדים), בקרה שלילית (ללא תבנית ו) 40 תאים בודדים (Cell 01-40). הערכים גלם CT קודדו צבע מבוסס כמתואר מפתח. Ef1a = 1a גורם התארכות, gata4 = חלבון 4 מחייב גאטה, nkx2.5 = homeobox 5 NK2, myl7 = אור שרירן שרשרת 7, vmhc = שרשרת כבדה שרירן חדרית, GAPDH = glyceraldehyde -3 ופוספט isl1 = ISL LIM homeobox 1. נא ללחוץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
| סף מחזור (CT) | ||||||||
| ef1a | gata4 | nkx25 | myl7 | vmhc | GAPDH | isl1 | ||
| עוּבָּר | 20.69 | 20.92 | 28.59 | 32.87 | 26.30 | 27.00 | 33.57 | |
| פּוּל | 26.4 | 22.4 | 27.7 | 27.4 | 24.5 | 29.2 | 40.0 | |
| NTC | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| סוג 1 | Cell-01 | 25.1 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 |
| Cell-02 | 25.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-03 | 26.3 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-04 | 27.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-05 | 28.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-06 | 28.2 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-07 | 29.3 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-08 | 30.7 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-09 | 34.3 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| סוג 2 | Cell-10 | 23.5 | 28.0 | 40.0 | 40.0 | 40.0 | 26.3 | 40.0 |
| Cell-11 | 23.5 | 27.2 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-12 | 23.7 | 17.9 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-13 | 24.1 | 32.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-14 | 24.9 | 27.5 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-15 | 25.9 | 28.6 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-16 | 26.0 | 28.1 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-17 | 27.0 | 27.7 | 40.0 | 40.0 | 40.0 | 38.5 | 40.0 | |
| Cell-18 | 27.0 | 27.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-19 | 27.0 | 30.2 | 40.0 | 40.0 | 40.0 | 38.2 | 40.0 | |
| Cell-20 | 27.0 | 28.2 | 40.0 | 40.0 | 40.0 | 39.2 | 40.0 | |
| Cell-21 | 27.7 | 30.3 | 40.0 | 40.0 | 40.0 | 38.6 | 40.0 | |
| Cell-22 | 30.4 | 26.2 | 40.0 | 40.0 | 40.0 | 38.3 | 40.0 | |
| Cell-23 | 31.3 | 22.4 | 40.0 | 40.0 | 40.0 | 39.7 | 40.0 | |
| Cell-24 | 31.5 | 28.8 | 40.0 | 40.0 | 40.0 | 39.5 | 40.0 | |
| Cell-25 | 33.8 | 27.4 | 40.0 | 40.0 | 40.0 | 37.3 | 40.0 | |
| Cell-26 | 40.0 | 27.4 | 40.0 | 40.0 | 40.0 | 36.6 | 40.0 | |
| סוג 3 | Cell-27 | 24.7 | 19.9 | 28.8 | 40.0 | 40.0 | 40.0 | 40.0 |
| Cell-28 | 24.8 | 28.4 | 26.3 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-29 | 25.1 | 23.5 | 27.6 | 40.0 | 40.0 | 32.5 | 40.0 | |
| Cell-30 | 26.3 | 21.4 | 27.5 | 40.0 | 40.0 | 39.9 | 40.0 | |
| Cell-31 | 26.9 | 24.8 | 28.2 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-32 | 27.0 | 22.5 | 23.8 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-33 | 27.8 | 27.6 | 27.7 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-34 | 28.1 | 21.9 | 26.6 | 40.0 | 40.0 | 37.7 | 40.0 | |
| Cell-35 | 29.3 | 26.5 | 28.1 | 40.0 | 40.0 | 38.3 | 40.0 | |
| סוג 4 | Cell-36 | 24.8 | 20.3 | 26.0 | 27.4 | 26.6 | 40.0 | 40.0 |
| Cell-37 | 28.7 | 22.3 | 25.9 | 28.9 | 22.9 | 38.5 | 40.0 | |
| סוג 5 | Cell-38 | 25.5 | 20.0 | 29.3 | 23.0 | 19.9 | 25.9 | 40.0 |
| Cell-39 | 25.9 | 21.8 | 28.6 | 25.1 | 21.7 | 25.7 | 40.0 | |
| Cell-40 | 28.9 | 22.0 | 27.0 | 23.0 | 21.3 | 27.6 | 40.0 | |
טבלה 1. ערכי הגלם CT.
Discussion
השיטה המתוארת במסמך זה השתמש ביטוי של חלבון פלואורסצנטי תחת השליטה של מקדם ספציפי תאים מהסוג להעשיר אוכלוסייה של ובתאים לב מעוברי דג זברה לשימוש במערכת ללכוד microfluidic בסיוע תא בודד להעריך ביטוי של תת-קבוצה של גני לב יחיד תאים. ובלבד יכולות עירור ופליטה לייזר FACS עולים בקנה אחד עם fluorophore (ים) של בחירה, שיטה זו יכולה לשמש לכל קו כתב ניאון. קווי כתב דג זברה רבים כבר קיים, ודגים מהונדסים נושאת לכתבי רומן יכולים להיוצר קטן כמו 3 חודשים. בנוסף, זרימת עבודה זו יכולה להתאים לייצר cDNA מן transcriptome השלם עבור סידור תא-רנ"א חד. לשם כך, על שלבי 5-7 יהיה צורך שונה במקצת לשימוש עם כימיות אופטימיזציה להכנת cDNA להפקת ספריות מתאימות רצף RNA. עם זאת, רצוי לאמת ההטרוגניות באוכלוסייהבאמצעות השיטה המתוארת כאן לפני כיווץ יישומי רצף תפוקה גבוהה.
חשוב לציין כי יש מספר מגבלות השיטה המתוארת. ראשית, השימוש במעגל microfluidic משולב (IFC) צלחות דורש ציוד מיוחד, יקר. עם זאת, שבבי microfluidic להציע יתרונות משמעותיים על פני 96 באר מסורתי 384 פורמטי צלחת גם עבור assaying תאים בודדים. מאת זורם תאים ריאגנטים דרך נתיבים בקנה מידה מיקרומטר, תאים בודדים מוצבים למתחמים ללכוד יחיד שבו הם יכולים לצפות ישירות לאשר כי הם תאים בודדים במצב בריאותי טוב. מיצוי RNA וסינתזה cDNA להתרחש באתרו על השבב IFC באמצעות בהיקפים נמוכים מאוד עבור כל תא. שבבי נוזליים משולבים, בעת כתיבת שורות אלה, הם זמינים מסחרית באמצעות מקור אחד בלבד. למרות ייצור שאינו מסחרי שבוצע על ידי מספר קבוצות, זה מחוץ הקיבולת של ברוב המעבדות. שלבים 5-7 רשאיצריך להיות שונה עבור פלטפורמות המיוצר על ידי חברות אחרות או בתוך הבית בדותות. שנית, למרות זמינות מסחרי צלחות IFC יכולות להכיל עד 96 גנים, הגנים שנבחרו מוגבל על ידי הזמינות של בדיקות גן fluorogenic שכותרתו תוקפות לשימוש דג זברה. תאים שלישיים, דג זברה הם בדרך כלל קטנים יותר מאשר בתאי יונקים, וגודל קטן זה מציב אתגרים לדגום עיבוד. שינוי קטן בקוטר תא מיתרגם שינוי גדול נפח תא, צמצום חומר זמין, ולהקטין את הסיכוי של גילוי נמוך לבטא גנים.
ישנם מספר שיקולים נוספים עבור פרוטוקול זה. בשלב 1, חשוב עובר לשימוש רק מופרה בתוך חלון זמן קצר. שמירה על טמפרטורה קבועה וללא הגבלת תנאי חמצן, עוברי דג הזברה לפתח באופן סינכרוני. את ההודעה הסופית של פרוטוקול זה (ביטוי גנים יחסי של תאים בודדים) אינה יכולה להפלות מקור heterogeneity בין תאים בודדים. מסיבה זו, פרשנות של ההטרוגניות אינטר מסתמכת על עוברים מבוימים בקפידה ופיתוח סינכרוני. בשלבים 2-3, קיימת תחלופה בין דיסוציאציה ואת הכדאיות. למרות צעד 4 בוחר עבור תאי קיימא, ואת השלב 3 מכיל מספר צעדי סינון המסייעים לכלול multimers, יתר העיכול יפחית את חומר הקיימא זמין עבור FACS ואפשרות להפחית תשואת תא ממוינת. צעדים כדי לפתור כדאיות נמוכה כוללת צמצום זמן עיכול, הפחתת עוצמת טחינה דקה, ואופטימיזציה של המספר העוברים בתוך החומר המוצא. עשרת FACS של אוכלוסיית התא של עניין (שלב 4) הוא לטעון את השלב הקריטי ביותר בפרוטוקול זה. קריטריוני מיון קפדניים חיוניים כדי ליצור הכנת תא יחיד המכילה רק לחיות תאים המבטאים את החלבון פלואורסצנטי של עניין. לזהם תאים מאוכלוסיות אחרות יכול להוביל מצג שווא של ההטרוגניות בין תאית. יש לציין,עשרת FACS של אוכלוסייה של העניין אינו מוגבלים את אסטרטגית הצבע שני לראות בפרוטוקול זה. אסטרטגיות נוספות מורכבות יכולות לייצר אוכלוסיות תאים מעודנות יותר הם דרך מהירה להאריך את המתודולוגיה שדווח ההוכחה במחקר עיקרון. בשלב 5, תאים עשויים להיות שנצפו ישירות כדי לאשר את מספר התאים בשבי, בריאות התא, ואת הביטוי של חלבון פלואורסצנטי. אבל, בהתאם לגודל תא בהיר, את fluorophore עניינים עשוי לא להיות ויזואלית לגילוי ואינו הודעה אמינה. אם תסיים לבצע בהצלחה את השלבים 6-8 דורש דבקות קפדנית פרוטוקולים של היצרן.
כהוכחה עקרונית, רמות ביטוי של תת-קבוצה של סמנים לבביים ידועים הוערכו ב יחיד נגזר nkx2.5 קו דג הזברה המהונדס BAC שתואר לעיל: ZsY לבחון סמני בידול לב nkx2.5 פרט: תאים חיוביים ZsY ב 18 שעות שלאחר הפריה (hpf). כדי לחקור את heterogeneity של סמני בידול מתבטאים nkx2.5 שנלכד: תאי מבטאי ZsY, חבילה של גנים היו assayed כוללת גני משק (ef1a, GAPDH), גורמי שעתוק הידועים להיות מופעלים מוקדם מפרט לב (gata4, nkx2.5) חברה סמן ובתאים שני בלב שדה (isl1), וגנים הידועים להיות מופעלים מאוחר יותר במהלך התמיינות לב (myl7, vmhc). השפע היחסי של כל גן נמדד על ידי qRT-PCR על ידי חישוב סף מחזור (CT). ערך CT הנמוך ביותר המחושב היה 17.9 ואת הגבוה ביותר היה 40, שאינו מהווה שום הגברה (טבלה 1). ערכי CT של> 35.0 נחשבו מתחת זיהוי.
של 46 התאים את מערכת הנתונים, 6 נפסלו עקב הגברה כושלת של כל גנים. התאים הנותרים היו תת-מסווג תאים לתוך 5 סוגים מבוססים על ביטוי של gata4, nkx2.5, myl7, ו vmhc, ו werדואר ממוין לפי ערך ef1a בתוך קבוצות. למרות GAPDH נכללה במקור גן משק אפשרי, הביטוי היה משתנה מאוד על פני התאים, מה שהופך אותו מתאים בתור גן משק. הביטוי GAPDH כנראה, עדיף מייצג שינויים במטבוליזם האנרגיה הקשורה בידול הלב סוג תא זה. מעניין, היה אחד התא שבו ef1a היה בלתי ניתן לגילוי אבל בגן אחר לא הורגש (gata1 בתאי 26). הדבר מצביע על כך בעוד ef1a הוא גן משק מספיק בדרך כלל להבחין בין תגובות שעתוק לאחור תא בודדים מוצלחות ולא מוצלחות, זה לא יכול להיות אידיאלי עבור כל היישומים. חוסר ביטוי ef1a בתא 26 יכול להיות בגלל הדינמיקה תעתיק תא בודד. מחקרים חד-תאים אחרונים מראים כי שעתוק גנים הוא סטוכסטיים ביסודו, לסירוגין בין שלבי פעילות תעתיק מהיר וזניחה 26. Tמודל התפוצצות התעתיק שלו רומז כי שימוש של גן משק עבור השוואות כמותיות בין תאים בודדים עשוי להיות לגמרי בלתי הולם.
בקבוצה סוג 1, ef1a הוא הגן רק לזיהוי. תאים אלה עשויים לכלול גם nkx2.5: ZsY תאים או תאים שליליים עם ביטוי נמוך מאוד של גנים האחרים בעלי עניין. הקלד 2 תאים, למעט תא-26, באה לידי ביטוי הן ef1a ו gata4 ללא מתאם ברמות הביטוי היחסי של הגנים האלה. יש לציין, ביטוי nkx2.5 ב סוג 1 ותאי סוג 2 יכול להיות בגלל חומרא FACS תת אופטימלית, התפוצצות תעתיק, leakiness transgene, או רמות ביטוי תת-הסף. סוג 3 תאים הביעו רמות לגילוי של ef1a, gata4, ו nkx-2.5. תאים אלה הם ובתאי לב סבירים והוא עשוי לכלול הבחנת cardiomyocytes פרוזדורים. סוג 4 תאים הביעו רמות לגילוי של ef1a, gata4, nkx2.5, myl7 </ em> ו vmhc והם תאים סבירים הבחנה לתוך cardiomyocytes חדרית. סוג 5 תאים הביע ef1a, gata4, nkx2.5, myl7, vmhc, ו GAPDH. התוספת של רמות לגילוי של GAPDH עולה כי תאים אלה יש את היכולת לחילוף חומרים glycolytic משופרת נמצא cardiomyocytes מובחן. זה היה מסקרן, כי למרות מיון תאים חיוביים ZsYellow מ Tg. (Nkx2.5: ZsYellow), עוברים, 21/40 תאים היו רמות תת-סף nkx2.5 בניתוח qRT-PCR שלנו זה יכול להיות בגלל התפוצצות תעתיק או יכול לשקף הבדלים בעיבוד תמליל בין nkx2.5 אנדוגני ZsYellow. חשוב לציין כי isl1 סמן שני לב השדה היה בלתי ניתן לגילוי בכל התאים או cDNA ונקווה מכל האתרים ללכוד שלנו. לסיכום, השוואת ברחבי דגימות, ההטרוגניות משמעותי ניכר ברמת התא הבודדה, דבר המצביע על כך ב 18 hpf, nkx2.5: ZsY + cells מהווה אבות לב כמו גם צאצאים בשלבים שונים של בידול.
Disclosures
The authors have nothing to disclose.
Acknowledgements
We thank Dr. C. Geoffrey Burns for fish stock. The authors are grateful to UNC Flow Cytometry Core Facility, UNC-CGIBD AAC core for resources enabling this project, and the ZAC facility for animal care. L.S. is supported by NIH T32 grant HL069768-13 (PI, Nobuyo Maeda). N.F. is supported by NSF Graduate Research Fellowship NSF-DGE-1144081. This study was supported by NIH P30DK034987 grant (to UNC Advanced Analytics Core), American Heart Association Scientist Development Grant 13SDG17060010 and Ellison Medical Foundation New Scholar Grant AG-NS-1064-13 (to Dr. Qian), and NIH R00 HL109079 grant (to Dr. Liu).
Materials
| Name | Company | Catalog Number | Comments |
| Supplies | |||
| Dumont #5 forceps | Fine Science Tools | 11254-20 | For removing the chorion from embryos |
| Microcentrifuge tube 2 ml | GeneMate | C-3261-1 | |
| 40 um cell strainer | Biobasic | SP104151 | |
| 35 mm culture dish | Falcon | 351008 | |
| FACS tubes topped with 35 um cell strainer | Falcon | 352235 | |
| P1000 and tips | Rainin | 17005089 | |
| P20 and tips | Rainin | 17005091 | |
| IFC chip manufacturer's protocol | Fluidigm | 100-6117 | Version 100-6117 E1 was used in representative experiment |
| Wide bore pastuer glass pippette | VWR | 14673-010 | For transferring embryos |
| Adult wild type zebrafish | N/A | We used AB line | |
| Adult transgenic zebrafish | N/A | We used Tg(nkx2.5:ZsYellow) | |
| Name | Company | Catalog Number | Comments |
| Reagents for cell dissociation | |||
| Double distilled water | N/A | ||
| Instant Ocean Sea Salt | Instant Ocean | SS15-10 | |
| NaCl | FisherScientific | S271-3 | make stock in water and use for de-yolking buffer |
| KCl | Sigma Aldrich | P5405 | make stock in water and use for de-yolking buffer |
| NaHCO3 | Sigma Aldrich | S6014 | make stock in water and use for de-yolking buffer |
| Leibovitz's L-15 | Gibco | 21083-027 | |
| FBS | FisherScientific | 03-600-511 | Heat inactivate; any brand of FBS should be fine |
| Cell Dissociation Reagent 1 -TrypLE | Life Technologies | 12605-010 | Store at room temperature. |
| Cell Dissociation Reagent 2 - FACSmax | Genlantis | T200100 | Store -20; thaw on ice; bring to room temp before use |
| pronase (optional) | Sigma | P5147 | |
| L/D Dye - Sytox Blue | Life Technologies | S34857 | Any live/dead stain suitable for flow cytometry will work |
| Trypan Blue | Gibco | 15250-061 | |
| Name | Company | Catalog Number | Comments |
| Reagents for IFC plate use and qRT-PCR | |||
| Gene-specific probes | Probes will vary by experiment | ||
| TaqMan Probe ef1a | Life Technologies | Dr03432748_m1 | |
| TaqMan Probe gata4 | Life Technologies | Dr03443262_g1 | |
| TaqMan Probe nk2-5 | Life Technologies | Dr03074126_m1 | |
| TaqMan Probe myl7 | Life Technologies | Dr03105700_m1 | |
| TaqMan Probe vmhc | Life Technologies | Dr03431136_m1 | |
| TaqMan Probe isl1 | Life Technologies | Dr03425734_m1 | |
| TaqMan Gene Expression Master Mix | Life Technologies | 4369016 | |
| Reagents listed in IFC manufacturer's protocol | |||
| C1 Reagent Kit | Fluidigm | 100-5319 | Reagents for loading cells onto IFC plate |
| Ambion Single Cell-to-CTTM | Life Technologies | 4458237 | Reagents for reverse transcription and pre-amplification steps |
| Molecular Biology Quality Water | Corning | 46-000CM | |
| C1 IFC for PreAmp (5-10 um) | Fluidigm | 100-5757 | IFC plate for small cells |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| C1 AutoPrep machine | Fluidigm | 100-5477 | For IFC plate use |
| Hemocytometer | Sigma Aldrich | Z359629 | For counting cells and assessing cell size |
| Dissecting microscope | For removing embryos from chorion | ||
| Tissue culture microscope | For assessing single cell digestion | ||
| FACS machine | For isolating cells of interest |
References
- Speicher, M. R. Single-cell analysis: toward the clinic. Genome medicine. 5, (2013).
- Macaulay, I. C., Voet, T. Single cell genomics: advances and future perspectives. PLoS genetics. 10, e1004126(2014).
- Marco, E., et al. Bifurcation analysis of single-cell gene expression data reveals epigenetic landscape. Proceedings of the National Academy of Sciences of the United States of America. 111, 5643-5650 (2014).
- Yin, H., Marshall, D. Microfluidics for single cell analysis. Current opinion in biotechnology. 23, 110-119 (2012).
- Garlanda, C., Dejana, E. Heterogeneity of endothelial cells. Specific markers. Arteriosclerosis, thrombosis, and vascular biology. 17, 1193-1202 (1997).
- Diaz-Cano, S. J. Tumor heterogeneity: mechanisms and bases for a reliable application of molecular marker design. International journal of molecular sciences. 13, 1951-2011 (2012).
- Guo, G., et al. Mapping cellular hierarchy by single-cell analysis of the cell surface repertoire. Cell stem cell. 13, 492-505 (2013).
- Guo, G., et al. Resolution of cell fate decisions revealed by single-cell gene expression analysis from zygote to blastocyst. Developmental cell. 18, 675-685 (2010).
- Treutlein, B., et al. Reconstructing lineage hierarchies of the distal lung epithelium using single-cell RNA-seq. Nature. 509, 371-375 (2014).
- Kimmel, C. B. Genetics and early development of zebrafish. Trends in genetics : TIG. 5, 283-288 (1989).
- Liu, J., Stainier, D. Y. Zebrafish in the study of early cardiac development. Circulation research. 110, 870-874 (2012).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Developmental dynamics : an official publication of the American Association of Anatomists. 203, 253-310 (1995).
- Zhao, S., Fung-Leung, W. P., Bittner, A., Ngo, K., Liu, X. Comparison of RNA-Seq and microarray in transcriptome profiling of activated T cells. PloS one. 9, e78644(2014).
- McCall, M. N., McMurray, H. R., Land, H., Almudevar, A. On non-detects in qPCR data. Bioinformatics. 30, 2310-2316 (2014).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature. 3, 1101-1108 (2008).
- Lyons, I., et al. Myogenic and morphogenetic defects in the heart tubes of murine embryos lacking the homeo box gene Nkx2-5. Genes & development. 9, 1654-1666 (1995).
- Chen, J. N., Fishman, M. C. Zebrafish tinman homolog demarcates the heart field and initiates myocardial differentiation. Development. 122, 3809-3816 (1996).
- Zhou, Y., et al. Latent TGF-beta binding protein 3 identifies a second heart field in zebrafish. Nature. 474, 645-648 (2011).
- Paffett-Lugassy, N., et al. Heart field origin of great vessel precursors relies on nkx2.5-mediated vasculogenesis. Nature cell biology. 15, 1362-1369 (2013).
- McCurley, A. T., Callard, G. V. Characterization of housekeeping genes in zebrafish: male-female differences and effects of tissue type, developmental stage and chemical treatment. BMC molecular biology. 9, 102(2008).
- Tang, R., Dodd, A., Lai, D., McNabb, W. C., Love, D. R. Validation of zebrafish (Danio rerio) reference genes for quantitative real-time RT-PCR normalization. Acta biochimica et biophysica Sinica. 39, 384-390 (2007).
- Serbedzija, G. N., Chen, J. N., Fishman, M. C. Regulation in the heart field of zebrafish. Development. 125, 1095-1101 (1998).
- de Pater, E., et al. Distinct phases of cardiomyocyte differentiation regulate growth of the zebrafish heart. Development. 136, 1633-1641 (2009).
- Hami, D., Grimes, A. C., Tsai, H. J., Kirby, M. L. Zebrafish cardiac development requires a conserved secondary heart field. Development. 138, 2389-2398 (2011).
- Yelon, D., Horne, S. A., Stainier, D. Y. Restricted expression of cardiac myosin genes reveals regulated aspects of heart tube assembly in zebrafish. Developmental biology. 214, 23-37 (1999).
- Molina, N., et al. Stimulus-induced modulation of transcriptional bursting in a single mammalian gene. Proceedings of the National Academy of Sciences of the United States of America. 110, 20563-20568 (2013).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved