Method Article
Isolierung und Charakterisierung von Einzelzellen aus Zebrafischembryonen
In diesem Artikel
Zusammenfassung
This protocol describes a method for isolating single cells from zebrafish embryos, enriching for cells of interest, capturing zebrafish cells in microfluidic based single cell multiplex systems, and assessing gene expression from single cells.
Zusammenfassung
The zebrafish (Danio rerio) is a powerful model organism to study vertebrate development. Though many aspects of zebrafish embryonic development have been described at the morphological level, little is known about the molecular basis of cellular changes that occur as the organism develops. With recent advancements in microfluidics and multiplexing technologies, it is now possible to characterize gene expression in single cells. This allows for investigation of heterogeneity between individual cells of specific cell populations to identify and classify cell subtypes, characterize intermediate states that occur during cell differentiation, and explore differential cellular responses to stimuli. This study describes a protocol to isolate viable, single cells from zebrafish embryos for high throughput multiplexing assays. This method may be rapidly applied to any zebrafish embryonic cell type with fluorescent markers. An extension of this method may also be used in combination with high throughput sequencing technologies to fully characterize the transcriptome of single cells. As proof of principle, the relative abundance of cardiac differentiation markers was assessed in isolated, single cells derived from nkx2.5 positive cardiac progenitors. By evaluation of gene expression at the single cell level and at a single time point, the data support a model in which cardiac progenitors coexist with differentiating progeny. The method and work flow described here is broadly applicable to the zebrafish research community, requiring only a labeled transgenic fish line and access to microfluidics technologies.
Einleitung
Die meisten aktuellen Studien der Zell- und Molekularbiologie sind auf Bevölkerungsdurchschnittswerten. Jedoch können wichtige biologische Ereignisse, die von diesen traditionellen populationsbasierten Analysen maskiert werden, da kleinere Populationen wichtige Rollen in biologischen Prozessen und Krankheitsverlauf spielen können. Genexpression in heterogenen Populationen auf Einzelzellebene zu verstehen , kann (und hat) führen zu relevanten biologischen und klinischen Erkenntnisse 1,2. Von Bedeutung für die embryonale Entwicklung Studien, in einer größeren Population von Zellen, sind Vorläuferzellen oft unterrepräsentiert, so dass es schwierig subtile Veränderungen in der Genexpression zu erkennen , die letztlich Entscheidungen Zellschicksal 3 einzuleiten. In ähnlicher Weise kann eine einzige Zelltyp unterschiedlich haben Expressionsprofile in Reaktion auf die Mikroumgebung 4. Zum Beispiel resident Endothelzellen im selben Organ oder in verschiedenen Organen (z. B. Aorta oder Niere) zeigen signifikante Heterogenität trotz teilen gemeinsame morp5 hological und funktionellen Eigenschaften. Darüber hinaus können die gleichen Tumor Bestücken Krebszellen haben auch molekularen Profile oder Mutationen auf Einzelzellebene 6 variiert.
In Modellsystemen hat Transkriptomik in einzelnen Zellen erfolgreich neue Zellpopulationen identifiziert, charakterisiert Zwischenzustände , die während der Zelldifferenzierung auftreten, und offenbarte Differential zelluläre Reaktionen 7.8.9 auf Reize. Solche Einsichten hätte in herkömmlichen populationsbasierten Studien maskiert. Zebrabärbling-Embryonen sind eine enorm in Anspruch genommenen Quelle von Stammzellen, Vorläufer und differenzierenden Zellen für Fragen der einzelnen Zelle Heterogenität und molekulare Regulation von zellulären Identitäten während der Entwicklung zu erforschen. Ihre hoch schablonenhaft , ex vivo Entwicklung und einfache genetische Manipulation machen sie ein ausgezeichnetes Modellsystem für diesen Ansatz 10,11. Insbesondere wird eine große Einschränkung der Interpretation von einzelnen Zelle gene - Expressionsdaten ist , dass eine zuverlässige Identifizierung neuer Zwischenzellenzustände während der Entwicklung sehr sorgfältige Timing von Gewebe erfordert Sammlung 9. Dies ist notwendig, um sicherzustellen, dass die Heterogenität zwischen gefangenen Zellen Heterogenität innerhalb eines Gewebes an einem einzigen Zeitpunkt anstatt Heterogenität in der Genexpression stellt durch altersabhängige Zelldifferenzierung dargestellt. Im Vergleich zu Mäusen, Zebrabärbling Embryonalentwicklung genau über eine große Anzahl von Embryos 12 synchronisiert werden kann. Zusätzlich mit großer Gelegegrößen, Genaktivität kann als ergiebige Quelle für Stamm- und Vorläuferzellen verwendet werden.
Dieses Protokoll beschreibt ein Verfahren, Zellen von Zebrafischembryos zu isolieren und Einzelzellen mit einem handelsüblichen integrierten Mikrofluidik Schaltung (IFC) Chip und Autoprep System für qRT-PCR Genexpressionsanalyse zu erfassen. Dieses Protokoll kann keinen hohen Durchsatz Multiplexassays einschließlich ganze schnell übertragbarTranskriptom - Sequenzierung , die 13 umfassendere Analyse der zellulären Heterogenität ermöglicht. Es bietet auch einige Vorteile zu herkömmlichen Genexpressionsassays. Die Einzelzellisolierung Protokoll ergibt eine hohe Lebensfähigkeit nach FACS, die den Anteil von kompromittierten Zellen verringert, die in Downstream-Anwendungen enthalten sind. Durch die Verwendung eines IFC kann gefangenen Zellen direkt Erfassungsraten beobachtet werden, um zu bewerten und Zellgesundheit morphologisch beurteilen. Darüber hinaus ist dieses Protokoll zu der Zebrabärbling Forschungsgemeinschaft breit anwendbar und erfordert nur einen markierten transgenen Fischen Linie und den Zugang zu mikrofluidischen Zellerfassungstechnologien.
Als Beweis für das Prinzip, von Herz-Vorläufern abgeleitet Einzelzellen wurden auf einem IFC Chip isoliert und gefangen genommen und dann die relative Häufigkeit von Herz-Differenzierungsmarker wurde durch qRT-PCR gemessen. Genexpressionsanalyse auf Einzelzellebene zeigt, dass Herz-Vorläufern mit ihren differe koexistierenntiating Nachkommen. Die Einsicht von Single-Cell-Profilierung von Herz-Vorläufern gewonnen können unter Herzvorläuferzellen während der Entwicklung von Wirbeltieren Licht auf die Heterogenität in Genexpressionsmuster Schuppen, die in der traditionellen populationsbasierten Analysen maskiert wurden.
Protokoll
Dieses Protokoll erfordert die Verwendung von lebenden, erwachsenen Zebrafischembryonen zu erzeugen. Die Embryonen werden für die Gewebesammlung geerntet. Es ist wichtig, die Genehmigung zuständigen Ethik Review Boards zu erhalten, dieses Experiment durchzuführen.
1. Besorgen Inszenierte Embry
- Am Tag vor dem Experiment, bereiten gesunde, erwachsene Zebrafisch für die Zucht. Setzen Sie ein Mann und eine Frau, die auf gegenüberliegenden Seiten eines klaren Teiler in einem Zuchtbecken.
- Wiederholen 1.1 für so viele Zuchtbecken als notwendig für eine ausreichende Embryo Produktion für die nachgeschaltete Anwendung. Erhalten Embryonen von beiden Wildtyp-Fisch und transgene Fische, die fluoreszierende Proteine in den Zelltyp von Interesse exprimieren.
HINWEIS: Die Anzahl der Embryonen für die Downstream-Anwendungen in den Schritten 2-8 benötigt wird, hängt von der relativen Häufigkeit der Zellen von Interesse zu dem Zeitpunkt, von Interesse. Obwohl dies nach Zelltyp variieren kann, produzieren 200 Embryonen 2000-5000 sortierten Zellen, wenn die cells von Interesse repräsentieren <1,0% der gesamten Zellen bei 24 HPF (Stunden nach der Befruchtung). - Am nächsten Morgen, ändern Sie das Wasser in der Panade Tank von Fisch zu einem frischen Zuchtbecken und entfernen Sie die Trennlinie zu übertragen. Kippen Sie den Tank in einem Winkel Zucht zu fördern.
- Sammeln Sie inszeniert Embryonen.
- Alle 15 min, sammeln Embryonen, die durch die Erwachsenen zu einem frischen Zuchtbecken zu übertragen und die Eier vorbei, die hinten durch ein Teesieb gelassen werden.
HINWEIS: Zebrabärbling-Embryonen synchron entwickeln, wenn bei vergleichbaren Dichten und Temperaturen gehalten. - Spülen Sie die Eier mit Ei Wasser (0,21 g / L Instant Ocean Salze in 1 l doppelt destilliertem Wasser) aufnehmen und in eine Petrischale. Übertragen Sie die Petrischale mit einem feuchten Inkubator bei 28,5 ° C mit Luftzirkulation.
- Alle 15 min, sammeln Embryonen, die durch die Erwachsenen zu einem frischen Zuchtbecken zu übertragen und die Eier vorbei, die hinten durch ein Teesieb gelassen werden.
- Zwei Stunden nach der letzten Erhebung, Art befruchtet, mehrzelligen Embryonen in 10 cm Petrischalen und reduzieren Dichte auf 50 Embryonen pro Schale. Wählen Embryonen aus einem einzigen, 15min Zeitfenster Sammlung für Downstream-Anwendung. Inkubieren Embryonen bei 28,5 ° C.
HINWEIS: Zum Beispiel sammeln Embryonen bei 08.30, 08.45, 09.00, 09.15, 09.30, 09.45, 10.00 und 10.15 Uhr. Vergleicht man über Zeitpunkte, wenn die größte Zahl der befruchteten Embryonen aus den Klauen um 9:00 gesammelt sind, dann verwenden Sie nur diese Embryonen für die Downstream-Anwendungen.
2. für Einzel Zelldissoziationsmedium Set Up
- Etwa 30 Minuten vor dem Zeitpunkt der Umgebung (18 HPF) entfernen Embryonen aus ihrem Chorion manuell mit einer feinen Pinzette.
- Sammeln und beschriften Sie die folgende für jede Bedingung: zwei 2 ml Mikrozentrifugenröhrchen, ein 40 & mgr; m Zelle Sieb, einer 35 mm Zellkulturschale und zwei FACS-Röhrchen mit einem Sieb 35 um Zelle gekrönt.
- Kühlen Sie die folgenden Reagenzien auf Eis: Ei Wasser 0,21 g / L Instant Ocean Salze in 1 l doppelt destilliertem Wasser enthält; De-yolking Puffer enthaltend 55 mM NaCl, 1,8 mM KCl und 1,25 mM NaHCO 3; und FACS-Puffer enthält, Leibovitz L-15-Medium, supplementiert mit 5% hitzeinaktiviertem fötalem Rinderserum.
- Bringt 1 ml pro Probe Zelldissoziationsmedium Reagenz 1 auf RT. Thaw 1 ml Zelldissoziationsmedium Reagenz 2 pro Probe auf Eis. Bringen Sie unmittelbar vor dem Gebrauch auf RT.
3. Single Zelldissoziationsmedium
- Unter Verwendung einer breiten Bohrungsglaspipette übertragen 100-300 Embryonen in ein 2 ml Mikrozentrifugenröhrchen in einem minimalen Volumen an Wasser Egg.
- Euthanize Embryonen, die durch Wasser mit 1 ml eiskaltem Ei Wasser ersetzt und Untertauchen Rohr in Eis für 20 min.
ACHTUNG! Es ist wichtig , die Zustimmung angemessenen institutionellen Tierpflege Aufsichtsgremium für diese Euthanasie Verfahren zu erhalten (auf Eis durch Zelldissoziationsmedium als sekundäre Euthanasie gefolgt Kühlen). Standard-Euthanasie erfordert in der Regel, dass Embryonen <3 Tage nach der Befruchtung gebleicht werden, nachdem sie gekühlt werden, die für den Erhalt der lebenden Zellen nicht geeignet ist. - Waschen Sie die Embryonen zweimal mit 1 ml eiskaltem Wasser Ei. Zum Waschen verwenden, um eine breite Bohrung Glaspipette Ei Wasser und P1000 zu entfernen Egg Wasser hinzuzufügen.
- Entfernen Sie das Eigelb durch den Embryo Wasser mit 1 ml Deyolking Buffer ersetzen und Verreiben 8-12 mal mit einer P1000 Spitze, oder bis das Eigelb gelöst und nur die Körper der Embryonen sind sichtbar.
- Sammeln Sie das Gewebe durch Zentrifugation bei 300 g für 1 min. Verwenden Sie eine Pipette vorsichtig den Überstand zu entfernen, ohne das Gewebe Pellet zu stören. Re-suspend in 1 ml Wasser Ei.
- Wiederholen Sie Schritt 3.5 für insgesamt drei Waschungen, aber auf dem letzten Waschen resuspendieren in 1 ml RT Zelldissoziation Reagenz 1.
- Inkubieren in Zelldissoziation Reagenz 1 10 min bei RT mit Rohren horizontal angeordnet. Alle 2-3 min, verreiben vorsichtig mit einem P1000 Pipette Verklumpen zu verhindern.
Hinweis: Behandeln Proben sanft während Verreiben Schritten. Irgendwann eine einzige, werden große Klumpen aus einem Gewirr von Embryo Körper in der Röhre bilden. Dieser Willezerstreuen mit zusätzlichen Verdauung durch vorsichtiges Zerreiben unterstützt. NICHT VORTEX. - Sammeln Sie Gewebe durch Zentrifugation bei 300 g für 3 min. Überstand entfernen und resuspendieren in 1 ml Zelldissoziationsmedium Reagenz 2.
- Inkubieren für 5-15 min bei RT. Die Röhrchen horizontal. Alle 2-3 min, verreiben sanft Verklumpen zu verhindern. Alle 5 min, zu bewerten Verdauung Fortschritt.
- Zur Beurteilung der Verdauung Fortschritt, verdünnen 2 ul Überstand in 18 & mgr; l FACS-Puffer und Pipette als ein Tröpfchen auf einer Zellkulturschale.
- Legen Sie ein Deckglas über die Probe und beobachten unter einer Gewebekultur Mikroskop bei 10X und 20X Vergrößerung.
HINWEIS: Die Zubereitung als eine Mischung aus einzelnen Zellen, kleine Cluster, große Cluster erscheinen soll, und gelegentlich meist intakten Embryo Körper. - Visuell beurteilen, um den Anteil der Zubereitung, die einzelne Zellen und kleine Cluster ist.
HINWEIS: Über Verdauung Lebensfähigkeit zu reduzieren; Unter Verdauung wird einzelne Zellausbeute zu reduzieren.
- Sammeln der Zellpräparation durch Zentrifugation bei 300 xg für 5 min. Überstand verwerfen und erneut zu suspendieren in 1 ml kaltem FACS-Puffer.
- Befeuchten Sie ein 40 & mgr; m Zellsieb mit FACS-Puffer. Übergeben Sie die Zellsuspension durch die 40 & mgr; m Zelle Sieb auf eine 35 mm-Zellkulturschale. Waschen Sie die Zelle Sieb einmal mit 1 ml FACS Sortierpuffer.
- Übertragung der Durchfluss in ein 2 ml-Mikrozentrifugenröhrchen. Sammle die Zellen durch Zentrifugation bei 300 xg für 5 Minuten und Resuspendieren in 100 & mgr; l FACS-Puffer.
- Graf Zellausbeute unter Verwendung einer Zählkammer. Verdünnen Weitere Proben bis 5x10 6 Zellen pro ml oder optimale Konzentration für die FACS - Maschine der Wahl empfohlen. Optional Wenn diese der Hemocytometer, Gegenfärbung toten Zellen mit Trypanblau Zellausbeute zählen Lebensfähigkeit der Zellpräparation Sortierung vor der FACS zu bestätigen.
HINWEIS: Die Vorbereitungen, die zu verdünnen sind, werden überschüssige Menge an Zeit in Anspruch nehmen zu sortieren. Die Vorbereitungen, die zu sind concentrated neigen zu Multimeren zu verklumpen und sind suboptimal für eine reine Population zu sortieren. - Reserve 10-20% jeder Probe als ungefärbten Kontrollen. Für restlichen Proben, Flecken tote Zellen durch Zugabe eines fluoreszierenden Live / Dead (L / D) Diskriminierung in jeder Probe zu färben. Nicht waschen.
Hinweis: alle Fluoreszenzfarbstoff, der Zellen mit beeinträchtigter Zellmembranen und Flecken Nukleinsäuren durchdringt, kann als L / D-Farbstoff verwendet werden, vorausgesetzt, dass die Fluoreszenzspektren mit der FACS-Maschine der Wahl und des Fluorophors Kennzeichnungs Zellen von Interesse vereinbar ist.- Nicht waschen die Herstellung nach den Farbstoff Zugabe, da einige Zellen im Transitverkehr zu FACS Reinigung sterben werden und sollte aus der sortierten Population ausgeschlossen werden.
- Befeuchten Sie die 35 & mgr; m Zellsieb capped FACS-Röhrchen mit 20 & mgr; l FACS-Puffer. In Zellen mit dem Sieb und sammeln durch die Schwerkraft. Lagern auf Eis.
4. FACS Anreicherung
- Verwenden Sie FACS für einzelne zu bereichern, Lebende Zellen, die fluoreszierende Marker exprimieren.
- Stellen Sie eine Gate-Zellen, die aus Trümmern in einem Streudiagramm der Vorwärtsstreuung (FSC-A) Amplitude vs Seitenstreuamplitude (SSC-A), die beide mit linearer Skalierung zu unterscheiden.
Achtung! Zebrabärbling - Zellen sind typischerweise kleiner als Maus oder menschliche Zellen. Dies ist in einem unteren basal Trennung zwischen den Zellen und Debris reflektiert. - Vom Tor in 4.1.1 festgelegt, setzen Sie ein Tor für einzelne Zellen zu bereichern und Multimere mit einem Streudiagramm Vorwärtsstreuung Höhe (FSC-H) und Low-Side-Streuhöhe (SSC-H), die beide mit linearer Skalierung auszuschließen.
Hinweis: Zellen mit überproportional hohen FSC-H und niedrigen SSC-H sind wahrscheinlich Multimere und werden von der Sortierung ausgeschlossen. - Vom Tor Satz in 4.1.2, mit einzelnen Farbsteuerung, setzen Sie ein Tor nur lebende Zellen enthalten ein Streudiagramm von FSA-A mit linearer Skalierung und Amplitude des Kanals verwendet, um L / D Fleck mit log Skalierung zu erkennen. Fügen L / D-negativen Zellen.
- Vondas Tor in 4.1.3 festgelegt, einzelne Farbsteuerung verwenden, stellen eine Gate nur lebende Zellen mit positiven Fluoreszenz unter Verwendung eines Streudiagramm von FSA-A mit linearer Skalierung und Amplitude des Kanals verwendet werden, zu fluoreszierendes Protein Zellmarker mit Log-Skalierung zu erkennen . Fügen Sie positive Zellen.
- Gesetzt keine Kompensationssteuerungen, falls erforderlich, für eine Interferenz zwischen dem L / D-Färbung und fluoreszierendes Protein Spektren zu berücksichtigen.
- Stellen Sie eine Gate-Zellen, die aus Trümmern in einem Streudiagramm der Vorwärtsstreuung (FSC-A) Amplitude vs Seitenstreuamplitude (SSC-A), die beide mit linearer Skalierung zu unterscheiden.
- Set Art Logik-Zellen, die in alle Tore in Schritt 4.1 gesetzt fallen.
- Mit Doppel-Zellen markiert, überprüfen Sie für Zellsortierung Gating.
HINWEIS: Die Zellen für die Sortierung werden die Zellen statt Schutt, einzelner Zellen anstatt Multimere, L / D-negativ und Fluoreszenz-positiven Zellen sein.
- Mit Doppel-Zellen markiert, überprüfen Sie für Zellsortierung Gating.
- Sortieren 2.000-4.000 Zellen aus der Population von Interesse in 5 ul kaltem FACS-Puffer in einem Mikrozentrifugenröhrchen auf Eis. Um Scher und Belastung auf Zellen während des Sortierens, verwenden Sie die niedrigsten Drücken möglich, dass die Zelle zu minimierenSorter.
HINWEIS: Die 5 ul Tröpfchen FACS Puffer dient als Puffer für Zellen die FACS-Maschine verlassen. 2.000-4.000 Zellen Sammeln direkt in FACS-Puffer beseitigt die Notwendigkeit für die Zentrifugation vor auf die IFC-Chips zu laden. Diese Strategie wird empfohlen, weil der Zentrifugation zur Bildung von Multimeren führen kann, wenn die Zellen miteinander in dem Pellet anhaften. - Beurteilen Sie die Post-Sortiertragfähigkeitsanalyse.
- Übertragung 1 & mgr; l Zellen in ein frisches FACS-Röhrchen sortiert mit 100 ul FACS Sortierpuffer und 1: 1000 Verdünnung von L / D-Fleck. Führen Sie die Zellen durch die FACS-Sorter mit der Gating-Strategie in Schritt 4.1. Verwenden Sie den Anteil von L / D negativ auf positiv Lebensfähigkeit von sortierten Zellen abzuschätzen.
5. Wägezellen auf Mikrofluidik-Chip
- Mit Hilfe einer Zählkammer, messen sowohl die Konzentration und Größe der sortierten Zellen.
- Verdünnte sortierten Zellen auf mindestens 10 & mgr; l. Einen aliquoten von 5 μ; L Zellen mit 5 ul Trypanblau. Legen Sie auf Hämozytometer und anwenden Deck.
- Sammeln Hellfeldbilder aller Zellen in 4 x 4 Gitter von Hemocytometer. Zählen Sie die Anzahl der lebenden Zellen und berechnen lebenden Zellen / ml. Lebende Zellen nehmen nicht Trypan blau.
HINWEIS: Verwenden Sie eine beliebige Standard-Bildanalyse-Software, den Durchmesser aller lebenden Zellen zu messen. Berechnen Sie den Mittelwert und Standardabweichung der Zellengröße, und wählen Sie von Interesse, eine IFC Platte geeignet für die Zellgrößenbereich. Wenn Zellgrößenbereich eine IFC Plattenzellgrößenbereich spreizt, verwenden Sie mehrere Platten, die das gesamte Spektrum von Zellen von Interesse zu erfassen.
- Wägezellen auf IFC Platte den Anweisungen des Herstellers nach (siehe Materialien).
- Verdünnte Zellen bis 1x10 6 Zellen / ml und Auftrieb Optimierung durchführen als den Anweisungen des Herstellers.
HINWEIS: weder Buoyancy Optimierung sorgt dafür, dass die Zellen auf den Boden sinken, noch schweben an die Spitze des Lade well für optimale Beladung auf IFC Chip. Das Verhältnis von Puffer zu Zellen, die durch Zelltyp kann variieren, aber liegt typischerweise im Bereich 6: 4-7: 3 von Zellen: Puffer. - In Zellen auf grundierte-IFC-Platte. Lastplatte in kompatible Fluidik-Maschine, und führen Sie Skript, um eine Zelle Ladezellen durch Mikrofluidik Schaltung zu schieben und in Capture-Bahnen.
- Verdünnte Zellen bis 1x10 6 Zellen / ml und Auftrieb Optimierung durchführen als den Anweisungen des Herstellers.
- Bestätigen Sie, dass Zellen in Fangstellen auf IFC Platte eingelegt werden.
- Entfernen IFC Platte von Fluidik-Maschine. Montieren Sie die IFC Platte auf einem Mikroskop mit einem Plattenadapter ausgestattet.
- Sammeln Sie Schnappschüsse von Hellfeld und Fluoreszenz für jede Einfangstelle bei 10-facher Vergrößerung.
HINWEIS: Hell Capture Zeiten können von 10 bis 50 ms liegen, je nach Lampenintensität. Die Fluoreszenz-Capture-Zeiten können 250 bis 750 ms liegen, abhängig von Fluorophor Helligkeit. Vermeiden Zellen überzubelichten Lichtschäden zu verhindern.
6. cDNA Synthesis
- Führen Zelllyse in situ gemäß IFC Platte Anweisungen des Herstellers.
- In Lysepuffern in die Vertiefungen auf IFC Platte. Lastplatte auf kompatible Fluidik-Maschine und Lauf Zelllyse Skript als den Anweisungen des Herstellers.
- Führen Sie die reverse Transkription nach IFC Platte Anweisungen des Herstellers.
- In der reversen Transkription Reagenzien IFC Platte. Lastplatte auf kompatible Fluidik-Maschine. Führen Sie die reverse Transkription-Skript. Probenzyklusbedingungen: 1 Zyklus bei 25 ° C für 10 min, 1 Zyklus bei 42 ° C für 1 h, dann 1 Zyklus bei 85 ° C für 5 min.
- Führen Sie Vorverstärkung mit Gen-spezifischen Sonden nach IFC Platte Anweisungen des Herstellers.
HINWEIS: Aufgrund ihrer höheren Spezifität bei niedriger Kopienzahlen verwenden fluorogenen-markierten Sonden anstatt für den Einsatz optimierte Sonden mit Interkalator Farbstoffen.- Pool-Primer und verdünnt auf endgültige 180 nM jeder. In gepoolten Primer an IFC Platte. Lastplatte auf kompatible FLUIDIcs Maschine. Führen Sie Vorverstärkung Skript.
- Führen Vorverstärkung mit den folgenden Zyklusbedingungen: 1 Zyklus bei 95 ° C für 10 min, dann 18 Zyklen mit 15 sec bei 95 ° C denaturieren dann Tempern und Verlängerung bei 60 ° C für 4 min. Store cDNA bei 4 ° C bis zur Ernte vorge amplifiziert. Ernte-cDNA aus mikrofluidischen Platte gemäß den Anweisungen des Herstellers und lagern bei -20 ° C bis zur Verwendung.
7. Wählen Sie cDNA von Einzelzellen für Einzelziel qRT-PCR
- Verdünnte cDNA in 25 & mgr; l Verdünnungs Reagens nach den Anweisungen des Herstellers.
HINWEIS: Die ungefähre endgültige Ausbeute beträgt 28 & mgr; l von vorverstärkten cDNA pro Zelle. Reservieren Sie einen aliquoten Teil des Verdünnungsmittels für die Verwendung als nicht-Template Kontrolle in qRT-PCR.- Mit Bezug auf Hellfeld und Fluoreszenz-in-Schritt aufgezeichneten Bilder 5.3.2, Score jede Einfangstelle. Manuell zu zählen und die Anzahl der Zellen aufzuzeichnen. Beurteilen und notieren, ob jede Zelle Grippeorescent.
HINWEIS: Jede einzelne Website enthalten 0, 1 oder> 1 Zelle, wo> 1 Zelleinfang Websites umfasst, die mehrere Zellen und / oder nicht identifizierbare Trümmer enthalten. Wenn die Bilder ausreichender Qualität, ein Pixelwert sind auch zugeordnet werden können, um die Intensität des Fluoreszenzsignals quantitativ zu bestimmen.
- Mit Bezug auf Hellfeld und Fluoreszenz-in-Schritt aufgezeichneten Bilder 5.3.2, Score jede Einfangstelle. Manuell zu zählen und die Anzahl der Zellen aufzuzeichnen. Beurteilen und notieren, ob jede Zelle Grippeorescent.
- Ausgewählte Proben für qRT-PCR-Analyse.
- Wählen Sie cDNA-Proben von Fangstellen identifiziert in Schritt 7.2, die genau 1 Zelle mit positiven Fluoreszenz enthalten. Pool 1 & mgr; l Aliquots von cDNA aus jeder Probe.
ANMERKUNG: Dies ist der "Pool" positive Kontrolle verwendet, um zu bestimmen, ob die Gene von Interesse in einer der ausgewählten Zellen exprimiert werden. - Wählen Sie cDNA aus mindestens einer Fangstelle, die genau 0-Zellen als negative Kontrolle enthält. Verwenden Sie Verdünnungsmittel aus Schritt 7.1.2 als No-Template-Steuerung.
- Wählen Sie cDNA-Proben von Fangstellen identifiziert in Schritt 7.2, die genau 1 Zelle mit positiven Fluoreszenz enthalten. Pool 1 & mgr; l Aliquots von cDNA aus jeder Probe.
- Führen Sie qRT-PCR-Assays.
- Verwenden Sie nur Sonden verwendet für die Prä-Amplifikation in Schritt 6.4.1. Laden Sie alle Proben in triplicate. Zyklusbedingungen für eine 10 & mgr; l-Reaktion auf einer 384-Well-Platte: 1 Zyklus 50 ° C 2 min, 1 Zyklus 95 ° C für 10 min, 40 Zyklen mit 95 ° C 15 sec Denaturierung dann Annealing und Extension bei 60 ° C für 1 Minute
8. Datenanalyse
- Validieren qRT-PCR-Produkte von Standard-Gelelektrophorese. Bestätigen Produkt eine einzelne Bande bei einem erwarteten Molekulargewicht (variiert für jede Sonde).
HINWEIS: Wenn eine Sonde mehrere Bänder oder eine einzelne Bande bei einem ungeeigneten Größe erzeugt, dann Spezifität ist fraglich, und das schließt das Gen aus der Analyse. - Überprüfen Sie alle Amplifikationskurven und Konto für alle Anomalien 14. Berechnen Sie den Mittelwert CT - Wert für jede Probe / Gen - Kombination 15.
HINWEIS: CT-Werte für positive Kontrollen liegen typischerweise im Bereich von 10 bis 30. CT-Werte für negative Kontrollen liegen typischerweise im Bereich von 35 bis 40 (ohne Verstärkung). CT-Werte von 30 bis 35 sind sehr niedrig Ausdruck betrachtetund sollten mit Vorsicht interpretiert werden 14. - Report-Daten
- Wenn Proben in der CT fallen = 10-30, Daten auch als fache Veränderung der Transkript Fülle relativ zu einer Kontrollprobe gemeldet werden kann.
HINWEIS: Falten Änderung gleich 2 ^ (- ΔΔCT) , wo ACt zu einem Housekeeping - Gen relativ ist , indem die durchschnittliche CT der Probe aus dem Housekeeping - Gen subtrahiert und ΔΔCT ist die Unterschiede in ACt zwischen der Probe und einer positiven Kontrolle 15.
- Wenn Proben in der CT fallen = 10-30, Daten auch als fache Veränderung der Transkript Fülle relativ zu einer Kontrollprobe gemeldet werden kann.
Ergebnisse
Als Beweis für das Prinzip wurde die Genexpression zu bewerten Differenzierung Dynamik während der Herzentwicklung erforschen. In Zebrabärbling entstehen Herzvorläuferzellen aus einer mesodermalen Population von Zellen, die an der vorderen Seitenplatte Mesoderm wandern, wo sie verschmelzen die lineare Herzrohr zu bilden. Vor der Fusion beginnen Herz Vorläufern den Transkriptionsfaktor Nkx2.5 (NK2 homeobox 5) zum Ausdruck bringen, die die früheste spezifischer Marker für Herz-Vorläufern 16,17 zu sein , gedacht wird. Hier wird eine vorher beschriebenen BAC transgener Fische Tg (Nkx2.5: ZsYellow) 18, abgekürzt Nkx2.5: ZSY wurde verwendet , um Herz-Differenzierungsmarker in einzelnen Zellen an der 18 somite Stufe, 18 Stunden nach der Befruchtung (HPF) 12 untersuchen. Dies war der früheste Zeitpunkt, an dem ZsYellow Signal war visuell detektierbar. Wie zuvor für diese transgene Linie beschrieben, Etiketten ZsYellow Herz Vorläufern, wie wirll als ein paar extra Herzzellen , die Anlass zu der Rachen geben Bogen Endothelzellen bei 28 HPF 19 (1A-B, Daten nicht gezeigt). Mit 18 HPF, Nkx2.5: ZSY Embryonen wurden in einer einzigen Zellsuspension dissoziiert dann mit Sytox Blau gefärbt tote Zellen auszuschließen. Live, ZsYellow positiv, Sytox Blau - negativen Zellen wurden FACS sortiert entweder eine MoFloXDP oder Sony SH800Z Sortierer ausgestattet mit 100 & mgr; m Düse (1C-F). Zur Beurteilung Reinheit der sortierten Population und Postsortier Lebensfähigkeit, ein Aliquot von sortierten Zellen wurden mit Sytox Blue gefärbt und bewertet den Prozentsatz von Ereignissen , die innerhalb der ursprünglichen Sortiergatter (1G-J) fiel.
Nach dem Sortieren wurden die Zellen in einem integrierten Mikrofluidik - Schaltkreis (IFC) -Chip Arbeitsablauf (2A) eingegeben gemäß den Anweisungen des Herstellers. Ein Hemocytometer, kombiniert mit Trypanblau exclusion wurde verwendet Zelldurchmesser zu messen, der Konzentration, und die Lebensfähigkeit (2B und Daten nicht gezeigt). Zell Auftrieb wurde optimiert (6,5: 3,5 Zellen: Puffer) gemäß den Anweisungen des Herstellers, und die Zellen auf eine IFC geladen wurden 5-10 & mgr; m Durchmesser Zellen zu erfassen. Um Fangeffizienz, Fluoreszenz und / oder Hellfeldsignale wurden auf allen Fangstellen abgebildet, eine Kachelfunktion in FIJI beurteilen wurde verwendet, um ein einzelnes Bild der Mikrofluidik Platte zu nähen. Jeder Capture Site 0, 1 oder> 1 einzelnen Zellen (2C-F). Wie erwartet von FACS - Anreicherung, ausgedrückt gefangenen Zellen ZsYellow (2G). Einfangeffizienz 90% in 5 Einzelversuchen überschritten Nkx2.5 Verwendung: ZSY Zellen sortiert, und mindestens 70% der Bindungsstellen wurden von einer einzelnen Zelle besetzt (Daten nicht gezeigt). Zellen wurden lysiert, RNA isoliert, cDNA synthetisiert und spezifische Zielgene wurden amplifiziert, alle entsprechend den Anweisungen des Herstellers.
Eine Untergruppe von Zellenfangstellen wurden für qRT-PCR-Analyse ausgewählt, wie in dem oben beschriebenen Protokoll. Insbesondere Elongationsfaktor 1A (efl1a) 20 und Glyceraldehyd-3 - Phosphat (GAPDH) 21 wurden als Housekeeping - Gene getestet zu erwarten in jeder Zelle exprimiert werden; GATA - Bindungsprotein 4 (GATA4) 22 und NK2 homeobox 5 (Nkx2.5) so früh Herzvorläufermarker; ISL LIM Homeobox 1 Box 1 (ISL1) als zweites Herz Feld Marker 23,24; und Myosin Light Chain (myl7) und ventrikulären Myosin schwere Kette (vmhc) 25 bis Kardiomyozyten markieren. Genspezifischen Sonden wurden unter Verwendung von cDNA aus 48 hpf Embryonen validiert (Abbildung 3). qRT-PCR wurde verwendet, um relative Genexpression dieser Gene aus 18 HPF Embryonen in 45 einzelnen Zellen zu bewerten, eine No-Template Negativkontrolle und einer gepoolten Population positive Kontrolle haltendeng cDNA aus allen 96 Fangstellen. Roh-CT-Werte für 40 repräsentative Zellen und Kontrollen sind in Tabelle 1 gezeigt Bemerkenswerter, aus dem Pool von 46 Zellen untersucht, 6 Zellen von der Analyse ausgeschlossen wurden, da alle Genexpression CT Werte überschritten CT = 35,0, und es ist nicht bekannt, ob dieser hohe CT Werte sind auf wahre, sehr niedrige Expressionsniveaus oder Probenabbau. Da der Bereich der CT - Werte für das Housekeeping - Gen, EF1A, war zu breit Genexpression zwischen den Proben zu vergleichen und viele CT - Werte überschritten 30 wurden CT - Werte als Heatmap visualisiert (Abbildung 3). Vergleicht man in Proben, erhebliche Heterogenität in der Genexpression beobachtet wurden , und die Zellen Zellen als Typ 1-5 basierend auf Expressionsmuster (Abbildung 3) eingestuft.

Abbildung 1. Einzelzellisolierung von Zebrabärbling Nkx2.5: ZSY positive Zellen bei 18 HPF Whole Mount Bilder von repräsentativen Tg (Nkx2.5: ZsYellow). Embryo bei 18 HPF mit (A) ZsYellow Fluoreszenz allein oder (B) fusioniert mit Hellfeldbild. (CF) Repräsentative FACS Gating - Strategie zur Anreicherung für ZsYellow positive Zellen und (GJ) Post-Sortieranalyse mit (C, G) FSC / SSC Größe Gating (D, H) Dublette Diskriminierung, (E, I) Live / Dead - Gating und (F, J) sortiert Bevölkerung. Maßstabsbalken ist 100 & mgr; m. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
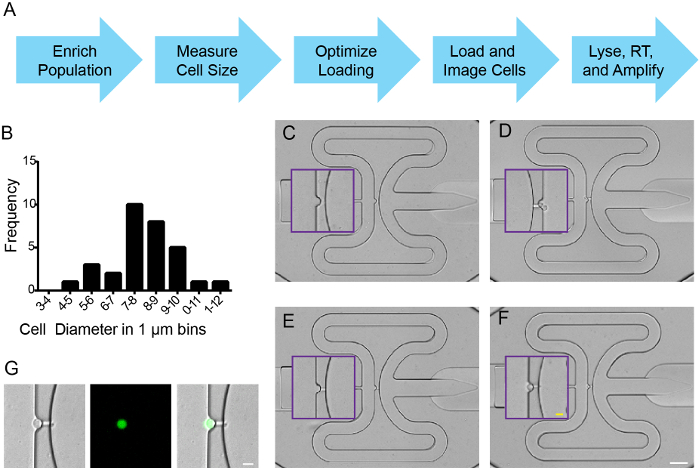
Abbildung 2. Einzelzelle Erfassung von Zebrabärbling Nkx2.5: ZSY Ong> positive Zellen. (A) Arbeitsablauf. (B) Zellgrößenverteilung für sortierten Zellen von Tg (Nkx2.5: ZsYellow) Embryonen mit 18 HPF isoliert. (CF) Repräsentative Zelleinfang Ereignisse auf IFC Platte , wo (C) ist eine leere gut, (D) enthält zwei Zellen, (E) hat eine einzelne Zelle in der Fluidik gestellten Kanal als "Kanal - Capture", und (F) eine einzelne Zelle erfasst. Lila-Boxen-Kennzeichnung für vergrößerte Ansicht Fangstellen nach innen versetzt. (G) Einzel Zelleinfang mit Hell-, ZsYellow und fusionierte Bilder. (CF) Weiß Maßstab ist 50 & mgr; m; Gelb-Skala bar beträgt 10 um. (G) Maßstabsbalken ist 10 & mgr; m. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
ild 3 "src =" / files / ftp_upload / 53877 / 53877fig3.jpg "/>
Abbildung 3. Genexpressionsanalyse von Capture einzigen Zebrabärbling Nkx2.5. ZSY positive Zellen Genexpression durch qRT-PCR in positive Kontrollen (Embryonen mit 2 Tagen post-Befruchtung, gepoolte cDNA aus einzelnen Zellen), Negativkontrolle (no-template ) und 40 Einzelzellen (Cell 01-40). Raw CT - Werte wurden farbkodiert basierend wie im Key beschrieben. EF1A = Elongationsfaktor 1a, GATA4 = GATA - Bindungsprotein 4, Nkx2.5 = NK2 homeobox 5, myl7 = Myosin leichte Kette 7, vmhc = ventrikulären Myosin schwere Kette, GAPDH = Glyceraldehyd-3 - Phosphat, und ISL1 = ISL LIM homeobox 1. Bitte hier klicken , um eine größere Version dieser Figur zu sehen.
| Cycle - Threshold (CT) | ||||||||
| EF1A | GATA4 | nkx25 | myl7 | vmhc | GAPDH | ISL1 | ||
| Embryo | 20.69 | 20.92 | 28,59 | 32,87 | 26,30 | 27,00 | 33,57 | |
| Schwimmbad | 26.4 | 22.4 | 27.7 | 27.4 | 24.5 | 29.2 | 40.0 | |
| NTC | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Typ 1 | Handy-01 | 25.1 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 |
| Handy-02 | 25.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Zell-03 | 26.3 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Handy-04 | 27.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Zell-05 | 28.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Handy-06 | 28.2 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Handy-07 | 29.3 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 400,0 | |
| Handy-08 | 30.7 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Handy-09 | 34.3 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Typ 2 | Zell-10 | 23.5 | 28.0 | 40.0 | 40.0 | 40.0 | 26.3 | 40.0 |
| Zell-11 | 23.5 | 27.2 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Zell-12 | 23.7 | 17.9 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Zell-13 | 24.1 | 32.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Zell-14 | 24.9 | 27.5 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Zell-15 | 25.9 | 28.6 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Zell-16 | 26.0 | 28.1 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Zell-17 | 27.0 | 27.7 | 40.0 | 40.0 | 40.0 | 38.5 | 40.0 | |
| Zell-18 | 27.0 | 27.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Zell-19 | 27.0 | 30.2 | 40.0 | 40.0 | 40.0 | 38.2 | 40.0 | |
| Zell-20 | 27.0 | 28.2 | 40.0 | 40.0 | 400,0 | 39.2 | 40.0 | |
| Zell-21 | 27.7 | 30.3 | 40.0 | 40.0 | 40.0 | 38.6 | 40.0 | |
| Zell-22 | 30.4 | 26.2 | 40.0 | 40.0 | 40.0 | 38.3 | 40.0 | |
| Zell-23 | 31.3 | 22.4 | 40.0 | 40.0 | 40.0 | 39.7 | 40.0 | |
| Zell-24 | 31.5 | 28.8 | 40.0 | 40.0 | 40.0 | 39.5 | 40.0 | |
| Zell-25 | 33.8 | 27.4 | 40.0 | 40.0 | 40.0 | 37.3 | 40.0 | |
| Zell-26 | 40.0 | 27.4 | 40.0 | 40.0 | 40.0 | 36.6 | 40.0 | |
| Typ 3 | Zell-27 | 24.7 | 19.9 | 28.8 | 40.0 | 40.0 | 40.0 | 40.0 |
| Zell-28 | 24.8 | 28.4 | 26.3 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Zell-29 | 25.1 | 23.5 | 27.6 | 40.0 | 40.0 | 32.5 | 40.0 | |
| Zell-30 | 26.3 | 21.4 | 27.5 | 40.0 | 40.0 | 39.9 | 40.0 | |
| Zell-31 | 26.9 | 24.8 | 28.2 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Zell-32 | 27.0 | 22.5 | 23.8 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Handy-33 | 27.8 | 27.6 | 27.7 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Zell-34 | 28.1 | 21.9 | 26.6 | 40.0 | 40.0 | 37.7 | 40.0 | |
| Handy-35 | 29.3 | 26.5 | 28.1 | 40.0 | 40.0 | 38.3 | 40.0 | |
| Typ 4 | Zell-36 | 24.8 | 20.3 | 26.0 | 27.4 | 26.6 | 40.0 | 40.0 |
| Zell-37 | 28.7 | 22.3 | 25.9 | 28.9 | 22.9 | 38.5 | 40.0 | |
| Typ 5 | Zell-38 | 25.5 | 20,0 | 29.3 | 23.0 | 19.9 | 25.9 | 40.0 |
| Handy-39 | 25.9 | 21.8 | 28.6 | 25.1 | 21.7 | 25.7 | 40.0 | |
| Zell-40 | 28.9 | 22.0 | 27.0 | 23.0 | 21.3 | 27.6 | 40.0 | |
Tabelle 1. Raw CT - Werte.
Diskussion
Das hier beschriebene Verfahren verwendet die Expression eines Fluoreszenzprotein unter der Kontrolle eines Zelltyp-spezifischen Promotor eine Population von Herzvorläuferzellen von Zebrabärbling-Embryonen in mikrofluidischen assistierten Einzelzellerfassungssystem zur Verwendung zur Anreicherung Expression einer Untergruppe von Herz Gene in einzelnen zu beurteilen Zellen. Vorausgesetzt, dass FACS Laseranregung und Emissionsfähigkeiten sind kompatibel mit dem Fluorophor (en) der Wahl kann dieses Verfahren für jeden fluoreszierenden Reporter Zeile verwendet werden. Viele Zebrabärbling Reporter Linien existieren bereits, und transgene Fische Roman Reporter tragen, können in weniger als 3 Monate generiert werden. Darüber hinaus kann dieses Arbeitsablauf angepasst werden, um cDNA für einzelne Zell-RNA-Sequenzierung aus dem gesamten Transkriptom zu erzeugen. Um dies zu tun, die Schritte 5-7 müssen geändert werden leicht für den Einsatz mit Chemikalien optimiert zur Herstellung von cDNA für Bibliotheken geeignet für RNA-Sequenzierung zu erzeugen. Allerdings ist es ratsam, Bevölkerungs Heterogenität zu validierenunter Verwendung des Verfahrens hier vor beschriebenen Hochdurchsatz-Sequenzierungsanwendungen pursing.
Es ist wichtig anzumerken, dass es einige Einschränkungen für das beschriebene Verfahren. Erstens, die Verwendung von integrierten mikrofluidischen Schaltung (IFC) Platten erfordert spezielle, teure Ausrüstung. Allerdings bieten Mikrofluidik-Chips erhebliche Vorteile gegenüber herkömmlichen 96-Well und 384-Well-Plattenformate für einzelne Testen von Zellen. Durch Zellen und Reagenzien durch Mikrometer-Skala Fahrspuren, einzelne Zellen positioniert sind, in einzelne Fangstellen fließen, wo sie direkt zu bestätigen beobachtet werden, dass sie einzelne Zellen bei guter Gesundheit sind. RNA - Extraktion und cDNA - Synthese auftreten , in situ auf dem IFC - Chip sehr geringen Mengen für jede Zelle. Integrierte Fluidik-Chips, die zum Zeitpunkt des Schreibens dieses Artikels, sind im Handel durch nur eine einzige Quelle zur Verfügung. Obwohl nicht-kommerzielle Fertigung wurde von mehreren Gruppen durchgeführt wurde, ist es außerhalb der Kapazität der meisten Laboratorien. Die Schritte 5-7 könnenmüssen für Plattformen, die von anderen Unternehmen oder im Haus Fabrikationen hergestellt werden modifiziert. Zweitens, obwohl im Handel erhältliche IFC Platten bis 96 Gene unterbringen kann, die Gene, ausgewählt durch die Verfügbarkeit von fluorogenen-markierten Gensonden validiert für die Verwendung in Zebrabärbling begrenzt. Drittens Zebrabärbling-Zellen sind typischerweise kleiner als Säugerzellen, und das kleine Größe stellt Herausforderungen Verarbeitung abzutasten. Eine kleine Veränderung in der Zelldurchmesser führt zu einer großen Änderung des Zellvolumens, erhältlich Materials verringert und die Wahrscheinlichkeit des Erfassens niedrigen exprimierenden Gene abnimmt.
Es gibt einige zusätzliche Überlegungen für dieses Protokoll. In Schritt 1 ist es wichtig, nur den Einsatz Embryonen innerhalb eines kurzen Zeitfensters gedüngt. Halten Sie die Temperatur konstant und ohne Sauerstoffgrenzbedingungen entwickeln Genaktivität synchron. Das ultimative Auslesen dieses Protokolls (relative Genexpression aus einzelnen Zellen) kann nicht die Quelle der Heterogenität diskriminierentät zwischen den einzelnen Zellen. Aus diesem Grund Interpretation der interzellulären Heterogenität beruht auf sorgfältig inszenierten Embryonen und synchrone Entwicklung. In den Schritten 2-3, gibt es einen Kompromiss zwischen Dissoziation und Lebensfähigkeit. Obwohl Schritt 4 für lebende Zellen auswählt, und Schritt 3 enthält Schritte, mehrere Filter, die Multimere helfen, Über Verdauung ausschließen das tragfähige Material für FACS reduzieren und möglicherweise sortierten Zellausbeute reduzieren. Schritte niedrige Rentabilität zu beheben Aufschlusszeit umfassen Reduzierung, die Verringerung Verreiben Intensität und die Anzahl der Embryonen im Ausgangsmaterial optimiert. FACS Anreicherung der Zellpopulation von Interesse (Schritt 4) ist wohl der wichtigste Schritt in diesem Protokoll. Strikte Sortierkriterien sind wichtig, eine einzige Zellpräparation nur zu erzeugen, die Zellen enthalten, leben das fluoreszierende Protein von Interesse exprimieren. Contaminating Zellen von anderen Populationen könnten falsche Darstellung der interzellulären Heterogenität führen. Vor allem,FACS Anreicherung einer Population von Interesse ist nicht auf die beiden Farbstrategie beschränkt in diesem Protokoll gezeigt. Weitere komplexe Strategien können feinere Zellpopulationen herzustellen und eine schnelle Möglichkeit, die Methodik im Proof of Principle-Studie berichtet, zu verlängern. In Schritt 5 können Zellen direkt beobachtet, um die Anzahl der Zellen erfasst, Zellgesundheit und die Expression der Fluoreszenz-Protein zu bestätigen. Aber je nach Zellgröße und Helligkeit, das Fluorophor von Interesse sein kann visuell nicht nachweisbar und ist keine zuverlässige Anzeige. Der erfolgreiche Abschluss der Schritte 6-8 erfordert eine sorgfältige Einhaltung der Herstellerprotokolle.
Als Beweis für das Prinzip, aus einem zuvor beschriebenen BAC transgenen Zebrabärbling Linie Nkx2.5 abgeleitet Ebenen Ausdruck einer Untergruppe von bekannten kardiale Marker in Einzel bewertet wurden: ZSY positive Zellen bei 18 Stunden nach: ZSY Herzdifferenzierungsmarker in einzelnen Nkx2.5 zu untersuchen Befruchtung (hPF). Zur Erkundung der heterogeneity von Differenzierungsmarker in gefangenen Nkx2.5 ausgedrückt: ZSY exprimierenden Zellen, wurden schon früh in der Herz - Spezifikation werden eine Reihe von Genen , einschließlich Housekeeping - Gene (EF1A, GAPDH), Transkriptionsfaktoren bekannt getestet eingeschaltet (GATA4, Nkx2.5), ein zweite Herzfeld Vorläufermarker (ISL1) und bekannte Gene später während der Herzdifferenzierung (myl7, vmhc) eingeschaltet werden. Die relative Abundanz jedes Gens wurde durch qRT-PCR gemessen durch Berechnen Zyklus Schwellenwert (CT). Die niedrigste berechnete CT-Wert betrug 17,9, und der höchste war 40, was keine Amplifikation (Tabelle 1). CT-Werte von> 35,0 wurden unter der Nachweisgrenze berücksichtigt.
Von den 46 Zellen in dem Datensatz, wurden 6 aufgrund fehlgeschlagenen Amplifikation beliebiger Gene ausgeschlossen. Die verbleibenden Zellen wurden den Unterklassen - Zellen in 5 Typen auf Basis von Expression von GATA4, Nkx2.5 myl7 und vmhc, und were von EF1A Wert innerhalb von Gruppen sortiert. Obwohl GAPDH ursprünglich als mögliche Housekeeping - Gen enthalten war, war die Expression in Zellen sehr variabel, es ungeeignet als Housekeeping - Gen macht. GAPDH Ausdruck wahrscheinlich besser repräsentiert Änderungen an den Energiestoffwechsel mit Herz-Differenzierung in diesem Zelltyp zugeordnet ist . Interessanterweise gab es eine Zelle , in der EF1A war un-nachweisbar , aber ein anderes Gen nachweisbar war (GATA1 in Zellen 26). Dies deutet darauf hin , dass während EF1A im Allgemeinen eine ausreichende Housekeeping - Gen ist zwischen erfolgreichen und nicht erfolgreichen Einzelzelle Reverse Transkriptionsreaktionen zu unterscheiden, ist es nicht für alle Anwendungen ideal sein kann. Der Mangel an EF1A Ausdruck in Zelle 26 könnte aufgrund einzelne Zelle Transkriptions Dynamik sein. Neue Single-Zellen Studien zeigen , dass Gen - Transkription grundsätzlich stochastischen ist, 26 zwischen den Phasen der schnellen und vernachlässigbare Transkriptionsaktivität abwechseln. Tseine Transkriptions Platzen Modell schlägt vor, dass die Verwendung eines Housekeeping-Gen für quantitative Vergleiche zwischen einzelnen Zellen können ganz ungeeignet sein.
In der Typ - 1 - Gruppe ist EF1A das einzige Gen nachweisbar. Diese Zelle kann umfassen entweder Nkx2.5: ZSY negativen Zellen oder Zellen mit einer sehr geringen Expression der anderen Gene von Interesse. Typ 2 - Zellen, mit Ausnahme von Zell-26, exprimiert sowohl EF1A und GATA4 ohne Korrelation in den relativen Expressionsniveaus dieser Gene. Bemerkenswert ist , Nkx2.5 Ausdruck in Typ 1 und Typ 2 Zellen können aufgrund suboptimaler FACS Stringenz, Transkriptions Platzen, Transgen leakiness oder Unterschwellenexpressionsniveaus. Typ 3 - Zellen exprimiert nachweisbare Mengen an EF1A, GATA4 und nkx-2.5. Diese Zellen wahrscheinlich Herzvorläuferzellen sind und umfassen können atriale Kardiomyozyten differenzieren. Typ 4 - Zellen exprimiert nachweisbare Mengen an EF1A, GATA4, Nkx2.5 myl7 </ em> und vmhc und Differenzierung wahrscheinlich Zellen in Kardiomyozyten. Typ 5 - Zellen exprimiert EF1A, GATA4, Nkx2.5 myl7, vmhc und GAPDH. Die Zugabe von nachweisbaren Mengen an GAPDH legt nahe , dass diese Zellen die Fähigkeit zur verbesserten glykolytischen Metabolismus in differenzierten Kardiomyozyten gefunden. Es war interessant , dass trotz Sortierung für ZsYellow positiven Zellen aus Tg. (Nkx2.5: ZsYellow), Embryonen, 21/40 Zellen Unterschwellenniveaus Nkx2.5 in unserer qRT-PCR - Analyse hatte Dies könnte aufgrund Transkriptions Platzen sein oder könnten Unterschiede in der Transkript Verarbeitung zwischen endogenen Nkx2.5 und ZsYellow reflektieren. Wichtig ist , dass das zweite Herzfeld Marker ISL1 nicht nachweisbar in Zellen oder cDNA von allen unseren Fangstellen gebündelt. In der Summe über Proben zu vergleichen, beträchtliche Heterogenität wurde auf Einzelzellebene klar, was darauf hindeutet , dass bei 18 HPF, Nkx2.5: ZSY + cells umfassen Herz Vorläufern sowie Nachkommen in verschiedenen Stadien der Differenzierung.
Offenlegungen
The authors have nothing to disclose.
Danksagungen
We thank Dr. C. Geoffrey Burns for fish stock. The authors are grateful to UNC Flow Cytometry Core Facility, UNC-CGIBD AAC core for resources enabling this project, and the ZAC facility for animal care. L.S. is supported by NIH T32 grant HL069768-13 (PI, Nobuyo Maeda). N.F. is supported by NSF Graduate Research Fellowship NSF-DGE-1144081. This study was supported by NIH P30DK034987 grant (to UNC Advanced Analytics Core), American Heart Association Scientist Development Grant 13SDG17060010 and Ellison Medical Foundation New Scholar Grant AG-NS-1064-13 (to Dr. Qian), and NIH R00 HL109079 grant (to Dr. Liu).
Materialien
| Name | Company | Catalog Number | Comments |
| Supplies | |||
| Dumont #5 forceps | Fine Science Tools | 11254-20 | For removing the chorion from embryos |
| Microcentrifuge tube 2 ml | GeneMate | C-3261-1 | |
| 40 um cell strainer | Biobasic | SP104151 | |
| 35 mm culture dish | Falcon | 351008 | |
| FACS tubes topped with 35 um cell strainer | Falcon | 352235 | |
| P1000 and tips | Rainin | 17005089 | |
| P20 and tips | Rainin | 17005091 | |
| IFC chip manufacturer's protocol | Fluidigm | 100-6117 | Version 100-6117 E1 was used in representative experiment |
| Wide bore pastuer glass pippette | VWR | 14673-010 | For transferring embryos |
| Adult wild type zebrafish | N/A | We used AB line | |
| Adult transgenic zebrafish | N/A | We used Tg(nkx2.5:ZsYellow) | |
| Name | Company | Catalog Number | Comments |
| Reagents for cell dissociation | |||
| Double distilled water | N/A | ||
| Instant Ocean Sea Salt | Instant Ocean | SS15-10 | |
| NaCl | FisherScientific | S271-3 | make stock in water and use for de-yolking buffer |
| KCl | Sigma Aldrich | P5405 | make stock in water and use for de-yolking buffer |
| NaHCO3 | Sigma Aldrich | S6014 | make stock in water and use for de-yolking buffer |
| Leibovitz's L-15 | Gibco | 21083-027 | |
| FBS | FisherScientific | 03-600-511 | Heat inactivate; any brand of FBS should be fine |
| Cell Dissociation Reagent 1 -TrypLE | Life Technologies | 12605-010 | Store at room temperature. |
| Cell Dissociation Reagent 2 - FACSmax | Genlantis | T200100 | Store -20; thaw on ice; bring to room temp before use |
| pronase (optional) | Sigma | P5147 | |
| L/D Dye - Sytox Blue | Life Technologies | S34857 | Any live/dead stain suitable for flow cytometry will work |
| Trypan Blue | Gibco | 15250-061 | |
| Name | Company | Catalog Number | Comments |
| Reagents for IFC plate use and qRT-PCR | |||
| Gene-specific probes | Probes will vary by experiment | ||
| TaqMan Probe ef1a | Life Technologies | Dr03432748_m1 | |
| TaqMan Probe gata4 | Life Technologies | Dr03443262_g1 | |
| TaqMan Probe nk2-5 | Life Technologies | Dr03074126_m1 | |
| TaqMan Probe myl7 | Life Technologies | Dr03105700_m1 | |
| TaqMan Probe vmhc | Life Technologies | Dr03431136_m1 | |
| TaqMan Probe isl1 | Life Technologies | Dr03425734_m1 | |
| TaqMan Gene Expression Master Mix | Life Technologies | 4369016 | |
| Reagents listed in IFC manufacturer's protocol | |||
| C1 Reagent Kit | Fluidigm | 100-5319 | Reagents for loading cells onto IFC plate |
| Ambion Single Cell-to-CTTM | Life Technologies | 4458237 | Reagents for reverse transcription and pre-amplification steps |
| Molecular Biology Quality Water | Corning | 46-000CM | |
| C1 IFC for PreAmp (5-10 um) | Fluidigm | 100-5757 | IFC plate for small cells |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| C1 AutoPrep machine | Fluidigm | 100-5477 | For IFC plate use |
| Hemocytometer | Sigma Aldrich | Z359629 | For counting cells and assessing cell size |
| Dissecting microscope | For removing embryos from chorion | ||
| Tissue culture microscope | For assessing single cell digestion | ||
| FACS machine | For isolating cells of interest |
Referenzen
- Speicher, M. R. Single-cell analysis: toward the clinic. Genome medicine. 5, (2013).
- Macaulay, I. C., Voet, T. Single cell genomics: advances and future perspectives. PLoS genetics. 10, e1004126 (2014).
- Marco, E., et al. Bifurcation analysis of single-cell gene expression data reveals epigenetic landscape. Proceedings of the National Academy of Sciences of the United States of America. 111, 5643-5650 (2014).
- Yin, H., Marshall, D. Microfluidics for single cell analysis. Current opinion in biotechnology. 23, 110-119 (2012).
- Garlanda, C., Dejana, E. Heterogeneity of endothelial cells. Specific markers. Arteriosclerosis, thrombosis, and vascular biology. 17, 1193-1202 (1997).
- Diaz-Cano, S. J. Tumor heterogeneity: mechanisms and bases for a reliable application of molecular marker design. International journal of molecular sciences. 13, 1951-2011 (2012).
- Guo, G., et al. Mapping cellular hierarchy by single-cell analysis of the cell surface repertoire. Cell stem cell. 13, 492-505 (2013).
- Guo, G., et al. Resolution of cell fate decisions revealed by single-cell gene expression analysis from zygote to blastocyst. Developmental cell. 18, 675-685 (2010).
- Treutlein, B., et al. Reconstructing lineage hierarchies of the distal lung epithelium using single-cell RNA-seq. Nature. 509, 371-375 (2014).
- Kimmel, C. B. Genetics and early development of zebrafish. Trends in genetics : TIG. 5, 283-288 (1989).
- Liu, J., Stainier, D. Y. Zebrafish in the study of early cardiac development. Circulation research. 110, 870-874 (2012).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Developmental dynamics : an official publication of the American Association of Anatomists. 203, 253-310 (1995).
- Zhao, S., Fung-Leung, W. P., Bittner, A., Ngo, K., Liu, X. Comparison of RNA-Seq and microarray in transcriptome profiling of activated T cells. PloS one. 9, e78644 (2014).
- McCall, M. N., McMurray, H. R., Land, H., Almudevar, A. On non-detects in qPCR data. Bioinformatics. 30, 2310-2316 (2014).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature. 3, 1101-1108 (2008).
- Lyons, I., et al. Myogenic and morphogenetic defects in the heart tubes of murine embryos lacking the homeo box gene Nkx2-5. Genes & development. 9, 1654-1666 (1995).
- Chen, J. N., Fishman, M. C. Zebrafish tinman homolog demarcates the heart field and initiates myocardial differentiation. Development. 122, 3809-3816 (1996).
- Zhou, Y., et al. Latent TGF-beta binding protein 3 identifies a second heart field in zebrafish. Nature. 474, 645-648 (2011).
- Paffett-Lugassy, N., et al. Heart field origin of great vessel precursors relies on nkx2.5-mediated vasculogenesis. Nature cell biology. 15, 1362-1369 (2013).
- McCurley, A. T., Callard, G. V. Characterization of housekeeping genes in zebrafish: male-female differences and effects of tissue type, developmental stage and chemical treatment. BMC molecular biology. 9, 102 (2008).
- Tang, R., Dodd, A., Lai, D., McNabb, W. C., Love, D. R. Validation of zebrafish (Danio rerio) reference genes for quantitative real-time RT-PCR normalization. Acta biochimica et biophysica Sinica. 39, 384-390 (2007).
- Serbedzija, G. N., Chen, J. N., Fishman, M. C. Regulation in the heart field of zebrafish. Development. 125, 1095-1101 (1998).
- de Pater, E., et al. Distinct phases of cardiomyocyte differentiation regulate growth of the zebrafish heart. Development. 136, 1633-1641 (2009).
- Hami, D., Grimes, A. C., Tsai, H. J., Kirby, M. L. Zebrafish cardiac development requires a conserved secondary heart field. Development. 138, 2389-2398 (2011).
- Yelon, D., Horne, S. A., Stainier, D. Y. Restricted expression of cardiac myosin genes reveals regulated aspects of heart tube assembly in zebrafish. Developmental biology. 214, 23-37 (1999).
- Molina, N., et al. Stimulus-induced modulation of transcriptional bursting in a single mammalian gene. Proceedings of the National Academy of Sciences of the United States of America. 110, 20563-20568 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenWeitere Artikel entdecken
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten