Method Article
Aislamiento y caracterización de células individuales a partir de embriones de pez cebra
En este artículo
Resumen
This protocol describes a method for isolating single cells from zebrafish embryos, enriching for cells of interest, capturing zebrafish cells in microfluidic based single cell multiplex systems, and assessing gene expression from single cells.
Resumen
The zebrafish (Danio rerio) is a powerful model organism to study vertebrate development. Though many aspects of zebrafish embryonic development have been described at the morphological level, little is known about the molecular basis of cellular changes that occur as the organism develops. With recent advancements in microfluidics and multiplexing technologies, it is now possible to characterize gene expression in single cells. This allows for investigation of heterogeneity between individual cells of specific cell populations to identify and classify cell subtypes, characterize intermediate states that occur during cell differentiation, and explore differential cellular responses to stimuli. This study describes a protocol to isolate viable, single cells from zebrafish embryos for high throughput multiplexing assays. This method may be rapidly applied to any zebrafish embryonic cell type with fluorescent markers. An extension of this method may also be used in combination with high throughput sequencing technologies to fully characterize the transcriptome of single cells. As proof of principle, the relative abundance of cardiac differentiation markers was assessed in isolated, single cells derived from nkx2.5 positive cardiac progenitors. By evaluation of gene expression at the single cell level and at a single time point, the data support a model in which cardiac progenitors coexist with differentiating progeny. The method and work flow described here is broadly applicable to the zebrafish research community, requiring only a labeled transgenic fish line and access to microfluidics technologies.
Introducción
La mayoría de los estudios actuales de la biología celular y molecular se basan en promedios de la población. Sin embargo, importantes eventos biológicos pueden estar enmascarados por estos análisis tradicionales basados en la población ya que las poblaciones de menor importancia pueden desempeñar un papel importante en los procesos biológicos y evolución de la enfermedad. La comprensión de la expresión de genes en poblaciones heterogéneas a nivel de una sola célula puede (y tiene) conducir a conocimientos biológicos y clínicos 1,2. De interés para los estudios de desarrollo embrionario, en una población mayor de células, las células progenitoras son a menudo poco representados, por lo que es difícil de detectar cambios sutiles en la expresión de genes que inician en última instancia, las decisiones del destino celular 3. Del mismo modo, un solo tipo de células puede tener diferentes perfiles de expresión en respuesta a la microambiente 4. Por ejemplo, las células endoteliales residentes en el mismo órgano o en diferentes órganos (por ejemplo., Aorta o riñón) presentan una heterogeneidad significativa a pesar de compartir morp comúncaracterísticas hological y funcionales 5. Además, las células cancerosas que pueblan el mismo tumor también pueden tener diversos perfiles moleculares o mutaciones en el nivel de células individuales 6.
En los sistemas modelo, transcriptómica en células individuales ha identificado con éxito nuevas poblaciones de células, que se caracteriza estados intermedios que se producen durante la diferenciación celular, y puso de manifiesto las respuestas celulares a los estímulos diferenciales 7.8.9. Estas ideas se han enmascarado en los estudios convencionales basados en la población. embriones de pez cebra son una fuente tremendamente infrautilizados del tallo, células progenitoras, y diferenciación de las células para explorar cuestiones de la heterogeneidad de células individuales y la regulación molecular de identidades celulares durante el desarrollo. Su altamente estereotipada, ex vivo el desarrollo y la facilidad de manipulación genética ellas un excelente sistema modelo para este enfoque 10,11 hacen. En concreto, una limitación importante a la interpretación de un solo gen celulardatos de correo expresión es que la identificación fiable de los nuevos estados intermedios de células durante el desarrollo requiere mucho cuidado el momento de la recogida de tejidos 9. Esto es necesario para asegurar que la heterogeneidad entre las células capturadas representa heterogeneidad dentro de un tejido en un solo punto de tiempo en lugar de la heterogeneidad en la expresión génica presentado por la diferenciación de células dependiente de la edad. En comparación con los ratones, el desarrollo del embrión de pez cebra se puede sincronizar con precisión a través de un gran número de embriones 12. Además, con tamaños grandes de embrague, embriones de pez cebra se pueden utilizar como una fuente abundante de células madre y progenitoras.
Este protocolo describe un método para aislar células de embriones de pez cebra y la captura de células individuales utilizando un chip y Autoprep sistema disponible comercialmente circuito microfluídica integrado (IFC) para el análisis de la expresión génica QRT-PCR. Este protocolo puede ser rápidamente transferible a ningún ensayos de alto rendimiento, incluyendo toda la multiplexaciónsecuenciación del transcriptoma que permite un análisis más exhaustivo de la heterogeneidad celular 13. También ofrece varias ventajas a los ensayos de expresión génica tradicionales. El protocolo de aislamiento de células individuales se obtiene una alta viabilidad después de FACS, lo que disminuye la proporción de células comprometidas que se incluyen en las aplicaciones posteriores. Mediante el uso de un CFI, células capturadas pueden ser observados directamente para evaluar las tasas de captura y evaluar la salud de células morfológicamente. Además, este protocolo es ampliamente aplicable a la comunidad de investigación pez cebra, que sólo requiere una línea de peces transgénicos etiquetados y el acceso a las tecnologías de captura de células de microfluidos.
Como prueba de principio, se aislaron células individuales derivadas de progenitores cardíacos y capturados en un chip IFC, y luego la abundancia relativa de marcadores de diferenciación cardíaco se midió mediante qRT-PCR. análisis de la expresión génica a nivel de células individuales demuestra que los progenitores cardíacos coexisten con su differeprogenie ntiating. El conocimiento que se obtiene a partir de perfiles de una sola célula de células progenitoras cardíacas puede arrojar luz sobre la heterogeneidad en los patrones de expresión génica entre células progenitoras cardiacas durante el desarrollo de vertebrados, que puede haber sido enmascarado en los análisis tradicionales basados en la población.
Protocolo
Este protocolo requiere el uso de vivo, el pez cebra adultos para producir embriones. Los embriones se cosechan para la recogida de tejidos. Es esencial para obtener la aprobación de ética apropiadas juntas de revisión para llevar a cabo este experimento.
1. obtener embriones por etapas
- El día antes del experimento, preparar sana pez cebra, adulto para la cría. Coloque un macho y una hembra en lados opuestos de una clara divisoria en un tanque de cría.
- Repita 1,1 para tantos tanques de cría como necesarios para la producción de embriones suficiente para la aplicación de aguas abajo. Obtener embriones de peces, tanto de tipo salvaje y peces transgénicos que expresan las proteínas fluorescentes en el tipo de célula de interés.
NOTA: El número de embriones necesarios para las aplicaciones posteriores en los Pasos 2-8 depende de la abundancia relativa de las células de interés en el lugar de interés tiempo. Aunque esto puede variar según el tipo de células, 200 embriones producen 2.000-5.000 células clasificadas cuando el cells de interés representan <1,0% del total de células en 24 HPF (horas después de la fecundación). - A la mañana siguiente, cambie el agua en el depósito de empanado por la transferencia de los peces a un tanque de cría fresco y eliminar el divisor. Incline el tanque en un ángulo para fomentar la cría.
- Recoger los embriones por etapas.
- Cada 15 minutos, recoger embriones mediante la transferencia de los adultos a un tanque de cría fresco y pasar los huevos que se quedan atrás a través de un colador de té.
NOTA: los embriones de pez cebra se desarrollan de forma sincrónica cuando se mantienen a densidades y temperaturas comparables. - Lavar los huevos con el huevo de agua (sales Instant Ocean 0,21 g / L en 1 l de agua doblemente destilada) y transferir a una placa de Petri. La transferencia de la placa Petri hasta una incubadora húmeda a 28,5 ° C con circulación de aire.
- Cada 15 minutos, recoger embriones mediante la transferencia de los adultos a un tanque de cría fresco y pasar los huevos que se quedan atrás a través de un colador de té.
- Dos horas después de la última recogida, tipo fertilizado, los embriones multicelulares en 10 cm placas de Petri y reducir la densidad a 50 embriones por placa. Seleccione los embriones procedentes de una sola, 15ventana de tiempo min de la recolección para la aplicación de aguas abajo. Incubar los embriones a 28,5 ° C.
NOTA: Por ejemplo, recoger los embriones a las 8:30, 08:45, 09:00, 09:15, 09:30, 09:45, 10:00 y 10:15 AM. Hacer comparaciones entre los puntos de tiempo, si el mayor número de embriones fecundados son de las garras recogidos a las 9:00, a continuación, utilizar sólo estos embriones para aplicaciones posteriores.
2. Configuración para las disociación de los organismos unicelulares
- Aproximadamente 30 minutos antes del punto de interés (18 HPF) Retire los embriones de su corion manualmente con unas pinzas finas.
- Recoger y etiquetar lo siguiente para cada condición: dos tubos de microcentrífuga de 2 ml, un colador de 40 micras de células, una placa de cultivo celular de 35 mm, y dos tubos de FACS rematados con un filtro de células de 35 micras.
- Enfriar los siguientes reactivos sobre hielo: contiene huevo Agua sales Instant Ocean 0,21 g / L en 1 l de agua doblemente destilada; De-yolking tampón que contiene NaCl 55 mM, KCl 1,8 mM, y mM NaHCO 3 1,25; y FACS tampón que contiene de Leibovitz L-15 medio suplementado con suero bovino fetal inactivado por calor al 5%.
- Llevar 1 ml por muestra de célula reactiva Disociación 1 a RT. Descongelar 1 ml de Disociación Celular reactivo 2 por muestra en hielo. Llevar a TA inmediatamente antes de su uso.
3. La disociación de los organismos unicelulares
- Usando una pipeta de vidrio de ancho diámetro interior, transferir 100-300 embriones a un tubo de 2 ml de microcentrífuga en un volumen mínimo de agua del huevo.
- La eutanasia a los embriones mediante la sustitución de agua con 1 ml de hielo-agua fría huevo y sumergiendo el tubo en hielo durante 20 min.
PRECAUCIÓN! Es fundamental para obtener la aprobación del comité de supervisión del cuidado animal institucional adecuado para este método de eutanasia (que se enfría en hielo seguido de disociación celular como la eutanasia secundaria). la eutanasia norma general requiere que los embriones <3 días después de la fertilización se blanqueada después que se enfríen, lo que no es apropiado para la obtención de células viables. - Lavar los embriones dos veces con 1 ml enfriado en hielo de agua del huevo. Para lavar, usar una amplia pipeta de vidrio para eliminar el orificio del huevo de agua y un P1000 añadir huevo agua.
- Retire la yema mediante la sustitución del agua de embriones con 1 ml de tampón deyolking y triturando 8-12 veces con una punta P1000, o hasta que la yema se disuelve y sólo los cuerpos de los embriones son visibles.
- Recoger el tejido por centrifugación a 300 xg durante 1 min. Utilizar una pipeta para eliminar suavemente el sobrenadante sin perturbar el sedimento de tejido. Vuelva a suspender en 1 ml de agua del huevo.
- Repita el paso 3.5 para un total de tres lavados, pero en el lavado final, re-suspender en 1 ml RT Cell disociación Reactivo 1.
- Incubar en la célula reactivo de disociación 1 durante 10 min a RT con los tubos en posición horizontal. Cada 2-3 minutos, se tritura suavemente con una pipeta P1000 para evitar la formación de grumos.
Nota: Maneje con cuidado las muestras durante las etapas de trituración. En algún momento un único grupo grande se formará en el tubo de una maraña de cuerpos embrionarios. Esta voluntaddispersar con la digestión adicional con la ayuda de trituración suave. NO HACER vórtice. - Recoger el tejido por centrifugación a 300 xg durante 3 min. Aspirar el sobrenadante y resuspender en 1 ml de Disociación Celular reactivo 2.
- Incubar durante 5-15 minutos a temperatura ambiente. Colocar los tubos en posición horizontal. Cada 2-3 minutos, triturar con cuidado para evitar la formación de grumos. Cada 5 minutos, evaluar el progreso digestión.
- Para evaluar el progreso de la digestión, se diluye el sobrenadante 2 l en 18 l de tampón FACS y la pipeta como una gota sobre una placa de cultivo celular.
- Colocar un cubreobjetos sobre la muestra y observar bajo un microscopio de cultivo de tejidos a 10X y 20X aumentos.
NOTA: La preparación debe aparecer como una mezcla de células individuales, pequeños grupos, grandes grupos, y de vez en cuando el cuerpo del embrión en su mayoría intacta. - evaluar visualmente la proporción de la preparación que es células individuales y pequeños grupos.
NOTA: Durante la digestión reducirá la viabilidad; bajo-digestión reducirá el rendimiento de una sola célula.
- Recoger la preparación de células por centrifugación a 300 xg durante 5 min. Desechar el sobrenadante y resuspender en 1 ml de tampón FACS frío.
- Humedecer un filtro de células de 40 m con tampón FACS. Pasar la suspensión de células a través del filtro celular 40 micras sobre una placa de cultivo celular 35 mm. Lavar el filtro de células una vez con 1 ml de tampón FACS clasificación.
- Transferir el flujo a través de un tubo de microcentrífuga de 2 ml. Recoger las células por centrifugación a 300 xg durante 5 min y volver a suspender en 100 l de tampón FACS.
- Contar el rendimiento de células usando un hemocitómetro. Diluir las muestras de 5x10 6 células por ml, o la concentración óptima recomendada para la máquina de FACS de elección. Opcional-Al contar rendimiento celular en el hemocitómetro, las células muertas de colorante de contraste con azul de tripano para confirmar la viabilidad de la preparación celular antes de la separación FACS.
NOTA: Las preparaciones que se diluyen también se llevará a cantidad excesiva de tiempo para ordenar. Las preparaciones que son demasiado concentrated tienden a agruparse en multímeros, y no son óptimas para clasificar una población pura. - Reserva un 10-20% de cada muestra para usar como controles sin teñir. Para las muestras restantes, las células muertas de la mancha mediante la adición de un / muerto (L / D) la discriminación en vivo tinte fluorescente en cada muestra. No lavar.
NOTA: Cualquier colorante fluorescente que penetra en las células con las membranas celulares comprometidas y manchas ácidos nucleicos se pueden utilizar como el colorante L / D, a condición de que los espectros de fluorescencia es compatible con la máquina de FACS de elección y de etiquetado fluoróforo células de interés.- No lave la preparación después de añadir el colorante ya que algunas células mueren durante el transporte a la purificación FACS y deben ser excluidos de la población clasificada.
- Humedecer el filtro de 35 micras de opérculo tubo de FACS con 20 l de tampón FACS. Añadir células para el colador y recoger por gravedad. Almacenar en hielo.
4. FACS Enriquecimiento
- Utilice FACS para enriquecer sola, Células vivas que expresan el marcador fluorescente.
- Establecer una puerta para distinguir las células de los escombros en un gráfico de dispersión de dispersión frontal (FSC-A) de amplitud vs amplitud de dispersión lateral (SSC-A), ambos con la escala lineal.
¡Precaución! Células de pez cebra son más pequeños que las células de ratón o humanos. Esto se refleja en una separación basal inferior entre las células y los desechos. - Desde la puerta fijado en el punto 4.1.1, establezca una puerta para enriquecer en células individuales y no incluyen multímeros utilizando un gráfico de dispersión de la altura de dispersión hacia adelante (FSC-H) y baja altura de dispersión lateral (SSC-H), tanto con la escala lineal.
NOTA: Las células con desproporcionadamente alto FSC-H y bajo SSC-H son probables multímeros y se excluyen de la clasificación. - A partir del conjunto de puerta en 4.1.2, utilizando los controles de un solo color, establecer una puerta para incluir sólo las células vivas utilizando un gráfico de dispersión de la FSA-A con la escala lineal y la amplitud del canal utilizado para la detección de manchas L / D con el escalamiento de registro. Incluir células negativas L / D.
- Dela puerta situado en 4.1.3, utilizando los controles de un solo color, establece una puerta para incluir sólo las células vivas con fluorescencia positiva utilizando un gráfico de dispersión de la FSA-A con la escala lineal y la amplitud del canal utilizado para detectar células marcador proteína fluorescente con el escalamiento de registro . Incluyen células positivas.
- Establecer los controles de compensación, si es necesario, para tener en cuenta la interferencia entre la L D mancha / y los espectros de la proteína fluorescente.
- Establecer una puerta para distinguir las células de los escombros en un gráfico de dispersión de dispersión frontal (FSC-A) de amplitud vs amplitud de dispersión lateral (SSC-A), ambos con la escala lineal.
- Juego de lógica tipo de células que se dividen en todas las puertas establecidas en el paso 4.1.
- El uso de células doblemente marcadas, verificar la compuerta de clasificación de células.
NOTA: Las células para la ordenación será células en lugar de los desechos, las células individuales en vez de multímeros, L / D células positivas y negativas de fluorescencia.
- El uso de células doblemente marcadas, verificar la compuerta de clasificación de células.
- Ordenar 2.000-4.000 células de la población de interés en 5 l de tampón FACS frío en un tubo de microcentrífuga en hielo. Para minimizar cizallamiento y la tensión en las células durante la clasificación, utilice las presiones más bajas posible que la célulaclasificador.
NOTA: La gota de 5 l de tampón FACS sirve como un amortiguador para las células que salen de la máquina de FACS. El cobro de 2.000-4.000 células directamente en tampón FACS elimina la necesidad de centrifugación antes de cargarlo en los chips de la IFC. Esta estrategia se recomienda porque la centrifugación se puede llevar a la formación de multímeros de si las células se adhieren el uno al otro en el sedimento. - Evaluar el análisis de viabilidad posterior a la especie.
- Transferencia de 1 l ordenados células a un tubo de FACS fresco con 100 l de tampón FACS y 1 de clasificación: 1000 dilución de L / D mancha. Pasar las células a través del clasificador FACS con la estrategia de compuerta en el paso 4.1. Utilice la proporción de L / D negativo a positivo para estimar la viabilidad de las células clasificadas.
5. Las células se cargan en microfluídica viruta
- El uso de un hemocitómetro, medir tanto la concentración y el tamaño de células clasificadas.
- Diluir las células ordenadas en al menos 10 l. Diluir una alícuota de 5 μ; L células con 5 l de azul de tripano. Cargar en hemocitómetro y aplicar cubreobjetos.
- Recoger las imágenes de campo claro de todas las células en 4 x 4 rejillas de hemocitómetro. Contar el número de células vivas y calcular vivo células / ml. Las células vivas no ocupan azul de tripano.
NOTA: Utilice cualquier software de análisis de imagen estándar para medir el diámetro de todas las células vivas. Se calcula la media y la desviación estándar del tamaño de la celda y seleccione una placa CFI adecuado para el rango de tamaño de célula de interés. Si el rango de tamaño de celda a caballo entre un rango de tamaño de celda placa de IFC, utilizar múltiples placas para capturar toda la gama de células de interés.
- Las celdas de carga en la placa de la CFI según las instrucciones del fabricante (ver Materiales).
- Diluir las células a 1x10 6 células / ml y llevar a cabo la optimización de la flotabilidad de acuerdo con las instrucciones del fabricante.
NOTA: optimización de la flotabilidad asegura que las células no se hunden hasta el fondo ni flotan en la parte superior de la carga well para la carga óptima en chip de la CFI. La relación de tampón a las células puede variar según el tipo de célula, pero típicamente oscila 6: tampón: 4-7: 3 de las células. - Añadir las células a la placa-CFI cebado. Carga de placa en la máquina de fluidos compatibles, y ejecutar un script de carga de las células para empujar las células a través del circuito de microfluidos y en carriles de captura.
- Diluir las células a 1x10 6 células / ml y llevar a cabo la optimización de la flotabilidad de acuerdo con las instrucciones del fabricante.
- Confirman que las células sean alojados en los sitios de captura en la placa de la CFI.
- Retire la placa de la CFI de la máquina de fluidos. Montar la placa del CFI en un microscopio equipado con un adaptador de la placa.
- Recoge instantáneas de campo claro y fluorescencia para cada sitio de captura en un aumento de 10X.
NOTA: Campo claro tiempos de captura pueden variar de 10-50 ms, dependiendo de la intensidad de la lámpara. los tiempos de captura de fluorescencia pueden oscilar entre 250-750 ms, dependiendo de la luminosidad fluoróforo. Evitar la sobreexposición de las células para prevenir el daño solar.
6. Síntesis de ADNc
- Realizar la lisis celular in situ de acuerdo con Ilas instrucciones del fabricante FC placa.
- Añadir tampones de lisis a los pocillos en la placa de la CFI. Carga de placa en la máquina de fluidos compatibles y escritura de la lisis celular de ejecución según las instrucciones del fabricante.
- Realizar la transcripción inversa de acuerdo con las instrucciones del fabricante CFI plato.
- Añadir los reactivos de transcripción inversa a la placa de la CFI. Carga de placa en la máquina de fluidos compatibles. Ejecutar script de la transcripción inversa. condiciones de los ciclos de la muestra: 1 ciclo a 25 ° C durante 10 min, 1 ciclo a 42 ° C durante 1 hora, a continuación, 1 ciclo a 85 ° C durante 5 min.
- Realizar pre-amplificación con sondas de genes específicos de acuerdo con las instrucciones del fabricante IFC placa.
NOTA: Debido a su mayor especificidad con los números de bajo número de copias, utilizar sondas-fluorogénicos marcado en lugar de sondas optimizadas para su uso con tintes intercalantes.- cebadores de la piscina y se diluye hasta final de 180 nM cada uno. Añadir cebadores agrupados a la placa de la CFI. Carga de placa en FLUIDI compatiblescs máquina. escritura de preamplificación ejecutar.
- Realizar pre-amplificación con las siguientes condiciones de ciclo: 1 ciclo a 95 ° C durante 10 min, luego 18 ciclos con desnaturalización a 95 ° C durante 15 s después de recocido y extensión a 60 ° C durante 4 min. Tienda de pre-amplificación de cDNA a 4 ° C hasta el momento de la cosecha. Cosecha de cDNA a partir de la placa de microfluidos acuerdo con las instrucciones del fabricante y almacenar a -20ºC hasta su uso.
7. Selección de ADNc a partir de células individuales para un solo objetivo QRT-PCR
- Diluir cDNA en 25 reactivo de dilución l de acuerdo con las instrucciones del fabricante.
NOTA: rendimiento final aproximado es de 28 l de cDNA pre-amplificado por célula. Reserva una alícuota de diluyente para su uso como un control sin plantilla en QRT-PCR.- Refiriéndose a campo brillante y las imágenes fluorescentes registrados en el paso 5.3.2, la puntuación de cada sitio de captura. contar y registrar manualmente el número de células. Evaluar y registrar si cada célula es la gripeorescent.
NOTA: Cada sitio individual puede contener 0, 1, o> 1 célula, donde> 1 célula incluye sitios de captura que contienen múltiples células y / o restos no identificables. Si las imágenes son suficientes calidad, un valor de píxel también puede ser asignado para cuantificar la intensidad de la señal de fluorescencia.
- Refiriéndose a campo brillante y las imágenes fluorescentes registrados en el paso 5.3.2, la puntuación de cada sitio de captura. contar y registrar manualmente el número de células. Evaluar y registrar si cada célula es la gripeorescent.
- Seleccionar muestras para el análisis de QRT-PCR.
- Seleccionar muestras de cDNA a partir de los lugares de captura identificadas en el paso 7.2 que contienen exactamente 1 célula con fluorescencia positiva. 1 piscina alícuotas de cDNA de cada muestra.
NOTA: Este es el control positivo "piscina" que se utiliza para determinar si los genes de interés se expresan en cualquiera de las celdas seleccionadas. - Seleccionar ADNc a partir de al menos un sitio de captura que contiene exactamente 0 células como un control negativo. Use diluyente de la etapa 7.1.2 como control sin molde.
- Seleccionar muestras de cDNA a partir de los lugares de captura identificadas en el paso 7.2 que contienen exactamente 1 célula con fluorescencia positiva. 1 piscina alícuotas de cDNA de cada muestra.
- Ejecutar QRT-PCR ensayo.
- Utilice sólo las sondas utilizadas para la pre-amplificación en el paso 6.4.1. Cargar todas las muestras en triplicatmi. Las condiciones de ciclación para una reacción de 10 l en una placa de 384: 1 ciclo de 50 ° C 2 min, 1 ciclo de 95 ° C durante 10 min, 40 ciclos con desnaturalización a 95 ° C 15 seg a continuación, recocido y extensión a 60 ° C durante 1 min.
Análisis de datos 8.
- Validar productos QRT-PCR por electroforesis en gel estándar. Confirmar producto es una sola banda a peso molecular esperado (varía para cada sonda).
NOTA: Si una sonda produce múltiples bandas o una sola banda a un tamaño apropiado, a continuación, la especificidad es cuestionable, y esto excluye el gen del análisis. - Examinar todas las curvas de amplificación y dar cuenta de cualquier anomalía 14. Calcular el valor medio de CT para cada combinación de muestra / gen 15.
NOTA: Los valores de CT para los controles positivos se extienden típicamente a partir de 10-30. CT valores para los controles negativos suelen oscilar entre un 35-40 (sin amplificación). valores CT 30-35 se consideran muy baja expresióny deben interpretarse con precaución 14. - informe de datos
- Si las muestras caen en el CT = 10-30, los datos pueden ser también registradas como factor de cambio en la abundancia de transcripción en relación con una muestra de control.
NOTA: doble cambio es igual a 2 ^ (- ΔΔCT) donde Ct es relativa a un gen de mantenimiento restando el CT promedio de la muestra de la limpieza de genes y ΔΔCT son las diferencias en Ct entre la muestra y un control positivo 15.
- Si las muestras caen en el CT = 10-30, los datos pueden ser también registradas como factor de cambio en la abundancia de transcripción en relación con una muestra de control.
Resultados
Como prueba de principio, la expresión génica se evaluó para explorar la dinámica de diferenciación durante el desarrollo cardíaco. En el pez cebra, surgen progenitores cardíacos de una población mesodérmico de células que migran a la anterior mesodermo de la placa lateral donde se fusionan para formar el tubo lineal corazón. Antes de la fusión, progenitores cardíacos comienzan a expresar el factor de transcripción nkx2.5 (NK2 homeobox 5), que se cree que es el primer marcador específico de células progenitoras cardíacas 16,17. Aquí, un descrito previamente BAC peces transgénicos Tg (nkx2.5: ZsYellow) 18, nkx2.5 abreviado: ZSY se utilizó para examinar marcadores de diferenciación cardíacos en células individuales en la etapa 18 somite, 18 h después de la fecundación (HPF) 12. Este fue el momento más temprano en el que la señal ZsYellow era perceptible a simple vista. Como se ha descrito anteriormente para esta línea transgénica, ZsYellow etiquetas progenitores cardíacos, como hemosll como unas pocas células extra-cardiaca que dan lugar a la arco faríngeo células endoteliales en 28 hpf 19 (Figura 1A-B, los datos no mostrados). En 18 hpf, nkx2.5: embriones ZSY se disociaron en una suspensión de células después se tiñeron con azul Sytox para excluir las células muertas. Live, ZsYellow positivo, las células negativas Sytox azules eran FACS ordenados usando un clasificador de MoFloXDP o Sony SH800Z equipado con boquilla de 100 micras (Figura 1C-F). Para evaluar la pureza de la población y después de la especie viabilidad ordenados, una alícuota de células clasificadas se tiñeron con azul y Sytox evaluó el porcentaje de eventos que cayó dentro de la puerta de orden original (Figura 1G-J).
Después de la clasificación, se introdujeron las células en un circuito integrado de microfluidos (CFI) del flujo de trabajo de chip (Figura 2 A) de acuerdo con las instrucciones del fabricante. Un hemocitómetro, combinado con azul de tripano exclusion se utilizó para medir el diámetro de la célula, la concentración, y la viabilidad (Figura 2B, y los datos no se muestra). flotabilidad celular se optimizó (6,5: 3,5: células de amortiguamiento) de acuerdo con las instrucciones del fabricante, y se cargaron las células sobre un IFC para capturar 5-10 micras de diámetro células. Para evaluar la eficiencia de la captura, la fluorescencia y / o señales de campo claro se obtuvieron imágenes en todos los lugares de captura, se utilizó una función de mosaico en FIJI para coser una sola imagen de la placa de la microfluídica. Cada sitio de captura contenía 0, 1 o> 1 en las células individuales (Figura 2C-F). Como se esperaba de FACS enriquecimiento, células capturadas expresaron ZsYellow (Figura 2G). Eficiencia de captura excedió 90% en 5 experimentos individuales utilizando nkx2.5: ZSY células clasificadas, y al menos 70% de los sitios de captura fueron ocupados por una sola célula (datos no mostrados). Las células se lisaron, el ARN aislado, ADNc sintetizado y genes diana específicos se amplificaron, todo de acuerdo con las instrucciones del fabricante.
Un subconjunto de los lugares de captura de células fueron seleccionados para el análisis de QRT-PCR como se describe en el protocolo anterior. Específicamente, el factor de elongación 1a (efl1a) 20 y gliceraldehido-3 fosfato (GAPDH) 21 se ensayaron como genes de limpieza se espera que se expresa en todas las células; Proteína de unión a GATA 4 (GATA4) 22 y NK2 homeobox 5 (nkx2.5) como marcadores tempranos progenitoras cardiacas; ISL LIM homeobox 1 caja 1 (Isl1) como marcador de campo de segundo corazón 23,24; y la cadena ligera de la miosina (myl7) y la cadena pesada de la miosina ventricular (vmhc) 25 para marcar los cardiomiocitos ventriculares. Sondas de genes específicos se validaron utilizando ADNc a partir de 48 embriones HPF (Figura 3). QRT-PCR se utilizó para evaluar la expresión génica relativa de estos genes en 45 células individuales a partir de 18 hpf embriones, un control negativo sin molde, y una población combinada containi de control positivong de cDNA de los 96 sitios de captura. Los valores en bruto CT para 40 células representativas y los controles se muestran en la Tabla 1. En particular, desde la piscina de 46 células examinadas, fueron excluidos del análisis 6 celdas porque todos los valores de CT de expresión génica superaron CT = 35,0, y se desconoce si estos altos CT Los valores son atribuibles a los verdaderos, muy bajos niveles de expresión o degradación de la muestra. Puesto que el rango de valores de CT para la limpieza de genes, ef1a, era demasiado amplia para comparar la expresión de genes entre las muestras, y muchos valores CT superaron 30, los valores de CT se visualizaron como un mapa de calor (Figura 3). La comparación entre las muestras, se observó heterogeneidad significativa en la expresión génica, las células y las células fueron como Tipo 1-5 basado en el patrón de expresión (Figura 3) clasificado.

Figura 1. Aislamiento de células individuales de pez cebra nkx2.5: zsy células positivas en 18 HPF imágenes todo el montaje de representante Tg (nkx2.5: ZsYellow). embrión en 18 HPF con (A) ZsYellow fluorescencia solo o (B) se fusionó con la imagen de campo claro. Gating estrategia (CF) Representante FACS para enriquecer en células positivas ZsYellow y (GJ) análisis post-tipo con (C, G) FSC / compuerta tamaño SSC, (D, H) la discriminación doblete, (E, I) en vivo gating / Muerto y (F, J) ordenados población. La barra de escala es de 100 micras. Por favor, haga clic aquí para ver una versión más grande de esta figura.
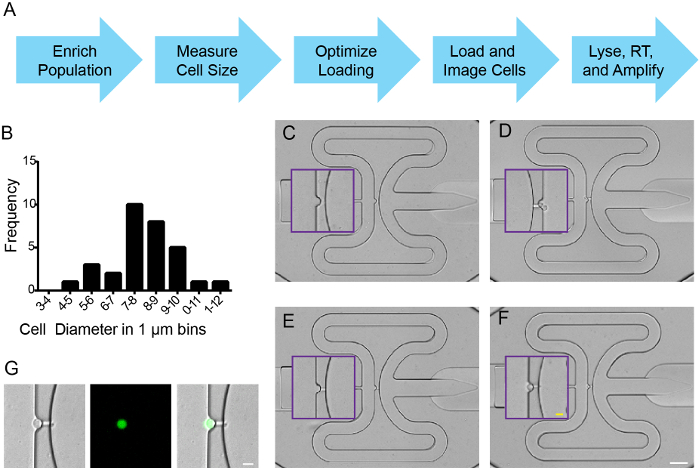
Figura 2. Captura de células individuales de nkx2.5 pez cebra: zsy ong> células positivas. (A) El flujo de trabajo. (B) la distribución del tamaño de la célula de células clasificadas de Tg (nkx2.5: ZsYellow) embriones aislados en 18 HPF. (CF) eventos de captura de células representativas en la placa de la CFI, donde (C) es un pozo vacío, (D) contiene dos células, (e) tiene una sola célula presentado en el canal de fluidos como una "captura de canal", y (F) es una sola célula capturada. cajas de color púrpura marcan Hundir en la vista ampliada de los lugares de captura. (G) la captura de células individual con campo claro, ZsYellow y se fusionó imágenes. (CF) Blanca barra de escala es de 50 micras; barra amarilla escala es de 10 micras. (G) La barra de escala es de 10 micras. Haga clic aquí para ver una versión más grande de esta figura.
igura 3 "src =" / files / ftp_upload / 53877 / 53877fig3.jpg "/>
Figura 3. Análisis de la expresión génica de la captura del pez cebra sola nkx2.5: zsy células positivas. La expresión de genes de QRT-PCR en los controles positivos (embriones a los 2 días post-fertilización, agrupados ADNc a partir de células individuales), control negativo (sin molde ) y 40 células individuales (Cell 01 a 40). Los valores en bruto TC fueron codificados por colores basan tal como se describe en la clave. Ef1a = factor de elongación 1a, GATA4 = proteína de unión a GATA 4, nkx2.5 = NK2 homeobox 5, la cadena ligera de la miosina myl7 = 7, vmhc = ventricular miosina de cadena pesada, GAPDH = gliceraldehido-3 fosfato, y Isl1 = ISL LIM homeobox 1. Haga clic aquí para ver una versión más grande de esta figura.
| Ciclo umbral (CT) | ||||||||
| ef1a | GATA4 | nkx25 | myl7 | vmhc | GAPDH | Isl1 | ||
| Embrión | 20.69 | 20.92 | 28.59 | 32.87 | 26.30 | 27.00 | 33.57 | |
| Piscina | 26.4 | 22.4 | 27.7 | 27.4 | 24.5 | 29.2 | 40.0 | |
| NTC | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Tipo 1 | Cell-01 | 25.1 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 |
| Cell-02 | 25.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-03 | 26.3 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-04 | 27.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-05 | 28.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-06 | 28.2 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-07 | 29.3 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 400.0 | |
| Cell-08 | 30.7 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-09 | 34.3 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Tipo 2 | Cell-10 | 23.5 | 28.0 | 40.0 | 40.0 | 40.0 | 26.3 | 40.0 |
| Cell-11 | 23.5 | 27.2 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-12 | 23.7 | 17.9 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-13 | 24.1 | 32.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-14 | 24.9 | 27.5 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-15 | 25.9 | 28.6 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-16 | 26.0 | 28.1 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-17 | 27.0 | 27.7 | 40.0 | 40.0 | 40.0 | 38.5 | 40.0 | |
| Cell-18 | 27.0 | 27.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-19 | 27.0 | 30.2 | 40.0 | 40.0 | 40.0 | 38.2 | 40.0 | |
| Cell-20 | 27.0 | 28.2 | 40.0 | 40.0 | 400.0 | 39.2 | 40.0 | |
| Cell-21 | 27.7 | 30.3 | 40.0 | 40.0 | 40.0 | 38.6 | 40.0 | |
| Cell-22 | 30.4 | 26.2 | 40.0 | 40.0 | 40.0 | 38.3 | 40.0 | |
| Cell-23 | 31.3 | 22.4 | 40.0 | 40.0 | 40.0 | 39.7 | 40.0 | |
| Cell-24 | 31.5 | 28.8 | 40.0 | 40.0 | 40.0 | 39.5 | 40.0 | |
| Cell-25 | 33.8 | 27.4 | 40.0 | 40.0 | 40.0 | 37.3 | 40.0 | |
| Cell-26 | 40.0 | 27.4 | 40.0 | 40.0 | 40.0 | 36.6 | 40.0 | |
| tipo 3 | Cell-27 | 24.7 | 19.9 | 28.8 | 40.0 | 40.0 | 40.0 | 40.0 |
| Cell-28 | 24.8 | 28.4 | 26.3 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-29 | 25.1 | 23.5 | 27.6 | 40.0 | 40.0 | 32.5 | 40.0 | |
| Cell-30 | 26.3 | 21.4 | 27.5 | 40.0 | 40.0 | 39.9 | 40.0 | |
| Cell-31 | 26.9 | 24.8 | 28.2 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-32 | 27.0 | 22.5 | 23.8 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-33 | 27.8 | 27.6 | 27.7 | 40.0 | 40.0 | 40.0 | 40.0 | |
| Cell-34 | 28.1 | 21.9 | 26.6 | 40.0 | 40.0 | 37.7 | 40.0 | |
| Cell-35 | 29.3 | 26.5 | 28.1 | 40.0 | 40.0 | 38.3 | 40.0 | |
| tipo 4 | Cell-36 | 24.8 | 20.3 | 26.0 | 27.4 | 26.6 | 40.0 | 40.0 |
| Cell-37 | 28.7 | 22.3 | 25.9 | 28.9 | 22.9 | 38.5 | 40.0 | |
| tipo 5 | Cell-38 | 25.5 | 20.0 | 29.3 | 23.0 | 19.9 | 25.9 | 40.0 |
| Cell-39 | 25.9 | 21.8 | 28.6 | 25.1 | 21.7 | 25.7 | 40.0 | |
| Cell-40 | 28.9 | 22.0 | 27.0 | 23.0 | 21.3 | 27.6 | 40.0 | |
Tabla 1. Valores de Raw CT.
Discusión
El método descrito en este documento utiliza la expresión de una proteína fluorescente bajo el control de un promotor específico de tipo celular para enriquecer una población de células progenitoras cardíacas a partir de embriones de pez cebra para su uso en sistema de captura única célula asistida de microfluidos para evaluar la expresión de un subconjunto de genes cardiacos en solo Células. A condición de que las capacidades de FACS de excitación láser y emisión son compatibles con el fluoróforo (s) de elección, este método puede ser utilizado para cualquier línea de indicador fluorescente. Muchos reportero líneas de pez cebra ya existen, y los peces transgénicos llevar a nuevos reporteros pueden ser generados en tan poco como 3 meses. Además, este flujo de trabajo puede estar adaptado para producir ADNc a partir de todo el transcriptoma para la secuenciación sola célula-RNA. Para ello, los pasos 5-7 tendrá que ser modificado ligeramente para su uso con las químicas optimizadas para la preparación de ADNc para la generación de bibliotecas adecuadas para la secuenciación de ARN. Sin embargo, es aconsejable para validar heterogeneidad de la poblaciónusando el método descrito aquí antes de fruncir aplicaciones de secuenciación de alto rendimiento.
Es importante tener en cuenta que hay algunas limitaciones en el método descrito. En primer lugar, el uso de placas de circuitos integrados de microfluidos (CFI) requiere un equipo especializado, costoso. Sin embargo, los chips de microfluidos ofrecen ventajas sustanciales sobre tradicional de 96 pocillos y formatos de placas de 384 pocillos para el ensayo de células individuales. Por fluye a través de las células y los reactivos carriles escala micrométrica, las células individuales están colocados en los lugares de captura individuales donde pueden ser observados directamente para confirmar que son células individuales en buen estado de salud. La extracción de RNA y la síntesis de cDNA se producen in situ en el chip IFC usando volúmenes muy bajos para cada celda. los chips integrados de fluidos, en el momento de escribir estas líneas, están disponibles comercialmente a través de una sola fuente. Aunque la fabricación no comercial se ha realizado por varios grupos, está fuera de la capacidad de la mayoría de los laboratorios. Los pasos 5-7 de mayonecesitar ser modificado para plataformas producidos por otras empresas o de la propia fabricaciones. En segundo lugar, aunque las placas IFC disponibles en el mercado tienen capacidad para 96 genes, los genes seleccionados se ven limitados por la disponibilidad de sondas de genes-fluorogénicos marcado validados para su uso en el pez cebra. En tercer lugar, las células de pez cebra son más pequeños que las células de mamífero, y este pequeño tamaño presenta desafíos procesamiento de la muestra. Un pequeño cambio en el diámetro celular se traduce en un gran cambio en volumen de la celda, la reducción de material disponible y la disminución de la probabilidad de detectar la expresión de genes bajo.
Hay varias consideraciones adicionales para este protocolo. En el paso 1, es importante sólo los embriones fertilizados de uso dentro de una ventana de tiempo corto. la temperatura se mantiene constante y sin limitar condiciones de oxígeno, embriones de pez cebra se desarrollan de forma sincrónica. La lectura final de este protocolo (la expresión génica relativa de las células individuales) no puede discriminar el origen de heterogeneidad entre las células individuales. Por esta razón, la interpretación de la heterogeneidad intercelular se basa en embriones cuidadosamente por etapas y el desarrollo sincrónico. En los pasos 2-3, hay una compensación entre la disociación y la viabilidad. Aunque el paso 4 elige células viables, y el paso 3 contiene varias etapas de filtrado que ayudan a excluir multímeros, el exceso de la digestión se reducirá el material viable disponible para FACS y reducir potencialmente el rendimiento de células ordenada. Pasos para solucionar los problemas de baja viabilidad incluye la reducción de tiempo de digestión, reducir la intensidad de trituración, y optimizando el número de embriones en el material de partida. FACS enriquecimiento de la población celular de interés (paso 4) es posiblemente el paso más crítico en este protocolo. criterios de clasificación estrictas son esenciales para generar una preparación de células única que contiene sólo viven las células que expresan la proteína fluorescente de interés. Contaminación de las células de otras poblaciones podría conducir a la falsa representación de la heterogeneidad intercelular. Notablemente,FACS enriquecimiento de una población de interés no se limita a la estrategia de dos colores que se muestra en este protocolo. Más estrategias complejas pueden producir poblaciones de células más refinados y son una manera rápida para extender la metodología se informó en el estudio de prueba de principio. En la etapa 5, las células pueden ser observados directamente para confirmar el número de células capturadas, la salud celular, y la expresión de la proteína de fluorescencia. Pero, en función de tamaño de celda y el brillo, el fluoróforo de interés puede no ser visualmente detectable y no es una lectura fiable. La conclusión con éxito de los pasos 6-8 requiere una cuidadosa adhesión a los protocolos del fabricante.
Como prueba de principio, los niveles de expresión de un subconjunto de los marcadores cardíacos conocidos se evaluaron en un solo derivado de un transgénico nkx2.5 línea de pez cebra BAC anteriormente descrito: zsy para examinar marcadores de diferenciación cardíacos en nkx2.5 individuo: zsy células positivas en el poste 18 hr la fecundación (HPF). Para explorar la heterogeneidad de marcadores de diferenciación expresados en nkx2.5 capturado: células que expresan zsy, un conjunto de genes se analizaron los genes de limpieza (incluyendo ef1a, GAPDH), factores de transcripción conocidos que estar encendido temprano en la especificación cardiaca (GATA4, nkx2.5), una segundo campo cardíaco marcador progenitor (Isl1), y los genes que se sabe que pueden activar más tarde durante la diferenciación cardiaca (myl7, vmhc). La abundancia relativa de cada gen se midió mediante qRT-PCR mediante el cálculo de ciclo umbral (CT). El valor CT calculada más baja fue de 17,9 y la más alta fue de 40, lo que supone ninguna amplificación (Tabla 1). CT valores de> 35,0 se consideraron debajo de la detección.
De las 46 células en el conjunto de datos, 6 se excluyeron debido a la amplificación fallida de cualquier genes. Las células restantes se células sub-clasifican en 5 tipos basados en la expresión de GATA4, nkx2.5, myl7, y vmhc, y were ordenados por valor ef1a dentro de los grupos. Aunque GAPDH se incluyeron originalmente como una posible limpieza de genes, expresión era altamente variable a través de las células, lo que es inadecuado como un gen de mantenimiento. Expresión de GAPDH probablemente representa mejor los cambios en el metabolismo de energía asociados con la diferenciación cardiaca en este tipo de células. Curiosamente, había una celda en la que ef1a era ONU-detectable pero otro gen fue detectable (GATA1 en las células 26). Esto sugiere que, si bien ef1a es generalmente un gen de mantenimiento adecuada para diferenciar entre las reacciones de transcripción inversa de células individuales con y sin éxito, puede que no sea ideal para todas las aplicaciones. La falta de expresión ef1a en la celda 26 podría ser debido a la dinámica de la transcripción sola célula. Recientes estudios demuestran que las células individuales de la transcripción de genes es fundamentalmente estocástico, alternando entre las fases de la rápida e insignificante actividad transcripcional 26. Tsu modelo de ruptura de la transcripción sugiere que el uso de un gen de mantenimiento para las comparaciones cuantitativas entre las células individuales puede ser totalmente inadecuado.
En el grupo de Tipo 1, ef1a es el único gen detectable. Estos celular puede comprender ya sea nkx2.5: ZSY células negativas o células con muy baja expresión de los otros genes de interés. Tipo 2 células, con la excepción de Cell-26, expresado tanto ef1a y GATA4 sin correlación en los niveles relativos de expresión de estos genes. En particular, la expresión en células nkx2.5 Tipo 1 y Tipo 2 puede ser debido a la rigurosidad subóptima FACS, estallido de la transcripción, permeabilidad transgén, o los niveles de expresión por debajo del umbral. Tipo 3 células expresaron niveles detectables de ef1a, GATA4, y NKX-2.5. Estas células son células progenitoras cardíacas probables y pueden incluir la diferenciación de los miocitos auriculares. Tipo 4 células expresaron niveles detectables de ef1a, GATA4, nkx2.5, myl7 </ em> y vmhc y son células que puedan diferenciarse en cardiomiocitos ventriculares. 5 células de tipo expresaron ef1a, GATA4, nkx2.5, myl7, vmhc, y GAPDH. La adición de niveles detectables de GAPDH sugiere que estas células tienen la capacidad de metabolismo glicolítico mejorada encontrado en cardiomiocitos diferenciados. Era interesante que, a pesar de la clasificación de células positivas ZsYellow de Tg. (Nkx2.5: ZsYellow), embriones, células habían 21/40 niveles por debajo del umbral de nkx2.5 en nuestro análisis qRT-PCR Esto podría ser debido al estallido de la transcripción o podría reflejar diferencias en el procesamiento de transcripción entre nkx2.5 endógeno y ZsYellow. Es importante destacar que el segundo marcador Isl1 campo del corazón era indetectable en cualquier célula o ADNc agrupados de todos nuestros sitios de captura. En suma, la comparación entre las muestras, heterogeneidad significativa fue evidente a nivel de células individuales, lo que sugiere que a los 18 HPF, nkx2.5: zsy + cells comprenden progenitores cardíacas, así como la progenie en diferentes etapas de la diferenciación.
Divulgaciones
The authors have nothing to disclose.
Agradecimientos
We thank Dr. C. Geoffrey Burns for fish stock. The authors are grateful to UNC Flow Cytometry Core Facility, UNC-CGIBD AAC core for resources enabling this project, and the ZAC facility for animal care. L.S. is supported by NIH T32 grant HL069768-13 (PI, Nobuyo Maeda). N.F. is supported by NSF Graduate Research Fellowship NSF-DGE-1144081. This study was supported by NIH P30DK034987 grant (to UNC Advanced Analytics Core), American Heart Association Scientist Development Grant 13SDG17060010 and Ellison Medical Foundation New Scholar Grant AG-NS-1064-13 (to Dr. Qian), and NIH R00 HL109079 grant (to Dr. Liu).
Materiales
| Name | Company | Catalog Number | Comments |
| Supplies | |||
| Dumont #5 forceps | Fine Science Tools | 11254-20 | For removing the chorion from embryos |
| Microcentrifuge tube 2 ml | GeneMate | C-3261-1 | |
| 40 um cell strainer | Biobasic | SP104151 | |
| 35 mm culture dish | Falcon | 351008 | |
| FACS tubes topped with 35 um cell strainer | Falcon | 352235 | |
| P1000 and tips | Rainin | 17005089 | |
| P20 and tips | Rainin | 17005091 | |
| IFC chip manufacturer's protocol | Fluidigm | 100-6117 | Version 100-6117 E1 was used in representative experiment |
| Wide bore pastuer glass pippette | VWR | 14673-010 | For transferring embryos |
| Adult wild type zebrafish | N/A | We used AB line | |
| Adult transgenic zebrafish | N/A | We used Tg(nkx2.5:ZsYellow) | |
| Name | Company | Catalog Number | Comments |
| Reagents for cell dissociation | |||
| Double distilled water | N/A | ||
| Instant Ocean Sea Salt | Instant Ocean | SS15-10 | |
| NaCl | FisherScientific | S271-3 | make stock in water and use for de-yolking buffer |
| KCl | Sigma Aldrich | P5405 | make stock in water and use for de-yolking buffer |
| NaHCO3 | Sigma Aldrich | S6014 | make stock in water and use for de-yolking buffer |
| Leibovitz's L-15 | Gibco | 21083-027 | |
| FBS | FisherScientific | 03-600-511 | Heat inactivate; any brand of FBS should be fine |
| Cell Dissociation Reagent 1 -TrypLE | Life Technologies | 12605-010 | Store at room temperature. |
| Cell Dissociation Reagent 2 - FACSmax | Genlantis | T200100 | Store -20; thaw on ice; bring to room temp before use |
| pronase (optional) | Sigma | P5147 | |
| L/D Dye - Sytox Blue | Life Technologies | S34857 | Any live/dead stain suitable for flow cytometry will work |
| Trypan Blue | Gibco | 15250-061 | |
| Name | Company | Catalog Number | Comments |
| Reagents for IFC plate use and qRT-PCR | |||
| Gene-specific probes | Probes will vary by experiment | ||
| TaqMan Probe ef1a | Life Technologies | Dr03432748_m1 | |
| TaqMan Probe gata4 | Life Technologies | Dr03443262_g1 | |
| TaqMan Probe nk2-5 | Life Technologies | Dr03074126_m1 | |
| TaqMan Probe myl7 | Life Technologies | Dr03105700_m1 | |
| TaqMan Probe vmhc | Life Technologies | Dr03431136_m1 | |
| TaqMan Probe isl1 | Life Technologies | Dr03425734_m1 | |
| TaqMan Gene Expression Master Mix | Life Technologies | 4369016 | |
| Reagents listed in IFC manufacturer's protocol | |||
| C1 Reagent Kit | Fluidigm | 100-5319 | Reagents for loading cells onto IFC plate |
| Ambion Single Cell-to-CTTM | Life Technologies | 4458237 | Reagents for reverse transcription and pre-amplification steps |
| Molecular Biology Quality Water | Corning | 46-000CM | |
| C1 IFC for PreAmp (5-10 um) | Fluidigm | 100-5757 | IFC plate for small cells |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| C1 AutoPrep machine | Fluidigm | 100-5477 | For IFC plate use |
| Hemocytometer | Sigma Aldrich | Z359629 | For counting cells and assessing cell size |
| Dissecting microscope | For removing embryos from chorion | ||
| Tissue culture microscope | For assessing single cell digestion | ||
| FACS machine | For isolating cells of interest |
Referencias
- Speicher, M. R. Single-cell analysis: toward the clinic. Genome medicine. 5, (2013).
- Macaulay, I. C., Voet, T. Single cell genomics: advances and future perspectives. PLoS genetics. 10, e1004126 (2014).
- Marco, E., et al. Bifurcation analysis of single-cell gene expression data reveals epigenetic landscape. Proceedings of the National Academy of Sciences of the United States of America. 111, 5643-5650 (2014).
- Yin, H., Marshall, D. Microfluidics for single cell analysis. Current opinion in biotechnology. 23, 110-119 (2012).
- Garlanda, C., Dejana, E. Heterogeneity of endothelial cells. Specific markers. Arteriosclerosis, thrombosis, and vascular biology. 17, 1193-1202 (1997).
- Diaz-Cano, S. J. Tumor heterogeneity: mechanisms and bases for a reliable application of molecular marker design. International journal of molecular sciences. 13, 1951-2011 (2012).
- Guo, G., et al. Mapping cellular hierarchy by single-cell analysis of the cell surface repertoire. Cell stem cell. 13, 492-505 (2013).
- Guo, G., et al. Resolution of cell fate decisions revealed by single-cell gene expression analysis from zygote to blastocyst. Developmental cell. 18, 675-685 (2010).
- Treutlein, B., et al. Reconstructing lineage hierarchies of the distal lung epithelium using single-cell RNA-seq. Nature. 509, 371-375 (2014).
- Kimmel, C. B. Genetics and early development of zebrafish. Trends in genetics : TIG. 5, 283-288 (1989).
- Liu, J., Stainier, D. Y. Zebrafish in the study of early cardiac development. Circulation research. 110, 870-874 (2012).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Developmental dynamics : an official publication of the American Association of Anatomists. 203, 253-310 (1995).
- Zhao, S., Fung-Leung, W. P., Bittner, A., Ngo, K., Liu, X. Comparison of RNA-Seq and microarray in transcriptome profiling of activated T cells. PloS one. 9, e78644 (2014).
- McCall, M. N., McMurray, H. R., Land, H., Almudevar, A. On non-detects in qPCR data. Bioinformatics. 30, 2310-2316 (2014).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature. 3, 1101-1108 (2008).
- Lyons, I., et al. Myogenic and morphogenetic defects in the heart tubes of murine embryos lacking the homeo box gene Nkx2-5. Genes & development. 9, 1654-1666 (1995).
- Chen, J. N., Fishman, M. C. Zebrafish tinman homolog demarcates the heart field and initiates myocardial differentiation. Development. 122, 3809-3816 (1996).
- Zhou, Y., et al. Latent TGF-beta binding protein 3 identifies a second heart field in zebrafish. Nature. 474, 645-648 (2011).
- Paffett-Lugassy, N., et al. Heart field origin of great vessel precursors relies on nkx2.5-mediated vasculogenesis. Nature cell biology. 15, 1362-1369 (2013).
- McCurley, A. T., Callard, G. V. Characterization of housekeeping genes in zebrafish: male-female differences and effects of tissue type, developmental stage and chemical treatment. BMC molecular biology. 9, 102 (2008).
- Tang, R., Dodd, A., Lai, D., McNabb, W. C., Love, D. R. Validation of zebrafish (Danio rerio) reference genes for quantitative real-time RT-PCR normalization. Acta biochimica et biophysica Sinica. 39, 384-390 (2007).
- Serbedzija, G. N., Chen, J. N., Fishman, M. C. Regulation in the heart field of zebrafish. Development. 125, 1095-1101 (1998).
- de Pater, E., et al. Distinct phases of cardiomyocyte differentiation regulate growth of the zebrafish heart. Development. 136, 1633-1641 (2009).
- Hami, D., Grimes, A. C., Tsai, H. J., Kirby, M. L. Zebrafish cardiac development requires a conserved secondary heart field. Development. 138, 2389-2398 (2011).
- Yelon, D., Horne, S. A., Stainier, D. Y. Restricted expression of cardiac myosin genes reveals regulated aspects of heart tube assembly in zebrafish. Developmental biology. 214, 23-37 (1999).
- Molina, N., et al. Stimulus-induced modulation of transcriptional bursting in a single mammalian gene. Proceedings of the National Academy of Sciences of the United States of America. 110, 20563-20568 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados