Method Article
ゼブラフィッシュ胚からの単一細胞の単離とキャラクタリゼーション
要約
This protocol describes a method for isolating single cells from zebrafish embryos, enriching for cells of interest, capturing zebrafish cells in microfluidic based single cell multiplex systems, and assessing gene expression from single cells.
要約
The zebrafish (Danio rerio) is a powerful model organism to study vertebrate development. Though many aspects of zebrafish embryonic development have been described at the morphological level, little is known about the molecular basis of cellular changes that occur as the organism develops. With recent advancements in microfluidics and multiplexing technologies, it is now possible to characterize gene expression in single cells. This allows for investigation of heterogeneity between individual cells of specific cell populations to identify and classify cell subtypes, characterize intermediate states that occur during cell differentiation, and explore differential cellular responses to stimuli. This study describes a protocol to isolate viable, single cells from zebrafish embryos for high throughput multiplexing assays. This method may be rapidly applied to any zebrafish embryonic cell type with fluorescent markers. An extension of this method may also be used in combination with high throughput sequencing technologies to fully characterize the transcriptome of single cells. As proof of principle, the relative abundance of cardiac differentiation markers was assessed in isolated, single cells derived from nkx2.5 positive cardiac progenitors. By evaluation of gene expression at the single cell level and at a single time point, the data support a model in which cardiac progenitors coexist with differentiating progeny. The method and work flow described here is broadly applicable to the zebrafish research community, requiring only a labeled transgenic fish line and access to microfluidics technologies.
概要
細胞及び分子生物学の最新の研究は、母集団平均に基づいています。マイナー集団は生物学的プロセスおよび疾患の結果で主要な役割を再生することができますので、重要な生物学的事象は、これらの伝統的な集団ベースの分析によってマスクすることができます。関連する生物学的および臨床的洞察1,2につながる(たと)単一細胞レベルでの不均一な集団における遺伝子発現をすることができます理解します。胚発生の研究に懸念されるのは、細胞の大きな集団において、前駆細胞は、それが困難な、最終的に細胞の運命の決定3を開始し 、遺伝子発現の微妙な変化を検出すること、しばしば過小評価されています。同様に、単一の細胞型は、微小環境4に対応して異なる発現プロファイルを有することができます。たとえば、同じ器官または異なる器官内に常駐内皮細胞( 例えば 、大動脈または腎臓)は、共通のmorpを共有するにもかかわらず、有意な異質性を示しますhologicalおよび機能的特徴5。また、同じ腫瘍を移植癌細胞は、単一細胞レベルでの6分子プロフィールまたは突然変異を変化させることができます。
モデル系では、単一細胞におけるトランスクリプトが正常に、新しい細胞集団を同定し、細胞分化の間に生じる中間状態を特徴づけ、そして7,8,9を刺激するために、差動細胞応答を明らかにしました。このような洞察は、従来の集団ベースの研究でマスクされていたであろう。ゼブラフィッシュの胚は、単一の細胞の不均一や開発中の細胞のアイデンティティの分子調節の質問を探索するための幹細胞、前駆細胞、および分化する細胞の途方もなく過小利用源です。彼らの非常にステレオタイプ、ex vivoでの開発や遺伝子操作の容易さは、それらのこのアプローチ10,11のための優れたモデル系にします。具体的には、単一のセル世代の解釈に大きな制限Eの発現データは、開発中の新規な中間体セル状態の信頼性の識別は、組織収集9の非常に慎重にタイミングを必要とすることです。これは、捕捉された細胞との間の不均一性は、年齢依存性細胞分化によって提示される遺伝子発現における単一の時点で組織内の不均一ではなく、不均一性を表していることを保証する必要があります。マウスと比較して、ゼブラフィッシュ胚発生を正確に胚12の多数にわたって同期させることができます。さらに、大規模なクラッチサイズで、ゼブラフィッシュの胚は、幹細胞および前駆細胞の豊富な供給源として使用することができます。
このプロトコルは、ゼブラフィッシュ胚からの細胞を単離し、市販の集積マイクロ流体回路のqRT-PCR遺伝子発現解析のために(IFC)チップとオートプレップシステムを用いて単一細胞を捕捉する方法を記載します。このプロトコルは、全体を含む任意の高スループット多重アッセイに急速に譲渡することができます細胞異質13のより包括的な分析を可能にするトランスクリプトーム配列決定。また、伝統的な遺伝子発現アッセイにいくつかの利点を提供しています。単一細胞単離プロトコールは、下流アプリケーションに含まれている妥協細胞の割合を減少さFACS、後に高い生存率をもたらします。 IFCを使用することにより、捕捉された細胞を直接捕捉率を評価し、形態学的細胞の健康を評価するために観察することができます。また、このプロトコルは、マイクロ流体細胞捕捉技術にのみラベルされたトランスジェニック魚のラインとのアクセスを必要とする、ゼブラフィッシュ研究コミュニティに広く適用可能です。
原理の証明として、心臓前駆細胞に由来する単一の細胞を単離し、IFCチップ上に捕捉し、次いで心臓分化マーカーの相対的存在量は、定量RT-PCRにより測定しました。単一細胞レベルでの遺伝子発現解析は、心臓前駆体が自分のdiffereと共存することを実証しますntiating子孫。心臓前駆体の単一細胞プロファイリングから得られた知見は、伝統的な集団ベースの解析でマスクされている可能性があり、脊椎動物の発生時に心臓前駆細胞間の遺伝子発現パターンの不均一性、に光を当てることがあります。
プロトコル
このプロトコルは、胚を生成するために、ライブ、大人のゼブラフィッシュを使用する必要があります。胚は組織収集のために回収されています。この実験を行うために、適切な倫理審査委員会の承認を得ることが不可欠です。
1.段階的な胚を入手
- 実験の前日には、繁殖のための健康、大人のゼブラフィッシュを準備します。 1雄と繁殖タンク内の明確な分周器の反対側に一人の女性を配置します。
- 下流側のアプリケーションのための十分な胚の生産のために必要な数の繁殖タンクのための1.1を繰り返します。野生型の魚と目的の細胞型に蛍光タンパク質を発現するトランスジェニック魚の両方から胚を入手します。
注:ステップ2-8の下流のアプリケーションに必要な胚の数は、関心のある時点で目的の細胞の相対的な量に依存します。これは、細胞の種類によって異なる場合がありますが、200胚は2,000〜5,000ソートされた細胞を生成するとき、C関心のellsは<24 HPF(時間受精後)における全細胞の1.0%を表します。 - 翌朝には、新鮮な繁殖タンクに魚を転送することにより、パン粉タンク内の水を変更し、仕切りを取り外します。繁殖を奨励する角度でタンクを傾けます。
- 段階的な胚を収集します。
- 15分ごとに、新鮮な繁殖タンクに大人を転送し、茶こしを通して残された卵を渡すことによって、胚を集めます。
注:同等の密度と温度に維持するときゼブラフィッシュの胚は、同期の開発します。 - (1 L二重蒸留水に0.21グラム/ Lインスタントオーシャン塩)卵水と卵をすすぎ、ペトリ皿に移します。空気循環と28.5℃の高温多湿インキュベーターにシャーレを転送します。
- 15分ごとに、新鮮な繁殖タンクに大人を転送し、茶こしを通して残された卵を渡すことによって、胚を集めます。
- 二時間最後の収集後、ソート10センチメートルペトリ皿に、多細胞胚を受精し、皿当たり50の胚に密度を低減。シングル、15から胚を選択下流側のアプリケーションのためのコレクションの分の時間ウィンドウ。 28.5℃で胚を培養します。
注:たとえば、午前8時30分、8時45、午前9時00分、午前9時15分、午前9時30分、9時45分、午前10時と10時15分AMに胚を収集します。受精胚の最大数は9時で収集クラッチからのものである場合、時間ポイント間で比較すると、その後、下流のアプリケーションのためにのみこれらの胚を使用しています。
2.単一細胞解離のための設定します
- 前関心(18 HPF)の時点まで約30分、微細な鉗子を使用して手動で自分の絨毛膜から胚を削除します。
- 収集し、各条件について、次のラベル:2 2ミリリットルのマイクロチューブ、140μmのセルストレーナー、1 35ミリメートルの細胞培養皿、および35μmのセルストレーナーをトッピング2 FACSチューブを。
- 氷上で以下の試薬を冷やす:卵、水は1L蒸留水に0.21グラム/ Lインスタントオーシャン塩を含みます。 55のNaCl、1.8mMのKClと1.25 mMの炭酸水素ナトリウムを含有する緩衝液を脱yolking;そしてFACS緩衝液を、5%熱不活化ウシ胎児血清を補充したリーボビッツのL-15培地を含みます。
- RTに細胞解離試薬1のサンプルあたり1ミリリットルを持参してください。氷上でサンプル当たり1ミリリットルの細胞解離試薬2を解凍します。使用直前に室温に持参してください。
3.単一細胞解離
- 広口ガラスピペットを用いて、卵の水の最小容量で2ミリリットルマイクロ遠心チューブに100から300の胚を移します。
- 1ミリリットルの氷冷卵水と水を交換し、20分間氷上でチューブを沈めることによって胚を安楽死させます。
注意!(二次安楽死などの細胞解離に続いて氷上で冷却)この安楽死法のための適切な制度上の動物のケア監督委員会の承認を得ることが重要です。標準的な安楽死は、典型的には冷蔵された後の胚<3日後に受精が生細胞を得るために適切ではないである、漂白されていること。必要で - 1ミリリットルの氷冷卵水で胚を2回洗浄します。洗浄するために、卵水と卵の水を追加するにはP1000を削除するには、広口ガラスピペットを使用しています。
- P1000チップで8-12回1ミリリットルDeyolking Bufferで胚の水を交換し、粉砕することにより、卵黄を削除するか、または卵黄が溶解し、されるまで、胚の唯一の体が表示されます。
- 1分間300×gで遠心分離することによって組織を収集します。優しく組織ペレットを中断することなく、上清を除去するために、ピペットを使用してください。 1ミリリットル卵水に再懸濁します。
- 繰り返し合計3回の洗浄のためのステップ3.5が、最終的な洗浄に、1ミリリットルRTの細胞解離試薬1中に再懸濁します。
- 水平に置い管を室温で10分間、細胞解離試薬1でインキュベートします。すべての2-3分、軽く凝集を防止するために、P1000ピペットで粉砕します。
注:摩砕工程中に静かにサンプルを処理します。いつか、単一の大きな塊は、胚体のもつれからチューブ内に形成することになります。この意志穏やかな粉砕により支援追加の消化を分散させます。 VORTEXしないでください。 - 3分間300×gで遠心分離することによって組織を収集します。 1ミリリットルの細胞解離試薬2に上清および再懸濁を削除します。
- RTで5-15分間インキュベートします。水平にチューブを入れます。すべての2-3分は、静かに凝集を防止するために粉砕します。 5分毎に、消化の進捗状況を評価します。
- 消化の進行状況を評価するために、18μlのFACS緩衝液および細胞培養皿上に液滴としてピペット内に2μlの上清を希釈。
- サンプルの上にカバースリップを置き、10Xと20X倍率で組織培養顕微鏡下で観察します。
注:準備は単一細胞、小さなクラスター、大規模なクラスタ、および臨時のほとんどが無傷胚体のミックスとして表示されます。 - 視覚的に単一細胞と小さなクラスターである準備の割合を評価します。
注:過剰消化の生存率が低下します。下消化単一細胞収量が減少します。
- 5分間、300×gの遠心分離によって細胞調製物を収集します。上清を捨て、冷たいFACS緩衝液1ml中に再懸濁します。
- FACS緩衝液で40μmのセルストレーナーを湿ら。 35mmの細胞培養皿上に40μmのセルストレーナーを介して細胞懸濁液を渡します。バッファをソートする1ミリリットルのFACSを用いて細胞ストレーナー1時間を洗ってください。
- 2ミリリットルのマイクロチューブにフロースルーを転送します。 5分間、300×gの遠心分離により細胞を回収し、100μlのFACS緩衝液に再懸濁します。
- 血球計数器を用いて細胞収量をカウントします。また、5×10 6ミリリットルあたりの細胞、または選択のFACSマシンの推奨される最適な濃度にサンプルを希釈します。血球計数器で細胞収率をカウントすると、オプションの-、トリパンブルーで対比染色死んだ細胞は、FACS選別の前に細胞調製の生存を確認しました。
注:あまりにも希薄されている準備がソートするために時間を過剰になります。あまりにcは準備oncentrated多量体に凝集する傾向があり、純粋な集団をソートするための次善のです。 - 無染色のコントロールとして使用するには、各サンプルの準備百分の10から20まで。各試料中に蛍光(L / D)デッド/ライブ判別色素を添加することにより、残りのサンプル、汚れ死んだ細胞について。洗浄しないでください。
注:核酸は、L / Dの染料として使用することができる損なわれた細胞膜や汚れを細胞に浸透する任意の蛍光染料は、蛍光スペクトルは、対象の選択及びフルオロフォア標識細胞のFACS機器と互換性があることを条件とします。- いくつかの細胞は、FACS精製に輸送中に死んでしまうと、ソートされた集団から除外されるべきである色素を添加した後の準備を洗浄しないでください。
- 20μlのFACS緩衝液でFACSチューブキャップさ35μmのセルストレーナーを湿ら。ストレーナーにセルを追加し、重力によって収集します。氷の上に保管してください。
4. FACS濃縮
- 単一のために豊かにするために、FACSを使用して、、蛍光マーカーを発現する細胞が住んでいます。
- 側方散乱振幅(SSC-A)、リニアスケーリングで両方の振幅対前方散乱(FSC-A)の散布図に破片から細胞を区別するためにゲートを設定します。
注意!ゼブラフィッシュ細胞は、典型的にはマウスまたはヒト細胞よりも小さいです。これは、細胞および破片間の低い基底の分離に反映されています。 - 4.1.1で設定されたゲートからは、単一の細胞を濃縮し、前方散乱の高さ(FSC-H)とロー側方散乱の高さ(SSC-H)の散布図を使用して多量体、リニアスケーリングとの両方を除外するためにゲートを設定します。
注:不相応に高いFSC-Hと低SSC-Hを有する細胞は、おそらく多量体であり、ソートの対象から除外されています。 - 4.1.2におけるゲートセットから、単一のカラーコントロールを使用して、チャネルのリニアスケーリングおよび振幅でFSA-Aの散布図を使用している唯一の生きた細胞が含まれるようにゲートを設定するには、対数スケールでのL / Dの汚れを検出するために使用されます。 L / D陰性細胞が含まれます。
- からゲートは、単一色のコントロールを使用して、4.1.3に設定されたログスケールで蛍光タンパク質の細胞マーカーを検出するために使用されるチャネルの線形スケーリングおよび振幅FSA-Aの散布図を使用して正の蛍光を有する唯一の生細胞を含むようにゲートを設定します。陽性細胞が含まれます。
- 必要に応じて、L / D染色および蛍光タンパク質のスペクトル間の干渉を考慮して、任意の補償制御を設定します。
- 側方散乱振幅(SSC-A)、リニアスケーリングで両方の振幅対前方散乱(FSC-A)の散布図に破片から細胞を区別するためにゲートを設定します。
- ステップ4.1で設定されたゲートのすべてに該当した細胞にソートロジックを設定します。
- 二重標識された細胞を用いて、細胞選別のためのゲーティングを確認してください。
注:ソート用の細胞は、細胞よりもむしろ破片、むしろ多量体よりも、単一細胞、L / D負および蛍光陽性細胞となります。
- 二重標識された細胞を用いて、細胞選別のためのゲーティングを確認してください。
- 氷上でマイクロ遠心チューブ内の冷たいFACSバッファーを5μlへの関心の人口からソート2,000〜4,000細胞。セルのために可能な限り最低の圧力を使用して、並べ替えの際に細胞上のせん断ひずみを最小限に抑えるために、ソーター。
注:FACSバッファー5μlの滴はFACS装置を出た細胞のためのクッションとして機能します。直接FACSバッファに2,000〜4,000細胞を収集することはIFCチップ上にロードする前に遠心分離する必要がなくなります。細胞はペレットに互いに接着した場合、遠心分離は、多量体の形成につながることができますので、この戦略が推奨されます。 - ポストソート生存率分析を評価します。
- L / D染色の千希釈:転送1μlをバッファと1を並べ替え100μlのFACS新鮮なFACSチューブに細胞を選別しました。ステップ4.1でのゲーティング戦略にFACSソーターを介して細胞を渡します。ソートされた細胞の生存率を推定するための正にL / Dの負の割合を使用してください。
マイクロフルイディクスチップ上に5.ロードセル
- 血球計数器を使用して、ソートされた細胞の濃度とサイズの両方を測定します。
- 少なくとも10μlにソートされた細胞を希釈します。 5μのアリコートを希釈; 5μlのトリパンブルーとL細胞。血球計数器上にロードし、カバースリップを適用します。
- 血球計数器の4×4のグリッド内のすべてのセルの明視野画像を収集します。生細胞の数をカウントし、生細胞/ mlを計算します。生細胞は、トリパンブルーを取ることはありません。
注:すべての生細胞の直径を測定するために、任意の標準的な画像解析ソフトウェアを使用してください。細胞の大きさの平均値と標準偏差を計算し、目的の細胞サイズの範囲に適したIFCプレートを選択します。セルサイズ範囲は、IFCプレートセルサイズ範囲にまたがっている場合、関心対象の細胞の全範囲を捕捉するために複数のプレートを使用します。
- IFCプレートへのロードセル製造業者の指示に従って(材料を参照されたいです)。
- 1×10 6細胞/ mlに細胞を希釈し、製造業者の説明書に従って浮力の最適化を行います。
注:浮力の最適化は、細胞が底に沈むことも、ローディングWELのトップにフロートもないことを保証しますIFCのチップに最適なロード用リットル。細胞へのバッファの割合は、細胞型によって異なりますが、一般的に6の範囲にあることがあります。4-7:細胞の3:バッファ。 - 下塗りされた-IFCプレートに細胞を追加します。ロード互換性流体工学のマシンにプレート、およびマイクロ流体回路を介して、キャプチャレーンに細胞をプッシュするセルロード・スクリプトを実行します。
- 1×10 6細胞/ mlに細胞を希釈し、製造業者の説明書に従って浮力の最適化を行います。
- 細胞はIFCプレート上の捕捉部位に留まっていることを確認してください。
- 流体工学マシンからIFCプレートを取り外します。プレートアダプターを備えた顕微鏡でIFCプレートをマウントします。
- 10X倍率で各捕捉部位のための明視野および蛍光のスナップショットを収集します。
注:明捕捉時間は、ランプの強度に応じて、10〜50ミリ秒の範囲であってもよいです。蛍光捕捉時間は、フルオロフォアの明るさに応じて、250〜750ミリ秒の範囲であってもよいです。光損傷を防ぐために、細胞をoverexposing避けてください。
6. cDNA合成
- Iに従ってin situで細胞溶解を行いますFCプレートの製造元の指示。
- IFCプレート上のウェルに溶解緩衝液を追加します。ロードプレート互換性流体工学のマシンにあたり、メーカーの指示通り細胞溶解スクリプトを実行します。
- IFCプレートの製造元の指示に従って逆転写を行ってください。
- IFC板に逆転写試薬を追加します。互換性のある流体のマシンにロードプレート。逆転写スクリプトを実行します。サンプルサイクリング条件:5分間85℃を1サイクル、10分間25°Cで1サイクル、1時間、42℃で1サイクル。
- IFCプレートの製造元の指示に従って、遺伝子特異的プローブを用いてプレ増幅を行います。
注:低コピー数とのより高い特異性のため、インターカレーター色素での使用に最適化された蛍光性標識プローブではなく、プローブを使用しています。- プールのプライマーと最終180 nMのそれぞれに希釈します。 IFCプレートにプールされたプライマーを追加します。互換性のfluidiにロードプレートCS機。プリアンプスクリプトを実行します。
- 以下のサイクル条件で予備増幅を行う:1サイクル、95℃で10分間、次いで18サイクルを4分間60℃で、15秒間、95℃でのアニーリングおよび伸長を変性して。ストアは、収穫まで4℃でのcDNAを予備増幅しました。メーカーの指示および使用するまで-20℃で保存によるマイクロ流体プレートから収穫するcDNA。
シングルターゲット定量RT-PCRのための単一細胞から7. [cDNAを
- 製造者の指示に従って25μlの希釈剤中のcDNAを希釈します。
注:おおよその最終収率は、セル当たりプレ増幅したcDNAの28μlです。定量RT-PCRで無鋳型対照として使用するための希釈剤の一定量を確保。- 明視野とステップ5.3.2に記録蛍光画像を参照すると、各捕捉部位をスコア。手動でカウントし、細胞数を記録。各セルはインフルエンザであるかどうかを評価し、記録しますorescent。
注:個々のサイトは、> 1のセルが複数のセルおよび/または識別不能な破片を含む捕捉部位を含む0,1、または> 1細胞を含有してもよいです。画像が十分な品質である場合、画素値はまた、蛍光シグナルの強度を定量化するために割り当てられてもよいです。
- 明視野とステップ5.3.2に記録蛍光画像を参照すると、各捕捉部位をスコア。手動でカウントし、細胞数を記録。各セルはインフルエンザであるかどうかを評価し、記録しますorescent。
- 定量RT-PCR分析のためのサンプルを選択します。
- 正の蛍光を有する正確な1セルが含まれているステップ7.2で識別された捕捉部位からcDNAサンプルを選択します。各サンプルからのcDNAのプール1μlのアリコート。
注:これは、目的の遺伝子が選択された細胞のいずれかにおいて発現されるかどうかを決定するために使用される「プール」陽性対照です。 - ネガティブコントロールとして正確に0細胞を含む少なくとも1つの捕捉部位からcDNAを選択します。無鋳型対照としてステップ7.1.2からの希釈剤を使用してください。
- 正の蛍光を有する正確な1セルが含まれているステップ7.2で識別された捕捉部位からcDNAサンプルを選択します。各サンプルからのcDNAのプール1μlのアリコート。
- 定量RT-PCRアッセイを実行します。
- ステップ6.4.1でプレ増幅のために使用されるプローブのみを使用してください。 triplicat内のすべてのサンプルをロードします電子。 384ウェルプレートに10μlの反応のためのサイクリング条件:10分間の1サイクル、50℃2分、1サイクル、95℃、1 60℃でのアニーリングおよび伸長後、95℃15秒で変性と40サイクル分。
8.データ解析
- 標準的なゲル電気泳動によって定量RT-PCR産物を検証します。製品を確認して予想される分子量(各プローブごとに異なる)で単一バンドです。
注:プローブは不適切なサイズで複数のバンドまたは単一バンドを生成した場合、特異性は疑問であり、これは解析から遺伝子を除外します。 - 異常14のすべての増幅曲線とアカウントを確認します。各サンプル/遺伝子の組み合わせ15の平均CT値を計算します。
注:ポジティブコントロールのCT値は、一般的に10から30の範囲です。ネガティブコントロールのCT値は、典型的には35〜40(増幅なし)の範囲です。 CT値は、30から35は、非常に低い発現と考えられていますそして、注意14と解釈されるべきです。 - レポートデータ
- サンプルはCT = 10から30に落ちた場合、データはまた、対照試料と比較し転写産物量の変化倍率として報告されることがあります。
注:( - ΔΔCT)の変化を折るは2 ^等しいΔCTは、ハウスキーピング遺伝子からのサンプルの平均CTを減算することにより、ハウスキーピング遺伝子に対して相対的であるとΔΔCTは、サンプルおよび陽性対照15との間の ΔCTの違いです。
- サンプルはCT = 10から30に落ちた場合、データはまた、対照試料と比較し転写産物量の変化倍率として報告されることがあります。
結果
原理の証明として、遺伝子発現は、心臓の開発中分化の動態を調査するために評価しました。ゼブラフィッシュでは、心臓前駆体は、彼らが線形心管を形成するために融合し、前方側板中胚葉に移動する細胞の中胚葉集団から生じます。融合の前に、心臓前駆体は、心臓前駆体16,17の最も初期の特異的なマーカーであると考えられる転写因子nkx2.5(NK2ホメオボックス5)、発現し始めます。ここでは、先に述べたBACトランスジェニック魚のTg(nkx2.5:ZsYellow)18、nkx2.5 を略記:ZsYは 18体節期、18時間後に受精(HPF)12で単一細胞における心臓分化マーカーを調べました。これはZsYellow信号が視覚的に検出した最も早い時点でした。以前にこのトランスジェニック系統のために説明したように、ZsYellowは、私たちのように、心臓前駆体を標識28 HPF 19における咽頭弓の内皮細胞を生じ得、いくつかの余分な心臓細胞のようなLL( 図1A-Bを 、データは示していません)。 18 HPFでは、nkx2.5:ZsY胚その後、死細胞を排除するためのSytoxブルーで染色した単一細胞懸濁液に解離しました。正、のSytoxブルー陰性細胞は、FACSが100μmのノズル( 図1C-F)を装備したMoFloXDPまたはソニーSH800Zソーターのいずれかを使用して選別したZsYellow、住んでいます。ソートされた集団とポストソート生存率の純度を評価するために、選別した細胞のアリコートのSytoxブルーで染色し、元の選別ゲート( 図1G-J)内に入る事象の割合を評価しました。
選別後、細胞を、製造者の指示に従って、集積マイクロ流体回路(IFC)チップワークフロー( 図2A)に入力しました。トリパンブルーexclusioと組み合わせる血球計数器、nが( 図2B、データは示していない)セル径、濃度、及び生存率を測定しました。製造者の指示に従って、細胞は5-10ミクロンの直径の細胞を捕捉するためにIFCにロードした(緩衝液:3.5細胞6.5)細胞浮力を最適化しました。捕捉効率、蛍光および/またはすべてのキャプチャサイトで撮像した明視野信号を評価するために、フィジーでのタイリング機能は、マイクロフルイディクスプレートの単一画像をステッチするために使用されました。各捕捉部位は0、1、又は> 1、個々の細胞( 図2C-F)を含有しました 。 FACS濃縮から予想されるように、捕獲された細胞はZsYellow( 図2G)を表明しました。捕捉効率は、nkx2.5を用いて、5個体の実験において、90%を超えて:ZsY細胞をソートし、捕捉部位の少なくとも70%が単一のセルによって占有された(データは示さず)。細胞を溶解し、RNA単離され、cDNAを合成し、特定の標的遺伝子は、すべての製造業者の指示に従って、増幅しました。
上記のプロトコールに記載のように細胞捕捉部位のサブセットは、定量RT-PCR分析のために選択しました。具体的には、伸長因子1A(efl1a)は 20とグリセルアルデヒド-3-リン酸(GAPDH)21は、全ての細胞において発現されることが予想されるハウスキーピング遺伝子としてアッセイしました。 GATA結合タンパク質4(GATA4)22及びNK2ホメオボックス5(nkx2.5)早けれ心臓前駆マーカー;第二の心臓フィールドマーカー23,24としてISL LIMホメオボックス1ボックス1(ISL1)。そして、ミオシン軽鎖(myl7)と心室ミオシン重鎖(vmhc)25は、心室心筋細胞をマークします。遺伝子特異的プローブは、48 HPFの胚( 図3)からのcDNAを用いて検証しました。定量RT-PCRは、18 HPF胚、無テンプレート陰性対照からの45の単一細胞におけるこれらの遺伝子の相対的な遺伝子発現を評価するために使用され、プールされた集団の陽性対照containiました全96捕捉部位からcDNAをngの。 40代表的な細胞およびコントロールの生のCT値は、すべての遺伝子発現CT値をCT = 35.0を超えたために調べ46細胞のプールから、6個の細胞を分析から除外した、特に表1に示され、それは未知であるこれらの高CTか値がtrueの場合、非常に低い発現レベルまたはサンプルの分解に起因するものです。ハウスキーピング遺伝子、EF1AためのCT値の範囲ので、試料間の遺伝子発現を比較することが広すぎであり、多くのCT値が30を超えて、CT値は、ヒートマップ( 図3)のように可視化しました。サンプルにわたって比較し、遺伝子発現の実質的な不均一性が観察され、細胞は、発現パターンに基づいて、タイプ1-5( 図3)のような細胞を分類しました。

ゼブラフィッシュの1.単一細胞の単離を図 nkx2.5:18 HPFで ZsY 陽性細胞全体が代表のTg(nkx2.5:ZsYellow)の画像をマウントします。(A)ZsYellow単独の蛍光または(B)で18 HPFで胚は、明視野像と合併しました。 (CF)代表FACSゲーティング戦略はZsYellow陽性細胞とを富化する(GJ)(C、G)FSC / SSCサイズ・ゲーティング、(D、H)ダブレット差別、(E、I)ライブ/デッド・ゲーティングとポストソート解析、及び(F、J)は、人口ソート。スケールバーは100μmである。 この図の拡大版をご覧になるにはこちらをクリックしてください。
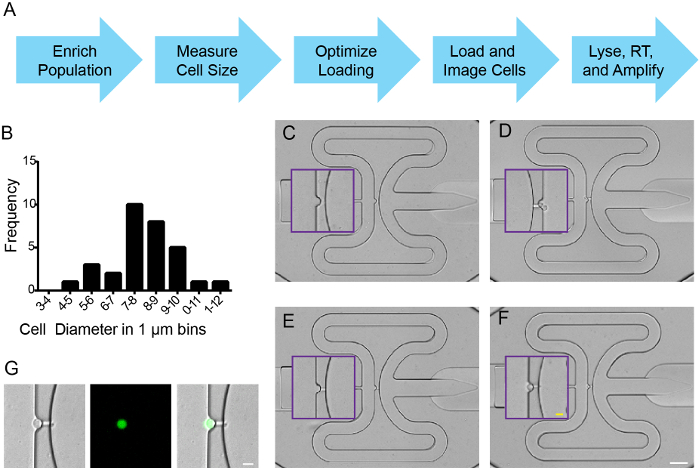
図2ゼブラフィッシュ nkx2.5 の単一細胞捕捉 :ZsY オング>陽性細胞。(A)作業の流れ。 (B) のTg(nkx2.5:ZsYellow)からソートされた細胞の細胞サイズ分布18 HPFで分離胚。 (CF)、(C)は空のウェルであるIFCプレート上の代表的な細胞捕捉イベント、(D)は、2つのセルを含む、(E)は、「チャネル捕捉」として流体チャネルに留まる1つのセルを有し、及び(F)であります単一捕捉された細胞。パープルボックスは、捕捉部位の拡大図のためのインセットマーク。明視野、ZsYellowとマージされた画像と(G)単一細胞の捕捉。 (CF)ホワイトスケールバーは50μmです。黄色のスケールバーは10μmです。 (G)スケールバーは10μmである。 この図の拡大版をご覧になるにはこちらをクリックしてください。
igure 3 "SRC =" /ファイル/ ftp_upload / 53877 / 53877fig3.jpg "/>
図キャプチャ単一ゼブラフィッシュ nkx2.5 の3遺伝子発現解析 :ZsY 陽性細胞の遺伝子陽性対照における定量RT-PCRによる発現(2日-受精後に胚、単一細胞からのcDNAをプールされた)、ネガティブコントロール(無テンプレート。 )と40単セル(セル01から40)。キーに記載されているように、生のCT値を色分け基づいていた。EF1A = 伸長因子1aと 、GATA4 = GATA結合タンパク質4、nkx2.5 = NK2ホメオボックス5、myl7 = ミオシン軽鎖7、vmhc = 心室ミオシン重鎖 、GAPDH = グリセルアルデヒド-3-リン酸 、およびISL1 = ISL LIMホメオボックス1。 この図の拡大版をご覧になるにはこちらをクリックしてください。
| サイクル閾値(CT) | ||||||||
| EF1A | GATA4 | nkx25 | myl7 | vmhc | GAPDH | ISL1 | ||
| 胚 | 20.69 | 20.92 | 28.59 | 32.87 | 26.30 | 27.00 | 33.57 | |
| プール | 26.4 | 22.4 | 27.7 | 27.4 | 24.5 | 29.2 | 40.0 | |
| NTC | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| タイプ1 | セル-01 | 25.1 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 |
| セル-02 | 25.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-03 | 26.3 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-04 | 27.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-05 | 28.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-06 | 28.2 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-07 | 29.3 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 400.0 | |
| セル-08 | 30.7 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-09 | 34.3 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| タイプ2 | セル-10 | 23.5 | 28.0 | 40.0 | 40.0 | 40.0 | 26.3 | 40.0 |
| セル-11 | 23.5 | 27.2 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-12 | 23.7 | 17.9 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-13 | 24.1 | 32.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-14 | 24.9 | 27.5 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-15 | 25.9 | 28.6 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-16 | 26.0 | 28.1 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-17 | 27.0 | 27.7 | 40.0 | 40.0 | 40.0 | 38.5 | 40.0 | |
| セル-18 | 27.0 | 27.4 | 40.0 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-19 | 27.0 | 30.2 | 40.0 | 40.0 | 40.0 | 38.2 | 40.0 | |
| セル-20 | 27.0 | 28.2 | 40.0 | 40.0 | 400.0 | 39.2 | 40.0 | |
| セル-21 | 27.7 | 30.3 | 40.0 | 40.0 | 40.0 | 38.6 | 40.0 | |
| セル-22 | 30.4 | 26.2 | 40.0 | 40.0 | 40.0 | 38.3 | 40.0 | |
| セル-23 | 31.3 | 22.4 | 40.0 | 40.0 | 40.0 | 39.7 | 40.0 | |
| セル-24 | 31.5 | 28.8 | 40.0 | 40.0 | 40.0 | 39.5 | 40.0 | |
| セル-25 | 33.8 | 27.4 | 40.0 | 40.0 | 40.0 | 37.3 | 40.0 | |
| セル-26 | 40.0 | 27.4 | 40.0 | 40.0 | 40.0 | 36.6 | 40.0 | |
| タイプ3 | セル-27 | 24.7 | 19.9 | 28.8 | 40.0 | 40.0 | 40.0 | 40.0 |
| セル-28 | 24.8 | 28.4 | 26.3 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-29 | 25.1 | 23.5 | 27.6 | 40.0 | 40.0 | 32.5 | 40.0 | |
| セル-30 | 26.3 | 21.4 | 27.5 | 40.0 | 40.0 | 39.9 | 40.0 | |
| セル-31 | 26.9 | 24.8 | 28.2 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-32 | 27.0 | 22.5 | 23.8 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-33 | 27.8 | 27.6 | 27。7 | 40.0 | 40.0 | 40.0 | 40.0 | |
| セル-34 | 28.1 | 21.9 | 26.6 | 40.0 | 40.0 | 37.7 | 40.0 | |
| セル-35 | 29.3 | 26.5 | 28.1 | 40.0 | 40.0 | 38.3 | 40.0 | |
| タイプ4 | セル-36 | 24.8 | 20.3 | 26.0 | 27.4 | 26.6 | 40.0 | 40.0 |
| セル-37 | 28.7 | 22.3 | 25.9 | 28.9 | 22.9 | 38.5 | 40.0 | |
| タイプ5 | セル-38 | 25.5 | 20.0 | 29.3 | 23.0 | 19.9 | 25.9 | 40.0 |
| セル-39 | 25.9 | 21.8 | 28.6 | 25.1 | 21.7 | 25.7 | 40.0 | |
| セル-40 | 28.9 | 22.0 | 27.0 | 23.0 | 21.3 | 27.6 | 40.0 | |
表1. 生のCT値。
ディスカッション
本明細書に記載される方法は、単一の心臓遺伝子のサブセットの発現を評価するために、マイクロ流体アシスト単一細胞捕捉システムで使用するためのゼブラフィッシュ胚からの心臓前駆細胞の集団を濃縮するために細胞型特異的プロモーターの制御下で蛍光タンパク質の発現を使用して細胞。 FACSレーザ励起と発光の機能が選択されたフルオロフォア(複数可)と適合することを、この方法は、任意の蛍光レポーターラインのために使用することができます。多くのゼブラフィッシュのレポーターラインがすでに存在しており、新たなレポーターを保有するトランスジェニック魚はわずか3ヶ月で生成することができます。また、このワークフローは、単一細胞RNA配列決定のための全トランスクリプトームからcDNAを生成するように適合されてもよいです。これを行うには、ステップ5-7 RNA配列決定に適したライブラリーを作製するためのcDNAを調製するために最適化された化学反応で使用するためにわずかに修正する必要があります。しかし、人口の異質性を検証することをお勧めしますハイスループットシークエンシングアプリケーションを追求する前に、ここで説明した方法を使用して。
記載された方法にはいくつかの制限があることに留意することが重要です。まず、統合されたマイクロ流体回路(IFC)プレートの使用は、特殊な、高価な装置を必要とします。しかし、マイクロ流体チップは、単一細胞をアッセイするための伝統的な96ウェルおよび384ウェルプレートフォーマットを超える実質的な利点を提供します。マイクロメートルスケールのレーンを介して細胞および試薬を流すことにより、単一細胞は、それらが直接、彼らが健康で単一細胞であることを確認するために観察することができる単一の捕捉部位に配置されています。 RNA抽出およびcDNA合成は、各セルのための非常に少量を使用したIFCチップ上にその場で起こります。統合された流体チップは、この記事の執筆時点では、唯一のソースを介して市販されています。非商業的な製造は、いくつかのグループによって行われていたが、それはほとんどの研究室の容量の外にあります。 5-7できるステップ他の企業や社内の捏造によって生成プラットフォーム用に変更する必要があります。市販のIFCプレートは96遺伝子まで収容できるものの第二に、選択された遺伝子は、ゼブラフィッシュでの使用のために検証発蛍光標識遺伝子プローブの利用可能性によって制限されています。第三に、ゼブラフィッシュ細胞は、典型的には、哺乳動物細胞よりも小さく、この小さなサイズは、サンプル処理の課題を提示します。セル径の小さな変化は、利用可能な材料を低減し、低検出遺伝子を発現する可能性を減少させる、細胞体積の大きな変化に変換されます。
このプロトコルのためのいくつかの追加の考慮事項があります。ステップ1では、それは短い時間窓内受精のみ使用胚にとって重要です。一定の酸素条件を限定することなく、温度を維持し、ゼブラフィッシュ胚は同期開発します。このプロトコル(単一細胞からの相対的な遺伝子発現)の最終的な読み出しがheterogeのソースを判別することはできません個々のセル間のneity。このため、細胞間不均一性の解釈は慎重に段階的な胚および同期の開発に依存してい。ステップ2-3で、解離および生存率との間にトレードオフが存在します。ステップ4は、生細胞のために選択し、ステップ3は、多量体を除外するに役立ついくつかのフィルタリングステップが含まれていますが、過剰消化FACSで使用可能な実行可能な材料を削減し、潜在的にソートされた細胞収量が減少します。手順は、低い生存率は、消化時間を短縮する粉砕強度を低減し、出発物質中の胚の数を最適化することが挙げられるトラブルシューティングします。関心(ステップ4)の細胞集団のFACS濃縮は、間違いなく、このプロトコルの中で最も重要なステップです。厳格なソート基準は、目的の蛍光タンパク質を発現する唯一の生きた細胞を含む単一細胞調製物を生成するために必須です。他の集団から細胞を汚染することは細胞間の異質の誤った表現につながる可能性があります。注目すべきは、関心の人口のFACS濃縮は、このプロトコルに示した2色の戦略に限定されるものではありません。より複雑な戦略は、より洗練された細胞集団を生成し、原則として研究の証拠で報告された方法論を拡張するための迅速な方法であることができます。ステップ5において、細胞は、直接捕捉された細胞の数、細胞の健康、および蛍光タンパク質の発現を確認するために観察することができます。しかし、細胞の大きさや明るさに応じて、関心のフルオロフォアは、視覚的に検出できないと信頼性の高い読み出していない場合があります。ステップ6-8の正常終了は、製造業者のプロトコルに慎重に遵守する必要があります。
原理の証明としては、既知の心臓マーカーのサブセットの発現レベルは、以前に記載さBACトランスジェニックゼブラフィッシュラインnkx2.5に由 来する単一に評価された:ZsYは 、個々のnkx2.5における心臓分化マーカーを調べた:ZsY陽性細胞18時間後に受精(HPF)。 heterogeneiを探求するために、捕獲さnkx2.5で表現分化マーカーのTY:、遺伝子のスイートはハウスキーピング遺伝子(EF1A、GAPDH)、早期の心臓仕様(GATA4、nkx2.5)でオンにすることが知られている転写因子を含むアッセイした ZsY 発現している細胞 、心臓分化(myl7、vmhc)中に、後にオンされることが知られている第二の心臓フィールド前駆マーカー(ISL1)、および遺伝子。各遺伝子の相対的存在量は、サイクル閾値(CT)を計算することにより定量RT-PCRにより測定しました。計算された最も低いCT値は17.9だった、最高には、増幅(表1)を表すない、40でした。 > 35.0のCT値が検出について考察しました。
データセット内の46細胞のうち、6は何らかの遺伝子の障害が発生した増幅を除外しました。残りの細胞はGATA4、nkx2.5、myl7、およびvmhc、およびWERの発現に基づいて5種類に細胞をサブ分類しましたeはグループ内のEF1A値によってソート。 GAPDHは、もともと可能なハウスキーピング遺伝子として含まれていたが、表情はハウスキーピング遺伝子として、それが不適当、細胞全体で非常に変動した。GAPDH発現の可能性が高い、より良い、この細胞型で心臓分化に関連したエネルギー代謝の変化を表しています。興味深いことに、未検出可能であったが、他の遺伝子は、(セル26におけるGATA1)検出可能であったEF1Aで一つのセルがありました。これはEF1Aは一般的に成功と失敗単細胞逆転写反応を区別するための十分なハウスキーピング遺伝子であるが、それはすべてのアプリケーションに理想的ではないかもしれないことを示唆しています。セル26におけるEF1A発現の欠如は、単一細胞の転写ダイナミクスが原因である可能性があります。最近の単一細胞研究は、迅速かつごくわずかな転写活性26の位相との間に交互に、遺伝子転写は、基本的に確率論的であることを示しています。 T彼の転写破裂モデルは、単一の細胞間の定量的比較のためのハウスキーピング遺伝子の使用は全く不適切であることを示唆しています。
タイプ1のグループでは、EF1Aは、検出可能な唯一の遺伝子です。関心のある他の遺伝子の非常に低い発現とZsY陰性細胞または細胞:これらの細胞は、nkx2.5のいずれかを含むことができます。 2細胞を入力し、セル-26を除いて、これらの遺伝子の相対的発現レベルの相関がないとEF1AとGATA4の両方を表明しました。なお、タイプ1とタイプ2細胞におけるnkx2.5式が原因で、次善のFACSジェンシー、転写破裂、導入遺伝子の漏出、またはサブスレッショルド発現レベルである可能性があります。タイプ3細胞はEF1A、GATA4、及びNKX-2.5の検出可能なレベルで発現した。これらの細胞は、おそらく心臓前駆細胞であり、心房心筋細胞を分化含んでいてもよいです。タイプ4細胞はEF1A、GATA4、nkx2.5、myl7の検出可能なレベルで発現し</ em>のとvmhcおよび心室心筋細胞に分化する可能性が高い細胞です。タイプ5細胞はEF1A、GATA4、nkx2.5、myl7、vmhc、およびGAPDHを表明しました 。 GAPDHの検出可能なレベルの添加は、これらの細胞が分化した心筋細胞において見出さ増強解糖代謝能力を有することを示唆しています。 、胚、40分の21細胞は私たちの定量RT-PCR解析でnkx2.5のサブスレッショルドレベルを有していた。これは、転写破裂が原因である可能性があります:それはTgは(ZsYellow nkx2.5)からZsYellow陽性細胞のソートにもかかわらず、ことは興味深いでした。または内因性nkx2.5とZsYellow間の転写物処理の違いを反映している可能性が。重要なのは、第二の心臓フィールドマーカーISL1は、当社の捕捉部位の全てからプールの任意の細胞またはcDNAで検出されませんでした。要するに、サンプル全体で比較すると、かなりの異質性が示唆され、単一細胞レベルで明らかになったその18 HPFで、nkx2.5:ZsY + CELlsコマンドは、分化の異なる段階で心臓前駆体だけでなく、子孫を含みます。
開示事項
The authors have nothing to disclose.
謝辞
We thank Dr. C. Geoffrey Burns for fish stock. The authors are grateful to UNC Flow Cytometry Core Facility, UNC-CGIBD AAC core for resources enabling this project, and the ZAC facility for animal care. L.S. is supported by NIH T32 grant HL069768-13 (PI, Nobuyo Maeda). N.F. is supported by NSF Graduate Research Fellowship NSF-DGE-1144081. This study was supported by NIH P30DK034987 grant (to UNC Advanced Analytics Core), American Heart Association Scientist Development Grant 13SDG17060010 and Ellison Medical Foundation New Scholar Grant AG-NS-1064-13 (to Dr. Qian), and NIH R00 HL109079 grant (to Dr. Liu).
資料
| Name | Company | Catalog Number | Comments |
| Supplies | |||
| Dumont #5 forceps | Fine Science Tools | 11254-20 | For removing the chorion from embryos |
| Microcentrifuge tube 2 ml | GeneMate | C-3261-1 | |
| 40 um cell strainer | Biobasic | SP104151 | |
| 35 mm culture dish | Falcon | 351008 | |
| FACS tubes topped with 35 um cell strainer | Falcon | 352235 | |
| P1000 and tips | Rainin | 17005089 | |
| P20 and tips | Rainin | 17005091 | |
| IFC chip manufacturer's protocol | Fluidigm | 100-6117 | Version 100-6117 E1 was used in representative experiment |
| Wide bore pastuer glass pippette | VWR | 14673-010 | For transferring embryos |
| Adult wild type zebrafish | N/A | We used AB line | |
| Adult transgenic zebrafish | N/A | We used Tg(nkx2.5:ZsYellow) | |
| Name | Company | Catalog Number | Comments |
| Reagents for cell dissociation | |||
| Double distilled water | N/A | ||
| Instant Ocean Sea Salt | Instant Ocean | SS15-10 | |
| NaCl | FisherScientific | S271-3 | make stock in water and use for de-yolking buffer |
| KCl | Sigma Aldrich | P5405 | make stock in water and use for de-yolking buffer |
| NaHCO3 | Sigma Aldrich | S6014 | make stock in water and use for de-yolking buffer |
| Leibovitz's L-15 | Gibco | 21083-027 | |
| FBS | FisherScientific | 03-600-511 | Heat inactivate; any brand of FBS should be fine |
| Cell Dissociation Reagent 1 -TrypLE | Life Technologies | 12605-010 | Store at room temperature. |
| Cell Dissociation Reagent 2 - FACSmax | Genlantis | T200100 | Store -20; thaw on ice; bring to room temp before use |
| pronase (optional) | Sigma | P5147 | |
| L/D Dye - Sytox Blue | Life Technologies | S34857 | Any live/dead stain suitable for flow cytometry will work |
| Trypan Blue | Gibco | 15250-061 | |
| Name | Company | Catalog Number | Comments |
| Reagents for IFC plate use and qRT-PCR | |||
| Gene-specific probes | Probes will vary by experiment | ||
| TaqMan Probe ef1a | Life Technologies | Dr03432748_m1 | |
| TaqMan Probe gata4 | Life Technologies | Dr03443262_g1 | |
| TaqMan Probe nk2-5 | Life Technologies | Dr03074126_m1 | |
| TaqMan Probe myl7 | Life Technologies | Dr03105700_m1 | |
| TaqMan Probe vmhc | Life Technologies | Dr03431136_m1 | |
| TaqMan Probe isl1 | Life Technologies | Dr03425734_m1 | |
| TaqMan Gene Expression Master Mix | Life Technologies | 4369016 | |
| Reagents listed in IFC manufacturer's protocol | |||
| C1 Reagent Kit | Fluidigm | 100-5319 | Reagents for loading cells onto IFC plate |
| Ambion Single Cell-to-CTTM | Life Technologies | 4458237 | Reagents for reverse transcription and pre-amplification steps |
| Molecular Biology Quality Water | Corning | 46-000CM | |
| C1 IFC for PreAmp (5-10 um) | Fluidigm | 100-5757 | IFC plate for small cells |
| Name | Company | Catalog Number | Comments |
| Equipment | |||
| C1 AutoPrep machine | Fluidigm | 100-5477 | For IFC plate use |
| Hemocytometer | Sigma Aldrich | Z359629 | For counting cells and assessing cell size |
| Dissecting microscope | For removing embryos from chorion | ||
| Tissue culture microscope | For assessing single cell digestion | ||
| FACS machine | For isolating cells of interest |
参考文献
- Speicher, M. R. Single-cell analysis: toward the clinic. Genome medicine. 5, (2013).
- Macaulay, I. C., Voet, T. Single cell genomics: advances and future perspectives. PLoS genetics. 10, e1004126(2014).
- Marco, E., et al. Bifurcation analysis of single-cell gene expression data reveals epigenetic landscape. Proceedings of the National Academy of Sciences of the United States of America. 111, 5643-5650 (2014).
- Yin, H., Marshall, D. Microfluidics for single cell analysis. Current opinion in biotechnology. 23, 110-119 (2012).
- Garlanda, C., Dejana, E. Heterogeneity of endothelial cells. Specific markers. Arteriosclerosis, thrombosis, and vascular biology. 17, 1193-1202 (1997).
- Diaz-Cano, S. J. Tumor heterogeneity: mechanisms and bases for a reliable application of molecular marker design. International journal of molecular sciences. 13, 1951-2011 (2012).
- Guo, G., et al. Mapping cellular hierarchy by single-cell analysis of the cell surface repertoire. Cell stem cell. 13, 492-505 (2013).
- Guo, G., et al. Resolution of cell fate decisions revealed by single-cell gene expression analysis from zygote to blastocyst. Developmental cell. 18, 675-685 (2010).
- Treutlein, B., et al. Reconstructing lineage hierarchies of the distal lung epithelium using single-cell RNA-seq. Nature. 509, 371-375 (2014).
- Kimmel, C. B. Genetics and early development of zebrafish. Trends in genetics : TIG. 5, 283-288 (1989).
- Liu, J., Stainier, D. Y. Zebrafish in the study of early cardiac development. Circulation research. 110, 870-874 (2012).
- Kimmel, C. B., Ballard, W. W., Kimmel, S. R., Ullmann, B., Schilling, T. F. Stages of embryonic development of the zebrafish. Developmental dynamics : an official publication of the American Association of Anatomists. 203, 253-310 (1995).
- Zhao, S., Fung-Leung, W. P., Bittner, A., Ngo, K., Liu, X. Comparison of RNA-Seq and microarray in transcriptome profiling of activated T cells. PloS one. 9, e78644(2014).
- McCall, M. N., McMurray, H. R., Land, H., Almudevar, A. On non-detects in qPCR data. Bioinformatics. 30, 2310-2316 (2014).
- Schmittgen, T. D., Livak, K. J. Analyzing real-time PCR data by the comparative C(T) method. Nature. 3, 1101-1108 (2008).
- Lyons, I., et al. Myogenic and morphogenetic defects in the heart tubes of murine embryos lacking the homeo box gene Nkx2-5. Genes & development. 9, 1654-1666 (1995).
- Chen, J. N., Fishman, M. C. Zebrafish tinman homolog demarcates the heart field and initiates myocardial differentiation. Development. 122, 3809-3816 (1996).
- Zhou, Y., et al. Latent TGF-beta binding protein 3 identifies a second heart field in zebrafish. Nature. 474, 645-648 (2011).
- Paffett-Lugassy, N., et al. Heart field origin of great vessel precursors relies on nkx2.5-mediated vasculogenesis. Nature cell biology. 15, 1362-1369 (2013).
- McCurley, A. T., Callard, G. V. Characterization of housekeeping genes in zebrafish: male-female differences and effects of tissue type, developmental stage and chemical treatment. BMC molecular biology. 9, 102(2008).
- Tang, R., Dodd, A., Lai, D., McNabb, W. C., Love, D. R. Validation of zebrafish (Danio rerio) reference genes for quantitative real-time RT-PCR normalization. Acta biochimica et biophysica Sinica. 39, 384-390 (2007).
- Serbedzija, G. N., Chen, J. N., Fishman, M. C. Regulation in the heart field of zebrafish. Development. 125, 1095-1101 (1998).
- de Pater, E., et al. Distinct phases of cardiomyocyte differentiation regulate growth of the zebrafish heart. Development. 136, 1633-1641 (2009).
- Hami, D., Grimes, A. C., Tsai, H. J., Kirby, M. L. Zebrafish cardiac development requires a conserved secondary heart field. Development. 138, 2389-2398 (2011).
- Yelon, D., Horne, S. A., Stainier, D. Y. Restricted expression of cardiac myosin genes reveals regulated aspects of heart tube assembly in zebrafish. Developmental biology. 214, 23-37 (1999).
- Molina, N., et al. Stimulus-induced modulation of transcriptional bursting in a single mammalian gene. Proceedings of the National Academy of Sciences of the United States of America. 110, 20563-20568 (2013).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
ISSN 2578-2746
Copyright © 2023 MyJoVE Corporation. All rights reserved
当社はcookieを使用しています。
「続行」をクリックすることで、当社のcookieへの同意となります。