Method Article
Высокопроизводительное и глубокое протеомное профилирование с помощью 16-плексного тандемного мечения масс-меток в сочетании с двумерной хроматографией и масс-спектрометрией
* Эти авторы внесли равный вклад
В этой статье
Резюме
Здесь представлен оптимизированный протокол с высокой пропускной способностью, разработанный с использованием 16-плексных тандемных реагентов-меток, позволяющих количественно протеомировать биологические образцы. Обширное базовое фракционирование pH и ЖХ-МС/МС высокого разрешения смягчают коэффициент сжатия и обеспечивают глубокое покрытие протеома.
Аннотация
Изобарическая тандемная метка массой (ТМТ) широко используется в протеомике из-за ее высокой мультиплексирующей способности и глубокого покрытия протеома. В последнее время был внедрен расширенный 16-плексный метод ТМТ, который еще больше увеличивает пропускную способность протеомных исследований. В этой рукописи мы представляем оптимизированный протокол для 16-плексного протеомного профилирования на основе ТМТ, включая подготовку образцов белка, ферментативное расщепление, реакцию мечения ТМТ, фракционирование двумерной обратной фазовой жидкостной хроматографии (LC/LC), тандемную масс-спектрометрию (MS/MS) и вычислительную обработку данных. Выделены важнейшие этапы контроля качества и усовершенствования процесса, характерные для 16-плексного анализа TMT. Этот мультиплексный процесс представляет собой мощный инструмент для профилирования различных сложных образцов, таких как клетки, ткани и клинические образцы. Более 10 000 белков и посттрансляционных модификаций, таких как фосфорилирование, метилирование, ацетилирование и убиквитинирование в очень сложных биологических образцах из 16 различных образцов, могут быть количественно определены в одном эксперименте, что является мощным инструментом для фундаментальных и клинических исследований.
Введение
Быстрое развитие технологии масс-спектрометрии позволило достичь высокой чувствительности и глубокого протеомного покрытия в протеомных приложениях 1,2. Несмотря на эти разработки, мультиплексирование выборки остается узким местом для исследователей, занимающихся анализом большой выборочной когорты.
Методы мультиплексного изобарического мечения широко используются для количественного определения больших партий образцов 3,4,5,6 в масштабах всего протеома. Количественное определение на основе тандемных массовых меток (TMT) является популярным выбором благодаря высокой возможности мультиплексирования 7,8. Реагенты TMT первоначально выпускались в виде 6-плексного набора, способного количественно определять до 6 образцов одновременно9. Эта технология была в дальнейшем расширена для количественного определения 10-11 образцов10,11. Недавно разработанные 16-плексные реагенты TMTpro (далее TMT16) еще больше увеличили емкость мультиплексирования до 16 образцов в одном эксперименте12,13. В реагентах TMT16 используется репортерная группа на основе пролина, в то время как в 11-plex TMT используется репортерная группа, полученная из диметилпиперидина. Как TMT11, так и TMT16 используют одну и ту же аминоспособную группу, но группа массового баланса TMT16 больше, чем у TMT11, что позволяет комбинировать 8 стабильных изотопов C13 и N15 в репортерных ионах для получения 16 репортеров (рис. 1).
Расширение возможностей мультиплексирования обеспечивает платформу для разработки экспериментов с достаточным количеством повторений для преодоления статистических проблем14. Кроме того, дополнительные каналы в 16-плексной ТМТ помогают уменьшить общее количество исходного материала на канал, что может помочь в развитии новой протеомики одиночныхклеток15. Высокая емкость мультиплексирования также будет ценна при количественном определении посттрансляционных модификаций, для которых обычно требуется большое количество исходного материала16,17.
Протеомные рабочие процессы с использованием технологии TMT были оптимизированы 18,19,20, и за последнее десятилетие они значительно претерпели изменения с точки зрения пробоподготовки, разделения жидкостной хроматографии, сбора масс-спектрометрических данных и вычислительного анализа 21,22,23,24,25,26. В нашей предыдущей статье представлен подробный обзор 10-плексной платформы TMT27. Описанный здесь протокол представляет собой подробный, оптимизированный метод для TMT16, включая экстракцию и разложение белка, мечение TMT16, объединение образцов и обессоливание, основной pH и кислотный pH в обратной фазе (RP) LC, МС с высоким разрешением и обработку данных (рис. 2). В протоколе также выделены ключевые этапы контроля качества, которые были включены для успешного завершения количественного эксперимента по протеомике. Этот протокол может быть рутинно использован для идентификации и количественного определения более 10 000 белков с высокой воспроизводимостью, для изучения биологических путей, клеточных процессов и прогрессирования заболевания 20,28,29,30.
протокол
Человеческие ткани для исследования были получены с одобрения Программы донорства мозга и тела в Научно-исследовательском институте здоровья Banner Sun.
1. Извлечение белка из ткани и контроль качества
ПРИМЕЧАНИЕ: Чтобы уменьшить воздействие сбора образцов на протеом, крайне важно собирать образцы за минимальное время при низкой температуре,если это возможно. Это особенно важно при анализе посттрансляционных модификаций, поскольку они, как правило, лабилизированы, например, некоторые события фосфорилирования имеют всего несколько секунд периода полураспада32,33.
- Вырезание и взвешивание образцов тканей
- Поместите микроцентрифужную пробирку объемом 1,5 мл с помощью аналитических весов и предварительно охладите пробирку над сухим льдом.
- Разрежьте замороженную ткань (например, ткань человеческого мозга, ~10 мг) из определенной области на мелкие кусочки и немедленно перенесите кусочки ткани в предварительно охлажденную трубку.
ПРИМЕЧАНИЕ: Чтобы уменьшить гетерогенность образца, важно использовать однородные размеры и анатомические области для всех 16 образцов. Количество белка, полученного из ткани, обычно составляет 5-10% от массы ткани. - Взвесьте трубку вместе с тканью и сразу положите трубку на сухой лед. Обработайте оставшиеся 15 образцов, используя ту же процедуру. Сразу после вскрытия образцы хранить в сухом льду при температуре ˗80 °C.
- Образцы лизисной ткани
- В день эксперимента приготовьте свежий буфер для лизиса (50 мМ HEPES pH 8,5, 8 М мочевины и 0,5% дезоксихолата натрия). Ингибиторы фосфатазы следует добавлять в буфер для лизиса для сохранения состояния фосфорилирования белков.
ПРИМЕЧАНИЕ: Перед использованием поместите буфер для лизиса при комнатной температуре, так как 8 М мочевины выпадают в осадок на льду, что может привести к неполной денатурации белка во время лизиса образца и снизить эффективность переваривания белка. - Добавьте буфер для лизиса (добавьте 100 мкл буфера для лизиса на 10 мг ткани для достижения конечной концентрации белка от 5 до 10 мкг/мкл) и стеклянные шарики (~20% от объема лизата, 0,5 мм в диаметре) к каждому образцу.
- Лизируйте ткань в блендере при температуре 4 °C со скоростью 8 в течение 30 с, отдыхайте 5 с, повторяйте до тех пор, пока образцы не станут гомогенизированными (~ 5 циклов).
- В день эксперимента приготовьте свежий буфер для лизиса (50 мМ HEPES pH 8,5, 8 М мочевины и 0,5% дезоксихолата натрия). Ингибиторы фосфатазы следует добавлять в буфер для лизиса для сохранения состояния фосфорилирования белков.
- Подготовьте аликвоты из лизатов.
- Подготовьте не менее двух аликвот для каждого образца. Небольшая аликвота (~10 мкл) используется для анализа концентрации белка и оценки качества белка (например, валидация по вестерн-блоттингу белков положительного контроля). Для анализа протеома используется более крупная аликвота (~50 μL).
- Немедленно заморозьте аликвоты на сухом льду и храните при температуре -80 °C до дальнейшего использования.
- Измерьте концентрацию белка
ПРИМЕЧАНИЕ: Концентрацию белка можно измерить с помощью анализа BCA или короткого метода окрашивания гелем SDS34 (Рисунок 3A). Поскольку небелково-редуцирующие компоненты в тканевом лизате могут влиять на измерение в анализе BCA, пользователи могут проверить концентрацию белка с помощью короткого метода окрашивания гелем SDS. Короткий метод окрашивания гелем SDS представлен здесь.- Разбавьте 16 аликвотированных образцов в 10 раз и приготовьте стандарт BSA (например, титрования BSA 0,15, 0,5, 1,5 и 3 мкг).
- Запустите образцы и стандарт BSA на 10% геле SDS-PAGE (26 лунок) с укладывающим гелем до тех пор, пока все белки не мигрируют примерно на 3 мм в гель.
- Окрашивайте гель синим Кумасси в течение 1 часа и удаляйте пятна с геля, пока фон в пустой области не станет чистым.
- Отсканируйте гель, чтобы измерить интенсивность окрашенных Кумасси белковых полос с помощью ImageJ и создайте стандартную кривую BSA в соответствии с измерениями.
- Рассчитайте абсолютную концентрацию белка по стандартной кривой.
ПРИМЕЧАНИЕ: Конечная концентрация белка для каждого образца в этом эксперименте составила ~5-10 мкг/мкл. Для протеомного анализа на основе TMT16 достаточно 50 мкг белка на образец (всего 0,8 мг белка для анализа всего протеома).
- Контроль качества образцов
ПРИМЕЧАНИЕ: Этот этап контроля качества имеет решающее значение для выявления образцов низкого качества перед проведением анализа TMT. Для образцов с известным изменением белка рекомендуется подтвердить изменение с помощью вестерн-блоттинга. Стандартный анализ SDS-PAGE также рекомендуется для изучения белковых структур и исключения любых образцов с высокой степенью деградации (рис. 3B).- Возьмите ~10 мкг каждого образца из маленькой аликвоты и запустите образцы на градиентном геле SDS-PAGE до тех пор, пока бромфенольный синий краситель не достигнет дна геля.
- Окрасьте гель синим Кумасси и снимите с него пятна. Проверьте качество белка, чтобы удалить сильно деградировавшие образцы белка.
ПРИМЕЧАНИЕ: Деградировавшие образцы могут быть идентифицированы как образцы с очень небольшим количеством белковых полос в области с высокой молекулярной массой и усиленными полосами в области с низкой молекулярной массой (Рисунок 3B).
2. Расщепление белка в растворе, восстановление пептидов и алкилирование, проверка эффективности разложения и обессоливание пептидов
- Переваривание белка с помощью Lys-C и трипсина
- Возьмите ~50 мкг белка из большой аликвоты каждого образца и добавьте буфер для лизиса до 50 мкл.
- Добавьте 100% ацетонитрил (ACN) до конечной концентрации 10%.
- Выполните расщепление Lys-C, добавив Lys-C в соотношении белок:Lys-C 100:1 (w/w) и инкубируя при комнатной температуре в течение 3 часов.
- Образцы разбавляют до конечной концентрации 2 М мочевины на 50 мМ HEPES (pH 8,5).
- Добавьте трипсин в каждый образец в соотношении белок:трипсин 50:1 (по весу) и выполняйте разложение при комнатной температуре в течение 3 часов или в течение ночи.
- Восстановление пептидов и алкилирование
- Добавьте свежеприготовленный раствор дитиотреитола (1 М DTT) до конечной концентрации 1 мМ и инкубируйте в течение 1 часа при комнатной температуре для уменьшения дисульфидных связей.
- Свежеприготовленный раствор йодоацетамида (1 М ИУК) добавить до конечной концентрации 10 мМ в течение 30 мин в темноте для алкилатирования остатков цистеина.
- Утолите непрореагировавшую IAA, добавив 1М DTT к конечной концентрации 30 мМ, и инкубируйте при комнатной температуре еще 30 минут.
- Изучите эффективность пищеварения
- Возьмите ~1 г каждого образца и обессолите с помощью наконечников для дозатора C18, покрытых смолой, в соответствии с протоколом производителя.
- Проанализируйте каждый образец с помощью короткого градиентного прогона LC-MS/MS (подробнее см. в шаге 5).
- Выполните поиск в базе данных по необработанным данным MS (подробнее см. в шаге 6). Рассчитайте процент идентифицированных пептидов с хотя бы одним сайтом расщепления трипсина. Процент обычно ниже 15%.
- Если процент больше 15%, добавьте в образцы дополнительный трипсин, чтобы повторить расщепление.
- После разложения подкислите образцы, добавив ТЖК до 0,5% (v/v). Проверьте pH с помощью полоски pH, чтобы убедиться, что pH меньше 3.
- Пептидное обессоливание
- Центрифугируйте подкисленные пептиды при давлении 21 000 x g в течение 10 минут. Переложите надосадочную жидкость в новую трубку.
- Обессоливающие колонны C18 (~25 мкл смолы) дважды промыть 250 мкл 100% метанола путем центрифугирования при 500 x g в течение 30 с.
ПРИМЕЧАНИЕ: Чтобы уменьшить потери пептидов в процессе обессоливания выбирайте обессоливающие колонны со связующей способностью, соответствующей объему входа. - Дважды промойте колонки, используя 250 мкл элюирующего буфера (60% ACN, 0,1% TFA) центрифугированием при 500 x g в течение 30 с.
- Дважды уравновесьте колонки с помощью 250 мкл уравновешивающего и промывочного буфера (0,1% TFA) путем центрифугирования при 500 x g в течение 30 с.
- Загрузите образцы на предварительно уравновешенные колонны. Дайте образцам привязаться к столбцам, вращая при 100 x g в течение 6 минут. Убедитесь, что весь раствор прошел через колонну.
- Трижды промыть колонки 250 мкл уравновешивающего вещества и промыть буфер центрифугированием при 500 x g в течение 30 с.
- Разбавьте пептиды, добавив 125 мкл элюирующего буфера в каждую колонку и вращая при 100 x g в течение 3 мин. Убедитесь, что столбец не содержит остатков раствора.
- Высушите элюированные пептиды в вакуумном концентраторе и храните пептиды при температуре -80 °C для будущей маркировки TMT.
3. Мечение пептидов TMT16, тест на эффективность мечения, объединение образцов и обессоливание меченых пептидов
- Мечение пептидов TMT16
- Ресуспендируйте каждый образец обессоленного пептида в 50 мкл 50 мМ HEPES (pH 8,5) путем многократного вортексирования или ультразвукового растворения с последующим использованием полоски pH для проверки pH.
ПРИМЕЧАНИЕ: Образец может быть кислым, если он не высушен полностью перед маркировкой, что отрицательно влияет на эффективность маркировки. Убедитесь, что pH находится в диапазоне от 7 до 8. - Возьмите ~1 г немеченых пептидов из каждого образца в качестве отрицательного контроля для теста эффективности мечения TMT.
- Растворите реагенты TMT16 в безводном АКН. Проведите реакцию мечения, добавив реагенты в соотношении ТМТ:белок 1,5:1 (масса/масса) и инкубируя при комнатной температуре в течение 30 минут.
ПРИМЕЧАНИЕ: Соотношение ТМТ:белок, используемое для ТМТ16, на 50% выше, чем соотношение, используемое для ТМТ11. Это небольшое расхождение может быть связано с тем, что молекулярная масса TMT16 больше (в 1,2 раза), чем у реагентов TMT11. Количество белка оценивается по образцам без учета потерь при обессоливании.
- Ресуспендируйте каждый образец обессоленного пептида в 50 мкл 50 мМ HEPES (pH 8,5) путем многократного вортексирования или ультразвукового растворения с последующим использованием полоски pH для проверки pH.
- Тест на эффективность маркировки
- Возьмите ~1 г меченых пептидов из каждого образца для теста эффективности мечения. Поместите оставшиеся образцы при температуру -80 °C, не гася реакцию.
- Обессолите ~1 мкг каждого из меченых и немеченных TMT16 образцов наконечниками для дозаторов, покрытыми смолой C18, в соответствии с протоколом производителя.
- Анализируйте образцы с помощью ЖХ-МС/МС (см. раздел 5, за исключением того случая, когда градиент составляет 10 минут).
- Оцените эффективность мечения, проанализировав снижение интенсивности MS1 немеченных пептидов между немечеными и мечеными образцами. Выберите от 6 до 10 различных пептидов, чтобы проверить эффективность мечения и убедиться, что все пептиды помечены. Для полной мечения немеченые пептиды не наблюдаются.
ПРИМЕЧАНИЕ: Важно обеспечить полную маркировку всех образцов для последующей точной идентификации белка и количественного определения. - Если мечение не завершено, добавьте дополнительные реагенты TMT для мечения оставшихся пептидов и еще раз проверьте эффективность мечения перед закалкой. После того, как образец будет полностью помечен, погасите реакцию при комнатной температуре, добавив гидроксиламин до конечной концентрации 0,5%, и инкубируйте при комнатной температуре в течение 15 минут.
- Объединение проб и обессоливание
- Объедините половину каждого образца, меченного TMT, чтобы получить смесь.
- Возьмите 1 г смеси и обессолите с помощью наконечников пипеток C18, покрытых смолой, затем проанализируйте с помощью ЖХ-МС/МС с использованием короткого градиента (~30 минут).
- Рассчитайте относительную концентрацию, используя среднюю интенсивность каждого иона репортера TMT16 и сравнив расхождения между 16 каналами. Чтобы добиться равномерного перемешивания каждого канала, добавьте в смесь оставшиеся образцы с маркировкой TMT в соответствии с рассчитанной средней интенсивностью. Повторяйте корректировку до тех пор, пока все образцы не будут одинаково смешаны. Репрезентативные данные, показывающие процесс объединения выборки, приведены в таблице 1.
ПРИМЕЧАНИЕ: Поскольку ошибки пипетирования могут повлиять на точность концентраций и количественное определение белка, важно обеспечить правильное объединение количества. Расхождения в интенсивности между 16 образцами должны составлять менее 5%.
- Обессоливание меченых пептидов
Примечание: Поскольку фоновые производные в реакции мечения TMT16 (например, TMTpro-NHOH в результате реакции гашения гидроксиламина и TMTpro-OH в результате гидроксилирования TMT) являются гидрофобными, для образцов, меченных TMT16, для эффективного удаления производных используют обширные условия промывки. Используется добавление 5% ACN в обычный буфер для стирки (0,1% TFA) и 10-кратный объем буфера для стирки.- Подкислите объединенный образец, добавив 10% ТЖК к pH < 3.
- Центрифугируйте объединенный образец при давлении 21 000 x g в течение 10 минут и поместите надосадочную жидкость в новую пробирку.
- Высушите образец с помощью вакуумного концентратора для удаления ACN.
- Предварительно подготовьте твердофазный экстракционный картридж, содержащий колонку сорбента 50 мг, промыв колонку 2 мл 100% метанола, затем 2 мл элюирующего буфера (60% ACN плюс 0,1% TFA) и, наконец, 2 мл промывочного буфера (0,1% TFA).
- Загрузите образец в столбец. Отрегулируйте скорость потока до ~100 μл/мин, чтобы обеспечить полную степень связывания пептида. Сохраните поток.
- Промойте колонку три раза с помощью буфера для промывки объемом 1 мл.
- Элюируйте пептиды с 1 мл элюирующего буфера.
- Высушите элюированные пептиды в вакуумном концентраторе и храните пептиды при температуре -80 °C для дальнейшего фракционирования.
4. Базовое предварительное фракционирование pH LC в автономном режиме
- Подготовка системы фракционирования
- Подготовьте буфер A (10 мМ формиат аммония, pH 8,0) и буфер B (10 мМ формиат аммония, 90% ACN, pH 8,0) для высокопроизводительной системы LC с микролитровым потоком.
- Установите колонку ВЭЖХ, содержащую мостиковые гибридные частицы этилена (размер частиц 3,5 мкм, 4,6 мм × 25 см) в высокопроизводительной системе LC с микролитровым потоком для фракционирования.
- Установите контур отбора проб объемом 100 мкл и последовательно промойте контур 300 мкл метанола, воды и буфера А.
- Используйте 100 мкл соотношения 1:1:1:1 изопропанол : метанол : ацетонитрил : вода для промывки колонки. Затем дополнительно уравновесить столбик в течение 0,5 ч в 95% буфера А.
- Подготовка образцов
- Растворите объединенный и обессоленный образец TMT16 в 70 мкл буфера А. Убедитесь, что pH образца составляет ~ 8,0. Если все еще кислый, отрегулируйте pH до 8,0, используя 28% гидроксид аммония (NH4OH).
ПРИМЕЧАНИЕ: Во избежание потери образца объем образца должен составлять менее 70% от объема контура. - Центрифугируйте образец при давлении 21 000 x g в течение 10 минут, чтобы удалить осадки.
- Растворите объединенный и обессоленный образец TMT16 в 70 мкл буфера А. Убедитесь, что pH образца составляет ~ 8,0. Если все еще кислый, отрегулируйте pH до 8,0, используя 28% гидроксид аммония (NH4OH).
- Фракционирование и конкатенация
ПРИМЕЧАНИЕ: Перед фактическим фракционированием образца настоятельно рекомендуется провести пилотный эксперимент, чтобы убедиться в хорошем состоянии системы LC. Это можно сделать с небольшим количеством фактического образца (~5%) или с помощью смеси пептидов, не меченной TMT.- Вводят образец и фракционируют его по следующему градиенту: 5% буфер В в течение 10 мин, 5–15% буфер В в течение 2 мин, 15–45% буфер В в течение 148 мин и 45–95% буфер В в течение 5 мин. Используйте расход 0,4 мл/мин.
- Настройте коллектор фракций на сбор дробей каждые 1 мин и объедините 160 дробей обратно в 40 дробей за 4 цикла.
ПРИМЕЧАНИЕ: Конкатенация осуществляется путем объединения ранних, средних и поздних фракций LC, элюированных из внутренних органов того же времени, в конкатенированную фракцию. Конкатенированные дроби имеют небольшое перекрытие в первом измерении LC, что увеличивает эффективное использование окна элюирования во втором измерении LC. Кроме того, благодаря нескольким раундам конкатенации пептиды могут быть равномерно распределены по всем конкатенированным фракциям. Показано, что такой подход увеличивает покрытие протеома по сравнению с анализом отдельных фракций35,36. - Высушите все конкатенированные фракции в вакуумном концентраторе и храните высушенные образцы при температуре -80 °C для дальнейшего анализа ЖХ-МС/МС.
5. Кислотный pH RPLC-MS/MS анализ
- Подготовка кислотной pH RPLC-MS/MS системы
- Упакуйте пустую колонку (внутренний диаметр 75 мкм с концевым отверстием 15 мкм) смолой C18 толщиной 1,9 мкм до 10-15 см в длину.
- Нагрейте колонну до 65 °C с помощью нагревателя для производства бабочек, чтобы уменьшить противодавление.
- Тщательно промойте колонку 95% буфером В (3% диметилсульфоксида, 0,2% муравьиной кислоты и 67% ACN). Затем полностью уравновесьте колонку в 95% буфере А (3% диметилсульфоксида и 0,2% муравьиной кислоты).
- Проверьте производительность системы ЖХ-МС/МС, запустив 100 нг пептидов мозга крысы или пептидов BSA перед анализом экспериментальных образцов.
- ЖХ-МС/МС анализ конкатенированных фракций
- Восстановите высушенные пептиды из основных фракций pH в 5% FA и центрифугируйте при 21 000 × г в течение 5 минут. Перенесите надосадочную жидкость каждого образца во вкладыш для флакона с ВЭЖХ.
- Загрузите в колонку ~1 мкг пептидов каждой фракции. Пептиды элюируют со скоростью потока 0,25 мкл/мин с 60-минутным градиентом 18–45% буфера В.
ПРИМЕЧАНИЕ: Чтобы получить высокие идентификационные номера, выполните одну дробь и скорректируйте градиент для остальных дробей на основе первого прогона. Наилучший градиент должен иметь равномерно распределенные пептиды по всему градиенту (рисунок 4А). - Работа масс-спектрометра со следующими параметрами для анализа образцов, меченных TMT16: сканирование MS1 (полный диапазон сканирования MS: 450-1600 м/з; Разрешение Orbitrap: 60 000; автоматическая регулировка усиления цели: 1 x 106; максимальное время ионирования: 50 мс) и 20 зависящих от данных сканирований MS2 (разрешение Orbitrap: 60 000; цель АРУ: 1 x 105; максимальное время иона: 110 мс; Нормированная энергия столкновения HCD: 32%; Изоляционное окно: 1,0 м/з; Смещение изоляции: 0,2 м/з; динамическое исключение: 10 с).
ПРИМЕЧАНИЕ: Используемые здесь параметры оптимизированы на одном типе масс-спектрометра (см. Таблицу материалов). Для различных приборов MS пользователи должны точно настроить параметры инструмента для достижения высококачественных результатов. Одной из настроек является мониторинг нормализованной энергии столкновения HCD, так как оптимальная энергия может варьироваться в зависимости от прибора, а также между TMT11 и TMT16.
6. Обработка данных
ПРИМЕЧАНИЕ: Анализ данных проводился с использованием программного комплекса JUMP 37,38,39, включающего гибридную поисковую систему базы данных (на основе шаблонов и тегов), фильтрующее программное обеспечение, которое контролирует частоту ложных обнаружений (FDR) идентифицированных пептидов/белков, и программное обеспечение для количественной оценки наборов данных TMT. В зависимости от ситуации пользователя, анализ данных может быть выполнен с помощью других коммерческих или свободно доступных программ.
- Поиск по базе данных
- Преобразуйте файлы .raw из прибора MS в файлы .mzXML и выполните поиск спектров MS2 по неизбыточной базе данных 40-мишени-приманки, созданной из последовательностей белков человека UniProt (или другой подходящей видоспецифичной базе данных) для расчета FDR идентифицированных белков.
Примечание: Сгенерируйте неизбыточную базу данных, комбинируя белковые последовательности из баз данных Swiss-Prot и TrEMBL. Можно также добавить специализированные белковые последовательности, не содержащиеся в этих справочных базах данных, включая белки, расщепленные протеазы, белки с однонуклеотидными полиморфизмами и общие загрязнители. - Проводите поиск по следующим параметрам. Допуск по массе прекурсора: 10 ppm; допуск по массе ионов продукта: 15 ppm; Максимальное количество пропущенных спайвений: 2; Максимальное количество модификаций сайтов: 3; статические модификации: 304.20715 Da для меток TMT16 на остатках Lys и N termini, 57.02146 Da для карбамидметилирования на остатках Cys; динамическая модификация: 15.99492 Да для окисления на Met.
- Преобразуйте файлы .raw из прибора MS в файлы .mzXML и выполните поиск спектров MS2 по неизбыточной базе данных 40-мишени-приманки, созданной из последовательностей белков человека UniProt (или другой подходящей видоспецифичной базе данных) для расчета FDR идентифицированных белков.
- Фильтрация результатов поиска
- Отфильтруйте полученные совпадения пептидного спектра (PSM) по длине пептида (>6 аминокислот), точности массы иона-предшественника и оценкам соответствия на основе JUMP (Jscore и ΔJn). Затем пептиды группируются по длине пептида, триптическим концам, модификациям, пропущенным сайтам расщепления и зарядовому состоянию.
- Отфильтруйте данные с помощью соответствующих оценок, чтобы достичь FDR ниже 1% на уровне белка (анализ цельного протеома) или пептида (анализ фосфопротеома).
Примечание: Если на этапах фильтрации отсутствуют пептиды/белки положительного контроля, то FDR может быть увеличен до разумного уровня, чтобы эти пептиды/белки могли быть спасены. - Для пептидов, общих для более чем одного члена семейства белков, сгруппируйте совместимых членов в одну группу.
Примечание: Согласно правилу экономии, группа представлена гомологичным белком с наибольшим числом общих пептидов и другими белками, соответствующими уникальным пептидам.
- Количественное определение белка
- Количественное определение белков с помощью встроенной программы статистического программного обеспечения для суммирования интенсивностей ионов репортеров TMT по всем согласованным PSM.
- Извлекайте интенсивности репортерных ионов TMT из каждого принятого PSM и корректируйте исходные интенсивности в соответствии с изотопным распределением каждого мечающего реагента (например, TMT16-126 генерирует 92,6%, 7,2% и 0,2% 126, 127C и 128C m/z ионов соответственно) и отфильтровывайте низкоинтенсивные и/или сильно зашумленные PSM на основе заданных пользователем пороговых значений. Нормализация количественных данных с использованием усеченных средних (или медианных) интенсивностей образцов для коррекции смещения нагрузки.
- Для каждого идентифицированного белка рассчитайте среднецентрированные интенсивности по выборкам (т.е. относительные интенсивности) совпадающих PSM и суммируйте относительные интенсивности PSM, взяв среднее значение по выборке. Преобразуйте относительные сигналы в абсолютные, умножив общую среднюю интенсивность трех наиболее распространенных совпадающих PSM.
- Корректировка количественной интерференции с использованием ранее описанного подхода37 к коррекции ионов y1, который предполагает, что интенсивность ионов y1 коррелирует с интенсивностью ионов репортера. Оценивая линейную зависимость между y1 и интенсивностью репортерных ионов при чистых сканированиях, выводится и корректируется уровень интерференции от загрязненной интенсивности ионов y1 при зашумленном сканировании.
Примечание: Для меченых ТМТ триптических пептидов остатки K-TMT и R представляют собой два репрезентативных иона y1 (376,27574 Да и 175,11895 Да, соответственно) в спектре MS2. Если обнаружен только один ион y1 и он согласуется с идентифицированным пептидом, то MS2 считается чистым сканированием. Если обнаружены оба иона y1, то MS2 считается зашумленным сканированием. - Перенесите количественные значения белка в таблицу для дальнейшего анализа. Используйте неконтролируемые методы анализа данных, такие как PCA или кластерный анализ, для изучения распределения выборок. Для идентификации дифференциально экспрессируемых белков используют статистические методы, такие как t-критерий и дисперсионный анализ (ANOVA).
7. Валидация данных MS
ПРИМЕЧАНИЕ: Прежде чем проводить трудоемкие биологические эксперименты, используйте по крайней мере один метод валидации для оценки качества данных МС.
- Вручную изучите спектры МС/МС интересующих белков для валидации пептидной последовательности и интенсивности репортерных ионов ТМТ.
- Используйте подходы, основанные на антителах (например, вестерн-блоттинг или иммуногистохимический анализ) для проверки изменений уровня белка. Чтобы подтвердить наличие нативных пептидов, используйте синтетические пептиды в качестве внутренних стандартов. Спектры МС/МС пептидов и время удержания во время ЖХ-МС/МС должны быть идентичны при одних и тех же условиях.
- Используйте целевой подход к РС для проверки изменений белка.
Результаты
Протокол для недавно разработанного TMT16, включая реакцию мечения, обессоливание и условия LC-MS, был систематически оптимизирован41. Кроме того, мы напрямую сравнили 11-плексный и 16-плексный методы, используя их для анализа одних и тех же образцов болезни Альцгеймера41. После оптимизации ключевых параметров для TMT16 оба метода TMT11 и TMT16 обеспечивают одинаковое покрытие протеома, идентификацию и количественную оценку > 100 000 пептидов в > 10 000 белков человека.
Поскольку реагенты TMT16 более гидрофобны, чем реагенты TMT11, меченые TMT16 пептиды, вероятно, будут более гидрофобными, чем меченые TMT11 пептиды, что может объяснять различное время удержания (RT) в RPLC. Таким образом, мы оценили влияние TMT16 на пептид RT по сравнению с TMT11 путем анализа смеси меченых TMT11 и TMT16 пептидов с использованием LC-MS/MS. Мы обнаружили, что TMT16 оказывает значительное влияние на RT к пептидам со средней гидрофобностью, но мало влияет на пептиды с чрезвычайно высокой или низкой гидрофобностью. Таким образом, аналогичные начальные и конечные концентрации буфера B в градиенте LC могут быть использованы для различных пептидов, меченных TMT.
Затем мы оптимизировали онлайн-градиент RPLC для образца, меченного TMT16. Градиент для TMT16 очень похож на градиент для TMT11. Процентное соотношение начального и конечного буфера B одинаково (например, от 18% до 45%). Но мы заметили, что количество идентифицированных пептидов в TMT16 быстро снижается на уровне около 40% буфера B при использовании того же градиента, который используется для TMT11. Таким образом, мы немного сократили время градиента между 40% и 45%. Мы также внесли незначительные корректировки в этот градиент для разных фракций и разных образцов. После оптимизации градиента идентифицированные пептиды были равномерно распределены по всему градиенту (рис. 4А).
Чтобы максимизировать количество белков, идентифицированных и точно количественно определенных с помощью метода TMT16, мы оптимизировали нормализованную энергию столкновения (NCE) для образцов, меченных TMT16, в нашем предыдущем отчете41. Различные НКЭ (от 20% до 40%) испытывали на масс-спектрометре во время прогонов ЖХ-МС/МС. Уравновешивая количество идентификаций белков и интенсивность репортерных ионов, NCE 30-32,5% был выбран в качестве оптимальной энергии столкновения HCD для образцов, меченных TMT16.
Соотношение-сжатие, вызванное соэлюированными интерферирующими ионами, является ограничением методов изобарического мечения для количественного определения белка. Ранее опубликованное исследование с использованием метода TMT11 показывает, что сжатие коэффициента может быть практически устранено за счет обширного предварительного фракционирования ЛК, оптимизированных настроек МСи стратегий коррекции данных после МС. Мы использовали эти стратегии, включая экстенсивное фракционирование перед МС (40 основных фракций pH LC), применение узкого изоляционного окна (1 м/z) в условиях МС и коррекцию ионов y1 в протеомных анализах TMT11 и TMT16 одних и тех же образцов. Изучив кривую корреляции изменения складки белка между наборами данных TMT11 и TMT16, мы обнаружили, что наклон был очень близок к 1, что указывает на то, что отношение сжатия в TMT16 не было заметно выше, чем в TMT11 в наших экспериментальных условиях41. Получены согласованные результаты, согласно которым отношение сжатия не имеет разницы при увеличении уровня мультиплексирования с 11 до 1613,45. Таким образом, ранее опубликованные стратегии могут быть использованы для смягчения сжатия отношений, тем самым значительно повышая точность количественной оценки 27,37,44,46.
Наконец, мы сравнили количество ПСМ, уникальных пептидов и уникальных белков, количественно определенных в образцах, меченных TMT11 и TMT16 (рис. 4B). Результаты показывают, что ПСМ обоих методов сопоставимы; тем не менее, количественно определенные белки и пептиды несколько ниже в способе TMT16, что согласуется с другими отчетами12,13. Наши результаты показывают, что усовершенствования в процессе TMT16 наряду с использованием оптимизированных параметров LC-MS обеспечивают высокопроизводительное глубокое протеомное профилирование биологических образцов.
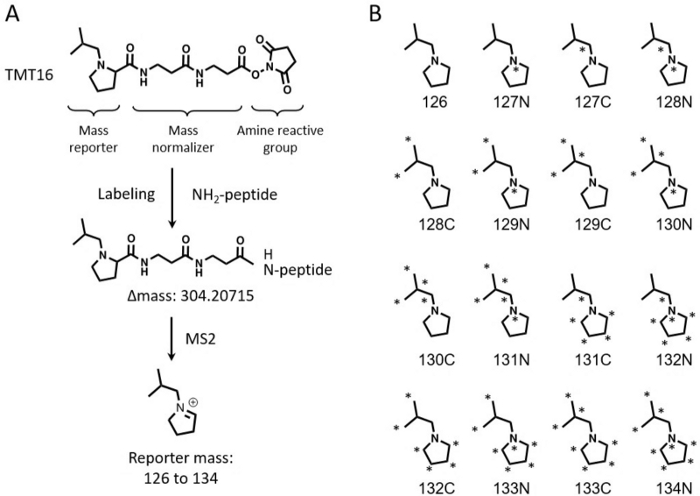
Иллюстрация 1: Структура 16-плексного реагента TMT. (A) Показана структура 16-плексного реагента TMT, процесс мечения, сдвиг массы после мечения и масса репортерного иона. (B) Тяжелые меченые изотопами структуры репортерных ионов реагентов TMT16. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
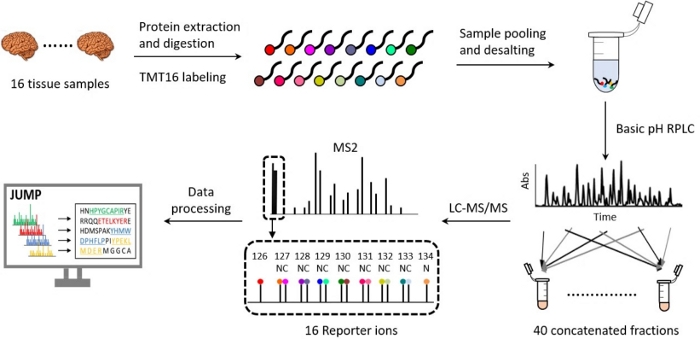
Рисунок 2: Схема протеомного профилирования с помощью 16-плексного TMT-LC/LC-MS/MS. Белок, извлеченный из 16 образцов биологических тканей, был расщеплен и помечен 16 различными метками TMT. Образцы из 16 каналов объединяются поровну, а смесь фракционируется и объединяется в 40 фракций с помощью автономной базовой обратной фазовой жидкостной хроматографии (RPLC) с обратной фазой pH. Каждая фракция дополнительно анализируется с помощью кислотного RPLC в сочетании с масс-спектрометрией высокого разрешения. Были обработаны необработанные файлы MS/MS. Картина тканей мозга приведена из Medium.com с некоторыми изменениями. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
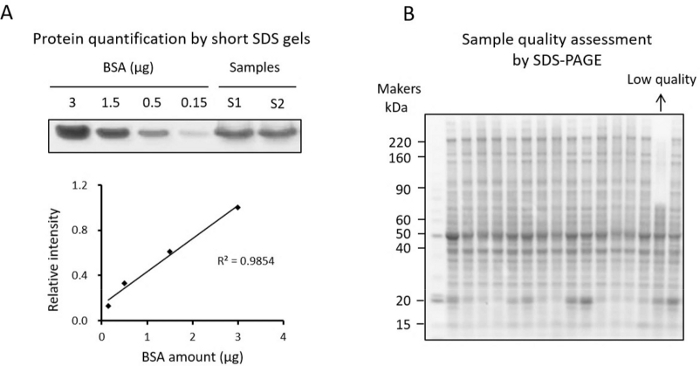
Рисунок 3: Контроль качества белка. (A) Количественное определение экстрагированного белка из ткани на коротком геле SDS с BSA в качестве стандарта. Стандартная кривая отображает концентрацию BSA и интенсивность полосы белка, окрашенного по методу Кумасси, используемую для количественного определения. (B) Гель SDS, используемый для анализа качества белка. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
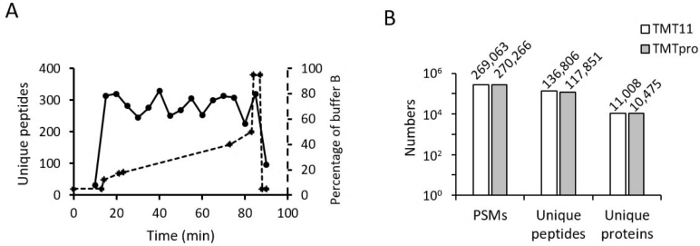
Рисунок 4: Репрезентативные результаты. (А) Распределение пептидов в кислых ЛХ. Оптимизированный градиент буфера B после коррекции мертвого объема выравнивается на этом же графике. (B) Гистограмма показывает количество количественно определенных PSM, уникального пептида и уникального белка в методах TMT11 и TMT16. Пожалуйста, нажмите здесь, чтобы просмотреть увеличенную версию этой цифры.
| 1-й (50 μл, использовать 50% в первой смеси) | 2-й (отрегулируйте смесь и сэкономьте 10%) | 3-й (окончательная корректировка) | |||||||||||
| Каналами | Журналистам | Объем смеси (μл) | Интенсивность (единицы) | (единица/мкл) | Ожидаемая интенсивность (единицы) | Добавленный объем (μл) | Общий объем (μл) | Интенсивность (единицы) | (единица/мкл) | Ожидаемая интенсивность (единицы) | Добавленный объем (μл) | Общий объем (μл) | Интенсивность (единицы) |
| 1 | СИГ126 | 25 | 94.7 | 3.8 | 122.1 | 7.2 | 32.2 | 99.6 | 3.1 | 105.3 | 1.8 | 34.1 | 100 |
| 2 | сиг127Н | 25 | 83 | 3.3 | 122.1 | 11.8 | 36.8 | 101.1 | 2.7 | 105.3 | 1.5 | 38.3 | 98 |
| 3 | sig127C | 25 | 86 | 3.4 | 122.1 | 10.5 | 35.5 | 99.9 | 2.8 | 105.3 | 1.9 | 37.4 | 99.9 |
| 4 | sig128N | 25 | 103.9 | 4.2 | 122.1 | 4.4 | 29.4 | 102.1 | 3.5 | 105.3 | 0.9 | 30.3 | 97.2 |
| 5 | sig128C | 25 | 90.8 | 3.6 | 122.1 | 8.6 | 33.6 | 103.3 | 3.1 | 105.3 | 0.7 | 34.3 | 98.3 |
| 6 | сиг129Н | 25 | 82.8 | 3.3 | 122.1 | 11.9 | 36.9 | 99 | 2.7 | 105.3 | 2.4 | 39.3 | 98.7 |
| 7 | sig129C | 25 | 101.3 | 4.1 | 122.1 | 5.1 | 30.1 | 98.5 | 3.3 | 105.3 | 2.1 | 32.2 | 102.1 |
| 8 | сиг130Н | 25 | 98.9 | 4 | 122.1 | 5.9 | 30.9 | 100.1 | 3.2 | 105.3 | 1.6 | 32.5 | 99.7 |
| 9 | сиг130К | 25 | 86.3 | 3.5 | 122.1 | 10.4 | 35.4 | 96 | 2.7 | 105.3 | 3.4 | 38.8 | 99.3 |
| 10 | сиг131Н | 25 | 87 | 3.5 | 122.1 | 10.1 | 35.1 | 95.3 | 2.7 | 105.3 | 3.7 | 38.8 | 101.5 |
| 11 | sig131C | 25 | 119.1 | 4.8 | 122.1 | 0.6 | 25.6 | 100.9 | 3.9 | 105.3 | 1.1 | 26.7 | 100.2 |
| 12 | сиг132Н | 25 | 86 | 3.4 | 122.1 | 10.5 | 35.5 | 95.3 | 2.7 | 105.3 | 3.7 | 39.2 | 99.6 |
| 13 | сиг132К | 25 | 119.1 | 4.8 | 122.1 | 0.6 | 25.6 | 101.2 | 3.9 | 105.3 | 1 | 26.7 | 100 |
| 14 | сиг133Н | 25 | 116.3 | 4.7 | 122.1 | 1.3 | 26.3 | 99.9 | 3.8 | 105.3 | 1.4 | 27.7 | 100.9 |
| 15 | sig133C | 25 | 122.1 | 4.9 | 122.1 | 0 | 25 | 101 | 4 | 105.3 | 1.1 | 26.1 | 101.9 |
| 16 | сиг134Н | 25 | 121.3 | 4.9 | 122.1 | 0.2 | 25.2 | 105.3 | 4.2 | 105.3 | 0 | 25.2 | 101.3 |
Таблица 1: Репрезентативные данные, показывающие процесс объединения образцов на шаге 3.3.
Обсуждение
Оптимизированный протокол для глубокого протеомного профилирования на основе TMT16 был успешно реализован в более ранних публикациях 12,13,41. С помощью этого протокола более 10 000 уникальных белков из 16 различных образцов могут быть регулярно количественно определены в одном эксперименте с высокой точностью.
Для получения качественных результатов важно уделять внимание критическим шагам на протяжении всего протокола. В дополнение ко всем шагам контроля качества, рассмотренным в нашей предыдущей статье27, мы включаем дополнительные важные шаги, специфичные для процесса TMT16. Эти шаги важны для обеспечения успешного эксперимента. Например, производные реакции TMT (например, TMTpro-NHOH в результате реакции гашения гидроксиламина и TMTpro-OH в результате гидроксилирования TMT) детектируются в виде заметных одиночно заряженных ионов перед обессоливанием с помощью анализа LC-MS/MS. Очень важно удалить их на этапе обессоливания . Мы протестировали различные условия обессоливания и обнаружили, что добавление 5% ACN в обычный буфер для промывки в сочетании с 10 × объемов промывки в три раза эффективно удаляет производные41. Кроме того, TMT16 имеет увеличенную массу по сравнению с TMT11, поэтому полный диапазон сканирования начинается с более высокого m/z (450 вместо 410) для образцов, меченных TMT16. Более того, поскольку оптимальная энергия столкновения для пептида зависит от массы заряда и зарядового состояния предшественника иона21, пептиды, помеченные различными химическими метками, могут иметь различные оптимальные энергии столкновения. Для TMT16 оптимальна энергия столкновения 30-32,5% для TMT16, что немного ниже, чем у TMT11.
Изобарическая маркировка является мощным методом, обеспечивающим высокую способность к мультиплексированию. Несмотря на то, что другие методы, такие как SILAC (мечение стабильных изотопов аминокислотами в клеточной культуре)47 и метод без меток, обеспечивают альтернативные стратегии количественного определениябелков48, они страдают от низкой пропускной способности. Теоретически TMT16 может количественно определять белки в 16 различных биологических образцах. Тем не менее, гораздо чаще некоторые из этих каналов используются в качестве биологических репликаций, что обеспечивает большую статистическую мощность и помогает генерировать надежные данные. Использование репликатов или даже трипликатов очень важно, особенно в системах, где ожидаемое изменение концентрации белка является номинальным. Важно понимать биологию системы, прежде чем планировать эксперимент, чтобы включить в него соответствующее количество реплик. Некоторые биологические системы не идеально подходят для некоторых этапов контроля качества, описанных в этом протоколе. Тест на коэффициент премикса не используется при использовании образцов иммунопреципитации для протокола из-за большого процента ожидаемых изменений белков. В этих случаях результаты будут искажены при тесте премикса. Это также верно в тех случаях, когда ожидается, что по крайней мере 1 из 10 образцов будет сильно различаться по экспрессии белка (пустой вектор, ингибирование протеасом и т. д.). Также предлагается использовать канал TMT в качестве «внутреннего эталона», который затем может быть использован для объединения нескольких партий экспериментов TMT1649.
Этот протокол может быть использован для высокопроизводительного глобального протеомного профилирования сложных биологических образцов, изучения дифференциально экспрессируемых белков и клеточных сигнальных путей, а также для понимания биологии заболевания. Кроме того, с небольшими изменениями в протоколе, он может быть использован для изучения посттрансляционных модификаций, таких как фосфорилирование, убиквитинирование, метилирование и ацетилирование. Применение комплексного подхода, сочетающего исчерпывающий крупномасштабный протеомный анализ с другими омиксными системами, такими как геномика, транскриптомика и метаболомика, может дать понимание, позволяющее расширить понимание сложных биологических систем 30,50.
Раскрытие информации
Авторам нечего раскрывать.
Благодарности
Эта работа была частично поддержана Национальными институтами здравоохранения (R01GM114260, R01AG047928, R01AG053987, RF1AG064909 и U54NS110435) и ALSAC (Американо-ливанско-сирийская ассоциированная благотворительная организация). Анализ на РС был проведен в Центре протеомики и метаболомики Детской исследовательской больницы Святого Иуды, который частично поддержан грантом поддержки онкологического центра NIH (P30CA021765). Ответственность за содержание лежит исключительно на авторах и не обязательно отражает официальную точку зрения Национальных институтов здравоохранения.
Материалы
| Name | Company | Catalog Number | Comments |
| 10% Criterion TGX Precast Midi Protein Gel | Biorad | 5671035 | |
| 10X TGS (Tris/Glycine/SDS) Buffer | BioRad | 161-0772 | |
| 4–20% Criterion TGX Precast Midi Protein Gel | Biorad | 5671095 | |
| 50% Hydroxylamine | Thermo Scientific | 90115 | |
| 6 X SDS Sample Loading Buffer | Boston Bioproducts Inc | BP-111R | |
| Ammonium Formate (NH4COOH) | Sigma | 70221-25G-F | |
| Ammonium Hydroxide, 28% | Sigma | 338818-100ml | |
| Bullet Blender | Next Advance | BB24-AU | |
| Butterfly Portfolio Heater | Phoenix S&T | PST-BPH-20 | |
| C18 Ziptips | Harvard Apparatus | 74-4607 | Used for desalting |
| Dithiothreitol (DTT) | Sigma | D5545 | |
| DMSO | Sigma | 41648 | |
| Formic Acid | Sigma | 94318 | |
| Fraction Collector | Gilson | FC203B | |
| Gel Code Blue Stain Reagent | Thermo | 24592 | |
| Glass Beads | Next Advance | GB05 | |
| HEPES | Sigma | H3375 | |
| HPLC Grade Acetonitrile | Burdick & Jackson | AH015-4 | |
| HPLC Grade Water | Burdick & Jackson | AH365-4 | |
| Iodoacetamide (IAA) | Sigma | I6125 | |
| Lys-C | Wako | 125-05061 | |
| Mass Spectrometer | Thermo Scientific | Q Exactive HF | |
| MassPrep BSA Digestion Standard | Waters | 186002329 | |
| Methanol | Burdick & Jackson | AH230-4 | |
| Nanoflow UPLC | Thermo Scientific | Ultimate 3000 | |
| Pierce BCA Protein Assay kit | Thermo Scientific | 23225 | |
| ReproSil-Pur C18 resin, 1.9um | Dr. Maisch GmbH | r119.aq.0003 | |
| Self-Pack Columns | New Objective | PF360-75-15-N-5 | |
| SepPak 1cc 50mg | Waters | WAT054960 | Used for desalting |
| Sodium Deoxycholate | Sigma | 30970 | |
| Speedvac | Thermo Scientific | SPD11V | |
| TMTpro 16plex Label Reagent Set | Thermo Scientific | A44520 | |
| Trifluoroacetic Acid (TFA) | Applied Biosystems | 400003 | |
| Trypsin | Promega | V511C | |
| Ultra-micro Spin Column,C18 | Harvard apparatus | 74-7206 | Used for desalting |
| Urea | Sigma | U5378 | |
| Xbridge Column C18 column | Waters | 186003943 | Used for basic pH LC |
Ссылки
- Levy, M. J., Washburn, M. P., Florens, L. Probing the sensitivity of the orbitrap lumos mass spectrometer using a standard reference protein in a complex background. Journal of Proteome Research. 17 (10), 3586-3592 (2018).
- Bekker-Jensen, D. B., et al. An optimized shotgun strategy for the rapid generation of comprehensive human proteomes. Cell Systems. 4 (6), 587-599 (2017).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Frost, D. C., Greer, T., Li, L. High-Resolution Enabled 12-Plex DiLeu Isobaric Tags for Quantitative Proteomics. Analytical Chemistry. 87 (3), 1646-1654 (2015).
- Moulder, R., Bhosale, S. D., Goodlett, D. R., Lahesmaa, R. Analysis of the plasma proteome using iTRAQ and TMT-based Isobaric labeling. Mass Spectrometry Reviews. 37 (5), 583-606 (2018).
- Wang, H., et al. Deep multiomics profiling of brain tumors identifies signaling networks downstream of cancer driver genes. Nature Communications. 10 (1), 3718(2019).
- Rauniyar, N., Yates, J. R. Isobaric labeling-based relative quantification in shotgun proteomics. Journal of Proteome Research. 13 (12), 5293-5309 (2014).
- Hogrebe, A., et al. Benchmarking common quantification strategies for large-scale phosphoproteomics. Nature Communications. 9 (1), 1045(2018).
- Dayon, L., et al. Relative quantification of proteins in human cerebrospinal fluids by MS/MS using 6-plex isobaric tags. Analytical Chemistry. 80 (8), 2921-2931 (2008).
- Stepanova, E., Gygi, S. P., Paulo, J. A. Filter-based protein digestion (FPD): A detergent-free and scaffold-based strategy for TMT workflows. Journal of Proteome Research. 17 (3), 1227-1234 (2018).
- McAlister, G. C., et al. Increasing the multiplexing capacity of TMTs using reporter ion isotopologues with isobaric masses. Analytical Chemistry. 84 (17), 7469-7478 (2012).
- Thompson, A., et al. TMTpro: Design, synthesis, and initial evaluation of a proline-based isobaric 16-plex tandem mass tag reagent set. Analytical Chemistry. 91 (24), 15941-15950 (2019).
- Li, J., et al. TMTpro reagents: a set of isobaric labeling mass tags enables simultaneous proteome-wide measurements across 16 samples. Nature Methods. 17 (4), 399-404 (2020).
- Arul, A. B., Robinson, R. A. S. Sample Multiplexing Strategies in Quantitative Proteomics. Analytical Chemistry. 91 (1), 178-189 (2019).
- Labib, M., Kelley, S. O. Single-cell analysis targeting the proteome. Nature Reviews Chemistry. 4 (3), 143-158 (2020).
- Ren, R. J., Dammer, E. B., Wang, G., Seyfried, N. T., Levey, A. I. Proteomics of protein post-translational modifications implicated in neurodegeneration. Translational Neurodegeneration. 3 (1), 23(2014).
- Pagel, O., Loroch, S., Sickmann, A., Zahedi, R. P. Current strategies and findings in clinically relevant post-translational modification-specific proteomics. Expert Review of Proteomics. 12 (3), 235-253 (2015).
- Mertins, P., et al. Reproducible workflow for multiplexed deep-scale proteome and phosphoproteome analysis of tumor tissues by liquid chromatography-mass spectrometry. Nature Protocols. 13 (7), 1632-1661 (2018).
- Aebersold, R., Mann, M. Mass-spectrometric exploration of proteome structure and function. Nature. 537 (7620), 347-355 (2016).
- Bai, B., et al. Deep multilayer brain proteomics identifies molecular networks in Alzheimer's disease progression. Neuron. 105 (6), 975-991 (2020).
- Kelstrup, C. D., et al. Rapid and deep proteomes by faster sequencing on a benchtop quadrupole ultra-high-field orbitrap mass spectrometer. Journal of Proteome Research. 13 (12), 6187-6195 (2014).
- Meier, F., et al. Online parallel accumulation - serial fragmentation (PASEF) with a novel trapped ion mobility mass spectrometer. Molecular & Cellular Proteomics. 17 (12), (2018).
- Schweppe, D. K., et al. Full-featured, real-time database searching platform enables fast and accurate multiplexed quantitative proteomics. Journal of Proteome Research. 19 (5), 2026-2034 (2020).
- Wang, H., et al. Systematic optimization of long gradient chromatography mass spectrometry for deep analysis of brain proteome. Journal of Proteome Research. 14 (2), 829-838 (2015).
- Dey, K. K., et al. Deep undepleted human serum proteome profiling toward biomarker discovery for Alzheimer's disease. Clinical Proteomics. 16, 16(2019).
- Bai, B., et al. Deep profiling of proteome and phosphoproteome by isobaric labeling, extensive liquid chromatography, and mass spectrometry. Methods in Enzymology. 585, 377-395 (2017).
- High, A. A., et al. Deep proteome profiling by isobaric labeling, extensive liquid chromatography, mass spectrometry, and software-assisted quantification. Journal of Visualized Experiments. (129), e56474(2017).
- Chick, J. M., et al. Defining the consequences of genetic variation on a proteome-wide scale. Nature. 534 (7608), 500-505 (2016).
- Wang, Z., et al. Quantitative phosphoproteomic analysis of the molecular substrates of sleep need. Nature. 558 (7710), 435-439 (2018).
- Tan, H., et al. Integrative proteomics and phosphoproteomics profiling reveals dynamic signaling networks and bioenergetics pathways underlying T cell activation. Immunity. 46 (3), 488-503 (2017).
- Eden, E., et al. Proteome Half-Life Dynamics in Living Human Cells. Science. 331 (6018), 764(2011).
- Kleiman, L. B., Maiwald, T., Conzelmann, H., Lauffenburger, D. A., Sorger, P. K. Rapid phospho-turnover by receptor tyrosine kinases impacts downstream signaling and drug binding. Molecular Cell. 43 (5), 723-737 (2011).
- Mertins, P., et al. Ischemia in tumors induces early and sustained phosphorylation changes in stress kinase pathways but does not affect global protein levels. Molecular & Cellular Proteomics. 13 (7), 1690(2014).
- Xu, P., Duong, D. M., Peng, J. Systematical optimization of reverse-phase chromatography for shotgun proteomics. Journal of Proteome Research. 8 (8), 3944-3950 (2009).
- Wang, Y., et al. Reversed-phase chromatography with multiple fraction concatenation strategy for proteome profiling of human MCF10A cells. Proteomics. 11 (10), 2019-2026 (2011).
- Yang, F., Shen, Y., Camp, D. G., Smith, R. D. High-pH reversed-phase chromatography with fraction concatenation for 2D proteomic analysis. Expert Review of Proteomics. 9 (2), 129-134 (2012).
- Niu, M., et al. Extensive peptide fractionation and y(1) ion-based interference detection method for enabling accurate quantification by isobaric labeling and mass spectrometry. Analytical Chemistry. 89 (1), 2956-2963 (2017).
- Wang, X., et al. A tag-based database search tool for peptide identification with high sensitivity and accuracy. Molecular & Cellular Proteomics. 13 (12), 3663(2014).
- Li, Y., et al. JUMPg: An integrative proteogenomics pipeline identifying unannotated proteins in human brain and cancer cells. Journal of Proteome Research. 15 (7), 2309-2320 (2016).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nature Methods. 4 (3), 207-214 (2007).
- Wang, Z., et al. 27-plex tandem mass tag mass spectrometry for profiling brain proteome in Alzheimer's disease. Analytical Chemistry. 92 (10), 7162-7170 (2020).
- Ow, S. Y., et al. iTRAQ underestimation in simple and complex mixtures: "The good, the bad and the ugly". Journal of Proteome Research. 8 (11), 5347-5355 (2009).
- Karp, N. A., et al. Addressing accuracy and precision issues in iTRAQ quantitation. Molecular & Cellular Proteomics. 9 (9), 1885-1897 (2010).
- Ting, L., Rad, R., Gygi, S. P., Haas, W. MS3 eliminates ratio distortion in isobaric multiplexed quantitative proteomics. Nature Methods. 8 (11), 937-940 (2011).
- Gygi, J. P., et al. A triple knockout isobaric-labeling quality control platform with an integrated online database search. Journal of The American Society for Mass Spectrometry. 31 (7), 1344-1349 (2020).
- Savitski, M. M., et al. Measuring and managing ratio compression for accurate iTRAQ/TMT quantification. Journal of Proteome Research. 12 (8), 3586-3598 (2013).
- Ong, S. E., et al. Stable isotope labeling by amino acids in cell culture, SILAC, as a simple and accurate approach to expression proteomics. Molecular & Cellular Proteomics. 1 (5), 376(2002).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513(2014).
- Brenes, A., Hukelmann, J., Bensaddek, D., Lamond, A. I. Multibatch TMT reveals false positives, batch effects and missing values. Molecular & Cellular Proteomics. 18 (10), 1967-1980 (2019).
- Yu, J., Peng, J., Chi, H. Systems immunology: Integrating multi-omics data to infer regulatory networks and hidden drivers of immunity. Current Opinion in Systems Biology. 15, 19-29 (2019).
Перепечатки и разрешения
Запросить разрешение на использование текста или рисунков этого JoVE статьи
Запросить разрешениеСмотреть дополнительные статьи
This article has been published
Video Coming Soon
Авторские права © 2025 MyJoVE Corporation. Все права защищены