Method Article
Profilage à haut débit et en profondeur du protéome par marquage de masse en tandem 16-plex couplé à la chromatographie bidimensionnelle et à la spectrométrie de masse
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Nous présentons ici un protocole optimisé à haut débit développé avec des réactifs de marquage de masse en tandem 16-plex, permettant le profilage quantitatif du protéome des échantillons biologiques. Le fractionnement de base du pH étendu et la LC-MS/MS haute résolution atténuent la compression du rapport et fournissent une couverture profonde du protéome.
Résumé
Le marquage isobare en tandem de masse (TMT) est largement utilisé en protéomique en raison de sa grande capacité de multiplexage et de sa couverture profonde du protéome. Récemment, une méthode TMT 16-plex élargie a été introduite, ce qui augmente encore le débit des études protéomiques. Dans ce manuscrit, nous présentons un protocole optimisé pour le profilage profond du protéome basé sur le TMT 16-plex, y compris la préparation d’échantillons de protéines, la digestion enzymatique, la réaction de marquage TMT, le fractionnement par chromatographie liquide en phase inverse (LC/LC) bidimensionnelle, la spectrométrie de masse en tandem (MS/MS) et le traitement des données informatiques. Les étapes cruciales du contrôle de la qualité et les améliorations du processus spécifiques à l’analyse TMT 16-plex sont mises en évidence. Ce processus multiplexé offre un outil puissant pour profiler une variété d’échantillons complexes tels que des cellules, des tissus et des échantillons cliniques. Plus de 10 000 protéines et modifications post-traductionnelles telles que la phosphorylation, la méthylation, l’acétylation et l’ubiquitination dans des échantillons biologiques très complexes provenant d’un maximum de 16 échantillons différents peuvent être quantifiées en une seule expérience, ce qui constitue un outil puissant pour la recherche fondamentale et clinique.
Introduction
Les développements rapides de la technologie de spectrométrie de masse ont permis d’obtenir une sensibilité élevée et une couverture protéomique profonde dans les applications de protéomique 1,2. Malgré ces développements, le multiplexage d’échantillons reste le goulot d’étranglement pour les chercheurs chargés de l’analyse d’une grande cohorte d’échantillons.
Les techniques de marquage isobare multiplexé sont largement utilisées pour la quantification relative à l’échelle du protéome de grands lots d’échantillons 3,4,5,6. La quantification basée sur les marqueurs de masse en tandem (TMT) est un choix populaire pour sa grande capacité de multiplexage 7,8. Les réactifs TMT ont été initialement lancés sous la forme d’un kit 6-plex capable de quantifier jusqu’à 6 échantillons simultanément9. Cette technologie a été étendue pour quantifier 10-11 échantillons10,11. Les réactifs TMTpro 16-plex récemment développés (appelés TMT16 ci-après) ont encore augmenté la capacité de multiplexage à 16 échantillons en une seule expérience12,13. Les réactifs TMT16 utilisent un groupe rapporteur à base de proline, tandis que le TMT 11-plex applique un groupe rapporteur dérivé de la diméthylpipéridine. Le TMT11 et le TMT16 utilisent tous deux le même groupe amine réactif, mais le groupe bilan massique du TMT16 est plus grand que celui du TMT11, ce qui permet à la combinaison de 8 isotopes stables C13 et N15 dans les ions rapporteurs d’obtenir 16 rapporteurs (Figure 1).
L’augmentation de la capacité de multiplexage fournit une plate-forme pour la conception d’expériences avec suffisamment de répétitions pour surmonter les défis statistiques14. De plus, les canaux supplémentaires dans le TMT 16-plex aident à réduire la quantité totale de matériel de départ par canal, ce qui peut aider au développement de la protéomique unicellulaire émergente15. La grande capacité de multiplexage sera également précieuse pour la quantification des modifications post-traductionnelles, qui nécessitent généralement de grandes quantités de matière première16,17.
Les flux de travail protéomiques utilisant la technologie TMT ont été rationalisés 18,19,20 et ont considérablement évolué au cours de la dernière décennie en termes de préparation d’échantillons, de séparation par chromatographie liquide, d’acquisition de données par spectrométrie de masse et d’analyse informatique 21,22,23,24,25,26. Notre précédent article donne un aperçu approfondi de la plate-forme TMT10-plex 27. Le protocole décrit ici présente une méthode détaillée et optimisée pour le TMT16, y compris l’extraction et la digestion des protéines, le marquage du TMT16, le regroupement et le dessalage des échantillons, le pH basique et le pH acide en phase inverse (RP) LC, la spectrométrie de masse haute résolution et le traitement des données (Figure 2). Le protocole met également en évidence les principales étapes de contrôle de la qualité qui ont été intégrées pour mener à bien une expérience de protéomique quantitative. Ce protocole peut être utilisé en routine pour identifier et quantifier plus de 10 000 protéines à haute reproductibilité, pour étudier les voies biologiques, les processus cellulaires et la progression de la maladie 20,28,29,30.
Protocole
Les tissus humains pour l’étude ont été obtenus avec l’approbation du programme de don de cerveau et de corps de l’Institut de recherche sur la santé Banner Sun.
1. Extraction de protéines à partir de tissus et contrôle de la qualité
REMARQUE : Pour réduire l’impact de la collecte d’échantillons sur le protéome, il est crucial de collecter des échantillons en un minimum de temps à basse température si possible31. Ceci est particulièrement important lors de l’analyse des modifications post-traductionnelles car elles sont généralement labiles, par exemple, certains événements de phosphorylation n’ont que quelques secondes de demi-vie32,33.
- Prélèvements de tissus d’accise et de pesée
- Tarez un tube de microcentrifugation de 1,5 mL à l’aide d’une balance analytique et prérefroidissez le tube sur de la glace sèche.
- Coupez un tissu congelé (par exemple, du tissu cérébral humain, ~10 mg) d’une région définie en petits morceaux et transférez immédiatement les morceaux de tissu dans le tube pré-refroidi.
REMARQUE : Pour réduire l’hétérogénéité de l’échantillon, il est important d’utiliser des tailles et des régions anatomiques homogènes pour les 16 échantillons. La quantité de protéines obtenue à partir du tissu est généralement de 5 à 10 % du poids du tissu. - Pesez le tube avec le mouchoir et placez immédiatement le tube sur de la glace sèche. Traitez les 15 échantillons restants en utilisant la même procédure. Conserver les échantillons dans de la glace carbonique immédiatement après la dissection et les conserver à ˗80 °C.
- Échantillons de tissus de lyse
- Le jour de l’expérience, préparez un tampon de lyse frais (50 mM de HEPES pH 8,5, 8 M d’urée et 0,5 % de désoxycholate de sodium). Des inhibiteurs de la phosphatase doivent être ajoutés dans le tampon de lyse pour préserver l’état de phosphorylation des protéines.
REMARQUE : Mettez le tampon de lyse à température ambiante avant de l’utiliser, car 8 M d’urée précipitera sur la glace, ce qui peut entraîner une dénaturation incomplète des protéines pendant la lyse de l’échantillon et réduire l’efficacité de la digestion des protéines. - Ajouter le tampon de lyse (ajouter 100 μL de tampon de lyse par 10 mg de tissu pour obtenir une concentration finale en protéines de 5 à 10 μg/μL) et des billes de verre (~20 % du volume du lysat, 0,5 mm de diamètre) à chaque échantillon.
- Lyser le tissu dans un mélangeur à 4 °C avec une vitesse de 8 pendant 30 s, laisser reposer pendant 5 s, répéter jusqu’à ce que les échantillons soient homogénéisés (~ 5 cycles).
- Le jour de l’expérience, préparez un tampon de lyse frais (50 mM de HEPES pH 8,5, 8 M d’urée et 0,5 % de désoxycholate de sodium). Des inhibiteurs de la phosphatase doivent être ajoutés dans le tampon de lyse pour préserver l’état de phosphorylation des protéines.
- Préparez des aliquotes des lysats.
- Préparez au moins deux aliquotes pour chaque échantillon. Une petite aliquote (~10 μL) est utilisée pour l’analyse de la concentration en protéines et l’évaluation de la qualité des protéines (p. ex., validation par transfert Western des protéines témoins positives). Une aliquote plus grande (~50 μL) est utilisée pour l’analyse du protéome.
- Congelez immédiatement les aliquotes sur de la glace carbonique et conservez-les à -80 °C jusqu’à une nouvelle utilisation.
- Mesurer la concentration en protéines
REMARQUE : La concentration en protéines peut être mesurée par le dosage BCA ou la méthode de coloration sur gel SDS34 (Figure 3A). Étant donné que les composants non réducteurs de protéines dans le lysat tissulaire peuvent affecter la mesure dans le test BCA, les utilisateurs peuvent valider la concentration en protéines par la méthode de coloration sur gel SDS courte. La méthode de coloration sur gel SDS courte est présentée ici.- Diluer 16 échantillons aliquotes par 10 et préparer l’étalon BSA (par exemple, titrages BSA de 0,15, 0,5, 1,5 et 3 μg).
- Analysez les échantillons et l’étalon BSA sur un gel SDS-PAGE à 10 % (26 puits) avec un gel d’empilement jusqu’à ce que toutes les protéines migrent d’environ 3 mm dans le gel.
- Teignez le gel avec du bleu Coomassie pendant 1 h et détachez le gel jusqu’à ce que le fond de la zone vierge soit clair.
- Scannez le gel pour mesurer l’intensité des bandes de protéines colorées à Coomassie par ImageJ et créez une courbe standard BSA en fonction des mesures.
- Calculez la concentration absolue en protéines par la courbe standard.
REMARQUE : La concentration finale en protéines de chaque échantillon dans cette expérience était de ~5-10 μg/μL. Pour l’analyse protéomique basée sur le TMT16, 50 μg de protéines par échantillon (0,8 mg de protéines totales pour l’analyse du protéome entier) sont suffisants.
- Contrôle de la qualité des échantillons
REMARQUE : Cette étape de contrôle de la qualité est essentielle pour identifier les échantillons de faible qualité avant d’effectuer l’analyse TMT. Pour les échantillons où l’on sait que l’on observe un changement protéique, il est suggéré de valider le changement par western blot. L’analyse standard SDS-PAGE est également recommandée pour examiner les profils protéiques et exclure tout échantillon présentant des degrés élevés de dégradation (figure 3B).- Prélever ~10 μg de chaque échantillon de la petite aliquote et analyser les échantillons sur un gel SDS-PAGE à gradient jusqu’à ce que le colorant bleu de bromophénol atteigne le fond du gel.
- Teignez le gel avec du bleu Coomassie et détachez le gel. Inspectez la qualité des protéines pour éliminer les échantillons de protéines fortement dégradés.
REMARQUE : Les échantillons dégradés peuvent être identifiés comme un échantillon qui comporte très peu de bandes protéiques dans la région de poids moléculaire élevé et des bandes intensifiées dans la région de faible poids moléculaire (figure 3B).
2. Digestion des protéines en solution, réduction et alkylation des peptides, test d’efficacité de la digestion et dessalage des peptides
- Digestion des protéines par Lys-C et trypsine
- Prélever ~50 μg de protéines dans la grande partie aliquote de chaque échantillon et ajouter un tampon de lyse à 50 μL.
- Ajouter 100 % d’acétonitrile (ACN) pour atteindre une concentration finale de 10 %.
- Effectuez la digestion Lys-C en ajoutant Lys-C à un rapport protéine :Lys-C de 100:1 (p/p) et en incubant à température ambiante pendant 3 h.
- Les échantillons sont dilués pour contenir une concentration finale de 2 M d’urée par 50 mM d’HEPES (pH 8,5).
- Ajoutez de la trypsine dans chaque échantillon à un rapport protéine :trypsine de 50:1 (p/p) et effectuez la digestion à température ambiante pendant 3 h ou toute la nuit.
- Réduction et alkylation des peptides
- Ajouter une solution de dithiothréitol (1 M DTT) fraîchement préparée à une concentration finale de 1 mM et incuber pendant 1 h à température ambiante pour réduire les liaisons disulfure.
- Ajouter une solution d’iodoacétamide (1 M IAA) fraîchement préparée à une concentration finale de 10 mM pendant 30 min dans l’obscurité pour alkylate les résidus de cystéine.
- Tremper l’AIA n’ayant pas réagi en ajoutant 1M de DTT à une concentration finale de 30 mM et incuber à température ambiante pendant 30 minutes supplémentaires.
- Examiner l’efficacité de la digestion
- Prélever ~1 μg de chaque échantillon et dessaler à l’aide d’embouts de pipette recouverts de résine C18 selon le protocole du fabricant.
- Analysez chaque échantillon par une courte exécution LC-MS/MS à gradient (voir plus de détails à l’étape 5).
- Effectuez une recherche dans la base de données pour les données brutes MS (voir plus de détails à l’étape 6). Calculer le pourcentage de peptides identifiés avec au moins un site de clivage de la trypsine. Le pourcentage est généralement inférieur à 15 %.
- Si le pourcentage est supérieur à 15 %, ajoutez de la trypsine supplémentaire aux échantillons pour répéter la digestion.
- Après la digestion, acidifiez les échantillons en ajoutant du TFA à 0,5 % (v/v). Vérifiez le pH à l’aide d’une bandelette de pH pour vous assurer que le pH est inférieur à 3.
- Dessalage des peptides
- Centrifuger les peptides acidifiés à 21 000 x g pendant 10 min. Transférez les surnageants dans un nouveau tube.
- Laver deux fois les colonnes de dessalage C18 (~25 μL de résine) avec 250 μL de méthanol à 100 % par centrifugation à 500 x g pendant 30 s.
REMARQUE : Pour réduire la perte de peptides pendant le processus de dessalage, choisissez des colonnes de dessalage avec une capacité de liaison correspondant à la quantité d’entrée. - Laver les colonnes deux fois avec 250 μL de tampon d’élution (60 % ACN, 0,1 % TFA) par centrifugation à 500 x g pendant 30 s.
- Équilibrer les colonnes deux fois avec 250 μL de tampon d’équilibrage et de lavage (0,1 % TFA) par centrifugation à 500 x g pendant 30 s.
- Chargez les échantillons sur les colonnes pré-équilibrées. Laissez les échantillons se lier aux colonnes en tournant à 100 x g pendant 6 min. Assurez-vous que toute la solution est passée par la colonne.
- Laver les colonnes trois fois avec 250 μL d’équilibrage et laver le tampon par centrifugation à 500 x g pendant 30 s.
- Éluer les peptides en ajoutant 125 μL de tampon d’élution dans chaque colonne et en tournant à 100 x g pendant 3 min. Vérifiez que la colonne ne contient pas de solution restante.
- Faites sécher les peptides élués dans un concentrateur sous vide et stockez les peptides à -80 °C pour un futur marquage TMT.
3. Marquage TMT16 des peptides, test d’efficacité de marquage, regroupement d’échantillons et dessalage des peptides marqués
- Marquage des peptides par le TMT16
- Remettre en suspension chaque échantillon de peptide dessalé dans 50 μL de HEPES 50 mM (pH 8,5) par vortex plusieurs fois ou dissolution par ultrasons suivie d’une bandelette de pH pour vérifier le pH.
REMARQUE : L’échantillon peut être acide s’il n’est pas complètement séché avant l’étiquetage, ce qui influence négativement l’efficacité de l’étiquetage. Assurez-vous que le pH est compris entre 7 et 8. - Prélever ~1 μg de peptides non marqués dans chaque échantillon comme témoins négatifs pour le test d’efficacité de marquage TMT.
- Dissoudre les réactifs TMT16 dans l’ACN anhydre. Effectuez la réaction de marquage en ajoutant les réactifs à un rapport TMT :protéine de 1,5:1 (p/p) et en incubant à température ambiante pendant 30 min.
REMARQUE : Le rapport TMT :protéines utilisé pour le TMT16 est 50 % plus élevé que le rapport utilisé pour le TMT11. Ce petit écart peut être dû au fait que la masse moléculaire du TMT16 est plus grande (1,2 fois) que celle des réactifs du TMT11. La quantité de protéines est estimée à partir des échantillons sans tenir compte de la perte lors du dessalage.
- Remettre en suspension chaque échantillon de peptide dessalé dans 50 μL de HEPES 50 mM (pH 8,5) par vortex plusieurs fois ou dissolution par ultrasons suivie d’une bandelette de pH pour vérifier le pH.
- Test d’efficacité de l’étiquetage
- Prélever ~1 μg de peptides marqués dans chaque échantillon pour le test d’efficacité du marquage. Mettez les échantillons restants à -80 °C sans éteindre la réaction.
- Dessaler ~1 μg d’échantillons marqués et non marqués par TMT16 à l’aide d’embouts de pipette recouverts de résine C18 conformément au protocole du fabricant.
- Analysez les échantillons par LC-MS/MS (voir section 5, à l’exception du gradient de 10 min).
- Estimez l’efficacité du marquage en analysant la réduction de l’intensité MS1 des peptides non marqués entre les échantillons non marqués et marqués. Sélectionnez 6 à 10 peptides différents pour vérifier l’efficacité de l’étiquetage afin de vous assurer que tous les peptides sont marqués. Pour un marquage complet, les peptides non marqués ne sont pas observés.
REMARQUE : Il est important d’assurer l’étiquetage complet de tous les échantillons pour une identification et une quantification précises des protéines en aval. - Si l’étiquetage n’est pas terminé, ajoutez des réactifs TMT supplémentaires pour marquer les peptides restants et vérifiez à nouveau l’efficacité de l’étiquetage avant la trempe. Une fois l’échantillon complètement marqué, trempez la réaction à température ambiante en ajoutant de l’hydroxylamine à une concentration finale de 0,5 % et incubez à température ambiante pendant 15 min.
- Regroupement et dessalage des échantillons
- Mettez en commun la moitié de chaque échantillon étiqueté TMT pour former un mélange.
- Prélever 1 μg du mélange et dessaler à l’aide d’embouts de pipette recouverts de résine C18, puis analyser par LC-MS/MS à l’aide d’un gradient court (~30 min).
- Calculez la concentration relative en utilisant l’intensité moyenne de chaque ion rapporteur TMT16 et en comparant les écarts entre les 16 canaux. Pour obtenir un mélange égal de chaque canal, ajoutez le reste des échantillons marqués TMT dans le mélange en fonction de l’intensité moyenne calculée. Répétez l’ajustement jusqu’à ce que tous les échantillons soient mélangés de manière égale. Le tableau 1 présente des données représentatives montrant le processus de regroupement des échantillons.
REMARQUE : Étant donné que les erreurs de pipetage peuvent affecter la précision des concentrations et la quantification des protéines, il est important de s’assurer que la quantité de regroupement est correcte. Les écarts d’intensité entre 16 échantillons doivent être inférieurs à 5 %.
- Dessalage des peptides étiquetés
REMARQUE : Étant donné que les dérivés de fond de la réaction de marquage du TMT16 (par exemple, le TMTpro-NHOH issu de la réaction d’extinction de l’hydroxylamine et le TMTpro-OH issu de l’hydroxylation du TMT) sont hydrophobes, une condition de lavage extensive est utilisée pour les échantillons marqués au TMT16 afin d’éliminer efficacement les dérivés. L’ajout de 5 % d’ACN dans le tampon de lavage régulier (0,1 % de TFA) et 10 volumes de lit de tampon de lavage sont utilisés.- Acidifiez l’échantillon groupé en ajoutant 10 % de TFA au pH < 3.
- Centrifuger l’échantillon groupé à 21 000 x g pendant 10 min et placer le surnageant dans un nouveau tube.
- Sécher l’échantillon à l’aide d’un concentrateur sous vide pour éliminer l’ACN.
- Préconditionner une cartouche d’extraction en phase solide contenant une colonne de sorbant de 50 mg en lavant la colonne avec 2 mL de méthanol à 100 %, puis 2 mL de tampon d’élution (60 % d’ACN plus 0,1 % d’AGT) et enfin 2 mL de tampon de lavage (0,1 % d’AGT).
- Chargez l’exemple sur la colonne. Ajustez le débit à ~100 μL/min pour assurer toute l’étendue de la liaison peptidique. Enregistrez le flux.
- Laver la colonne trois fois avec un tampon de lavage de 1 ml.
- Éluer les peptides avec un tampon d’élution de 1 mL.
- Faites sécher les peptides élués dans un concentrateur sous vide et stockez les peptides à -80 °C pour un fractionnement ultérieur.
4. Pré-fractionnement du pH LC de base hors ligne
- Préparation du système de fractionnement
- Préparez le tampon A (10 mM de formiate d’ammonium, pH 8,0) et le tampon B (10 mM de formiate d’ammonium, 90 % d’ACN, pH 8,0) pour un système LC haute performance à débit d’un microlitre.
- Mise en place d’une colonne HPLC contenant des particules hybrides d’éthylène pontées (taille des particules de 3,5 μm, 4,6 mm × 25 cm) dans un système LC haute performance à débit d’un microlitre pour le fractionnement.
- Installez une boucle d’échantillonnage de 100 μL et lavez-la avec 300 μL de méthanol, d’eau et de tampon A, séquentiellement.
- Utiliser 100 μL de rapport 1:1:1:1 isopropanol : méthanol : acétonitrile : eau pour laver la colonne. Ensuite, équilibrez davantage la colonne pendant 0,5 h dans 95 % du tampon A.
- Préparation des échantillons
- Dissoudre l’échantillon de TMT16 mélangé et dessalé dans 70 μL de tampon A. Confirmer que le pH de l’échantillon est de ~ 8,0. S’il est toujours acide, ajustez le pH à 8,0 en utilisant de l’hydroxyde d’ammonium à 28 % (NH4OH).
REMARQUE : Pour éviter la perte d’échantillon, le volume de l’échantillon doit être inférieur à 70 % du volume de la boucle. - Centrifuger l’échantillon à 21 000 x g pendant 10 min pour éliminer les précipités.
- Dissoudre l’échantillon de TMT16 mélangé et dessalé dans 70 μL de tampon A. Confirmer que le pH de l’échantillon est de ~ 8,0. S’il est toujours acide, ajustez le pH à 8,0 en utilisant de l’hydroxyde d’ammonium à 28 % (NH4OH).
- Fractionnement et concaténation
REMARQUE : Avant le fractionnement réel de l’échantillon, une expérience pilote est fortement recommandée pour s’assurer que le système LC est en bon état. Cela peut être effectué avec une petite quantité de votre échantillon réel (~5 %) ou avec un mélange de peptides non marqué TMT.- Injectez l’échantillon et fractionnez-le selon le gradient suivant : 5 % de tampon B pendant 10 min, 5-15 % de tampon B pendant 2 min, 15-45 % de tampon B pendant 148 min et 45-95 % de tampon B pendant 5 min. Utilisez un débit de 0,4 mL/min.
- Réglez le collecteur de fractions pour collecter des fractions toutes les 1 min et concaténez 160 fractions en 40 fractions en 4 cycles.
REMARQUE : La concaténation est effectuée en combinant des fractions LC précoces, moyennes et tardives éluées à partir des mêmes internes temporels en une fraction concaténée. Les fractions concaténées ont peu de chevauchement dans la première dimension de LC, ce qui augmente l’utilisation efficace de la fenêtre d’élution dans la deuxième dimension de LC. De plus, grâce à plusieurs cycles de concaténation, les peptides peuvent être répartis uniformément sur toutes les fractions concaténées. Il a été démontré que cette approche augmente la couverture protéomique par rapport à l’analyse des fractions individuelles35,36. - Séchez toutes les fractions concaténées dans un concentrateur sous vide et stockez les échantillons séchés à -80 °C pour une analyse LC-MS/MS plus poussée.
5. Analyse du pH acide RPLC-MS/MS
- Préparation du système RPLC-MS/MS à pH acide
- Emballez une colonne vide (75 μm de diamètre intérieur avec un orifice de pointe de 15 μm) avec de la résine C18 de 1,9 μm sur une longueur de 10 à 15 cm.
- Chauffez la colonne à 65 °C à l’aide d’un réchauffeur de portefeuille papillon pour réduire la contre-pression.
- Lavez soigneusement la colonne avec 95 % de tampon B (3 % de diméthylsulfoxyde, 0,2 % d’acide formique et 67 % d’ACN). Ensuite, équilibrez complètement la colonne dans le tampon A à 95 % (3 % de diméthylsulfoxyde et 0,2 % d’acide formique).
- Vérifiez les performances du système LC-MS/MS en analysant 100 ng de peptides de cerveau de rat ou de peptides BSA avant d’analyser les échantillons expérimentaux.
- Analyse LC-MS/MS de fractions concaténées
- Reconstituer les peptides séchés à partir des fractions de pH basiques dans 5 % d’acide fa et centrifuger à 21 000 × g pendant 5 min. Transférez le surnageant de chaque échantillon dans un insert de flacon HPLC.
- Chargez ~1 μg de peptides de chaque fraction sur la colonne. Les peptides sont élués à un débit de 0,25 μL/min avec un gradient de 60 min de 18 à 45 % de tampon B.
REMARQUE : Pour obtenir des numéros d’identification élevés, exécutez une fraction et ajustez le gradient des fractions restantes en fonction de la première exécution. Le meilleur dégradé doit avoir des peptides uniformément répartis sur l’ensemble du dégradé (Figure 4A). - Faire fonctionner le spectromètre de masse avec les paramètres suivants pour l’analyse d’échantillons marqués TMT16 : balayages MS1 (plage de balayage MS complète : 450-1600 m/z ; Résolution Orbitrap : 60 000 ; Cible de contrôle automatique du gain : 1 x 106 ; temps ionique maximal : 50 ms) et 20 balayages MS2 dépendants des données (résolution Orbitrap : 60 000 ; Cible AGC : 1 x 105 ; temps ionique maximum : 110 ms ; Énergie de collision normalisée HCD : 32 % ; fenêtre d’isolation : 1,0 m/z ; Décalage d’isolation : 0,2 m/z ; Exclusion dynamique : 10 s).
REMARQUE : Les paramètres utilisés ici sont optimisés sur un type de spectromètre de masse (voir Tableau des matériaux). Pour différents instruments MS, les utilisateurs doivent affiner les paramètres de l’instrument pour obtenir des résultats de haute qualité. L’un des paramètres consiste à surveiller l’énergie de collision normalisée du HCD, car l’énergie optimale peut varier d’un instrument à l’autre ainsi qu’entre TMT11 et TMT16.
6. Traitement des données
REMARQUE : L’analyse des données a été effectuée à l’aide d’une suite logicielle JUMP 37,38,39 comprenant un moteur de recherche de base de données hybride (basé sur des motifs et des étiquettes), un logiciel de filtrage qui contrôle le taux de fausses découvertes (FDR) des peptides et protéines identifiés, et un logiciel de quantification des ensembles de données TMT. Selon la situation de l’utilisateur, l’analyse des données peut être effectuée à l’aide d’autres programmes commerciaux ou disponibles gratuitement.
- Recherche dans la base de données
- Convertissez les fichiers .raw de l’instrument MS en fichiers .mzXML, et effectuez des recherches dans les spectres MS2 par rapport à une base de données de leurres cibles non redondante40 générée à partir de séquences de protéines humaines UniProt (ou d’une autre base de données appropriée spécifique à l’espèce) pour calculer le FDR des protéines identifiées.
REMARQUE : Générez la base de données non redondante en combinant les séquences de protéines des bases de données Swiss-Prot et TrEMBL. On peut également ajouter des séquences protéiques personnalisées qui ne figurent pas dans ces bases de données de référence, y compris des protéines clivées par la protéase, des protéines avec des polymorphismes nucléotidiques simples et des contaminants communs. - Effectuez des recherches à l’aide des paramètres suivants. Tolérance de masse pour le précurseur : 10 ppm ; tolérance massique pour les ions produits : 15 ppm ; clivages manqués maximaux : 2 ; sites de modification maximale : 3 ; modifications statiques : 304.20715 Da pour les marqueurs TMT16 sur les résidus de Lys et les terminaisons N, 57.02146 Da pour la carbamidométhylation sur les résidus de Cys ; modification dynamique : 15.99492 Da pour l’oxydation sur Met.
- Convertissez les fichiers .raw de l’instrument MS en fichiers .mzXML, et effectuez des recherches dans les spectres MS2 par rapport à une base de données de leurres cibles non redondante40 générée à partir de séquences de protéines humaines UniProt (ou d’une autre base de données appropriée spécifique à l’espèce) pour calculer le FDR des protéines identifiées.
- Filtrer les résultats de la recherche
- Filtrez les correspondances peptidiques résultantes (PSM) par longueur peptidique (>6 acides aminés), la précision de masse de l’ion précurseur et les scores d’appariement basés sur JUMP (Jscore et ΔJn). Les peptides sont ensuite regroupés par longueur de peptide, extrémités tryptiques, modifications, sites de clivage manqués et état de charge.
- Filtrez davantage les données avec les scores correspondants pour obtenir un FDR inférieur à 1 % au niveau de la protéine (analyse du protéome entier) ou du peptide (analyse du phosphoprotéome).
REMARQUE : Si des peptides/protéines de contrôle positif sont manquants aux étapes de filtration, le FDR peut être augmenté à un niveau raisonnable afin que ces peptides/protéines puissent être sauvés. - Pour les peptides partagés par plus d’un membre d’une famille de protéines, regroupez les membres appariés en un seul groupe.
REMARQUE : Avec la règle de parcimonie, le groupe est représenté par la protéine homologue avec le plus grand nombre de peptides partagés et d’autres protéines appariées par des peptides uniques.
- Quantification des protéines
- Quantifiez les protéines à l’aide d’un programme intégré d’une suite logicielle statistique pour résumer les intensités ioniques rapporteures du TMT sur tous les PSM appariés.
- Extraire les intensités ioniques rapporteures TMT de chaque PSM accepté et corriger les intensités brutes en fonction de la distribution isotopique de chaque réactif de marquage (par exemple, TMT16-126 génère 92,6 %, 7,2 % et 0,2 % des ions m/z 126, 127C et 128C, respectivement) et filtrer les PSM de faible intensité et/ou très bruyants sur la base de seuils définis par l’utilisateur. Normalisez les données de quantification à l’aide de la moyenne ajustée (ou médiane) des intensités des échantillons pour corriger le biais de chargement.
- Pour chaque protéine identifiée, calculez les intensités moyennes centrées sur les échantillons (c’est-à-dire les intensités relatives) des PSM appariés et résumez les intensités relatives des PSM en prenant la moyenne par échantillon. Convertissez les signaux relatifs en signaux absolus en multipliant l’intensité moyenne globale des trois PSM appariés les plus abondants.
- Quantification correcte du brouillage à l’aide d’une approche de correction de l’ion y1 précédemment rapportée37 qui suppose que l’intensité de l’ion y1 est corrélée à l’intensité de l’ion rapporteur. En estimant la relation linéaire entre les intensités d’ions y1 et rapporteurs à partir de balayages propres, le niveau d’interférence dû à l’intensité d’ions y1 contaminés dans les balayages bruyants est dérivé et corrigé.
REMARQUE : Pour les peptides tryptiques marqués au TMT, les résidus K-TMT et R sont deux ions y1 représentatifs (376,27574 Da et 175,11895 Da, respectivement) dans un spectre MS2. Si un seul ion y1 est détecté et qu’il correspond au peptide identifié, le MS2 est considéré comme un balayage propre. Si les deux ions y1 sont détectés, le MS2 est considéré comme un balayage bruyant. - Transférez les valeurs de quantification des protéines dans une feuille de calcul pour une analyse plus approfondie. Utilisez des méthodes d’analyse de données non supervisées telles que l’ACP ou l’analyse par clustering pour explorer la distribution des échantillons. Pour identifier les protéines exprimées de manière différentielle, utilisez des méthodes statistiques telles que le test t et l’analyse de variance (ANOVA).
7. Validation des données MS
REMARQUE : Avant d’effectuer des expériences biologiques qui prennent beaucoup de temps, utilisez au moins une méthode de validation pour évaluer la qualité des données de la MS.
- Inspectez manuellement les spectres MS/MS des protéines d’intérêt pour valider la séquence peptidique et l’intensité des ions rapporteurs TMT.
- Utiliser des approches basées sur les anticorps (p. ex. transfert Western ou analyse immunohistochimique) pour vérifier les changements dans les taux de protéines. Pour confirmer la présence de peptides natifs, utilisez des peptides synthétiques comme étalons internes. Les spectres MS/MS des peptides et le temps de rétention pendant LC-MS/MS doivent être identiques dans les mêmes conditions.
- Utiliser une approche SP ciblée pour vérifier les changements protéiques.
Résultats
Le protocole du TMT16 nouvellement développé, y compris la réaction de marquage, le dessalage et les conditions LC-MS, a été systématiquement optimisé41. De plus, nous avons comparé directement les méthodes 11-plex et 16-plex en les utilisant pour analyser les mêmes échantillons humains de MA41. Après optimisation des paramètres clés du TMT16, les méthodes TMT11 et TMT16 produisent une couverture, une identification et une quantification similaires du protéome > 100 000 peptides dans > 10 000 protéines humaines.
Étant donné que les réactifs TMT16 sont plus hydrophobes que les réactifs TMT11, les peptides marqués au TMT16 sont susceptibles d’être plus hydrophobes que les peptides marqués au TMT11, ce qui peut expliquer le temps de rétention (RT) différent dans la RPLC. Ainsi, nous avons évalué l’impact du TMT16 sur le peptide RT par rapport au TMT11 en analysant le mélange peptidique marqué TMT11 et TMT16 à l’aide de LC-MS/MS. Nous avons constaté que le TMT16 a une influence significative sur la RT aux peptides d’hydrophobicité moyenne, mais a peu d’effet sur les peptides d’hydrophobicité extrêmement élevée ou faible. Par conséquent, les concentrations similaires de début et de fin du tampon B dans le gradient LC peuvent être utilisées pour différents peptides marqués au TMT.
Nous avons ensuite optimisé le gradient RPLC en ligne pour l’échantillon marqué TMT16. La pente du TMT16 est très similaire à celle du TMT11. Le pourcentage de la réserve tampon de début et de fin B est le même (p. ex., de 18 % à 45 %). Mais nous avons remarqué que le nombre de peptides identifiés dans le TMT16 a chuté rapidement à environ 40 % du tampon B en utilisant le même gradient que celui utilisé pour le TMT11. Ainsi, nous avons légèrement réduit le temps de la pente entre 40 % et 45 %. Nous avons également apporté des ajustements mineurs à ce gradient pour différentes fractions et différents échantillons. Après l’optimisation du gradient, les peptides identifiés ont été répartis uniformément dans tout le gradient (Figure 4A).
Afin de maximiser le nombre de protéines identifiées et quantifiées avec précision à l’aide de la méthode TMT16, nous avons optimisé l’énergie de collision normalisée (NCE) pour les échantillons marqués au TMT16 dans notre précédent rapport41. Différents NCE (de 20 % à 40 %) ont été testés sur le spectromètre de masse lors d’exécutions LC-MS/MS. En équilibrant le nombre d’identifications de protéines et l’intensité de l’ion rapporteur, une NCE de 30 à 32,5 % a été choisie comme énergie de collision HCD optimale à utiliser pour les échantillons marqués au TMT16.
La compression du rapport causée par les ions interférents co-élutés a été une limitation des techniques de marquage isobare pour la quantification des protéines. Une étude précédemment publiée utilisant la méthode TMT11 montre que la compression du rapport peut être presque éliminée par une pré-fractionnement étendue de la LC, des paramètres MS optimisés et des stratégies de correction des données post-MS37. Nous avons utilisé ces stratégies, y compris le fractionnement extensif pré-MS (40 fractions de pH LC basiques), l’application d’une fenêtre d’isolement étroite (1 m/z) dans le cadre de la MS et la correction de l’ion y1 dans les analyses du protéome TMT11 et TMT16 des mêmes échantillons. Après avoir examiné la courbe de corrélation du changement de plissement des protéines entre les ensembles de données TMT11 et TMT16, nous avons constaté que la pente était très proche de 1, indiquant que la compression du rapport dans TMT16 n’était pas visiblement plus élevée que celle dans TMT11 dans notre condition expérimentale41. Les résultats cohérents ont montré que la compression du rapport n’a pas de différence lorsque le niveau de multiplexage est passé de 11 à 1613,45. Ainsi, les stratégies précédemment publiées peuvent être utilisées pour atténuer la compression du rapport, améliorant ainsi considérablement la précision de la quantification 27,37,44,46.
Enfin, nous avons comparé le nombre de PSM, de peptides uniques et de protéines uniques quantifiés dans des échantillons marqués au TMT11 par rapport au TMT16 (Figure 4B). Les résultats montrent que les MSP des deux méthodes sont comparables ; cependant, les protéines et les peptides quantifiés sont légèrement plus faibles dans la méthode TMT16, ce qui est cohérent avec d’autres rapports12,13. Nos résultats indiquent que les améliorations apportées au procédé TMT16 ainsi que l’utilisation de paramètres LC-MS optimisés permettent un profilage protéomique profond à haut débit des échantillons biologiques.
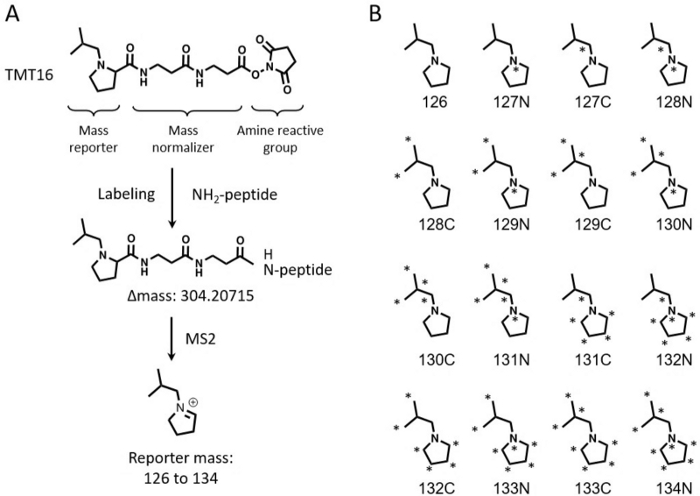
Figure 1 : Structure du réactif TMT 16-plex. (A) La structure du réactif TMT 16-plex, le processus de marquage, le décalage de masse après marquage et la masse de l’ion rapporteur sont montrés. (B) Structures marquées par des isotopes lourds des ions rapporteurs des réactifs TMT16. Veuillez cliquer ici pour voir une version agrandie de cette figure.
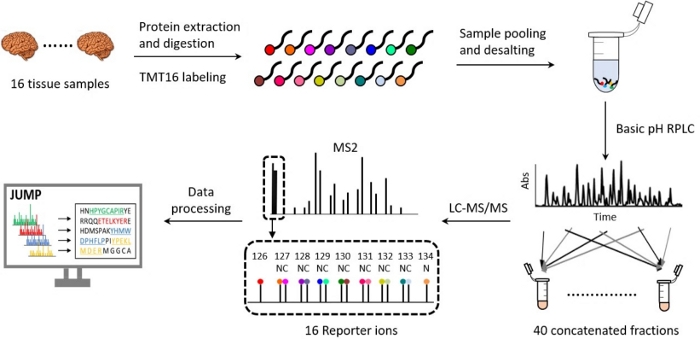
Figure 2 : Flux de travail du profilage du protéome par TMT-LC/LC-LC/MS 16-plex. Les protéines extraites de 16 échantillons de tissus biologiques ont été digérées et marquées avec 16 étiquettes TMT différentes. Les échantillons de 16 canaux sont regroupés de manière égale, et le mélange est fractionné et concaténé en 40 fractions par chromatographie liquide en phase inverse (RPLC) à pH basique hors ligne. Chaque fraction est analysée par RPLC acide couplé à une spectrométrie de masse à haute résolution. Les fichiers bruts MS/MS ont été traités. L’image du tissu cérébral est citée à partir de Medium.com avec quelques modifications. Veuillez cliquer ici pour voir une version agrandie de cette figure.
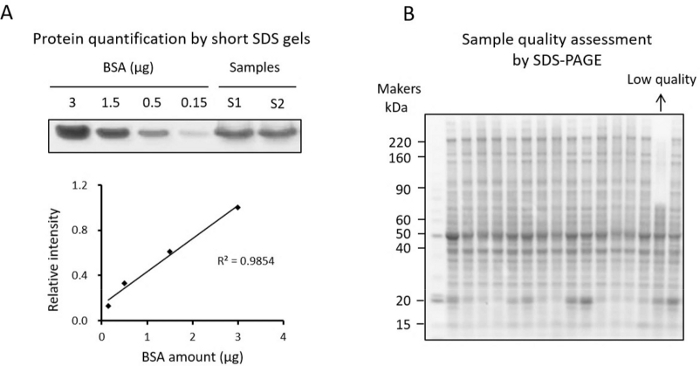
Figure 3 : Contrôle de la qualité des protéines. (A) Quantification de la protéine extraite du tissu sur un gel SDS court avec BSA comme norme. La courbe standard représente la concentration de BSA et l’intensité de la bande protéique colorée à Coomassie utilisées pour la quantification. (B) Gel SDS utilisé pour le dosage de la qualité des protéines. Veuillez cliquer ici pour voir une version agrandie de cette figure.
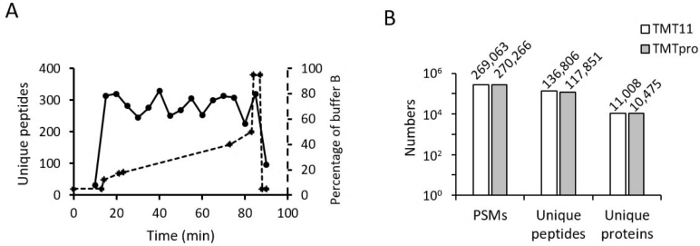
Figure 4 : Résultats représentatifs. (A) Distribution peptidique dans les LC acides. Le gradient optimisé du tampon B après correction du volume mort est aligné dans le même graphique. (B) L’histogramme montre le nombre de PSM quantifiés, de peptides uniques et de protéines uniques dans les méthodes TMT11 et TMT16. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| 1ère (50 μl, utiliser 50 % dans le premier mélange) | 2ème (ajuster le mélange et économiser 10 %) | 3ème (réglage final) | |||||||||||
| Canaux | Journalistes | Mélange Vol (μL) | Intensité (unités) | Conc. (unité/μL) | Intensité attendue (unités) | Vol ajouté (μL) | Vol total (μL) | Intensité (unités) | Conc. (unité/μL) | Intensité attendue (unités) | Vol ajouté (μL) | Vol total (μL) | Intensité (unités) |
| 1 | sig126 | 25 | 94.7 | 3.8 | 122.1 | 7.2 | 32.2 | 99.6 | 3.1 | 105.3 | 1.8 | 34.1 | 100 |
| 2 | sig127N | 25 | 83 | 3.3 | 122.1 | 11.8 | 36.8 | 101.1 | 2.7 | 105.3 | 1.5 | 38.3 | 98 |
| 3 | sig127C | 25 | 86 | 3.4 | 122.1 | 10.5 | 35.5 | 99.9 | 2.8 | 105.3 | 1.9 | 37.4 | 99.9 |
| 4 | sig128N | 25 | 103.9 | 4.2 | 122.1 | 4.4 | 29.4 | 102.1 | 3.5 | 105.3 | 0.9 | 30.3 | 97.2 |
| 5 | sig128C | 25 | 90.8 | 3.6 | 122.1 | 8.6 | 33.6 | 103.3 | 3.1 | 105.3 | 0.7 | 34.3 | 98.3 |
| 6 | sig129N | 25 | 82.8 | 3.3 | 122.1 | 11.9 | 36.9 | 99 | 2.7 | 105.3 | 2.4 | 39.3 | 98.7 |
| 7 | sig129C | 25 | 101.3 | 4.1 | 122.1 | 5.1 | 30.1 | 98.5 | 3.3 | 105.3 | 2.1 | 32.2 | 102.1 |
| 8 | sig130N | 25 | 98.9 | 4 | 122.1 | 5.9 | 30.9 | 100.1 | 3.2 | 105.3 | 1.6 | 32.5 | 99.7 |
| 9 | sig130C | 25 | 86.3 | 3.5 | 122.1 | 10.4 | 35.4 | 96 | 2.7 | 105.3 | 3.4 | 38.8 | 99.3 |
| 10 | sig131N | 25 | 87 | 3.5 | 122.1 | 10.1 | 35.1 | 95.3 | 2.7 | 105.3 | 3.7 | 38.8 | 101.5 |
| 11 | sig131C | 25 | 119.1 | 4.8 | 122.1 | 0.6 | 25.6 | 100.9 | 3.9 | 105.3 | 1.1 | 26.7 | 100.2 |
| 12 | sig132N | 25 | 86 | 3.4 | 122.1 | 10.5 | 35.5 | 95.3 | 2.7 | 105.3 | 3.7 | 39.2 | 99.6 |
| 13 | sig132C | 25 | 119.1 | 4.8 | 122.1 | 0.6 | 25.6 | 101.2 | 3.9 | 105.3 | 1 | 26.7 | 100 |
| 14 | sig133N | 25 | 116.3 | 4.7 | 122.1 | 1.3 | 26.3 | 99.9 | 3.8 | 105.3 | 1.4 | 27.7 | 100.9 |
| 15 | sig133C | 25 | 122.1 | 4.9 | 122.1 | 0 | 25 | 101 | 4 | 105.3 | 1.1 | 26.1 | 101.9 |
| 16 | sig134N | 25 | 121.3 | 4.9 | 122.1 | 0.2 | 25.2 | 105.3 | 4.2 | 105.3 | 0 | 25.2 | 101.3 |
Tableau 1 : Données représentatives montrant le processus de regroupement des échantillons à l’étape 3.3.
Discussion
Un protocole optimisé pour le profilage profond du protéome basé sur TMT16 a été mis en œuvre avec succès dans des publications antérieures 12,13,41. Avec ce protocole actuel, plus de 10 000 protéines uniques provenant d’un maximum de 16 échantillons différents peuvent être quantifiées de manière routinière en une seule expérience avec une grande précision.
Pour obtenir des résultats de haute qualité, il est important de prêter attention aux étapes critiques tout au long du protocole. En plus de toutes les étapes de contrôle qualité abordées dans notre précédent article27, nous incluons des étapes essentielles supplémentaires spécifiques au processus TMT16. Ces étapes sont importantes pour assurer la réussite d’une expérience. Par exemple, les dérivés de la réaction TMT (par exemple, TMTpro-NHOH de la réaction d’extinction de l’hydroxylamine et TMTpro-OH de l’hydroxylation du TMT) sont détectés comme des ions monochargeables proéminents avant le dessalage par l’analyse LC-MS/MS. Il est essentiel de les retirer lors de l’étape de dessalage. Nous avons testé différentes conditions de dessalage et avons constaté que l’ajout de 5 % d’ACN dans le tampon de lavage régulier combiné à 10 volumes de lit de × pendant trois lavages efficaces éliminait efficacement les dérivés41. De plus, le TMT16 a une masse accrue par rapport au TMT11, donc la plage de balayage complète commence à partir d’un m/z plus élevé (450 au lieu de 410) pour les échantillons marqués au TMT16. De plus, comme l’énergie de collision optimale pour un peptide dépend de la masse à la charge et de l’état de charge de l’ionprécurseur 21, les peptides marqués avec différentes étiquettes de marquage chimique peuvent avoir des énergies de collision optimales différentes. Pour le TMT16, l’énergie de collision de 30 à 32,5 % est optimale pour le TMT16, qui est légèrement inférieur à celui du TMT11.
Le marquage isobare est une technique puissante qui offre une capacité de multiplexage élevée. Bien que d’autres techniques telles que SILAC (stable isotope marking by amino acids in cell culture)47 et le mark-free offrent des stratégies alternatives pour quantifier lesprotéines48, elles souffrent d’un faible débit. En théorie, le TMT16 peut quantifier les protéines de 16 échantillons biologiques différents. Cependant, il est beaucoup plus courant d’utiliser certains de ces canaux comme réplicats biologiques, ce qui fournit plus de puissance statistique et aide à générer des données fiables. L’utilisation de répétitions, voire de triplets, est très critique, en particulier dans les systèmes où le changement attendu de la concentration en protéines est minime. Il est important de comprendre la biologie du système avant de concevoir l’expérience afin d’inclure le nombre approprié de répétitions. Certains systèmes biologiques ne sont pas parfaitement adaptés à certaines des étapes de contrôle de la qualité de ce protocole. Le test du rapport de prémélange n’est pas utilisé lors de l’utilisation d’échantillons d’immunoprécipitation pour le protocole en raison du pourcentage élevé de protéines qui devraient changer. Dans ces cas, les résultats seraient faussés par le test de prémélange. Cela est également vrai dans les cas où l’expression protéique d’au moins 1 des 10 échantillons est susceptible de varier considérablement (vecteur vide, inhibition du protéasome, etc.). Il est également suggéré d’utiliser un canal TMT comme « référence interne » qui peut ensuite être utilisé pour combiner plusieurs lots d’expériences TMT1649.
Ce protocole peut être utilisé pour le profilage protéomique mondial à haut débit d’échantillons biologiques complexes, pour étudier les protéines exprimées de manière différentielle et les voies de signalisation cellulaire, et pour comprendre la biologie des maladies. De plus, avec de légères modifications du protocole, il peut être utilisé pour étudier les modifications post-traductionnelles telles que la phosphorylation, l’ubiquitination, la méthylation et l’acétylation. L’adoption d’une approche intégrée combinant une analyse protéomique exhaustive à grande échelle avec d’autres pipelines -omiques tels que la génomique, la transcriptomique et la métabolomique peut fournir des informations permettant d’élargir la compréhension des systèmes biologiques complexes30,50.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été partiellement soutenu par les National Institutes of Health (R01GM114260, R01AG047928, R01AG053987, RF1AG064909 et U54NS110435) et l’ALSAC (American Lebanese Syrian Associated Charities). L’analyse de la SEP a été réalisée au Centre de protéomique et de métabolomique du St. Jude Children’s Research Hospital, qui est partiellement soutenu par la subvention de soutien du NIH Cancer Center (P30CA021765). Le contenu relève de la seule responsabilité des auteurs et ne représente pas nécessairement les opinions officielles des National Institutes of Health.
matériels
| Name | Company | Catalog Number | Comments |
| 10% Criterion TGX Precast Midi Protein Gel | Biorad | 5671035 | |
| 10X TGS (Tris/Glycine/SDS) Buffer | BioRad | 161-0772 | |
| 4–20% Criterion TGX Precast Midi Protein Gel | Biorad | 5671095 | |
| 50% Hydroxylamine | Thermo Scientific | 90115 | |
| 6 X SDS Sample Loading Buffer | Boston Bioproducts Inc | BP-111R | |
| Ammonium Formate (NH4COOH) | Sigma | 70221-25G-F | |
| Ammonium Hydroxide, 28% | Sigma | 338818-100ml | |
| Bullet Blender | Next Advance | BB24-AU | |
| Butterfly Portfolio Heater | Phoenix S&T | PST-BPH-20 | |
| C18 Ziptips | Harvard Apparatus | 74-4607 | Used for desalting |
| Dithiothreitol (DTT) | Sigma | D5545 | |
| DMSO | Sigma | 41648 | |
| Formic Acid | Sigma | 94318 | |
| Fraction Collector | Gilson | FC203B | |
| Gel Code Blue Stain Reagent | Thermo | 24592 | |
| Glass Beads | Next Advance | GB05 | |
| HEPES | Sigma | H3375 | |
| HPLC Grade Acetonitrile | Burdick & Jackson | AH015-4 | |
| HPLC Grade Water | Burdick & Jackson | AH365-4 | |
| Iodoacetamide (IAA) | Sigma | I6125 | |
| Lys-C | Wako | 125-05061 | |
| Mass Spectrometer | Thermo Scientific | Q Exactive HF | |
| MassPrep BSA Digestion Standard | Waters | 186002329 | |
| Methanol | Burdick & Jackson | AH230-4 | |
| Nanoflow UPLC | Thermo Scientific | Ultimate 3000 | |
| Pierce BCA Protein Assay kit | Thermo Scientific | 23225 | |
| ReproSil-Pur C18 resin, 1.9um | Dr. Maisch GmbH | r119.aq.0003 | |
| Self-Pack Columns | New Objective | PF360-75-15-N-5 | |
| SepPak 1cc 50mg | Waters | WAT054960 | Used for desalting |
| Sodium Deoxycholate | Sigma | 30970 | |
| Speedvac | Thermo Scientific | SPD11V | |
| TMTpro 16plex Label Reagent Set | Thermo Scientific | A44520 | |
| Trifluoroacetic Acid (TFA) | Applied Biosystems | 400003 | |
| Trypsin | Promega | V511C | |
| Ultra-micro Spin Column,C18 | Harvard apparatus | 74-7206 | Used for desalting |
| Urea | Sigma | U5378 | |
| Xbridge Column C18 column | Waters | 186003943 | Used for basic pH LC |
Références
- Levy, M. J., Washburn, M. P., Florens, L. Probing the sensitivity of the orbitrap lumos mass spectrometer using a standard reference protein in a complex background. Journal of Proteome Research. 17 (10), 3586-3592 (2018).
- Bekker-Jensen, D. B., et al. An optimized shotgun strategy for the rapid generation of comprehensive human proteomes. Cell Systems. 4 (6), 587-599 (2017).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Frost, D. C., Greer, T., Li, L. High-Resolution Enabled 12-Plex DiLeu Isobaric Tags for Quantitative Proteomics. Analytical Chemistry. 87 (3), 1646-1654 (2015).
- Moulder, R., Bhosale, S. D., Goodlett, D. R., Lahesmaa, R. Analysis of the plasma proteome using iTRAQ and TMT-based Isobaric labeling. Mass Spectrometry Reviews. 37 (5), 583-606 (2018).
- Wang, H., et al. Deep multiomics profiling of brain tumors identifies signaling networks downstream of cancer driver genes. Nature Communications. 10 (1), 3718(2019).
- Rauniyar, N., Yates, J. R. Isobaric labeling-based relative quantification in shotgun proteomics. Journal of Proteome Research. 13 (12), 5293-5309 (2014).
- Hogrebe, A., et al. Benchmarking common quantification strategies for large-scale phosphoproteomics. Nature Communications. 9 (1), 1045(2018).
- Dayon, L., et al. Relative quantification of proteins in human cerebrospinal fluids by MS/MS using 6-plex isobaric tags. Analytical Chemistry. 80 (8), 2921-2931 (2008).
- Stepanova, E., Gygi, S. P., Paulo, J. A. Filter-based protein digestion (FPD): A detergent-free and scaffold-based strategy for TMT workflows. Journal of Proteome Research. 17 (3), 1227-1234 (2018).
- McAlister, G. C., et al. Increasing the multiplexing capacity of TMTs using reporter ion isotopologues with isobaric masses. Analytical Chemistry. 84 (17), 7469-7478 (2012).
- Thompson, A., et al. TMTpro: Design, synthesis, and initial evaluation of a proline-based isobaric 16-plex tandem mass tag reagent set. Analytical Chemistry. 91 (24), 15941-15950 (2019).
- Li, J., et al. TMTpro reagents: a set of isobaric labeling mass tags enables simultaneous proteome-wide measurements across 16 samples. Nature Methods. 17 (4), 399-404 (2020).
- Arul, A. B., Robinson, R. A. S. Sample Multiplexing Strategies in Quantitative Proteomics. Analytical Chemistry. 91 (1), 178-189 (2019).
- Labib, M., Kelley, S. O. Single-cell analysis targeting the proteome. Nature Reviews Chemistry. 4 (3), 143-158 (2020).
- Ren, R. J., Dammer, E. B., Wang, G., Seyfried, N. T., Levey, A. I. Proteomics of protein post-translational modifications implicated in neurodegeneration. Translational Neurodegeneration. 3 (1), 23(2014).
- Pagel, O., Loroch, S., Sickmann, A., Zahedi, R. P. Current strategies and findings in clinically relevant post-translational modification-specific proteomics. Expert Review of Proteomics. 12 (3), 235-253 (2015).
- Mertins, P., et al. Reproducible workflow for multiplexed deep-scale proteome and phosphoproteome analysis of tumor tissues by liquid chromatography-mass spectrometry. Nature Protocols. 13 (7), 1632-1661 (2018).
- Aebersold, R., Mann, M. Mass-spectrometric exploration of proteome structure and function. Nature. 537 (7620), 347-355 (2016).
- Bai, B., et al. Deep multilayer brain proteomics identifies molecular networks in Alzheimer's disease progression. Neuron. 105 (6), 975-991 (2020).
- Kelstrup, C. D., et al. Rapid and deep proteomes by faster sequencing on a benchtop quadrupole ultra-high-field orbitrap mass spectrometer. Journal of Proteome Research. 13 (12), 6187-6195 (2014).
- Meier, F., et al. Online parallel accumulation - serial fragmentation (PASEF) with a novel trapped ion mobility mass spectrometer. Molecular & Cellular Proteomics. 17 (12), (2018).
- Schweppe, D. K., et al. Full-featured, real-time database searching platform enables fast and accurate multiplexed quantitative proteomics. Journal of Proteome Research. 19 (5), 2026-2034 (2020).
- Wang, H., et al. Systematic optimization of long gradient chromatography mass spectrometry for deep analysis of brain proteome. Journal of Proteome Research. 14 (2), 829-838 (2015).
- Dey, K. K., et al. Deep undepleted human serum proteome profiling toward biomarker discovery for Alzheimer's disease. Clinical Proteomics. 16, 16(2019).
- Bai, B., et al. Deep profiling of proteome and phosphoproteome by isobaric labeling, extensive liquid chromatography, and mass spectrometry. Methods in Enzymology. 585, 377-395 (2017).
- High, A. A., et al. Deep proteome profiling by isobaric labeling, extensive liquid chromatography, mass spectrometry, and software-assisted quantification. Journal of Visualized Experiments. (129), e56474(2017).
- Chick, J. M., et al. Defining the consequences of genetic variation on a proteome-wide scale. Nature. 534 (7608), 500-505 (2016).
- Wang, Z., et al. Quantitative phosphoproteomic analysis of the molecular substrates of sleep need. Nature. 558 (7710), 435-439 (2018).
- Tan, H., et al. Integrative proteomics and phosphoproteomics profiling reveals dynamic signaling networks and bioenergetics pathways underlying T cell activation. Immunity. 46 (3), 488-503 (2017).
- Eden, E., et al. Proteome Half-Life Dynamics in Living Human Cells. Science. 331 (6018), 764(2011).
- Kleiman, L. B., Maiwald, T., Conzelmann, H., Lauffenburger, D. A., Sorger, P. K. Rapid phospho-turnover by receptor tyrosine kinases impacts downstream signaling and drug binding. Molecular Cell. 43 (5), 723-737 (2011).
- Mertins, P., et al. Ischemia in tumors induces early and sustained phosphorylation changes in stress kinase pathways but does not affect global protein levels. Molecular & Cellular Proteomics. 13 (7), 1690(2014).
- Xu, P., Duong, D. M., Peng, J. Systematical optimization of reverse-phase chromatography for shotgun proteomics. Journal of Proteome Research. 8 (8), 3944-3950 (2009).
- Wang, Y., et al. Reversed-phase chromatography with multiple fraction concatenation strategy for proteome profiling of human MCF10A cells. Proteomics. 11 (10), 2019-2026 (2011).
- Yang, F., Shen, Y., Camp, D. G., Smith, R. D. High-pH reversed-phase chromatography with fraction concatenation for 2D proteomic analysis. Expert Review of Proteomics. 9 (2), 129-134 (2012).
- Niu, M., et al. Extensive peptide fractionation and y(1) ion-based interference detection method for enabling accurate quantification by isobaric labeling and mass spectrometry. Analytical Chemistry. 89 (1), 2956-2963 (2017).
- Wang, X., et al. A tag-based database search tool for peptide identification with high sensitivity and accuracy. Molecular & Cellular Proteomics. 13 (12), 3663(2014).
- Li, Y., et al. JUMPg: An integrative proteogenomics pipeline identifying unannotated proteins in human brain and cancer cells. Journal of Proteome Research. 15 (7), 2309-2320 (2016).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nature Methods. 4 (3), 207-214 (2007).
- Wang, Z., et al. 27-plex tandem mass tag mass spectrometry for profiling brain proteome in Alzheimer's disease. Analytical Chemistry. 92 (10), 7162-7170 (2020).
- Ow, S. Y., et al. iTRAQ underestimation in simple and complex mixtures: "The good, the bad and the ugly". Journal of Proteome Research. 8 (11), 5347-5355 (2009).
- Karp, N. A., et al. Addressing accuracy and precision issues in iTRAQ quantitation. Molecular & Cellular Proteomics. 9 (9), 1885-1897 (2010).
- Ting, L., Rad, R., Gygi, S. P., Haas, W. MS3 eliminates ratio distortion in isobaric multiplexed quantitative proteomics. Nature Methods. 8 (11), 937-940 (2011).
- Gygi, J. P., et al. A triple knockout isobaric-labeling quality control platform with an integrated online database search. Journal of The American Society for Mass Spectrometry. 31 (7), 1344-1349 (2020).
- Savitski, M. M., et al. Measuring and managing ratio compression for accurate iTRAQ/TMT quantification. Journal of Proteome Research. 12 (8), 3586-3598 (2013).
- Ong, S. E., et al. Stable isotope labeling by amino acids in cell culture, SILAC, as a simple and accurate approach to expression proteomics. Molecular & Cellular Proteomics. 1 (5), 376(2002).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513(2014).
- Brenes, A., Hukelmann, J., Bensaddek, D., Lamond, A. I. Multibatch TMT reveals false positives, batch effects and missing values. Molecular & Cellular Proteomics. 18 (10), 1967-1980 (2019).
- Yu, J., Peng, J., Chi, H. Systems immunology: Integrating multi-omics data to infer regulatory networks and hidden drivers of immunity. Current Opinion in Systems Biology. 15, 19-29 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon