Method Article
Profilazione del proteoma profondo e ad alta produttività mediante etichettatura di tag di massa tandem a 16 plex abbinata a cromatografia bidimensionale e spettrometria di massa
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui viene presentato un protocollo ottimizzato ad alto rendimento sviluppato con reagenti per tag di massa tandem a 16 plex, che consente la profilazione quantitativa del proteoma di campioni biologici. L'ampio frazionamento del pH basico e l'LC-MS/MS ad alta risoluzione mitigano la compressione del rapporto e forniscono una copertura profonda del proteoma.
Abstract
La marcatura con tag di massa tandem isobarico (TMT) è ampiamente utilizzata in proteomica grazie alla sua elevata capacità di multiplexing e alla profonda copertura del proteoma. Recentemente, è stato introdotto un metodo TMT espanso a 16 plex, che aumenta ulteriormente la produttività degli studi proteomici. In questo manoscritto, presentiamo un protocollo ottimizzato per la profilazione del proteoma profondo basata su TMT a 16 plex, che include la preparazione di campioni proteici, la digestione enzimatica, la reazione di marcatura TMT, il frazionamento della cromatografia liquida bidimensionale in fase inversa (LC/LC), la spettrometria di massa tandem (MS/MS) e l'elaborazione computazionale dei dati. Vengono evidenziate le fasi cruciali del controllo di qualità e i miglioramenti nel processo specifico per l'analisi TMT a 16 plessi. Questo processo multiplex offre un potente strumento per la profilazione di una varietà di campioni complessi come cellule, tessuti e campioni clinici. Più di 10.000 proteine e modifiche post-traduzionali come fosforilazione, metilazione, acetilazione e ubiquitinazione in campioni biologici altamente complessi da un massimo di 16 campioni diversi possono essere quantificate in un singolo esperimento, fornendo un potente strumento per la ricerca di base e clinica.
Introduzione
I rapidi sviluppi della tecnologia della spettrometria di massa hanno permesso di ottenere un'elevata sensibilità e una copertura profonda del proteoma nelle applicazioni di proteomica 1,2. Nonostante questi sviluppi, il multiplexing dei campioni rimane il collo di bottiglia per i ricercatori che gestiscono l'analisi di un'ampia coorte di campioni.
Le tecniche di marcatura isobarica multiplexata sono ampiamente utilizzate per la quantificazione relativa a livello di proteoma di grandi lotti di campioni 3,4,5,6. La quantificazione basata su tag di massa tandem (TMT) è una scelta popolare per la sua elevata capacità di multiplexing 7,8. I reagenti TMT sono stati inizialmente lanciati come kit a 6 plex in grado di quantificare fino a 6 campioni contemporaneamente9. Questa tecnologia è stata ulteriormente ampliata per quantificare 10-11 campioni10,11. I reagenti TMTpro a 16 plex di recente sviluppo (di seguito denominati TMT16) hanno ulteriormente aumentato la capacità di multiplexing a 16 campioni in un singolo esperimento12,13. I reagenti TMT16 utilizzano un gruppo reporter a base di prolina, mentre il TMT a 11-plex applica un gruppo reporter derivato dalla dimetilpiperidina. Sia TMT11 che TMT16 utilizzano lo stesso gruppo reattivo amminico, ma il gruppo di bilancio di massa di TMT16 è più grande di quello di TMT11, consentendo la combinazione di 8 isotopi stabili C13 e N15 negli ioni reporter per ottenere 16 reporter (Figura 1).
L'aumento della capacità di multiplexing fornisce una piattaforma per la progettazione di esperimenti con repliche sufficienti per superare le sfide statistiche14. Inoltre, i canali aggiuntivi nel TMT a 16 plessi aiutano a ridurre la quantità totale di materiale di partenza per canale, il che può aiutare nello sviluppo della proteomica emergente a singola cellula15. L'elevata capacità di multiplexing sarà preziosa anche per la quantificazione delle modifiche post-traduzionali, che tipicamente richiede elevate quantità di materiale di partenza16,17.
I flussi di lavoro proteomici che utilizzano la tecnologia TMT sono stati semplificati 18,19,20 e si sono evoluti in modo significativo nell'ultimo decennio in termini di preparazione dei campioni, separazione con cromatografia liquida, acquisizione di dati con spettrometria di massa e analisi computazionale 21,22,23,24,25,26. Il nostro precedente articolo fornisce una panoramica approfondita della piattaforma TMT a 10 plessi27. Il protocollo qui descritto introduce un metodo dettagliato e ottimizzato per TMT16, che include l'estrazione e la digestione delle proteine, la marcatura TMT16, il pooling e la desalinizzazione dei campioni, il pH basico e la LC in fase inversa (RP) del pH acido, la MS ad alta risoluzione e l'elaborazione dei dati (Figura 2). Il protocollo evidenzia anche le fasi chiave del controllo di qualità che sono state incorporate per completare con successo un esperimento di proteomica quantitativa. Questo protocollo può essere utilizzato di routine per identificare e quantificare più di 10.000 proteine con elevata riproducibilità, per studiare percorsi biologici, processi cellulari e progressione della malattia 20,28,29,30.
Protocollo
I tessuti umani per lo studio sono stati ottenuti con l'approvazione del Brain and Body Donation Program presso il Banner Sun Health Research Institute.
1. Estrazione di proteine dai tessuti e controllo qualità
NOTA: Per ridurre l'impatto della raccolta dei campioni sul proteoma, è fondamentale raccogliere i campioni in un tempo minimo a bassa temperatura, se possibile31. Ciò è particolarmente importante quando si analizzano le modificazioni post-traduzionali poiché sono tipicamente labili, ad esempio, alcuni eventi di fosforilazione hanno solo pochi secondi di emivita32,33.
- Accisa e pesa campioni di tessuto
- Tarare una provetta da microcentrifuga da 1,5 mL utilizzando una bilancia analitica e pre-raffreddare la provetta su ghiaccio secco.
- Tagliare un tessuto congelato (ad es. tessuto cerebrale umano, ~10 mg) da una regione definita in piccoli pezzi e trasferire immediatamente i pezzi di tessuto nella provetta pre-raffreddata.
NOTA: Per ridurre l'eterogeneità del campione, è importante utilizzare dimensioni omogenee e regioni anatomiche per tutti i 16 campioni. La quantità di proteine ottenute dal tessuto è in genere il 5-10% del peso del tessuto. - Pesare il tubo insieme al fazzoletto e mettere immediatamente il tubo sul ghiaccio secco. Elaborare i restanti 15 campioni utilizzando la stessa procedura. Conservare i campioni in ghiaccio secco subito dopo la dissezione e conservarli a ˗80 °C.
- Campioni di tessuto di lisi
- Preparare un tampone di lisi fresco (50 mM HEPES pH 8,5, 8 M urea e sodio desossicolato allo 0,5%) il giorno dell'esperimento. Gli inibitori della fosfatasi devono essere aggiunti al tampone di lisi per preservare lo stato di fosforilazione delle proteine.
NOTA: Mettere il tampone di lisi a temperatura ambiente prima di utilizzarlo, poiché l'urea 8 M precipiterà sul ghiaccio, il che può causare una denaturazione incompleta delle proteine durante la lisi del campione e ridurre l'efficienza della digestione delle proteine. - Aggiungere il tampone di lisi (aggiungere 100 μL di tampone di lisi per 10 mg di tessuto per ottenere una concentrazione proteica finale da 5 a 10 μg/μL) e le perle di vetro (~20% del volume del lisato, 0,5 mm di diametro) a ciascun campione.
- Lisare il fazzoletto in un frullatore a 4 °C con una velocità di 8 per 30 s, riposare per 5 s, ripetere fino a quando i campioni non sono omogeneizzati (~ 5 cicli).
- Preparare un tampone di lisi fresco (50 mM HEPES pH 8,5, 8 M urea e sodio desossicolato allo 0,5%) il giorno dell'esperimento. Gli inibitori della fosfatasi devono essere aggiunti al tampone di lisi per preservare lo stato di fosforilazione delle proteine.
- Preparare aliquote dei lisati.
- Preparare almeno due aliquote per ogni campione. Una piccola aliquota (~10 μL) viene utilizzata per l'analisi della concentrazione proteica e la valutazione della qualità delle proteine (ad esempio, la validazione del western blotting di proteine a controllo positivo). Per l'analisi del proteoma viene utilizzata un'aliquota più grande (~50 μL).
- Congelare immediatamente le aliquote su ghiaccio secco e conservarle a -80 °C fino a nuovo utilizzo.
- Misura la concentrazione di proteine
NOTA: La concentrazione proteica può essere misurata con il saggio BCA o con il metodo di colorazione con gel SDS breve34 (Figura 3A). Poiché i componenti non riducenti le proteine nel lisato tissutale possono influenzare la misurazione nel saggio BCA, gli utenti possono convalidare la concentrazione proteica con il metodo di colorazione con gel SDS corto. Il metodo di colorazione con gel SDS corto è presentato qui.- Diluire 16 campioni aliquotati per 10 volte e preparare lo standard BSA (ad esempio, titolazioni BSA di 0,15, 0,5, 1,5 e 3 μg).
- Eseguire i campioni e lo standard BSA su un gel SDS-PAGE al 10% (26 pozzetti) con un gel di impilamento fino a quando tutte le proteine migrano di circa 3 mm nel gel.
- Colorare il gel con il blu Coomassie per 1 ora e smacchiare il gel fino a quando lo sfondo nella regione vuota è chiaro.
- Scansiona il gel per misurare l'intensità delle bande proteiche colorate con Coomassie con ImageJ e crea una curva standard BSA in base alle misurazioni.
- Calcola la concentrazione proteica assoluta con la curva standard.
NOTA: La concentrazione proteica finale per ogni campione in questo esperimento era di ~5-10 μg/μL. Per l'analisi proteomica basata su TMT16, sono sufficienti 50 μg di proteine per campione (0,8 mg totali di proteine per l'analisi dell'intero proteoma).
- Controllo qualità dei campioni
NOTA: Questa fase del controllo qualità è fondamentale per identificare campioni di bassa qualità prima di eseguire l'analisi TMT. Per i campioni con cambiamento proteico noto, si suggerisce di convalidare il cambiamento mediante western blotting. Si raccomanda inoltre l'analisi SDS-PAGE standard per esaminare i pattern proteici ed escludere eventuali campioni con alti gradi di degradazione (Figura 3B).- Prelevare ~10 μg di ciascun campione dall'aliquota piccola ed eseguire i campioni su un gel SDS-PAGE a gradiente fino a quando il colorante blu di bromofenolo raggiunge il fondo del gel.
- Colorare il gel con il blu Coomassie e smacchiare il gel. Ispezionare la qualità delle proteine per rimuovere i campioni proteici altamente degradati.
NOTA: I campioni degradati possono essere identificati come campioni che hanno pochissime bande proteiche nella regione ad alto peso molecolare e bande intensificate nella regione a basso peso molecolare (Figura 3B).
2. Digestione delle proteine in soluzione, riduzione e alchilazione dei peptidi, test di efficienza della digestione e desalinizzazione dei peptidi
- Digestione delle proteine da parte di Lys-C e tripsina
- Prelevare ~50 μg di proteine dall'aliquota grande di ciascun campione e aggiungere il tampone di lisi a 50 μl.
- Aggiungere il 100% di acetonitrile (ACN) per raggiungere una concentrazione finale del 10%.
- Eseguire la digestione di Lys-C aggiungendo Lys-C a un rapporto proteine:Lys-C di 100:1 (p/p) e incubando a temperatura ambiente per 3 ore.
- I campioni vengono diluiti per contenere una concentrazione finale di 2 M di urea per 50 mM di HEPES (pH 8,5).
- Aggiungere la tripsina in ogni campione con un rapporto proteine:tripsina di 50:1 (p/p) ed eseguire la digestione a temperatura ambiente per 3 ore o durante la notte.
- Riduzione e alchilazione dei peptidi
- Aggiungere la soluzione di ditiotreitolo (1 M DTT) appena preparata a una concentrazione finale di 1 mM e incubare per 1 ora a temperatura ambiente per ridurre i legami disolfuro.
- Aggiungere la soluzione di iodoacetammide (1 M IAA) appena preparata a una concentrazione finale di 10 mM per 30 minuti al buio per ottenere residui di cisteina alchilata.
- Estinguere l'IAA non reagito aggiungendo 1M DTT a una concentrazione finale di 30 mM e incubare a temperatura ambiente per altri 30 minuti.
- Esaminare l'efficienza della digestione
- Prelevare ~1 μg di ciascun campione e dissalare utilizzando puntali per pipette rivestiti di resina C18 secondo il protocollo del produttore.
- Analizzare ogni campione con una breve corsa LC-MS/MS a gradiente (vedere maggiori dettagli nel passaggio 5).
- Eseguire una ricerca nel database per i dati grezzi MS (vedere ulteriori dettagli nel passaggio 6). Calcolare la percentuale di peptidi identificati con almeno un sito di misclivage della tripsina. La percentuale è solitamente inferiore al 15%.
- Se la percentuale è superiore al 15%, aggiungere altra tripsina ai campioni per ripetere la digestione.
- Dopo la digestione, acidificare i campioni aggiungendo TFA allo 0,5% (v/v). Controllare il pH con una striscia di pH per assicurarsi che il pH sia inferiore a 3.
- Desalinizzazione dei peptidi
- Centrifugare i peptidi acidificati a 21.000 x g per 10 minuti. Trasferire i surnatanti in una nuova provetta.
- Lavare due volte le colonne di desalinizzazione C18 (~25 μL di resina) con 250 μL di metanolo al 100% centrifugando a 500 x g per 30 s.
NOTA: Per ridurre la perdita di peptidi durante il processo di desalinizzazione, scegliere colonne di desalinizzazione con capacità di legante corrispondente alla quantità di input. - Lavare le colonne due volte utilizzando 250 μL di tampone di eluizione (60% ACN, 0,1% TFA) centrifugando a 500 x g per 30 s.
- Equilibrare le colonne due volte con 250 μL di tampone di equilibratura e lavaggio (0,1% TFA) centrifugando a 500 x g per 30 s.
- Caricare i campioni sulle colonne pre-equilibrate. Lasciare che i campioni si leghino alle colonne ruotando a 100 x g per 6 minuti. Assicurarsi che tutta la soluzione sia passata attraverso la colonna.
- Lavare le colonne tre volte con 250 μL di equilibrante e tampone di lavaggio centrifugando a 500 x g per 30 s.
- Eluire i peptidi aggiungendo 125 μL di tampone di eluizione a ciascuna colonna e centrifugare a 100 x g per 3 minuti. Verificare che la colonna non contenga alcuna soluzione avanzata.
- Essiccare i peptidi eluiti in un concentratore sottovuoto e conservare i peptidi a -80 °C per la futura marcatura TMT.
3. Marcatura TMT16 dei peptidi, test di efficienza dell'etichettatura, raggruppamento dei campioni e desalinizzazione dei peptidi marcati
- Marcatura TMT16 di peptidi
- Risospendere ogni campione di peptide desalinizzato in 50 μL di 50 mM HEPES (pH 8,5) mediante vortice più volte o dissolvenza a ultrasuoni, seguita dall'uso di una striscia di pH per verificare il pH.
NOTA: Il campione può essere acido se non viene asciugato completamente prima dell'etichettatura, il che influisce negativamente sull'efficienza dell'etichettatura. Assicurarsi che il pH sia compreso tra 7 e 8. - Prelevare ~1 μg di peptidi non marcati da ciascun campione come controlli negativi per il test di efficienza di marcatura TMT.
- Sciogliere i reagenti TMT16 in ACN anidro. Eseguire la reazione di marcatura aggiungendo i reagenti a un rapporto TMT:proteina di 1,5:1 (p/p) e incubando a temperatura ambiente per 30 minuti.
NOTA: Il rapporto TMT:proteine utilizzato per TMT16 è superiore del 50% rispetto al rapporto utilizzato per TMT11. Questa piccola discrepanza può essere dovuta al fatto che la massa molecolare di TMT16 è maggiore (1,2 volte) di quella dei reagenti TMT11. La quantità di proteine viene stimata dai campioni senza considerare la perdita durante la desalinizzazione.
- Risospendere ogni campione di peptide desalinizzato in 50 μL di 50 mM HEPES (pH 8,5) mediante vortice più volte o dissolvenza a ultrasuoni, seguita dall'uso di una striscia di pH per verificare il pH.
- Test di efficienza dell'etichettatura
- Prelevare ~1 μg di peptidi marcati da ciascun campione per il test di efficienza della marcatura. Mettere i campioni rimanenti a -80 °C senza spegnere la reazione.
- Dissalare ~1 μg di campioni marcati con TMT16 e non marcati mediante puntali per pipette rivestiti di resina C18 secondo il protocollo del produttore.
- Analizzare i campioni mediante LC-MS/MS (vedere la sezione 5, con l'eccezione che il gradiente è di 10 minuti).
- Stimare l'efficienza della marcatura analizzando la riduzione dell'intensità MS1 dei peptidi non marcati tra campioni non marcati e marcati. Seleziona da 6 a 10 peptidi diversi per verificare l'efficienza dell'etichettatura e assicurarti che tutti i peptidi siano etichettati. Per la marcatura completa, non si osservano peptidi non marcati.
NOTA: È importante garantire l'etichettatura completa di tutti i campioni per un'identificazione e una quantificazione accurate delle proteine a valle. - Se l'etichettatura non è completa, aggiungere ulteriori reagenti TMT per etichettare i peptidi rimanenti e controllare nuovamente l'efficienza di marcatura prima di estinguerla. Dopo che il campione è stato completamente etichettato, estinguere la reazione a temperatura ambiente aggiungendo idrossilammina a una concentrazione finale dello 0,5% e incubare a temperatura ambiente per 15 minuti.
- Pooling e desalinizzazione dei campioni
- Raggruppare metà di ciascun campione marcato con TMT per ottenere una miscela.
- Prelevare 1 μg dalla miscela e dissalare con puntali per pipette rivestiti di resina C18, quindi analizzare con LC-MS/MS utilizzando un gradiente breve (~30 min).
- Calcolare la concentrazione relativa utilizzando l'intensità media di ogni ione reporter TMT16 e confrontando le discrepanze tra i 16 canali. Per ottenere una miscelazione uniforme di ciascun canale, aggiungere il resto dei campioni marcati con TMT nella miscela in base all'intensità media calcolata. Ripetere la regolazione fino a quando tutti i campioni non sono miscelati allo stesso modo. I dati rappresentativi che mostrano il processo di raggruppamento dei campioni sono mostrati nella Tabella 1.
NOTA: Poiché gli errori di pipettaggio possono influire sull'accuratezza delle concentrazioni e sulla quantificazione delle proteine, è importante garantire la corretta quantità di pooling. Le discrepanze di intensità tra 16 campioni devono essere inferiori al 5%.
- Desalinizzazione di peptidi etichettati
NOTA: Poiché i derivati di fondo nella reazione di marcatura TMT16 (ad esempio, TMTpro-NHOH dalla reazione di estinzione dell'idrossilammina e TMTpro-OH dall'idrossilazione TMT) sono idrofobici, viene utilizzata una condizione di lavaggio estesa per i campioni marcati con TMT16 per rimuovere efficacemente i derivati. Viene utilizzata l'aggiunta del 5% di ACN nel normale tampone di lavaggio (0,1% TFA) e 10 volte i volumi del letto di tampone di lavaggio.- Acidificare il campione aggregato aggiungendo il 10% di TFA a pH < 3.
- Centrifugare il campione aggregato a 21.000 x g per 10 minuti e mettere il surnatante in una nuova provetta.
- Asciugare il campione utilizzando un concentratore sottovuoto per rimuovere l'ACN.
- Precondizionare una cartuccia di estrazione in fase solida contenente 50 mg di colonna assorbente lavando la colonna con 2 mL di metanolo al 100%, seguiti da 2 mL di tampone di eluizione (60% ACN più 0,1% TFA) e infine 2 mL di tampone di lavaggio (0,1% TFA).
- Caricare l'esempio nella colonna. Regolare la portata a ~100 μL/min per garantire la piena estensione del legame peptidico. Salvare il flusso.
- Lavare la colonna tre volte con 1 mL di tampone di lavaggio.
- Eluire i peptidi con 1 mL di tampone di eluizione.
- Essiccare i peptidi eluiti in un concentratore sottovuoto e conservare i peptidi a -80 °C per un ulteriore frazionamento.
4. Pre-frazionamento LC di pH basico offline
- Preparazione del sistema di frazionamento
- Preparare il tampone A (10 mM di formiato di ammonio, pH 8,0) e il tampone B (10 mM di formiato di ammonio, 90% ACN, pH 8,0) per un sistema LC ad alte prestazioni con flusso di microlitri.
- Installazione di una colonna HPLC contenente particelle ibride di etilene a ponte (dimensione delle particelle di 3,5 μm, 4,6 mm × 25 cm) in un sistema LC ad alte prestazioni con flusso di microlitri per il frazionamento.
- Installare un circuito di campionamento da 100 μl e lavare il circuito con 300 μl di metanolo, acqua e tampone A, in sequenza.
- Utilizzare 100 μl di rapporto 1:1:1:1 di isopropanolo : metanolo : acetonitrile : acqua per lavare la colonna. Quindi, equilibrare ulteriormente la colonna per 0,5 ore al 95% del tampone A.
- Preparazione del campione
- Sciogliere il campione di TMT16 raccolto e desalinizzato in 70 μl di tampone A. Verificare che il pH del campione sia ~ 8,0. Se ancora acido, regolare il pH a 8,0 utilizzando idrossido di ammonio al 28% (NH4OH).
NOTA: Per evitare la perdita di campione, il volume del campione deve essere inferiore al 70% del volume del ciclo. - Centrifugare il campione a 21.000 x g per 10 minuti per rimuovere i precipitati.
- Sciogliere il campione di TMT16 raccolto e desalinizzato in 70 μl di tampone A. Verificare che il pH del campione sia ~ 8,0. Se ancora acido, regolare il pH a 8,0 utilizzando idrossido di ammonio al 28% (NH4OH).
- Frazionamento e concatenazione
NOTA: Prima del frazionamento del campione reale, si consiglia vivamente un esperimento pilota per garantire che il sistema LC sia in buone condizioni. Questo può essere eseguito con una piccola quantità del campione effettivo (~5%) o con una miscela di peptidi non marcata con TMT.- Iniettare il campione e frazionarlo con il seguente gradiente: 5% di tampone B per 10 minuti, 5-15% di tampone B per 2 minuti, 15-45% di tampone B per 148 minuti e 45-95% di tampone B per 5 minuti. Utilizzare una velocità di flusso di 0,4 mL/min.
- Imposta il raccoglitore di frazioni per raccogliere le frazioni ogni 1 minuto e concatenare 160 frazioni a 40 frazioni in 4 cicli.
NOTA: La concatenazione viene eseguita combinando frazioni LC precoci, medie e tardive eluite dagli interni dello stesso tempo in una frazione concatenata. Le frazioni concatenate hanno poca sovrapposizione nella prima dimensione della LC, aumentando così l'uso efficiente della finestra di eluizione nella seconda dimensione LC. Inoltre, attraverso diversi cicli di concatenazione, i peptidi possono essere distribuiti uniformemente su tutte le frazioni concatenate. Questo approccio ha dimostrato di aumentare la copertura del proteoma rispetto all'analisi delle singole frazioni 35,36. - Essiccare tutte le frazioni concatenate in un concentratore sottovuoto e conservare i campioni essiccati a -80 °C per ulteriori analisi LC-MS/MS.
5. Analisi del pH acido RPLC-MS/MS
- Preparazione del sistema RPLC-MS/MS con pH acido
- Imballare una colonna vuota (75 μm ID con orifizio della punta da 15 μm) con resina C18 da 1,9 μm a 10-15 cm di lunghezza.
- Riscaldare la colonna a 65 °C utilizzando un riscaldatore a portafoglio a farfalla per ridurre la contropressione.
- Lavare accuratamente la colonna con tampone B al 95% (3% dimetilsolfossido, 0,2% acido formico e 67% ACN). Quindi, equilibrare completamente la colonna nel tampone A al 95% (3% di dimetilsolfossido e 0,2% di acido formico).
- Controllare le prestazioni del sistema LC-MS/MS eseguendo 100 ng di peptidi cerebrali di ratto o peptidi BSA prima di analizzare i campioni sperimentali.
- Analisi LC-MS/MS di frazioni concatenate
- Ricostituire i peptidi essiccati dalle frazioni basiche del pH in 5% di acido f.a. e centrifugare a 21.000 × g per 5 minuti. Trasferire il surnatante di ciascun campione in un foglietto per fiala HPLC.
- Caricare ~1 μg di peptidi di ciascuna frazione sulla colonna. I peptidi vengono eluiti a una velocità di flusso di 0,25 μL/min con un gradiente di 60 min del 18-45% del tampone B.
NOTA: Per ottenere numeri di identificazione elevati, eseguire una frazione e regolare il gradiente per le frazioni rimanenti in base alla prima esecuzione. Il gradiente migliore dovrebbe avere peptidi distribuiti uniformemente su tutto il gradiente (Figura 4A). - Utilizzare lo spettrometro di massa con i seguenti parametri per l'analisi di campioni marcati con TMT16: Scansioni MS1 (intervallo di scansione MS completo: 450-1600 m/z; Risoluzione Orbitrap: 60.000; Target di controllo automatico del guadagno: 1 x 106; tempo massimo di utilizzo dello ione: 50 ms) e 20 scansioni MS2 dipendenti dai dati (risoluzione Orbitrap: 60.000; Bersaglio AGC: 1 x 105; tempo massimo di ione: 110 ms; Energia di collisione normalizzata HCD: 32%; finestra di isolamento: 1,0 m/z; Offset di isolamento: 0,2 m/z; esclusione dinamica: 10 s).
NOTA: I parametri utilizzati qui sono ottimizzati su un tipo di spettrometro di massa (vedi Tabella dei materiali). Per i diversi strumenti MS, gli utenti devono mettere a punto i parametri dello strumento per ottenere risultati di alta qualità. Un'impostazione consiste nel monitorare l'energia di collisione HCD normalizzata, poiché l'energia ottimale può variare tra gli strumenti e tra TMT11 e TMT16.
6. Trattamento dei dati
NOTA: L'analisi dei dati è stata eseguita utilizzando una suite software JUMP 37,38,39 che include un motore di ricerca di database ibrido (basato su pattern e tag), un software di filtraggio che controlla il tasso di falsa scoperta (FDR) di peptidi/proteine identificati e un software di quantificazione per i set di dati TMT. A seconda della situazione dell'utente, l'analisi dei dati può essere eseguita utilizzando altri programmi commerciali o disponibili gratuitamente.
- Ricerca nel database
- Converti i file .raw dallo strumento MS in file .mzXML e cerca gli spettri MS2 in un database di esche target non ridondante40 generato dalle sequenze proteiche umane UniProt (o da un altro database specie-specifico appropriato) per calcolare l'FDR delle proteine identificate.
NOTA: Generare il database non ridondante combinando sequenze proteiche dai database Swiss-Prot e TrEMBL. Si possono anche aggiungere sequenze proteiche personalizzate non contenute in quei database di riferimento, comprese le proteine scisse della proteasi, le proteine con polimorfismi a singolo nucleotide e i contaminanti comuni. - Effettuare ricerche utilizzando i seguenti parametri. Tolleranza di massa per il precursore: 10 ppm; tolleranza di massa per gli ioni del prodotto: 15 ppm; scollature mancate massime: 2; siti di modifica massimali: 3; modifiche statiche: 304.20715 Da per i tag TMT16 su residui di Lys e N terminali, 57.02146 Da per carbamidometilazione su residui di Cys; modifica dinamica: 15,99492 Da per ossidazione su Met.
- Converti i file .raw dallo strumento MS in file .mzXML e cerca gli spettri MS2 in un database di esche target non ridondante40 generato dalle sequenze proteiche umane UniProt (o da un altro database specie-specifico appropriato) per calcolare l'FDR delle proteine identificate.
- Filtrare i risultati della ricerca
- Filtra le corrispondenze dello spettro peptidico (PSM) risultanti in base alla lunghezza del peptide (>6 amminoacidi), all'accuratezza della massa dello ione precursore e ai punteggi di corrispondenza basati su JUMP (Jscore e ΔJn). I peptidi vengono quindi raggruppati in base alla lunghezza del peptide, alle estremità triptiche, alle modifiche, ai siti di scissione mancati e allo stato di carica.
- Filtra ulteriormente i dati con i punteggi corrispondenti per ottenere un FDR inferiore all'1% a livello di proteina (analisi dell'intero proteoma) o di peptide (analisi del fosfoproteoma).
NOTA: Se i peptidi/proteine a controllo positivo mancano nelle fasi di filtraggio, l'FDR può essere aumentato a un livello ragionevole in modo che tali peptidi/proteine possano essere recuperati. - Per i peptidi condivisi da più di un membro di una famiglia di proteine, raggruppare i membri abbinati in un unico gruppo.
NOTA: Con la regola della parsimonia, il gruppo è rappresentato dalla proteina omologa con il maggior numero di peptidi condivisi e altre proteine abbinate da peptidi unici.
- Quantificazione delle proteine
- Quantifica le proteine utilizzando un programma integrato di una suite di software statistici per riassumere le intensità degli ioni reporter TMT su tutti i PSM abbinati.
- Estrarre le intensità degli ioni reporter TMT da ciascun PSM accettato e correggere le intensità grezze in base alla distribuzione isotopica di ciascun reagente di marcatura (ad esempio, TMT16-126 genera rispettivamente il 92,6%, il 7,2% e lo 0,2% degli ioni m/z 126, 127C e 128C) e filtrare i PSM a bassa intensità e/o altamente rumorosi sulla base di soglie definite dall'utente. Normalizzare i dati di quantificazione utilizzando le intensità medie (o mediane) dei campioni per correggere la distorsione di caricamento.
- Per ogni proteina identificata, calcolare le intensità medie centrate tra i campioni (cioè le intensità relative) dei PSM abbinati e riassumere le intensità relative dei PSM prendendo la media per campione. Converti i segnali relativi in quelli assoluti moltiplicando l'intensità media complessiva dei tre PSM abbinati più abbondanti.
- Corretta quantificazione dell'interferenza utilizzando un approccio di correzione ionica y1 precedentemente riportato37 che presuppone che l'intensità dello ione y1 sia correlata all'intensità dello ione reporter. Stimando la relazione lineare tra l'intensità degli ioni y1 e quella del reporter dalle scansioni pulite, viene derivato e corretto il livello di interferenza dall'intensità degli ioni y1 contaminati nelle scansioni rumorose.
NOTA: Per i peptidi triptici marcati con TMT, i residui K-TMT e R sono due ioni y1 rappresentativi (376,27574 Da e 175,11895 Da, rispettivamente) in uno spettro MS2. Se viene rilevato un solo ione y1 ed è coerente con il peptide identificato, l'MS2 è considerato una scansione pulita. Se vengono rilevati entrambi gli ioni y1, l'MS2 è considerato una scansione rumorosa. - Trasferisci i valori di quantificazione delle proteine su un foglio di calcolo per ulteriori analisi. Utilizza metodi di analisi dei dati non supervisionati come la PCA o l'analisi di clustering per esplorare la distribuzione dei campioni. Per identificare le proteine differenzialmente espresse, utilizzare metodi statistici come il t-test e l'analisi della varianza (ANOVA).
7. Convalida dei dati MS
NOTA: Prima di eseguire esperimenti biologici che richiedono molto tempo, utilizzare almeno un metodo di convalida per valutare la qualità dei dati MS.
- Ispezionare manualmente gli spettri MS/MS delle proteine di interesse per convalidare la sequenza peptidica e l'intensità degli ioni reporter TMT.
- Utilizzare approcci basati su anticorpi (ad esempio, western blotting o analisi immunoistochimica) per verificare le variazioni dei livelli proteici. Per confermare la presenza di peptidi nativi, utilizzare i peptidi sintetici come standard interni. Gli spettri MS/MS e il tempo di ritenzione dei peptidi durante la LC-MS/MS dovrebbero essere identici nelle stesse condizioni.
- Utilizzare un approccio mirato alla SM per verificare i cambiamenti delle proteine.
Risultati
Il protocollo per il TMT16 di nuova concezione, che include la reazione di marcatura, la desalinizzazione e le condizioni LC-MS, è stato sistematicamente ottimizzato41. Inoltre, abbiamo confrontato direttamente i metodi 11-plex e 16-plex utilizzandoli per analizzare gli stessi campioni di AD umano41. Dopo l'ottimizzazione dei parametri chiave per TMT16, entrambi i metodi TMT11 e TMT16 producono una copertura, un'identificazione e una quantificazione del proteoma simili > 100.000 peptidi in > 10.000 proteine umane.
Poiché i reagenti TMT16 sono più idrofobici dei reagenti TMT11, è probabile che i peptidi marcati con TMT16 siano più idrofobici dei peptidi marcati con TMT11, il che può spiegare il diverso tempo di ritenzione (RT) nell'RPLC. Pertanto, abbiamo valutato l'impatto di TMT16 sul peptide RT rispetto a TMT11 analizzando la miscela peptidica marcata con TMT11 e TMT16 utilizzando LC-MS/MS. Abbiamo scoperto che TMT16 ha un'influenza significativa sulla RT per i peptidi con idrofobicità media, ma ha scarso effetto sui peptidi di idrofobicità estremamente alta o bassa. Pertanto, le concentrazioni simili di tampone B iniziale e finale nel gradiente LC possono essere utilizzate per diversi peptidi marcati con TMT.
Abbiamo quindi ottimizzato il gradiente RPLC online per il campione etichettato con TMT16. Il gradiente per TMT16 è molto simile a quello di TMT11. La percentuale del buffer iniziale e finale B è la stessa (ad esempio, dal 18% al 45%). Ma abbiamo notato che il numero di peptidi identificati in TMT16 è diminuito rapidamente di circa il 40% del tampone B quando si utilizza lo stesso gradiente utilizzato per TMT11. Pertanto, abbiamo leggermente ridotto il tempo di pendenza tra il 40% e il 45%. Abbiamo anche apportato piccole modifiche a questo gradiente per diverse frazioni e diversi campioni. Dopo l'ottimizzazione del gradiente, i peptidi identificati sono stati distribuiti uniformemente in tutto il gradiente (Figura 4A).
Per massimizzare il numero di proteine identificate e quantificate con precisione utilizzando il metodo TMT16, abbiamo ottimizzato l'energia di collisione normalizzata (NCE) per i campioni marcati con TMT16 nel nostro precedente rapporto41. Diversi NCE (dal 20% al 40%) sono stati testati sullo spettrometro di massa durante le esecuzioni LC-MS/MS. Bilanciando il numero di identificazioni proteiche e l'intensità dello ione reporter, è stato scelto un NCE del 30-32,5% come energia di collisione HCD ottimale da utilizzare per i campioni marcati con TMT16.
La compressione del rapporto causata da ioni interferenti co-eluiti è stata una limitazione delle tecniche di marcatura isobarica per la quantificazione delle proteine. Uno studio pubblicato in precedenza che utilizza il metodo TMT11 mostra che la compressione del rapporto può essere quasi eliminata da un'estesa pre-frazionamento LC, impostazioni MS ottimizzate e strategie di correzione dei dati post-MS37. Abbiamo utilizzato queste strategie, tra cui il frazionamento estensivo pre-MS (40 frazioni di LC a pH basico), l'applicazione di una finestra di isolamento ristretta (1 m/z) nell'impostazione MS e la correzione ionica y1 nelle analisi del proteoma TMT11 e TMT16 degli stessi campioni. Dopo aver esaminato la curva di correlazione del cambiamento di ripiegamento proteico tra i set di dati TMT11 e TMT16, abbiamo scoperto che la pendenza era molto vicina a 1, indicando che la compressione del rapporto in TMT16 non era visibilmente superiore a quella in TMT11 nella nostra condizione sperimentale41. I risultati coerenti sono stati riportati che il rapporto di compressione non ha differenze quando il livello di multiplexing è stato aumentato da 11 a16 13,45. Pertanto, le strategie pubblicate in precedenza possono essere utilizzate per alleviare la compressione del rapporto, migliorando così significativamente l'accuratezza della quantificazione 27,37,44,46.
Infine, abbiamo confrontato il numero di PSM, peptidi unici e proteine uniche quantificati in campioni marcati con TMT11 rispetto a campioni marcati con TMT16 (Figura 4B). I risultati mostrano che i PSM di entrambi i metodi sono comparabili; tuttavia, le proteine e i peptidi quantificati sono leggermente inferiori nel metodo TMT16, il che è coerente con altri rapporti12,13. I nostri risultati indicano che i miglioramenti nel processo TMT16 insieme all'uso di parametri LC-MS ottimizzati forniscono una profilazione del proteoma profonda e ad alto rendimento dei campioni biologici.
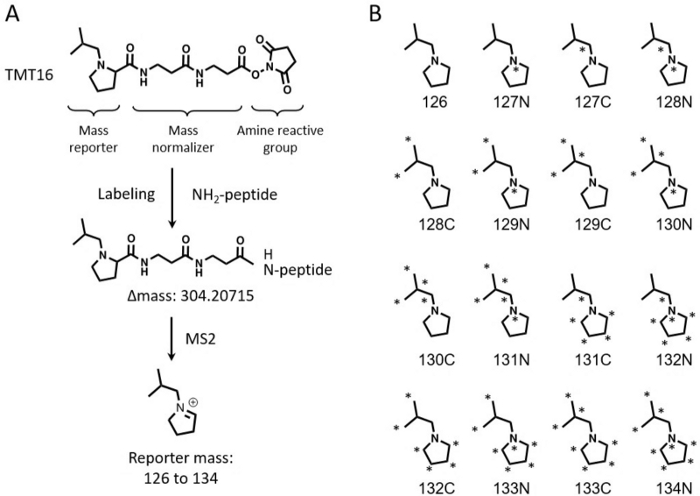
Figura 1: Struttura del reagente TMT a 16 plessi. (A) Vengono mostrati la struttura del reagente TMT a 16 plessi, il processo di marcatura, lo spostamento di massa dopo la marcatura e la massa dello ione reporter. (B) Strutture marcate con isotopi pesanti degli ioni reporter dei reagenti TMT16. Clicca qui per visualizzare una versione più grande di questa figura.
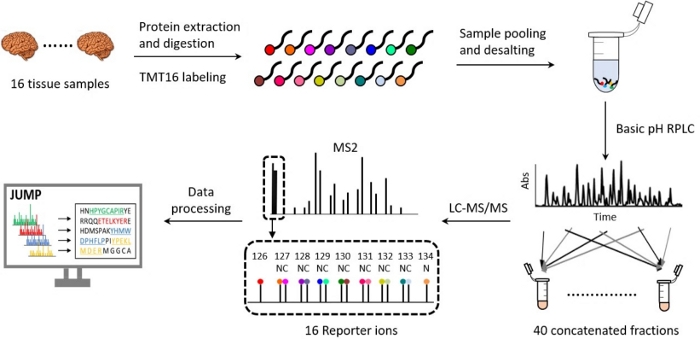
Figura 2: Flusso di lavoro della profilazione del proteoma mediante 16-plex TMT-LC/LC-MS/MS. Le proteine estratte da 16 campioni di tessuto biologico sono state digerite ed etichettate con 16 diversi tag TMT. I campioni provenienti da 16 canali vengono raggruppati in modo uniforme e la miscela viene frazionata e concatenata in 40 frazioni mediante cromatografia liquida in fase inversa (RPLC) di base a pH offline. Ogni frazione viene ulteriormente analizzata mediante RPLC acido accoppiato con spettrometria di massa ad alta risoluzione. I file non elaborati MS/MS sono stati elaborati. L'immagine del tessuto cerebrale è citata da Medium.com con alcune modifiche. Clicca qui per visualizzare una versione più grande di questa figura.
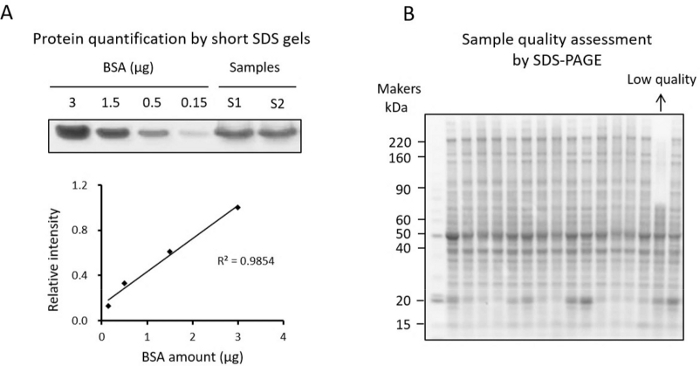
Figura 3: Controllo della qualità delle proteine. (A) Quantificazione delle proteine estratte dal tessuto su un gel SDS corto con BSA come standard. La curva standard rappresenta la concentrazione di BSA e l'intensità della banda proteica colorata con Coomassie utilizzata per la quantificazione. (B) Gel SDS utilizzato per il test della qualità delle proteine. Clicca qui per visualizzare una versione più grande di questa figura.
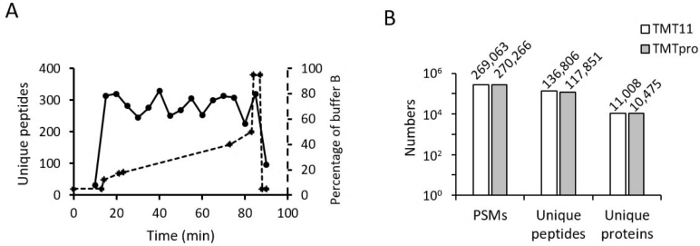
Figura 4: Risultati rappresentativi. (A) Distribuzione peptidica in LC acido. Il gradiente ottimizzato del buffer B dopo la correzione del volume morto è allineato nello stesso grafico. (B) L'istogramma mostra il numero di PSM quantificato, peptide unico e proteina unica nei metodi TMT11 e TMT16. Clicca qui per visualizzare una versione più grande di questa figura.
| 1° (50 μl, utilizzare il 50% nella prima miscela) | 2° (aggiusta la miscela e risparmia il 10%) | 3a (regolazione finale) | |||||||||||
| Canali | Giornalisti | Miscela Vol (μL) | Intensità (unità) | Conc. (unità/μL) | Intensità prevista (unità) | Vol aggiunto (μL) | Vol totale (μL) | Intensità (unità) | Conc. (unità/μL) | Intensità prevista (unità) | Vol aggiunto (μL) | Vol totale (μL) | Intensità (unità) |
| 1 | Sig126 | 25 | 94.7 | 3.8 | 122.1 | 7.2 | 32.2 | 99.6 | 3.1 | 105.3 | 1.8 | 34.1 | 100 |
| 2 | sig127N | 25 | 83 | 3.3 | 122.1 | 11.8 | 36.8 | 101.1 | 2.7 | 105.3 | 1.5 | 38.3 | 98 |
| 3 | sig127C | 25 | 86 | 3.4 | 122.1 | 10.5 | 35.5 | 99.9 | 2.8 | 105.3 | 1.9 | 37.4 | 99.9 |
| 4 | sig128N | 25 | 103.9 | 4.2 | 122.1 | 4.4 | 29.4 | 102.1 | 3.5 | 105.3 | 0.9 | 30.3 | 97.2 |
| 5 | sig128C | 25 | 90.8 | 3.6 | 122.1 | 8.6 | 33.6 | 103.3 | 3.1 | 105.3 | 0.7 | 34.3 | 98.3 |
| 6 | sig129N | 25 | 82.8 | 3.3 | 122.1 | 11.9 | 36.9 | 99 | 2.7 | 105.3 | 2.4 | 39.3 | 98.7 |
| 7 | sig129C | 25 | 101.3 | 4.1 | 122.1 | 5.1 | 30.1 | 98.5 | 3.3 | 105.3 | 2.1 | 32.2 | 102.1 |
| 8 | sig130N | 25 | 98.9 | 4 | 122.1 | 5.9 | 30.9 | 100.1 | 3.2 | 105.3 | 1.6 | 32.5 | 99.7 |
| 9 | sig130C | 25 | 86.3 | 3.5 | 122.1 | 10.4 | 35.4 | 96 | 2.7 | 105.3 | 3.4 | 38.8 | 99.3 |
| 10 | sig131N | 25 | 87 | 3.5 | 122.1 | 10.1 | 35.1 | 95.3 | 2.7 | 105.3 | 3.7 | 38.8 | 101.5 |
| 11 | sig131C | 25 | 119.1 | 4.8 | 122.1 | 0.6 | 25.6 | 100.9 | 3.9 | 105.3 | 1.1 | 26.7 | 100.2 |
| 12 | sig132N | 25 | 86 | 3.4 | 122.1 | 10.5 | 35.5 | 95.3 | 2.7 | 105.3 | 3.7 | 39.2 | 99.6 |
| 13 | sig132C | 25 | 119.1 | 4.8 | 122.1 | 0.6 | 25.6 | 101.2 | 3.9 | 105.3 | 1 | 26.7 | 100 |
| 14 | sig133N | 25 | 116.3 | 4.7 | 122.1 | 1.3 | 26.3 | 99.9 | 3.8 | 105.3 | 1.4 | 27.7 | 100.9 |
| 15 | sig133C | 25 | 122.1 | 4.9 | 122.1 | 0 | 25 | 101 | 4 | 105.3 | 1.1 | 26.1 | 101.9 |
| 16 | sig134N | 25 | 121.3 | 4.9 | 122.1 | 0.2 | 25.2 | 105.3 | 4.2 | 105.3 | 0 | 25.2 | 101.3 |
Tabella 1: Dati rappresentativi che mostrano il processo di raggruppamento dei campioni nella fase 3.3.
Discussione
Un protocollo ottimizzato per la profilazione profonda del proteoma basata su TMT16 è stato implementato con successo in precedenti pubblicazioni 12,13,41. Con questo protocollo attuale, più di 10.000 proteine uniche provenienti da un massimo di 16 campioni diversi possono essere quantificate di routine in un singolo esperimento con alta precisione.
Per ottenere risultati di alta qualità, è importante prestare attenzione ai passaggi critici durante il protocollo. Oltre a tutte le fasi del controllo qualità discusse nel nostro precedente articolo27, includiamo ulteriori fasi essenziali specifiche per il processo TMT16. Questi passaggi sono importanti per assicurare il successo dell'esperimento. Ad esempio, i derivati della reazione TMT (ad esempio, TMTpro-NHOH dalla reazione di quenching dell'idrossilammina e TMTpro-OH dall'idrossilazione del TMT) vengono rilevati come ioni caricati singolarmente prominenti prima della desalinizzazione mediante l'analisi LC-MS/MS. È fondamentale rimuoverli durante la fase di desalinizzazione. Abbiamo testato diverse condizioni di desalinizzazione e abbiamo scoperto che l'aggiunta del 5% di ACN nel normale tampone di lavaggio combinato con 10 volumi di letto × lavaggio per tre volte ha rimosso efficacemente i derivati41. Inoltre, TMT16 ha una massa maggiore rispetto a TMT11, quindi l'intero intervallo di scansione parte da un m/z più alto (450 invece di 410) per i campioni marcati con TMT16. Inoltre, poiché l'energia di collisione ottimale per un peptide dipende dalla massa da caricare e dallo stato di carica dello ione precursore21, i peptidi marcati con diversi tag di etichettatura chimica possono avere diverse energie di collisione ottimali. Per TMT16, l'energia di collisione del 30-32,5% è ottimale per TMT16, che è leggermente inferiore a TMT11.
La marcatura isobarica è una tecnica potente che fornisce un'elevata capacità di multiplexing. Sebbene altre tecniche come SILAC (marcatura di isotopi stabili da parte di amminoacidi in colture cellulari)47 e label-free forniscano strategie alternative per quantificare le proteine48, esse soffrono di una bassa produttività. In teoria, TMT16 è in grado di quantificare le proteine in 16 diversi campioni biologici. Tuttavia, è molto più comune utilizzare alcuni di questi canali come repliche biologiche, fornendo più potenza statistica e aiutando a generare dati affidabili. L'uso di repliche o addirittura triplicati è molto critico, specialmente nei sistemi in cui la variazione attesa nella concentrazione proteica è nominale. È importante comprendere la biologia del sistema prima di progettare l'esperimento per includere il numero appropriato di repliche. Alcuni sistemi biologici non sono ideali per alcune delle fasi di controllo della qualità di questo protocollo. Il test del rapporto di premiscelazione non viene utilizzato quando si utilizzano campioni di immunoprecipitazione per il protocollo a causa dell'elevata percentuale di proteine che si prevede cambi. In questi casi, i risultati verrebbero distorti con il test della premiscela. Questo è vero anche nei casi in cui si prevede che almeno 1 dei 10 campioni vari notevolmente nell'espressione proteica (vettore vuoto, inibizione del proteasoma, ecc.). Si suggerisce inoltre di utilizzare un canale TMT come "riferimento interno" che può quindi essere utilizzato per combinare più lotti di esperimenti TMT1649.
Questo protocollo può essere utilizzato per la profilazione del proteoma globale ad alto rendimento di campioni biologici complessi per studiare le proteine differenzialmente espresse e le vie di segnalazione cellulare e per comprendere la biologia delle malattie. Inoltre, con lievi modifiche al protocollo, può essere utilizzato per studiare le modifiche post-traduzionali come la fosforilazione, l'ubiquitinazione, la metilazione e l'acetilazione. L'adozione di un approccio integrato che combini un'analisi proteomica esaustiva su larga scala insieme ad altre pipeline -omiche come la genomica, la trascrittomica e la metabolomica può fornire informazioni per ampliare la comprensione di complessi sistemi biologici30,50.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato parzialmente sostenuto dal National Institutes of Health (R01GM114260, R01AG047928, R01AG053987, RF1AG064909 e U54NS110435) e dall'ALSAC (American Lebanese Syrian Associated Charities). L'analisi della SM è stata eseguita presso il Centro di Proteomica e Metabolomica del St. Jude Children's Research Hospital, che è parzialmente supportato dal NIH Cancer Center Support Grant (P30CA021765). Il contenuto è di esclusiva responsabilità degli autori e non rappresenta necessariamente le opinioni ufficiali del National Institutes of Health.
Materiali
| Name | Company | Catalog Number | Comments |
| 10% Criterion TGX Precast Midi Protein Gel | Biorad | 5671035 | |
| 10X TGS (Tris/Glycine/SDS) Buffer | BioRad | 161-0772 | |
| 4–20% Criterion TGX Precast Midi Protein Gel | Biorad | 5671095 | |
| 50% Hydroxylamine | Thermo Scientific | 90115 | |
| 6 X SDS Sample Loading Buffer | Boston Bioproducts Inc | BP-111R | |
| Ammonium Formate (NH4COOH) | Sigma | 70221-25G-F | |
| Ammonium Hydroxide, 28% | Sigma | 338818-100ml | |
| Bullet Blender | Next Advance | BB24-AU | |
| Butterfly Portfolio Heater | Phoenix S&T | PST-BPH-20 | |
| C18 Ziptips | Harvard Apparatus | 74-4607 | Used for desalting |
| Dithiothreitol (DTT) | Sigma | D5545 | |
| DMSO | Sigma | 41648 | |
| Formic Acid | Sigma | 94318 | |
| Fraction Collector | Gilson | FC203B | |
| Gel Code Blue Stain Reagent | Thermo | 24592 | |
| Glass Beads | Next Advance | GB05 | |
| HEPES | Sigma | H3375 | |
| HPLC Grade Acetonitrile | Burdick & Jackson | AH015-4 | |
| HPLC Grade Water | Burdick & Jackson | AH365-4 | |
| Iodoacetamide (IAA) | Sigma | I6125 | |
| Lys-C | Wako | 125-05061 | |
| Mass Spectrometer | Thermo Scientific | Q Exactive HF | |
| MassPrep BSA Digestion Standard | Waters | 186002329 | |
| Methanol | Burdick & Jackson | AH230-4 | |
| Nanoflow UPLC | Thermo Scientific | Ultimate 3000 | |
| Pierce BCA Protein Assay kit | Thermo Scientific | 23225 | |
| ReproSil-Pur C18 resin, 1.9um | Dr. Maisch GmbH | r119.aq.0003 | |
| Self-Pack Columns | New Objective | PF360-75-15-N-5 | |
| SepPak 1cc 50mg | Waters | WAT054960 | Used for desalting |
| Sodium Deoxycholate | Sigma | 30970 | |
| Speedvac | Thermo Scientific | SPD11V | |
| TMTpro 16plex Label Reagent Set | Thermo Scientific | A44520 | |
| Trifluoroacetic Acid (TFA) | Applied Biosystems | 400003 | |
| Trypsin | Promega | V511C | |
| Ultra-micro Spin Column,C18 | Harvard apparatus | 74-7206 | Used for desalting |
| Urea | Sigma | U5378 | |
| Xbridge Column C18 column | Waters | 186003943 | Used for basic pH LC |
Riferimenti
- Levy, M. J., Washburn, M. P., Florens, L. Probing the sensitivity of the orbitrap lumos mass spectrometer using a standard reference protein in a complex background. Journal of Proteome Research. 17 (10), 3586-3592 (2018).
- Bekker-Jensen, D. B., et al. An optimized shotgun strategy for the rapid generation of comprehensive human proteomes. Cell Systems. 4 (6), 587-599 (2017).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Frost, D. C., Greer, T., Li, L. High-Resolution Enabled 12-Plex DiLeu Isobaric Tags for Quantitative Proteomics. Analytical Chemistry. 87 (3), 1646-1654 (2015).
- Moulder, R., Bhosale, S. D., Goodlett, D. R., Lahesmaa, R. Analysis of the plasma proteome using iTRAQ and TMT-based Isobaric labeling. Mass Spectrometry Reviews. 37 (5), 583-606 (2018).
- Wang, H., et al. Deep multiomics profiling of brain tumors identifies signaling networks downstream of cancer driver genes. Nature Communications. 10 (1), 3718(2019).
- Rauniyar, N., Yates, J. R. Isobaric labeling-based relative quantification in shotgun proteomics. Journal of Proteome Research. 13 (12), 5293-5309 (2014).
- Hogrebe, A., et al. Benchmarking common quantification strategies for large-scale phosphoproteomics. Nature Communications. 9 (1), 1045(2018).
- Dayon, L., et al. Relative quantification of proteins in human cerebrospinal fluids by MS/MS using 6-plex isobaric tags. Analytical Chemistry. 80 (8), 2921-2931 (2008).
- Stepanova, E., Gygi, S. P., Paulo, J. A. Filter-based protein digestion (FPD): A detergent-free and scaffold-based strategy for TMT workflows. Journal of Proteome Research. 17 (3), 1227-1234 (2018).
- McAlister, G. C., et al. Increasing the multiplexing capacity of TMTs using reporter ion isotopologues with isobaric masses. Analytical Chemistry. 84 (17), 7469-7478 (2012).
- Thompson, A., et al. TMTpro: Design, synthesis, and initial evaluation of a proline-based isobaric 16-plex tandem mass tag reagent set. Analytical Chemistry. 91 (24), 15941-15950 (2019).
- Li, J., et al. TMTpro reagents: a set of isobaric labeling mass tags enables simultaneous proteome-wide measurements across 16 samples. Nature Methods. 17 (4), 399-404 (2020).
- Arul, A. B., Robinson, R. A. S. Sample Multiplexing Strategies in Quantitative Proteomics. Analytical Chemistry. 91 (1), 178-189 (2019).
- Labib, M., Kelley, S. O. Single-cell analysis targeting the proteome. Nature Reviews Chemistry. 4 (3), 143-158 (2020).
- Ren, R. J., Dammer, E. B., Wang, G., Seyfried, N. T., Levey, A. I. Proteomics of protein post-translational modifications implicated in neurodegeneration. Translational Neurodegeneration. 3 (1), 23(2014).
- Pagel, O., Loroch, S., Sickmann, A., Zahedi, R. P. Current strategies and findings in clinically relevant post-translational modification-specific proteomics. Expert Review of Proteomics. 12 (3), 235-253 (2015).
- Mertins, P., et al. Reproducible workflow for multiplexed deep-scale proteome and phosphoproteome analysis of tumor tissues by liquid chromatography-mass spectrometry. Nature Protocols. 13 (7), 1632-1661 (2018).
- Aebersold, R., Mann, M. Mass-spectrometric exploration of proteome structure and function. Nature. 537 (7620), 347-355 (2016).
- Bai, B., et al. Deep multilayer brain proteomics identifies molecular networks in Alzheimer's disease progression. Neuron. 105 (6), 975-991 (2020).
- Kelstrup, C. D., et al. Rapid and deep proteomes by faster sequencing on a benchtop quadrupole ultra-high-field orbitrap mass spectrometer. Journal of Proteome Research. 13 (12), 6187-6195 (2014).
- Meier, F., et al. Online parallel accumulation - serial fragmentation (PASEF) with a novel trapped ion mobility mass spectrometer. Molecular & Cellular Proteomics. 17 (12), (2018).
- Schweppe, D. K., et al. Full-featured, real-time database searching platform enables fast and accurate multiplexed quantitative proteomics. Journal of Proteome Research. 19 (5), 2026-2034 (2020).
- Wang, H., et al. Systematic optimization of long gradient chromatography mass spectrometry for deep analysis of brain proteome. Journal of Proteome Research. 14 (2), 829-838 (2015).
- Dey, K. K., et al. Deep undepleted human serum proteome profiling toward biomarker discovery for Alzheimer's disease. Clinical Proteomics. 16, 16(2019).
- Bai, B., et al. Deep profiling of proteome and phosphoproteome by isobaric labeling, extensive liquid chromatography, and mass spectrometry. Methods in Enzymology. 585, 377-395 (2017).
- High, A. A., et al. Deep proteome profiling by isobaric labeling, extensive liquid chromatography, mass spectrometry, and software-assisted quantification. Journal of Visualized Experiments. (129), e56474(2017).
- Chick, J. M., et al. Defining the consequences of genetic variation on a proteome-wide scale. Nature. 534 (7608), 500-505 (2016).
- Wang, Z., et al. Quantitative phosphoproteomic analysis of the molecular substrates of sleep need. Nature. 558 (7710), 435-439 (2018).
- Tan, H., et al. Integrative proteomics and phosphoproteomics profiling reveals dynamic signaling networks and bioenergetics pathways underlying T cell activation. Immunity. 46 (3), 488-503 (2017).
- Eden, E., et al. Proteome Half-Life Dynamics in Living Human Cells. Science. 331 (6018), 764(2011).
- Kleiman, L. B., Maiwald, T., Conzelmann, H., Lauffenburger, D. A., Sorger, P. K. Rapid phospho-turnover by receptor tyrosine kinases impacts downstream signaling and drug binding. Molecular Cell. 43 (5), 723-737 (2011).
- Mertins, P., et al. Ischemia in tumors induces early and sustained phosphorylation changes in stress kinase pathways but does not affect global protein levels. Molecular & Cellular Proteomics. 13 (7), 1690(2014).
- Xu, P., Duong, D. M., Peng, J. Systematical optimization of reverse-phase chromatography for shotgun proteomics. Journal of Proteome Research. 8 (8), 3944-3950 (2009).
- Wang, Y., et al. Reversed-phase chromatography with multiple fraction concatenation strategy for proteome profiling of human MCF10A cells. Proteomics. 11 (10), 2019-2026 (2011).
- Yang, F., Shen, Y., Camp, D. G., Smith, R. D. High-pH reversed-phase chromatography with fraction concatenation for 2D proteomic analysis. Expert Review of Proteomics. 9 (2), 129-134 (2012).
- Niu, M., et al. Extensive peptide fractionation and y(1) ion-based interference detection method for enabling accurate quantification by isobaric labeling and mass spectrometry. Analytical Chemistry. 89 (1), 2956-2963 (2017).
- Wang, X., et al. A tag-based database search tool for peptide identification with high sensitivity and accuracy. Molecular & Cellular Proteomics. 13 (12), 3663(2014).
- Li, Y., et al. JUMPg: An integrative proteogenomics pipeline identifying unannotated proteins in human brain and cancer cells. Journal of Proteome Research. 15 (7), 2309-2320 (2016).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nature Methods. 4 (3), 207-214 (2007).
- Wang, Z., et al. 27-plex tandem mass tag mass spectrometry for profiling brain proteome in Alzheimer's disease. Analytical Chemistry. 92 (10), 7162-7170 (2020).
- Ow, S. Y., et al. iTRAQ underestimation in simple and complex mixtures: "The good, the bad and the ugly". Journal of Proteome Research. 8 (11), 5347-5355 (2009).
- Karp, N. A., et al. Addressing accuracy and precision issues in iTRAQ quantitation. Molecular & Cellular Proteomics. 9 (9), 1885-1897 (2010).
- Ting, L., Rad, R., Gygi, S. P., Haas, W. MS3 eliminates ratio distortion in isobaric multiplexed quantitative proteomics. Nature Methods. 8 (11), 937-940 (2011).
- Gygi, J. P., et al. A triple knockout isobaric-labeling quality control platform with an integrated online database search. Journal of The American Society for Mass Spectrometry. 31 (7), 1344-1349 (2020).
- Savitski, M. M., et al. Measuring and managing ratio compression for accurate iTRAQ/TMT quantification. Journal of Proteome Research. 12 (8), 3586-3598 (2013).
- Ong, S. E., et al. Stable isotope labeling by amino acids in cell culture, SILAC, as a simple and accurate approach to expression proteomics. Molecular & Cellular Proteomics. 1 (5), 376(2002).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513(2014).
- Brenes, A., Hukelmann, J., Bensaddek, D., Lamond, A. I. Multibatch TMT reveals false positives, batch effects and missing values. Molecular & Cellular Proteomics. 18 (10), 1967-1980 (2019).
- Yu, J., Peng, J., Chi, H. Systems immunology: Integrating multi-omics data to infer regulatory networks and hidden drivers of immunity. Current Opinion in Systems Biology. 15, 19-29 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon