Method Article
Perfilado de proteoma profundo y de alto rendimiento mediante etiquetado de etiquetas de masas en tándem de 16 complejos junto con cromatografía bidimensional y espectrometría de masas
* Estos autores han contribuido por igual
En este artículo
Resumen
Aquí se presenta un protocolo optimizado de alto rendimiento desarrollado con reactivos de marcado masivo en tándem 16-plex, que permite la creación de perfiles cuantitativos de proteomas de muestras biológicas. El amplio fraccionamiento básico del pH y el LC-MS/MS de alta resolución mitigan la compresión de la relación y proporcionan una cobertura profunda del proteoma.
Resumen
El marcaje isobárico con etiquetas de masa en tándem (TMT) se usa ampliamente en proteómica debido a su alta capacidad de multiplexación y su profunda cobertura de proteoma. Recientemente, se ha introducido un método TMT ampliado de 16 plex, que aumenta aún más el rendimiento de los estudios proteómicos. En este manuscrito, presentamos un protocolo optimizado para el perfilado de proteoma profundo basado en TMT 16-plex, que incluye la preparación de muestras de proteínas, la digestión enzimática, la reacción de marcaje de TMT, el fraccionamiento de cromatografía líquida de fase inversa bidimensional (LC/LC), la espectrometría de masas en tándem (MS/MS) y el procesamiento de datos computacional. Se destacan los pasos cruciales del control de calidad y las mejoras en el proceso específico para el análisis TMT de 16 complejos. Este proceso multiplexado ofrece una poderosa herramienta para perfilar una variedad de muestras complejas, como células, tejidos y muestras clínicas. Más de 10.000 proteínas y modificaciones postraduccionales como la fosforilación, la metilación, la acetilación y la ubiquitinación en muestras biológicas altamente complejas de hasta 16 muestras diferentes se pueden cuantificar en un solo experimento, lo que proporciona una potente herramienta para la investigación básica y clínica.
Introducción
Los rápidos desarrollos en la tecnología de espectrometría de masas han permitido lograr una alta sensibilidad y una cobertura profunda del proteoma en aplicaciones de proteómica 1,2. A pesar de estos avances, la multiplexación de muestras sigue siendo el cuello de botella para los investigadores que manejan el análisis de una gran cohorte de muestras.
Las técnicas de marcaje isobárico multiplexado se utilizan ampliamente para la cuantificación relativa de todo el proteoma de grandes lotes de muestras 3,4,5,6. La cuantificación basada en etiquetas de masa en tándem (TMT) es una opción popular por su alta capacidad de multiplexación 7,8. Los reactivos TMT se lanzaron inicialmente como un kit de 6 complejos capaz de cuantificar hasta 6 muestras simultáneamente9. Esta tecnología se amplió aún más para cuantificar 10-11 muestras 10,11. Los reactivos TMTpro 16-plex (denominados TMT16 en adelante) desarrollados recientemente han aumentado aún más la capacidad de multiplexación a 16 muestras en un solo experimento12,13. Los reactivos TMT16 utilizan un grupo reportero a base de prolina, mientras que el TMT 11-plex aplica un grupo reportero derivado de la dimetilpiperidina. Tanto TMT11 como TMT16 utilizan el mismo grupo reactivo a aminas, pero el grupo de balance de masa de TMT16 es mayor que el de TMT11, lo que permite la combinación de 8 isótopos estables C13 y N15 en los iones reporteros para lograr 16 reporteros (Figura 1).
El aumento de la capacidad de multiplexación proporciona una plataforma para diseñar experimentos con suficientes réplicas para superar los desafíos estadísticos14. Además, los canales adicionales en el TMT de 16 complejos ayudan a reducir la cantidad total de material de partida por canal, lo que puede ayudar en el desarrollo de la proteómica unicelular emergente15. La alta capacidad de multiplexación también será valiosa en la cuantificación de modificaciones postraduccionales, que normalmente requieren grandes cantidades de material de partida 16,17.
Los flujos de trabajo proteómicos que emplean la tecnología TMTse han simplificado 18,19,20 y han evolucionado significativamente en la última década en términos de preparación de muestras, separación por cromatografía líquida, adquisición de datos de espectrometría de masas y análisis computacional 21,22,23,24,25,26. Nuestro artículo anterior proporciona una descripción detallada de la plataforma TMT 10-plex27. El protocolo descrito aquí presenta un método detallado y optimizado para TMT16, que incluye la extracción y digestión de proteínas, el etiquetado de TMT16, la agrupación y desalinización de muestras, el pH básico y la fase inversa del pH ácido (RP) LC, la MS de alta resolución y el procesamiento de datos (Figura 2). El protocolo también destaca los pasos clave de control de calidad que se han incorporado para completar con éxito un experimento de proteómica cuantitativa. Este protocolo puede ser utilizado de forma rutinaria para identificar y cuantificar más de 10.000 proteínas con alta reproducibilidad, para estudiar vías biológicas, procesos celulares y progresión de la enfermedad 20,28,29,30.
Protocolo
Los tejidos humanos para el estudio se obtuvieron con la aprobación del Programa de Donación de Cerebro y Cuerpo del Instituto de Investigación de Salud Banner Sun.
1. Extracción de proteínas de tejidos y control de calidad
NOTA: Para reducir el impacto de la recolección de muestras en el proteoma, es crucial recolectar muestras en un tiempo mínimo a baja temperatura si es posible31. Esto es especialmente importante cuando se analizan modificaciones postraduccionales, ya que suelen ser lábiles, por ejemplo, algunos eventos de fosforilación solo tienen unos pocos segundos de vida media32,33.
- Extirpar y pesar muestras de tejido
- Tara un tubo de microcentrífuga de 1,5 mL utilizando una balanza analítica y preenfría el tubo sobre hielo seco.
- Corte un tejido congelado (por ejemplo, tejido cerebral humano, ~ 10 mg) de una región definida en pedazos pequeños y transfiera los pedazos de tejido al tubo preenfriado inmediatamente.
NOTA: Para reducir la heterogeneidad de las muestras, es importante utilizar tamaños y regiones anatómicas homogéneos para las 16 muestras. La cantidad de proteína que se obtiene del tejido suele ser del 5-10% del peso del tejido. - Pese el tubo junto con el pañuelo y colóquelo inmediatamente sobre hielo seco. Procese las 15 muestras restantes utilizando el mismo procedimiento. Mantenga las muestras en hielo seco inmediatamente después de la disección y guárdelas a una temperatura de 80 °C.
- Lisar muestras de tejido
- Prepare un tampón de lisis fresco (50 mM HEPES pH 8,5, 8 M de urea y 0,5% de desoxicolato de sodio) el día del experimento. Los inhibidores de la fosfatasa deben añadirse al tampón de lisis para preservar el estado de fosforilación de las proteínas.
NOTA: Ponga el tampón de lisis a temperatura ambiente antes de usarlo, ya que la urea 8 M se precipitará en el hielo, lo que puede resultar en una desnaturalización incompleta de las proteínas durante la lisis de la muestra y reducir la eficiencia de la digestión de las proteínas. - Agregue el tampón de lisis (agregue 100 μL de tampón de lisis por 10 mg de tejido para lograr una concentración final de proteína de 5 a 10 μg/μL) y perlas de vidrio (~20% del volumen de lisado, 0,5 mm de diámetro) a cada muestra.
- Lisear el tejido en una licuadora a 4 °C con un ajuste de velocidad 8 durante 30 s, descansar durante 5 s, repetir hasta homogeneizar las muestras (~ 5 ciclos).
- Prepare un tampón de lisis fresco (50 mM HEPES pH 8,5, 8 M de urea y 0,5% de desoxicolato de sodio) el día del experimento. Los inhibidores de la fosfatasa deben añadirse al tampón de lisis para preservar el estado de fosforilación de las proteínas.
- Prepara las alícuotas de los lisados.
- Prepare al menos dos alícuotas para cada muestra. Se utiliza una pequeña alícuota (~10 μL) para el análisis de la concentración de proteínas y la evaluación de la calidad de las proteínas (por ejemplo, la validación de Western blot de proteínas de control positivo). Se utiliza una alícuota más grande (~50 μL) para el análisis del proteoma.
- Congele las alícuotas inmediatamente en hielo seco y almacene a -80 °C hasta su uso posterior.
- Medir la concentración de proteínas
NOTA: La concentración de proteínas se puede medir mediante el ensayo BCA o el método de tinción en gel SDS corto34 (Figura 3A). Debido a que los componentes no reductores de proteínas en el lisado tisular pueden afectar la medición en el ensayo BCA, los usuarios pueden validar la concentración de proteínas mediante el método de tinción en gel SDS corto. Aquí se presenta el método corto de tinción en gel SDS.- Diluya 16 muestras alícuotas por 10 veces y prepare el estándar BSA (por ejemplo, valoraciones BSA de 0,15, 0,5, 1,5 y 3 μg).
- Ejecute las muestras y el estándar BSA en un gel SDS-PAGE al 10% (26 pocillos) con un gel apilador hasta que todas las proteínas migren aproximadamente 3 mm en el gel.
- Tiñe el gel con azul Coomassie durante 1 h y quita las manchas del gel hasta que el fondo de la región en blanco quede claro.
- Escanee el gel para medir las intensidades de las bandas de proteínas teñidas con Coomassie por ImageJ y cree una curva estándar BSA de acuerdo con las mediciones.
- Calcule la concentración absoluta de proteínas mediante la curva estándar.
NOTA: La concentración final de proteína para cada muestra en este experimento fue de ~5-10 μg/μL. Para el análisis proteómico basado en TMT16, 50 μg de proteína por muestra (0,8 mg de proteína total para el análisis de proteoma completo) es suficiente.
- Control de calidad de la muestra
NOTA: Este paso de control de calidad es fundamental para identificar muestras de baja calidad antes de realizar el análisis TMT. En el caso de las muestras con cambios proteicos conocidos, se sugiere validar el cambio mediante Western blot. También se recomienda el análisis estándar SDS-PAGE para examinar los patrones de proteínas y excluir cualquier muestra con altos grados de degradación (Figura 3B).- Tome ~ 10 μg de cada muestra de la alícuota pequeña y ejecute las muestras en un gel SDS-PAGE degradado hasta que el tinte azul de bromofenol llegue al fondo del gel.
- Tiñe el gel con azul Coomassie y quita las manchas del gel. Inspeccione la calidad de la proteína para eliminar las muestras de proteína altamente degradadas.
NOTA: Las muestras degradadas pueden identificarse como aquellas que tienen muy pocas bandas de proteínas en la región de alto peso molecular y bandas intensificadas en la región de bajo peso molecular (Figura 3B).
2. Digestión de proteínas en solución, reducción y alquilación de péptidos, prueba de eficiencia de digestión y desalinización de péptidos
- Digestión de proteínas por Lys-C y tripsina
- Tome ~50 μg de proteína de la alícuota grande de cada muestra y agregue tampón de lisis a 50 μL.
- Agregue acetonitrilo al 100% (ACN) para alcanzar una concentración final del 10%.
- Realice la digestión de Lys-C añadiendo Lys-C en una proporción proteína:Lys-C de 100:1 (p/p) e incubando a temperatura ambiente durante 3 h.
- Las muestras se diluyen para contener una concentración final de 2 M de urea por 50 mM de HEPES (pH 8,5).
- Agregue tripsina en cada muestra en una proporción proteína:tripsina de 50:1 (p/p) y realice la digestión a temperatura ambiente durante 3 h o toda la noche.
- Reducción de péptidos y alquilación
- Agregue la solución de ditiotreitol (DTT 1 M) recién preparada a una concentración final de 1 mM e incube durante 1 h a temperatura ambiente para reducir los enlaces disulfuro.
- Agregue la solución de yodoacetamida (1 M IAA) recién preparada a una concentración final de 10 mM durante 30 min en la oscuridad para alalquilar los residuos de cisteína.
- Apague el IAA no reaccionado agregando 1M de DTT a una concentración final de 30 mM e incube a temperatura ambiente durante otros 30 minutos.
- Examinar la eficiencia de la digestión
- Tome ~1 μg de cada muestra y desalar con puntas de pipeta recubiertas de resina C18 de acuerdo con el protocolo del fabricante.
- Analice cada muestra mediante una ejecución de LC-MS/MS de gradiente corto (consulte más detalles en el paso 5).
- Realice una búsqueda en la base de datos de datos brutos de MS (consulte más detalles en el paso 6). Calcule el porcentaje de péptidos identificados con al menos un sitio de escisión de tripsina. El porcentaje suele estar por debajo del 15%.
- Si el porcentaje es superior al 15%, agregue tripsina adicional a las muestras para repetir la digestión.
- Después de la digestión, acidificar las muestras añadiendo TFA al 0,5% (v/v). Verifique el pH con una tira de pH para asegurarse de que el pH sea inferior a 3.
- Desalinización de péptidos
- Centrifugar los péptidos acidificados a 21.000 x g durante 10 min. Transfiera los sobrenadantes a un tubo nuevo.
- Lave las columnas de desalinización C18 (~25 μL de resina) dos veces con 250 μL de metanol al 100% centrifugando a 500 x g durante 30 s.
NOTA: Para reducir la pérdida de péptidos durante el proceso de desalinización, elija columnas de desalinización con una capacidad de unión que coincida con la cantidad de entrada. - Lavar las columnas dos veces con 250 μL de tampón de elución (60% ACN, 0,1% TFA) centrifugando a 500 x g durante 30 s.
- Equilibrar las columnas dos veces con 250 μL de tampón de equilibrio y lavado (0,1% TFA) centrifugando a 500 x g durante 30 s.
- Cargue las muestras en las columnas preequilibradas. Deje que las muestras se unan a las columnas girando a 100 x g durante 6 min. Asegúrese de que toda la solución haya pasado a través de la columna.
- Lavar las columnas tres veces con 250 μL de tampón de equilibrio y lavar centrifugando a 500 x g durante 30 s.
- Eluir los péptidos añadiendo 125 μL de tampón de elución a cada columna y girando a 100 x g durante 3 min. Compruebe que la columna no contiene ninguna solución sobrante.
- Seque los péptidos eluidos en un concentrador de vacío y almacene los péptidos a -80 °C para el futuro etiquetado de TMT.
3. Etiquetado de péptidos TMT16, prueba de eficiencia de etiquetado, agrupación de muestras y desalinización de péptidos marcados
- Marcaje de péptidos TMT16
- Vuelva a suspender cada muestra de péptido desalado en 50 μL de 50 mM HEPES (pH 8,5) mediante vórtice varias veces o disolución ultrasónica seguida de una tira de pH para verificar el pH.
NOTA: La muestra puede ser ácida si no se seca completamente antes del etiquetado, lo que influye negativamente en la eficiencia del etiquetado. Asegúrese de que el pH esté entre 7 y 8. - Tome ~ 1 μg de péptidos no marcados de cada muestra como controles negativos para la prueba de eficiencia de etiquetado TMT.
- Disuelva los reactivos de TMT16 en ACN anhidro. Realice la reacción de etiquetado añadiendo los reactivos en una proporción TMT:proteína de 1,5:1 (p/p) e incubando a temperatura ambiente durante 30 min.
NOTA: La proporción TMT:proteína utilizada para TMT16 es un 50% más alta que la proporción utilizada para TMT11. Esta pequeña discrepancia puede deberse a que la masa molecular de TMT16 es mayor (1,2 veces) que la de los reactivos de TMT11. La cantidad de proteína se estima a partir de las muestras sin tener en cuenta la pérdida durante la desalinización.
- Vuelva a suspender cada muestra de péptido desalado en 50 μL de 50 mM HEPES (pH 8,5) mediante vórtice varias veces o disolución ultrasónica seguida de una tira de pH para verificar el pH.
- Prueba de eficiencia de etiquetado
- Tome ~ 1 μg de péptidos marcados de cada muestra para la prueba de eficiencia de etiquetado. Coloque las muestras restantes a -80 °C sin apagar la reacción.
- Desala ~1 μg de cada una de las muestras marcadas y no marcadas con TMT16 con puntas de pipeta recubiertas de resina C18 de acuerdo con el protocolo del fabricante.
- Analice las muestras por LC-MS/MS (consulte la sección 5, con la excepción de que el gradiente es de 10 min).
- Estime la eficiencia del etiquetado mediante el análisis de la reducción de la intensidad de MS1 de los péptidos no marcados entre muestras no marcadas y marcadas. Seleccione de 6 a 10 péptidos diferentes para verificar la eficiencia del etiquetado y asegurarse de que todos los péptidos estén etiquetados. Para un etiquetado completo, no se observan péptidos no marcados.
NOTA: Es importante garantizar el etiquetado completo de todas las muestras para la identificación y cuantificación precisas de las proteínas posteriores. - Si el etiquetado no está completo, agregue reactivos TMT adicionales para etiquetar los péptidos restantes y verifique la eficiencia del etiquetado nuevamente antes de enfriar. Una vez que la muestra esté completamente etiquetada, apague la reacción a temperatura ambiente agregando hidroxilamina hasta una concentración final del 0,5% e incube a temperatura ambiente durante 15 minutos.
- Agrupación y desalinización de muestras
- Agrupe la mitad de cada muestra etiquetada con TMT para hacer una mezcla.
- Tome 1 μg de la mezcla y desale con puntas de pipeta recubiertas de resina C18, luego analice mediante LC-MS/MS usando un gradiente corto (~30 min).
- Calcule la concentración relativa utilizando la intensidad media de cada ion reportero TMT16 y comparando las discrepancias entre los 16 canales. Para lograr una mezcla uniforme de cada canal, agregue el resto de las muestras marcadas con TMT a la mezcla de acuerdo con la intensidad promedio calculada. Repita el ajuste hasta que todas las muestras se mezclen por igual. En la Tabla 1 se muestran datos representativos que muestran el proceso de agrupación de muestras.
NOTA: Debido a que los errores de pipeteo pueden afectar la precisión de las concentraciones y la cuantificación de proteínas, es importante asegurarse de que la cantidad de agrupación sea correcta. Las discrepancias en la intensidad entre 16 muestras deben ser inferiores al 5%.
- Desalinización de péptidos etiquetados
NOTA: Debido a que los derivados de fondo en la reacción de etiquetado de TMT16 (por ejemplo, TMTpro-NHOH de la reacción de enfriamiento de hidroxilamina y TMTpro-OH de la hidroxilación de TMT) son hidrofóbicos, se utiliza una condición de lavado extensa para las muestras marcadas con TMT16 para eliminar eficazmente los derivados. Se utiliza la adición de un 5% de ACN en el tampón de lavado normal (0,1% de TFA) y volúmenes de lecho 10x de tampón de lavado.- Acidificar la muestra agrupada añadiendo un 10% de TFA al pH < 3.
- Centrifugar la muestra agrupada a 21.000 x g durante 10 min y colocar el sobrenadante en un tubo nuevo.
- Seque la muestra con un concentrador de vacío para eliminar el ACN.
- Preacondicionar un cartucho de extracción en fase sólida que contenga una columna de sorbente de 50 mg lavando la columna con 2 mL de metanol al 100%, seguido de 2 mL de tampón de elución (60% ACN más 0,1% TFA) y, por último, 2 mL de tampón de lavado (0,1% TFA).
- Cargue la muestra en la columna. Ajuste el caudal a ~100 μL/min para garantizar la extensión total de la unión de péptidos. Guarde el flujo.
- Lave la columna tres veces con 1 ml de tampón de lavado.
- Péptidos eluidos con tampón de elución de 1 mL.
- Seque los péptidos eluidos en un concentrador de vacío y almacene los péptidos a -80 °C para su posterior fraccionamiento.
4. Prefraccionamiento básico de pH LC fuera de línea
- Preparación del sistema de fraccionamiento
- Prepare el tampón A (10 mM de formiato de amonio, pH 8,0) y el tampón B (10 mM de formiato de amonio, 90% ACN, pH 8,0) para un sistema LC de alto rendimiento y flujo de microlitros.
- Configure una columna de HPLC que contenga partículas híbridas de etileno puenteadas (tamaño de partícula de 3,5 μm, 4,6 mm × 25 cm) en un sistema LC de alto rendimiento de flujo de microlitros para el fraccionamiento.
- Instale un bucle de muestra de 100 μL y lave el bucle con 300 μL de metanol, agua y tampón A, secuencialmente.
- Utilice 100 μL de proporción 1:1:1:1 de isopropanol : metanol : acetonitrilo : agua para lavar la columna. A continuación, equilibre aún más la columna durante 0,5 h en el 95% del tampón A.
- Preparación de la muestra
- Disuelva la muestra de TMT16 agrupada y desalada en 70 μL de tampón A. Confirme que el pH de la muestra es ~ 8,0. Si todavía es ácido, ajuste el pH a 8.0 usando hidróxido de amonio al 28% (NH4OH).
NOTA: Para evitar la pérdida de muestra, el volumen de la muestra debe ser inferior al 70% del volumen del bucle. - Centrifugar la muestra a 21.000 x g durante 10 minutos para eliminar los precipitados.
- Disuelva la muestra de TMT16 agrupada y desalada en 70 μL de tampón A. Confirme que el pH de la muestra es ~ 8,0. Si todavía es ácido, ajuste el pH a 8.0 usando hidróxido de amonio al 28% (NH4OH).
- Fraccionamiento y concatenación
NOTA: Antes del fraccionamiento real de la muestra, se recomienda encarecidamente un experimento piloto para asegurarse de que el sistema LC esté en buenas condiciones. Esto se puede realizar con una pequeña cantidad de su muestra real (~5%) o con una mezcla de péptidos no marcada con TMT.- Inyecte la muestra y fraccione mediante el siguiente gradiente: 5% de tampón B durante 10 min, 5-15% tampón B durante 2 min, 15-45% tampón B durante 148 min y 45-95% tampón B durante 5 min. Utilice un caudal de 0,4 mL/min.
- Configure el colector de fracciones para recolectar fracciones cada 1 minuto y concatenar 160 fracciones a 40 fracciones en 4 ciclos.
NOTA: La concatenación se realiza combinando fracciones LC tempranas, medias y tardías eluidas de los mismos internos de tiempo en una fracción concatenada. Las fracciones concatenadas tienen poca superposición en la primera dimensión de LC, por lo que aumentan el uso eficiente de la ventana de elución en la LC de segunda dimensión. Además, a través de varias rondas de concatenación, los péptidos se pueden distribuir uniformemente en todas las fracciones concatenadas. Se ha demostrado que este enfoque aumenta la cobertura del proteoma en comparación con el análisis de fracciones individuales35,36. - Seque todas las fracciones concatenadas en un concentrador de vacío y almacene las muestras secas a -80 °C para su posterior análisis LC-MS/MS.
5. Análisis de pH RPLC-MS/MS ácido
- Preparación del sistema RPLC-MS/MS de pH ácido
- Empaque una columna vacía (75 μm de diámetro interior con un orificio de punta de 15 μm) con resina C18 de 1,9 μm de 10-15 cm de longitud.
- Caliente la columna a 65 °C con un calentador de mariposa para reducir la contrapresión.
- Lave la columna a fondo con un 95% de tampón B (3% de dimetilsulfóxido, 0,2% de ácido fórmico y 67% de ACN). A continuación, equilibre completamente la columna en un 95% de tampón A (3% de dimetilsulfóxido y 0,2% de ácido fórmico).
- Compruebe el rendimiento del sistema LC-MS/MS ejecutando 100 ng de péptidos cerebrales de rata o péptidos BSA antes de analizar las muestras experimentales.
- Análisis LC-MS/MS de fracciones concatenadas
- Reconstituya los péptidos secos a partir de las fracciones básicas de pH en AG al 5% y centrifugue a 21.000 × g durante 5 min. Transfiera el sobrenadante de cada muestra a un inserto de vial de HPLC.
- Cargue ~1 μg de péptidos de cada fracción en la columna. Los péptidos se eluyen a un caudal de 0,25 μL/min con un gradiente de 60 min de 18-45% de tampón B.
NOTA: Para obtener números de identificación altos, ejecute una fracción y ajuste el gradiente de las fracciones restantes en función de la primera ejecución. El mejor gradiente debe tener péptidos distribuidos uniformemente a lo largo de todo el gradiente (Figura 4A). - Opere el espectrómetro de masas con los siguientes parámetros para el análisis de muestras marcadas con TMT16: Escaneos MS1 (rango de escaneo MS completo: 450-1600 m/z; Resolución Orbitrap: 60.000; objetivo de control automático de ganancia: 1 x 106; tiempo máximo de iones: 50 ms) y 20 escaneos MS2 dependientes de los datos (resolución Orbitrap: 60.000; Objetivo AGC: 1 x 105; Tiempo máximo de iones: 110 ms; Energía de colisión normalizada HCD: 32%; ventana de aislamiento: 1,0 m/z; Desplazamiento de aislamiento: 0,2 m/z; Exclusión dinámica: 10 s).
NOTA: Los parámetros utilizados aquí están optimizados en un tipo de espectrómetro de masas (consulte la Tabla de materiales). Para diferentes instrumentos MS, los usuarios deben ajustar los parámetros del instrumento para lograr resultados de alta calidad. Una configuración es monitorear la energía de colisión HCD normalizada, ya que la energía óptima puede variar entre instrumentos, así como entre TMT11 y TMT16.
6. Tratamiento de datos
NOTA: El análisis de datos se realizó utilizando un paquete de software JUMP 37,38,39 que incluye un motor de búsqueda de base de datos híbrido (basado en patrones y etiquetas), software de filtrado que controla la tasa de falsos descubrimientos (FDR) de péptidos/proteínas identificados y software de cuantificación para conjuntos de datos TMT. Dependiendo de la situación del usuario, el análisis de datos se puede realizar utilizando otros programas comerciales o de libre acceso.
- Búsqueda en bases de datos
- Convierta los archivos .raw del instrumento MS a archivos .mzXML y busque espectros MS2 en una base de datos de señuelo de objetivos no redundante40 generada a partir de secuencias de proteínas humanas UniProt (u otra base de datos específica de la especie apropiada) para calcular la FDR de las proteínas identificadas.
NOTA: Genere la base de datos no redundante combinando secuencias de proteínas de las bases de datos Swiss-Prot y TrEMBL. También se pueden agregar secuencias de proteínas personalizadas que no están contenidas en esas bases de datos de referencia, incluidas las proteínas escindidas con proteasa, las proteínas con polimorfismos de un solo nucleótido y los contaminantes comunes. - Realice búsquedas utilizando los siguientes parámetros. Tolerancia de masa para precursor: 10 ppm; tolerancia de masa para iones de producto: 15 ppm; Escotes máximos perdidos: 2; Sitios de modificación máximos: 3; modificaciones estáticas: 304.20715 Da para las etiquetas TMT16 en residuos de Lys y N termini, 57.02146 Da para la carbamidometilación en residuos de Cys; modificación dinámica: 15.99492 Da para oxidación en Met.
- Convierta los archivos .raw del instrumento MS a archivos .mzXML y busque espectros MS2 en una base de datos de señuelo de objetivos no redundante40 generada a partir de secuencias de proteínas humanas UniProt (u otra base de datos específica de la especie apropiada) para calcular la FDR de las proteínas identificadas.
- Filtrar los resultados de la búsqueda
- Filtre las coincidencias de espectro peptídico (PSM) resultantes por longitud del péptido (>6 aminoácidos), precisión de masa del ion precursor y puntuaciones de coincidencia basadas en JUMP (Jscore y ΔJn). A continuación, los péptidos se agrupan por longitud peptídica, extremos trípticos, modificaciones, sitios de escisión perdidos y estado de carga.
- Filtre aún más los datos con las puntuaciones de coincidencia para lograr una FDR inferior al 1 % a nivel de proteína (análisis de proteoma completo) o de péptido (análisis de fosfoproteoma).
NOTA: Si faltan péptidos/proteínas de control positivo en las etapas de filtrado, entonces FDR puede aumentarse a un nivel razonable para que esos péptidos/proteínas puedan ser rescatados. - En el caso de los péptidos compartidos por más de un miembro de una familia de proteínas, agrupe los miembros emparejados en un grupo.
NOTA: Con la regla de la parsimonia, el grupo está representado por la proteína homóloga con el mayor número de péptidos compartidos y otras proteínas emparejadas por péptidos únicos.
- Cuantificación de proteínas
- Cuantifique las proteínas utilizando un programa incorporado de un paquete de software estadístico para resumir las intensidades de los iones reporteros de TMT en todos los PSM coincidentes.
- Extraiga las intensidades de los iones indicadores de TMT de cada PSM aceptado y corrija las intensidades brutas de acuerdo con la distribución isotópica de cada reactivo de etiquetado (por ejemplo, TMT16-126 genera el 92,6 %, el 7,2 % y el 0,2 % de los iones m/z de 126, 127C y 128C, respectivamente) y filtre los PSM de baja intensidad y/o muy ruidosos sobre la base de umbrales definidos por el usuario. Normalice los datos de cuantificación utilizando las intensidades medias recortadas (o medianas) de las muestras para corregir el sesgo de carga.
- Para cada proteína identificada, calcule las intensidades centradas en la media en las muestras (es decir, las intensidades relativas) de los PSM emparejados y resuma las intensidades relativas de los PSM tomando el promedio de la muestra. Convierta las señales relativas en absolutas multiplicando la intensidad media general de los tres PSM coincidentes más abundantes.
- Corrección de la interferencia de cuantificación utilizando un enfoque de corrección de iones y137 previamente informado, que supone que la intensidad del ion y1 está correlacionada con la intensidad del ion reportero. Al estimar la relación lineal entre las intensidades de iones y1 y reporteros de escaneos limpios, se deriva y corrige el nivel de interferencia de la intensidad de iones y1 contaminados en escaneos ruidosos.
NOTA: Para los péptidos trípticos marcados con TMT, los residuos de K-TMT y R son dos iones y1 representativos (376,27574 Da y 175,11895 Da, respectivamente) en un espectro MS2. Si solo se detecta un ion y1 y es consistente con el péptido identificado, entonces el MS2 se considera un escaneo limpio. Si se detectan ambos iones y1, entonces se considera que el MS2 es un escaneo ruidoso. - Transfiera los valores de cuantificación de proteínas a una hoja de cálculo para su posterior análisis. Utilice métodos de análisis de datos no supervisados, como PCA o análisis de agrupamiento, para explorar la distribución de las muestras. Para identificar las proteínas expresadas diferencialmente, utilice métodos estadísticos como la prueba t y el análisis de varianza (ANOVA).
7. Validación de datos de los Estados miembros
NOTA: Antes de realizar experimentos biológicos que requieren mucho tiempo, utilice al menos un método de validación para evaluar la calidad de los datos de EM.
- Inspeccione manualmente los espectros MS/MS de las proteínas de interés para validar la secuencia peptídica y las intensidades de los iones reporteros TMT.
- Usar enfoques basados en anticuerpos (por ejemplo, Western blot o análisis de inmunohistoquímica) para verificar los cambios en los niveles de proteínas. Para confirmar la presencia de péptidos nativos, utilice péptidos sintéticos como patrones internos. Los espectros MS/MS y el tiempo de retención de los péptidos durante la LC-MS/MS deben ser idénticos en las mismas condiciones.
- Utilice un enfoque de EM dirigido para verificar los cambios en las proteínas.
Resultados
El protocolo para el TMT16 recientemente desarrollado, que incluye la reacción de etiquetado, la desalinización y las condiciones de LC-MS, se ha optimizado sistemáticamente41. Además, comparamos directamente los métodos 11-plex y 16-plex utilizándolos para analizar las mismas muestras humanas de EA41. Después de la optimización de los parámetros clave para TMT16, los métodos TMT11 y TMT16 producen una cobertura, identificación y cuantificación de proteomas similares > 100.000 péptidos en > 10.000 proteínas humanas.
Debido a que los reactivos TMT16 son más hidrofóbicos que los reactivos TMT11, es probable que los péptidos marcados con TMT16 sean más hidrofóbicos que los péptidos marcados con TMT11, lo que puede explicar diferentes tiempos de retención (RT) en RPLC. Por lo tanto, evaluamos el impacto de TMT16 en el péptido RT en comparación con TMT11 mediante el análisis de la mezcla de péptidos marcados con TMT11 y TMT16 utilizando LC-MS/MS. Encontramos que TMT16 tiene una influencia significativa en RT a los péptidos con hidrofobicidad media, pero tiene poco efecto sobre los péptidos de hidrofobicidad extremadamente alta o baja. Por lo tanto, las concentraciones similares de tampón B inicial y final en el gradiente LC se pueden utilizar para diferentes péptidos marcados con TMT.
A continuación, optimizamos el gradiente RPLC en línea para la muestra marcada con TMT16. El gradiente de TMT16 es muy similar al de TMT11. El porcentaje de búfer inicial y final B es el mismo (por ejemplo, del 18 % al 45 %). Pero notamos que el número de péptidos identificados en TMT16 disminuyó rápidamente a alrededor del 40% del tampón B cuando se usó el mismo gradiente que se usa para TMT11. Así, redujimos ligeramente el tiempo de la pendiente entre un 40% y un 45%. También hicimos pequeños ajustes a este gradiente para diferentes fracciones y diferentes muestras. Después de la optimización del gradiente, los péptidos identificados se distribuyeron uniformemente a lo largo del gradiente (Figura 4A).
Para maximizar el número de proteínas identificadas y cuantificadas con precisión utilizando el método TMT16, optimizamos la energía de colisión normalizada (NCE) para las muestras marcadas con TMT16 en nuestro informe anterior41. Se probaron diferentes NCE (del 20% al 40%) en el espectrómetro de masas durante las ejecuciones de LC-MS/MS. Equilibrando el número de identificaciones de proteínas y la intensidad de los iones reporteros, se eligió un NCE de 30-32,5% como la energía óptima de colisión de HCD que se utilizará para las muestras marcadas con TMT16.
La compresión de la relación causada por iones interferentes coeluidos ha sido una limitación de las técnicas de marcaje isobárico para la cuantificación de proteínas. Un estudio publicado anteriormente utilizando el método TMT11 muestra que la compresión de la relación puede eliminarse casi por completo mediante un prefraccionamiento extensivo de LC, configuraciones optimizadas de MS y estrategias de corrección de datos posteriores a MS37. Utilizamos estas estrategias, incluyendo el fraccionamiento extensivo previo a la MS (40 fracciones básicas de pH LC), la aplicación de una ventana de aislamiento estrecha (1 m/z) en el entorno de MS y la corrección de iones y1 en los análisis del proteoma TMT11 y TMT16 de las mismas muestras. Después de examinar la curva de correlación del cambio en el pliegue de la proteína entre los conjuntos de datos TMT11 y TMT16, encontramos que la pendiente estaba muy cerca de 1, lo que indica que la compresión de la relación en TMT16 no fue visiblemente mayor que la de TMT11 en nuestra condición experimental41. Los resultados consistentes fueron reportados que la relación de compresión no tiene diferencia cuando el nivel de multiplexación se incrementó de 11 a 1613,45. Por lo tanto, las estrategias publicadas anteriormente pueden utilizarse para aliviar la compresión de la relación, mejorando significativamente la precisión de la cuantificación 27,37,44,46.
Por último, comparamos el número de PSMs, péptidos únicos y proteínas únicas cuantificados en muestras marcadas con TMT11 frente a TMT16 (Figura 4B). Los resultados muestran que los PSM de ambos métodos son comparables; sin embargo, las proteínas y péptidos cuantificados son ligeramente inferiores en el método TMT16, lo que es consistente con otros relatos12,13. Nuestros resultados indican que las mejoras en el proceso TMT16, junto con el uso de parámetros LC-MS optimizados, proporcionan un perfil de proteoma profundo y de alto rendimiento de muestras biológicas.
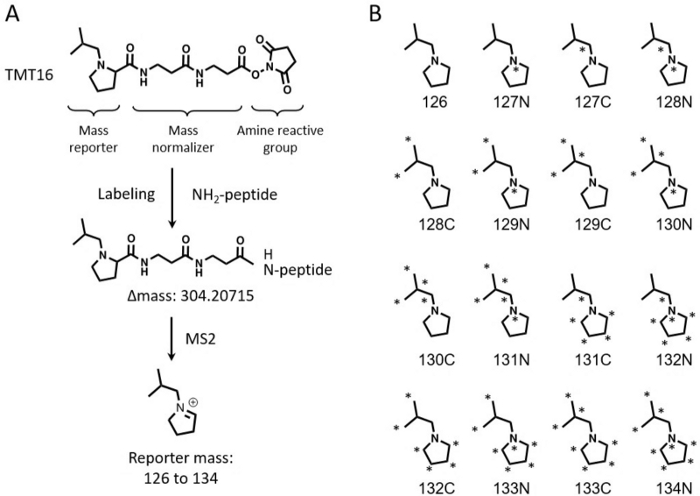
Figura 1: Estructura del reactivo TMT 16-plex. (A) Se muestra la estructura del reactivo TMT de 16 complejos, el proceso de etiquetado, el cambio de masa después del etiquetado y la masa del ion reportero. (B) Estructuras marcadas con isótopos pesados de los iones reporteros de los reactivos TMT16. Haga clic aquí para ver una versión más grande de esta figura.
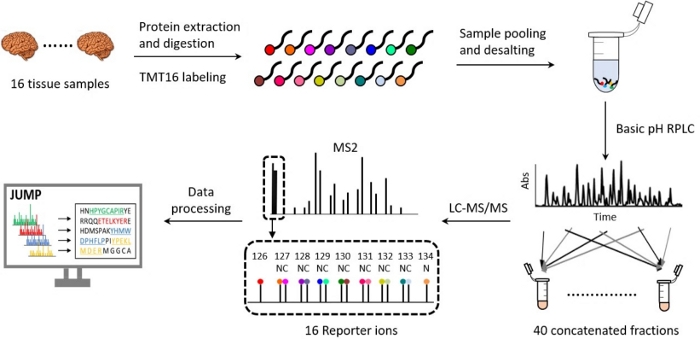
Figura 2: Flujo de trabajo de la creación de perfiles de proteoma mediante TMT-LC/LC-MS/MS de 16 complejos. La proteína extraída de 16 muestras de tejido biológico se digierió y se etiquetó con 16 etiquetas TMT diferentes. Las muestras de 16 canales se agrupan por igual, y la mezcla se fracciona y concatena en 40 fracciones mediante cromatografía líquida de fase inversa de pH básico (RPLC) fuera de línea. Cada fracción se analiza posteriormente mediante RPLC ácido junto con espectrometría de masas de alta resolución. Se procesaron los archivos RAW de MS/MS. La imagen del tejido cerebral se cita de Medium.com con algunas modificaciones. Haga clic aquí para ver una versión más grande de esta figura.
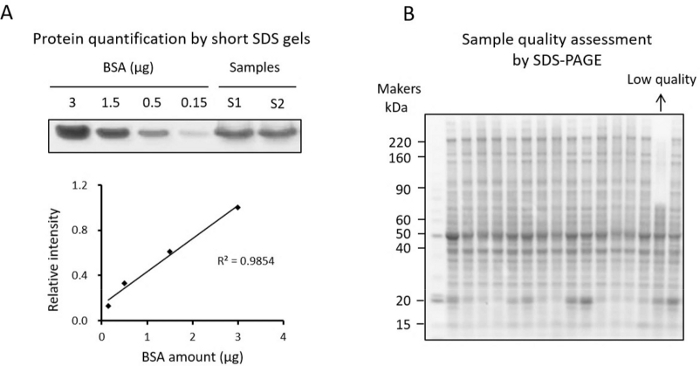
Figura 3: Control de calidad de proteínas. (A) Cuantificación de la proteína extraída del tejido en un gel corto de SDS con BSA como estándar. La curva estándar representa la concentración de BSA y la intensidad de la banda de proteínas teñida con Coomassie utilizada para la cuantificación. (B) Gel SDS utilizado para el ensayo de calidad de proteínas. Haga clic aquí para ver una versión más grande de esta figura.
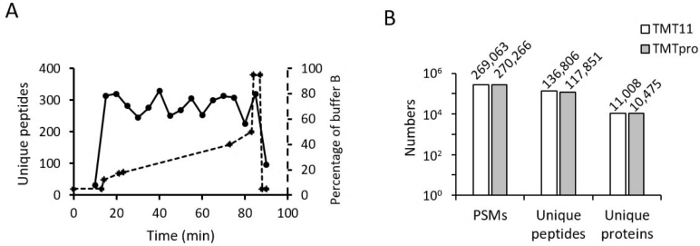
Figura 4: Resultados representativos. (A) Distribución de péptidos en LC ácidos. El gradiente optimizado del búfer B después de la corrección del volumen muerto se alinea en el mismo gráfico. (B) El histograma muestra el número de PSM cuantificados, péptido único y proteína única en los métodos TMT11 y TMT16. Haga clic aquí para ver una versión más grande de esta figura.
| 1º (50 μl, usar el 50% en la primera mezcla) | 2º (ajustar la mezcla y ahorrar un 10%) | 3º (ajuste final) | |||||||||||
| Canales | Reporteros | Volumen de mezcla (μL) | Intensidad (unidades) | Conc. (unidad/μL) | Intensidad esperada (unidades) | Volumen añadido (μL) | Volumen total (μL) | Intensidad (unidades) | Conc. (unidad/μL) | Intensidad esperada (unidades) | Volumen añadido (μL) | Volumen total (μL) | Intensidad (unidades) |
| 1 | SIG126 | 25 | 94.7 | 3.8 | 122.1 | 7.2 | 32.2 | 99.6 | 3.1 | 105.3 | 1.8 | 34.1 | 100 |
| 2 | sig127N | 25 | 83 | 3.3 | 122.1 | 11.8 | 36.8 | 101.1 | 2.7 | 105.3 | 1.5 | 38.3 | 98 |
| 3 | SIG127C | 25 | 86 | 3.4 | 122.1 | 10.5 | 35.5 | 99.9 | 2.8 | 105.3 | 1.9 | 37.4 | 99.9 |
| 4 | sig128N | 25 | 103.9 | 4.2 | 122.1 | 4.4 | 29.4 | 102.1 | 3.5 | 105.3 | 0.9 | 30.3 | 97.2 |
| 5 | SIG128C | 25 | 90.8 | 3.6 | 122.1 | 8.6 | 33.6 | 103.3 | 3.1 | 105.3 | 0.7 | 34.3 | 98.3 |
| 6 | sig129N | 25 | 82.8 | 3.3 | 122.1 | 11.9 | 36.9 | 99 | 2.7 | 105.3 | 2.4 | 39.3 | 98.7 |
| 7 | sig129C | 25 | 101.3 | 4.1 | 122.1 | 5.1 | 30.1 | 98.5 | 3.3 | 105.3 | 2.1 | 32.2 | 102.1 |
| 8 | sig130N | 25 | 98.9 | 4 | 122.1 | 5.9 | 30.9 | 100.1 | 3.2 | 105.3 | 1.6 | 32.5 | 99.7 |
| 9 | sig130C | 25 | 86.3 | 3.5 | 122.1 | 10.4 | 35.4 | 96 | 2.7 | 105.3 | 3.4 | 38.8 | 99.3 |
| 10 | sig131N | 25 | 87 | 3.5 | 122.1 | 10.1 | 35.1 | 95.3 | 2.7 | 105.3 | 3.7 | 38.8 | 101.5 |
| 11 | sig131C | 25 | 119.1 | 4.8 | 122.1 | 0.6 | 25.6 | 100.9 | 3.9 | 105.3 | 1.1 | 26.7 | 100.2 |
| 12 | sig132N | 25 | 86 | 3.4 | 122.1 | 10.5 | 35.5 | 95.3 | 2.7 | 105.3 | 3.7 | 39.2 | 99.6 |
| 13 | sig132C | 25 | 119.1 | 4.8 | 122.1 | 0.6 | 25.6 | 101.2 | 3.9 | 105.3 | 1 | 26.7 | 100 |
| 14 | sig133N | 25 | 116.3 | 4.7 | 122.1 | 1.3 | 26.3 | 99.9 | 3.8 | 105.3 | 1.4 | 27.7 | 100.9 |
| 15 | SIG133C | 25 | 122.1 | 4.9 | 122.1 | 0 | 25 | 101 | 4 | 105.3 | 1.1 | 26.1 | 101.9 |
| 16 | sig134N | 25 | 121.3 | 4.9 | 122.1 | 0.2 | 25.2 | 105.3 | 4.2 | 105.3 | 0 | 25.2 | 101.3 |
Tabla 1: Datos representativos que muestran el proceso de agrupación de muestras en el paso 3.3.
Discusión
Un protocolo optimizado para el perfil de proteoma profundo basado en TMT16 se ha implementado con éxito en publicaciones anteriores 12,13,41. Con este protocolo actual, más de 10.000 proteínas únicas de hasta 16 muestras diferentes se pueden cuantificar de forma rutinaria en un solo experimento con alta precisión.
Para obtener resultados de alta calidad, es importante prestar atención a los pasos críticos a lo largo del protocolo. Además de todos los pasos de control de calidad discutidos en nuestro artículo27 anterior, incluimos pasos esenciales adicionales específicos para el proceso TMT16. Estos pasos son importantes para asegurar el éxito de un experimento. Por ejemplo, los derivados de la reacción TMT (por ejemplo, TMTpro-NHOH de la reacción de enfriamiento de hidroxilamina y TMTpro-OH de la hidroxilación de TMT) se detectan como iones prominentes cargados individualmente antes de la desalinización mediante el análisis LC-MS/MS. Es fundamental eliminarlos durante la etapa de desalinización. Hemos probado diferentes condiciones de desalinización y hemos comprobado que la adición de un 5% de ACN en el tampón de lavado regular combinado con volúmenes de lecho de 10 × lavado tres veces eliminó eficazmente los derivados41. Además, TMT16 tiene una masa mayor en comparación con TMT11, por lo tanto, el rango de escaneo completo comienza desde un m/z más alto (450 en lugar de 410) para muestras marcadas con TMT16. Además, como la energía de colisión óptima para un péptido depende de la masa a la carga y del estado de carga del ionprecursor 21, los péptidos marcados con diferentes etiquetas de etiquetado químico pueden tener diferentes energías de colisión óptimas. Para TMT16, la energía de colisión 30-32.5% es óptima para TMT16, que es ligeramente más baja que TMT11.
El etiquetado isobárico es una técnica poderosa que proporciona una alta capacidad de multiplexación. Aunque otras técnicas como SILAC (marcaje de isótopos estables por aminoácidos en cultivo celular)47 y sin marcaje proporcionan estrategias alternativas para cuantificar proteínas48, adolecen de un bajo rendimiento. En teoría, TMT16 puede cuantificar proteínas en 16 muestras biológicas diferentes. Sin embargo, es mucho más común utilizar algunos de estos canales como réplicas biológicas, proporcionando más potencia estadística y ayudando a generar datos fiables. El uso de réplicas o incluso triplicados es muy crítico, especialmente en sistemas donde el cambio esperado en la concentración de proteínas es nominal. Es importante comprender la biología del sistema antes de diseñar el experimento para incluir el número adecuado de réplicas. Ciertos sistemas biológicos no son ideales para algunos de los pasos de control de calidad de este protocolo. La prueba de proporción de premezcla no se utiliza cuando se utilizan muestras de inmunoprecipitación para el protocolo debido al gran porcentaje de proteínas que se espera que cambien. En estos casos, los resultados se sesgarían con la prueba de premezcla. Esto también es cierto en los casos en los que se espera que al menos 1 de las 10 muestras varíe mucho en la expresión de proteínas (vector vacío, inhibición del proteasoma, etc.). También se sugiere utilizar un canal TMT como "referencia interna" que luego se puede utilizar para combinar múltiples lotes de experimentos TMT1649.
Este protocolo se puede utilizar para el perfil de proteoma global de alto rendimiento de muestras biológicas complejas para estudiar proteínas expresadas diferencialmente y vías de señalización celular y para comprender la biología de la enfermedad. Además, con ligeras modificaciones en el protocolo, se puede utilizar para estudiar modificaciones postraduccionales como la fosforilación, la ubiquitinación, la metilación y la acetilación. Adoptar un enfoque integrado que combine el análisis proteómico exhaustivo a gran escala junto con otras vías ómicas como la genómica, la transcriptómica y la metabolómica puede proporcionar información para ampliar la comprensión de los sistemas biológicos intrincados 30,50.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Este trabajo fue parcialmente apoyado por los Institutos Nacionales de Salud (R01GM114260, R01AG047928, R01AG053987, RF1AG064909 y U54NS110435) y ALSAC (American Lebanese Syrian Associated Charities). El análisis de la EM se realizó en el Centro de Proteómica y Metabolómica del St. Jude Children's Research Hospital, que cuenta con el apoyo parcial de la Subvención (P30CA021765) del Centro de Cáncer de los NIH. El contenido es responsabilidad exclusiva de los autores y no representa necesariamente los puntos de vista oficiales de los Institutos Nacionales de Salud.
Materiales
| Name | Company | Catalog Number | Comments |
| 10% Criterion TGX Precast Midi Protein Gel | Biorad | 5671035 | |
| 10X TGS (Tris/Glycine/SDS) Buffer | BioRad | 161-0772 | |
| 4–20% Criterion TGX Precast Midi Protein Gel | Biorad | 5671095 | |
| 50% Hydroxylamine | Thermo Scientific | 90115 | |
| 6 X SDS Sample Loading Buffer | Boston Bioproducts Inc | BP-111R | |
| Ammonium Formate (NH4COOH) | Sigma | 70221-25G-F | |
| Ammonium Hydroxide, 28% | Sigma | 338818-100ml | |
| Bullet Blender | Next Advance | BB24-AU | |
| Butterfly Portfolio Heater | Phoenix S&T | PST-BPH-20 | |
| C18 Ziptips | Harvard Apparatus | 74-4607 | Used for desalting |
| Dithiothreitol (DTT) | Sigma | D5545 | |
| DMSO | Sigma | 41648 | |
| Formic Acid | Sigma | 94318 | |
| Fraction Collector | Gilson | FC203B | |
| Gel Code Blue Stain Reagent | Thermo | 24592 | |
| Glass Beads | Next Advance | GB05 | |
| HEPES | Sigma | H3375 | |
| HPLC Grade Acetonitrile | Burdick & Jackson | AH015-4 | |
| HPLC Grade Water | Burdick & Jackson | AH365-4 | |
| Iodoacetamide (IAA) | Sigma | I6125 | |
| Lys-C | Wako | 125-05061 | |
| Mass Spectrometer | Thermo Scientific | Q Exactive HF | |
| MassPrep BSA Digestion Standard | Waters | 186002329 | |
| Methanol | Burdick & Jackson | AH230-4 | |
| Nanoflow UPLC | Thermo Scientific | Ultimate 3000 | |
| Pierce BCA Protein Assay kit | Thermo Scientific | 23225 | |
| ReproSil-Pur C18 resin, 1.9um | Dr. Maisch GmbH | r119.aq.0003 | |
| Self-Pack Columns | New Objective | PF360-75-15-N-5 | |
| SepPak 1cc 50mg | Waters | WAT054960 | Used for desalting |
| Sodium Deoxycholate | Sigma | 30970 | |
| Speedvac | Thermo Scientific | SPD11V | |
| TMTpro 16plex Label Reagent Set | Thermo Scientific | A44520 | |
| Trifluoroacetic Acid (TFA) | Applied Biosystems | 400003 | |
| Trypsin | Promega | V511C | |
| Ultra-micro Spin Column,C18 | Harvard apparatus | 74-7206 | Used for desalting |
| Urea | Sigma | U5378 | |
| Xbridge Column C18 column | Waters | 186003943 | Used for basic pH LC |
Referencias
- Levy, M. J., Washburn, M. P., Florens, L. Probing the sensitivity of the orbitrap lumos mass spectrometer using a standard reference protein in a complex background. Journal of Proteome Research. 17 (10), 3586-3592 (2018).
- Bekker-Jensen, D. B., et al. An optimized shotgun strategy for the rapid generation of comprehensive human proteomes. Cell Systems. 4 (6), 587-599 (2017).
- Mertins, P., et al. Proteogenomics connects somatic mutations to signalling in breast cancer. Nature. 534 (7605), 55-62 (2016).
- Frost, D. C., Greer, T., Li, L. High-Resolution Enabled 12-Plex DiLeu Isobaric Tags for Quantitative Proteomics. Analytical Chemistry. 87 (3), 1646-1654 (2015).
- Moulder, R., Bhosale, S. D., Goodlett, D. R., Lahesmaa, R. Analysis of the plasma proteome using iTRAQ and TMT-based Isobaric labeling. Mass Spectrometry Reviews. 37 (5), 583-606 (2018).
- Wang, H., et al. Deep multiomics profiling of brain tumors identifies signaling networks downstream of cancer driver genes. Nature Communications. 10 (1), 3718(2019).
- Rauniyar, N., Yates, J. R. Isobaric labeling-based relative quantification in shotgun proteomics. Journal of Proteome Research. 13 (12), 5293-5309 (2014).
- Hogrebe, A., et al. Benchmarking common quantification strategies for large-scale phosphoproteomics. Nature Communications. 9 (1), 1045(2018).
- Dayon, L., et al. Relative quantification of proteins in human cerebrospinal fluids by MS/MS using 6-plex isobaric tags. Analytical Chemistry. 80 (8), 2921-2931 (2008).
- Stepanova, E., Gygi, S. P., Paulo, J. A. Filter-based protein digestion (FPD): A detergent-free and scaffold-based strategy for TMT workflows. Journal of Proteome Research. 17 (3), 1227-1234 (2018).
- McAlister, G. C., et al. Increasing the multiplexing capacity of TMTs using reporter ion isotopologues with isobaric masses. Analytical Chemistry. 84 (17), 7469-7478 (2012).
- Thompson, A., et al. TMTpro: Design, synthesis, and initial evaluation of a proline-based isobaric 16-plex tandem mass tag reagent set. Analytical Chemistry. 91 (24), 15941-15950 (2019).
- Li, J., et al. TMTpro reagents: a set of isobaric labeling mass tags enables simultaneous proteome-wide measurements across 16 samples. Nature Methods. 17 (4), 399-404 (2020).
- Arul, A. B., Robinson, R. A. S. Sample Multiplexing Strategies in Quantitative Proteomics. Analytical Chemistry. 91 (1), 178-189 (2019).
- Labib, M., Kelley, S. O. Single-cell analysis targeting the proteome. Nature Reviews Chemistry. 4 (3), 143-158 (2020).
- Ren, R. J., Dammer, E. B., Wang, G., Seyfried, N. T., Levey, A. I. Proteomics of protein post-translational modifications implicated in neurodegeneration. Translational Neurodegeneration. 3 (1), 23(2014).
- Pagel, O., Loroch, S., Sickmann, A., Zahedi, R. P. Current strategies and findings in clinically relevant post-translational modification-specific proteomics. Expert Review of Proteomics. 12 (3), 235-253 (2015).
- Mertins, P., et al. Reproducible workflow for multiplexed deep-scale proteome and phosphoproteome analysis of tumor tissues by liquid chromatography-mass spectrometry. Nature Protocols. 13 (7), 1632-1661 (2018).
- Aebersold, R., Mann, M. Mass-spectrometric exploration of proteome structure and function. Nature. 537 (7620), 347-355 (2016).
- Bai, B., et al. Deep multilayer brain proteomics identifies molecular networks in Alzheimer's disease progression. Neuron. 105 (6), 975-991 (2020).
- Kelstrup, C. D., et al. Rapid and deep proteomes by faster sequencing on a benchtop quadrupole ultra-high-field orbitrap mass spectrometer. Journal of Proteome Research. 13 (12), 6187-6195 (2014).
- Meier, F., et al. Online parallel accumulation - serial fragmentation (PASEF) with a novel trapped ion mobility mass spectrometer. Molecular & Cellular Proteomics. 17 (12), (2018).
- Schweppe, D. K., et al. Full-featured, real-time database searching platform enables fast and accurate multiplexed quantitative proteomics. Journal of Proteome Research. 19 (5), 2026-2034 (2020).
- Wang, H., et al. Systematic optimization of long gradient chromatography mass spectrometry for deep analysis of brain proteome. Journal of Proteome Research. 14 (2), 829-838 (2015).
- Dey, K. K., et al. Deep undepleted human serum proteome profiling toward biomarker discovery for Alzheimer's disease. Clinical Proteomics. 16, 16(2019).
- Bai, B., et al. Deep profiling of proteome and phosphoproteome by isobaric labeling, extensive liquid chromatography, and mass spectrometry. Methods in Enzymology. 585, 377-395 (2017).
- High, A. A., et al. Deep proteome profiling by isobaric labeling, extensive liquid chromatography, mass spectrometry, and software-assisted quantification. Journal of Visualized Experiments. (129), e56474(2017).
- Chick, J. M., et al. Defining the consequences of genetic variation on a proteome-wide scale. Nature. 534 (7608), 500-505 (2016).
- Wang, Z., et al. Quantitative phosphoproteomic analysis of the molecular substrates of sleep need. Nature. 558 (7710), 435-439 (2018).
- Tan, H., et al. Integrative proteomics and phosphoproteomics profiling reveals dynamic signaling networks and bioenergetics pathways underlying T cell activation. Immunity. 46 (3), 488-503 (2017).
- Eden, E., et al. Proteome Half-Life Dynamics in Living Human Cells. Science. 331 (6018), 764(2011).
- Kleiman, L. B., Maiwald, T., Conzelmann, H., Lauffenburger, D. A., Sorger, P. K. Rapid phospho-turnover by receptor tyrosine kinases impacts downstream signaling and drug binding. Molecular Cell. 43 (5), 723-737 (2011).
- Mertins, P., et al. Ischemia in tumors induces early and sustained phosphorylation changes in stress kinase pathways but does not affect global protein levels. Molecular & Cellular Proteomics. 13 (7), 1690(2014).
- Xu, P., Duong, D. M., Peng, J. Systematical optimization of reverse-phase chromatography for shotgun proteomics. Journal of Proteome Research. 8 (8), 3944-3950 (2009).
- Wang, Y., et al. Reversed-phase chromatography with multiple fraction concatenation strategy for proteome profiling of human MCF10A cells. Proteomics. 11 (10), 2019-2026 (2011).
- Yang, F., Shen, Y., Camp, D. G., Smith, R. D. High-pH reversed-phase chromatography with fraction concatenation for 2D proteomic analysis. Expert Review of Proteomics. 9 (2), 129-134 (2012).
- Niu, M., et al. Extensive peptide fractionation and y(1) ion-based interference detection method for enabling accurate quantification by isobaric labeling and mass spectrometry. Analytical Chemistry. 89 (1), 2956-2963 (2017).
- Wang, X., et al. A tag-based database search tool for peptide identification with high sensitivity and accuracy. Molecular & Cellular Proteomics. 13 (12), 3663(2014).
- Li, Y., et al. JUMPg: An integrative proteogenomics pipeline identifying unannotated proteins in human brain and cancer cells. Journal of Proteome Research. 15 (7), 2309-2320 (2016).
- Elias, J. E., Gygi, S. P. Target-decoy search strategy for increased confidence in large-scale protein identifications by mass spectrometry. Nature Methods. 4 (3), 207-214 (2007).
- Wang, Z., et al. 27-plex tandem mass tag mass spectrometry for profiling brain proteome in Alzheimer's disease. Analytical Chemistry. 92 (10), 7162-7170 (2020).
- Ow, S. Y., et al. iTRAQ underestimation in simple and complex mixtures: "The good, the bad and the ugly". Journal of Proteome Research. 8 (11), 5347-5355 (2009).
- Karp, N. A., et al. Addressing accuracy and precision issues in iTRAQ quantitation. Molecular & Cellular Proteomics. 9 (9), 1885-1897 (2010).
- Ting, L., Rad, R., Gygi, S. P., Haas, W. MS3 eliminates ratio distortion in isobaric multiplexed quantitative proteomics. Nature Methods. 8 (11), 937-940 (2011).
- Gygi, J. P., et al. A triple knockout isobaric-labeling quality control platform with an integrated online database search. Journal of The American Society for Mass Spectrometry. 31 (7), 1344-1349 (2020).
- Savitski, M. M., et al. Measuring and managing ratio compression for accurate iTRAQ/TMT quantification. Journal of Proteome Research. 12 (8), 3586-3598 (2013).
- Ong, S. E., et al. Stable isotope labeling by amino acids in cell culture, SILAC, as a simple and accurate approach to expression proteomics. Molecular & Cellular Proteomics. 1 (5), 376(2002).
- Cox, J., et al. Accurate proteome-wide label-free quantification by delayed normalization and maximal peptide ratio extraction, termed MaxLFQ. Molecular & Cellular Proteomics. 13 (9), 2513(2014).
- Brenes, A., Hukelmann, J., Bensaddek, D., Lamond, A. I. Multibatch TMT reveals false positives, batch effects and missing values. Molecular & Cellular Proteomics. 18 (10), 1967-1980 (2019).
- Yu, J., Peng, J., Chi, H. Systems immunology: Integrating multi-omics data to infer regulatory networks and hidden drivers of immunity. Current Opinion in Systems Biology. 15, 19-29 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados